一种基于二代测序数据对HLA-DRB基因进行分型的方法及应用与流程
本发明属于生物,涉及一种基于二代测序数据对hla-drb基因进行分型的方法及应用。
背景技术:
1、人类白细胞抗原(human leukocyte antigen,hla)是由hla基因复合体所编码的产物,位于第6染色体短臂上,其主要功能是识别和呈递抗原给免疫系统,从而引发免疫反应。hla基因分为ⅰ、ⅱ、ⅲ3类基因,其中hlaⅰ类基因包括经典的a、b、c基因座位,非经典hlaⅰ类基因包括e、f、g、h、mica、micb等基因座位;hlaⅱ类基因主要包括dr、dp和dq基因座位;hlaⅲ类基因包括补体基因和炎症递质基因。hla分型是指检测每个hla基因座上的等位基因,在器官移植、输血和疾病诊断上都有很大的临床价值。《人类白细胞抗原基因分型技术平台规范化建设及临床应用专家共识》中专家建议首选a、b、c、drb1、dqb1、dpb1、dpa1、dqa1、drb3/4/5位点相合的供者,其次选择a、b、c、drb1、dqb1位点相合的供者。
2、hla-drb等位基因编码hla-dr分子的β链,一共有9个等位基因,其中drb1、drb3、drb4和drb5(drb3/4/5)是功能基因,drb2、drb6、drb7、drb8和drb9是假基因。drb1和drb9基因存在于所有个体中,但是其它的等位基因不一定存在,主要取决于单倍型。例如,drb1*01、*10带有drb6;drb1*15、*16通常带有drb5和drb6;drb1*03、*11、*12、*13、*14带有drb2和drb3;drb1*04、*07、*09带有drb7,drb8和drb4,少数样本不带有drb4;drb1*08基本上不带有除drb9以外的drb等位基因,极少数样本存在drb3。
3、现有研究表明drb3/4/5配型可以改善无关造血干细胞移植的疗效。在对3410例接受了来自hla9/10或10/10等位基因相合非血缘造血干细胞移植的德国患者的回顾性分析中发现,drb3/4/5不相容可能会增加不良结局的风险,特别是那些有并发症和比较虚弱的患者。在另一项研究中发现,drb3/4/5位点相合可能有助于降低移植相关的发病率和死亡率。对1975例因血液恶性肿瘤接受了10/10匹配的非血缘供体的患者和供体分别进行高分辨率分型,发现接受来自drb3/4/5不相合供体的患者发生ii-iv级急性移植物抗宿主病(agvhd)的风险显著增加。此外,drb3/4/5与疾病的发生也有一定的相关性。2016年的一项研究发现drb3/4/5等位基因可能与胰岛自身抗体和儿童1型糖尿病风险相关。1型糖尿病和drb1*03:01:01之间的关联受到drb3*01:01:02和drb3*02:02:01的影响。
4、hla主要的分型方法有血清学分型、细胞学分型和基于dna的分型。基于dna的分型方法包括低分辨水平的序列特异性引物法(sequence specific primer,ssp)中分辨水平的序列特异性寡核苷酸探针法(sequence specific oligonucleotide probes,ssop)、高分辨水平的基于测序分型法(sequencing based typing,sbt)以及等位基因水平的二代测序法(next generation sequencing,ngs)。sbt技术是是现世界卫生组织(who)推荐的hla分型方法的金标准,具有分辨率高,特异性强及准确性高等特点。但是,这项技术只针对hla-ⅰ类基因的2、3、4外显子以及hla-ⅱ类基因2、3外显子的关键区域进行扩增和测序,无法检测全长,因此在分型过程中会出现摸棱两可的结果,即多个分型组合(不同的等位基因组合具有相同的外显子序列)。近年来,国内外已开始使用高通量测序技术对hla基因分型,通过对hla基因全长序列进行靶向捕获和上机测序,具有检测范围广、分辨率高、特异性强、通量高等特点。除了可以检测经典a、b、c、drb1/3/4/5、dqb1、dpb1、dpa1、dqa1位点外,还可检测非经典e、f、g、h位点,以及mica、micb位点的等位基因分型结果,且98%以上样本可直接获得唯一的分型结果。
5、目前,国内外已经开发了很多基于高通量测序数据进行hla分型的软件,例如hla-hd、athlates、optitype、hlascan、hlareporter、xhla和hla-la。由于drb3/4/5并非像drb1以及其它的hla等位基因一样存在于所有的个体中,再加上其重要性不及a、b、c、drb1、dqb1,很多方法都忽略了对它们的分型。即使有些软件能对drb3/4/5分型,也没有考虑到drb3/4/5的拷贝数情况,分型的准确性很低。例如,optitype只能用于a、b、c基因分型且精度只有4位;hlascan忽略了drb3/4的分型;hla-la软件也没有对drb3/4/5的拷贝数进行评估;athlates能对所有的hla基因进行分型,但准确率不高,部分基因的分型结果无法进行判定;hla-hd主要是基于reads的权重打分和等位基因对的筛选进行分型,整体来说分型效果很好,但依然存在错误分型的可能,需要合适的方法进行校正,特别是drb3/4/5,没有考虑到其拷贝数的特殊性,导致较高的分型错误率。
6、综上所述,目前基于高通量测序数据进行hla分型的软件忽略了对drb3/4/5的分型,没有考虑到drb3/4/5的拷贝数情况,分型的准确性低,如何提供一种能够考虑到drb3/4/5拷贝数的特殊性,校正错误分型的drb3/4/5分型方法,已成为目前生物技术领域亟待解决的问题之一。
技术实现思路
1、针对现有技术的不足和实际需求,本发明提供一种基于二代测序数据对hla-drb基因进行分型的方法及应用,解决了现有drb3/4/5分型方法忽略了drb3/4/5拷贝数,准确性低的问题,能够准确判断包括drb3/4/5在内的15个基因的拷贝数,与已知分型结果100%一致,再结合等位基因对的打分和筛选规则,drb3/4/5六位分型结果的准确率分别从85%,98%,96%提升至100%。
2、为达到此发明目的,本发明采用以下技术方案:
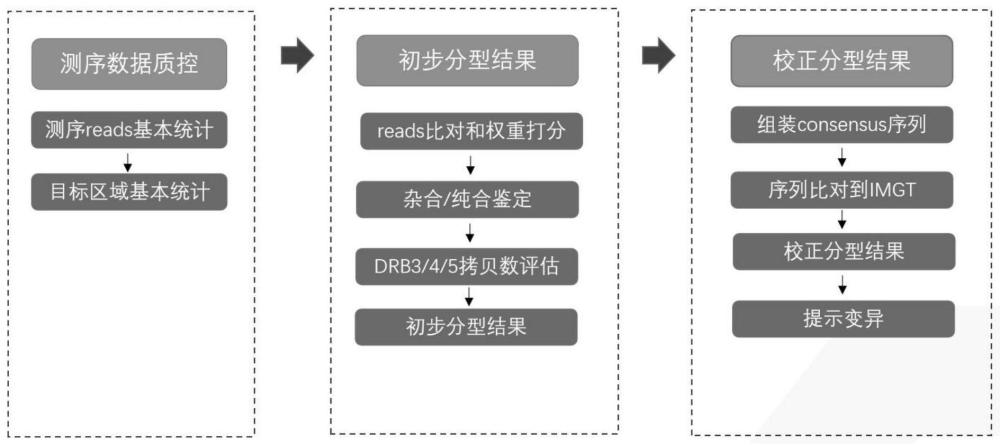
3、第一方面,本发明提供了一种基于二代测序数据对hla-drb基因进行分型的方法,所述方法包括:测序数据质控,初步分型和校正分型;
4、所述数据质控包括测序reads统计和目标区域统计;
5、所述初步分型包括构建参考基因组,reads比对和权重打分,hla-drb基因拷贝数评估,单个等位基因的筛选和等位基因对的筛选;
6、所述校正分型包括组装consensus序列,序列比对,校正分型结果和提示变异;
7、所述hla-drb基因包括hla-drb3基因、hla-drb4基因和hla-drb5基因中的任意一种或至少两种的组合。
8、本发明中能够准确判断包括drb3/4/5在内的15个基因的拷贝数,与已知分型结果100%一致,再结合等位基因对的打分和筛选规则,drb3/4/5六位分型结果的准确率分别从85%,98%,96%提升至100%,此外,在初步分型统计每个utr、外显子、内含子的错配情况,根据错配位置和类型进行罚分,给出最优的匹配,能够将错误的分型结果进行纠正,提示变异,并进行可视化。
9、优选地,所述目标区域统计的指标包括碱基数、q20、q30、gc含量、插入片段长度、靶标区域比对率、捕获效率和hla基因平均测序深度。
10、优选地,所述质控的标准为:有效reads数大于500k,插入片段长度大于400bp,q30大于0.85和hla基因平均测序深度大于200,所述有效reads数为编辑距离小于等于2的reads数量。
11、优选地,所述初步分型包括:
12、(1)提取数据库所有等位基因的外显子和/或内含子参考序列进行去重和编号,每个外显子和/或内含子序列前后加上100-200个n碱基组成新的参考序列并建立索引文件;
13、(2)将筛选的reads分别比对到外显子和内含子构建好的参考基因组,根据reads的比对情况对每条read进行权重打分;
14、(3)统计包括hla-drb3基因、hla-drb4基因和hla-drb5基因在内的15个基因关键外显子exon2和exon3的reads数并计算reads数占比;
15、(4)筛选单个等位基因,统计比对到每个基因的第2,3个外显子的reads数,选择前20个reads最高的exon2和exon3,exon2+exon3组合对应的等位基因作为候选等位基因进行筛选;
16、(5)筛选等位基因对,针对候选的等位基因,具有相同外显子序列的等位基因做为一个cluster,cluster和cluster之间两两组合进行打分,筛选最高得分组合,判断其基因型为纯合或杂合。
17、优选地,步骤(1)中所述数据库包括ipd-imgt/hla数据库。
18、优选地,步骤(2)中所述权重打分的标准包括hla-hd reads权重打分标准。
19、优选地,步骤(3)中所述15个基因hla-a、hla-b、hla-c、hla-dqa1、hla-dqb1、hla-dpa1、hla-dpb1、hla-drb1、hla-drb3、hla-drb4、hla-drb5、hla-e、hla-f、hla-g和hla-h。
20、优选地,步骤(3)中所述计算reads数占比的公式如式(1)所示:
21、
22、其中,ratiog表示基因g第2,3外显子reads数占比;ng,e2表示比对到基因g第2个外显子的reads数;ng,e3表示比对到基因g第3个外显子的reads数;ng,e2/e3表示同时比对到基因g第2,3外显子的reads数;t表示有效reads数。
23、优选地,步骤(5)中所述打分的公式如式(2)所示:
24、
25、其中,scoreallele为等位基因的得分;wr为reads的权重打分;eallele为等位基因allele的外显子集合;lene为等位基因allele的外显子e的长度;medlene为allele所属基因对应外显子e的长度中位数。
26、优选地,步骤(5)中所述判断其基因型为纯合或杂合的公式如式(3)和式(4)所示:
27、
28、
29、其中,clustera和cluster b为两组不同外显子序列的等位基因集合;delta1表示clustera与cluster b打分的比值;delta2表示clustera特有的打分与cluster b特有的打分的比值;scorea表示比对到clustera的reads打分;scoreb表示比对到cluster b的reads打分;scoreab表示比对到clustera未比对到cluster b的reads打分;scoreab表示比对到clusterb未比对到clustera的reads打分;pscoreab表示比对到clustera未比对到clusterb的paired reads打分;pscoreab表示比对到clusterb未比对到clustera的pairedreads打分。
30、优选地,步骤(5)中所述判断其基因型为纯合或杂合的标准为:delta1大于等于4或小于等于0.25,delta2大于等于4或小于等于0.25,则为纯合,否则为杂合;
31、若clustera和cluster b组合判定为杂合,则按式(5)计算等位基因对的得分:
32、cscoreab=scoreab+pscoreab+scoreab+pscoreab+scoreab+pscoreab(a≠b,杂合)式(5);
33、其中,cscoreab表示等位基因对clustera和clusterb的总分;scoreab表示同时比对到clustera和cluster b的reads打分;scoreab表示比对到clustera未比对到cluster b的reads打分;scoreab表示比对到cluster b未比对到clustera的reads打分;pscoreab表示同时比对到clustera和cluster b的paired reads打分;pscoreab表示比对到clustera未比对到clusterb的pairedreads打分;pscoreab表示比对到clusterb未比对到clustera的paired reads打分。
34、若clustera和cluster b组合判定为杂合,则按式(6)计算等位基因对的得分:
35、cscoreab=2*max(scorea,scoreb)(a≠b,纯合)式(6);
36、其中,cscoreab表示等位基因对clustera和clusterb的总分;scorea表示比对到clustera的reads打分;scoreb表示比对到clusterb的reads打分。
37、优选地,所述校正分型结果包括计算候选等位基因的得分;所述计算候选等位基因的得分的公式如式(7)所示:
38、
39、其中,e为allelea所属基因g的所有外显子,内含子和utr的集合,t为错配、插入、删除的集合;u为未覆盖和数据库中未收录的集合,scorec,a为consensus c和allelea的得分;ne,x表示在基因元件e中出现不一致碱基的个数;we,x表示在基因元件e中出现错配、插入或者删除的罚分权重;we,y表示在基因元件e中出现碱基未覆盖或者该元件未被数据库收录的罚分权重。
40、本发明流程图如图1所示。
41、第二方面,本发明提供了根据第一方面所述的基于二代测序数据对hla-drb基因进行分型的方法在hla基因分型中的应用。
42、与现有技术相比,本发明具有如下有益效果:
43、(1)本发明能够准确判断包括drb3/4/5在内的15个基因的拷贝数,与已知分型结果100%一致,再结合等位基因对的打分和筛选规则,drb3/4/5六位分型结果的准确率分别从85%,98%,96%提升至100%;
44、(2)本发明在初步分型统计每个utr、外显子、内含子的错配情况,根据错配位置和类型进行罚分,给出最优的匹配,能够将错误的分型结果进行纠正,提示变异,并进行可视化。
- 还没有人留言评论。精彩留言会获得点赞!