一种基于序列同步优化的RNA序列表示增强方法
本发明属于生物,具体涉及一种rna序列表示增强方法。
背景技术:
1、目前,对rna序列的分析方法主要分为传统方法和深度学习方法两大类。传统的rna序列分析方法主要依赖于生物化学领域的专业知识,包括功能注释和理化性质等。具体而言,这些方法涉及rna序列中碱基的反应活性、稳定性等。通过构建相关的理化性质矩阵来对rna序列进行表征。深度学习模型,如e2efold,首先将rna序列进行one-hot编码,然后进行嵌入表示作为rna序列的表征。而模型ltpconstraint则使用1、2、3、4对rna序列进行编码,然后进行嵌入表示,通过不断地迭代训练来获取可靠的表征。
技术实现思路
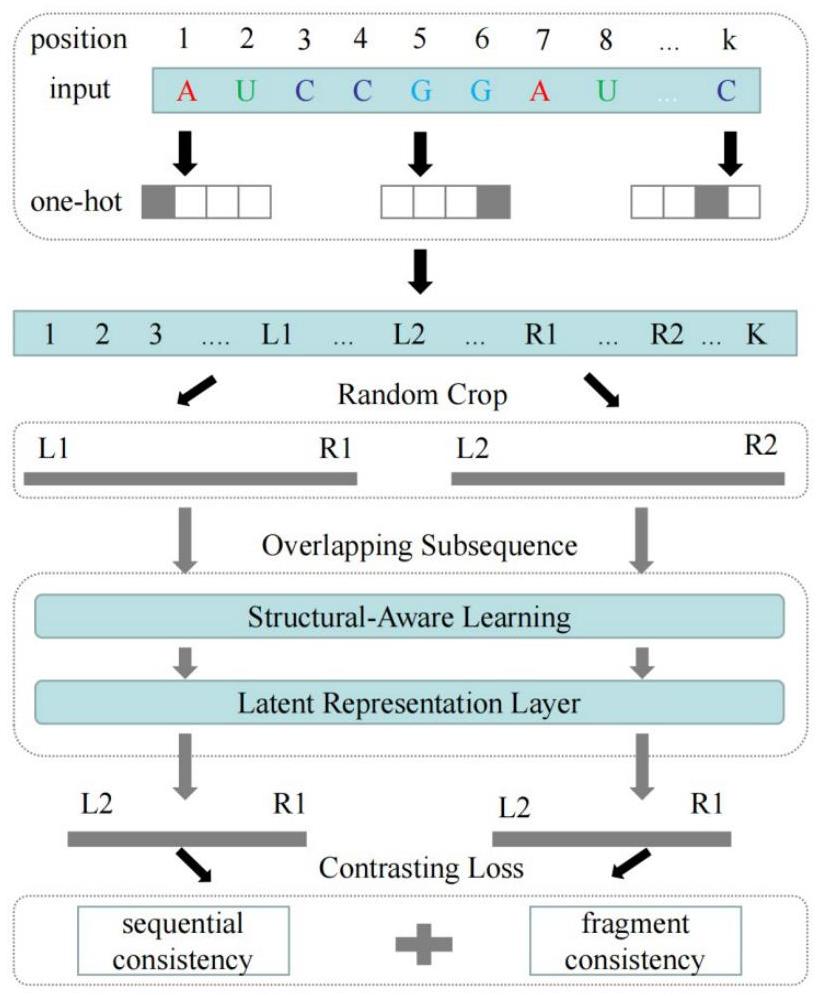
1、为了克服现有技术的不足,本发明提供了一种基于序列同步优化的rna序列表示增强方法,包括one-hot编码、随机裁剪、结构感知学习、序列表征、分层对比损失计算五个过程。本发明方法可以对rna序列进行可靠表征,很好的保留了rna序列的特征信息。
2、本发明解决其技术问题所采用的技术方案包括如下步骤:
3、步骤1:one-hot编码;
4、对于长度为l的线性rna序列x,通过one-hot编码后得到x:
5、x∈rl·4
6、步骤2:随机裁剪;
7、对编码后的rna序列进行随机裁剪,得到sm和sn,其中sm和sn满足下式:
8、
9、设置超参数λ对重叠序列的最小长度进行限制:
10、|sm∩sn≥λ|
11、步骤3:结构感知学习;
12、对两个重叠子序列通过线性层进行高维映射,得到rna序列的潜在向量v;w和b分别表示需要优化的参数和向量偏差参数;
13、v=wx+b
14、步骤4:序列表征;
15、rna序列的潜在向量v中的每一个元素都将经过一个概率掩码mask∈{0,1}操作;
16、步骤5:分层对比损失计算;
17、分层对比损失计算包括序列一致性和片段一致性;其中序列一致性用于比较rna序列中不同位置碱基的信息损失,片段一致性用于比较不同rna序列中相同位置碱基的信息损失。
18、优选地,所述步骤5具体为:
19、碱基之间的相似性计算公式:
20、sim(γ1,γ2)=γ1γ2
21、其中γ1和γ2分别代表两个重叠子序列中的碱基所对应的高维向量;
22、相似性度量公式:
23、
24、在上述相似性度量的基础上,计算序列一致性信息损失:
25、
26、其中m、n均为为rna序列索引,u、v均为为rna序列中碱基的索引;
27、计算片段一致性信息损失:
28、
29、最终,损失函数综合上述两种损失,总损失函数如下:
30、
31、上式中s表示样本集,ps表示该样本的位置集;|s|和|ps|分别表示各自集合中元素的数量。
32、本发明的有益效果如下:
33、本发明方法构建的线性rna序列表征模型能够很好地对rna序列进行表征。验证结果表明:表征向量很好的保留了rna序列的特征信息。
技术特征:
1.一种基于序列同步优化的rna序列表示增强方法,其特征在于,包括如下步骤:
2.根据权利要求1所述的一种基于序列同步优化的rna序列表示增强方法,其特征在于,所述步骤5具体为:
技术总结
本发明公开了一种基于序列同步优化的RNA序列表示增强方法,包括one‑hot编码、随机裁剪、结构感知学习、序列表征、分层对比损失计算五个过程。本发明方法可以对RNA序列进行可靠表征,很好的保留了RNA序列的特征信息。
技术研发人员:王永天,申烨玮,尚学群
受保护的技术使用者:西北工业大学
技术研发日:
技术公布日:2024/3/21
- 还没有人留言评论。精彩留言会获得点赞!