一种基于深度对比学习的蛋白质与多肽绑定位点预测方法与流程
本发明涉及药物制备,尤其是指一种基于深度对比学习的蛋白质与多肽绑定位点预测方法。
背景技术:
1、蛋白质与多肽之间的相互作用在生命活动中普遍存在且至关重要,它们之间的相互作用位点亦是药物重要靶向表位之一。因此,精确识别蛋白质与多肽绑定位点对理解蛋白质与多肽间相互作用机理、创新药物设计都具有重要的指导意义。尽管基于生物实验来识别蛋白质与多肽绑定位点是目前最为精准的方法,但其耗时、费力且成本高昂,严重阻碍了相互作用机理研究与药物研发的进程。
2、基于计算的蛋白质与多肽绑定位点预测方法受到了越来越多的关注。利用机器学习与深度学习等算法,可以从蛋白质与多肽序列与结构信息中挖掘鉴别信息并训练预测模型,来进行蛋白质与多肽绑定位点的识别。蛋白质的序列数据远远超过它的结构数据,然而序列信息的鉴别性要低于结构信息。如何借助高鉴别性的结构数据来提升海量序列数据的鉴别性,是基于计算的蛋白质与多肽绑定位点识别的关键问题之一,亦是目前方法被忽略的问题之一。
3、需要说明的是,在上述背景技术部分公开的信息仅用于加强对本公开的背景的理解,因此可以包括不构成对本领域普通技术人员已知的现有技术的信息。
技术实现思路
1、为此,本发明所要解决的技术问题在于克服现有技术中未充分利用蛋白质结构数据来提升序列信息鉴别性的不足。
2、为解决上述技术问题,本发明的第一方面提供了一种基于深度对比学习的蛋白质与多肽绑定位点预测方法,所述方法包括:
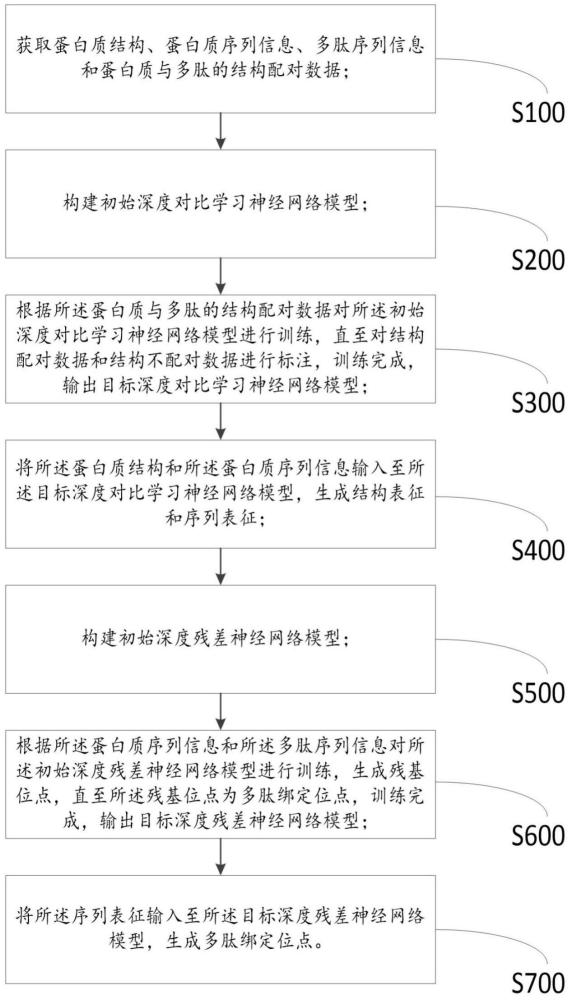
3、获取蛋白质结构、蛋白质序列信息、多肽序列信息和蛋白质与多肽的结构配对数据;
4、构建初始深度对比学习神经网络模型;
5、根据所述蛋白质与多肽的结构配对数据对所述初始深度对比学习神经网络模型进行训练,直至对结构配对数据和结构不配对数据进行标注,训练完成,输出目标深度对比学习神经网络模型;
6、将所述蛋白质结构和所述蛋白质序列信息输入至所述目标深度对比学习神经网络模型,生成结构表征和序列表征;
7、构建初始深度残差神经网络模型;
8、根据所述蛋白质序列信息和所述多肽序列信息对所述初始深度残差神经网络模型进行训练,生成残基位点,直至所述残基位点为多肽绑定位点,训练完成,输出目标深度残差神经网络模型;
9、将所述序列表征输入至所述目标深度残差神经网络模型,生成多肽绑定位点。
10、在本发明的一个实施例中,所述初始深度对比学习神经网络模型包括:结构表征模块、序列表征模块以及对比操作模块;
11、将所述蛋白质结构输入至所述结构表征模块,生成结构表征;
12、将所述蛋白质序列信息输入至所述序列表征模块,生成序列表征;
13、将所述结构表征和所述序列表征输入至所述对比操作模块生成相似度。
14、在本发明的一个实施例中,所述结构表征模块包括:独热编码层、位置编码层,蛋白质接触距离图提取层、2d残差卷积层和按行取最大值层;
15、将所述蛋白质结构输入至所述独热编码层,生成第一编码特征;
16、将所述第一编码特征输入至所述位置编码层,生成第二编码特征;
17、将所述第二编码特征输入至蛋白质接触距离图提取层,生成提取特征;
18、将所述提取特征输入至2d残差卷积层,生成卷积特征;
19、将所述卷积特征输入至所述按行取最大值层,生成结构表征。
20、在本发明的一个实施例中,所述序列表征模块包括:蛋白质语言预训练单元和1d残差卷积层;
21、将所述蛋白质序列信息输入至所述蛋白质语言预训练单元,生成训练序列信息;
22、将所述训练序列信息输入至所述1d残差卷积层,生成序列表征。
23、在本发明的一个实施例中,所述对比操作模块包括:根据余弦相似度算法对所述结构表征和所述序列表征进行计算,生成相似度。
24、在本发明的一个实施例中,所述初始深度残差神经网络模型包括:残差卷积层和全连接层;
25、将所述序列表征输入至所述残差卷积层,生成初始表征;
26、将所述初始表征输入至全连接层,生成多肽绑定位点。
27、本发明的第二方面提供了一种电子设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现上述第一方面或第一方面中任一种可能的实施方式中所述的方法。
28、本发明的第三方面提供了一种非暂态计算机可读存储介质,其上存储有计算机程序,该计算机程序被处理器执行时实现上述第一方面或第一方面中任一种可能的实施方式中所述的方法。
29、本发明的上述技术方案相比现有技术具有以下优点:
30、本发明所述的一种基于深度对比学习的蛋白质与多肽绑定位点预测方法,通过初始深度对比学习神经网络模型,对蛋白质结构和蛋白质序列进行一致性表征,从而提升海量蛋白质序列表征的鉴别性;从蛋白质序列出发,充分利用了海量蛋白质序列数据,并训练目标深度残差神经网络模型,进一步地提升了蛋白质与多肽绑定位点的预测性能。
技术特征:
1.一种基于深度对比学习的蛋白质与多肽绑定位点预测方法,其特征在于,所述方法包括:
2.根据权利要求1所述的一种基于深度对比学习的蛋白质与多肽绑定位点预测方法,其特征在于,所述初始深度对比学习神经网络模型包括:结构表征模块、序列表征模块以及对比操作模块;
3.根据权利要求2所述的一种基于深度对比学习的蛋白质与多肽绑定位点预测方法,其特征在于,所述结构表征模块包括:独热编码层、位置编码层,蛋白质接触距离图提取层、2d残差卷积层和按行取最大值层;
4.根据权利要求2所述的一种基于深度对比学习的蛋白质与多肽绑定位点预测方法,其特征在于,所述序列表征模块包括:蛋白质语言预训练单元和1d残差卷积层;
5.根据权利要求2所述的一种基于深度对比学习的蛋白质与多肽绑定位点预测方法,其特征在于,所述对比操作模块包括:根据余弦相似度算法对所述结构表征和所述序列表征进行计算,生成相似度。
6.根据权利要求1所述的一种基于深度对比学习的蛋白质与多肽绑定位点预测方法,其特征在于,所述初始深度残差神经网络模型包括:残差卷积层和全连接层;
7.一种电子设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,其特征在于,所述处理器执行所述程序时实现如权利要求1至6任一项所述一种基于深度对比学习的蛋白质与多肽绑定位点预测方法的步骤。
8.一种非暂态计算机可读存储介质,其上存储有计算机程序,其特征在于,该计算机程序被处理器执行时实现如权利要求1至6任一项所述一种基于深度对比学习的蛋白质与多肽绑定位点预测方法的步骤。
技术总结
本发明涉及一种基于深度对比学习的蛋白质与多肽绑定位点预测方法,通过初始深度对比学习神经网络模型,对蛋白质结构和蛋白质序列进行一致性表征,从而提升海量蛋白质序列表征的鉴别性;从蛋白质序列出发,充分利用了海量蛋白质序列数据,并训练目标深度残差神经网络模型,进一步地提升了蛋白质与多肽绑定位点的预测性能。
技术研发人员:胡俊,张阳
受保护的技术使用者:深药科技(苏州)有限公司
技术研发日:
技术公布日:2024/4/22
- 还没有人留言评论。精彩留言会获得点赞!