基于GWO算法在GWAS数据上检测与复杂疾病相关SNP组合的方法
本发明属于计算机科学技术与生物学的交叉领域,尤其涉及一种基于灰狼优化(grey wolf optimizer ,gwo)算法在全基因组关联分析数据上检测与复杂疾病相关snp组合的方法。
背景技术:
1、复杂疾病包含心脏病、高血压、糖尿病等常见疾病,它们是由多基因、多因素共同引起的疾病,它们很难被预防,一旦发现,也很难治愈,很多甚至无法治愈,只能依靠药物来维持,影响着患者的生活。全基因组关联分析(genome-wide association study ,gwas)是一种遗传学研究方法,用于揭示基因与复杂疾病之间的关系,gwas一般针对某一种复杂疾病,收集数千的正常人与患者,通过检测他们dna上几十万的snp(single-nucleotidepolymorphism)的基因型,探索snp与某种复杂疾病之间的相关性,其结果对于复杂疾病致病机理的探索,复杂疾病的预防与治疗都有着重要的意义。然而,gwas数据上普遍存在的上位性现象为我们检测与复杂疾病相关的snp带来了巨大的困难。上位性的原意指的是一种基因相互作用的类型,其中一个基因能够掩盖或支配另一个基因的表现。当在gwas数据上使用统计的方法检测与疾病相关的snp时,上位性表现为,很多snp在单独分析时,其与疾病没有显著的相关性,但当它们与其他的一些snp组合在一起进行分析时,它们与复杂疾病之间产生了非常显著的相关性,这种现象为检测与疾病相关的snp带来了巨大的问题。其原因是,复杂疾病是由多基因、多因素共同引起的,往往snp以组合的方式共同与疾病的发生有关系,上位性普遍存在于gwas数据中,为了充分地分析复杂疾病的致病机理,就需要去分析数据中每两个、每三个(甚至更多)snp与疾病之间的相关性,其计算量随着上位性阶数的升高成指数级增长。因此,提出高效、精确的在gwas数据上检测与复杂疾病相关snp组合的方法是当前的研究热点之一。
2、近年来,针对这一问题,提出了非常多的算法,antepiseeker(ant代表蚂蚁,因为它是一种蚁群算法,epi代表上位性,因为它是在数据中找上位性snp组合,seeker是表示它是一种搜索算法)是一种基于蚁群优化算法检测snp的方法,通过蚁群算法探索与疾病显著相关的snp组合,再对蚁群算法的结果做进一步的穷举搜索,获取结果,它提出了用蚁群优化算法检测snp的方法,证明了基于群智能优化算法检测与复杂疾病相关的snp的可行性。macoed(a multi-objective ant colony optimization algorithm for snp epistasisdetection)是一种多目标启发式优化方法,它将逻辑回归和贝叶斯网络方法结合作为蚁群优化算法的目标函数,其实验结果证明了在群智能优化算法中引入多个目标函数可以达到互补的效果,从而提升群智能算法检测致病snp组合的效率。boost(boolean operation-based screening and testing)是一种快速检测二阶snp组合与疾病关系的方法,它提出了一种基于统计模型检测显著与疾病相关的snp组合的下界函数,这个下界函数可以快速地用穷举的方法检测每一对snp是否有可能与疾病具有显著的相关性,只需要对下界函数的值高于指定阈值的snp组合进行分析就可以在考虑二阶上位性的前提下检测到与疾病相关的snp,boost还提出了一种在计算机中gwas数据的二进制存储与计算的方法,可以提高算法在gwas数据上的计算速度。
3、现有的在gwas数据上检测与复杂疾病相关的snp组合的方法主要有以下缺点:
4、1.多数基于群智能优化算法的检测方法都是基于一个目标函数设计的,而复杂疾病的潜在致病模型往往非常复杂,而一个目标函数无法充分、合理地度量snp组合与疾病之间的相关性;
5、2.绝大多数算法都需要用户指定考虑的上位性的阶数,而复杂疾病的致病机理非常复杂,用户无法预判一个真实的复杂疾病潜在的上位性阶数,为用户的使用带来了不便;
6、3.绝大多数算法在设计的时候着重考虑了二阶上位性的情况,而忽略了高阶(>=3)上位性snp组合,针对算法的实验也只在蕴含二阶上位性snp致病关系的模拟数据上进行,因此绝大多数算法在检测高阶上位性snp组合的能力上表现欠佳,从而导致无法检测出致病的高阶snp组合;
7、4.该领域普遍使用的k2、ce、gini等用于评测snp组合与疾病关系的目标函数,普遍存在“样本分的份数越多,计算结果越相关”的问题,导致评测函数上的不公,阻碍了算法对相关snp组合的检测。
技术实现思路
1、本发明目的在于提供一种基于gwo算法在gwas数据上检测与复杂疾病相关snp组合的方法,以解决在gwas数据上检测与疾病相关的snp需要非常大的计算量的技术问题,本发明的宗旨是在同等计算量下,追求检测的精度。
2、为实现上述目的,本发明的一种基于gwo算法在gwas数据上检测与复杂疾病相关snp组合的方法的具体技术方案如下:
3、复杂疾病是由多基因、多因素共同引起的常见疾病,全基因组关联分析(gwas,genome-wide association study)数据蕴含着单核苷酸多态性(snp, single-nucleotidepolymorphism)与复杂疾病之间的相关性,但由于上位性现象的存在,在gwas数据上检测与疾病相关的snp需要非常大的计算量,提出高效、精确的检测算法是当前的研究热点之一。
4、本发明提出了一种基于gwo算法在gwas数据上检测与复杂疾病相关snp组合的方法,针对现有方法存在的缺陷,主要的解决方案如下:
5、1.本发明提出的方法是一种基于多个目标函数实现的灰狼优化算法,同时使用k2、ce、gini三个函数作为算法优化的目标,并最后使用g-test检测snp组合与疾病是否显著相关;
6、2.本发明提出了一种自动根据数据中的样本数目推测计算过程中最大上位性阶数的方法,而不需要用户指定上位性阶数,算法考虑了小于或等于最大阶数的所有的上位性;
7、3.本发明提出了一种基于k2和g-test在高阶snp组合上检测显著相关的snp组合的方法,该方法使算法可以检测到与gwas数据中的疾病显著相关的所有小于或等于最大上位性阶数的snp组合;
8、4.针对k2、ce、gini等函数评测不公的问题,本发明提出了一种对计算过程中的列联表进行合并的方案,可以有效地消除函数计算中的不公现象。
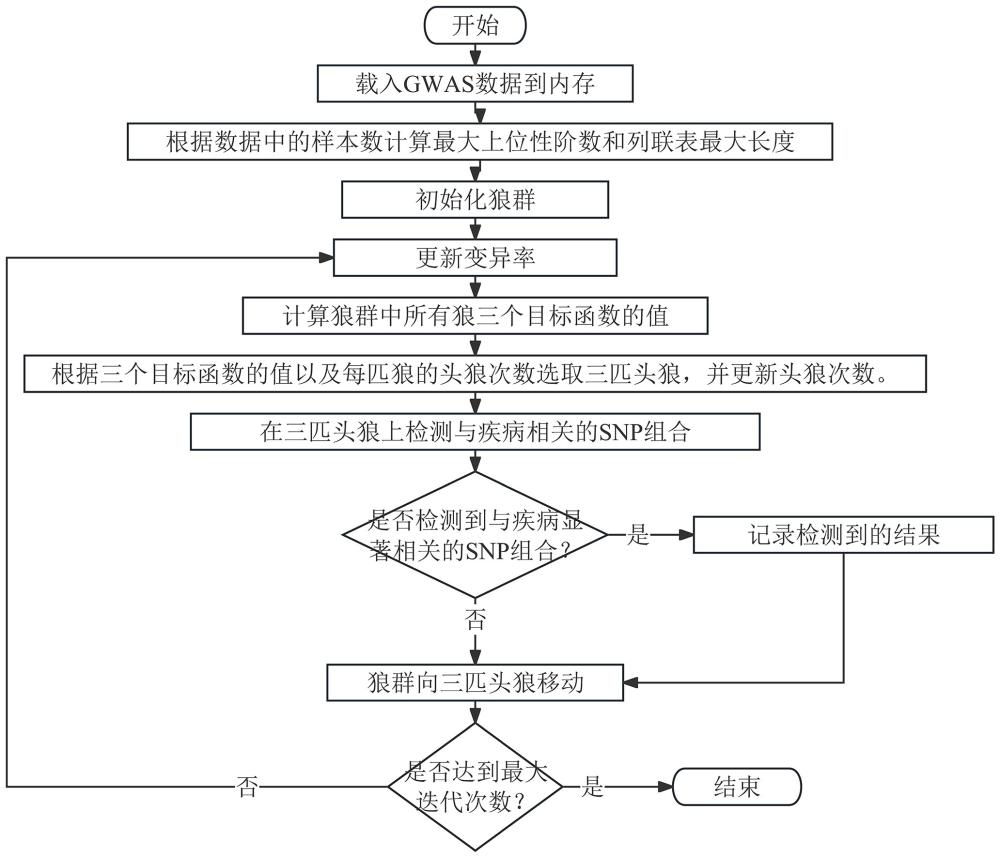
9、一种基于gwo算法在gwas数据上检测与复杂疾病相关snp组合的方法,包括以下步骤,且以下步骤顺次进行:
10、步骤s1:载入全基因组关联分析genome-wide association study ,gwas数据到内存
11、使用与boolean operation-based screening and testing,boost算法类似的基于二进制的存储方式;
12、步骤s2:根据数据中的样本数计算最大上位性阶数和列联表最大长度
13、步骤s3:初始化狼群
14、初始化狼群中的狼,狼的数目由用户通过参数指定,每一只狼是一个长度为mo的整型向量,向量上的每一个数字代表gwas数据中某一个single-nucleotide polymorphism,snp的索引下标,而每一只狼对应的就是一个snp组合;
15、步骤s4:更新变异率
16、变异率的计算方式如式(3)所示,它是0到最大变异率mr之间的一个值;
17、 (3)
18、其中,vr是变异率,ns是狼群中所有snp去重之后的数目,代表了狼群的复杂性,nw是狼群中狼的数目,mr是算法可以接受的最大的变异率,mr变量的值由用户通过参数指定;
19、步骤s5:计算狼群中所有狼的k2、ce、gini目标函数的值
20、
21、其中,x代表一个snp组合,y代表疾病状态,k2( x, y)是snp组合x与疾病状态y之间的k2值,xg是snp组合的组合基因型集合,yg代表疾病状态的集合,对于gwas研究,通常样本的状态只有患病与正常,因此yg通常为{0,1},一般用0代表正常样本,1代表患病样本,mx是样本中组合基因型为x的样本的数目,mx,y是样本中组合基因型为x并且样本状态为y的样本数目;ce( x, y)是x与y之间的ce值,p( x, y)是snp组合基因型为x并且样本状态为y的样本的数目与样本总数的比例,p(x)是snp组合基因型为x的数目与样本总数的比例; gini( x, y)是x与y之间的 gini值,p( y| x)是在所有snp组合基因型为x的样本中,样本状态为y的样本的占比;
22、步骤s6:根据k2、ce、gini目标函数的值以及每匹狼的头狼次数选取三匹头狼,并更新头狼次数;
23、步骤s7:在三匹头狼上检测与疾病相关的snp组合
24、在每一次狼群中选出三匹头狼之后,针对每一匹头狼,基于k2值反复移除头狼中的噪声snp,直到不存在噪声,如果最后剩余的snp的数目大于1,则算法找到了一个snp组合,并且这个组合中的snp彼此联系且与疾病状态相关;
25、步骤s8:狼群向三匹头狼移动
26、狼是一个长度为mo的整型向量,每一个整数代表了gwas数据中一个snp的下标,检测完三匹头狼之后,对于狼群中其它的狼,向三匹头狼移动,以达到狼群寻优的目的;
27、步骤s9:移动狼群中的每一匹头狼之外的狼,从而完成了一次狼群的移动,而后判断灰狼算法的迭代次数,如果迭代次数到达最大迭代次数,则算法执行完成,将算法记录的结果输出到结果文件,如果未达到最大迭代次数,更新变异率,进入下一次循环。
28、进一步,所述步骤s1中,在内存中使用两个三维数组分别存储患病样本与正常样本的基因型数据,对于每一个数组,第一维度代表数据中不同的snp,第二维度代表对应的snp不同的基因型,第三个维度代表对应snp与其对应的基因型下有哪些样本。
29、进一步,所述步骤s8具体包括以下步骤:
30、对于一匹狼a和一匹头狼b,a向b移动后的狼为c,它们都是长度为mo的整型向量,在生成c中的每一个元素的时候,依变异率随机从gwas数据的所有snp中随机选择一个填入c中。
31、进一步,所述步骤s2中,最大上位性阶数的计算方法如式(1)所示:
32、 (1)
33、其中,mo是最大上位性阶数,log是以自然常数为底的对数函数,m0是gwas数据中的正常样本数,m1是gwas中的患病样本数,min是取两个数最小值的函数;
34、列联表最大长度的计算如式(2)所示:
35、(2)
36、其中, ml是列联表最大长度,min、 m0、m1的定义同公式1。进一步,所述步骤s6中,根据式(7)的计算结果进行头狼的选择:
37、 (7)
38、其中,x是一个snp组合,即狼群中的一匹狼, ls(x)是选择头狼时依据的值,在狼群中计算每一匹狼的 ls值,选择值最小的狼作为头狼, lw(x)是x已经做过头狼的次数,在算法开始运行时,将所有snp组合的头狼次数置0,当任意一匹狼x被选择为头狼时,对应的 lw(x)的计数加一, f(x)是x的k2、ce、gini值之一,在狼群初始化之后,或每一次向三匹头狼进行移动之后,针对每一个目标函数,都根据公式7在狼群中选择一匹头狼,选出三匹头狼之后,狼群参照三匹头狼的位置进行移动。
39、进一步,所述步骤s7中,根据公式8计算该snp组合与疾病状态之间关系的显著性,如果其显著性超过用户指定的阈值,保存这个组合作为算法的输出结果之一;
40、
41、其中,g( x, y)是snp组合x与样本状态y进行g-test独立性检验的p-value值,f( x, y)是独立性检验的自由度,mx是snp组合基因型为x的样本的数目,my是样本状态为y的样本数目,m为样本总数,e( x, y)是当假设x与y之间独立时,snp组合基因型为x且样本状态为y的期望样本数,s(x,y)是一个统计量,用于综合度量不同组合基因型下真实样本数与期望样本数之间的差距,根据独立向检验的知识,当x与y独立时,s(x,y)服从自由度为f(x,y)的卡方分布,pvalueofg(x,y)是根据统计量s(x,y)计算自由度为f(x,y)的卡方分布下的pvalue值的函数,g(x,y)的值越低,代表x与y越不可能是互相独立的,其他符号的定义与前文相同。
42、本发明的一种基于gwo算法在gwas数据上检测与复杂疾病相关snp组合的方法具有以下优点:
43、1.本发明提出的方法是一种基于多个目标函数实现的灰狼优化算法,以不同的角度度量与疾病状态相关的snp组合,针对gwas数据具有更好的检测能力;
44、2.本发明提出的方法不需要用户在运行算法时指定上位性阶数,可以检测到与疾病状态显著相关的任意的小于或等于最大上位性阶数的snp组合,更加方便用户使用的同时,也更加地符合gwas研究的实际情况;
45、3.本发明提出了一种在度量snp组合与疾病状态相关性时,合并列联表的方法,解决了k2、ce、gini等函数对列联表长度的偏性问题,从而使算法中的目标函数的计算更加的合理,提升了算法检测的能力。
- 还没有人留言评论。精彩留言会获得点赞!