一种基于深度学习的基因错义突变致病性预测系统
本发明涉及深度学习和生物医学的,尤其是指一种基于深度学习的基因错义突变致病性预测系统。
背景技术:
1、在现代精准医疗中,基因错义突变扮演着至关重要的角色,涵盖了疾病机制研究、临床诊断、药物设计以及个性化治疗等多个领域。然而,并非所有的基因错义突变都表现出致病性,有些变异对蛋白质功能的影响较小,甚至在临床表现上呈现良性。由于对功能影响的不确定性,许多在临床基因检测中发现的错义突变都被划归为“不确定性”。这种分类的不确定性可能导致精准医疗中临床诊断的不确定性,过度治疗,甚至错失临床干预的良机,进而带来不良后果。因此,对于基因错义突变的准确预测成为亟待解决的研究课题。
2、然而,在基因错义突变准确预测领域仍然存在一些挑战。目前的方法主要依赖于已知疾病标签,其中大多数是基于监督学习的。然而,对标签的高要求使得网络的性能容易受到标签数据的稀疏性、有偏差性和质量可变性的影响。这一问题可能导致网络在处理未标签数据时表现不佳,特别是在面对罕见疾病或新发现的基因错义突变时。此外,收集有标签的数据成本昂贵,这限制了数据规模的扩大,使得训练数据的数量相对较小,可能导致网络过度拟合已有数据,难以泛化到新的数据集。
技术实现思路
1、本发明的目的在于克服现有技术的缺点与不足,提出了一种基于深度学习的基因错义突变致病性预测系统,采用无监督学习的方式,突破传统基因错义突变致病性预测系统过度依赖已知的疾病标签的弊端,避免标签稀疏、偏差和质量不一的问题,从而增强疾病的智能诊断能力,同时,蛋白质高质量多序列比对数据能够揭示蛋白质序列之间的保守区域和保守模式,有助于进行蛋白质功能的注释和预测,为诊断结果提供了更好的可解释性说明,增强了诊断结果的可信度。
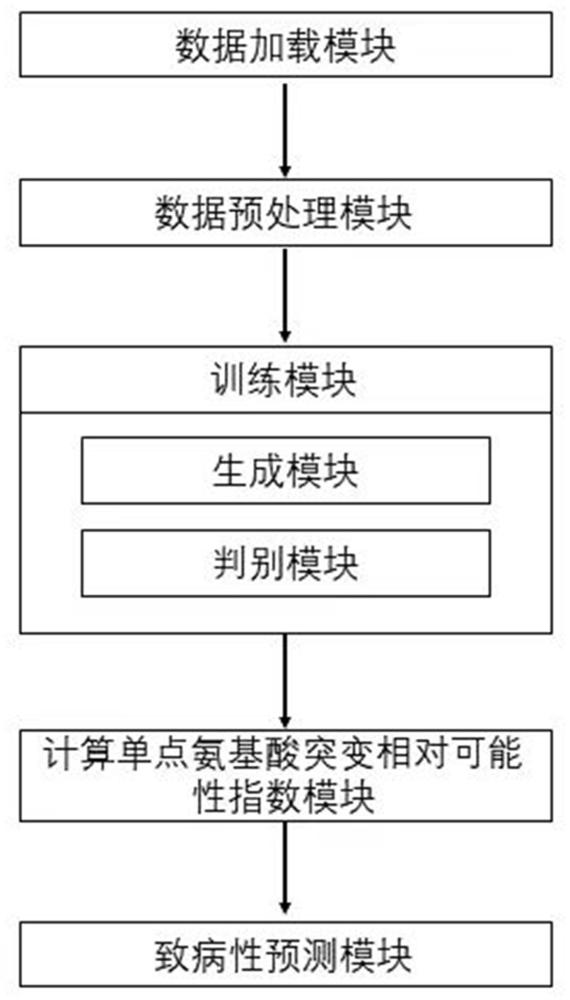
2、为实现上述目的,本发明所提供的技术方案为:一种基于深度学习的基因错义突变致病性预测系统,包括:
3、数据加载模块,用于加载蛋白质高质量多序列比对数据;
4、数据预处理模块,用于对蛋白质高质量多序列比对数据进行预处理,得到蛋白质高质量多序列比对数据对应的野生型氨基酸、蛋白质序列独热编码数据以及每个氨基酸可能有效的单点氨基酸突变数据,其中蛋白质序列独热编码数据需要计算权重,用于提高改进生成对抗网络对关键序列的关注以及改进生成对抗网络泛化能力;
5、训练模块,基于改进生成对抗网络从蛋白质序列独热编码数据学习每个蛋白质的氨基酸序列分布,捕获蛋白质的氨基酸序列的突变信息,学习其中突变的约束条件以及空间上的复杂依赖性,最终得到训练好的改进生成对抗网络;其中,该改进生成对抗网络是对传统生成对抗网络的生成模块和判别模块进行改进;对生成模块的改进是:引入一对变分自动编码器,其中一个变分自动编码器与生成器平行,另一个变分自动编码器引入在生成器生成的数据之后;对判别模块的改进是:将原来的单一判别器改成两个判别器,其中一个判别器接收生成数据与原始数据,另一个判别器接收原始数据经过变分自动编码器输出的数据和生成数据经过另一个变分自动编码器输出的数据;
6、计算单点氨基酸突变相对可能性指数模块,利用训练好的改进生成对抗网络学习每个蛋白质的氨基酸序列分布,构建蛋白质突变预测矩阵,用于计算所有单点氨基酸突变相对野生型氨基酸的相对可能性指数;
7、致病性预测模块,通过在单点氨基酸突变的相对可能性指数分布上引入高斯混合模型,并应用变分贝叶斯估计进行拟合,以提升致病性预测的准确性;其中,该致病性预测模块将突变分为三个关键类别,分别为良性、不确定和致病,并提供单点氨基酸突变得分来解释基因错义突变致病性。
8、进一步,所述数据加载模块使用deepmsa获取蛋白质高质量多序列比对数据,格式为a2m,其中,对于蛋白质单体,通过dmsa、qmsa和mmsa三个迭代msa搜索流程,涵盖全基因组和宏基因组数据库,随后利用折叠模型对msa进行评分和排序;对于蛋白质多体,通过将组分链的单体msa配对生成一系列混合msa,然后根据msa深度和单体链折叠得分的组合评分选择最优多体msa,其中蛋白质高质量多序列比对数据包括蛋白质序列名称、蛋白质焦点序列和蛋白质序列数据,蛋白质序列数据和蛋白质焦点序列可能来自同一家族、同一亚型或不同物种。
9、进一步,所述数据预处理模块执行以下操作:
10、生成野生型氨基酸:对蛋白质焦点序列进行独热编码,根据氨基酸字母的转换生成野生型氨基酸;
11、生成独热编码数据:将蛋白质高质量多序列比对数据中蛋白质序列名称与蛋白质序列数据建立映射关系,然后针对每一个蛋白质序列数据进行以下处理:将缺失字符替换为短划线、删除野生型氨基酸中的缺失位点对应的列、识别蛋白质序列数据中含有过多缺失位点的片段、识别焦点列、将非焦点列转为小写和过滤片段序列,预处理后的蛋白质序列数据根据氨基酸字母转换为对应的蛋白质序列独热编码数据,通过计算每个蛋白质序列独热编码数据非空位置的数量和相似性,并针对相似性进行筛选,生成权重值,相似性越大,则权重值越大,这有助于强调那些在任务中更为关键的蛋白质序列特征;
12、生成单点氨基酸突变数据:遍历蛋白质焦点序列中每个氨基酸字母,为每个氨基酸生成可能有效的单点氨基酸突变数据。
13、进一步,所述训练模块执行以下操作:
14、a、将蛋白质高质量多序列比对数据的蛋白质序列独热编码数据输入到改进生成对抗网络的生成模块,所述生成模块包括生成器和两个变分自动编码器,其中一个变分自动编码器用于接收原始数据,另一个变分自动编码器用于接收生成数据,所述生成器试图生成与真实数据相似的数据,接收原始数据的变分自动编码器负责加强蛋白质序列数据之间关联性的学习,有利于增强不同蛋白质序列数据的关键突变位置识别,接收生成数据的变分自动编码器是为了重构生成数据,有利于增强生成数据的真实性,减少噪声带来的干扰;
15、所述变分自动编码器包括改进的编码器和解码器,其中,对于编码器和解码器的改进是:由三层结构变成四层结构,并增加每一层数的神经元,这有利于捕捉到蛋白质序列数据的复杂模式和关系,并加速改进生成对抗网络的训练过程;
16、b、使用反向传播训练生成模块;
17、c、将真实数据和生成模块输出的数据输入到改进生成对抗网络的判别模块进行真伪分类,所述判别模块包括两个判别器,其中一个判别器接收生成数据与原始数据,另一个判别器接收原始数据经过变分自动编码器输出的数据和生成数据经过另一个变分自动编码器输出的数据;两个判别器的引入适应了生成模块的数据多样性和增强了判别模块的区分能力,生成模块和判别模块相互竞争,同时不断优化,最终生成模块能够生成更加真实和有意义的数据,这意味着能够捕获更多蛋白质序列数据的突变信息,学习其中突变的约束条件以及空间上的复杂依赖性;
18、d、使用反向传播训练判别模块,最终得到训练好的改进生成对抗网络。
19、进一步,改进的编码器由四层全连接神经网络组成,第一层为输入层,节点个数为5000,第二层为隐藏层,节点个数为512,第三层为隐藏层,节点个数为128,第四层为表征层,节点个数为64,激活函数均为relu;改进的解码器由四层全连接神经网络组成,第一层为输入层,节点个数为64,第二层为隐藏层,节点个数为128,第三层为隐藏层,节点个数为512,第四层为重构层,节点个数为5000,激活函数均为relu。
20、进一步,所述计算单点氨基酸突变相对可能性指数模块执行以下操作:
21、a、对每个氨基酸所有可能有效的单点氨基酸突变数据的有效性进行遍历验证,将有效单点氨基酸突变数据和野生型氨基酸添加到有效单点氨基酸突变列表中,对有效单点氨基酸突变列表的每一个单点氨基酸突变进行独热编码,所得独热编码单点氨基酸突变序列转换为python机器学习库中pytorch张量,用于批量加载独热编码单点氨基酸突变序列;
22、b、利用训练好的改进生成对抗网络对每个批次的独热编码单点氨基酸突变序列进行预测,将预测结果储存到蛋白质氨基酸突变预测矩阵,用于计算野生型氨基酸的平均预测值和单点氨基酸突变的平均预测值;
23、c、计算蛋白质氨基酸突变预测矩阵每一行的均值生成一维数组,该一维数组的第一个元素是野生型氨基酸的平均预测值,其它元素是单点氨基酸突变的平均预测值;
24、d、计算单点氨基酸突变的平均预测值相对于野生型氨基酸的平均预测值的差值,最终得到所有单点氨基酸突变相对野生型氨基酸的相对可能性指数是这些差值的负数。
25、进一步,所述致病性预测模块执行以下操作:
26、a、读取计算单点氨基酸突变相对可能性指数模块中生成的单点氨基酸突变的相对可能性指数,存储在数组array中;
27、b、使用python机器学习库中scikit-learn库的bayesiangaussianmixture类进行高斯混合模型的变分贝叶斯估计时,先将数据分为两个聚类:良性和致病,利用输入步骤a中生成的数组array拟合,实现原理如下:变分贝叶斯估计通过引入一个分布来逼近后验分布,以便简化计算,在高斯混合模型中,假设数据由多个高斯分布组成,每个高斯分布对应一个聚类,通过变分贝叶斯估计,在高斯混合模型训练过程中学习每个高斯分布的参数,同时学习每个数据点属于每个聚类的概率;
28、c、使用步骤a生成的数组array对高斯混合模型进行拟合,获取每个单点氨基酸突变属于每个聚类的概率p,最后使用python机器学习库中numpy库的argmax函数获得每个单点氨基酸突变概率最大的聚类标签,并将这些标签存储到聚类标签列表list中;
29、d、在高斯混合模型中,能够根据概率阈值实现重分类,阈值的范围属于0到1,遍历高斯混合模型得到的聚类标签列表list中的每个突变,如果概率大于阈值,则认为分类在置信水平上是能够接受的,否则认为置信度不够,将其分配到不确定类中,将重分类的重分类标签储存到一个新的列表newlist中;
30、e、在完成重分类和得到每个单点氨基酸突变属于每个聚类的概率p的基础上,计算单点氨基酸突变得分s,s∈[0,1],其中0认为是良性,1认为是致病,如果某个单点氨基酸突变的聚类标签为致病性,则s=p,如果聚类标签为良性,则s=1-p;
31、f、最后根据单点氨基酸突变得分s来解释基因错义突变致病性。
32、本发明与现有技术相比,具有如下优点与有益效果:
33、1、deepmsa通过对全基因组和宏基因组数据库的搜索,系统可以涵盖更广泛的数据源,进一步丰富了训练网络的数据。
34、2、通过深度学习方法分析蛋白质高质量多序列比对数据,增强了智能诊断系统的准确性。
35、3、改进生成对抗网络实现了对蛋白质氨基酸序列更准确的建模,有效捕获了突变信息和复杂依赖性。这种改进不仅提高了生成器的性能,使其学习更具真实性和合理性的蛋白质序列分布,同时也增强了判别器的监测和评估能力。
36、4、变分自动编码器的四层结构有利于捕捉到蛋白质序列数据的复杂模式和关系,并加速改进生成对抗网络的训练过程,更有效地学习蛋白质序列数据的抽象表示,也不容易受到训练数据中的噪声或异常值的影响。
37、5、本发明能够在基因错义突变的预测中灵活而精准地判定致病性,提供可解释性强且置信水平可调的综合预测,增强了对基因错义突变的准确性和可信度。
38、6、本发明对基因错义突变致病性的准确挖掘不仅可以为精准诊断提供有效的临床标记物,还可对疾病致病机理的理解和个性化精准用药提供理论指导。
39、7、作为科学发现的独立手段以及临床和实验方法的重要补充,本发明可为基因突变致病性位点的发现提供新思路。
- 还没有人留言评论。精彩留言会获得点赞!