生产6‑碳单体的方法和材料与流程
相关申请的交叉引用
本申请要求2014年11月14日提交的美国申请号62/079,903,和2015年11月13日提交的美国申请号62/255,276的权益,其公开内容通过提述以其整体并入本文。
发明领域
本发明提供了用于生产6碳单体的非天然存在的方法。本发明提供了使用具有β-酮硫解酶活性的多肽生物合成3-氧代-6-羟基己酰基coa,并且使用一种或多种具有以下酶活性的多肽,或者使用一种或多种表达此类酶的重组宿主将3-氧代-6-羟基己酰基coa酶促转化为6-羟基己酸:具有3-羟基酰基coa脱氢酶活性的多肽、具有3-酮酰基coa还原酶活性的多肽、具有烯酰coa水合酶活性的多肽、具有反式-2-烯酰coa还原酶活性的多肽,和具有硫酯酶活性的多肽。本发明还涉及使用一种或多种分离的酶或者使用表达一种或多种此类酶的重组宿主细胞将6-羟基己酸转化为己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,和1,6-己二醇的一种或多种的方法,所述分离的酶诸如具有脱氢酶活性的多肽、具有还原酶活性的多肽、具有水合酶活性的多肽、具有硫酯酶活性的多肽、具有单加氧酶活性的多肽、具有转氨酶活性的多肽。
发明背景
尼龙是聚酰胺,其一般通过二胺与二羧酸的缩合聚合(condensationpolymerization)合成。类似地,可以通过内酰胺的缩合聚合生成尼龙。一种普遍存在的尼龙是尼龙6,6,其通过六亚甲基二胺(hmd)和己二酸的缩合聚合生成。可以通过己内酰胺的开环聚合生成尼龙6。因此,己二酸,六亚甲基二胺和己内酰胺是尼龙生产中重要的中间体(anton&baird,polyamidesfibers,encyclopediaofpolymerscienceandtechnology,2001)。
工业上,己二酸和己内酰胺通过环己烷的空气氧化产生。环己烷的空气氧化在一系列步骤中产生环己酮(k)和环己醇(a)和混合物,命名为ka油。ka油的硝酸氧化产生己二酸(musser,adipicacid,ullmann'sencyclopediaofindustrialchemistry,2000)。己内酰胺由环己酮经由其肟和随后的酸重排产生(fuchs,kieczkaandmoran,caprolactam,ullmann'sencyclopediaoflndustrialchemistry,2000)。
工业上,通过将c6构建快氢氰化成己二腈,然后氢化成hmd来制备六亚甲基二胺(hmd)(herzogandsmiley,hexamethylenediamine,ullmann'sencyclopediaofindustrialchemistry,2012)。
鉴于对石油化学原料的依赖;生物技术通过生物催化提供了一种替代方法。生物催化是使用生物催化剂(如酶)来进行有机化合物的生物化学转化。
生物来源的原料和石油化学原料都是用于生物催化过程的可行的原料。
发明概述
因此,针对这种背景,显然需要生产己二酸、己内酰胺、6-氨基己酸、六亚甲基二胺和1,6-己二醇(下文称为“c6构建快(c6buildingblock)”)的一种或多种的可持续方法,其中所述方法是基于生物催化剂的。
本文至少部分基于这样的发现,即可以构建生物化学途径,其(除其他外)使用β-酮硫解酶以生产6-羟基己酸,6-羟基己酸可以在一个或多个酶促步骤中转化为己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,或1,6-己二醇。己二酸(adipicacid)和己二酸盐/酯、6-羟基己酸(6-hydroxyhexanoicacid)和6-羟基己酸盐/酯,以及6-氨基己酸(6-aminohexanoicacid)和6-氨基己酸盐/酯在本文中可互换使用,指任何其中性或离子化形式的化合物,包括其任何盐形式。本领域技术人员应当理解,具体形式将取决于ph。
令人惊奇地,在面对最优性原则下,已经发现可以组合适当的非天然途径、原料、宿主微生物、对宿主生物化学网络的弱化策略,和培养策略以有效地生产作为c6构建块的6-羟基己酸,或将6-羟基己酸转化为其他c6构建块,如己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,或1,6-己二醇。
在一些实施方案中,可以使用硫酯酶、醛脱氢酶、6-氧代己酸脱氢酶、7-氧代庚酸脱氢酶,或单加氧酶(例如与氧化还原酶和铁氧还蛋白组合)酶促形成末端羧基。参见图1和图2。
在一些实施方案中,可以使用ω-转氨酶或脱酰基酶酶促形成末端氨基。参见图4。ω-转氨酶可以与seqidnos.7-12中所列的任一个氨基酸序列具有至少70%序列同一性。
在一些实施方案中,可以使用醇脱氢酶酶促形成末端羟基。参见图1和图5。
一方面,本文件特征在于生产3-氧代-6-羟基己酰基coa的方法。所述方法包括使用归类于ec.2.3.1.-(如ec2.3.1.16或ec2.3.1.174)下的具有β-酮硫解酶活性的多肽将4-羟基丁酰基coa酶促转化为3-氧代-6-羟基己酰基coa。所述具有β-酮硫解酶活性多肽可以与seqidno:1、seqidno:13或seqidno:14中所列的氨基酸序列具有至少70%序列同一性。所述方法可以包括使用以下的酶将3-氧代-6-羟基己酰基coa酶促转化为6-羟基己酸:3-羟基酰基coa脱氢酶或3-酮酰基coa还原酶、烯酰coa水合酶、反式-2-烯酰coa还原酶,和硫酯酶或coa转移酶。所述3-羟基酰基coa脱氢酶或3-酮酰基coa还原酶可以归类于ec1.1.1.35、ec1.1.1.36、ec1.1.1.100,或ec1.1.1.157下。所述烯酰coa水合酶可以被归类于ec4.2.1.17或ec4.2.1.119下。所述反式-2-烯酰coa还原酶可以被归类于ec1.3.1.38、ec1.3.1.44,或ec1.3.1.8下。所述反式-烯酰coa还原酶可以与seqidno:15或seqidno:16中所列的氨基酸序列具有至少70%序列同一性。
一方面,本文件特征在于生物合成6-羟基己酸的方法,所述方法包括使用归类于ec.2.3.1.-(例如ec2.3.1.16或ec2.3.1.174)下的β-酮硫解酶从4-羟基丁酰基coa酶促合成3-氧代-6-羟基己酰基coa,并且将3-氧代-6-羟基己酰基coa酶促转化为6-羟基己酸。所述β-酮硫解酶可以与seqidno:1、seqidno:13或seqidno:14中所列的氨基酸序列具有至少70%序列同一性。可以使用3-羟基酰基coa脱氢酶或3-酮酰基coa还原酶将3-氧代-6-羟基己酰基coa转化为3-羟基-6-羟基己酰基coa,可以使用烯酰coa水合酶将3-羟基-6-羟基己酰基coa转化为2,3-脱氢-6-羟基己酰基coa,可以使用反式-2-烯酰coa还原酶将2,3-脱氢-6-羟基己酰基coa转化为6-羟基己酰基coa,以及可以使用硫酯酶或coa转移酶将6-羟基己酰基coa转化为6-羟基己酸。
任何方法还可以包括在一个或多个步骤中将6-羟基己酸酶促转化为己二酸、6-氨基己酸、己内酰胺,六亚甲基二胺,或1,6-己二醇。
例如,可以使用一种或多种以下的酶将6-羟基己酸转化为己二酸:单加氧酶、醇脱氢酶、4-羟基丁酸脱氢酶、5-羟基戊酸脱氢酶、6-羟基己酸脱氢酶、7-氧代庚酸脱氢酶、6-氧代己酸脱氢酶、5-氧代戊酸脱氢酶,或醛脱氢酶。
例如,可以使用一种或多种以下的酶将6-羟基己酸转化为6-氨基己酸:醇脱氢酶、6-羟基己酸脱氢酶、5-羟基戊酸脱氢酶、4-羟基丁酸脱氢酶,和ω-转氨酶。所述ω-转氨酶可以与seqidnos:7-12中所列的任一个氨基酸序列具有至少70%序列同一性。
例如,可以使用一种或多种以下的酶将6-羟基己酸转化为己内酰胺:醇脱氢酶、6-羟基己酸脱氢酶、5-羟基戊酸脱氢酶、4-羟基丁酸脱氢酶、ω-转氨酶,和酰胺水解酶。所述ω-转氨酶可以与seqidnos:7-12中所列的任一个氨基酸序列具有至少70%序列同一性。
例如,可以使用一种或多种以下的酶将6-羟基己酸转化为六亚甲基二胺:羧酸还原酶、ω-转氨酶、醇脱氢酶、n-乙酰基转移酶,和乙酰腐胺脱酰基酶。所述ω-转氨酶可以与seqidnos:7-12中所列的任一个氨基酸序列具有至少70%序列同一性。
例如,可以使用羧酸还原酶和醇脱氢酶将6-羟基己酸转化为1,6-己二醇。所述羧酸还原酶可以与seqidnos:2-6中所列的任一个氨基酸序列具有至少70%序列同一性。
在任何方法中,可以从2-酮戊二酸酶促生产4-羟基丁酰基coa。例如,可以使用以下酶的一种或多种从2-酮戊二酸酶促生产4-羟基丁酰基coa:谷氨酸合酶;2-酮戊二酸脱羧酶;支链脱羧酶;谷氨酸脱羧酶;ω-转氨酶、coa转移酶、coa连接酶,和醇脱氢酶。
在本文描述的任何方法中,可以使用以下的酶通过在己二酸半醛(也称为6-氧代己酸)中形成第二末端官能团来生产己二酸:(i)归类于ec1.2.1.3下的醛脱氢酶,(ii)归类于ec1.2.1.63下的6-氧代己酸脱氢酶如由chne编码,或归类于ec1.2.1.-下的7-氧代庚酸脱氢酶(例如thng的基因产物)或者(iii)细胞色素p450家族中的单加氧酶。
在本文描述的任何方法中,可以使用归类于ec2.61.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82下的ω-转氨酶通过在己二酸半醛中形成第二末端官能团来生产6-氨基己酸。
在本文描述的任何方法中,可以使用归类于ec3.5.2.-下的酰胺水解酶从6-氨基己酸生产己内酰胺。从6-氨基己酸的末端羧基和末端氨基生产与己内酰胺相关的酰胺键。
在本文描述的任何方法中,可以通过以下(i)或者(ii)在6-氨基己醛中形成第二末端官能团:(i)使用归类于ec2.61.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48或ec2.6.1.82下的ω-转氨酶,或者(ii)使用例如归类于ec3.5.1.17下的脱酰基酶。
在本文描述的任何方法中,可以使用归类于ec1.1.1.-(例如ec1.1.1.1、1.1.1.2、1.1.1.21,或1.1.1.184)下的醇脱氢酶,如由ymr318c、yqhd或caa81612.1编码的醇脱氢酶,在6-羟基己醛中形成第二末端官能团来生产1,6己二醇。
在一些实施方案中,生物原料可以是或可以源自单糖、二糖、木质纤维素、半纤维素、纤维素、木质素、乙酰丙酸和甲酸、甘油三酯、甘油、脂肪酸、农业废物、浓缩酒糟(condenseddistillers'solubles),或城市废物。
在一些实施方案中,非生物原料可以是或可以源自天然气、合成气、co2/h2、甲醇、乙醇、苯甲酸盐、非挥发性残留物(nvr)或来自环己烷氧化过程的碱洗液(causticwash)废物流、或对苯二甲酸/异酞酸混合物废物流。
在一些实施方案中,通过在选择的环境中连续培养来改善宿主对高浓度的一种或多种c6构件块的耐受。
在一些实施方案中,所述宿主微生物的生物化学网络是弱化的或增强的以(1)确保乙酰coa和4-羟基丁酰基coa的细胞内利用度,(2)创建nadh或nadph不平衡,其仅可经由形成一种或多种c6构件块来平衡,(3)防止导致并包括c6构件块的中心代谢物、中心前体的降解和(4)确保从细胞有效的流出。
在一些实施方案中,培养策略用于实现厌氧、微需氧,或需氧培养条件。
在一些实施方案中,培养策略包括限制营养物,如限制氮、磷酸盐或氧。
在一些实施方案中,例如使用发酵策略,通过单一类型的微生物(例如含有一种或多种外源核酸的重组宿主)来生产一种或多种c6构建块。
另一方面,本文件特征在于包括至少一种编码以下酶的外源核酸的重组宿主:(i)β-酮硫解酶,(ii)硫酯酶或coa转移酶,以及一种或多种(iii)3-羟基酰基coa脱氢酶或3-酮酰基coa还原酶,(iv)烯酰coa水合酶,和(v)反式-2-烯酰coa还原酶,所述宿主生产6-羟基己酸。
生产6-羟基己酸的宿主还可以包括一种或多种以下的外源酶:单加氧酶、醇脱氢酶、4-羟基丁酸脱氢酶、5-羟基戊酸脱氢酶、6-羟基己酸脱氢酶、7-氧代庚酸脱氢酶、6-氧代己酸脱氢酶、5-氧代戊酸脱氢酶,或醛脱氢酶,所述宿主进一步生产己二酸。
生产6-羟基己酸的宿主还可以包括一种或多种以下的外源酶:单加氧酶、转氨酶、6-羟基己酸脱氢酶、5-羟基戊酸脱氢酶、4-羟基丁酸脱氢酶,和醇脱氢酶,所述宿主进一步生产6-氨基己酸。此类宿主还可以包括外源酰胺水解酶,所述宿主进一步生产己内酰胺。
生产6-羟基己酸的宿主还可以包括一种或多种以下的外源酶:羧酸还原酶、ω-转氨酶、脱酰基酶、n-乙酰基转移酶,或醇脱氢酶,所述宿主进一步生产六亚甲基二胺。
生产6-羟基己酸的宿主还可以包括外源羧酸还原酶和外源醇脱氢酶,所述宿主进一步生产1,6-己二醇。
本文所述的任何重组宿主还可以包括一种或多种以下的外源酶:谷氨酸合酶、2-酮戊二酸脱羧酶、支链脱羧酶、谷氨酸脱羧酶、ω-转氨酶、coa-连接酶、coa-转移酶,和醇脱氢酶。
任何重组宿主可以是原核生物,如选自下组的原核生物:埃希氏菌属(escherichia);梭菌属(clostridia);棒状杆菌属(corynebacteria);贪铜菌属(cupriavidus);假单胞菌属(pseudomonas);代尔夫特菌属(delftia);芽孢杆菌属(bacillus);乳杆菌属(lactobacillus);乳球菌属(lactococcus);和红球菌属(rhodococcus)。例如,所述原核生物可以选自下组:大肠杆菌(escherichiacoli)、杨氏梭菌(clostridiumljungdahlii)、自产乙醇梭菌(clostridiumautoethanogenum)、克鲁佛梭菌(clostridiumkluyveri)、谷氨酸棒状杆菌(corynebacteriumglutamicum)、钩虫贪铜菌(cupriavidusnecator)、耐金属贪铜菌(cupriavidusmetallidurans)、荧光假单胞菌(pseudomonasfluorescens)、恶臭假单胞菌(pseudomonasputida)、食油假单胞菌(pseudomonasoleavorans)、食酸代尔夫特菌(delftiaacidovorans)、枯草芽胞杆菌(bacillussubtillis)、德氏乳杆菌(lactobacillusdelbrueckii)、乳酸乳球菌(lactococcuslactis),和马红球菌(rhodococcusequi)。此类原核生物还可以是用于构建本文中所述的能够生产c6构件块的重组宿主细胞的基因的。
任何重组宿主可以是真核生物,如选自下组的真核生物:曲霉属(aspergillus)、酵母属(saccharomyces)、毕赤氏酵属(pichia)、耶罗维亚酵母属(yarrowia)、伊萨酵母属(issatchenkia)、德巴利酵母属(debaryomyces)、arxula属和克鲁维酵母菌属(kluyveromyces)。例如,所述真核生物可以选自下组:黑曲霉(aspergillusniger)、酿酒酵母(saccharomycescerevisiae)、巴斯德毕赤酵母(pichiapastoris)、解脂耶罗维亚酵母(yarrowialipolytica)、东方伊萨酵母(issathenkiaorientalis)、汉逊德巴利酵母(debaryomyceshansenii)、arxulaadenoinivorans和乳酸克鲁维酵母(kluyveromyceslactis)。此类真核生物还可以是用于构建本文中所述的能够生产c6构件块的重组宿主细胞的基因的来源。
本文所述的任何重组宿主还可以包括一种或多种以下酶的弱化:聚羟基烷酸酯(polyhydroxyalkanoate)合酶、乙酰coa硫酯酶、形成乙酸的磷酸转乙酰酶、乙酸激酶、乳酸脱氢酶、menaquinol-富马酸氧化还原酶、形成乙醇的醇脱氢酶、丙糖磷酸异构酶、丙酮酸脱羧酶、葡萄糖-6-磷酸异构酶、消耗nadh的转氢酶、nadh特异性谷氨酸脱氢酶,和利用nadh/nadph的谷氨酸脱氢酶、庚二酰-coa脱氢酶;接受c6构件块和中心前体作为底物的的乙酰coa脱氢酶;丁酰基coa脱氢酶,或己二酰coa合成酶。
本文所述的任何重组宿主还可以过表达一种或多种编码以下的基因:乙酰coa合成酶;6-磷酸葡萄糖酸脱氢酶;转酮醇酶;嘌呤核苷酸转氢酶;甘油醛-3p-脱氢酶;苹果酸酶;葡萄糖-6-磷酸脱氢酶;葡萄糖脱氢酶;果糖1,6二磷酸酶;l-丙氨酸脱氢酶;l-谷氨酸脱氢酶;甲酸脱氢酶;l-谷氨酰胺合成酶;二胺转运蛋白;二羧酸转运蛋白,和/或多药转运蛋白。
本文所述的许多酶催化可逆反应,并且感兴趣的反应可以是所述反应的逆反应。示于图1至5的示意途径说明了每种中间体的感兴趣的反应。
在一些方面,本文件特征在于核酸构建体和/或表达载体,其包含(a)多核苷酸,其编码具有β-酮硫解酶活性的多肽,其中所述多核苷酸可操作地连接于一种或多种指导多肽生产的异源控制序列,并且其中所述具有β-酮硫解酶活性的多肽选自下组:(a)与seqidnos:1、13或14的多肽具有至少70%序列同一性的多肽;(b)多核苷酸,其编码具有ω-转氨酶活性的多肽,其中所述多核苷酸可操作地连接于一种或多种指导多肽生产的异源控制序列,并且其中所述具有ω-转氨酶活性的多肽选自下组:(a)与seqidnos:7-12的多肽具有至少70%序列同一性的多肽;多核苷酸,其编码具有羧酸还原酶活性的多肽,其中所述多核苷酸可操作地连接于一种或多种指导多肽生产的异源控制序列,并且其中所述具有羧酸还原酶活性的多肽选自下组:(a)与seqidnos:2-6的多肽具有至少70%序列同一性的多肽;或者(d)多核苷酸,其编码具有以下酶活性的多肽:3-羟基酰基coa脱氢酶活性、3-酮酰基coa还原酶活性、烯酰coa水合酶活性、反式-2-烯酰coa还原酶活性、硫酯酶coa转移酶活性、单加氧酶活性、醇脱氢酶活性、4-羟基丁酸脱氢酶活性、5-羟基戊酸脱氢酶活性、6-羟基己酸脱氢酶活性、7-氧代庚酸脱氢酶活性、6-氧代己酸脱氢酶活性、5-氧代戊酸脱氢酶活性、醛脱氢酶活性、6-羟基己酸脱氢酶活性、5-羟基戊酸脱氢酶活性、ω-转氨酶活性、酰胺水解酶活性、谷氨酸合成酶活性;2-酮戊二酸脱羧酶活性;支链脱羧酶活性;谷氨酸脱羧酶活性;ω-转氨酶活性活性;coa转移酶活性、coa连接酶活性。在一些实施方案中,本公开提供了含有上述核酸构建体或表达载体的组合物。
一方面,本文件特征在于生产生物衍生的六碳化合物的方法。所述生产生物衍生的六碳化合物的方法可以包括在条件下培养或生长本文所述的宿主达足够的时间段以生产生物衍生的六碳化合物,其中,任选地,所述生物衍生的六碳化合物选自下组:己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,或1,6-己二醇,及它们的组合。
一方面,本文件特征在于组合物,其包含如本文所述的生物衍生的六碳化合物以及除生物衍生的六碳化合物以外的化合物,其中所述生物衍生的六碳化合物选自下组:己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,或1,6-己二醇,及它们的组合。例如,所述生物衍生的六碳化合物是宿主细胞或生物体的细胞部分。
本文件特征还在于生物基聚合物,其包含生物衍生的己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,或1,6-己二醇,及它们的组合。
本文件特征还在于生物基树脂,其包含生物衍生的己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,或1,6-己二醇,及它们的组合,以及通过模制生物基树脂获得的模制的产品。
另一方面,本文件特征在于生产生物基聚合物的方法,其包括在聚合物生产反应中将生物衍生的己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,或1,6-己二醇与其自身或另一化合物发生化学反应。
另一方面,本文件特征在于生产生物基树脂的方法,其包括在树脂生产反应中将生物衍生的己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,或1,6-己二醇与其自身或另一化合物发生化学反应。
此外,描述的是包含具有β-酮硫解酶活性的多肽的生物化学网络,其中所述具有β-酮硫解酶活性的多肽将4-羟基丁酰基coa酶促转化为3-氧代-6-羟基己酰基coa。
所述生物化学网络还可以包括具有3-羟基酰基coa脱氢酶活性的多肽或具有3-酮酰基coa还原酶活性的多肽、具有烯酰coa水合酶活性的多肽、具有反式-2-烯酰coa还原酶活性的多肽、具有硫酯酶或coa转移酶活性的多肽,用于将3-氧代-6-羟基己酰基coa酶促转化为6-羟基己酸。
生物化学网络还可以包括一种或多种具有以下酶活性的多肽将6-羟基己酸酶促转化为己二酸:单加氧酶活性、醇脱氢酶活性、4-羟基丁酸脱氢酶活性、5-羟基戊酸脱氢酶活性、6-羟基己酸脱氢酶活性、7-氧代庚酸脱氢酶活性、6-氧代己酸脱氢酶活性、5-氧代戊酸脱氢酶活性,或醛脱氢酶活性。
生物化学网络还可以包括具有ω-转氨酶活性的多肽将6-羟基己酸酶促转化为6-氨基己酸。
生物化学网络还可以包括具有酰胺水解酶活性的多肽将6-氨基己酸转化为己内酰胺。
生物化学网络还可以包括一种或多种具有ω-转氨酶或脱酰基酶活性的多肽将6-羟基己酸酶促转化为六亚甲基二胺。
生物化学网络还可以包括一种或多种具有醇脱氢酶活性的多肽,通过在6-羟基己烷中形成第二末端官能团1,6己二醇。
一方面,生物化学网络是非天然存在的生物化学网络,其包含至少一种图1至图5的底物、至少一种编码具有至少一种图1至图5的活性的多肽的外源核酸,以及至少一种图1至图5的产物。
本发明的一方面,描述的是用于形成至少一种图1至图5化合物的步骤。本发明的一方面,描述的是用于形成至少一种图1至图5化合物的手段。还描述的是使用一种或多种具有以下酶活性的多肽获得己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,或1,6-己二醇的手段:β-酮硫解酶活性、3-羟基酰基coa脱氢酶活性、3-酮酰基coa还原酶活性、烯酰coa水合酶活性、反式-2-烯酰coa还原酶活性、硫酯酶或coa转移酶活性、单加氧酶活性、醇脱氢酶活性、4-羟基丁酸脱氢酶活性、5-羟基戊酸脱氢酶活性、6-羟基己酸脱氢酶活性、7-氧代庚酸脱氢酶活性、6-氧代己酸脱氢酶活性、5-氧代戊酸脱氢酶活性、醛脱氢酶活性、ω-转氨酶活性、酰胺水解酶活性、ω-转氨酶或脱酰基酶活性。
另一方面,本文件特征在于组合物,其包含一种或多种具有以下酶活性的多肽以及己二酸、6-氨基己酸、六亚甲基二胺、己内酰胺,或1,6-己二醇的至少一种:β-酮硫解酶活性、3-羟基酰基coa脱氢酶活性、3-酮酰基coa还原酶活性、烯酰coa水合酶活性、反式-2-烯酰coa还原酶活性、硫酯酶或coa转移酶活性、单加氧酶活性、醇脱氢酶活性、4-羟基丁酸脱氢酶活性、5-羟基戊酸脱氢酶活性、6-羟基己酸脱氢酶活性、7-氧代庚酸脱氢酶活性、6-氧代己酸脱氢酶活性、5-氧代戊酸脱氢酶活性、醛脱氢酶活性、ω-转氨酶活性、酰胺水解酶活性、ω-转氨酶或脱酰基酶活性。所述组合物可以是细胞的。
本领域技术人员理解,当存在于亲本化合物中的酸性质子被金属离子,例如碱金属离子,碱土金属离子或铝离子替代;或与有机碱配位时,含有羧酸基团的化合物(包括但不限于有机一元酸,羟基酸,氨基酸和二羧酸)得以形成或被转化成其离子盐形式。可接受的有机碱包括但不限于乙醇胺,二乙醇胺,三乙醇胺,氨丁三醇,n-甲基葡糖胺等。可接受的无机碱包括但不限于氢氧化铝,氢氧化钙,氢氧化钾,碳酸钠,氢氧化钠等。将本发明的盐分离为盐或通过经由添加酸或用酸性离子交换树脂处理将ph降至pka以下而转化成游离酸。
本领域技术人员理解,含有胺基团的化合物(包括但不限于有机胺,氨基酸和二胺)得以形成或被转化为它们的离子盐形式,例如通过向胺中加入酸性质子形成铵盐,与无机酸如盐酸,氢溴酸,硫酸,硝酸,磷酸等形成;或与有机酸形成,包括但不限于乙酸,丙酸,己酸,环戊烷丙酸,乙醇酸,丙酮酸,乳酸,丙二酸,琥珀酸,苹果酸,马来酸,富马酸,酒石酸,柠檬酸,苯甲酸,3-(4-羟基苯甲酰基)苯甲酸,肉桂酸,扁桃酸,甲磺酸,乙磺酸,1,2-乙二磺酸,2-羟基乙磺酸,苯磺酸,2-萘磺酸,4-甲基双环-[2.2.2]辛-2-烯-1-羧酸,葡庚糖酸,4,4'-亚甲基双-(3-羟基-2-烯-1-羧酸),3-苯基丙酸,三甲基乙酸,叔丁基乙酸,月桂基硫酸,葡糖酸,谷氨酸,羟基萘甲酸,水杨酸,硬脂酸,粘康酸等。可接受的无机碱包括但不限于氢氧化铝,氢氧化钙,氢氧化钾,碳酸钠,氢氧化钠等。本发明的盐以盐的形式分离或通过经由添加碱或用碱性离子交换树脂处理将ph至高于pkb转化成游离胺。
本领域技术人员理解,含有胺基团和羧酸基团两者的化合物(包括但不限于氨基酸)通过以下形成或转化成它们的离子盐形式:通过1)与无机酸形成的酸加成盐,所述无机酸包括但不限于盐酸,氢溴酸,硫酸,硝酸,磷酸等;或与有机酸形成的酸加成盐,所述有机酸包括但不限于乙酸,丙酸,己酸,环戊烷丙酸,乙醇酸,丙酮酸,乳酸,丙二酸,琥珀酸,苹果酸,马来酸,富马酸,酒石酸,柠檬酸,苯甲酸,3-(4-羟基苯甲酰基)苯甲酸,肉桂酸,扁桃酸,甲磺酸,乙磺酸,1,2-乙二磺酸,2-羟基乙磺酸,苯磺酸,2-萘磺酸,4-甲基双环-[2.2.2]辛-2-烯-1-羧酸,葡庚糖酸,4,4'-亚甲基双-(3-羟基-2-烯-1-羧酸),3-苯基丙酸,三甲基乙酸酸,叔丁基乙酸,月桂基硫酸,葡糖酸,谷氨酸,羟基萘甲酸,水杨酸,硬脂酸,粘康酸等。可接受的无机碱包括但不限于氢氧化铝,氢氧化钙,氢氧化钾,碳酸钠,氢氧化钠等,或2)当存在于亲本化合物中的酸性质子被金属离子,例如碱金属离子,碱土金属离子或铝离子置换时;或与有机碱配位。可接受的有机碱包括但不限于乙醇胺,二乙醇胺,三乙醇胺,氨丁三醇,n-甲基葡糖胺等。可接受的无机碱包括但不限于氢氧化铝,氢氧化钙,氢氧化钾,碳酸钠,氢氧化钠等。本发明的盐可以作为盐分离或通过经由添加酸或用酸性离子交换树脂处理将ph降至pka以下而转化为游离酸。
除非另有定义,本文中使用的所有技术和科学术语与本发明所属领域的普通技术人员的通常理解具有相同的含义。尽管与本文中所述的方法和材料类似或等同的方法和材料可用于实施本发明,但下文描述了合适的方法和材料。本文中提及的所有出版物,专利申请,专利和其它参考文献,包括具有登录号的genbank和ncbi提交物通过引用整体并入。在冲突的情况下,以本说明书(包括定义)为准。此外,材料,该方法和实施例仅仅是说明性的,而不是限制性的。
在下面的附图和描述中阐述了本发明的一个或多个实施例的细节。本发明的其它特征,目的和优点将从说明书和附图以及权利要求书中显而易见。根据专利法中的标准实践,权利要求书中的词语“包括”可以被“基本上由...组成”或由“由...组成”替代。
附图简述
图1是使用2-酮戊二酸作为中心代谢物导致6-羟基己酸的示例性生物化学途径的示意图。
图2是使用6-羟基己酸作为中心前体导致己二酸的示例性生物化学途径的示意图。
图3是使用6-羟基己酸作为中心前体导致6-氨基己酸的示例性生物化学途径的示意图,以及从6-氨基己酸导致己内酰胺的示例性生物化学途径的示意图。
图4是使用6-氨基己酸、6-羟基己酸、己二酸半醛,或1,6-己二醇作为中心前体导致六亚甲基二胺的示例性生物化学途径的示意图。
图5是使用6-羟基己酸作为中心前体导致1,6-己二醇的示例性生物化学途径的示意图。
图6包含以下酶的氨基酸序列:钩虫贪铜菌(cupriavidusnecator)β-酮硫解酶(参见genbank登录号aac38322.1,seqidno:1)、海分枝杆菌(mycobacteriummarinum)羧酸还原酶(参见genbank登录号acc40567.1,seqidno:2)、耻垢分枝杆菌(mycobacteriumsmegmatis)羧酸还原酶(参见genbank登录号abk71854.1,seqidno:3)、segniliparusrugosus羧酸还原酶(参见genbank登录号efv11917.1,seqidno:4)、马赛分枝杆菌(mycobacteriummassiliense)羧酸还原酶(参见genbank登录号eivl1143.1,seqidno:5)、segniliparusrotundus羧酸还原酶(参见genbank登录号adg98140.1,seqidno:6)、紫色色杆菌(chromobacteriumviolaceum)ω-转氨酶(参见genbank登录号aaq59697.1,seqidno:7)、铜绿假单胞菌(pseudomonasaeruginosa)ω-转氨酶(参见genbank登录号aag08191.1,seqidno:8)、丁香假单胞菌(pseudomonassyringae)ω-转氨酶(参见genbank登录号aay39893.1,seqidno:9)、球形红杆菌(rhodobactersphaeroides)ω-转氨酶(参见genbank登录号aba81135.1,seqidno:10)、大肠杆菌ω-转氨酶(参见genbank登录号aaa57874.1,seqidno:11)、河流弧菌(vibriofluvialis)ω-转氨酶(参见genbank登录号aea39183.1,seqidno:12)、大肠杆菌β-酮硫解酶(参见genbank登录号aac74479.1,seqidno:13)、氨基丁酸羧菌(clostridiumaminobutyricum)coa转移酶(参见genbank登录号cab60036.2,seqidno:14)、齿垢密螺旋体(treponemadenticola)烯酰coa还原酶(参见genbank登录号aas11092.1,seqidno:15)、euglenagracilis烯酰coa还原酶(参见genbank登录号aaw66853.1,seqidno:16)以及鼠伤寒沙门氏菌(salmonellatyphimurium)脱羧酶(参见genbank登录号cac48239.1,seqidno:17)。
图7是总结了20分钟后在340nm处吸光度变化的柱状图,其是仅酶对照(无底物)的nadph消耗和羧酸还原酶活性的量度。
图8是20分钟后在340nm处吸光度变化的柱状图,其是相对于空载体对照的nadph消耗和将6-羟基己酸转化为6-羟基己醛的羧酸还原酶活性的量度。
图9是20分钟后在340nm处吸光度变化的柱状图,其是相对于空载体对照的nadph消耗和将n6-乙酰基-6-氨基己酸转化为n6-乙酰基-6-氨基己醛的羧酸还原酶活性的量度。
图10是20分钟后在340nm处吸光度变化的柱状图,其是相对于空载体对照的nadph消耗和将己二酸半醛转化为己二醛的羧酸还原酶活性的量度。
图11是总结了4小时后丙酮酸至l-丙氨酸的转化百分比(mol/mol)的柱状图,其作为仅酶的对照(无底物)的ω-转氨酶活性的量度。
图12是24小时后丙酮酸至l-丙氨酸的转化百分比(mol/mol)的柱状图,其作为相对于空载体对照的将6-氨基己酸转化为己二酸半醛的ω-转氨酶活性的量度。
图13是4小时后l-丙氨酸至丙酮酸的转化百分比(mol/mol)的柱状图,其作为相对于空载体对照的将己二酸半醛转化为6-氨基己酸的ω-转氨酶活性的量度。
图14是4小时后丙酮酸至l-丙氨酸的转化百分比(mol/mol)的柱状图,其作为相对于空载体对照的将六亚甲基二胺转化为6-氨基己醛的ω-转氨酶活性的量度。
图15是4小时后丙酮酸至l-丙氨酸的转化百分比(mol/mol)的柱状图,其作为相对于空载体对照的将n6-乙酰基-1,6-二氨基己烷转化为n6-乙酰基-6-氨基己醛的ω-转氨酶活性的量度。
图16是4小时后丙酮酸至l-丙氨酸的转化百分比(mol/mol)的柱状图,其作为相对于空载体对照的将6-氨基己醇转化为6-氧代己醇的ω-转氨酶活性的量度。
发明详述
一般而言,本文件提供了用于生产6-羟基己酸或己二酸、己内酰胺、6-氨基己酸、六亚甲基二胺,或1,6-己二醇的一种或多种(所有这些都被称为c6构件块)的酶、非天然途径、培养策略、原料、宿主微生物和宿主生物化学网络的弱化(attenuation)。如本文所用,术语“中心前体”用于表示本文所示的任何代谢途径中的任何代谢物,其导致c6构件块的合成。术语“中心代谢物”在本文中用于表示在所有微生物中产生以支持生长的代谢物。
本文所述的宿主微生物可以包括能够被操作,使得可以产生6-羟基己酸或者一种或多种其它c6构件块的内源途径。在内源途径中,宿主微生物天然表达催化途径中的反应的所有酶。包含工程化途径的宿主微生物不天然表达催化途径中的反应的所有酶,但已经被工程化改造,从而在宿主中表达途径中所有的酶。
如本文中提及核酸(或蛋白质)和宿主使用的,术语“外源”指不像其在自然界中被发现一样存在于特定类型细胞中(并且不能从特定类型细胞获得)的核酸或由所述核酸编码的蛋白质。如此,非天然存在的核酸一旦在宿主中即视为对于宿主而言外源的。重要的是注意非天然存在的核酸可含有在自然界中发现的核酸序列的核酸亚序列或片段,只要该核酸作为整体不存在于自然界中。例如,表达载体内含有基因组dna序列的核酸分子是非天然存在的核酸,如此一旦导入宿主中对于宿主细胞而言是外源的,因为该核酸分子作为整体(基因组dna加载体dna)不存在于自然界中。如此,作为整体不存在于自然界中的任何载体、自主复制的质粒或病毒(例如逆转录病毒、腺病毒、或疱疹病毒)视为非天然存在的核酸。由此得出结论通过pcr或限制内切性核酸酶处理产生的基因组dna片段以及cdna也视为非天然存在的核酸,因为它们作为未见于自然界的分开的分子存在。由此还得出结论任何以未见于自然界中的排布含有启动子序列和多肽编码序列(例如cdna或基因组dna)的任何核酸也是非天然存在的核酸。天然存在的核酸可以是对于特定宿主微生物而言外源的。例如,从酵母x的细胞分离的完整染色体一旦将该染色体导入酵母y的细胞中就酵母y细胞而言是外源核酸。
比较而言,如本文中提及核酸(例如基因)(或蛋白质)和宿主使用的,术语“内源的”指就像其在自然界中被发现一样的确存在于特定宿主中(并且可以从特定宿主获得)的核酸(或蛋白质)。此外,“内源表达”核酸(或蛋白质)的细胞就像其在自然界中被发现时相同特定类型的宿主那样表达所述核酸(或蛋白质)。此外,“内源生成”核酸、蛋白质、或其它化合物的宿主就像其在自然界中被发现时相同特定类型的宿主那样生成所述核酸、蛋白质、或化合物。
例如,根据宿主和宿主产生的化合物,除了β-酮硫解酶外,还可以在宿主中表达以下酶的一种或多种:3-羟基酰基coa脱氢酶、3-酮酰基coa还原酶、烯酰coa水合酶、反式-2-烯酰coa还原酶、硫酯酶、coa转移酶、醛脱氢酶、单加氧酶、醇脱氢酶、6-氧代己酸脱氢酶、7-氧代庚酸脱氢酶、ω-转氨酶、6-羟基己酸脱氢酶、5-羟基戊酸脱氢酶、4-羟基丁酸脱氢酶、羧酸还原酶、脱酰基酶、n-乙酰基转移酶、ω-转氨酶,或酰胺水解酶。在表达羧酸还原酶的重组宿主中,也可以表达磷酸泛酰巯基乙胺基转移酶,因为它增强羧酸还原酶的活性。在表达单加氧酶的重组宿主中,也可以表达电子转移链蛋白,如氧化还原酶或铁氧还蛋白多肽。
例如,重组宿主可以包括外源β-酮硫解酶并生产可以被转化为6-羟基己酸的3-氧代-6-羟基己酰基coa。
例如,重组宿主可以包括外源β-酮硫解酶和外源硫酯酶或coa转移酶,以及以下外源酶的一种或多种:3-羟基酰基coa脱氢酶或3-酮酰基coa还原酶、烯酰coa水合酶,和反式-2-烯酰coa还原酶,并生产6-羟基己酸。例如,重组宿主可以包括外源β-酮硫解酶、外源硫酯酶或coa转移酶、烯酰coa水合酶、外源反式-2-烯酰coa还原酶,和外源3-羟基酰基coa脱氢酶或外源3-酮酰基coa还原酶,并生产6-羟基己酸。
例如,生产6-羟基己酸的重组宿主可以包括以下外源酶的一种或多种:单加氧酶、醇脱氢酶、4-羟基丁酸脱氢酶、5-羟基戊酸脱氢酶、6-羟基己酸脱氢酶、7-氧代庚酸脱氢酶、5-氧代戊酸脱氢酶、6-氧代己酸脱氢酶,或醛脱氢酶,并进一步生产己二酸。例如生产6-羟基己酸的重组宿主可以包括外源单加氧酶并生产己二酸。例如,生产6-羟基己酸的重组宿主可以包括外源6-羟基己酸脱氢酶和醛脱氢酶并生产己二酸。例如,生产6-羟基己酸的重组宿主可以包括外源醇脱氢酶和以下外源酶之一:5-氧代戊酸脱氢酶、6-氧代己酸脱氢酶,或7-氧代庚酸脱氢酶,并生产己二酸。
例如,生产6-羟基己酸的重组宿主可以包括以下外源酶的一种或多种:醇脱氢酶、6-羟基己酸脱氢酶,或转氨酶,并进一步生产6-氨基己酸。例如,生产6-羟基己酸的重组宿主可以包括外源醇脱氢酶和外源转氨酶并生产6-氨基己酸。例如,生产6-羟基己酸的重组宿主可以包括外源6-羟基己酸脱氢酶和外源转氨酶并生产6-氨基己酸。任何此类宿主还可以包括外源酰胺水解酶并进一步生产己内酰胺。
例如,生产6-羟基己酸的重组宿主可以包括以下外源酶的一种或多种:羧酸还原酶、ω-转氨酶、脱酰基酶、n-乙酰基转移酶,或醇脱氢酶,并生产六亚甲基二胺。例如,生产6-羟基己酸的重组宿主可以包括外源羧酸还原酶、外源醇脱氢酶,以及一种或多种外源转氨酶(例如一种转氨酶或两种不同的转氨酶),并生产六亚甲基二胺。例如,生产6-羟基己酸的重组宿主可以包括外源羧酸还原酶和一种或多种外源转氨酶(例如一种转氨酶或两种不同的转氨酶),并生产六亚甲基二胺。例如,生产6-羟基己酸的重组宿主可以包括外源醇脱氢酶、外源羧酸还原酶,以及一种或多种外源转氨酶(例如一种转氨酶或两种不同的转氨酶),并生产六亚甲基二胺。例如,生产6-羟基己酸的重组宿主可以包括外源醇脱氢酶、外源n-乙酰基转移酶、羧酸还原酶、脱酰基酶,以及一种或多种外源转氨酶(例如一种转氨酶或两种不同的转氨酶),并生产六亚甲基二胺。
例如,生产6-羟基己酸的重组宿主可以包括一种或多种以下的外源酶:羧酸还原酶以及外源醇脱氢酶,并进一步生产1,6-己二醇。
在任何重组宿主中,重组宿主还可以包括一种或多种(例如,一种、两种、三种,或四种)用于将2-酮戊二酸转化为4-羟基丁酰基coa的以下的外源酶:谷氨酸合酶;2-酮戊二酸脱羧酶;支链脱羧酶;谷氨酸脱羧酶;coa连接酶;coa-转移酶;ω-转氨酶;和醇脱氢酶。例如,重组宿主可以包括外源谷氨酸合酶,谷氨酸脱羧酶;coa连接酶或coa-转移酶;ω-转氨酶;和醇脱氢酶。例如,重组宿主可以包括外源2-酮戊二酸脱羧酶或支链脱羧酶;coa连接酶;coa-转移酶;和醇脱氢酶。
在工程化途径中,酶可以来自单一来源,即来自一个物种或属,或可来自多个来源,即不同的物种或属。已经从各种生物体鉴定了编码本文所述酶的核酸,并且可容易地在公众可获得的数据库,如genbank或embl中获得。
如本文所用,提及特定的酶(例如β-酮硫解酶)意指具有特定酶活性的多肽(例如具有β-酮硫解酶活性的多肽)。
本文描述的可用于生产一种或多种c6构建块的任何酶可与相应的野生型酶的氨基酸序列具有至少70%序列同一性(同源性)(例如,至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)。应当理解,可以基于成熟酶(例如,除去任何信号序列)或基于未成熟酶(例如,包括任何信号序列)确定序列同一性。还应当理解,起始甲硫氨酸残基可以存在于或可以不存在于本文所述的任何酶序列上。
例如,本文所述的具有β-酮硫解酶活性的多肽可以与钩虫贪铜菌β-酮硫解酶(参见genbank登录号aac38322.1,seqidno:1)、大肠杆菌β-酮硫解酶(参见genbank登录号aac74479.1,seqidno:13)或氨基丁酸羧菌(参见genbank登录号no.cab60036.2,seqidno:14)的氨基酸序列具有至少70%序列同一性(同源性)(例如,至少75%、80%、85%、90%、95%、97%、98%、99%或100%)。参见图6。
例如,本文所述的羧酸还原酶可以与海分枝杆菌(参见genbank登录号acc40567.1,seqidno:2)、耻垢分枝杆菌(参见genbank登录号abk71854.1,seqidno:3)、segniliparusrugosus(参见genbank登录号efv11917.1,seqidno:4)、马赛分枝杆菌(参见genbank登录号eiv11143.1,seqidno:5),或segniliparusrotundus(参见genbank登录号adg98140.1,seqidno:6)羧酸还原酶的氨基酸序列具有至少70%序列同一性(同源性)(例如,至少75%、80%、85%、90%、95%、97%、98%、99%或100%)。参见图6。
例如,本文所述的ω-转氨酶可以与紫色色杆菌(参见genbank登录号aaq59697.1,seqidno:7)、铜绿假单胞菌(参见genbank登录号aag08191.1,seqidno:8)、丁香假单胞菌(参见genbank登录号aay39893.1,seqidno:9)、球形红杆菌(参见genbank登录号aba81135.1,seqidno:10)、大肠杆菌(参见genbank登录号aaa57874.1,seqidno:11),或河流弧菌(参见genbank登录号aea39183.1,seqidno:12)ω-转氨酶的氨基酸序列具有至少70%序列同一性(同源性)(例如,至少75%、80%、85%、90%、95%、97%、98%、99%或100%)。这些ω-转氨酶中的一些是二胺ω-转氨酶。参见图6。
例如,本文所述的烯酰coa还原酶可以与齿垢密螺旋体(参见genbank登录号aas11092.1,seqidno:15)或euglenagracilis(参见genbank登录号aaw66853.1,seqidno:16)的氨基酸序列具有至少70%序列同一性(同源性)(例如,至少75%、80%、85%、90%、95%、97%、98%、99%或100%)。参见图6。
例如,本文所述的脱羧酶可以与鼠伤寒沙门氏菌的氨基酸序列(参见genbankaccessionno.cac48239.1,seqidno:17)具有至少70%序列同一性(同源性)(例如,至少75%、80%、85%、90%、95%、97%、98%、99%或100%)。参见图6。
可以如下测定两种氨基酸序列间的百分比同一性(同源性)。首先,使用来自含有blastp第2.0.14版的单机版blastz的blast2sequences(bl2seq)程序比对氨基酸序列。此单机版blastz可以获自fish&richardson的网站(例如www.fr.com/blast/)或美国政府国立生物技术信息中心网站(www.ncbi.nlm.nih.gov)。解释如何使用bl2seq程序的用法说明可以参见伴随blastz的自述文件。bl2seq使用blastp算法实施两种氨基酸序列之间的比较。为了比较两种氨基酸序列,如下设置bl2seq的选项:-i设置为含有要比较的第一氨基酸序列的文件(例如c:\seq1.txt);-j设置为含有要比较的第二氨基酸序列的文件(例如c:\seq2.txt);-p设置为blastp;-o设置为任何期望的文件名称(例如c:\output.txt);并且所有其它选项保持为其缺省设置。例如,可以使用以下命令来产生含有两种氨基酸序列间的比较的输出文件:c:\bl2seq–ic:\seq1.txt–jc:\seq2.txt–pblastp–oc:\output.txt。如果两种比较序列共享同源性(同一性),那么指定的输出文件会呈现那些同源性区作为比对序列。如果两种比较序列不共享同源性(同一性),那么指定的输出文件不会呈现比对序列。可以对核酸序列遵循相似的规程,只是使用blastn。
一旦比对,通过计算相同氨基酸残基在这两种序列中呈现的位置的数目确定匹配数目。通过用匹配数目除以全长多肽氨基酸序列的长度,接着将所得的数值乘以100来确定百分比同一性(同源性)。注意到百分比同一性(同源性)值被四舍五入到最近的十分位。例如,78.11、78.12、78.13、和78.14被向下四舍五入到78.1,而78.15、78.16、78.17、78.18、和78.19被向上四舍五入到78.2。还注意到长度值会总是整数。
应当领会,许多核酸可以编码具有特定氨基酸序列的多肽。遗传密码的简并性是本领域中公知的;即对于许多氨基酸,存在有超过一种充当氨基酸密码子的核苷酸三联体。例如,可以修饰给定酶的编码序列中的密码子,从而获得特定物种(例如细菌或真菌)中的最佳表达,这使用适合于所述物种的密码子偏爱表进行。
也可以在本文件的方法中使用本文中描述的任何酶的功能性片段。如本文中使用的,术语“功能性片段”指与相应的成熟、全长、野生型蛋白质的活性具有至少25%(例如至少30%;40%;50%;60%;70%;75%;80%;85%;90%;95%;98%;99%;100%;或甚至大于100%)的蛋白质的肽片段。功能性片段一般但不总是可以由蛋白质的连续区构成,其中该区具有功能性活性。
此文件还提供了(i)本文件的方法中使用的酶的功能性变体和(ii)上文描述的功能性片段的功能性变体。相对于相应的野生型序列,酶和功能性片段的功能性变体可以含有添加、缺失、或取代。具有取代的酶一般会具有不超过50(例如不超过1、2、3、4、5、6、7、8、9、10、12、15、20、25、30、35、40、或50)处氨基酸取代(例如保守取代)。这适用于本文中描述的任何酶和功能性片段。保守取代是用一种氨基酸取代具有相似特征的另一种。保守取代包括下列组内的取代:缬氨酸、丙氨酸和甘氨酸;亮氨酸、缬氨酸、和异亮氨酸;天冬氨酸和谷氨酸;天冬酰胺和谷氨酰胺;丝氨酸、半胱氨酸、和苏氨酸;赖氨酸和精氨酸;和苯丙氨酸和酪氨酸。非极性疏水性氨基酸包括丙氨酸、亮氨酸、异亮氨酸、缬氨酸、脯氨酸、苯丙氨酸、色氨酸和甲硫氨酸。极性中性氨基酸包括甘氨酸、丝氨酸、苏氨酸、半胱氨酸、酪氨酸、天冬酰胺和谷氨酰胺。带正电荷的(碱性)氨基酸包括精氨酸、赖氨酸和组氨酸。带负电荷的(酸性)氨基酸包括天冬氨酸和谷氨酸。用上文提及的极性、碱性或酸性组的一种成员被相同组的另一种成员的任何取代可以视为保守取代。比较而言,非保守取代是用一种氨基酸取代具有不同特征的另一种。
缺失变体可以缺乏1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、或20个氨基酸的区段(具有两个或更多个氨基酸)或非连续的单一氨基酸。添加(添加变体)包括融合蛋白,其含有:(a)本文中描述的任何酶或其片段;和(b)内部或末端(c或n)无关或外源氨基酸序列。在此类融合蛋白的背景中,术语“外源氨基酸序列”指与(a)不同的氨基酸序列。外源序列可以是例如用于纯化重组蛋白的序列(例如flag、多组氨酸(例如六组氨酸)、凝集素(ha)、谷胱甘肽-s-转移酶(gst)、或麦芽糖结合蛋白(mbp))。外源序列也可以是可用作可检测标志物的蛋白质,例如萤光素酶、绿色荧光蛋白(gfp)、或氯霉素乙酰基转移酶(cat)。在一些实施方案中,融合蛋白含有来自另一种蛋白质的信号序列。在某些宿主细胞(例如酵母宿主细胞)中,可以经由使用外源信号序列提高靶蛋白的表达和/或分泌。在一些实施方案中,融合蛋白可以含有可用于例如引发免疫应答以生成抗体的载体(例如klh)或er或高尔基体保留信号。外源序列可以是不同长度的,并且在一些情况中可以是比与外源序列附接的全长靶蛋白质更长的序列。
工程化宿主可天然表达本文所述途径的酶的无一(none)或一些(例如,一种或多种、两种或多种、三种或多种、四种或多种、五种或多种,或六种或多种)。因此,工程化宿主内的途径可以包括所有外源酶,或者可以包括内源和外源酶两者。工程化宿主的内源基因也可以被破坏以防止不期望的代谢物的形成或防止通过作用于此类中间体的其他酶导致途径中的中间体的损失。工程化宿主可以称为重组宿主或重组宿主细胞。如本文所述,重组宿主可以包括编码如下的一种或多种的核酸:如本文所述的β-酮硫解酶、脱氢酶、合酶、脱羧酶、还原酶、水合酶、硫酯酶、单加氧酶、硫酯酶、酰胺水解酶,和转氨酶。
此外,可以使用本文所述的分离的酶,使用来自宿主微生物的裂解物(例如,细胞裂解物)作为酶的来源,或使用多种来自不同宿主微生物的裂解物作为酶的来源,体外进行c6构件块的生产。
本文中所述的途径的反应可以在一种或多种宿主菌株中进行,所述宿主菌株(a)天然表达一种或多种的相关酶,(b)经遗传工程化以表达一种或多种相关的酶,或(c)天然表达一种或多种相关的酶并经遗传工程化以表达一种或多种相关的酶。或者,可以从上述类型的宿主细胞中提取相关的酶,并以纯化或半纯化形式使用。此外,此类提取物包括可用作相关酶来源的裂解物(例如细胞裂解物)。在由本文件所提供的方法中,所有步骤可以在宿主细胞中进行,所有步骤可以使用提取的酶进行,或者一些步骤可以在细胞中进行,而其它步骤可以使用提取的酶进行。
产生6-羟基己酸的酶
如图1所示,可以从2-酮戊二酸通过中间体3-氧代-6-羟基己酰基coa(其可以使用β-酮硫解酶从4-羟基丁酰基coa生产)生物合成6-羟基己酸。可以使用以下的酶将3-氧代-6-羟基己酰基coa转化为6-羟基己酸:3-羟基酰基coa脱氢酶或3-酮酰基coa脱氢酶、烯酰coa水合酶、反式-2-烯酰coa还原酶,和硫酯酶或coa转移酶。
在一些实施方案中β-酮硫解酶可以被归类于ec2.3.1.16下,如bktb的基因产物,或者可以被归类于ec2.3.1.174下,如paaj的基因产物。来自钩虫贪铜菌由bktb编码的β-酮硫解酶接受乙酰coa和丙酰coa作为底物,形成coa活化的脂肪族骨架(参见例如haywoodetal.,femsmicrobiologyletters,1988,52:91-96;slateretal.,j.bacteriol.,1998,180(8):1979-1987)。来自大肠杆菌由paaj编码的β-酮硫解酶接受琥珀酰coa和乙酰coa作为底物,形成coa活化的骨架(nogalesetal.,microbiology,2007,153,357-365)。参见,例如图6中的seqidno:1和seqidno:13。
在一些实施方案中,3-羟基酰基coa脱氢酶或3-酮酰基coa脱氢酶可以被归类于ec1.1.1.-下。例如,3-羟基酰基coa脱氢酶可以被归类于ec1.1.1.35下,如fadb的基因产物;归类于ec1.1.1.157下,如hbd的基因产物(也可称为3-羟基丁酰基coa脱氢酶);或归类于ec1.1.1.36下,如phab的乙酰乙酰基coa还原酶基因产物(liu&chen,appl.microbiol.biotechnol,2007,76(5):1153-1159;shenetal,appl.environ.microbiol,2011,77(9):2905-2915;buddeetal,j.bacteriol,2010,192(20):5319-5328)。
在一些实施方案中,3-酮酰基coa还原酶可以被归类于ec1.1.1.100下,如fabg的基因产物(buddeetal,j.bacteriol,2010,192(20):5319-5328;nomuraetal,appl.environ.microbiol,2005,71(8):4297-4306)。
在一些实施方案中,烯酰coa水合酶可以被归类于ec4.2.1.17下,如crt的基因产物,或归类于ec4.2.1.119下,如phaj的基因产物(shenetal,2011,同上;fukuietal,j.bacteriol,1998,180(3):667-673)。
在一些实施方案中,反式-2-烯酰coa还原酶可以被归类于ec1.3.1.38或ec1.3.1.44下,如egter的基因产物(nishimakietal,j.biochem.,1984,95:1315-1321;shenetal,2011,同上),或者tdter的基因产物(bond-wattsetal,biochemistry,2012,51:6827-6837),或者被归类于ec1.3.1.8下(inuietal,eur.j.biochem.,1984,142,121-126)。
在一些实施方案中,通过归类于ec3.1.2.-下的硫酯酶,在6-羟基己酰基coa中酶促形成导致合成6-羟基己酸的末端羧基,导致6-羟基己酸的产生。硫酯酶可以是ycia或acot13的基因产物(cantuetal,proteinscience,2010,19,1281-1295;zhuangetal,biochemistry,2008,47(9):2789-2796;naggertetal.,jbiol.chem.,1991,266(17):11044-11050)。
在一些实施方案中,通过coa转移酶在6-羟基己酰基coa中酶促形成导致合成6-羟基己酸的末端羧基,所述coa转移酶例如归类于ec2.8.3-下,如来自克鲁佛梭菌cat2的基因产物,来自氨基丁酸羧菌abft的基因产物,或来自clostridiumviride的5-羟基戊酸coa转移酶。
在己二酸的生物合成中产生末端羧基的酶
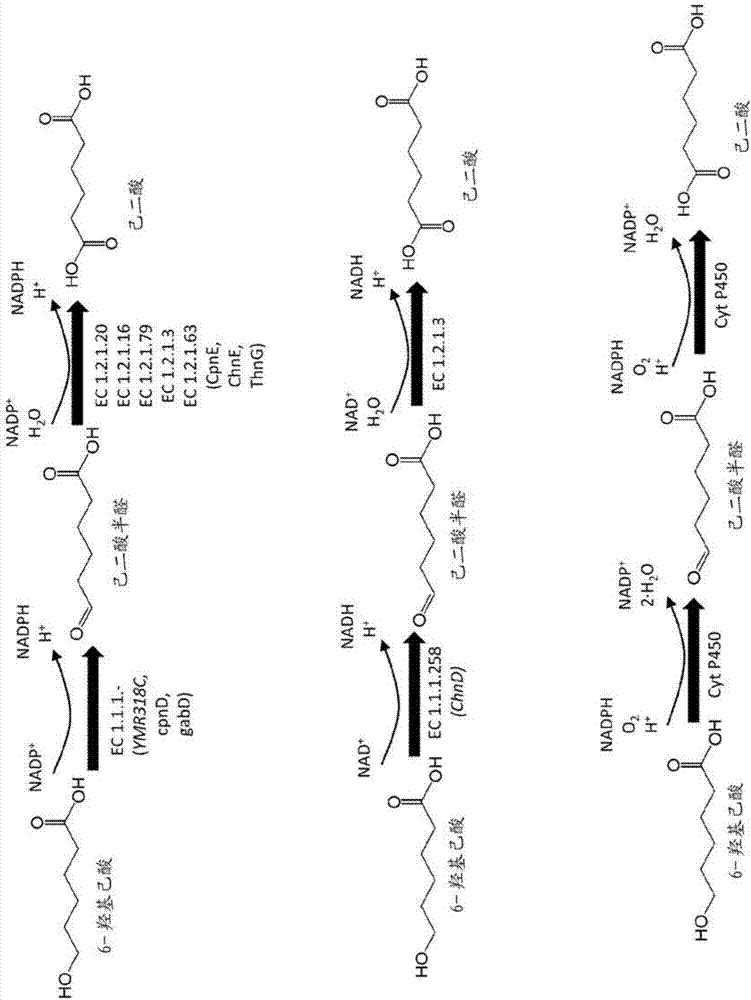
如描绘于图2中,可以使用醛脱氢酶、5-氧代戊酸脱氢酶、6-氧代己酸脱氢酶、7-氧代庚酸脱氢酶,或单加氧酶来酶促形成导致己二酸生产的末端羧基。
在一些实施方案中,可以通过归类于ec1.2.1.3下的醛脱氢酶,在己二酸半醛中酶促形成导致己二酸合成的第二末端羧基(guerrillot&vandecasteele,eur.j.biochem.,1977,81,185-192)。参见图2。
在一些实施方案中,通过以下的酶,在己二酸半醛中酶促形成导致己二酸合成的第二末端羧基:ec1.2.1.-如5-氧代戊酸脱氢酶(例如归类于ec1.2.1.20下,如cpne的基因产物),6-氧代己酸脱氢酶(例如归类于ec1.2.1.63下,如来自不动杆菌属(acinetobactersp.)chne的基因产物),或7-氧代庚酸脱氢酶(如来自鞘氨醇单胞菌(sphingomonasmacrogolitabida)thng的基因产物)(iwakietal,appl.environ.microbiol,1999,65(11),5158-5162;lopez-sanchezetal.,αppl.environ.microbiol,2010,76(1),110-118))。参见图2。
在一些实施方案中,通过细胞色素p450家族中的单加氧酶(如cyp4f3b),在己二酸半醛中酶促形成导致己二酸合成的第二末端羧基(参见例如,sandersetal,j.lipidresearch,2005,46(5):1001-1008;sandersetal,thefasebjournal,2008,22(6):2064-2071)。参见图2。
在六亚甲基二胺或6-氨基己酸的生物合成中产生末端胺基的酶
如描绘与图3和图4中,可以使用ω-转氨酶或脱酰基酶酶促形成末端胺基。
在一些实施方案中,通过ω-转氨酶,在己二酸半醛中酶促形成导致6-氨基己酸合成的末端胺基,所述ω-转氨酶例如归类于ec2.6.1.-下,如ec2.6.1.18,ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82下,诸如获得自紫色色杆菌(参见genbank登录号aaq59697.1,seqidno:7)、铜绿假单胞菌(参见genbank登录号aag08191.1,seqidno:8)、丁香假单胞菌(参见genbank登录号aay39893.1,seqidno:9)、球形红杆菌(参见genbank登录号aba81135.1,seqidno:10)、河流弧菌(参见genbank登录号aea39183.1,seqidno:12)、灰色链霉菌(streptomycesgriseus)或clostridiumviride。参见图3。
可被用于本文所述方法和宿主中的另外的ω-转氨酶来自大肠杆菌(参见genbank登录号aaa57874.1,seqidno:11)。一些例如归类于ec2.6.1.29或ec2.6.1.82下的ω-转氨酶是二胺ω-转氨酶(例如seqidno:11)。
来自紫色色杆菌的可逆的ω-转氨酶(genbank登录号aaq59697.1,seqidno:13)已经证明接受6-氨基己酸作为氨基供体的类似的活性,从而在己二酸半醛中形成第一末端氨基(kaulmannetal.,enzymeandmicrobialtechnology,2007,41,628-637)。
来自灰色链霉菌的可逆的4-氨基丁酸:2-酮戊二酸转氨酶已经证明对于6-氨基己酸到己二酸半醛的转化的类似的活性(yonahaetal.,eur.j.biochem.,1985,146,101-106)。
来自clostridiumviride的可逆的5-氨基戊酸转氨酶已经证明对于6-氨基己酸到己二酸半醛的转化的类似的活性(barkeretal,j.biol.chem.,1987,262(19),8994-9003)。
在一些实施方案中,通过二胺转氨酶,在6-氨基己醛中酶促形成导致六亚甲基二胺合成的第二末端胺基,所述二胺转氨酶例如归类于ec2.6.1.29下或例如归类于ec2.6.1.82下,如来自大肠杆菌的ygjg的基因产物(genbank登录号aaa57874.1,seqidno:12)。还可以使用seqidnos:7-10和11中所列的转氨酶来生产六亚甲基二胺。参见图4。
ygjg的基因产物接受宽范围的二胺碳链长度底物,如腐胺,尸胺和亚精胺(samsonovaetal,bmcmicrobiology,2003,3:2)。
来自大肠杆菌菌株b的二胺ω-转氨酶已经证明针对1,7二氨基庚烷的活性(kim,thejournalofchemistry,1964,239(3),783-786)。
在一些实施方案中,通过脱酰基酶,在n6-乙酰基-1,6-二氨基己烷中酶促形成导致六亚甲基二胺合成的第二末端胺基,所述脱酰基酶例如归类于ec3.5.1.17下,如酰基赖氨酸脱羧酶。
在1,6己二醇的生物合成中产生末端羟基的酶
如描绘于图5,可以使用醇脱氢酶来酶促形成末端羟基。例如,通过醇脱氢酶,在6-羟基己烷中酶促形成导致1,6己二醇合成的末端羟基,所述醇脱氢酶归类于ec1.1.1.-(例如ec1.1.1.1、1.1.1.2、1.1.1.21,或1.1.1.184)下,如ymr318c或yqhd的基因产物(liuetal,microbiology,2009,155,2078-2085;larroyetah,2002,biochemj.,361(pt1),163-172;jarboe,2011,appl.microbiol.biotechnol,89(2),249-257)或者具有genbank序列号caa81612.1的蛋白质。
生物化学途径
导致6-羟基己酸的途径
在一些实施方案中,如下从中心代谢物2-酮戊二酸合成6-羟基己酸:通过谷氨酸合酶(例如归类于ec1.4.1.13下)或者α-氨基转移酶(例如归类于ec2.6.1.-,如ec2.6.1.39下)将2-酮戊二酸转化为l-谷氨酸;然后通过谷氨酸脱羧酶(例如归类于ec4.1.1.15或ec4.1.1.18下)将l-谷氨酸转化为4-氨基丁酸;然后通过ω-转氨酶(例如归类于ec2.6.1.18,ec2.6.1.19,ec2.6.1.48,或ec2.6.1.96下),如来自大肠杆菌gabt的基因产物,将4-氨基丁酸转化为琥珀酸半醛(bartschetal,j.bacteriol,1990,172(12),7035);然后通过醇脱氢酶(例如归类于ec1.1.1.61下),如gbd(例如来自纤维素堆囊菌(sorangiumcellulosu))、gabd(bartschetal,j.bacteriol,1990,172(12),7035)或者yihu(saitoetal,j.biol.chem.,2009,284(24),16442-16452)的基因产物,或者通过5-羟基戊酸脱氢酶(如cpnd的基因产物(例如参见,iwakietal,2002,appl.environ.microbiol,68(11):5671-5684))将琥珀酸半醛转化为4-羟基丁酸;然后通过使用coa连接酶(例如归类于ec6.2.1-(例如ec6.2.1.40))或coa转移酶(例如归类于ec2.8.3-)如来自克鲁佛梭菌cat2的基因产物、来自氨基丁酸羧菌abft的基因产物或者来自clostridiumviride5-羟基戊酸coa转移酶,将4-羟基丁酸转化为4-羟基丁酰基coa;然后通过使用β-酮硫解酶(例如归类于ec2.3.1.16或ec2.3.1.174下)如bktb或paaj的基因产物(例如seqidno:1或13),或者由cab60036.2编码的β-酮硫解酶活性(例如seqidno:14)将4-羟基丁酰基coa转化为3-氧代-6-羟基己酰基coa;然后使用3-羟基酰基coa脱氢酶(例如归类于ec1.1.1.-下)如ec1.1.1.35(例如fadb的基因产物),ec1.1.1.36(例如phab的基因产物),或ec1.1.1.157(例如hbd的基因产物),或者通过3-酮酰基coa还原酶(例如归类于ec1.1.1.100下)如fabg的基因产物,转化为3-羟基-6-羟基己酰基coa;然后使用烯酰coa水合酶(例如归类于ec4.2.1.17下如crt的基因产物,或者归类于ec4.2.1.119下,如phaj的基因产物)将3-羟基-6-羟基己酰基coa转化为2,3-脱氢-6-羟基己酰基coa;然后通过反式-2-烯酰coa还原酶(例如归类于ec1.3.1.38、ec1.3.1.44,或ec1.3.1.8下)如egter或tdter的基因产物,将2,3-脱氢-6-羟基己酰基coa转化为6-羟基己酰基coa;然后通过硫酯酶(例如归类于ec3.1.2.-下)如ycia或acot13的基因产物,或者coa转移酶(例如归类于ec2.8.3.-下)将6-羟基己酰基coa转化为6-羟基己酸。参见图1。
在一些实施方案中,如下将2-酮戊二酸转化为琥珀酸半醛:使用羧基裂解酶(例如归类于ec.4.1.1.-下)像是2-酮戊二酸脱羧酶(例如归类于ec4.1.1.71下)或支链脱羧酶(例如归类于ec4.1.1.72下)如kdca或kivd的基因产物,或者吲哚丙酮酸脱羧酶(例如归类于ec4.1.1.74下)或者苯基丙酮酸脱羧酶(例如归类于ec4.1.1.43下)。以这种方式生产的琥珀酸半醛可以如上所述转化为6-羟基己酸。参见图1。
使用6-羟基己酸作为中心前体至己二酸的途径
在一些实施方案中,如下从6-羟基己酸合成己二酸:通过醇脱氢酶(例如归类于ec1.1.1.-下)如ymr318c的基因产物(例如归类于ec1.1.1.2下,参见genbank登录号caa90836.1)(larroyetal,2002,biochemj.,361(pt1),163-172),cpnd的基因产物(iwakietal,2002,appl.environ.microbiol,68(11):5671-5684)或gabd的基因产物(lutke-eversloh&steinbüchel,1999,femsmicrobiologyletters,181(1):63-71),或者通过6-羟基己酸脱氢酶(例如归类于ec1.1.1.258下)如chnd的基因产物(iwakietal,αρρι.environ.microbiol.,1999,65(11):5158-5162)将6-羟基己酸转化为己二酸半醛;然后通过脱氢酶(例如归类于ec1.2.1.-下)如7-氧代庚酸脱氢酶(例如,thng的基因产物)、6-氧代己酸脱氢酶(例如,chne的基因产物),通过谷氨酸半醛脱氢酶(例如归类于ec1.2.1.20下),通过5-氧代戊酸脱氢酶(如cpne的基因产物),或醛脱氢酶(归类于ec1.2.1.3下),将己二酸半醛转化为己二酸。参见图2。由ymr318c编码的醇脱氢酶具有广泛的底物特异性,包括c6醇的氧化。
在一些实施方案中,如下从中心前体6-羟基己酸合成己二酸:通过细胞色素p450将6-羟基己酸转化为己二酸半醛(sandersetal,j.lipidresearch,2005,46(5),1001-1008;sandersetal,thefasebjournal,2008,22(6),2064-2071);然后通过细胞色素p450家族中的单加氧酶(如cyp4f3b)将己二酸半醛转化为己二酸。参见图2。
使用6-羟基己酸作为中心前体至6-氨基己酸和ε-己内酰胺的途径
在一些实施方案中,如下从中心前体6-羟基己酸合成6-氨基己酸:通过醇脱氢酶(例如归类于ec1.1.1.2下)如ymr318c的基因产物,6-羟基己酸脱氢酶(例如归类于ec1.1.1.258下),5-羟基戊酸脱氢酶(例如归类于ec1.1.1.-下)如cpnd的基因产物,或4-羟基丁酸脱氢酶(例如归类于ec1.1.1.-)如gabd的基因产物,将6-羟基己酸转化为己二酸半醛;然后通过ω-转氨酶(ec2.6.1.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82如seqidnos:7-10或12中的一个,见上)将己二酸半醛转化为6-氨基己酸。参见图3。
在一些实施方案中,如下从中心前体6-羟基己酸合成ε-己内酰胺:通过醇脱氢酶(例如归类于ec1.1.1.2下)如ymr318c的基因产物,6-羟基己酸脱氢酶(例如归类于ec1.1.1.258下),5-羟基戊酸脱氢酶(例如归类于ec1.1.1.-)如cpnd的基因产物,或者4-羟基丁酸脱氢酶(例如归类于ec1.1.1.-)如gabd的基因产物,将6-羟基己酸转化为己二酸半醛;然后通过ω-转氨酶(ec2.6.1.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82)将己二酸半醛转化为6-氨基己酸;然后通过酰胺水解酶(ec3.5.2.-)将6-氨基己酸转化为ε-己内酰胺。参见图3。
在一些实施方案中,通过上述最后一步(即通过使用酰胺水解酶如ec.3.5.2.-中的一个酶转化)从中心前体6-氨基己酸合成ε-己内酰胺。参见图3。
使用6-氨基己酸、6-羟基己酸、己二酸半醛,或1,6-己二醇作为中心前体至六亚甲基二胺的途径
在一些实施方案中,如下从中心前体6-氨基己酸合成六亚甲基二胺:通过羧酸还原酶(例如归类于ec1.2.99.6下)如car的基因产物与磷酸泛酰巯基乙胺转移酶增强剂(enhancer)(例如由来自枯草芽孢杆菌的sfp基因编码或来自诺卡氏菌的npt基因编码)或来自灰色链霉菌的gric和grid的基因产物(suzukietal.,j.antibiot.,2007,60(6),380-387)组合,将6-氨基己酸转化为6-氨基己醛;然后通过ω-转氨酶(例如归类于ec2.6.1.-下(如ec2.6.1.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82,如seqidnos:7-12))将6-氨基己醛转化为六亚甲基二胺。例如,所述羧酸还原酶可以获得自海分枝杆菌(genbankaccessionno.acc40567.1,seqidno:2)、耻垢分枝杆菌(genbank登录号abk71854.1,seqidno:3)、segniliparusrugosus(genbank登录号efv11917.1,seqidno:4)、马赛分枝杆菌(genbank登录号eiv11143.1,seqidno:5),或者segniliparusrugosus(genbank登录号adg98140.1,seqidno:6)。参见图4。
由car的基因产物编码的羧酸还原酶和磷酸泛酰巯基乙胺转移酶增强剂npt或sfp具有广泛的底物特异性,其包括末端双官能的c4和c5羧酸(venkitasubramanianetal.,enzymeandmicrobialtechnology,2008,42,130-137)。
在一些实施方案中,如下从中心前体6-羟基己酸(其可以如图1中描述的产生)合成六亚甲基二胺:通过羧酸还原酶(例如归类于ec1.2.99.6下)如car的基因产物(见上)与磷酸泛酰巯基乙胺转移酶增强剂(例如由来自枯草芽孢杆菌的sfp基因编码或来自诺卡氏菌的npt基因编码)或gric和grid的基因产物(suzukietal,2001,见上)组合,将6-羟基己酸转化为6-羟基己醛;然后通过ω-转氨酶(例如归类于ec2.6.1.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82下)如seqidnos:7-12(见上),将6-氨基己醛转化为6-氨基己醇;然后通过醇脱氢酶或者具有genbank登录号caa81612.1的蛋白质转化为7-氨基己醛,所述醇脱氢酶例如归类于ec1.1.1.-(如ec1.1.1.1,ec1.1.1.2,ec1.1.1.21,或ec1.1.1.184)下,如ymr318c或yqhd的基因产物(liuetal,microbiology,2009,155,2078-2085;larroyetal,2002,biochemj.,361(pt1),163-172;jarboe,2011,appl.microbiol.biotechnol,89(2),249-257);然后通过ω-转氨酶转化为六亚甲基二胺,所述ω-转氨酶例如归类于ec2.6.1.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82下,如seqidnos:7-12,见上。参见图4。
在一些实施方案中,如下从中心前体6-氨基己酸合成六亚甲基二胺:通过n-乙酰基转移酶(如例如归类于ec2.3.1.32下的赖氨酸n-乙酰基转移酶)将6-氨基己酸转化为n6-乙酰基-6-氨基己酸;然后通过羧酸还原酶(例如归类于ec1.2.99.6下)如car的基因产物(见上,例如seqidno:4、5,或6)与磷酸泛酰巯基乙胺转移酶增强剂(例如由来自枯草芽孢杆菌的sfp基因编码或来自诺卡氏菌的npt基因编码)或gric和grid的基因产物组合,转化为n6-乙酰基-6-氨基己醛;然后通过ω-转氨酶转化为为n6-乙酰基-1,6-氨基己烷,所述ω-转氨酶例如归类于ec2.6.1.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82下,如seqidnos:7-12,见上;然后通过酰基赖氨酸脱酰基,酶(例如归类于ec3.5.1.17下)转化为六亚甲基二胺。参见图4。
在一些实施方案中,如下从中心前体己二酸半醛合成六亚甲基二胺:通过羧酸还原酶(例如归类于ec1.2.99.6下)如car的基因产物(见上,例如seqidno:6)与磷酸泛酰巯基乙胺转移酶增强剂(例如由来自枯草芽孢杆菌的sfp基因编码或来自诺卡氏菌的npt基因编码)或gric和grid的基因产物组合,将己二酸半醛转化为己二醛;然后通过ω-转氨酶转化为6-氨基己醛,所述ω-转氨酶例如归类于ec2.6.1.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82下;然后通过ω-转氨酶转化为六亚甲基二胺,所述ω-转氨酶例如归类于ec2.6.1.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82下,如seqidnos:7-12。参见图4。
在一些实施方案中,如下从1,6-己二醇合成六亚甲基二胺:使用醇脱氢酶将1,6-己二酮转化为6-羟基己烷,所述醇脱氢酶例如归类于ec1.1.1.-(例如ec1.1.1.1、ec1.1.1.2、ec1.1.1.21,或ec1.1.1.184)下,如ymr318c或yqhd的基因产物或者具有genbank登录号caa81612.1的蛋白质;然后通过ω-转氨酶转化为6-氨基己醇,所述ω-转氨酶例如归类于ec2.6.1.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82下,如seqidnos:7-12;然后通过醇脱氢酶转化为6-氨基己醛,所述醇脱氢酶例如归类于ec1.1.1.-(例如ec1.1.1.1、ec1.1.1.2、ec1.1.1.21,或ec1.1.1.184)下,如ymr318c或yqhd的基因产物或者具有genbank登录号caa81612.1的蛋白质;然后通过ω-转氨酶转化为六亚甲基二胺,所述ω-转氨酶例如归类于ec2.6.1.18、ec2.6.1.19、ec2.6.1.29、ec2.6.1.48,或ec2.6.1.82下,如seqidnos:7-12。参见图4。
使用6-羟基己酸作为中心前体至1,6-己二醇的途径
在一些实施方案中,如下从中心前体6-羟基己酸合成1,6己二醇:通过羧酸还原酶(例如归类于ec1.2.99.6下)如car的基因产物(见上,例如seqidno:2,3,4,5,或6)与磷酸泛酰巯基乙胺转移酶增强剂(例如由来自枯草芽孢杆菌的sfp基因编码或来自诺卡氏菌的npt基因编码)或来自灰色链霉菌的gric和grid的基因产物组合(suzukietah,j.antibiot,2007,60(6),380-387),将6-羟基己酸转化为6-羟基己醛;然后通过醇脱氢酶将6-羟基己醛转化为1,6-己二醇,所述醇脱氢酶例如归类于ec1.1.1.-(例如ec1.1.1.1、ec1.1.1.2、ec1.1.1.21,或ec1.1.1.184)下,如ymr318c或yqhd的基因产物(来自大肠杆菌,genbank登录号aaa69178.1)(参见例如liuetah,microbiology,2009,155,2078-2085;larroyetal,2002,biochemj.,361(pt1),163-172;或者jarboe,2011,appl.microbiol.biotechnol.,89(2),249-257)或者具有genbank登录号caa81612.1(来自嗜热脂肪地芽孢杆菌(geobacillusstearothermophilus))的蛋白质。参见图5。
培养策略
在一些实施方案中,在重组宿主中使用厌氧、需氧或微需氧培养条件生物合成一种或多种c6构建块。在一些实施方案中,培养策略需要营养限制,如氮、磷酸盐或氧限制。
在一些实施方案中,可以采用使用例如陶瓷膜的细胞保留策略来实现并维持补料分批或连续发酵期间的高细胞密度。
在一些实施方案中,在一种或多种c6构件块的合成中对发酵补料的主要碳源可以源自生物或非生物原料。
在一些实施方案中,生物原料可以是或可以源自单糖,二糖,木质纤维素,半纤维素,纤维素,木质素,乙酰丙酸和甲酸,甘油三酯,甘油,脂肪酸,农业废物,浓缩的酒糟可溶物(condenseddistillers'solubles),或城市废物。
已经在几种微生物(诸如大肠杆菌、钩虫贪铜菌、食油假单胞菌、恶臭假单胞菌和解脂耶罗维亚酵母)中证明了源自生物柴油产生的粗制甘油的有效分解代谢(leeetal,appl.biochem.biotechnol.,2012,166:1801-1813;yangetal,biotechnologyforbiofuels,2012,5:13;meijnenetal,appl.microbiol.biotechnol,2011,90:885-893)。
已经在几种生物体(诸如钩虫贪铜菌和恶臭假单胞菌)中在经由前体丙酰基-coa合成3-羟基戊酸中证明了木质纤维素衍生的乙酰丙酸的有效分解代谢(jaremkoandyu,2011,同上;martinandprather,j.biotechnol,2009,139:61-67)。
已经在几种微生物(诸如恶臭假单胞菌、钩虫贪铜菌)中证明了木质素衍生的芳香族化合物诸如苯甲酸类似物的有效分解代谢(buggetal.,currentopinioninbiotechnology,2011,22,394-400;pérez-pantojaetal,femsmicrobiol.rev.,2008,32,736-794)。
已经在几种微生物(包括解脂耶罗维亚酵母)中证明了农业废物(诸如橄榄磨坊废水)的有效利用(papanikolaouetal,bioresour.technol,2008,99(7):2419-2428)。
已经对几种微生物(诸如大肠杆菌、谷氨酸棒状杆菌和德氏乳杆菌和乳酸乳球菌)证明了可发酵糖类(诸如源自纤维素、半纤维素、甘蔗和甜菜糖蜜、木薯、玉米和其它农业来源的单糖和二糖)的有效利用(参见例如hermannetal,j.biotechnol,2003,104:155-172;weeetal,foodtechnol.biotechnol,2006,44(2):163-172;ohashietal,j.bioscienceandbioengineering,1999,87(5):647-654)。
已经针对钩虫贪铜菌证明了源自多种农业木质纤维素来源的糠醛的有效利用(lietal,biodegradation,2011,22:1215-1225)。
在一些实施方案中,非生物原料可以是或者可以源自天然气,合成气,co2/h2,甲醇,乙醇,苯甲酸酯,非挥发性残留物(nvr),或来自环己烷氧化过程的碱洗液(causticwash)废物流,或对苯二甲酸/间苯二甲酸混合物废物流。
已经对甲基营养型酵母巴斯德毕赤酵母证明了甲醇的有效分解代谢。
已经对克鲁佛梭菌证明了乙醇的有效分解代谢(seedorfetal,proc.natlacad.sci.usa,2008,105(6)2128-2133)。
已经对钩虫贪铜菌证明了co2和h2(其可以源自天然气和其它化学和石油化学来源)的有效分解代谢(prybylskietal,energy,sustainabilityandsociety,2012,2:11)。
已经对多种微生物(诸如杨氏梭菌和自产乙醇梭菌)证明了合成气的有效分解代谢(
已经对多种微生物(诸如食酸代尔夫特菌和钩虫贪铜菌)证明了来自环己烷过程的非挥发性残留物废物流的有效分解代谢(ramsayetal,appliedandenvironmentalmicrobiology,1986,52(1):152-156)。
在一些实施方案中,宿主微生物可以是原核生物。例如,原核生物可以是来自以下的细菌:埃希氏菌属如大肠杆菌;梭菌属如杨氏梭菌、自产乙醇梭菌或者克鲁佛梭菌;棒状杆菌属如谷氨酸棒状杆菌;贪铜菌属如钩虫贪铜菌或者耐金属贪铜菌;假单胞菌属如荧光假单胞菌、恶臭假单胞菌或者食油假单胞菌;代尔夫特菌属如食酸代尔夫特菌;芽孢杆菌属如枯草芽胞杆菌;乳杆菌属如德氏乳杆菌;或者乳球菌属如乳酸乳球菌。此类原核生物也可以是构建能够生成一种或多种c6构件块的本文中描述的重组宿主细胞的基因来源。
在一些实施方案中,宿主微生物可以是真核生物。例如,真核生物可以是丝状真菌,例如来自曲霉属如黑曲霉的。另外,真核生物可以是酵母,例如来自酵母属如酿酒酵母;来自毕赤氏酵属如巴斯德毕赤酵母;来自耶罗维亚酵母属如解脂耶罗维亚酵母;来自伊萨酵母属如东方伊萨酵母;来自德巴利酵母属如汉逊德巴利酵母;来自arxula属如arxulaadenoinivorans;或者来自克鲁维酵母菌属如乳酸克鲁维酵母菌的酵母。此类真核生物也可以是构建能够生成一种或多种c6构件块的本文中描述的重组宿主细胞的基因来源。
代谢工程
本文件提供方法,所述方法牵涉对所有上述途径描述的少于所有的步骤。此类方法可牵涉例如此类步骤中的1,2,3,4,5,6,7,8,9,10,11,12或者更多个。在此类方法包含少于所有步骤的情况下,第一个步骤(且仅在一些实施方案中)可为所列步骤中的任一个。
此外,本文所述的重组宿主可以包括以上酶的任何组合,使得可以在重组宿主中进行一个或多个步骤,例如此类步骤中的1,2,3,4,5,6,7,8,9,10或者更多个。本文件提供了列出的任何属和种的宿主细胞,并遗传工程化以表达一种或多种(例如,2,3,4,5,6,7,8,9,10,11,12或者更多种)本文件所述的任何酶的重组形式。因此,例如,宿主细胞可以含有编码催化本文所述的任意途径的一个或多个步骤的酶的核酸。
另外,本文件认识到,在酶已经被描述为接受coa活化的底物的情况下,存在与[acp]结合底物相关的相似的酶活性,其不一定为相同的酶种类。
此外,本文件认识到,在酶已经被描述为接受底物的(r)-对映异构体的情况下,存在与底物的(s)-对映异构体相关的相似的酶活性,其不一定为相同的酶种类。
本文件还认识到,在已经显示酶接受特定辅因子如nadph或者共底物如乙酰coa的情况下,许多酶在催化特定酶活性中在接受大量不同辅因子或者共底物方面是泛宿主性的(promiscuous)。此外,本文件认识到,在酶对例如特定辅因子如nadh具有高特异性的情况下,对辅助因子nadph具有高特异性的具有相似或者相同活性的酶可为不同的酶种类。
在一些实施方案中,概述于本文的途径中的酶是经由非直接或者合理酶设计方法的酶工程的结果,目的在于改善活性、改善特异性、降低反馈抑制、降低阻抑、改善酶溶解度、改变立体特异性,或者改变辅因子特异性。
在一些实施方案中,将概述于本文的途径中的酶经由附加型或者染色体整合方法基因投入(genedose)(即,通过在宿主生物中具有多个拷贝的基因的过表达)至所得的经遗传修饰的生物体中。
在一些实施方案中,可以使用基因组级别(genome-scale)系统生物学技术如通量平衡分析(fluxbalanceanalysis)来设计用于将碳流量引导至c6构件块的基因组级别的弱化或者敲除策略。
弱化策略包括但不限于使用转座子,同源重组(双交叉方法),诱变,酶抑制剂和rnai干扰。
在一些实施方案中,可以利用通量组(fluxomic)、代谢物组(metabolomic)和转录物组(transcriptomal)数据来告知或者支持基因组级别的系统生物学技术,由此在将碳流量导向c6构件块中设计基因组级别的弱化或者敲除策略。
在一些实施方案中,可以通过在选择性环境中连续培养,改善宿主微生物对高浓度c6构件块的耐受性。
在一些实施方案中,弱化或增强宿主微生物的生物化学网络以(1)确保乙酰coa和4-羟基丁酰基coa的细胞内利用度,(2)创建nadh或nadph不平衡,其仅可通过形成c6构件块来平衡,(3)防止导致并包括c6构件块的中心代谢物、中心前体的降解和(4)确保从细胞有效的流出。
在需要用于c6构建块合成的乙酰coa细胞内利用度的一些实施方案中,可以在宿主生物体中弱化催化乙酰coa水解的内源性酶,如短链长度的硫酯酶。
在需要用于c6构建块合成的乙酰coa细胞内利用度的一些实施方案中,可以弱化产生乙酸的内源磷酸转乙酰化酶,如pta(shenetal,αppl.environ.microbiol,2011,77(9):2905-2915)。
在需要用于c6构建块合成的乙酰coa细胞内利用度的一些实施方案中,可以弱化乙酸合成途径中编码乙酸激酶的内源基因,如ack。
在需要用于c6构建块合成的乙酰coa和nadh细胞内利用度的一些实施方案中,可以弱化编码催化丙酮酸降解为乳酸的酶(如由ldha编码的乳酸脱氢酶)的内源基因(shenetal.,2011,同上)。
在一些实施方案中,可以在宿主中过表达催化添补反应(anapleroticreactions)的酶,如pep羧化酶和/或丙酮酸羧化酶。
在需要用于c6构建块合成的乙酰coa和nadh细胞内利用度的一些实施方案中,可以弱化例如编码催化磷酸烯醇式丙酮酸降解至琥珀酸的酶(如menaquinol-富马酸氧化还原酶)的内源基因,如frdbc(参见例如shenetal.,2011,同上)。
在需要用于c6构建块合成的乙酰coa和nadh细胞内利用度的一些实施方案中,可以弱化编码催化乙酰coa降解至乙醇的酶(如由adhe编码的醇脱氢酶)的内源基因(shenetal.,2011,同上)。
在一些实施方案中,其中途径需要过量的nadh辅因子用于c6构件块合成,可以在宿主生物体中过表达重组甲酸脱氢酶基因(shenetal.,2011,同上)。
在一些实施方案中,其中途径需要过量的nadh辅因子用于c6构件块合成,可以弱化消耗nadh的重组转氢酶。
在一些实施方案中,可以弱化编码催化丙酮酸降解至乙醇的酶(如丙酮酸脱羧酶)的内源基因。
在需要用于c6构建块合成的乙酰coa细胞内利用度的一些实施方案中,可以在微生物中过表达重组乙酰coa合成酶如acs的基因产物(satohetal,j.bioscienceandbioengineering,2003,95(4):335-341)。
在一些实施方案中,可以通过弱化内源葡萄糖-6-磷酸异构酶(ec5.3.1.9)将碳流引导入戊糖磷酸循环来增加nadph的供应。
在一些实施方案中,可以通过过表达6-磷酸葡糖酸脱氢酶和/或转酮醇酶将碳流重新引导入戊糖磷酸循环来增加nadph的供应(leeetal.,2003,biotechnologyprogress,19(5),1444-1449)。
在一些实施方案中,其中途径在c6构件块的合成中需要过量的nadph辅因子,可以在宿主生物体中过表达编码吡啶(puridine)核苷酸转氢酶的基因,如udha(brighametal.,advancedbiofuelsandbioproducts,2012,chapter39,1065-1090)。
在一些实施方案中,其中途径在c6构件块的合成中需要过量的nadph辅因子,可以在宿主生物体中过表达重组甘油醛-3-磷酸脱氢酶基因如gapn(brighametal,2012,同上)。
在一些实施方案中,其中途径在c6构件块的合成中需要过量的nadph辅因子,可以在宿主生物体中过表达重组苹果酸酶基因如maea或maeb(brighametal,2012,同上)。
在一些实施方案中,其中途径在c6构件块的合成中需要过量的nadph辅因子,可以在宿主生物体中过表达重组葡萄糖-6-磷酸脱氢酶基因如zwf(limetal.,j.bioscienceandbioengineering,2002,93(6),543-549)。
在一些实施方案中,其中途径在c6构件块的合成中需要过量的nadph辅因子,可以在宿主生物体中过表达重组果糖1,6二磷酸酶基因如fbp(beckeretal.,j.biotechnol,2007,132:99-109)。
在一些实施方案中,其中途径在c6构件块的合成中需要过量的nadph辅因子,可以弱化内源性丙糖磷酸异构酶(ec5.3.1.1)。
在一些实施方案中,其中途径在c7构件块的合成中需要过量的nadph辅因子,可以在宿主生物体中过表达重组葡萄糖脱氢酶如gdh的基因产物(satohetal,j.bioscienceandbioengineering,2003,95(4):335-341)。
在一些实施方案中,可以弱化促进将nadph转化为nadh的内源性酶,如可经由ec1.4.1.2(nadh-特异的)和ec1.4.1.4(nadph-特异的)下分类的谷氨酸脱氢酶的相互转换产生的nadh产生循环。
在一些实施方案中,可以弱化利用nadh和nadph两者作为辅因子的内源谷氨酸脱氢酶(ec1.4.1.3)。
在一些实施方案中,可以通过仅表达胞质域而不是锚定p450到内质网的n端区域溶解膜结合的细胞色素p450如cyp4f3b(schelleretal,j.biol.chem.,1994,269(17):12779-12783)。
在一些实施方案中,可以通过表达作为与小的可溶性蛋白(例如麦芽糖结合蛋白)的融合蛋白来溶解烯酰coa还原酶(gloerichetal,febsletters,2006,580,2092-2096)。
在使用天然累积聚羟基链烷酸的宿主的一些实施方案中,可以在宿主菌株中弱化内源聚合物合酶。
在一些实施方案中,可以在宿主中过表达l-丙氨酸脱氢酶以从丙酮酸再生l-丙氨酸作为氨基供体用于ω-转氨酶反应。
在一些实施方案中,可以在宿主中过表达l-谷氨酸脱氢酶、l-谷氨酰胺合成酶,或谷氨酸合成酶以从2-酮戊二酸再生l-谷氨酸作为氨基供体用于ω-转氨酶反应。
在一些实施方案中,可以弱化酶如分类于ec1.3.1.62下的庚二酰coa脱氢酶;例如归类于ec1.3.8.7、ec1.3.8.1,或ec1.3.99.-下的酰基coa脱氢酶;和/或归类于例如ec1.3.8.6下的丁酰coa脱氢酶,其降解导致并包括c6构件块的中心代谢物和中心前体。
在一些实施方案中,可以弱化经由辅酶a酯化活化c6构件块的内源性酶,诸如例如归类于ec6.2.1.14下的coa连接酶(例如己二酰coa合成酶)。
在一些实施方案中,可以通过对细胞膜的基因工程结构修饰或增加任何与c6构件块相关的转运蛋白活性来增强或放大跨越细胞膜至细胞外培养基的c6构件块流出。
可以通过过表达宽底物范围多药物转运蛋白,诸如来自枯草芽孢杆菌的blt(woolridgeetal,1997,j.biol.chem.,272(14):8864-8866);来自大肠杆菌的acrb和acrd(elkins&nikaido,2002,j.bacteriol.,184(23),6490–6499),来自金黄色葡萄球菌(staphylococcusaereus)的nora(ngetal,1994,antimicrobagentschemother,38(6),1345-1355),或来自枯草芽孢杆菌的bmr(neyfakh,1992,antimicrobagentschemother,36(2),484-485)增强或放大六亚甲基二胺的流出。
可以通过过表达溶质转运蛋白诸如来自谷氨酸棒杆菌(corynebacteriumglutamicum)的lyse转运蛋白增强或放大6-氨基己酸和七亚甲基二胺的流出(bellmannetal,2001,microbiology,147,1765-1774)。
通过过表达二羧酸转运蛋白诸如来自谷氨酸棒杆菌的suce转运蛋白增强或放大己二酸的流出(huhnetal.,appl.microbiol&biotech.,89(2),327-335)。
使用重组宿主生产c6构件块
通常,可以通过提供宿主微生物,并且用含有如上文描述的合适的碳源的培养基培养提供的微生物生成一种或多种c7构件块。一般地,培养基和/或培养可以使得微生物生长到足够的密度并且有效产生c7构件块。对于大规模生产过程,可以使用任何方法,诸如别处描述的(manualofindustrialmicrobiologyandbiotechnology,2ndedition,editors:a.l.demainandj.e.davies,asmpress;和principlesoffermentationtechnology,p.f.stanburyanda.whitaker,pergamon)。简言之,用特定微生物接种含有合适的培养基的大罐(例如,100加仑、200加仑、500加仑、或更多的罐)。在接种后,培养微生物以容许产生生物量。一旦达到期望的生物量,可以将含有微生物的培养基转移到第二个罐。此第二个罐可以是任何大小。例如,第二个罐可以是较大的,较小的,或者与第一个罐相同大小的。通常,第二个罐大于第一个,使得可以对来自第一个罐的培养基添加额外的培养基。另外,此第二个罐内的培养基可以与第一个罐中使用的培养基相同或不同。
一旦转移,可以温育微生物以容许生成c6构件块。一旦生成,可以使用任何方法来分离c6构件块。例如,可以经由吸附方法从发酵液选择性回收c6构件块。在己二酸和6-氨基己酸的情况下,可以经由蒸发进一步浓缩所得的洗脱液,经由蒸发和/或冷却结晶来结晶,并且经由离心回收晶体。在六亚甲基二胺和1,6-己二醇的情况下,可以采用蒸馏实现期望的产物纯度。
将在以下实施例中进一步描述本发明,其并不限制权利要求中描述的发明的范围。
实施例
实施例1
使用己二酸半醛作为底物并形成6-氨基己酸的ω-转氨酶的酶活性
将编码n末端his-标签的核苷酸序列添加到分别编码seqidno:7、8、9、10和12的ω-转氨酶的来自紫色色杆菌、铜绿假单胞菌、丁香假单胞菌、球形红杆菌和河流弧菌的核酸序列(参见图6),使得可以产生n末端加his标签的ω-转氨酶。将得到的每个修饰的基因在t7启动子控制下克隆到pet21a表达载体中,并将每个表达载体转化到bl21[de3]大肠杆菌宿主中。所得的重组大肠杆菌菌株在含有50mllb培养基和抗生素选择压力的250ml摇瓶培养物中于37℃在以230rpm振荡下培养。使用1mmiptg在16℃诱导每种培养物过夜。
通过离心收获来自每个诱导的摇瓶培养物的团粒。重悬每个团粒并通过超声处理裂解。通过离心从上清液中分离细胞碎片,并将无细胞提取物立即用于酶活性测定。
在由最终浓度为50mmhepes缓冲液(ph=7.5),10mm6-氨基己酸,10mm丙酮酸和100μμ吡哆5’磷酸盐(pyridoxyl5’phosphate)组成的缓冲液中进行反向(reversedirection)(即6-氨基己酸至己二酸半醛)酶活性测定。通过将ω-转氨酶基因产物或空载体对照的无细胞提取物加入到含有6-氨基己酸的测定缓冲液中来开始每个酶活性测定反应,并在25℃在250rpm振荡的情况下温育24h。通过rp-hplc对由丙酮酸形成的l-丙氨酸定量。
不含6-氨基己酸的每个仅酶的对照表明丙酮酸向l-丙氨酸的低基线转化。参见图11。如针对空载体对照所证实的,seqidno7、seqidno9、seqidno10和seqidno12的基因产物接受6-氨基己酸作为底物。参见图12。
对于seqidno7、seqidno8、seqidno9、seqidno10和seqidno12的转氨酶,证实了正向(forwarddirection)(即己二酸半醛至6-氨基己酸)酶活性。酶活性测定在由最终浓度为50mmhepes缓冲液(ph=7.5),10mm己二酸半醛,10mml-丙氨酸和100μm吡哆醇5'磷酸盐组成的缓冲液中进行。通过将ω-转氨酶基因产物或空载体对照的无细胞提取物加入到含有己二酸半醛的测定缓冲液中来开始每个酶活性测定反应,并在25℃在250rpm振荡的情况下温育4h。通过rp-hplc定量丙酮酸的形成。
如针对空载体对照所证实的,seqidno7、seqidno8、seqidno9、seqidno10和seqidno12的基因产物接受己二酸半醛作为底物。参见图13。证实了ω-转氨酶活性的可逆性,这表明seqidno7、seqidno8、seqidno9、seqidno10和seqidno12的ω-转氨基酶接受己二酸半醛作为底物,并合成6-氨基己酸作为反应产物。
实施例2
使用6-羟基己酸作为底物并形成6-羟基己醛的羧酸还原酶活性
将编码n末端his-标签的核苷酸序列添加到分别编码seqidno:2-6的羧酸还原酶的来自海分枝杆菌、耻垢分枝杆菌、segniliparusrugosus、马赛分枝杆菌和segniliparusrotundus的核酸序列(参见图6),使得可以产生n末端加his标签的羧酸还原酶。将每个修饰的基因与编码加his标签的来自枯草芽孢杆菌的磷酸泛酰巯基乙胺转移酶的sfp基因一同克隆到petduet表达载体中,均在t7启动子下。将每个表达载体与来自实施例3的表达载体一同转化入bl21[de3]大肠杆菌宿主中。在含有50mllb培养基和抗生素选择压力的250ml摇瓶培养物中于37℃在230rpm振荡的情况下培养每个所得的重组大肠杆菌菌株。使用自诱导培养基在37℃诱导每种培养物过夜。
通过离心收获来自每个诱导的摇瓶培养物的团粒。重悬每个团粒并通过超声处理裂解。通过离心从上清液中分离细胞碎片。使用ni亲和层析从上清液纯化羧酸还原酶和磷酸泛酰巯基乙胺转移酶,以10倍稀释到50mmhepes缓冲液(ph=7.5)中,并通过超滤浓缩。
在由最终浓度为50mmhepes缓冲液(ph=7.5),2mm6-羟基己烷,10mmmgcl2,1mmatp和1mmnadph组成的缓冲液中一式三份进行酶活性测定(即从6-羟基己酸到6-羟基己醛)。通过将纯化的羧酸还原酶和磷酸泛酰巯基乙胺转移酶或空载体对照加入到含有6-羟基己酸的测定缓冲液中,开始每个酶活性测定反应,然后在室温温育20分钟。通过在340nm处的吸光度监测nadph的消耗。不含6-羟基己酸的每个仅酶的对照表明nadph的低基线消耗。参见图7。
如针对空载体对照所证实的,通过sfp的基因产物增强的seqidno:2-6的基因产物接受6-羟基己酸作为底物(参见图13),并合成6-羟基己醛。
实施例3
用于7-氨基己醇,形成7-氧代己醇的ω-转氨酶活性
将编码n-末端his-标签的核苷酸序列添加到分别编码seqidno:7-12的ω-转氨酶的紫色色杆菌、铜绿假单胞菌、丁香假单胞菌、球形红杆菌、大肠杆菌和河流弧菌的核酸序列(参见图6),使得可以产生n末端加his标签的ω-转氨酶。将修饰的基因在t7启动子下克隆到pet21a表达载体中。将每个表达载体转化到bl21[de3]大肠杆菌宿主中。将每个所得的重组大肠杆菌菌株在含有50mllb培养基和抗生素选择压力的250ml摇瓶中于37℃在230rpm振荡情况下培养。使用1mmiptg在16℃诱导每种培养物过夜。
通过离心收获来自每个诱导的摇瓶培养物的团粒。重悬每个团粒并通过超声处理裂解。通过离心从上清液中分离细胞碎片,并将无细胞提取物立即用于酶活性测定。
在由最终浓度为50mmhepes缓冲液(ph=7.5),10mm6-氨基己醇,10mm丙酮酸和100μm吡哆5’磷酸盐组成的缓冲液中进行反向(即6-氨基己醇至6-氧代己醇)酶活性测定。通过将ω-转氨酶基因产物或空载体对照的无细胞提取物加入到含有6-氨基己醇的测定缓冲液中来开始每个酶活性测定反应,并在25℃在250rpm振荡的情况下温育4小时。通过rp-hplc定量l-丙氨酸的形成。
不含7-氨基己醇的每个仅酶的对照具有丙酮酸向l-丙氨酸的低基线转化。参见图11。
如针对空载体对照所证实的,seqidno7-12的基因产物接受6-氨基己醇作为底物(参见图16)并合成6-氧代己醇作为反应产物。鉴于ω-转氨酶活性的可逆性(参见实施例1),可以得出结论,seqidno7-12的基因产物接受6-氨基己醇作为底物并形成6-氧代己醇。
实施例4
使用六亚甲基二胺作为底物并形成6-氨基己醛的ω-转氨酶的酶活性
将编码n末端his-标签的核苷酸序列添加到分别编码seqidno:7-12的ω-转氨酶的来自紫色色杆菌、铜绿假单胞菌、丁香假单胞菌、球形红杆菌、大肠杆菌和河流弧菌的核酸序列(参见图6),使得可以产生n末端加his标签的ω-转氨酶。将修饰的基因在t7启动子下克隆到pet21a表达载体中。将每个表达载体转化到bl21[de3]大肠杆菌宿主中。所得的重组大肠杆菌菌株在含有50mllb培养基和抗生素选择压力的250ml摇瓶培养物中于37℃在以230rpm振荡下培养。使用1mmiptg在16℃诱导每种培养物过夜。
通过离心收获来自每个诱导的摇瓶培养物的团粒。重悬每个团粒并通过超声处理裂解。通过离心从上清液中分离细胞碎片,并将无细胞提取物立即用于酶活性测定。
在由最终浓度为50mmhepes缓冲液(ph=7.5),10mm六亚甲基二胺,10mm丙酮酸和100μμ吡哆5’磷酸盐(pyridoxyl5’phosphate)组成的缓冲液中进行反向(reversedirection)(即六亚甲基二胺至6-氨基己醛)酶活性测定。通过将ω-转氨酶基因产物或空载体对照的无细胞提取物加入到含有六亚甲基二胺的测定缓冲液中来开始每个酶活性测定反应,并在25℃在250rpm振荡的情况下温育4h。通过rp-hplc定量l-丙氨酸的形成。
不含六亚甲基二胺的每个仅酶的对照具有丙酮酸向l-丙氨酸的低基线转化。参见图11。
如针对空载体对照所证实的,seqidno7-12的基因产物接受六亚甲基二胺作为底物并合成6-氨基己醛作为反应产物。鉴于ω-转氨酶活性的可逆性(参见实施例1),可以得出结论,seqidnos:7-12的基因产物接受6-氨基己醛作为底物并形成六亚甲基二胺。
实施例5
用于n6-乙酰基-6-氨基己酸,形成n6-乙酰基-6-氨基己醛的羧酸还原酶活性
在由最终浓度为50mmhepes缓冲液(ph=7.5),2mmn6-乙酰基-6-氨基己酸,10mmmgcl2,1mmatp和1mmnadph组成的缓冲液中一式三份测定用于将n6-乙酰基-6-氨基己酸转化为n6-乙酰基-6-氨基己醛的每个n末端加his标签的seqidnos:4-6的羧酸还原酶的活性(参见实施例2,和图6)。通过将纯化的羧酸还原酶和磷酸泛酰巯基乙胺转移酶或空载体对照加入到含有n6-乙酰基-6-氨基己酸的测定缓冲液中来开始测定,然后在室温温育20min。通过340nm处的吸光度监测nadph的消耗。不含n6-乙酰基-6-氨基己酸的每个仅酶的对照表明nadph的低基线消耗。参见图7。
如针对空载体对照所证实的,通过sfp的基因产物增强的seqidno4-6的基因产物接受n6-乙酰基-6-氨基己酸作为底物(参见图9),并合成n6-乙酰基-6-氨基己醛。
实施例6
使用n6-乙酰基-1,6-二氨基己烷,并形成n6-乙酰基-6-氨基己醛的ω-转氨酶的酶活性
使用由最终浓度为50mmhepes缓冲液(ph=7.5),10mmn6-乙酰基-1,6-二氨基己烷,10mm丙酮酸和100μm吡哆5’磷酸盐组成的缓冲液测定用于将n6-乙酰基-1,6-二氨基己烷转化为n6-乙酰基-6-氨基己醛的n末端加his标签的seqidno:7-12的ω-转氨酶(参见实施例4,和图6)的活性。通过将ω-转氨酶的无细胞提取物或空载体对照加入到含有n6-乙酰基-1,6-二氨基己酸的测定缓冲液中来开始每个酶活性测定反应,然后在25℃在250rpm振荡的情况下温育4h。通过rp-hplc定量l-丙氨酸的形成。
不含n6-乙酰基-1,6-二氨基己酸的每个仅酶的对照表明丙酮酸到l-丙氨酸的低基线转化。参见图11。
如针对空载体对照所证实的,seqidno7-12的基因产物接受n6-乙酰基-1,6-二氨基己烷作为底物(参见图15)并合成n6-乙酰基-6-氨基己醛作为反应产物。
鉴于ω-转氨酶活性的可逆性(参见实施例1),seqidnos:7-12的基因产物接受n6-乙酰基-6-氨基己醛作为底物,形成n6-乙酰基-1,6-二氨基己烷。
实施例7
使用己二酸半醛作为底物并形成己二醛的羧酸还原酶的酶活性
使用己二酸半醛作为底物测定n-末端加his标签的seqidno6的羧酸还原酶(参见实施例2和图6)。在由最终浓度为50mmhepes缓冲液(ph=7.5),2mm己二酸半醛,10mmmgcl2,1mmatp和1mmnadph组成的缓冲液中一式三份进行酶活性测定。通过将纯化的羧酸还原酶和磷酸泛酰巯基乙胺转移酶或空载体对照加入到含有己二酸半醛的测定缓冲液中来开始酶活性测定反应,并然后在室温温育20min。通过340nm处的吸光度监测nadph的消耗。不含己二酸半醛的每个仅酶的对照表明nadph的低基线消耗。参见图7。
如针对空载体对照所证实的,通过sfp的基因产物增强的seqidno:6的基因产物接受己二酸半醛作为底物(参见图10),并合成己二醛。
实施例8
使用4-羟基丁酰基coa和乙酰coa作为底物并形成3-氧代-6-羟基己酰基coa的β-酮硫解酶活性
将编码n末端his-标签的核苷酸序列添加到来自氨基丁酸羧菌的编码seqidno:14的β-酮硫解酶活性的基因(参见图6),使得可以产生n末端加his标签的酶。将所得的修饰的基因在t7启动子控制下克隆到pet15b表达载体中并将该表达载体转化到bl21[de3]大肠杆菌宿主中。将所得的重组大肠杆菌菌株在含有350mllb培养基和抗生素选择压力的1l摇瓶培养物中于37℃在以230rpm振荡下培养。使用1mmiptg在25℃诱导每个培养物过夜。
通过离心收获来自每个诱导的摇瓶培养物的团粒。重悬每个团粒并通过超声处理裂解。通过离心从上清液中分离细胞碎片。使用ni亲和层析从上清液纯化酶,交换缓冲液并通过超滤浓缩至50mm磷酸钾缓冲液(ph=6.8)中。
在由最终浓度为50mm磷酸钾缓冲液(ph=6.8),75μμzncl2,10mmγ-丁内酯和5mm乙酰coa组成的缓冲液中一式三份进行将4-羟基丁酰基coa和乙酰coa转化为3-氧代-6-羟基己酰基coa的酶活性测定。通过将分别至5[μμ]的终浓度的seqidno:14和由来自不动杆菌属chnc编码的内酯酶加入到含有10mmγ-丁内酯和5mm乙酰coa组成的测定缓冲液中,并在30℃在180rpm震荡下温育3小时。通过lc-ms确定3-氧代-6-羟基己酰基-coa的形成。
省略一种底物或一种酶的阴性对照表明没有转化为3-氧代-6-羟基己酰基coa。通过lc-ms,确认了seqidno:14接受4-羟基丁酰基coa和乙酰coa作为底物并合成3-氧代-6-羟基己酰基coa作为产物。
其它实施方案
应理解的是,尽管本发明已经结合其详细说明进行描述,但是前面描述意图说明并且不限制发明的范围,本发明的范围由所附权利要求书的范围限定。其它方面、优点和修改也在权利要求书的范围内。
序列表
<110>英威达技术有限责任公司
<120>使用beta-酮硫酯酶生产6-碳单体的方法
<130>35643-0078wo1
<150>us62/079,903
<151>2014-11-14
<160>17
<170>fastseqforwindowsversion4.0
<210>1
<211>394
<212>prt
<213>钩虫贪铜菌
<400>1
metthrarggluvalvalvalvalserglyvalargthralailegly
151015
thrpheglyglyserleulysaspvalalaproalagluleuglyala
202530
leuvalvalargglualaleualaargalaglnvalserglyaspasp
354045
valglyhisvalvalpheglyasnvalileglnthrgluproargasp
505560
mettyrleuglyargvalalaalavalasnglyglyvalthrileasn
65707580
alaproalaleuthrvalasnargleucysglyserglyleuglnala
859095
ilevalseralaalaglnthrileleuleuglyaspthraspvalala
100105110
ileglyglyglyalaglusermetserargalaprotyrleualapro
115120125
alaalaargtrpglyalaargmetglyaspalaglyleuvalaspmet
130135140
metleuglyalaleuhisaspprophehisargilehismetglyval
145150155160
thralagluasnvalalalysglutyraspileserargalaglngln
165170175
aspglualaalaleugluserhisargargalaseralaalailelys
180185190
alaglytyrphelysaspglnilevalprovalvalserlysglyarg
195200205
lysglyaspvalthrpheaspthraspgluhisvalarghisaspala
210215220
thrileaspaspmetthrlysleuargprovalphevallysgluasn
225230235240
glythrvalthralaglyasnalaserglyleuasnaspalaalaala
245250255
alavalvalmetmetgluargalaglualagluargargglyleulys
260265270
proleualaargleuvalsertyrglyhisalaglyvalaspprolys
275280285
alametglyileglyprovalproalathrlysilealaleugluarg
290295300
alaglyleuglnvalseraspleuaspvalileglualaasngluala
305310315320
phealaalaglnalacysalavalthrlysalaleuglyleuasppro
325330335
alalysvalasnproasnglyserglyileserleuglyhisproile
340345350
glyalathrglyalaleuilethrvallysalaleuhisgluleuasn
355360365
argvalglnglyargtyralaleuvalthrmetcysileglyglygly
370375380
glnglyilealaalailephegluargile
385390
<210>2
<211>1174
<212>prt
<213>海分枝杆菌
<400>2
metserproilethrargglugluargleugluargargileglnasp
151015
leutyralaasnaspproglnphealaalaalalysproalathrala
202530
ilethralaalailegluargproglyleuproleuproglnileile
354045
gluthrvalmetthrglytyralaaspargproalaleualaglnarg
505560
servalgluphevalthraspalaglythrglyhisthrthrleuarg
65707580
leuleuprohisphegluthrilesertyrglygluleutrpasparg
859095
ileseralaleualaaspvalleuserthrgluglnthrvallyspro
100105110
glyaspargvalcysleuleuglypheasnservalasptyralathr
115120125
ileaspmetthrleualaargleuglyalavalalavalproleugln
130135140
thrseralaalailethrglnleuglnproilevalalagluthrgln
145150155160
prothrmetilealaalaservalaspalaleualaaspalathrglu
165170175
leualaleuserglyglnthralathrargvalleuvalpheasphis
180185190
hisargglnvalaspalahisargalaalavalgluseralaargglu
195200205
argleualaglyseralavalvalgluthrleualaglualaileala
210215220
argglyaspvalproargglyalaseralaglyseralaproglythr
225230235240
aspvalseraspaspserleualaleuleuiletyrthrserglyser
245250255
thrglyalaprolysglyalamettyrproargargasnvalalathr
260265270
phetrparglysargthrtrpphegluglyglytyrgluproserile
275280285
thrleuasnphemetprometserhisvalmetglyargglnileleu
290295300
tyrglythrleucysasnglyglythralatyrphevalalalysser
305310315320
aspleuserthrleuphegluaspleualaleuvalargprothrglu
325330335
leuthrphevalproargvaltrpaspmetvalpheaspgluphegln
340345350
sergluvalaspargargleuvalaspglyalaaspargvalalaleu
355360365
glualaglnvallysalagluileargasnaspvalleuglyglyarg
370375380
tyrthrseralaleuthrglyseralaproileseraspglumetlys
385390395400
alatrpvalglugluleuleuaspmethisleuvalgluglytyrgly
405410415
serthrglualaglymetileleuileaspglyalaileargargpro
420425430
alavalleuasptyrlysleuvalaspvalproaspleuglytyrphe
435440445
leuthraspargprohisproargglygluleuleuvallysthrasp
450455460
serleupheproglytyrtyrglnargalagluvalthralaaspval
465470475480
pheaspalaaspglyphetyrargthrglyaspilemetalagluval
485490495
glyprogluglnphevaltyrleuaspargargasnasnvalleulys
500505510
leuserglnglygluphevalthrvalserlysleuglualavalphe
515520525
glyaspserproleuvalargglniletyriletyrglyasnserala
530535540
argalatyrleuleualavalilevalprothrglnglualaleuasp
545550555560
alavalprovalglugluleulysalaargleuglyaspserleugln
565570575
gluvalalalysalaalaglyleuglnsertyrgluileproargasp
580585590
pheileilegluthrthrprotrpthrleugluasnglyleuleuthr
595600605
glyilearglysleualaargproglnleulyslyshistyrglyglu
610615620
leuleugluglniletyrthraspleualahisglyglnalaaspglu
625630635640
leuargserleuargglnserglyalaaspalaprovalleuvalthr
645650655
valcysargalaalaalaalaleuleuglyglyseralaseraspval
660665670
glnproaspalahisphethraspleuglyglyaspserleuserala
675680685
leuserphethrasnleuleuhisgluilepheaspilegluvalpro
690695700
valglyvalilevalserproalaasnaspleuglnalaleualaasp
705710715720
tyrvalglualaalaarglysproglyserserargprothrpheala
725730735
servalhisglyalaserasnglyglnvalthrgluvalhisalagly
740745750
aspleuserleuasplyspheileaspalaalathrleualagluala
755760765
proargleuproalaalaasnthrglnvalargthrvalleuleuthr
770775780
glyalathrglypheleuglyargtyrleualaleuglutrpleuglu
785790795800
argmetaspleuvalaspglylysleuilecysleuvalargalalys
805810815
seraspthrglualaargalaargleuasplysthrpheaspsergly
820825830
aspprogluleuleualahistyrargalaleualaglyasphisleu
835840845
gluvalleualaglyasplysglyglualaaspleuglyleuasparg
850855860
glnthrtrpglnargleualaaspthrvalaspleuilevalasppro
865870875880
alaalaleuvalasnhisvalleuprotyrserglnleupheglypro
885890895
asnalaleuglythralagluleuleuargleualaleuthrserlys
900905910
ilelysprotyrsertyrthrserthrileglyvalalaaspglnile
915920925
proproseralaphethrgluaspalaaspileargvalileserala
930935940
thrargalavalaspaspsertyralaasnglytyrserasnserlys
945950955960
trpalaglygluvalleuleuargglualahisaspleucysglyleu
965970975
provalalavalpheargcysaspmetileleualaaspthrthrtrp
980985990
alaglyglnleuasnvalproaspmetphethrargmetileleuser
99510001005
leualaalathrglyilealaproglyserphetyrgluleualaala
101010151020
aspglyalaargglnargalahistyraspglyleuprovalgluphe
1025103010351040
ilealaglualaileserthrleuglyalaglnserglnaspglyphe
104510501055
histhrtyrhisvalmetasnprotyraspaspglyileglyleuasp
106010651070
gluphevalasptrpleuasngluserglycysproileglnargile
107510801085
alaasptyrglyasptrpleuglnargphegluthralaleuargala
109010951100
leuproaspargglnarghisserserleuleuproleuleuhisasn
1105111011151120
tyrargglnprogluargprovalargglyserilealaprothrasp
112511301135
argpheargalaalavalglnglualalysileglyproasplysasp
114011451150
ileprohisvalglyalaproileilevallystyrvalseraspleu
115511601165
argleuleuglyleuleu
1170
<210>3
<211>1173
<212>prt
<213>耻垢分枝杆菌
<400>3
metthrseraspvalhisaspalathraspglyvalthrgluthrala
151015
leuaspaspgluglnserthrargargilealagluleutyralathr
202530
aspprogluphealaalaalaalaproleuproalavalvalaspala
354045
alahislysproglyleuargleualagluileleuglnthrleuphe
505560
thrglytyrglyaspargproalaleuglytyrargalaarggluleu
65707580
alathraspgluglyglyargthrvalthrargleuleuproargphe
859095
aspthrleuthrtyralaglnvaltrpserargvalglnalavalala
100105110
alaalaleuarghisasnphealaglnproiletyrproglyaspala
115120125
valalathrileglyphealaserproasptyrleuthrleuaspleu
130135140
valcysalatyrleuglyleuvalservalproleuglnhisasnala
145150155160
provalserargleualaproileleualagluvalgluproargile
165170175
leuthrvalseralaglutyrleuaspleualavalgluservalarg
180185190
aspvalasnservalserglnleuvalvalpheasphishisproglu
195200205
valaspasphisargaspalaleualaargalaarggluglnleuala
210215220
glylysglyilealavalthrthrleuaspalailealaaspglugly
225230235240
alaglyleuproalagluproiletyrthralaasphisaspglnarg
245250255
leualametileleutyrthrserglyserthrglyalaprolysgly
260265270
alamettyrthrglualametvalalaargleutrpthrmetserphe
275280285
ilethrglyaspprothrprovalileasnvalasnphemetproleu
290295300
asnhisleuglyglyargileproileserthralavalglnasngly
305310315320
glythrsertyrphevalprogluseraspmetserthrleupheglu
325330335
aspleualaleuvalargprothrgluleuglyleuvalproargval
340345350
alaaspmetleutyrglnhishisleualathrvalaspargleuval
355360365
thrglnglyalaaspgluleuthralaglulysglnalaglyalaglu
370375380
leuarggluglnvalleuglyglyargvalilethrglyphevalser
385390395400
thralaproleualaalaglumetargalapheleuaspilethrleu
405410415
glyalahisilevalaspglytyrglyleuthrgluthrglyalaval
420425430
thrargaspglyvalilevalargproprovalileasptyrlysleu
435440445
ileaspvalprogluleuglytyrpheserthrasplysprotyrpro
450455460
argglygluleuleuvalargserglnthrleuthrproglytyrtyr
465470475480
lysargprogluvalthralaservalpheaspargaspglytyrtyr
485490495
histhrglyaspvalmetalagluthralaproasphisleuvaltyr
500505510
valaspargargasnasnvalleulysleualaglnglyglupheval
515520525
alavalalaasnleuglualavalpheserglyalaalaleuvalarg
530535540
glnilephevaltyrglyasnsergluargserpheleuleualaval
545550555560
valvalprothrproglualaleugluglntyraspproalaalaleu
565570575
lysalaalaleualaaspserleuglnargthralaargaspalaglu
580585590
leuglnsertyrgluvalproalaasppheilevalgluthrglupro
595600605
pheseralaalaasnglyleuleuserglyvalglylysleuleuarg
610615620
proasnleulysaspargtyrglyglnargleugluglnmettyrala
625630635640
aspilealaalathrglnalaasnglnleuarggluleuargargala
645650655
alaalathrglnprovalileaspthrleuthrglnalaalaalathr
660665670
ileleuglythrglysergluvalalaseraspalahisphethrasp
675680685
leuglyglyaspserleuseralaleuthrleuserasnleuleuser
690695700
aspphepheglyphegluvalprovalglythrilevalasnproala
705710715720
thrasnleualaglnleualaglnhisileglualaglnargthrala
725730735
glyaspargargproserphethrthrvalhisglyalaaspalathr
740745750
gluileargalasergluleuthrleuasplyspheileaspalaglu
755760765
thrleuargalaalaproglyleuprolysvalthrthrgluproarg
770775780
thrvalleuleuserglyalaasnglytrpleuglyargpheleuthr
785790795800
leuglntrpleugluargleualaprovalglyglythrleuilethr
805810815
ilevalargglyargaspaspalaalaalaargalaargleuthrgln
820825830
alatyraspthraspprogluleuserargargphealagluleuala
835840845
asparghisleuargvalvalalaglyaspileglyaspproasnleu
850855860
glyleuthrprogluiletrphisargleualaalagluvalaspleu
865870875880
valvalhisproalaalaleuvalasnhisvalleuprotyrarggln
885890895
leupheglyproasnvalvalglythralagluvalilelysleuala
900905910
leuthrgluargilelysprovalthrtyrleuserthrvalserval
915920925
alametglyileproasppheglugluaspglyaspileargthrval
930935940
serprovalargproleuaspglyglytyralaasnglytyrglyasn
945950955960
serlystrpalaglygluvalleuleuargglualahisaspleucys
965970975
glyleuprovalalathrpheargseraspmetileleualahispro
980985990
argtyrargglyglnvalasnvalproaspmetphethrargleuleu
99510001005
leuserleuleuilethrglyvalalaproargserphetyrilegly
101010151020
aspglygluargproargalahistyrproglyleuthrvalaspphe
1025103010351040
valalaglualavalthrthrleuglyalaglnglnarggluglytyr
104510501055
valsertyraspvalmetasnprohisaspaspglyileserleuasp
106010651070
valphevalasptrpleuileargalaglyhisproileaspargval
107510801085
aspasptyraspasptrpvalargargphegluthralaleuthrala
109010951100
leuproglulysargargalaglnthrvalleuproleuleuhisala
1105111011151120
pheargalaproglnalaproleuargglyalaprogluprothrglu
112511301135
valphehisalaalavalargthralalysvalglyproglyaspile
114011451150
prohisleuaspglualaleuileasplystyrileargaspleuarg
115511601165
glupheglyleuile
1170
<210>4
<211>1148
<212>prt
<213>segniliparusrugosus
<400>4
metglyaspglyglugluargalalysargphepheglnargilegly
151015
gluleuseralathraspproglnphealaalaalaalaproasppro
202530
alavalvalglualavalseraspproserleuserphethrargtyr
354045
leuaspthrleumetargglytyralagluargproalaleualahis
505560
argvalglyalaglytyrgluthrilesertyrglygluleutrpala
65707580
argvalglyalailealaalaalatrpglnalaaspglyleualapro
859095
glyaspphevalalathrvalglyphethrserproasptyrvalala
100105110
valaspleualaalaalaargserglyleuvalservalproleugln
115120125
alaglyalaserleualaglnleuvalglyileleuglugluthrglu
130135140
prolysvalleualaalaseralaserserleugluglyalavalala
145150155160
cysalaleualaalaproservalglnargleuvalvalpheaspleu
165170175
argglyproaspalasergluseralaalaaspgluargargglyala
180185190
leualaaspalaglugluglnleualaargalaglyargalavalval
195200205
valgluthrleualaaspleualaalaargglyglualaleuproglu
210215220
alaproleuphegluproalagluglygluaspproleualaleuleu
225230235240
iletyrthrserglyserthrglyalaprolysglyalamettyrser
245250255
glnargleuvalserglnleutrpglyargthrprovalvalprogly
260265270
metproasnileserleuhistyrmetproleuserhissertyrgly
275280285
argalavalleualaglyalaleuseralaglyglythralahisphe
290295300
thralaasnseraspleuserthrleuphegluaspilealaleuala
305310315320
argprothrpheleualaleuvalproargvalcysglumetleuphe
325330335
glngluserglnargglyglnaspvalalagluleuarggluargval
340345350
leuglyglyargleuleuvalalavalcysglyseralaproleuser
355360365
proglumetargalaphemetglugluvalleuglypheproleuleu
370375380
aspglytyrglyserthrglualaleuglyvalmetargasnglyile
385390395400
ileglnargproprovalileasptyrlysleuvalaspvalproglu
405410415
leuglytyrargthrthrasplysprotyrproargglygluleucys
420425430
ileargserthrserleuileserglytyrtyrlysargprogluile
435440445
thralagluvalpheaspalaglnglytyrtyrlysthrglyaspval
450455460
metalagluilealaproasphisleuvaltyrvalaspargserlys
465470475480
asnvalleulysleuserglnglygluphevalalavalalalysleu
485490495
glualaalatyrglythrserprotyrvallysglnilephevaltyr
500505510
glyasnsergluargserpheleuleualavalvalvalproasnala
515520525
gluvalleuglyalaargaspglngluglualalysproleuileala
530535540
alaserleuglnlysilealalysglualaglyleuglnsertyrglu
545550555560
valproargasppheleuilegluthrgluprophethrthrglnasn
565570575
glyleuleusergluvalglylysleuleuargprolysleulysala
580585590
argtyrglyglualaleuglualaargtyraspgluilealahisgly
595600605
glnalaaspgluleuargalaleuargaspglyalaglyglnargpro
610615620
valvalgluthrvalvalargalaalavalalaileserglyserglu
625630635640
glyalagluvalglyproglualaasnphealaaspleuglyglyasp
645650655
serleuseralaleuserleualaasnleuleuhisaspvalpheglu
660665670
valgluvalprovalargileileileglyprothralaserleuala
675680685
glyilealalyshisileglualagluargalaglyalaseralapro
690695700
thralaalaservalhisglyalaglyalathrargileargalaser
705710715720
gluleuthrleuglulyspheleuprogluaspleuleualaalaala
725730735
lysglyleuproalaalaaspglnvalargthrvalleuleuthrgly
740745750
alaasnglytrpleuglyargpheleualaleugluglnleugluarg
755760765
leualaargserglyglnaspglyglylysleuilecysleuvalarg
770775780
glylysaspalaalaalaalaargargargileglugluthrleugly
785790795800
thraspproalaleualaalaargphealagluleualagluglyarg
805810815
leugluvalvalproglyaspvalglygluprolyspheglyleuasp
820825830
aspalaalatrpaspargleualaglugluvalaspvalilevalhis
835840845
proalaalaleuvalasnhisvalleuprotyrhisglnleuphegly
850855860
proasnvalvalglythralagluileileargleualailethrala
865870875880
lysarglysprovalthrtyrleuserthrvalalavalalaalagly
885890895
valgluproserserpheglugluaspglyaspileargalavalval
900905910
progluargproleuglyaspglytyralaasnglytyrglyasnser
915920925
lystrpalaglygluvalleuleuargglualahisgluleuvalgly
930935940
leuprovalalavalpheargseraspmetileleualahisthrarg
945950955960
tyrthrglyglnleuasnvalproaspglnphethrargleuvalleu
965970975
serleuleualathrglyilealaprolysserphetyrglnglngly
980985990
alaalaglygluargglnargalahistyraspglyileprovalasp
99510001005
phethralaglualailethrthrleuglyalagluprosertrpphe
101010151020
aspglyglyalaglypheargserpheaspvalpheasnprohishis
1025103010351040
aspglyvalglyleuaspgluphevalasptrpleuileglualagly
104510501055
hisproileserargileaspasphislysglutrpphealaargphe
106010651070
gluthralavalargglyleuproglualaglnargglnhisserleu
107510801085
leuproleuleuargalatyrserpheprohisproprovalaspgly
109010951100
servaltyrprothrglylyspheglnglyalavallysalaalagln
1105111011151120
valglyserasphisaspvalprohisleuglylysalaleuileval
112511301135
lystyralaaspaspleulysalaleuglyleuleu
11401145
<210>5
<211>1185
<212>prt
<213>马赛分枝杆菌
<400>5
metthrasngluthrasnproglnglngluglnleuserargargile
151015
gluserleuarggluseraspproglnpheargalaalaglnproasp
202530
proalavalalagluglnvalleuargproglyleuhisleuserglu
354045
alailealaalaleumetthrglytyralagluargproalaleugly
505560
gluargalaarggluleuvalileaspglnaspglyargthrthrleu
65707580
argleuleuproargpheaspthrthrthrtyrglygluleutrpser
859095
argthrthrservalalaalaalatrphishisaspalathrhispro
100105110
vallysalaglyaspleuvalalathrleuglyphethrserileasp
115120125
tyrthrvalleuaspleualailemetileleuglyglyvalalaval
130135140
proleuglnthrseralaproalaserglntrpthrthrileleuala
145150155160
glualagluproasnthrleualavalserilegluleuileglyala
165170175
alametgluservalargalathrproserilelysglnvalvalval
180185190
pheasptyrthrprogluvalaspaspglnargglualaphegluala
195200205
alaserthrglnleualaglythrglyilealaleugluthrleuasp
210215220
alavalilealaargglyalaalaleuproalaalaproleutyrala
225230235240
proseralaglyaspaspproleualaleuleuiletyrthrsergly
245250255
serthrglyalaprolysglyalamethissergluasnilevalarg
260265270
argtrptrpilearggluaspvalmetalaglythrgluasnleupro
275280285
metileglyleuasnphemetprometserhisilemetglyarggly
290295300
thrleuthrserthrleuserthrglyglythrglytyrphealaala
305310315320
serseraspmetserthrleuphegluaspmetgluleuileargpro
325330335
thralaleualaleuvalproargvalcysaspmetvalpheglnarg
340345350
pheglnthrgluvalaspargargleualaserglyaspthralaser
355360365
alaglualavalalaalagluvallysalaaspileargaspasnleu
370375380
pheglyglyargvalseralavalmetvalglyseralaproleuser
385390395400
glugluleuglyglupheileglusercysphegluleuasnleuthr
405410415
aspglytyrglyserthrglualaglymetvalpheargaspglyile
420425430
valglnargproprovalileasptyrlysleuvalaspvalproglu
435440445
leuglytyrpheserthrasplysprohisproargglygluleuleu
450455460
leulysthraspglymetpheleuglytyrtyrlysargprogluval
465470475480
thralaservalpheaspalaaspglyphetyrmetthrglyaspile
485490495
valalagluleualahisaspasnilegluileileaspargargasn
500505510
asnvalleulysleuserglnglygluphevalalavalalathrleu
515520525
glualaglutyralaasnserprovalvalhisglniletyrvaltyr
530535540
glysersergluargsertyrleuleualavalvalvalprothrpro
545550555560
glualavalalaalaalalysglyaspalaalaalaleulysthrthr
565570575
ilealaaspserleuglnaspilealalysgluileglnleuglnser
580585590
tyrgluvalproargasppheileilegluproglnprophethrgln
595600605
glyasnglyleuleuthrglyilealalysleualaargproasnleu
610615620
lysalahistyrglyproargleugluglnmettyralagluileala
625630635640
gluglnglnalaalagluleuargalaleuhisglyvalaspproasp
645650655
lysproalaleugluthrvalleulysalaalaglnalaleuleugly
660665670
valserseralagluleualaalaaspalahisphethraspleugly
675680685
glyaspserleuseralaleuserpheseraspleuleuargaspile
690695700
phealavalgluvalprovalglyvalilevalseralaalaasnasp
705710715720
leuglyglyvalalalysphevalaspgluglnarghisserglygly
725730735
thrargprothralagluthrvalhisglyalaglyhisthrgluile
740745750
argalaalaaspleuthrleuasplyspheileaspglualathrleu
755760765
hisalaalaproserleuprolysalaalaglyileprohisthrval
770775780
leuleuthrglyserasnglytyrleuglyhistyrleualaleuglu
785790795800
trpleugluargleuasplysthraspglylysleuilevalileval
805810815
argglylysasnalaglualaalatyrglyargleugluglualaphe
820825830
aspthrglyaspthrgluleuleualahispheargserleualaasp
835840845
lyshisleugluvalleualaglyaspileglyaspproasnleugly
850855860
leuaspalaaspthrtrpglnargleualaaspthrvalaspvalile
865870875880
valhisproalaalaleuvalasnhisvalleuprotyrasnglnleu
885890895
pheglyproasnvalvalglythralagluileilelysleualaile
900905910
thrthrlysilelysprovalthrtyrleuserthrvalalavalala
915920925
alatyrvalaspprothrthrpheaspglugluseraspileargleu
930935940
ileseralavalargproileaspaspglytyralaasnglytyrgly
945950955960
asnalalystrpalaglygluvalleuleuargglualahisaspleu
965970975
cysglyleuprovalalavalpheargseraspmetileleualahis
980985990
serargtyrthrglyglnleuasnvalproaspglnphethrargleu
99510001005
ileleuserleuilealathrglyilealaproglyserphetyrgln
101010151020
alaglnthrthrglygluargproleualahistyraspglyleupro
1025103010351040
glyaspphethralaglualailethrthrleuglythrglnvalpro
104510501055
gluglysergluglyphevalthrtyraspcysvalasnprohisala
106010651070
aspglyileserleuaspasnphevalasptrpleuileglualagly
107510801085
tyrproilealaargileaspasntyrthrglutrpphethrargphe
109010951100
aspthralaileargglyleuserglulysglnlysglnhisserleu
1105111011151120
leuproleuleuhisalaphegluglnproseralaalagluasnhis
112511301135
glyvalvalproalalysargpheglnhisalavalglnalaalagly
114011451150
ileglyprovalglyglnaspglythrthraspileprohisleuser
115511601165
argargleuilevallystyralalysaspleugluglnleuglyleu
117011751180
leu
1185
<210>6
<211>1186
<212>prt
<213>segniliparusrotundus
<400>6
metthrglnserhisthrglnglyproglnalaseralaalahisser
151015
argleualaargargalaalagluleuleualathraspproglnala
202530
alaalathrleuproaspprogluvalvalargglnalathrargpro
354045
glyleuargleualagluargvalaspalaileleuserglytyrala
505560
aspargproalaleuglyglnargserpheglnthrvallysasppro
65707580
ilethrglyargserservalgluleuleuprothrpheaspthrile
859095
thrtyrarggluleuarggluargalathralailealaseraspleu
100105110
alahishisproglnalaproalalysproglyasppheleualaser
115120125
ileglypheileservalasptyrvalalaileaspilealaglyval
130135140
phealaglyleuthralavalproleuglnthrglyalathrleuala
145150155160
thrleuthralailethralagluthralaprothrleuphealaala
165170175
serilegluhisleuprothralavalaspalavalleualathrpro
180185190
servalargargleuleuvalpheasptyrargalaglyseraspglu
195200205
aspargglualavalglualaalalysarglysilealaaspalagly
210215220
serservalleuvalaspvalleuaspgluvalilealaargglylys
225230235240
seralaprolysalaproleuproproalathraspalaglyaspasp
245250255
serleuserleuleuiletyrthrserglyserthrglythrprolys
260265270
glyalamettyrprogluargasnvalalahisphetrpglyglyval
275280285
trpalaalaalapheaspgluaspalaalaproprovalproalaile
290295300
asnilethrpheleuproleuserhisvalalaserargleuserleu
305310315320
metprothrleualaargglyglyleumethisphevalalalysser
325330335
aspleuserthrleuphegluaspleulysleualaargprothrasn
340345350
leupheleuvalproargvalvalglumetleutyrglnhistyrgln
355360365
sergluleuaspargargglyvalglnaspglythrargglualaglu
370375380
alavallysaspaspleuargthrglyleuleuglyglyargileleu
385390395400
thralaglypheglyseralaproleuseralagluleualaglyphe
405410415
ilegluserleuleuglnilehisleuvalaspglytyrglyserthr
420425430
glualaglyprovaltrpargaspglytyrleuvallysproproval
435440445
thrasptyrlysleuileaspvalprogluleuglytyrpheserthr
450455460
aspserprohisproargglygluleualailelysthrglnthrile
465470475480
leuproglytyrtyrlysargprogluthrthralagluvalpheasp
485490495
gluaspglyphetyrleuthrglyaspvalvalalaglnileglypro
500505510
gluglnphealatyrvalaspargarglysasnvalleulysleuser
515520525
glnglygluphevalthrleualalysleuglualaalatyrserser
530535540
serproleuvalargglnleuphevaltyrglysersergluargser
545550555560
tyrleuleualavalilevalprothrproaspalaleulyslysphe
565570575
glyvalglyglualaalalysalaalaleuglygluserleuglnlys
580585590
ilealaargaspgluglyleuglnsertyrgluvalproargaspphe
595600605
ileilegluthraspprophethrvalgluasnglyleuleuserasp
610615620
alaarglysserleuargprolysleulysgluhistyrglygluarg
625630635640
leuglualamettyrlysgluleualaaspglyglnalaasngluleu
645650655
argaspileargargglyvalglnglnargprothrleugluthrval
660665670
argargalaalaalaalametleuglyalaseralaalagluilelys
675680685
proaspalahisphethraspleuglyglyaspserleuseralaleu
690695700
thrpheserasnpheleuhisaspleuphegluvalaspvalproval
705710715720
glyvalilevalseralaalaasnthrleuglyservalalagluhis
725730735
ileaspalaglnleualaglyglyargalaargprothrphealathr
740745750
valhisglylysglyserthrthrilelysalaseraspleuthrleu
755760765
asplyspheileaspgluglnthrleuglualaalalyshisleupro
770775780
lysproalaaspproproargthrvalleuleuthrglyalaasngly
785790795800
trpleuglyargpheleualaleuglutrpleugluargleualapro
805810815
alaglyglylysleuilethrilevalargglylysaspalaalagln
820825830
alalysalaargleuaspalaalatyrgluserglyaspprolysleu
835840845
alaglyhistyrglnaspleualaalathrthrleugluvalleuala
850855860
glyaspphesergluproargleuglyleuaspglualathrtrpasn
865870875880
argleualaaspgluvalasppheileserhisproglyalaleuval
885890895
asnhisvalleuprotyrasnglnleupheglyproasnvalalagly
900905910
valalagluileilelysleualailethrthrargilelysproval
915920925
thrtyrleuserthrvalalavalalaalaglyvalgluproserala
930935940
leuaspgluaspglyaspileargthrvalseralagluargserval
945950955960
aspgluglytyralaasnglytyrglyasnserlystrpglyglyglu
965970975
valleuleuargglualahisaspargthrglyleuprovalargval
980985990
pheargseraspmetileleualahisglnlystyrthrglyglnval
99510001005
asnalathraspglnphethrargleuvalglnserleuleualathr
101010151020
glyleualaprolysserphetyrgluleuaspalaglnglyasnarg
1025103010351040
glnargalahistyraspglyileprovalaspphethralagluser
104510501055
ilethrthrleuglyglyaspglyleugluglytyrargsertyrasn
106010651070
valpheasnprohisargaspglyvalglyleuaspgluphevalasp
107510801085
trpleuileglualaglyhisproilethrargileaspasptyrasp
109010951100
glntrpleuserargphegluthrserleuargglyleuprogluser
1105111011151120
lysargglnalaservalleuproleuleuhisalaphealaargpro
112511301135
glyproalavalaspglyserpropheargasnthrvalpheargthr
114011451150
aspvalglnlysalalysileglyalagluhisaspileprohisleu
115511601165
glylysalaleuvalleulystyralaaspaspilelysglnleugly
117011751180
leuleu
1185
<210>7
<211>459
<212>prt
<213>紫色色杆菌
<400>7
metglnlysglnargthrthrserglntrparggluleuaspalaala
151015
hishisleuhisprophethraspthralaserleuasnglnalagly
202530
alaargvalmetthrargglygluglyvaltyrleutrpaspserglu
354045
glyasnlysileileaspglymetalaglyleutrpcysvalasnval
505560
glytyrglyarglysaspphealaglualaalaargargglnmetglu
65707580
gluleuprophetyrasnthrphephelysthrthrhisproalaval
859095
valgluleuserserleuleualagluvalthrproalaglypheasp
100105110
argvalphetyrthrasnserglysergluservalaspthrmetile
115120125
argmetvalargargtyrtrpaspvalglnglylysproglulyslys
130135140
thrleuileglyargtrpasnglytyrhisglyserthrileglygly
145150155160
alaserleuglyglymetlystyrmethisgluglnglyaspleupro
165170175
ileproglymetalahisilegluglnprotrptrptyrlyshisgly
180185190
lysaspmetthrproaspglupheglyvalvalalaalaargtrpleu
195200205
gluglulysileleugluileglyalaasplysvalalaalapheval
210215220
glygluproileglnglyalaglyglyvalilevalproproalathr
225230235240
tyrtrpprogluilegluargilecysarglystyraspvalleuleu
245250255
valalaaspgluvalilecysglypheglyargthrglyglutrpphe
260265270
glyhisglnhispheglypheglnproaspleuphethralaalalys
275280285
glyleuserserglytyrleuproileglyalavalphevalglylys
290295300
argvalalagluglyleuilealaglyglyasppheasnhisglyphe
305310315320
thrtyrserglyhisprovalcysalaalavalalahisalaasnval
325330335
alaalaleuargaspgluglyilevalglnargvallysaspaspile
340345350
glyprotyrmetglnlysargtrparggluthrpheserargpheglu
355360365
hisvalaspaspvalargglyvalglymetvalglnalaphethrleu
370375380
vallysasnlysalalysarggluleupheproasppheglygluile
385390395400
glythrleucysargaspilephepheargasnasnleuilemetarg
405410415
alacysglyasphisilevalseralaproproleuvalmetthrarg
420425430
alagluvalaspglumetleualavalalagluargcysleugluglu
435440445
phegluglnthrleulysalaargglyleuala
450455
<210>8
<211>468
<212>prt
<213>铜绿假单胞菌
<400>8
metasnalaargleuhisalathrserproleuglyaspalaaspleu
151015
valargalaaspglnalahistyrmethisglytyrhisvalpheasp
202530
asphisargvalasnglyserleuasnilealaalaglyaspglyala
354045
tyriletyraspthralaglyasnargtyrleuaspalavalglygly
505560
mettrpcysthrasnileglyleuglyarggluglumetalaargthr
65707580
valalagluglnthrargleuleualatyrserasnprophecysasp
859095
metalaasnproargalailegluleucysarglysleualagluleu
100105110
alaproglyaspleuasphisvalpheleuthrthrglyglyserthr
115120125
alavalaspthralaileargleumethistyrtyrglnasncysarg
130135140
glylysargalalyslyshisvalilethrargileasnalatyrhis
145150155160
glyserthrpheleuglymetserleuglyglylysseralaasparg
165170175
proalaglupheasppheleuaspgluargilehishisleualacys
180185190
protyrtyrtyrargalaprogluglyleuglyglualaglupheleu
195200205
aspglyleuvalaspgluphegluarglysileleugluleuglyala
210215220
aspargvalglyalapheilesergluprovalpheglyserglygly
225230235240
valilevalproproalaglytyrhisargargmettrpgluleucys
245250255
glnargtyraspvalleutyrileseraspgluvalvalthrserphe
260265270
glyargleuglyhisphephealaserglnalavalpheglyvalgln
275280285
proaspileileleuthralalysglyleuthrserglytyrglnpro
290295300
leuglyalacysilepheserargargiletrpgluvalilealaglu
305310315320
proasplysglyargcyspheserhisglyphethrtyrserglyhis
325330335
provalalacysalaalaalaleulysasnilegluileilegluarg
340345350
gluglyleuleualahisalaaspgluvalglyargtyrphegluglu
355360365
argleuglnserleuargaspleuproilevalglyaspvalarggly
370375380
metargphemetalacysvalgluphevalalaasplysalaserlys
385390395400
alaleupheprogluserleuasnileglyglutrpvalhisleuarg
405410415
alaglnlysargglyleuleuvalargproilevalhisleuasnval
420425430
metserproproleuileleuthrarggluglnvalaspthrvalval
435440445
argvalleuarggluserileglugluthrvalgluaspleuvalarg
450455460
alaglyhisarg
465
<210>9
<211>454
<212>prt
<213>丁香假单胞菌
<400>9
metseralaasnasnproglnthrleuglutrpglnalaleuserser
151015
gluhishisleualapropheserasptyrlysglnleulysglulys
202530
glyproargileilethrargalagluglyvaltyrleutrpaspser
354045
gluglyasnlysileleuaspglymetserglyleutrpcysvalala
505560
ileglytyrglyargglugluleualaaspalaalaserlysglnmet
65707580
arggluleuprotyrtyrasnleuphepheglnthralahispropro
859095
valleugluleualalysalaileseraspilealaprogluglymet
100105110
asnhisvalphephethrglyserglysergluglyasnaspthrmet
115120125
leuargmetvalarghistyrtrpalaleulysglyglnproasnlys
130135140
lysthrileileserargvalasnglytyrhisglyserthrvalala
145150155160
glyalaserleuglyglymetthrtyrmethisgluglnglyaspleu
165170175
proileproglyvalvalhisileproglnprotyrtrppheglyglu
180185190
glyglyaspmetthrproaspglupheglyiletrpalaalaglugln
195200205
leuglulyslysileleugluleuglyvalgluasnvalglyalaphe
210215220
ilealagluproileglnglyalaglyglyvalilevalproproasp
225230235240
sertyrtrpprolysilelysgluileleuserargtyraspileleu
245250255
phealaalaaspgluvalilecysglypheglyargthrserglutrp
260265270
pheglyseraspphetyrglyleuargproaspmetmetthrileala
275280285
lysglyleuthrserglytyrvalprometglyglyleuilevalarg
290295300
aspgluilevalalavalleuasngluglyglyasppheasnhisgly
305310315320
phethrtyrserglyhisprovalalaalaalavalalaleugluasn
325330335
ileargileleuarggluglulysilevalgluargvalargserglu
340345350
thralaprotyrleuglnlysargleuarggluleuserasphispro
355360365
leuvalglygluvalargglyvalglyleuleuglyalailegluleu
370375380
vallysasplysthrthrarggluargtyrthrasplysglyalagly
385390395400
metilecysargthrphecyspheaspasnglyleuilemetargala
405410415
valglyaspthrmetileilealaproproleuvalileserpheala
420425430
glnileaspgluleuvalglulysalaargthrcysleuaspleuthr
435440445
leualavalleuglngly
450
<210>10
<211>467
<212>prt
<213>球形红杆菌
<400>10
metthrargasnaspalathrasnalaalaglyalavalglyalaala
151015
metargasphisileleuleuproalaglnglumetalalysleugly
202530
lysseralaglnprovalleuthrhisalagluglyiletyrvalhis
354045
thrgluaspglyargargleuileaspglyproalaglymettrpcys
505560
alaglnvalglytyrglyargarggluilevalaspalametalahis
65707580
glnalametvalleuprotyralaserprotrptyrmetalathrser
859095
proalaalaargleualaglulysilealathrleuthrproglyasp
100105110
leuasnargilephephethrthrglyglyserthralavalaspser
115120125
alaleuargphesergluphetyrasnasnvalleuglyargprogln
130135140
lyslysargileilevalargtyraspglytyrhisglyserthrala
145150155160
leuthralaalacysthrglyargthrglyasntrpproasnpheasp
165170175
ilealaglnaspargileserpheleuserserproasnproarghis
180185190
alaglyasnargserglnglualapheleuaspaspleuvalglnglu
195200205
phegluaspargilegluserleuglyproaspthrilealaalaphe
210215220
leualagluproileleualaserglyglyvalileileproproala
225230235240
glytyrhisalaargphelysalailecysglulyshisaspileleu
245250255
tyrileseraspgluvalvalthrglypheglyargcysglyglutrp
260265270
phealaserglulysvalpheglyvalvalproaspileilethrphe
275280285
alalysglyvalthrserglytyrvalproleuglyglyleualaile
290295300
serglualavalleualaargileserglygluasnalalysglyser
305310315320
trpphethrasnglytyrthrtyrserasnglnprovalalacysala
325330335
alaalaleualaasnilegluleumetgluarggluglyilevalasp
340345350
glnalaargglumetalaasptyrphealaalaalaleualaserleu
355360365
argaspleuproglyvalalagluthrargservalglyleuvalgly
370375380
cysvalglncysleuleuaspprothrargalaaspglythralaglu
385390395400
asplysalaphethrleulysileaspgluargcysphegluleugly
405410415
leuilevalargproleuglyaspleucysvalileserproproleu
420425430
ileileserargalaglnileaspglumetvalalailemetarggln
435440445
alailethrgluvalseralaalahisglyleuthralalysglupro
450455460
alaalaval
465
<210>11
<211>459
<212>prt
<213>大肠杆菌
<400>11
metasnargleuproserseralaseralaleualacysseralahis
151015
alaleuasnleuileglulysargthrleuasphisgluglumetlys
202530
alaleuasnarggluvalileglutyrphelysgluhisvalasnpro
354045
glypheleuglutyrarglysservalthralaglyglyasptyrgly
505560
alavalglutrpglnalaglyserleuasnthrleuvalaspthrgln
65707580
glyglnglupheileaspcysleuglyglypheglyilepheasnval
859095
glyhisargasnprovalvalvalseralavalglnasnglnleuala
100105110
lysglnproleuhisserglngluleuleuaspproleuargalamet
115120125
leualalysthrleualaalaleuthrproglylysleulystyrser
130135140
phephecysasnserglythrgluservalglualaalaleulysleu
145150155160
alalysalatyrglnserproargglylysphethrpheilealathr
165170175
serglyalaphehisglylysserleuglyalaleuseralathrala
180185190
lysserthrphearglysprophemetproleuleuproglyphearg
195200205
hisvalpropheglyasnileglualametargthralaleuasnglu
210215220
cyslyslysthrglyaspaspvalalaalavalileleugluproile
225230235240
glnglygluglyglyvalileleuproproproglytyrleuthrala
245250255
valarglysleucysaspglupheglyalaleumetileleuaspglu
260265270
valglnthrglymetglyargthrglylysmetphealacysgluhis
275280285
gluasnvalglnproaspileleucysleualalysalaleuglygly
290295300
glyvalmetproileglyalathrilealathrglugluvalpheser
305310315320
valleupheaspasnpropheleuhisthrthrthrpheglyglyasn
325330335
proleualacysalaalaalaleualathrileasnvalleuleuglu
340345350
glnasnleuproalaglnalagluglnlysglyaspmetleuleuasp
355360365
glypheargglnleualaargglutyrproaspleuvalglngluala
370375380
argglylysglymetleumetalailegluphevalaspasngluile
385390395400
glytyrasnphealaserglumetpheargglnargvalleuvalala
405410415
glythrleuasnasnalalysthrileargilegluproproleuthr
420425430
leuthrilegluglncysgluleuvalilelysalaalaarglysala
435440445
leualaalametargvalservalglugluala
450455
<210>12
<211>453
<212>prt
<213>河流弧菌
<400>12
metasnlysproglnsertrpglualaargalagluthrtyrserleu
151015
tyrglyphethraspmetproserleuhisglnargglythrvalval
202530
valthrhisglygluglyprotyrilevalaspvalasnglyargarg
354045
tyrleuaspalaasnserglyleutrpasnmetvalalaglypheasp
505560
hislysglyleuileaspalaalalysalaglntyrgluargphepro
65707580
glytyrhisalaphepheglyargmetseraspglnthrvalmetleu
859095
serglulysleuvalgluvalserpropheaspserglyargvalphe
100105110
tyrthrasnserglyserglualaasnaspthrmetvallysmetleu
115120125
trppheleuhisalaalagluglylysproglnlysarglysileleu
130135140
thrargtrpasnalatyrhisglyvalthralavalseralasermet
145150155160
thrglylysprotyrasnservalpheglyleuproleuproglyphe
165170175
valhisleuthrcysprohistyrtrpargtyrglyglugluglyglu
180185190
thrglugluglnphevalalaargleualaarggluleuglugluthr
195200205
ileglnarggluglyalaaspthrilealaglyphephealaglupro
210215220
valmetglyalaglyglyvalileproproalalysglytyrphegln
225230235240
alaileleuproileleuarglystyraspileprovalileserasp
245250255
gluvalilecysglypheglyargthrglyasnthrtrpglycysval
260265270
thrtyraspphethrproaspalaileileserserlysasnleuthr
275280285
alaglyphepheprometglyalavalileleuglyprogluleuser
290295300
lysargleugluthralaileglualailegluglupheprohisgly
305310315320
phethralaserglyhisprovalglycysalailealaleulysala
325330335
ileaspvalvalmetasngluglyleualagluasnvalargargleu
340345350
alaproargpheglugluargleulyshisilealagluargproasn
355360365
ileglyglutyrargglyileglyphemettrpalaleuglualaval
370375380
lysasplysalaserlysthrpropheaspglyasnleuservalser
385390395400
gluargilealaasnthrcysthraspleuglyleuilecysargpro
405410415
leuglyglnservalvalleucyspropropheileleuthrgluala
420425430
glnmetaspglumetpheasplysleuglulysalaleuasplysval
435440445
phealagluvalala
450
<210>13
<211>401
<212>prt
<213>大肠杆菌
<400>13
metargglualapheilecysaspglyileargthrproileglyarg
151015
tyrglyglyalaleuserservalargalaaspaspleualaalaile
202530
proleuarggluleuleuvalargasnproargleuaspalaglucys
354045
ileaspaspvalileleuglycysalaasnglnalaglygluaspasn
505560
argasnvalalaargmetalathrleuleualaglyleuproglnser
65707580
valserglythrthrileasnargleucysglyserglyleuaspala
859095
leuglyphealaalaargalailelysalaglyaspglyaspleuleu
100105110
ilealaglyglyvalglusermetserargalaprophevalmetgly
115120125
lysalaalaseralapheserargglnalaglumetpheaspthrthr
130135140
ileglytrpargphevalasnproleumetalaglnglnpheglythr
145150155160
aspsermetprogluthralagluasnvalalagluleuleulysile
165170175
serarggluaspglnaspserphealaleuargserglnglnargthr
180185190
alalysalaglnserserglyileleualaglugluilevalproval
195200205
valleulysasnlyslysglyvalvalthrgluileglnhisaspglu
210215220
hisleuargprogluthrthrleugluglnleuargglyleulysala
225230235240
propheargalaasnglyvalilethralaglyasnalaserglyval
245250255
asnaspglyalaalaalaleuileilealasergluglnmetalaala
260265270
alaglnglyleuthrproargalaargilevalalametalathrala
275280285
glyvalgluproargleumetglyleuglyprovalproalathrarg
290295300
argvalleugluargalaglyleuserilehisaspmetaspvalile
305310315320
gluleuasnglualaphealaalaglnalaleuglyvalleuargglu
325330335
leuglyleuproaspaspalaprohisvalasnproasnglyglyala
340345350
ilealaleuglyhisproleuglymetserglyalaargleualaleu
355360365
alaalaserhisgluleuhisargargasnglyargtyralaleucys
370375380
thrmetcysileglyvalglyglnglyilealametileleugluarg
385390395400
val
<210>14
<211>438
<212>prt
<213>氨基丁酸羧菌
<400>14
metasptrplyslysiletyrgluaspargthrcysthralaaspglu
151015
alavallysserilelysserglyaspargvalleuphealahiscys
202530
valalagluproprovalleuvalglualametvalalaasnalaala
354045
alatyrlysasnvalthrvalserhismetvalthrleuglylysgly
505560
glutyrserlysproglutyrlysgluasnphethrphegluglytrp
65707580
phethrserproserthrargglyserilealagluglyhisglygln
859095
phevalprovalphephehisgluvalproserleuilearglysasp
100105110
ilephehisvalaspvalphemetvalmetvalserproproasphis
115120125
asnglyphecyscysvalglyvalserserasptyrthrmetglnala
130135140
ilelysseralalysilevalleualagluvalasnaspglnvalpro
145150155160
valvaltyrglyaspthrphevalhisvalsergluileasplysphe
165170175
valgluthrserhisproleuprogluileglyleuprolysilegly
180185190
gluvalglualaalaileglylyshiscysalaserleuilegluasp
195200205
glyserthrleuglnleuglyileglyalaileproaspalavalleu
210215220
serglnleulysasplyslyshisleuglyilehisserglumetile
225230235240
seraspglyvalvalaspleutyrglualaglyvalileaspcysser
245250255
glnlysserileasplysglylysmetalailethrpheleumetgly
260265270
thrlysargleutyraspphealaalaasnasnprolysvalgluleu
275280285
lysprovalasptyrileasnhisproservalvalalaglncysser
290295300
lysmetvalcysileasnalacysleuglnvalaspphemetglygln
305310315320
ilevalseraspserileglythrlysglnpheserglyvalglygly
325330335
glnvalaspphevalargglyalasermetserileaspglylysgly
340345350
lysalaileilealametproservalalalyslyslysaspglyser
355360365
metileserlysilevalpropheileasphisglyalaalavalthr
370375380
thrserargasnaspalaasptyrvalvalthrglutyrglyileala
385390395400
glumetlysglylysserleuglnaspargalaargalaleuileasn
405410415
ilealahisproaspphelysaspgluleulysalaglupheglulys
420425430
argpheasnalaalaphe
435
<210>15
<211>397
<212>prt
<213>齿垢密螺旋体
<400>15
metilevallysprometvalargasnasnilecysleuasnalahis
151015
proglnglycyslyslysglyvalgluaspglnileglutyrthrlys
202530
lysargilethralagluvallysalaglyalalysalaprolysasn
354045
valleuvalleuglycysserasnglytyrglyleualaserargile
505560
thralaalapheglytyrglyalaalathrileglyvalserpheglu
65707580
lysalaglysergluthrlystyrglythrproglytrptyrasnasn
859095
leualapheaspglualaalalysarggluglyleutyrservalthr
100105110
ileaspglyaspalapheseraspgluilelysalaglnvalileglu
115120125
glualalyslyslysglyilelyspheaspleuilevaltyrserleu
130135140
alaserprovalargthraspproaspthrglyilemethislysser
145150155160
valleulyspropheglylysthrphethrglylysthrvalasppro
165170175
phethrglygluleulysgluileseralagluproalaasnaspglu
180185190
glualaalaalathrvallysvalmetglyglygluasptrpgluarg
195200205
trpilelysglnleuserlysgluglyleuleuglugluglycysile
210215220
thrleualatyrsertyrileglyproglualathrglnalaleutyr
225230235240
arglysglythrileglylysalalysgluhisleuglualathrala
245250255
hisargleuasnlysgluasnproserileargalaphevalserval
260265270
asnlysglyleuvalthrargalaseralavalileprovalilepro
275280285
leutyrleualaserleuphelysvalmetlysglulysglyasnhis
290295300
gluglycysilegluglnilethrargleutyralagluargleutyr
305310315320
arglysaspglythrileprovalaspglugluasnargileargile
325330335
aspasptrpgluleuglugluaspvalglnlysalavalseralaleu
340345350
metglulysvalthrglygluasnalagluserleuthraspleuala
355360365
glytyrarghisasppheleualaserasnglypheaspvalglugly
370375380
ileasntyrglualagluvalgluargpheaspargile
385390395
<210>16
<211>539
<212>prt
<213>euglenagracilis
<400>16
metsercysproalaserproseralaalavalvalseralaglyala
151015
leucysleucysvalalathrvalleuleualathrglyserasnpro
202530
thralaleuserthralaserthrargserprothrserleuvalarg
354045
glyvalaspargglyleumetargprothrthralaalaalaleuthr
505560
thrmetarggluvalproglnmetalagluglypheserglygluala
65707580
thrseralatrpalaalaalaglyproglntrpalaalaproleuval
859095
alaalaalaserseralaleualaleutrptrptrpalaalaargarg
100105110
servalargargproleualaalaleualagluleuprothralaval
115120125
thrhisleualaproprometalametphethrthrthralalysval
130135140
ileglnprolysileargglypheilecysthrthrthrhisproile
145150155160
glycysglulysargvalglnglugluilealatyralaargalahis
165170175
proprothrserproglyprolysargvalleuvalileglycysser
180185190
thrglytyrglyleuserthrargilethralaalapheglytyrgln
195200205
alaalathrleuglyvalpheleualaglyproprothrlysglyarg
210215220
proalaalaalaglytrptyrasnthrvalalapheglulysalaala
225230235240
leuglualaglyleutyralaargserleuasnglyaspalapheasp
245250255
serthrthrlysalaargthrvalglualailelysargaspleugly
260265270
thrvalaspleuvalvaltyrserilealaalaprolysargthrasp
275280285
proalathrglyvalleuhislysalacysleulysproileglyala
290295300
thrtyrthrasnargthrvalasnthrasplysalagluvalthrasp
305310315320
valserilegluproalaserproglugluilealaaspthrvallys
325330335
valmetglyglygluasptrpgluleutrpileglnalaleuserglu
340345350
alaglyvalleualagluglyalalysthrvalalatyrsertyrile
355360365
glyproglumetthrtrpprovaltyrtrpserglythrileglyglu
370375380
alalyslysaspvalglulysalaalalysargilethrglnglntyr
385390395400
glycysproalatyrprovalvalalalysalaleuvalthrglnala
405410415
serseralaileprovalvalproleutyrilecysleuleutyrarg
420425430
valmetlysglulysglythrhisgluglycysilegluglnmetval
435440445
argleuleuthrthrlysleutyrprogluasnglyalaproileval
450455460
aspglualaglyargvalargvalaspasptrpglumetalagluasp
465470475480
valglnglnalavallysaspleutrpserglnvalserthralaasn
485490495
leulysaspileseraspphealaglytyrglnthrglupheleuarg
500505510
leupheglypheglyileaspglyvalasptyraspglnprovalasp
515520525
valglualaaspleuproseralaalaglngln
530535
<210>17
<211>550
<212>prt
<213>鼠伤寒沙门氏菌
<400>17
metglnasnprotyrthrvalalaasptyrleuleuaspargleuala
151015
glycysglyileglyhisleupheglyvalproglyasptyrasnleu
202530
glnpheleuasphisvalileasphisprothrleuargtrpvalgly
354045
cysalaasngluleuasnalaalatyralaalaaspglytyralaarg
505560
metserglyalaglyalaleuleuthrthrpheglyvalglygluleu
65707580
seralaileasnglyilealaglysertyralaglutyrvalproval
859095
leuhisilevalglyalaprocysseralaalaglnglnargglyglu
100105110
leumethishisthrleuglyaspglyaspphearghisphetyrarg
115120125
metserglnalaileseralaalaseralaileleuaspgluglnasn
130135140
alacysphegluileaspargvalleuglyglumetleualaalaarg
145150155160
argproglytyrilemetleuproalaaspvalalalyslysthrala
165170175
ileproprothrglnalaleualaleuprovalhisglualaglnser
180185190
glyvalgluthralapheargtyrhisalaargglncysleumetasn
195200205
serargargilealaleuleualaasppheleualaglyargphegly
210215220
leuargproleuleuglnargtrpmetalagluthrproilealahis
225230235240
alathrleuleumetglylysglyleupheaspgluglnhisproasn
245250255
phevalglythrtyrseralaglyalaserserlysgluvalarggln
260265270
alailegluaspalaaspargvalilecysvalglythrargpheval
275280285
aspthrleuthralaglyphethrglnglnleuproalagluargthr
290295300
leugluileglnprotyralaserargileglygluthrtrppheasn
305310315320
leuprometalaglnalavalserthrleuarggluleucysleuglu
325330335
cysalaphealaproproprothrargseralaglyglnprovalarg
340345350
ileasplysglygluleuthrglngluserphetrpglnthrleugln
355360365
glntyrleulysproglyaspileileleuvalaspglnglythrala
370375380
alapheglyalaalaalaleuserleuproaspglyalagluvalval
385390395400
leuglnproleutrpglyserileglytyrserleuproalaalaphe
405410415
glyalaglnthralacysproaspargargvalileleuileilegly
420425430
aspglyalaalaglnleuthrileglnglumetglysermetleuarg
435440445
aspglyglnalaprovalileleuleuleuasnasnaspglytyrthr
450455460
valgluargalailehisglyalaalaglnargtyrasnaspileala
465470475480
sertrpasntrpthrglnileproproalaleuasnalaalaglngln
485490495
alaglucystrpargvalthrglnalaileglnleualagluvalleu
500505510
gluargleualaargproglnargleuserpheilegluvalmetleu
515520525
prolysalaaspleuprogluleuleuargthrvalthrargalaleu
530535540
glualaargasnglygly
545550
- 还没有人留言评论。精彩留言会获得点赞!