一种与桃果实甜酸风味性状相关的SNP标记及其应用的制作方法
本发明涉及生物技术领域,属于桃分子育种和生物技术领域,尤其涉及一种与桃果实甜酸风味性状相关的SNP标记及其应用。
背景技术:
桃(Prunus persica)的果实富含蛋白质、糖类、膳食纤维、维生素等多种营养物质,深受消费者的喜爱。桃果实的甜酸风味是桃的重要经济性状之一,根据品质的不同主要分为酸和非酸的桃。桃起源于中国,因此我国在桃资源的种类及数量上远高于其他的国家,但现有经济价值较高的品种都为育成性品种,因此新品种的选育是提高我国桃品质及产量的重要影响因素。现在桃的育种主要是常规的杂交产生新品种,种植后3-4年才能开花结果,育种周期长、成本高。近年来开发了与甜酸风味性状相关的RAPD、AFLP、SSR等分子标记辅助育种,但是限于材料的数量、标记的密度、研究的手段等众多因素的限制,导致检测的准确率较低。因此为了有效降低错误率和提高检测效率,需要鉴定鉴别出甜酸相关标记或者基因,为桃果树的优良品种的选育提供技术支撑。
单核苷酸多态性(Single Nucleotide Polymorphism,SNP)是指基因组上单碱基的多态性,即一个位点有多种不同的碱基型,多态性丰富,是造成物种多样性的重要原因。SNP作为新一代最高密度的分子标记,可与个体的表型进行关联分析,是研究农艺性状的遗传机制的重要数据依据。目前对桃SNP分型的高通量方法主要有两种,高通量测序技术及基因芯片技术。
技术实现要素:
本发明的一个目的是提供检测待测桃树基因组的g.1495589位点的基因型的物质的用途。
本发明提供的检测待测桃树基因组的g.1495589位点的基因型的物质在鉴定或辅助鉴定待测桃树的果实是否为酸性表型中的应用;
所述g.1495589位点为5号染色体的第1495589位或序列1第601位核苷酸。
本发明的另一个目的是提供检测待测桃树基因组的g.1495589位点的基因型的物质。
本发明提供的检测待测桃树基因组的g.1495589位点的基因型的物质在制备鉴定或辅助鉴定待测桃树的果实是否为酸性表型产品中的应用;
所述g.1495589位点为5号染色体的第1495589位或序列1第601位核苷酸。
本发明第三个目的是提供一种鉴定或辅助鉴定待测桃树果实的酸性表型的方法。
本发明提供的方法,包括如下步骤:检测待测桃树基因组的g.1495589位点的基因型为TT、TC还是CC,
若所述待测桃树基因组的g.1495589位点的基因型为TT,则该待测桃树的果实为或候选为酸性表型;
若所述待测桃树基因组的g.1495589位点的基因型为TC或者CC,则该待测桃树的果实为或候选为非酸性表型;
所述g.1495589位点为5号染色体的第1495589位或序列1第601位核苷酸。
上述方法中,所述检测待测桃树基因组的g.1495589位点的基因型的方法为采用Axiom芯片平台上进行桃基因组170KSNP的基因型判定,具体为基因芯片检测,
所述基因芯片上固定有探针1和探针2;
所述探针1的核苷酸序列为序列2,且5’末端标记荧光蛋白A;
所述探针2的核苷酸序列为序列3,且5’末端标记荧光蛋白B;
所述荧光蛋白A和所述荧光蛋白B为不同荧光颜色。
上述荧光蛋白A具体为藻蓝蛋白(APC),发出蓝色荧光;
上述荧光蛋白B具体为藻红蛋白(PE),发出红色荧光。
本发明第四个目的是提供一种鉴定或辅助鉴定待测桃树果实是否为酸性表型产品。
本发明提供的产品,为上述的应用中的检测待测桃树基因组的g.1495589位点的基因型的物质。
上述的方法在鉴定或辅助鉴定待测桃树的果实是否为酸性表型中的应用也是本发明保护的范围。
上述的方法或上述的产品在培育果实酸性表型的桃树中的应用也是本发明保护的范围。
上述的方法或上述的产品在培育果实非酸性表型的桃树中的应用也是本发明保护的范围。
本发明第五个目的是提供一种筛选或培育果实酸性表型桃树的方法。
本发明提供的方法,包括如下步骤:筛选或培育g.1495589位点的基因型为TT的桃树。
本发明第六个目的是提供一种筛选或培育果实非酸性表型桃树的方法。
本发明提供的方法,包括如下步骤:筛选或培育g.1495589位点的基因型为TC或者CC的桃树。
本发明相对于现有技术具有如下的优点及效果:本发明基于现有的284份测序及芯片的桃SNP标记数据,开展了与表型性状的关联分析,成功在5号染色体上定位到一个与甜酸相关的SNP标记,相较于之前的RAPD、AFLP、SSR标记,可将预测的准确性提高到85%以上,使得预测的结果更加可靠。在种子时期进行个体甜酸性表型的预测,提高选种效率、节约育种成本。
附图说明
图1为与桃果实甜酸风味性状相关的SNP标记的获得过程。
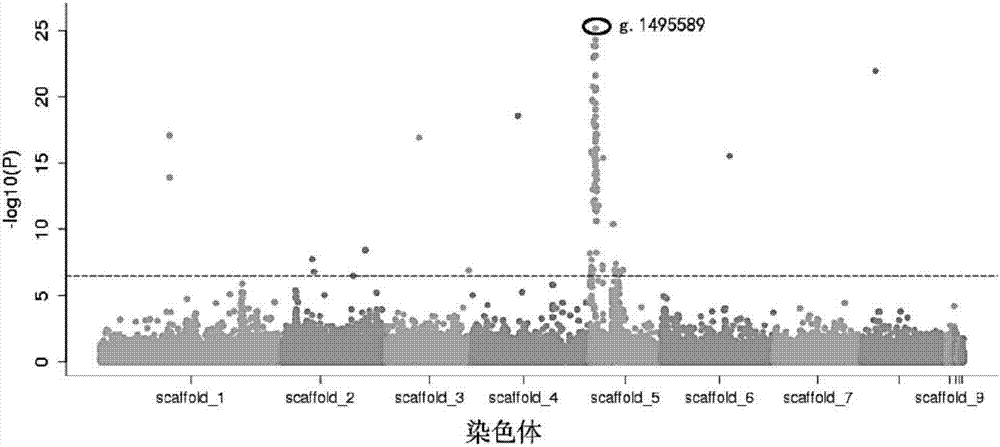
图2为桃果实甜酸风味性状的全基因组关联分析结果图;横坐标为不同染色体上的位置,纵坐标为-logPvalue。
图3为SNP标记位点g.1495589的基因分型结果;四方形表示基因型为TT,正三角表示基因型为CC。
具体实施方式
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
以下的实施例便于更好地理解本发明,但并不限定本发明。
实施例1、与桃果实甜酸风味性状相关的SNP标记的获得
图1为与桃果实甜酸风味性状相关的SNP标记的获得过程。
本发明所使用的实验材料来源于国家果树种质北京桃、草莓圃资源群体。
本实验共选取该资源群体中的192株桃树,根据果实表面酸覆盖情况进行表型的鉴定,其中91个酸桃(果实酸性表型)、101个非酸桃(果实非酸性表型)。
果实酸性表型为果实总酸含量大于等于0.4%,且果实pH值小于等于4;
果实非酸性表型为果实总酸含量小于0.4%,且果实pH值大于4。
上述果实含酸量测定方法如下:
1)取10~50g精确至0.001g,置于100ml烧杯中。用80℃热蒸馏水将烧杯中的内容物转移到250mL容量瓶中(总体积约150ml)。置于沸水浴中煮沸30min(摇动2~3次,使固体中的有机酸全部溶解于溶液中),取出,冷却至室温(约20℃),用快速滤纸过滤,收集滤液备测。
2)取25.00~50.00mL上述试液,使之含0.035~0.070g酸,置于250mL三角瓶中。加40~60mL水及0.2mL 1%酚酞指示剂,用0.1mol/L氢氧化钠标准滴定溶液(如样品酸度较低,可用0.01mol/L或0.05mol/L氢氧化钠标准滴定溶液)滴定至微红色30s不褪色。记录消耗0.1mol/L氢氧化钠标准滴定溶液的毫升数(V1)。
3)用水代替试液。以下按第2)条操作。记录消耗0.1mol/L氢氧化钠标准滴定溶液的毫升数(V2)。
总酸以每公斤(或每斤)样品中酸的克数表示,按式(1)计算:
X=c(V1-V2)×K×F×1000/m(V0)——————(1)
式中:X——每公斤(或每斤)样品中酸的克数,g/kg(或g/L);
c——氢氧化钠标准滴定溶液的浓度,mol/L;
V1——滴定试液时消耗氢氧化钠标准滴定溶液的体积,mL;
V2——空白试验时消耗氢氧化钠标准滴定溶液的体积,mL;
F——试液的稀释倍数;
m(V0)——试样的取样量,g或mL;
K——酸换算系数。
一、提取基因组DNA
采集192株桃树的幼叶组织,放置于锡箔纸中,-80℃冰箱内保存备用;用如下方法提取基因组DNA,得到192株桃树的基因组DNA。
1)新鲜叶片于2ml EP管中液氮研磨;
2)水浴65℃预热DNA提取液;
3)每管加入600μl提取液,10μlβ-巯基乙醇,2μl蛋白酶K,混匀;
4)65℃水浴加热,期间每10min温和混匀一次,40min后取出,室温冷却10min;
5)加入600μl酚:氯仿:异戊醇(25:24:1),剧烈震荡混匀,静置2-3min;
6)12000rpm,离心10min,上清液移入新管中;
7)重复5),6)步骤;
8)加入2/3体积的异丙醇,混匀,-20℃放置大于30min;
9)4℃12000rpm,离心20min;
10)加200μl TE重溶,加2μl RNase A,37℃水浴30min;
11)加200μl氯仿:异戊醇(24:1)混匀,12000rpm,离心10min;
12)上清移入新管中,加100μl 7.5M乙酸铵,混匀,加400μl无水乙醇,混匀,-20℃放置大于30min;
13)4℃12000rpm,离心20min;
14)70%乙醇洗涤3次,晾干;
15)加50μl TE重溶。
500ml提取液配方10g CTAB,50ml 1M Tris.HCl(pH8.0),20ml 0.5M EDTA,140ml 5M NaCl和290ml H2O组成。
二、与桃树表型相关的SNP位点的获得
1、SNP分型及位点过滤
上述192株桃树的基因组DNA在Affymetrix公司生产的Axiom芯片平台上进行桃基因组620KSNP的基因型判定,芯片信号的分析采用Affymetrix的SNPolisher package及Genotyping ConsoleTM Software(GTC)软件包聚类进行基因分型,根据聚类的效果筛选多态性的位点。其中四个样本的位点检出率低于97%,未计入后续的分析。剩余188份样本(89个酸桃、99个非酸桃)最终获得189,568个多态性的SNP位点。
2、不同样本的亲缘关系分析
基于188份样本(89个酸桃、99个非酸桃)的SNP位点运用GEMMA(genome-wide efficient mixed model association)软件进行不同个体之间的亲缘关系分析,得到亲缘关系系数矩阵。
3、全基因组关联分析
使用GEMMA软件的混合线性模型(LMM)进行188份样本(89个酸桃、99个非酸桃)基因型和表型的关联分析,将第2步中获得的亲缘关系矩阵作为协变量计入模型,以消除群体分层的影响。采用Boferroni法确定SNP与果实甜酸风味性状关联程度的阈值,计算方法为0.05除以有效的SNP位点数量,之后取负对数【-log(0.05/189568)】,即显著关联水平的阈值为6.58。
分析结果如图2所示,从图2中可知桃栽培品种果实表皮的甜酸性状与5号染色体的第1495589(染色体的位置)个核苷酸(即g.1495589位点)显著相关(P=25.17);g.1495589位点的基因型为TT、TC和CC三种形式。
统计表型和基因型的关系,结果如表1可知,g.1495589位点为TT基因型的桃为酸桃,果实为酸性表型;该位点具有TC或者CC基因型的桃为非酸桃,果实为非酸性表型。其中酸桃的预测准确度为95.51%,非酸桃的预测准确度为90.91%。
表1为SNP标记位点g.1495589(T/C)与桃果实甜酸风味的相关性
*阈值为6.58
因此,可以根据5号染色体的g.1495589位点的基因型为TT或TC/CC鉴定或辅助鉴定待测桃树的果实为酸性表型还是非酸性表型;
若待测桃树基因组的5号染色体的g.1495589位点的基因型为TT,则该待测桃树的果实为或候选为酸性表型;
若待测桃树基因组的5号染色体的g.1495589位点的基因型为TC或CC,则该待测桃树的果实为或候选为非酸性表型。
实施例2、SNP标记g.1495589在鉴定桃果实甜酸风味性状中的应用
一、提取待测样本的基因组DNA
采集已经鉴定果实酸性表型和非酸性表型的262个桃树幼叶组织,放置于锡箔纸中,提取每个桃树基因组DNA,与实施例1的一提取方法相同,得到各个样本的基因组DNA。
二、SNP标记位点基因分型
将上述每个桃树基因组DNA在GeneTitan平台的Axiom基因芯片上根据公司的标准流程进行桃基因组170K SNP(Affymetrix,美国昂飞)的基因型判定,芯片信号的分析采用Affymetrix的SNPolisher package及Genotyping ConsoleTM Software(GTC)软件包聚类进行基因分型,根据聚类的效果确定g.1495589的基因型,具体方法如下:
采用的探针为2条,序列分别如下:
C基因型的探针:
TGTTGATCTTGGCTGTTGGGGCAAAGGTTTCCTAGCAGTTCACCCCATACCTCTATGTGTACCCCTTCGCT(序列2),5’末端标记荧光蛋白藻蓝蛋白(APC),发出蓝色荧光;
T基因型的探针:
TGTTGATCTTGGCTGTTGGGGCAAAGGTTTCCTAGTAGTTCACCCCATACCTCTATGTGTACCCCTTCGCT(序列3),5’末端标记荧光蛋白藻红蛋白(PE),发出红色荧光;
荧光判读:根据扫描的SNP两个等位基因的荧光信号强度,采用Affymetrix分型算法Axiom GT1进行计算和判读,并给出相应的基因型。
根据g.1495589的基因型为TT或TC/CC判断待测桃树的果实为酸性表型还是非酸性表型;
若待测桃树基因组的5号染色体的g.1495589位点的基因型为TT,则该待测桃树的果实为或候选为酸性表型;
若待测桃树基因组的5号染色体的g.1495589位点的基因型为TC或CC,则该待测桃树的果实为或候选为非酸性表型。
检测结果见表2和图3,可以看出果实非酸性表型的桃树(非酸桃)的预测准确度为93.43%,果实酸性表型桃树(酸桃)的预测准确度为86.40%,证明本发明的方法正确。
表2SNP标记位点g.1495589(T/C)基因型与桃果实甜酸风味的性状分布
*预测准确性:Y表示正确,N表示错误。
序列表
<110>北京市农林科学院
<120>一种与桃果实甜酸风味性状相关的SNP标记及其应用
<160> 3
<170> PatentIn version 3.5
<210> 1
<211> 18
<212> DNA
<213>人工序列
<220>
<221>misc_feature
<222>601
<223>y=t或c
<400> 1
cgtacaactt atgattcatc tcaatgggtg tgtcagtagg tttacatcat agcattcctg 60
tttcagtgag tagatccatc acatacttcc tttgagacaa aaaataccag tttttgactt 120
tgctacttca attctgagga agtatttcag agtacataaa tccttcatct caaattcatg 180
agataaatac ttttgtagcg tatgtttctc tcatgtgtcg ttcccagtta cgaccatgtc 240
atccacataa acaattaatg cagtgatttt tcctccatca cgtatcaaga acagggtgtg 300
gtatgaatta ctttgagtgt atctaaaatt cttcatggac ttagtaaatc ttccaaacca 360
tggcatgaga gatttttttt taacccatat aatgactttc tgagcttgca aacttgattt 420
ttcttatcat aagctcaatt atatcctggt ggtaaatcca tgtataactc tttttctaag 480
tctccatgta agaagacatt tttaacatca aactgatgta gtggccaatc tagatttgct 540
gctacagaca atagaacttt gatagtgttg atcttggctg ttggggcaaa ggtttcctag 600
yagttcaccc catacctcta tgtgtacccc ttcgctacca atctggcttt atatatttca 660
atggatccat ctgctttgag tttcatagta taggtccacc tatatcccac tatcttcttt 720
tcgtgaggta aggggacgag ctcccatgtg gcattcttct ggagagctcg catttcttca 780
ttcttagctt ctttccattt tgagtcttct agtgtttcat tcacactatt tgggacagat 840
atatcagcca attgcttcac aaagtgtaca tgtaactttg ataatctgct tagagatata 900
taattattga tgagatattt tcctttgact ttaagatcta cttcatactg tactcgaggt 960
ttaccactgt tcttcctttc tggaagttgg tagatagagg tatctgtatc ttgtatgctt 1020
tctgaaatca aatcatcatg acatatttca taagtgttat tagtttcaag gagagatgtt 1080
acctgaatga catcttcagc aggcgattgg tatagttcta gagctgaaga cgggttcgaa 1140
gtgggcaaaa tctcagaaaa aaggatcaac tgtctcctcg tgattttgga aactggaagg 1200
c 1201
<210> 2
<211>71
<212> DNA
<213>人工序列
<220>
<223>
<400>2
tgttgatctt ggctgttggg gcaaaggttt cctagcagtt caccccatac ctctatgtgt 60
accccttcgc t 71
<210> 3
<211> 71
<212> DNA
<213>人工序列
<220>
<223>
<400> 3
tgttgatctt ggctgttggg gcaaaggttt cctagtagtt caccccatac ctctatgtgt 60
accccttcgc t 71
- 还没有人留言评论。精彩留言会获得点赞!