改良的用于生产丙酸的丙酸杆菌菌株的制作方法
本申请要求2015年9月30日提交的美国临时专利申请no.62/234,900的优先权,将其通过提述完整并入。
通过提述并入以电子方式提交的材料
通过提述将其完整并入的是与此同时提交的计算机可读核苷酸/氨基酸序列列表,其标识如下:建立于2016年9月26日的名称为“258544_seqlisting.txt”的一个76千字节的ascii文本(文本)文件”。
背景
丙酸(pa)在食品工业中广泛用作食品防腐剂。pa还用作合成聚合物的前体,包括但不限于聚丙烯和丙酸乙烯酯。另外,丙醇和其他有价值的化学品可以从pa衍生而来。pa传统上一直来源于化石燃料,直到最近,日益严重的环境问题已经使得终端用户的兴趣转向可持续性替代品。为了探索这种可持续替代品,细菌发酵重新成为了c3化学品生产的替代方案。
丙酸杆菌(propionibacterium)是多形杆状革兰氏阳性细菌,它们通过wood-werckman循环天然地产生pa作为主要发酵产物。天然状况下,pa与其他有机酸(乳酸、琥珀酸和乙酸)一起产生,导致生产力低,产率有限,从而导致下游工艺耗费巨大。直到最近为止,产酸丙酸杆菌的代谢工程一直是一项挑战,原因主要在于七个成簇规律间隔短回文重复序列(crispr)提供了针对接合质粒和噬菌体的抗性。如本文中公开的,可以使用基因组改组(gs)来提高产酸丙酸杆菌的生长速率和pa生产量。
gs最初在70年代中期作为原生质体融合被描述的,现已经广泛用于工业。实例包括在弗氏链霉菌(streptomycesfradiae)中产生泰乐菌素(zhang等人,nature,2002,415,644-646),在酿酒酵母中产生乙醇(l.hou,appl.biochem.biotechnol.,2010,160,1084-1093),在谢氏丙酸杆菌(p.shermanii)中产生维生素b12(zhang等人,j.biotechnol.,2010,148,139-143),在乳酸菌中产生乳酸(patnaik等人,nat.biotechnol.,2002,20,707-12),在二元醇梭菌(clostridiumdiolis)中产生1,3-丙二醇(otte等人,appl.environ.microbiol.,2009,75,7610-7616),以及在产酸丙酸杆菌中产生pa(guan等人,″genome-shufflingimprovesacidtolerance...″收录于advancesinchemistryresearch,vol15,chapter8(2012)novasciencepublishers,inc.,pp143-152)及其他。
为了获得具有较高丙酸产率和较低副产物的丙酸杆菌菌株,人们利用了基因组改组(gs)方案在两个丙酸杆菌菌株之间转移遗传物质,从而生成具有提高的丙酸生产潜力的新菌株。gs结合了通过dna交换促进的多亲本杂交的优点,其中供体提供少量的dna材料,而受体的其余部分保持完整。gs的最终结果是具有潜在的新途径和调控机制的、遗传上独特的菌株。
先前使用gs来增强pa生产的尝试未能产生具有期望表型的菌株。这部分地是由于在这样的程序中使用的已知菌株缺乏基因组多样性。因此,以前的努力未能产生能够利用葡萄糖或蔗糖发酵产生超过0.54g/g丙酸的丙酸杆菌菌株,而且保留了相当量的多种副产物(stowers等人,j.ind.microbiol.biotechnol.,2014,41,837-852)。
因此,使用gs来增强pa生产能够获得成功的前提是能够选择两种这样的菌株,它们具有期望表型,如果这些表型结合就会产生有利的丙酸生产菌株。申请人已经选择了一些丙酸杆菌菌株,经他们鉴定,这些菌株具有高的丙酸生产潜力。选出的这些菌株现在已被用于生产生长速率提高、丙酸产生增强(例如,超过0.54g/g)、和任选地不需要的副产物如乙酸和琥珀酸的产生减少的新丙酸杆菌菌株。
概述
根据一个实施方案,提供了用于丙酸的生物合成生产的组合物和方法。根据一个实施方案,提供了新的丙酸杆菌菌株,其相对于天然丙酸杆菌和其他已知的衍生菌株具有提高的利用葡萄糖或蔗糖作为碳源和能量源以增加丙酸产率的能力。在一个实施方案中,提供了新的具有增强的丙酸生产的丙酸杆菌菌株。在一个实施方案中,新的菌株通过在第一和第二丙酸杆菌菌株之间转移遗传物质产生,任选地通过基因组改组,其中所述第一和第二菌株具有不同的基因组。随后从基因组改组后生成的菌株中筛选相对于亲本菌株具有提高的利用葡萄糖/蔗糖作为碳源和能量源的能力、从而增加丙酸的产率的菌株。在一个实施方案中,用于基因组改组的丙酸杆菌菌株选自下列菌株:产酸丙酸杆菌atcc55737、产酸丙酸杆菌atcc4875、产酸丙酸杆菌atcc4965、中间型丙酸杆菌(p.intermedium)atcc14072和詹氏丙酸杆菌(p.jensenii)atcc9617。在一个实施方案中,新的具有增强的丙酸生产的丙酸杆菌菌株是产酸丙酸杆菌菌株。
根据一个实施方案,产酸丙酸杆菌菌株是通过在产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737之间交换遗传物质、任选地通过基因组改组交换遗传物质而产生的,其中,相对于亲本菌株产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737以及其他已知的产酸丙酸杆菌菌株,所得菌株的生长速率以及在新的菌株中丙酸的产生增加。在另一个实施方案中,产酸丙酸杆菌菌株是通过产酸丙酸杆菌atcc4875和产酸丙酸杆菌菌株f3e8之间交换遗传物质而产生的,所述菌株f3e8在2015年6月25日以atcc登录号pta-122267保藏于美国典型培养物保藏中心(atcc),其中,当在相同条件下培养时,新生成的菌株相对于通过产酸丙酸杆菌atcc55737具有增加的丙酸总产率(g/g)。
根据一个实施方案,提供了一种生产丙酸杆菌新菌株的方法。所述方法包括以下步骤:通过在两种不同的已知菌株的各种组合之间进行单独的基因组改组反应来产生丙酸杆菌菌株的文库;并且将从每个反应生成的所得菌株组合以形成相对于亲本菌株具有增加的基因组多样性的丙酸杆菌菌株的文库。
在一个实施方案中,使用下列菌株创建丙酸杆菌菌株的文库:产酸丙酸杆菌atcc4875、产酸丙酸杆菌atcc4965、中间型丙酸杆菌atcc14072和詹氏丙酸杆菌atcc9617。在一个实施方案中,为了获得不同菌株的文库,在三个单独的基因组改组反应中,使用产酸丙酸杆菌atcc4875与其他三个丙酸杆菌菌株进行基因组改组。在一个实施方案中,用每组菌株进行1、2或3轮基因组改组。将从三组基因组改组反应生成的菌株组合而产生丙酸杆菌菌株的文库。
然后可以使用该菌株文库进行第二轮基因组改组,第二轮基因组改组在菌株文库与选定的丙酸杆菌菌株之间进行,这些包括例如产酸丙酸杆菌atcc4875或产酸丙酸杆菌菌株f3e8(于2015年6月25日以atcc登录号pta-122267保藏于美国典型培养物保藏中心(atcc)),以产生第二组gs生成的菌株。然后测量在该第二组gs生成的菌株中产生的丙酸的量,以鉴定那些相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有提高的丙酸产率的新菌株。在一个实施方案中,在测量丙酸生产以鉴定相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有增加的生长速率的那些菌株的步骤之前,培养第二组gs生成的菌株。选择第二组gs生成的菌株中相对于亲本菌株具有增加的生长速率的成员,并且仅对选出的菌株加以分析以鉴定相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有提高的丙酸产率的新菌株。
根据一个实施方案,提供了分离的丙酸杆菌菌株,其中所述菌株具有大于0.18/小时、任选至少0.24/小时的最大生长速率,最大生长速率针对在32℃、ph6.5下pam培养基中培养的细胞的指数期计算。在一个实施方案中,提供了新的丙酸杆菌菌株,该菌株的最大生长速率是相同条件下生长的产酸丙酸杆菌atcc菌株4875的至少1.5倍,所述条件包括例如在32℃厌氧条件下,ph6.5的适于生长的培养基(包括例如pam培养基)中。在一个实施方案中,提供了具有约0.24/hr至约0.26/hr的生长速率的产酸丙酸杆菌菌株,所述生长速率针对细胞的指数期计算,所述细胞包括例如在32℃、ph6.5下在pam培养基中培养的细胞。
本公开的新丙酸杆菌菌株相对于产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737也具有提高的丙酸产率。在一个实施方案中,当在相同条件下(例如,在32℃厌氧条件下,ph6.5,在pam培养基中)培养时,本公开的新丙酸杆菌菌株的丙酸总产率(g/g)相对于产酸丙酸杆菌atcc菌株4875或atcc菌株55737具有至少10%,并且任选地10%至15%、15%至20%,或20%至25%或30%至40%的增加。在一个实施方案中,新丙酸杆菌菌株产生的丙酸产率为约0.49至约0.8g/g、约0.5至约0.7g/g、或约0.5至约0.66、或约0.66g/g。在一个实施方案中,新产酸丙酸杆菌菌株在最佳培养条件下产生0.77至约0.92g/l/hr或约0.84g/l/hr或约0.95g/l/hr的丙酸,所述最佳培养条件包括例如在32℃厌氧条件下、ph6.5的pam培养基中培养。在一个实施方案中,新产酸丙酸杆菌菌株在最佳培养条件下产生约0.8至约1.1g/l/hr或约0.95g/l/hr的丙酸,所述最佳培养条件包括例如在32℃厌氧条件下、ph6.5的pam培养基中培养。
在一个实施方案中,提供了分离的丙酸杆菌菌株,其中所述菌株比产酸丙酸杆菌菌株atcc55737产生更高的丙酸产率。更具体而言,相对于产酸丙酸杆菌菌株atcc55737,分离的菌株的进一步特征在于包含修饰基因,所述修饰基因选自abc极性氨基酸转运蛋白基因、细胞色素c生物发生基因或abc多重糖转运蛋白基因。在一个实施方案中,分离的丙酸杆菌菌株编码abc极性氨基酸转运蛋白和/或细胞色素c生物发生蛋白的经改变的基因产物。在一个实施方案中,分离的菌株包含abc极性氨基酸转运蛋白基因、细胞色素c生物发生基因和abc多重糖转运蛋白基因中每一者的修饰基因。
在一个实施方案中,提供了分离或纯化的丙酸杆菌菌株,其中所述菌株具有提高的生长速率(例如,大于约0.24/hr)和/或提高的丙酸产率(例如,约0.55g/g或更大),并且包含
(i)修饰的大核糖体rna基因(seqidno15),其在相对于产酸丙酸杆菌atcc55737中的相同基因的位置1441处、以及在相对于产酸丙酸杆菌atcc4875中的相同基因的位置1425、1270和1271处包含核苷酸取代;和/或
(ii)修饰的长链脂酰辅酶a合成酶基因(seqidno:16),其相对于产酸丙酸杆菌atcc55737中的相同基因在位置2269处包含缺失且在位置1500之后包含无义密码子,以及在相对于产酸丙酸杆菌atcc4875中相同基因的位置264、1650和2295处。编码蛋白质中的氨基酸变化位于参考产酸丙酸杆菌atcc55737的位置500和757处、以及相对于产酸丙酸杆菌atcc4875中的相同蛋白质的位置88和757处(参见seqidno:16;编码的截短肽提供为seqidno:17);和/或
(iii)修饰的阳离子扩散协助蛋白基因(seqidno:13),其相对于产酸丙酸杆菌atcc55737中相同的基因在位置953处、以及在相对于产酸丙酸杆菌atcc4875中的相同基因在位置18、19、56、211、334、340、406、451、484、574和953处包含核苷酸置换。编码蛋白质(seqidno:14)的氨基酸变化包括在相对于产酸丙酸杆菌atcc55737中相同蛋白质的位置318处、以及在相对于产酸丙酸杆菌atcc4875中相同蛋白质的位置7、60、71、86、112、114、136、151、162、192和318处的氨基酸置换。
(iv)整个核糖体rna基因(seqidno:18)相对于两个亲本菌株(产酸丙酸杆菌atcc55737和产酸丙酸杆菌atcc4875)的额外拷贝;和/或
(v)精氨酸脱亚胺酶调节子(seqidno:21)相对于两个亲本菌株(产酸丙酸杆菌atcc55737和产酸丙酸杆菌atcc4875)的额外拷贝。在一个实施方案中,所述额外拷贝相对于两个亲本菌株(产酸丙酸杆菌atcc55737和产酸丙酸杆菌atcc4875)在位置37处包含突变。
根据一个实施方案,提供了新菌株产酸丙酸杆菌f3e8,其于2015年6月25日保藏于美国典型培养物保藏中心(atcc),10801universityboulevard,manassas,va,20110-2209usa,atcc登录号为pta-122267。本公开还涵盖所述保藏菌株的变体,其中所述变体已经过进一步的遗传操作。这样的变体包括菌株f3e8的衍生物,其通过将f3e8菌株与不同的产酸丙酸杆菌菌株进行基因组改组而生成。根据一个实施方案,提供了新菌株产酸丙酸杆菌wgs7,其于2016年9月2日保藏于美国典型培养物保藏中心(atcc),10801universityboulevard,manassas,va,20110-2209usa,atcc登录号为_____。产酸丙酸杆菌f3e8是使用葡萄糖作为碳源和能源生成的,而wgs7是使用蔗糖作为碳源和能源生成的。
根据一个实施方案,提供了生产具有提高的丙酸生产产率的丙酸杆菌菌株的方法,其中所述方法包括将选自产酸丙酸杆菌f3e8、产酸丙酸杆菌wgs7、产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737的菌株与选自产酸丙酸杆菌f3e8、产酸丙酸杆菌wgs7、产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737的不同菌株进行基因组改组,并且回收所得到的相对于亲本菌株具有增加的生长速率或提高的丙酸产率的修饰菌株。在一个实施方案中,提供了生产具有提高的丙酸生产产率的新产酸丙酸杆菌菌株的方法,其中所述方法包括将第一和第二产酸丙酸杆菌菌株进行基因组改组。更具体地说,在一个实施方案中,第一菌株选自产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737,第二菌株是产酸丙酸杆菌菌株f3e8,所述菌株f3e8于2015年6月25日以atcc登录号pta-122267保藏于美国典型培养物保藏中心(atcc)。然后培养通过将第一和第二产酸丙酸杆菌菌株进行基因组改组而产生的新菌株,以鉴定相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有增加的生长速率或丙酸生产量的菌株。在一个实施方案中,所述方法包括以下步骤:首先选择所得到的相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有增加的生长速率的新菌株;然后测量选定菌株中的丙酸生产量,以鉴定相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有提高的丙酸产率的新菌株。
在一个实施方案中,修饰的菌株在细胞组分和/或编码细胞组分的基因中具有一个、二个、三个、四个或五个修饰,其中在一个实施方案中的细胞组分选自abc极性氨基酸转运蛋白、细胞色素c生物发生蛋白、abc多重糖转运蛋白、核糖体rna的大亚基、长链脂酰辅酶a合成酶、阳离子扩散协助蛋白、整个核糖体rna操纵子、精氨酸脱亚胺酶调节子。在一个实施方案中,修饰的菌株在细胞组分和/或编码细胞组分的基因中具有一个、两个或三个修饰,其中在一个实施方案中的细胞组分选自核糖体rna的大亚基、长链脂酰辅酶a合成酶和阳离子扩散协助蛋白。
在一个实施方案中,提供了一种生产丙酸的方法。在一个实施方案中,所述方法包括在适于产酸丙酸杆菌菌株生长的条件下培养所述菌株并回收由所述菌株产生的丙酸的步骤。在一个实施方案中,提供了具有至少0.24/hr最大生长速率的产酸丙酸杆菌菌株,所述最大生长速率是针对在32℃、ph6.5下在pam培养基中培养的细胞的指数期计算的。在一个另外的实施方案中,丙酸杆菌菌株产生约0.49至约0.8g/g、约7.0至约8.0g/g、约0.5至约0.7g/g、或约0.5至约0.66、或约0.66g/g的丙酸产率。在一个实施方案中,用于生产乳酸的产酸丙酸杆菌菌株是菌株f3e8,其于2015年6月25日保藏于美国典型培养物保藏中心(atcc),10801universityboulevard,manassas,va,20110-2209usa,指定atcc登录号为pta-122267。本公开还涵盖通过在产酸丙酸杆菌atcc4875与产酸丙酸杆菌atcc55737之间转移遗传物质而产生的新产酸丙酸杆菌菌株,包括产酸丙酸杆菌f3e8与产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737的基因组改组过程中、和/或产酸丙酸杆菌菌株f3e8与不同的产酸丙酸杆菌菌株的基因组改组的过程中所产生的菌株。在一个实施方案中,用于生产乳酸的产酸丙酸杆菌菌株是菌株wgs7,其于2016年9月2日保藏于美国典型培养物保藏中心(atcc),10801universityboulevard,manassas,va,20110-2209usa,atcc登录号为____。
附图简述
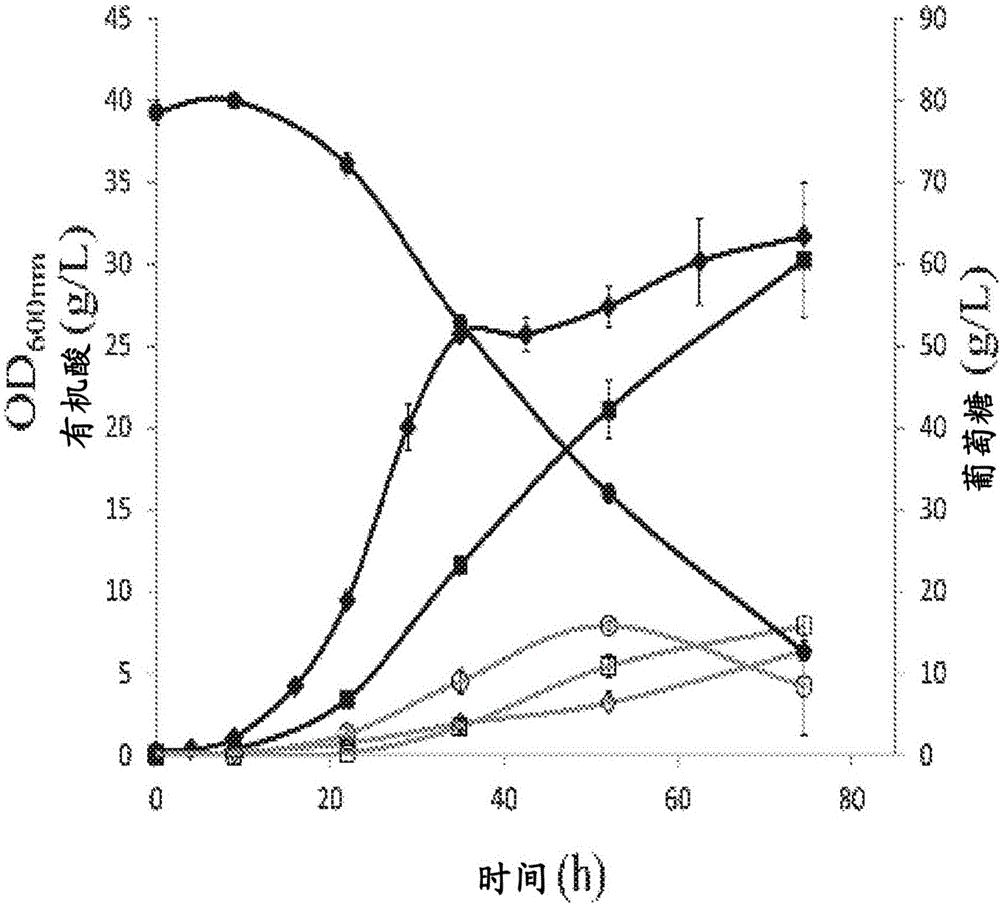
图1a-1c是显示产酸丙酸杆菌atcc4875(图1a)、产酸丙酸杆菌atcc55737(图1b)和产酸丙酸杆菌f3e8菌株(图1c)在2l生物反应器中的发酵特性的曲线图。绘图显示了两个生物学重复的平均值。测量在600nm处的光密度:黑线和◆;丙酸:黑线和■;葡萄糖:黑线和●;琥珀酸:灰线和□;乙酸:灰线和◇;丙酮酸:灰线和○。
图2是显示产酸丙酸杆菌f3e8在血清瓶(三个生物学重复)中的发酵生长特性的曲线图。
图3a和3b是显示不同ph(○:ph6.5.□:ph5.5.δ:ph4.5)下pa抑制动力学的曲线图。图3a是产酸丙酸杆菌atcc4875抑制的曲线图,而图3b提供了产酸丙酸杆菌f3e8抑制的数据。
图4a-4d显示了在2l生物反应器中的发酵特性及其外部代谢物特异性速率(metabolitespecificrate)。图4a:产酸丙酸杆菌atcc55737,图4b:从gs获得的产酸丙酸杆菌wgs7菌株。光密度:实心菱形;丙酸:实心方形;葡萄糖:实心圆形;琥珀酸:空心方形;乙酸:空心菱形;丙酮酸:空心圆形。图4c是显示蔗糖的比消耗速率(qs)、pa的比生产速率(qp)和比生长速率(μ)的条形图。图4d是显示在pam培养基中呈现的游离氨基酸的比消耗速率的条形图。阴影条:产酸丙酸杆菌atcc55737;空心条:产酸丙酸杆菌wgs7。数据代表每个菌株的两个生物学重复的平均值。比速率是在指数中期(15至25小时)计算的。
图5显示了在产酸丙酸杆菌atcc55737(阴影条)和产酸丙酸杆菌wgs7(空心条)中外源添加50mm乳酸盐(lac)、10mm延胡索酸盐(fum)、10mm精氨酸(arg)、10mm赖氨酸(lys)或10mm脯氨酸(pro)的效果。通过含有cdm的培养基的重复血清瓶发酵进行发酵。
发明详述
定义
在描述和要求本发明时,将根据下文陈述的定义使用下列术语。
如本文中使用的术语“大约”意指比陈述的值或值的范围大或小10%,但不意图针对仅仅这个更宽的定义指定任何值或值的范围。在每个值或值的范围之前的术语“大约”同样意图涵盖所陈述的绝对值或值的范围的实施方案。
如本文中使用的,术语“纯化的”等术语定义了细菌或化合物的分离,所述细菌或化合物处于基本上不含通常与细菌相关的污染物、或天然或自然环境中的化合物的形式。如本文中使用的,术语“纯化的”并不要求绝对纯度,而是意图为相对定义。术语“纯化的细菌”在本文中用于描述已经从其他细菌分离并且以同源群体存在的细菌群体。
术语“分离的”要求将参考材料从其原始环境(例如,如果它是天然存在的,则为天然环境)中移出。
如本文中使用的,“(经)改变的基因产物”定义了由修饰的基因编码的rna或氨基酸序列,其中所述rna或氨基酸序列相对于由野生型基因/参照基因产生的相应基因产物具有不同的一级序列。
如本文中使用的,“拷贝数变异”指某一特定基因的数目相对于野生型菌株中的相同基因的数目不同。拷贝数变异可以是编码序列的重复或缺失,所述编码序列编码rna或氨基酸序列。额外的拷贝相对于菌株的序列可以是完全相同的,也可以是有修饰的。
如本文中使用的,“氨基酸修饰”涵盖(i)将氨基酸用不同的氨基酸取代,(ii)添加/插入氨基酸,或(iii)缺失一个或多个氨基酸。
如本文中使用的,氨基酸“取代”是指一个氨基酸残基被不同的氨基酸残基置换。
如本文中使用的,术语“保守氨基酸取代”在本文中定义为在以下五组之一中的交换:
i.小的脂族、非极性或微极性残基:
ala、ser、thr、pro、gly;
ii.极性、带负电荷的残基及其酰胺:
asp、asn、glu、gln;
iii.极性、带正电荷的残基:
his、arg、lys;
iv.大的脂族、非极性残基:
met、leu、ile、val、cys
v.大的芳香族残基:
phe、tyr、trp。
“非保守氨基酸取代”定义为第一个氨基酸与第二个氨基酸的交换,其中所述第二个氨基酸选自所述第一个氨基酸所在的组之外的上述五个组之一。
“启动子”为指导多核苷酸转录的dna序列。通常,启动子位于待转录的多核苷酸的5′区域中,靠近这个多核苷酸的转录起始位点。
如本文中使用的“基因”描述了这样的核酸分子,其包含编码基因产物(即,rna转录物和/或多肽)的开放阅读框以及控制基因产物表达的相关调控区和非编码转录区(例如,内含子以及5′和3′非翻译区)。
如本文中使用的“修饰的基因”是相对于参考菌株(例如,产酸丙酸杆菌atcc菌株4875或55737)中发现的相应基因已经以某种方式被改变的基因。改变可以包括整个基因的重复或缺失、基因的多个部分的重复或缺失、或基因序列中核苷酸的插入或取代。
如本文中使用的术语“同一性”定义了在两个或更多个序列之间的相似性。通过将相同残基的数目除以残基总数并乘以100而得到的百分比来量度同一性。因此,完全相同序列的两个拷贝具有100%的同一性,而具有相对于彼此的氨基酸缺失、添加或取代的两个序列具有较低的同一性程度。本领域技术人员将认识到,若干计算机程序,比如采用诸如blast(基本局部比对搜索工具,altschul等人,(1993)j.mol.biol.215:403-410)的算法的那些计算机程序可以用于确定序列同一性。
“培养基”是指可用于支持在本发明方法中使用的微生物的生长的任何液体、半固体或固体培养基。在一些实施方案中,微生物是细菌,例如产酸丙酸杆菌。用于培养微生物的培养基是熟知的,参见,例如,sambrook等人以及currentprotocolsinmolecularbiology,f.m.ausubel等人编辑,currentprotocols,ajointventurebetweengreenepublishingassociates,inc.andjohnwiley&sons,inc.,(1998年增刊)(ausubel)。
如本文中使用的术语“基因组改组”或“gs”是在两个细菌菌株之间转移遗传物质以创建新的包含来自两个初始菌株的元件的重组菌株的过程。
如本文中使用的缺乏任何进一步限定的术语“生长速率”是指基于指数期中的细胞密度变化计算的最大比生长速率(μmax),正如通过基于在600nm处的光吸收的光密度变化所测量。使用公式n=n0eμt以每时间的单位数或1/时间来表示生长速率,其中“n”是在od600测量的细胞密度,“n0”是原始或先前的细胞密度,“t”是时间,“e”是常数(欧拉数,e=2.71828),μ是生长速率。
如本文中使用的,术语“产率”定义为所产生的感兴趣的产物的量除以所消耗的总底物的量(g/g)。
如本文中使用的,术语“丙酸的生产速率”表示每小时每升产生的丙酸的总克数。
如本文中使用的,术语“效价”表示每升发酵液所产生的产物的总克数。
术语“发酵”是指通过将一种底物转化为另一种底物的生物进行的代谢过程,在此过程中细胞能够获得细胞能量,例如当生物利用葡萄糖并将其转化为丙酸时。
实施方案
根据一个实施方案,提供了用于丙酸的生物合成生产的组合物和方法。根据一个实施方案,提供了新的丙酸杆菌菌株,其相对于天然丙酸杆菌和其他已知的衍生菌株具有提高的利用葡萄糖作为碳源和能量源以增加丙酸产率的能力。根据一个实施方案,新的丙酸杆菌菌株是产酸丙酸杆菌菌株。
具有所需的提高的生长速率和/或丙酸产率的新菌株可以任何适合的丙酸杆菌菌株之间的基因组改组来产生。在一个实施方案中,通过在产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737之间交换遗传物质,任选地通过基因组改组,来产生具有增加的丙酸产率的菌株。然后可以将得到的菌株(包括例如本文中公开的产酸丙酸杆菌菌株f3e8、f3g8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1、f3b9、wgs.1、wgs.2、wgs.3、wgs.4、wgs.5、wgs.6、wgs7、wgs.8、wgs.9、wgs.10、wgs.11、wgs.12和wgs.13)与不同的产酸丙酸杆菌菌株进行进一步的基因组改组,来进一步操作得到的菌株。根据一个实施方案,通过在产酸丙酸杆菌菌株f3e8与另一种不同的产酸丙酸杆菌菌株之间交换遗传物质来产生新的产酸丙酸杆菌菌株。在一个实施方案中,新的产酸丙酸杆菌菌株通过第一菌株与第二菌株交换遗传物质(任选地通过基因组改组)而产生,所述第一菌株选自产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737,其中所述第二菌株选自2015年6月25日保藏于美国典型培养物保藏中心(atcc)的atcc登录号为pta-122267的产酸丙酸杆菌菌株f3e8或2016年9月2日保藏于美国典型培养物保藏中心(atcc)的atcc登录号为____的产酸丙酸杆菌菌株wgs7,其中,当在相同条件下培养时,新菌株相对于产酸丙酸杆菌atcc55737具有增加的丙酸总产率(g/g)。在一个实施方案中,通过在产酸丙酸杆菌菌株f3e8和产酸丙酸杆菌菌株wgs7之间交换遗传物质(任选地通过基因组改组)来产生新的产酸丙酸杆菌菌株。
在一个实施方案中,得到的菌株将具有至少0.24/hr的最大生长速率和/或约0.55g/g或更高的丙酸产率,其中最大生长速率是针对在最佳条件下培养的细胞的指数生长期计算的,所述最佳条件包括例如在32℃厌氧条件下,ph6.5,在pam培养基中。在一个实施方案中,新菌株将以约0.49至约0.8g/g、约0.5至约0.7g/g、或约0.5至约0.66、或约0.66g/g的产率产生丙酸。
根据一个实施方案,通过在两个亲本产酸丙酸杆菌菌株之间进行基因组改组来生成新菌株。简言之,单独培养两个亲本菌株,然后通过离心从培养物收集亲本菌株,并进行酶处理(溶菌酶)以生成原生质体。在原生质体形成缓冲液中,将来自每个群体的相等数量的原生质体混合在一起。将聚乙二醇和cacl2加入到悬浮液中以诱导原生质体的融合,随后将融合的原生质体离心,洗涤并重悬在再生培养基中。然后将细胞接种在生长培养基平板上,选择显示出至少0.19/hr的生长速率的细胞用于进一步分析。
根据一个实施方案,提供了产生相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有增加的生长速率或丙酸生产量的新丙酸杆菌菌株的方法。该方法包括在第一组和第二组丙酸杆菌菌株之间交换遗传物质(任选地通过基因组改组)的步骤。更具体而言,第一组丙酸杆菌菌株选自产酸丙酸杆菌atcc4875;产酸丙酸杆菌atcc55737;或自产酸丙酸杆菌atcc4875与产酸丙酸杆菌atcc55737、产酸丙酸杆菌atcc4965、中间型丙酸杆菌atcc14072或詹氏丙酸杆菌atcc9617之间的基因组改组生成的菌株。第二组丙酸杆菌菌株是产酸丙酸杆菌菌株f3e8,其于2015年6月25日以atcc登录号pta-122267保藏于美国典型培养物保藏中心(atcc)。然后培养通过第一组和第二组丙酸杆菌菌株之间的基因组改组而生成的菌株,以鉴定相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有增加的生长速率或丙酸生产量的新丙酸杆菌菌株。在一个实施方案中,首先培养通过第一组和第二组丙酸杆菌菌株之间的基因组改组而生成的菌株,以鉴定相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737显示出增加的生长速率的菌株。然后选择表现出相对增加的生长速率的菌株,然后各别培养选出的菌株,以鉴定哪些选择的菌株相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有提高的丙酸产率。根据一个实施方案,第一组菌株通过产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737之间的基因组改组而生成。在一个实施方案中,第一组菌株通过产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc14072之间的基因组改组而生成。在一个实施方案中,第一组菌株通过产酸丙酸杆菌atcc4875和詹氏丙酸杆菌atcc9617之间的基因组改组而生成。在一个实施方案中,第一组菌株代表通过单独的基因组改组反应而生成的两个或更多个菌株的组合所产生的菌株文库。在一个实施方案中,在公开的生产相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有增加的生长速率或丙酸生产量的新丙酸杆菌菌株的方法中,第一组菌株包括来自2个、3个或4个以下单独的基因组改组反应的菌株的混合物:
i)产酸丙酸杆菌atcc4875与产酸丙酸杆菌atcc55737之间的基因组改组;
ii)产酸丙酸杆菌atcc4875与产酸丙酸杆菌atcc4965之间的基因组改组;
iii)产酸丙酸杆菌atcc4875与中间型丙酸杆菌atcc14072之间的基因组改组;以及
iv)产酸丙酸杆菌atcc4875与詹氏丙酸杆菌atcc9617之间的基因组改组。在一个实施方案中,第一组菌株代表通过i)和ii);或ii)和iii);或iii)和iv);或ii)和iv)或ii)、iii)和iv);或i)、ii)、iii)和iv)而生成的菌株的组合。
根据一个实施方案,提供了生产具有提高的丙酸生产产率的新产酸丙酸杆菌菌株的方法,所述方法包括以下步骤:
将第一和第二产酸丙酸杆菌菌株进行基因组改组以产生新的菌株,其中所述第一菌株选自产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737,所述第二菌株是产酸丙酸杆菌菌株f3e8,所述菌株f3e8于2015年6月25日以atcc登录号pta-122267保藏于美国典型培养物保藏中心(atcc)。
任选地培养所述新菌株,以鉴定相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有增加的生长速率的菌株,并选择得到的具有增加的生长速率的新菌株;和
测量在所述新菌株中的丙酸生产量,以鉴定相对于产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737具有提高的丙酸产率的菌株。
本公开还涵盖根据本文中公开的方法产生的任何新产酸丙酸杆菌菌株。
根据一个实施方案,提供了分离或纯化的新丙酸杆菌菌株,其中所述新菌株相对于产酸丙酸杆菌菌株f3e8、产酸丙酸杆菌atcc4875、产酸丙酸杆菌atcc4875和/或产酸丙酸杆菌atcc55737表现出更快的生长速率和/或增加的丙酸产率。在一个实施方案中,当在相同条件下生长时,本公开的新菌株相对于产酸丙酸杆菌atcc菌株4875具有至少1.5倍的最大生长速率和/或增加15%至20%的总产率(g/g),所述相同条件包括例如在32℃厌氧条件下,ph6.5,在pam培养基中。在一个实施方案中,本公开的丙酸杆菌菌株具有约0.24/hr至约0.26/hr的生长速率,生长速率是针对在32℃、ph6.5下在pam培养基中培养的细胞的指数期计算的。在一个实施方案中,丙酸杆菌菌株是产酸丙酸杆菌菌株。
在一个实施方案中,当在相同条件下(例如,在32℃厌氧条件下,ph6.5,在pam培养基中)培养时,本公开的丙酸杆菌菌株的丙酸产率(g/g)相对于产酸丙酸杆菌atcc菌株4875具有至少10%,任选地10%至15%、15%至20%、或20%至25%的增加。在一个实施方案中,新丙酸杆菌菌株以约0.49至约0.7g/g、约0.5至约0.7g/g、或约0.5至约0.66、或约0.66g/g产率产生丙酸。在一个实施方案中,新产酸丙酸杆菌菌株在最佳培养条件下产生至少约0.77至约0.92g/l/hr或约0.84g/l/hr的丙酸,所述最佳培养条件包括例如在32℃厌氧条件下,ph6.5,在pam培养基中。在一个实施方案中,当在相同条件下(例如,在32℃厌氧条件下,ph6.5,在pam培养基中)培养时,本公开的丙酸杆菌菌株的丙酸产率(g/g)相对于产酸丙酸杆菌菌株atcc55737具有至少10%,并且任选地10%至15%、15%至20%、或20%至25%的增加。
在一个实施方案中,产酸丙酸杆菌菌株是分离或纯化的产酸丙酸杆菌菌株,其中当在相同条件下培养时,所述菌株的丙酸产率(g/g)相对于产酸丙酸杆菌atcc菌株4875具有10%至15%的增加,并且任选地,相对于产酸丙酸杆菌atcc菌株4875具有至少1.5倍的最大生长速率,所述相同条件任选地为在32℃厌氧条件下,ph6.5,在诸如pam的可接受的培养基中。在一个实施方案中,产酸丙酸杆菌菌株是分离或纯化的产酸丙酸杆菌菌株,其中所述菌株具有约0.49至约0.7g/g、约0.6至约0.8g/g、约0.5至约0.7g/g、或约0.5至约0.66、或约0.66g/g、或约0.75g/g的丙酸产率,并且任选地,当在相同条件下培养时,所述菌株相对于产酸丙酸杆菌atcc菌株具有至少1.5倍的最大生长速率,所述相同条件任选地为在32℃厌氧条件下,ph6.5,在诸如pam的可接受的培养基中。在一个实施方案中,提供了新的产酸丙酸杆菌菌株,其中所述菌株是分离或纯化的产酸丙酸杆菌菌株,当在相同条件下生长时,所述菌株具有比产酸丙酸杆菌菌株atcc55737高20%、30%或40%的丙酸产率,所述相同条件任选地为在32℃厌氧条件下,ph6.5,在诸如pam的可接受的培养基中。在一个实施方案中,产酸丙酸杆菌菌株是分离或纯化的产酸丙酸杆菌菌株,其中所述菌株具有约0.49至约0.8g/g、约0.5至约0.7g/g、或约0.5至约0.66、或约0.55或0.66g/g的丙酸产率,并且任选地具有约0.24/hr至约0.26/hr的最大生长速率,所述最大生长速率是在32℃、ph6.5下在pam培养基中培养的细胞的指数期计算的。在一个实施方案中,培养的产酸丙酸杆菌菌株是分离的或纯化的产酸丙酸杆菌菌株,其中所述菌株产生约0.84g/l/hr至约0.95g/l/hr的丙酸,并且任选地具有至少为0.24/hrod600的最大生长速率,所述最大生长速率是在32℃、ph6.5下在可接受的培养基(如pam)中培养的细胞的指数期计算的。在一个实施方案中,培养的产酸丙酸杆菌菌株是分离或纯化的产酸丙酸杆菌菌株,其中所述菌株在最佳培养条件下产生至少约0.77至约0.95g/g或约0.84g/g的丙酸,并且任选地具有约0.24/hr至约0.26/hr的生长速率,所述生长速率是针对在32℃、ph6.5下在pam培养基中培养的细胞的指数期计算的。
根据一个实施方案,任何上述丙酸杆菌菌株相对于产酸丙酸杆菌atcc4875和atcc55737的进一步特征在于包含一个或多个根据表1和表7的修饰基因。在一个实施方案中,相对于菌株产酸丙酸杆菌atcc4875和atcc55737,所述丙酸杆菌菌株包含编码大亚基核糖体、长链脂酰coa合成酶或阳离子扩散协助蛋白的修饰基因(表1)。在一个实施方案中,相对于产酸丙酸杆菌菌株atcc55737,所述丙酸杆菌菌株包含修饰基因,所述修饰基因选自abc极性氨基酸转运蛋白基因、细胞色素c生物发生基因和abc多重糖转运蛋白基因(表7)。在一个实施方案中,相对于在产酸丙酸杆菌菌株atcc55737或atcc4875中发现的相应基因,所述修饰基因包含1、2、3、4、5、6、7、8、9或10个修饰,其中每个修饰代表单核苷酸插入、缺失或取代。在一个实施方案中,提供了相对于产酸丙酸杆菌atcc55737或atcc4875包含修饰基因的丙酸杆菌菌株,所述修饰基因各自针对大亚基核糖体、长链脂酰coa合成酶和阳离子扩散协助蛋白。
根据一个实施方案,提供了分离的丙酸杆菌菌株,任选地为产酸丙酸杆菌菌株,其中所述菌株具有至少0.24/hr的最大生长速率,所述最大生长速率是针对在32℃厌氧条件下ph为6.5在pam培养基中培养的细胞的指数期计算的,并且所述菌株包含相对于菌株产酸丙酸杆菌atcc55737或atcc4875的修饰基因,所述修饰基因编码针对abc极性氨基酸转运蛋白、细胞色素c生物发生蛋白、大亚基核糖体rna、长链脂酰coa合成酶或阳离子扩散协助蛋白的改变的基因产物。
根据一个实施方案,提供了分离的丙酸杆菌菌株,任选地为产酸丙酸杆菌菌株,其相对于产酸丙酸杆菌atcc菌株4875和atcc55737具有提高的丙酸产率,其中所述菌株包含相对于产酸丙酸杆菌菌株atcc4875和atcc55737的修饰基因,所述修饰基因针对abc极性氨基酸转运蛋白基因、细胞色素c生物发生基因、abc多重糖转运蛋白基因、大亚基核糖体、长链脂酰coa合成酶或阳离子扩散协助蛋白。在一个实施方案中,当在相同条件下培养时,所述包含修饰的大亚基核糖体、长链脂酰coa合成酶或阳离子扩散协助蛋白的丙酸杆菌菌株相对于产酸丙酸杆菌菌株atcc4875具有至少15%至20%的丙酸总产率(g/g)增加,并且在一个实施方案中,其产生至少0.55或0.66g/g的丙酸产率。在一个实施方案中,所述菌株包含修饰的大亚基核糖体、长链脂酰coa合成酶和阳离子扩散协助蛋白基因。在一个实施方案中,所述修饰基因编码经改变的基因产物。
根据本公开的一个实施方案,初步选择出了产生增加的丙酸产率的分离的丙酸杆菌菌株。在分出10个选定菌株之后,有3个变体基因在所有10个选定菌株中是保守的(表1)。变异存在于:
i)阳离子扩散协助蛋白中,
ii)大亚基核糖体rna(23s)中,
iii)长链脂酰coa合成酶中。
变体的位置在表1中详细说明。
表1:在10个新菌株中发现的保守基因变体的位置参考产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737。所有10个菌株在其基因组中都具有保守突变。
*新菌株:10个菌株通过基因组改组产生
atcc4875:产酸丙酸杆菌atcc4875
atcc55737:产酸丙酸杆菌atcc55737
snp:单核苷酸多态性
indel:插入或缺失
x:表示无法定义氨基酸
在一个实施方案中,提供了分离的丙酸杆菌菌株,任选地为产酸丙酸杆菌菌株,其包含编码大亚基核糖体rna(seqidno:15)的修饰的大亚基核糖体基因,其中,相对于产酸丙酸杆菌atcc55737或atcc4875中的相同基因,所述基因在其一级序列中被改变。在一个实施方案中,目前公开的丙酸杆菌菌株的大亚基核糖体基因通过1、2或3个核苷酸取代而被改变。根据一个实施方案,本发明的丙酸杆菌菌株的大亚基核糖体(23s)基因与产酸丙酸杆菌atcc4875的大亚基核糖体基因由于在位置1270、1271和1425处的1、2或3个核苷酸取代而不同,并且更具体地在一个实施方案中,所述核苷酸取代是g1270a、t1271c和g1425t。在一个实施方案中,所述大亚基核糖体(23s)基因包含相对于产酸丙酸杆菌atcc55737中的相同基因具有99%序列同一性并在位置1425处具有取代的序列,并且更具体地,在一个实施方案中,核苷酸取代是a1441g。
在一个实施方案中,提供了分离的丙酸杆菌菌株,任选地为产酸丙酸杆菌菌株,其中所述菌株包含相对于产酸丙酸杆菌atcc55737或atcc4875的修饰的长链脂酰coa合成酶基因。在一个实施方案中,对长链脂酰coa合成酶基因的修饰阻止了功能性长链脂酰coa合成酶的表达。在一个实施方案中,所述基因包含引起移码突变的核苷酸插入或缺失。在一个实施方案中,所述插入或缺失将终止密码子引入转录的mrna的阅读框中,导致产生截短的肽。在一个实施方案中,编码长链脂酰coa合成酶的基因包含在核苷酸位置1500之后的无义密码子以及在相对于产酸丙酸杆菌atcc55737中相同基因的位置2269处以及在相对于产酸丙酸杆菌菌株atcc4875的位置264、1271和2295处的缺失或插入。在一个实施方案中,编码的长链脂酰coa合成酶在天然蛋白质的氨基酸位置500附近和相对于产酸丙酸杆菌atcc55737的位置757处被截短,并且具有相对于丙酸杆菌atcc4875中的相同氨基酸序列在位置88处的氨基酸取代和在位置757处的无义氨基酸取代。在一个实施方案中,修饰的长链脂酰coa合成酶基因包含seqidno:16的序列或编码截短肽seqidno:17的序列。
在一个实施方案中,提供了分离的丙酸杆菌菌株,任选地为产酸丙酸杆菌菌株,其中所述菌株包含修饰的阳离子扩散协助蛋白基因。根据一个实施方案,本发明的丙酸杆菌菌株的阳离子扩散协助蛋白基因(seqidno:13)与产酸丙酸杆菌atcc4875中的阳离子扩散协助蛋白基因的区别在于1、2、3、4、5、6、7、8、9、10、11个或更多个在位置19、178、256、211、334、340、406、451、484、574和953处的核苷酸取代。具体的取代是g19a、t178g、g211a、a256g、g334a、c340g、a406c、c451a、t484c、a574g和g953a。在一个实施方案中,修饰的阳离子扩散协助蛋白基因包含相对于产酸丙酸杆菌atcc55737中的相同基因具有99.5%或99%序列同一性并且任选地在位置953处具有腺苷的序列。在一个实施方案中,修饰的基因编码改变的基因产物,其中编码的多肽包含一个或多个相对于由产酸丙酸杆菌atcc4875编码的阳离子扩散协助蛋白的氨基酸取代。根据一个实施方案,本发明的丙酸杆菌菌株的阳离子扩散协助蛋白(seqidno:14)与产酸丙酸杆菌atcc4875中编码的阳离子扩散协助蛋白的区别在于1、2、3、4、5、6、7、8、9、10、11个或更多个在位置7、60、71、86、112、114、136、151、162、192和318处的分别为氨基酸残基i、a、n、v、i、a、h、t、p、a和y的氨基酸取代。在一个实施方案中,改变的阳离子扩散协助蛋白与由产酸丙酸杆菌atcc55737编码的阳离子扩散协助蛋白的区别在于1、2或3个非保守氨基酸取代。在一个实施方案中,相对于由产酸丙酸杆菌atcc4875编码的阳离子扩散协助蛋白,本发明菌株的阳离子扩散协助蛋白的改变的基因产物包含在氨基酸位置318处的氨基酸取代,并且更具体地所述氨基酸取代是非保守氨基酸取代。在一个实施方案中,本公开的分离的丙酸杆菌菌株编码这样的阳离子扩散协助蛋白,其中氨基酸位置318处的天然c被f、y和w取代。在一个实施方案中,本公开的分离的丙酸杆菌菌株编码这样的阳离子扩散协助蛋白,其中氨基酸位置318处的天然c被y取代。在一个实施方案中,修饰的阳离子扩散协助蛋白基因包含seqidno:13的序列或编码肽seqidno:14的基因。seqidno:13在序列上以11个不同的核苷酸(在位置19、178、211、256、334、340、406、451、484、574和953处)与产酸丙酸杆菌atcc4875的阳离子扩散协助蛋白基因不同。在一个实施方案中,编码的阳离子扩散促进蛋白包含seqidno:14的序列或与产酸丙酸杆菌atcc4875或atcc55737中的相同蛋白质序列以在表1中提到的位置处的1、2、3个或更多个氨基酸取代而不同的多肽。
在一个实施方案中,新菌株具有整个核糖体rna的额外拷贝和精氨酸脱亚胺酶调节子(argr)的额外拷贝。argr的额外拷贝在基因的位置37处有一个突变(gc的del)。所述突变存在于蛋白质的两个活性结构域之一中。所述突变引起基因发生移码。这个基因与精氨酸脱亚胺酶途径(参与耐酸机制)的调节相关。这里报道的突变对新菌株产酸丙酸杆菌f3e8的改进有积极作用。
在一个实施方案中,提供了分离的丙酸杆菌菌株,任选地为产酸丙酸杆菌菌株,其中所述菌株包含:
(i)修饰的大核糖体rna基因,其包含在相对于产酸丙酸杆菌atcc55737中相同基因的位置1441处以及在相对于产酸丙酸杆菌atcc4875中相同基因的位置1270、1271和1425处的大核糖体基因的核苷酸取代;和/或
(ii)修饰的长链脂酰辅酶a合成酶基因,其包含在相对于产酸丙酸杆菌atcc55737中相同基因的核苷酸位置1500和位置2269之后以及在相对于产酸丙酸杆菌atcc4875中相同基因的位置264、1650和2295处的无义密码子;和/或
(iii)修饰的阳离子扩散协助蛋白基因,其编码包含根据表1的在相对于产酸丙酸杆菌atcc55737中的编码蛋白质的氨基酸位置318处和相对于产酸丙酸杆菌atcc4875的位置的氨基酸取代的基因产物;和/或
(iv)相对于产酸丙酸杆菌atcc4875或atcc55737的整个rna操纵子(seqidno:18)的拷贝数变异(例如,额外拷贝)。
(v)具有相对于产酸丙酸杆菌atcc4875或atcc55737的在位置37处(seqidno:21)的缺失的精氨酸脱亚胺酶调节子(seqidno:19)的拷贝数变异(例如,额外拷贝)。
在一个实施方案中,提供了分离的丙酸杆菌菌株,任选地为产酸丙酸杆菌菌株,其包含相对于选自seqidno:1、seqidno:3和seqidno:4的基因序列的修饰的基因序列,或与seqidno:1、seqidno:3和seqidno:4共有85%、90%、95%或99%序列同一性但小于100%序列同一性的序列。
在一个实施方案中,提供了分离的丙酸杆菌菌株,任选地为产酸丙酸杆菌菌株,其中所述菌株包含:
(i)修饰的大核糖体rna,其包含seqidno:15的序列或与产酸丙酸杆菌atcc4875或atcc55737中的相同序列具有95%或99%序列同一性的序列;和/或
(ii)修饰的长链脂酰coa合成酶,其包含seqidno:17的序列或与seqidno:17因1、2或3个氨基酸取代而不同的多肽;和/或
(iii)修饰的阳离子扩散协助蛋白,其包含seqidno:14的序列或与seqidno:14因1、2或3个氨基酸取代而不同的多肽;和/或
(iv)相对于产酸丙酸杆菌atcc4875或atcc55737的整个rna操纵子(seqidno:17)的拷贝数变异(例如,额外拷贝);和/或
(v)具有相对于产酸丙酸杆菌atcc4875或atcc55737的在位置37处的引起移码的缺失的精氨酸脱亚胺酶调节子(seqidno:20)的拷贝数变异(例如,额外拷贝)。所述缺失位于两个结构域之一中。
在一个实施方案中,提供了分离的丙酸杆菌菌株,任选地为产酸丙酸杆菌菌株,其包含大亚基核糖体基因序列seqidno:15、长链脂酰coa合成酶基因序列seqidno:16和阳离子扩散协助蛋白基因序列seqidno:13。在一个实施方案中,提供了分离的产酸丙酸杆菌菌株,其包含编码包含seqidno:15的大亚基核糖体rna、包含seqidno:17的长链脂酰coa合成酶和包含seqidno:2的阳离子扩散协助蛋白的基因序列。
在一个实施方案中,提供了分离的丙酸杆菌菌株,任选地为产酸丙酸杆菌菌株,其中所述菌株包含一个或多个负责调控精氨酸脱亚胺酶途径的蛋白质的重复,其任选地与一个或多个相对于产酸丙酸杆菌菌株atcc4875的修饰基因结合,所述修饰基因编码上述公开的大亚基核糖体、长链脂酰coa合成酶或阳离子扩散协助蛋白。在一个实施方案中,所述菌株包括表1中列出的任何变异。这些变异包括例如针对转录调节子、转运和与耐酸机制有关的基因的修饰。例如,在一些实施方案中,本公开的另外的丙酸杆菌菌株包括在转录调节子merr基因、deor家族的转录调节子和/或fis家族的σ54特异性转录调节子中的突变。与转运有关的突变的实例包括在na+/h+逆向转运蛋白、abc转运蛋白结合蛋白、abc型硝酸盐/磺酸盐/碳酸氢盐转运系统、砷泵出蛋白和寡肽转运atp结合蛋白中的突变。在一些实施方案中,本公开的丙酸杆菌菌株包括与耐酸机制有关的基因的突变,比如在麦芽寡糖基海藻糖海藻糖水解酶(malto-oligotreahalosetrehalohydrolase)中的突变和在磷酸葡萄糖酸脱氢酶脱羧基因中的突变。其他突变包括2-酮戊二酸脱氢酶复合物的二氢硫辛酰胺琥珀酰转移酶组分e2/2-酮戊二酸脱氢酶e1组分和d-3-磷酸甘油酸脱氢酶以及在过氧化氢酶/过氧化物酶基因中的突变。
根据一个实施方案,提供了选自下组菌株的新产酸丙酸杆菌菌株:f3e8、f3g8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1、f3b9、wgs.1、wgs.2、wgs.3、wgs.4、wgs.5、wgs.6、wgs7、wgs.8、wgs.9、wgs.10、wgs.11、wgs.12和wgs.13。在一个实施方案中,新产酸丙酸杆菌菌株选自下组的菌株:f3e8、f3g8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1和f3b9。在一个实施方案中,新产酸丙酸杆菌菌株选自下组的菌株:f3e8和f3f6。根据一个实施方案,提供了选自下组菌株的新产酸丙酸杆菌菌株:wgs.1、wgs.2、wgs.3、wgs.4、wgs.5、wgs.6、wgs7、wgs.8、wgs.9、wgs.10、wgs.11、wgs.12和wgs.13。
根据一个实施方案,所述新菌株是产酸丙酸杆菌菌株f3e8,其于2015年6月25日保藏于美国典型培养物保藏中心(atcc),10801universityboulevard,manassas,va,20110-2209usa,atcc登录号指定为122267。根据一个实施方案,所述新菌株是产酸丙酸杆菌菌株wgs7,其于____保藏于美国典型培养物保藏中心(atcc),atcc登录号pta-___;在本申请未决期间,专利商标局局长以及由局长决定的有资格的人在提出请求之后可以获取这些保藏物。这些保藏物将在atcc保藏所(其为公共保藏所)保存,期限为30年或最近一次请求后的5年,或为专利的可实施期,以较长者为准,并且如果保藏物在该期限期间无法存活,将会予以更换。申请人不会放弃追究对本专利授予的权利的任何侵犯。
本公开还涵盖产酸丙酸杆菌菌株f3e8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1、f3b9、wgs.1、wgs.2、wgs.3、wgs.4、wgs.5、wgs.6、wgs7、wgs.8、wgs.9、wgs.10、wgs.11、wgs.12和wgs.13的衍生物,所述衍生物通过与其他已知的产酸丙酸杆菌菌株进行基因组改组而形成,或者,其中一个或多个亲本菌株基因已被修饰以便产生所述衍生物。在一个实施方案中,将菌株f3e8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1和f3b9用不同的产酸丙酸杆菌菌株进行一轮或多轮遗传改组。在一个实施方案中,通过对选自菌株f3e8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1、f3b9、wgs.1、wgs.2、wgs.3、wgs.4、wgs.5、wgs.6、wgs7、wgs.8、wgs.9、wgs.10、wgs.11、wgs.12和wgs.13的一个菌株与选自产酸丙酸杆菌atcc4875或产酸丙酸杆菌atcc55737的第二菌株进行基因组改组而形成衍生物。在另一个实施方案中,通过在选自菌株f3e8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1、f3b9、wgs.1、wgs.2、wgs.3、wgs.4、wgs.5、wgs.6、wgs7、wgs.8、wgs.9、wgs.10、wgs.11、wgs.12和wgs.13的一个菌株与选自f3e8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1、f3b9、wgs.1、wgs.2、wgs.3、wgs.4、wgs.5、wgs.6、wgs7、wgs.8、wgs.9、wgs.10、wgs.11、wgs.12和wgs.13的一个不同的菌株之间进行基因组改组而形成衍生物。在一个实施方案中,通过在选自菌株f3e8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1和f3b9的一个菌株与第二个不同的产酸丙酸杆菌菌株(即,不是atcc4875、atcc55737、f3e8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1或f3b9)之间进行基因组改组而形成衍生物。在一个实施方案中,通过在菌株f3e8与第二个不同的产酸丙酸杆菌菌株之间进行基因组改组而形成衍生物。
根据一个实施方案,菌株f3e8、f3g8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1和f3b9被进一步修饰以减少副产物如乙酸盐和琥珀酸盐的产生。这些副产物的产生不仅降低了主要发酵产物的产率,而且还引起产物纯化困难。
根据一个实施方案,提供了丙酸或其衍生物之一如丙醇或丙烯的商业生产方法,所述方法包括利用本文中公开的新产酸丙酸杆菌进行分批发酵。根据一个实施方案,用于发酵的主要底物是蔗糖和/或葡萄糖。在一个实施方案中,主要氮源将是补充有氨基酸/蛋白质混合物的气态氨,所述混合物来自植物衍生的粉末(大豆粉、棉籽粉、玉米浆粉等)。然而,其他氮源对于本领域技术人员来说是已知的并且适合于在公开的方法中使用。
在一个实施方案中,提供了生产丙酸或其衍生物之一如丙醇或丙烯的方法。所述方法包括在适于如本文中公开的产酸丙酸杆菌菌株生长的条件下培养所述菌株并回收由所述菌株产生的丙酸的步骤。在一个实施方案中,培养的产酸丙酸杆菌菌株是分离的或纯化的产酸丙酸杆菌菌株,其中所述菌株具有至少0.24/hr的最大生长速率,所述最大生长速率是针对在32℃、ph6.5下在诸如pam的可接受培养基中培养的细胞的指数期计算的。在一个实施方案中,培养的产酸丙酸杆菌菌株是分离的或纯化的产酸丙酸杆菌菌株,其中所述菌株的最大生长速率是产酸丙酸杆菌atcc菌株4875的至少1.5倍。在一个实施方案中,培养的产酸丙酸杆菌菌株是分离或纯化的产酸丙酸杆菌菌株,其中当在相同的厌氧条件下培养时,所述菌株的丙酸总产率(g/g)相对于产酸丙酸杆菌atcc菌株4875具有10%至15%的增加,并且任选地,所述菌株相对于产酸丙酸杆菌atcc菌株4875具有至少1.5倍的最大生长速率,所述条件任选地为在32℃,ph6.5,在诸如pam的可接受的培养基中。在一个实施方案中,培养的产酸丙酸杆菌菌株是分离或纯化的产酸丙酸杆菌菌株,其中所述菌株具有约0.55或0.66g/g的丙酸产率,并且任选地,当在相同的厌氧条件下培养时,所述菌株具有至少1.5倍于产酸丙酸杆菌atcc菌株4875的最大生长速率,所述条件任选地为在32℃,ph6.5,在诸如pam的可接受的培养基中。在一个实施方案中,培养的产酸丙酸杆菌菌株是分离的或纯化的产酸丙酸杆菌菌株,其中所述菌株产生约0.84g/l/hr的丙酸,并且任选地具有至少0.24/hr的最大生长速率,所述最大生长速率是针对在32℃、ph6.5下在诸如pam的可接受培养基中培养的细胞的指数期计算的。′
在一个实施方案中,培养的丙酸杆菌菌株(任选地产酸丙酸杆菌菌株)包含:
(i)修饰的大核糖体rna,其包含在根据表1的位置处的大核糖体基因的核苷酸置换;
(ii)修饰的长链脂酰coa合成酶基因,其包含根据表1的无义密码子;和
(iii)修饰的阳离子扩散协助蛋白基因,其编码包含在相对于产酸丙酸杆菌atcc55737中的相同基因的氨基酸位置318处和在相对于产酸丙酸杆菌atcc4875的如表1所示的位置处的氨基酸取代的基因产物。在一个实施方案中,培养的丙酸杆菌菌株(任选地产酸丙酸杆菌菌株)包含相对于选自seqidno:1、seqidno:3和seqidno:4的基因序列的修饰的基因序列,或与seqidno:1、seqidno:3和seqidno:4有85%、90%、95%或99%序列同一性但小于100%序列同一性的序列。在一个实施方案中,培养的产酸丙酸杆菌菌株包含相对于基因序列seqidno:1、seqidno:3和seqidno:4的每一个的修饰的基因序列,或与seqidno:1、seqidno:3和seqidno:4共有85%、90%、95%或99%序列同一性但小于100%序列同一性的序列;
(iv)相对于产酸丙酸杆菌atcc4875或atcc55737的整个rna操纵子(seqidno:18)的拷贝数变异(例如,额外的拷贝);和/或
(v)具有相对于产酸丙酸杆菌atcc4875或atcc55737的在位置37处的引起移码的缺失的精氨酸脱亚胺酶调节子(seqidno:20)的拷贝数变异(例如,额外拷贝)。所述缺失位于两个结构域之一中。
在一个实施方案中,培养的产酸丙酸杆菌菌株是分离的或纯化的产酸丙酸杆菌菌株,其选自下组菌株:f3e8、f3c8、f3h8、f3e9、f3f8、f3f6、f3d9、f3c1、f3b9、wgs.1、wgs.2、wgs.3、wgs.4、wgs.5、wgs.6、wgs7、wgs.8、wgs.9、wgs.10、wgs.11、wgs.12和wgs.13,或选自菌株f3e8、f3g8、f3c8、f3h8、f3e9、f3f6、f3d9和f3c1,或选自wgs7、f3e8和f3f6。在一个实施方案中,培养的产酸丙酸杆菌菌株是分离或纯化的产酸丙酸杆菌菌株f3e8,其在atcc登录号122267下保藏。根据一个实施方案,所述新菌株是产酸丙酸杆菌菌株wgs7,其于2016年9月2日保藏于美国典型培养物保藏中心(atcc),atcc登录号为___。
实施例1
利用基因组改组(gs)制备产酸丙酸杆菌新菌株
丙酸杆菌属物种是多形杆状革兰氏阳性细菌,其通过wood-werckman循环天然地产生作为其主要发酵产物的pa。在本质上,pa与其他有机酸(乳酸、琥珀酸和乙酸)一起产生,导致生产率低和产率不高,产生昂贵的下游工艺。直到最近才证明产酸丙酸杆菌的代谢工程是具有挑战性的,原因主要在于有七个成簇规律间隔短回文重复序列(crispr)提供了对接合质粒和噬菌体的抗性。因此使用基因组改组(gs)来提高产酸丙酸杆菌的生长速率和pa生产量,从而获得能够实现提高的产率和更高的生长速率的菌株。
另外,为了理解导致改善的表型的基因组变化,使用下一代测序(ngs)来表征基因型变化。使用clonalframe(didelot和d.falush,genetics,2007,175,1251-66)发现,基因组中的改变与高重组概率的区域对应,这支持了gs期间的基因转化机制。人们主要把细菌中的gs与基因转化(不对称贡献)联系起来,在不对称贡献中,供体提供少量的dna材料,使得受体的其余部分保持完整。
材料和方法
细菌.产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737选自17个菌株的集合(stowers等人,j.ind.microbiol.biotechnol.,2014,41,837-852),它们快速生长并且能够实现高pa产率。使用甘油(20%)作为冷冻保护剂将所述菌株保持在-80℃。
培养基.用于预接种物制备的培养基(pam)由酵母提取物(10g/l)、胰蛋白酶解酪蛋白大豆(5g/l)、k2hpo4(0.05g/l)、mnso4(0.05g/l)和葡萄糖(40g/l)组成。仅添加琼脂(15g/l)来制备pam板。使用葡萄糖(100g/l)作为碳源。将培养基组分和碳源分别在121℃下灭菌20分钟。
原生质体形成缓冲液(pfb).这种缓冲液由溶解在1升的0.05mol/l的tris-hcl中的琥珀酸钠(40.5g/l)、蔗糖(42.75g/l)和mgcl2(1.9g/l)组成,ph7.1。
再生缓冲液(rb).这种缓冲液由酵母提取物(10g/l)、胰蛋白酶解酪蛋白大豆(5g/l)、kh2po4(1.5g/l)、k2hpo4(2.5g/l)和牛血清白蛋白(bsa)(5g/l)组成,调节至ph7。
分析方法.使用biochromlibras12uv/vis分光光度计测量在600nm处的培养物的光密度。使用agilent1200hplc系统和具有保护柱(securityguardcarbo-h,phenomenexpn:ajo-4490)的agilenthiplexh柱(300x7.7mm,pl1170-6830),通过离子排斥色谱法对有机酸、碳水化合物和醇进行定量。使用折射率检测器(agilentrid,g1362a)监测糖和醇,所述折射率检测器设置在正极性和40℃的光学单元温度,同时在210nm(agilentmwd,g1365b)处监测有机酸。使用自动进样器(agilenthip-als,g1367b)将30μl样品注射到柱上并使用柱温箱(agilenttcc,g1316a)将柱温保持在65℃。将分析物用4mmh2so4以0.6ml/min进行等强度洗脱26分钟。使用chemstation(revb.03.02[341])集成色谱图。
接种物制备.在无菌条件下,在含有1ml接种有0.8%(v/v)甘油储液的pam培养基的1.5mleppendorf管中进行细菌活化。允许此培养物在32℃生长24小时。将培养物转移到含有14mlpam培养基的15mlfalcon管中,允许其进一步生长24小时。使用5%(v/v)的这一培养物接种含有100mlpam培养基的250ml血清瓶,允许其进一步生长24小时。使用来自血清瓶的处于指数中期的细胞以0.3的初始od600nm接种发酵罐。
原生质体制备和再生.如guan等人(guan等人,j.biotechnol.,2013,167,56-63)中所述制备具有微小修饰的原生质体。使细胞在补充有40g/l葡萄糖和1%甘氨酸的pam培养基中生长24小时。然后将细胞在含有1%甘氨酸和120g/l葡萄糖的pam培养基中再适应24小时。在至少十代之后,使用pbs将细胞洗涤两次,并在含有15mg/ml-600,000u/ml的溶菌酶溶液-在pfb中的溶菌酶溶液中固定至od600nm=0.2。将细胞壁在120rpm和40℃下消化2小时。使用利用100x油浸物镜的光学显微镜检测原生质体,并使用血球计数器计数。在适当时,使原生质体在rb中再生(ph=7,在32℃,48小时)。
基因组改组(gs).使用来自guan等人的方案(guan等人,j.biotechnol.,2013,167,56-63)并进行了一些修改。将原生质体用紫外线处理0.5分钟或在60℃加热2小时。将细胞混合,离心,并重悬于500μl的pfb中。然后加入500μl含20mmol/lcacl2的peg6000(80%)。在ph7.4、32℃的融合条件下进行30分钟。在融合后,加入5mlpfb,并以2500rpm将悬浮液离心5分钟。将原生质体用5mlpfb洗涤两次并重悬于1mlrb中。
筛选.在第一阶段,使用pam培养基平板筛选具有提高的生长速率的菌株。在32℃的厌氧室中温育pam板,监测24小时,每6小时一次。标记平板上最早的菌落(firstcolonies)并选择用于下一轮gs。在三轮gs之后,随机选择分离的菌落,并且在含有100μlpam培养基的96孔板中筛选。使用酶标仪omega
仪表化发酵罐.使用具有工作体积为1l的2lapplikon发酵罐进行发酵。发酵罐配备了针对ph、溶解氧、温度和搅拌的探头和控制器。用两个rushton叶轮以300rpm控制搅拌速度。使用10mnaoh将ph控制在6.5。使用电热套将培养物的温度保持在32℃。在接种之前,发酵罐用n2喷射至少15分钟。整个发酵过程中保持恒定的n2流量,流率为0.3l/min。
发酵计算.在指数期计算最大比生长速率(μmax)。为了一致性,计算了相同时间间隔(从22.5到52小时)的容积生产率(pv)。利用在消耗的总底物上所产生的总pa,计算出产率(yps)。最后,利用总有机酸生产量计算pa∶乙酸比率和pa∶琥珀酸比率。
dna测序和从头组装.使用
生物信息学工具.使用rast服务器来注释不同的组装的基因组(aziz等人,bmcgenomics,2008,9,75)。使用mauve软件来执行多个全基因组比对。还使用mauve来移动来自gs的菌株的重叠群作为参考,即,基因组关闭的产酸丙酸杆菌f3e8(darling等人,genomeres.,2004,14,1394-403)。使用gril来发现mauve比对中的重排(darling等人,bioinformatics,2003,20,122-124)。使用基因组到基因组距离计算器(ggdc2.0)来计算两个基因组之间的基因组距离(auch等人,genomicsci.,2010,2,142-148)。使用bowtie2和samtool或
结果
筛选优良菌株.
为了利用gs增加pa生产量,选择产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737作为出发菌株。在gs之后,利用高通量微观动力学选择菌落,以选择具有较高生长速率的菌株。将新菌落的生长速率与亲本菌株(在同一个平板上生长)的生长速率进行比较。将每个平板的生长最快的10个菌落在血清瓶中培养。在生物反应器上表征了这组10个菌株中生长最快的菌株之一(以下称为f3e8)(图1c)。与亲本菌株0.15h-1和0.18h-1的生长速率(表2)相比,新菌株显示出0.26h-1的生长速率,与血清瓶中观察到的生长速率一致(表3)。
表2用于测试亲本菌株产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737以及来自gs的新菌株产酸丙酸杆菌f3e8的稳定性的2l仪表化发酵的动力学参数。pa∶sa是发酵结束时的pa滴度除以sa滴度。pa∶aa是发酵结束时的pa滴度除以aa滴度。
*时间范围:225-52h
表3丙酸杆菌菌株的基因组和动力学特征
1.从ncbi网页下载基因组序列(nc_019395.1)。2.内部测序、组装和注释的菌株。3.从产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737之间的基因改组获得的重组体。比生长速率数据是血清瓶中的两个生物学重复的平均值。●基因组关闭
在发酵结束时,与产生30g/l(72小时发酵)的亲本菌株(图1a-1c)相比,f3e8产生40g/l的pa,获得25%的pa生产量的总体提高。就容积生产率而言,相对于最佳的亲本菌株,新菌株显示出27%的提高。就产率而言(yps),与最佳亲本代表的0.44g/g产率相比,新菌株具有0.55g/g的产率,产率增加了20%。
讨论
基因组改组在工业中已经广泛用于增加细菌表型。如本文中报道的,将表现出强基因组差异的两个不同的菌株用作用于基因组改组(gs)的亲本菌株。由gs产生的菌株的比较分析显示,重组仅仅发生在这两个菌株的高度保守基因之间。在使用产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737进行三轮gs后,获得了10个生长速率和pa生产量增加的突变体。
实施例2
产酸丙酸杆菌f3e8血清瓶发酵
材料和方法
细菌.使用实施例1中公开的基因组改组获得的产酸丙酸杆菌f3e8进行在血清瓶中的动力学评价。所述菌株从保持在-80℃的甘油(20%,v/v)储液中取得。
培养基.用于预接种物制备的培养基(pam)由酵母提取物(10g/l)、胰蛋白酶解酪蛋白大豆(5g/l)、k2hpo4(0.05g/l)、mnso4(0.05g/l)和葡萄糖(40g/l)组成。将培养基组分和碳源分别在高压灭菌器中在121℃下灭菌20分钟。
血清瓶.使用具有pam培养基的工作体积为100ml的250ml血清瓶。碳源和培养基分别在血清瓶内消毒并在无菌条件下混合。在高压灭菌之后,用无菌氮气清洗血清瓶的顶部空间。将血清瓶在32℃和90rpm的回转式培养箱中温育96小时。
接种物制备.在无菌条件下用0.8%(v/v)f3e8甘油储液接种含有1mlpam培养基的1.5mleppendorf管,以进行培养的第一步。允许此培养物在32℃生长24小时。然后将培养物转移到含有14mlpam培养基的15mlfalcon管中,允许其进一步生长24小时。使用5%(v/v)的这一培养物接种含有100mlpam培养基的250ml血清瓶,允许其进一步生长24小时。使用来自血清瓶的处于指数中期的细胞以0.3的初始od600nm接种其他血清瓶。
分析方法.使用biochromlibras12uv/vis分光光度计测量在600nm处的培养物的光密度。使用agilent1200hplc系统和具有保护柱(securityguardcarbo-h,phenomenexpn:ajo-4490)的agilenthiplexh柱(300x7.7mm,pl1170-6830),通过离子排斥色谱法对有机酸和碳水化合物进行定量。
结果.图2显示了产酸丙酸杆菌f3e8的生长曲线,表4显示了相应的发酵性能参数。可以看出,最大比生长速率约为0.26h-1,最终的pa生产量约为10.35g/l。pa转化率(yps产率)约为0.66g/g。作为副产品,检测到约0.23g/l的琥珀酸(sa)和1.29g/l的乙酸(aa)。
表4测试来自产酸丙酸杆菌f3e8基因组改组的突变体的生长速率和生产量的血清瓶动力学参数。
讨论.结果表明,产酸丙酸杆菌f3e8将葡萄糖转化为pa的效率在0.6g/g以上。
实施例4
产酸丙酸杆菌atcc4875和产酸丙酸杆菌f3e8中的p值和丙酸耐受性
为了比较产酸丙酸杆菌atcc4875和产酸丙酸杆菌f3e8的耐酸性,使细胞在不同ph值(6.5、5.5和4.5)和不同丙酸(pa)浓度(100、50、25、12.5、6.25、3.125、1.56、0.78和0)的96孔板中生长。在酶标仪中持续监测生长三天。
材料和方法
细菌.使用产酸丙酸杆菌atcc4875和产酸丙酸杆菌f3e8。使用甘油(20%,v/v)作为冷冻保护剂将所述菌株保持在-80℃。
培养基.用于预接种物制备的培养基(pam)由酵母提取物(10g/l)、胰蛋白酶解酪蛋白大豆(5g/l)、k2hpo4(0.05g/l)、mnso4(0.05g/l)和葡萄糖(40g/l)组成。将培养基组分和碳源分别在高压灭菌器中在121℃下灭菌20分钟。使用5mhcl调节ph并使用丙酸钠制备100g/lpa储液。
接种物制备.在无菌条件下用0.8%(v/v)f3e8甘油储液接种含有1mlpam培养基的1.5mleppendorf管,以进行培养的第一步。允许此培养物在32℃生长24小时。将培养物转移到含有14mlpam培养基的15mlfalcon管中,允许其进一步生长24小时。使用处于指数中期的细胞接种微孔板,达到0.2至0.3的od600nm。
微观动力学.使用96孔板测试菌株在不同酸性条件下的生长速率。每孔的工作溶容积为100μl。以一式两份进行动力学研究。在不同pa酸浓度(100、50、25、12.5、6.25、3.125、1.56、0.78和0)下测试三个ph(6.5、5.5和4.5)。首先,各排填充不同ph的100μl的pam。通过用100g/l的储备pa溶液进行双倍稀释来建立pa浓度。将微孔板在酶标仪omegafluostar中温育,所述酶标仪被适配为维持厌氧条件。将微孔板在32℃温育72小时。最后,使用growthrates程序(hall等人,mol.biol.evol.,2014,31,232-238)计算最大比生长速率。
结果.图3a-3b显示了菌株在不同酸性条件下的比生长速率曲线。从图中可以看出,随着pa浓度的增加,两个菌株的生长速率均受到抑制。尽管如此,产酸丙酸杆菌atcc4875(图3a)的抑制模式比突变菌株产酸丙酸杆菌f3e8(图3b)的抑制模式更严重。产酸丙酸杆菌f3e8的最小抑制条件(mic)为ph4.5和pag/l,而产酸丙酸杆菌atcc4875的mic为6.5和50g/l。
实施例5
丙酸杆菌测序、从头组装.和注释.
产酸丙酸杆菌atcc4875的基因组先前已被测序和注释(parizzi等人,bmcgenomics,2012,13,562)。使用truseqillimina300碱基配对末端测序对产酸丙酸杆菌atcc55737和10个gs菌株进行测序。为了关闭基因组,也使用pacbiorsii对重组产酸丙酸杆菌f3e8和亲本产酸丙酸杆菌atcc55737进行测序。使用rast和seed查看器进行基因组注释(aziz等人,bmcgenomics,2008,9,75;overbeek等人,nucleicacidsres.,2005,33,5691-5702)。
产酸丙酸杆菌atcc55737具有3.71mb的基因组、68.7%的gc含量、3406个cds(子系统中的42%)和65个rna。命名为产酸丙酸杆菌f3e8的来自gs的新菌株具有3.63mb大小、68.7%的gc含量、3350个cds(子系统中的43%)和73个rna。来自gs的其他九个菌株具有与菌株产酸丙酸杆菌f3e8相似的基因组特征(表3)。可以看出,来自gs的菌株的基因组大小与亲本菌株产酸丙酸杆菌atcc4875相似。令人惊讶的是,重组体中的rna数量比亲本菌株高出11%。
产酸丙酸杆菌atcc4875与产酸丙酸杆菌atcc55737之间的基因组关联.
为了阐明表型和基因型之间的关系,进行了产酸丙酸杆菌atcc4875和产酸丙酸杆菌atcc55737之间的系统基因组比较。这两个菌株显示出不同的比生长速率。首先,进行基因间比较以确定基因存在、不存在和相似性。这个比较表明,产酸丙酸杆菌atcc4875具有345个独特基因,产酸丙酸杆菌atcc55737具有423个独特基因(e.值<0.0001)。所有参与pa产生的基因的98%之内都是保守的。(e值<0.0001)。pa通过wood-werckman循环而产生,其涉及十个基因:甲基丙二酸单酰coa羧基转移酶(两个亚基)、苹果酸脱氢酶、延胡索酸水合酶、琥珀酸脱氢酶(两个亚基)、丙酰coa:琥珀酸coa转移酶、甲基丙二酰coa差向异构酶和甲基丙二酰coa变位酶(两个亚基)。
观察到两个菌株之间的主要变化是子系统“原噬菌体”。原噬菌体是可移动的元件,可帮助细菌应对不利的环境条件,如亚致死浓度的抗生素,或抵抗渗透压、氧化和酸胁迫。先前已经报道,原噬菌体的数目增加对生长和生物膜形成具有影响。产酸丙酸杆菌atcc55737呈现总大小33,000bp的30种原噬菌体相关蛋白,而产酸丙酸杆菌atcc4875仅有总大小20,143bp的17种原噬菌体相关蛋白;其中11种是在这两个菌株之间共有的。
基因组比较
为了比较通过gs获得的新菌株,我们使用mauve将新菌株的基因组进行了比对;选择了产酸丙酸杆菌atcc4875作为参比。分析显示38个lcb,其中4个倒位,11个重排。比对显示,相比于产酸丙酸杆菌atcc4875,与亲本产酸丙酸杆菌atcc55737的相似性增加。为了计算相似性,使用ggdc2.0,利用基因组距离来计算的重组菌株与亲本之间的差异。24这个比较表明,以产酸丙酸杆菌atcc4875为参考时,重组体的基因组距离为0.1090±0.0013,以产酸丙酸杆菌atcc55737为参考时,其为0.0156±0.0019。
变体分析
使用bowtie2和samtools或
在所有的单独的变异中,在所有10个菌株中只有3个是保守的。第一个snp在大亚基核糖体rna中鉴定出,第二个snp在阳离子扩散协助蛋白中鉴定出,最后一个snp在长链脂酰coa合成酶中鉴定出。在使基因组关闭的情况下,使用pacbio测序也能够鉴定菌株f3e8中的重复元件。与亲本菌株相比,在f3e8中发现了整个核糖体rna操纵子的额外拷贝和精氨酸调节子阻抑物的一个额外拷贝。
导致基因组变异的重组机制的分析
clonalframe是一种贝叶斯系统发育方法,在考虑到同源重组的影响的情况下,它在在进化模型下进行推理(x.didelot和d.falush,genetics,2007,175,1251-66)。为了确定基因组中同源重组高度可能性的区域,使用了clonalframe。首先,使用stripsubsetlcb从单独的mauve比对中除去重组体和亲本之间的变异区。这个脚本由mauve发布,作为补充主要脚本(mauve)的次要程序。将核心区域提交到10,000次clonalframe迭代,其中前半部分作为“预烧”(“bum-in”)(在概率确定开始时用于建立定常迭代的数据)弃去。我们在全部重组基因组中检测到305±42个重组事件,这些重组事件对应于发现变体的区域。另外,我们观察到重组体中的变异位于亲本菌株之间非常保守的基因中,这表明重组事件是发现于新菌株的变体的原因。
讨论
为了解通过基因组改组生成的新菌株的基因型和表型之间的关系,对生长速率和pa生产量提高最大的10个菌株进行了dna测序。
两个亲本菌株之间的比较显示30个原噬菌体的存在是两个菌株之间最显著的差异。其他研究先前已经报道,除其他重要的表型之外,细菌中存在的隐匿性原噬菌体可以提供生长速率的增加。gs菌株有29个与原噬菌体相关的基因,其中47%与亲本菌株产酸丙酸杆菌atcc4875共有。为了解释相对于亲本的生长的增加,进行了变体和多拷贝变异分析。三个突变是所有菌株共有的。
第一个突变是在大亚基核糖体rna中。将突变的核糖体rna与本研究中使用的所有菌株进行比对。先前在其他gs研究中报道过类似的突变。绿色产色链霉菌(streptomycesviridochmogenes)对阿维霉素的抗性与核糖体蛋白s12中的突变有关。第二个共有的突变发现于长链脂酰coa合成酶。该酶是激活复合脂肪酸分解的连接酶家族的成员。该酶通过产生影响蛋白质转运、酶活化、蛋白质酰化、细胞信号转导和转录调节的长链脂酰coa酯而在各种细胞功能的生理调节中发挥作用。我们推测该酶在wood-werkman循环中起到有效利用coa的作用。第三个共有的突变是在阳离子扩散协助蛋白中。该蛋白质被认为是利用质子逆向转运从细胞中去除离子以驱动底物跨膜易位的外排泵。该酶可能在细胞稳态中起着重要作用。
除了共有的突变外,我们还发现与调控、耐酸性和转运相关的基因的变化。例如,组蛋白h1样蛋白hc2具有在五个重组体中的五个位置的突变。先前已经报道了在弗氏链霉菌中使用gs的类似结果,其中在三个突变体中发现了调控基因的突变。在该调查研究中,作者还观察到有关基因的突变的保护,用于实现表型改善。
pacbio测序允许关闭f3e8菌株的基因组,这进而允许检测可能有助于表型改善的基因重复。发现了核糖体rna操纵子的额外拷贝。通过信号识别颗粒修饰,先前已经将核糖体rna的变化与羧酸耐受性相关联。在改良的细胞中新的核糖体机器的存在将允许新的翻译程序,这可能导致新的基因表达程序。这类似于使用全局转录机器工程或核糖体工程获得的菌株,其中整个基因表达改变导致表型改善。同样,还发现编码负责调控精氨酸脱亚胺酶途径的蛋白质的基因的额外拷贝。该途径与产酸丙酸杆菌的耐酸性有关。精氨酸脱亚胺酶途径转运在细胞内的精氨酸,将精氨酸转化为瓜氨酸和氨,以产生鸟氨酸和氨甲酰磷酸。最后,氨基甲酸激酶可逆地将氨甲酰磷酸和adp转化为atp、co2和氨。这个系统不仅可用于产生能源,还可产生用于碱化介质的氨。使用组装的重组基因组和亲本基因组,我们在重组基因组中检测到约300个重组事件,其与新菌株中鉴定的变体的位置强相关。在两个亲本菌株之间的非常保守的基因中发现变异。在亲本菌株的独特基因中没有发现变体,这进一步提供了在gs中的重组机制的证据。
实施例6
使用后续轮的基因组改组进一步改良产酸丙酸杆菌菌株
为了进一步增加生长速率、ph耐受性、丙酸生产率和产率,使用来自原始轮基因组改组的性能最高的菌株f3e8进行另一轮基因组改组。在基因组改组中包括四个另外的丙酸杆菌菌株以确保遗传多样性:产酸丙酸杆菌atcc4875、产酸丙酸杆菌atcc4965、中间型丙酸杆菌atcc14072、詹氏丙酸杆菌atcc9617。
材料和方法
细菌.为了获得基因组多样性,将四个丙酸杆菌野生型菌株和一个突变体用于基因组改组。从17个野生型菌株(stowersetal.,j.ind.microbiol.biotechnol.,2014,41,837-852)的集合中,选择以下四个菌株:产酸丙酸杆菌atcc4875、产酸丙酸杆菌atcc4965、中间型丙酸杆菌atcc14072、詹氏丙酸杆菌atcc9617。我们还使用了本公开中描述的衍生菌株产酸丙酸杆菌f3e8。使用甘油(20%)作为冷冻保护剂将所述菌株保持在-80℃。
培养基.如实施例2中所述,用于预接种物制备的培养基是pam。添加琼脂(15g/l)来制备pam板。添加蔗糖(80g/l),在仪表化发酵罐中进行发酵。将培养基组分和碳源(即,蔗糖)分别在121℃下灭菌20分钟。使用限定化学成分培养基(cdm)来测试外源代谢物添加的作用。基础cdm培养基的组成为(mg/l):蔗糖(20000)、feso4·7h2o(10)、fe(no3)2·9h2o(1)、k2hpo4(100)、kh2po4(500)、mgso4·7h2o(500)、mnso4(10)、cacl2·6h2o(10)、nah2po4·h2o(1597.5)、cocl2·6h2o(10)、na2hpo4(3675)、精氨酸(200)、天冬酰胺(2000)、半胱氨酸(200)、谷氨酰胺(200)、组氨酸(200)、亮氨酸(200)、甲硫氨酸(200)、苯丙氨酸(200)、脯氨酸(200)、丝氨酸(200)、色氨酸(200)、酪氨酸(200)、生物素(0.2)、核黄素(2)、盐酸硫胺素(1)、维生素b12(0.2)和泛酸(2)。
ph梯度平板.使用低ph(3)或接近中性ph(6.5)的补充有5g/lpa盐的pam琼脂制备pa/ph梯度平板。为了产生梯度,将方形平板(100x100mm)的侧面抬高0.5em,并将富含ph6.5的pa的琼脂倒入平板中。一旦该层固化,将平板水平放置,倒入ph为3的第二层补充有pa的琼脂。最后,将平板在室温下放置过夜以形成pa/ph梯度。使用ph试纸条证实ph梯度。
内部ph(phi)的测量.测量内部ph值的方案改编自(guanetal.,j.biotechnol.,2013,167,56-63)。使用2’,7’-双-(2-羧乙基)-5(和6)-羧基荧光素乙酰氧基甲基酯(bcecfam)的荧光方法确定phi。将细胞(对于2ml的工作体积,od600=2)离心(12,000rpm,1min),并用50mmhepes-k缓冲液(ph8)洗涤。将沉淀重悬于2ml相同的hepes-k缓冲液中,并在32℃与1μl1μmbcecfam一起温育20分钟。之后,用50mm磷酸钾缓冲液(ph7)洗涤细胞。将沉淀重悬于hepes-k缓冲液中,过滤一半悬浮液。使用具有490nm(ph敏感)和440nm(ph不敏感)的激发光谱的荧光分光光度计测量荧光强度。在535nm处发射。确定在490和440处的悬浮液(s)和滤液(f)的发射强度的比率:r=(s490-s440)/(f490-f440)。使用该比率和校准曲线来计算所有后续样品的phi。
如下确定每个菌株的校准曲线。将缬氨霉素和尼日利亚菌素(sigma)加入到每个菌株至终浓度为50μm,以维持phi与胞外ph(phex)的平衡。然后将培养物在32℃温育20分钟。将细胞(对于工作体积2ml,a600)离心,洗涤,并重悬于2mlph4、5、6、7或8的缓冲液中(50mm柠檬酸盐缓冲液,ph4和5;50mm磷酸盐缓冲液,ph6、7和8)。之后,加入1μl1μm的bcecfam,在32℃温育20分钟。温育之后,使用对应的缓冲液将细胞洗涤并重悬。最后,过滤一半悬浮液并如上所述进行荧光测定和比率计算。
分析方法.使用biochromlibras12uv/vis分光光度计测量在600nm处的培养物的光密度。使用agilent1200hplc系统和具有保护柱(securityguardcarbo-h,phenomenexpn:ajo-4490)的agilenthiplexh柱(300x7.7mm,pl1170-6830),通过离子排斥色谱法对有机酸、碳水化合物和醇进行定量。使用折射率检测器(agilentrid,g1362a)监测糖和醇,所述折射率检测器设置在正极性和40℃的光学单元温度,同时在210nm(agilentmwd,g1365b)处监测有机酸。使用自动进样器(agilenthip-als,g1367b)将30μl样品注射到柱中并使用柱温箱(agilenttcc,g1316a)将柱温保持在65℃。将分析物用4mmh2so4以0.6ml/min进行等强度洗脱26分钟。使用chemstation(revb.03.02[341])集成色谱图。
如(chacko等人,mol.microbiol.,2014,93(4),797-813)所述将氨基酸定量。简言之,用内标将样品1∶1稀释,并通过rp-hplc分析衍生的氨基酸。在高性能自动进样器(agilenthip-alssl,g1367c)中进行衍生化。在2.5μl硼酸盐缓冲液(0.4n,ph10.2,agilentpn:5061-3339)中加入0.5μl含250μm内标、肌氨酸和2-氨基丁酸的样品,混合,在4℃温育20秒。然后加入1μlopa试剂(在3-巯基丙酸中的10mg邻苯二甲醛/ml,agilentpn:5061-3335),以初步衍生一级氨基酸。将反应物混合并在4℃温育20秒。然后加入0.4μlfmoc试剂(在乙腈中的2.5mg9-芴甲基氯甲酸酯/ml,agilentpn:5061-3337),混合,在4℃温育20秒以衍生其他氨基酸。加入45.6μl缓冲液a(40mmna2hpo4、0.02%nan3,ph7.8)以降低反应的ph,然后将50μl反应混合物注入到具有保护柱(securityguardgeminic18,phenomenexpn:ajo-7597)的agilentzorbaxextendc-18柱(3.5μm,4.6x150mm,agilentpn:763953-902)上。在柱温箱(agilenttcc,g1316b)中将柱温保持在37℃。使用配备有主动密封清洗和脱气装置(agilentdegasser,g1379b)的agilent1200-slhplc系统进行色谱法。hplc梯度在0-18分钟为2-45%b,在18.1-20分钟为50-60%b,在20.1-24分钟为100%b,在24.1-27分钟为2%b-使用二元泵(agilentbinpumpsl,g1312b)。缓冲液b的组成为45%的乙腈、45%的甲醇和10%的水。流率为2ml/min。使用荧光检测器监测衍生化的氨基酸(agilentfld,g1321a)。在1-18分钟在340ex和450emnm处检测到opa衍生的氨基酸,并且在18-27分钟在266ex和305emnm处检测到fmoc衍生的氨基酸。定量的依据是来源于氨基酸标准品(sigma,aas18-10ml)和氨基酸补充(agilent,5062-2478)试剂盒的连续稀释的标准曲线。使用chemstation(revb.03.02[341])集成色谱图。
细胞内代谢物提取.将在仪表化发酵罐的指数中期取样的细胞用于细胞内代谢组学分析。用50%乙腈(acn)进行代谢物提取。简言之,收集对应于1-20光密度单位(od)的细胞体积,并在室温下在20,172g下离心2分钟。然后弃去上清液,将沉淀重悬于50%acn中。将该溶液涡旋10秒,每2分钟一次,共三次,并在4℃、20,172g下离心3分钟。之后,将上清液放入管中并在-80℃冷冻,然后冷冻干燥。最后,将粉末重悬于0.5mlmilliq水中。通过lc-ms分析中心碳代谢的细胞内代谢物,并通过hplc(上述方法)分析细胞内氨基酸。使用细胞干重值将代谢物浓度标准化。对于将产酸丙酸杆菌atcc55737的od600转化为细胞干重(g/l)的因数为0.25,对于产酸丙酸杆菌wgs7为0.29。
接种物制备.在无菌条件下,在含有1ml接种有0.8%(v/v)甘油储液的pam培养基的1.5mleppendorf管中进行细菌活化。允许此培养物在32℃生长24小时。将培养物转移到含有14mlpam培养基的15mlfalcon管中,允许其在32℃再生长24小时。使用5%(vol/vol)的这一培养物接种含有100mlpam培养基的250ml血清瓶,允许其进一步生长24小时。使用来自血清瓶的处于指数中期的细胞以在600nm处测量的0.3的初始光密度接种发酵罐。
原生质体形成、融合和再生.由琥珀酸钠(40.5)、蔗糖(42.75)和mgcl2(1.9)制备原生质体形成缓冲液(pfb)(g/l)。将pfb溶解于1升的ph7.1的0.05mol/ltris-hcl中(guanetal.,″genome-shufflingimprovesacidtolerance...″inadvancesinchemistryresearch,vol15,chapter8(2012)novasciencepublishers,inc.,pp143-152)。由酵母提取物(10)、胰蛋白酶解酪蛋白大豆(5)、kh2po4(1.5)、k2hpo4(2.5)和bsa(5)制备再生缓冲液(rb)(g/l)。将ph调节至7(guanetal.,″genome-shufflingimprovesacidtolerance...″inadvancesinchemistryresearch,vol15,chapter8(2012)novasciencepublishers,inc.,pp143-152)。按照随后所述的一些修改,如(guanetal.,″genome-shufflingimprovesacidtolerance...″inadvancesinchemistryresearch,vol15,chapter8(2012)novasciencepublishers,inc.,pp143-152)中所述制备原生质体。使细胞在补充有40g/l蔗糖和1%甘氨酸的pam培养基中生长24小时。然后将细胞在含有1%甘氨酸和120g/l蔗糖的pam培养基中再适应24小时。在至少十代之后,使用pbs将细胞洗涤两次,并在pfb中的含有15mg/ml(600,000u/ml)的溶菌酶溶液中固定至od600nm=0.2。在125ml烧瓶中在振荡培养箱中以120rpm和40℃将细胞壁消化2小时。在使用100x油浸物镜的光学显微镜中检测原生质体,并使用血球计数器计数。在适当时,使原生质体在rb中再生(ph=7,在32℃,48小时)。对于原生质体融合,使用来自guanetal.,″genome-shufflingimprovesacidtolerance...″inadvancesinchemistryresearch,vol15,chapter8(2012)novasciencepublishers,inc.,pp143-152)的具有小修改的方案。将原生质体用紫外线处理1分钟或在60℃加热2小时。将细胞混合,离心,并重悬于500μl的pfb中。然后加入500μl含20mmol/lcacl2的peg6000(80%)。融合条件为ph7.1,时间30分钟,温度32℃。在融合后,加入5mlpfb,并以3500rpm将样品离心5分钟。将原生质体用5mlpfb洗涤两次并重悬于1mlrb中。
获得高pa产率的重组体.使用下列野生型菌株产生菌株多样性:产酸丙酸杆菌atcc4875、产酸丙酸杆菌atcc4965、中间型丙酸杆菌atcc14072和詹氏丙酸杆菌atcc9617。为了获得这个文库,使用产酸丙酸杆菌atcc4875分别与其他三个野生型丙酸杆菌菌株进行gs。对每组菌株总共进行三轮基因组改组。后续gs轮的细胞选自ph/pa梯度平板的酸性侧。接下来,用获得的丙酸杆菌菌株文库、亲本菌株(产酸丙酸杆菌atcc4875、产酸丙酸杆菌atcc4965、中间型丙酸杆菌atcc14072和詹氏丙酸杆菌atcc9617)和先前产生的突变体产酸丙酸杆菌f3e8进行另外三轮gs。最后,通过在pam培养基琼脂平板上连续稀释来分离新的重组体。随机选择单独的重组体,并在含有ph为5的100μlpam培养基和25g/lpa的96孔板中筛选。使用酶标仪(fluostaromega,bmglabtech,momington,victoria,australia)监测生长,所述酶标仪被适配为通过恒定注入氮气保持厌氧条件。
选择策略的根据是耐酸性提高,其通过新菌株与野生型之间的酸性比率的比较加以确定-用酸性条件下的比生长速率除以非酸性条件下的比生长速率计算出单独的比率。将表现最好的菌株在具有工作体积100ml的250ml血清瓶中放大培养。使用回转式振荡培养箱(multitron,infors-ht,bottmingen,switzerland)以100rpm(2.5cm轨道)的搅拌速度和32℃的工作温度将血清瓶温育96小时。
仪表化发酵罐.使用具有工作体积为1l的2lapplikon发酵罐进行发酵。发酵罐配备了针对ph、溶解氧、温度和搅拌的探头和控制器。用两个rushton叶轮以300rpm控制搅拌速度。使用10mnaoh将ph控制在6.5。使用电热套将培养物的温度保持在32℃。在接种之前,发酵罐用n2喷射至少15分钟。在发酵期间保持恒定的n2流量,流率为0.3l/min。在指数中期获取用于代谢组学分析的样品。
生长表征.使用对数法在指数中期计算比生长速率(μ)。使用程序growthrates(halletal.,mol.biol.evol.,2014,31,232-238进行96孔板中的动力学的比生长速率计算。为了一致性,计算了相同时间间隔(范围从15到30小时)的容积生产率(pv)。利用在消耗的总底物上所产生的总pa,计算出产率(yps)。利用总有机酸生产量计算pa:乙酸比率和pa:琥珀酸比率。通过在指数中期的比生长速率乘以糖或pa与生物量的线性相关计算出蔗糖的比消耗速率(qs)和pa的比生产速率(qp)。
结果
获得高pa产率的菌株.
使用在材料和方法中描述的基因组改组方法,生成并筛选丙酸杆菌菌株。筛选之后,获得13个耐酸性明显提高的菌株。为了证实结果,将选定菌株通过血清瓶发酵加以筛选。从表5中可以看出,新菌株在生长速率和pa产率方面表现出多样性。重组体呈现出平均最终pa滴度增加38%,pa产率平均提高41%,平均生长密度提高27%。而且,平均最终ph值比野生型菌株研究期间观察到的低0.05-0.15个ph单位。其中一个菌株wgs7的丙酸产率达到0.75+/-0.05g/g,为迄今报道的最高值。为了验证血清瓶发酵的结果,我们选择了产率最高的重组体(产酸丙酸杆菌wgs7)用于在2l发酵罐中进行进一步研究。
从表6和图4a-4d可以看出,新菌株(产酸丙酸杆菌wgs7)在其产生pa的能力方面呈现出显著提高。产率增加了37%,pa∶aa比率约85%,容积生产率约80%。另外,对于发酵的指数中期的内部δph(phi-phext),如表6中可见,在wgs7中δph(phi-phext)为1.27,在野生型(atcc55737)中为0.43。从图4d还可以看出,wgs7和atcc55737之间的氨基酸消耗率显著不同。
实施例7
从基因组改组获得的wgs7突变体的基因组学和代谢组学表征
为了更好地了解wgs7菌株性能改善的原因,进行了一系列基因组学、代谢组学和转录组学分析,从而将所述菌株相对于亲本菌株加以表征。
材料和方法
rna提取、测序和分析
将在仪表化发酵罐的指数中期取样的细胞用于rna提取。收获50个od并在室温下以4000g离心10分钟。移除上清液,并向沉淀中加入5ml的
dna测序、从头组装和注释.
使用purelink基因组dna小量提取试剂盒(invitrogen目录号k1820-01)提取不同丙酸杆菌菌株的基因组dna,并使用nanodrop1000(thermoscientific)和qubitdsdnabr测定试剂盒(lifetechnologies目录号q32850)定量。通过用dna凝胶染剂sybrtmsafe(lifetechnologies目录号s33102)在1%琼脂糖凝胶上运行来确定dna的质量。使凝胶在chemidocmp系统(bio-rad)中可视化。使用pacbio获得新的重组菌株的全基因组。使用bluepippin系统所选择的20kb方案进行pacbio文库制备。使用来自pacbio的p6-c4化学制备文库,并使用磁珠进行加载。用smrtportal进行基因组组装。还使用该portal来比对读段,并调用经过测序的基因组的变体。最后,分别使用rast和seed查看器服务器对组装的基因组进行注释和可视化(azizetal.,bmcgenomics,2008,9,75;etal.,methodsmol.biol.,2013,985,17-45)。
结果
产酸丙酸杆菌wgs7的测序、从头组装、注释和变体检测
使用rast服务器注释wgs7基因组(aziz等人,bmcgenomics,2008,9,75)。发现基因组具有3.61mb大小、3333个cds和65个rna。在针对野生型菌株产酸丙酸杆菌atcc55737将wgs7基因组作图后,鉴定出17个snp和7个插入/缺失,如表7所示。这些变体的注释描绘了10个假定蛋白和子系统内的14个蛋白质(表7)。似乎可能影响丙酸生产量的三个突变是abc极性氨基酸转运蛋白基因中的snpc2293187t、细胞色素c生物发生基因中的snpc1487806t和abc多重糖转运蛋白基因启动子中的snpg1917729a。
细胞外代谢物
测量用wgs7和atcc55737的2l发酵中的胞外代谢物(图4a和4b)。在指数中期计算比生长率、蔗糖的比消耗率、游离氨基酸的比消耗率和pa的比生产率。如图4c可以看出,相对于atcc55737,wgs7表现出蔗糖的比消耗率增加96%,pa的比生产率增加216%。有趣的是,wgs7耗尽了培养基中的蔗糖,而atcc55737仅消耗了总糖的69%。这一观察结果支持这样的假设:在推定的abc多重糖转运蛋白基因的启动子中鉴定的snpg1917729a上调了其表达并提高了蔗糖消耗。还检测到游离氨基酸的比消耗率的变化。如图4d可以看出,wgs7具有下列氨基酸的比消耗率的增加:丝氨酸、精氨酸、酪氨酸、缬氨酸、甲硫氨酸、色氨酸、脯氨酸、苯丙氨酸、异亮氨酸、亮氨酸和赖氨酸。
差异性rnaseq分析
评价了野生型菌株产酸丙酸杆菌atcc55737和改组的菌株产酸丙酸杆菌wgs7之间的差异表达。分析鉴定出2406个转录的基因,其中76个表达显著(q<0.05)不同-13个表达下调,63个表达上调。
在显著差异表达的基因中,新菌株呈现出8个abc糖转运蛋白的上调。在这些转运蛋白中,abc糖转运蛋白xloc_000592具有g1917729a突变,其位于该基因的启动子中。改组的菌株也呈现出三个abc氨基酸转运蛋白和三个abc寡肽转运蛋白的显著上调。在这些类别的基因中,abc氨基酸转运蛋白xloc_000834呈现出突变c2293187t(见表7)。关于电子传递基因,鉴定了一氧化氮还原酶xloc_002350(细胞色素c)的上调,其可能与细胞色素c生物发生基因中的snpc1487806t有关。有趣的是,还发现甲基乙二醛途径也在新菌株中过度表达。这个途径通过形成l-乳酸和d-乳酸而辅助降解甲基乙二醛化合物,所述l-乳酸和d-乳酸最终转化为丙酮酸(weberetal.,microbiology+,2005,151(3),707-716)。另外,鉴定了涉及戊糖和葡糖醛酸酯相互转换的三个基因的显著上调:阿卓糖酸脱水酶(uxaa)、阿卓糖酸氧化还原酶(uxab)和unronate异构酶(uxac)。发现磷酸戊糖途径的氧化反应过度表达。在wgs7中与醋酸相互转换有关的反应的表达也高于atcc5573。通过柠檬酸合酶(glta)反应,乙酸异化或同化的中间化合物,即乙酰coa被利用进入tca循环。该基因的转录也发现在wgs7中过度表达。这表明tca循环在改组的菌株中比在wgs7中更活跃。
测试代谢物的单独作用的在cdm上的实验
基于基因组变体和代谢组学概况分析,进行实验来测试单独的代谢物在pa产生中的作用。使用cdm在血清瓶中进行实验。如图5可以看出,当将乳酸盐、延胡索酸盐、脯氨酸、赖氨酸或精氨酸添加至cdm时,产酸丙酸杆菌wgs7使pa生产量分别增加大约112%、50%、38%、38%和25%。相反,野生型菌株产酸丙酸杆菌atcc55737只有在将乳酸盐添加至cdm培养基中时,pa生产量才增加66%。该数据与基因组测序数据一致,揭示了在基因(xloc_000834)中的snp(c2293187t),其被注释为极性氨基酸的推定的abc转运蛋白。这似乎导致了几种极性氨基酸的消耗增加。rnaseq数据证实了这一主张,正如xloc_000834的上调所指示的那样。此外,从图5中可以看出,某些氨基酸的进料可以显著提高菌株wgs7的pa滴度,远远超过用atcc55737所观察到的提高。加入10mm的丝氨酸使wgs7中的滴度提高75%,并且添加10mm的丙氨酸使滴度提高25%。
如前面提到的,wgs7的基因组学分析揭示了位于细胞色素c生物发生基因内的snpc1487806t。相应地,rnaseq分析显示一氧化氮还原酶-xloc_000683(细胞色素c)显著上调(q<0.05,log21.30倍),表明在细胞色素c生物发生中的c1487806t突变可能在c型细胞色素的生成中起着重要作用。这个突变也可以间接地辅助中和细胞内的酸性环境,因为最终的硝酸盐降解产物是氨。另外,据报道,乳酸需要电子受体而被氧化并通过乳酸脱氢酶的反应产生丙酮酸。一些作者认为细胞色素c可以充当乳酸-丙酮酸反应中的天然电子传递(ogataetal.,j.biochem.,1981,89(5),1423-1431;yoshimuraetal.,biochimbiophysacta,1977,492,331-339)。因此,上调的xloc_000683基因可以改善乳酸-丙酮酸反应中的电子传递。为了检验这一假设,进行了富含50mm外源乳酸盐的cdm血清瓶发酵。有趣的是,如图5可以看出,通过添加乳酸盐,wgs7比atcc55737多产生63%的pa。这表明突变c1487806t在产酸丙酸杆菌wgs7的电子传递链中具有重要作用。产酸丙酸杆菌中的电子传递体系还负责woodwerkman循环中延胡索酸盐的还原(parizzietal.,bmcgenomics,2012,13,562)。黄素腺嘌呤二核苷酸(fad)形式的黄素已被鉴定为延胡索酸还原酶(ingledewetal.,microbiol.rev.,1984,48(3),222-71)。关于这个系统,在我们的转录组数据中,我们发现黄素还原酶基因(xloc_001914)显著上调(q<0.05,log21.31)。这表明延胡索酸-琥珀酸步骤有助于pa生产量的增加,因为这与atp生成相关(ingledewetal.,microbiol.rev.,1984,48(3),222-71)。为了评价我们的假设,我们在cdm培养基上添加了外源延胡索酸(10mm),并在血清瓶中培养这两个菌株。如图5可以看出,新菌株的pa滴度比野生型菌株高两倍。后一种菌株对外源添加延胡索酸盐没有呈现任何益处这一事实加强了我们对延胡索酸盐-琥珀酸盐反应中的电子传递链可能改善的假设(图5)。电子传递链中的修饰似乎也影响tca循环中的代谢物池。新菌株仅呈现出柠檬酸盐增加1.13倍和琥珀酸盐增加1.65倍;细胞内苹果酸盐没有呈现任何变化。与我们的结果相反,guanetal.(guanetal.,metabolomics,2015,11(5),1106-1116)在其突变株产酸丙酸杆菌wsh1105中发现超过野生型产酸丙酸杆菌cgmcc1.2230两个数量级的细胞内柠檬酸盐、琥珀酸盐、延胡索酸盐或苹果酸盐。在该研究中,为了测试延胡索酸盐的单独作用,将0-50mm的这种代谢物添加到缓冲的pam培养基中;30mm延胡索酸盐的添加使pa生产量增加了10.52%(guanetal.,metabolomics,2015,11(5),1106-1116)。
序列表
<110>stowers,chris
martins,bianca
cox,brad
luna-flores,carlos
nielsen,lars
marcellin,esteban
<120>改良的用于生产丙酸的丙酸杆菌菌株
<130>9138-258544
<150>62/234900
<151>2015-09-30
<160>22
<170>patentinversion3.5
<210>1
<211>978
<212>dna
<213>propionibacteriumacidi-propionici
<400>1
tcagacgtccagatcctcataggaccgggggtcctcgatcggttccaggtggcccatcac60
ccgcagatcgggccacttgtcgaggagttcgtcgatgaggtcctccatgacgtcgtggcc120
gtgctgaacggtccactcaccggggaccagcatgtggaactccaggaaccgccgatagcc180
ggcctcccgggtgcggatggcgtggaaggcaacccgctcgctgctgtgctggtccaggaa240
ctcccggatggcggcgttgtcggccttcggcagggagacgtccatcagccctgcgcccga300
ctggccgatgagtcgggccccggtgacgaggatattcaggcccaccgcgaaggccacgat360
cgggtccagtcgctgccagccggtgagccacaccaggccgactcccacgaccacgccgat420
cgaggtgacgacgtcggtccacaggtgccgaccgtcggccagcagggtcatcgagcgacg480
acgccggccgttgcgcatgaggaccgcggccaccgagccgttgatgaccgaggcgaccgc540
cgagaccaccaggcccagtcccagattctccaggccctgggggtgcaggaagcggtcgac600
ggccgagaccaggatgacggccgccgcgacgaagatcatgatgccctcgaccgccgcgga660
gaagtactcggccttcgagtgcccgaactggtggttcttgtccggcggcttgatggacac720
cttgagggccaccagggcgacgatcgcggccaccaggttcaccaccgactcggcggcgtc780
cgagagcaggcccaccgatccggtgagaagccacgctccggtcttgaggaggatggtggc840
gatcgccgccgcgatcgacagccaggcgaaacggctcaggtcctccggaggttcgtggcg900
cggggccgagttggctgtcggggggatccgggggctgctcaccgcgccaattatgccagc960
gtccgggcgatcggccac978
<210>2
<211>326
<212>prt
<213>propionibacteriumacidi-propionici
<400>2
seraspvalglnileleuileglyproglyvalleuaspargphegln
151015
valalahishisproglnileglyproleuvalglugluphevalasp
202530
gluvalleuhisaspvalvalalavalleuasnglyproleuthrgly
354045
aspglnhisvalgluleuglngluproproilealaglyleuprogly
505560
alaaspglyvalgluglyasnproleualaalavalleuvalglnglu
65707580
leuproaspglyglyvalvalglyleuargglnglyaspvalhisgln
859095
procysalaargleualaaspgluserglyproglyaspgluaspile
100105110
glnalahisarggluglyhisaspargvalglnserleuproalagly
115120125
gluprohisglnalaaspserhisasphisalaaspargglyaspasp
130135140
valglyproglnvalprothrvalglyglnglnglyhisargalathr
145150155160
thrproalavalalahisgluaspargglyhisargalavalaspasp
165170175
argglyaspargargasphisglnalaglnserglnileleuglnala
180185190
leuglyvalglnglualavalaspglyargaspglnaspaspglyarg
195200205
argaspgluasphisaspalaleuaspargargglygluvalleugly
210215220
leuargvalprogluleuvalvalleuvalargargleuaspglyhis
225230235240
leugluglyhisglnglyaspaspargglyhisglnvalhishisarg
245250255
leuglyglyvalarggluglnalahisargserglyglulysproarg
260265270
serglyleuglugluaspglyglyaspargargargaspargglnpro
275280285
glygluthralaglnvalleuargargphevalalaargglyargval
290295300
glycysargglyaspproglyalaalahisargalaasntyralaser
305310315320
valargalaileglyhis
325
<210>3
<211>3103
<212>dna
<213>propionibacteriumacidi-propionici
<400>3
gttgcaagctactaagtgcggtcggtggatgccttggcaccaagagccgatgaaggacgt60
tgtaacctgcgataagccccggggagttggttcacgagctgtgatccgggggtgtccgaa120
tggggaaaccttgaattgccggagtcatgtccggtgaccctgccctgaatgtataggggt180
gtgggagggaacgtggggaagtgaaacatctcagtacccgcaggaagagaaaacaatatg240
tgattccgtgagtagtggcgagcgaaagcggatgaggccaaaccgtgtgtgtgttcaaac300
cggcaggtgttgcacgtgcggggttgtggggtcttctgggatcgactgccgtcggtccgt360
ccagtgataaatggtgtgttgaagtcgaagcgtctgggaaggcgtaccggagtgggtgag420
agtcccgtagacgtggatgcaccactggtggaggataccccaagtagcgcgggactcgtg480
gaatttcgcgtgaatctggcgggaccacccgtcaagcctgaatactccttggtgaccgat540
agtggatagtaccgtgagggaatggtgaaaagtaccccgggaggggagtgaaagagtacc600
tgaaaccggccgcatacaatccgtcggagcctgcctgtacgggtgggtgacggcgtgcct660
attgaagaatgagcctgcgagttagtggcatgtggcgaggttaacccgtgtggggtagtc720
gtagcgaaagcgagtccgataagggcgccagtcgcgtgttctagacccgaagcggtgtga780
tctatccatggccaggatgaagcgacggtaagacgtcgtggaggtccgcacccacttcag840
ttgaaaatggaggggatgagctgtggataggggtgaaaggccaatcaaacactgtgatag900
ctggttctccccgaaatgcatttaggtgcagcgtcgcgtggttcttgctggaggtagagc960
actggatgatctagggggcctatcagcttaccgaaatcagccaaactccgaatgccggca1020
agtggagcgtggcagtgagacggcgggggataagcttcgtcgtcgagagggaaacagccc1080
agatcatcagctaaggcccctaagcggtggctaagtggaaaaggatgtggagttgcggtg1140
acaaccaggaggttggcttggaagcagccatccttgaaagagtgcgtaatagctcactgg1200
tcaagtgattccgcgccgacaatgtagcggggctcaagccatccgccgaagctgtggcaa1260
tcaggatttactcctggttgggtaggggagcgtcgtgttctcggtgaagcggtccggtga1320
cgggtcgtggaggggatgcgagtgagaatgcaggcatgagtagcgaatgacgggtgagaa1380
acccgtccgccgaatatccaagggttccagggtcaagttaatcttccctgggtgagtcgg1440
gacctaaggcgaggccgacaggcgtagtcgatggacgaccagttgatattctggtaccgg1500
tgtagcaccgtccgtgtcgaggtgtgtgatgctaagcatgcgagtccctgttccgcgggc1560
ctttggtctgtggggtgggtggtgagtgtgtgaaccgatcatgtagtaggcaagctgcgg1620
agggacgcagggaggtagctcaaccccagcgatggttgtctggggctaaacgtgtggacc1680
gtccggtaggtaaatccgccgggcatgatggttgaggcgtgatggcgagcccactgtgtg1740
ggtgagtgagtgatcctgtactgccgagaaaagcttcgtgagcgaggtgtgagccgcccg1800
taccctaaaccgacactggtggattggtagagtataccgaggcgatcgagagaatcatgg1860
tgaaggaactcggcaaaatgaccccgtaacttcgggataaggggtgcccgaaccgtccgg1920
ctgtttactggctgggggcggtgagggtcgcagagtccagggggaaacgactgtttacta1980
aaaacacaggtccgtgcgaagttgtaagacgatgtatacggactgactcctgcccggtgc2040
tggaaggttaaggggaactgtcagggccttcgggttcgaagcggtgaacttaagccccag2100
taaacggcggtggtaactataaccatcctaaggtagcgaaattccttgtcgggtaagttc2160
cgacctgcacgaatggagtaacgatttccctactgtctccaccatgaactcggtgaaatt2220
gcattacgagtaaagatgctcgttacgcgcagcaggacggaaagaccccgggacctttac2280
tatagtttggtattggtgatcggtgcgacttgtgtaggataggtgggagacggtgaagcg2340
gccacgccagtggttgtggagtcattgttgaaataccactctggtcgttctggttacctc2400
acctcggaccgtgatccggttcagggacagtgcctgatgggtagtttgactggggcggtc2460
gcctcctaaaaggtaacggaggcgcccaaaggttccctcagcctggttggtaatcaggtg2520
tcgagtgtaagtgcacaagggagcttgactgtgagacaggcatgtcgagcagggacgaga2580
gtcgggactagtgatctgacggtggcttgtggaagtgccgtcactcaacggataaaaggt2640
accccggggataacaggctgatcttgcccgagcgctcacagcgacggcatggtttggcac2700
ctcgatgtcggctcgtcgcatcctggggctggagtcggtcccaagggttgggctgttcgc2760
ccattaaagcggcacgcgagctgggtttagaacgtcgtgagacagttcggtccctatccg2820
ctgcgcgcacaggaatcttgagaagagctgtctctagtacgagaggaccgagacggactg2880
acctctggtgtgccagttgttccgccaggagcatggctggttggctacgtcgggtcgtga2940
taaccgctgaaagcatctaagcgggaagcacgcttcaagatgagggttcccacagatgaa3000
tctggtaaggcccccgagagatgatcgggttgataggccggacgtggacgcactgcaagg3060
tgcggagctgaccggtactaataggccgagggcttgccccaca3103
<210>4
<211>2335
<212>dna
<213>propionibacteriumacidi-propionici
<400>4
atgaacacggccgagtatctggtcggcgacggcgagtcgggatcggtggcggtggtggac60
tccgacggggtgcacagctatgccgacctccgcgcgcaggcgtcgaggtgccgtgccgcc120
ctcgaggaactcgggttgcgcgccggcgaccgcgtcgggctgatcggcgcgaactgcttc180
ggctgggtggccgcctacctgggggtgctggccgccggcatggtcgccgtgccgatcgcc240
cataccctgcgccccgacgagatcgtctccagaatccggtggctcggcgtccaggccgcc300
ttcctgggcccgatggagtcgcgccgactcgccggacaggtcccggggggtcttcctatg360
gccgccgagggtccgtcgccggcgggccccccgctgcgattcgtcgatcggcccgccgat420
ctggacgccgccctggtgttcacctcgggcaccaccggccggccgcacgtcgtgcggctc480
acccacgccaacctgcaggccaacaccggctccatcctgagctatctgccgctggcagat540
tcggatcgagtccttgtcgtgctgcccttcagctacgtcttcggggcctccctgctgcac600
acccacctgagggtgggagcggccctggtggtgcagccgaacgccgccttcccccagcag660
atggtcgagcggatggccgccgagcgatgcaccggcctcgcgggggtgccctccaccttc720
tcggtgctgctgcgcaacagcaccttcggctcccgtcggctgcccgacctgcgcatcatc780
cagcaggccggcggcaggctcgcgccgacgatggtcgaggagttgcgcggggcccagccc840
caggcgcaggtgttcgtgatgtacgggcagaccgaggccaccgcgcggctgtcctacctg900
ccgccccaggagctggatcgccgtccggggtcgatcggccggggaataccgggagtggac960
ctgcgcgtggtcggcgaggacggctcgcaggtcgccgccggacagatcggcgagatcgtg1020
gccggcggggacaatatctcgcccggctacctcgacgaccccgaggagaccgcccggcgg1080
atgcccggaggggtactgcggaccggcgatctggcgacggtcgacgaggacggttacatc1140
tacgtcgtcgatcgcagggaggacttcatcaagtcgtggggggtgaggatctcgagtcag1200
gacatcgaggcggtggcgctgcagctcaccgatctcgtgtcggtggctgcggtgggagtg1260
cccgacgaggccgccggggagcgggtcgagttggtggccgtgccccgggaggggtcgcgg1320
ctcaccgagggccagatcatcgaccactgccgggcccggctggctaggaccatggtgccg1380
caggccgtgcatcttgtcccgttgatgcccctcaacggcaacggtaaaatctccaagaca1440
gcggtccgcgaactctgcgtggatctggccagagccgaccagtccccaggaggatcatca1500
tgaggcggaaaccagtcgtcgtcctgatcgcaggcgtcctcgcggtggtggtcgccagcg1560
gcgcatgggtgctgcagggcaggcccgacccgctgccctcctcggtggtgaacacggccg1620
ccggcgacgacgagctcgccgatttcagcgggcggaggatcttcttcggtcaccagtccg1680
tcggggcgaacatcatcgatggtctgaaagccgcctattcgggccgggagggctccggcc1740
tcgacgttgtcgagacgcgcaccgatccaggcaggagcggcggatacctggcccatgccg1800
cgatgggggtcaacggcgacccgctcggcaaactcgccgatttcgagaaggtgctggccg1860
gctcgatgggcggcgcggtcgacctcgccgtgctcaagctgtgctacatcgacgtcacgg1920
ccgacaccgacgtcgacgcgctgttcacggcgtactcgcagaccatgaccaggctcgagg1980
ccgcccatccgggggtcaccttcatctacacgacagtcccgctgaccaccgaccggacct2040
ggaagcagaccgtcaagtcgtggatcgggcgcgacgagcagacggggccggacgacaacg2100
ccgcccgccagcgttacaaccggctcgtgcgcgagcggtacggcgattccgacaggctct2160
tcgacatcgccgcggtccaggccacgatggacagctccccgacctcgcggacccgtgacg2220
gatccacctactacgtccttcacgaccggctggcggccgaccccgggccctcaacgcgct2280
ggggtcacgggtgaccgccgcccgcctcgtgcacctggtggccgcacagcactag2335
<210>5
<211>779
<212>prt
<213>propionibacteriumacidi-propionici
<220>
<221>misc_feature
<222>(501)..(501)
<223>501位的xaa是未知的
<220>
<221>misc_feature
<222>(509)..(509)
<223>xaa可为任何天然氨基酸
<220>
<221>misc_feature
<222>(537)..(537)
<223>xaa可为任何天然氨基酸
<220>
<221>misc_feature
<222>(569)..(569)
<223>xaa可为任何天然氨基酸
<220>
<221>misc_feature
<222>(656)..(656)
<223>xaa可为任何天然氨基酸
<220>
<221>misc_feature
<222>(675)..(675)
<223>xaa可为任何天然氨基酸
<220>
<221>misc_feature
<222>(779)..(779)
<223>xaa可为任何天然氨基酸
<400>5
metasnthralaglutyrleuvalglyaspglygluserglyserval
151015
alavalvalaspseraspglyvalhissertyralaaspleuargala
202530
glnalaserargcysargalaalaleuglugluleuglyleuargala
354045
glyaspargvalglyleuileglyalaasncyspheglytrpvalala
505560
alatyrleuglyvalleualaalaglymetvalalavalproileala
65707580
histhrleuargproaspgluilevalserargileargtrpleugly
859095
valglnalaalapheleuglyprometgluserargargleualagly
100105110
glnvalproglyglyleuprometalaalagluglyproserproala
115120125
glyproproleuargphevalaspargproalaaspleuaspalaala
130135140
leuvalphethrserglythrthrglyargprohisvalvalargleu
145150155160
thrhisalaasnleuglnalaasnthrglyserileleusertyrleu
165170175
proleualaaspseraspargvalleuvalvalleuprophesertyr
180185190
valpheglyalaserleuleuhisthrhisleuargvalglyalaala
195200205
leuvalvalglnproasnalaalapheproglnglnmetvalgluarg
210215220
metalaalagluargcysthrglyleualaglyvalproserthrphe
225230235240
servalleuleuargasnserthrpheglyserargargleuproasp
245250255
leuargileileglnglnalaglyglyargleualaprothrmetval
260265270
glugluleuargglyalaglnproglnalaglnvalphevalmettyr
275280285
glyglnthrglualathralaargleusertyrleuproproglnglu
290295300
leuaspargargproglyserileglyargglyileproglyvalasp
305310315320
leuargvalvalglygluaspglyserglnvalalaalaglyglnile
325330335
glygluilevalalaglyglyaspasnileserproglytyrleuasp
340345350
aspproglugluthralaargargmetproglyglyvalleuargthr
355360365
glyaspleualathrvalaspgluaspglytyriletyrvalvalasp
370375380
argarggluasppheilelyssertrpglyvalargilesersergln
385390395400
aspileglualavalalaleuglnleuthraspleuvalservalala
405410415
alavalglyvalproaspglualaalaglygluargvalgluleuval
420425430
alavalproarggluglyserargleuthrgluglyglnileileasp
435440445
hiscysargalaargleualaargthrmetvalproglnalavalhis
450455460
leuvalproleumetproleuasnglyasnglylysileserlysthr
465470475480
alavalarggluleucysvalaspleualaargalaaspglnserpro
485490495
glyglyserserxaaglyglyasnglnserserserxaaserglnala
500505510
serserargtrptrpserproalaalahisglycyscysargalagly
515520525
prothrargcysproproargtrpxaathrargproproalathrthr
530535540
serserproileseralaglyglyglyserserservalthrserpro
545550555560
serglyargthrsersermetvalxaalysproproileargalagly
565570575
argalaproalaserthrleuserargargalaproileglnalagly
580585590
alaalaaspthrtrpprometproargtrpglyserthralathrarg
595600605
seralaasnserproileserargargcystrpproalaargtrpala
610615620
alaargserthrserprocyssersercysalathrserthrserarg
625630635640
prothrprothrserthrargcysserargargthrargargproxaa
645650655
proglyserargproproileargglyserproserserthrarggln
660665670
serargxaaproprothrglyproglyserargproserserarggly
675680685
serglyalathrserargargglyargthrthrthrproproalaser
690695700
valthrthrglysercysalaserglythralaileprothrglyser
705710715720
serthrserproargserargproargtrpthralaproargproarg
725730735
glyprovalthraspproprothrthrserphethrthrglytrparg
740745750
prothrproglyproglnargalaglyvalthrglyaspargargpro
755760765
proargalaproglyglyargthralaleuxaa
770775
<210>6
<211>3103
<212>dna
<213>propionibacteriumacidi-propionici
<400>6
gttgcaagctactaagtgcggtcggtggatgccttggcaccaagagccgatgaaggacgt60
tgtaacctgcgataagccccggggagttggttcacgagctgtgatccgggggtgtccgaa120
tggggaaaccttgaattgccggagtcatgtccggtgaccctgccctgaatgtataggggt180
gtgggagggaacgtggggaagtgaaacatctcagtacccgcaggaagagaaaacaatatg240
tgattccgtgagtagtggcgagcgaaagcggatgaggccaaaccgtgtgtgtgttcaaac300
cggcaggtgttgcacgtgcggggttgtggggtcttctgggatcgactgccgtcggtccgt360
ccagtgataaatggtgtgttgaagtcgaagcgtctgggaaggcgtaccggagtgggtgag420
agtcccgtagacgtggatgcaccactggtggaggataccccaagtagcgcgggactcgtg480
gaatttcgcgtgaatctggcgggaccacccgtcaagcctgaatactccttggtgaccgat540
agtggatagtaccgtgagggaatggtgaaaagtaccccgggaggggagtgaaagagtacc600
tgaaaccggccgcatacaatccgtcggagcctgcctgtacgggtgggtgacggcgtgcct660
attgaagaatgagcctgcgagttagtggcatgtggcgaggttaacccgtgtggggtagtc720
gtagcgaaagcgagtccgataagggcgccagtcgcgtgttctagacccgaagcggtgtga780
tctatccatggccaggatgaagcgacggtaagacgtcgtggaggtccgcacccacttcag840
ttgaaaatggaggggatgagctgtggataggggtgaaaggccaatcaaacactgtgatag900
ctggttctccccgaaatgcatttaggtgcagcgtcgcgtggttcttgctggaggtagagc960
actggatgatctagggggcctatcagcttaccgaaatcagccaaactccgaatgccggca1020
agtggagcgtggcagtgagacggcgggggataagcttcgtcgtcgagagggaaacagccc1080
agatcatcagctaaggcccctaagcggtggctaagtggaaaaggatgtggagttgcggtg1140
acaaccaggaggttggcttggaagcagccatccttgaaagagtgcgtaatagctcactgg1200
tcaagtgattccgcgccgacaatgtagcggggctcaagccatccgccgaagctgtggcaa1260
tcaggatttactcctggttgggtaggggagcgtcgtgttctcggtgaagcggtccggtga1320
cgggtcgtggaggggatgcgagtgagaatgcaggcatgagtagcgaatgacgggtgagaa1380
acccgtccgccgaatatccaagggttccagggtcaagttaatcttccctgggtgagtcgg1440
aacctaaggcgaggccgacaggcgtagtcgatggacgaccagttgatattctggtaccgg1500
tgtagcaccgtccgtgtcgaggtgtgtgatgctaagcatgcgagtccctgttccgcgggc1560
ctttggtctgtggggtgggtggtgagtgtgtgaaccgatcatgtagtaggcaagctgcgg1620
agggacgcagggaggtagctcaaccccagcgatggttgtctggggctaaacgtgtggacc1680
gtccggtaggtaaatccgccgggcatgatggttgaggcgtgatggcgagcccactgtgtg1740
ggtgagtgagtgatcctgtactgccgagaaaagcttcgtgagcgaggtgtgagccgcccg1800
taccctaaaccgacactggtggattggtagagtataccgaggcgatcgagagaatcatgg1860
tgaaggaactcggcaaaatgaccccgtaacttcgggataaggggtgcccgaaccgtccgg1920
ctgtttactggctgggggcggtgagggtcgcagagtccagggggaaacgactgtttacta1980
aaaacacaggtccgtgcgaagttgtaagacgatgtatacggactgactcctgcccggtgc2040
tggaaggttaaggggaactgtcagggccttcgggttcgaagcggtgaacttaagccccag2100
taaacggcggtggtaactataaccatcctaaggtagcgaaattccttgtcgggtaagttc2160
cgacctgcacgaatggagtaacgatttccctactgtctccaccatgaactcggtgaaatt2220
gcattacgagtaaagatgctcgttacgcgcagcaggacggaaagaccccgggacctttac2280
tatagtttggtattggtgatcggtgcgacttgtgtaggataggtgggagacggtgaagcg2340
gccacgccagtggttgtggagtcattgttgaaataccactctggtcgttctggttacctc2400
acctcggaccgtgatccggttcagggacagtgcctgatgggtagtttgactggggcggtc2460
gcctcctaaaaggtaacggaggcgcccaaaggttccctcagcctggttggtaatcaggtg2520
tcgagtgtaagtgcacaagggagcttgactgtgagacaggcatgtcgagcagggacgaga2580
gtcgggactagtgatctgacggtggcttgtggaagtgccgtcactcaacggataaaaggt2640
accccggggataacaggctgatcttgcccgagcgctcacagcgacggcatggtttggcac2700
ctcgatgtcggctcgtcgcatcctggggctggagtcggtcccaagggttgggctgttcgc2760
ccattaaagcggcacgcgagctgggtttagaacgtcgtgagacagttcggtccctatccg2820
ctgcgcgcacaggaatcttgagaagagctgtctctagtacgagaggaccgagacggactg2880
acctctggtgtgccagttgttccgccaggagcatggctggttggctacgtcgggtcgtga2940
taaccgctgaaagcatctaagcgggaagcacgcttcaagatgagggttcccacagatgaa3000
tctggtaaggcccccgagagatgatcgggttgataggccggacgtggacgcactgcaagg3060
tgcggagctgaccggtactaataggccgagggcttgccccaca3103
<210>7
<211>2336
<212>dna
<213>propionibacteriumacidi-propionici
<400>7
atgaacacggccgagtatctggtcggcgacggcgagtcgggatcggtggcggtggtggac60
tccgacggggtgcacagctatgccgacctccgcgcgcaggcgtcgaggtgccgtgccgcc120
ctcgaggaactcgggttgcgcgccggcgaccgcgtcgggctgatcggcgcgaactgcttc180
ggctgggtggccgcctacctgggggtgctggccgccggcatggtcgccgtgccgatcgcc240
cataccctgcgccccgacgagatcgtctccagaatccggtggctcggcgtccaggccgcc300
ttcctgggcccgatggagtcgcgccgactcgccggacaggtcccggggggtcttcctatg360
gccgccgagggtccgtcgccggcgggccccccgctgcgattcgtcgatcggcccgccgat420
ctggacgccgccctggtgttcacctcgggcaccaccggccggccgcacgtcgtgcggctc480
acccacgccaacctgcaggccaacaccggctccatcctgagctatctgccgctggcagat540
tcggatcgagtccttgtcgtgctgcccttcagctacgtcttcggggcctccctgctgcac600
acccacctgagggtgggagcggccctggtggtgcagccgaacgccgccttcccccagcag660
atggtcgagcggatggccgccgagcgatgcaccggcctcgcgggggtgccctccaccttc720
tcggtgctgctgcgcaacagcaccttcggctcccgtcggctgcccgacctgcgcatcatc780
cagcaggccggcggcaggctcgcgccgacgatggtcgaggagttgcgcggggcccagccc840
caggcgcaggtgttcgtgatgtacgggcagaccgaggccaccgcgcggctgtcctacctg900
ccgccccaggagctggatcgccgtccggggtcgatcggccggggaataccgggagtggac960
ctgcgcgtggtcggcgaggacggctcgcaggtcgccgccggacagatcggcgagatcgtg1020
gccggcggggacaatatctcgcccggctacctcgacgaccccgaggagaccgcccggcgg1080
atgcccggaggggtactgcggaccggcgatctggcgacggtcgacgaggacggttacatc1140
tacgtcgtcgatcgcagggaggacttcatcaagtcgtggggggtgaggatctcgagtcag1200
gacatcgaggcggtggcgctgcagctcaccgatctcgtgtcggtggctgcggtgggagtg1260
cccgacgaggccgccggggagcgggtcgagttggtggccgtgccccgggaggggtcgcgg1320
ctcaccgagggccagatcatcgaccactgccgggcccggctggctaggaccatggtgccg1380
caggccgtgcatcttgtcccgttgatgcccctcaacggcaacggtaaaatctccaagaca1440
gcggtccgcgaactctgcgtggatctggccagagccgaccagtccccaggaggatcatca1500
atgaggcggaaaccagtcgtcgtcctgatcgcaggcgtcctcgcggtggtggtcgccagc1560
ggcgcatgggtgctgcagggcaggcccgacccgctgccctcctcggtggtgaacacggcc1620
gccggcgacgacgagctcgccgatttcagcgggcggaggatcttcttcggtcaccagtcc1680
gtcggggcgaacatcatcgatggtctgaaagccgcctattcgggccgggagggctccggc1740
ctcgacgttgtcgagacgcgcaccgatccaggcaggagcggcggatacctggcccatgcc1800
gcgatgggggtcaacggcgacccgctcggcaaactcgccgatttcgagaaggtgctggcc1860
ggctcgatgggcggcgcggtcgacctcgccgtgctcaagctgtgctacatcgacgtcacg1920
gccgacaccgacgtcgacgcgctgttcacggcgtactcgcagaccatgaccaggctcgag1980
gccgcccatccgggggtcaccttcatctacacgacagtcccgctgaccaccgaccggacc2040
tggaagcagaccgtcaagtcgtggatcgggcgcgacgagcagacggggccggacgacaac2100
gccgcccgccagcgttacaaccggctcgtgcgcgagcggtacggcgattccgacaggctc2160
ttcgacatcgccgcggtccaggccacgatggacagctccccgacctcgcggacccgtgac2220
ggatccacctactacgtccttcacgaccggctggcggccgaccccgggccctcaacgcgc2280
tggggtcacgggtgaccgccgcccgcctcgtgcacctggtggccgcacagcactag2336
<210>8
<211>500
<212>prt
<213>propionibacteriumacidi-propionici
<400>8
metasnthralaglutyrleuvalglyaspglygluserglyserval
151015
alavalvalaspseraspglyvalhissertyralaaspleuargala
202530
glnalaserargcysargalaalaleuglugluleuglyleuargala
354045
glyaspargvalglyleuileglyalaasncyspheglytrpvalala
505560
alatyrleuglyvalleualaalaglymetvalalavalproileala
65707580
histhrleuargproaspgluilevalserargileargtrpleugly
859095
valglnalaalapheleuglyprometgluserargargleualagly
100105110
glnvalproglyglyleuprometalaalagluglyproserproala
115120125
glyproproleuargphevalaspargproalaaspleuaspalaala
130135140
leuvalphethrserglythrthrglyargprohisvalvalargleu
145150155160
thrhisalaasnleuglnalaasnthrglyserileleusertyrleu
165170175
proleualaaspseraspargvalleuvalvalleuprophesertyr
180185190
valpheglyalaserleuleuhisthrhisleuargvalglyalaala
195200205
leuvalvalglnproasnalaalapheproglnglnmetvalgluarg
210215220
metalaalagluargcysthrglyleualaglyvalproserthrphe
225230235240
servalleuleuargasnserthrpheglyserargargleuproasp
245250255
leuargileileglnglnalaglyglyargleualaprothrmetval
260265270
glugluleuargglyalaglnproglnalaglnvalphevalmettyr
275280285
glyglnthrglualathralaargleusertyrleuproproglnglu
290295300
leuaspargargproglyserileglyargglyileproglyvalasp
305310315320
leuargvalvalglygluaspglyserglnvalalaalaglyglnile
325330335
glygluilevalalaglyglyaspasnileserproglytyrleuasp
340345350
aspproglugluthralaargargmetproglyglyvalleuargthr
355360365
glyaspleualathrvalaspgluaspglytyriletyrvalvalasp
370375380
argarggluasppheilelyssertrpglyvalargilesersergln
385390395400
aspileglualavalalaleuglnleuthraspleuvalservalala
405410415
alavalglyvalproaspglualaalaglygluargvalgluleuval
420425430
alavalproarggluglyserargleuthrgluglyglnileileasp
435440445
hiscysargalaargleualaargthrmetvalproglnalavalhis
450455460
leuvalproleumetproleuasnglyasnglylysileserlysthr
465470475480
alavalarggluleucysvalaspleualaargalaaspglnserpro
485490495
glyglyserser
500
<210>9
<211>3103
<212>rna
<213>propionibacteriumacidi-propionici
<400>9
guugcaagcuacuaagugcggucgguggaugccuuggcaccaagagccgaugaaggacgu60
uguaaccugcgauaagccccggggaguugguucacgagcugugauccggggguguccgaa120
uggggaaaccuugaauugccggagucauguccggugacccugcccugaauguauaggggu180
gugggagggaacguggggaagugaaacaucucaguacccgcaggaagagaaaacaauaug240
ugauuccgugaguaguggcgagcgaaagcggaugaggccaaaccguguguguguucaaac300
cggcagguguugcacgugcgggguuguggggucuucugggaucgacugccgucgguccgu360
ccagugauaaaugguguguugaagucgaagcgucugggaaggcguaccggagugggugag420
agucccguagacguggaugcaccacugguggaggauaccccaaguagcgcgggacucgug480
gaauuucgcgugaaucuggcgggaccacccgucaagccugaauacuccuuggugaccgau540
aguggauaguaccgugagggaauggugaaaaguaccccgggaggggagugaaagaguacc600
ugaaaccggccgcauacaauccgucggagccugccuguacgggugggugacggcgugccu660
auugaagaaugagccugcgaguuaguggcauguggcgagguuaacccgugugggguaguc720
guagcgaaagcgaguccgauaagggcgccagucgcguguucuagacccgaagcgguguga780
ucuauccauggccaggaugaagcgacgguaagacgucguggagguccgcacccacuucag840
uugaaaauggaggggaugagcuguggauaggggugaaaggccaaucaaacacugugauag900
cugguucuccccgaaaugcauuuaggugcagcgucgcgugguucuugcuggagguagagc960
acuggaugaucuagggggccuaucagcuuaccgaaaucagccaaacuccgaaugccggca1020
aguggagcguggcagugagacggcgggggauaagcuucgucgucgagagggaaacagccc1080
agaucaucagcuaaggccccuaagcgguggcuaaguggaaaaggauguggaguugcggug1140
acaaccaggagguuggcuuggaagcagccauccuugaaagagugcguaauagcucacugg1200
ucaagugauuccgcgccgacaauguagcggggcucaagccauccgccgaagcuguggcaa1260
ucaggauuuacuccugguuggguaggggagcgucguguucucggugaagcgguccgguga1320
cgggucguggaggggaugcgagugagaaugcaggcaugaguagcgaaugacgggugagaa1380
acccguccgccgaauauccaaggguuccagggucaaguuaaucuucccugggugagucgg1440
gaccuaaggcgaggccgacaggcguagucgauggacgaccaguugauauucugguaccgg1500
uguagcaccguccgugucgaggugugugaugcuaagcaugcgagucccuguuccgcgggc1560
cuuuggucugugggguggguggugagugugugaaccgaucauguaguaggcaagcugcgg1620
agggacgcagggagguagcucaaccccagcgaugguugucuggggcuaaacguguggacc1680
guccgguagguaaauccgccgggcaugaugguugaggcgugauggcgagcccacugugug1740
ggugagugagugauccuguacugccgagaaaagcuucgugagcgaggugugagccgcccg1800
uacccuaaaccgacacugguggauugguagaguauaccgaggcgaucgagagaaucaugg1860
ugaaggaacucggcaaaaugaccccguaacuucgggauaaggggugcccgaaccguccgg1920
cuguuuacuggcugggggcggugagggucgcagaguccagggggaaacgacuguuuacua1980
aaaacacagguccgugcgaaguuguaagacgauguauacggacugacuccugcccggugc2040
uggaagguuaaggggaacugucagggccuucggguucgaagcggugaacuuaagccccag2100
uaaacggcggugguaacuauaaccauccuaagguagcgaaauuccuugucggguaaguuc2160
cgaccugcacgaauggaguaacgauuucccuacugucuccaccaugaacucggugaaauu2220
gcauuacgaguaaagaugcucguuacgcgcagcaggacggaaagaccccgggaccuuuac2280
uauaguuugguauuggugaucggugcgacuuguguaggauaggugggagacggugaagcg2340
gccacgccagugguuguggagucauuguugaaauaccacucuggucguucugguuaccuc2400
accucggaccgugauccgguucagggacagugccugauggguaguuugacuggggcgguc2460
gccuccuaaaagguaacggaggcgcccaaagguucccucagccugguugguaaucaggug2520
ucgaguguaagugcacaagggagcuugacugugagacaggcaugucgagcagggacgaga2580
gucgggacuagugaucugacgguggcuuguggaagugccgucacucaacggauaaaaggu2640
accccggggauaacaggcugaucuugcccgagcgcucacagcgacggcaugguuuggcac2700
cucgaugucggcucgucgcauccuggggcuggagucggucccaaggguugggcuguucgc2760
ccauuaaagcggcacgcgagcuggguuuagaacgucgugagacaguucggucccuauccg2820
cugcgcgcacaggaaucuugagaagagcugucucuaguacgagaggaccgagacggacug2880
accucuggugugccaguuguuccgccaggagcauggcugguuggcuacgucgggucguga2940
uaaccgcugaaagcaucuaagcgggaagcacgcuucaagaugaggguucccacagaugaa3000
ucugguaaggcccccgagagaugaucggguugauaggccggacguggacgcacugcaagg3060
ugcggagcugaccgguacuaauaggccgagggcuugccccaca3103
<210>10
<211>326
<212>prt
<213>propionibacteriumacidi-propionici
<400>10
seraspvalglnileleuileglyproglyvalleuaspargphegln
151015
valalahishisproglnileglyproleuvalglugluphevalasp
202530
gluvalleuhisaspvalvalalavalleuasnglyproleuthrgly
354045
aspglnhisvalgluleuglngluproproilealaglyleuprogly
505560
alaaspglyvalgluglyasnproleualaalavalleuvalglnglu
65707580
leuproaspglyglyvalvalglyleuargglnglyaspvalhisgln
859095
procysalaargleualaaspgluserglyproglyaspgluaspile
100105110
glnalahisarggluglyhisaspargvalglnserleuproalagly
115120125
gluprohisglnalaaspserhisasphisalaaspargglyaspasp
130135140
valglyproglnvalprothrvalglyglnglnglyhisargalathr
145150155160
thrproalavalalahisgluaspargglyhisargalavalaspasp
165170175
argglyaspargargasphisglnalaglnserglnileleuglnala
180185190
leuglyvalglnglualavalaspglyargaspglnaspaspglyarg
195200205
argaspgluasphisaspalaleuaspargargglygluvalleugly
210215220
leuargvalprogluleuvalvalleuvalargargleuaspglyhis
225230235240
leugluglyhisglnglyaspaspargglyhisglnvalhishisarg
245250255
leuglyglyvalarggluglnalahisargserglyglulysproarg
260265270
serglyleuglugluaspglyglyaspargargargaspargglnpro
275280285
glygluthralaglnvalleuargargphevalalaargglyargval
290295300
glycysargglyaspproglyalaalahisargalaasncysalaser
305310315320
valargalaileglyhis
325
<210>11
<211>978
<212>dna
<213>propionibacteriumacidi-propionici
<400>11
tcagacgtccagatcctcgtaggaccgggggtcctcgatcggttccaggtggcccatcac60
ccgcagatcgggccacttgtcgaggagttcgtcgatgaggtcctccatgacgtcgtggcc120
gtgctgaacggtccactcaccggggaccagcatgtggaactccaggaaccgccgatatcc180
ggcctcccgggtgcggatggcgtggaaggcgacccgctcgctgctgtgctggtccaggaa240
ctcccggatggcggcattgtcggccttcggcagggagacgtccatcagccctgcgcccga300
ctggccgatgagtcgggccccggtgacgaggatgttcagccccaccgcgaaggccacgat360
cgggtccagtcgctgccagccggtgagccacaccaggccgactccaacgaccacgccgat420
cgaggtgacgacgtcggtccacaggtgccgcccgtcggccagcagggtcatcgagcgacg480
acgtcggccgttgcgcatgaggaccgcggccaccgagccgttgatgaccgaggcgaccgc540
cgagaccaccaggcccagtcccagattctccagaccctgggggtgcaggaagcggtcgac600
ggccgagaccaggatgacggccgccgcgacgaagatcatgatgccctcgaccgccgcgga660
gaagtactcggccttcgagtgcccgaactggtggttcttgtccggcggcttgatggacac720
cttgagggccaccagggcgacgatcgcggccaccaggttcaccaccgactcggcggcgtc780
cgagagcaggcccaccgatccggtgagaagccacgctccggtcttgaggaggatggtggc840
gatcgccgccgcgatcgacagccaggcgaaacggctcaggtcctccggaggttcgtggcg900
cggggccgagttggctgtcggggggatccgggggctgctcaccgcgccaattgtgccagc960
gtccgggcgatcggccac978
<210>12
<211>3103
<212>dna
<213>propionibacteriumacidi-propionici
<400>12
gttgcaagctactaagtgcggtcggtggatgccttggcaccaagagccgatgaaggacgt60
tgtaacctgcgataagccccggggagttggttcacgagctgtgatccgggggtgtccgaa120
tggggaaaccttgaattgccggagtcatgtccggtgaccctgccctgaatgtataggggt180
gtgggagggaacgtggggaagtgaaacatctcagtacccgcaggaagagaaaacaatatg240
tgattccgtgagtagtggcgagcgaaagcggatgaggccaaaccgtgtgtgtgttcaaac300
cggcaggtgttgcacgtgcggggttgtggggtcttctgggatcgactgccgtcggtccgt360
ccagtgataaatggtgtgttgaagtcgaagcgtctgggaaggcgtaccggagtgggtgag420
agtcccgtagacgtggatgcaccactggtggaggataccccaagtagcgcgggactcgtg480
gaatttcgcgtgaatctggcgggaccacccgtcaagcctgaatactccttggtgaccgat540
agtggatagtaccgtgagggaatggtgaaaagtaccccgggaggggagtgaaagagtacc600
tgaaaccggccgcatacaatccgtcggagcctgcctgtacgggtgggtgacggcgtgcct660
attgaagaatgagcctgcgagttagtggcatgtggcgaggttaacccgtgtggggtagtc720
gtagcgaaagcgagtccgataagggcgccagtcgcgtgttctagacccgaagcggtgtga780
tctatccatggccaggatgaagcgacggtaagacgtcgtggaggtccgcacccacttcag840
ttgaaaatggaggggatgagctgtggataggggtgaaaggccaatcaaacactgtgatag900
ctggttctccccgaaatgcatttaggtgcagcgtcgcgtggttcttgctggaggtagagc960
actggatgatctagggggcctatcagcttaccgaaatcagccaaactccgaatgccggca1020
agtggagcgtggcagtgagacggcgggggataagcttcgtcgtcgagagggaaacagccc1080
agatcatcagctaaggcccctaagcggtggctaagtggaaaaggatgtggagttgcggtg1140
acaaccaggaggttggcttggaagcagccatccttgaaagagtgcgtaatagctcactgg1200
tcaagtgattccgcgccgacaatgtagcggggctcaagccatccgccgaagctgtggcaa1260
tcaggatttgttcctggttgggtaggggagcgtcgtgttctcggtgaagcggtccggtga1320
cgggtcgtggaggggatgcgagtgagaatgcaggcatgagtagcgaatgacgggtgagaa1380
acccgtccgccgaatatccaagggttccagggtcaagttaatctgccctgggtgagtcgg1440
gacctaaggcgaggccgacaggcgtagtcgatggacgaccagttgatattctggtaccgg1500
tgtagcaccgtccgtgtcgaggtgtgtgatgctaagcatgcgagtccctgttccgcgggc1560
ctttggtctgtggggtgggtggtgagtgtgtgaaccgatcatgtagtaggcaagctgcgg1620
agggacgcagggaggtagctcaaccccagcgatggttgtctggggctaaacgtgtggacc1680
gtccggtaggtaaatccgccgggcatgatggttgaggcgtgatggcgagcccactgtgtg1740
ggtgagtgagtgatcctgtactgccgagaaaagcttcgtgagcgaggtgtgagccgcccg1800
taccctaaaccgacactggtggattggtagagtataccgaggcgatcgagagaatcatgg1860
tgaaggaactcggcaaaatgaccccgtaacttcgggataaggggtgcccgaaccgtccgg1920
ctgtttactggctgggggcggtgagggtcgcagagtccagggggaaacgactgtttacta1980
aaaacacaggtccgtgcgaagttgtaagacgatgtatacggactgactcctgcccggtgc2040
tggaaggttaaggggaactgtcagggccttcgggttcgaagcggtgaacttaagccccag2100
taaacggcggtggtaactataaccatcctaaggtagcgaaattccttgtcgggtaagttc2160
cgacctgcacgaatggagtaacgatttccctactgtctccaccatgaactcggtgaaatt2220
gcattacgagtaaagatgctcgttacgcgcagcaggacggaaagaccccgggacctttac2280
tatagtttggtattggtgatcggtgcgacttgtgtaggataggtgggagacggtgaagcg2340
gccacgccagtggttgtggagtcattgttgaaataccactctggtcgttctggttacctc2400
acctcggaccgtgatccggttcagggacagtgcctgatgggtagtttgactggggcggtc2460
gcctcctaaaaggtaacggaggcgcccaaaggttccctcagcctggttggtaatcaggtg2520
tcgagtgtaagtgcacaagggagcttgactgtgagacaggcatgtcgagcagggacgaga2580
gtcgggactagtgatctgacggtggcttgtggaagtgccgtcactcaacggataaaaggt2640
accccggggataacaggctgatcttgcccgagcgctcacagcgacggcatggtttggcac2700
ctcgatgtcggctcgtcgcatcctggggctggagtcggtcccaagggttgggctgttcgc2760
ccattaaagcggcacgcgagctgggtttagaacgtcgtgagacagttcggtccctatccg2820
ctgcgcgcacaggaatcttgagaagagctgtctctagtacgagaggaccgagacggactg2880
acctctggtgtgccagttgttccgccaggagcatggctggttggctacgtcgggtcgtga2940
taaccgctgaaagcatctaagcgggaagcacgcttcaagatgagggttcccacagatgaa3000
tctggtaaggcccccgagagatgatcgggttgataggccggacgtggacgcactgcaagg3060
tgcggagctgaccggtactaataggccgagggcttgccccaca3103
<210>13
<211>978
<212>dna
<213>propionibacteriumacidi-propionici
<400>13
tcagacgtccagatcctcataggaccgggggtcctcgatcggttccaggtggcccatcac60
ccgcagatcgggccacttgtcgaggagttcgtcgatgaggtcctccatgacgtcgtggcc120
gtgctgaacggtccactcaccggggaccagcatgtggaactccaggaaccgccgatagcc180
ggcctcccgggtgcggatggcgtggaaggcaacccgctcgctgctgtgctggtccaggaa240
ctcccggatggcggcgttgtcggccttcggcagggagacgtccatcagccctgcgcccga300
ctggccgatgagtcgggccccggtgacgaggatattcaggcccaccgcgaaggccacgat360
cgggtccagtcgctgccagccggtgagccacaccaggccgactcccacgaccacgccgat420
cgaggtgacgacgtcggtccacaggtgccgaccgtcggccagcagggtcatcgagcgacg480
acgccggccgttgcgcatgaggaccgcggccaccgagccgttgatgaccgaggcgaccgc540
cgagaccaccaggcccagtcccagattctccaggccctgggggtgcaggaagcggtcgac600
ggccgagaccaggatgacggccgccgcgacgaagatcatgatgccctcgaccgccgcgga660
gaagtactcggccttcgagtgcccgaactggtggttcttgtccggcggcttgatggacac720
cttgagggccaccagggcgacgatcgcggccaccaggttcaccaccgactcggcggcgtc780
cgagagcaggcccaccgatccggtgagaagccacgctccggtcttgaggaggatggtggc840
gatcgccgccgcgatcgacagccaggcgaaacggctcaggtcctccggaggttcgtggcg900
cggggccgagttggctgtcggggggatccgggggctgctcaccgcgccaattatgccagc960
gtccgggcgatcggccac978
<210>14
<211>326
<212>prt
<213>propionibacteriumacidi-propionici
<400>14
seraspvalglnileleuileglyproglyvalleuaspargphegln
151015
valalahishisproglnileglyproleuvalglugluphevalasp
202530
gluvalleuhisaspvalvalalavalleuasnglyproleuthrgly
354045
aspglnhisvalgluleuglngluproproilealaglyleuprogly
505560
alaaspglyvalgluglyasnproleualaalavalleuvalglnglu
65707580
leuproaspglyglyvalvalglyleuargglnglyaspvalhisgln
859095
procysalaargleualaaspgluserglyproglyaspgluaspile
100105110
glnalahisarggluglyhisaspargvalglnserleuproalagly
115120125
gluprohisglnalaaspserhisasphisalaaspargglyaspasp
130135140
valglyproglnvalprothrvalglyglnglnglyhisargalathr
145150155160
thrproalavalalahisgluaspargglyhisargalavalaspasp
165170175
argglyaspargargasphisglnalaglnserglnileleuglnala
180185190
leuglyvalglnglualavalaspglyargaspglnaspaspglyarg
195200205
argaspgluasphisaspalaleuaspargargglygluvalleugly
210215220
leuargvalprogluleuvalvalleuvalargargleuaspglyhis
225230235240
leugluglyhisglnglyaspaspargglyhisglnvalhishisarg
245250255
leuglyglyvalarggluglnalahisargserglyglulysproarg
260265270
serglyleuglugluaspglyglyaspargargargaspargglnpro
275280285
glygluthralaglnvalleuargargphevalalaargglyargval
290295300
glycysargglyaspproglyalaalahisargalaasntyralaser
305310315320
valargalaileglyhis
325
<210>15
<211>3103
<212>dna
<213>propionibacteriumacidi-propionici
<400>15
gttgcaagctactaagtgcggtcggtggatgccttggcaccaagagccgatgaaggacgt60
tgtaacctgcgataagccccggggagttggttcacgagctgtgatccgggggtgtccgaa120
tggggaaaccttgaattgccggagtcatgtccggtgaccctgccctgaatgtataggggt180
gtgggagggaacgtggggaagtgaaacatctcagtacccgcaggaagagaaaacaatatg240
tgattccgtgagtagtggcgagcgaaagcggatgaggccaaaccgtgtgtgtgttcaaac300
cggcaggtgttgcacgtgcggggttgtggggtcttctgggatcgactgccgtcggtccgt360
ccagtgataaatggtgtgttgaagtcgaagcgtctgggaaggcgtaccggagtgggtgag420
agtcccgtagacgtggatgcaccactggtggaggataccccaagtagcgcgggactcgtg480
gaatttcgcgtgaatctggcgggaccacccgtcaagcctgaatactccttggtgaccgat540
agtggatagtaccgtgagggaatggtgaaaagtaccccgggaggggagtgaaagagtacc600
tgaaaccggccgcatacaatccgtcggagcctgcctgtacgggtgggtgacggcgtgcct660
attgaagaatgagcctgcgagttagtggcatgtggcgaggttaacccgtgtggggtagtc720
gtagcgaaagcgagtccgataagggcgccagtcgcgtgttctagacccgaagcggtgtga780
tctatccatggccaggatgaagcgacggtaagacgtcgtggaggtccgcacccacttcag840
ttgaaaatggaggggatgagctgtggataggggtgaaaggccaatcaaacactgtgatag900
ctggttctccccgaaatgcatttaggtgcagcgtcgcgtggttcttgctggaggtagagc960
actggatgatctagggggcctatcagcttaccgaaatcagccaaactccgaatgccggca1020
agtggagcgtggcagtgagacggcgggggataagcttcgtcgtcgagagggaaacagccc1080
agatcatcagctaaggcccctaagcggtggctaagtggaaaaggatgtggagttgcggtg1140
acaaccaggaggttggcttggaagcagccatccttgaaagagtgcgtaatagctcactgg1200
tcaagtgattccgcgccgacaatgtagcggggctcaagccatccgccgaagctgtggcaa1260
tcaggatttactcctggttgggtaggggagcgtcgtgttctcggtgaagcggtccggtga1320
cgggtcgtggaggggatgcgagtgagaatgcaggcatgagtagcgaatgacgggtgagaa1380
acccgtccgccgaatatccaagggttccagggtcaagttaatcttccctgggtgagtcgg1440
gacctaaggcgaggccgacaggcgtagtcgatggacgaccagttgatattctggtaccgg1500
tgtagcaccgtccgtgtcgaggtgtgtgatgctaagcatgcgagtccctgttccgcgggc1560
ctttggtctgtggggtgggtggtgagtgtgtgaaccgatcatgtagtaggcaagctgcgg1620
agggacgcagggaggtagctcaaccccagcgatggttgtctggggctaaacgtgtggacc1680
gtccggtaggtaaatccgccgggcatgatggttgaggcgtgatggcgagcccactgtgtg1740
ggtgagtgagtgatcctgtactgccgagaaaagcttcgtgagcgaggtgtgagccgcccg1800
taccctaaaccgacactggtggattggtagagtataccgaggcgatcgagagaatcatgg1860
tgaaggaactcggcaaaatgaccccgtaacttcgggataaggggtgcccgaaccgtccgg1920
ctgtttactggctgggggcggtgagggtcgcagagtccagggggaaacgactgtttacta1980
aaaacacaggtccgtgcgaagttgtaagacgatgtatacggactgactcctgcccggtgc2040
tggaaggttaaggggaactgtcagggccttcgggttcgaagcggtgaacttaagccccag2100
taaacggcggtggtaactataaccatcctaaggtagcgaaattccttgtcgggtaagttc2160
cgacctgcacgaatggagtaacgatttccctactgtctccaccatgaactcggtgaaatt2220
gcattacgagtaaagatgctcgttacgcgcagcaggacggaaagaccccgggacctttac2280
tatagtttggtattggtgatcggtgcgacttgtgtaggataggtgggagacggtgaagcg2340
gccacgccagtggttgtggagtcattgttgaaataccactctggtcgttctggttacctc2400
acctcggaccgtgatccggttcagggacagtgcctgatgggtagtttgactggggcggtc2460
gcctcctaaaaggtaacggaggcgcccaaaggttccctcagcctggttggtaatcaggtg2520
tcgagtgtaagtgcacaagggagcttgactgtgagacaggcatgtcgagcagggacgaga2580
gtcgggactagtgatctgacggtggcttgtggaagtgccgtcactcaacggataaaaggt2640
accccggggataacaggctgatcttgcccgagcgctcacagcgacggcatggtttggcac2700
ctcgatgtcggctcgtcgcatcctggggctggagtcggtcccaagggttgggctgttcgc2760
ccattaaagcggcacgcgagctgggtttagaacgtcgtgagacagttcggtccctatccg2820
ctgcgcgcacaggaatcttgagaagagctgtctctagtacgagaggaccgagacggactg2880
acctctggtgtgccagttgttccgccaggagcatggctggttggctacgtcgggtcgtga2940
taaccgctgaaagcatctaagcgggaagcacgcttcaagatgagggttcccacagatgaa3000
tctggtaaggcccccgagagatgatcgggttgataggccggacgtggacgcactgcaagg3060
tgcggagctgaccggtactaataggccgagggcttgccccaca3103
<210>16
<211>2335
<212>dna
<213>propionibacteriumacidi-propionici
<220>
<221>cds
<222>(1)..(1500)
<400>16
atgaacacggccgagtatctggtcggcgacggcgagtcgggatcggtg48
metasnthralaglutyrleuvalglyaspglygluserglyserval
151015
gcggtggtggactccgacggggtgcacagctatgccgacctccgcgcg96
alavalvalaspseraspglyvalhissertyralaaspleuargala
202530
caggcgtcgaggtgccgtgccgccctcgaggaactcgggttgcgcgcc144
glnalaserargcysargalaalaleuglugluleuglyleuargala
354045
ggcgaccgcgtcgggctgatcggcgcgaactgcttcggctgggtggcc192
glyaspargvalglyleuileglyalaasncyspheglytrpvalala
505560
gcctacctgggggtgctggccgccggcatggtcgccgtgccgatcgcc240
alatyrleuglyvalleualaalaglymetvalalavalproileala
65707580
cataccctgcgccccgacgagatcgtctccagaatccggtggctcggc288
histhrleuargproaspgluilevalserargileargtrpleugly
859095
gtccaggccgccttcctgggcccgatggagtcgcgccgactcgccgga336
valglnalaalapheleuglyprometgluserargargleualagly
100105110
caggtcccggggggtcttcctatggccgccgagggtccgtcgccggcg384
glnvalproglyglyleuprometalaalagluglyproserproala
115120125
ggccccccgctgcgattcgtcgatcggcccgccgatctggacgccgcc432
glyproproleuargphevalaspargproalaaspleuaspalaala
130135140
ctggtgttcacctcgggcaccaccggccggccgcacgtcgtgcggctc480
leuvalphethrserglythrthrglyargprohisvalvalargleu
145150155160
acccacgccaacctgcaggccaacaccggctccatcctgagctatctg528
thrhisalaasnleuglnalaasnthrglyserileleusertyrleu
165170175
ccgctggcagattcggatcgagtccttgtcgtgctgcccttcagctac576
proleualaaspseraspargvalleuvalvalleuprophesertyr
180185190
gtcttcggggcctccctgctgcacacccacctgagggtgggagcggcc624
valpheglyalaserleuleuhisthrhisleuargvalglyalaala
195200205
ctggtggtgcagccgaacgccgccttcccccagcagatggtcgagcgg672
leuvalvalglnproasnalaalapheproglnglnmetvalgluarg
210215220
atggccgccgagcgatgcaccggcctcgcgggggtgccctccaccttc720
metalaalagluargcysthrglyleualaglyvalproserthrphe
225230235240
tcggtgctgctgcgcaacagcaccttcggctcccgtcggctgcccgac768
servalleuleuargasnserthrpheglyserargargleuproasp
245250255
ctgcgcatcatccagcaggccggcggcaggctcgcgccgacgatggtc816
leuargileileglnglnalaglyglyargleualaprothrmetval
260265270
gaggagttgcgcggggcccagccccaggcgcaggtgttcgtgatgtac864
glugluleuargglyalaglnproglnalaglnvalphevalmettyr
275280285
gggcagaccgaggccaccgcgcggctgtcctacctgccgccccaggag912
glyglnthrglualathralaargleusertyrleuproproglnglu
290295300
ctggatcgccgtccggggtcgatcggccggggaataccgggagtggac960
leuaspargargproglyserileglyargglyileproglyvalasp
305310315320
ctgcgcgtggtcggcgaggacggctcgcaggtcgccgccggacagatc1008
leuargvalvalglygluaspglyserglnvalalaalaglyglnile
325330335
ggcgagatcgtggccggcggggacaatatctcgcccggctacctcgac1056
glygluilevalalaglyglyaspasnileserproglytyrleuasp
340345350
gaccccgaggagaccgcccggcggatgcccggaggggtactgcggacc1104
aspproglugluthralaargargmetproglyglyvalleuargthr
355360365
ggcgatctggcgacggtcgacgaggacggttacatctacgtcgtcgat1152
glyaspleualathrvalaspgluaspglytyriletyrvalvalasp
370375380
cgcagggaggacttcatcaagtcgtggggggtgaggatctcgagtcag1200
argarggluasppheilelyssertrpglyvalargilesersergln
385390395400
gacatcgaggcggtggcgctgcagctcaccgatctcgtgtcggtggct1248
aspileglualavalalaleuglnleuthraspleuvalservalala
405410415
gcggtgggagtgcccgacgaggccgccggggagcgggtcgagttggtg1296
alavalglyvalproaspglualaalaglygluargvalgluleuval
420425430
gccgtgccccgggaggggtcgcggctcaccgagggccagatcatcgac1344
alavalproarggluglyserargleuthrgluglyglnileileasp
435440445
cactgccgggcccggctggctaggaccatggtgccgcaggccgtgcat1392
hiscysargalaargleualaargthrmetvalproglnalavalhis
450455460
cttgtcccgttgatgcccctcaacggcaacggtaaaatctccaagaca1440
leuvalproleumetproleuasnglyasnglylysileserlysthr
465470475480
gcggtccgcgaactctgcgtggatctggccagagccgaccagtcccca1488
alavalarggluleucysvalaspleualaargalaaspglnserpro
485490495
ggaggatcatcatgaggcggaaaccagtcgtcgtcctgatcgcaggcgtcct1540
glyglyserser
500
cgcggtggtggtcgccagcggcgcatgggtgctgcagggcaggcccgacccgctgccctc1600
ctcggtggtgaacacggccgccggcgacgacgagctcgccgatttcagcgggcggaggat1660
cttcttcggtcaccagtccgtcggggcgaacatcatcgatggtctgaaagccgcctattc1720
gggccgggagggctccggcctcgacgttgtcgagacgcgcaccgatccaggcaggagcgg1780
cggatacctggcccatgccgcgatgggggtcaacggcgacccgctcggcaaactcgccga1840
tttcgagaaggtgctggccggctcgatgggcggcgcggtcgacctcgccgtgctcaagct1900
gtgctacatcgacgtcacggccgacaccgacgtcgacgcgctgttcacggcgtactcgca1960
gaccatgaccaggctcgaggccgcccatccgggggtcaccttcatctacacgacagtccc2020
gctgaccaccgaccggacctggaagcagaccgtcaagtcgtggatcgggcgcgacgagca2080
gacggggccggacgacaacgccgcccgccagcgttacaaccggctcgtgcgcgagcggta2140
cggcgattccgacaggctcttcgacatcgccgcggtccaggccacgatggacagctcccc2200
gacctcgcggacccgtgacggatccacctactacgtccttcacgaccggctggcggccga2260
ccccgggccctcaacgcgctggggtcacgggtgaccgccgcccgcctcgtgcacctggtg2320
gccgcacagcactag2335
<210>17
<211>500
<212>prt
<213>propionibacteriumacidi-propionici
<400>17
metasnthralaglutyrleuvalglyaspglygluserglyserval
151015
alavalvalaspseraspglyvalhissertyralaaspleuargala
202530
glnalaserargcysargalaalaleuglugluleuglyleuargala
354045
glyaspargvalglyleuileglyalaasncyspheglytrpvalala
505560
alatyrleuglyvalleualaalaglymetvalalavalproileala
65707580
histhrleuargproaspgluilevalserargileargtrpleugly
859095
valglnalaalapheleuglyprometgluserargargleualagly
100105110
glnvalproglyglyleuprometalaalagluglyproserproala
115120125
glyproproleuargphevalaspargproalaaspleuaspalaala
130135140
leuvalphethrserglythrthrglyargprohisvalvalargleu
145150155160
thrhisalaasnleuglnalaasnthrglyserileleusertyrleu
165170175
proleualaaspseraspargvalleuvalvalleuprophesertyr
180185190
valpheglyalaserleuleuhisthrhisleuargvalglyalaala
195200205
leuvalvalglnproasnalaalapheproglnglnmetvalgluarg
210215220
metalaalagluargcysthrglyleualaglyvalproserthrphe
225230235240
servalleuleuargasnserthrpheglyserargargleuproasp
245250255
leuargileileglnglnalaglyglyargleualaprothrmetval
260265270
glugluleuargglyalaglnproglnalaglnvalphevalmettyr
275280285
glyglnthrglualathralaargleusertyrleuproproglnglu
290295300
leuaspargargproglyserileglyargglyileproglyvalasp
305310315320
leuargvalvalglygluaspglyserglnvalalaalaglyglnile
325330335
glygluilevalalaglyglyaspasnileserproglytyrleuasp
340345350
aspproglugluthralaargargmetproglyglyvalleuargthr
355360365
glyaspleualathrvalaspgluaspglytyriletyrvalvalasp
370375380
argarggluasppheilelyssertrpglyvalargilesersergln
385390395400
aspileglualavalalaleuglnleuthraspleuvalservalala
405410415
alavalglyvalproaspglualaalaglygluargvalgluleuval
420425430
alavalproarggluglyserargleuthrgluglyglnileileasp
435440445
hiscysargalaargleualaargthrmetvalproglnalavalhis
450455460
leuvalproleumetproleuasnglyasnglylysileserlysthr
465470475480
alavalarggluleucysvalaspleualaargalaaspglnserpro
485490495
glyglyserser
500
<210>18
<211>5205
<212>dna
<213>propionibacteriumacidi-propionici
<400>18
ttttcattggagagtttgatcctggctcaggacgaacgctggcggcgtgcttaacacatg60
caagtcgaacggtaaggccctttcgggggtacacgagtggcgaacgggtgagtaacacgt120
gagtaacctgcccacttcttcgggataacgctaggaaactggtgctaataccggatatga180
gctcctgccgcatggtgggggttggaaagtgtttgtggtggtggatggactcgcggccta240
tcagcttgttggtgaggtagtggctcaccaaggcggtgacgggtagccggcctgagaggg300
tgaccggccacattgggactgagatacggcccagactcctacgggaggcagcagtgggga360
atattgcacaatgggcggaagcctgatgcagcaacgccgcgtgcgggatgacggccttcg420
ggttgtaaaccgctttcaccaggggcgaaggcatccttttggggtgttgacggtacctgg480
agaagaagcaccggctaactacgtgccagcagccgcggtgatacgtagggtgcgagcgtt540
gtccggatttattgggcgtaaagggctcgtaggcggttgatcgcgtcggaagtgaaaact600
tggggcttaaccctgagcgtgctttcgatacgggttgacttgaggaaggtaggggagaat660
ggaattcctggtggagcggtggaatgcgcagatatcaggaggaacaccagtggcgaaggc720
ggttctctggacctttcctgacgctgaggagcgaaagcgtggggagcaaacaggcttaga780
taccctggtagtccacgctgtaaacggtgggtactaggtgtggggtccattccacggatt840
ccgtgccgtagctaacgcattaagtaccccgcctggggagtacggccgcaaggctaaaac900
tcaaaggaattgacggggccccgcacaagcggcggagcatgcggattaattcgatgcaac960
gcgaagaaccttacctgggtttgacatggattggtaacggtcagagatggccgcccccct1020
tgtgggccggttcacaggtggtgcatggctgtcgtcagctcgtgtcgtgagatgttgggt1080
taagtcccgcaacgagcgcaaccctcgtccactgttgccagcatttggttggggactcag1140
tggagaccgccggggtcaactcggaggaaggtggggatgacgtcaagtcatcatgcccct1200
tatgtccagggcttcacgcatgctacaatggccggtacaaagagtggcgacatcgtgagg1260
tggagcgaatctcagaaagccggtctcagttcggattggggtctgcaactcgaccccatg1320
aagtcggagtcgctagtaatcgcagatcagcaacgctgcggtgaatacgttcccggggct1380
tgtacacaccgcccgtcaagtcatgaaagtcggtaacacccgaagccggtggcccaacac1440
gttctgcgtgggggagtcgtcgaaggtgggactggtaattaggactaagtcgtaacaagg1500
tagccgtaccggaaggtgcggctggatcacctcctttctaaggagctttctggaagccgg1560
tcgtcgcccgagtgtggtgatggttcggttcaggctgtcgggtccttgcgggtccggtgg1620
tgcttctgagtggaatgttggctatggacgcttctggtcttcgggtcgggggtgggatgc1680
actgttggggttctggggtatcacctgcacaggtggtggcctggtgtggcggacatcgct1740
gccggctggcctggttgtcgggttggtgtggtgggtgtctcgtgtggtggttgagaactg1800
tacagtggatatgagcatctttgtagattttttgtaatgtgttgtgttgcaagctactaa1860
gtgcggtcggtggatgccttggcaccaagagccgatgaaggacgttgtaacctgcgataa1920
gccccggggagttggttcacgagctgtgatccgggggtgtccgaatggggaaaccttgaa1980
ttgccggagtcatgtccggtgaccctgccctgaatgtataggggtgtgggagggaacgtg2040
gggaagtgaaacatctcagtacccgcaggaagagaaaacaatatgtgattccgtgagtag2100
tggcgagcgaaagcggatgaggccaaaccgtgtgtgtgttcaaaccggcaggtgttgcac2160
gtgcggggttgtggggtcttctgggatcgactgccgtcggtccgtccagtgataaatggt2220
gtgttgaagtcgaagcgtctgggaaggcgtaccggagtgggtgagagtcccgtagacgtg2280
gatgcaccactggtggaggataccccaagtagcgcgggactcgtggaatttcgcgtgaat2340
ctggcgggaccacccgtcaagcctgaatactccttggtgaccgatagtggatagtaccgt2400
gagggaatggtgaaaagtaccccgggaggggagtgaaagagtacctgaaaccggccgcat2460
acaatccgtcggagcctgcctgtacgggtgggtgacggcgtgcctattgaagaatgagcc2520
tgcgagttagtggcatgtggcgaggttaacccgtgtggggtagtcgtagcgaaagcgagt2580
ccgataagggcgccagtcgcgtgttctagacccgaagcggtgtgatctatccatggccag2640
gatgaagcgacggtaagacgtcgtggaggtccgcacccacttcagttgaaaatggagggg2700
atgagctgtggataggggtgaaaggccaatcaaacactgtgatagctggttctccccgaa2760
atgcatttaggtgcagcgtcgcgtggttcttgctggaggtagagcactggatgatctagg2820
gggcctatcagcttaccgaaatcagccaaactccgaatgccggcaagtggagcgtggcag2880
tgagacggcgggggataagcttcgtcgtcgagagggaaacagcccagatcatcagctaag2940
gcccctaagcggtggctaagtggaaaaggatgtggagttgcggtgacaaccaggaggttg3000
gcttggaagcagccatccttgaaagagtgcgtaatagctcactggtcaagtgattccgcg3060
ccgacaatgtagcggggctcaagccatccgccgaagctgtggcaatcaggatttactcct3120
ggttgggtaggggagcgtcgtgttctcggtgaagcggtccggtgacgggtcgtggagggg3180
atgcgagtgagaatgcaggcatgagtagcgaatgacgggtgagaaacccgtccgccgaat3240
atccaagggttccagggtcaagttaatcttccctgggtgagtcgggacctaaggcgaggc3300
cgacaggcgtagtcgatggacgaccagttgatattctggtaccggtgtagcaccgtccgt3360
gtcgaggtgtgtgatgctaagcatgcgagtccctgttccgcgggcctttggtctgtgggg3420
tgggtggtgagtgtgtgaaccgatcatgtagtaggcaagctgcggagggacgcagggagg3480
tagctcaaccccagcgatggttgtctggggctaaacgtgtggaccgtccggtaggtaaat3540
ccgccgggcatgatggttgaggcgtgatggcgagcccactgtgtgggtgagtgagtgatc3600
ctgtactgccgagaaaagcttcgtgagcgaggtgtgagccgcccgtaccctaaaccgaca3660
ctggtggattggtagagtataccgaggcgatcgagagaatcatggtgaaggaactcggca3720
aaatgaccccgtaacttcgggataaggggtgcccgaaccgtccggctgtttactggctgg3780
gggcggtgagggtcgcagagtccagggggaaacgactgtttactaaaaacacaggtccgt3840
gcgaagttgtaagacgatgtatacggactgactcctgcccggtgctggaaggttaagggg3900
aactgtcagggccttcgggttcgaagcggtgaacttaagccccagtaaacggcggtggta3960
actataaccatcctaaggtagcgaaattccttgtcgggtaagttccgacctgcacgaatg4020
gagtaacgatttccctactgtctccaccatgaactcggtgaaattgcattacgagtaaag4080
atgctcgttacgcgcagcaggacggaaagaccccgggacctttactatagtttggtattg4140
gtgatcggtgcgacttgtgtaggataggtgggagacggtgaagcggccacgccagtggtt4200
gtggagtcattgttgaaataccactctggtcgttctggttacctcacctcggaccgtgat4260
ccggttcagggacagtgcctgatgggtagtttgactggggcggtcgcctcctaaaaggta4320
acggaggcgcccaaaggttccctcagcctggttggtaatcaggtgtcgagtgtaagtgca4380
caagggagcttgactgtgagacaggcatgtcgagcagggacgagagtcgggactagtgat4440
ctgacggtggcttgtggaagtgccgtcactcaacggataaaaggtaccccggggataaca4500
ggctgatcttgcccgagcgctcacagcgacggcatggtttggcacctcgatgtcggctcg4560
tcgcatcctggggctggagtcggtcccaagggttgggctgttcgcccattaaagcggcac4620
gcgagctgggtttagaacgtcgtgagacagttcggtccctatccgctgcgcgcacaggaa4680
tcttgagaagagctgtctctagtacgagaggaccgagacggactgacctctggtgtgcca4740
gttgttccgccaggagcatggctggttggctacgtcgggtcgtgataaccgctgaaagca4800
tctaagcgggaagcacgcttcaagatgagggttcccacagatgaatctggtaaggccccc4860
gagagatgatcgggttgataggccggacgtggacgcactgcaaggtgcggagctgaccgg4920
tactaataggccgagggcttgccccacacaccatcatcgacaattacgcgcatatccact4980
gtacggctcccggccaccacacccccccacacgggggctggtgcgccacacatgtgagtc5040
acaacacaatacgagttccgtctggaacaccccaagattctggtgttccggtggccatgg5100
ctggagggaaacacccggtcccattccgaacccggaagttaagcctccacacgctgatgg5160
tactgcccacgccagtgggtgggagagtaagacgccgccggaaca5205
<210>19
<211>471
<212>dna
<213>propionibacteriumacidi-propionici
<220>
<221>cds
<222>(1)..(471)
<400>19
gtgtcctcgggccacgtctcctcccagtcggagctcggcgcctccctg48
valserserglyhisvalserserglnsergluleuglyalaserleu
151015
gcggccgaaggaatggcggtgtcccaggggaccctgtcacgggacctg96
alaalagluglymetalavalserglnglythrleuserargaspleu
202530
gtggagatcggcgcggtgcgcggacgggacaagaacgggaacccctgc144
valgluileglyalavalargglyargasplysasnglyasnprocys
354045
tacaccatccctgagggggagcatccgtccgacgtgacgaccggatca192
tyrthrileprogluglygluhisproseraspvalthrthrglyser
505560
ccggcatggagcagactggcccgcctcacccgcgaactgtgcaccggc240
proalatrpserargleualaargleuthrarggluleucysthrgly
65707580
gtccagcacaatgacaccctcgtcgtcctgaagaccccgccgggggcc288
valglnhisasnaspthrleuvalvalleulysthrproproglyala
859095
gcgcagtacttcggttcggcgatcgaccgttccggatcgagggcgatc336
alaglntyrpheglyseralaileaspargserglyserargalaile
100105110
ctgggaacgatagccggggacgacacgatcgccctcatctgcgccgcg384
leuglythrilealaglyaspaspthrilealaleuilecysalaala
115120125
gaggtgagcgcggacgcgctggccgacgcgttcaggcagatggccgag432
gluvalseralaaspalaleualaaspalapheargglnmetalaglu
130135140
accggcttcccggcgccactgctgtccgaggggagctga471
thrglypheproalaproleuleusergluglyser
145150155
<210>20
<211>156
<212>prt
<213>propionibacteriumacidi-propionici
<400>20
valserserglyhisvalserserglnsergluleuglyalaserleu
151015
alaalagluglymetalavalserglnglythrleuserargaspleu
202530
valgluileglyalavalargglyargasplysasnglyasnprocys
354045
tyrthrileprogluglygluhisproseraspvalthrthrglyser
505560
proalatrpserargleualaargleuthrarggluleucysthrgly
65707580
valglnhisasnaspthrleuvalvalleulysthrproproglyala
859095
alaglntyrpheglyseralaileaspargserglyserargalaile
100105110
leuglythrilealaglyaspaspthrilealaleuilecysalaala
115120125
gluvalseralaaspalaleualaaspalapheargglnmetalaglu
130135140
thrglypheproalaproleuleusergluglyser
145150155
<210>21
<211>469
<212>dna
<213>propionibacteriumacidi-propionici
<220>
<221>cds
<222>(1)..(153)
<400>21
gtgtcctcgggccacgtctcctcccagtcggagctcggcctccctggc48
valserserglyhisvalserserglnsergluleuglyleuprogly
151015
ggccgaaggaatggcggtgtcccaggggaccctgtcacgggacctggt96
glyargargasnglyglyvalproglyaspprovalthrglyprogly
202530
ggagatcggcgcggtgcgcggacgggacaagaacgggaacccctgcta144
glyaspargargglyalaargthrglyglngluarggluproleuleu
354045
caccatccctgagggggagcatccgtccgacgtgacgaccggatcaccg193
hishispro
50
gcatggagcagactggcccgcctcacccgcgaactgtgcaccggcgtccagcacaatgac253
accctcgtcgtcctgaagaccccgccgggggccgcgcagtacttcggttcggcgatcgac313
cgttccggatcgagggcgatcctgggaacgatagccggggacgacacgatcgccctcatc373
tgcgccgcggaggtgagcgcggacgcgctggccgacgcgttcaggcagatggccgagacc433
ggcttcccggcgccactgctgtccgaggggagctga469
<210>22
<211>51
<212>prt
<213>propionibacteriumacidi-propionici
<400>22
valserserglyhisvalserserglnsergluleuglyleuprogly
151015
glyargargasnglyglyvalproglyaspprovalthrglyprogly
202530
glyaspargargglyalaargthrglyglngluarggluproleuleu
354045
hishispro
50
pct/ro/134表
- 还没有人留言评论。精彩留言会获得点赞!