一种提高光滑球拟酵母酸耐受能力的方法与流程
本发明涉及一种提高光滑球拟酵母酸耐受能力的方法,属于生物工程领域。
背景技术:
中介体复合物(mediatorcomplex)是多亚基蛋白质复合物,是转录因子与rna聚合酶ii之间信息传递的桥梁。细胞中不同信号转导通路通过激发其控制的特异转录因子与中介体特定的亚基发生相互作用,调控下游基因的表达,进而影响细胞的生长及各种生理功能。
光滑球拟酵母(candidaglabrata)可以用于生产丙酮酸、富马酸和苹果酸等多种有机酸。在有机酸发酵过程中,c.glabrata会面临低ph胁迫。低ph环境对使微生物的胞内环境酸化,致使菌体生长受损、发酵产物的积累减缓甚至停止。目前主要通过外源添加辅助底物、诱变育种、基因工程和适应性进化等策略提高菌体的酸耐受性和有机酸产量。但这些策略存在增大产物提纯复杂度、工作量大和周期长等问题。通过中介体调控作用进行耐酸机制的研究是从基因转录水平上进行的研究,可从根本上指导解决这一问题,但此机制尚不清楚。因此,探索一种提高c.glabrata酸耐受机制的方法对提高有机酸产量具有指导意义。
技术实现要素:
本发明的第一个目的是提供一种基因工程菌,是以光滑球拟酵母为宿主,过表达seqidno.1所示的蛋白亚基。
在本发明的一种实施方式中,以py26为载体过表达所述蛋白亚基。
在本发明的一种实施方式中,所述光滑球拟酵母是光滑球拟酵母atcc55。
本发明的第二个目的是提供所述基因工程菌的构建方法,所述方法是扩展seqidno.1所示蛋白亚基的基因序列,与载体连接,转化至宿主细胞中。
在本发明的一种实施方式中,所述基因序列如seqidno.2所示。
在本发明的一种实施方式中,所述方法包括以下步骤:
(1)扩增seqidno.2所示cgmed15b基因序列,(2)通过t4连接酶将cgmed15b基因经酶切位点noti和sacii克隆到质粒py26上(3)将重组质粒py26-cgmed15b电击转化到c.glabrataatcc55。
本发明的第三个目的是提供一种提高c.glabrata酸耐受能力的方法,所述方法是在光滑球拟酵母中过表达中介体亚基med15b。
本发明还提供所述基因工程菌在有机酸生产领域的应用。
在本发明的一种实施方式中,所述应用包括以过表达seqidno.1所示中介体亚基的光滑球拟酵母为发酵微生物,发酵生产有机酸。
在本发明的一种实施方式中,所述方法是将所述光滑球拟酵母接种至添加40g/l的caco3的发酵培养基中,28~30℃,200~250rpm,培养40~72h。
在本发明的一种实施方式中,所述接种是接种经富集培养的种子液。
在本发明的一种实施方式中,所述方法是将菌株经种子培养基富集培养24h,转接至添加40g/l的caco3ph缓冲液的发酵培养基,30℃,200rpm,培养52h。
有益效果:本发明利用过表达中介体亚基med15b,使光滑球拟酵母在ph2.0条件下最终生物量提高59.2%,在ph2.0条件下,12h时细胞生长量较出发菌株增加73.8%,细胞膜酵母甾醇、粪甾醇和麦角甾醇的含量分别提高了970%、16.6%和94.5%,细胞膜完整性和流动性增强,并使丙酮酸发酵能力提高100%。
附图说明
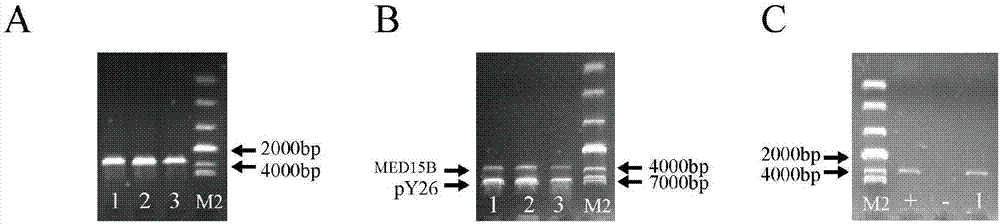
图1为基因过表达菌株的构建及验证,其中,a为目的基因的扩增结果,1~3表示cgmed15b基因扩增序列,m2表示dl10000dnamarker;b为过表达质粒双酶切验证,1~3表示过表达质粒,m2表示dl10000dnamarker;c为基因过表达菌株的验证,+表示质粒py26-cgmed15b为模板的阳性对照,-表示未转化入质粒py26-cgmed15b的c.glabrataatcc55菌落做模板的阴性对照,1表示以转化入质粒py26-cgmed15b的c.glabrataatcc55菌落做模板的过表达转化子;
图2为基因缺失菌株的构建及验证,a为敲除框的构建,b为基因缺失菌株的验证;其中,l表示目的基因的上游500bp的左臂,m表示敲除标记基因his3,r表示目的基因下游500bp的右臂,lm表示左臂和his3的融合片段,rm表示右臂和his3的融合片段,lmr表示左臂和右臂中间融合入his3的敲除框片段;-表示未加入dna模板的阴性对照;+表示加入c.glabrata野生型基因组为模板的阳性对照,1和2表示c.glabrata野生型基因组,3、4分别表示cgmed15b基因没有被敲除的菌株、cgmed15b基因成功被敲除的菌株。
图3为c.glabrata生长性能的测定,a不同ph下的平板生长实验,b不同ph下生长曲线测定,c细胞生长活性的测定;
图4为细胞膜性能测定结果,a细胞膜甾醇含量测定,b细胞膜完整性测定,c细胞膜流动性测定。
图5丙酮酸发酵能力测定结果;
图6为对照例实施方式下的基因工程菌生长性能。
具体实施方式
具体实施方式中涉及的测定方法:
(1)菌体平板生长情况记录:将待测菌株的单菌落接种于20ml的ynb(0.67%酵母提取物无氨基酸的基本氮源,2%葡萄糖,ph6.0)液体培养基中,将对数生长期的菌体经适当梯度稀释点种在ynb固体培养基,30℃培养并观察菌体的生长情况并拍照,具体方法参见2016年公开的《crz1pregulatesphhomeostasisincandidaglabratabyalteringmembranelipidcomposition》。
(2)生长曲线测定:将对数期的待测菌株接种于ph6.0和ph2.0的ynb液体培养基,初始od660=0.1,30℃摇床培养,每隔一定的时间取菌液,稀释适当倍数,660nm测定od值,绘制生长曲线;
(3)生长活性测定:将活化后的菌株接种于ph2.0的ynb液体培养基,初始od660=0.1,30℃摇床培养,每隔一定的时间取菌液,测定细胞的生长活性,方法参见2015年公开的《transcriptionfactorsasg1pandhal9pregulatephhomeostasisincandidaglabrata》。
(4)细胞膜甾醇含量测定:分别收集ynb培养基,ph6.0和ph2.0,30℃,200rpm,培养8h的酵母细胞。用pbs缓冲液洗涤三遍,然后冷冻干燥后备用。称取30mg冷冻干燥的酵母细胞,用于细胞膜甾醇提取,方法参见2016年公开的《转录因子crz1p调控光滑球拟酵母应答酸胁迫的生理机制》。
甾醇含量质谱分析方法:气相色谱—飞行时间质谱(tof)联用仪(waters,usa)。固定相:硅胶毛细管柱(30m×0.25mm,0.25μmdb-5msstationaryphase,j&wscientific,folsom,ca)。具体条件参见2009年公开的《单双倍体酵母细胞对乙醇响应的比较脂组学研究》。
(5)细胞膜完整性检测:分别收集ynb培养基,ph6.0和ph2.0,30℃,200rpm,培养8h的酵母细胞,pbs缓冲液洗涤细胞并调节菌体浓度至od660=0.5,pi标记,利用流式细胞仪检测细胞膜的完整性,方法参见2015年公开的《twonon-exclusivestrategiesemployedtoprotecttorulopsisglabrataagainsthyperosmoticstress》。
(6)细胞膜流动性检测:分别收集ynb培养基,ph6.0和ph2.0,30℃,200rpm,培养8h的酵母细胞,磷酸钾缓冲液洗涤并调节菌体浓度至od660=0.5,以tma-dph为荧光探针,采用荧光分光光度计检测细胞膜的流动性,方法参见2012年公开的《lactobacilluscaseicombatsacidstressbymaintainingcellmembranefunctionality》。
(7)丙酮酸发酵能力检测:将各菌株经种子培养基富集培养24h,转接至添加40g/l的caco3ph缓冲液的发酵培养基,30℃,200rpm,培养52h,利用高效液相色谱法检测丙酮酸的发酵能力,方法参见2016年公开的《crz1pregulatesphhomeostasisincandidaglabratabyalteringmembranelipidcomposition》。
实施例1:过表达菌株的构建
以c.glabrataatcc2001(以下简写为wt)基因组为模板,以p1/p2为引物,将扩增出的带有酶切位点noti和sacii的片段cgmed15b(图1a,3.3kb)。经双酶切后连接到质粒py26上,构建重组质粒py26-cgmed15b,双酶切验证py26质粒(7.2kb)、cgmed15b(3.3kb)这两个片段(图1b)。以c.glabrataatcc55(后文简写做htuδ)为出发菌株,将重组质粒py26-cgmed15b导入其感受态细胞,涂布mm筛选培养基,获得阳性转化子。出发菌株因缺失ura3基因不能在mm筛选培养基上生长,而质粒py26上的ura3基因可使过表达菌株获得这一生长能力。以p3/p4为引物,通过菌落pcr筛选验证发现,作为对照的出发菌株htuδ无条带产生,而阳性转化子扩增得到特异性片段(图1c,3.3kb),将此菌株命名为htuδ/cgmed15b。
p1:ataagaatgcggccgcatgtctagcaaagagaccattcc(noti)
p2:tccccgcggttactctatacttgtccagaaattcc(sacⅱ)
p3:aagttttctagaactagcgcg
p4:gtgtcaacaacgtatctaccaac
实施例2:敲除菌株的构建
以wt基因组为模板,分别以p5/p6、p7/p8、p9/p10、为引物,扩增出cgmed15b的左臂(以l表示)、右臂(以r表示)和his3基因(以m表示),经融合pcr构建敲除框cgmed15b-lmr(图2a)。将敲除框导入出发菌株htuδ,经同源重组获得标记基因his3替换掉目的基因cgmed15b的突变菌株。出发菌株htuδ因缺失his3基因不能在筛选培养基上生长,而突变菌株因标记基因his3表达而能在mm培养基上生长。以p11/p12为引物,对转化子进行菌落和基因组pcr验证,如图1b所示,发现出发菌株htuδ产生4.0kb左右基因片段,而转化子获得1.7kb左右的基因his3及基因cgmed15b的左右臂。将验证正确的突变菌株命名为med15bδ。
p5:cctgtgacagctaaatggaag
p6:accctcttaacaaacgccattgtttcattgtcagttgtgtattt
p7:acacaactgacaatgaaacaatggcgtttgttaagagggtt
p8:tcatccctgtcatttacagtctatgctaggacacccttagtgg
p9:ccactaagggtgtcctagcatagactgtaaatgacagggatgactt
p10:cgaaattaaaaacgacacaactc
p11:ccatgctaccactacaataacg
p12:tggattccttcccattctta
实施例3:突变菌的生长性能测定
通过平板生长情况记录、生长曲线和生长活性测定对med15b提高c.glabrata酸耐受性的功能进行评定:
平板生长实验分析不同ph条件对出发菌株htuδ、过表达菌株htuδ/cgmed15b和敲除菌株med15bδ酸耐受性能的影响,如图3a,发现以ph6.0为对照,ph值由6.0上升到8.0,c.glabrata的生长都会受到抑制,但相同ph条件下三株菌的生长情况大致相同;但随着ph由6.0降低到2.0,过表达med15b会小幅度促进菌株的生长,缺失med15b会显著抑制菌株的生长。进一步对菌体在ph2.0条件下的生长曲线进行测定,如图3b,发现过表达菌株的最终生物量较出发菌株提高59.2%,敲除菌株的最终生物量较出发菌株降低30.4%。ph2.0条件下,12h时三株菌之间的细胞活性测定如图3c,发现过表达菌株的细胞生长量较出发菌株增加73.8%,敲除菌株的细胞生长量较出发菌株降低55.9%。以上结果表明,缺失med15b会降低c.glabrata的生长能力和对酸性环境的耐受能力,过表达med15b能够提高c.glabrata的生长能力和对酸性环境的耐受能力。
实施例4:突变菌的细胞膜性能测定
通过测定细胞膜甾醇含量和细胞膜完整性、流动性,对c.glabrata细胞膜性能进行评价。
ph2.0时出发菌株htuδ、过表达菌株htuδ/cgmed15b和敲除菌株med15bδ细胞膜含量测定结果如图4a,与出发菌株htuδ相比,过表达菌株htuδ/cgmed15b细胞膜酵母甾醇、粪甾醇和麦角甾醇的含量分别提高了970%、16.6%和94.5%,敲除菌株med15bδ中酵母甾醇含量大量积累比出发菌株提高60.3倍,但是无法合成粪甾醇和麦角甾醇。对三株菌的细胞膜完整性测定结果如图4b,发现在ph2.0条件下,过表达med15b的菌株能够增强膜完整性,pi染色的过表达菌株细胞数比出发菌株降低30.2%,缺失med15b导致膜完整性损坏,pi染色的敲除菌株细胞数比出发菌株增多69.2%。对三株菌的细胞膜流动性测定结果如图4c,发现在ph2.0条件下,过表达med15b能够增强膜流动性,过表达菌株细胞膜流动性比出发菌株提高6.9%,缺失med15b使敲除菌株细胞膜流动性降低了11.6%。
实施例5:突变菌的丙酮酸发酵能力测定
通过测定丙酮酸含量,对c.glabrata丙酮酸发酵能力进行评价。
对出发菌株htuδ、过表达菌株htuδ/cgmed15b和敲除菌株med15bδ发酵52h时丙酮酸含量测定结果如图5,与出发菌株htuδ相比,过表达菌株htuδ/cgmed15b丙酮酸含量提高了100%,敲除菌株med15bδ中丙酮酸含量比出发菌株下降了39.7%。上述结果表明过表达med15b能够提高c.glabrata的丙酮酸发酵能力。
对照例1
采用实施例1-2相同的策略,区别在于,将seqidno.1所示序列替换为中介体亚基med3a(序列如seqidno.15)、med3b(序列如seqidno.16)。过表达med3a的菌株表示为htuδ/cgmed3a,缺失的菌株表示为med3aδ;过表达med3b的菌株表示为htuδ/cgmed3b,缺失的菌株表示为med3bδ;对相应的过表达和缺失突变株在酸性条件下的生长性能进行检测,结果显示如图6,发现亚基med3a、med3b的过表达和缺失对细胞在酸性条件下的耐受能力无明显影响。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
sequencelisting
<110>江南大学
<120>一种提高光滑球拟酵母酸耐受能力的方法
<160>16
<170>patentinversion3.3
<210>1
<211>1095
<212>prt
<213>人工序列
<400>1
metserserlysgluthrilepromethisglnargserglnasnval
151015
alagluleuleuthrvalleumetaspileasnlysileasnglygly
202530
aspserthrthralaglulysmetlysvalhisalalysserpheglu
354045
alaalaleupheglulysserserserlysgluglutyrglnlysthr
505560
metlysserlysileaspalametargserthrargasplysarglys
65707580
arggluservalglyseralasermetmetalaasnleuglyglnasp
859095
glythrasnasnasnasnasnasnasnasnasnasnasnasnasnleu
100105110
asnmetalaalaserphemetglyglyaspmetpheglyargasngln
115120125
serproalaglnasnserasnalaasnthrasnleuasnthrasnval
130135140
glyproglyvalasnglyproasnglyasnaspglythralaasnpro
145150155160
glnmetphemetasnglnglnalaglnalaargglnglnalaalaala
165170175
argglnleulysasnargglnmetglyglyserseralaglnglngln
180185190
glnleuthrglnglnglnglnglnleuleuasnglnmetargvalala
195200205
proileprolysgluleuleuglnargileproasnleuproprogly
210215220
valthrthrtrpgluglnvalthralaleualaglnglnasnargleu
225230235240
seralaglnaspmetserilealalysaspiletyrlysilehisgln
245250255
glntyrleuilelysalalysleuglnglnglnglnglnargglngln
260265270
glnglnargglnglnglyasnproaspvalasnasnasnmetalagly
275280285
serasnasnasnasnasnasnasnleuprometalaglnglnglnmet
290295300
glnglnargglnglnglnglnglnglnserglnglnglnglnasnarg
305310315320
asnproasnglnarghisasnvalleuserglnileasnglnmetphe
325330335
thralaaspgluglnargalaleuleuglnglualametglualacys
340345350
lysasnpheglnlysthrhispheglyglyglnmetseraspalaasn
355360365
lysglnalapheilelyslyspheileasnserlysalaleulyslys
370375380
leuglualametargmetalaglnglyglyasnasnasnalaasnleu
385390395400
asnlysglyglnalaaspmetleuglnargglnglnalaasnmetgln
405410415
metasnglnglnglnglnargalaalaglnasnglnargargglypro
420425430
valmetasnaspalavalserglnglytyrasnasnglnmetasnser
435440445
alaalaaspserthrmetasnasnserasnglnprometasnilegly
450455460
asnasnglyvalasnmetileproasnglnserglnglnglnglngln
465470475480
thrasnargprolysgluglnthrproglnglnproglnglnargile
485490495
glnserasnargservalprometleuasnprothrprogluaspval
500505510
gluvalvalargargileseralaglualaalalysthrglnleuarg
515520525
leuthraspleuthrasnserleuthrproglngluargaspgluile
530535540
lyslysargleuglnlysasnglnglnleuphealaglnvalserser
545550555560
tyralaproglnvaltyrleuphethrlyssergluserpheleulys
565570575
gluvalleuglnleuargilepheilelysgluileleuglulyscys
580585590
serlysglyiletyrvalvallysleuaspthrvalasplysleuval
595600605
ilelystyrglnlystyrtrpglusermetlysileglnleuleuarg
610615620
argglnglnleuleuglnglnglnglnglnglnglnglnglnglymet
625630635640
aspproasnargalaglnasnserglnglnglnglnglnglnasngln
645650655
alaasnmetglnglnalaargasnarglysprothrlysasnglnthr
660665670
thrproalailealaalaservalalametasnmetasnasplysgly
675680685
alasermetserproalaleuglnlysalaglyseralavalproasn
690695700
phealaglnglnmetserproasnmetthrproglythrilepropro
705710715720
thrasnvalleuserprohisserglnserhisileprometvalser
725730735
prothrmetalalysalaalaseralaalaalaleulysasnaspthr
740745750
alaserserserargargglyserthrlysproargglylysserthr
755760765
alaprovalthrglylyslysthrserasnalaprothrproglnval
770775780
valproalathrvalproserthrthrasnleuseralaalaglythr
785790795800
proasnileargasnlysseralathrproleuthralaglyleuser
805810815
prolysserthrileargserasnserasnthralaleualaserala
820825830
lysthrproserprometthrvalserileproglnproglyasnser
835840845
servalphelyslysglugluglutyrleuserlysleuglnleuarg
850855860
lysglugluileargpheargglnlysglnargleuaspileleuser
865870875880
serserprovalaspleupheleuthrthrvalalaaspcysleugly
885890895
ileasnaspglugluilegluleuileasnlysileprogluthrthr
900905910
alaaspasnileasnasnthrglylyslyslysleuthrlysalaala
915920925
glnlysleuargasplysgluileleuasnvalserileglnvalgly
930935940
glulysasplysleuilemetserserlysalaproasplysvalmet
945950955960
asptyrserileseralametserleualaalavalphelysasnleu
965970975
serserthrglyserleuasnasnilealaleuserglyserasnala
980985990
thrthrserlysaspileglyasniletyrserhisthrglyglyval
99510001005
lysarglyspheaspgluvalgluileserproasnserasngly
101010151020
serproseralaserilemetsergluserlyslysilelysile
102510301035
aspserprogluaspmetphevalthrhisserserglualaala
104010451050
lysglythrasnasnserserleumetaspserglylysglugly
105510601065
sercyslyssermetalaglyseralathrgluvalasnaspthr
107010751080
seriletrpasptrpasnphetrpthrserileglu
108510901095
<210>2
<211>3288
<212>dna
<213>人工序列
<400>2
atgtctagcaaagagaccattccaatgcaccagcggtctcagaatgtcgctgaactatta60
actgtcctcatggacatcaacaagattaatggcggcgactctaccaccgcagagaagatg120
aaagtgcacgcaaaaagcttcgaagccgcactgttcgagaaatcatcctccaaggaggag180
taccaaaagactatgaaatcgaaaattgacgcaatgagaagcacaagagataaacgcaag240
agagagtccgtgggttctgcctctatgatggcaaacctagggcaagatggtaccaataac300
aacaacaacaacaacaacaacaacaacaacaacctgaacatggccgctagcttcatgggt360
ggggatatgttcggaagaaatcagtcacctgctcagaacagtaacgcgaacactaactta420
aacacaaacgttgggcccggcgtaaacggccctaatggaaacgacggcaccgcgaacccc480
cagatgttcatgaaccaacaggcgcaggcaagacaacaagctgcagccagacaactaaaa540
aacagacagatgggaggctcatctgcacagcaacagcaacttacgcaacagcaacagcag600
ctgctaaatcagatgcgtgtagcaccaattccaaaagagctactccaacgtattccaaac660
ttaccaccaggtgtcaccacttgggagcaagttactgccttggctcaacaaaacagacta720
tcagcacaggatatgagcattgctaaagatatctataagatacatcagcagtatctgatc780
aaagcgaagttgcaacagcagcagcaacgccaacagcaacaacgtcaacagggaaaccct840
gacgtgaataataatatggccggatctaacaacaataataacaataatctgcccatggct900
cagcagcaaatgcaacagagacagcagcagcagcaacagtcacagcagcaacagaacaga960
aaccctaaccaaagacataacgtgctatcacagataaaccaaatgttcaccgcagatgag1020
caaagagctctattgcaagaagctatggaggcttgtaaaaacttccagaagactcacttc1080
ggtggtcaaatgtctgatgctaacaagcaggcttttatcaagaagttcataaattcaaag1140
gctttgaagaaactagaagctatgagaatggctcaaggtggaaacaataatgctaatttg1200
aataaagggcaagccgatatgctacagagacagcaagctaacatgcaaatgaaccagcaa1260
caacaaagagcagctcagaaccaacgtagaggtccagtaatgaacgatgctgttagtcaa1320
gggtataacaaccaaatgaactctgccgctgattccacaatgaataactctaaccagcca1380
atgaatattggaaataatggtgttaatatgataccaaaccaatctcagcagcagcagcaa1440
acaaatagaccaaaggagcaaaccccacagcaacctcagcagcgtatacagtcaaacaga1500
tctgtaccgatgcttaacccaacgcctgaggatgtagaggttgtacgaaggatatctgca1560
gaagccgccaagactcaactaagactcactgatttaactaactctttaactcctcaagaa1620
agagatgaaataaagaagagacttcaaaagaatcagcagttatttgcccaagttagtagt1680
tatgcacctcaagtttacctcttcactaagagtgaaagctttttgaaggaagttttacaa1740
ttgaggatctttatcaaagagattctagagaagtgctccaagggaatctatgtagtcaag1800
cttgatactgtagataaattggttatcaaatatcagaagtattgggaaagcatgaaaatt1860
cagttgttgagaagacagcaattattacagcagcagcaacaacaacaacaacaaggcatg1920
gaccctaacagagctcaaaactctcaacagcaacagcaacagaaccaggcaaatatgcaa1980
caggcacgtaatagaaagccaactaaaaaccaaaccactcctgctattgcagctagtgtt2040
gcaatgaacatgaatgacaagggcgcttcaatgtcacccgctttacaaaaggctggatct2100
gctgtaccgaactttgcacaacaaatgtcacctaacatgacaccaggaaccatccctcca2160
acaaatgtgttatcacctcactctcaaagccatataccaatggtgtctccaacgatggca2220
aaagcagcatctgctgcagccttaaagaatgacactgctagtagcagtcgccgtggaagc2280
acaaaacctagaggcaagtcaactgcaccggttactggtaaaaagacatctaatgcacca2340
acaccccaagttgttcctgctaccgtcccaagcacaacaaacttgtcggctgccggtaca2400
ccaaatatcaggaataaatcagctaccccattaactgcaggcttgtcaccaaagtccacg2460
attcgttccaattctaacacagcattagcttctgcaaaaactccatcaccaatgactgta2520
tccattccacaacctgggaatagctcggttttcaagaaagaagaggagtatttgtctaag2580
ttacaattaagaaaagaagagattcgattccgccaaaagcagagactagatattctaagt2640
agttctccggttgatttatttttgactactgttgctgattgtttgggtattaatgatgaa2700
gaaattgaattgataaacaaaataccagaaaccactgctgacaacattaataatacaggc2760
aagaagaaattaactaaagctgctcaaaagctaagagacaaggagatattgaatgtgtcg2820
atacaggttggtgaaaaggataaacttatcatgtcgagcaaagcacctgataaggttatg2880
gactactccattagtgcaatgtcgctggcggcagttttcaaaaacttatctagtacaggt2940
agcctgaataatatcgcactttctggcagtaatgcaactacttccaaggacatcggaaat3000
atctattcacacacaggtggtgtgaaaagaaaatttgatgaagttgagattagtccaaat3060
tctaatggatctccttctgcttcgataatgagtgaatcaaagaaaattaaaattgactca3120
cctgaagacatgtttgtaacgcattcttctgaagccgccaaaggcactaacaattcatca3180
ctgatggattcaggtaaagaaggctcctgcaagagtatggctggttccgcaactgaggtc3240
aatgacactagcatttgggattggaatttctggacaagtatagagtaa3288
<210>3
<211>39
<212>dna
<213>人工序列
<400>3
ataagaatgcggccgcatgtctagcaaagagaccattcc39
<210>4
<211>35
<212>dna
<213>人工序列
<400>4
tccccgcggttactctatacttgtccagaaattcc35
<210>5
<211>21
<212>dna
<213>人工序列
<400>5
aagttttctagaactagcgcg21
<210>6
<211>23
<212>dna
<213>人工序列
<400>6
gtgtcaacaacgtatctaccaac23
<210>7
<211>21
<212>dna
<213>人工序列
<400>7
cctgtgacagctaaatggaag21
<210>8
<211>44
<212>dna
<213>人工序列
<400>8
accctcttaacaaacgccattgtttcattgtcagttgtgtattt44
<210>9
<211>41
<212>dna
<213>人工序列
<400>9
acacaactgacaatgaaacaatggcgtttgttaagagggtt41
<210>10
<211>43
<212>dna
<213>人工序列
<400>10
tcatccctgtcatttacagtctatgctaggacacccttagtgg43
<210>11
<211>46
<212>dna
<213>人工序列
<400>11
ccactaagggtgtcctagcatagactgtaaatgacagggatgactt46
<210>12
<211>23
<212>dna
<213>人工序列
<400>12
cgaaattaaaaacgacacaactc23
<210>13
<211>22
<212>dna
<213>人工序列
<400>13
ccatgctaccactacaataacg22
<210>14
<211>20
<212>dna
<213>人工序列
<400>14
tggattccttcccattctta20
<210>15
<211>477
<212>prt
<213>人工序列
<400>15
metalathrlysglnglugluglnalaasnleuseraspvalleuthr
151015
prosermetserleuthrgluleuglumetlysphealaaspgluthr
202530
aspglylysalalysaspvalvalglnalaargilelyslysalaglu
354045
aspglyileleuproleuargleuglnpheasnaspphethrglnile
505560
metserserleuaspglugluargtyralaasnvalserlysglnglu
65707580
lyspheglnmetileargserlysvalleuglyleuthrgluargleu
859095
glngluleuserasnasppheglugluleuglnproleuphealathr
100105110
valglyglutyrserlysthrtyrlysasnlysasnpheglnvalleu
115120125
gluasnleualasertyrasnhisargglylysalaglyalaserile
130135140
serasnserthrprothrproalaalaalathrprothrthralapro
145150155160
thrproglyalaglythrlyslysalaalalysthralaprothrpro
165170175
thralathralathrileglythrproserasnasnalaprothrpro
180185190
alathrthralathrthrproglythrglnalalyslysproarglys
195200205
proargglnthrlyslysglnglnglnalaalaalaalaalaalaala
210215220
valalaglnalaglnalaglnalaglnalaglnalaglnasnglnasn
225230235240
glnasnasnmetglnasnlysasnileserasnproglymetasnser
245250255
asnmetglythrprovalmetglyasnproasnmetlysglnmetgln
260265270
serproileproalaasnalametasnasnmetasnvalprohisasn
275280285
glyalametargproservalproasnglyasnmetglyasnproser
290295300
metglyasnleuasnmetasnalaproasnmetglyasnproasnmet
305310315320
asnasnproasnmetasnasnproasnalametmetserprometala
325330335
glyglnglyglnleuasnglnmetpheprometglnasnhisasngln
340345350
asnglyhisphemetglyglnglnserproglyproasnileglygln
355360365
metglnpheproproasnasnglyasnmetasnglymetproglythr
370375380
seraspmetasnleuglymetasnprosermetasnmetasnmetgly
385390395400
metasnmetasnglnilethrproalaasnileleusermetasnthr
405410415
lysglylysaspaspglnmetglnasnileglymetaspglnasngln
420425430
asnglnasnglnasnglnasnglnasnglnsermetasnmetasnmet
435440445
asnasnaspserasnasnprolysseralatyraspleuvalaspphe
450455460
asnserleuaspleuserserleuasnmetasppheleu
465470475
<210>16
<211>481
<212>prt
<213>人工序列
<400>16
metalathrlysglnglugluglnalaasnleuseraspvalleuthr
151015
prosermetserleuthrgluleuglumetlysphealaaspgluthr
202530
aspglylysalalysaspvalvalglnalaargilelyslysalaglu
354045
aspglyileleuproleuargleuglnpheasnaspphethrglnile
505560
metserserleuaspglugluargtyralaasnvalserlysglnglu
65707580
lyspheglnmetileargserlysvalleuglyleuthrgluargleu
859095
glngluleuserasnasppheglugluleuglnproleuphealathr
100105110
valglyglutyrserlysthrtyrlysasnlysasnpheglnvalleu
115120125
gluasnleualasertyrasnhisargglylysalaglyalaserile
130135140
serasnserthrprothrproalaalaalathrprothrthralapro
145150155160
thrproglyalaglythrlyslysalaalalysthralaprothrpro
165170175
thralathralathrileglythrproserasnasnalaprothrpro
180185190
alathrthralathrthrproglythrglnalalyslysproarglys
195200205
proargglnthrlyslysglnglnglnalaalaalaalaalaalaala
210215220
valalaglnalaglnalaglnalaglnalaglnalaglnasnglnasn
225230235240
glnasnasnmetglnasnlysasnileserasnproglymetasnser
245250255
asnmetglythrprovalmetglyasnproasnmetlysglnmetgln
260265270
serproileproalaasnalametasnasnmetasnvalprohisasn
275280285
glyalametargproservalproasnglyasnmetglyasnproser
290295300
metglyasnleuasnmetasnalaproasnmetglyasnproasnmet
305310315320
asnasnproasnmetasnasnproasnalametmetserprometala
325330335
glyglnglyglnleuasnglnmetpheprometglnasnhisasngln
340345350
asnglyglnphemetglyglnglnserproglyproasnileglygln
355360365
metglnpheproproasnasnglyasnmetasnglymetproglythr
370375380
seraspmetasnleuglymetasnprosermetasnmetasnmetgly
385390395400
metasnleuasnglnilethrproalaasnileleusermetasnthr
405410415
lysglylysaspaspglnmetglnasnileglymetaspglnasngln
420425430
asnglnasnglnserglnasnglnserglnasnglnasnglnsermet
435440445
asnmetasnmetasnasnaspserasnasnprolysseralatyrasp
450455460
leuvalasppheasnserleuaspleuserserleuasnmetaspphe
465470475480
leu
- 还没有人留言评论。精彩留言会获得点赞!