新型脂肪酸脱羧酶及其用途的制作方法
本发明涉及酶学领域,特别涉及脂肪酸脱羧酶。
发明背景
烷烃和烯烃是化石燃料的主要组分,但它们也天然存在于植物、昆虫、蓝细菌和一些细菌中。还已知硅藻和集落绿藻纲(chlorophyceae)布朗葡萄藻(botryococcusbraunii)产生具有超过21个碳的烷(烯)烃。
大多数烷(烯)烃生物合成途径涉及活化的脂肪酸(酰基-acp或酰基-coa)转化为醛中间体并最终脱羰基为烷(烯)烃。这些反应由多种酶催化。在植物中,通过两种同源推定的氧化还原酶eceriferum1(cer1)和eceriferum3(cer3)的作用,由脂肪酸产生27至33个碳的非常长链烷烃(bernard等,2012,theplantcell,24(7)),3106-18;bourdenx等,2011,plantphysiology,156(1),29-45)。在昆虫中,21至37个碳的角质层烷(烯)烃的合成涉及酰基-酯还原酶和细胞色素p450(qiu等,2012,pnas,109,14858-14863)。在蓝细菌中,该途径由酰基-acp还原酶和醛去甲酰化加氧酶组成(li等,2012,biochemistry,51(40),7908-16;rude等,2011,appliedandenvironmentalmicrobiology,77(5),1718-27)。只有细菌中半乳糖球菌属(jeotgalicoccusspp)和假单胞菌属(pseudomonassp)能够通过游离脂肪酸的直接脱羧产生烃(grant等2015,journaloftheamericanchemicalsociety,137(15),4940-3;rui等,2014,pnas,111,18237-18242;rude等,2011,appliedandenvironmentalmicrobiology,77(5),1718-27)。然而,这些细菌脂肪酸脱羧酶能产生烯烃(带有末端不饱和度)但不能产生烷烃。在微藻中,已知没有催化烃合成的酶。已有尝试去纯化微藻布朗葡萄藻的烷烃合成系统,并且已经表明通过钴-卟啉酶(cobalt-porphyrinenzyme)使脂肪醛中间体脱羰进行该合成(dennis和kolattukudy,1992,pnas,89(12),5306-10)。然而,这种蛋白质从未被鉴定。
因此,对于适合于由脂肪酸产生烷烃/烯烃的酶任仍然有强烈的需求。
发明简述
本发明涉及用于合成烷烃和烯烃的酶的新类别的鉴定,其在微藻中鉴定。这是首次在这些微生物中鉴定出烷烃合成酶。所述酶通过脱羧催化游离脂肪酸转化为烷烃和/或烯烃。所述酶属于存在于原核生物和真核生物中的fad依赖性蛋白质的超家族(gmc(glucose-methanol-choline,葡萄糖-甲醇-胆碱)氧化还原酶),并且其包括多种酶(主要是醇氧化酶)。到目前为止,从生物化学观点来看,没有从微藻中鉴定出gmc氧化还原酶,并且没有其他gmc氧化还原酶超家族成员被鉴定为烷烃合成酶。因此,这是一种应用于生物燃料、绿色化学、诊断和营养的用于合成烷烃和烯烃的新酶。
因此,本发明涉及将多肽或表达所述多肽的细胞在由脂肪酸产生烷烃和/烯烃中的用途,其中所述多肽具有脂肪酸脱羧酶活性并且包含与seqidno1具有至少40%相同性的序列。
优选地,所述多肽包含共有序列g-x-l-(x)4-c-[d/e]-x-g-[a/g]-f-x-[k/r](seqidno4),x是任何氨基酸。可选地或另外地,seqidno1中的c372、r391、y406、q426、h512和n515位中的一个、两个、三个、四个、五个或六个氨基酸是保守的。可选地或另外地,位置388-428之间的至少40%的氨基酸残基是疏水性残基,所述疏水性残基选自由v、i、l、m、f、w、ca和y组成的组。
在更具体的实施方案中,所述多肽包含或基本上由选自以下的氨基酸序列组成:seqidno1-3和5-14以及与它们中的一个具有至少80%相同性的氨基酸序列。
优选地,所述多肽来自藻类,优选微藻或蓝细菌。
优选地,所述脂肪酸和相应的脱羧烷烃和/烯烃包含8至24个碳原子,优选12至22个碳原子。
任选地,所述脂肪酸和相应的脱羧烷烃和/烯烃被一个或几个官能团取代和/或中断。优选地,所述脂肪酸和相应的脱羧烷烃和/烯烃被一个或多个基团取代,所述基团例如羟基、c1-c3醇、c1-c3酰基、c1-c3酯、c1-c3胺、氨基酸基团、c1-c3酰胺、羧基、醛、环氧基、卤素、c1-c3烷氧基、c1-c3硫代烷基、c1-c3亚胺、腈、含硫基团(如c1-c3硫基砜或c1-c3亚砜)、硫醇、硝基、氰基、c1-c3卤代烷基,或可被杂原子如o、n或s、炔基基团、醚(如二乙烯基醚基团)、或者是氧代基团中断。
本发明还涉及由脂肪酸产生烷烃和/烯烃的方法,其中如上定义的多肽与脂肪酸接触并在多肽的fad辅因子存在下进行光照。优选地,所述光的波长在300-540nm之间,更优选在400-520nm之间。例如,所述光是白光或包含蓝色或uv光子的任何光(例如从400到520nm)。
本发明进一步涉及重组宿主细胞,其包含编码如上定义的异源多肽的核酸序列。优选地,所述宿主细胞是细菌、微藻、丝状真菌或酵母。
在第一个实施方案中,所述异源多肽与硫酯酶共表达,优选在微藻、蓝细菌或大肠杆菌中。在另一个实施方案中,所述异源多肽与脂肪酶共表达,优选在细菌或微藻中。
本发明涉及由脂肪酸产生烷烃和/烯烃的方法,其中培养根据本发明的重组宿主细胞并回收所述烷烃和/烯烃。
此外,本发明涉及将如上定义的多肽用于样品中游离脂肪酸的定量(dosage)的用途。更具体地,用于在样品中定量(dosing)游离脂肪酸的方法包括(a)在适于将脂肪酸转化为烷烃/烯烃的条件下,使样品与本发明的脂肪酸脱羧酶接触,(b)回收烷烃/烯烃和/或二氧化碳;和(c)量化烷烃/烯烃和/或co2。可选地,用于在样品中定量游离脂肪酸的方法包括(a)在适于将脂肪酸转化为烷(烯)烃的条件下,使样品与本发明的脂肪酸脱羧酶接触,和(b)测量由所述脂肪酸脱羧酶发出的荧光。任选地,所述方法包括使样品与脂肪酶接触的前置步骤,特别是在适于将脂肪酸转化为游离脂肪酸的条件下。
本发明还涉及将如上定义的多肽用于脂肪酸的脱羧的用途。
发明详述
本发明人鉴定新的一类酶,其能够通过脂肪酸脱羧活性将游离脂肪酸转化为烷烃和烯烃。
定义
约:当在本文中使用时,“约”表示多于或少于10%,优选多于或少于5%。例如,约100意味着在90和110之间,优选地在95和105之间。
编码序列:术语“编码序列”是指多核苷酸,其直接指定多肽的氨基酸序列。编码序列的边界通常由开放阅读框确定,其以起始密码子(如atg,gtg或ttg)开始,并以终止密码子(如taa,tag或tga)结束。编码序列可以是基因组dna,cdna,合成dna或其组合。遗传密码子能针对宿主细胞进行优化。
控制序列:术语“控制序列”是指表达编码本发明酶的多核苷酸所必需的核酸序列。控制序列相对于编码此酶的多核苷酸可以是天然的(即来自相同的基因)或异源的(即来自不同的基因和/或不同的物种)。优选地,控制序列是异源的。众所周知的控制序列并且本领域技术人员目前使用的将是优选的。此类控制序列包括但不限于前导序列,多腺苷酸化序列,前肽序列,启动子,信号肽序列和转录终止子。控制序列至少包括启动子和转录及翻译终止信号。可以向控制序列提供接头,从而达到引入特定的限制性位点,以促进控制序列与编码该酶的多核苷酸的编码区的连接的目的。控制序列和编码序列的功能性组合能称为表达盒。
表达:术语“表达”包括涉及多肽的产生的任何步骤,包括但不限于转录、转录后修饰、翻译、翻译后修饰和分泌。
表达载体:术语“表达载体”是指线性或环状dna分子,其包含编码本发明酶的多核苷酸,并且与为其表达而提供的控制序列可操作地连接。此外,表达载体包含适于表达本发明酶的表达盒。
分离的:术语“分离的”是指在以自然界中不存在的形式或自然界中不存在的环境中的物质。分离的物质的非限制性实例包括(1)任何非天然存在的物质,(2)任何物质,包括但不限于任何酶、变体、核酸、蛋白质、肽或辅因子,其至少部分地从与其天然相关的一种或多种或所有天然存在的成分中除去;(3)任何相对于在自然界中发现物质的人工修饰的物质;或(4)通过增加相对于与其天然相关的其他组分的物质的量来修饰的任何物质(例如,编码该物质的基因的多个拷贝;使用比编码该物质基因天然相关的启动子的更强启动子)。
重组体:重组体是指通过基因工程产生的核酸构建体、载体和蛋白质。
异源的:在宿主细胞、载体或核酸构建体的上下文中,其指的是通过基因工程引入宿主细胞、载体或核酸构建体的酶的编码序列。在宿主细胞的上下文中,其意味着酶的编码序列源自与其所引入的细胞不同的来源,或者酶的编码序列来自与其所引入的细胞相同但由于其环境不是天然存在的而被认为是异源的物种,例如因为它在不是其天然启动子的启动子的控制下,或者在不同于其天然位置的位置引入。
核酸构建体:术语“核酸构建体”是指单链或双链的核酸分子,其被修饰或合成为以天然本来不存在的方式包含核酸区段,所述核酸分子包含一个或多个控制序列。
可操作地连接:术语“可操作地连接”是指这样的结构,其中以控制序列指导编码序列的表达的方式,将控制序列置于相对于编码序列的适当位置。
过表送:术语过表达是指在细胞中表达或导致将表达核酸或多肽,其浓度比通常在相应的野生型细胞中表达的更高。
序列相同性:两个氨基酸序列之间的序列相同性由参数“序列相同性”描述。出于本发明的目的,两个氨基酸序列(a)和(b)之间的“百分比相同性”是通过比较窗口,比较以最佳方式比对的两个序列来确定。所述序列的比对能通过众所周知的方法进行,例如使用针对needleman-wunsch的全局比对的算法。蛋白质分析软件使用分配给多种取代、缺失和其它修饰(包括保守氨基酸取代)的相似性测量,来匹配相似的序列。一旦获得总比对,通过将序列(a)和(b)之间比对的相同氨基酸残基的总数除以两者间最长序列中包含的残基总数,能够获得相同性百分比。
通常使用序列分析软件确定序列相同性。为了比较两个氨基酸序列,能够使用例如用于蛋白质成对序列比对的工具“embossneedle”,其通过embl-ebi提供并在以下可获得:www.ebi.ac.uk/tools/services/web/toolform.ebi?tool=emboss_needle&context=protein,使用默认设置:(i)matrix:blosum62,(ii)gapopen:10,(iii)gapextend:0.5,(iv)outputformat:pair,(v)endgappenalty:false,(vi)endgapopen:10,(vii)endgapextend:0.5。
可选地,序列相同性通常也能使用序列分析软件clustalomega运用hhalign算法及其默认设置作为其核心比对引擎来确定。该算法在
氨基酸:本文定义的氨基酸序列由单字母符号表示,如下所示:a,ala,(丙氨酸);r,arg,(精氨酸);n,asn,(天冬酰胺);d,asp,(天冬氨酸);c,cys,(半胱氨酸);q,gln,(谷氨酰胺);e,glu,(谷氨酸);g,gly,(甘氨酸);h,his,(组氨酸);i,ile,(异亮氨酸);l,leu,(亮氨酸);k,lys,(赖氨酸);m,met,(蛋氨酸);f,phe,(苯丙氨酸);p,pro,(脯氨酸);s,ser,(丝氨酸);t,thr,(苏氨酸);w,trp,(色氨酸);y,tyr,(酪氨酸);和v,val,(缬氨酸)。
通过“基本组成”是指多肽具有指示的seqidno,并且能进一步包含不超过20个氨基酸,优选不超过10个氨基酸的改变(即取代、插入和/或缺失)。特别地,多肽可以具有不超过11、10、9、8、7、6、5、4、3、2或1个氨基酸的改变,例如,可以具有1、2、3、4、5、6、7、8、9或10个氨基酸的取代、插入和/或缺失。取代是指用不同的氨基酸替换占据位置的氨基酸;缺失意味着去除占据位置的氨基酸;并且插入是指在占据位置的氨基酸的附近和紧随其后添加氨基酸。取代能是保守取代。保守取代的实例在以下的组内:碱性氨基酸(精氨酸,赖氨酸和组氨酸)、酸性氨基酸(谷氨酸和天冬氨酸)、极性氨基酸(谷氨酰胺和天冬氨酸)、疏水性氨基酸(亮氨酸、异亮氨酸和缬氨酸)、芳香族氨基酸(苯丙氨酸,色氨酸和酪氨酸)和小氨基酸(甘氨酸、丙氨酸、丝氨酸、苏氨酸和蛋氨酸)。通常不改变比活性的氨基酸取代是本领域已知的并被例如由h.neurath和r.l.hill(1979,in,theproteins,academicpress,newyork)所描述。常见的取代如下:ala/ser、val/lle、asp/glu、thr/ser、ala/gly、ala/thr、ser/asn、ala/val、ser/gly、tyr/phe、ala/pro、lys/arg、asp/asn、leu/lle、leuaal,ala/glu和asp/gly。
可选地,氨基酸变化具有改变多肽理化性质的特性。例如,氨基酸改变可以改善多肽的热稳定性、改变底物特异性、改变最适ph等。多肽中的必需氨基酸能根据本领域已知的方法鉴定,例如定点诱变或丙氨酸扫描诱变(cunningham和wells,1989,science244:1081-1085)。在后一种技术中,在分子中的每个残基处引入单个丙氨酸突变,并且测试所得突变分子从l-酪氨酸产生4-hba的能力,以鉴定对分子活性起决定性的氨基酸残基。还参见,hilton等,1996,j.biol.chem.271:4699-4708。酶的活性位点或其它生物相互作用也能通过结构的物理分析来确定,例如通过诸如核磁共振、结晶学、电子衍射或光亲和标记技术,结合所推定的接触位点氨基酸的突变来确定。参见,例如,devos等,1992,science255:306-312;smith等,1992,j.mol.biol.224:899-904;wlodaver等,1992,febslett.309:59-64。必需氨基酸的身份也能从与相关多肽的比对中推断。
使用已知诱变、重组和/或改组(shuffling)的方法能制备和测试单个或多个氨基酸取代、缺失和/或插入,后续进行有关的筛选步骤,例如由以下所公开的那些,reidhaar-olson和sauer,1988,science241:53-57;bowieandsauer,1989,proc.natl.acad.sci.usa86:2152-2156;wo95/17413;或wo95/22625。能使用的其它方法包括易错pcr、噬菌体展示(例如,lowman等,1991,biochemistry30:10832-10837;美国专利号5,223,409;wo92/06204)和区域定向诱变(derbyshire等,1986,gene46:145;ner等,1988,dna7:127)。诱变/改组方法能与高通量、自动化筛选方法组合以检测由宿主细胞表达的克隆的、突变的多肽的活性(ness等,1999,naturebiotechnology17:893-896)。能从宿主细胞中回收编码活性多肽的突变dna分子,并使用本领域的标准方法快速测序。这些方法允许快速确定多肽中单个氨基酸残基的重要性。
保守:通过保守氨基酸意指将定义的序列与参考序列比对,并且对应于参考序列中所示位置的定义序列中的残基与参考序列中存在的残基相同。能通过任何可用的方法进行比对,并且特别是通过上面所公开的用于相同性鉴定的方法,更优选通过clustalomega进行。残基位置在参考序列中指示。
纯化或纯化的:如本文所用,术语“纯化”、“纯化的”或“提纯”是指通过例如隔离或分离从其环境中去除或分离分子。“充分纯化的”分子至少约60%不含,优选至少约75%不含,和更优选至少约90%不含与它们相关的其它组分。如本文所用,这些术语还指从样品中去除污染物。例如,污染物的去除能导致样品中烷烃/烯烃的百分比增加。例如,当在宿主细胞中产生烷烃/烯烃时,能通过去除宿主细胞蛋白来纯化烷烃/烯烃。纯化后,样品中烷烃/烯烃的百分比增加。术语“纯化”,“纯化的”和“提纯”不需要绝对纯度。它们是相对的术语。因此,例如,当在宿主细胞中产生链烯时,纯化的烷烃/烯烃是充分与其它细胞组分(例如,核酸,多肽,脂质,碳水化合物或其他烃)分离的烷烃/烯烃。在另一个实例中,纯化的烷烃/烯烃制剂是其中烷烃/烯烃充分不含污染物(例如在发酵后可能存在的污染物)的制剂。在一些实施方案中,当样品的重量至少约50%由烷烃/烯烃组成时,烷烃/烯烃是纯化的。在其它实施方案中,当样品的重量至少约60%、70%、80%、85%、90%、92%、95%、98%或99%或更多由烷烃/烯烃组成时,链烯是纯化的。
脂肪酸脱羧酶活性:
“脂肪酸脱羧酶活性”是指从脂肪酸中去除羧酸基团,特别是在没有醛中间体且不引入末端不饱和度而直接去除。脂肪酸脱羧酶活性能通过本领域技术人员可用的任何方法测量。更优选地,活性能通过以下方法测量。
在体外酶促测定中,反应在密封的小瓶中进行,小瓶中含有溶解在适当溶剂(乙醇或二甲基亚砜)中的100nmol的底物(通常为c16:0游离脂肪酸),作为内参标准的2nmolc16烷烃,和具有500μl活性缓冲液(50mmtris-hclph8.2,含100mmnacl)的5至10μg纯化的酶(含有其fad辅因子),或总蛋白质提取物。将小瓶在25℃下在白光(或任何含有蓝光子的光)存在下,在2000μmole.photon.m-2.s-1下在旋转搅拌器上以250rpm孵育。然后将浓缩的氢氧化钠(10μl,10m)注入小瓶中以停止反应,并将样品在冰上冷却。用己烷提取烃并通过与火焰离子化检测器和质谱联用的气相色谱法(gc-fid-ms)进行量化。
脂肪酸脱羧酶多肽:
本发明涉及具有脂肪酸脱羧酶活性的多肽,在本文中称为“脂肪酸脱羧酶”,并且包含,基本上由或由与seqidno1,seqidno5或seqidno7,优选seqidno1,具有至少40%相同性的序列所组成。
例如,所述脂肪酸脱羧酶包含,基本上由或由与seqidno1,seqidno5或seqidno7,优选seqidno1,具有至少42%、45%、50%或55%相同性的序列所组成。
在另一方面,所述脂肪酸脱羧酶包含,基本上由或由与seqidno1-3和5-14任一个序列具有至少55、55、60、65、70、75、80、85、90、95或99%相同性的序列组成。
在另一方面,所述脂肪酸脱羧酶包含,基本上由或由seqidno1-3和5-14任一个的序列组成。在一个优选的实施方案中,所述脂肪酸脱羧酶包含,基本上由或由seqidno1、5、7-14任一个的序列所组成,更优选的为seqidno为1、5和7的任一个。
在一个实施方案中,所述脂肪酸脱羧酶包含共有序列g-x1-l-(x)4-c-[d/e]-x2-g-[a/g]-f-x3-[k/r/s/e](seqidno4),x是任意氨基酸(seqidno26)。在优选的实施方案中,所述脂肪酸脱羧酶包含共有序列g-x1-l-(x)4-c-[d/e]-x2-g-[a/g]-f-x3-[k/r](seqidno4),x是任意氨基酸。
优选地,x1能选自由p、l和g组成的组中。优选地,(x)4可以更具体地为[t/a]-[t/s/c]-[p/t/a]-[g/a]。优选地,x2可以选自由h、n和r组成的组中。优选地,x3可以是疏水性氨基酸,尤其选自由l、va和f组成的组中。
在一个具体实施方案中,所述脂肪酸脱羧酶包含共有序列g-x1-l-(x)4-c-[d/e]-x2-g-[a/g]-f-x3-[k/r/s/e](seqidno26),其中
-x1可以选自由p、l和g组成的组中;
-(x)4可以更具体地为[t/a]-[t/s/c]-[p/t/a]-[g/a];
-x2可以选自由h、n和r组成的组中;以及
x3可以是疏水性氨基酸,尤其选自由l、va和f组成的组中。
在一个非常具体的实施方案中,所述脂肪酸脱羧酶包含共有序列g-x1-l-(x)4-c-[d/e]-x2-g-[a/g]-f-x3-[k/r](seqidno4),其中
-x1可以选自由p、l和g组成的组中;
-(x)4可以更具体地为[t/a]-[t/s/c]-[p/t/a]-[g/a];
-x2可以选自由h、n和r组成的组中;以及
-x3可以是疏水性氨基酸,尤其选自由l、va和f组成的组中。
在另一个或另外的优选实施方案中,所述脂肪酸脱羧酶包含参考seqidno1相关的保守氨基酸。基于seqidno1的参考序列,seqidno1中残基c372、sr391、y406、q426、h512和n515中的一个、两个、三个、四个、五个或所有是保守的。在另一种脂肪酸脱羧酶中对应于seqidno1中的这些位置的位置上的相应残基也是如此。
在另一个或另外的优选实施方案中,所述脂肪酸脱羧酶包含形成疏水通道的区域,其中底物、游离脂肪酸能够进入。该区域位于seqidno1的第391至426位残基之间。因此,位置388-428之间的氨基酸残基的至少40%是疏水残基,优选选自由v、i、l、m、f、w、c、a和y组成的组。
所述脂肪酸脱羧酶有任何来源,例如来自细菌或藻类。
优选地,所述脂肪酸脱羧酶来自藻类,优选微藻或蓝细菌。例如,所述脂肪酸脱羧酶是具有脂肪酸脱羧酶活性的gmc氧化还原酶,其来自小球藻(chlorella)、衣藻(chlamydomonas)、褐指藻(phaeodactylum)、胶球藻(coccomyxa)、团藻(volvox)、褐藻(ectocarpus)、圆石藻(emiliania)、褐潮藻(aureococcus)、爱尔兰海藻(chondrus)、红藻(galdieria)或微拟球藻(nannochloropsis)。许多微藻的物种是已知的,并且能在例如数据库algaebase(www.algaebase.org/)上找到。
在一个具体实施方案中,所述脂肪酸脱羧酶来自变异小球藻(chlorellavariabilis),特指变异小球藻nc64a。在另一个具体实施方案中,所述脂肪酸脱羧酶来自莱茵衣藻(chlamydomonasreinhardtii)。在另一个具体实施方案中,所述脂肪酸脱羧酶来自三角褐指藻(phaeodactylumtricornutum)。在另一个具体的实施方案中,所述脂肪酸脱羧酶来自胶球藻(coccomyxasubellipsoidea),特别是胶球藻c-169。在另一个具体实施方案中,所述脂肪酸脱羧酶来自强壮团藻(volvoxcarteri)。在另一个具体实施方案中,所述脂肪酸脱羧酶来自长囊水云(ectocarpussiliculosus)。在另一个具体的实施方案中,所述脂肪酸脱羧酶来自赫氏圆石藻(emilianiahuxleyi)。在另一个具体实施方案中,所述脂肪酸脱羧酶来自抑食金球藻(aureococcusaophagefferens)。在另一个具体的实施方案中,所述脂肪酸脱羧酶来自海洋富油微拟球藻。
优选地,所述脂肪酸脱羧酶的长度不超过约750、700、650或600个氨基酸残基。
本发明还涉及杂合多肽或融合多肽,其中如上定义的脂肪酸脱羧酶的氨基酸序列与另一多肽的区域的n末端或c末端融合。脂肪酸脱羧酶活性在杂合多肽或融合多肽中保留。优选地,另一多肽的区域在所述脂肪酸脱羧酶的n末端融合。可选地,另一多肽的区域在所述脂肪酸脱羧酶的c末端融合。用于产生融合多肽的技术是本领域已知的,并且包括连接编码酶的编码序列和另一多肽的添加区域,使得它们在表达框内并且融合多肽的表达在相同启动子和终止子的控制下。融合多肽也可以使用内含肽技术构建,其中融合多肽在翻译后产生(cooper等,1993,emboj.12:2575-2583;dawson等,1994,science266:776-779)。
可以选择融合多肽的添加区域以增强根据本发明的酶的稳定性,以促进来自细胞(例如细菌细胞或酵母细胞)的所述融合蛋白的分泌(例如n-末端疏水信号肽),或协助所述融合蛋白的纯化。更具体地,所述添加区域可以是用于所述酶的纯化或固定的标签。这种标签是本领域技术人员公知的,例如his标签(his6)、flag标签、ha标签(源自流感蛋白血细胞凝集素的表位)、麦芽糖结合蛋白(mpb)、myc标签(源自人原癌蛋白myc的表位)、strep标签或gst标签(小谷胱甘肽-s-转移酶)。所述添加区域可以是硫氧还蛋白。
融合多肽可以进一步包含酶和添加区域之间的切割位点。在分泌或纯化所述融合蛋白时,所述位点被切割而释放两种多肽。切割位点的实例包括但不限于公开在martin等,2003,j.ind.microbiol.biotechnol.3:568-576;svetina等,2000,j.biotechnol.76:245-251;rasmussen-wilson等,1997,appl.environ.microbiol.63:3488-3493;ward等,1995,biotechnology13:498-50;和contreras等,1991,biotechnology9:378-381;eaton等,1986,biochemistry25:505-512;collins-racie等,1995,biotechnology13:982-987;carter等,1989,proteins:structure,function,andgenetics6:240-24;和stevens,2003,drugdiscoveryworld4:35-48。例如,所述切割位点能是tev(烟草蚀纹病毒,tobaccoetchvirus)切割位点。其他切割位点是本领域技术人员所公知的。
在一个具体实施方案中,本发明涉及包含如上定义的组氨酸标签、硫氧还蛋白、切割位点和所述脂肪酸脱羧酶的多肽(例如参见seqidno3,此构建体具有小球藻脂肪酸脱羧酶)。
本发明进一步涉及重组核酸构建体或载体,其包含编码如上定义的脂肪酸脱羧酶的核酸序列。更具体地,所述核酸构建体或载体适合于表达所述脂肪酸脱羧酶。另外地,提供了包含含有编码如上定义的脂肪酸脱羧酶的核酸序列的核酸、重组核酸构建体或重组载体的重组宿主细胞。
编码脂肪酸脱羧酶的核酸和核酸构建体
本发明涉及编码本发明脂肪酸脱羧酶的多核苷酸。核酸可以是dna(cdna或gdna)、rna或两者的混合物。它可以是单链形式或双链形式或两者的混合物。它可以包含修饰的核苷酸,包括例如修饰的键、修饰的嘌呤或嘧啶碱基、或修饰的糖。它可以通过本领域技术人员已知的任何方法制备,包括化学合成、重组和诱变。
任选地,为了宿主细胞表达能优化编码序列。特别地,由于本发明的脂肪酸脱羧酶来自藻类,特别是微藻,编码酶的核酸序列可以针对细菌宿主(例如大肠杆菌,酵母宿主或甚至是与酶来源不同的微藻)进行优化。
本发明还涉及核酸构建体,其包含与一个或多个控制序列可操作地连接的编码根据本发明公开的脂肪酸脱羧酶的多核苷酸,所述控制序列在与控制序列相容的条件下在合适的宿主细胞中指导编码序列的表达。可以用多种方式操控多核苷酸以提供脂肪酸脱羧酶的表达。根据表达载体,在将多核苷酸插入载体之前对其进行操控可以是预期的或必需的。利用重组dna方法修饰多核苷酸的技术是本领域熟知的。
控制序列可包括被宿主细胞或体外表达系统识别的启动子,用于表达编码本发明的脂肪酸脱羧酶的多核苷酸。所述启动子含有介导脂肪酸脱羧酶表达的转录控制序列。所述启动子可以是在宿主细胞中显示转录活性的任何多核苷酸,包括突变的、截短的和杂合启动子,并且可以从编码与宿主细胞同源或异源的细胞外或细胞内多肽的基因获得。任选地,所述启动子能是诱导型的。任选地,所述启动子是强启动子,允许脂肪酸脱羧酶的过表达。任选地,所述启动子是强诱导型启动子。
细菌宿主细胞中合适启动子的实例是从以下获得的启动子:解淀粉芽孢杆菌(bacillusamyloliquefaciens)α-淀粉酶基因(amyq)、地衣芽孢杆菌α-淀粉酶基因(amyl)、地衣芽孢杆菌青霉素酶基因(penp)、嗜热脂肪芽孢杆菌(bacillusstearothermophilus)产麦芽糖淀粉酶基因(amym)、枯草芽孢杆菌左聚糖蔗糖酶基因(sacb)、枯草芽孢杆菌xyla和xylb基因、苏云金芽孢杆菌(bacillusthuringiensis)cryllla基因(agaisse和lereclus,1994,molecularmicrobiology13:97-107)、大肠杆菌lac操纵子、大肠杆菌trc启动子(egonetal.,1988,gene69:301-315)、天蓝色链霉菌(streptomycescoelicolor)琼脂水解酶基因(daga)和原核β-内酰胺酶基因(villa-kamaroff等,1978,proc.natl.acad.sci.usa75:3727-3731),以及tac启动子(deboer等,1983,proc.natl.acad.sci.usa80:21-25)。其它启动子描述于“usefulproteinsfromrecombinantbacteria”gilbert等,1980,scientificamerican242:74-94;和sambrook等,1989。串联启动子的实例公开在wo99/43835中。
丝状真菌宿主细胞中合适启动子的实例是从以下基因获得的启动子:构巢曲霉乙酰胺酶、黑曲霉(aspergillusniger)中性α-淀粉酶、黑曲霉酸稳定的α-淀粉酶、黑曲霉或泡盛曲霉(aspergillusawamori)葡糖淀粉酶(glaa)、米曲霉(aspergillusoryzae)taka淀粉酶、米曲霉碱性蛋白酶、米曲霉磷酸丙糖异构酶、尖孢镰刀菌(fusariumoxysporum)胰蛋白酶样蛋白酶(wo96/00787)、镰孢霉(fusariumvenenatum)淀粉转葡糖苷酶(wo00/56900)、镰孢霉daria(wo00/56900)、镰孢霉quinn(wo00/56900)、米赫根毛霉(rhizomucormiehei)脂肪酶、米赫根毛霉天冬氨酸蛋白酶、里氏木霉(trichodermareesei)β-葡萄糖苷酶、里氏木霉纤维二糖水解酶i、里氏木霉纤维二糖水解酶ii、里氏木霉内切葡聚糖酶i、里氏木霉内切葡聚糖酶ii、里氏木霉内切葡聚糖酶iii、里氏木霉内切葡聚糖酶iv、里氏木霉内切葡聚糖酶v、里氏木霉木聚糖酶i、里氏木霉木聚糖酶ii、里氏木霉β-木糖苷酶,以及na2-tpi启动子(来自曲霉(aspergillus)中性α-淀粉酶基因的修饰启动子,其中非翻译前导序列已被来自曲霉磷酸异构酶基因的非翻译前导序列替换);非限制性实例包括来自黑曲霉中性α-淀粉酶基因的修饰启动子,其中非翻译前导序列已被来自构巢曲霉或米曲霉磷酸丙糖异构酶基因的非翻译前导序列替换;以及以上的突变的、截短的和杂合的启动子。
在酵母宿主中,有用的启动子获自以下基因:酿酒酵母(saccharomycescerevisiae)烯醇酶(eno-1)、酿酒酵母半乳糖激酶(gal1)、酿酒酵母乙醇脱氢酶/甘油醛-3-磷酸脱氢酶(adh1、adh2/gap)、酿酒酵母磷酸丙糖异构酶(tpi)、酿酒酵母金属硫蛋白(cup1)和酿酒酵母3-磷酸甘油酸激酶。酵母宿主细胞中其它有用的启动子在romanos等,1992,yeast8:423-488中有所描述。
诱导型启动子能响应于例如光强度或温度高低,和/或能响应于特定化合物。诱导型启动子可以是,例如,激素响应性启动子(例如,蜕皮激素响应性启动子,如us6,379,945中所述)、金属硫蛋白启动子(us6,410,828),能响应于化学物质例如水杨酸、乙烯、硫胺素和/或bth(us5,689,044)等发病机理相关(pr)启动子或其某些组合。诱导型启动子也能响应于光或暗(us8,318,482;us5,750,385;us5,639,952)、金属(eukaryoticcell2:995-1002(2003))或温度(us5,447,858;abe等,plantcellphysiol.49:625-632(2008);shroda等,plantj.21:121-131(2000))。前述实施例不是限制可以使用的启动子或特异性启动子的类型。启动子序列能来自任何生物体,只要它在宿主生物体中有功能。在某些实施方案中,通过将已知的诱导型启动子的一个或多个部分或结构域,与能在宿主细胞中起作用(例如赋予在宿主物种中起作用的启动子的诱导能力)的不同启动子的至少一部分融合,形成诱导型启动子。
本领域技术人员将容易理解,能有效地将多种已知的启动子序列用于微藻物种。例如,通常用于驱动微藻中转基因表达的启动子包括花椰菜花叶病毒启动子35s(camv35s)的各种形式,其已用于甲藻(dinoflagellates)和绿藻(chlorophyta)(chow等,plantcellrep.,18:778-780,1999;jarvis和brown,curr.genet.,317-321,1991;lohuis和miller,plantj.,13:427-435,1998)。来自猿猴病毒的sv40启动子也已报道在几种藻类中具有活性(gan等,j.appl.phycol.,151345-349,2003;qin等,hydrobiologia398-399,469-472,1999)。来自衣藻的rbcs2(核酮糖二磷酸羧化酶,小亚基)的启动子(fuhrmann等,plantj.,19:353-361,1999)和psad(光系统i复合物的丰富蛋白;fischer和rochaix,febslett.581:5555-5560,2001)启动子也能是有用的。hsp70a/rbcs2和hsp70a/p2tub(微管蛋白)的融合启动子(schroda等,plantj.,21:121-131,2000)也可用于改善转基因的表达,其中hsp70a启动子当置于其它启动子的上游可用作转录激活因子。感兴趣的基因的高水平表达也能在例如硅藻物种中,在编码硅藻岩黄素-叶绿素a/b结合蛋白的fcp基因的启动子(falciatore等,mar.biotechnol,1:239-251,1999;zaslavskaia等,j.phycol.36:379-386,2000)或编码eustigmatophyteviolaxanthin-叶绿素a/b结合蛋白的vcp基因的启动子(参见美国专利号8,318,482)的控制下实现。如果需要,诱导型启动子能在转基因微藻中提供基因的快速且严格控制的表达。例如,编码硝酸还原酶的nr基因的启动子区域能用作这种诱导型启动子。nr启动子活性通常被铵抑制并且当铵被硝酸盐取代时被诱导(poulsen和kroger,febslett272:3413-3423,2005),因此当微藻细胞在铵/硝酸盐存在下生长时能关闭或开启基因表达。多种藻类启动子是已知的并且能够使用,包括在us2013/0023035;us2013-0323780,us2014-0154806;和us2014-0363892中公开的那些。
控制序列也可以是转录终止子,其被宿主细胞识别以终止转录。终止子可操作地连接到编码多肽的多核苷酸的3′-端。任何在宿主细胞中有功能的终止子可以用于本发明。
用于细菌宿主细胞的优选终止子获自以下基因:克劳氏芽孢杆菌(bacillusclausii)碱性蛋白酶(aprh)、地衣芽孢杆菌α-淀粉酶(amyl)、和大肠杆菌核糖体rna(rrnb)。
用于丝状真菌宿主细胞的优选终止子获自以下基因:构巢曲霉邻氨基苯甲酸合成酶、黑曲霉葡糖淀粉酶、黑曲霉α-葡糖苷酶、米曲霉taka淀粉酶、和尖孢镰刀菌胰蛋白酶样蛋白酶。
用于酵母宿主细胞的优选终止子获自以下基因:酿酒酵母烯醇酶、酿酒酵母细胞色素c(cyc1)和酿酒酵母甘油醛-3-磷酸脱氢酶。对于酵母宿主细胞其它有用的终止子在上述romanos等,1992中描述了。
控制序列还可以是启动子下游和基因编码序列上游的mrna稳定子(stabilizer)区域,其增加基因的表达。
合适的mrna稳定子区域的实例获自苏云金芽孢杆菌cryllla基因(wo94/25612),和枯草芽孢杆菌sp82基因(hue等,1995,journalofbacteriology177:3465-3471)。
控制序列也可以是前导序列,是对宿主细胞的翻译重要的mrna的非翻译区。前导序列与编码脂肪酸脱羧酶的多核苷酸的5′-端可操作地连接。可以使用在宿主细胞中有功能的任何前导序列。
丝状真菌宿主细胞的优选前导序列获自以下基因:米曲霉taka淀粉酶和构巢曲霉丙糖磷酸异构酶。
酵母宿主细胞的合适前导序列获自以下基因:酿酒酵母烯醇酶(eno-1)、酿酒酵母3-磷酸甘油酸激酶、酿酒酵母α-因子、和酿酒酵母乙醇脱氢酶/甘油醛-3-磷酸脱氢酶(adh2/gap)。
控制序列还可以是多聚腺苷酸化序列,可操作地连接到编码脂肪酸脱羧酶的多核苷酸的3′-端的序列,并且当转录时,被宿主细胞识别为将多聚腺苷残基添加到转录的mrna的信号。可以使用在宿主细胞中有功能的任何多聚腺苷酸化序列。
用于丝状真菌宿主细胞的优选多聚腺苷酸化序列获自以下基因:构巢曲霉邻氨基苯甲酸合成酶、黑曲霉葡糖淀粉酶、黑曲霉α-葡糖苷酶、米曲霉taka淀粉酶、和尖孢镰刀菌胰蛋白酶样蛋白酶。
用于酵母宿主细胞的有用的多聚腺苷酸化序列在guo和sherman,1995,mol.cellularbiol.15:5983-5990中有所描述。
控制序列还可以是信号肽编码区,其编码与脂肪酸脱羧酶n-末端相连接的信号肽,并指导脂肪酸脱羧酶进入细胞的分泌途径。多核苷酸编码序列的5′-端可以固有地含有信号肽编码序列,所述信号肽编码序列在翻译阅读框中与编码脂肪酸脱羧酶的编码序列的区段天然连接。可选地,编码序列的5′-端可含有外源于编码序列的信号肽编码序列。当编码序列不天然含有信号肽编码序列时,可需要外源信号肽编码序列。可选地,外源信号肽编码序列可以简单地取代天然信号肽编码序列,以增强多肽的分泌。然而,可以使用任何指导表达的多肽进入宿主细胞分泌途径的信号肽编码序列。信号肽也能是叶绿体转运肽、脂肪酸脱羧酶的叶绿体转运肽或任何其他叶绿体转运肽。
细菌宿主细胞的有效信号肽编码序列是从芽孢杆菌(bacillusncib11837)产麦芽糖淀粉酶、地衣芽孢杆菌枯草杆菌蛋白酶、地衣芽孢杆菌β-内酰胺酶、嗜热脂肪芽孢杆菌(bacillusstearothermophilus)α-淀粉酶、嗜热脂肪芽孢杆菌(bacillusstearothermophilus)中性蛋白酶(nprt、nprs、nprm)、和枯草芽孢杆菌prsa的基因获得的信号肽编码序列。其它信号肽在simonen和palva,1993,microbiologicalreviews57:109-137中有所描述。
丝状真菌宿主细胞的有效信号肽编码序列是获自以下基因的信号肽编码序列:黑曲霉中性淀粉酶、黑曲霉葡糖淀粉酶、米曲霉taka淀粉酶、特异腐质霉(humicolainsolens)纤维素酶、特异腐质霉内切葡聚糖酶v、柔毛腐质霉(humicolalanuginosa)脂肪酶、和米赫根毛霉天冬氨酸蛋白酶。
用于酵母宿主细胞的有用信号肽获自以下基因:酿酒酵母α-因子和酿酒酵母转化酶。其它有用的信号肽编码序列在上述romanos等,1992中有所描述。
还可以期望添加调节序列,其调节多肽相对于宿主细胞生长的表达。调节系统的实例是那些导致基因表达响应化学或物理刺激而开启或关闭的系统,包括调节化合物的存在。原核系统中的调节系统包括lac、tac和trp操纵子系统。在酵母中,可以使用adh2系统或gal1系统。在丝状真菌中,可以使用黑曲霉葡糖淀粉酶启动子、米曲霉takaα-淀粉酶启动子和米曲霉葡糖淀粉酶启动子。调节序列的其它实例是允许基因扩增的那些。在真核系统中,这些调节序列包括在甲氨蝶呤存在下扩增的二氢叶酸还原酶基因,以及在重金属存在下扩增的金属硫蛋白基因。在这些情况下,编码多肽的多核苷酸将与调节序列可操作地连接。
表达载体
本发明还涉及重组表达载体,其包含如上公开的核酸构建体、或编码本发明的脂肪酸脱羧酶的多核苷酸、启动子、和转录及翻译终止信号。多种核苷酸和控制序列可以连接在一起以产生重组表达载体,所述重组表达载体可以包括一个或多个方便的限制性位点,以允许在这些位点插入或取代编码脂肪酸脱羧酶的多核苷酸。可选地,可以通过将多核苷酸或包含所述多核苷酸的核酸构建体插入合适的表达载体中来表达多核苷酸。
在构建表达载体时,编码序列位于载体中,使得编码序列与适当的控制序列可操作地连接以进行表达。
重组表达载体可以是任何载体(例如质粒或病毒),其能方便地进行重组dna步骤并能引起多核苷酸的表达。载体的选择通常取决于载体与将被引入载体的宿主细胞的相容性。载体可以是线性或闭合的环状质粒。
载体可以是自主复制载体,即作为染色体外实体存在的载体,其复制独立于染色体复制,例如质粒、染色体外元件、微型染色体、或人工染色体。载体可以包含用于确保自我复制的任何元件。可选地,载体可以是当其被引入宿主细胞时整合到基因组中并与其整合进入的染色体一起复制的载体。此外,可以使用单个载体或质粒、或一起含有待引入宿主细胞基因组的总dna的两个或多个载体或质粒、或转座子。
载体优选含有一个或多个选择性标记,其允许对被转化的、被转染的、被转导的或类似的细胞的简单选择。选择性标记是一种基因,其产物提供杀菌性或病毒抗性、对重金属的抗性、对营养缺陷型的原养型等。
细菌选择性标记的实例是地衣芽孢杆菌或枯草芽孢杆菌基因或标记,其赋予抗生素抗性(例如氨苄青霉素、氯霉素、卡那霉素、新霉素、壮观霉素或四环素抗性)。用于酵母宿主细胞的合适标记包括但不限于ade2、his3、leu2、lys2、met3、trp1和ura3。用于丝状真菌宿主细胞的选择标记包括但不限于amds(乙酰胺酶)、argb(鸟氨酸氨基甲酰基转移酶)、bar(草丁膦乙酰转移酶)、hph(潮霉素磷酸转移酶)、niad(硝酸盐还原酶)、pyrg(orotidine)-5′-磷酸脱羧酶)、sc(硫酸盐腺苷转移酶)和trpc(邻氨基苯甲酸合成酶),以及它们的等价物。优选用于曲霉属(aspergillus)细胞的是构巢曲霉或米曲霉amds和pyrg基因以及吸水链霉菌(streptomyceshygroscopicus)基因。
载体优选含有允许载体整合到宿主细胞基因组中或载体在细胞中独立于基因组而自主复制的元件。
当整合于宿主细胞基因组中发生时,序列整合到基因组中可能依赖于同源或非同源重组。可选地,载体可以含有额外的多核苷酸,用于通过同源重组引导在精确位置整合入宿主细胞基因组染色体。为了增加在精确位置整合的可能性,整合元件应包含足够数量的核酸,例如100至10000个碱基对,400至10000个碱基对,以及800至10000个碱基对,其具有与相应的靶序列高度的序列相同性,以增强同源重组的可能性。整合元件可以是与宿主细胞基因组中的靶序列同源的任何序列。此外,整合元件可以是非编码或编码多核苷酸。另一方面,载体可以通过非同源重组整合到宿主细胞的基因组中。
对于自主复制,载体可以进一步包含复制起点,所述复制起点使载体能够在所讨论的宿主细胞中自主复制。复制起点可以是介导在细胞中起作用的自主复制的任何质粒复制子。术语“复制起点”或“质粒复制子”是指使质粒或载体能够在体内复制的多核苷酸。细菌复制起点的实例是以下的复制起点:允许在大肠杆菌中复制的质粒pbr322、puc19、pacyc177、和pacyc184,以及允许在芽孢杆菌中复制的pub110、pe194、pta1060,和pamβ1。用于酵母宿主细胞的复制起点的实例是2微米复制起点、ars1、ars4、ars1和cen3的组合、以及ars4和cen6的组合。用于丝状真菌细胞的复制起点的实例是ama1和ansi(gems等,1991,gene98:61-67;cullen等,1987,nucleicacidsres.15:9163-9175;wo00/24883)。能根据wo00/24883中公开的方法完成ama1基因的分离和包含该基因的质粒或载体的构建。
可以将多于一个拷贝的本发明多核苷酸插入宿主细胞中以增加多肽的产生。通过将至少一个额外的序列拷贝整合到宿主细胞基因组中,或通过和多核苷酸一起包含可扩增的选择性标记基因使细胞含有该选择性标记基因的扩增拷贝,能获得多核苷酸的拷贝数的增加,且因此能通过在合适的选择剂存在下培养细胞来选择多核苷酸的额外拷贝。
用于连接上述元件以构建本发明的重组表达载体的步骤是本领域技术人员熟知的(参见例如,上述的sambrook等,1989)。
宿主细胞
本发明涉及表达本发明中脂肪酸脱羧酶的重组宿主细胞,更具体地涉及工程化以产生烷烃/烯烃的重组宿主细胞。重组宿主细胞可表达内源性脂肪酸脱羧酶但具有它的过表达(例如通过强异源启动子控制表达和/或通过增加细胞中编码脂肪酸脱羧酶的基因数)。可选地,重组宿主细胞可以表达本发明中的异源脂肪酸脱羧酶。另外地或在另一个可选方案中,重组宿主细胞已经过基因工程改造以增加脂肪酸的产生或促进优选脂肪酸的产生。例如,宿主细胞可以是与产生脂肪酸脱羧酶的物种不同的细菌(包括光合细菌)、丝状真菌、酵母或微藻物种。
因此,本发明涉及重组宿主细胞,其包含编码根据本发明公开的脂肪酸脱羧酶的多核苷酸,其可操作地连接于一个或多个控制序列,所述控制序列指导本发明的脂肪酸脱羧酶的产生。编码根据本发明的脂肪酸脱羧酶的多核苷酸的构建体或载体引入宿主细胞中,使得构建体或载体维持为染色体整合体或如前所述的自我复制的染色体外载体。
术语“宿主细胞”包括由于在复制过程中发生突变而与亲本细胞不同的亲本细胞的任何后代。宿主细胞的选择在很大程度上取决于编码多肽的基因及其来源。
宿主细胞可以是用于重组产生本发明的脂肪酸脱羧酶的任何细胞,例如原核生物或真核生物。
原核宿主细胞可以是任何革兰氏阳性或革兰氏阴性细菌。革兰氏阳性细菌包括但不限于芽孢杆菌(bacillus)、梭菌(clostridium)、肠球菌(enterococcus)、地芽孢杆菌(geobacillus)、乳杆菌(lactobacillus)、乳球菌(lactococcus)、海洋杆菌(oceanobacillus)、葡萄球菌(staphylococcus)、链球菌(streptococcus)和链霉菌(streptomyces)。革兰氏阴性细菌包括但不限于弯曲杆菌(campylobacter)、大肠杆菌、黄杆菌(flavobacterium)、梭杆菌(fusobacterium)、螺杆菌(helicobacter)、泥杆菌(ilyobacter)、奈瑟球菌(neisseria)、假单胞菌(pseudomonas)、沙门氏菌(salmonella)和脲原体(ureaplasma)。细菌宿主细胞可以是任何芽孢杆菌(bacillus)细胞,包括但不限于嗜碱芽孢杆菌(bacillusalkalophilus)、解淀粉芽孢杆菌(bacillusamyloliquefaciens)、短杆菌(bacillusbrevis)、环状芽胞杆菌(bacilluscirculans)、克劳氏芽孢杆菌、凝结芽孢杆菌(bacilluscoagulans)、坚强芽孢杆菌(bacillusfirmus)、灿烂芽胞杆菌(bacilluslautu)、迟缓芽孢杆菌(bacilluslentus)、地衣芽孢杆菌、巨大芽胞杆菌(bacillusmegateriu)、短小芽胞杆菌(bacilluspumilus)、嗜热脂肪芽孢杆菌(bacillusstearothermophilus)、枯草芽孢杆菌和苏云金芽孢杆菌细胞。细菌宿主细胞也可以是任何链球菌(streptococcus)细胞,包括但不限于似马链球菌(streptococcusequisimilis)、酿脓链球菌(streptococcuspyogenes)、乳房链球菌(streptococcusuberis)、马链球菌(streptococcusequi)和兽疫链球菌(streptococcuszooepidemicus)细胞。细菌宿主细胞还可以是任何链霉菌(streptomyces)细胞,包括但不限于不产色链霉菌(streptomycesachromogenes)、除虫链霉菌(streptomycesavermitilis)、天蓝色链霉菌(streptomycescoelicolor)、灰色链霉菌(streptomycesgriseus)和浅青紫链霉菌(streptomyceslividans)细胞。在一个具体实施方案中,宿主细胞是蓝细菌。蓝细菌包括但不限于聚球藻属(synechococcus)、集胞藻属(synechocystis)、鱼腥藻(anabaena)、螺旋藻属(spirulina)的物种。
将dna引入芽孢杆菌细胞可以通过原生质体转化(参见例如chang和cohen,1979,mol.gen.genet.168:111-115)、感受态细胞转化(参见例如young和spizizen,1961,j.bacteriol.81:823-829,或dubnau和davidoff-abelson,1971,j.mol.biol.56:209-221)、电穿孔(参见例如,shigekawa和dower,1988,biotechniques6:742-751)或接合(参见例如,koehler和thorne,1987,j.bacteriol.169:5271-5278)来实现。将dna引入大肠杆菌细胞可以通过原生质体转化(参见例如,hanahan,1983,j.mol.biol.166:557-580)或电穿孔(参见例如dower等,1988,nucleicacidsres.16:6127-6145)来实现。将dna引入链霉菌细胞可以通过原生质体转化、电穿孔(参见例如,gong等,2004,foliamicrobiol.(praha)49:399-405)、接合(参见例如,mazodier等,1989,j.bacteriol.171:3583-3585)、或转导(参见例如,burke等,2001,proc.natl.acad.sci.usa98:6289-6294)来实现。将dna导入假单胞菌细胞可以通过电穿孔(参见例如,choi等,2006,j.microbiol.methods64:391-397)或缀合(参见例如,pinedo和smets,2005,appl.environ.microbiol.71:51-57)来实现。将dna引入链球菌细胞可以通过自然感受态(参见例如,perry和kuramitsu,1981,infect.immun.32:1295-1297)、原生质体转化(参见例如,catt和jollick,1991,microbios68:189-207)、电穿孔(参见例如,buckley等,1999,appl.environ.microbiol.65:3800-3804)、或接合(参见例如,clewell,1981,microbiol.rev.45:409-436)来实现。然而,本领域已知的用于将dna导入宿主细胞的任何方法皆能使用。
宿主细胞也可以是真核细胞,例如哺乳动物、昆虫、植物或真菌细胞。宿主细胞可以是真菌细胞。如本文所用的“真菌”包括子囊菌(ascomycota)门、担子菌(basidiomycota)门、壶菌(chytridiomycota)门、和接合菌(zygomycota)门以及卵菌(oomycota)门和所有有丝分裂真菌(如hawksworth等人,in,ainsworthandaisby′sdictionaryofthefungi,第8版,1995,cabinternational,universitypress,cambridge,uk中所定义的)。真菌宿主细胞可以是酵母细胞。如本文所用的“酵母”包括子囊孢子(ascosporogenous)酵母(内孢霉目(endomycetales))、产担子(basidiosporogenous)酵母和属于不完全菌纲(fungiimperfecti)(芽孢纲(blastomycetes))的酵母。由于酵母的分类可能在将来发生变化,为了本发明的目的,酵母可如biologyandactivitiesofyeast(skinner、passmore、和davenport,editors,soc.app.bacteriol.symposiumseriesno.9,1980)所定义。酵母宿主细胞可以是假丝酵母属(candida)、汉逊酵母属(hansenula)、克鲁维酵母属(kluyveromyces)、毕赤酵母属(pichia)、酵母菌属(saccharomyces)、裂殖酵母属(schizosaccharomyces)或耶氏酵母属(yarrowia)细胞,例如乳酸克鲁维酵母(kluyveromyceslactis)、卡尔酵母(saccharomycescarlsbergensis)、酿酒酵母、糖化酵母(saccharomycesdiastaticus)、道格拉氏酵母(saccharomycesdouglasii)、克鲁氏酵母(saccharomyceskluyveri)、诺氏酵母(saccharomycesnorbensis)、卵形酵母(saccharomycesoviformis)或解脂耶氏酵母(yarrowialipolytica)细胞。真菌宿主细胞可以是丝状真菌细胞。“丝状真菌”包括亚门真菌门(eumycota)和卵菌门(oomycota)的所有丝状形式(如上述hawksworth等,1995所定义)。丝状真菌的特征通常在于由几丁质、纤维素、葡聚糖、壳聚糖、甘露聚糖和其他复合多糖组成的菌丝体壁。丝状真菌宿主细胞可以是支顶孢属(acremonium)、曲霉属(aspergillus)、金担子菌属(aureobasidium)、烟管菌属(bjerkandera)、拟蜡孔菌属(ceriporiopsis)、金孢子菌属(chrysosporium)、鬼伞属(coprinus)、革盖菌属(coriolus)、隐球菌属(cryptococcus)、线黑粉菌科(filibasidium)、镰刀菌属(fusarium)、腐质霉属(humicola)、稻瘟菌属(magnaporthe)、毛霉属(mucor)、毁丝霉属(myceliophthora)、新美鞭菌属(neocallimastix)、脉孢菌属(neurospora)、拟青霉属(paecilomyces)、青霉菌属(penicillium)、平革菌属(phanerochaete)、射脉菌属(phlebia)、梨囊鞭菌属(piromyces)、侧耳属(pleurotus)、裂褶菌属(schizophyllum)、踝节菌属(talaromyces)、热子囊菌属(thermoascus)、梭孢壳属(thielavia)、弯颈霉属(tolypocladium)、栓菌属(trametes)或木霉属(trichoderma)细胞。例如,丝状真菌宿主细胞可以是泡盛曲霉、臭曲霉(aspergillusfoetidus)、烟曲霉(aspergillusfumigatus)、日本曲霉(aspergillusjaponicus)、构巢曲霉、黑曲霉(aspergillusniger)、米曲霉、烟管菌(bjerkanderaadusta)、干拟蜡菌(ceriporiopsisaneirina)、卡内基拟蜡菌(ceriporiopsiscaregiea)、浅黄拟蜡孔菌(ceriporiopsisgilvescens)、潘诺希塔拟蜡菌(ceriporiopsispannocinta)、环带拟蜡菌(ceriporiopsisrivulosa)、微红拟蜡菌(ceriporiopsissubrufa)、虫拟蜡菌(ceriporiopsissubvermispora)、黄孢霉(chrysosporiuminops)、嗜角质金孢子菌(chrysosporiumkeratinophilum)、拉克淖金孢子菌(chrysosporiumlucknowense)、粪状金孢子菌(chrysosporiummerdarium)、毡金孢子菌(chrysosporiumpannicola)、昆士兰金孢子菌(chrysosporiumqueenslandicum)、热带金孢子菌(chrysosporiumtropicum)、带纹金孢子菌(chrysosporiumzonatum)、灰盖鬼伞(coprinuscinereus)、毛云芝菌(coriolushirsutus)、孢状镰孢(fusariumbactridioides)、禾谷镰孢(fusariumcerealis)、克地镰刀菌(fusariumcrookwellense)、黄色镰刀菌(fusariumculmorum)、禾谷镰刀菌(fusariumgraminearum)、禾赤镰孢(fusariumgraminum)、异孢镰孢(fusariumheterosporum)、合欢木镰孢(fusariumnegundi)、尖孢镰刀菌、多枝镰孢(fusariumreticulatum)、粉红镰孢菌(fusariumroseum)、接骨木镰孢(fusariumsambucinum)、肤色镰孢(fusariumsarcochroum)、拟枝孢镰刀菌(fusariumsporotrichioides)、硫色镰刀菌(fusariumsulphureum)、圆镰孢(fusariumtorulosum)、拟丝孢镰刀菌(fusariumtrichothecioides)、镶片镰孢菌(fusariumvenenatum)、特异腐质霉、柔毛腐质霉、米黑毛霉(mucormiehei)、嗜热毁丝霉(myceliophthorathermophila)、粗糙脉孢菌(neurosporacrassa)、产紫青霉(penicilliumpurpurogenum)、黄孢原毛平革菌(phanerochaetechrysosporium)、射脉侧菌(phlebiaradiata)、杏鲍菇(pleurotuseryngii)、土生梭孢壳(thielaviaterrestris)、长绒毛栓菌(trametesvillosa)、变色栓菌(trametesversicolor)、哈茨木霉(trichodermaharzianum)、康宁木霉(trichodermakoningii)、长枝木霉(trichodermalongibrachiatum)、里氏木霉或绿色木霉(trichodermaviride)细胞。
真菌细胞可以通过本身已知的涉及原生质体形成、原生质体转化和细胞壁再生的方法转化。用于转化曲霉属和木霉属宿主细胞的合适方法描述于ep238023,yelton等,1984,proc.natl.acad.sci.usa81:1470-1474和christensen等,1988,bio/technology6:1419-1422。转化镰孢属物种的合适方法描述于malardier等,1989,gene78:147-156和wo96/00787。可以使用文中所描述的方法转化酵母(becker和guarente、inabelson,j.n.和simon.m.i.,editors,yeastgeneticsandmolecularbiology,methodsinenzymology,volume194,pp182-187,academicpress,inc.,newyork;ito等,1983,j.bacteriol.153:163;和hinnen等,1978,proc.natl.acad.sci.usa75:1920)。
细胞也可以是哺乳动物细胞,例如cos、cho(us4889803;us5047335)。在一个具体实施方案中,细胞是非人的和非胚胎的。另外地,本发明的脂肪酸脱羧酶能通过非人转基因动物产生,例如通过动物产生的乳汁。
适用于本发明方法的藻类物种包括微藻类如,例如以下属的物种:曲壳藻属(achnanthes)、茧形藻属(amphiprora)、双眉藻属(amphora)、纤维藻属(ankistrodesmus)、星胞藻属(asteromonas)、破囊壶菌(aurantiochytrium)、黄金色藻属(boekelovia)、扭鞘藻属(bolidomonas)、包特氏菌属(borodinella)、气球藻属(botrydium)、葡萄藻属(botryococcus)、荀球藻属(bracteococcus)、角毛藻属(chaetoceros)、四鞭藻属(carteria)、衣藻属(chlamydomonas)、绿球藻属(chlorococcum)、绿梭藻属(chlorogonium)、小球藻属(chlorella)、蓝隐藻属(chroomonas)、金球藻属(chrysosphaera)、球丐板藻属(cricosphaera)、隐甲藻属(crypthecodinium)、隐藻属(cryptomonas)、小环藻属(cyclotella)、链带藻属(desmodesmus)、杜氏藻属(dunaliella)、后棘藻属(elipsoidon)、圆石藻属(emiliania)、空球藻属(eudorina)、独球藻属(eremosphaera)、衣迪斯藻属(ernodesmius)、眼虫属(euglena)、真眼点藻属(eustigmatos)、伏氏藻属(franceia)、脆杆藻属(fragilaria)、拟脆杆藻属(fragilaropsis)、丝藻属(gloeothamnion)、盘藻属(gonium)、红球藻属(haematococcus)、菱板藻属(hantzschia)、异弯藻属(heterosigma)、膜胞藻属(hymenomonas)、等鞭金藻(isochrysis)、鳞孔藻属(lepocinclis)、缺刻叶球藻属(lobosphaera)、微芒藻属(micractinium)、微星鼓藻属(micrasterias)、蒜头藻属(monodus)、单针藻属(monoraphidium)、微球藻属(nannochloris)、微拟球藻属(nannochloropsis)、舟形藻属(navicula)、新绿藻属(neochloris)、异鞭藻(nephrochloris)、肾藻属(nephroselmis)、菱形藻属(nitzschia)、棕鞭藻属(ochromonas)、鞘藻属(oedogonium)、卵囊藻(oocystis)、鸵球藻属(ostreococcus)、实球藻属(pandorina)、拟小球藻属(parachlorella)、缘绿藻属(parietochloris)、parietichytrium、盐藻属(pascheria)、巴夫藻属(pavlova)、pelagomonas、褐指藻属(phaiodactylum)、噬菌体属(phagus)、混营性微藻属(picochlorum)、扁藻属(platymonas)、颗石藻属(pleurochrysis)、球藻属(pleurococcus)、原壁藻属(prototheca)、拟绿球藻属(pseudochlorella)、微细绿藻属(pseudochoricystis)、拟新绿藻属(pseudoneochloris)、假十字趾藻属(pseudostaurastrum)、塔胞藻属(pyramimonas)、桑葚藻(pyrobotrys)、栅藻属(scenedesmus)、schizochlamydella、裂壶藻属(schizochytrium)、骨条藻属(skeletonema)、水绵藻属(spyrogyra)、裂丝藻属(stichococcus)、四球藻属(tetrachlorella)、四爿藻属(tetraselmis)、海链藻属(thalassiosira)、破囊壶菌属(thrautochytrium)、黄丝藻属(tribonema)、石莼属(ulva)、无隔藻属(vaucheria)、鲜绿球藻属(viridiella)、紲夏藻属(vischeria)、和团藻属。特别合适的物种的非限制性实例包括,例如硅藻如,例如,以下任何属的物种:双眉藻属、角毛藻属、小环藻属、细柱藻属(cylindrotheca)、管状藻属、脆杆藻属、拟脆杆藻属、舟形藻属、菱形藻属、褐指藻、pseudo-nitzia、或海链藻属、或真眼点藻纲(eustigmatophytes)(例如真眼点藻属、蒜头藻属、微拟球藻属或紲夏藻属)。
更具体地,可以使用的微藻包括但不限于东方曲壳藻(achnanthesorientalis)、阿格门氏藻属(agmenellum)、透明苗形藻属(amphiprorahyaline)、咖啡形双眉藻(amphoracojfeiformis)、咖啡形双眉藻线形(amphoracojfeiformislinea)、咖啡形双眉藻斑点(amphoracoffeijbrmispunctata)、咖啡形双眉藻泰勒氏(amphoracojfeiformistaylori)。咖啡形双眉藻细薄(amphoracojfeiformistenuis)、优美双眉藻(amphoradelicatissima)、优美双眉藻头状(amphoradelicatissimacapitaia)、双眉藻某种(amphorasp.)、鱼腥藻属(anahaena)、变异鱼腥藻(anabaenavariabilis)、纤维藻属(ankistrodesrnus)、镰形纤维藻(ankistrodesrnusjalcatus)、黄金色藻(boekeloviahooglandii)、包特氏菌属某种(borodinellasp.)、布朗尼葡萄藻(botryococciisbraunii)、苏台德克斯葡萄藻(botryococcussudeiicus)、小型片球藻(bracteococcusminor)、中核片球藻(bracteococcusmedionucleatus)、四鞭藻(carteria)、纤细角毛藻(chaetocerosgracilis)、牟勒氏角毛藻(chaetocerosmuelleri)、牟勒氏角毛藻亚盐(chaetocerosmuellerisuhsalsum)、角毛藻某种(chaetocerossp.)、莱茵衣藻、(chlamydomonasemoewusi)、雪衣藻(chlamydomonasnivalis)、尾状衣藻(chlamydomonascaudate)。无硝小球藻(chlorellaanitrata)、南极洲小球藻(chlorellaantarctica)、黄绿小球藻(chlorellaaureoviridis)、念珠菌小球藻(chlorellacandida)、囊状小球藻(chlorellacapsulate)、干燥小球藻(chlorelladesiccate)、捕圆小球藻(chlorellaellipsoidea)、浮水小球藻(chlorellaemersonii)、淡揭小球藻(chlorellafusca)、淡褐小球藻空泡变种(chlorellafuscavar.vacuolata)、谷小球藻(chlorellaglucotropha)、水溪小球藻(chlorellainfusionum)、融合小球藻海岸螺变种(chlorellainfusionumvar.actophila)、融合小球藻异物变种(chlorellainfusionurnvar.auxenophila)、凯氏小球藻(chlorellakessleri)、路氏小球藻(chlorellalobophora)(sag37.88株)、路特小球藻(chlorellaluteoviriais)、路特小球藻维斯变种(chlorellaluteoviridisvar.aureoviridis)。路特小球藻路斯变种(chlorellaluteoviridisvar.lutescens)、微小小球藻(chlorellaminiata)、极微小球藻(chlorellaminutissima)、突变小球藻(chlorellamutabilis)、夜间小球藻(chlorellanocturna)、卵圆形小球藻(chlorellaovalis)、小形小球藻(chlorellaparva)、嗜光小球藻(chlorellaphotophila)、毛球小球藻(chlorellapringsheimii)、原始小球藻酸居变种(chlorellaprotothecoidesvar.acidicola)、规则小球藻(chlorellaregularis)、规则小球藻小型变种(chlorellaregularisvar.minima)、瓦状寻常小球藻变种(chlorellaregularisvar.umhricata)、瑞小球藻(chloreilareisiglii)、嗜糖小球藻(chlorellasaccharophila)、海洋小球藻捕圆变种(chlorellasaccharophilavar.ellipsoidea)。海水小球藻(chlorellasalina)、单纯小球藻(chlorellasimplex)。耐热性小球藻(chlorellasorokiniana)、小球藻某种(chlorellasp.)、球抱小球藻(chlorellasphaerica)。红皮小球藻(chlorellastigmaiophora)、变异小球藻、万尼氏小球藻(chlorellavanniellii)、普通小球藻(chlorellavulgaris)、普通小球藻粗皮型(chlorellavulgarisf.tenia)、普通小球藻自养变种(chlorellavulgarisvar.autotrophica)、普通小球藻绿色变种(chlorellavulgarisvar.viridis)、普通小球藻普通变种(chlorellavulgarisvar.vulgaris)、普通小球藻普通变种粗皮型(chlorellavulgarisvar.vulgarisftertia)、普通小球藻普通变种翠绿型(chlorellavulgarisvar.vulgarisf.viridis)、仙氏小球藻(chlorellaxanthella)、佐氏小球藻(chlorellazojingiensis)、梯氏小球藻(chlorellatrehouxioides)、普通小球藻(chlorellavulgaris)、水溪绿球藻(chlorococcuminfusionum)、绿球藻某种(chlorococcumsp.)、绿梭藻属(chlorogonium)、蓝隐藻属某种(chroomonassp.)、金球藻属某种(chrysosphaerasp.)、coccomyxasuhellipsoideac-169、球钙板藻属某种(cricosphaerasp.)、隐甲藻(crypihecodiniumcohnii)。隐藻属某种(cryptomonassp.)、隐秘小环藻(cyclotellacryptica)、梅尼小环藻(cyclotellameneghiniana)、小环藻属某种(cyclotellasp.)、杜氏藻某种(dunaiiellasp.)、拜尔代维勒杜氏藻(dimaliellabardawil)、桕氏杜氏藻(dunaiiellahioculata)、颗粒状杜氏藻(dunaiiellagranulate)、海生杜氏藻(dunaiiellamaritime)、微小杜氏藻(dunaiiellaminuta)、巴夫杜氏藻(dunaiiellaparva)、皮氏杜氏藻(dunaiiellapeircei)、普氏杜氏藻(dunaliellaprimolecta)、盐生杜氏藻(dunaliellasaiina)、陆生杜氏藻(dimaliellaterricola)、杜氏盐藻(dunaliellatertiolecta)、绿色杜氏藻(dimaliellaviridis)、杜氏盐藻(dunaliellatertiolecta)、绿色独球藻(eremosphaeraviridis)、独球藻属某种(eremosphaerasp.)、后棘藻属某种(ellipsoidonsp.)。裸藻属某些种(euglena)。披剌藻某种(franceiasp.)、克罗脆杆藻(fragilariacrotonensis)、脆杆藻属(fragilariasp.)、蓝鼓藻属某种(gleocapsasp.)、丝藻属某种(gloeothamnionsp.)、雨生红球藻(haematococcusplwialis)、膜胞藻属某种(hymenomonassp.)、球等大鞭金藻(isochrysisaffgalhana)、球等鞭金藻(isochrysisgalbana)、鳞孔藻属(lepocinclis)、微星藻属(micractinium)、微星藻藻(micractinium)、微胞藻(micromonas)、细小微胞藻(micromonaspusilla)、微小单针藻(monoraphidiumminuium)、单针藻属某种(monoraphiaiumsp.)、侏囊菌属某种(nannochlorissp.)、盐生微拟球藻(nannochloropsissaiina)、微拟球藻某种(nannochloropsissp.)、截形舟形藻(naviculaacceptata)、拜氏舟形藻(naviculahiskanterae)、假舟形藻(naviculapseudotenelloides)、具膜舟形藻(naviculapelliculosa)、腐生舟形藻(naviculasaprophila)、舟形藻某种(naviculasp.)、肾鞭藻某种(nephrochlorissp.)、肾藻属某种(nephroselmissp.)、普通菱形藻(nitschiacommunis)、亚历山大菱形藻(nitzschiaalexandfjna)、普通菱形藻(nitzschiacommunis)、细端菱形藻(nitzschiadissipata)、碎片菱形藻(nitzschiafrustulum)、汉氏菱形藻(nitzschiahantzschiana)。平庸菱形藻(nitzschiainconspicua)、中型菱形藻(nitzschiaintermedia)、小头菱形藻(nitzschiamicrocephalia)、微小菱形藻(nitzschiapusilla)、微小菱形捕圆藻(nitzschiapusillaelliptica)、微小菱形单藻(nitzschiapusillamonoensis)、四角形菱形藻(nitzschiaquadrangular)、菱形藻属某种(nitzschiasp.)、念珠藻属某种(nostocsp.)、点型念珠藻(nostocpimctiforme)、掠鞭藻属某种(ochromonassp)、微细卵囊藻(oocystisparva)、卵泡藻(oocystispusilla)、卵囊藻属某种(oocystissp.)、沼泽颤藻(oscllatorialimnetica)、颤藻属某种(oscillatoriasp)、拟短形颤藻(oscillatoriasuborevis)、鞭毛藻(osterococcus)、绿色鞭毛藻(osterococcuslucimariniis)、微小鞭毛藻(osterococcustauri)、凯氏拟小球藻(parachlorellakessleri)、酸性杜氏亚属盐藻(pascheriaacidophila)、巴夫藻属某种(pavlovasp.)、噬菌体属(phagus)、三角褐指藻(phaeodactylumtricomutum)、席藻属(phormidium)、扁藻属某种(piaiymonassp.)、颗石藻(pleurochrysiscarterae)、紫石藻(pleurochrysisdentate)、颗石藻属某种(pleurochrysissp.)、原绿球藻(prochlorococcusmarinus)、威克海姆原壁藻(protothecawickerhamii)、大型原壁藻(protothecastagnant)、波多黎各原壁藻(protothecaportoricensis)、无色微小藻(protothecamoirforrms)、饶氏原藻(protothecazopfii)、水生假小球藻(pseudochlorellaaquatica)、塔胞藻属某种(pyramirnoriassp.)、桑堪藻属(pyroboirys)、不透明红球菌(rhodococcusopacus)。克瑞类斯菌(sarcinoidchrysophyte)、被甲栅藻(scenedesmusarmatus)、集胞藻属某种(scynechocystissp.)、聚球藻属(scynechococcus)、裂殖壶菌(schizochytrim)、水绵属(spirogyra)、钝顶螺旋藻(spirulinaplatensis)、裂丝藻属某种(stichococcussp.)、聚球藻属某种(synechococcussp.)、四角藻属(tetraedron)、假微型海链藻(thalassiosirapseudonana)、四月藻某种(tetraselmissp.)、瑞典四爿藻(tetraselmissuecica)、威氏海链藻(thalassiosiraweissflogii)、弗德小藻(viridiellafridericiana)和强壮团藻。在一些实施方案中,微拟球藻属(nannochloropsis)的成员选自(n.gaditana)、颗粒状微拟球藻(n.granulate)、沼泽微拟球藻(n.limnetica)、海洋微拟球藻(n.oceanica)、(n.oculata)、和海水微拟球藻(n.salina)。
适用于本发明方法的藻类物种还包括但不限于以下门的藻类物种:红藻门(rhodophyta),如原始红藻属(cyanidioschyzon)、江蓠属(gracilaria)、珊瑚藻属(kappaphycus)、紫球藻属(porphyridium)和紫菜属(porphyra);轮藻门(charophyta),如新月藻属(closterium)和柱形鼓藻属(penium);绿藻门(chlorophyta),如空球藻属(eudorina)、盘藻属、红球藻属、缺刻叶球藻属、微星鼓藻属、鸵球藻属、实球藻属、拟小球藻属、扁藻属、微细绿藻、栅藻属、石莼属和团藻属;褐藻门(phaeophyta),如昆布属(laminaria)。
宿主细胞还能选自甲藻门(dinophyta)的物种,例如前沟藻属(amphidinium)和虫黄藻属(symbiodinium);绿角藻门(chlorarachniophyta)的物种,例如lotharella;眼虫门(euglenozoa)的物种,例如眼虫属(euglena)。
根据本发明,具有特定支化模式、饱和水平和碳链长度的烷烃/烯烃,能由具有那些特定特征的脂肪酸底物和本发明的脂肪酸脱羧酶制备。因此,能修饰脂肪酸生物合成途径中的每个步骤以产生或过量产生感兴趣的脂肪酸底物。例如,参与脂肪酸生物合成途径的已知基因能在宿主细胞中表达、过表达或减弱,以产生期望的脂肪酸底物(参见例如wo08/119082、us8183028)。示例性基因在wo08/119082的图1或us8183028的表1和第25-30栏中(该公开内容通过引用并入本文)提供。
然后,能修饰宿主细胞以增加或指导脂质产生和/或脂肪酸产生。修饰的宿主细胞能依据产生游离脂肪酸或游离脂肪酸的特定性质的能力被选择(例如在随机诱变后),或者能通过基因工程引入新基因或增加某些基因(参与游离脂肪酸分解代谢的基因)的表达,和/或通过缺失其他基因或减少其表达(参与游离脂肪酸合成代谢的基因)来制备。另外地,能修饰宿主细胞以增加优选脂肪酸的产生并减少不需要的脂肪酸的产生。
例如,为了产生脂质,宿主细胞(例如但不限于藻类或异形鞭毛类宿主细胞)能任选地包括一种或多种编码多肽的非天然基因,所述多肽在脂质生物合成中起作用,包括但不限于编码用于产生脂肪酸、脂肪酸衍生物和/或甘油脂(包括但不限于二酰基甘油酰基转移酶(dgat)基因、甘油磷酸酰基转移酶(gpat)基因、溶血磷脂酸酰基转移酶(脱氢酶)(lpaat)基因、磷脂酸磷酸酶(pap)基因、和/或单酰基甘油酰基转移酶(mgat)基因)的酶的多肽。
在优选的实施方案中,所述宿主细胞已被修饰用于表达或过表达脂肪酶。脂肪酶水解酰基脂质(例如甘油三酯或甘油磷脂或甘油半乳糖脂)以产生游离脂肪酸和甘油。优选地,脂肪酶是具有广泛的三酰基甘油、磷脂酶和半乳糖脂酶活性的脂肪分解酶。实例包括豚鼠胰脂肪酶相关蛋白2型、镰刀菌角质酶或猪葡萄球菌脂肪酶(liu等,2011,pnas108:6905)。在一个优选的实施方案中,所述宿主细胞是细菌并且表达(优选过表达)脂肪酶。因此,本发明的一个目的是表达(优选过表达)本发明的脂肪酸脱羧酶和脂肪酶(其中一种或两种对宿主细胞是异源的)的重组宿主细胞。在优选的实施方案中,所述宿主细胞是细菌。
在一个优选的实施方案中,所述宿主细胞已工程化以表达、过表达或减弱表达硫酯酶,以增加脂肪酸的产生或有利于优选脂肪酸的产生。优选地,所述宿主细胞已被修饰用于表达或过表达硫酯酶。因此,本发明的一个目的是表达(优选过表达)本发明的脂肪酸脱羧酶和硫酯酶(其中一种或两种对宿主细胞是异源的)的重组宿主细胞。在优选的实施方案中,所述宿主细胞是微藻或蓝细菌。
通过终止脂肪酸生物合成,酰基-酰基载体蛋白(acp)硫酯酶在功能上决定脂肪酸终产物的长度和身份(salas等,(2002)archivesofbiochemistryandbiophysics403:25-34)。基于氨基酸序列比对,植物硫酯酶已显示聚集成两个家族,fatas,其显示出对18:1-acp的显著偏好,对18:0-和16:0-acp具有较小活性;和fatbs,其水解链长在8-16个碳之间变化的初级饱和的酰基-acp(voelker,ingeneticengineeringvolume18.editedby:setlowjk.newyork,plenumpress;1996:111-133;ginalski等,nucl.acids.res.(2003)31:3291-3292;和jones等,(1995)plantcell7:359-371)。
所述硫酯酶属于来自ec3.1.2的酶,更具体地来自ec3.1.2.2(棕榈酰-coa水解酶)、ec3.1.2.14(油酰-[酰基-载体-蛋白]水解酶)、ec3.1.2.18(adp-依赖性短链酰基辅酶a水解酶)、ec3.1.2.19(adp依赖性中链酰基辅酶a水解酶)、ec3.1.2.20、ec3.1.2.21(十二烷酰基-[酰基-载体-蛋白质]水解酶)、ec3.1.2.22(棕榈酰-蛋白水解酶)的酶。
硫酯酶的实例已在wo14120829和wo16044779中公开。它们还包括由tesb编码的大肠杆菌(escherichiacoli)硫酯酶(参见genbank登录号aaa24665.1)、短乳杆菌(lactobacillusbrevis)硫酯酶(genbank登录号abj63754.1)和植物乳杆菌(lactobacillusplantarum)酯酶(genbank登录号ccc78182.1)。在radakovits等,2011,metabolicengineering13(1):89-95中公开了适于制备中链脂肪酸的来自植物的源硫酯酶的另一个实例。
在一个优选的实施方案中,为了利于短链和中链脂肪酸而选择硫酯酶。
在本发明的一个具体方面,还提供了一种产生根据本发明的脂肪酸脱羧酶的方法,包括在有助于产生所述脂肪酸脱羧酶的条件下培养如上定义的宿主细胞,并回收和/或纯化所述脂肪酸脱羧酶。可选地,还提供了产生根据本发明的脂肪酸脱羧酶的方法,包括用编码如上定义的脂肪酸脱羧酶的核酸在体外表达所述脂肪酸脱羧酶。任选地,所述方法进一步包括将脂肪酸脱羧酶固定在固体支持物上的步骤。
可以使用本领域已知的方法回收所述酶。例如,可以通过常规方法(包括但不限于收集、离心、过滤、提取、喷雾干燥、蒸发或沉淀)从营养培养基中回收所述酶。
所述酶可以通过本领域已知的多种方法包括但不限于层析(例如,离子交换、亲和、疏水、层析聚焦和尺寸排阻)、电泳过程(例如,制备等电聚焦)、差异溶解度(例如,硫酸铵沉淀)、sds-page或提取(参见例如,proteinpurification,janson和ryden,editors,vchpublishers,newyork,1989)纯化,以获得基本上纯的多肽。在可选的一方面,不回收所述酶而是将表达所述酶的本发明宿主细胞用作酶的来源。
用于产生烷烃/烯烃的用途
本发明还涉及如上定义的脂肪酸脱羧酶、或包含所述脂肪酸脱羧酶的固体支持物、或包含含有编码如上定义的脂肪酸脱羧酶的核酸序列的核酸、重组核酸构建体或重组载体的重组宿主细胞,用于从脂肪酸产生烷烃/烯烃的用途。
脂肪酸底物:
烷烃/烯烃由根据本发明的酶从脂肪酸产生。特别地,所述脂肪酸是包含脂肪链或支链和羧酸基团的游离脂肪酸。它们能是饱和的或不饱和的。它们能是单不饱和的或多不饱和的。它们可包含2-36个碳原子。通常,基于脂肪链的长度将所述脂肪酸分为四组:(1)短链脂肪酸(scfa)是脂肪族尾部少于六个碳的脂肪酸(例如丁酸);(2)中链脂肪酸(mcfa)是脂肪族尾部6-12个碳的脂肪酸,其能在中链甘油三酯中发现;(3)长链脂肪酸(lcfa)是脂肪族尾部13至21个碳的脂肪酸;(4)特长链脂肪酸(vlcfa)是脂肪尾部长于22个碳的脂肪酸。本发明与mcfa和lcfa高度相关,更具体地与lcfa高度相关。在优选的实施方案中,所述脂肪酸包含从8或10至24个碳原子,优选从12至22个碳原子。在一些实施方案中,所述脂肪酸底物是c4、c5、c6、c7、c8、c9、c10、c11、c12、c13、c14、c15、c16、c17、c18、c19、c20、c21或c22脂肪酸,优选c10、c11、c12、c13、c14、c15、c16、c17、c18、c19、c20、c21或c22脂肪酸,还更优选c12、c13、c14、c15、c16、c17、c18、c19、c20、c21或c22脂肪酸。更优选地,所述脂肪酸包含8至20个碳原子,优选12至18个碳原子,还更优选14至18个碳原子,并且甚至更优选16或17个碳原子。在一个具体方面,所述脂肪酸底物是c12、c13、c14、c15、c16、c17、c18、c19、c20或c21脂肪酸,优选c12、c13、c14、c15、c16、c17、c18,或c19脂肪酸,还更优选c12、c14、c16、c17或c18脂肪酸。脂肪链能是直链、支链或甚至包括环状部分。
在一个具体实施方案中,所述脂肪酸包含支链。所述支链可包括具有c1-c3烷基取代的主链,优选甲基取代。特别地,所述脂肪酸可以是萜类脂肪酸,例如植烷酸和降植烷酸。
另外地,所述脂肪酸的所述脂肪链可以被一个或几个基团(例如羟基、c1-c3醇、c1-c3酰基、c1-c3酯、c1-c3胺、氨基、c1-c3酰胺、羧基、醛、环氧基、卤素、c1-c3烷氧基、c1-c3硫代烷基、c1-c3亚胺、腈、c1-c3砜等硫基或c1-c3亚砜、硫醇、硝基、氰基、c1-c3卤代烷基)取代,或可以被杂原子(如o、n或s、炔基、醚如二乙醚或氧代基团)中断。优选地,所述脂肪酸的所述脂肪链可以被一个或几个基团(例如羟基、羧基、环氧基、卤素、硫醇或腈)取代,或者可以被s原子、炔或醚如二乙醚中断。优选地,所述脂肪链被一个基团取代,特别是在所述脂肪链的末端。在一个非常具体的实施方案中,所述脂肪酸的所述脂肪链被羟基取代,特别是在脂肪链的末端。
在另一个实施方案中,所述脂肪酸的所述脂肪链是未被取代的。
所述脂肪酸底物能是分离的或纯化的脂肪酸或如上所述的脂肪酸的混合物。
通过“游离脂肪酸”是意在指其中脂肪酸为其带有羧基形式的形式,即不是酯化形式或脂肪酸的其它衍生物。
烷烃/烯烃:
本发明的烷烃/烯烃是通过相应脂肪酸的脱羧获得的。因此,烷烃由饱和脂肪酸和烯烃由不饱和脂肪酸获得。本发明的酶不引入末端不饱和度。因此,在优选的实施方案中,所获得的烯烃不存在末端不饱和度。
通过本发明的用途和方法获得的烷烃/烯烃包含8或10至24个碳原子,优选12至22个碳原子。在一些实施方案中,所述烷烃/烯烃是c4、c5、c6、c7、c8、c9、c10、c11、c12、c13、c14、c15、c16、c17、c18、c19、c20、c21或c22的烷烃/烯烃,优选c10、c11、c12、c13、c14、c15、c16、c17、c18、c19、c20、c21或c22的烷烃/烯烃,还更优选c12、c13、c14、c15、c16、c17、c18、c19、c20、c21或c22的烷烃/烯烃。更优选地,所述烷烃/烯烃包含8至20个碳原子,优选12至18个碳原子,还更优选14至18个碳原子,并且甚至更优选16至17个碳原子。在一个具体方面,所述烷烃/烯烃是c12、c13、c14、c15、c16、c17、c18、c19、c20或c21的烷烃/烯烃,优选c12、c13、c14、c15、c16、c17、c18或c19的烷烃/烯烃,还更优选c12、c14、c16、c17或c18的烷烃/烯烃。脂肪/烃链能是直链、支链或甚至包括环状部分。
在一个具体实施方案中,所述烷烃/烯烃包含支链。所述支链可包括具有c1-c3烷基取代的主链,优选甲基取代。特别地,所述脂肪酸可以是萜类烷烃,例如植烷和降植烷。
另外地,由于脂肪酸的脂肪链可以被如上详述的一个或几个基团取代或中断,脱羧后相应的烷烃/烯烃也能被一个或几个基团取代或中断。因此,烷烃/烯烃能被一个或几个基团(例如羟基、c1-c3醇、c1-c3酰基、c1-c3酯、c1-c3胺、氨基、c1-c3酰胺、羧基、醛、环氧基、卤素、c1-c3烷氧基、c1-c3硫代烷基、c1-c3亚胺、腈、硫基如c1-c3砜或c1-c3亚砜硫醇、硝基、氰基、c1-c3卤代烷基)取代,或可以被杂原子(如o、n或s、炔基、醚如二乙醚、或氧代基)中断。优选地,所述烷烃/烯烃可以被一个或几个基团(例如羟基、羧基、环氧基、卤素、硫醇或腈)取代,或者可以被s原子、炔基或醚如二乙醚基团中断。优选地,所述烷烃/烯烃被一个基团取代,特别是在脂肪链的末端。在一个非常具体的实施方案中,所述烷烃/烯烃被羟基取代,特别是在脂肪链的末端,并且是醇。
所述烷烃/烯烃能是分离的或纯化的烷烃/烯烃或如上所述的烷烃/烯烃的混合物。在一个非常具体的实施方案中,所述酶能产生c13-c17烷烃和烯烃,特别是当在细菌(如大肠杆菌)中表达时。
在无细胞系统中产生烷烃/烯烃
在第一方面,本发明涉及如上定义的脂肪酸脱羧酶用于从脂肪酸,尤其是游离脂肪酸产生烷烃/烯烃的用途。因此,本发明涉及一种制备烷烃/烯烃的方法,其中将如上定义的脂肪酸脱羧酶与脂肪酸或脂肪酸的混合物接触并暴露于光下,从而转化所述脂肪酸或脂肪酸的混合物为相应的烷烃/烯烃。所述脂肪酸或脂肪酸混合物能是纯化的形式或能存在于未加工组合物/产物中。所述脂肪酸脱羧酶能被纯化、分离或存在于蛋白质提取物中,特别是总蛋白质提取物中。通常,fad辅因子已经与脂肪酸脱羧酶一起作为复合物存在。任选地,所述fad辅因子能与所述脂肪酸脱羧酶一起添加。
所述酶为了其活性需要暴露于含蓝光子的光中。在优选的实施方案中,蓝光具有波长为400-520nm,优选450nm-495nm,特别是约450nm。所提供的光量能是例如10至3000μmole.photon.m-2.s-1,优选约2000μmole.photon.m-2.s-1。
然而,所述光可以具有在300至540nm之间的更宽波长范围。实际上,本发明人观察到所述酶的辅因子fad能够吸收波长范围为300-540nm的光,并且所述酶应该能够在该范围内使脂肪酸脱羧。实际上,所述fad吸收光谱包含在300-540nm的波长范围内。
所述酶还需要fad(黄素腺嘌呤二核苷酸)作为辅因子,其通常结合至从微藻提取物或从异源表达物中纯化的酶。
所述方法可包括回收烷烃/烯烃的额外步骤。所述烷烃/烯烃能通过技术人员可用的任何方法提取或纯化。所述烷烃/烯烃能在有机相中回收。例如,它们能以有机溶剂(例如以己烷)萃取。但是烷烃/烯烃也可以通过冷凝从培养物的气相中回收。
在重组宿主细胞系统中产生烷烃/烯烃
本发明涉及如上所述的重组宿主细胞用于由脂肪酸(尤其是游离脂肪酸)产生烷烃/烯烃的用途。它还涉及由脂肪酸产生烷烃/烯烃的方法,其中在有效表达本发明的脂肪酸脱羧酶的条件下培养如上所述的重组宿主细胞。
因此,本发明涉及细胞培养物,其包含在有效表达本发明的脂肪酸脱羧酶的条件下培养的如上所述的重组宿主细胞。
任选地,所述重组宿主细胞能在包含脂肪酸的培养基中培养。可选地或另外地,所述重组宿主细胞产生或包含脂肪酸。
在所述方法中,所述重组宿主细胞优选在获得生物质的条件下培养,优选富含脂质(尤其是脂肪酸)的生物质。然后,在第二步中,开始将所述脂肪酸转化为烷烃/烯烃。例如,如果脂肪酸脱羧酶已经在重组宿主细胞中表达,则能通过向重组宿主细胞施加含蓝光子的光以允许所述脂肪酸脱羧酶的活性来启动第二步骤。实际上,发明人表明所述烷烃的产生能通过光(尤其是其波长)来控制。蓝光(>400nm和<530nm)允许产生烷烃,而红光(>600nm和<700nm)则不能。然后,在本发明的方法中,能通过光在体内调节烷烃的产生。可选地,如果所述脂肪酸脱羧酶的表达受诱导型启动子控制,则能通过诱导脂肪酸脱羧酶的表达并使细胞接收光子(300-540nm)来启动第二步。此外,光强度(光子通量)调节酶活性。高光子通量增加酶活性,而低光子通量降低酶活性。
可选地,所述方法能包括在共同促进生物质(优选富含脂质(尤其是脂肪酸))的产生的条件下培养重组宿主细胞,以及将所述脂肪酸转化为烷烃/烯烃。
通过“富含”意指生物质的干重包含至少20、30、40、50、60或70%的脂质(尤其是脂肪酸)重量。
所述方法可包括从重组宿主细胞或从培养基中分离或回收烷烃/烯烃的附加步骤。
所述烷烃/烯烃能通过本领域技术人员可用的任何方法提取或纯化。例如,它们能以有机溶剂(例如以己烷)萃取。所述烷烃/烯烃能在有机相中回收。
一个示例性分离方法是两相(双相)分离方法。该方法包括在足以产生烷烃/烯烃的条件下发酵基因工程宿主细胞,使烷烃/烯烃收集在有机相中,并将有机相与含水发酵液分离。该方法能在分批和连续发酵设置中实施。
根据本发明的用于产生烷烃/烯烃的方法和用途能用于制备生物燃料、有用于化学的材料,特别是化妆品和其它领域(例如塑料、树脂、纤维、弹性体、药物、润滑剂或凝胶)。
本文描述的烯烃能用作或转化为燃料。本领域普通技术人员将理解,根据燃料的预期目的,能产生和使用不同的烯烃。例如,对于旨在用于寒冷气候的汽车燃料,可能期望是支链烯烃。此外,当本文所述的烯烃用作燃料产生的原料时,本领域普通技术人员将理解,链烯原料的特性将影响所产生的燃料的特性。因此,能通过产生用作原料的特定烯烃来选择燃料产品的特性。
使用本文所述的方法,能由烯烃产生具有期望的燃料品质的生物燃料。生物产生的烯烃代表生物燃料的新来源,其能用作喷气燃料、柴油或汽油。使用烯烃制造的一些生物燃料不是由可再生资源产生的,而是新的物质组合物。
脂肪酸定量
本发明的脂肪酸脱羧酶也有用于量化游离脂肪酸,并且能包括在用于量化游离脂肪酸的试剂盒中。特别地,本发明涉及一种用于量化样品中脂肪酸的方法,包括在适于将脂肪酸转化为烷烃/烯烃、回收烷烃/烯烃和量化烷烃/烯烃的条件下,使样品与本发明的脂肪酸脱羧酶接触。所述样品能是食物样品或生物样品,特别是生物流体样品(例如血液、血清、血浆、尿液等)。生成的烷烃/烯烃能以有机溶剂萃取并量化,特别是通过与火焰离子化检测器和质谱法偶联的气相色谱法(gc-fid-ms)量化。
在一个具体实施方案中,本发明的脂肪酸脱羧酶也有用于量化总脂肪酸,并且能包括在用于量化总脂肪酸的试剂盒中。特别地,本发明涉及一种用于量化样品中脂肪酸的方法,包括在适于将脂肪酸转化为游离脂肪酸和量化如上详述的游离脂肪酸的条件下,使样品与脂肪酶接触。因此,所述方法可包括在适于将脂肪酸转化为游离脂肪酸的条件下使样品与脂肪酶接触,在适于将脂肪酸转化为烷烃/烯烃、回收烷烃/烯烃并量化烷烃/烯烃的条件下使样品与本发明的脂肪酸脱羧酶接触。产生的烷烃/烯烃能以有机溶剂萃取并量化。
当脂肪酸脱羧酶将脂肪酸转化为烷烃/烯烃时,它产生co2作为副产物。然后,在一种可选方法中,本发明涉及一种定量样品中脂肪酸的方法,包括在适于将脂肪酸转化为烷烃/烯烃、回收co2和定量co2的条件下,使样品与本发明的脂肪酸脱羧酶接触。能通过本领域技术人员已知的任何方法(例如气相色谱-质谱(gc-mc))测量co2的量。
另一方面,本发明人观察到脂肪酸脱羧酶的荧光随底物浓度的变化而变化。因此,荧光变化提供了用于量化样品中脂肪酸的额外手段。然后,在另一可选方法中,本发明涉及一种用于量化样品中脂肪酸的方法,包括在适于将脂肪酸转化为烷烃/烯烃的条件下使样品与本发明的脂肪酸脱羧酶接触,并测量由脂肪酸脱羧酶发出的荧光。优选地,所述荧光在500至700的波长下测量,动力学优选在约540nm处进行。
当然,用于量化样品中脂肪酸的方法能组合如上详述的几种方法,例如,量化烷烃/烯烃和co2、量化烷烃/烯烃并测量酶荧光、量化co2和测量酶荧光、或三个参数的组合。
脂肪酸定量能用于诊断疾病,例如肝脏疾病(wo15089102)、糖尿病和先兆子痫(wo13170369)。
去除脂肪酸
本发明的脂肪酸脱羧酶还能用于从组合物(例如从营养油,特别是精制的营养油)中去除游离脂肪酸。实际上,本发明的脂肪酸脱羧酶的用途能允许在没有任何化学过程的情况下使脂肪酸脱羧。因此,本发明涉及用如上定义的多肽用于脂肪酸脱羧从而去除脂肪酸的用途;或涉及从组合物中去除游离脂肪酸的方法,包括在适于使游离脂肪酸脱羧的条件下,使本发明的脂肪酸脱羧酶与组合物接触。
在以下实验部分中公开了本发明的其它方面和优势,该实验部分应被视为说明性的而非限制本申请的范围。
附图简述
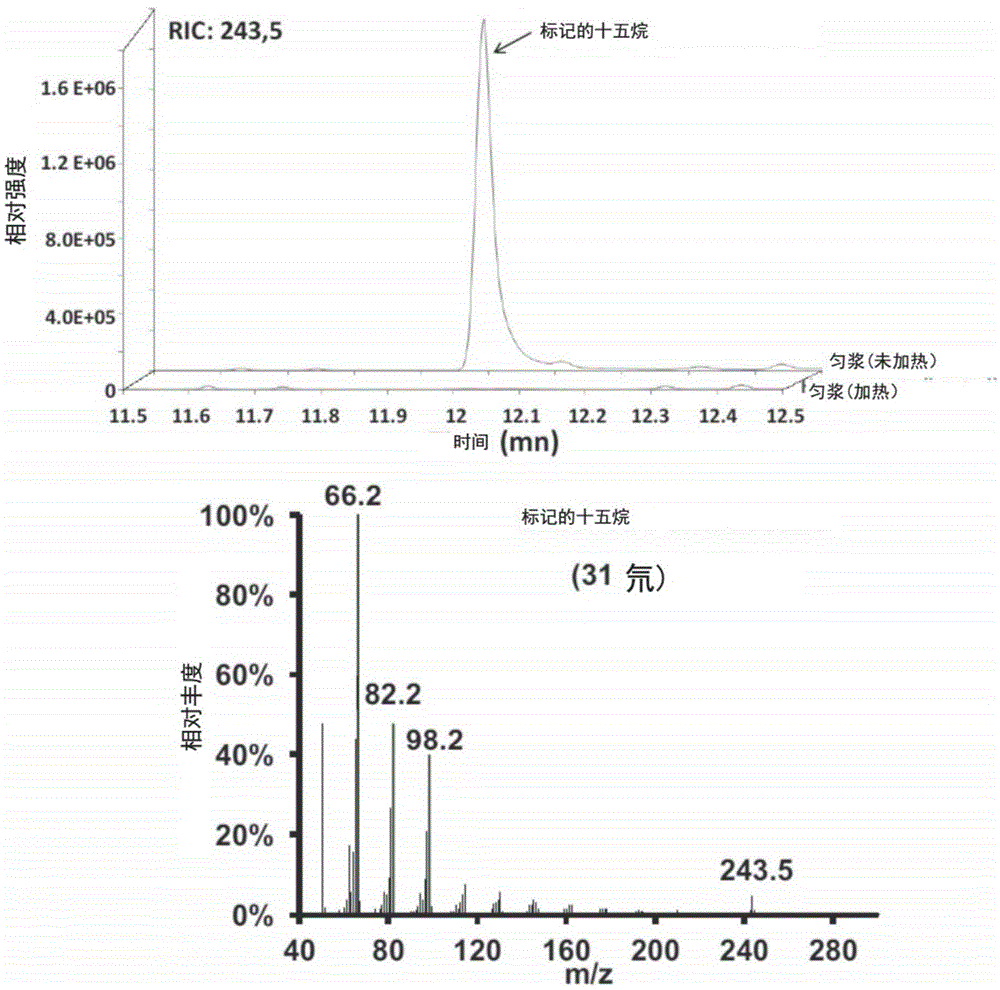
图1.小球藻匀浆中烷烃合成酶活性的检测。将变异小球藻nc64a的细胞匀浆在密封小瓶中与全氘化(d31)棕榈酸一起孵育过夜。通过固相微萃取(spme)萃取气相的烃,并通过气相色谱与质谱联用(gc-ms)分析。提取对应于m/z=243,5±0.5的离子。
上图:对应于标记的十五烷产物的层析图的一部分;对照:将匀浆在95℃下预热30分钟。下图:标记的十五烷的质谱。
图2.烷烃合成酶的部分纯化步骤和纯化中多个步骤的蛋白谱分析的总结。图1所示的细胞匀浆的细胞级分和洗脱级分进行烷烃合成酶活性测定。蛋白质电泳是在变性条件下在10%凝胶丙烯酰胺上进行。1:凝胶滤过后;2:fastflowq后;3和4:monoq后(用于蛋白质组学分析的级分)。
图3.在3次纯化中的每次后通过蛋白质组学分析检测的蛋白质的数量和10种常见蛋白质的列表。进行三次独立的纯化(a、b、c)。
图4.小球藻烷烃合成酶是预测的叶绿体gmc氧化还原酶。从pfam和protparam检索数据。氨基酸:654;分子量:69070da;理论等电点:9.075。用predalgo预测位于叶绿体中。
图5.表达小球藻gmc氧化还原酶的大肠杆菌细胞中蛋白质的分析。小球藻酶表达为c末端his标签的蛋白质。左:通过sds聚丙烯酰胺凝胶电泳观察到的总蛋白谱。右:使用抗his抗体的蛋白质印迹。c:具有空载体的对照菌株;gmc:表达小球藻gmc氧化还原酶的菌株。
图6.表达小球藻gmc氧化还原酶的大肠杆菌细胞中烃含量的分析。将细胞皂化并通过spme和gcms分析烃含量。应该强调的是,没有添加脂肪酸底物。上图:对应于烃的层析图的区域。下图:检测到的烷烃的质谱。对照:具有空载体的菌株;gmc:表达小球藻gmc氧化还原酶的菌株。量化数据显示平均值±s.d.(n=3)。
图7.表达gmc氧化还原酶的转基因微藻中的烃的分析。通过gc-ms在微藻莱茵衣藻的菌株中分析烃,其中用编码gmc氧化还原酶的基因转化叶绿体基因组。上图:gc层析图的一部分,显示烃峰。下图:烃的定量。wt:野生型菌株。gmcoe:表达小球藻gmc氧化还原酶的菌株。
图8.gmc氧化还原酶家族的多重比对(a)和系统发育树(b)。使用来自小球藻(seqidno1)、衣藻(seqidno5)、胶球藻(seqidno9)、团藻(seqidno10)、褐藻(seqidno11)、圆石藻(seqidno12)、褐潮藻(seqidno13)、褐指藻(seqidno7)、微拟球藻(seqidno14)的序列,用clustalo(1.2.1)构建多重比对。使用多个来源的56个gmc氧化还原酶蛋白质序列的组来构建无根系统发育树(邻接法)。方框指示小球藻gmc氧化还原酶。
图9.重组小球藻gmc氧化还原酶的纯化的级分。该酶在大肠杆菌中表达为n-末端具有his-标签的蛋白质并在镍柱上纯化。f1至f4是洗脱级分。l:分子量梯。
图10.小球藻gmc氧化还原酶对多种脂肪酸的相对活性。在白光下将纯化的重组酶与多种链长的脂肪酸一起孵育,并通过gc-ms分析产物。量化数据显示平均值±s.d.(n=3)
图11.co2作为烷烃合成酶催化的脱羧副产物。1-13c-棕榈酸酯与纯化的重组酶孵育后释放的13co2的相对定量。量化数据显示平均值±s.d.(n=3)。
图12.小球藻gmc氧化还原酶将萜烯酸转化为甲基烷(烯)烃。将纯化的重组酶在白光下与植烷酸一起孵育,并通过gc-ms分析产物。
图13.小球藻gmc氧化还原酶将羟基脂肪酸转化为烷醇。将纯化的重组酶在白光下与羟基棕榈酸酯一起孵育,并通过gc-ms分析产物。
图14.使用gmc氧化还原酶与脂肪酶组合由脂肪酸体外产生烷烃。将纯化的小球藻gmc还原酶和根霉属菌(rhizopus)脂肪酶与三十五烷酸甘油酯一起孵育,并通过gc-ms分析产物。量化数据显示平均值±s.d.(n=3)。
图15.小球藻gmc氧化还原酶的吸收光谱。
图16.藻类gmc氧化还原酶的光依赖性。a,小球藻gmc氧化还原酶的吸收光谱和作用光谱。b,在1-13c-棕榈酸酯与小球藻酶孵育后,通过膜入口质谱法监测13co2释放。将反应混合物暴露于850μmolesphotonsm-2s-1的连续光(蓝色或红色)条件下。c,小球藻gmc氧化还原酶活性与光照强度的相关性。d,在蓝光然后在红光(30μmolesphotonsm-2s-1)培养的过程中,衣藻细胞中总烃的变化。量化数据显示平均值±s.d.(n=3)。
图17.表达小球藻烷烃合成酶的大肠杆菌细胞中的烃的量化,并在黑暗或光照下培养(1000μmolphotonsm-2s-1的白光,含有400-800nm的光子)。量化数据显示平均值±s.d.(n=3)。nd,未检测到。
图18.在底物存在下gmc氧化还原酶荧光的动力学。激发在450nm±5且发射在540nm。使用多种浓度的棕榈酸。
图19.表达衣藻或褐指藻gmc氧化还原酶的大肠杆菌细胞中的烃的量化。将细胞转甲基化并通过溶剂提取和gcms分析烃含量。应该强调的是,没有添加脂肪酸底物。在用空载体转化的大肠杆菌细胞中未检测到烷烃。tr,痕迹。量化数据显示平均值±s.d.(n=3)。
图20.共表达藻类gmc氧化还原酶和脂肪酶的大肠杆菌细胞中的烃的量化。将细胞转甲基化并通过溶剂提取和gcms分析烃含量。应该强调的是,没有添加脂肪酸底物。阴性对照:用空载体转化的大肠杆菌细胞。nd,未检测到。cr:莱茵衣藻;cv:变异小球藻。脂肪酶来自细菌猪葡萄球菌(uniprotp04635)。量化数据显示平均值±s.d.(n=3)。
序列表简述
实施例
在此,发明人在模式微藻变异小球藻nc64a中鉴定了催化烷(烯)烃合成的酶。使用氘标记的棕榈酸作为底物和固相微萃取以捕获十五烷产物来部分纯化酶。通过三次独立部分纯化的蛋白质组学分析鉴定属于葡萄糖-甲醇-胆碱氧化还原酶家族的候选蛋白质。该小球藻候选基因在大肠杆菌中的异源表达导致13至17个碳的线性烃的产生,表明单一酶足以产生类似燃料的烷(烯)烃。使用fad作为辅因子,小球藻烷烃合成酶是69kda预测的叶绿体蛋白质。体外试验表明它能使用c12至c22脂肪酸生成烷(烯)烃。发现该酶的活性严格依赖于400至540nm的光子的存在,但其也可以在300至400nm的范围内起作用。因此,这些结果扩展了关于葡萄糖-甲醇-胆碱氧化还原酶家族的催化库的现有知识,并为可再生和光驱动的烷(烯)烃在微生物中的产生开辟了新的途径。
结果
从变异小球藻nc64a中部分纯化烷烃合成酶活性
发明人已经表明,包括变异小球藻nc64a在内的多种微藻具有合成c15-c17烷烃和烯烃的能力。在同样的工作中,他们还表明,外源添加到小球藻培养物中的氘代棕榈酸能转化为烷烃和烯烃。为了鉴定微藻中烷烃合成的酶促途径,他们选择了基于使用氘标记的棕榈酸作为底物的传统纯化方法。
第一步是确认能在小球藻细胞匀浆中测量酶活性。将氘代棕榈酸加入细胞匀浆中并在密封小瓶中孵育过夜。通过固相微萃取(spme)提取预期的十五烷产物,并用气相色谱与质谱联用(gc-ms)分析。能在完整细胞上检测到对应于标记的十五烷的12.03分钟的峰,但是在预热的对照匀浆上不存在(图1)。
因此,该实验表明小球藻匀浆具有烷烃合成酶活性。因为在大多数生物中鉴定的烷烃合成途径具有醛中间体,本发明人使用标记的c16醛进行相同的实验,但是未检测到标记的十五烷。
因此,标记的棕榈酸用于测定所有纯化过程中的活性(图2)。当细胞以50000g离心时,在上清液级分中发现大多数活性。但在105000g第二次离心后,活性主要在沉淀物中发现(微粒体级分)。测试了不同的洗涤剂以溶解该活性,并且发现效率最高的是tritonx100。
然后在进一步纯化活性之前进行一些初步测试。在溶解的微粒体级分上以不同的组合添加几种辅因子如铁氧还蛋白、铁氧还蛋白还原酶、nadp、nadph和atp。没有发现它们增加活性,并且它们没有被添加到纯化级分的测定中。本发明人还观察到,在3天内,在4℃下储存的溶解的微粒体级分活性降低了90%,表明整个纯化过程必须在几天内进行。
溶解活性的部分纯化包括用制备柱superdex200凝胶过滤的第一步,然后是两个阴离子交换柱、fastflowq和最终更具溶解性的monoq。使用先前描述过的测定法测定大多数级分的烷烃合成酶活性。通过在变性条件下在丙烯酰胺凝胶上电泳来分析大部分活性级分的蛋白质含量(图2)。
进行三次独立的部分纯化。将最终纯化步骤后具有最高活性的级分送去进行蛋白质组学分析。通过至少2个肽计数的截断,在三种纯化之间仅共有10种蛋白质(图3)。这些蛋白中的9种是与充分表征的酶的明确同源物。属于具有多种活性的酶的一组的唯一候选物是来自葡萄糖-甲醇-胆碱(gmc)氧化还原酶家族的推定酶。该候选物也是具有最高肽计数的候选物。因此选择它用于异源表达。
小球藻烷烃合成酶是gmc氧化还原酶家族的成员
编码小球藻gmc氧化还原酶的基因未被公众可获得的est完全覆盖。使用来自小球藻的总rna提取物克隆约2kb的cdna。它编码69kda蛋白质(图4),并且通过微藻序列适应软件predalgo预测其定位于叶绿体。预测n-末端的叶绿体靶向信号长50个残基。tmhmm软件预测没有跨膜结构域。该cdna在大肠杆菌中表达为具有c末端his-标签的蛋白质(图5)。通过蛋白质印迹检查重组蛋白的存在。通过spme和gc-ms分析表达gmc氧化还原酶的大肠杆菌细胞的挥发性产物,显示存在13至17个碳的长链烷烃,其在用空载体转化的大肠杆菌细胞中不存在(图6)。因此,这些结果证明小球藻gmc氧化还原酶的表达足以在大肠杆菌中产生烷烃和烯烃。此外,在用编码小球藻酶的cdna转化其叶绿体基因组的衣藻属株系中,注意到十七烯含量增加,并且还出现十七烷(图7)。因此,该结果表明小球藻酶在叶绿体中是有功能的。
使用小球藻gmc氧化还原酶或来自其他物种的其他生化表征的gmc氧化还原酶的blastp搜索在公共数据库中进行以检索多种gmc氧化还原酶蛋白质序列。藻类序列的多重比对表明一些小球藻残基如c372、r391、y406、q426、h512和n515在其它藻类中高度保守(图8a),并且进行了无根系统发育树(图8b)。blast搜索表明,在大多数生物中,gmc氧化还原酶是单基因家族。在变异小球藻nc64a中,小球藻烷烃合成酶是唯一的gmc氧化还原酶。系统发育树显示gmc氧化还原酶家族由至少7组组成,其中一组仍然没有生化表征的成员。有趣的是,棕色大型藻类(ectocarpus)、红色大型藻类(chondrus)和来自不同来源的微藻(共球藻纲(trebouxiophyceae)小球藻、绿藻纲(chlorophyceae)的衣藻和团藻、胶球藻科(coccomyxaceae)胶球藻、真眼点藻纲微拟球藻、颗石藻纲(coccolithophyceae)圆石藻、硅藻门(diatom)褐指藻)的序列分组在一起。小球藻酶是第一个在该gmc氧化还原酶的藻类分组中具有功能表征的。
小球藻烷合成酶是一种作用于各种脂肪酸的光驱动的光酶
为了进一步表征其活性,小球藻gmc氧化还原酶在大肠杆菌中表达为his-标签的蛋白质并在镍柱上纯化(图9)。发现纯化的重组体对范围在c12至c22个碳的多种饱和游离脂肪酸具有活性(图10),这解释了大肠杆菌中形成的脂肪酸的广泛特征(图6)。
为了确定反应的副产物,本发明人使用在羧基上用13c标记的棕榈酸。他们观察到13co2的产生,证明酶释放co2作为副产物,即其是脂肪酸脱羧酶。(图11)
为了表征进一步的烷烃合成酶活性,使用取代的脂肪酸作为底物。发现花生酸(3,7,11,15-四甲基十六烷酸)转化为支链烷烃,表明烷烃合成酶对萜烯酸具有活性(图12)。使用16-oh棕榈酸作为底物导致十五烷醇的产生,表明小球藻gmc氧化还原酶能用于产生脂肪醇(图13)。
在体外,烷烃合成酶不能直接使用tag(三酰基甘油)作为底物(图14)。然而,在存在释放脂肪酸的tag(三酰基甘油)脂肪酶的情况下,观察到烃的产生。该观察显示,gmc氧化还原酶能用于从富含脂质的油和废物中产生烃。
图15显示fad能吸收320至540nm的光子。这与酶的作用光谱一致(图16a)。
为了确定烷烃合成酶是光驱动酶(光酶)还是光活化酶,在反应过程中监测co2的产生。酶的活性由光子的存在驱动,并在光关闭时立即停止。(图16b)。十五烷的产生随光强度线性增加,证实了小球藻gmc氧化还原酶是光酶的事实(图16c)。藻类烷烃合成酶的光依赖性能用于使用在莱茵衣藻上观察到的光质(蓝色或红色)调节藻类细胞的烃产生(图16d)。微藻在蓝色光子存在下产生烃,而在红色中产生停止并且烷烃含量减少。在表达烷烃光合成酶的大肠杆菌细胞中,光也能用作诱导剂以选择最佳产生烷烃和烯烃的时间(图17)。
底物浓度改变酶荧光
基于酶的光依赖性,我们怀疑在具有或不具有底物的酶活性期间酶荧光能发生变化。为此,我们制作了荧光光谱和动力学(图18)。发明人观察到荧光和动力学随着底物浓度而急剧变化。使用荧光参数来确定脂肪酸浓度能是有用的。
来自衣藻属和褐指藻属的gmc氧化还原酶也是烷烃合成酶
为了研究其他藻类gmc氧化还原酶也具有脂肪酸脱羧酶活性并因此是烷烃合成酶的可能性,另一种衣藻(莱茵衣藻)和硅藻(三角褐指藻)的gmc氧化还原酶在大肠杆菌中表达并通过gc-ms-fid分析细菌细胞的总脂肪酸和烃。衣藻(酶引起在大肠杆菌细胞的十五烷和十七烷以及它们的单不饱和类似物中的形成(图19)。尽管含量较低,褐指藻酶形成相同的化合物。
共表达具有gmc氧化还原酶的脂肪酶在大肠杆菌中促进烷烃合成
游离脂肪酸库通常在活细胞中很小,因为游离脂肪酸对膜结构是有害的。为了通过增加可用的游离脂肪酸的量来观察在大肠杆菌中是否能增加烷烃和烯烃的产生,将细菌脂肪酶(uniprotp04635)与小球藻或衣藻的烷烃合成酶共表达(图20)。与仅表达gmc氧化还原酶的大肠杆菌菌株相比,这导致大大部分烃增加2至3倍。
讨论
烷烃和烯烃是生物燃料产生的感兴趣的化合物,并且烯烃对化学工业特别有意义。在该工作中,使用部分纯化和蛋白质组学分析,发明人能够鉴定来自小球藻的微藻烷基合成酶。它是gmc氧化还原酶家族的成员。当在大肠杆菌中表达时,该蛋白质单独能够产生烷烃和烯烃。该酶的主要意义在于其催化游离脂肪酸的正式脱羧形成饱和烃的明显的能力以及它是光酶的事实。预期机制和可能的辅因子需求不同于细菌细胞色素p450烷烃合成酶。因此,小球藻gmc氧化还原酶扩展烷烃合成酶的库,并为生物技术应用提供了新的可能性。
gmc氧化还原酶的藻类分组
gmc氧化还原酶家族于1992年首次被描述。来自多种生物(分别为:黑腹果蝇(drosophilamelanogaster)、大肠杆菌、黑曲霉和多形汉逊酵母(hansenulapolymorpha))的葡萄糖脱氢酶、胆碱脱氢酶、葡萄糖氧化酶和甲醇氧化酶的蛋白质序列的比较显示较低的相似性但有保守的基序。这些酶含有黄素酶位点和靠近其氨基末端的典型adp结合βαβ折叠。结构研究证实这些蛋白质由n-末端fad结合结构域和c-末端底物结合结构域组成。fad结合结构域形成二核苷酸结合蛋白典型的α-b折叠,而底物结合结构域由α螺旋包围的b折叠组成。尽管插入的结构元件存在于胆碱脱氢酶和醇脱氢酶中,但这些蛋白质的一般拓扑结构是保守的。
gmc氧化还原酶家族的成员催化多种反应,主要是将醇氧化成醛。该家族包括葡萄糖和甲醇氧化酶、脂肪醇氧化酶、胆碱脱氢酶。但该家族还包括来自扁桃的裂解酶,其作用于羟基甘露腈,表明该家族具有非常多样化的催化机制。活性对光的严格依赖性可能是由发现与重组酶相关的fad辅因子介导的。fad的存在与小球藻酶具有像所有gmc氧化还原酶一样的fad结合结构域的事实是一致的(图15)。
有趣的是,所有已经显示产生长链或非常长链烷(烯)烃的微藻物种都与小球藻gmc氧化还原酶具有同源性,但是没有可检测到的烷(烯)烃的唯一物种鸵球藻没有gmc同系物。因此,gmc氧化还原酶的藻类分组的成员很可能都是烷烃合成酶。该想法被来自莱茵衣藻和三角褐指藻的gmc氧化酶具有脂肪酸脱羧酶活性的证据所支持(图19)。
微藻烷烃合成酶的可能的生物技术应用
用于烷烃合成的微藻途径的发现具有生物技术意义,因为微藻是用于脂质产生而非得到生物质的有希望的平台,油的提取和转化为生物柴油是非常昂贵的。在微藻中产生类似燃料的挥发性烷烃,其能被容易的从培养基中回收而因此规避这些问题。
在体外,小球藻酶能够作用于多种脂肪酸,包括中链脂肪酸(图10)。与作用于脂肪醛的植物、蓝细菌或昆虫酶相比,酶作用于游离脂肪酸的事实是明显的优势,因为这些中间体能被许多内源性还原酶降解(rodriguez&atsumi,2014,metabolicengineering,25,227-37)。此外,游离脂肪酸能通过多种类型的脂肪酶产生,其中一些脂肪酶具有高活性并且能使用多种脂质底物(andersson,1996,biochimbiophysacta,1302,236-240)。通过共表达脂肪酶增加游离脂肪酸的可用性来促进烷烃的产生(图20)。在大肠杆菌中,当表达小球藻gmc氧化还原酶时产生一些十三烷(图6)。该烷烃比在藻类中观察到的那些更短,并且是由于在大肠杆菌中存在用于酶的新底物(肉豆蔻酸)。因此,微藻gmc氧化还原酶能用于产生大量的中至短链烷烃,特别是当与积聚短链至中链脂肪酸的微生物中的脂肪酶一起使用时。
由于烷烃合成酶是光酶,光能用于在体外和体内精细调节烷烃的产生。首先,320-540的光子的存在或不存在能用于选择烷烃产生的时刻。其次,光强度能用于增加或降低烷烃合成的速率。这里的实验是在连续光照下进行的,但其他条件如闪光能够对烃类合成是有意义的。
最后,产物(烷烃和烯烃)、副产物(co2)和酶荧光能用于估计样品中游离脂肪酸(或与脂肪酶组合使用时的总脂肪酸)的浓度。
材料与方法
菌株和培养条件
使用莱茵衣藻野生型菌株cc124(nit1nit2;mt-)和cc125(nit1nit2;415mt+)。变异小球藻nc64a来自j.l.vanetten(内布拉斯加大学)的实验室。在富含2%(v/v)co2的空气中,在140rpm下的摇动并且在120μmolphotonsm-2s-1(衣藻)和70μmolphotonsm-2s-1(小球藻)的光强下。在25℃的孵育摇床(inforsht)中将所有株系常规地在锥形瓶中生长,。衣藻和小球藻在tris-醋酸盐-磷酸盐(tap)培养基和基本培养基中培养。使用multisizertm3(coulter)常规计数细胞。
天然烷烃合成酶的纯化
使用fastproteinliquidchromatography系统(aktapurifier900,gehealthcare)。烷烃合成酶活性测定在下一部分中描述。将小球藻细胞(200.109)以6000g离心1小时,并将细胞沉淀在液氮中冷冻并在负80℃下储存1小时。将细胞重悬于含有20mmtris(ph8.0)、100mmnacl和1mmedta(缓冲液a)的裂解缓冲液中,并使用celldisruption(恒定的)在2kbar压力下破碎。将匀浆物以50000g离心两次,每次40分钟。收集上清液并以105000g离心90分钟。将得到的微粒体沉淀在4℃下在缓冲液a中摇动重悬过夜,缓冲液a中加入2.7mmtritonx100。超速离心在105000g下进行90分钟,并将上清液上样到凝胶过滤柱上。合并大多数活性级分,使用30kda
蛋白质纯化的活性测定
在使用具有隔膜的帽密封的透明玻璃小瓶中进行酶测定。反应混合物含有500μl的每个纯化级分、200μm的全氘化棕榈酸(在乙醇中的10mm储备溶液)和45nmol十六烷作为内标(在氯仿中的4.5mm储备溶液)。将小瓶在25℃下在白光下(强度120μmolphotonsm-2s-1)在120rpm摇动过夜。通过使用注射器通过隔膜添加10μlnaoh10m来终止反应。通过在小瓶的顶部空间中孵育固定在支架上的固相微萃取(spme)纤维(dvbpdms熔融石英,65μm双极,supelco)来分析产生的烃。在室温下孵育15分钟后,立即将spme纤维插入gc-ms的注射器中并在250℃下解吸。如下所述进行gc-ms分析。
蛋白质组学分析
如前所述进行蛋白质制备、凝胶内消化和nanolc-ms/ms分析。简而言之,将溶解在laemmli缓冲液中的蛋白质堆叠在4-12%(w/v)nupage凝胶(invitrogen)的顶部,并用r-250考马斯蓝(biorad)染色。然后切下凝胶条带并使用胰蛋白酶(promega)在凝胶中消化蛋白质。通过使用120分钟梯度的偶联至串联质谱(ultimate3000偶联至ltq-orbitrapvelospro,thermoscientific)的纳米液相色谱分析所得到的肽。通过使用mascot(版本2.5.1)对uniprot(chlorellavariabilistaxonomy,2016年9月版)、典型污染物(自制)和相应的反向数据库进行同时搜索来鉴定肽和蛋白质。在进行汇编、分组和比较来自不同样品的蛋白质组之前,使用proline软件过滤结果(1级肽的保守性,通过采用反向数据库策略,肽长度≥7,每个鉴定的蛋白质分组最少1个特定肽,肽评分计算的肽鉴定fdr<1%)。仅考虑鉴定的具有最小特异性光谱计数为2的蛋白质进行进一步比较。
蛋白质分析和蛋白质印迹
向蛋白质提取物中加入ldsnupage上样染料至1×终浓度,在95℃下煮沸10分钟,用mops跑样缓冲液以还原性10%(w/v)sds-page分离并用硝酸银染色。为了检测带his标签的蛋白质,使用半干印迹系统将通过sds-page分离的多肽转移到硝酸纤维素膜上,并使用兔抗his抗体、辣根过氧化物酶缀合的抗兔抗体和ecl底物(amershambiosciences)以显示his-标签。
烷烃合成酶cdna的克隆及重组烷烃合成酶的纯化
通过酚-氯仿法从小球藻细胞中提取总rna,并使用
变异小球藻(、莱茵衣藻和三角褐指藻gmc氧化还原酶在大肠杆菌中的表达
莱茵衣藻和三角褐指藻的烷烃合成酶,使用莱茵衣藻的正向引物5′-tacttccaatcaatgatgctgggtccgaaaacc-3′(seqidno29),和反向引物5′-tatccacctttactgttctactaaactgcaaccggctgacg-3′(seqidno30),从针对大肠杆菌密码子优化的合成基因扩增。。对于三角褐指藻,正向引物为5′-tacttccaatcaatgaaaaaaagcctgcgtagc-3′(seqidno31),反向引物为5′-tatccacctttactgttctactatgcgcttgcggtg-3′(seqidno32)。将基因克隆到plic07载体中,该载体在atg起始密码子的下游引入编码6his-标签的硫氧还蛋白和烟草蚀刻病毒(tev)蛋白酶切割位点的盒。表达来自变异小球藻nc64a、莱茵衣藻或三角褐指藻的gmc氧化酶的大肠杆菌菌株在37℃下生长,同时以180rpm的速度并且在100μmole.photon.m-2.s-1的光下摇动。当od达到0.6时,加入500μm异丙基β-d-1-硫代半乳糖吡喃糖苷。然后将细胞在37℃下生长24小时,收获,用添加有5%硫酸的甲醇进行转甲基化,并用己烷萃取烃,并如前所述通过gc-ms分析。
在大肠杆菌中共表达gmc氧化还原酶和脂肪酶
用表达来自变异小球藻nc64a(或莱茵衣藻)的gmc氧化酶的载体,和/或表达来自细菌猪葡萄球菌的脂肪酶的载体,转化大肠杆菌菌株并在37℃下在tb培养基中生长。用1mm异丙基β-d-1-硫代半乳糖吡喃糖苷(加入0.2%阿拉伯糖以共表达)诱导表达。然后将细胞在25℃,100μmole.photon.m-2.s-1下培养过夜和6小时,2000μmole.photon.m-2.s-1下培养。收获细胞(4ml,od=5),用添加有5%硫酸的甲醇进行转甲基化,并如前所述通过spme和gc-ms分析烃。
用纯化的酶进行酶促测定
所有测定均在使用具有隔膜的帽所密封的透明玻璃小瓶中进行。使用teorellstenhagen通用缓冲液(33mm柠檬酸一水合物、33mm磷酸、100mmnaoh、16.7mm硼酸,用1nhcl调节到ph8.5)确定最佳ph。其它测定在100mmtrishclph8.5、100mmnacl中进行。反应混合物(500μl)通常含有160nm纯化的酶(储备溶液2.5mgml-1)和400μm底物(在乙醇中的储备溶液10mm)。在一些测定中,脂肪酶与烷烃合成酶一起使用。在这种情况下,底物是三酰基甘油。通常,在led制造的2000μmolphotonsm-2s-1的白光下,样品在200rpm下摇动15分钟。孵育后,将样品在95℃加热15分钟以停止酶促反应。冷却样品并加入内标(十六烷)(45nm,来自在氯仿中的4.5mm储备溶液)。然后将naoh加入到反应混合物中(10μl取自10m的储备溶液中),并将样品涡旋5分钟。然后加入250μl己烷并将样品涡旋5分钟以萃取烷烃和烯烃。通过离心收集己烷相并通过gc-ms-fid分析。通过将100μl的顶部空间直接注入gc-ms进行分析。在图11中,通过使用膜入口质谱法释放13co2来监测1-13c-棕榈酸酯的活性。照明是由cbt-120led(luminous,billerica)提供,强度为850μmolphotonsm-2s-1的蓝色(峰值在460nm,25nmfwhm)或红色(峰值在635nm,15nmfwhm)均是。
烷烃合成酶的荧光
通过uv-vis光谱法(来自secomam的uvikonxs分光光度计)分析酶。在含有100mmtrisph8.5和100mmnacl的缓冲液中,在纯化的酶上测量吸收光谱。使用450nm的激发通量和10nm狭缝在variancaryeclipse上测量荧光光谱(500至700nm)。对于动力学,使用450nm的激发通量和10nm狭缝在540nm处测量荧光。
膜进样质谱(mims)
使用质谱法(型号primab,thermoscientific)监测12co2(m/z=44)和13co2(m/z=45)的在线测量。膜入口系统由热调节氧电极室(hansatech)组成,其通过透气性薄teflon膜(0.001英寸厚度,ysiinc.)(其密封室的底部)连接到质谱仪的真空管线。对于分析,将20μl纯化的酶(2.5mgml-1)和30μl底物(10mm,在二甲基亚砜(13c-棕榈酸)中)加入到1.45ml含有100mmnacl的100mm,ph6.5的tris/acetate/borate缓冲液中,放入测量室,在25℃下进行热调节,并连续搅拌。溶解在介质中的气体通过teflon膜扩散到质谱仪的离子源。
光生物反应器中的培养物
莱茵衣藻cc124(nit1nit2;mt-)和过表达烷烃合成酶基因的莱茵衣藻在基本培养基(harris,1989)中,在作为恒浊器使用的1升光生物反应器(biostataplus,sartoriusstedimbiotech)中培养。使用生物质探针(excellprobe,exner)连续测量a880,并通过注射新鲜培养基将培养物维持在恒定的a880。通过注入koh(0.2n)或hcl(0.2n)将ph维持在恒定值7。使用金属螺旋桨(250rpm)搅拌培养物。将气体流速调节至0.5lmin-1。使用质量流量计(elflow,bronkhorst)产生富含2%(v/v)co2的空气。通过放射状放置在光生物反应器周围的八个荧光管(osramduluxl18w)提供白光。我们使用蓝色滤光片(363specialmedium,leefilters,usa)和红色滤光片(113magenta,leefilters,usa)分别提供蓝光和红光。两种光都具有相同的强度(35μmolphotonsm-2s-1)。
转甲基化
为了将烃与脂肪酸一起量化,使用全细胞的转甲基化。简而言之,向细胞沉淀(1亿个衣藻细胞,2亿个小球藻nc64a细胞,20mlod-1单位的大肠杆菌)中加入2ml含有5%(v/v)硫酸和25μg三十五烷酸酯(来自在氯仿中的2.5mg/ml储备溶液)的甲醇的溶液,以及包括5μg16:0-烷烃(在氯仿中的1mg/ml储备溶液)作为内标。将样品在密封的玻璃管中于85℃孵育90分钟。冷却后,通过加入250μl己烷和500μl0.9%nacl(w/v)来萃取fame和烃。将样品涡旋10分钟,并通过在3000g下离心2分钟将有机相与水相分离。回收己烷相,并在gc-ms/fid中注入1μl。
gc-ms分析
使用以下设置进行在固相微萃取(spme)之后进行的气相色谱-质谱联用(gc-ms)分析。将thermo-fischer气相色谱仪focus系列与thermo-fischerdsqii质谱仪(简单四极杆)偶联并与db-5ht(agilent)非极性毛细管柱(长30m,内径0.25mm,膜厚0.1μm)一起使用。氦载气为1mlmin-1。将烘箱温度设定为在50℃下最初2分钟的保持时间,然后在10℃min-1下从50℃升温至300℃,并在300℃最终3分钟的保持时间。在250℃下以不分流模式(2分钟)注入样品。ms在40-500amu(电子碰撞电离,70ev)下进行全扫描,并使用内标基于总离子电流量化峰值。对于共底物确定,使用柱hp-plotq(直径0.32mm×30m),并在40℃的烘箱温度和单离子监测(m/z40、44、45)下分析co2、13co2和氩气。
gc-ms/fid分析
通过与质谱和火焰离子化检测(gc-ms/fid)联用气相色谱仅在转甲基化反应后进行分析,以便一起定量脂肪酸和烃。在agilent7890a气相色谱仪上进行分析,该仪器与agilent5975c质谱仪(简单四极杆)偶联。使用zebron7hg-g007-11(phenomenex)极性毛细管柱(长30m,内径0.25mm,膜厚0.25μm)。氢载气为1mlmin-1。烘箱温度设定为以60℃最初2分钟的保持时间为,第一个斜坡以20℃min-1从60℃到150℃,在150℃时5分钟的保持时间,然后是第二个斜坡以6℃min-1从150℃至240℃,在240℃下最后3分钟的保持时间。在250℃下以不分流模式(1分钟)注入样品。在40-350amu(70ev的电子碰撞电离)下进行ms全扫描,并使用内标基于fid信号量化峰值。
系统发育
为了构建系统发育树,从cyanobase(http://genome.kazusa.or.jp/cyanobase/)、ncbi(http://www.ncbi.nlm.nih.gov/)、phytozome(https://phytozome.jgi.doe.gov)或cyanidioschyzonmerolae(http://merolae.biol.s.u-tokyo.ac.jp/)中检提取了来自原核生物和真核生物的gmc氧化还原酶的56个氨基酸序列。序列与mafft第7版程序比对。将所得的比对用seaview第4版手动精制,并且从进一步分析中去除同源性令人怀疑的区域。保留了总共266个氨基酸位置用于系统发育分析。该树是使用neighbor-joining(nj)获得的,用到phylogeneticinferencepackagephylip版本3.69中的方法。protdist程序用于创建距离矩阵。neighbor程序用于nj分析,序列输入顺序随机化(20个jumbles)。seqboot和consense程序分别用于100次重复和一致树重建的自举值计算。使用dendroscope第3版绘制系统发育树。
序列表
<110>cea
cnrs
<120>新型脂肪酸脱羧酶及其用途
<130>b2241pc
<160>32
<170>patentinversion3.5
<210>1
<211>594
<212>prt
<213>变异小球藻
<400>1
argalaseralavalgluaspilearglysvalleuseraspserser
151015
serprovalalaglyglnlystyrasptyrileleuvalglyglygly
202530
thralaalacysvalleualaasnargleuseralaaspglyserlys
354045
argvalleuvalleuglualaglyproaspasnthrserargaspval
505560
lysileproalaalailethrargleupheargserproleuasptrp
65707580
asnleuphesergluleuglngluglnleualagluargglniletyr
859095
metalaargglyargleuleuglyglyserseralathrasnalathr
100105110
leutyrhisargglyalaalaglyasptyraspalatrpglyvalglu
115120125
glytrpsersergluaspvalleusertrpphevalglnalagluthr
130135140
asnalaasppheglyproglyalatyrhisglyserglyglypromet
145150155160
argvalgluasnproargtyrthrasnlysglnleuhisthralaphe
165170175
phelysalaalaglugluvalglyleuthrproasnserasppheasn
180185190
asptrpserhisasphisalaglytyrglythrpheglnvalmetgln
195200205
asplysglythrargalaaspmettyrargglntyrleulysproval
210215220
leuglyargargasnleuglnvalleuthrglyalaalavalthrlys
225230235240
valasnileaspglnalaalaglylysalaglnalaleuglyvalglu
245250255
pheserthraspglyprothrglygluargleuseralagluleuala
260265270
proglyglygluvalilemetcysalaglyalavalhisthrprophe
275280285
leuleulyshisserglyvalglyproseralagluleulysgluphe
290295300
glyileprovalvalserasnleualaglyvalglyglnasnleugln
305310315320
aspglnproalacysleuthralaalaprovallysglulystyrasp
325330335
glyilealaileserasphisiletyrasnglulysglyglnilearg
340345350
lysargalailealasertyrleuleuglyglyargglyglyleuthr
355360365
serthrglycysaspargglyalaphevalargthralaglyglnala
370375380
leuproaspleuglnvalargphevalproglymetalaleuasppro
385390395400
aspglyvalserthrtyrvalargphealalyspheglnserglngly
405410415
leulystrpproserglyilethrmetglnleuilealacysargpro
420425430
glnserthrglyservalglyleulysseralaaspprophealapro
435440445
prolysleuserproglytyrleuthrasplysaspglyalaaspleu
450455460
alathrleuarglysglyilehistrpalaargaspvalalaargser
465470475480
seralaleuserglutyrleuaspglygluleupheproglysergly
485490495
valvalseraspaspglnileaspglutyrileargargserilehis
500505510
serserasnalailethrglythrcyslysmetglyasnalaglyasp
515520525
serserservalvalaspasnglnleuargvalhisglyvalglugly
530535540
leuargvalvalaspalaservalvalprolysileproglyglygln
545550555560
thrglyalaprovalvalmetilealagluargalaalaalaleuleu
565570575
thrglylysalathrileglyalaseralaalaalaproalathrval
580585590
alaala
<210>2
<211>654
<212>prt
<213>变异小球藻
<400>2
metalaserilethrserargalaseralaargalasercyssergln
151015
alaasnthrargalaglyargvalalaleuserglyglyalaleuleu
202530
argproalaargproalaargserphevalproalaarglysglngln
354045
glnglyalavalargargglyglyalaleuseralaargalaserala
505560
valgluaspilearglysvalleuseraspserserserprovalala
65707580
glyglnlystyrasptyrileleuvalglyglyglythralaalacys
859095
valleualaasnargleuseralaaspglyserlysargvalleuval
100105110
leuglualaglyproaspasnthrserargaspvallysileproala
115120125
alailethrargleupheargserproleuasptrpasnleupheser
130135140
gluleuglngluglnleualagluargglniletyrmetalaarggly
145150155160
argleuleuglyglyserseralathrasnalathrleutyrhisarg
165170175
glyalaalaglyasptyraspalatrpglyvalgluglytrpserser
180185190
gluaspvalleusertrpphevalglnalagluthrasnalaaspphe
195200205
glyproglyalatyrhisglyserglyglyprometargvalgluasn
210215220
proargtyrthrasnlysglnleuhisthralaphephelysalaala
225230235240
glugluvalglyleuthrproasnserasppheasnasptrpserhis
245250255
asphisalaglytyrglythrpheglnvalmetglnasplysglythr
260265270
argalaaspmettyrargglntyrleulysprovalleuglyargarg
275280285
asnleuglnvalleuthrglyalaalavalthrlysvalasnileasp
290295300
glnalaalaglylysalaglnalaleuglyvalglupheserthrasp
305310315320
glyprothrglygluargleuseralagluleualaproglyglyglu
325330335
valilemetcysalaglyalavalhisthrpropheleuleulyshis
340345350
serglyvalglyproseralagluleulysglupheglyileproval
355360365
valserasnleualaglyvalglyglnasnleuglnaspglnproala
370375380
cysleuthralaalaprovallysglulystyraspglyilealaile
385390395400
serasphisiletyrasnglulysglyglnilearglysargalaile
405410415
alasertyrleuleuglyglyargglyglyleuthrserthrglycys
420425430
aspargglyalaphevalargthralaglyglnalaleuproaspleu
435440445
glnvalargphevalproglymetalaleuaspproaspglyvalser
450455460
thrtyrvalargphealalyspheglnserglnglyleulystrppro
465470475480
serglyilethrmetglnleuilealacysargproglnserthrgly
485490495
servalglyleulysseralaaspprophealaproprolysleuser
500505510
proglytyrleuthrasplysaspglyalaaspleualathrleuarg
515520525
lysglyilehistrpalaargaspvalalaargserseralaleuser
530535540
glutyrleuaspglygluleupheproglyserglyvalvalserasp
545550555560
aspglnileaspglutyrileargargserilehisserserasnala
565570575
ilethrglythrcyslysmetglyasnalaglyaspserserserval
580585590
valaspasnglnleuargvalhisglyvalgluglyleuargvalval
595600605
aspalaservalvalprolysileproglyglyglnthrglyalapro
610615620
valvalmetilealagluargalaalaalaleuleuthrglylysala
625630635640
thrileglyalaseralaalaalaproalathrvalalaala
645650
<210>3
<211>729
<212>prt
<213>人工序列
<220>
<223>shgmc
<400>3
metglyhishishishishishisserasplysileilehisleuthr
151015
aspaspserpheaspthraspvalleulysalaaspglyalaileleu
202530
valaspphetrpalaglutrpcysglyprocyslysmetilealapro
354045
ileleuaspgluilealaaspglutyrglnglylysleuthrvalala
505560
lysleuasnileaspglnasnproglythralaprolystyrglyile
65707580
argglyileprothrleuleuleuphelysasnglygluvalalaala
859095
thrlysvalglyalaleuserlysglyglnleulysglupheleuasp
100105110
alaasnleualaglyserglyserglyvalserglyvalaspleugly
115120125
thrgluasnleutyrpheglnseralaseralavalgluaspilearg
130135140
lysvalleuseraspserserserprovalalaglyglnlystyrasp
145150155160
tyrileleuvalglyglyglythralaalacysvalleualaasnarg
165170175
leuseralaaspglyserlysargvalleuvalleuglualaglypro
180185190
aspasnthrserargaspvallysileproalaalailethrargleu
195200205
pheargserproleuasptrpasnleuphesergluleuglnglugln
210215220
leualagluargglniletyrmetalaargglyargleuleuglygly
225230235240
serseralathrasnalathrleutyrhisargglyalaalaglyasp
245250255
tyraspalatrpglyvalgluglytrpsersergluaspvalleuser
260265270
trpphevalglnalagluthrasnalaasppheglyproglyalatyr
275280285
hisglyserglyglyprometargvalgluasnproargtyrthrasn
290295300
lysglnleuhisthralaphephelysalaalaglugluvalglyleu
305310315320
thrproasnserasppheasnasptrpserhisasphisalaglytyr
325330335
glythrpheglnvalmetglnasplysglythrargalaaspmettyr
340345350
argglntyrleulysprovalleuglyargargasnleuglnvalleu
355360365
thrglyalaalavalthrlysvalasnileaspglnalaalaglylys
370375380
alaglnalaleuglyvalglupheserthraspglyprothrglyglu
385390395400
argleuseralagluleualaproglyglygluvalilemetcysala
405410415
glyalavalhisthrpropheleuleulyshisserglyvalglypro
420425430
seralagluleulysglupheglyileprovalvalserasnleuala
435440445
glyvalglyglnasnleuglnaspglnproalacysleuthralaala
450455460
provallysglulystyraspglyilealaileserasphisiletyr
465470475480
asnglulysglyglnilearglysargalailealasertyrleuleu
485490495
glyglyargglyglyleuthrserthrglycysaspargglyalaphe
500505510
valargthralaglyglnalaleuproaspleuglnvalargpheval
515520525
proglymetalaleuaspproaspglyvalserthrtyrvalargphe
530535540
alalyspheglnserglnglyleulystrpproserglyilethrmet
545550555560
glnleuilealacysargproglnserthrglyservalglyleulys
565570575
seralaaspprophealaproprolysleuserproglytyrleuthr
580585590
asplysaspglyalaaspleualathrleuarglysglyilehistrp
595600605
alaargaspvalalaargserseralaleuserglutyrleuaspgly
610615620
gluleupheproglyserglyvalvalseraspaspglnileaspglu
625630635640
tyrileargargserilehisserserasnalailethrglythrcys
645650655
lysmetglyasnalaglyaspserserservalvalaspasnglnleu
660665670
argvalhisglyvalgluglyleuargvalvalaspalaservalval
675680685
prolysileproglyglyglnthrglyalaprovalvalmetileala
690695700
gluargalaalaalaleuleuthrglylysalathrileglyalaser
705710715720
alaalaalaproalathrvalalaala
725
<210>4
<211>15
<212>prt
<213>人工序列
<220>
<223>共有序列
<220>
<221>misc_feature
<222>(2)..(2)
<223>x是任意氨基酸
<220>
<221>misc_feature
<222>(4)..(7)
<223>x是任意氨基酸
<220>
<221>misc_feature
<222>(9)..(9)
<223>x是d或e
<220>
<221>misc_feature
<222>(10)..(10)
<223>x是任意氨基酸
<220>
<221>misc_feature
<222>(12)..(12)
<223>xisaorg
<220>
<221>misc_feature
<222>(14)..(14)
<223>x是任意氨基酸
<220>
<221>misc_feature
<222>(15)..(15)
<223>x是k或r
<400>4
glyxaaleuxaaxaaxaaxaacysxaaxaaglyxaaphexaaxaa
151015
<210>5
<211>580
<212>prt
<213>莱茵衣藻
<400>5
leuservalargalaalaalaglyproalaglyserglulyspheasp
151015
tyrvalleuvalglyglyglythralasercysvalleualaasnlys
202530
leuseralaaspglyasnlyslysvalleuvalleuglualaglypro
354045
thrglyaspalametgluvalalavalproalaglyilethrargleu
505560
phealahisprovalmetasptrpglymetserserleuthrglnlys
65707580
glnleuvalalaarggluiletyrleualaargglyargmetleugly
859095
glyserserglyserasnalathrleutyrhisargglyseralaala
100105110
asptyraspalatrpglyleugluglytrpserserlysaspvalleu
115120125
asptrpphevallysalaglucystyralaaspglyprolysprotyr
130135140
hisglythrglyglysermetasnthrgluglnproargtyrgluasn
145150155160
valleuhisaspgluphephelysalaalaalaalathrglyleupro
165170175
alaasnproasppheasnasptrpserhisproglnaspglyphegly
180185190
glupheglnvalserglnlyslysglyglnargalaaspthrtyrarg
195200205
thrtyrleulysproalametalaargglyasnleulysvalvalile
210215220
glyalaargalathrlysvalasnileglulysglyserserglyala
225230235240
argthrthrglyvalglutyralametglnglnpheglyaspargphe
245250255
thralagluleualaproglyglygluvalleumetcysserglyala
260265270
valhisthrprohisleuleumetleuserglyvalglyproalaala
275280285
thrleulysgluhisglyileaspvalvalseraspleuserglyval
290295300
glyglnasnleuglnasphisproalaalavalleualaalaargala
305310315320
lysproglupheglulysleuservalthrsergluvaltyraspasp
325330335
lyscysasnilelysleuglyalavalalaglntyrleupheglnarg
340345350
argglyproleualathrthrglycysasphisglyalaphevalarg
355360365
thrserserserleuserglnproaspleuglnmetargphevalpro
370375380
glycysalaleuaspproaspglyvallyssertyrilevalphegly
385390395400
gluleulyslysglnglyargalatrpproglyglyilethrleugln
405410415
leuleualaileargalalysserlysglyserileglyleulysala
420425430
alaasppropheileasnproalaileasnileasntyrpheserasp
435440445
proalaaspleualathrleuvalasnalavallysmetalaarglys
450455460
ilealaalaglngluproleulyslystyrleuglnglugluthrphe
465470475480
proglygluargalaserserasplysaspleugluglutyrilearg
485490495
argthrvalhisserglyasnalaleuvalglythralaalametgly
500505510
alaserproalaalaglyalavalvalserseralaaspleulysval
515520525
pheglyvalgluglyleuargvalvalaspalaservalleuproarg
530535540
ileproglyglyglnthrglyalaalathrvalmetvalalagluarg
545550555560
alaalaalaleuleuargglyglnalathrilealaproserarggln
565570575
provalalaval
580
<210>6
<211>611
<212>prt
<213>莱茵衣藻
<400>6
metmetleuglyprolysthrvalthrargglyalathrlysglyala
151015
alaproargsermetalaalaargargvalglyglyalaargargleu
202530
servalargalaalaalaglyproalaglyserglulyspheasptyr
354045
valleuvalglyglyglythralasercysvalleualaasnlysleu
505560
seralaaspglyasnlyslysvalleuvalleuglualaglyprothr
65707580
glyaspalametgluvalalavalproalaglyilethrargleuphe
859095
alahisprovalmetasptrpglymetserserleuthrglnlysgln
100105110
leuvalalaarggluiletyrleualaargglyargmetleuglygly
115120125
serserglyserasnalathrleutyrhisargglyseralaalaasp
130135140
tyraspalatrpglyleugluglytrpserserlysaspvalleuasp
145150155160
trpphevallysalaglucystyralaaspglyprolysprotyrhis
165170175
glythrglyglysermetasnthrgluglnproargtyrgluasnval
180185190
leuhisaspgluphephelysalaalaalaalathrglyleuproala
195200205
asnproasppheasnasptrpserhisproglnaspglypheglyglu
210215220
pheglnvalserglnlyslysglyglnargalaaspthrtyrargthr
225230235240
tyrleulysproalametalaargglyasnleulysvalvalilegly
245250255
alaargalathrlysvalasnileglulysglyserserglyalaarg
260265270
thrthrglyvalglutyralametglnglnpheglyaspargphethr
275280285
alagluleualaproglyglygluvalleumetcysserglyalaval
290295300
histhrprohisleuleumetleuserglyvalglyproalaalathr
305310315320
leulysgluhisglyileaspvalvalseraspleuserglyvalgly
325330335
glnasnleuglnasphisproalaalavalleualaalaargalalys
340345350
proglupheglulysleuservalthrsergluvaltyraspasplys
355360365
cysasnilelysleuglyalavalalaglntyrleupheglnargarg
370375380
glyproleualathrthrglycysasphisglyalaphevalargthr
385390395400
serserserleuserglnproaspleuglnmetargphevalprogly
405410415
cysalaleuaspproaspglyvallyssertyrilevalpheglyglu
420425430
leulyslysglnglyargalatrpproglyglyilethrleuglnleu
435440445
leualaileargalalysserlysglyserileglyleulysalaala
450455460
asppropheileasnproalaileasnileasntyrpheserasppro
465470475480
alaaspleualathrleuvalasnalavallysmetalaarglysile
485490495
alaalaglngluproleulyslystyrleuglnglugluthrphepro
500505510
glygluargalaserserasplysaspleugluglutyrileargarg
515520525
thrvalhisserglyasnalaleuvalglythralaalametglyala
530535540
serproalaalaglyalavalvalserseralaaspleulysvalphe
545550555560
glyvalgluglyleuargvalvalaspalaservalleuproargile
565570575
proglyglyglnthrglyalaalathrvalmetvalalagluargala
580585590
alaalaleuleuargglyglnalathrilealaproserargglnpro
595600605
valalaval
610
<210>7
<211>568
<212>prt
<213>三角褐指藻
<400>7
tyrasptyrileilecysglyglyglyleualaglycysvalleuala
151015
gluargleuserglnaspgluserlysargvalleuvalleugluala
202530
glyglyserasptyrlysserleupheileargileproalaglyval
354045
leuargleupheargserlystyrasptrpglnhisgluthrglygly
505560
glulysglycysasnglyargasnvalpheleuglnargglylysile
65707580
leuglyglysersercysthrasnvalcysleuhishisargglyser
859095
alagluasptyrasnsertrpasnileproglytrpthralathrasp
100105110
valleuprophephelysglnserglnlysaspgluthrglyargasp
115120125
alathrphehisglyalaaspglyglutrpvalmetaspgluvalarg
130135140
tyrglnasnproleuserlysleupheleugluvalglyglualaala
145150155160
glyleuglythrasnaspasppheasnasntrpserhisproglnasp
165170175
glyvalglyargpheglnvalsergluvalasnglygluargcysser
180185190
glyalathralapheleuserlysalaalalysargserasnvalile
195200205
valargthrglythrmetvalargargileasppheaspgluthrlys
210215220
thralalysglyilethrtyraspleumetglyaspaspthrcysthr
225230235240
thrpheglnalacysleulysgluglyglygluvalleuvalthrgly
245250255
glyalailealaserproglnleuleumetcysserglyileglypro
260265270
glylyshisleuargserleuglyileprovalvalhisaspasnser
275280285
alavalglygluasnleuglnasphisproalaalavalvalserphe
290295300
lysthrproglnlysglyvalservalthrserlysleuargleuphe
305310315320
glylysthrasnproileprovalpheglntrpleuphephelysser
325330335
glyleuleuthrserthrglycysasphisglyalaphevalargthr
340345350
seraspserleugluglnproaspleuglnileargpheleualaala
355360365
argalaleuglyproaspglymetthrthrtyrthrlyspheargthr
370375380
metlysthrvalgluaspglytyrserpheglnservalalacysarg
385390395400
alalysserlysglyargileargleuserserserasnserhisval
405410415
lysprometileaspglyglytyrleuserasnglnaspaspleuala
420425430
thrleuargalaglyilelysleuglyargmetleuglyasnargpro
435440445
glutrpglyglutyrleuglyglngluvaltyrproglyproaspval
450455460
glnthraspglugluileaspglutyrileargasnserleuhisthr
465470475480
alaasnalaleuthrglythrcyslysmetglythrglyargglyala
485490495
valvalglyproaspleuargvalileglyvalasnglyvalargval
500505510
glyaspserservalpheprocysileproglyglyglnthralathr
515520525
prothrvalmetileglyaspargalaalavalphevalargglnpro
530535540
valserglnleuasnilegluilepheargglulysglyglythrhis
545550555560
proglyalathrthralaserala
565
<210>8
<211>614
<212>prt
<213>三角褐指藻
<400>8
metlyslysserleuargserleuleuvaltrpthrleuservalser
151015
seralaseralathrthrmetalaglyalalysproleuglyasnval
202530
proargalaglngluaspgluarglysvalvalalagluprotyrasp
354045
tyrileilecysglyglyglyleualaglycysvalleualagluarg
505560
leuserglnaspgluserlysargvalleuvalleuglualaglygly
65707580
serasptyrlysserleupheileargileproalaglyvalleuarg
859095
leupheargserlystyrasptrpglnhisgluthrglyglyglulys
100105110
glycysasnglyargasnvalpheleuglnargglylysileleugly
115120125
glysersercysthrasnvalcysleuhishisargglyseralaglu
130135140
asptyrasnsertrpasnileproglytrpthralathraspvalleu
145150155160
prophephelysglnserglnlysaspgluthrglyargaspalathr
165170175
phehisglyalaaspglyglutrpvalmetaspgluvalargtyrgln
180185190
asnproleuserlysleupheleugluvalglyglualaalaglyleu
195200205
glythrasnaspasppheasnasntrpserhisproglnaspglyval
210215220
glyargpheglnvalsergluvalasnglygluargcysserglyala
225230235240
thralapheleuserlysalaalalysargserasnvalilevalarg
245250255
thrglythrmetvalargargileasppheaspgluthrlysthrala
260265270
lysglyilethrtyraspleumetglyaspaspthrcysthrthrphe
275280285
glnalacysleulysgluglyglygluvalleuvalthrglyglyala
290295300
ilealaserproglnleuleumetcysserglyileglyproglylys
305310315320
hisleuargserleuglyileprovalvalhisaspasnseralaval
325330335
glygluasnleuglnasphisproalaalavalvalserphelysthr
340345350
proglnlysglyvalservalthrserlysleuargleupheglylys
355360365
thrasnproileprovalpheglntrpleuphephelysserglyleu
370375380
leuthrserthrglycysasphisglyalaphevalargthrserasp
385390395400
serleugluglnproaspleuglnileargpheleualaalaargala
405410415
leuglyproaspglymetthrthrtyrthrlyspheargthrmetlys
420425430
thrvalgluaspglytyrserpheglnservalalacysargalalys
435440445
serlysglyargileargleuserserserasnserhisvallyspro
450455460
metileaspglyglytyrleuserasnglnaspaspleualathrleu
465470475480
argalaglyilelysleuglyargmetleuglyasnargproglutrp
485490495
glyglutyrleuglyglngluvaltyrproglyproaspvalglnthr
500505510
aspglugluileaspglutyrileargasnserleuhisthralaasn
515520525
alaleuthrglythrcyslysmetglythrglyargglyalavalval
530535540
glyproaspleuargvalileglyvalasnglyvalargvalglyasp
545550555560
serservalpheprocysileproglyglyglnthralathrprothr
565570575
valmetileglyaspargalaalavalphevalargglnprovalser
580585590
glnleuasnilegluilepheargglulysglyglythrhisprogly
595600605
alathrthralaserala
610
<210>9
<211>585
<212>prt
<213>胶球藻
<400>9
alaleuargvalargalaileilelysseraspasnproalaalaasp
151015
lystyrasppheileleuvalglyglyglythralaglycysvalleu
202530
alaasnargleuthralaaspglyserlyslysvalleuleuleuglu
354045
alaglyglyalaasnlysalaarggluvalargthrproalaglyleu
505560
proargleuphelysseralaleuasptrpasnleutyrserserleu
65707580
glnglnalaalaseraspargseriletyrleualaargglylysleu
859095
leuglyglyserseralathrasnalathrleutyrhisargglythr
100105110
alaalaasptyraspalatrpglyvalproglytrpthrserglnasp
115120125
alaleuargtrppheileglnalagluasnasncysargglyileglu
130135140
aspglyvalhisglythrglyglyleumetargvalgluasnproarg
145150155160
tyrasnasnproleuhisgluvalphepheglnalaalalysglnala
165170175
glyleuprogluasnaspasnpheasnasntrpglyargserglnala
180185190
glytyrglyglupheglnvalthrhisserlysglygluargalaasp
195200205
cyspheargmettyrleugluprovalmetglyargserasnleuthr
210215220
valleuthrglyalalysthrleulysilegluthrglulyssergly
225230235240
glyalathrvalserargglyvalthrpheglnvalasnglyglnasp
245250255
glyserlyshisseralagluleualaalaglyglygluvalvalleu
260265270
cysalaglyserilehisserproglnileleuglnleuserglyile
275280285
glyproglnalagluleuargserlysaspileprovalvalalaasp
290295300
leuproglyvalglyglnasnmetglnasphisproalacysleuser
305310315320
alaphetyrleulysgluseralaglyproileservalthraspglu
325330335
leuleuhisthrasnglyargileargalaargalaileleulystyr
340345350
leuleuphelyslysglyproleualathrthrglycysasphisgly
355360365
alaphevallysthralaglyglnsergluproaspleuglnilearg
370375380
phevalproglyleualaleuaspproaspglyileglysertyrthr
385390395400
alapheglylysmetlysaspglnlystrpproserglyilethrphe
405410415
glnleuleuglyvalargprolysserargglyservalglyleuarg
420425430
seraspaspprotrpaspalaprolysleuaspileglypheleuthr
435440445
asplysgluglyalaaspleualathrleuargserglyilelysleu
450455460
serarggluilealaalagluproalapheglyalatyrvalglyasn
465470475480
gluleuhisproglyalaalaalaserseraspseralaileaspser
485490495
pheileargaspthrvalhisserglyasnalaasnvalglythrcys
500505510
sermetglyvalasnglyasnalavalvalaspproserleuargval
515520525
pheglyileargglyleuargvalalaaspalaservalileproval
530535540
ileproglyglyglnthrglyalaalathrvalmetvalalagluarg
545550555560
alaalagluileleuleuglyserasnglnlysglnproalaalaala
565570575
valproalaalaglnproalaleuala
580585
<210>10
<211>580
<212>prt
<213>强壮团藻
<400>10
provalalavallysalaalaalaservalglyserglulyspheasp
151015
tyrileleuvalglyglyglythralaglycysvalleualaasnlys
202530
leuseralaasnglyserlyslysvalleuvalleuglualaglypro
354045
thrglyaspalametgluvalalavalproalaglyilealaargleu
505560
phealahisprovalpheasptrpglymetserserleuthrglngln
65707580
glnleuvalalaarggluiletyrleualaargglyargleuleugly
859095
glyserserglythrasnalathrleutyrhisargglythrproala
100105110
asptyraspsertrpglyleugluglytrpthrserlysaspleuleu
115120125
asptrpphevallysalaglucystyrglyaspglyproargalaphe
130135140
hisglyglnserglysermetasnvalgluglnproargtyrglnasn
145150155160
valleuhisaspgluphepheargalaalaalaalaalaglyleupro
165170175
alaasngluasppheasnasptrpserargproglngluglytyrgly
180185190
glupheglnvalalaglnlysasnglygluargalaaspthrtyrarg
195200205
thrtyrleulysproalametglyargaspasnleulysvalmetthr
210215220
glyalaargthrthrlysvalhisileglulysserserthrglypro
225230235240
argalaargglyvalglutyralathrglnglnpheglygluargtyr
245250255
thralagluleuthrproglyglygluvalleumetcysthrglyala
260265270
valhisthrprohisleuleumetleuserglyileglyproalapro
275280285
thrleuleugluhisglyleuaspvalileserserleuproglyval
290295300
glyalaasnleuglnasphisproalaalavalleualavalargala
305310315320
lysprogluphegluglyleuservalthrsergluiletyraspser
325330335
lyscysasnileargleuglyalavalmetlystyrleupheglyarg
340345350
argglyproleualathrthrglycysasphisglyalaphevalarg
355360365
thrseralaserhisserglnproaspleuglnmetargphevalpro
370375380
glycysalaleuaspproaspglyvallyssertyrilevalphegly
385390395400
gluleulyslysglnglyargalatrpproglyglyilethrleugln
405410415
leuleuglyileargalalysserargglyserileglyleulysala
420425430
alaasppropheileasnproalaileasnileasntyrpheserasp
435440445
progluaspleualathrleulysasnglyvalargilealaargglu
450455460
ilevalalaglngluproleuarglystyrleuleuglugluthrphe
465470475480
proglygluargalaasnthrasplysaspilegluglutyrvalarg
485490495
argthrvalhisserglyasnalaleuvalglythrcysalametgly
500505510
thrthrproalaserglyalavalvalserseralaaspleulysval
515520525
pheglyvalaspglyleuargvalvalaspalaservalleuproarg
530535540
ileproglyglyglnthrglyalaalathrvalmetvalalagluarg
545550555560
alaalaalametleuleuglyglnalathrilethrserargargglu
565570575
proalaalaval
580
<210>11
<211>557
<212>prt
<213>长囊水云
<400>11
valproalaalaargtyralathrserservalsermetservalala
151015
glugluglyhislyspheileileileglyglyglythralaglycys
202530
valleualaasnargleuseralaasplysaspasnservalleuval
354045
leuglualaglyserglulyspheasnaspargasnilelysmetpro
505560
ilealaileleuargleuphelysservalpheasptrpglyphegln
65707580
sergluasnglulysphealathrglyaspglyiletyrleucysarg
859095
glylysvalleuglyglysersercysthrasnvalmetleutyrhis
100105110
argglygluglualaasptyraspalatrpglyvalaspglytrplys
115120125
glylysaspvalleuprotyrphelyslysalagluasnasnargser
130135140
lyslyslysglygluphehisglylysglyglyleumetglnvalglu
145150155160
asnalaargtyrmetasnproleuthrlysleuphephelysalacys
165170175
gluglnalaglyleusergluasngluasppheasnasptrpserhis
180185190
serglngluglypheglyargpheglnvalalaglnlysargglylys
195200205
argcysseralaalasersertyrleulysglualametglyarglys
210215220
asnleuaspvalglnthrseralaglnilethrlysvalleuileglu
225230235240
asnglyglyalaileglyvalglutyrvalargaspglyglulyslys
245250255
ilealalysleualavalglyglygluileleuleualaglyglyala
260265270
ileserserproglnvalleumetleuserglyvalglyproalaglu
275280285
hisleuargserlysglyilegluvallysserasnvalproglyval
290295300
glylysasnleuargasphisproalavalthrvalmetalaaspile
305310315320
asnlysproileserilethrasplysvalleulysgluglysergly
325330335
aspvalasnlysilethralaleuglntrpleuleuthrglythrgly
340345350
proleuthrserproglycysgluasnglyalaphephelysthrthr
355360365
proasplysalaalaalaaspleuglnleuargphevalproglyarg
370375380
serthrthrproaspglyvallysalatyrasnthrileglythrlys
385390395400
glyargproproserglyvalthrvalglnvalvalglyileargpro
405410415
glnsergluglyhisvalgluleuargserserasppropheasplys
420425430
prohisilevalthrasntyrleugluserglygluaspmetalaser
435440445
leuthrasnglyileglumetalaarglysleupheaspglngluala
450455460
pheglyglumetvalasplysgluvalpheproglyargaspasnlys
465470475480
gluileserglutyrilelysserthrvalhisseralaasnalaleu
485490495
valglythrcyslysmetglyglugluseraspasnmetservalval
500505510
asnseralaleulysvallysglyvalalaglyleuargvalileasp
515520525
serservalmetproserileproglyglyglnthralaalaprothr
530535540
ilemetilealaglulysalaalaaspmetleumetala
545550555
<210>12
<211>601
<212>prt
<213>赫氏圆石藻
<400>12
leuargglyglyserglyvalthrglyglyserleuglyargglygly
151015
glyserproalaileaspglyglupheasptyrileilevalglygly
202530
glyalaalaglycysvalleualaasnargleuseralaaspproala
354045
hisargvalleuleuileglualaglyglyaspalaserargasplys
505560
argalaglnvalprotrpalaphethrlysleuleuargserglutyr
65707580
asptrpaspphehisvalglualaglualaalavalasnglnglnglu
859095
valtyrleucysargglylysalaleuglyglyserservalthrasn
100105110
valmetleutyrhisargglyserproalaasptyraspalatrpglu
115120125
glualaglyalaargglytrpglyalalysaspvalleuprotyrtyr
130135140
leuargvalgluasptyrglyaspglyalaserglntyrhisalaval
145150155160
glyglyhisvalservalglngluvalprotyrglnasnglnleuser
165170175
alathrpheleuargalametglyglnleuglypheargproasngly
180185190
asppheasnasptrpserserproglngluglytyrglyargtyrlys
195200205
valthrglnargalaglyargargcysthralaalaaspglytyrleu
210215220
alaalaalaarggluargalaasnleuvalvalvalthrglyalagln
225230235240
alathrargleualaleuaspseralatyraspglyalaglyargleu
245250255
glnvalserglyvalgluphealaargglyaspgluarggluprocys
260265270
servalargleualaargglyglyglualavalleucysalaglyala
275280285
valglnthrprohisleuleuleuleuserglyileglyproalaglu
290295300
hisleuarggluvalglyvalprovalargalaaspleuproglyval
305310315320
glyserglyleuglnasphisproalavalvalvalsertyrgluser
325330335
lyslysalavalalaalathraspaspalaleuleulysglytyrala
340345350
serleuvalasnproleualametleuargtrpleuleupheglyarg
355360365
glyproleualacysalaalacysasphisglyglyphevalargser
370375380
serproaspleuaspglnproaspvalglnileargphevalproala
385390395400
argalaserseralaserglymetasnthrleuilegluleuglyarg
405410415
argalaargpheleuproglypheserthrglnvalvalalacysarg
420425430
proargsergluglyargvalargleuargseralaasppropheala
435440445
lysproileilegluglyilehisleuglyalaalagluaspvalala
450455460
serleuarghisglyileargleuglyargglnvalcysalaalaala
465470475480
alapheaspglutyrargglyglugluvalpheproglyalaalaval
485490495
glnseraspgluglnileaspglutyrileargserservalhisser
500505510
alaasnalaleuthrsersercysargmetglyaspproserasppro
515520525
alaalavalleuaspserhisleuargvalargglyvalglyglyleu
530535540
argvalalaaspalaseralametproargileileglyglyglnthr
545550555560
glnalaprothrtyrmetleualagluargalaalaaspileleuleu
565570575
hisalaargleuglnalahisgluproalathrgluservalsergln
580585590
argleugluvalalaalaalaalaleu
595600
<210>13
<211>533
<212>prt
<213>抑食金球藻
<400>13
leusergluaspproserlyslysvalleuvalleuglualaglyasp
151015
argglyproasnserproleuvallysileprovalalaileleulys
202530
leuphelysseralatyrasptrpasnphealathrargproserglu
354045
alavalalaaspargserleutyrvalcysargglylysglyleugly
505560
glyserserleuthrasnvalmetleutyrasnargglyseralaasn
65707580
asptyraspalatrpalaalaalacysglyaspaspsertrpglyala
859095
gluglumetleuglytyrphelyslysalagluaspcysleuvalpro
100105110
alahisargalaasnhistyrhisglyvalglyglyprotyralaser
115120125
serhisvalprotyrthrasnglumetserthralaphevalgluala
130135140
alavalgluaspglyglyvalargasnglyasppheasnasptrpser
145150155160
thrserglnvalglypheglyargphealavalserglnarglysgly
165170175
alaargvalaspalaalathralatyrleuproarglysvalargarg
180185190
argalaasnleuaspvalvalargglyalaalaleuserglyvalthr
195200205
trpasnalaasnlysalathrglyvalgluphealapheglyglyval
210215220
serglyilealacysglyglygluvalileleuserglyglyalaval
225230235240
hisserproglnmetleumetleuserglyvalglyalalysalagln
245250255
leugluglupheglyileprovalvalalaaspargproglyvalgly
260265270
lysasnleuglnasphisproalacysleuvalsertrpargglyser
275280285
alalysalaglnglylysserhisserthrglnleuargileprogly
290295300
thrthrlysthrserprolysalaleuleuglntrpleupheleugly
305310315320
argglyproleualaserproglycysasphisglyglyphealalys
325330335
valglyalaglyaspglyaspcysaspvalglnpheargpheleuala
340345350
thrlysserilethrproaspglymetserthrileseraspsertyr
355360365
glualaalavalasphisproaspglyleuthrileglnthrileval
370375380
alaargprolysserargalaglygluvallysleualaserargasp
385390395400
proalaalalysprovalilegluasnalatyrleuseraspgluala
405410415
aspvalmetthrmetvallysalaleuglnlysalaargserileala
420425430
serargalaproleuseralatyralaglyhisgluglupheprogly
435440445
gluaspvalalaaspgluargglnleualaalatyrvalargasnthr
450455460
alahisthralaasnalavalvalglythrcyslysmetglygluser
465470475480
seraspalaleualavalvalaspasnhisleulysvalileglyval
485490495
serasnleuargvalvalaspalaservalmetprothrleuprogly
500505510
glyglnthralaalaserthrvalalaleualaglulysalaalaasp
515520525
leuilelysglygly
530
<210>14
<211>1236
<212>prt
<213>海洋富油微拟球藻
<400>14
leuserlysthrthrileglyleuglnserphevalthralaasntyr
151015
glyvalargargalaileserleuargglyglyleuglnservalser
202530
metlysalaproalaalavalalaserserthrtyrasptyrileile
354045
valglyglyglyileglyglycysvalleualaasnargleuthrglu
505560
serglyargphelysvalleuleuleuglualaglylysseralaglu
65707580
argasnprotyrvalasnileproalaglyvalvalargleuphelys
859095
seralaleuasptrpglnphegluseralaprogluarghisleuasp
100105110
glylysgluvaltyrleuvalargglylysalametglyglyserser
115120125
alavalasnvalmetleuvalhisargglyseralaserasptyrala
130135140
lystrpglualagluglyalaglnglytrpglyprogluglualaleu
145150155160
argtyrphelyslysmetgluaspasnleuvalglyglygluglyarg
165170175
trphisglyglnglyglymettyrprovalaspaspvallystyrgln
180185190
asnproleuserlysargpheleuglnalacysgluglutyrglytrp
195200205
argalaasnproasppheasnasptrpserhisproglnaspglytyr
210215220
glyserphelysvalalaglnlyshisglylysargvalthralaala
225230235240
serglytyrleuasnlysalavalargargargproasnleuaspile
245250255
leuserglualaleuvalthrargvalleuleugluglygluglyasp
260265270
vallysalavalglyvalgluphethrglylysaspglylysthrhis
275280285
glnvalargthrthrglylysalaglygluvalleuleualaglygly
290295300
alavalasnserproglnleuleumetleuserglyileglyproglu
305310315320
alaaspleuglnalavalglyilealathrlysvalasnargprogly
325330335
valglygluasnleuglnasphisproalavalthrilealahisasn
340345350
ilethrargproileserleucysaspaspleupheleuphehisthr
355360365
provalprolysprohisglnvalleuargtrpthrleuthrglyser
370375380
glyproleuthrthrproglycysasphisglyalapheleulysthr
385390395400
arggluaspleuglngluproasnvalglnpheargpheilealagly
405410415
argglyseraspproaspglyvalargsertyrilemetglyglyser
420425430
alaargproleuserglyleuthrleuglnvalvalasnileargpro
435440445
lysserlysglylysleuthrleualaserlysaspproleulyslys
450455460
proargilegluvalargtyrleuseralaalagluaspleuglnala
465470475480
leuargthrglymetargileglyargaspleuilelysglnargala
485490495
phealaaspileleuaspglugluvalpheproglyproalaalagln
500505510
thraspglugluleuaspalatyrileargaspserleuhisthrala
515520525
asnalaleuvalglythrcyslysmetglyservalgluaspargasn
530535540
alavalvalaspproglucysargvalileglyvalglyglyleuarg
545550555560
valvalaspalaservalmetprovalileproglyglyglnthrgly
565570575
serglythrthrmetleualaglulysalaalaaspleuvalargala
580585590
hisalaglyaspleuvalglumetglyvalglnaspglugluarglys
595600605
glyglytrppheasnglyleuleuglyarglysglnlysvalalathr
610615620
glulysgluargglygluargglylyssergluargphevalserglu
625630635640
valilearghismetglyargvalphevalglnvalserargalaarg
645650655
argalaglnthrcysmetargvalglylysglyleuasparggluarg
660665670
glnleuglucysalametarglysgluleuthrilealaleuphetyr
675680685
alametleuphethrmetarghisserglypheleuserthrthrgly
690695700
argalasertyrlysaspleuglytyrleuthrglysercysargala
705710715720
hisprocysthrserproserserleucysleupheproglulyspro
725730735
phemetlysleuserproalaleualavalvalglyphecyspheasn
740745750
serileasnvalglnglypheleuleuserasnleualaglyargser
755760765
leulyshisprovalproglnlysglyleutyrserargileglutyr
770775780
aspalaarggluproargleuaspglupheglyleuproleuasppro
785790795800
alaaspleumetglulysproargvalproleulysaspargvaltyr
805810815
hisileileaspmetthrasnasptrpvalaspalavalserarggly
820825830
argarggluglugluthrargargileileglnargargargalaala
835840845
alalysalametalailelysasplysvalleuileserleuasptyr
850855860
valphehisprovallysalatrpargthrphevalalaaspproleu
865870875880
glualaarghisglnargglnleuargglnglnalaglulysargala
885890895
argleugluargtyrleuglnargtyrasnthrvallysasnargphe
900905910
hisaspthrleuaspleuleugluserthrthrargthrservallys
915920925
valalalysservalserseralavalvalglyalaproglythrval
930935940
thrargthrvallysgluvallysserglnalaglnglythralaglu
945950955960
alavalalalysvalserserservalserservalvalserlysile
965970975
thrservalilearglysgluaspglyalaleualaglyalalysgly
980985990
lyslysaspproargsergluaspgluglylysalaaspprovallys
99510001005
valarggluiletrpgluthrlysgluglnthralaileargthr
101010151020
iletrpglualaaspgluleuvalthrprovalthrproproala
102510301035
thralametalaserthrvalservalsergluproglnaspglu
104010451050
asnglualaserileserglnglyalaalaproserproserthr
105510601065
serserproserserprogluprovalthrargleuserphearg
107010751080
alaargvalglualaaspglulysgluargpheglyserargarg
108510901095
leulysileserglyasnvalproprothralaserprothrarg
110011051110
glyalaserserleuproleuaspthrleuserserseralathr
111511201125
glnthrphegluargserlysvalglyproproileargthrser
113011351140
lysalaargcysileglylyscysvalhisasnglytrplysgly
114511501155
ilecysgluglutrpphevalhisileserpheprothrtyrala
116011651170
valserilevalargpropromethisvalhisasnphelysval
117511801185
ilecyscysvalleualavalarghisalaargarglyslysglu
119011951200
metserthralaleuserthrhisleuiletyrleuleuleulys
120512101215
thrvallysmetleuglnaspleuproglnleuargarglysgly
122012251230
lysthrasn
1235
<210>15
<211>1785
<212>dna
<213>变异小球藻
<400>15
cgcgcctctgcggtggaggacatccgcaaggtgctgtctgacagcagcagcccggtggcg60
gggcagaagtacgactacattctggtcggcggcggcaccgcggcctgcgtgctggccaat120
cgcctgtccgccgacggctccaagcgtgtgctggtgctggaagccggccccgacaacacc180
tcgcgcgacgtcaagatccccgccgccatcacccgcctcttccgcagccccctcgactgg240
aacctcttctccgagctgcaggagcagctggcggagcgccagatctacatggcgcgcggc300
cggctgctgggcggcagcagcgccaccaatgccacgctgtaccaccgcggcgcggcgggc360
gactatgatgcctggggcgtggagggctggagctcggaggacgtgctcagctggtttgtg420
caggcggagaccaacgccgactttggccctggcgcgtaccacggcagcggcggccctatg480
cgtgtcgagaacccacgctacaccaacaagcagctgcacaccgccttcttcaaggccgca540
gaggaggtggggcttaccccaaacagcgatttcaacgactggagccacgatcatgctggg600
tacggtaccttccaggtcatgcaggacaagggcacacgtgccgacatgtaccggcagtac660
ctcaagcccgtgctgggccgcagaaacctgcaggtgctgaccggcgcggccgtgaccaag720
gtcaacattgaccaggctgcaggcaaggcgcaggctctgggtgtggagttctcaaccgac780
gggcccacaggtgagcggctgagcgctgagctggcgccaggcggcgaggtgatcatgtgt840
gccggcgccgtgcacacgcccttcctgctcaagcacagcggcgtggggccctcggcggag900
ctcaaggagtttggcatcccggtggtttccaacctggcaggtgtgggtcagaacctgcag960
gaccagcccgcctgcctgaccgcggcgccggtcaaggaaaagtatgatggcatcgccatc1020
agcgaccacatctacaacgagaagggccagatccgcaagcgcgccattgccagctacctg1080
ctgggcggccgcggcggcctcacatccaccggatgcgaccgcggcgcctttgtgcgcacg1140
gcaggccaggcgctgccagatctgcaggtgcgctttgtgccgggcatggcgctggacccg1200
gatggcgtgtccacgtatgtgcgctttgccaagttccagtcccaggggctcaagtggccc1260
agcggcatcaccatgcagctgatcgcctgccgcccgcaaagcacgggcagcgtgggcctc1320
aagagtgccgacccctttgcgccgcccaagctgagccccggctatctgacggacaaggac1380
ggcgctgacctggccacgctccgcaagggcatccactgggcgcgcgacgtggcgcgcagc1440
agcgcgctcagcgagtacctggatggcgagctgttccccggcagcggagtggtgagcgat1500
gatcagattgatgagtacatccgccgcagcatccatagctccaacgccatcacaggcaca1560
tgcaagatgggcaatgccggcgacagcagcagcgtggtggacaaccagctgcgggtgcac1620
ggtgtggagggcctgcgggtggtggatgcctcggtggtgcccaagatcccaggtggccag1680
acgggcgcgccagtggtgatgattgcggagcgcgccgccgcgctgttgactggcaaggcg1740
acgatcggggcgagcgctgcggccccggcgacagtggcagcatga1785
<210>16
<211>1743
<212>dna
<213>莱茵衣藻
<400>16
ctgtcggtgcgtgctgctgcgggcccagctggctcggagaagttcgactacgtgcttgtc60
gggggtggcactgcctcgtgcgtgcttgctaacaagctgtcggctgatggaaacaagaag120
gttctggttctggaggctggccctaccggtgacgccatggaggtggctgtgcccgccggc180
atcacccgcttgttcgcgcacccagtgatggactggggcatgtcgtcgctgacacagaag240
cagctggtggcccgcgagatctacctggcccgcggccgcatgctgggcggctcctccggc300
tctaacgccacgctgtaccaccgcggcagcgccgccgactacgacgcttggggcctggag360
ggctggagcagtaaggacgtgctggactggttcgtcaaggcggagtgctacgccgacggc420
cccaagccctaccacggcactgggggcagcatgaacacggagcagccgcgctacgagaac480
gtgctgcacgatgagttcttcaaggccgccgccgccaccggcctgcccgccaaccccgat540
ttcaacgactggagccacccgcaggacggcttcggtgagttccaggtgtctcagaagaag600
ggccagcgcgccgacacctaccgcacctacctcaagcccgccatggcgcgcggcaacctc660
aaggtggtgattggtgcccgggccaccaaggtcaacattgagaagggctcgtcgggcgca720
cgcacgaccggcgtggagtacgccatgcagcagttcggcgaccgcttcaccgccgagctg780
gcccctggcggcgaggtgctgatgtgctcgggcgcggtgcacacgccgcacctgctcatg840
ctgtcgggcgtgggccccgccgccacgctgaaggagcacggcatcgatgtggtgtcggac900
ctgtcgggtgtggggcagaacctgcaggaccaccccgcggcggtgctggcggcgcgcgcc960
aagccagagtttgagaagctgtcggtgaccagcgaggtgtatgacgacaagtgcaacatc1020
aagctgggcgccgtggcgcagtacctgttccagcgccgcggcccgctggccaccaccggc1080
tgcgaccacggagccttcgtgcgcacctcctcctcgctctcgcagcccgacctgcagatg1140
cgcttcgtgccgggctgcgccctggaccccgatggcgtcaagtcctacatcgtgtttggc1200
gagctcaagaagcagggccgcgcatggcccggcggcatcacgctgcagctgctggccatc1260
cgcgccaagagcaagggcagcatcggcctgaaggcggctgacccgttcatcaaccccgcc1320
atcaacatcaactacttctccgaccccgccgacctggccacgctggtcaacgccgtcaag1380
atggcgcgcaagatcgcggcgcaggagccgctcaagaagtacctgcaggaggagacgttc1440
cccggcgagcgcgcctccagcgacaaggacctggaggagtacatccgccgcaccgtgcac1500
tcgggcaacgcgttggtgggcaccgcggccatgggcgcctcgcccgcggccggcgccgtg1560
gtgtcctccgccgacctcaaggtcttcggcgtggagggcctgcgtgtggtggacgcctcg1620
gtgctgccgcgcatccccggcggccagaccggcgccgccactgtcatggtggcggagcgc1680
gcggcggcgctgctgcgcggccaggccaccatcgcgcccagccgccagcccgtggccgtg1740
taa1743
<210>17
<211>1707
<212>dna
<213>三角褐指藻
<400>17
tacgactacattatctgcgggggagggttggctgggtgcgttttggctgaacgcctttcg60
caggacgagtccaagcgtgtgctcgtactagaggctggcggctcggactacaaaagtctt120
ttcatcaggattccggctggagtcttacggctctttcgcagtaaatacgattggcaacat180
gaaacgggcggtgaaaagggttgcaacggacggaatgtgtttttgcaacgcggcaagatt240
ctcggcggatcttcgtgcaccaatgtatgtctgcatcaccgtgggtcagcggaagattac300
aacagctggaacattcccgggtggacggctactgatgtgcttcctttcttcaagcaatcg360
caaaaggatgagacgggccgggatgccacgttccacggtgcggatggggagtgggtcatg420
gatgaggtgcgataccaaaatccattgagcaagttattcctggaggtaggggaggcagct480
ggcctcggaaccaatgatgatttcaacaactggtcgcatccgcaggatggcgttgggcgc540
ttccaggtttcagaagtaaacggagaacgatgttcgggagccacagcatttctaagtaaa600
gcggccaagcgctcaaatgtcatagttcgaactggcactatggtgagaaggatcgacttt660
gatgagacaaaaacagcgaaagggattacctacgatttgatgggcgatgacacatgtacg720
acatttcaagcttgtttgaaagagggtggcgaagtgctggtgacaggtggagcaattgca780
tcccctcagcttctcatgtgctccggaattggacctggcaagcacttacggtcgctgggc840
attccagtagtccacgacaactcggcagttggtgagaacctgcaggatcacccagctgct900
gtcgtttcctttaaaacccctcaaaagggagtttctgttacttctaaattgcggctcttc960
ggtaagacaaaccccattccagtcttccagtggctattttttaaaagtggtcttttgacc1020
tccactggatgcgatcatggcgctttcgttcggacatcggattcactcgaacaaccggac1080
ttgcagattagattcttggcagctcgcgctcttggacccgacggaatgaccacgtatacc1140
aaattccgtacgatgaaaacagttgaagatggttattcgttccagagtgttgcatgtcga1200
gcaaaaagcaagggaaggattcggttatcttcgtccaactcgcacgtaaagcctatgatt1260
gatggcggatacctctctaaccaggatgatcttgccacactacgtgctggtatcaagctt1320
ggacgtatgctcggtaaccgaccggaatggggcgagtaccttggtcaagaggtgtatcca1380
gggccagatgtgcaaacggatgaagagattgacgaatacattcgtaactcgctccatacc1440
gccaatgctttaacggggacatgcaaaatgggaacgggtcgtggggctgttgttgggccc1500
gatttacgtgtaattggggtcaatggtgtacgtgtgggggattcatctgttttcccctgc1560
attccagggggacaaacggcaacaccgaccgtcatgataggggatcgggccgctgttttt1620
gttcggcaacccgtatcccagctgaacattgaaattttcagagaaaaagggggcacacat1680
cctggggcaacgaccgcctcagcctag1707
<210>18
<211>1758
<212>dna
<213>胶球藻
<400>18
gcactgcgggtcagggcgataatcaaaagcgacaatccagctgcagataagtatgacttc60
atcctggtcggcggcggcacagcaggctgcgtgctggccaacaggctgacagcagatgga120
tccaagaaggtcctgctgctcgaggcgggaggcgccaacaaggcgcgggaggtacgcaca180
ccggccgggctgccgcgcctgttcaagagcgccctcgactggaacctctactcctccctg240
cagcaggcagccagcgacagatcgatctacctggcacggggaaagcttctgggtggcagc300
tccgcaactaatgccacgctgtaccaccgggggactgcggcagactacgacgcgtggggt360
gtgccggggtggacctcccaggacgccctgcgctggtttattcaggcagagaacaactgc420
agagggattgaggacggggtgcatgggaccgggggattgatgcgcgtggagaaccctcgc480
tacaacaaccccctgcacgaggtctttttccaggcggccaagcaggccggcctcccagag540
aacgacaactttaacaactggggcagatcacaggcggggtacggggagttccaggtgact600
cacagcaagggtgagcgcgccgactgcttccggatgtacctggagccagtcatggggcgt660
tccaacctcacagtgctcaccggagccaaaacgctcaaaatcgaaactgaaaagtccggc720
ggcgccacggtctcgcgcggcgtcaccttccaggtcaacggccaggacggcagcaagcac780
tcagctgagctggcggctggcggcgaggtggtgctgtgcgcggggtcaatccactcgccc840
cagatcctgcagctctccggcatcggcccccaggcggagctgcgctccaaggacatccct900
gtcgtcgcagacctgcccggcgtcggccagaatatgcaggaccacccggcgtgcctgtca960
gccttctacctcaaagagagcgcgggtcccatcagtgtgactgatgagttgctgcacacc1020
aatggccgaatccgcgcgcgcgcaatcctaaagtacctgctcttcaaaaaggggcccctc1080
gccaccacaggatgcgaccacggcgcctttgtgaagactgcaggtcagagtgagccggac1140
ctgcagatccgctttgtgccgggtctggcgcttgaccctgacggcatcggatcctacacg1200
gcctttggcaagatgaaggaccagaagtggccctccggaatcaccttccagctgctcgga1260
gtgcggcccaagagccgcgggtcggtgggtcttcgcagcgacgacccctgggacgcgcca1320
aaactggacatcggcttcctgactgacaaggagggcgctgacctggccacgctgaggagc1380
ggcatcaagctctcgcgcgagatcgcagcggagccggcattcggagcatatgtaggcaac1440
gaacttcacccgggcgcagccgcctcgtccgattctgcgatcgactcattcatccgtgac1500
acggtgcactcgggcaatgccaacgtgggcacatgctcgatgggggtgaatgggaacgcc1560
gtcgtggacccgtcactcagagtgttcggcatccgcggcctgcgagtggccgacgcatca1620
gtcatccccgtcatcccaggaggccagaccggcgctgcaacggtgatggtggcagagagg1680
gccgcggagattctcctcggctcgaaccagaagcagccggccgccgccgtccccgccgcg1740
cagccggccctggcttga1758
<210>19
<211>1744
<212>dna
<213>强壮团藻
<400>19
tccagttgcagtcaaagctgcggcaagcgtggggtcggagaagttcgactacatccttgt60
gggaggagggacggcaggttgcgttctggcaaacaaattgtccgctaatggcagcaagaa120
ggttcttgttcttgaggctggtccaaccggcgatgctatggaagtggccgtaccggccgg180
tattgcccgtctctttgcacatccagtttttgactggggtatgtccagtttgacgcagca240
gcagctggttgcacgcgagatctaccttgctcgcggtcgccttctgggcggttcctctgg300
gaccaacgcaacgctgtatcatcgcggcacccccgctgactacgacagttggggcttgga360
gggctggacgagcaaggatctacttgattggttcgttaaggccgaatgctatggagacgg420
tccccgggccttccatggccagtccggttctatgaatgtggagcaaccgcgttaccagaa480
cgtcctgcacgacgagtttttcagggctgctgcggcagctggattacctgccaacgagga540
tttcaatgactggagccggccgcaggagggctatggggagttccaggtggcccagaagaa600
cggtgagcgcgcggacacgtatcggacctacctcaagccggctatgggccgcgacaacct660
gaaggtgatgaccggcgcgcgtacgaccaaagtgcatatcgagaagagctcgacgggtcc720
acgtgcccgcggtgtcgagtatgctacccagcaatttggggagcgctacaccgccgagct780
gactcctggtggcgaggttctcatgtgcaccggtgccgtgcacacaccgcacctactgat840
gctttcgggcattgggccggcgccgaccctccttgagcacggactcgacgtgatatcaag900
cctgcctggcgtgggtgccaacctgcaggatcacccagccgcggtgcttgctgtccgggc960
taagcccgagttcgagggactgtcggtcacttcggaaatctatgacagcaagtgcaacat1020
ccggttgggcgccgttatgaagtacctgttcggacgccgcgggccacttgcaaccacagg1080
ctgtgatcatggcgcttttgtgcgcacttcggcctcgcattcgcagccggacctgcagat1140
gcgattcgtgcccggttgcgcgctggaccccgacggcgtgaagtcgtacattgtgttcgg1200
cgagctgaagaagcagggccgtgcttggcccggcggtatcacgttgcagctgctgggcat1260
ccgcgccaagagcaggggcagcatcggtctcaaggccgcggatcccttcatcaaccccgc1320
cattaacataaactatttctccgatcctgaggaccttgccacgcttaagaacggtgtgcg1380
cattgcgcgcgaaatcgttgcccaggagccattgcgcaaataccttctggaggagacctt1440
ccccggggagcgggcgaacactgacaaggacattgaggagtatgtccggcgcacggtcca1500
cagcggcaacgcgcttgtgggcacctgcgcgatgggtaccacgccagccagtggcgccgt1560
ggtgtcttctgctgacctcaaggtgttcggtgttgacgggctgcgtgtcgtggatgcgtc1620
tgtgctgccacgcattccaggcgggcaaacaggagcggccactgtcatggttgcagagcg1680
ggcggcggccatgctgcttggacaggcgacgattacttctcgtcgcgagcctgcggctgt1740
ttag1744
<210>20
<211>1674
<212>dna
<213>长囊水云
<400>20
gtgcctgcagcacgttatgcgacgtcttcggtgagcatgtcggtggcggaagaaggccac60
aagttcatcatcatcggcgggggtacggctgggtgtgttctggcgaaccgcctctctgcc120
gacaaggacaattcagtgctggtgttggaggccggctcggaaaaattcaatgacaggaat180
atcaagatgcccatcgcaatcctaaggttgttcaagtctgtcttcgactggggcttccag240
tcggaaaacgagaagtttgccaccggcgacggcatctacctctgccgtggcaaggtcctc300
ggcgggtctagctgcaccaacgtcatgctttaccatcgcggagaggaggctgactatgac360
gcgtggggggtggatggatggaaaggcaaggacgtgctcccctacttcaagaaggcggag420
aacaaccgatccaagaaaaagggggagttccacggcaagggaggcctgatgcaggtggaa480
aacgcgcgttacatgaaccctttgacgaagttgttcttcaaggcgtgcgagcaggcaggc540
ctgtcggagaacgaggacttcaacgattggtcccactcgcaggagggcttcggtcgcttc600
caggttgcacagaagagaggcaagcgctgttcggcagcctcctcctacctcaaggaagcc660
atggggcgcaaaaacctggacgtgcagaccagcgcccagatcaccaaggtcttgatcgag720
aacggcggtgccataggcgttgagtacgtgcgggatggcgaaaagaagatcgccaagctt780
gccgtgggcggagagatcctgctagcaggaggtgccatcagctcaccgcaggtgctgatg840
ctcagtggggtgggtccggccgagcacttgaggtccaagggcatcgaggtgaagagcaat900
gtgcctggggtcggaaagaacctccgcgaccatcccgctgtcaccgtgatggccgacatc960
aacaagcctatctccatcaccgacaaggttctcaaggagggctctggggacgtgaacaaa1020
attacggcccttcagtggctgttgacgggaactgggccgctgacctcgccagggtgcgag1080
aacggcgcgttcttcaagaccacgcctgacaaagcggcggcagatctgcagttgcgattt1140
gtccctggaagatccactaccccagacggggtgaaggcgtacaacactattggcaccaag1200
gggaggcctccttcgggtgtcaccgtgcaggtggtggggatccggccgcagagtgaaggc1260
cacgtcgagctgcgttcttcggacccgttcgacaagccacacatcgtcactaactacctt1320
gagagtggggaggacatggcgtctttgacgaacggaatcgaaatggcacgaaagctgttc1380
gaccaggaggccttcggagaaatggtggacaaagaagtcttccccggcagagacaacaag1440
gagatctcggagtacatcaagtctaccgtgcactccgccaacgccttggttggcacttgc1500
aagatgggcgaagaatcagacaacatgtctgttgtgaactcggcactcaaggtaaagggt1560
gttgccgggctgagggtgatcgattcttcggtgatgccttcaatccccggcggtcaaaca1620
gcggcaccgacgatcatgatcgcagagaaagcggcggacatgctcatggcttag1674
<210>21
<211>1808
<212>dna
<213>赫氏圆石藻
<400>21
gccttcgcggcggcagcggcgtcactggcggcagcctcggccgcggcggcggctcgccgg60
cgatcgacggcgagtttgactacatcatcgttggaggcggcgccgcgggctgcgtgctcg120
ccaatcggctgagcgccgacccggcgcaccgggtgctgctgatcgaggccggaggcgacg180
cgtcgcgcgacaagcgtgcgcaggtgccgtgggccttcaccaagctgctgcgttccgagt240
acgactgggactttcacgtggaggcggaggcggcggtgaaccagcaggaggtgtacctct300
gccgcggcaaggcgctgggcggctcttcggtcacaaacgtgatgctctaccaccggggca360
gccccgcggactacgacgcttgggaggaggctggcgcgcgagggtggggcgcgaaggacg420
tgctcccgtattacctccgcgtcgaggactacggcgacggcgcctcgcagtaccacgccg480
tcggcgggcacgtctcggtgcaggaggtgccgtaccagaaccaactctcggcaaccttcc540
tgcgggcgatggggcagctgggcttccggccgaacggagacttcaacgactggtccagcc600
cgcaggaggggtacggccgctacaaggtgacccagcgggccggccgccgctgcaccgctg660
ccgacggctacctggcggcggcgagagagcgcgcgaacctggtggtggtgaccggcgcgc720
aggcgacccgcctcgccctcgacagtgcgtacgacggcgcgggccggctgcaggtctccg780
gcgtcgagtttgcacgcggcgacgagcgcgagccgtgctccgtccggctcgcgcgcggcg840
gagaggccgtcctctgcgccggcgcggtccagacgccgcacttgctgctcctctccggga900
tcggcccggcggagcacctgcgcgaggttggcgtgccggtgcgggcggacctgcctgggg960
tggggtccggcctgcaggaccaccccgcggtggtcgtctcgtacgagagcaagaaagcgg1020
tcgccgcgacggacgacgcgctcctcaagggctacgcgtcgctggtcaacccgctcgcga1080
tgctgcgctggctcctgtttgggcgcgggccgctggcgtgcgccgcgtgcgaccacggcg1140
gcttcgtccgctcctcgcccgacctcgaccagcccgacgtccagatccgcttcgtgcctg1200
cgcgcgcctcgtccgcctccgggatgaacacgctgatcgagctgggcaggagagcgcgct1260
tcctgccgggcttctcgacgcaggtcgtcgcttgccgcccgcggagcgaggggcgcgtgc1320
ggctgcggtcggccgacccgttcgccaagccgatcatcgagggcatccacttgggcgcag1380
cggaggacgtcgcgtcgctgcgccacggcatccggctcggccggcaggtttgcgccgcgg1440
cagccttcgacgagtaccgcggagaggaggtcttccccggcgcggcggtgcagtccgacg1500
agcagatcgacgagtacatccgctcgtcggtgcactctgcgaacgcgctcacctcgtcct1560
gccgcatgggcgacccgtccgacccggccgcagtcctcgactcccacttgcgcgtgcgcg1620
gcgtcggcggcttgcgcgtggccgacgcctccgccatgcctcgcatcatcggagggcaga1680
cgcaggcgcccacgtacatgctcgccgaacgtgccgccgacatcctgctgcacgcgcgcc1740
tgcaggcgcacgagccagccaccgagagcgtctcgcagcggctcgaggttgcggcggccg1800
cactctag1808
<210>22
<211>1605
<212>dna
<213>抑食金球藻
<400>22
cgcctcagcgaggacccgagcaagaaggtgctcgtgctcgaggccggcgaccggggcccg60
aactcgccgctcgtgaagatccccgtggccatcctcaagctcttcaagtccgcgtacgat120
tggaacttcgcgacgaggccgtccgaggccgtcgcggaccggagcctctacgtctgccgg180
ggcaagggcctgggcggcagctccctgacgaacgtcatgctctacaaccggggctcggcc240
aacgactacgacgcgtgggcggccgcgtgcggcgacgactcgtggggggcggaagagatg300
ctcggctacttcaagaaggccgaggactgcctcgtgccggcgcaccgagcgaaccactac360
cacggcgtcggcgggccctacgcgtcgagccacgtgccctacacgaacgagatgtcgacg420
gcgttcgtcgaggccgccgtcgaggacggcggcgtgcgcaacggcgacttcaacgactgg480
tccacgtcccaggtcggcttcggccgcttcgccgtgagccagcgcaagggggcaagggtg540
gacgcggcgacggcctacctcccgcggaaggtccggcggcgcgcgaacttggacgtggtc600
cgcggcgcggcgctctccggcgtcacgtggaacgcgaacaaggccacgggcgtcgagttc660
gccttcggcggcgtctcgggcatcgcctgcggcggcgaggtgatcctctccggcggcgcg720
gtccactcgccgcagatgctcatgctctccggcgtcggcgcgaaagcgcagctcgaggag780
ttcggcatccccgtcgtcgcggaccggccgggcgtgggcaagaatttgcaggaccacccg840
gcctgcctcgtgtcctggcgcgggtcggccaaggcccagggcaagagccactcgacgcag900
ctccggatccccgggacgacgaagacctcgcccaaggcgctcctccagtggctcttcctc960
ggccggggccccttggcctcgccgggctgcgaccacggcggcttcgccaaggtcggcgcc1020
ggcgacggcgactgcgacgtccagttccggttcctcgcgacgaagtcgatcacgcccgac1080
ggcatgtctaccatctccgactcctacgaggccgccgtggaccaccccgacggcttgacg1140
atccagaccatcgtcgcccggcccaagagccgcgcgggcgaggtgaaactcgcgtcccgg1200
gacccggcggcgaagcccgtcatcgagaacgcgtacctctccgacgaggccgacgtcatg1260
accatggtcaaggcgctccagaaggcgcggtccatcgcgagccgggcgccgttgagcgcc1320
tacgcgggacacgaggagttcccgggggaggacgtcgcggacgagcgccaactggcggcc1380
tacgtgcggaacaccgctcataccgcgaacgcggtcgtcgggacctgcaagatgggcgag1440
tccagcgacgccctcgccgtcgtcgacaaccacctcaaggtcatcggcgtctcgaacctg1500
cgggtggtggacgcgtcggtgatgccgacgctgccgggcggccagacggccgcgtcgacc1560
gtcgcgctcgcggagaaggccgcggacctcatcaaggggggctga1605
<210>23
<211>3711
<212>dna
<213>海洋富油微拟球藻
<400>23
ctctccaagaccaccatcggcctccaatccttcgtgacagcaaattatggagtgaggaga60
gcaatctctctccgaggggggctacaatctgtttcgatgaaggcaccggctgccgttgct120
tcgtccacctacgactacatcatagtcggtggcggcatcggcggttgtgttctcgccaac180
cgactgacagaaagcgggcgtttcaaggtccttctcctggaggccggaaaatcagcagag240
cgaaacccgtacgtgaacatccccgcgggcgtggtccgcctctttaaatcggccttggat300
tggcagtttgagagcgcccccgaaagacacctggacgggaaggaggtgtacttggtcagg360
gggaaggccatgggaggctccagtgcggtgaatgtgatgctcgtacaccgcggctcggcc420
tccgactacgcgaagtgggaggcggagggagctcagggatggggcccggaggaagcctta480
cgctacttcaagaaaatggaagacaacttggtgggcggggaagggcggtggcacggccag540
gggggcatgtacccggtggacgacgtcaagtaccaaaaccctttgtccaagcgcttttta600
caggcgtgcgaggagtacgggtggcgggcgaacccggattttaacgactggtcccatccc660
caggacggatacgggagcttcaaagtggcgcagaagcacggcaaacgggtgacggccgcc720
tccgggtacttgaacaaggccgttcgacgtcgaccaaacctagacattctgtcagaggcc780
cttgtgacccgggttttgcttgagggagagggggatgtgaaggcggtcggggtggagttc840
acgggcaaagatgggaagacgcaccaggttcggaccacggggaaggcgggagaagttctc900
ttggcgggcggcgcggtcaacagcccccaactcctgatgctgagtgggatcggacccgag960
gcagacctccaggcagtaggcatcgcgaccaaagtaaaccgtccgggcgtgggggagaac1020
ttgcaagaccatcccgccgtcactattgcgcacaacatcacgcgccctatctcgctctgc1080
gacgacctcttccttttccacacacctgtccctaagccccaccaggtgctccggtggacc1140
ttgaccggatcgggcccgctgaccaccccaggctgcgaccacggcgcgttcctgaagacc1200
cgagaagacctccaggaacccaacgtacagtttcgtttcatagccgggcggggttcggac1260
cctgacggcgtgcgctcctacatcatgggaggctcagcacgccccctgtcgggattgacg1320
cttcaagtcgtcaacatccgccccaaaagcaaagggaagctgaccttggcgagcaaggac1380
ccgttgaagaagccccggatcgaagtccggtacttgtccgcggccgaagacttgcaggcc1440
cttcgcacgggcatgaggattggccgggatctgatcaagcaacgggcttttgcggatatc1500
ttggacgaggaagtgttcccggggcccgccgctcagacggacgaggaactggacgcctac1560
atccgggatagcttgcacacggccaacgccctggtaggcacctgcaagatggggagtgtg1620
gaggatcggaacgcagtggtggacccggagtgccgggtcatcggcgtgggaggcctgcgg1680
gtggtggatgcaagcgtgatgcccgttatccccggagggcaaacgggaagtggcacgacc1740
atgctggccgaaaaagcggccgacctggtcagggcccacgctggggatttggtggagatg1800
ggagtgcaagatgaggagaggaagggaggatggtttaacggcctgttagggcgcaaacag1860
aaggtggcgacagaaaaggagcgaggcgaacgtggtaaaagcgaacgatttgtttctgag1920
gtgattaggcatatgggtcgggttttcgtacaagtttcgagggcaaggcgtgcgcaaacg1980
tgcatgagggtagggaagggtctcgatagggaacggcagttggagtgtgccatgcgcaag2040
gaattgacgattgcgcttttttacgcaatgctttttacaatgagacatagtggatttctt2100
tcgacgactggtcgcgcctcatataaggaccttggatacttgacagggtcctgtcgcgcc2160
cacccttgtacatctccatcctccttgtgcctgtttccagaaaaacctttcatgaagcta2220
agccctgctttggcggtcgttggcttttgctttaattctatcaatgtccaaggtttccta2280
ctcagcaaccttgccggtcggagcctgaagcatccggtcccacaaaagggcttgtatagt2340
cgcattgagtacgatgcacgggagccacgtctcgatgaattcggcctccctcttgatccg2400
gcggatttgatggaaaaaccccgagtgcccctgaaggaccgagtttatcacatcattgac2460
atgacaaacgactgggtcgatgctgtgagccgcggacggcgagaggaggagacgcgtcgc2520
atcatccaacgacgtcgtgccgctgccaaggccatggcaatcaaggacaaggtcttgatt2580
tctcttgactacgtcttccatccagtcaaggcctggaggaccttcgtggcggaccccttg2640
gaagcacggcatcagcgccagctgcgtcagcaggcagaaaagcgagcgagactagaacgg2700
tatctgcaacgctataacacggtcaagaatcgcttccatgatactctggatcttttggag2760
tcgacgacccggacatcggtgaaagtggccaagtccgtgagttccgcagtggtgggggcg2820
cctgggacggtgacgcggaccgtgaaggaggtgaaaagccaggcacagggcacagcggag2880
gcagtggcgaaggtgtcttcctccgtctcctcggtggtgtccaagatcacgtcggtgatc2940
aggaaggaagacggggctctggcgggggccaaggggaaaaaggacccgagatcagaagac3000
gaaggaaaagccgatccagtcaaggtccgggagatctgggaaacgaaagagcagaccgcg3060
attcgcaccatctgggaagcggatgagcttgtcacgccggtaacaccgccagccacggct3120
atggcgtcgacggtttcggtgtcagagcctcaggacgagaacgaagcttccatctctcag3180
ggagcggcgccttcccccagcacctcatcaccatcctcccccgagcccgtcacacgcctc3240
tctttccgggcacgtgtggaggcggacgagaaggaacgctttgggtcccgaagattgaag3300
atatcggggaatgtcccgccgaccgcttctcccactcggggggcttcttctctccctttg3360
gacacgctttcctcctccgccacccaaacctttgagcgttccaaagtggggccgcccata3420
cgaacaagtaaggcgagatgcattggaaaatgtgtacataacgggtggaaagggatatgt3480
gaggaatggtttgtgcacattagttttccaacgtatgcggtctctatcgtccgtccaccc3540
atgcacgtgcataattttaaagtgatttgttgtgttctagcggtccgacatgcaagaagg3600
aagaaggagatgagcacggctttatctacacatttaatttatttactgctgaaaacggta3660
aaaatgttgcaagatcttccccagctacggagaaagggcaaaacaaactag3711
<210>24
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>24
atggcgtcaattacatcgcg20
<210>25
<211>18
<212>dna
<213>人工序列
<220>
<223>引物
<400>25
tcatgctgccactgtcgc18
<210>26
<211>15
<212>prt
<213>人工序列
<220>
<223>共有序列
<220>
<221>misc_feature
<222>(2)..(2)
<223>x是任意氨基酸
<220>
<221>misc_feature
<222>(4)..(7)
<223>x是任意氨基酸
<220>
<221>misc_feature
<222>(9)..(9)
<223>x是d或e
<220>
<221>misc_feature
<222>(10)..(10)
<223>x是任意氨基酸
<220>
<221>misc_feature
<222>(12)..(12)
<223>x是a或g
<220>
<221>misc_feature
<222>(14)..(14)
<223>x是任意氨基酸
<220>
<221>misc_feature
<222>(15)..(15)
<223>x是k,r,s或e
<400>26
glyxaaleuxaaxaaxaaxaacysxaaxaaglyxaaphexaaxaa
151015
<210>27
<211>37
<212>dna
<213>人工序列
<220>
<223>引物
<400>27
ctgtacttccaatcagccagcgcagttgaagatattc37
<210>28
<211>41
<212>dna
<213>人工序列
<220>
<223>引物
<400>28
tatccacctttactgttatcatgctgcaacggttgccggtg41
<210>29
<211>33
<212>dna
<213>人工序列
<220>
<223>引物
<400>29
tacttccaatcaatgatgctgggtccgaaaacc33
<210>30
<211>41
<212>dna
<213>人工序列
<220>
<223>引物
<400>30
tatccacctttactgttctactaaactgcaaccggctgacg41
<210>31
<211>33
<212>dna
<213>人工序列
<220>
<223>引物
<400>31
tacttccaatcaatgaaaaaaagcctgcgtagc33
<210>32
<211>36
<212>dna
<213>人工序列
<220>
<223>引物
<400>32
tatccacctttactgttctactatgcgcttgcggtg36
- 还没有人留言评论。精彩留言会获得点赞!