用于个体细胞的配对mRNA捕获和测序蝶形条码的高通量油-乳液合成的制作方法
本申请要求2016年8月19日提交的美国临时申请号62/377,123的优先权和权益,其通过引用纳入本文。
关于联邦资助研究的声明
本发明是在政府支持下由国家卫生研究院授予的基金号r21ca196460和r21ai12587资助完成。政府对本发明拥有一定的权利。
背景技术:
有许多生物学问题需要多基因序列的单细胞分析,包括淋巴细胞上克隆性分布的二聚免疫受体的分析和多克隆肿瘤中驱动/辅助突变的积累。大量细胞群的裂解导致基因序列的混合,使得知道哪对基因序列来源于任何特定细胞成为不可能,并且混淆了大细胞群中稀有序列的分析。尽管目前的单细胞分选技术可被用于处理其中的一些问题,此类方法昂贵、要求专门设备、以及缺乏进行大样本综合分析所必要的高通量能力。因此,在本领域中仍然需要用于个体细胞的遗传分析的改进方法。
技术实现要素:
在第一方面,本文提供一种将单链dna(ssdna)条码化的多核苷酸纳入核酸折纸纳米结构的方法。所述方法可基本上包括或由以下组成:(a)进行条码核酸的第一链合成以产生双链条码核酸,其中条码核酸包含至少一个与单链5’-5’蝶形接头核酸的一侧互补的引发位点,所述单链5’-5’蝶形接头核酸包含中心5’-5’磷酸二酯接头,该中心5’-5’磷酸二酯接头的任一侧与互补的条码核酸和与核酸折纸纳米结构互补的序列侧接,所述合成可以在随后的油-乳液扩增步骤之前或期间进行;(b)在包含用于扩增靶核酸的试剂的油-乳液液滴中合并双链条码核酸(或单链条码和互补的引物对)和5’-5’蝶形接头核酸;(c)使包含双链(或单链)条码核酸引物和5’-5’蝶形接头核酸的油-乳液液滴热循环,所述热循环足以导致每条链自双链条码核酸退火至5'-5'蝶形接头核酸上的互补序列,并延伸ssdna条码化的5'-5'蝶形多核苷酸,从而获得产物,该产物在5'-5'磷酸二酯键蝶形链的任一侧含有条码化的mrna捕获序列;(d)自经热循环的液滴中提取延伸产物;(e)自提取的延伸产物纯化ssdna条码化的5’-5’蝶形多核苷酸;以及(f)将纯化的ssdna条码化的5’-5’蝶形多核苷酸纳入核酸折纸纳米结构。纳入可包括将纯化的ssdna条码化的5’-5’蝶形多核苷酸退火至核酸折纸纳米结构。所述核酸折纸纳米结构可以是dna纳米结构。
在另一方面,本文提供一种在单细胞水平上检测靶核酸序列的方法。所述方法可基本上包括或由以下组成:(a)使根据权利要求1的方法获得的核酸折纸纳米结构与分离自单细胞的核酸接触,其中所述纳米结构包括ssdna条码化的多核苷酸,所述ssdna条码化的多核苷酸具有与靶核酸序列互补的条码化的序列;并且其中如果单细胞中存在靶核酸序列,则所述接触在适合条码化的序列与靶核酸序列结合的条件下发生;(b)回收与条码化的序列结合的靶核酸序列;以及(c)使用所述ssdna条码化的多核苷酸作为逆转录的基因特异性引物,对回收的靶核酸序列进行逆转录,由此如果单细胞中存在靶核酸,则靶核酸在没有单细胞分选步骤的情况下被检测。所述核酸折纸纳米结构可以是dna纳米结构。
在又一方面,本文提供一种在单细胞水平上检测b细胞受体的方法。所述方法可基本上包括或由以下组成:(a)转染至抗原特异性b细胞表达独特重链(igh)和轻链(igl)bcrmrna;(b)与包含多条码化的mrna捕获序列的dna折纸纳米结构接触,以在被转染的抗原特异性b细胞中捕获和保护免疫球蛋白重链(igh)和轻链(igl)bcrmrna;(c)分离经接触的dna折纸纳米结构以回收与条码化的mrna捕获序列结合的igh和iglmrna;以及(d)使用所述条码化的mrna捕获探针作为逆转录的基因特异性引物,对回收的igh和iglmrna进行逆转录,由此靶bcr序列在没有单细胞分选步骤的情况下被检测。所述dna折纸纳米结构可包括完整的生物素标签,且其中分离经接触的纳米结构包括亲和素柱纯化。
通过以下描述可以清楚地了解本发明的上述内容和其他优势。在以下说明书中,参照构成说明书的一部分的附图,其中以说明性方式显示了本发明的优选实施方式。这类实施方式不必然代表本发明的全部范围,而是因此对权利要求进行说明并在本文中解释本发明的范围。
附图说明
当考虑以下的详细描述时,本发明将被更好地理解,并且除了上述内容之外的特征,方面和优点将变得显而易见。这样的详细描述参考了以下附图,其中:
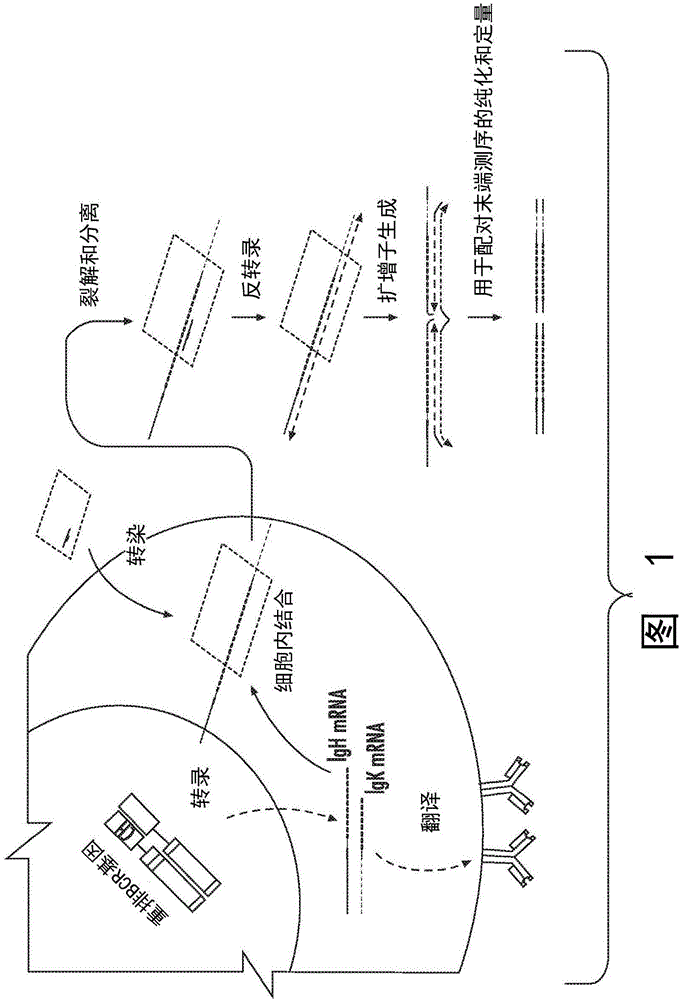
图1是无需单细胞分选、使用dna折纸纳米结构从单细胞的bcr/抗体mrna获得链接序列信息的示例性方法的示意图。抗原反应性b细胞获自使用荧光标记的抗原的流式细胞术细胞分选,并用包含与igh和igl(即或)恒定区mrna二者互补的延伸序列的dna折纸纳米结构通过电穿孔进行转染。dna折纸分子结合并保护个体细胞内的胞内igh和iglmrna,然后该细胞被裂解,然后与mrna结合的折纸被重新分离和纯化。作为逆转录引物使用mrna捕获探针,igh和igl基因序列在折纸捕获探针上被延伸。然后通过标准igh/iglv-基因多重pcr来扩增cdna,以获得适合亿明达配对末端测序的扩增产物池。每个扩增子包含12聚体条码,条码可与其互补物配对,从而提供来自个体细胞的igh和iglmrna的序列信息而无需单细胞分选。
图2a-2c显示了dna折纸设计和合成。(a)5’-5’蝶形mrna捕获探针的组织。各种区域可用于纳入dna折纸纳米结构、下游pcr扩增、条码配对、以及igh/iglmrna捕获。(b)当与bcrigh和iglmrna退火时,自折纸纳米结构的表面延伸的5’-5’蝶形mrna捕获探针的可视化示意图(注意:5’-5’蝶形捕获探针的长度被放大,以允许结构的可视化)。(c)通过afm可视化显示预期的“晶片”形状,来验证dna折纸分子的正确折叠(探针过于挠性,无法通过afm来可视化)。每个折纸分子的规模约为60×90nm。
图3a-3c显示包含互补条码序列的5’-5’蝶形mrna捕获探针的构建。(a)10聚体条码链的第一链合成后,建立1:1:1的条码:5’-5’链:油-水乳液液滴反应。(b)使用条码链作为模板的5’-5’链的重叠延伸允许互补条码被纳入到5’-5’链的任一末端上。(c)在变性page纯化后,使用衔接体链进行模块化t4dna连接反应,以将基因特异性互补mrna捕获序列附着到5’-5’链的任一末端上。在最终变性page纯化和定量后,所述条码5’-5’mrna捕获探针可被纳入到个体dna折纸纳米结构中。
图4a-4c展示了dna折纸纳米结构与靶mrna的结合。(a)靶mrna与dna折纸结构特异性结合:使用报告荧光素分子,靶mrna的特异性结合导致的dna折纸的移动被可视化。泳道1-3:用体外转录的靶mrna孵育的不含有靶特异性延伸的主要序列(staplesequences)的dna折纸(1)、单独孵育的不含有靶特异性延伸的主要序列的dna折纸(2)、或用无义(乱序)mrna孵育的不含有靶特异性延伸的主要序列的dna折纸(3)。泳道4:体外转录的靶mrna(不可见),泳道5:mw标志物(不可见),泳道6-7:dna折纸与延伸的靶特异性主要序列单独孵育(6)或与体外转录的靶mrna孵育(7)。特异性fret信号识别dna折纸结构。(b)选择的afm图像显示了与具有靶特异性延伸的主要序列(而不是非靶主要序列)的折纸结合的体外转录的靶mrna的特异性(mrna仅在折纸分子的一侧可见)。(c)选择的afm图像显示了两个体外转录的靶mrna的结合,用含有α和β主要序列的个体折纸捕获(mrna结合在折纸分子的两侧)。
图5a-5b展示了用dna折纸纳米结构转染原代淋巴细胞。(a)来自小鼠的淋巴细胞被模拟转染(左)或用fitc-标记的dna折纸纳米结构转染(中间)。基于通过流式细胞术检测fitc标记,被转染的细胞被可视化。为了确保折纸结构被细胞吸收并且没有与细胞表面结合,在转染后用dna酶处理细胞。在dna酶处理之后,自dna折纸检测到类似的fitc信号(右)。(b)如凝胶电泳所证实的,dna酶消化在孵育后破坏了细胞表面上的dna折纸。
图6是显示示例性的下一代测序(ngs)分析方案的流程图。将运行的来自个体的序列解析为fastq文件,用于使用bwa-mem软件的比对。生成分析文件用于映射、读数计数、以及结点检测,并且进行进一步处理,用于序列和扩增偏差的质量控制。
尽管本发明可以进行大量的改变和采用替代形式,在附图和以下详述中以示例的方式显示了本发明的示例性实施方式。但是应当理解,附图和详述并不是将本发明的范围限制在所揭示的示例性形式,本发明应包括所附权利要求书所揭示的本发明范围内的所有变化、等价形式和替代形式。
具体说明
说明书中所引用的所有出版物,包括但不限于专利和专利申请通过引用纳入本文,如同它们完全示于本申请中那样。
发明人之前开发了新的dna折纸纳米结构,其具有自被转染的细胞捕获和保护mrna序列的能力。dna折纸上的完整的荧光标记促进了被转染的细胞的识别,并且与mrna结合的纳米结构的重新分离使用整合的生物素标签和亲和素柱纯化来实现。本文提供的方法至少部分基于发明人对以下稳健的方法的发现:该方法用于配对来自个体细胞的多个感兴趣的基因,而无需单细胞分选或特定的设备(图1)。本公开提供了用于将“蝶形条码”技术纳入dna折纸纳米结构以及其它基于核酸的纳米结构的新的方法,以允许来自数百万个细胞的mrna的同时配对和捕获。微孔化(使用微升体积进行细胞电穿孔)技术中的额外的改进提高了转染效率,并改善了细胞活力。最终,蝶形条码捕获方法和基于系统的油-水乳液液滴的组合以及重叠延伸pcr能够创造数百万个配对的随机序列,所述配对的随机序列可被用作条码以在测序中将个体遗传序列相互连接。通过用独特的匹配条码化的序列分析来连接mrna,发明人开发了一种成本效益高、对分析大细胞群有用且可容易地适用于探测来自几乎所有的异质性细胞群的配对基因的序列多样性的方法。
对于其它可转染的核酸杂交平台,对于需要连接多种mrna种类的分析,不存在其他成功的分子方法。当尝试单链寡核苷酸的转染时,单链寡核苷酸激活risc复合物并被降解,阻断下游分析。不受任何特定的机理或理论所束缚,正如重新捕获和分离与mrna结合的被转染的折纸分子的能力所证明的那样,认为折纸分子的半超螺旋性质抑制了由risc复合物导致的降解。另外,关于标准的单细胞的基于乳液的系统,它们不适于需要连接多种mrna种类的分析。尽管基于乳液的系统允许输入比单细胞分选更高的细胞数,但现有的单细胞的基于乳液的系统仅捕获7对序列(>0.000001%),且在浓度小于1000/1000000时无法检测到它们的对照的存在。虽然认为大多数克隆以人均单克隆的方式存在,但这种灵敏度水平对于发现稀有细胞是不可接受的。
因此,在第一方面,本文提供一种将单链dna(ssdna)条码化的多核苷酸纳入核酸折纸纳米结构的方法。所述方法基本上包括或由以下步骤组成:(a)(在随后的油-乳液步骤之前或期间)进行条码核酸的第一链合成以产生互补的双链条码核酸,其中条码核酸包括至少一个与单链蝶形接头dna链的一部分互补的引发位点,所述单链蝶形接头dna链包括与条码核酸互补的序列和与核酸折纸纳米结构互补的序列侧接的中心5’-5’磷酸二酯接头;(b)在包含用于延伸靶核酸的试剂的油-乳液液滴中合并双链条码核酸(或单链条码和互补的引物组)和5’-5’蝶形接头核酸;(c)使包含双链条码核酸(或单链条码核酸引物以及互补引物)和5'-5'蝶形接头核酸的油-乳液液滴热循环,所述热循环足以导致每条链自双链条码核酸退火至5'-5'蝶形接头核酸上的互补序列,并延伸单链dna(ssdna)条码化的5'-5'蝶形多核苷酸,从而产生延伸产物液滴;(d)从液滴中提取延伸产物;(e)由提取的延伸产物纯化ssdna条码化的5’-5’蝶形多核苷酸;以及(f)将纯化的ssdna条码化的5’-5’蝶形多核苷酸纳入核酸折纸纳米结构。
如本文所用,术语“条码”是指独特的核苷酸序列,其可用于区分、配对、以及唯一地识别来自相同的细胞的核酸。在一些情况下,条码可被用于区分例如源于不同细胞或其它来源的数十、数百、或甚至数千个核酸。如本文所用,术语“蝶形条码”是指一组通过独特的5’-5’非链磷酸二酯dna“蝶形”结构(指中心磷酸二酯接头两侧侧接有互补的条码核酸序列)连接在一起的互补的条码序列。
如本文所用,术语“核酸纳米结构”只是为了方便而使用,应理解本发明通常涵盖核酸纳米结构。本发明的纳米结构可以是线性(例如,纳米棒)或非线性(例如,星形,三角形等)的。核酸如dna或rna可使用各种技术(如dna或rna折纸)被折叠为预定的一维、二维、或三维纳米结构。
术语“寡核苷酸”、“核苷酸”和“核酸”可互换地使用,是指含有与磷酸基团和可替换的有机碱基连接的糖(例如,核糖或脱氧核糖)的分子,所述有机碱基是取代的嘧啶(例如胞嘧啶(c),胸苷(t)或尿嘧啶(u))或取代的嘌呤(例如腺嘌呤(a)或鸟嘌呤(g))。因此,该术语包括dna和rna寡核苷酸。该术语还应当包括多聚核苷(即,多核苷酸减去磷酸)和任何其他包含有机碱基的聚合物。寡核苷酸可获自现有的核酸源(例如,基因组或cdna),但优选是合成的(例如,通过核酸合成生成)。
条码化的5’-5’蝶形接头捕获探针的构建:条码化的多核苷酸是长ssdna链,以包含中心5’-5’“蝶形”连接的方式构建,以允许两个末端均为5'→3'。所述蝶形连接可包括预合成的dna序列(例如,从商业供应商订购)。优选地,条码化的多核苷酸的任一末端可额外地包含与m13mp18噬菌体dna折纸骨架互补的特异性序列。该序列可以变化,并且使条码适于纳入到任何靶纳米结构中。根据图2a,任一末端可额外地包括用于下游扩增的保守pcr引物位点、与链的相对端上的条码互补的独特的条码、第二保守引发位点、以及与感兴趣的基因的保守区域互补的mrna捕获位点。捕获序列的一个重要设计特征是mrna通过包含在5'-5'蝶形链各端的一组独特的互补核苷酸条码来相互配对。
长条码化的蝶形多核苷酸然后被纳入到dna折纸主混合物(或其它所需的纳米结构)中,并且条码蝶形中的m13mp18互补序列与折纸纳米结构自组装。根据图2b,蝶形的mrna捕获序列区域延伸自折纸纳米结构的表面。在一些情况下,将ssdna条码化的5’-5’蝶形多核苷酸纳入核酸纳米结构可包括退火。
如本文所述,油-乳液液滴可被用于将一组独特的互补条码纳入数百万(或更多)蝶形链中。包含中心5’-5’非传统磷酸二酯键的基础(base)蝶形链可包括与核酸折纸纳米结构的序列互补的序列以及购自商业供应商(例如,整合dna技术公司(integrateddnatechnologies,inc.))的两个不同的保守引发位点。在一些情况下,所述5’-5’蝶形链例如具有如下序列:
3’-cgagtccctttatcgggaac-5’-5’-gaacgtggcgagaaaggaagggaacaaactatggacagcaaagacagcacct-3’(seqidno:1)。
在一些情况下,所述条码链具有如下序列:
5’caccgactttgactcccaaatcaatgtgcggacagcaaagacagcacctnnnnnnnnnnnngctcagggaaatagcccttggggtagccttttgtttgtttgcaatctctg3’(seqidno:2),其中“n”可以是dna中发现的四个氮碱基中的任何一个(腺嘌呤、胞嘧啶、鸟嘌呤或胸腺嘧啶)。
在另一些情况下,条码标记的链包含与一侧5’-5’蝶形链互补的一个引发位点;随机的10聚体核苷酸条码(410=1048576个独特的条码,如果要分析>106个序列,可使用11聚体或12聚体条码);以及与另一侧5’-5’蝶形链互补的第二引发位点,其具有与条码链两端侧接的特异性mrna(互补)捕获序列。短条码链可在第一链合成反应中使用,以创造具有互补条码的dsdna产物,或者短条码链可在油-乳液延伸步骤期间在标准聚合酶链式反应(pcr)的下游使用,以直接在乳液液滴中合成dsdna条码链。根据图3a,>1010相同的ssdna5’-5’蝶形链和独特的dsdna条码链(或具有互补引物的ssdna条码链)之后在油-水乳液液滴重叠延伸伸长系统中以1:1:1的分子:分子:液滴的比例被纳入。标准pcr循环可被用于使dsdna条码链变性(或在变性后扩增ssdna条码链),以使自dsdna条码链的两条ssdna链与5’-5’蝶形的任一末端退火,并将ssdna条码链用作模板来延伸蝶形。
在变性后,随着来自dsdna条码的每个链与5’-5’蝶形接头上的其互补的引发位点(作为重叠延伸的引物和模板起作用)退火,重叠延伸在每个液滴中进行,并因此纳入5’-5’蝶形链的任一末端上的互补的10聚体条码和蝶形链3’末端上的mrna特异性捕获序列(图3b)。虽然该系统不是指数pcr扩增,但采用多个解离/退火/延伸循环以确保每个5'-5'接头的两端都经延伸。然后使用标准乙醚/乙酸乙酯萃取方案自油-水乳液系统中提取pcr产物,并通过标准的变性page纯化法自残留的核酸中纯化ssdna条码化的5’-5’蝶形链。此时产物将包括(以从5'-5'中心蝶形连接向两个3'末端延伸为序):折纸互补序列(可以是仅一侧),保守pcr引发位点,与5’-5’连接的相对臂上的条码互补的随机10聚体条码,以及第二保守引发位点,以及基因特异性mrna捕获序列(图3b)。在最终变性page纯化之后,最终产物将包含必需的区域:1)纳入dna折纸结构,2)用于下游pcr扩增的保守引发位点,3)用于配对捕获的mrna的互补条码,以及4)与感兴趣的基因的保守区域互补的mrna捕获序列,其可用作基因特异性逆转录引物(图3c)。
这些独特的蝶形条码可通过简单改变链的“折纸退火序列”部分的特征而被纳入个体折纸分子或任何感兴趣的核酸结构。由于条码化的mrna捕获序列独特地附着到每个蝶形链,该方法允许极高水平的mrna配对特异性。
使用捕获链上的保守序列作为基因特异性rt引物,将回收的mrna逆转录(rt)为cdna,从而自蝶形条码的3'末端延伸基因特异性cdna。由于蝶形条码上的保守基因特异性序列是引发rt反应所必需的,因此任何未结合的mrna都不会被扩增,因而可改善灵敏度和避免错误配对。
逆转录和条码连接的mrna扩增子生成(图1):一旦mrna结合的折纸被从细胞裂解物中纯化,就进行逆转录(rt)。使用蝶形条码的mrna互补区域作为rt引物,延伸可通过简单添加任何商业获得的rt主混合物实现,并且根据制造商的建议进行孵育。然后mrna可通过添加商业rna酶h混合物来移除,并且条码化的cdna作为来自条码蝶形的3’末端的延伸产物而获得。我们预先使用体外转录的靶mrna结合的折纸验证了该方法,并确认了通过pcr分离基因特异性cdna的能力。最终,多重pcr(针对蝶形条码的任一末端上的两个保守引发序列使用单一引物),并取决于技术的应用,1)可进行用于tcrα/tcrβv-基因或igh/iglv家族的成熟多重引物组,以产生具有免疫受体相应cdr3区域的扩增子池或2)针对已知癌症驱动因子/共同驱动基因中感兴趣的突变之外的保守区域的单一引物,将产生条码配对的pcr产物。通过在引物组中容入亿明达特异性衔接子序列,可获得具有cdr3序列的条码化的扩增子、癌症突变序列、或任何来自异源细胞群的感兴趣的配对的基因序列,它们直接适用标准亿明达配对末端测序。
在另一方面,本文提供用于自大量单细胞平行捕获、条码化、和定量数十至数百或更多特异性dna和/或rna序列的方法,例如用于分析细胞群或其他目的的方法。在优选的实施方式中,用于在单细胞水平上检测靶核酸序列的方法包括或基本上由以下步骤组成:(a)使根据权利要求1的方法获得的核酸折纸纳米结构与分离自单细胞的核酸接触,其中所述纳米结构包括具有与靶核酸序列互补的条码序列的ssdna条码化的多核苷酸;并且其中如果单细胞中存在靶核酸序列,则接触在适合条码序列与靶核酸序列结合的条件下发生;(b)回收与条码序列结合的靶核酸序列;以及(c)使用ssdna条码化的多核苷酸作为逆转录的基因特异性引物,对回收的靶核酸序列进行逆转录,由此如果单细胞中存在靶核酸,则靶核酸在没有单细胞分选步骤的情况下被检测。
在又一方面,本文提供一种在单细胞水平上检测b细胞受体的方法。所述方法基本上包括或由以下步骤组成:(a)转染至抗原特异性b细胞;(b)与包含一组或多组mrna捕获序列的dna折纸纳米结构接触,以在被转染的抗原特异性b细胞中捕获和保护免疫球蛋白重链(igh)和轻链(igl)bcrmrna;(c)分离经接触的dna折纸纳米结构以回收与一个或多个mrna捕获序列结合的igh和iglmrna;以及(d)使用一组或多组mrna捕获探针作为逆转录的基因特异性引物,对回收的igh和iglmrna进行逆转录,由此靶bcr序列在没有单细胞分选步骤的情况下被检测。
在一些情况下,dna折纸纳米结构包括完整的生物素标签,且分离经接触的纳米结构步骤可包括亲和素柱纯化。
如本文所用,核酸或核酸分子可包括任何感兴趣的核酸。在一些实施方式中,核酸包括但不限于dna、rna、肽核酸、吗啉代核酸、锁核酸、乙二醇核酸、苏糖核酸、它们的混合物、以及它们的杂合体、或具有内部碳骨架间隔区的核酸。在一些方面,核酸是“引物”,当条件适合于引物延伸产物的合成时,该“引物”能够作为沿着核酸互补链的合成起始点起作用。核酸可以是单链、双链、也可以是三链。在一些方面,核酸作为合成互补核酸的模板,例如,通过核苷酸单元的碱基互补纳入。例如,在一些方面,核酸包括天然存在的dna(包括基因组dna)、rna(包括mrna)、和/或包括合成分子(包括但不限于互补dna(cdna)和以任何方式产生的重组分子)。在一些方面,核酸产自化学合成、逆转录、dna复制或这些生产方法的组合。
核酸可使用任何合适的方法获得,包括maniatis等在molecularcloning:alaboratorymanual(《分子克隆:实验室手册》),纽约冷泉港,280-281页(1982)中描述的哪些。在一些方面,核酸以美国专利申请公开号2002/0190663中描述的方式获得。从生物样品中获得的核酸通常被片段化以产生适用于分析的片段。
本发明的核酸和/或其他部分可以分离。如本文所用,“分离”意指与其通常关联的至少一些组分分开,无论其是衍生自天然存在的来源还是全部或部分地合成制备。本发明的核酸和/或其他部分可以纯化。如本文所用,“纯化”意指与大多数其他化合物或实体分开。化合物或部分可以是部分纯化的或基本上纯化的。纯度可以通过重量测量来表示,并且可以使用各种分析技术来确定,例如但不限于质谱、hplc等。
除非另有限定,在此使用的所有技术和科学术语均与本发明所属领域技术人员的通常理解一致。如本文定义和使用的所有定义应理解为控制在字典定义、通过引用并入的文献中的定义、和/或定义的术语的普通含义。
在本申请的说明书和权利要求书中,除非有明确的相反说明,不定冠词“一个”和“一种”应理解为“至少一个(种)”。
本说明书中和权利要求中所用的短语“和/或”应理解为指相关联要素“之一或两者”,即,在一些情况中联合存在而在其它情况中非联合存在的要素。采用“和/或”列出的多种要素应以同一种方式解释,即如此连述的“一种或多种”所述要素。由“和/或”从句特定指出的要素以外的其它要素可选存在,不论是否与那些特定指出的要素相关。因此,作为一个非限定性例子,提及“a和/或b”与开放式语句例如“包括”联用时可以在一个实施方式中仅指a(可选地包括除了b以外的要素);而在其它实施方式中仅指b(可选地包括除了a以外的要素);而在另一实施方式中既指a又指b(可选地包括其它要素);等等。
本文的说明书和权利要求所用的“或”应理解为与如上定义的“和/或”有相同的含意。例如,当在一个列表中隔离项目时,“或”或者“和/或”应解释为包括性,即,包括至少一个,但也包括多于一个的一些要素或要素列表,以及可选的额外未列出项目。仅清楚指示相反的术语例如“其中仅一个”或“确定的一个”或在权利要求中使用时的“由……组成”,指包含一些要素或要素列表中确定的一个要素。一般而言,当其之前有排他性术语时例如“任一”、“其中之一”、“仅其中之一”或“明确的其中之一”,本文所用的术语“或”应仅解释为指示排他性选择(即,“一个或另一个但非两者”)。
“基本由……组成”用于权利要求中时,应具有其在专利法领域中使用的普通含意。
如本文所用,关于数字的术语“近似”或“约”通常被认为包括在任一方向(大于或小于)数字的5%范围内的数字,除非另有说明或从上下文中明显看出(除非此类数字超过可能值的100%)。在陈述范围的情况下,除非另有说明或从上下文中显而易见,否则端点包括在该范围内。
已通过一个或多个优选的实施方式描述了本发明,但应理解除了明确描述的那些方案之外,许多等同方案、替代方案、变化方案和修改方案是可能实现的并在本发明范围内。在考虑以下非限制性实施例的基础上,能够更完整地理解本发明。
实施例
实施例1-逆转录和条码连接的mrna扩增子生成
一旦mrna结合的折纸被从细胞裂解物中纯化,则进行逆转录(rt)。使用蝶形条码的mrna互补区域作为rt引物,通过添加任何商业获得的rt主混合物实现延伸,并且根据制造商的建议进行孵育。然后mrna通过添加商业rna酶h混合物来移除,并且条码化的cdna作为自条码蝶形的3’末端的延伸产物而获得。我们使用体外转录的靶mrna结合的折纸预先验证了该方法,并确认了通过pcr分离基因特异性cdna的能力。最终,对于蝴形条码的任一末端上的每个保守引发序列,使用单一引物进行多重pcr。取决于技术的应用,(1)用于tcrα/tcrβ基因或bcrigh/iglv家族的成熟多重引物组用以产生具有免疫受体相应cdr3区域的扩增子池;或(2)已知癌症驱动/共驱动基因中感兴趣的突变之外的保守区域的单一引物用于产生条码配对的pcr产物;或(3)单一引物,用于与不同生物物种或细胞群内的异源基因相邻的保守区域。通过在引物组中容入亿明达特异性衔接子序列,获得具有cdr3序列的条码化扩增子,其直接适用于标准亿明达配对末端测序。
实施例2-dna折纸与免疫受体mrna结合
我们预先开发了用于异二聚体的t细胞受体(tcr)的分析的方法,其在多样性和异质细胞群方面存在类似的问题。图4a-4c显示了在已知转基因tcr(p14转基因t细胞)的分析的背景下的初步数据。折纸纳米结构被设计为每个单独折纸分子中包含多个mrna捕获探针。我们改变了我们的纳米结构设计以在每个折纸分子中纳入单个5’-5’条码化的mrna捕获链。虽然假定mrna结合动力学相似,但将进行优化实验以评估mrna结合效率。之前,当包含tcrα-和tcrβ-特异性探针两者的折纸仅用tcrβmrna孵育时,mrna仅被折纸分子的一侧捕获(推测可能是tcrβ-特异性探针)(图4b)。当包含tcrα-和tcrβ-特异性探针两者的折纸用tcrα和tcrβmrna两者孵育时,折纸的两侧捕获mrna,表明两种类型的tcrmrna与各个折纸分子任一侧的相应探针结合(图4c),证实正确mrna结合的能力和特异性。再次,对于我们先前分析tcrmrna结合的实验,我们使用折纸分子任一侧上的个体探针链,其分别与tcrα或tcrβ序列互补。尽管mrna的捕获将以略微不同的取向发生,但预期折纸-mrna捕获不会发生显著变化。
实施例3-dna折纸纳米结构的转染
之前的研究已经通过窖蛋白依赖性途径利用dna纳米结构的受体介导的内吞作用,导致微管转运至溶酶体并因此导致纳米结构的分解。该策略的突出问题是为了mrna捕获和随后的重新分离,纳米结构必须绕过内吞作用途径,而穿过膜直接至细胞质。电穿孔是进入细胞质的最简单的方法,并且因此可避免内体降解的有害影响。但是该方法通常伴随高细胞死亡率。微孔化(使用μl体积电穿孔)技术的最新进展已经显示了高转染效率(>93%)以及高细胞活力(>86%)。对于b细胞转染实验,我们将使用表达与先前产生的杂交瘤相同的kl25序列bcr的kl25igl/igh转基因小鼠[34]。通过机械破碎和在0.83%nh4cl中的红细胞裂解制备4-6周龄kl25igl/igh转基因小鼠的脾细胞。用磁性细胞分选来纯化igm+b细胞并用流式细胞术确认>95%纯度的分选群。通过离心(1200rpm,5分钟,4℃)使细胞沉淀,用opti-memtm培养基(吉布可公司(gibco))洗涤,并以1×107个细胞/ml重悬于opti-memtm培养基中。对于电穿孔,使用具有100μl注射器头的氖注射器转染系统(赛默飞世尔科技公司(thermoscientific)),使用以下设置:2000v,10ms,1脉冲。样品将由100μl细胞悬浮液和25μl(20nm)dna折纸悬浮液(在1xtae-mg2+中)或25μl1xtae-mg2+缓冲液的模拟转染对照组成。在电穿孔之后,立即将细胞转移到含有10%胎牛血清的新鲜rpmi-1640培养基中,并在96孔板的单独孔中于37℃孵育12-24小时。为了评估转染效率,将在lsrfortessa流式细胞仪上观察细胞;纳入每个纳米结构中的异硫氰酸荧光素(fitc;488nm激发,518nm发射)标签允许通过流式细胞术鉴定成功转染的细胞。
我们预先开发了针对小鼠原代t淋巴细胞的该转染方案(图5a),并预期类似的设置将为b细胞转染提供初始出发点。在我们先前的实验中,我们验证了dna折纸纳米结构进入被转染的淋巴细胞而不是非特异性地结合到细胞表面(通过用浓缩的dna酶(turbodna酶,ambion,生命科技公司(lifetechnologies))处理被转染的细胞将产生假阳性读数),然后通过facs分析法分析细胞的成功转染(图5a)。我们还确认了浓缩的dna酶处理导致了dna纳米结构的成功破坏(图5b)。因此,dna折纸对初级淋巴细胞具有优异的转染效率。应当注意,turbodna酶是dna酶i的专有工程版本,其对dna的亲和力明显高于野生型dna酶i,我们发现野生型dna酶i在折纸纳米结构降解方面效果较差。另外,我们小组之前已经证明dna折纸在细胞裂解液中高度稳定12小时[33],证实这些结构对细胞溶质核酸酶降解具有抗性。
实施例4-序列数据管理、处理和分析(预测)
该项目的成功取决于经验证的数据处理流程(pipeline)和负责管理、处理和共享大量数据的经验丰富的信息学团队。因此,使用用于分析配对的全基因组和转录组下一代测序数据的经验证的流程,如下一代数据处理和分析流程,其利用经验证的软件工具并产生标准的非平台依赖性格式(图6)。在流程中的每个步骤,创建统计文件以确保所有过程已经完成并且文件未被破坏。统计文件包含“被比对的碱基”、“错配率”和“md5sum”。在流程的早期,调用了几个额外的检查,包括对整体文库覆盖率、文库一致性、碱基质量和污染检查的评估。该流程围绕以下构建:(1)标准化的文件格式,(2)多个质量控制检查,(3)自动处理,(4)序列数据、测序比对和变体识别的计划性发布,以及(5)集中式主数据处理。
为了量化任何给定碱基处的偏差,读数经历一系列质量控制步骤,然后被解析成独立的fastq文件以使用bwa-mem进行比对,以进行独特的cdr3结构的精确拆分读取比对。每个序列具有与vκ或vh基因区段之一匹配的12个核苷酸(对应于来自处于v区段3'末端的第二个保守半胱氨酸的cass共有氨基酸序列)和与保守的苯丙氨酸相对应的匹配j区段的6个核苷酸。这些密码子之间的核苷酸总数决定了cdr3区域的长度并因此确定了cdr3区域的阅读框。处理后的序列数据将存储在asu安全关系数据库管理系统中,该系统允许webapp前端通过jaspersoftserver,以及安全mongodb实例,该实例允许adhoc查询。来自个体细胞的igh和igl序列的配对将通过基本的“if-then”算法进行,在每个读数的10聚体条码序列延伸处搜索互补碱基配对。
参考文献
1.arstila,t.p.,等,adirectestimateofthehumanalphabetatcellreceptordiversity(《人类的αβt细胞受体多样性的直接估计》)。science,1999年286(5441):958-61页。
2.bartok,i.,等,tcellreceptorcdr3loopsinfluencealphabetapairing(《t细胞受体cdr3环影响αβ配对》)。molimmunol,2010年47(7-8):1613-8页。
3.corthay,a.,k.s.nandakumar,和r.holmdahl,evaluationofthepercentageofperipheraltcellswithtwodifferenttcellreceptoralpha-chainsandoftheirpotentialroleinautoimmunity(《具有两种不同t细胞受体α链的外周t细胞的百分比的评估及其在自身免疫中的潜在作用的评估》)。jautoimmun,2001年16(4):423-9页。
4.rani,l.,等,immunoglobulinheavychainvariableregiongenerepertoireandb-cellreceptorstereotypesinindianpatientswithchroniclymphocyticleukemia(《印度慢性淋巴细胞白血病患者的免疫球蛋白重链可变区基因库和b细胞受体定型》)。leuklymphoma,2016年1-12页。
5.li,x.,dynamicchangesofdrivergenes'mutationsacrossclinicalstagesinninecancertypes(《9种癌症类型的临床期期间驱动基因突变的动态变化》)。cancermed,2016年。
6.newdrivermutationsdetectedinnsclc(《在nsclc中检测到的新的驱动突变》)。cancerdiscov,2016年。
7.anoosha,p.,等,discriminationofdriverandpassengermutationsinepidermalgrowthfactorreceptorincancer(《癌症中表皮生长因子受体的驱动突变和乘客突变的区分》)。mutatres,2015年780:24-34页。
8.bozic,i.,等,accumulationofdriverandpassengermutationsduringtumorprogression(《在肿瘤进展期间驱动突变和乘客突变的累积》)。procnatlacadsciusa,2010年107(43):18545-50页。
9.busse,c.e.,等,single-cellbasedhigh-throughputsequencingoffull-lengthimmunoglobulinheavyandlightchaingenes(《基于单细胞的全长免疫球蛋白重链和轻链基因的高通量测序》)。eurjimmunol,2014年44(2):597-603页。
10.dash,p.,等,pairedanalysisoftcralphaandtcrbetachainsatthesingle-celllevelinmice(《小鼠的单细胞水平上的tcrα和tcrβ链配对分析》)。jclininvest,2011年121(1):288-95页。
11.dekosky,b.j.,等,high-throughputsequencingofthepairedhumanimmunoglobulinheavyandlightchainrepertoire(《配对的人免疫球蛋白重链和轻链谱的高通量测序》)。natbiotechnol,2013年31(2):166-9页。
12.chen,a.k.,等,ratiometricbimolecularbeaconsforthesensitivedetectionofrnainsinglelivingcells(《用于在单个活细胞中灵敏检测rna的比率双分子信标》)。nucleicacidsres,2010年38(14):e148页。
13.tyagi,s.和f.r.kramer,molecularbeacons:probesthatfluoresceuponhybridization(《分子信标:杂交后发荧光的探针》)。natbiotechnol,1996年14(3):303-8页。
14.mhlanga,m.m.,等,trna-linkedmolecularbeaconsforimagingmrnasinthecytoplasmoflivingcells(《用于在活细胞的细胞质中成像mrna的trna连锁的分子信标》)。nucleicacidsres,2005年33(6):1902-12页。
15.dittmer,w.u.,a.reuter,和f.c.simmel,adna-basedmachinethatcancyclicallybindandreleasethrombin(《可循环结合和释放凝血酶的基于dna的机器》)。angewchemintedengl,2004年43(27):3550-3页。
16.benenson,y.,等,anautonomousmolecularcomputerforlogicalcontrolofgeneexpression(《用于基因表达逻辑控制的自主分子计算机》)。nature,2004年429(6990):423-9页。
17.ko,s.,等,dnananotubesascombinatorialvehiclesforcellulardelivery(《dna纳米管作为细胞传递的组合载体》)。biomacromolecules,2008年9(11):3039-43页。
18.chen,y.j.,等,dnananotechnologyfromthetesttubetothecell(《dna纳米技术从试管到细胞》)。natnanotechnol,2015年10(9):748-60页。
19.zhang,x.,等,quantitativeassessmentofratiometricbimolecularbeaconsasatoolforimagingsingleengineeredrnatranscriptsandmeasuringgeneexpressioninlivingcells(《作为成像单个工程rna转录物和测量活细胞中基因表达工具的比率双分子信标的定量评估》)。nucleicacidsres,2013年41(15):e152页。
20.kelso,g.f.,等,impactonmonoclonalantibodyproductioninmurinehybridomacellculturesofadenosinereceptorantagonistsandphosphodiesteraseinhibitors(《在腺苷受体拮抗剂和磷酸二酯酶抑制剂的鼠杂交瘤的细胞培养物中单克隆抗体生产的影响》)。bioorgmedchemlett,2016年26(2):540-4页。
21.frame,k.k.和w.s.hu,thelossofantibodyproductivityincontinuouscultureofhybridomacells(《杂交瘤细胞连续培养中抗体生产力的丧失》)。biotechnolbioeng,1990年35(5):469-76页。
22.franek,f.和j.dolnikova,hybridomagrowthandmonoclonalantibodyproductioniniron-richprotein-freemedium:effectofnutrientconcentration(《富含铁的无蛋白培养基中杂交瘤的生长和单克隆抗体的产生:营养浓度的影响》)。cytotechnology,1991年7(1):33-8页。
23.han,d.,等,dnaorigamiwithcomplexcurvaturesinthree-dimensionalspace(《三维空间中具有复杂的曲率的dna折纸》)。science,2011年332(6027):342-6页。
24.ke,y.,等,scaffoldeddnaorigamiofadnatetrahedronmolecularcontainer(《dna四面体分子容器的支架dna折纸》)。nanolett,2009年9(6):2445-7页。
25.han,d.,等,dnagridironnanostructuresbasedonfour-armjunctions(《基于四臂连接的dna网格纳米结构》)。science,2013年339(6126):1412-5页。
26.rinker,s.,等,self-assembleddnananostructuresfordistance-dependentmultivalentligand-proteinbinding(《用于距离依赖性多价配体-蛋白质结合的自组装dna纳米结构》)。natnanotechnol,2008年3(7):418-22页。
27.sharma,j.,等,controlofself-assemblyofdnatubulesthroughintegrationofgoldnanoparticles(《通过整合金纳米粒子的dna小管的自组装的控制》)。science,2009年323(5910):112-6页。
28.ke,y.,等,self-assembledwater-solublenucleicacidprobetilesforlabel-freernahybridizationassays(《用于无标记rna杂交测定的自组装水溶性核酸探针片》)。science,2008年319(5860):180-3页。
29.blattman,j.n.,等,evolutionofthetcellrepertoireduringprimary,memory,andrecallresponsestoviralinfection(《在对病毒感染的原发性、记忆性和回忆性应答期间t细胞库的演化》)。jimmunol,2000年165(11):6081-90页。
30.blattman,j.n.,等,therapeuticuseofil-2toenhanceantiviralt-cellresponsesinvivo(《在体内增强抗病毒t细胞应答中的il-2的治疗用途》)。natmed,2003年9(5):540-7页。
31.schietinger,a.,等,rescuedtolerantcd8tcellsarepreprogrammedtoreestablishthetolerantstate(《被拯救的耐受cd8t细胞被预编程以重建耐受状态》)。science,2012年335(6069):723-7页。
32.rothemund,p.w.,foldingdnatocreatenanoscaleshapesandpatterns(《折叠dna以创建纳米级形状和图案》)。nature,2006年440(7082):297-302页。
33.mei,q.,等,stabilityofdnaorigaminanoarraysincelllysate(《dna折纸纳米阵列在细胞裂解液中的稳定性》)。nanolett,2011年11(4):1477-82页。
34.seiler,p.,等,enhancedvirusclearancebyearlyinduciblelymphocyticchoriomeningitisvirus-neutralizingantibodiesinimmunoglobulin-transgenicmice(《通过免疫球蛋白转基因小鼠中的早期诱导型淋巴细胞脉络丛脑膜炎病毒中和抗体增强的病毒清除率》)。jvirol,1998年72(3):2253-8页。
35.miconnet,i.,probingthet-cellreceptorrepertoirewithdeepsequencing(《用深度测序探测t细胞受体库》)。curropinhivaids,2012年7(1):64-70页。
36.venturi,v.,等,methodforassessingthesimilaritybetweensubsetsofthetcellreceptorrepertoire(《用于评估t细胞受体库的子集之间的相似性的方法》)。jimmunolmethods,2008年329(1-2):67-80页。
37.brandle,d.,等,tcelldevelopmentandrepertoireofmiceexpressingasingletcellreceptoralphachain(《t细胞发育和表达单个t细胞受体α链的小鼠的所有组成成分》)。eurjimmunol,1995年25(9):2650-5页。
38.burns,r.p.,jr.,等,molecularanalysisofskewedtcra-vgeneuseint-cellreceptorbeta-chaintransgenicmice(《用于t细胞受体β链转基因小鼠的偏斜tcra-v基因的分子分析》)。immunogenetics,1998年47(2):107-14页。
39.cabaniols,j.p.,等,mostalpha/betatcellreceptordiversityisduetoterminaldeoxynucleotidyltransferase(《大多数α/βt细胞受体多样性归因于末端脱氧核苷酸转移酶》)。jexpmed,2001年194(9):1385-90页。
40.ewing,c.,等,virus-specificcd8+t-cellresponsesinmicetransgenicforat-cellreceptorbetachainselectedatrandom(《在随机选择的t细胞受体β链转基因小鼠中的病毒特异性cd8+t细胞应答》)。jvirol,1994年68(5):3065-70页。
41.turner,s.j.,s.c.cose,和f.r.carbone,tcralpha-chainusagecandetermineantigen-selectedtcrbeta-chainrepertoirediversity(《tcralpha链使用可以确定抗原选择的tcrβ链谱系多样性》)。jimmunol,1996年157(11):4979-85页。
42.hayakawa,k.,等,isolationofhigh-affinitymemorybcells:phycoerythrinasaprobeforantigen-bindingcells(《高亲和力记忆b细胞的分离:藻红蛋白作为抗原结合细胞的探针》)。procnatlacadsciusa,1987年84(5):1379-83页。
43.venturi,v.,等,methodsforcomparingthediversityofsamplesofthetcellreceptorrepertoire(《用于比较t细胞受体库样品多样性的方法》)。jimmunolmethods,2007年321(1-2):182-95页。
44.hurlbert,s.h.,thenonconceptofspeciesdiversity:acritiqueandalternativeparameters(《物种多样性的非概念:批判与替代参数》)。ecology,1971年52(4):577页。
45.preston,f.w.,thecommonness,andrarity,ofspecies(《物种的共性和稀有性》)。ecology,1948年29(3):254-283页。
46.futschik,a.和c.schlotterer,thenextgenerationofmolecularmarkersfrommassivelyparallelsequencingofpooleddnasamples(《来自合并dna样品的大规模平行测序的下一代分子标记》)。genetics,2010年186(1):207-18页。
47.johnson,p.l.和m.slatkin,inferenceofpopulationgeneticparametersinmetagenomics:acleanlookatmessydata(《宏基因组学中群体遗传参数的推断:清晰地看待杂乱的数据》)。genomeres,2006年16(10):1320-7页。
48.casrouge,a.,等,sizeestimateofthealphabetatcrrepertoireofnaivemousesplenocytes(《未处理的小鼠脾细胞的αβtcr谱的大小估计》)。jimmunol,2000年164(11):5782-7页。
49.lynch,m.,estimationofnucleotidediversity,disequilibriumcoefficients,andmutationratesfromhigh-coveragegenome-sequencingprojects(《由高覆盖率的基因组测序项目的核苷酸多样性、不平衡系数和突变率的估计》)。molbiolevol,2008年25(11):2409-19页。
50.robins,h.s.,等,comprehensiveassessmentoft-cellreceptorbeta-chaindiversityinalphabetatcells(《αβt细胞中t细胞受体β链多样性的综合评估》)。blood,2009年114(19):4099-107页。
序列表
<110>代表亚利桑那州立大学的亚利桑那校董会(thearizonaboardofregentsonbehalfofarizonastate
university)
<120>用于个体细胞的配对mrna捕获和测序蝶形条码的高通量油-乳液合成
<130>m17-010l/112624.00928
<150>62/377,123
<151>2016-08-19
<160>2
<170>patentinversion3.5
<210>1
<211>72
<212>dna
<213>人工序列
<220>
<223>合成的
<400>1
cgagtccctttatcgggaacgaacgtggcgagaaaggaagggaacaaactatggacagca60
aagacagcacct72
<210>2
<211>111
<212>dna
<213>人工序列
<220>
<223>合成的
<220>
<221>misc_feature
<222>(50)..(61)
<223>n是a,c,g,或t
<400>2
caccgactttgactcccaaatcaatgtgcggacagcaaagacagcacctnnnnnnnnnnn60
ngctcagggaaatagcccttggggtagccttttgtttgtttgcaatctctg111
- 还没有人留言评论。精彩留言会获得点赞!