一种检测染色体重复片段断裂位点和定位信息的方法与流程
本发明涉及检验医学技术领域,具体地说,是一种检测染色体重复片段断裂位点和定位信息的方法。
背景技术:
拷贝数变异是与人类遗传病相关的一种重要的基因组变异,已经被证明与许多遗传病特别是神经发育相关疾病相关。以往针对拷贝数变异的检测技术包括染色体核型,荧光原位杂交(fish)、荧光定量pcr(q-pcr)和染色体芯片等。染色体芯片分析技术又被称为“分子核型分析”,能够在全基因组水平进行扫描,可检测染色体不平衡的拷贝数变异。染色体芯片检测周期快、通量高、成本合理,被认为是目前针对拷贝数变异的检测中,最适用于临床患者检测的技术,但目前以后的检测技术包括染色体芯片,都是定量或半定量的检测,对拷贝数变异的分析不够全面。
临床检测中所有检测到的拷贝数变异都应进行分类和判读,对于拷贝数缺失,根据染色体芯片的结果可以明确缺失基因的数量和断裂位点的具体位置,从而针对性地进行致病机制分析,为判断拷贝数缺失的致病性提供依据。
相较于拷贝数缺失,拷贝数重复与临床表型的关系更难解释,因为拷贝数增加可能是串联重复(tandemduplication)或是插入重复(dispersedduplication),而染色体芯片的局限性在于仅根据探针杂交信号提供的拷贝数变异结果不能确定其断裂位点,更无法判断拷贝数增加片段可能插入基因组的位置和影响的基因,因此许多的拷贝数变异被分类为“临床意义未知的拷贝数重复”,目前临床上还没有一种可应用于检测拷贝数增加断裂位点和定位信息的技术,以帮助判断这些拷贝数重复的致病性。
目前已经被广泛接受的mate-pair和跳跃文库(jumpinglibrary)等高通量测序文库构建技术,已经可以成功地检测全基因组范围内的平衡性染色体结构变异,但这些技术仍然存在一些严重的缺陷,如步骤繁琐、成本较高,起始dna需要量大,不利于常规应用。近年来高通量测序技术(next-generationsequencing,ngs)的发展,为我们进一步研究这些原来无法明确解释的拷贝数变异提供了新的途径。所以,基于高通量测序技术建立一套用于检测拷贝数重复片段断裂位点和定位信息,帮助临床判断拷贝数重复致病性的检测技术,对于拷贝数变异检测的临床诊断应用有着重要的意义。
技术实现要素:
现有的染色体拷贝数重复的致病性较难评估,目前的检测技术均无法检测拷贝数重复片段的断裂位点和定位信息,从而无法分类为串联重复或插入重复,评估染色体重复片段对染色体上其他基因的影响及重复片段的致病性。本发明基于高通量测序技术,开发了一种靶向捕获建库技术,用于特异性扩增拷贝数重复断裂位点附近区域的序列,在获得测序数据后通过生物信息学分析得到拷贝数重复片段的所在位置,可帮助临床更加准确地判断拷贝数重复片段的致病性,提高患者的诊断效率。
本发明的目的是针对现有技术中的不足,提供一种检测染色体重复片段断裂位点和定位信息的方法。
为实现上述目的,本发明采取的技术方案是:
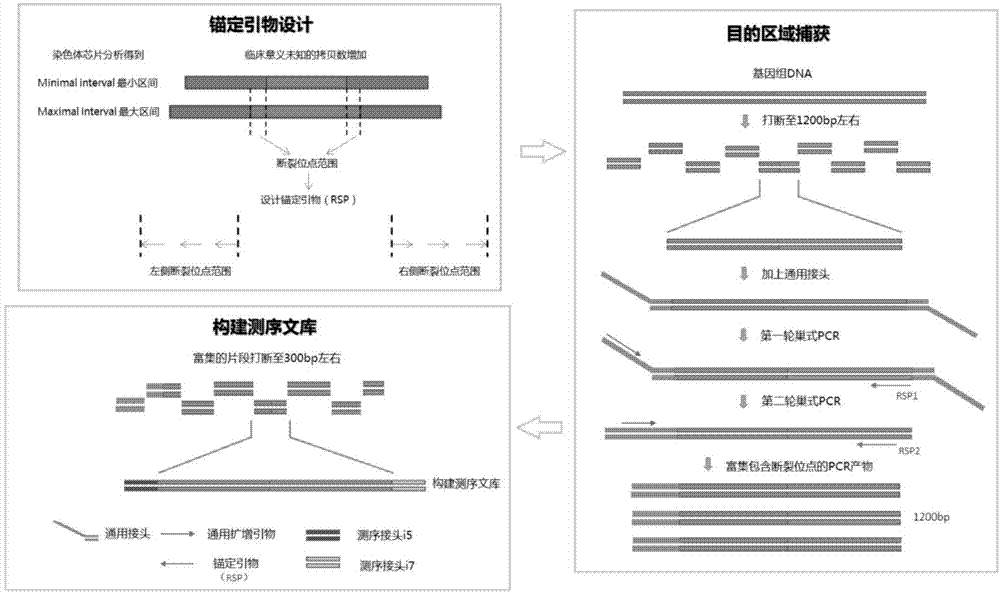
一种检测染色体重复片段断裂位点和定位信息的方法,在染色体芯片分析得到拷贝数重复片段基础信息后,设计靶向捕获引物,特异性扩增拷贝数重复断裂位点附近区域的序列,靶向捕获断裂位点可能所在大致区域的序列,富集包含断裂位点的基因序列并构建文库进行高通量测序,通过生物信息学分析得到拷贝数重复片段的所在位置。
在上述方法中,作为一个优选方案,包括如下步骤:
(1)准备通用接头;
通用接头序列1:5’pccatctcatccctgcgtgtctccgactcagctggcg*t
通用接头序列2:5’cgccaggttccagtca3’
(2)设计断裂位点区域特异性引物;
(3)锚定引物靶向捕获;
a.利用超声破碎仪covariss220对样本进行处理,使基因组dna在处理后最大片段峰值为1200bp;b.末端修复和加a尾;c.连接通用接头;d.第一轮锚定巢式pcr;e.第二轮引物巢式pcr;f.混合所有锚定pcr产物作为捕获区域扩增子;g.将混合后的pcr产物破碎至适合文库构建的大小;
(4)对捕获的扩增子进行建库并测序,通过下游生物信息学分析得到断裂位点的具体位置和附近序列信息。
在上述方法中,作为一个优选方案,所述断裂位点区域特异性引物设计包括如下步骤:
(1):根据染色体芯片检测结果,导出断裂位点可能所在区域染色体坐标;
(2):利用primer3设计区域特异性引物,避开与测序平台接头序列冲突的序列;
(3):利用blat将区域特异性引物和全基因组进行匹配,确保引物的特异性;
(4):对两轮巢式pcr的区域特异性引物进行检查,保证相邻的引物不会互相影响;
(5):检查所有引物3’端12个碱基的特异性。
在上述方法中,作为一个优选方案,所述检测染色体重复片段断裂位点和定位信息方法具体如下:
(1)准备通用接头
通用接头序列1:5’pccatctcatccctgcgtgtctccgactcagctggcg*t
通用接头序列2:5’cgccaggttccagtca3’
通用接头序列1和2同时稀释到100pmol/l浓度,在一个pcr反应管分别加入50ul去离子水,以及100pmol/l的通用接头序列1和2各25ul;95℃2分钟,取出放置室温20分钟后,-20℃保存;
(2)设计断裂位点区域特异性引物
根据染色体芯片检测结果,导出断裂位点可能所在区域染色体坐标;
利用primer3设计区域特异性引物,避开与测序平台接头序列冲突的序列;
利用blat将区域特异性引物和全基因组进行匹配,确保引物的特异性;
对两轮巢式pcr的区域特异性引物进行检查,保证相邻的引物不会互相影响;
检查所有引物3’端12个碱基的特异性;
(3)锚定引物靶向捕获
a.利用超声破碎仪对样本进行处理,使基因组dna在处理后最大片段峰值为1200bp;agencourtampurexp磁珠纯化破碎到适当长度的样本dna,洗脱体积为27ul;
b.末端修复和加a尾
末端修复:破碎并纯化后dna25ul,nebendrepairbuffer10ul,nebendrepairenzymemix5ul,水60ul,总量为100ul;20℃30分钟后取出,agencourtampurexp磁珠纯化,洗脱体积为22ul;
加a尾:末端修复并纯化后dna20ul,nebdatailingbuffer5ul,klenow3ul,水22ul,总量50ul;37℃30分钟后取出,agencourtampurexp磁珠纯化,洗脱体积为53ul;nanodrop定量纯化后样本作为质控,如起始样本量为2ug,此时纯化后浓度约为20ng/ul;
c.连接通用接头
上步中纯化后样本50ul,10xligationbuffer10ul,通用接头0.5ul,t4dnaligase5ul,水34.5ul,总量为100ul;混匀后室温1小时,qiagenpcrpurificationkit纯化,洗脱体积为23ul;
d.第一轮锚定巢式pcr
反应体系为25ul;10xhighfidelitypcrbuffer2.5ul,50mgmgso41ul,10mmdntp0.5ul,pcr1引物0.5ul,锚定引物0.5ul,模板1ul,
pcr条件:94℃预变性2分钟;94℃变性30秒,58℃退火30秒,68℃延伸1分钟,25个循环;68℃延伸2分钟;4℃保存;
agencourtampurexp磁珠纯化,洗脱体积为15ul;
e.第二轮引物巢式pcr
反应体系为50ul;10xhighfidelitypcrbuffer5ul,50mgmgso42ul,10mmdntp1ul,pcr1引物1ul,锚定引物1ul,模板2ul,
pcr条件:94℃预变性2分钟;94℃变性30秒,58℃退火30秒,68℃延伸1分钟,25个循环;68℃延伸2分钟;4℃保存;
agencourtampurexp磁珠纯化,洗脱体积为22ul;nanodrop定量纯化后pcr产物作为质控,浓度应在90ng/ul左右;
f.混合所有锚定pcr产物作为捕获区域扩增子
跟读naonodrop对每个pcr产物纯化后浓度,等比例混合所有pcr产物;
g.将混合后的pcr产物破碎至适合文库构建的大小
选用microtubeafafibersnap-cap处理样本,容量为130ul;程序设置为:peakincidentpower(w):50;dutyfactor:20%;cyclesperburst:200;treatmenttime(s):75;使基因组dna在处理后最大片段峰值为300bp;agencourtampurexp磁珠纯化样本dna,洗脱体积为45ul;
(4)对捕获的扩增子进行建库并测序,通过下游生物信息学分析得到断裂位点的具体位置和附近序列信息。
本发明优点在于:
1、建立了一种新的高通量测序文库构建方法来检测拷贝数重复的断裂位点及定位信息,该方法在染色体芯片得到拷贝数重复片段基础信息后,靶向捕获断裂位点可能所在大致区域的序列,通过成熟的生物信息学分析未知来源的序列以判断其定位。
2、不需要获得基因组上其他不相关区域的序列信息,就能富集包含断裂位点的基因序列并构建文库进行高通量测序,从而更加高效、快速、低成本地分析得到这些临床意义未知的拷贝数增加的具体断裂位点以及定位信息。
3、本发明将对拷贝数变异相关疾病的遗传诊断和咨询起到重要的作用。
附图说明
附图1为本发明操作流程示意图。
附图2为靶向捕获结果。
附图3为7号染色体重复患者断裂位点分析结果。
具体实施方式
下面结合具体实施方式,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。此外应理解,在阅读了本发明记载的内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
实施例
一、材料
dnaligationkit和t4dnaligase,购自宝日医生物技术(北京)有限公司,货号分别为:6022和2011a;pcr扩增试剂platinumtmtaqdnapolymerasehighfidelity购自美国thermofisherscientific公司,货号11304011;高通量测序文库建库试剂
二、方法
根据染色体芯片检测提供的信息设计靶向捕获引物
1.准备通用接头
通用接头序列1:5’pccatctcatccctgcgtgtctccgactcagctggcg*t
通用接头序列2:5’cgccaggttccagtca3’
通用接头序列1和2同时稀释到100pmol/l浓度,在一个pcr反应管分别加入50ul去离子水,以及100pmol/l的通用接头序列1和2各25ul。95℃2分钟,取出放置室温约20分钟后,-20℃保存。
2.设计断裂位点区域特异性引物
区域特异性引物的设计包括5个主要步骤:
(1):根据染色体芯片检测结果,导出断裂位点可能所在区域染色体坐标。
(2):利用primer3设计区域特异性引物,避开与测序平台接头序列冲突的序列。
(3):利用blat将区域特异性引物和全基因组进行匹配,确保引物的特异性。
(4):对两轮巢式pcr的区域特异性引物进行检查,保证相邻的引物不会互相影响。
(5):检查所有引物3’端12个碱基的特异性。
3.锚定引物靶向捕获
a.利用超声破碎仪covariss220对样本进行处理
初始样本量建议>2ug(至少1ug),选用microtubeafafibersnap-cap处理样本,容量为130ul,程序设置为:peakincidentpower(w):50;dutyfactor:2%;cyclesperburst:200;treatmenttime(s):60,使基因组dna在处理后最大片段峰值在1200bp左右。agencourtampurexp磁珠纯化破碎到适当长度的样本dna,洗脱体积为27ul。
b.末端修复和加a尾
试剂盒为
末端修复:破碎并纯化后dna25ul,nebendrepairbuffer10ul,nebendrepairenzymemix5ul,水60ul,总量为100ul。20℃30分钟后取出,agencourtampurexp磁珠纯化,洗脱体积为22ul。
加a尾:末端修复并纯化后dna20ul,nebdatailingbuffer5ul,klenow3ul,水22ul,总量50ul。37℃30分钟后取出,agencourtampurexp磁珠纯化,洗脱体积为53ul。nanodrop定量纯化后样本作为质控,如起始样本量为2ug,此时纯化后浓度约为20ng/ul。
c.连接通用接头
上步中纯化后样本50ul,10xligationbuffer10ul,通用接头(15um)0.5ul,t4dnaligase5ul,水34.5ul,总量为100ul。混匀后室温1小时,qiagenpcrpurificationkit纯化,洗脱体积为23ul(根据不同样本所涉及的锚定引物数量适当调整洗脱体积)。
d.第一轮锚定巢式pcr
反应体系为25ul。10xhighfidelitypcrbuffer2.5ul,50mgmgso41ul,10mmdntp0.5ul,pcr1引物0.5ul,锚定引物0.5ul,模板1ul,
pcr条件:94℃预变性2分钟;94℃变性30秒,58℃退火30秒,68℃延伸1分钟,25个循环;68℃延伸2分钟;4℃保存。
agencourtampurexp磁珠纯化,洗脱体积为15ul。
e.第二轮引物巢式pcr
反应体系为50ul。10xhighfidelitypcrbuffer5ul,50mgmgso42ul,10mmdntp1ul,pcr1引物1ul,锚定引物1ul,模板2ul,
pcr条件:94℃预变性2分钟;94℃变性30秒,58℃退火30秒,68℃延伸1分钟,25个循环;68℃延伸2分钟;4℃保存。
agencourtampurexp磁珠纯化,洗脱体积为22ul。nanodrop定量纯化后pcr产物作为质控,浓度应在90ng/ul左右。
f.混合所有锚定pcr产物作为捕获区域扩增子
跟读naonodrop对每个pcr产物纯化后浓度,等比例混合所有pcr产物。
g.将混合后的pcr产物破碎至适合文库构建的大小
选用microtubeafafibersnap-cap处理样本,容量为130ul。程序设置为:peakincidentpower(w):50;dutyfactor:20%;cyclesperburst:200;treatmenttime(s):75。以对照样本在相同条件下的破碎片段大小为参考进行参数调整,适当增加或减少处理时间(treatmenttime),使基因组dna在处理后最大片段峰值在300bp左右。agencourtampurexp磁珠纯化对破碎到适当长度的样本dna,洗脱体积为45ul。
具体操作流程如图1所示。
对捕获的扩增子进行建库并测序及生物信息学分析
建库试剂盒为:
三、结果
靶向捕获效率。由于基因组打断的过程是随机的,打断后的片段在街上通用接头之后,进行pcr反应时,序列特异性引物结合的位置和通用接头的位置从小到大呈递减式的分布,因此,每个pcr反应的产物大小并不均已,距离通用接头越远,pcr反应可以延伸到的可能性越小,表现在高通量测序上时就是测序深度降低。通过多次条件的摸索和尝试,我们发现,将基因组打断至1200bp左右,并控制pcr条件在相应范围内,可以保证锚定pcr最低覆盖度(>500x)仍能够满足捕获跨越断裂位点的要求(见图2)。
实施例27号染色体重复患者断裂位点分析
本发明所建立的t-abc技术为特异性检测方法,针对不同患者必须设计不同的靶向捕获引物,对目的区域富集后进行测序及分析,这里以一例7号染色体上348kb的重复为例,展示检测结果。通过在重复片段两侧的断裂位点附近设计锚定引物,扩增可能包含断裂位点的序列。测序数据分析后,得到跨越染色体重复片段5’端的断裂位点的测序读长,分析不同起始位置的测序读长发现,该重复片段正向插入10号染色体上,具体位置为chr10:16774602(图3)。
本发明建立的断裂位点靶向锚定捕获技术(targetedanchoredbreakpointcapture,t-abc),通过对拷贝数重复片段断裂位点附近区域进行扩增富集,之后通过高通量测序及生物信息学分析,可快速、高效、低成本地得到拷贝数重复片段的具体断裂位点和定位信息,可以帮助对这些拷贝数变异的致病性进行评估。因此,本发明对拷贝数变异相关疾病的遗传诊断和咨询起到重要的作用。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员,在不脱离本发明方法的前提下,还可以做出若干改进和补充,这些改进和补充也应视为本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!