链特异性检测亚硫酸氢盐转化的双链体的制作方法
本申请要求2017年3月24日提交的美国临时专利申请序列号62/476,234的权益,其公开通过引用全文纳入本文。
本发明在美国国立卫生研究院(nationalinstitutesofhealth)授予的ca62924下于政府资助下完成。政府对本发明拥有一定的权利。
本发明涉及核酸分析领域。具体地,其涉及灵敏度和准确性增强的核酸序列分析。
背景技术:
现在可以获得有关癌症基因改变的广泛知识,这为患者的管理开辟了新的机会(1-3)。这些机会中最重要的一些涉及“液体活检”,即评估血液和其他体液中从肿瘤细胞释放到这些液体中的突变dna模板分子。虽然液体活检潜在的价值早在二十多年前已被认识到(4-6),但是测序技术的最新进展使这种方法变得切实可行。例如,最近已经证明血液的液体活检可以检测患有早期结肠直肠癌的患者的最小量的疾病,从而提供可以显著影响其存活的证据(7)。其他研究已经证明可以在患有其他恶性肿瘤的患者的血液中检测到循环肿瘤dna(ctdna),也可以在其他体液如胰腺囊肿、子宫颈抹片和唾液中检测到ctdna(8-16)。
用于检测稀有突变的绝大多数现有技术采用数字方法,其中逐个评估每个模板分子,以确定它是野生型还是突变体(17)。数字化可以在孔(17)、在通过乳化或微流体形成的微小液滴(18,19)或在簇(20)中进行。这些方法中的最强大方法采用大规模平行测序来同时分析数亿个单独扩增的模板分子的整个序列(21)。然而,所有目前可用的测序仪器具有相对高的错误率,将许多核苷酸位置的灵敏度限制在100个野生型(wt)模板分子中一个突变体,即便使用具有最佳质量的dna模板(21)。临床样本的dna质量往往远低于最佳,使问题更加复杂。通过在测序前预处理dna以去除受损碱基(22、23)并通过生物信息学和统计学方法来增强测序后碱基识别(base-call)可以增强灵敏度(24,25)虽然可用于各种目的,但对于最具挑战性的应用通过这些改进可获得的灵敏度通常并不足够高,如液体活检,其可能需要在数千种wt分子中检测一种突变分子(9)。
提高灵敏度的另一个重要方法是使用“分子条码”,其中各模板与独特的鉴定序列(uid)共价连接。分子条码最初用于计算单个模板分子(26),但随后被纳入一种强大的方法,称为safeseqs,用于减少错误(27)。在纳入uid后,后续扩增步骤产生各uid连接的模板的多个拷贝。通过扩增产生的各子分子含有相同的uid,形成uid家族。为了被认为是一个真正的突变,被称之为超突变体(supermutant),uid家族的每个成员必需在每个查询位置具有相同的序列(27)。
将分子条码分配给模板dna分子有两种通用方法。一种方法用于使用一组基因座特异性引物的pcr扩增特定基因座,而另一种用于在扩增整个基因组之前连接衔接子,从而产生文库。pcr方法使用包含一段随机(n)碱基的引物来区分各单独的模板分子(外源条码)(27、28)。这种方法的优点是它适用于非常少量的dna,并且实际上唯一扩增的序列是所需的序列,减少了评估特定突变所需的测序量。缺点是在uid纳入循环期间引入一条链的错误将产生超突变体。因此,该方法仍将消除测序期间的错误,但不会消除pcr初始循环期间产生的错误。连接方法采用用于连接的衔接子中的随机序列(27-29),或者使用衔接子连接的随机剪切的模板dna的末端作为“内源性uid”(27、30)。虽然在用连接方法的pcr步骤中仍然引入错误,但其优点是可以从测序数据中鉴定出两条链(双链体测序(duplexsequencing))。将相同的互补突变纳入两条链的概率很低(突变概率的平方只出现在一条链中)。这种方法的缺点是它需要文库制备和捕获待查询的序列,这两种方法都不是高效的。
本领域一直需要以高效的方式灵敏且特异地测定序列变异。
发明概述
根据本发明的一个方面,提供了一种用于检测dna分子群中罕见突变的方法。用亚硫酸氢盐处理dna分子群以将dna分子中的胞嘧啶碱基转化为尿嘧啶碱基,形成转化的dna分子群。使用与分子条码连接的过量靶特异性扩增引物,将分子条码连接到转化的dna分子群的两条链上,形成扩增的条码化经转化dna分子群。将扩增的条码化经转化dna分子在扩增反应中扩增,以形成扩增的条码化经转化dna分子家族,其中共有相同分子条码的扩增的条码化经转化dna分子形成dna分子家族。对家族的多个成员进行测序反应,以获得所述家族所述多个成员的两条链的核苷酸序列。比较家族的多个成员的核苷酸序列,并鉴定其中>90%的成员含有所选突变的家族。比较扩增的条码化经转化dna分子两条互补链的核苷酸序列,并在两条互补链中鉴定所选突变。
根据本发明的另一方面,提供了一种用于同时检测正(plus)链和负(minus)链中cpg二核苷酸处甲基化的方法。用亚硫酸氢盐处理dna分子群以将dna分子中的胞嘧啶碱基转化为尿嘧啶碱基,形成转化的dna分子群。使用与分子条码连接的过量靶特异性扩增引物,将分子条码连接到转化的dna分子群的两条链上,形成扩增的、条码化经转化dna分子群。将扩增的条码化经转化dna分子在扩增反应中扩增,以形成扩增的条码化经转化dna分子家族,其中共有相同分子条码的扩增的条码化经转化dna分子形成dna分子家族。对家族的多个成员进行测序反应,以获得所述家族所述多个成员的两条链的核苷酸序列。比较家族的多个成员的核苷酸序列,并鉴定其中>90%的成员在cpg二核苷酸处包含选定的甲基化c的家族。比较扩增的条码化经转化dna分子两条互补链的核苷酸序列,并在两条互补链中鉴定在cpg二核苷酸处的甲基化c。
在本发明的另一个方面,提供了扩增引物,其包含选自下述组的序列:seqidno:1-32。
本发明的另一方面提供了包含一组或多组四种扩增引物的试剂盒。一组中的每个引物与亚硫酸氢盐转化的dna的双链体片段的四个末端之一互补
本发明的另一方面是用于检测dna分子群中多态性的方法。用亚硫酸氢盐处理dna分子群以将dna分子中的胞嘧啶碱基转化为尿嘧啶碱基,形成转化的dna分子群。使用与分子条码连接的过量靶特异性扩增引物,将分子条码连接到转化的dna分子群的两条链上,形成扩增的条码化经转化dna分子群。将扩增的条码化经转化dna分子在扩增反应中扩增,以形成扩增的条码化经转化dna分子家族,其中共有相同分子条码的扩增的条码化经转化dna分子形成dna分子家族。对家族的多个成员进行测序反应,以获得所述家族所述多个成员的两条链的核苷酸序列。比较家族的多个成员的核苷酸序列,并鉴定其中>90%的成员含有所选多态性的家族。比较扩增的条码化经转化dna分子两条互补链的核苷酸序列,并在两条互补链中鉴定所选多态性。
阅读本说明书后,对于本领域技术人员显而易见的是本发明的这些和其他方面提供了用于灵敏且特异性地分析dna变异和修饰的技术和工具。
附图说明
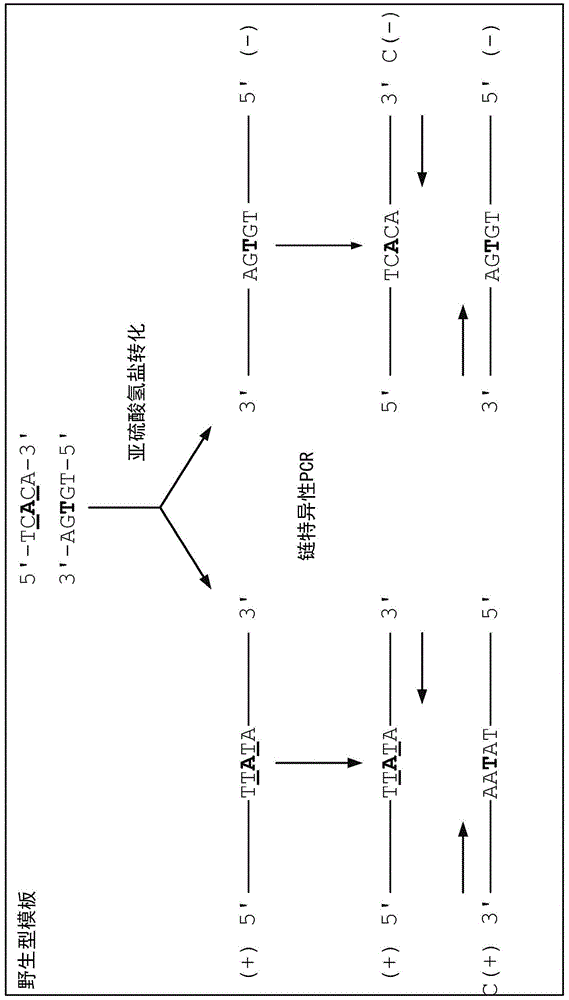
图1a-1b.biseqs方法概述。亚硫酸氢盐转化在各链的独特位置产生c>t转变。用是扩增子并具有链特异性的引物扩增(+)和(-)链允许靶向扩增和添加分子条码。分析两条链允许在第一pcr循环中产生的pcr错误显著地减少,因为在两条链上相同基因组位置处极不可能产生互补突变。野生型序列的转化和扩增示于图a,而a>c颠换的转化和扩增示于图b。
图2a-2c.biseqs显著降低扩增的基因座上单碱基取代突变的突变等位基因频率(maf)。所有扩增子上每个位置突变的maf(图2a)。所有扩增子上每个位置的超突变体的maf(图2b)。所有扩增子上每个位置的sdm的maf(图2c)。
图3.biseqs保持基于pcr分子条码化固有的灵敏度。将突变dna以0.20%或0.02%靶突变等位基因频率掺入正常dna中,并通过标准ngs、分子条码和biseqs评估测序数据。
图4a-4b.(图s1)未甲基化(图4a)和甲基化(图4b)基因座处biseqs平台的详细示意图。通过亚硫酸氢盐转化将未甲基化的c转化为t(步骤i),并且基于链特异性pcr的分子条码化将独特的标识符添加到分子的末端(步骤ii)。样品条码化(步骤iii)扩增分子条码化的dna,然后进行dna测序和分析(步骤iv),这允许序列与两条参照序列比对,一条针对(+)链而另一条针对(-)链。通用扩增引物允许指数扩增所有条码模板,而不管uid序列如何。移植序列代表illuminamiseq平台上所有配对末端读数所需的全长p5和p7序列。
图5.(图s2)针对8个基因组基因座制备的biseqs扩增子的代表性示例。引物长度的差异通常在一条链上产生更长的产物,允许容易地区分两条链的等摩尔扩增。
图6a-6c.(图s3)biseqs显著减少单碱基取代突变的数量。所有扩增子上每个位置突变的数量(图6a)。所有扩增子上每个位置超突变体的数量(图6b)。所有扩增子上每个位置sdm的数量(图6c)。注意图a和c中y轴比例相差三个数量级。
图7a-7c.(图s4)biseqs显著减少扩增的基因座上插入缺失突变的数量。所有扩增子上每个位置突变的数量(图7a)。所有扩增子上每个位置超突变体的数量(图7b)。所有扩增子上每个位置sdm的数量(图7c)。
图8a-8c.(图s5)biseqs显著降低扩增的基因座上插入缺失突变的突变等位基因频率(maf)。所有扩增子上每个位置突变体的maf(图8a)。所有扩增子上每个位置的超突变体的maf(图8b)。所有扩增子上每个位置的sdm的maf(图8c)。
图9.(图s6)标称突变等位基因分数(mutantallelefraction,maf)0.20%和0.02%的所有其他扩增子上biseqs的灵敏度。通过以与ngs和基于分子条码的测序类似的频率检测突变,biseqs保持基于pcr的分子条码化固有的灵敏度。
图10a-10b.(图s7)信噪比图显示了biseqs允许稳健检测双链突变。(图10a)nras中的c>a颠换的maf为0.20%。(图10b)tp53中的t>缺失的maf为0.20%。使用biseqs方法,可以极大超过其他位置的背景检测到预期位置的实际突变。
发明详述
发明人开发了一种结合了上述基于pcr和基于连接的方法的优点的方法。该方法利用亚硫酸氢盐处理可以高效地将dna中的dc碱基转化为u碱基这一事实的优势。这种转化使得两条dna链可以区分,并且之前用于区分拷贝自两种可能的dna模板链中各条的rna转录本(31)。亚硫酸氢盐转化也被广泛用于区分未转化为t碱基的甲基化c-残基与未甲基化的c碱基,从而阐明表观遗传变化(32)。也已经证明,dc碱基可以部分转化为t碱基,从而使每个单独的模板dna分子可以通过其c到t变化的独特模式与其他模板dna分子区分开,从而产生类似于藉由外部添加uid可以实现的内在条码(33)。其中所有c碱基已完全转化为t碱基的dna可用作pcr模板,其具有与外源条码连接的特殊设计的引物。这允许以可靠的方式在两条链上评估单个突变(双链体测序),而不产生文库并且具有相对少量的测序读数。
检测临床样本中的罕见突变对于癌症的筛查、诊断和治疗至关重要。虽然下一代测序大大提高了检测突变的灵敏度,但这些平台相对较高的错误率限制了它们的整体临床效用。消除测序假象(sequencingartifact)可以促进早期癌症的检测,并提供针对肿瘤遗传概况定制的改进的治疗建议。biseqs是一种基于亚硫酸氢盐转化的测序方法,可用于稀有突变的链特异性检测和定量。biseqs消除了三种常见类型突变中几乎所有的测序假象,从而大大提高了诊断分析的信噪比。
biseqs中使用两种类型的条码。分子条码用于在条码化和扩增之前鉴定原始样品中的各个模板分子。各单独的模板分子将具有独特的分子条码。样品条码用于鉴定反应样品或原始样品的等分试样;反应样品或等分试样中的所有模板分子共有鉴定反应样品或等分试样的条码。条码可以是,例如,随机产生的核苷酸延伸物(runs)或有意选择的核苷酸延伸物。特别是为了连接分子条码,反应混合物中单独的分子条码的数量将超过模板分子的数量。在组成本申请一部分的序列表中,条码表示为一串n。
亚硫酸氢盐转化接近于完全转化。因此,旨在扩增亚硫酸氢盐转化的双链体寡核苷酸的引物利用与转化的序列的互补性。引物被设计成以至少四个的组使用,因此原始双链体模板的两条链被扩增、测序和鉴定。
扩增条码化的序列产生类似条码化的模板的家族。各家族共有一种分子条码,表示其源自单个模板分子。对扩增的模板群(包括家族的多个成员)进行测序,允许比较单个家族多个成员的核苷酸序列并评估含有特定突变的家族成员的分数(fraction)。高分数诸如大于50、60、70、80、90或95%的家族具有特定突变表明突变在扩增之前存在于原始样品中。然而,一些鉴定出的突变可能仍然是由于体外酶促错误而在加工过程中导入的突变。通过比较由两个互补链家族获得的序列,可以进一步减少检测因这种错误而引起的突变。要求突变存在于由两条链产生的家族上显著减少了假象性表观突变。
核酸片段可任选地使用随机片段形成技术获得,如机械剪切、超声处理或使核酸经受其他物理或化学应激。片段可能并不是严格随机的,因为一些位点可能比其他位点对应激更易感。随机或特异性片段化的内切核酸酶也可用于产生片段。片段的大小可能变化,但理想的是在30至5,000个碱基对、100至2,000个碱基对、150至1,000个碱基对之间,或在具有这些端点的不同组合的范围内。核酸可以是例如rna或dna。也可以使用rna或dna的修饰形式。
分子条码与分析物核酸片段的连接可以通过本领域已知的任何方法进行,包括酶促的、化学的或生物学的。一种方法采用聚合酶链式反应。另一种方法使用连接酶。例如,酶可以是哺乳动物或细菌的。在使用其他酶如t4dna聚合酶的klenow片段连接之前,可以修复片段的末端。可用于连接的其他酶是其他聚合酶。可以将分子条码添加到片段的一端或两端,优选添加到两端。分子条码可以包含在核酸分子内,该核酸分子包含用于其他预期功能的其他区域。例如,可以添加通用引发位点以允许稍后的扩增。另一个额外的位点可以是与分析物核酸中特定区域或基因互补的区域。例如,分子条码的长度可以是2至4,000、100至1000、4至400个碱基。
可以使用随机添加核苷酸来制备分子条码,以形成用作标识符的短序列。在添加的各位置处,可以使用来自四种脱氧核糖核苷酸之一的选择。或者,可以使用三种、两种或一种脱氧核糖核苷酸之一的选择。因此,分子条码在某些位置可以是完全随机的、部分随机的或非随机的。制备分子条码的另一种方式利用组装在芯片上的预定核苷酸。在这种制备方式中,复杂性以有计划的方式达到。
用于添加外源性分子条码的聚合酶链式反应循环是指双链分子的热变性,杂交第一引物与所得单链,延伸该引物以形成与原始单链杂交的新的第二链。第二个循环是指使来自原始单链的新的第二链变性,杂交第二引物与新的第二链,和延伸第二引物以形成新的第三链,与新的第二链杂交。可能需要多个循环来提高效率,例如,当分析物被稀释或存在抑制物时。
可以根据已知技术进行含有分子条码的片段的扩增,以生成片段家族。可以使用聚合酶链式反应。为方便起见,也可以使用其他扩增方法。可以使用反向pcr,也可以使用滚环扩增。片段的扩增通常使用与引发位点互补的引物进行,且与分子条码同时与片段连接。引发位点位于分子条码的远端,因此扩增包括分子条码。扩增形成片段的家族,家族的各成员共有相同的分子条码。因为分子条码的多样性大大超过了片段的多样性,所以各家族应当源自分析物中的单个片段分子。用于扩增的引物可以经化学修饰,以使它们对外切核酸酶更具抗性。一种这样的修饰是在一个或多个3'核苷酸之间使用硫代磷酸酯连接。另一种采用硼烷磷酸酯。另外,lna(锁核酸)碱基可用于引物中;它们可以增加含有它们的寡核苷酸的tm。
对家庭成员进行测序和比较,以确定家庭中的任何分歧。测序优选在大规模平行测序平台上进行,这些平台中的许多可商购获得。如果测序平台需要“移植”序列,即连接到测序装置,那么可以在添加分子条码期间或单独添加这种序列。移植序列可以是分子条码化的引物、通用引物、基因靶特异性引物、用于制备家族的扩增引物、样品条码化引物或分开的一部分。冗余测序指单个家族的多个成员的测序。
可以设置阈值用于识别分析物中的突变。如果“突变”出现在家族的所有成员中,那么它来自分析物。如果其出现在少于所有成员中,那么其可能是在分析期间被导入的。可以设定用于识别突变的阈值为,例如,1%、5%、10%、20%、30%、40%、50%、60%、70%、80%、90%、95%、97%、98%或100%。阈值将根据被测序的家庭成员的数量以及特定的目的和情况来设置。
根据本文公开的方法检测、监测和/或分析的突变可在癌症驱动基因或癌症乘客基因(passengergene)中。它们可能在其他致病或疾病相关基因中。它们可能只是没有已知功能结果的种系多态性或体细胞突变。可以分析的驱动基因的示例包括nras、pik3r1、pten、rnf43和tp53。但这些方法绝不仅限于这些基因。相似地,该方法可用于检测双链核酸分子两条链上的甲基化。
可以用于该方法扩增步骤的聚合酶可以是具有特定扩增所需特征的任何聚合酶。我们在实施例中使用了赛默飞世尔公司(thermofisher)phusionuhotstarttm聚合酶,但我们还测试了其他聚合酶和酶的组合。它们包括恩佐公司(enzo)ampigenehstaqtm聚合酶;伯乐公司(biorad)itaqhotstartdnapolymerasetm;赛默飞世尔公司(thermofisher)phusionhotstartiidnatm聚合酶;和西格玛奥德里奇公司(sigmaaldrich)faststarttmdna聚合酶以及所述这些聚合酶的组合。
扩增引物可以单独包装或组合包装。它们可以是液体或干燥的。包装或试剂盒可任选地包含关于引物的分析信息和/或用于实施根据本发明的方法的说明书。试剂盒可以任选地包含其他组分,如聚合酶、扩增缓冲液、反应容器或其他工具,以便于实施该方法。
实施例中所述结果表明,biseqs可以以高度灵敏和特异性的方式准确地定量稀有突变。我们设想其主要用途将是监测原发肿瘤已被测序的癌症患者。已经表明,液体活检可以用于此目的,并且可以准确地识别临床缓解但注定要复发的患者(7、11、44)。许多这样的患者,特别是当他们的疾病残余负担很小并且因此最有可能通过辅助治疗治愈时(45),在10ml血浆中仅具有一个或两个突变dna分子。在这种情况下,可以有效地使用所有模板分子同时保持高特异性的技术,像biseqs,可以证明特别有用的。
biseqs的一个缺点是它不能应用于大多数转换型突变,因为c到u的亚硫酸氢盐转化所引起的模糊性,模仿这种转变。虽然一条链仍然对biseqs易感,但是该技术的力量在于它能够检测两条链中的突变,因此它对这种突变的分子条码化没有任何优势。例如,kras密码子12、13和61中的单个碱基取代通常在结肠、直肠和胰腺腺癌中发生突变(46)。biseqs可用于分别定量38.7%、43.4%和47.6%的这些癌症中的kras突变(47)。在iarctp53数据库中编目的所有癌症和突变中,大约44%的所有突变(即sbs和插入缺失)适合biseqs分析(iarctp53数据库,r18)。
此外,取决于孵育时间和试剂浓度,亚硫酸氢盐处理可导致甲基化c碱基在极少数情况下转化为u(48)。用于biseqs的方案采用降低的孵育温度,这似乎使这种可能性最小化(48),但甲基化cpg位点处的序列异质性可能提高背景,并且此类位点不优选用于突变评估。
然而,对于监测中的液体活检,单个基因固有的局限性不是主要问题,因为在癌症的全基因组测序后普遍观察到几种不同的突变,包括颠换和插入缺失(1-3),并且任何鉴定的突变都可能原则上适用于这种临床情况。最近对3,281个癌症样本进行的一项研究强调了93%的癌症在至少一个驱动基因中具有至少有一个非同义突变(49)。虽然小插入缺失和点突变的平均数量在肿瘤类型中不同,但大多数的癌症至少有一个驱动基因突变应该适合biseqs分析(49)。值得注意的是,克隆的乘客基因突变也可用于诊断评估(50)。因为在几乎所有癌症中,乘客突变的数量是驱动基因突变的至少10倍,所以很可能绝大多数的癌症都会有可以通过biseqs评估的几种体细胞突变。例如,在一项对乳腺癌中检测到的1157个单碱基取代的研究中,我们计算出54.7%的取代适用于biseqs分析,此外还有7.4%包含插入或缺失突变的肿瘤,总共适用于62.1%的肿瘤(51)。
biseqs的作用在于其能够大幅减少背景错误的能力。因此,biseqs还可以补充筛选其他基因组改变,如结构变体(sv),用于罕见的等位基因检测和监测(52)。结构变体(sv)为癌症提供了可用于液体活检的精确特异性标志物(9、50)。简单的聚合酶错误不会产生结构变体,提供了优于单碱基取代作为诊断靶标的优势。另一方面,使用sv作为诊断标志物存在缺点。首先,对于其初始检测,sv检测需要肿瘤的全基因组测序,而不是肿瘤的靶向测序;而后者目前比前者便宜得多。其次,并且更重要的是,结构变体是“私人的”,即通常局限于一个或少数患者。为了被用作肿瘤标志物,特异性扩增易位连接的引物必须在患者的肿瘤上被设计和测试,以确保结构变体是体细胞的并且扩增子是特异性的。虽然这种方法在研究环境中是可行的,但在大规模环境中并不容易实现。相反,在许多独立的肿瘤中观察到驱动基因中的单个碱基取代和插入缺失,并且可以使用一小组“现成的”引物来评估大多数患者。例如,我们估计>98%患有结直肠癌的患者具有可以通过130个预先设计的引物对中的一个进行扩增而可检测的突变。
在将来,当必须分析转换型突变时,将a:tbp(而不是c:g)bp转化为其他bp的dna化学处理可能替代亚硫酸氢盐。未来研究的另一个途径是多重化,其允许在筛选情景中同时评估各种扩增子中的突变。这种多重化比正常情况更困难,因为必须为每个感兴趣的区域设计两个扩增子,同时在所有感兴趣的区域中实现每个扩增子均一的效率。
上述公开内容总体上描述了本发明。本文所公开的所有参考文献通过引用明确纳入本文。通过参考下述具体实施例可以获得更完整的理解,所述实施例仅出于说明的目的而提供,而非旨在限制本发明的范围。
实施例1
材料和方法
简言之,提取来自宏观解剖的福尔马林固定石蜡包埋(ffpe)肿瘤切片的dna,并用ezdna甲基化试剂盒(zymo研究公司(zymoresearch),目录号d5001)进行亚硫酸氢盐处理。使用含有独特标识符(uid)和扩增子特异性序列的定制引物扩增dna的两条链,并在illuminamiseq仪器上对所得产物进行测序。为了表征biseqs的特异性,对分离自一个正常组织的dna进行亚硫酸氢盐处理并通过biseqs管线(pipeline)处理以查询单碱基取代和插入缺失。为了表征biseqs的灵敏度,用来自正常wbc的dna稀释具有已知maf的宏观切除的肿瘤样品,以获得0.02%至0.20%的最终肿瘤细胞含量,经亚硫酸氢盐处理并通过biseqs管线处理。如下提供更多的细节。
人组织
将福尔马林固定石蜡包埋的(ffpe)肿瘤切片在解剖显微镜下进行宏观解剖,以确保肿瘤细胞性>30%。用凯杰公司(qiagen)ffpe试剂盒(凯杰公司,目录号56494)纯化dna。用来自正常wbc的dna稀释具有已知maf的肿瘤样品,以获得0.02%至0.20%的最终肿瘤细胞含量。为了精确定量正常dna样品和肿瘤的dna浓度,用揭示最终扩增子内正常的单核苷酸多态性的引物来扩增肿瘤和正常dna的各种混合物。然后使用ngs来定量各种测试混合物中肿瘤细胞的分数,然后将相同的混合物用作biseqs的模板dna,如下所述。所有组织均来自约翰霍普金斯医院的患者,并有约翰霍普金斯大学机构审查委员会(johnshopkinsinstitutionalreviewboard)的批准。
用于biseqs的纯化dna的亚硫酸氢盐处理和pcr扩增
在对各种市售可及的亚硫酸氢盐转化试剂盒进行大量测试后,我们选择ezdna甲基化试剂盒(zymo研究公司,目录号d5001)按照生产商推荐的方案对dna样品进行亚硫酸氢盐处理和脱磺酸盐纯化。将dna在10μl洗脱缓冲液中洗脱并储存在-20℃。针对扩增的基因座处dna双螺旋的各亚硫酸氢盐转化的链设计定制的hplc纯化的pcr引物(idt)(序列表)。相较于传统的pcr引物,定制引物更长,以针对亚硫酸氢盐转化的dna降低的序列复杂性。各正向引物含有5'末端孔条码扩增所必需的序列,随后是作为唯一标识符(uid)的一串14个随机核苷酸,以及3'末端的扩增子特异性引物序列(图4a和4b)。各反向引物含有5'末端孔条码扩增所必需的序列,随后是扩增子特异性引物序列。为了使亚硫酸氢盐转化的dna退火,重要的是替换各种野生型扩增子特异性引物序列中的特定核苷酸。t取代了正链正向引物中的c,而a取代了正链反向引物中的g。a取代了负链正向引物中的g,而t取代了负链反向引物中的c。
分子条码化pcr循环包括在25μl反应中的12.5μl的2xphusionuhotstartpcr主混合物(赛默飞世尔公司,目录号f533s),以及各种正向和反向引物的优化浓度,对于每孔总共四个引物,范围从0.125μm到4μm的各种正向和各种反向引物。使用下述循环条件:95℃持续3分钟的1个循环,95℃持续10秒、63℃持续2分钟和72℃持续2分钟的20个循环。
使用ampurexp(贝克曼库尔特公司(beckmancoulter),目录号a63881)去除用于uid分配的引物。将由uid循环产生的0.025%pcr产物用于孔条码化(wbc)循环。用于孔条码步骤的引物与之前所述的引物相同并且示于图4a和4b(28)。在25μl反应中进行wbc循环,所述25μl反应包含11.8μl的水(赛默飞世尔公司ultrapure,目录号10977-023),5μl的5xphusionhf缓冲液(赛默飞世尔公司,目录号f518l),0.5μl的10mmdntps(neb公司,目录号n0447l)和0.25μl的phusionhotstartiidna聚合酶(赛默飞世尔公司,目录号f549l)。使用下述循环条件:98℃持续2分钟的1个循环,98℃持续10秒、65℃持续2分钟和72℃持续2分钟的24个循环。
测序
使用illuminamiseq仪器对上述所有扩增子进行测序。用于各仪器的读数的总长度在79-130个碱基之间变化。将通过illuminacasavachastity过滤器的读数用于后续分析。
biseqs管线
使用safeseqs管线(28)处理高质量读数以生成比对数据,然后将这些数据组织成针对各biseqs分析的表格。这些表格各自包含:(i)链信息,(ii)孔条码和uid序列,(iii)列出与参照扩增子所有差异的信息,和(iv)各uid家族对应于相对于每个扩增子所有uid家族变化的流行度。为了确定正链和负链变化的组合是否构成双链突变体,将在特定基因组基因座处检测到的各种突变从下述方面进行比较:(i)样品类型,(ii)染色体,(iii)基因组位置,和(iv)突变类型。当(i)在正链和负链上都出现变化,以及(ii)当对应于+和-链的maf相差小于10倍时,将变化被称为真正的突变。
biseqs特异性的表征
为了表征biseqs的特异性,对分离自一个正常组织的dna进行亚硫酸氢盐处理并通过biseqs管线处理以查询单碱基取代和插入缺失。使用经针对插入缺失的608个碱基和8个扩增子的ngs进行分析,在正链上鉴定了907个独特突变并在负链上鉴定了958个独特突变,其最终适合通过biseqs进行分析。对于各扩增子的各条链,我们通过将含有>2个突变读数/uid(uid家族计数>2)的uid数或读取数分别除以总uid数或总读取数来计算突变等位基因频率(maf)。使用分子条码将读数分组到家族中,将独特突变数量减少到正链92个和负链上71个(数据未显示)。匹配正链和负链扩增子并对正链上观察到的突变比率相对负链上观察到的突变比率(反之亦然)施加小于10的过滤器后,鉴定了了四个突变(数据未显示)。sdm的数量被认为是对应突变的正链和负链上超突变体数量的最小值,因为这是可检测的双链超突变分子的极限数量。相似地,双链分子的总数被认为是正链或负链上全部uid的最小值,因为这是检测到的双链模板分子的极限数量。以标准ngs分别检测正链和负链上197和167个插入缺失。分子条码的使用将检测到的插入缺失的数量分别减少到正链和负链各6个和5个,而biseqs双链分析将插入缺失的数量减少到0。
实施例2
biseqs工作流程
biseqs的主要特征是同时检测经亚硫酸氢盐处理和分子条码化的dna模板正链和负链的突变。我们将ucsc定义的参照序列称为正(+)链,将其反向互补物称为负(-)链。在进行测序数据的专门生物信息学分析之前,可以进行三个简单的实验步骤(亚硫酸氢盐转化、分子条码化和样品条码化),如下所述(图1和图4a-b)。
步骤i:亚硫酸氢盐转化。在高温和低ph下用亚硫酸氢钠孵育dna使胞嘧啶脱氨基形成5,6-二氢胞嘧啶-6-磺酸盐(34)。随后在高ph下水解脱氨去除磺酸盐,产生尿嘧啶(35)。已经描述了该碱性反应的许多修饰并且主要用于区分胞嘧啶和5-甲基胞嘧啶(5-mc),后者对亚硫酸氢盐转化并不易感。除了将c转化为u外,亚硫酸氢盐处理使dna变性并且可以降解dna。虽然这种降解不限于亚硫酸氢盐处理的标准应用,但对于在转化前已经降解的临床样品中涉及突变检测的应用而言是至关重要的(36-38)。在当前的研究中,我们评估了转化dna的许多方法,并纯化了转化的链。使用材料和方法中所述试剂、条件和孵育时间获得了最佳结果。如图5所示,在这些条件下的处理不会抑制pcr产物的扩增,其大小可达285bp。这些产物的测序表明,平均而言,>99.8%的c碱基在两条链上被转化为t碱基(除了5'-cpg位点的c碱基,其对亚硫酸氢盐转化具有抗性,因为它们经甲基化或羟甲基化)。
步骤ii:分子条码化。亚硫酸氢盐处理的目标是创建一个区分两条dna链的代码。相较于用于标准化扩增dna的步骤,这使得需要采用特定步骤进行分子条码化的模板数量增加了一倍。首先,必须设计四个引物以扩增各感兴趣的区域,各链两个引物。其次,引物必须与dna的转化形式互补,强调完全转化的重要性——否则,一些模板分子将不会被扩增,因为它们不能与引物完全互补。第三,在我们采用的条件下,亚硫酸氢盐处理实际上将所有未经修饰的c残基转化为t,从而总体上降低了引物退火位点和扩增子的解链温度。因为必须等同地并且在相同的反应中扩增两条链,所以必须选择引物,使得相同的pcr循环条件可以用于以高度特异性方式扩增两条链。对于已经具有低c:g碱基对含量的区域,引物必须足够长以允许在相对高温的退火条件下进行特异性扩增。在不产生大量引物二聚体的情况下这被证明是困难的,并且为了克服这些挑战,我们评估了几种引物设计。最终,引物长度、位置、组成和c:g含量的变化允许对各属意靶区域的两条链进行特异性和稳健扩增。
亚硫酸氢盐转化的dna的扩增所面临的另一个问题是许多聚合酶不能有效复制含有尿嘧啶碱基的dna。我们测试了7种市售可及的聚合酶和各种反应条件以在使用四种引物时优化模板使用效率和扩增两条链的均一性(表1)。虽然ampigenehotstarttaq聚合酶和itaq聚合酶的组合扩增了最多数量的模板分子,但其缺乏3'→5'外切核酸酶活性被证明了对特异性有所限制,因为pcr期间的错误数量高得令人无法接受。最终,我们选择了phusionuhotstart聚合酶(一种具有3'→5'外切核酸酶活性的聚合酶),因该酶在保持灵敏度同时以最高特异性扩增含尿嘧啶模板。
步骤iii:样品条码化。大规模平行测序仪器的部分功能是它们可以用于一次分析多种样品。为了使biseqs具有这种能力,我们在纯化分子条码化的pcr产物后纳入了样品条码pcr循环(图4,步骤iii。)此外,在分子条码化步骤之前,将转化的样品dna分到pcr板的2-6个孔。然后为各孔分配不同的样品条码。这种分布有两个目的。首先,藉由浓缩的dna模板,可以提供独立复制具有小突变等位基因分数的突变。其次,藉由稀释的dna模板,如常常存在于临床样品诸如血浆(9)、尿液(39)和脑脊液(12)中的那样,提供了测试更多模板分子的机会,增加了鉴定突变模板的机会。
实施例3
biseqs数据处理管线
将高质量的碱基识别与亚硫酸氢盐转化的参照序列比对,并将比对的数据组织成各样品的表格,其中将各孔的每条链中观察到的各突变列在单独的行中。该表格中的列包括各突变的读出数、uid和超突变体(数据未显示)。超突变体被定义为uid家族中的突变,其中在该家族中>90%的家族成员含有该突变。例如,如果uid家族的所有三个成员都包含相同的突变,那么它被认为是超突变体。将超突变等位基因部分定义为超突变体数量除以单独孔中的uid数量。
比较正链和负链中的个体突变以确定在两条链中是否发现相同的超突变体。如果在两条链中发现突变,那么比较各条链中的超突等位基因分数。各条链上超突变等位基因分数提供了额外的特异性水平,因为如果在转化和扩增之前模板dna中存在突变碱基对,那么预期这些分数是相似的。鉴于pcr期间出现的突变相对罕见,在两条链中相同位置出现相同突变甚至将更为罕见。转化后,当两条链含有明显不同的核苷酸环境(context)时,尤其如此。如果各条链中超突变等位基因分数的差异<10倍,那么该突变被认为是超强突变体(super-dupermutant,sdm)。定义sdm等位基因分数为sdm数量除以包含最少uid的链中的uid数量。例如,如果sdm数量是10,而正链和负链中的uid数量分别是10,000和20,000,那么sdm等位基因分数将是0.1%(即10,000分之10)。
转化dna中的突变分析包括以下特征。在测序中注意到的c>t的转变可能是由于将c:gbp变为t:abp的单碱基取代突变或由于将一条链上的c亚硫酸氢盐转化成t所导致。鉴于这种模糊性,c到t的突变不能被认为是含有c的链中的超突变体,虽然在含有g的链中的那个位置上仍然可以看到超突变体。在双链dna中共存在6种可能的单碱基取代:c:gbp可以突变成a:t、g:c或t:abp之一,而a:tbp可以突变成c:g、g:c或t:a之一。在这6种单碱基取代中,所有都在至少一条链上导致超突变体,并且有4种在两条链上导致超突变体(即,sdm)。此外,可以在两条链上评估产生其中c被甲基化的cpg二核苷酸的转换。扩增序列内的所有插入或缺失都可以形成sdm。甲基化也引入复杂性,因为甲基化或羟甲基化的c碱基不通过亚硫酸氢盐处理转化为u碱基。biseqs管线在其分析数据时考虑到这一点,不假设任何特定的c是甲基化的或未甲基化的(或者每个未甲基化的c都通过亚硫酸氢盐处理转化为t)。相反,其考虑转化和甲基化的可能影响,并且仅将没有歧义的突变标记为超突变或sdm。在下表1中提供了在任一条链上所有可能的单碱基取代的列表,其位于三联体环境内并突变的碱基在中间。
对于各单碱基替换,此表中还提供了biseqs鉴定sdm的能力。一般而言,所有的颠换、所有插入和缺失和一小亚类转换都可以被明确地评分为sdm(表1)。因为biseqs的能力在于sdm,所以以下仅考虑在两条链中可判断的突变。
实施例4
biseqs增加了突变识别的特异性
我们选择了原型癌驱动基因中的8个扩增子来评估biseqs的表现。对于8个扩增子中的每一个,合成针对每条链的2个正向引物和2个反向引物,并使用上述以及材料和方法中所述原则进行测试。对于所有扩增子,通过聚丙烯酰胺凝胶分析判断,发现每条链的至少一个引物对能够高效地特异性扩增预期的链(图5)。这些引物的序列列于序列表中。
对于8个扩增子中的每一个,我们比较了biseqs与常规下一代测序(ngs)和分子条码辅助测序(即safeseqs)的特异性。我们只考虑了可以在两条链中识别的潜在突变,如上所述。这些扩增子总共有608bp,可能总共产生1550个单碱基取代。在这1550个潜在的sbs中,1252个(80.8%)可以被评分为sdm;其余部分是由于上述原因而无法评估的转变。在测序数据中可以观察到各位置也有许多可能的插入缺失,所有这些插入缺失可以被评分为sdm。
在实际实验中,由于亚硫酸氢盐转化,我们可以区分用作测序仪器中模板的链。鉴于此,实际上有2504个突变(bp数量的2倍)可以对常规和分子条码辅助测序进行评分。对于这些2504个潜在sbs,1865(总可能突变的74.5%)实际上是在常规测序中观察到的(25),突出显示观察到的相对大量的误差,除非应用safeseqs或biseqs进行误差校正(数据未显示)。对于观察到的突变数量方面,两条链之间没有可辨别的差异,分别在正链和负链上观察到907和958个突变。常规ngs也观察到298个小插入或缺失。
将分子条码化方法应用于这些数据显著地减少了突变的数量,如图6a和6b的比较所示(注意,图6b中的y轴标度降低了两个数量级)。这种减少最相关的量度是比较应用分子条码化之前和之后的突变等位基因频率(maf)。在应用分子条码化之前,正链中sbs的中值突变等位基因频率(maf)为0.0233%(平均值0.0720%,95%ci为0.0627%至0.0813%;图2a-c)。负链中与之相似:中值为0.0185%,平均值为0.0751%,95%ci为0.0643%至0.0859%。如图2b所示,在分子条码化后,正链中的maf减少了8倍,中值为0.0000%,平均值为0.0091%(95%ci为0.0062%至0.0119%;p<10-12,配对双尾斯氏t检验)。需注意的是,分子条码化后的maf是超突变等位基因频率(smaf)的量度,但为简单起见,在图2b中标记为maf。通过分子条码化,负链的maf减少了9倍(中值为0.0000%,平均值为0.0080%,95%ci为0.0047%至0.0113%;p<10-12,配对双尾斯氏t检验)。safeseqs实现的减少幅度与对未经亚硫酸氢盐处理的天然dna的实验预期一致(27)。
将biseqs应用于这些数据可以进一步显著减少误差。在所有8个测序的扩增子中仅观察到4个sdm,这与分别未经分子条码化和经分子条码化的1865和163个突变相反(图6;需注意的是图6c的y轴相较于图6b又减少了一个数量级)。这反映在maf中,如图2c所示,通过biseqs的maf与ngs相比减少了1217倍,而与分子条码相比减少了141倍(中值为0.0000%,平均值为0.0001%,95%ci为0.0000%至0.0001%;p<10-12,配对双尾斯氏t检验)。
biseqs还减少了插入缺失处的误差:在8个扩增子中观察到364个突变体,11个超突变体和0个sdm(图7和8)。因此,maf的平均值由使用ngs的0.0041%降至使用分子条码化的0.0011%至使用bisqs的0.0000%(对于与针对正链的分子条码化比较的ngs,p<1.2x10-6,对于与针对负链的分子条码化比较的ngs,p<7.5x10-4,对于与biseqs比较的分子条码化,p<1.3x10-2)。
实施例5
biseqs的灵敏度
大规模平行测序允许同时评估数以亿计的扩增子,获得针对扩增子内任何碱基的>10亿个wt模板中1个突变的理论灵敏度。临床样品中的实际灵敏度仅受输入dna的量和特异性的限制。在许多类型的液体活检样品中,如来自血浆、胰腺囊肿、csf和尿液的活检样品,可用的总dna通常<33ng(7、9、12、39)。因此,0.01%的灵敏度足以检测可能存在于这类样品中的33ng人dna所包含的约10,000个模板之中的一个或两个突变体分子。这种检测的可靠性受到生物学和技术特异性的限制,其中所查询的突变必需要以比用于与肿瘤比较的正常对照样品中低非常多的频率存在。虽然可能无法规避可能导致正常样本中突变的生物学问题(40),但是可以通过方法进步如biseqs来解决和克服技术问题。
为了解决biseqs的灵敏度,我们评估了在上述8个扩增子内含有10个双链突变(如果对各链分开计数,那么有20个突变)的肿瘤样品(数据未显示)。通过ngs定义各肿瘤样品中的突变比例。我们使用来自这些肿瘤的dna来产生液体活检样品的特征,其中来自肿瘤细胞的少量dna与来自患者正常细胞的更大量的dna混合。更具体地,我们用正常白细胞稀释该肿瘤dna以获得0.02%和0.20%的次要等位基因分数,然后使用亚硫酸氢盐处理来转化混合物。在将输入dna保持在各孔5,000个模板分子的所有情况下,当用标准ngs、用分子条码或用biseqs分析时,我们确定了各肿瘤来源的突变的突变等位基因分数,并在6个孔中进行各个实验。我们发现这3种分析方法中各自产生了与对稀释物预期的类似突变等位基因分数(图3中的示例)。该实验证明,biseqs中各步骤的效率(从亚硫酸氢盐转化到扩增和测序步骤)都很高。
虽然扩增的效率因此总是足以检测突变体模板,但是正常对照的maf限制了对测序数据的解释。当信号噪声比(snr)(定义snr为肿瘤样品中的maf除以正常细胞中的maf)>10时,我们将突变体称为真正的突变。当考虑标准ngs或分子条码辅助的ngs时,我们对这两个链中的maf求平均值用于该计算。图3和图9显示了对0.20%和0.02%稀释物检测的maf。标准ngs仅对处于0.20%肿瘤细胞含量的8个突变中的2个产生>10的snr,而对处于0.02%肿瘤细胞含量的3个突变中的1个产生>10的snr。分子条码化对处于这些肿瘤细胞含量的10个突变中的7个产生>10的snr。与之相反,biseqs对处于所有测试的肿瘤细胞分数的所有10个突变产生>10的snr(图3、图9)对于nras和tp53中突变的maf的代表性snr图分别示于图10a和10b。
实施例6
biseqs同时检测两条链的甲基化状态
在亚硫酸氢盐处理期间,5'-cpg二核苷酸中甲基化的胞嘧啶碱基被保护免于转化为尿嘧啶,从而允许biseqs同时检测正链和负链的甲基化状态。虽然并非biseqs的主要目的,但这种鉴别对于分析低水平的甲基化而言是有用的,无论是用于基础研究还是临床目的。虽然亚硫酸氢盐处理和特别设计的引物过去通常被用于评估甲基化以用于各种临床目的(41-43),但是分子条码化与同时扩增两条链的组合在这种类型的分析中提供了前所未有的灵敏度。
为了证明biseqs同时区分两条链上甲基化状态的能力,我们评估了tp53基因的一个区域,该区域在hg19位置7,572,973至4含有已知甲基化的cpg。发现两条链上大于90%的uid在位于位置7,572,973正链的c处以及与位于位置7,572,974处负链上的与g相对的c处被甲基化。发现大于99.8%不在该扩增子内5'-cpg二核苷酸处的c残基被转化为t',这为解释甲基化的程度提供了必要的对照。然后,我们在正常wbc中在本研究中评估的所有8个扩增子中搜寻双链甲基化的证据。在608bp内有2个可以评估的5'-cpg残基。在它们当中,我们发现两条链上的cpg都被甲基化,甲基化的等位基因的分数为92.10%至96.10%(数据未显示)。
参考文献
所引用的每篇参考文献的公开内容明确地并入本文。
1.garrawayla和landeres(2013)来自癌症基因组的教训(lessonsfromthecancergenome).cell153(1):17-37.
2.strattonmr,campbellpj和futrealpa(2009)癌症基因组(thecancergenome).nature458(7239):719-724.
3.vogelsteinb,等(2013)癌症基因组特征(cancergenomelandscapes).science339(6127):1546-1558.
4.sidranskyd,等(1992)鉴定可治愈结肠直肠肿瘤患者粪便中的ras致癌基因突变(identificationofrasoncogenemutationsinthestoolofpatientswithcurablecolorectaltumors).science256(5053):102-105.
5.sidranskyd,等(1991)鉴定膀胱癌和尿液样本中的p53基因突变(identificationofp53genemutationsinbladdercancersandurinesamples).science252(5006):706-709.
6.hrubanrh,vanderrietp,erozanys和sidranskyd(1994)简报:膀胱癌的分子生物学和早期检测-huberthhumphrey例(briefreport:molecularbiologyandtheearlydetectionofcarcinomaofthebladder--thecaseofhuberth)..nengljmed330(18):1276-1278.
7.tiej,等(2016)循环肿瘤dna分析检测微小残留疾病并预测ii期结肠癌患者的复发(circulatingtumordnaanalysisdetectsminimalresidualdiseaseandpredictsrecurrenceinpatientswithstageiicoloncancer).scitranslmed8(346):346ra392.
8.dawsonsj,等(2013)分析循环肿瘤dna以监测转移性乳腺癌(analysisofcirculatingtumordnatomonitormetastaticbreastcancer).nengljmed368(13):1199-1209.
9.bettegowdac,等早期和晚期人类恶性肿瘤中循环肿瘤dna的检测(detectionofcirculatingtumordnainearly-andlate-stagehumanmalignancies).scitranslmed6(224):224ra224.
10.kindei,等(2013)评估来自巴氏测试的dna以检测卵巢和子宫内膜癌(evaluationofdnafromthepapanicolaoutesttodetectovarianandendometrialcancers).scitranslmed5(167):167ra164.
11.wangy,等(2015)检测头颈部鳞状细胞癌患者的唾液和血浆中的体细胞突变和hpv(detectionofsomaticmutationsandhpvinthesalivaandplasmaofpatientswithheadandnecksquamouscellcarcinomas).scitranslmed7(293):293ra104.
12.wangy,等(2015)检测患有脑和脊髓原发性肿瘤患者的脑脊液中的肿瘤来源的dna(detectionoftumor-deriveddnaincerebrospinalfluidofpatientswithprimarytumorsofthebrainandspinalcord).procnatlacadsciusa112(31):9704-9709.
13.wangy,等(2016)卵巢囊肿液中肿瘤dna的诊断潜力(diagnosticpotentialoftumordnafromovariancystfluid).elife5.
14.springers,等(2015)分子标记和临床特征的组合改善了胰腺囊肿的分类(acombinationofmolecularmarkersandclinicalfeaturesimprovetheclassificationofpancreaticcysts).gastroenterology149(6):1501-1510.
15.forshewt,等(2012)通过血浆dna的靶向深度测序无创识别和监测癌症突变(noninvasiveidentificationandmonitoringofcancermutationsbytargeteddeepsequencingofplasmadna).scitranslmed4(136):136ra168.
16.demattos-arrudal和caldasc(2016)无细胞循环肿瘤dna作为乳腺癌的液体活检(cell-freecirculatingtumourdnaasaliquidbiopsyinbreastcancer).moloncol10(3):464-474.
17.vogelsteinb和kinzlerkw(1999)数字pcr(digitalpcr).procnatlacadsciusa96(16):9236-9241.
18.dressmand,yanh,traversog,kinzlerkw和vogelsteinb(2003)转化单个dna分子为荧光磁性颗粒用于检测和计算遗传变异(transformingsinglednamoleculesintofluorescentmagneticparticlesfordetectionandenumerationofgeneticvariations).procnatlacadsciusa100(15):8817-8822.
19.marguliesm,等(2005)微制造的高密度皮升反应器中的基因组测序(genomesequencinginmicrofabricatedhigh-densitypicolitrereactors).nature437(7057):376-380.
20.mitrard和churchgm(1999)原位局部扩增和接触复制许多单独的dna分子(insitulocalizedamplificationandcontactreplicationofmanyindividualdnamolecules).nucleicacidsres27(24):e34.
21.shendurej和jih(2008)下一代dna测序(next-generationdnasequencing).natbiotechnol26(10):1135-1145.
22.doh和dobrovica(2012)通过用尿嘧啶-dna糖基化酶处理显著减少从福尔马林固定的癌症活组织检查中分离的dna中的序列假象(dramaticreductionofsequenceartefactsfromdnaisolatedfromformalin-fixedcancerbiopsiesbytreatmentwithuracil-dnaglycosylase).oncotarget3(5):546-558.
23.doh,wongsq,lij和dobrovica(2013)通过酶促耗尽含尿嘧啶模板减少福尔马林固定石蜡包埋的dna的基于扩增子的大规模平行测序中的序列假象(reducingsequenceartifactsinamplicon-basedmassivelyparallelsequencingofformalin-fixedparaffin-embeddeddnabyenzymaticdepletionofuracil-containingtemplates).clinchem59(9):1376-1383.
24.bratmansv,newmanam,alizadehaa和diehnm(2015)用capp-seq的超灵敏循环肿瘤dna检测的潜在临床效用(potentialclinicalutilityofultrasensitivecirculatingtumordnadetectionwithcapp-seq.expertrevmoldiagn15(6):715-719.
25.bokulichna,等(2013)质量过滤极大地改善了illumina扩增子测序的多样性估计(quality-filteringvastlyimprovesdiversityestimatesfromilluminaampliconsequencing).natmethods10(1):57-59.
26.sykespj,等(1992)通过使用有限稀释来定量pcr的靶标(quantitationoftargetsforpcrbyuseoflimitingdilution).biotechniques13(3):444-449.
27.kindei,wuj,papadopoulosn,kinzlerkw和vogelsteinb(2011)使用大规模平行测序检测和定量稀有突变(detectionandquantificationofraremutationswithmassivelyparallelsequencing).procnatlacadsciusa108(23):9530-9535.
28.casbonja,osbornerj,brenners和lichtensteincp(2011)一种用于与应用到下一代测序计数pcr模板分子的方法(amethodforcountingpcrtemplatemoleculeswithapplicationtonext-generationsequencing).nucleicacidsres39(12):e81.
29.schmittmw,等(2012)通过下一代测序检测超罕见突变(detectionofultra-raremutationsbynext-generationsequencing).procnatlacadsciusa109(36):14508-14513.
30.hoangml,等(2016)使用大规模平行测序法对正常人组织中罕见的体细胞突变进行全基因组定量(genome-widequantificationofraresomaticmutationsinnormalhumantissuesusingmassivelyparallelsequencing).procnatlacadsciusa113(35):9846-9851.
31.hey,vogelsteinb,velculescuve,papadopoulosn和kinzlerkw(2008)人类细胞的反义转录物组(theantisensetranscriptomesofhumancells).science322(5909):1855-1857.
32.frommerm,等(1992)实现单个dna链中5-甲基胞嘧啶残基的正展示的一种基因组测序方案(agenomicsequencingprotocolthatyieldsapositivedisplayof5-methylcytosineresiduesinindividualdnastrands).procnatlacadsciusa89(5):1827-1831.
33.levyd和wiglerm(2014)通过模板诱变促进序列计数和组装(facilitatedsequencecountingandassemblybytemplatemutagenesis).procnatlacadsciusa111(43):e4632-4637.
34.hayatsuh,watayay,kaik和iidas(1970)亚硫酸氢钠与尿嘧啶、胞嘧啶和其衍生物的反应(reactionofsodiumbisulfitewithuracil,cytosine,andtheirderivatives).biochemistry9(14):2858-2865.
35.clarksj,stathama,stirzakerc,molloypl和frommerm(2006)dna甲基化:亚硫酸氢盐修饰和分析(dnamethylation:bisulphitemodificationandanalysis).natprotoc1(5):2353-2364.
36.lim,等(2009)临床样品中dna甲基化的灵敏数字定量(sensitivedigitalquantificationofdnamethylationinclinicalsamples).natbiotechnol27(9):858-863.
37.lewisf,maughannj,smithv,hillank和quirkep(2001)解锁福尔马林包埋的组织中封存的基因(unlockingthearchive--geneexpressioninparaffin-embeddedtissue).jpathol195(1):66-71.
38.kochi,等(2006)实时定量rt-pcr显示对福尔马林固定的可变、试验依赖性灵敏度:对石蜡包埋组织中转录物水平的直接比较的意义(real-timequantitativert-pcrshowsvariable,assay-dependentsensitivitytoformalinfixation:implicationsfordirectcomparisonoftranscriptlevelsinparaffin-embeddedtissues).diagnmolpathol15(3):149-156.
39.kindei,等(2013)tert启动子突变在尿路上皮瘤形成早期发生并且是尿液中早期疾病和疾病复发的生物标志物(tertpromotermutationsoccurearlyinurothelialneoplasiaandarebiomarkersofearlydiseaseanddiseaserecurrenceinurine).cancerres73(24):7162-7167.
40.krimmeljd,等(2016)超深度测序检测腹膜液中的卵巢癌细胞并揭示了非癌组织中的体细胞tp53突变(ultra-deepsequencingdetectsovariancancercellsinperitonealfluidandrevealssomatictp53mutationsinnoncanceroustissues).procnatlacadsciusa113(21):6005-6010.
41.chungw,等(2011)使用尿沉渣中的新型dna甲基化生物标志物检测膀胱癌(detectionofbladdercancerusingnoveldnamethylationbiomarkersinurinesediments).cancerepidemiolbiomarkersprev20(7):1483-1491.
42.tabyr和issajp(2010)癌症表观遗传学(cancerepigenetics).cacancerjclin60(6):376-392.
43.issajp(2012)dna甲基化作为肿瘤学的临床标志物(dnamethylationasaclinicalmarkerinoncology).jclinoncol30(20):2566-2568.
44.harrisfr,等(2016)来自卵巢癌的循环无细胞dna中体细胞染色体重排的定量(quantificationofsomaticchromosomalrearrangementsincirculatingcell-freednafromovariancancers).scirep6:29831.
45.bozici,等(2013)响应靶向联合治疗的癌症进化动力学(evolutionarydynamicsofcancerinresponsetotargetedcombinationtherapy.elife2:e00747.
46.fearoner和vogelsteinb(1990)结肠直肠肿瘤发生的遗传模型(ageneticmodelforcolorectaltumorigenesis).cell61(5):759-767.
47.prioria,lewispd和mattosc(2012)对癌症中ras突变的全面调查(acomprehensivesurveyofrasmutationsincancer).cancerres72(10):2457-2467.
48.shiraishim和hayatsuh(2004)dna甲基化的亚硫酸氢盐基因组测序分析中将胞嘧啶高速转化为尿嘧啶(high-speedconversionofcytosinetouracilinbisulfitegenomicsequencinganalysisofdnamethylation.dnares11(6):409-415.
49.kandothc,等(2013)12种主要癌症类型的突变特征和意义(mutationallandscapeandsignificanceacross12majorcancertypes).nature502(7471):333-339.
50.learyrj,等(2012)用全基因组测序检测癌症患者循环中的染色体改变(detectionofchromosomalalterationsinthecirculationofcancerpatientswithwhole-genomesequencing).scitranslmed4(162):162ra154.
51.woodld,等(2007)人乳腺癌和结肠直肠癌的基因组特征(thegenomiclandscapesofhumanbreastandcolorectalcancers).science318(5853):1108-1113.
52.macintyreg,ylstrab和brentonjd(2016)用于精确治疗的癌症中的结构变异测序(sequencingstructuralvariantsincancerforprecisiontherapeutics).trendsgenet32(9):530-542.
序列表
<110>约翰斯霍普金斯大学(thejohnshopkinsuniversity)
<120>链特异性检测亚硫酸氢盐转化的双链体
<130>44807-0125wo1
<150>62/476、234
<151>2017-03-24
<160>48
<170>fastseqforwindowsversion4.0
<210>1
<211>65
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(65)
<223>n=a、t、c或g
<400>1
cgacgtaaaacgacggccagtnnnnnnnnnnnnnnggattagttggattgttagtgtgtt60
ttttt65
<210>2
<211>77
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(77)
<223>n=a、t、c或g
<400>2
cgacgtaaaacgacggccagtnnnnnnnnnnnnnncaaaataattctaaattaactaaat60
tatcaatacacttttcc77
<210>3
<211>78
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(78)
<223>n=a、t、c或g
<400>3
cgacgtaaaacgacggccagtnnnnnnnnnnnnnnagtattgagaaattgataaatgtat60
gaatagtattaaattaga78
<210>4
<211>73
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(73)
<223>n=a、t、c或g
<400>4
cgacgtaaaacgacggccagtnnnnnnnnnnnnnncaaaaaattaacaaacatataaaca60
acattaaaccaaa73
<210>5
<211>83
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(83)
<223>n=a、t、c或g
<400>5
cgacgtaaaacgacggccagtnnnnnnnnnnnnnnatttgttttgatgtaataaatatgt60
atatattattatattagtttgtt83
<210>6
<211>76
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(76)
<223>n=a、t、c或g
<400>6
cgacgtaaaacgacggccagtnnnnnnnnnnnnnnccaatataataaatatacacatatc60
attacaccaattcatc76
<210>7
<211>71
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(71)
<223>n=a、t、c或g
<400>7
cgacgtaaaacgacggccagtnnnnnnnnnnnnnngggaatttaaagtatatgaatttgt60
ttttttgttgt71
<210>8
<211>67
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(67)
<223>n=a、t、c或g
<400>8
cgacgtaaaacgacggccagtnnnnnnnnnnnnnnactcaaaatacataaacttatcttc60
ccatcat67
<210>9
<211>74
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(74)
<223>n=a、t、c或g
<400>9
cgacgtaaaacgacggccagtnnnnnnnnnnnnnngaagataagtttatgtattttgagt60
ttttttagttgtta74
<210>10
<211>69
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(69)
<223>n=a、t、c或g
<400>10
cgacgtaaaacgacggccagtnnnnnnnnnnnnnncaaattcatatactttaaattccct60
caaccatta69
<210>11
<211>64
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(64)
<223>n=a、t、c或g
<400>11
cgacgtaaaacgacggccagtnnnnnnnnnnnnnnggagaaaagtattggttggttttgt60
tttt64
<210>12
<211>80
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(80)
<223>n=a、t、c或g
<400>12
cgacgtaaaacgacggccagtnnnnnnnnnnnnnnactaaccttaaaatttaaaaaaaaa60
tatcaattaactttatcttt80
<210>13
<211>56
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(56)
<223>n=a、t、c或g
<400>13
cgacgtaaaacgacggccagtnnnnnnnnnnnnnngtgtgtagggtgaagtgtgag56
<210>14
<211>80
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(80)
<223>n=a、t、c或g
<400>14
cgacgtaaaacgacggccagtnnnnnnnnnnnnnnccaaaactaaataaaactccaaata60
tatataaaacaaaatataaa80
<210>15
<211>66
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(66)
<223>n=a、t、c或g
<400>15
cgacgtaaaacgacggccagtnnnnnnnnnnnnnnggagaatgttagtttgagttaggtt60
tttttg66
<210>16
<211>74
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<221>misc_feature
<222>(1)...(74)
<223>n=a、t、c或g
<400>16
cgacgtaaaacgacggccagtnnnnnnnnnnnnnncaaaaaataaaaaatatcaatctaa60
atcaaacccttcta74
<210>17
<211>71
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>17
cacacaggaaacagctatgaccatgattataaaaaactttaaaatactataaatataact60
caccaattaac71
<210>18
<211>62
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>18
cacacaggaaacagctatgaccatgagttttaaagtattgtagatgtggtttgttaatta60
at62
<210>19
<211>58
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>19
cacacaggaaacagctatgaccatgcattaactcatcctaaattataacaatcaccaa58
<210>20
<211>70
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>20
cacacaggaaacagctatgaccatggaaaagagaatgtattaatttattttgaattgtag60
taattattaa70
<210>21
<211>63
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>21
cacacaggaaacagctatgaccatgcaactaaaacttatcaaacccttttataaaaatct60
taa63
<210>22
<211>69
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>22
cacacaggaaacagctatgaccatgttattatagttagaatttattaaatttttttgtga60
agattttga69
<210>23
<211>70
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>23
cacacaggaaacagctatgaccatgtttaacaatttaacaattaaaaacatttcctataa60
aataatacta70
<210>24
<211>64
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>24
cacacaggaaacagctatgaccatgagtttgatagttaaaggtattttttgtgaaataat60
attg64
<210>25
<211>66
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>25
cacacaggaaacagctatgaccatgttttaatctatccttattttaaatatttctcccaa60
taaaaa66
<210>26
<211>69
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>26
cacacaggaaacagctatgaccatgagtttttaatttgtttttattttggatattttttt60
taatgaaag69
<210>27
<211>73
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>27
cacacaggaaacagctatgaccatgcaaaaaaaataaaaaataaaaatctatataatcaa60
aaaatcaataaca73
<210>28
<211>60
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>28
cacacaggaaacagctatgaccatgaaatggaagtttatgtgattaagaaattgatagta60
<210>29
<211>60
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>29
cacacaggaaacagctatgaccatgccactatacctatacaataccaataataacaacaa60
<210>30
<211>58
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>30
cacacaggaaacagctatgaccatggttgtatttgtgtaatgttagtgatgatgataa58
<210>31
<211>54
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>31
cacacaggaaacagctatgaccatgctcatataatatcatctctcctccctact54
<210>32
<211>69
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>32
cacacaggaaacagctatgaccatggtatagatttttttatttatgtgatgttatttttt60
ttttttgtt69
<210>33
<211>158
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>33
ggattagttggattgttagtgtgttttttttaatattatttgttttaattattattagtt60
tgtatttagttattttatattagtaagaatttgttggaaattagtaattagggttaattg120
gtgagttatatttatagtattttaaagttttttataat158
<210>34
<211>161
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>34
caaaataattctaaattaactaaattatcaatacacttttcccaacaccacctactccaa60
ccaccaccaatttatactcaatcatttcacaccaacaaaaacctattaaaaaccaataat120
caaaattaattaacaaaccacatctacaatactttaaaact161
<210>35
<211>150
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>35
agtattgagaaattgataaatgtatgaatagtattaaattagattttatttagttgagaa60
agatgagagattaatatttgatgtaagtatttgaaatggaattttatatatgaataattg120
gtgattgttataatttaggatgagttaatg150
<210>36
<211>157
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>36
caaaaaattaacaaacatataaacaacattaaaccaaaccttatccaactaaaaaaaaca60
aaaaaccaatacttaatataaatatttaaaataaaatcctatacataaataattaataat120
tactacaattcaaaataaattaatacattctcttttc157
<210>37
<211>145
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>37
atttgttttgatgtaataaatatgtatatattattatattagtttgtttttttttagttt60
tatagtgaattgttgtaatatgattgttatttttatttagttattggttaagatttttat120
aaaagggtttgataagttttagttg145
<210>38
<211>144
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>38
ccaatataataaatatacacatatcattacaccaattcatccctttccaactttacaata60
aattactacaacataattatcatcttcacttaaccattaatcaaaatcttcacaaaaaaa120
tttaataaattctaactataataa144
<210>39
<211>162
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>39
gggaatttaaagtatatgaatttgtttttttgttgtgtgggttttgaattggaggaatat60
atttttatttttagttggtagattataaattgaggatttgtatggttaaatatatattag120
tattattttataggaaatgtttttaattgttaaattgttaaa162
<210>40
<211>152
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>40
actcaaaatacataaacttatcttcccatcatataaatcctaaattaaaaaaatatatct60
tcacctttaactaacaaaccacaaactaaaaatctacataattaaatacataccaatatt120
atttcacaaaaaatacctttaactatcaaact152
<210>41
<211>152
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>41
gaagataagtttatgtattttgagtttttttagttgttatttgtgtgtggtgatattaaa60
gtagagtttttttataaatagaataagatgttaaaaaaggtttgtattttatttttattg120
ggagaaatatttaaaataaggatagattaaaa152
<210>42
<211>150
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>42
caaattcatatactttaaattccctcaaccattacctatatataataatatcaaaataaa60
attcttccacaaacaaaacaaaatactaaaaaaaatttatactttactttcattaaaaaa120
aatatccaaaataaaaacaaattaaaaact150
<210>43
<211>157
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>43
ggagaaaagtattggttggttttgtttttatttgttttgttaagattattttttgttaaa60
gtaagtattagatattttttgttattatttgtatgttttatattgtaaatgttattgatt120
ttttgattatatagatttttattttttattttttttg157
<210>44
<211>160
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>44
actaaccttaaaatttaaaaaaaaatatcaattaactttatctttatttactttatcaaa60
atcattttttattaaaataaatactaaatattccttatcattatctacacactctatact120
acaaatactatcaatttcttaatcacataaacttccattt160
<210>45
<211>143
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>45
gtgtgtagggtgaagtgtgagtttattttgttagttagtgataggtaggggtggggggtt60
tgttgaggattttttagtttgatgatgttgatgaatttaggttttagattgttgttatta120
ttggtattgtataggtatagtgg143
<210>46
<211>165
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>46
ccaaaactaaataaaactccaaatatatataaaacaaaatataaatctaccttactaacc60
aataacaaacaaaaacaaaaaacccatcaaaaactctccaacttaacaatactaataaat120
ccaaactccaaattatcatcatcactaacattacacaaatacaac165
<210>47
<211>133
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>47
ggagaatgttagtttgagttaggtttttttgttttgaatatgagttttttatggtgggag60
gtagattgattttttttggattttaggtggttgtaggagatagaagtagggaggagagat120
gatattatatgag133
<210>48
<211>156
<212>dna
<213>人工序列
<220>
<223>化学修饰的序列
<400>48
caaaaaataaaaaatatcaatctaaatcaaacccttctatcttaaacataaattttttat60
aacaaaaaataaactaaccctttttaaacttcaaataactataaaaaacaaaaacaaaaa120
aaaaaaataacatcacataaataaaaaaatctatac156
- 还没有人留言评论。精彩留言会获得点赞!