用于基因表达调节的人工基因组操纵的制作方法
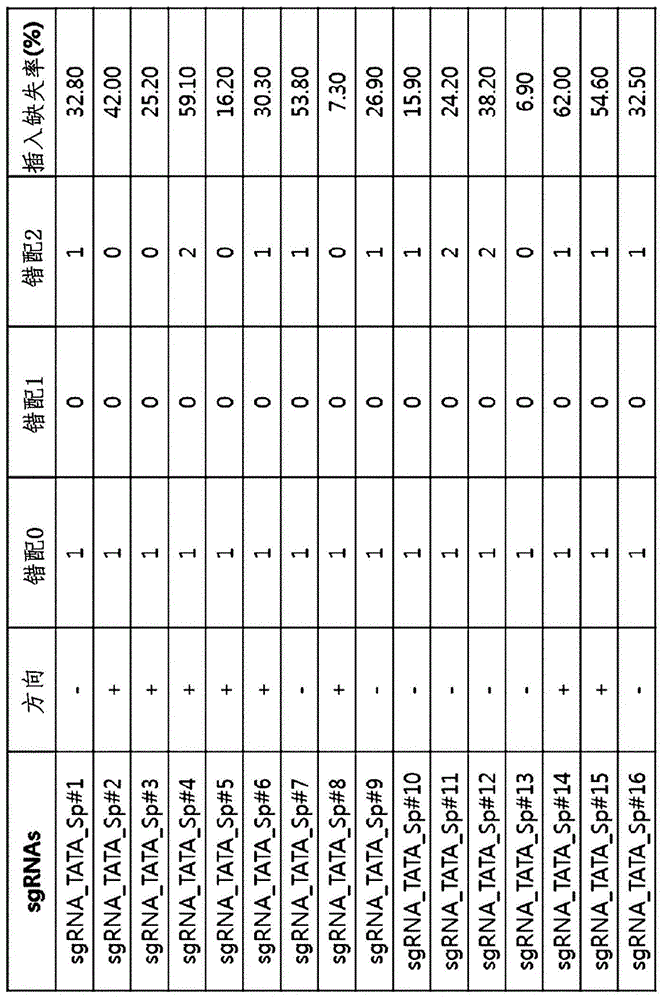
本发明涉及用于调控重复基因的表达的表达调控组合物,或者使用所述表达调控组合物的方法。更具体而言,本发明涉及表达调控组合物,所述表达调控组合物包含能够靶向重复基因的转录调节区域的引导核酸;以及对重复基因的表达进行调控的方法,所述方法通过使用所述表达调控组合物对所述重复基因的转录调节区域进行人工操纵和/或修饰来进行。此外,本发明涉及使用用于调控重复基因的表达的表达调控组合物治疗或改善由基因重复引起的疾病的方法。
背景技术:
:基因重复是染色体的遗传重组中产生的错误之一,是使染色体的部分区域重复的复制现象。基因重复是传递给下一代的一种突变类型。基因重复(以及由于染色体的部分区域的非复制导致的基因删除)影响基因表达。基因重复也引起遗传性疾病。具有代表性的是charcot-marie-tooth(cmt)1a型,其由于染色体的特定区域中发生的基因重复而引起,并且由于基因重复而导致手和足的周围神经发育有关的基因的过表达,从而造成手和足的畸形。因此,对于基因在恰当的位置和正确的时间表达以使生物学过程正常执行(例如细胞增殖、死亡、衰老和分化)非常重要。当基因在不适当的时间和位置不当表达,尤其是由基因重复引起的异常基因表达可能导致疾病时,由此有必要了解调控每个基因表达的分子机制,并且鉴别与每个基因有关的转录调节因子是重要的。存在能够精确地调控基因表达的多种转录调节因子,例如启动子、远端调控元件、以及转录因子、激活剂以及辅激活剂,它们参与基因表达的调控。可通过转录调节因子的改变来调控基因表达,并且转录调节因子的异常改变可引起基因的异常表达,从而诱发疾病。因此,转录调节因子的改变可引起多种疾病,或者改善和治疗疾病。然而,目前调控转录调节因子的方法仅调控瞬时基因表达,而难以有持续的基因表达调节。因此,没有从根本上的治疗方法来治疗由基因表达异常或困难引起的疾病。因此,需要一种通过转录调节因子的遗传编辑或修饰而表现出更持续的治疗效果的方法。[现有技术文件]非专利文件1.hamdan,h.,kockara,n.t.,jolly,l.a.,haun,s.,andwight,p.a.(2015).controlofhumanplp1expressionthroughtranscriptionalregulatoryelementsandalternativelysplicedexonsinintron1.asnneuro7.2.hamdan,h.,patyal,p.,kockara,n.t.,andwight,p.a.(2018).thewmn1enhancerregioninintron1isrequiredforexpressionofhumanplp1.glia.3.meng,f.,zolova,o.,kokorina,n.a.,dobretsova,a.,andwight,p.a.(2005).characterizationofanintronicenhancerthatregulatesmyelinproteolipidprotein(plp)geneexpressioninoligodendrocytes.jneuroscires82,346-356.4.tuason,m.c.,rastikerdar,a.,kuhlmann,t.,goujet-zalc,c.,zalc,b.,dib,s.,friedman,h.,andpeterson,a.(2008).separateproteolipidprotein/dm20enhancersservedifferentlineagesandstagesofdevelopment.jneurosci28,6895-6903.5.wight.p.a.(2017).effectsofintron1sequencesonhumanplp1expression:implicationsforplp1-relateddisorders.asnneuro9,1759091417720583.技术实现要素:[技术问题]本发明涉及提供用于调控细胞基因组中存在的重复基因的表达的表达调控组合物。本发明还涉及提供调控真核细胞的基因组中存在的重复基因的表达的方法。本发明还涉及使用表达调控组合物治疗基因重复疾病的方法。[技术方案]为实现上述目的,本发明涉及用于调控细胞的基因组中的重复基因表达的表达调控组合物。更具体而言,本发明涉及表达调控组合物,所述表达调控组合物包含能够靶向重复基因的转录调节区域的引导核酸;以及调控重复基因的表达的方法,所述方法通过使用所述表达调控组合物对所述重复基因的转录调节区域进行人工操纵和/或修饰来进行。此外,本发明涉及使用用于调控重复基因的表达的表达调控组合物治疗或改善由基因重复引起的疾病的方法。本发明提供了表达调控组合物,以用于调控细胞的基因组中存在的重复基因的表达。一方面,所述表达调控组合物可包含:引导核酸或者编码所述引导核酸的核酸,所述引导核酸能够靶向所述重复基因的转录调节区域中的靶序列;以及一种或多种编辑蛋白或者编码所述编辑蛋白的核酸。所述引导核酸可包含引导结构域,所述引导结构域能够靶向存在于所述重复基因的转录调节区域中的靶序列。此处,所述引导结构域可包含能够与存在于所述重复基因的转录调节区域中的靶序列的引导核酸结合序列形成互补结合的核苷酸序列。此处,所述引导结构域能够与所述重复基因的转录调节区域中的靶序列的引导核酸结合序列形成互补结合。此处,所述互补结合可包含0-5个错配结合。所述引导核酸可包含选自于由以下结构域所组成的组中的一个或多个结构域:第一互补结构域、接头结构域、第二互补结构域、近端(proximal)结构域和尾部(tail)结构域。所述编辑蛋白可为crispr酶。所述引导核酸和所述编辑蛋白可形成引导核酸-编辑蛋白复合体。此处,所述引导核酸-编辑蛋白复合体可通过所述引导核酸的部分核酸与所述编辑蛋白的部分氨基酸的相互作用而形成。所述转录调节区域可为选自于由以下区域所组成的组中的一个或多个区域:启动子区域、增强子区域、沉默子区域、绝缘子区域和基因座控制区域(lcr)。所述靶序列可为位于所述重复基因的转录调节区域中的10nt-25nt(核苷酸)的连续序列。所述靶序列可为位于所述重复基因的启动子区域或临近所述重复基因的启动子区域的10nt-25nt的连续序列。此处,所述靶序列可为位于所述重复基因的核心启动子区域或临近所述重复基因的核心启动子区域的10nt-25nt的连续序列。此处,所述靶序列可为包含所述重复基因的核心启动子区域的tata盒的10nt-25nt的连续序列,或者位于临近所述tata盒区域的10nt-25nt的连续序列。此处,所述靶序列可为包含所述重复基因的核心启动子区域存在的5′-tata-3′序列的全部或部分的10nt-25nt的连续序列。此处,所述靶序列可为包含所述重复基因的核心启动子区域中存在的5′-tatawaw-3′(w=a或t)序列的全部或部分的10nt-25nt的连续序列。此处,所述靶序列可为包含所述重复基因的核心启动子区域中存在的5′-tatawawr-3′(w=a或t,r=a或g)序列的全部或部分的10nt-25nt的连续序列。此处,所述靶序列可为包含选自于由以下序列所组成的组中的一个或多个序列的全部或部分的10nt-25nt的连续序列:5′-cataaaa-3′序列、5′-cataaaa-3′序列、5′-tataa-3′序列、5′-tataaaa-3′序列、5′-cataaata-3′序列、5′-tatataa-3′序列、5′-tatatatatatataa-3′序列、5′-tatattata-3′序列、5′-tataaa-3′序列、5′-tataaaata-3′序列、5′-tatata-3′序列、5′-gattaaaaa-3′序列、5′-tataaaaa-3′序列、5′-ttataa-3′序列、5′-ttttaaaa-3′序列、5′-tctttaaaa-3′序列、5′-gacatttaa-3′序列、5′-tgatatcaa-3′序列、5′-tataaata-3′序列、5′-tataaga-3′序列、5′-aataaa-3′序列、5′-tttata-3′序列、5′-cataaaaa-3′序列、5′-tataca-3′序列、5′-tttaaga-3′序列、5′-gataaag-3′序列、5′-tataaca-3′序列、5′-tcttatctt-3′序列、5′-ttgtacttt-3′序列、5′-catataa-3′序列、5′-tataaat-3′序列、5′-tatatataaaaaaaa-3′序列和5′-cataaataaaaaaaatta-3′序列。此处,所述靶序列可为位于选自于由以下序列所组成的组中的一个或多个序列的5′端或3′端的10nt-25nt的连续序列:5′-tata-3′序列、5′-cataaaa-3′序列、5′-cataaaa-3′序列、5′-tataa-3′序列、5′-tataaaa-3′序列、5′-cataaata-3′序列、5′-tatataa-3′序列、5′-tatatatatatataa-3′序列、5′-tatattata-3′序列、5′-tataaa-3′序列、5′-tataaaata-3′序列、5′-tatata-3′序列、5′-gattaaaaa-3′序列、5′-tataaaaa-3′序列、5′-ttataa-3′序列、5′-ttttaaaa-3′序列、5′-tctttaaaa-3′序列、5′-gacatttaa-3′序列、5′-tgatatcaa-3′序列、5′-tataaata-3′序列、5′-tataaga-3′序列、5′-aataaa-3′序列、5′-tttata-3′序列、5′-cataaaaa-3′序列、5′-tataca-3′序列、5′-tttaaga-3′序列、5′-gataaag-3′序列、5′-tataaca-3′序列、5′-tcttatctt-3′序列、5′-ttgtacttt-3′序列、5′-catataa-3′序列、5′-tataaat-3′序列、5′-tatatataaaaaaaa-3′序列和5′-cataaataaaaaaaatta-3′序列。所述靶序列可为位于所述重复基因的增强子区域中的10nt-25nt的连续序列。所述靶序列可为临近所述重复基因的增强子区域的10nt-25nt的连续序列。所述靶序列可为临近所述重复基因的转录调节区域的核酸序列的pam(proto-spacer-adjacentmotif,前间区序列邻近基序)序列的5′端和/或3′端的10nt-25nt的连续序列。此处,可根据所述crispr酶确定所述pam序列。所述crispr酶可为cas9蛋白或cpf1蛋白。此处,cas9蛋白可为选自于由以下cas9蛋白所组成的组中的一种或多种cas9蛋白:酿脓链球菌(streptococcuspyogenes)衍生而来的cas9蛋白、空肠弯曲杆菌(campylobacterjejuni)衍生而来的cas9蛋白、嗜热链球菌(streptococcusthermophilus)衍生而来的cas9蛋白、金黄色葡萄球菌(staphylococcusaureus)衍生而来的cas9蛋白、以及脑膜炎奈瑟菌(neisseriameningitidis)衍生而来的cas9蛋白。所述重复基因可为选自于由以下基因所组成的组中的一种或多种基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。所述重复基因可为癌基因(oncogene)。此处,所述癌基因可为选自于由以下基因所组成的组中的一种或多种基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。所述细胞可为真核细胞。所述真核细胞可为哺乳动物细胞。所述引导核酸和所述编辑蛋白可分别以核酸序列的形式存在于一个或多个载体中。此处,所述载体可为质粒或病毒载体。此处,所述病毒载体可为选自于由以下病毒载体所组成的组中的一种或多种病毒载体:逆转录病毒、慢病毒、腺病毒、腺相关病毒(aav)、痘苗病毒、痘病毒和单纯疱疹病毒。所述表达调控组合物可包含所述处于引导核酸-编辑蛋白复合体形式的引导核酸和编辑蛋白。所述表达调控组合物可进一步包含供体。本发明提供了一种调控真核细胞的基因组中存在的重复基因的表达的方法。一方面,用于调控真核细胞的基因组中存在的重复基因的表达的方法可包括将表达调控组合物导入真核细胞。所述表达调控组合物可包含以下:引导核酸或者编码所述引导核酸的核酸,所述引导核酸能够靶向存在于所述重复基因的转录调节区域中的靶序列;以及一种或多种编辑蛋白或者编码所述编辑蛋白的核酸。所述真核细胞可为哺乳动物细胞。所述引导核酸可包含能够靶向存在于所述重复基因的转录调节区域中的靶序列的引导结构域。此处,所述引导结构域可包含能够与存在于所述重复基因的转录调节区域中的靶序列的引导核酸结合序列形成互补结合的核苷酸序列。此处,所述引导结构域可与所述重复基因的转录调节区域中的靶序列的引导核酸结合序列形成互补结合。此处,所述互补结合可包含0-5个错配结合。所述引导核酸可包含选自于由以下结构域所组成的组中的一个或多个结构域:第一互补结构域、接头结构域、第二互补结构域、近端结构域和尾部结构域。所述编辑蛋白可为crispr酶。所述引导核酸和所述编辑蛋白可形成引导核酸-编辑蛋白复合体。此处,所述引导核酸-编辑蛋白复合体可通过引导核酸的部分核酸和编辑蛋白的部分氨基酸的相互作用而形成。所述表达调控组合物可包含处于引导核酸-编辑蛋白复合体形式的引导核酸和编辑蛋白。所述表达调控组合物可包含一种或多种载体,所述载体中分别包含处于核酸形式的引导核酸和编辑蛋白。可通过选自于由以下方法所组成的组中的一种或多种方法进行导入:电穿孔、脂质体、质粒、病毒载体、纳米粒子和蛋白易位结构域(ptd)融合蛋白法。本发明提供了用于治疗基因重复疾病的方法。一方面,用于治疗基因重复疾病的方法可包含将表达调控组合物给予待治疗的受试者。所述表达调控组合物可包含以下:引导核酸或者编码所述引导核酸的核酸,所述引导核酸能够靶向存在于重复基因的转录调节区域中的靶序列;以及一种或多种编辑蛋白或者编码所述编辑蛋白的核酸。所述引导核酸可包含能够靶向存在于所述重复基因的转录调节区域中的靶序列的引导结构域。此处,所述引导结构域可包含能够与存在于所述重复基因的转录调节区域中的靶序列的引导核酸结合序列形成互补结合的核苷酸序列。此处,所述引导结构域可与所述重复基因的转录调节区域中的靶序列的引导核酸结合序列形成互补结合。此处,所述互补结合可包含0-5个错配结合。所述引导核酸可包含选自于由以下结构域所组成的组中的一个或多个结构域:第一互补结构域、接头结构域、第二互补结构域、近端结构域和尾部结构域。所述编辑蛋白可为crispr酶。所述引导核酸和所述编辑蛋白可形成引导核酸-编辑蛋白复合体。此处,所述引导核酸-编辑蛋白复合体可通过引导核酸的部分核酸和编辑蛋白的部分氨基酸相互作用而形成。所述基因重复疾病可为charcot-marie-tooth1a(cmt1a)、dejerine-sottas病(dsd)、先天性髓鞘形成不良神经病(congenitalhypomyelinationneuropathy,chn)、roussy-levy综合征(rls)、pelizaeus-merzbacher病(pmd)、mecp2重复综合征、x连锁垂体机能减退(xlhp)、potocki-lupski综合征(ptls)、腭心面综合征(velocardiofacialsyndrome,vcfs)、williamsbeuren综合征(wbs)、alagille综合征(as)、生长障碍综合征、颅缝早闭、常染色体显性脑白质营养不良(adld)、帕金森病或阿尔茨海默病。所述基因重复疾病可为由癌基因重复引起的癌症。此处,所述由癌基因重复引起的癌症可为乳腺癌、宫颈癌、结直肠癌、食管癌、胃癌、胶质母细胞瘤、头颈癌、肝细胞癌、神经母细胞瘤、卵巢癌、肉瘤或小细胞肺癌。所述待治疗的受试者可为哺乳动物,包括人、猴、小鼠和大鼠。给予可通过注射、输注、植入或移植进行。[有益效果]本发明可通过表达调控组合物来调控重复基因的表达。更具体而言,可通过使用表达调控组合物对重复基因的转录调节区域进行人工操纵和/或修饰来对所述重复基因的表达进行调控,所述表达调控组合物包含能够靶向所述重复基因的转录调节区域的引导核酸。也可使用用于调控重复基因的表达的表达调控组合物改善或治疗由所述基因重复引起的疾病。附图说明图1为说明了由于spcas9-sgrna介导的基因操纵而来的插入缺失(indel)频率(%)的一组结果,并说明了(a)tata盒和(b)增强子(其中sgrna的靶位点被分割开)各自的插入缺失频率。图2为说明了由于cjcas9-sgrna介导的基因操纵而来的插入缺失频率(%)的一组结果,并说明了(a)tata盒和(b)增强子(其中sgrna的靶位点被分割开)各自的插入缺失频率。图3说明了靶向雪旺样细胞中人pmp22基因的调节元件的spcas9-sgrna的基因操纵效果。图4说明了由靶向人pmp22的cds的spcas9-sgrna诱导的移码突变率。图5说明了由双sgrna的处理导致的人pmp22的小部分的删除。图6为说明了人雪旺样细胞中由于spcas9-sgrna导致的人pmp22的mrna表达减少的图表。图7为说明了人原代雪旺细胞中人pmp22基因的每个靶位点处由spcas9-sgrna引起的pmp22的有效的特异性表达减少的图表,(a)说明了由spcas9-sgrna导致的每个靶位点处的插入缺失频率测量结果;(b)说明了pmp22的相对mrna表达比较结果,通过qrt-pcr在有或无用于每个靶位点的rnp复合体和髓鞘形成信号因子的处理的情况下进行测量(n=3,单因素anova和tukeypost-hoc检验:*p<0.05);以及(c)说明了由靶向远端增强子位点(远端增强子区域)b和c的spcas9-sgrna引起的插入缺失频率测量结果。图8为说明了借助靶向人pmp22基因的tata盒位点的crispr-cas9而来的pmp22的体外有效的特异性表达降低的图表,(a)说明了靶向人pmp22位置的启动子区域的靶序列;以及(b)中最左图、中图和最右图分别说明了在人原代雪旺细胞中使用靶向深度测序的插入缺失频率测量结果、总插入缺失频率中的tata盒1突变频率测量结果(n=3)以及pmp22的相对mrna表达比较结果,在人原代雪旺细胞中通过qrt-pcr在有或无rnp复合体和髓鞘形成信号因子的处理的情况下测量(n=3,单因素anova和tukeypost-hoc检验:*p<0.05)。图9示出了在通过人原代雪旺细胞中的靶向深度测序借助计算机脱靶分析发现脱靶和中靶中,由pmp22-tatarnp的插入缺失频率,(a)为说明了插入缺失频率的图表;(b)说明了高频率的插入缺失模式;以及(c)示出了借助计算机脱靶分析发现的脱靶位点。图10为说明了在人的整个基因组中被pmp22-tatarnp切割的位点的一组结果,(a)说明了全基因组circos图;(b)说明了借助计算机脱靶分析发现的脱靶位点中的通过digenome-seq显现的脱靶位点;以及(c)为说明了脱靶位点中插入缺失频率的图表。图11示意性地说明了在c22小鼠中使用pmp22-tatarna疗法的治疗性方法。图12为说明了在cmt1a小鼠中借助由crispr/cas9对pmp22的表达抑制来缓解疾病表型的一组结果,(a)为说明了在经mrosa26或pmp22-tatarnp复合体处理的坐骨神经中使用靶向深度测序的插入缺失频率的图表(n=3);(b)为总插入缺失频率中tata盒1突变频率测量结果(n=3);以及(c)为比较使用qrt-pcr从经mrosa26或pmp22-tatarnp复合体处理的坐骨神经得到的pmp22的mrna表达的相对量的图。图13为说明了通过计算机分析的小鼠基因组中pmp22-tatasgrna的插入缺失频率和脱靶位点的一组结果,(a)说明了脱靶位点;以及(b)为说明了每个脱靶位点处的插入缺失频率的图表。图14为说明了在cmt1a小鼠中借助由crispr/cas9对pmp22的表达抑制来缓解疾病表型的一组结果,(a)为经mrosa26或pmp22-tatarnp复合体处理的坐骨神经组织的半薄切片的一组图像;以及(b)中的上图和下图分别为说明了在经pmp22-tatarnp处理的小鼠中g比增加的散点图以及说明了在经mp22-tatarnp处理的小鼠中有髓鞘的轴突的直径增加的图表。图15为说明了在cmt1a小鼠中借助由crispr/cas9对pmp22的表达抑制引起的电生理学变化的一组结果,(a)为说明了远端潜伏期(dl)的变化的图表;(b)为说明了运动神经传导速度(ncv)的变化的图表;以及(c)为说明了复合肌肉动作电位(cmap)的变化的图表(对于mrosa26rnp,n=7;对于pmp22-tata,n=10)。图16为在cmt1a小鼠中由于由crispr/cas9对pmp22的表达抑制引起的运动行为的一组分析结果,(a)中的上图和下图分别为旋转杆测试结果(对于mrosa26rnp,n=7;对于pmp22-tata,n=11)以及每周测量的旋转杆测试结果,直至小鼠达到8周龄-16周龄(对于mrosa26rnp,n=7;对于pmp22-tata,n=11);(b)中的上图和下图分别为说明了经mrosa26或pmp22-tatarnp复合体处理的c22小鼠腓肠肌重量/体重的比的图表,以及一组经mrosa26或pmp22-tatarnp复合体处理的c22小鼠的腓肠肌图像。图17为说明了pmd治疗策略的示意图,其中设计了靶向plp1基因的tata盒区域和增强子区域的sgrna。在靶向增强子区域的sgrna的情况下,示出了使用两种sgrna去除增强子的策略。此处,靶向增强子区域的上游的sgrna表示为“上(up)”,靶向增强子区域的下游的sgrna表示为“下(down)”,“上”和“下”也根据表5和表6中的位置表示。图18说明了在示例性实施方式中使用的cjcas9质粒。图19为示出了靶向mplp1的tata盒区域的spcas9-sgrna的筛选结果的一组图表。(a)示出了nih-3t3细胞中确认的插入缺失频率(%);以及(b)示出了n20.1细胞中确认的插入缺失频率(%)。此处,使用的sgrna为靶向mplp1-tata-sp-01的sgrna,由图表上的靶序列中示出的数字来区分。图20为示出了靶向mplp1的tata盒区域的cjcas9-sgrna的筛选结果的一组图表。(a)示出了nih-3t3细胞中确认的插入缺失频率(%);以及(b)示出了n20.1细胞中确认的插入缺失频率(%)。此处,使用的sgrna为mplp1-tata-cj-01至mplp1-tata-cj-04,由图表上的靶序列中示出的数字来区分。图21为示出了靶向mplp1的增强子(wmn1增强子)区域的spcas9-sgrna的筛选结果的一组图表。(a)示出了nih-3t3细胞中确认的插入缺失频率(%);以及(b)示出了n20.1细胞中确认的插入缺失频率(%)。此处,使用的sgrna为mplp1-wmn1-sp-01至mplp1-wmn1-sp-36,由图表上的靶序列中示出的数字来区分。图22为示出了靶向mplp1的增强子(wmn1增强子)区域的cjcas9-sgrna的筛选结果的一组图表。(a)示出了nih-3t3细胞中确认的插入缺失频率(%);以及(b)示出了n20.1细胞中确认的插入缺失频率(%)。此处,使用的sgrna为mplp1-wmn1-cj-01至mplp1-wmn1-sp-28,由图表上的靶序列中示出的数字来区分。图23为示出了根据靶向mplp1的tata盒和增强子(wmn1增强子)区域的spcas9-sgrna和cjcas9-sgrna的plp的mrna表达水平的一组图表。(a)示出了根据spcas9-sgrna的plp的mrna表达水平,并且此处,靶向tata盒区域的mplp1-tata-sp-01以及靶向增强子的mplp1-wmn1-sp-07+mplp1-wmn1-sp-27和mplp1-wmn1-sp-08+mplp1-wmn1-sp-27用作sgrna。(b)示出了根据cjcas9-sgrna的plp的mrna表达水平,并且此处,靶向tata盒区域的mplp1-tata-cj-02和mplp1-tata-cj-03以及靶向增强子的mplp1-wmn1-cj-06+mplp1-wmn1-cj-09、mplp1-wmn1-cj-06+mplp1-wmn1-cj-10和mplp1-wmn1-cj-06+mplp1-wmn1-cj-19用作sgrna。mrosa26用作对照。图24为示出了靶向hplp1的增强子(wmn1增强子)区域的spcas9-sgrna的筛选结果的图表,示出了jurkat细胞中确认的插入缺失频率(%),使用的sgrna为hplp1-wmn1-sp-01至hplp1-wmn1-sp-36,由图表上的靶序列中示出的数字来区分。图25为示出了靶向hplp1的增强子(wmn1增强子)区域的cjcas9-sgrna的筛选结果的图表,示出了293t细胞中确认的插入缺失频率(%),使用的sgrna为hplp1-wmn1-cj-01至hplp1-wmn1-cj-36,由图表上的靶序列中示出的数字来区分。具体实施方式除非另有定义,本文使用的全部技术术语和科学术语具有与本发明所属领域的普通技术人员通常理解的含义相同的含义。尽管与本文所述的方法和材料类似或相同的方法和材料可用于本发明的实践或测试中,适合的方法和材料在下文中描述。本文提及的所有出版物、专利申请、专利和其它参考文献都以引用的方式将它们整体并入。此外,材料、方法和实例仅为说明性的,而不旨在为限制性的。本说明书中公开的一个方面涉及表达调控组合物。所述表达调控组合物是用于调控由基因重复引起的重复基因的表达的组合物。“基因重复”意为基因组中存在两个以上相同的基因。基因重复还包括在基因组中具有两个以上的相同基因的部分。例如,基因重复可意为在基因组中存在两个以上全长a基因,或者在基因组中存在一个全长a基因以及a基因的一个或多个部分(例如外显子1)。例如,基因重复可意为在基因组中存在两个全长b基因以及b基因的一个或多个部分(例如外显子1和外显子2)。基因重复的类型可改变,并且基因重复包括基因组中全长基因和/或基因的部分序列的重复(即两个以上)。此外,基因重复包括使染色体的部分区域重复的复制现象,其在染色体的遗传重组过程中发生。此类基因重复是一种基因突变类型,并且可传递给下一代。基因重复以及由于基因的部分区域未复制而发生的基因删除影响基因表达。此处,基因重复的对象(即以两个以上数目存在的基因)称为“重复基因(duplicationgene)”。重复基因可为由于基因重复而在基因组中总拷贝数目增加的基因。重复基因可为由于基因重复而仅使部分区域重复的突变基因。此处,突变基因可为基因的整个序列中的一个或多个核苷酸序列重复的基因。或者,突变基因可为由于基因重复而使基因的部分核酸片段重复的基因。此处,核酸片段可具有50bp以上的核苷酸序列。基因重复包括整个基因组重复。基因重复包括靶基因重复。此处,靶基因重复是在新物种应对环境变化的分化和适应中扩增相关基因或相关基因消失以适合于特定环境的基因重复类型,并且多数复制通过转座子完成。基因重复包括异位重组(ectopicrecombination)。此处,异位重组根据由于同源染色体的减数分裂过程中的不等交换引起的复制所造成的两个染色体之间的重复序列的程度而发生。出现了交换点处的重复和相互删除。异位重组由典型的重复遗传元件(例如转座元件)介导,并且引起由重组导致的复制。基因重复包括复制滑动(replicationslippage)。此处,复制滑动是由于dna复制过程中的错误引起的短遗传序列的复制,并且在dna聚合酶不正确地附着于变性的dna链上并再次复制dna链时发生。复制滑动也经常由重复的遗传元件介导。基因重复包括逆转录转座(retrotransposition)。此处,逆转录转座是由逆转录病毒或逆转录元件侵入细胞介导的复制,其中进行基因的逆转录以形成逆基因(retrogene),并且由于逆基因的重组而使基因复制实现。逆转录转座由遗传元件(例如可逆转录转座元件)介导。基因重复可增加由重复基因转录的mrna的表达。此处,与未发生基因重复的状态相比,转录的mrna的表达可能增加。基因重复可增加由重复基因编码的蛋白的表达。此处,与未发生基因重复的状态相比,蛋白的表达可能增加。基因重复可导致重复基因编码的蛋白的功能障碍。此处,功能障碍可为蛋白的功能过度、被抑制的功能以及第三功能。基因重复可引起基因重复疾病。“基因重复疾病”是由基因重复导致的疾病,并且包括由于重复基因的异常扩增引起遗传异常并由因此过表达或异常产生的蛋白诱发病理特征的所有疾病或病症。此处,“病理特征”是指由于疾病引起的生物体的细胞水平以及组织、器官和个体水平的变化。基因重复疾病可为charcot-marie-tooth1a型(cmt1a)、dejerine-sottas病(dsd)、先天性髓鞘形成不良神经病(chn)、roussy-levy综合征(rls)、pelizaeus-merzbacher病(pmd)、mecp2重复综合征、x连锁垂体机能减退(xlhp)、potocki-lupski综合征(ptls)、腭心面综合征(vcfs)、williamsbeuren综合征(wbs)、alagille综合征(as)、生长障碍综合征、颅缝早闭、常染色体显性脑白质营养不良(adld)、帕金森病或阿尔茨海默病。基因重复疾病可为由癌基因重复导致的癌症。此处,癌症可为乳腺癌、宫颈癌、结直肠癌、食管癌、胃癌、胶质母细胞瘤、头颈癌、肝细胞癌、神经母细胞瘤、卵巢癌、肉瘤或小细胞肺癌。基因重复疾病可为由pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因或app基因的重复导致的疾病。基因重复疾病可为由myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因或akt2基因的重复导致的疾病。基因重复疾病可为由重复基因转录的mrna的表达异常增加导致的疾病。基因重复疾病可为由重复基因编码的蛋白的表达异常增加导致的疾病。表达调控组合物可用于调控由重复基因的转录产生的mrna的表达。表达调控组合物可用于调控由重复基因编码的蛋白的表达。表达调控组合物可用于重复基因的人工修饰或操纵。此处,“人工修饰或操纵(人工修饰、操纵或工程化)”是指人工修饰的状态而不是天然发生的状态。下文可将非天然的、人工修饰或操纵的重复基因与人工重复基因互换使用。“表达调控系统”是包括由于人工操纵的重复基因的表达的调控引起的所有现象,以及表达调控系统中直接或间接涉及的所有材料、成分、方法和用途在内的术语。表达调控组合物可用于重复基因的转录调节区域的人工操纵或修饰。此处,“转录调节区域(转录调控区域)”是调控基于基因的dna合成rna的整体过程的区域,包括与基因的近端dna序列和/或基因的dna序列中的转录因子相互作用的所有区域。此处,转录因子是当被激活时结合至dna的特定区域(即,靠近基因的反应元件)从而促进或抑制基因表达的蛋白,并且该反应元件包含在转录调节区域中。转录调节区域的类型和位置可根据基因而改变,并且甚至在同一物种中,个体之间的核酸序列可存在差异。转录调节区域可为启动子、增强子、沉默子、绝缘子和/或基因座控制区域(lcr)。启动子可为核心启动子(corepromoter)、近端启动子(proximalpromoter)和/或远端启动子(distalpromoter)。此处,核心启动子可包含转录起始位点(tss)、rna聚合酶结合位点、转录因子结合位点和/或tata盒。tata盒可为位于用于起始rpb4/rbp7的转录的起始位点上游的25个碱基对的区域。tata盒可为位于tss上游的30个碱基对的区域。tata盒可为位于tss上游的40个-100个碱基对的区域。例如,tata盒可为包含启动子和/或核心启动子中存在的5′-tata(a/t)a(a/t)-3′序列的区域。或者,tata盒可为包含启动子和/或核心启动子中存在的5′-tata(a/t)a(a/t)(a/g)-3′序列的区域。例如,tata盒可为包含启动子和/或核心启动子中存在的选自于由以下序列所组成的组中的一个或多个序列:5′-cataaaa-3′序列、5′-cataaaa-3′序列、5′-tataa-3′序列、5′-tataaaa-3′序列、5′-cataaata-3′序列、5′-tatataa-3′序列、5′-tatatatatatataa-3′序列、5′-tatattata-3′序列、5′-tataaa-3′序列、5′-tataaaata-3′序列、5′-tatata-3′序列、5′-gattaaaaa-3′序列、5′-tataaaaa-3′序列、5′-ttataa-3′序列、5′-ttttaaaa-3′序列、5′-tctttaaaa-3′序列、5′-gacatttaa-3′序列、5′-tgatatcaa-3′序列、5′-tataaata-3′序列、5′-tataaga-3′序列、5′-aataaa-3′序列、5′-tttata-3′序列、5′-cataaaaa-3′序列、5′-tataca-3′序列、5′-tttaaga-3′序列、5′-gataaag-3′序列、5′-tataaca-3′序列、5′-tcttatctt-3′序列、5′-ttgtacttt-3′序列、5′-catataa-3′序列、5′-tataaat-3′序列、5′-tatatataaaaaaaa-3′序列和5′-cataaataaaaaaaatta-3′序列。例如,tata盒可为启动子和/或核心启动子中存在的tata结合蛋白(tbp)结合的区域。此处,近端启动子可包含tss上游的1-300bp的区域、cpg位点和/或特异性转录因子结合位点。增强子可包含增强子盒(e盒)。绝缘子可为抑制增强子与启动子之间的相互作用或防止被抑制的染色质的扩增的区域。基因座控制区域(lcr)可为其中存在许多顺式作用因子(例如增强子、沉默子、绝缘子、mar和sar)的区域。作为说明书中公开的一个方面,表达调控组合物可包含引导核酸。表达调控组合物可包含靶向重复基因的引导核酸或编码该引导核酸的核酸序列。“引导核酸”是指识别靶核酸、基因或染色体并且与编辑蛋白相互作用的核苷酸序列。此处,引导核酸可互补结合至靶核酸、基因或染色体中的部分核苷酸序列。此外,引导核酸的部分核苷酸序列可与编辑蛋白的一些氨基酸相互作用,从而形成引导核酸-编辑蛋白复合体。引导核酸可执行诱导引导核酸-编辑蛋白复合体定位于靶核酸、基因或染色体的靶区域的功能。引导核酸可以以dna、rna或dna/rna混合体的形式存在,并且可具有5nt-150nt的核酸序列。引导核酸可具有一条连续的核酸序列。例如,所述一条连续的核酸序列可为(n)m,其中n代表a、t、c或g,或为a、u、c或g;m为1-150的整数。引导核酸可具有两条以上连续的核酸序列。例如,所述两条以上连续的核酸序列可为(n)m以及(n)o,其中n代表a、t、c或g,或代表a、u、c或g;m和o为1-150的整数,并且m和o可彼此相同或彼此不同。引导核酸可包含一个或多个结构域。所述结构域可为引导结构域、第一互补结构域、接头结构域、第二互补结构域、近端结构域或尾部结构域,但不限于此。此处,一个引导核酸可具有两个以上功能结构域。此处,两个以上功能结构域可彼此不同。例如,一个引导核酸可具有引导结构域和第一互补结构域。又例如,一个引导核酸可具有第二互补结构域、近端结构域和尾部结构域。再例如,一个引导核酸可具有引导结构域、第一互补结构域、第二互补结构域、近端结构域和尾部结构域。或者,一个引导核酸中包含的两个以上功能结构域可彼此相同。例如,一个引导核酸可具有两个以上近端结构域。又例如,一个引导核酸可具有两个以上尾部结构域。但是,一个引导核酸中包含的功能结构域是相同的结构域的描述并不意为两个功能结构域的序列是相同的。即使序列不同,在功能上执行相同功能时,两个功能结构域也可为相同结构域。将在下面详细描述功能结构域。i)引导结构域术语“引导结构域”是能够与靶基因的转录调节区域中的核酸的双链的任一条链的部分序列互补结合的结构域,并与靶基因的转录调节区域的核酸特异性相互作用。例如,引导结构域可执行诱导引导核酸-编辑蛋白复合体定位于靶基因的转录调节区域的核酸中的特定核苷酸序列的功能。引导结构域可为10个-35个核苷酸的序列。在实例中,引导结构域可为10个-35个核苷酸、15个-35个核苷酸、20个-35个核苷酸、25个-35个核苷酸或30个-35个核苷酸的序列。在另一实例中,引导结构域可为10个-15个核苷酸、15个-20个核苷酸、20个-25个核苷酸、25个-30个核苷酸或30个-35个核苷酸的序列。引导结构域可具有引导序列。术语“引导序列”为与靶基因的转录调节区域中的核酸的双链的任一条链的部分序列互补的核苷酸序列。此处,引导序列可为具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%或更高互补性或完全互补性的核苷酸序列。引导序列可为10个-25个核苷酸的序列。在实例中,引导序列可为10个-25个核苷酸、15个-25个核苷酸或20个-25个核苷酸的序列。在另一实例中,引导序列可为10个-15个核苷酸、15个-20个核苷酸或20个-25个核苷酸的序列。此外,引导结构域可进一步包含额外核苷酸序列。额外核苷酸序列可用于改善或降低引导结构域的功能。额外核苷酸序列可用于改善或降低引导序列的功能。额外核苷酸序列可为1个-10个核苷酸的序列。在一个实例中,额外核苷酸序列可为2个-10个核苷酸、4个-10个核苷酸、6个-10个核苷酸或8个-10个核苷酸的序列。在另一实例中,额外核苷酸序列可为1-3个核苷酸、3-6个核苷酸或7-10个核苷酸的序列。在一个实施方式中,额外核苷酸序列可为1个、2个、3个、4个、5个、6个、7个、8个、9个或10个核苷酸的序列。例如,额外核苷酸序列可为一个核苷酸的序列(g(鸟嘌呤))或者两个核苷酸的序列(gg)。额外核苷酸序列可位于引导序列的5'端。额外核苷酸序列可位于引导序列的3'端。ii)第一互补结构域术语“第一互补结构域”是包含与下文描述的第二互补结构域互补的核苷酸序列的结构域,其具有足够的互补性以与第二互补结构域形成双链。例如,第一互补结构域可为与第二互补结构域具有至少50%、55%、60%、65%、70%、75%、80%、85%,90%、95%或更高的互补性或完全互补性的核苷酸序列。第一互补结构域可通过互补结合与第二互补结构域形成双链。此处,所形成的双链可通过与编辑蛋白的一些氨基酸相互作用,从而形成引导核酸-编辑蛋白复合体。第一互补结构域可为5个-35个核苷酸的序列。在实例中,第一互补结构域可为5个-35个核苷酸、10个-35个核苷酸、15个-35个核苷酸、20个-35个核苷酸、25个-35个核苷酸或30个-35个核苷酸的序列。在另一实例中,第一互补结构域可为1个-5个核苷酸、5个-10个核苷酸、10个-15个核苷酸、15个-20个核苷酸、20个-25个核苷酸、25个-30个核苷酸或30个-35个核苷酸的序列。iii)接头结构域术语“接头结构域”是连接两个以上结构域(两个以上相同或不同的结构域)的核苷酸序列。接头结构域可借助共价键或非共价键与两个以上结构域连接,或可借助共价键或非共价键连接两个以上结构域。接头结构域可为1个-30个核苷酸的序列。在一个实例中,接头结构域可为1个-5个核苷酸、5个-10个核苷酸、10个-15个核苷酸、15个-20个核苷酸、20个-25个核苷酸或25个-30个核苷酸的序列。在另一实例中,接头结构域可为1个-30个核苷酸、5个-30个核苷酸、10个-30个核苷酸、15个-30个核苷酸、20个-30个核苷酸或25个-30个核苷酸的序列。iv)第二互补结构域术语“第二互补结构域”是包含与上述第一互补结构域互补的核苷酸序列的结构域,其具有足够的互补性以与第一互补结构域形成双链。例如,第二互补结构域可为与第一互补结构域具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%或更高的互补性或完全互补性的核苷酸序列。第二互补结构域可通过互补结合与第一互补结构域形成双链。此处,所形成的双链可通过与编辑蛋白的一些氨基酸相互作用,从而形成引导核酸-编辑蛋白复合体。第二互补结构域可具有与第一互补结构域互补的核苷酸序列,以及与第一互补结构域没有互补性的核苷酸序列(例如不与第一互补结构域形成双链的核苷酸序列),并可具有比第一互补结构域更长的碱基序列。第二互补结构域可具有5个-35个核苷酸的序列。在实例中,第二互补结构域可为1个-35个核苷酸、5个-35个核苷酸、10个-35个核苷酸、15个-35个核苷酸、20个-35个核苷酸、25个-35个核苷酸或30个-35个核苷酸的序列。在另一实例中,第二互补结构域可为1个-5个核苷酸、5个-10个核苷酸、10个-15个核苷酸、15个-20个核苷酸、20个-25个核苷酸、25个-30个核苷酸或30个-35个核苷酸的序列。v)近端结构域术语“近端结构域”是其位置临近第二互补结构域的核苷酸序列。近端结构域中可具有互补核苷酸序列,可基于互补核苷酸序列形成双链。近端结构域可为1个-20个核苷酸的序列。在一个实例中,近端结构域可为1个-20个核苷酸、5个-20个核苷酸、10个-20个核苷酸或15个-20个核苷酸的序列。在另一实例中,近端结构域可为1个-5个核苷酸、5个-10个核苷酸、10-15个核苷酸或15个-20个核苷酸的序列。vi)尾部结构域术语“尾部结构域”为位于引导核酸两个末端中的一个或多个末端处的核苷酸序列。尾部结构域中可具有互补核苷酸序列,并可基于互补核苷酸序列形成双链。尾部结构域可为1个-50个核苷酸的序列。在实例中,尾部结构域可为5个-50个核苷酸、10个-50个核苷酸、15个-50个核苷酸、20个-50个核苷酸、25个-50个核苷酸、30个-50个核苷酸、35个-50个核苷酸、40个-50个核苷酸或45个-50个核苷酸的序列。在另一实例中,尾部结构域可为1个-5个核苷酸、5个-10个核苷酸、10个-15个核苷酸、15个-20个核苷酸、20个-25个核苷酸、25个-30个核苷酸、30个-35个核苷酸、35个-40个核苷酸、40个-45个核苷酸或45个-50个核苷酸的序列。同时,所述结构域(即引导结构域、第一互补结构域、接头结构域、第二互补结构域、近端结构域和尾部结构域)中包含的部分或全部核酸序列可任选地或额外地包含化学修饰。化学修饰可为但不限于甲基化、乙酰化、磷酸化、硫代磷酸酯连接、锁核酸(lna)、2'-o-甲基3'硫代磷酸酯(ms)或2'-o-甲基3'硫代pace(msp)。引导核酸包含一个或多个结构域。引导核酸可包含引导结构域。引导核酸可包含第一互补结构域。引导核酸可包含接头结构域。引导核酸可包含第二互补结构域。引导核酸可包含近端结构域。引导核酸可包含尾部结构域。此处,可以存在1、2、3、4、5、6个或更多个结构域。引导核酸可包含1、2、3、4、5、6个或更多个引导结构域。引导核酸可包含1、2、3、4、5、6个或更多个第一互补结构域。引导核酸可包含1、2、3、4、5、6个或更多个接头结构域。引导核酸可包含1、2、3、4、5、6个或更多个第二互补结构域。引导核酸可包含1、2、3、4、5、6个或更多个近端结构域。引导核酸可包含1、2、3、4、5、6个或更多个尾部结构域。此处,在引导核酸中,一种类型的结构域可以是重复的。引导核酸可包含具有或不具有重复的数个结构域。引导核酸可包含相同类型的结构域。此处,相同类型的结构域可具有相同的核酸序列或不同的核酸序列。引导核酸可包含两种类型的结构域。此处,两种不同类型的结构域可具有不同的核酸序列或相同的核酸序列。引导核酸可包含三种类型的结构域。此处,三种不同类型的结构域可具有不同的核酸序列或相同的核酸序列。引导核酸可包含四种类型的结构域。此处,四种不同类型的结构域可具有不同的核酸序列或相同的核酸序列。引导核酸可包含五种类型的结构域。此处,五种不同类型的结构域可具有不同的核酸序列或相同的核酸序列。引导核酸可包含六种类型的结构域。此处,六种不同类型的结构域可具有不同的核酸序列或相同的核酸序列。例如,引导核酸可由[引导结构域]-[第一互补结构域]-[接头结构域]-[第二互补结构域]-[接头结构域]-[引导结构域]-[第一互补结构域]-[接头结构域]-[第二互补结构域]组成。此处,两个引导结构域可包含针对不同或相同靶标的引导序列;两个第一互补结构域和两个第二互补结构域可具有相同或不同的核酸序列。当引导结构域包含针对不同靶标的引导序列时,引导核酸可与两种不同靶标特异性结合;此处,该特异性结合可以同时进行或顺序进行。此外,接头结构域可被特定的酶切割,在特定的酶的存在下,引导核酸可被分为两个或三个部分。在本说明书中公开的一个示例性实施方式中,引导核酸可为grna。术语“grna”指能够使grna-crispr酶复合体(即,crispr复合体)特异性靶向靶基因的转录调节区域中的核酸的rna。此外,grna是对靶基因的转录调节区域中的核酸而言特异的rna,其可结合至crispr酶并将crispr酶引导至靶基因的转录调节区域。grna可包含多个结构域。基于各结构域,相互作用可出现在三维结构或者grna的活性形式的链中或者这些链之间。grna可称为单链grna(单个rna分子、单个grna或sgrna)或者双链grna(包含多于一个rna分子,通常为两个独立的rna分子)。在一个示例性实施方式中,单链grna从5'至3'方向可包含引导结构域(即,包含能够与靶基因的转录调节区域的核酸形成互补结合的引导序列的结构域)、第一互补结构域、接头结构域、第二互补结构域(该结构域具有与第一互补结构域序列互补的序列,因此与第一互补结构域形成双链核酸)、近端结构域以及任选的尾部结构域。在另一实施方式中,双链grna可包含第一链和第二链,所述第一链从5'至3'方向包含引导结构域(即,包含能够与靶基因的转录调节区域的核酸形成互补结合的引导序列的结构域)以及第一互补结构域;所述第二链包含第二互补结构域(该结构域具有与第一互补结构域序列互补的序列,因此与第一互补结构域形成双链核酸)、近端结构域以及任选的尾部结构域。此处,第一链可指crrna,第二链可指tracrrna。crrna可包含引导结构域和第一互补结构域;tracrrna可包含第二互补结构域、近端结构域和任选的尾部结构域。在又一实施方式中,单链grna从5'至3'方向可包含引导结构域(即,包含能够与靶基因的转录调节区域的核酸形成互补结合的引导序列的结构域)、第一互补结构域、和第二互补结构域(该结构域具有与第一互补结构域序列互补的序列,因此与第一互补结构域形成双链核酸)。此处,第一互补结构域可与天然第一互补结构域具有同源性,或可由天然第一互补结构域衍生而来。此外,第一互补结构域可取决于天然存在的物种而在第一互补结构域的核苷酸序列中存在差异、可由天然存在的物种中含有的第一互补结构域衍生而来、或可与天然存在的物种中含有的第一互补结构域具有部分或完全同源性。在一个示例性实施方式中,第一互补结构域可与酿脓链球菌(streptococcuspyogenes)、空肠弯曲杆菌(campylobacterjejuni)、嗜热链球菌(campylobacterjejuni)、金黄色葡萄球菌(staphylococcusaureus)或脑膜炎奈瑟菌(neisseriameningitides)的第一互补结构域或由它们衍生而来的第一互补结构域具有部分(即至少50%以上)或完全同源性。例如,当第一互补结构域是酿脓链球菌的第一互补结构域或由其衍生而来的第一互补结构域时,第一互补结构域可为5′-guuuuagagcua-3′或与5′-guuuuagagcua-3′具有部分(即至少50%以上)或完全同源性的核苷酸序列。此处,第一互补结构域可进一步包含(x)n,使得其为5′-guuuuagagcua(x)n-3′。x可选自于由碱基a、t、u和g所组成的组;n可表示核苷酸数,其为5-15的整数。此处,(x)n可为相同核苷酸的n个重复,或者为n个核苷酸a、t、u和g的混合。在另一实施方式中,当第一互补结构域为空肠弯曲杆菌的第一互补结构域或由其衍生而来的第一互补结构域时,第一互补结构域可为5′-guuuuagucccuuuuuaaauuucuu-3′或5′-guuuuagucccuu-3′或者与5′-guuuuagucccuuuuuaaauuucuu-3′或5′-guuuuagucccuu-3′具有部分(即至少50%以上)或完全同源性的核苷酸序列。此处,第一互补结构域可进一步包含(x)n,使得其为5′-guuuuagucccuuuuuaaauuucuu(x)n-3′或5′-guuuuagucccuu(x)n-3′。x可选自于由核苷酸a、t、u和g所组成的组;n可表示核苷酸数,其为5-15的整数。此处,(x)n可表示相同核苷酸的n个重复,或者表示n个核苷酸a、t、u和g的混合。在另一实施方式中,第一互补结构域可与如下菌的第一互补结构域或由其衍生而来的第一互补结构域具有部分(即至少50%以上)或完全同源性:俭菌(parcubacteriabacterium)(gwc2011_gwc2_44_17)、毛螺菌(lachnospiraceaebacterium)(mc2017)、butyrivibrioproteoclasiicus、peregrinibacteriabacterium(gw2011_gwa_33_10)、氨基酸球菌属(acidaminococcussp.)(bv3l6)、猕猴卟啉单胞菌(porphyromonasmacacae)、毛螺菌(nd2006)、porphyromonascrevioricanis、解糖胨普雷沃菌(prevotelladisiens)、moraxellabovoculi(237)、smiihellasp.(sc_ko8d17)、稻田钩端螺旋体(leptospirainadai)、毛螺菌(ma2020)、新凶手弗朗西斯菌(francisellanovicida)(u112)、candidatusmethanoplasmatermitum或挑剔真杆菌(eubacteriumeligens)。例如,当第一互补结构域是俭菌的第一互补结构域或由其衍生而来的第一互补结构域时,第一互补结构域可为5'-uuuguagau-3'或与5'-uuuguagau-3'具有部分(即至少50%以上)同源性的核苷酸序列。此处,第一互补结构域可进一步包含(x)n,使得其为5'-(x)nuuuguagau-3'。x可选自于由核苷酸a、t、u和g所组成的组;n可表示核苷酸数,其为1-5的整数。此处,(x)n可表示相同核苷酸的n个重复,或者表示n个核苷酸a、t、u和g的混合。此处,接头结构域可为使得第一互补结构域与第二互补结构域连接的核苷酸序列。接头结构域可分别与第一互补结构域和第二互补结构域形成共价键或非共价键。接头结构域可将第一互补结构域与第二互补结构域共价或非共价连接。接头结构域适合用于单链grna分子中,并可用于借助共价或非共价键与双链grna的第一链和第二链连接或者连接第一链和第二链来产生单链grna。接头结构域可用于借助共价或非共价键与双链grna的crrna和tracrrna连接或者连接crrna和tracrrna来产生单链grna。此处,第二互补结构域可与天然第二互补结构域具有同源性,或可由天然第二互补结构域衍生而来。此外,第二互补结构域可取决于天然存在的物种而在第二互补结构域的核苷酸序列中存在差异、可由天然存在的物种中含有的第二互补结构域衍生而来、或可与天然存在的物种中含有的第二互补结构域具有部分或完全同源性。在示例性实施方式中,第二互补结构域可与酿脓链球菌、空肠弯曲杆菌、嗜热链球菌、金黄色葡萄球菌或脑膜炎奈瑟菌的第二互补结构域或由它们衍生而来的第二互补结构域具有部分(即至少50%以上)或完全同源性。例如,当第二互补结构域是酿脓链球菌的第二互补结构域或由其衍生而来的第二互补结构域时,第二互补结构域可为5′-uagcaaguuaaaau-3′或与5′-uagcaaguuaaaau-3′具有部分(即至少50%以上)同源性的核苷酸序列(下划线标出与第一互补结构域形成双链的核苷酸序列)。此处,第二互补结构域可进一步包含(x)n和/或(x)m,使得其为5′-(x)nuagcaaguuaaaau(x)m-3′。x可选自于由核苷酸a、t、u和g所组成的组;n和m各自可表示核苷酸数,其中n可为1-15的整数,m可为1-6的整数。此处,(x)n可表示相同核苷酸的n个重复,或者表示n个核苷酸a、t、u和g的混合。此外,(x)m可表示相同核苷酸的m个重复,或者表示m个核苷酸a、t、u和g的混合。在另一实例中,当第二互补结构域是空肠弯曲杆菌的第二互补结构域或由其衍生而来的第二互补结构域时,第二互补结构域可为5′-aagaaauuuaaaaagggacuaaaau-3′或5′-aagggacuaaaau-3′或者与5′-aagaaauuuaaaaagggacuaaaau-3′或5′-aagggacuaaaau-3′具有部分(即至少50%以上)同源性的核苷酸序列(下划线标出与第一互补结构域形成双链的核苷酸序列)。此处,第二互补结构域可进一步包含(x)n和/或(x)m,使得其为5′-(x)naagaaauuuaaaaagggacuaaaau(x)m-3′或5′-(x)naagggacuaaaau(x)m-3′。x可选自于由核苷酸a、t、u和g所组成的组;n和m各自可表示核苷酸数,其中n可为1-15的整数,m可为1-6的整数。此处,(x)n可表示相同核苷酸的n个重复,或者表示n个核苷酸a、t、u和g的混合。此外,(x)m可表示相同核苷酸的m个重复,或者表示m个核苷酸a、t、u和g的混合。在另一实施方式中,第二互补结构域可与如下菌的第二互补结构域或由其衍生而来的第二互补结构域具有部分(即至少50%以上)或完全同源性:俭菌(gwc2011_gwc2_44_17)、毛螺菌(mc2017)、butyrivibrioproteoclasiicus、peregrinibacteriabacterium(gw2011_gwa_33_10)、氨基酸球菌属(bv3l6)、猕猴卟啉单胞菌、毛螺菌(nd2006)、porphyromonascrevioricanis、解糖胨普雷沃菌、moraxellabovoculi(237)、smiihellasp.(sc_ko8d17)、稻田钩端螺旋体、毛螺菌(ma2020)、新凶手弗朗西斯菌(u112)、candidatusmethanoplasmatermitum或挑剔真杆菌。例如,当第二互补结构域是俭菌的第二互补结构域或由其衍生而来的第二互补结构域时,第二互补结构域可为5′-aaauuucuacu-3′或与5'-aaauuucuacu-7'具有部分(即至少50%以上)同源性的核苷酸序列(下划线标出与第一互补结构域形成双链的核苷酸序列)。此处,第二互补结构域可进一步包含(x)n和/或(x)m,使得其为5'-(x)naaauuucuacu(x)m-3'。x可选自于由核苷酸a、t、u和g所组成的组;n和m各自可表示核苷酸数,其中n可为1-10的整数,m可为1-6的整数。此处,(x)n可表示相同核苷酸的n个重复,或者表示n个核苷酸a、t、u和g的混合。此外,(x)m可表示相同核苷酸的m个重复,或者表示m个核苷酸a、t、u和g的混合。此处,第一互补结构域和第二互补结构域可彼此互补地结合。第一互补结构域和第二互补结构域可通过互补结合形成双链。形成的双链可与crispr酶相互作用。任选地,第一互补结构域可包含不互补结合至第二链的第二互补结构域的额外核苷酸序列。此处,额外核苷酸序列可为1个-15个核苷酸的序列。例如,额外核苷酸序列可为1个-5个核苷酸、5个-10个核苷酸或10个-15个核苷酸的序列。此处,近端结构域可为位于第二互补结构域的3'端方向的结构域。近端结构域可与天然近端结构域具有同源性,或可由天然近端结构域衍生而来。此外,近端结构域可取决于天然存在的物种而在核苷酸序列中存在差异、可由天然存在的物种中含有的近端结构域衍生而来、或可与天然存在的物种中含有的近端结构域具有部分或完全同源性。在示例性实施方式中,近端结构域可与酿脓链球菌、空肠弯曲杆菌、嗜热链球菌、金黄色葡萄球菌或脑膜炎奈瑟菌的近端结构域或由它们衍生而来的近端结构域具有部分(即至少50%以上)或完全同源性。例如,当近端结构域是酿脓链球菌的近端结构域或由其衍生而来的近端结构域时,近端结构域可为5′-aaggcuaguccg-3′或与5′-aaggcuaguccg-3′具有部分(即至少50%以上)同源性的核苷酸序列。此处,近端结构域可进一步包含(x)n,使其为5′-aaggcuaguccg(x)n-3′。x可选自于由核苷酸a、t、u和g所组成的组;n可表示核苷酸数,其可为1-15的整数。此处,(x)n可表示相同核苷酸的n个重复,或者表示n个核苷酸a、t、u和g的混合。在又一实施方式中,当近端结构域是空肠弯曲杆菌的近端结构域或由其衍生而来的近端结构域时,近端结构域可为5′-aaagaguuugc-3′或与5′-aaagaguuugc-3′具有至少50%或更高同源性的核苷酸序列。此处,近端结构域可进一步包含(x)n,使其为5′-aaagaguuugc(x)n-3′。x可选自于由核苷酸a、t、u和g所组成的组;n可表示核苷酸数,其可为1-40的整数。此处,(x)n可表示相同核苷酸的n个重复,或者表示n个核苷酸a、t、u和g的混合。此处,尾部结构域为能够被任选添加至单链grna的3′端或双链grna的第一链或第二链的结构域。尾部结构域可与天然尾部结构域具有同源性,或可由天然尾部结构域衍生而来。此外,尾部结构域可取决于天然存在的物种而在核苷酸序列中存在差异、可由天然存在的物种中含有的尾部结构域衍生而来、或可与天然存在的物种中含有的尾部结构域具有部分或完全同源性。在一个示例性实施方式中,尾部结构域可与酿脓链球菌、空肠弯曲杆菌、嗜热链球菌、金黄色葡萄球菌或脑膜炎奈瑟菌的尾部结构域或由它们衍生而来的尾部结构域具有部分(即至少50%以上)或完全同源性。例如,当尾部结构域是酿脓链球菌的尾部结构域或由其衍生而来的尾部结构域时,尾部结构域可为5′-uuaucaacuugaaaaaguggcaccgagucggugc-3′或与5′-uuaucaacuugaaaaaguggcaccgagucggugc-3′具有部分(即至少50%以上)同源性的核苷酸序列。此处,尾部结构域可进一步包含(x)n,使其为5′-uuaucaacuugaaaaaguggcaccgagucggugc(x)n-3′。x可选自于由核苷酸a、t、u和g所组成的组;n可表示核苷酸数,其为1-15的整数。此处,(x)n可表示相同核苷酸的n个重复,或者表示n个核苷酸(如a、t、u和g)的混合。在另一实例中,当尾部结构域是空肠弯曲杆菌的尾部结构域或由其衍生而来的尾部结构域时,尾部结构域可为5′-gggacucugcgggguuacaauccccuaaaaccgcuuuu-3′或与5′-gggacucugcgggguuacaauccccuaaaaccgcuuuu-3′具有部分(即至少50%以上)同源性的核苷酸序列。此处,尾部结构域可进一步包含(x)n,使其为5′-gggacucugcgggguuacaauccccuaaaaccgcuuuu(x)n-3′。x可选自于由核苷酸a、t、u和g所组成的组;n可表示核苷酸数,其为1-15的整数。此处,(x)n可表示相同核苷酸的n个重复,或者表示n个核苷酸a、t、u和g的混合。在另一实施方式中,尾部结构域可在3′端包含参与体外或体内转录方法的1nt-10nt的序列。例如,当将t7启动子用于grna的体外转录时,尾部结构域可为存在于dna模板3′端的任意核苷酸序列。此外,当将u6启动子用于体内转录时,尾部结构域可为uuuuuu;当将h1启动子用于转录时,尾部结构域可为uuuu;并且当使用pol-iii启动子时,尾部结构域可包含数个尿嘧啶核苷酸或可替代的核苷酸。grna可包含上文所述的多个结构域,因此可根据grna中含有的结构域来调整核酸序列的长度;基于各结构域,相互作用可出现在三维结构或者grna的活性形式的链中或者这些链之间。grna可指单链grna(单个rna分子)或者双链grna(包含多于一个rna分子,通常为两个独立的rna分子)。双链grna双链grna由第一链和第二链组成。此处,第一链可由5′-[引导结构域]-[第一互补结构域]-3′组成;以及第二链可由5′-[第二互补结构域]-[近端结构域]-3′或者5′-[第二互补结构域]-[近端结构域]-[尾部结构域]-3′组成。此处,第一链可以指crrna,第二链可以指tracrrna。此处,第一链和第二链可任选地包含额外核苷酸序列。在一个实例中,第一链可为5′-(n靶标)-(q)m-3′;或者5′-(x)a-(n靶标)-(x)b-(q)m-(x)c-3′。此处,n靶标是与靶基因的转录调节区域的核酸的双链的任一条链的部分序列互补的核苷酸序列,并且是可根据靶基因的转录调节区域的核酸上的靶序列进行改变的核苷酸序列区域。此处,(q)m是包含第一互补结构域的核苷酸序列,其能够与第二链的第二互补结构域形成互补结合。(q)m可为与天然存在的物种的第一互补结构域具有部分或完全同源性的序列;根据来源的物种,可对第一互补结构域的核苷酸序列进行改变。q可各自独立地选自于由a、u、c和g所组成的组;m可为核苷酸数,其为5-35的整数。例如,当第一互补结构域与酿脓链球菌的第一互补结构域或由酿脓链球菌衍生而来的第一互补结构域具有部分或完全同源性时,(q)m可为5′-guuuuagagcua-3′或与5′-guuuuagagcua-3′具有至少50%或更高同源性的核苷酸序列。在另一实例中,当第一互补结构域与空肠弯曲杆菌的第一互补结构域或由空肠弯曲杆菌衍生而来的第一互补结构域具有部分或完全同源性时,(q)m可为5′-guuuuagucccuuuuuaaauuucuu-3′或5′-guuuuagucccuu-3′或者与5′-guuuuagucccuuuuuaaauuucuu-3′或5′-guuuuagucccuu-3′具有至少50%或更高同源性的核苷酸序列。在又一实例中,当第一互补结构域与嗜热链球菌的第一互补结构域或由嗜热链球菌衍生而来的第一互补结构域具有部分或完全同源性时,(q)m可为5′-guuuuagagcuguguuguuucg-3′或与5′-guuuuagagcuguguuguuucg-3′具有至少50%或更高同源性的核苷酸序列。此外,(x)a、(x)b、(x)c各自为任选的额外核苷酸序列,其中x可各自独立地选自于由a、u、c和g所组成的组;a、b、c各自可为核苷酸数,其为0或1-20的整数。在一个示例性实施方式中,第二链可为5′-(z)h-(p)k-3′;或者5′-(x)d-(z)h-(x)e-(p)k-(x)f-3′。在另一实施方式中,第二链可为5′-(z)h-(p)k-(f)i-3′;或者5′-(x)d-(z)h-(x)e-(p)k-(x)f-(f)i-3′。此处,(z)h是包含第二互补结构域的核苷酸序列,其能够与第一链的第一互补结构域形成互补结合。(z)h可为与天然存在的物种的第二互补结构域具有部分或完全同源性的序列;根据来源的物种,可对第二互补结构域的核苷酸序列进行修饰。z可各自独立地选自于由a、u、c和g所组成的组;h可为核苷酸数,其可为5-50的整数。例如,当第二互补结构域与酿脓链球菌的第二互补结构域或由酿脓链球菌衍生而来的第二互补结构域具有部分或完全同源性时,(z)h可为5′-uagcaaguuaaaau-3′或与5′-uagcaaguuaaaau-3′具有至少50%或更高同源性的核苷酸序列。在另一实例中,当第二互补结构域与空肠弯曲杆菌的第二互补结构域或由其衍生而来的第二互补结构域具有部分或完全同源性时,(z)h可为5′-aagaaauuuaaaaagggacuaaaau-3′或5′-aagggacuaaaau-3′或者与5′-aagaaauuuaaaaagggacuaaaau-3′或5′-aagggacuaaaau-3′具有至少50%或更高同源性的核苷酸序列。在又一实例中,当第二互补结构域与嗜热链球菌的第二互补结构域或由其衍生而来的第二互补结构域具有部分或完全同源性时,(z)h可为5′-cgaaacaacacagcgaguuaaaau-3′或与5′-cgaaacaacacagcgaguuaaaau-3′具有至少50%或更高同源性的核苷酸序列。(p)k是包含近端结构域的核苷酸序列,其可与天然存在的物种的近端结构域具有部分或完全同源性;根据来源的物种,可对近端结构域的核苷酸序列进行修饰。p可各自独立地选自于由a、u、c和g所组成的组;k可为核苷酸数,其为1-20的整数。例如,当近端结构域与酿脓链球菌的近端结构域或由其衍生而来的近端结构域具有部分或完全同源性时,(p)k可为5′-aaggcuaguccg-3′或与5′-aaggcuaguccg-3′具有至少50%或更高同源性的碱基序列。在另一实例中,当近端结构域与空肠弯曲杆菌的近端结构域或由其衍生而来的近端结构域具有部分或完全同源性时,(p)k可为5′-aaagaguuugc-3′或与5′-aaagaguuugc-3′具有至少50%或更高同源性的核苷酸序列。在又一实例中,当近端结构域与嗜热链球菌的近端结构域或由其衍生而来的近端结构域具有部分或完全同源性时,(p)k可为5′-aaggcuuaguccg-3或与5′-aaggcuuaguccg-3′具有至少50%或更高同源性的核苷酸序列。(f)i可为包含尾部结构域的核苷酸序列,其可与天然存在的物种的尾部结构域具有部分或完全同源性;根据来源的物种,可对尾部结构域的核苷酸序列进行修饰。f可各自独立地选自于由a、u、c和g所组成的组;i可为核苷酸数,其为1-50的整数。例如,当尾部结构域与酿脓链球菌的尾部结构域或由其衍生而来的尾部结构域具有部分或完全同源性时,(f)i可为5′-uuaucaacuugaaaaaguggcaccgagucggugc-3′或与5′-uuaucaacuugaaaaaguggcaccgagucggugc-3′具有至少50%或更高同源性的核苷酸序列。在另一实例中,当尾部结构域与空肠弯曲杆菌的尾部结构域或由其衍生而来的尾部结构域具有部分或完全同源性时,(f)i可为5′-gggacucugcgggguuacaauccccuaaaaccgcuuuu-3′或与5′-gggacucugcgggguuacaauccccuaaaaccgcuuuu-3′具有至少50%或更高同源性的核苷酸序列。在又一实施方式中,当尾部结构域与嗜热链球菌的尾部结构域或由其衍生而来的尾部结构域具有部分或完全同源性时,(f)i可为5′-uacucaacuugaaaagguggcaccgauucgguguuuuu-3′或与5′-uacucaacuugaaaagguggcaccgauucgguguuuuu-3′具有至少50%或更高同源性的核苷酸序列。此外,(f)i可在3′端包含参与体外或体内转录方法的1-10个核苷酸的序列。例如,当将t7启动子用于grna的体外转录时,尾部结构域可为存在于dna模板3′端的任意核苷酸序列。此外,当将u6启动子用于体内转录时,尾部结构域可为uuuuuu;当将h1启动子用于转录时,尾部结构域可为uuuu;并且当使用pol-iii启动子时,尾部结构域可包含数个尿嘧啶核苷酸或可替代的核苷酸。此外,(x)d、(x)e和(x)f可为任选添加的核苷酸序列,其中x可各自独立地选自于由a、u、c和g所组成的组;d、e、f各自可为核苷酸数,其为0或1-20的整数。单链grna单链grna可分为第一单链grna和第二单链grna。第一单链grna第一单链grna为其中借助接头结构域使得双链grna的第一链或第二链连接的单链grna。具体而言,单链grna可由5′-[引导结构域]-[第一互补结构域]-[接头结构域]-[第二互补结构域]-3′,5′-[引导结构域]-[第一互补结构域]-[接头结构域]-[第二互补结构域]-[近端结构域]-3′,或5′-[引导结构域]-[第一互补结构域]-[接头结构域]-[第二互补结构域]-[近端结构域]-[尾部结构域]-3′组成。第一单链grna可任选包含额外核苷酸序列。在一个示例性实施方式中,第一单链grna可为5′-(n靶标)-(q)m-(l)j-(z)h-3′;5′-(n靶标)-(q)m-(l)j-(z)h-(p)k-3′;或者5′-(n靶标)-(q)m-(l)j-(z)h-(p)k-(f)i-3′。在另一实施方式中,第一单链grna可为5′-(x)a-(n靶标)-(x)b-(q)m-(x)c-(l)j-(x)d-(z)h-(x)e-3′;5′-(x)a-(n靶标)-(x)b-(q)m-(x)c-(l)j-(x)d-(z)h-(x)e-(p)k-(x)f-3′;或者5′-(x)a-(n靶标)-(x)b-(q)m-(x)c-(l)j-(x)d-(z)h-(x)e-(p)k-(x)f-(f)i-3′。此处,n靶标是与靶基因的转录调节区域的核酸的双链的任一条链的部分序列互补的核苷酸序列,并且是能够根据靶基因的转录调节区域上的靶序列进行改变的核苷酸序列区域。(q)m含有包含第一互补结构域的核苷酸序列,其能够与第二互补结构域形成互补结合。(q)m可为与天然存在的物种的第一互补结构域具有部分或完全同源性的序列;根据来源的物种,可对第一互补结构域的核苷酸序列进行改变。q可各自独立地选自于由a、u、c和g所组成的组;m可为核苷酸数,其可为5-35的整数。例如,当第一互补结构域与酿脓链球菌的第一互补结构域或由其衍生而来的第一互补结构域具有部分或完全同源性时,(q)m可为5′-guuuuagagcua-3′或与5′-guuuuagagcua-3′具有至少50%或更高同源性的核苷酸序列。在另一实例中,当第一互补结构域与空肠弯曲杆菌的第一互补结构域或由其衍生而来的第一互补结构域具有部分或完全同源性时,(q)m可为5′-guuuuagucccuuuuuaaauuucuu-3′或5′-guuuuagucccuu-3′或者与5′-guuuuagucccuuuuuaaauuucuu-3′或5′-guuuuagucccuu-3′具有至少50%或更高同源性的核苷酸序列。在又一实例中,当第一互补结构域与嗜热链球菌的第一互补结构域或由其衍生而来的第一互补结构域具有部分或完全同源性时,(q)m可为5′-guuuuagagcuguguuguuucg-3′或与5′-guuuuagagcuguguuguuucg-3′具有至少50%或更高同源性的核苷酸序列。此外,(l)j是包含接头结构域的核苷酸序列,它连接第一互补结构域和第二互补结构域,由此产生单链grna。此处,l可各自独立地选自于由a、u、c和g所组成的组;j可为核苷酸数,其为1-30的整数。(z)h是包含第二互补结构域的核苷酸序列,并且包含能够与第一互补结构域形成互补结合的核苷酸序列。(z)h可为与天然存在的物种的第二互补结构域具有部分或完全同源性的序列;根据来源的物种,可对第二互补结构域的核苷酸序列进行改变。z可各自独立地选自于由a、u、c和g所组成的组;h为核苷酸数,其可为5-50的整数。例如,当第二互补结构域与酿脓链球菌的第二互补结构域或由其衍生而来的第二互补结构域具有部分或完全同源性时,(z)h可为5′-uagcaaguuaaaau-3′或与5′-uagcaaguuaaaau-3′具有至少50%或更高同源性的核苷酸序列。在另一实例中,当第二互补结构域与空肠弯曲杆菌的第二互补结构域或由其衍生而来的第二互补结构域具有部分或完全同源性时,(z)h可为5′-aagaaauuuaaaaagggacuaaaau-3′或5′-aagggacuaaaau-3′或者与5′-aagaaauuuaaaaagggacuaaaau-3′或5′-aagggacuaaaau-3′具有至少50%或更高同源性的核苷酸序列。在又一实例中,当第二互补结构域与嗜热链球菌的第二互补结构域或由其衍生而来的第二互补结构域具有部分或完全同源性时,(z)h可为5′-cgaaacaacacagcgaguuaaaau-3′或与5′-cgaaacaacacagcgaguuaaaau-3′具有至少50%或更高同源性的核苷酸序列。(p)k是包含近端结构域的核苷酸序列,其可与天然存在的物种的近端结构域具有部分或完全同源性;根据来源的物种,可对近端结构域的核苷酸序列进行修饰。p可各自独立地选自于由a、u、c和g所组成的组;k可为核苷酸数,其为1-20的整数。例如,当近端结构域与酿脓链球菌的近端结构域或由其衍生而来的近端结构域具有部分或完全同源性时,(p)k可为5′-aaggcuaguccg-3′或与5′-aaggcuaguccg-3′具有至少50%或更高同源性的核苷酸序列。在另一实例中,当近端结构域与空肠弯曲杆菌的近端结构域或由其衍生而来的近端结构域具有部分或完全同源性时,(p)k可为5′-aaagaguuugc-3′或与5′-aaagaguuugc-3′具有至少50%或更高同源性的核苷酸序列。在又一实例中,当近端结构域与嗜热链球菌的近端结构域或由其衍生而来的近端结构域具有部分或完全同源性时,(p)k可为5′-aaggcuuaguccg-3′或与5′-aaggcuuaguccg-3′具有至少50%或更高同源性的核苷酸序列。(f)i可以是包含尾部结构域的核苷酸序列,其可与天然存在的物种的尾部结构域具有部分或完全同源性;根据来源的物种,可对尾部结构域的核苷酸序列进行修饰。f可各自独立地选自于由a、u、c和g所组成的组;i可为核苷酸数,其为1-50的整数。例如,当尾部结构域与酿脓链球菌的尾部结构域或由其衍生而来的尾部结构域具有部分或完全同源性时,(f)i可为5′-uuaucaacuugaaaaaguggcaccgagucggugc-3′或与5′-uuaucaacuugaaaaaguggcaccgagucggugc-3′具有至少50%或更高同源性的核苷酸序列。在另一实例中,当尾部结构域与空肠弯曲杆菌的尾部结构域或由其衍生而来的尾部结构域具有部分或完全同源性时,(f)i可为5′-gggacucugcgggguuacaauccccuaaaaccgcuuuu-3′或与5′-gggacucugcgggguuacaauccccuaaaaccgcuuuu-3′具有至少50%或更高同源性的核苷酸序列。在又一实例中,当尾部结构域与嗜热链球菌的尾部结构域或由其衍生而来的尾部结构域具有部分或完全同源性时,(f)i可为5′-uacucaacuugaaaagguggcaccgauucgguguuuuu-3′或与5′-uacucaacuugaaaagguggcaccgauucgguguuuuu-3′具有至少50%或更高同源性的核苷酸序列。此外,(f)i可在3′端包含参与体外或体内转录方法的1-10个核苷酸的序列。例如,当将t7启动子用于grna的体外转录时,尾部结构域可为存在于dna模板3'端的任意核苷酸序列。此外,当将u6启动子用于体内转录时,尾部结构域可为uuuuuu;当将h1启动子用于转录时,尾部结构域可为uuuu;并且当使用pol-iii启动子时,尾部结构域可包含数个尿嘧啶核苷酸或可替代的核苷酸。此外,(x)a、(x)b、(x)c、(x)d、(x)e和(x)f可为任选添加的核苷酸序列,其中x可各自独立地选自于由a、u、c和g所组成的组;a、b、c、d、e、f各自可为核苷酸数,其为0或1-20的整数。第二单链grna第二单链grna可为由引导结构域、第一互补结构域和第二互补结构域组成的单链grna。此处,第二单链grna可由5'-[第二互补结构域]-[第一互补结构域]-[引导结构域]-3';或者5'-[第二互补结构域]-[接头结构域]-[第一互补结构域]-[引导结构域]-3'组成。第二单链grna可任选地包含额外核苷酸序列。在一个示例性实施方式中,第二单链grna可为5'-(z)h-(q)m-(n靶标)-3';或者5'-(x)a-(z)h-(x)b-(q)m-(x)c-(n靶标)-3'。在另一实施方式中,单链grna可为5'-(z)h-(l)j-(q)m-(n靶标)-3';或者5'-(x)a-(z)h-(l)j-(q)m-(x)c-(n靶标)-3'。此处,n靶标是与靶基因的转录调节区域的核酸的双链的任一条链的部分序列互补的核苷酸序列,并且是能够根据靶基因的转录调节区域上的靶序列进行改变的核苷酸序列区域。(q)m是包含第一互补结构域的核苷酸序列,并且包含能够与第二互补结构域互补结合的核苷酸序列。(q)m可为与天然存在的物种的第一互补结构域具有部分或完全同源性的序列;根据来源的物种,可对第一互补结构域的核苷酸序列进行改变。q可各自独立地选自于由a、u、c和g所组成的组;m可为核苷酸数,其为5-35的整数。例如,当第一互补结构域与俭菌的第一互补结构域或由其衍生而来的第一互补结构域具有部分或完全同源性时,(q)m可为5′-uuuguagau-3'或与5'-uuuguagau-3'具有至少50%或更高同源性的核苷酸序列。(z)h是包含第一互补结构域的核苷酸序列,并且包含能够与第二互补结构域互补结合的核苷酸序列。(z)h可为与天然存在的物种的第二互补结构域具有部分或完全同源性的序列;根据来源的物种,可对第二互补结构域的核苷酸序列进行修饰。z可各自独立地选自于由a、u、c和g所组成的组;h可为核苷酸数,其为5-50的整数。例如,当第二互补结构域与俭菌的第二互补结构域或由俭菌衍生而来的第二互补结构域具有部分或完全同源性时,(z)h可为5′-aaauuucuacu-3′或与5'-aaauuucuacu-3'具有至少50%或更高同源性的核苷酸序列。此外,(l)j是包含接头结构域的核苷酸序列,它连接第一互补结构域和第二互补结构域。此处,l可各自独立地选自于由a、u、c和g所组成的组;j可为核苷酸数,其为1-30的整数。此外,(x)a、(x)b和(x)c各自为任选的额外核苷酸序列,其中x可各自独立地选自于由a、u、c和g所组成的组;a、b和c可为核苷酸数,其为0或1-20的整数。在本说明书的一个示例性实施方式中,引导核酸可为互补结合至重复基因的转录调节区域中的靶序列的grna。“靶序列”是靶基因的转录调节区域中存在的核苷酸序列,并且具体而言,是靶基因的转录调节区域的靶区域中的部分核苷酸序列;此处,“靶区域”是靶基因的转录调节区域中可被引导核酸-编辑蛋白修饰的区域。在下文中,靶序列可用于指两种类型的核苷酸序列信息。例如,在靶基因的情况下,靶序列可指靶基因dna的转录链的核苷酸序列信息,或非转录链的核苷酸序列信息。例如,靶序列可指靶基因a的靶区域中的部分核苷酸序列(转录链),即5′-atcattggcagactagttcg-3′,以及与其互补的核苷酸序列(非转录链),即5′-cgaactagtctgccaatgat-3′。靶序列可为5nt-50nt的序列。在一个示例性实施方式中,靶序列可为16nt的序列、17nt的序列、18nt的序列、19nt的序列、20nt的序列、21nt的序列、22nt的序列、23nt的序列、24nt的序列或25nt的序列。靶序列包含引导核酸结合序列或引导核酸非结合序列。“引导核酸结合序列”是与包含在引导核酸的引导结构域中的引导序列具有部分或完全互补性的核苷酸序列,并且可与包含在引导核酸的引导结构域中的引导序列互补结合。靶序列和引导核酸结合序列可为可根据待遗传工程化或编辑的靶标(取决于靶基因的转录调节区域)而改变的核苷酸序列,并且可根据靶基因的转录调节区域中的核酸序列被其设计为多种形式。“引导核酸非结合序列”是与包含在引导核酸的引导结构域中的引导序列具有部分或完全同源性的核苷酸序列,并且可不与包含在引导核酸的引导结构域中的引导序列互补结合。此外,引导核酸非结合序列可为与引导核酸结合序列具有互补性的核苷酸序列,并且可与引导核酸结合序列互补结合。引导核酸结合序列可为靶序列的部分核苷酸序列,其为与靶序列中包含的彼此具有不同的序列顺序的两个核苷酸序列的一个核苷酸序列,即能够彼此互补结合的两个核苷酸序列之一。此处,引导核酸非结合序列可为与引导核酸结合序列不同的靶序列的核苷酸序列。例如,当靶基因a的转录调节区域中的靶区域的部分核苷酸序列,即5′-atcattggcagactagttcg-3′,和与其互补的核苷酸序列,即5′-cgaactagtctgccaatgat-3′,用作靶序列时,引导核酸结合序列可为两个靶序列之一,即5′-atcattggcagactagttcg-3′或5′-cgaactagtctgccaatgat-3′。此处,当引导核酸结合序列为5′-atcattggcagactagttcg-3′时,引导核酸非结合序列可为5′-cgaactagtctgccaatgat-3′;或当引导核酸结合序列为5′-cgaactagtctgccaatgat-3′时,引导核酸非结合序列可为5′-atcattggcagactagttcg-3′。引导核酸结合序列可为靶序列之一,即与转录链相同的核苷酸序列以及与非转录链相同的核苷酸序列。此处,引导核酸非结合序列可为与靶序列的引导核酸结合序列不同的核苷酸序列,即选自与转录链相同的核苷酸序列和与非转录链相同的核苷酸序列的核苷酸序列。引导核酸结合序列可具有与靶序列相同的长度。引导核酸非结合序列可具有与靶序列或引导核酸结合序列相同的长度。引导核酸结合序列可为5nt-50nt的序列。在一个示例性实施方式中,引导核酸结合序列可为16nt的序列、17nt的序列、18nt的序列、19nt的序列、20nt的序列、21nt的序列、22nt的序列、23nt的序列、24nt的序列或25nt的序列。引导核酸非结合序列可为5nt-50nt的序列。在一个示例性实施方式中,引导核酸非结合序列可为16nt的序列、17nt的序列、18nt的序列、19nt的序列、20nt的序列、21nt的序列、22nt的序列、23nt的序列、24nt的序列或25nt的序列。引导核酸结合序列可部分或完全互补结合至引导核酸的引导结构域中包含的引导序列,并且引导核酸结合序列的长度可与引导序列的长度相同。引导核酸结合序列可为与引导核酸的引导结构域中包含的引导序列互补的核苷酸序列,例如具有至少70%、75%、80%、85%、90%、95%或更高的互补性或完全互补性的核苷酸序列。作为实例,引导核酸结合序列可具有或包含不与引导核酸的引导结构域中包含的引导序列互补的1nt-8nt的序列。引导核酸非结合序列可与引导核酸的引导结构域中包含的引导序列具有部分或完全同源性,并且引导核酸非结合序列的长度可与引导序列的长度相同。引导核酸非结合序列可为与引导核酸的引导结构域中包含的引导序列具有同源性的核苷酸序列,例如具有至少70%、75%、80%、85%、90%、95%或更高的同源性或完全同源性的核苷酸序列。在一个实例中,引导核酸非结合序列可具有或包含不与引导核酸的引导结构域中包含的引导序列同源的1nt-8nt的序列。引导核酸非结合序列可与引导核酸结合序列互补结合,并且引导核酸非结合序列可具有与引导核酸结合序列相同的长度。引导核酸非结合序列可为与引导核酸结合序列互补的核苷酸序列,例如具有至少90%、95%或更高的互补性或完全互补性的核苷酸序列。在一个实例中,引导核酸非结合序列可具有或包含不与引导核酸结合序列互补的1nt-2nt的序列。此外,引导核酸结合序列可为位于被编辑蛋白识别的核苷酸序列附近的核苷酸序列。在一个实例中,引导核酸结合序列可为位于临近被编辑蛋白识别的核苷酸序列的5'端和/或3'端的连续的5nt-50nt的序列。此外,引导核酸非结合序列可为位于被编辑蛋白识别的核苷酸序列附近的核苷酸序列。在一个实例中,引导核酸非结合序列可为位于临近被编辑蛋白识别的核苷酸序列的5'端和/或3'端的5nt-50nt的连续序列。“靶向”指与靶基因的转录调节区域中存在的靶序列的引导核酸结合序列互补结合。此处,互补结合可为100%完全互补结合,或70%以上且小于100%的不完全互补结合。因此,“靶向grna”是指与靶基因的转录调节区域中存在的靶序列的引导核酸结合序列互补结合的grna。本说明书中公开的靶基因可为重复基因。本说明书中公开的靶基因可为pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和/或app基因。本说明书中公开的靶基因可为癌基因。此处,癌基因可为myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和/或akt2基因。在一个示例性实施例中,本说明书中公开的靶序列可为位于重复基因的启动子区域的10nt-35nt的连续序列。此处,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。或者,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。靶序列可为10nt-35nt的序列、15nt-35nt的序列、20nt-35nt的序列、25nt-35nt的序列或30nt-35nt的序列。或者,靶序列可为10nt-15nt的序列、15nt-20nt的序列、20nt-25nt的序列、25nt-30nt的序列或30nt-35nt的序列。在一个实例中,靶序列可为位于重复基因的核心启动子区域中的10nt-25nt的连续序列。例如,靶序列可为位于包含或靠近重复基因的tss的区域中的10nt-25nt的连续序列。例如,靶序列可为位于包含或靠近重复基因的rna聚合酶结合区域的区域中的10nt-25nt的连续序列。例如,靶序列可为位于包含或靠近重复基因的转录因子结合区域的区域中的10nt-25nt的连续序列。例如,靶序列可为位于包含或靠近重复基因的tata盒的区域中的10nt-25nt的连续序列。例如,靶序列可为包含重复基因的核心启动子区域中存在的5′-tata(a/t)a(a/t)-3′序列的全部或部分的10nt-25nt的连续序列。例如,靶序列可为包含重复基因的核心启动子区域中存在的5′-tata(a/t)a(a/t)(a/g)-3′序列的全部或部分的10nt-25nt的连续序列。例如,靶序列可为10nt-25nt的连续序列,其包含选自于由以下序列所组成的组中的一个或多个序列(其存在于重复基因的核心启动子区域)的全部或部分:5′-cataaaa-3′序列、5′-cataaaa-3′序列、5′-tataa-3′序列、5′-tataaaa-3′序列、5′-cataaata-3′序列、5′-tatataa-3′序列、5′-tatatatatatataa-3′序列、5′-tatattata-3′序列、5′-tataaa-3′序列、5′-tataaaata-3′序列、5′-tatata-3′序列、5′-gattaaaaa-3′序列、5′-tataaaaa-3′序列、5′-ttataa-3′序列、5′-ttttaaaa-3′序列、5′-tctttaaaa-3′序列、5′-gacatttaa-3′序列、5′-tgatatcaa-3′序列、5′-tataaata-3′序列、5′-tataaga-3′序列、5′-aataaa-3′序列、5′-tttata-3′序列、5′-cataaaaa-3′序列、5′-tataca-3′5′-tttaaga-3′序列、5′-gataaag-3′序列、5′-tataaca-3′序列、5′-tcttatctt-3′序列、5′-ttgtacttt-3′序列、5′-catataa-3′序列、5′-tataaat-3′序列、5′-tatatataaaaaaaa-3′序列和5′-cataaataaaaaaaatta-3′序列。例如,靶序列可为包含重复基因的核心启动子区域中存在的tata结合蛋白(tbp)结合核酸序列的全部或部分的10nt-25nt的连续序列。在另一实例中,靶序列可为位于重复基因的近端启动子区域的10nt-25nt的连续序列。例如,靶序列可为位于重复基因的tss的1bp-300bp上游区域中的10nt-25nt的连续序列。在另一实例中,靶序列可为位于重复基因的远端启动子区域中的10nt-25nt的连续序列。本说明书中公开的靶序列可为位于重复基因的增强子区域中的10nt-35nt的连续序列。此处,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。或者,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。靶序列可为10nt-35nt的序列、15nt-35nt的序列、20nt-35nt的序列、25nt-35nt的序列或30nt-35nt的序列。或者,靶序列可为10nt-15nt的序列、15nt-20nt的序列、20nt-25nt的序列、25nt-30nt的序列或30nt-35nt的序列。例如,靶序列可为位于包含或靠近重复基因的增强子盒(e盒)的区域中的10nt-25nt的连续序列。例如,靶序列可为位于重复基因的内含子中存在的增强子区域中的10nt-35nt的连续序列。本说明书中公开的靶序列可为位于重复基因的绝缘子区域中的10nt-35nt的连续序列。此处,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。或者,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。靶序列可为10nt-35nt的序列、15nt-35nt的序列、20nt-35nt的序列、25nt-35nt的序列或30nt-35nt的序列。或者,靶序列可为10nt-15nt的序列、15nt-20nt的序列、20nt-25nt的序列、25nt-30nt的序列或30nt-35nt的序列。本说明书中公开的靶序列可为位于重复基因的沉默子区域中的10nt-35nt的连续序列。此处,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。或者,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。靶序列可为10nt-35nt的序列、15nt-35nt的序列、20nt-35nt的序列、25nt-35nt的序列或30nt-35nt的序列。或者,靶序列可为10nt-15nt的序列、15nt-20nt的序列、20nt-25nt的序列、25nt-30nt的序列或30nt-35nt的序列。本说明书中公开的靶序列可为位于重复基因的基因座控制区域(lcr)中的10nt-35nt的连续序列。此处,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。或者,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。靶序列可为10nt-35nt的序列、15nt-35nt的序列、20nt-35nt的序列、25nt-35nt的序列或30nt-35nt的序列。或者,靶序列可为10nt-15nt的序列、15nt-20nt的序列、20nt-25nt的序列、25nt-30nt的序列或30nt-35nt的序列。本说明书中公开的靶序列可为临近位于重复基因的转录调节区域中的前间区序列邻近基序(pam)序列的5'端和/或3'端的10nt-35nt的连续序列。此处,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。或者,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。此处,重复基因的转录调节区域可为重复基因的启动子、增强子、沉默子、绝缘子或基因座控制区域(lcr)。术语“前间区序列邻近基序(pam)序列”是可被编辑蛋白识别的核苷酸序列。此处,根据编辑蛋白的类型和编辑蛋白来源物种,pam序列可具有不同的核苷酸序列。此处,pam序列可为例如如下序列中的一种或多种(以5'至3'方向来描述):ngg(n为a、t、c或g);nnnnryac(n各自独立地为a、t、c或g;r为a或g;y为c或t);nnagaaw(n各自独立地为a、t、c或g;w为a或t);nnnngatt(n各自独立地为a、t、c或g);nngrr(t)(n各自独立地为a、t、c或g;r为a或g);以及ttn(n为a、t、c或g)。靶序列可为10nt-35nt的序列、15nt-35nt的序列、20nt-35nt的序列、25nt-35nt的序列或30nt-35nt的序列。或者,靶序列可为10nt-15nt的序列、15nt-20nt的序列、20nt-25nt的序列、25nt-30nt的序列或30nt-35nt的序列。在示例性实施方式中,当编辑蛋白识别的pam序列为5′-ngg-3′、5′-nag-3′和/或5′-nga-3′(n=a、t、g或c;或a、u、g或c)时,靶序列可为位于临近重复基因的转录调节区域中的5′-ngg-3′、5′-nag-3′和/或5′-nga-3′(n=a、t、g或c;或a、u、g或c)序列的5′端或/和3′端的10nt-25nt的连续序列。在另一示例性实施方式中,当编辑蛋白识别的pam序列为5′-nggng-3′和/或5′-nnagaaw-3(w=a或t;n=a、t、g或c,或a、u、g或c)时,靶序列可为位于临近重复基因的转录调节区域中的5′-nggng-3′和/或5′-nnagaaw-3′(w=a或t;n=a、t、g或c,或a、u、g或c)序列的5′端和/或3′端的10nt-25nt的连续序列。在另一示例性实施方式中,当编辑蛋白识别的pam序列为5′-nnnngatt-3′和/或5′-nnngctt-3′(n=a、t、g或c;或a、u、g或c)时,靶序列可为位于临近重复基因的转录调节区域中的5′-nnnngatt-3′和/或5′-nnngctt-3′(n=a、t、g或c;或a、u、g或c)序列的5′端和/或3′端的10nt-25nt的连续序列。在一个示例性实施方式中,当编辑蛋白识别的pam序列为5′-nnnvryac-3′(v=g、c或a;r=a或g;y=c或t;n=a、t、g或c,或a、u、g或c)时,靶序列可为位于临近重复基因的转录调节区域中的5′-nnnvryac-3′(v=g、c或a;r=a或g;y=c或t;n=a、t、g或c,或a、u、g或c)序列的5′端和/或3′端的10nt-25nt的连续序列。在另一示例性实施方式中,当编辑蛋白识别的pam序列为5′-naar-3′(r=a或g;n=a、t、g或c,或a、u、g或c)时,靶序列可为位于临近重复基因的转录调节区域中的5′-naar-3′(r=a或g;n=a、t、g或c,或a、u、g或c)序列的5′端和/或3′端的10nt-25nt的连续序列。在另一示例性实施方式中,当编辑蛋白识别的pam序列为5′-nngrr-3′、5′-nngrrt-3′和/或5′-nngrrv-3′(r=a或g;v=g、c或a;n=a、t、g或c,或a、u、g或c)时,靶序列可为位于临近重复基因的转录调节区域中的5′-nngrr-3′、5′-nngrrt-3′和/或5′-nngrrv-3′(r=a或g;v=g、c或a;n=a、t、g或c,或a、u、g或c)序列的5′端和/或3′端的10nt-25nt的连续序列。在一个示例性实施方式中,当编辑蛋白识别的pam序列为5′-ttn-3′(n=a、t、g或c;或a、u、g或c)时,靶序列可为位于临近重复基因的转录调节区域中的5′-ttn-3′(n=a、t、g或c;或a、u、g或c)序列的5′端和/或3′端的10nt-25nt的连续序列。下文中,表1、表2、表3、表4、表5和表6中列出了可用于本说明书中公开的示例性实施方式中的靶序列的实例。表1、表2、表3、表4、表5和表6中公开的靶序列为引导核酸非结合序列及其互补序列,即可从表中列出的序列预测引导核酸结合序列。此外,表1、表2、表3、表4、表5和表6中示出的sgrna根据编辑蛋白命名,关于spcas9命名为sp,关于cjcas9命名为cj。[表1]人pmp22基因的靶序列(关于spcas9)sgrnano.靶标(5′→3′)seqidnohpmp22-tata-sp#11ggaccagcccctgaataaacseqidno:1hpmp22-tata-sp#22ggcgtctttccagtttattcseqidno:2hpmp22-tata-sp#33gcgtctttccagtttattcaseqidno:3hpmp22-tata-sp#44cgtctttccagtttattcagseqidno:4hpmp22-tata-sp#55ttcaggggctggtccaatgcseqidno:5hpmp22-tata-sp#66tcaggggctggtccaatgctseqidno:6hpmp22-tata-sp#77accatgacatatcccagcatseqidno:7hpmp22-tata-sp#88tttccagtttattcaggggcseqidno:8hpmp22-tata-sp#99cagttacagggagcaccaccseqidno:9hpmp22-tata-sp#1010ctggtctggcttcagttacaseqidno:10hpmp22-tata-sp#1111cctggtctggcttcagttacseqidno:11hpmp22-tata-sp#1212aactggaaagacgcctggtcseqidno:12hpmp22-tata-sp#1313gaataaactggaaagacgccseqidno:13hpmp22-tata-sp#1414tccaatgctgggatatgtcaseqidno:14hpmp22-tata-sp#1515aatgctgggatatgtcatggseqidno:15hpmp22-tata-sp#1616atagaggctgagaacctctcseqidno:16hpmp22-enh-sp#117ttgggcatgtttgagctggtseqidno:17hpmp22-enh-sp#218tttgggcatgtttgagctggseqidno:18hpmp22-enh-sp#319gagctggtgggcgaagcataseqidno:19hpmp22-enh-sp#420agctggtgggcgaagcatatseqidno:20hpmp22-enh-sp#521tgggcgaagcatatgggcaaseqidno:21hpmp22-enh-sp#622ggcctccatcctaaacaatgseqidno:22hpmp22-enh-sp#1023gggttgggaggtttgggcgtseqidno:23hpmp22-enh-dp#1124aggtttgggcgtgggagtccseqidno:24hpmp22-enh-sp#1225ttcagagactcagctatttseqidno:25hpmp22-enh-sp#1326ggccacattgtttaggatgseqidno:26hpmp22-enh-sp#1427ggctttgggcatgtttgagseqidno:27hpmp22-fnh-dp#1528aacatgcccaaagcccagcsfqidno:28hpmp22-enh-sp#1629acatgcccaaagcccagcgseqidno:29hpmp22-cds-sp#130cgatgatactcagcaacaggseqidno:30hpmp22-cds-sp#331atggacacgcaactgatctcseqidno:31[表2]人pmp22基因的靶序列(关于cjcas9)sgrnano.靶标(5′→3′)seqidnohpmp22-tata-cj#11gccctctgaatctccagtcaatseqidno:32hpmp22-tata-cj#22aatctccagtcaattccaacacseqidno:33hpmp22-tata-cj#33aattaggcaattcttgtaaagcseqidno:34hpmp22-tata-cj#44ttaggcaattcttgtaaagcatseqidno:35hpmp22-tata-cj#55aaagcataggcacacatcacccseqidno:36hpmp22-tata-cj#66gcctggtctggcttcagttacaseqidno:37hpmp22-tata-cj#77gtgtccaactttgtttgctttcseqidno:38hpmp22-tata-cj#88gtattctggaaagcaaacaaagseqidno:39hpmp22-tata-cj#99cagtcttggcatcacaggcttcseqidno:40hpmp22-tata-cj#1010ggacctcttggctattacacagseqidno:41hpmp22-tata-cj#1111ggagccagtgggacctcttggcseqidno:42hpmp22-enh-cj#112taaatcacagaggcaaagagttseqidno:43hpmp22-enh-cj#213ttgcatagtgctagactgttttseqidno:44hpmp22-enh-cj#314gggtcatgtgttttgaaaacagseqidno:45hpmp22-enh-cj#415cccaaacctcccaacccacaacseqidno:46hpmp22-enh-cj#516actcagctatttctggaatgacseqidno:47hpmp22-enh-cj#617tcatcgcctttgtgagctccatseqidno:48hpmp22-enh-cj#718cagacacaggctttgctctagcseqidno:49hpmp22-enh-cj#319caaagcctgtgtctggccactaseqidno:50hpmp22-enh-cj#920agcagtttgtgcccactagtggseqidno:51hpmp22-enh-cj#1021atgtcaaggtattccagctaacseqidno:52hpmp22-enh-cj#1122gaataactgtatcaaagttagcseqidno:53hpmp22-enh-cj#1223ttcctaattaagaggctttgtgseqidno:54hpmp22-enh-cj#1324gagctagtttgtcagggtctagseqidno:55[表3]人plp1基因的靶序列(关于spcas9)[表4]人plp1基因的靶序列(关于cjcas9)[表5]小鼠plp1基因的靶序列(关于spcas9)[表6]小鼠plp1基因的靶序列(关于cjcas9)作为本说明书中公开的一方面,表达调控组合物可包含引导核酸和编辑蛋白。表达调控组合物可包含:(a)引导核酸或者编码所述引导核酸的核酸序列,所述引导核酸能够靶向位于重复基因的转录调节区域中的靶序列;以及(b)一种或多种编辑蛋白或者编码所述编辑蛋白的核酸序列。与重复基因有关的描述如上所述。与转录调节区域有关的描述如上所述。与靶序列有关的描述如上所述。表达调控组合物可包含引导核酸-编辑蛋白复合体。术语“引导核酸-编辑蛋白复合体”是指由引导核酸与编辑蛋白之间的相互作用形成的复合体。与引导核酸有关的描述如上所述。术语“编辑蛋白”指能够与核酸直接结合或无需直接结合而与核酸相互作用的肽、多肽或蛋白。此处,核酸可为靶核酸、基因或染色体中含有的核酸。此处,核酸可为引导核酸。编辑蛋白可为酶。此处,术语“酶”是指含有能够切割核酸、基因或染色体的结构域的多肽或蛋白。酶可为核酸酶或限制性酶。编辑蛋白可包括具有完全活性的酶。此处,“具有完全活性的酶”是指具有与野生型酶的核酸、基因或染色体切割功能相同的功能的酶。例如,切割双链dna的野生型酶可为将双链dna全部切割的具有完全活性的酶。作为另一实例,当切割双链dna的野生型酶由于人工工程化而经历氨基酸序列的部分序列的删除或置换时,人工工程化的酶变体与野生型酶一样切割双链dna,该人工工程化的酶变体可为具有完全活性的酶。此外,具有完全活性的酶可包括与野生型酶相比具有改善的功能的酶。例如,切割双链dna的野生型酶的特定修饰或经操纵的形式可具有高于野生型酶强的完全酶活性,即增加的切割双链dna的活性。编辑蛋白可包括具有不完全或部分活性的酶。此处,“具有不完全或部分活性的酶”是指具有野生型酶的核酸、基因或染色体切割功能的部分的酶。例如,野生型酶(切割双链dna)的特定修饰或经操纵的形式可为具有第一功能的形式或具有第二功能的形式。此处,第一功能为切割双链dna的第一链的功能,第二功能可为切割双链dna的第二链的功能。此处,具有第一功能的酶或具有第二功能的酶可为具有不完全或部分活性的酶。编辑蛋白可包括失活的酶。此处,“失活的酶”是指其中野生型酶的核酸、基因或染色体切割功能完全失活的酶。例如,野生型酶的特定修饰或经操纵的形式可为同时丧失第一功能和第二功能的形式,即切割双链dna的第一链的第一功能和切割其第二链的第二功能均丧失。此处,第一功能和第二功能均丧失的酶可为失活的酶。编辑蛋白可为融合蛋白。此处,术语“融合蛋白”是指通过将酶与额外的结构域、肽、多肽或蛋白融合而产生的蛋白。所述额外的结构域、肽、多肽或蛋白可为具有与所述酶相同或不同的功能的功能结构域、肽、多肽或蛋白。融合蛋白可处于以下形式:在酶的氨基末端或其附近、酶的羧基末端或其附近、酶的中间部分或它们的组合中的一个或多个处添加功能结构域、肽、多肽或蛋白。此处,功能结构域、肽、多肽或蛋白可为具有甲基化酶活性、去甲基化酶活性、转录激活活性、转录阻遏活性、转录释放因子活性、组蛋白修饰活性、rna切割活性或核酸结合活性的结构域、肽、多肽或蛋白,或者为用于纯化和分离蛋白(包括肽)的标签或报告基因,但本发明不限于此。功能结构域、肽、多肽或蛋白可为脱氨酶。标签包括组氨酸(his)标签、v5标签、flag标签、流感血凝素(ha)标签、myc标签、vsv-g标签和硫氧还蛋白(trx)标签;报告基因包括谷胱甘肽硫转移酶(gst)、辣根过氧化物酶(hrp)、氯霉素乙酰转移酶(cat)、β-半乳糖苷酶、β-葡萄糖醛酸酶、萤光素酶、自发荧光蛋白(包括绿色荧光蛋白(gfp)、hcred、dsred、青色荧光蛋白(cfp)、黄色荧光蛋白(yfp)和蓝色荧光蛋白(bfp)),但本发明不限于此。此外,功能结构域、肽、多肽或蛋白可为核定位序列或信号(nls)或者核输出序列或信号(nuclearexportsequenceorsignal,nes)。nls可为:具有氨基酸序列pkkkrkv的sv40病毒大t抗原的nls;由核质蛋白衍生而来的nls(例如具有序列krpaatkkagqakkkk的双分型核质蛋白(nucleoplasminbipartite)nls);具有氨基酸序列paakrvkld或rqrrnelkrsp的c-mycnls;具有序列nqssnfgpmkggnfggrssgpyggggqyfakprnqggy的hrnpa1m9nls;由输入蛋白α(importin-α)衍生而来的ibb结构域序列rmrizfknkgkdtaelrrrrvevsvelrkakkdeqilkrrnv;肌瘤t蛋白序列vsrkrprp和ppkkared;人p53序列popkkkpl;小鼠c-abliv序列salikkkkkmap;流感病毒ns1序列drlrr和pkqkkrk;肝炎病毒δ抗原序列rklkkkikkl;小鼠mx1蛋白序列rekkkflkrr;人多聚(adp-核糖)聚合酶序列krkgdevdgvdevakkkskk;或者类固醇激素受体(人)糖皮质激素序列rkclqagmnlearktkk,但本发明不限于此。额外的结构域、肽、多肽或蛋白可为不执行特定功能的非功能结构域、肽、多肽或蛋白。此处,非功能结构域、肽、多肽或蛋白可为不影响酶功能的结构域、肽、多肽或蛋白。融合蛋白可处于以下形式:在酶的氨基末端或其附近、酶的羧基末端或其附近、酶的中间部分或它们的组合中的一个或多个处添加非功能结构域、肽、多肽或蛋白。编辑蛋白可为天然的酶或融合蛋白。编辑蛋白可以以部分修饰的天然酶或融合蛋白的形式存在。编辑蛋白可为在天然状态下不存在的人工产生的酶或融合蛋白。编辑蛋白可以以在天然状态下不存在的部分修饰的人工酶或融合蛋白的形式存在。此处,修饰可为对编辑蛋白中含有的氨基酸进行置换、去除、添加或上述修饰的组合。或者,修饰可为对编码编辑蛋白的核苷酸序列中的一些核苷酸进行置换、去除、添加或或上述修饰的组合。此外,任选地,表达调控组合物可进一步包含具有待插入的期望的特定核苷酸序列的供体(donor),或编码所述供体的核酸序列。此处,待插入的核酸序列可为重复基因的转录调节区域中的部分核苷酸序列。此处,待插入的核酸序列可为用于将突变导入重复基因的转录调节区域中的核酸序列。此处,突变可为干扰重复基因的转录的突变。术语“供体”是指有助于经破坏的基因或核酸的基于同源重组(hr)的修复的核苷酸序列。供体可为双链核酸或单链核酸。供体可以线性或环状存在。供体可包含与靶基因的转录调节区域中的核酸具有同源性的核苷酸序列。例如,供体可包含与待插入特定核苷酸序列的位置(例如经破坏的核酸的上游(左侧)和下游(右侧))处的各核苷酸序列具有同源性的核苷酸序列。此处,待插入的特定核苷酸序列可位于与经破坏生物核酸的下游核苷酸序列具有同源性的核苷酸序列和与经破坏的核酸的上游核苷酸序列具有同源性的核苷酸序列之间。此处,同源的核苷酸序列可具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%或95%或更高的同源性或完全同源性。供体可包含特定的核酸序列。此处,特定的核酸序列可为靶基因的部分核苷酸序列或与其相似的核苷酸序列。靶基因的部分核苷酸序列可包含例如其中编辑了用于编辑具有突变的靶基因的突变的正常核酸序列。或者,靶基因的相似的部分核苷酸序列可包含突变诱导的核酸序列,其中用于对正常靶基因进行突变的靶基因的部分正常核酸序列的部分被修饰。此处,特定的核酸序列可为外源性核酸序列。例如,外源性核酸序列可为期望在具有靶基因的细胞中表达的外源性基因。此处,特定的核酸序列可为期望在具有靶基因的细胞中表达的核酸序列。例如,特定的核酸序列可为在具有靶基因的细胞中表达的特定基因;并且在此情况下,由于具有供体的表达调控组合物,特定基因可在细胞中的拷贝数增加,从而高度表达。任选地,供体可包含额外核苷酸序列。此处,额外核酸序列可用于增强供体的稳定性、插入靶标中的效率或同源重组效率。例如,额外核苷酸序列可为富含a和t核苷酸的核酸序列(即富a-t结构域)。例如,额外核苷酸序列可为支架/基质附着区(smar)。可以以多种方式将本说明书公开的引导核酸、编辑蛋白或引导核酸-编辑蛋白复合体递送入或导入受试者。此处,术语“受试者”是指导入引导核酸、编辑蛋白或引导核酸-编辑蛋白复合体的有机体,其中运转(operates)引导核酸、编辑蛋白或引导核酸-编辑蛋白复合体的有机体;或获取自所述有机体的试样或样本。受试者可为包含引导核酸-编辑蛋白复合体的靶基因或染色体的有机体。有机体可为动物、动物组织或动物细胞。有机体可为人、人组织或人细胞。组织可为眼球、皮肤、肝、肾、心脏、肺、脑、肌肉组织或血液。细胞可为成纤维细胞、雪旺细胞、神经细胞、少突胶质细胞、成肌细胞、胶质细胞、巨噬细胞、免疫细胞、肝细胞、视网膜色素上皮细胞、癌细胞或干细胞。试样或样本可从包含靶基因或染色体的有机体获取,并且可为唾液、血液、视网膜组织、脑组织、雪旺细胞、少突胶质细胞、成肌细胞、成纤维细胞、神经元、胶质细胞、巨噬细胞、肝细胞、免疫细胞、癌细胞或干细胞。优选地,受试者可为包含重复基因的有机体。此处,受试者可为其中的重复基因处于基因重复状态的有机体。可以以dna、rna的形式或其混合形式将引导核酸、编辑蛋白或引导核酸-编辑蛋白复合体递送入或导入受试者。此处,可通过本领域已知的方法以dna、rna或其混合物的形式将引导核酸和/或编辑蛋白递送入或导入受试者。或者,可借助载体、非载体或它们的组合将编码引导核酸和/或编辑蛋白的dna、rna或其混合物的形式递送入或导入受试者。载体可为病毒载体或非病毒载体(例如质粒)。非载体可为裸dna、dna复合体或mrna。在一个示例性实施方式中,可借助载体将编码引导核酸和/或编辑蛋白的核酸序列递送入或导入受试者。载体可包含编码引导核酸和/或编辑蛋白的核酸序列。在一个实例中,载体可同时包含分别编码引导核酸和编辑蛋白的核酸序列。在另一实例中,载体可包含编码引导核酸的核酸序列。作为实例,引导核酸中包含的结构域可全部包含于一个载体中,或可将其分割并随后包含于不同载体中。在另一实例中,载体可包含编码编辑蛋白的核酸序列。作为实例,在编辑蛋白的情况下,编码编辑蛋白的核酸序列可包含于一个载体中,或可将其分割并随后包含于数个载体中。载体可含有一种或多种调节/调控成分。此处,调节/调控成分可包括启动子、增强子、内含子、多聚腺苷酸信号、kozak共有序列、内部核糖体进入位点(ires)、剪接受体和/或2a序列。启动子可为由rna聚合酶ii识别的启动子。启动子可为由rna聚合酶iii识别的启动子。启动子可为诱导型启动子。启动子可为受试者特异性启动子。启动子可为病毒或非病毒启动子。就启动子而言,可根据控制区(即,编码引导核酸或编辑蛋白的核酸序列)而使用适当的启动子。例如,可用于引导核酸的启动子可为h1、ef-1a、trna或u6启动子。例如,可用于编辑蛋白的启动子可为cmv、ef-1a、efs、mscv、pgk或cag启动子。载体可为病毒载体或重组病毒载体。病毒可为dna病毒或rna病毒。此处,dna病毒可为双链dna(dsdna)病毒或单链dna(ssdna)病毒。此处,rna病毒可为单链rna(ssrna)病毒。病毒可为逆转录病毒、慢病毒、腺病毒、腺相关病毒(aav)、痘苗病毒、痘病毒或单纯疱疹病毒,但本发明不限于此。一般说来,病毒可感染宿主(例如细胞),由此将编码病毒遗传信息的核酸导入宿主或将编码遗传信息的核酸插入宿主基因组。可使用具有此类特征的病毒将引导核酸和/或编辑蛋白导入受试者。借助病毒导入的引导核酸和/或编辑蛋白可在受试者(例如细胞)中瞬时表达。或者,借助病毒导入的引导核酸和/或编辑蛋白可在受试者(例如细胞)中长时间持续表达(例如1、2或3周,1、2、3、6或9个月,1或2年,或永久)。根据病毒的类型,病毒的包装能力可在至少2kb至50kb间变化。取决于此类包装能力,可设计包含引导核酸或编辑蛋白的病毒载体或者包含引导核酸和编辑蛋白二者的病毒载体。或者,可设计包含引导核酸、编辑蛋白和额外成分的病毒载体。在一个实例中,可借助重组慢病毒对编码引导核酸和/或编辑蛋白的核酸序列进行递送或导入。在另一实例中,可借助重组腺病毒对编码引导核酸和/或编辑蛋白的核酸序列进行递送或导入。在又一实例中,可借助重组aav对编码引导核酸和/或编辑蛋白的核酸序列进行递送或导入。在又一实例中,可借助混合病毒(例如本文列出的病毒中的一种以上的混合)对编码引导核酸和/或编辑蛋白的核酸序列进行递送或导入。在另一示例性实施方式中,可借助非载体将编码引导核酸和/或编辑蛋白的核酸序列递送入或导入受试者。非载体可包含编码引导核酸和/或编辑蛋白的核酸序列。非载体可为裸dna、dna复合体、mrna或它们的混合物。可借助电穿孔、基因枪、声致穿孔(sonoporation)、磁性转染、瞬时细胞压缩或挤压(transientcellcompressionorsqueezing)(例如在文献“lee等(2012)nanolett.,12,6322-6327”中所记载的)、脂质介导的转染、树枝状大分子、纳米粒子、磷酸钙、二氧化硅、硅酸盐(ormosil)或它们的组合将非载体递送入或导入受试者。在一个实例中,可通过将细胞与编码引导核酸和/或编辑蛋白的核酸序列在卡盒(cartridge)、腔室(chamber)或比色皿(cuvette)中混合并以预定的持续时间和振幅将电刺激施加至细胞来实施借助电穿孔的递送。在另一实例中,可使用纳米粒子实施非载体递送。纳米粒子可为无机纳米粒子(例如磁性纳米粒子、二氧化硅等)或有机纳米粒子(例如聚乙二醇(peg)包覆的脂质等)。纳米粒子的外表面可缀合有能够进行附着的带正电的聚合物(例如聚乙烯亚胺、聚赖氨酸、聚丝氨酸等)。在某些实施方式中,可使用脂质壳递送非载体。在某些实施方式中,可使用外泌体(exosome)递送非载体。外泌体是用于转移蛋白和rna的内源性纳米囊泡,可将rna递送至脑和另一靶器官。在某些实施方式中,可使用脂质体递送非载体。脂质体为球形囊泡结构,由围绕内部水室的单个或多个层状脂质双层以及相对不透明的外部亲脂磷脂双层组成。尽管脂质体可由数种不同类型的脂质制成,最常使用磷脂来生产作为药物运载体的脂质体。此外,用于非载体递送的组合物可包含其它添加剂。可将编辑蛋白以肽、多肽或蛋白的形式递送入或导入受试者。可借助本领域已知的方法以肽、多肽或蛋白的形式将编辑蛋白递送入或导入受试者。可借助电穿孔、显微注射、瞬时细胞压缩或挤压(例如在文献“lee等(2012)nanolett.,12,6322-6327”中所记载的)、脂质介导的转染、纳米粒子、脂质体、肽介导的递送或它们的组合将肽、多肽或蛋白形式递送入或导入受试者。肽、多肽或蛋白可与编码引导核酸的核酸序列一起递送。在一个实例中,可通过将待导入编辑蛋白的细胞与引导核酸一起(或不与引导核酸一起)在卡盒、腔室或比色皿中混合并以预定的持续时间和振幅将电刺激施加至细胞来实施借助电穿孔的递送。可将引导核酸和编辑蛋白以核酸和蛋白混合的形式递送入或导入受试者。可以以引导核酸-编辑蛋白复合体的形式将引导核酸和编辑蛋白递送入或导入受试者。例如,引导核酸可为dna、rna或其混合物。编辑蛋白可为肽、多肽或蛋白。在一个实例中,可以以包含rna型引导核酸和蛋白型编辑蛋白的引导核酸-编辑蛋白复合体(即核糖核蛋白(rnp))的形式将引导核酸和编辑蛋白递送入或导入受试者。本说明书公开的引导核酸-编辑蛋白复合体可对靶核酸、基因或染色体进行修饰。例如,引导核酸-编辑蛋白复合体诱导靶核酸、基因或染色体的序列的修饰。作为结果,可对由靶核酸、基因或染色体表达的蛋白的结构和/或功能进行修饰,或可调控或抑制该蛋白的表达。引导核酸-编辑蛋白复合体可在dna、rna、基因或染色体水平发挥作用。在一个实例中,引导核酸-编辑蛋白复合体可操纵或修饰靶基因的转录调节区域,来对靶基因编码的蛋白的表达进行调控(例如抑制、阻遏、降低、增加或促进)、或表达其活性受到调控(例如抑制、阻遏、降低、增加或促进)或修饰的蛋白。引导核酸-编辑蛋白复合体可在基因转录和翻译阶段发挥作用。在一个实例中,引导核酸-编辑蛋白复合体可促进或阻遏靶基因的转录,从而调控(例如抑制、阻遏、降低、增加或促进)靶基因编码的蛋白的表达。在另一实例中,引导核酸-编辑蛋白复合体可促进或阻遏靶基因的翻译,从而调控(例如抑制、阻遏、降低、增加或促进)靶基因编码的蛋白的表达。在本说明书中公开的一个示例性实施方式中,表达调控组合物可包含grna和crispr酶。表达调控成分可包含以下:(a)grna或者编码所述grna的核酸序列,所述grna可靶向位于重复基因的转录调节区域中的靶序列;以及(b)一种或多种crispr酶或者编码所述crispr酶的核酸序列。与重复基因有关的描述如上所述。与转录调节区域有关的描述如上所述。与靶序列有关的描述如上所述。表达调控组合物可包含grna-crispr酶复合体。术语“grna-crispr酶复合体”是指由grna和crispr酶之间的相互作用形成的复合体。与grna有关的描述如上所述。术语“crispr酶”是crispr-cas系统的主要蛋白组分,并且与grna形成复合体,从而形成crispr-cas系统。crispr酶可为具有编码crispr酶的序列的核酸或多肽(或蛋白)。crispr酶可为ii型crispr酶。根据对两种以上类型的天然微生物ii型crispr酶分子的研究(jinek等,science,343(6176):1247997,2014)以及对酿脓链球菌cas9(spcas9)与grna复合的研究(nishimasu等,cell,156:935-949,2014;和anders等,nature,2014,doi:10.1038/nature13579)确定了ii型crispr酶的晶体结构。ii型crispr酶包含两个叶(lobes),即识别(rec)叶和核酸酶(nuc)叶,并且各叶包含数个结构域。rec叶包含富含精氨酸的螺旋桥(bh)结构域、rec1结构域和rec2结构域。此处,bh结构域为长的α螺旋并且为富含精氨酸的区域,而rec1结构域和rec2结构域在识别grna(例如单链grna、双链grna或tracrrna)中形成的双链中起重要作用。nuc叶包含ruvc结构域、hnh结构域和pam相互作用(pi)结构域。此处,ruvc结构域涵盖ruvc样结构域,hnh结构域涵盖hnh样结构域。此处,ruvc结构域与具有ii型crispr酶的天然存在的微生物家族的成员共享结构相似性,并切割单链(例如靶基因的转录调节区域中的核酸的非互补链,即不与grna形成互补结合的链)。在本领域中,ruvc结构域有时指ruvci结构域、ruvcii结构域或ruvciii结构域,一般称为ruvci、ruvcii或ruvciii。hnh结构域与hnh核酸内切酶共享结构相似性,并切割单链(例如靶核酸分子的互补链,即与grna形成互补结合的链)。hnh结构域位于ruvcii和ruvciii基序之间。pi结构域识别靶基因的转录调节区域中的特定核苷酸序列(即前间区序列邻近基序(pam))或与pam相互作用。此处,pam可根据ii型crispr酶的来源而变化。例如,当crispr酶是spcas9时,pam可为5′-ngg-3';当crispr酶是嗜热链球菌cas9(stcas9)时,pam可为5'-nnagaaw-3'(w=a或t);当crispr酶是脑膜炎奈瑟菌cas9(nmcas9)时,pam可为5'-nnnngatt-3';当crispr酶是空肠弯曲杆菌cas9(cjcas9)时,pam可为5′-nnnvryac-3′(v=g或c或a;r=a或g;y=c或t),其中,n为a、t、g或c,或a、u、g或c。然而,尽管通常理解根据如上所述的酶的来源来确定pam,pam可随着由相应来源衍生而来的酶的突变体的研究进展而改变。ii型crispr酶可为cas9。cas9可由多种微生物衍生而来,例如,酿脓链球菌(streptococcuspyogenes)、嗜热链球菌(streptococcusthermophilus)、链球菌属(streptococcussp.)、金黄色葡萄球菌(staphylococcusaureus)、空肠弯曲杆菌、达松维尔拟诺卡式菌(nocardiopsisdassonvillei)、始旋链霉菌(streptomycespristinaespiralis)、产绿色链霉菌(streptomycesviridochromogenes)、产绿色链霉菌、粉红链孢囊菌(streptosporangiumroseum)、粉红链孢囊菌、酸热脂环酸芽孢杆菌(alicyclobachlusacidocaldarius)、假真菌样芽孢杆菌(bacilluspseudomycoides)、bacillusselenitireducens、exiguobacteriumsibiricum、德氏乳杆菌(lactobacillusdelbrueckii)、唾液乳杆菌(lactobacillussalivarius)、microscillamarina、burkholderialesbacterium、polaromonasnaphthalenivorans、极单孢菌属(polaromonassp.)、crocosphaerawatsonii、蓝杆藻属(cyanothecesp.)、铜绿微囊藻(microcystisaeruginosa)、聚球藻属(synechococcussp.)、阿拉伯糖醋杆菌(acetohalobiumarabaticum)、ammonifexdegensii、caldicelulosiruptorbescii、candidatusdesulforudis、肉毒梭状芽胞杆菌(clostridiumbotulinum)、艰难梭状芽胞杆菌(clostridiumdifficile)、微小微单胞菌(finegoldiamagna)、natranaerobiusthermophilus、pelotomaculumthermopropionicum、喜温嗜酸硫杆菌(acidithiobacilluscaldus)、嗜酸氧化亚铁硫杆菌(acidithiobacillusferrooxidans)、allochromatiumvinosum、海杆菌属(marinobactersp.)、nitrosococcushalophilus、nitrosococcuswatsonii、pseudoalteromonashaloplanktis、ktedonobacterracemifer、methanohalobiumevestigatum、多变鱼腥藻(anabaenavariabilis)、泡沫节球藻(nodulariaspumigena)、念珠藻属(nostocsp.)、极大节旋藻(arthrospiramaxima)、钝顶节旋藻(arthrospiraplatensis)、节旋藻属(arthrospirasp.)、鞘丝藻属(lyngbyasp.)、原型微鞘藻(microcoleuschthonoplastes)、颤藻属(oscillatoriasp.)、运动石袍菌(petrotogamobilis)、非洲栖热腔菌(thermosiphoafricanus)以及acaryochlorismarina。cas9为与grna结合从而切割或修饰靶基因的转录调节区域上的靶序列或位置的酶,cas9可由hnh结构域(能够切割与grna形成互补结合的核酸链)、ruvc结构域(能够切割与grna形成非互补结合的核酸链)、rec结构域(与靶标相互作用)以及pi结构域(识别pam)组成。对于cas9的具体结构特征,可参见hiroshinishimasu等,(2014)cell156:935-949。cas9可从天然存在的微生物中分离或者通过重组或合成方法非天然地产生。此外,crispr酶可为v型crispr酶。v型crispr酶包含类似的ruvc结构域(对应于ii型crispr酶的ruvc结构域),并可由nuc结构域(而不是ii型crispr酶的hnh结构域)、rec结构域和wed结构域(识别靶标)以及pi结构域(识别pam)组成。对于v型crispr酶的具体结构特征,可参见takashiyamano等(2016)cell165:949-962。v型crispr酶可与grna相互作用,从而形成grna-crispr酶复合体,即crispr复合体,并且可在grna的协作下允许引导序列接近包含pam序列的靶序列。此处,v型crispr酶与靶基因的转录调节区域中的核酸相互作用的能力依赖于pam序列。pam序列可为靶基因的转录调节区域中存在的序列,其被v型crispr酶的pi结构域识别。pam序列可根据v型crispr酶的来源而具有不同的序列。即每个种类具有能够被特异性识别的pam序列。例如,由cpf1识别的pam序列可为5′-ttn-3′(n为a、t、c或g)。然而,尽管通常理解pam根据如上所述的酶的来源来确定,但pam可随着由相应来源衍生而来的酶的突变体的研究进展而改变。v型crispr酶可为cpf1。cpf1可为由以下衍生而来:链球菌(streptococcus)、弯曲杆菌(campylobacter)、nitratifractor、葡萄球菌(staphylococcus)、parvibaculum、罗斯氏菌(roseburia)、奈瑟菌(neisseria)、葡糖醋杆菌(gluconacetobacter)、固氮螺菌(azospirillum)、sphaerochaeta、乳杆菌(lactobacillus)、真杆菌(eubacterium)、棒状杆菌(corynebacter)、肉食杆菌(carnobacterium)、红细菌(rhodobacter)、李斯特菌(listeria)、paludibacter、梭菌(clostridium)、毛螺菌(lachnospiraceae)、clostridiaridium、纤毛菌(leptotrichia)、弗朗西斯氏菌属(francisella)、军团杆菌(legionella)、脂环酸芽孢杆菌(alicyclobacillus)、methanomethyophilus、卟啉单胞菌(porphyromonas)、普雷沃菌(prevotella)、拟杆菌(bacteroidetes)、创伤球菌(helcococcus)、钩端螺旋体(letospira)、脱硫弧菌(desulfovibrio)、desulfonatronum、丰佑菌(opitutaceae)、肿块芽孢杆菌(tuberibacillus)、芽孢杆菌(bacillus)、短芽孢杆菌(brevibacilus)、甲基杆菌(methylobacterium)或氨基酸球菌(acidaminococcus)。cpf1可由ruvc样结构域(对应于cas9的ruvc结构域)、nuc结构域(而不是cas9的hnh结构域)、rec结构域和wed结构域(识别靶标)以及pi结构域(识别pam)组成。对于cpf1的具体结构特征,可参见takashiyamano等(2016)cell165:949-962。cpf1可从天然存在的微生物分离或者通过重组或合成方法非天然地产生。crispr酶可为具有切割靶基因的转录调节区域中的双链核酸的功能的核酸酶或限制性酶。crispr酶可为具有完全活性的crispr酶。术语“具有完全活性”是指酶具有与野生型crispr酶相同的功能的状态,此状态下的crispr酶被称为具有完全活性的crispr酶。此处,“野生型crispr酶的功能”是指酶具有切割双链dna的功能的状态,即具有切割双链dna的第一链的第一功能和切割双链dna的第二链的第二功能的状态。具有完全活性的crispr酶可为切割双链dna的野生型crispr酶。具有完全活性的crispr酶可为通过对切割双链dna的野生型crispr酶进行修饰或操纵形成的crispr酶变体。crispr酶变体可为其中野生型crispr酶的氨基酸序列的一个或多个氨基酸被置换为其它氨基酸或一个或多个氨基酸去除的酶。crispr酶变体可为野生型crispr酶的氨基酸序列添加了一个或多个氨基酸的酶。此处,添加的氨基酸的位置可为野生型酶的n端、c端或氨基酸序列。crispr酶变体可为与野生型crispr酶相比具有改善的功能的具有完全活性的酶。例如,野生型crispr酶的特定修饰或经操纵的形式(即crispr酶变体)可切割双链dna而不与待切割的双链dna结合或与其保持一定距离。在这种情况下,修饰或经操纵的形式可为与野生型crispr酶相比具有改善的功能活性的具有完全活性的crispr酶。crispr酶变体可为与野生型crispr酶相比功能降低的具有完全活性的酶。例如,野生型crispr酶的特定修饰或经操纵的形式(即crispr酶变体)可在非常接近待切割的双链dna或与其形成特异性结合的情况下切割双链dna。此处,特异性结合可为例如crispr酶的特定区域处的氨基酸与切割位置处的dna序列之间的结合。在这种情况下,修饰或经操纵的形式可为与野生型crispr酶相比功能活性降低的具有完全活性的crispr酶。crispr酶可为具有不完全或部分活性的crispr酶。术语“具有不完全或部分活性”是指酶具有选自野生型crispr酶的功能(即,切割双链dna的第一链的第一功能,以及切割双链dna的第二链的第二功能)之一的状态。处于此状态下的crispr酶称为具有不完全或部分活性的crispr酶。此外,具有不完全或部分活性的crispr酶可称为切口酶(nickase)。术语“切口酶”是指经操纵或修饰而仅切割靶基因的转录调节区域中的核酸的双链的一条链的crispr酶,切口酶具有切割单链(例如与靶基因的转录调节区域中的核酸的grna互补的链或不与其互补的链)的核酸酶活性。因此,为了切割双链需要两种切口酶的核酸酶活性。切口酶可具有ruvc结构域的核酸酶活性。即,切口酶可不包含hnh结构域的核酸酶活性,为此可对hnh结构域进行操纵或修饰。在一个实例中,当crispr酶是ii型crispr酶时,切口酶可为包含修饰的hnh结构域的ii型crispr酶。例如,在ii型crispr酶为野生型spcas9的情况下,切口酶可为其中通过突变使hnh结构域的核酸酶活性失活的spcas9变体,所述突变为将野生型spcas9的氨基酸序列中的第840位氨基酸由组氨酸突变为丙氨酸。由于由此产生的切口酶(即spcas9变体)具有ruvc结构域的核酸酶活性,因此它能够切割靶基因的转录调节区域中的核酸的非互补链,即不与grna形成互补结合的链。又例如,在ii型crispr酶为野生型cjcas9的情况下,切口酶可为其中通过突变使hnh结构域的核酸酶活性失活的cjcas9变体,所述突变为野生型cjcas9的氨基酸序列中的第559位氨基酸由组氨酸突变为丙氨酸。由于由此产生的切口酶(即cjcas9变体)具有ruvc结构域的核酸酶活性,因此它能够切割靶基因的转录调节区域中的核酸的非互补链,即不与grna形成互补结合的链。此外,切口酶可具有通过crispr酶的hnh结构域的核酸酶活性。即,切口酶可不包含ruvc结构域的核酸酶活性,为此可对ruvc结构域进行操纵或修饰。在一个实例中,当crispr酶是ii型crispr酶时,切口酶可为包含经修饰的ruvc结构域的ii型crispr酶。例如,在ii型crispr酶为野生型spcas9的情况下,切口酶可为其中通过突变使ruvc结构域的核酸酶活性失活的spcas9变体,所述突变为野生型spcas9的氨基酸序列中的第10位氨基酸由天冬氨酸突变为丙氨酸。由于由此产生的切口酶(即spcas9变体)具有hnh结构域的核酸酶活性,因此它能够切割靶基因的转录调节区域中的核酸的互补链,即与grna形成互补结合的链。又例如,在ii型crispr酶为野生型cjcas9的情况下,切口酶可为其中通过突变使ruvc结构域的核酸酶活性失活的cjcas9变体,所述突变为野生型cjcas9的氨基酸序列中的第8位氨基酸由天冬氨酸突变为丙氨酸。由于由此产生的切口酶(即cjcas9变体)具有hnh结构域的核酸酶活性,因此它能够切割靶基因的转录调节区域中的核酸的互补链,即与grna形成互补结合的链。crispr酶可为失活的crispr酶。术语“失活”是指野生型crispr酶的功能(即切割双链dna的第一链的第一功能和切割双链dna的第二链的第二功能)均丧失的状态。处于此类状态下的crispr酶被称为失活的crispr酶。由于野生型crispr酶的具有核酸酶活性的结构域中的突变,失活的crispr酶可不具有核酸酶活性。失活的crispr酶可能由于ruvc结构域和hnh结构域的突变而不具有核酸酶活性。即,失活的crispr酶可不具有由crispr酶的ruvc结构域和hnh结构域生成的核酸酶活性,并且为此可对ruvc结构域和hnh结构域进行操纵或修饰。在一个实例中,当crispr酶为ii型crispr酶时,失活的crispr酶可为具有修饰的ruvc结构域和hnh结构域的ii型crispr酶。例如,当ii型crispr酶为野生型spcas9时,失活的crispr酶可为其中通过野生型spcas9的氨基酸序列中的第10位天冬氨酸和第840位组氨酸均突变为丙氨酸而使ruvc结构域和hnh结构域的核酸酶活性均失活的spcas9变体。此处,由于所产生的失活的crispr酶(即spcas9变体)中ruvc结构域和hnh结构域的核酸酶活性失活,可完全不切割靶基因的转录调节区域中的双链核酸。在另一实例中,当ii型crispr酶为野生型cjcas9时,失活的crispr酶可为其中通过野生型cjcas9的氨基酸序列中的第8位天冬氨酸和第559位组氨酸均突变为丙氨酸而使ruvc结构域和hnh结构域的核酸酶活性均失活的cjcas9变体。此处,由于所产生的失活的crispr酶(即cjcas9变体)中ruvc结构域和hnh结构域的核酸酶活性失活,可完全不切割靶基因的转录调节区域中的双链核酸。除上述核酸酶活性外,crispr酶可具有解旋酶活性,即,使得双链核酸的螺旋结构解旋的能力。此外,可对crispr酶进行修饰以使其具有完全的、不完全或部分的、或失活的解旋酶活性。crispr酶可为通过对野生型crispr酶进行人工操纵或修饰产生的crispr酶变体。crispr酶变体可为经人工操纵或修饰的crispr酶变体以修饰野生型crispr酶的功能(即,切割双链dna的第一链的第一功能,和/或切割双链dna的第二链的第二功能)。例如,crispr酶变体可为其中野生型crispr酶功能的第一功能丧失的形式。或者,crispr酶变体可为其中野生型crispr酶功能的第二功能丧失的形式。例如,crispr酶变体可为其中野生型crispr酶的两种功能(即第一功能和第二功能)均丧失的形式。crispr酶变体可通过与grna相互作用而形成grna-crispr酶复合体。crispr酶变体可为经人工操纵或修饰以修饰野生型crispr酶的与grna相互作用的功能的crispr酶变体。例如,crispr酶变体可为具有与野生型crispr酶相比降低的与grna的相互作用的形式。或者,crispr酶变体可为具有与野生型crispr酶相比增加的与grna的相互作用的形式。例如,crispr酶变体可为具有野生型crispr酶的第一功能以及降低的与grna的相互作用的形式。或者,crispr酶变体可为具有野生型crispr酶的第一功能以及增加的与grna的相互作用的形式。例如,crispr酶变体可为具有野生型crispr酶的第二功能以及降低的与grna的相互作用的形式。或者,crispr酶变体可为具有野生型crispr酶的第二功能以及增加的与grna的相互作用的形式。例如,crispr酶变体可为不具有野生型crispr酶的第一功能和第二功能并具有降低的与grna的相互作用的形式。或者,crispr酶变体可为不具有野生型crispr酶的第一功能和第二功能并具有增加的与grna的相互作用的形式。此处,可根据grna与crispr酶变体之间的相互作用强度来形成多种grna-crispr酶复合体,并且接近或切割靶序列的功能可根据crispr酶变体而存在差异。例如,仅当非常接近或定位于与grna完全互补结合的靶序列时,由与grna的相互作用降低的crispr酶变体形成的grna-crispr酶复合体才能够切割靶序列的双链或单链。crispr酶变体可为其中野生型crispr酶的氨基酸序列的至少一个氨基酸被修饰的形式。作为实例,crispr酶变体可为其中野生型crispr酶的氨基酸序列的至少一个氨基酸被置换的形式。作为另一实例,crispr酶变体可为其中野生型crispr酶的氨基酸序列的至少一个氨基酸被删除的形式。作为又一实例,crispr酶变体可为对野生型crispr酶的氨基酸序列的至少一个氨基酸进行添加的形式。在一个实例中,crispr酶变体可为其中野生型crispr酶的氨基酸序列的至少一个氨基酸被置换、删除和/或添加的形式。此外,任选地,除了野生型crispr酶的原始功能(即,切割双链dna的第一链的第一功能以及切割其第二链的第二功能)外,crispr酶变体可进一步包含功能结构域。此处,除了野生型crispr酶的原始功能外,crispr酶变体可具有额外功能。功能结构域可为具有甲基化酶活性、去甲基化酶活性、转录激活活性、转录阻遏活性、转录释放因子活性、组蛋白修饰活性、rna切割活性或核酸结合活性的结构域,或者为用于分离和纯化蛋白(包括肽)的标签或报告基因,但本发明不限于此。标签包括组氨酸(his)标签、v5标签、flag标签、流感血凝素(ha)标签、myc标签、vsv-g标签和硫氧还蛋白(trx)标签;报告基因包括谷胱甘肽-s-转移酶(gst)、辣根过氧化物酶(hrp)、氯霉素乙酰转移酶(cat)、β-半乳糖苷酶、β-葡萄糖醛酸酶、萤光素酶、自发荧光蛋白(包括绿色荧光蛋白(gfp)、hcred、dsred、青色荧光蛋白(cfp)、黄色荧光蛋白(yfp)和蓝色荧光蛋白(bfp)),但本发明不限于此。功能结构域可为脱氨酶。例如,具有不完全或部分活性的crispr酶可进一步包含胞苷脱氨酶作为功能结构域。在一个示例性实施方式中,可将胞苷脱氨酶(例如载脂蛋白b编辑复合体1(apobec1))添加至spcas9切口酶,从而产生融合蛋白。如上所述形成的[spcas9切口酶]-[apobec1]可用于由c到t或u的核苷酸编辑或者由g到a的核苷酸编辑中。在另一实例中,具有不完全或部分活性的crispr酶可进一步包含腺嘌呤脱氨酶作为功能结构域。在一个示例性实施方式中,可将腺嘌呤脱氨酶(例如tada变体、adar2变体或adat2变体等)添加至spcas9切口酶,从而产生融合蛋白。如上所述形成的[spcas9切口酶]-[tada变体]、[spcas9切口酶]-[adar2变体]或[spcas9切口酶]-[adat2变体]可用于由a到g的核苷酸编辑或者由t到c的核苷酸编辑中,由于融合蛋白将核苷酸a修饰为肌苷,修饰的肌苷被聚合酶识别为核苷酸g,从而实质上表现出由a到g的核苷酸编辑。功能结构域可为核定位序列或信号(nls)或者核输出序列或信号(nes)。在一个实例中,crispr酶可包含一个或多个nls。此处,一个或多个nls可包含于cripsr酶的n端或其附近、所述酶的c端或其附近,或者它们的组合。nls可为由如下nls衍生而来的nls序列,但本发明不限于此:具有氨基酸序列pkkkrkv的sv40病毒大t抗原的nls;来自核质蛋白的nls(例如具有序列krpaatkkagqakkkk的双分型核质蛋白nls);具有氨基酸序列paakrvkld或rqrrnelkrsp的c-mycnls;具有序列nqssnfgpmkggnfggrssgpyggggqyfakprnqggy的hrnpa1m9nls;来自输入蛋白α的ibb结构域的序列rmrizfknkgkdtaelrrrrvevsvelrkakkdeqilkrrnv;肌瘤t蛋白的序列vsrkrprp和ppkkared;人p53的序列popkkkpl;小鼠c-abliv的序列salikkkkkmap;流感病毒ns1的序列drlrr和pkqkkrk;肝炎病毒δ抗原的序列rklkkkikkl;小鼠mx1蛋白的序列rekkkflkrr;人多聚(adp-核糖)聚合酶的序列krkgdevdgvdevakkkskk;或者由类固醇激素受体(人)糖皮质激素的序列衍生而来的nls序列rkclqagmnlearktkk。此外,crispr酶突变体可包括通过将crispr酶分为两个以上部分而制备的拆分型(split-type)crispr酶。术语“拆分”是指对蛋白进行功能或结构性划分,或者将蛋白随机划分为两个以上部分。拆分型crispr酶可为具有完全活性的酶、具有不完全或部分活性的酶或失活的酶。例如,当crispr酶为spcas9时,可在第656位残基(酪氨酸)和第657位残基(苏氨酸)之间将spcas9分为两部分来生成拆分型spcas9。拆分型crispr酶可任选包含用于重构(reconstitution)的额外的结构域、肽、多肽或蛋白。用于重构的额外的结构域、肽、多肽或蛋白可组装以形成在结构上与野生型crispr酶相同或类似的拆分型crispr酶。用于重构的额外的结构域、肽、多肽或蛋白可为frb和fkbp二聚化结构域;内含肽(intein);ert和vpr结构域;或者在特定条件下形成异二聚体的结构域。例如,当crispr酶为spcas9时,可在第713位残基(丝氨酸)和第714位残基(甘氨酸)之间将spcas9分为两部分,从而生成拆分型spcas9。可将frb结构域连接至两部分中的一个部分,并将fkbp结构域连接至另一部分。在由此产生的拆分型spcas9中,frb结构域和fkbp结构域可以在存在雷帕霉素的环境中形成二聚体,从而生成重构的crispr酶。本说明书中公开的crispr酶或crispr酶变体可为多肽、蛋白或者具有编码所述多肽、蛋白的序列的核酸,并可针对待导入所述crispr酶或crispr酶变体的受试者实施密码子优化。术语“密码子优化”是指对核酸序列的修饰过程,该修饰过程通过在保持天然氨基酸序列的同时将天然序列中的至少一个密码子替换为在宿主细胞中更常或最常使用的密码子来改善在宿主细胞中的表达。多种物种对特定氨基酸的特定密码子具有特定偏好,该密码子偏好(不同生物体间密码子使用的差别)通常与mrna的翻译效率相关,认为这取决于所翻译的密码子的特征和特定trna分子的可获得性。细胞中选择的优势trna通常反映了肽合成中最常使用的密码子。因此,可基于密码子优化在给定生物体中通过优化基因表达来对基因进行定制化。可以借助多种递送方法和多种形式将本说明书中公开的grna、crispr酶或grna-crispr酶复合体递送入或导入受试者。关于受试者的描述如上所述。在一个示例性实施方式中,可通过载体将编码grna和/或crispr酶的核酸序列递送入或导入受试者。载体可包含编码grna和/或crispr酶的核酸序列。在一个实例中,载体可同时包含编码grna和crispr酶的核酸序列。在另一实例中,载体可包含编码grna的核酸序列。例如,grna中包含的结构域可包含在一个载体中,或者可将其分割并随后包含在不同载体中。在另一实例中,载体可包含编码crispr酶的核酸序列。例如,在crispr酶的情况下,编码crispr酶的核酸序列可包含在一个载体中,或者可将其分割并随后包含在数个载体中。载体可包含一种或多种调节/调控成分。此处,调节/调控成分可包括:启动子、增强子、内含子、多聚腺苷酸信号、kozak共有序列、内部核糖体进入位点(ires)、剪接受体和/或2a序列。启动子可为由rna聚合酶ii识别的启动子。启动子可为由rna聚合酶iii识别的启动子。启动子可为诱导型启动子。启动子可为受试者特异性启动子。启动子可为病毒启动子或非病毒启动子。就启动子而言,可根据控制区(即,编码grna和/或crispr酶的核酸序列)而使用恰当的启动子。例如,可用于grna的启动子可为h1、ef-1a、trna或u6启动子。例如,可用于crispr酶的启动子可为cmv、ef-1a、efs、mscv、pgk或cag启动子。载体可为病毒载体或重组病毒载体。病毒可为dna病毒或rna病毒。此处,dna病毒可为双链dna(dsdna)病毒或单链dna(ssdna)病毒。此处,rna病毒可为单链rna(ssrna)病毒。所述病毒可为逆转录病毒、慢病毒、腺病毒、腺相关病毒(aav)、痘苗病毒、痘病毒或单纯疱疹病毒,但本发明不限于此。在一个实例中,可通过重组慢病毒对编码grna和/或crispr酶的核酸序列进行递送或导入。在另一实例中,可通过重组腺病毒对编码grna和/或crispr酶的核酸序列进行递送或导入。在又一实例中,可通过重组aav对编码grna和/或crispr酶的核酸序列进行递送或导入。在又一实例中,可通过混合病毒(例如本文所述的病毒中的一种以上的混合)对编码grna和/或crispr酶的核酸序列进行递送或导入。在一个示例性实施方式中,可将grna-crispr酶复合体递送入或导入受试者。例如,grna可以以dna、rna或其混合物的形式存在。crispr酶可以以肽、多肽或蛋白的形式存在。在一个实例中,可以以包含rna型grna和蛋白型crispr的grna-crispr酶复合体(即核糖核蛋白(rnp))的形式将grna和crispr酶递送入或导入受试者。可借助电穿孔、显微注射、瞬时细胞压缩或挤压(例如在文献“lee等(2012)nanolett.,12,6322-6327”中所描述的)、脂质介导的转染、纳米粒子、脂质体、肽介导的递送或它们的组合将grna-crispr酶复合体递送入或导入受试者。本说明书中公开的grna-crispr酶复合体可用于对靶基因(即重复基因的转录调节区域)进行人工操纵或修饰。可使用上述所述grna-crispr酶复合体(即crispr复合体)对靶基因的转录调节区域进行操纵或修饰。此处,靶基因的转录调节区域的操纵或修饰可包括以下二者:i)对靶基因进行切割或损伤;以及ii)对损伤的转录调节区域进行修复。i)靶基因的转录调节区域的切割或损伤可为使用crispr复合体对靶基因的转录调节区域进行切割或损伤,具体而言对转录调节区域中的靶序列进行切割或损伤。靶序列可成为grna-crispr酶复合体的靶标,并且靶序列可包含由crispr酶识别的pam序列或者可不包含由crispr酶识别的pam序列。此类靶序列可为参与grna设计的人提供关键标准。靶序列可由grna-crispr酶复合体的grna特异性识别,因此,grna-crispr酶复合体可位于所识别的靶序列附近。靶位点处的“切割”是指多聚核苷酸共价骨架的断裂。切割包括但不限于磷酸二酯键的酶促水解或化学水解,但本发明不限于此。除此之外,可通过多种方法进行切割。单链切割和双链切割都是可能的,其中双链切割可以作为两条不同的单链切割的结果而发生。双链切割可产生平末端或交错(staggered)末端(或粘性末端)。在一个实例中,使用crispr复合体对靶基因的转录调节区进行切割或损伤可为将靶序列的双链完全切割或损伤。在一个示例性实施方式中,当crispr酶为野生型spcas9时,与grna形成互补结合的靶序列的双链可被crispr复合体完全切割。在另一示例性实施方式中,当crispr酶为spcas9切口酶(d10a)和spcas9切口酶(h840a)时,与grna形成互补结合的靶序列的两条单链可分别被每种crispr复合体切割。即,spcas9切口酶(d10a)可切割与grna形成互补结合的靶序列的互补单链,而spcas9切口酶(h840a)可切割与grna形成互补结合的靶序列的非互补单链,并且可顺序或同时实施切割。在另一实例中,使用crispr复合体对靶基因的转录调节区域进行切割或损伤可为仅对靶序列的双链中的单链进行切割或损伤。此处,单链可为与grna互补结合的靶序列中的引导核酸结合序列(即互补单链),或不与grna互补结合的引导核酸非结合序列(即与grna非互补的单链)。在一个示例性实施方式中,当crispr酶为spcas9切口酶(d10a)时,crispr复合体可通过spcas9切口酶(d10a)切割与grna互补结合的靶序列的引导核酸结合序列(即互补单链),而可不切割不与grna互补结合的非引导核酸结合序列(即与grna非互补的单链)。在另一示例性实施方式中,当crispr酶为spcas9切口酶(h840a)时,crispr复合体可通过spcas9切口酶(h840a)切割与grna不互补结合的靶序列的非引导核酸结合序列(即与grna非互补的单链),而可不切割与grna互补结合的靶序列的引导核酸结合序列(即互补单链)。在又一实例中,使用crispr复合体对靶基因的转录调节区域的切割或损伤可为部分去除核酸片段。在一个示例性实施方式中,当crispr复合体由野生型spcas9和具有不同靶序列的两个grna组成时,可切割与第一grna形成互补结合的靶序列的双链,并可切割与第二grna形成互补结合的靶序列的双链,从而导致借助第一grna和第二grna以及spcas9去除核酸片段。例如,当两个crispr复合体由与不同靶序列互补结合的两个grna(例如,与增强子的上游存在的靶序列互补结合的一个grna,以及与增强子的下游存在的靶序列互补结合的另一grna)以及野生型spcas9组成时,可切割与第一grna互补结合的增强子的上游存在的靶序列的双链,并可切割与第二grna互补结合的增强子的下游存在的靶序列的双链,从而借助第一grna、第二grna以及spcas9去除核酸片段(即增强子区域)。ii)损伤的转录调节区域的修复可为借助非同源末端接合(nhej)和同源介导的修复(hdr)进行的修复或恢复。非同源末端接合(nhej)是通过将被切割的双链或单链的两端进行连接对dna中的双链断裂进行恢复或修复的方法,一般而言,当将通过双链断裂(例如切割)形成的两个相容末端持续彼此接触使得两个末端完全相接,损伤的双链得以修复。nhej是能够用于整个细胞周期的恢复方法,并且通常在细胞中无同源基因组用作模板时(例如g1期)发生。在使用nhej对损伤的基因或核酸进行修复的过程中,nhej修复区中的核酸序列出现一些插入和/或删除(插入缺失,indel),此类插入和/或删除导致读码框位移,产生移码的转录组mrna。其结果是,由于无义介导的衰变(nonsense-mediateddecay)或正常蛋白无法合成,造成固有功能丧失。此外,即使读码框保持不变,序列中相当数量的插入或删除造成的突变也可导致蛋白功能破坏。由于相比蛋白中非重要区域的突变,对重要功能结构域中的突变可能耐受性更低,突变为基因座依赖型的。由于不能预测在天然状态下由nhej产生的插入缺失突变,特定的插入缺失序列优选位于指定的受损区中,并可来自于微同源的小区域。常规地,删除的长度范围为1bp-50bp,插入趋向于更短,并通常包含直接包围受损区的短重复序列。此外,nhej是造成突变的过程,当不必须生成特定的最终序列时,可将nhej用于对短序列基序进行删除。可使用该nhej进行crispr复合体所靶向的表达受转录调节区域调控的基因的特异性敲除。可使用crispr酶(例如cas9或cpf1)切割靶基因的转录调节区域的双链或两条单链,并可借助nhej使得受损的双链或两条单链具有插入缺失,从而诱导表达受转录调节区域调控的靶基因的特异性敲除。在一个实例中,可使用crispr复合体切割靶基因的转录调节区域的双链,并且可通过借助nhej的修复在修复区生成多种插入缺失(插入和删除)。术语“插入缺失”是指通过在dna的核苷酸序列中插入部分核苷酸或从dna的核苷酸序列中删除部分核苷酸而形成的变异。当grna-crispr酶复合体如上所述切割靶基因的转录调节区域中的靶序列时,在通过hdr或nhej进行修复期间可在靶序列中引入插入缺失。同源定向修复(hdr)是无错修正方法,其使用同源序列作为模板对损伤的转录调节区域进行修复或恢复,一般而言,为修复或恢复受损dna(即,恢复细胞的固有信息),利用未被修饰的互补核苷酸序列的信息或者姐妹染色单体的信息对受损dna进行修复。hdr最常见的类型为同源重组(hr)。hdr是通常出现在活跃分裂的细胞的s期或g2/m期的修复或恢复方法。为使用hdr而不使用细胞的姐妹染色单体或互补核苷酸序列对损伤的dna进行修复或恢复,可将使用互补核苷酸序列或同源核苷酸序列的信息人工合成的dna模板(即,包含互补核苷酸序列或同源核苷酸序列的核酸模板)提供至细胞,从而修复受损dna。此处,当进一步将核酸序列或核酸片段添加至核酸模板来修复受损dna时,可将进一步添加至受损dna的核酸序列或核酸片段敲入。进一步添加的核酸序列或核酸片段可为对由正常基因的突变修饰的靶基因的转录调节区域进行修正的核酸序列或核酸片段,或者为待在细胞中表达的基因或核酸,但本发明不限于此。在一个实例中,可使用crispr复合体切割靶基因的转录调节区域的双链或单链,可将核酸模板(该核酸模板包含与临近切割位点的核苷酸序列互补的核苷酸序列)提供至细胞,并可借助hdr修复或恢复靶基因的转录调节区域的被切割的核苷酸序列。此处,包含互补核苷酸序列的核酸模板可具有受损dna(即,被切割的双链或单链)的互补核苷酸序列,并进一步包含期望被插入至受损dna的核酸序列或核酸片段。可使用包含互补核苷酸序列和待插入的核酸序列或核酸片段的核酸模板将额外核酸序列或核酸片段插入至受损dna(即,靶基因的转录调节区域的切割位点)。此处,待插入的核酸序列或核酸片段以及额外核酸序列或核酸片段可为对由正常基因的突变修饰的靶基因的转录调节区域进行修正的核酸序列或核酸片段,或者为待在细胞中表达的基因或核酸。互补核苷酸序列可为与受损dna具有互补结合的核苷酸序列(即,靶基因的转录调节区域被切割的双链或单链的右侧和左侧的核苷酸序列)。或者,互补核苷酸序列可为与受损dna具有互补结合的核苷酸序列(即,靶基因的转录调节区域被切割的双链或单链的3'和5'端)。互补核苷酸序列可为15nt-3000nt的序列,可根据靶基因的转录调节区域或核酸模板的大小对互补核苷酸序列的长度或大小进行适当设计。此处,核酸模板可为双链或单链的核酸,并可为线性或环状,但本发明不限于此。在另一实例中,使用crispr复合体切割靶基因的转录调节区域的双链或单链,将核酸模板(该核酸模板包含临近切割位点的核苷酸序列的同源核苷酸序列)提供至细胞,并可通过hdr修复或恢复靶基因的转录调节区域被切割的核苷酸序列。此处,包含同源核苷酸序列的核酸模板可具有受损dna(即,被切割的双链或单链)的同源核苷酸序列,并进一步包含待插入至受损dna的核酸序列或核酸片段。可使用包含同源核苷酸序列和待插入的核酸序列或核酸片段的核酸模板将额外核酸序列或核酸片段插入至受损dna(即,靶基因的转录调节区域的切割位点)。此处,待插入的核酸序列或核酸片段以及额外核酸序列或核酸片段可为对由正常基因的突变修饰的靶基因的转录调节区域进行修正的核酸序列或核酸片段,或者为待在细胞中表达的基因或核酸。同源核苷酸序列可为与受损dna具有同源性的核苷酸序列(即,转录调节区域被切割的双链的右侧和左侧的核苷酸序列)。或者,同源核苷酸序列可为与受损dna具有同源性的核苷酸序列(即,转录调节区域被切割的双链或单链的3'和5'端)。同源核苷酸序列可为15nt-3000nt的序列,可根据靶基因的转录调节区域或核酸模板的大小对同源核苷酸序列的长度或大小进行适当设计。此处,核酸模板可为双链或单链的核酸,并可为线性或环状,但本发明不限于此。除了nhej和hdr,存在多种对损伤的转录调节区域进行修复或恢复的方法。例如,对损伤的转录调节区域进行修复或恢复的方法可为单链退火、单链断裂修复、错配修复、核苷酸切割修复或使用核苷酸切割修复的方法。单链退火(ssa)是对靶核酸中存在的两个重复序列间的双链断裂进行修复的方法,一般使用多于30个核苷酸的重复序列。对重复序列进行切割(以产生粘性末端),从而在靶核酸双链的各断裂端产生单链,并且在切割后用rpa蛋白对含有重复序列的单链垂悬部分(overhang)进行包覆,来防止重复序列彼此的不适当退火。rad52结合至垂悬部分上的各重复序列,并排列能够对互补重复序列进行退火的序列。退火后,垂悬部分的单链悬垂(flap)被切割,合成新dna来填充特定缺口,从而恢复dna双链。该修复的结果是两个重复间的dna序列被删除,并且删除长度可取决于多种因素(包括本文使用的两个重复的位置以及切割过程的路径或程度)。就对靶核酸序列进行修饰或修正而言,与hdr类似,ssa利用互补序列(即互补重复序列);与hdr不同,ssa不需要核酸模板。可通过单链断裂修复(ssbr,与上述修复机制不同的机制)对基因组中的单链断裂进行修复。在单链dna断裂的情况下,parp1和/或parp2识别断裂并动员修复机制。parp1对dna断裂的结合和活性是暂时的,通过促进损伤区域中ssbr蛋白复合体的稳定性来促进ssbr。ssbr复合体中最重要的蛋白是xrcc1,它与促进dna的3'和5'端加工的蛋白相互作用来稳定dna。末端加工通常涉及将损伤的3'端修复为羟基化状态和/或将损伤的5'端修复为磷酸部分,并在末端加工后发生dna缺口填充。存在两种dna缺口填充方法,即短补丁(patch)修复和长补丁修复,而短补丁修复涉及单核苷酸的插入。在dna缺口填充后,dna连接酶促进末端连接。错配修复(mmr)作用于错配的dna核苷酸。msh2/6或msh2/3复合体各自具有atpase活性,并因此在识别错配和引发修复中起到重要作用,并且msh2/6主要识别核苷酸-核苷酸错配并识别一个或两个核苷酸的错配,而msh2/3主要识别更长的错配。碱基切除修复(ber)是在整个细胞周期中均活跃的修复方法,其用于从基因组中去除较小的非螺旋扭曲碱基损伤区。在损伤的dna中,通过切割连接核苷酸与脱氧核糖-磷酸骨架的n-糖苷键去除损伤的核苷酸,随后切割磷酸二酯键骨架,从而生成单链dna断裂。去除由此形成的受损单链末端,并利用新的互补核苷酸填充由于单链去除而造成的缺口,随后通过dna连接酶将新填充的互补核苷酸的末端连接至骨架,引起对损伤dna的修复或恢复。核苷酸切除修复(ner)是对于从dna中去除大的螺旋扭曲损伤而言重要的切除机制,当识别到损伤时,去除含有损伤区域的短单链dna片段,产生22-30个核苷酸的单链缺口。利用新的互补碱基填充生成的缺口,并通过dna连接酶将新填充的互补核苷酸的末端与骨架连接,引起对损伤dna的修复或恢复。通过grna-crispr复合体对靶基因的转录调节区域进行人工操纵的效果可主要为敲除、敲减、敲入和增加的表达。术语“敲除”是指靶基因或核酸的失活,而“靶基因或核酸的失活”是指不发生靶基因或核酸的转录和/或翻译的状态。通过敲除可对造成疾病的基因或具有异常功能的基因的转录和翻译进行抑制,阻止蛋白表达。例如,当使用grna-crispr酶复合体(即crispr复合体)对靶基因的转录调节区域进行编辑时,可使用crispr复合体对靶基因的转录调节区域进行切割。可通过nhej对使用crispr复合体损伤的转录调节区域进行修复。nhej在损伤的转录调节区域中生成了插入缺失,因此损伤的转录调节区域失活,从而诱导靶基因或染色体的特异性敲除。在另一实例中,当使用grna-crispr酶复合体(即crispr复合体)和供体对靶基因的转录调节区域进行编辑时,可使用crispr复合体对靶基因的转录调节区域进行切割。由crispr复合体损伤的转录调节区域可使用供体借助hdr进行修复。此处,供体包含同源核苷酸序列和期望插入的核苷酸序列。此处,可根据插入的位置或目的来对待插入的核苷酸序列的数目进行改变。当使用供体对损伤的转录调节区域进行修复时,将待插入的核苷酸序列插入到损伤的核苷酸序列区域,因此转录调节区域失活,从而诱导靶基因或染色体的特异性敲除。术语“敲减”是指靶基因或核酸的转录和/或翻译或靶蛋白的表达降低。通过借助敲减对基因或蛋白的过表达进行调节,可预防发病或可治疗疾病。例如,当使用grna-crispr酶复合体(即crispr复合体)对靶基因的转录调节区域进行编辑时,可使用crispr复合体对靶基因的转录调节区域进行切割。可通过nhej对使用crispr复合体损伤的转录调节区域进行修复。nhej在损伤的转录调节区域中生成了插入缺失,因此损伤的转录调节区域失活,从而诱导靶基因或染色体的特异性敲减。在另一实例中,当使用grna-crispr酶复合体(即crispr复合体)和供体对靶基因的转录调节区域进行编辑时,可使用crispr复合体对靶基因的转录调节区域进行切割。使用crispr复合体损伤的转录调节区域可使用供体借助hdr进行修复。此处,供体包含同源核苷酸序列和期望插入的核苷酸序列。此处,可根据插入的位置或目的来对待插入的核苷酸序列的数目进行改变。当使用供体对损伤的转录调节区域进行修复时,将待插入的核苷酸序列插入到损伤的核苷酸序列区域,因此转录调节区域失活,从而诱导靶基因或染色体的特异性敲除。例如,当使用grna-crispr失活酶-转录抑制活性结构域复合体(即,包含转录抑制活性结构域的crispr失活复合体)对靶基因的转录调节区域进行编辑时,crispr失活复合体可特异性地结合至靶基因的转录调节区域,并通过crispr失活复合体中包含的转录抑制活性结构域对转录调节区域的活性进行抑制,从而诱导敲减(其中靶基因或染色体的表达被抑制)。术语“敲入”是指将特定核酸或基因插入至靶基因或核酸,此处,“特定核酸或基因”是指待插入或待表达的感兴趣的核酸或基因。借助敲入通过修正至正常或插入正常基因来诱导正常基因的表达,可将造成疾病的突变基因用于疾病治疗。此外,敲入可进一步需要供体。例如,当使用grna-crispr酶复合体(即crispr复合体)和供体对靶基因或核酸进行编辑时,可使用crispr复合体对靶基因或核酸进行切割。可使用供体借助hdr对使用crispr复合体损伤的靶基因或核酸进行修复。此处,供体可包含特定核酸或基因,并可用于将特定核酸或基因插入至损伤的基因或染色体。此处,插入的特定核酸或基因可诱导蛋白的表达。“增加的表达”是指与人工操纵前相比,靶基因或核酸的转录和/或翻译或靶蛋白的表达增加。可通过调控表达不足或非表达的基因或蛋白的表达来预防或治疗疾病。例如,当使用grna-crispr酶复合体(即crispr复合体)对靶基因的转录调节区域进行编辑时,可使用crispr复合体对靶基因的转录调节区域进行切割。使用crispr复合体损伤的转录调节区域可由nhej进行修复。nhej在损伤的转录调节区域中生成了插入缺失,从而增加了转录调节区域的活性并诱导了正常靶基因或染色体的表达。在本说明书中公开的一个示例性实施方式中,grna-crispr酶复合体可将人工操纵或修饰添加至重复基因的转录调节区域。grna-crispr酶复合体可特异性识别重复基因的转录调节区域中的靶序列。此处,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。或者,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。转录调节区域的描述如上所述。靶序列可被grna-crispr酶复合体的grna特异性识别,因此,grna-crispr酶复合体可位于所识别的靶序列附近。靶序列可为重复基因的转录调节区域中发生人工修饰的位点或区域。靶序列的描述如上所述。在一个示例性实施方式中,靶序列可为表1、表2、表3和表4中列出的一个或多个核苷酸序列。grna-crispr酶复合体可由grna和crispr酶组成。grna可包含能够与重复基因的转录调节区域中的靶序列的引导核酸结合序列部分或完全互补结合的引导结构域。引导结构域可与引导核酸结合序列具有至少70%、75%、80%、85%、90%、95%或更高的互补性或完全互补性。引导结构域可包含与重复基因的转录调节区域中的靶序列的引导核酸结合序列互补的核苷酸序列。此处,互补的核苷酸序列可包含0-5、0-4、0-3或0-2个错配。grna可包含选自于由第一互补结构域、接头结构域、第二互补结构域、近端结构域和尾部结构域所组成的组中的一个或多个结构域。crispr酶可为选自于由以下所组成的组中的一种或多种:酿脓链球菌衍生而来的cas9蛋白、空肠弯曲杆菌衍生而来的cas9蛋白、嗜热链球菌衍生而来的cas9蛋白、金黄色葡萄球菌衍生而来的cas9蛋白、脑膜炎奈瑟菌衍生而来的cas9蛋白以及cpf1蛋白。在一个实例中,编辑蛋白可为空肠弯曲杆菌衍生而来的cas9蛋白或金黄色葡萄球菌衍生而来的cas9蛋白。grna-crispr酶复合体可将多种人工操纵或修饰添加至重复基因的转录调节区域。重复基因的经人工操纵或修饰的转录调节区域可包含对位于靶序列中或临近靶序列的5'端和/或3'端的1bp-50bp的连续核苷酸序列区域的以下一个或多个修饰:i)一个或多个核苷酸的缺失;ii)将一个或多个核苷酸置换为不同于野生型基因的核苷酸;iii)一个或多个核苷酸的插入;或iv)选自于i)-iii)的两种以上的组合。例如,重复基因的经人工操纵或修饰的转录调节区域可在位于靶序列或临近靶序列的5'端和/或3'端的1bp-50bp连续核苷酸序列区域中包含一个或多个核苷酸的删除。在一个实例中,删除的核苷酸可为1个、2个、3个、4个或5个连续或非连续碱基对。在另一实例中,删除的核苷酸可为由2bp以上连续核苷酸组成的核苷酸片段。此处,核苷酸片段可为2个-5个、6个-10个、11个-15个、16个-20个、21个-25个、26个-30个、31个-35个、36个-40个、41个-45个或46个-50个碱基对。在另一实例中,删除的核苷酸可为两个以上核苷酸片段。此处,两个以上核苷酸片段可为各自具有非连续核苷酸序列(即一个或多个核苷酸序列缺口)的核苷酸片段,并且由于两个以上的删除的核苷酸片段而可具有两个以上删除位点。或者,例如,重复基因的经人工操纵或修饰的转录调节区域可在位于靶序列或临近靶序列的5'端和/或3'端的1bp-50bp连续核苷酸序列区域中包含一个或多个核苷酸的插入。在一个实例中,插入的核苷酸可为1个、2个、3个、4个或5个连续碱基对。在另一实例中,插入的核苷酸可为由5个以上连续碱基对组成的核苷酸片段。此处,核苷酸片段可为5个-10个、11个-50个、50个-100个、100个-200个、200个-300个、300个-400个、400个-500个、500个-750个或750个-1000个碱基对。在又一实例中,插入的核苷酸可为特定基因的部分或全部核苷酸序列。此处,特定基因可为受试者(例如人细胞)中不包含的从外部输入的具有重复基因的基因。或者,特定基因可为受试者(例如人细胞)中包含的具有重复基因的基因,例如人细胞的基因组中存在的基因。或者,例如,重复基因的经人工操纵或修饰的转录调节区域可在位于靶序列或临近靶序列的5'端和/或3'端的1bp-50bp的连续核苷酸序列区域中包含一个或多个核苷酸的删除和插入。在一个实例中,删除的核苷酸可为1个、2个、3个、4个或5个连续或非连续碱基对。此处,插入的核苷酸可为1个、2个、3个、4个或5个碱基对,核苷酸片段,或特定基因的部分或全部核苷酸序列;删除和插入可顺序发生或同时发生。此处,插入的核苷酸片段可为5个-10个、11个-50个、50个-100个、100个-200个、200个-300个、300个-400个、400个-500个、500个-750个或750个-1000个碱基对。此处,特定基因可为从受试者(例如人细胞)外部输入的具有重复基因的基因。或者,特定基因可为受试者(例如人细胞)中包含的具有重复基因的基因,例如人细胞的基因组中存在的基因。在另一实例中,删除的核苷酸可为由2个碱基对或以上组成的核苷酸片段。此处,删除的核苷酸片段可为2个-5个、6个-10个、11个-15个、16个-20个、21个-25个、26个-30个、31个-35个、36个-40个、41个-45个或46个-50个碱基对。此处,插入的核苷酸可为1个、2个、3个、4个或5个碱基对,核苷酸片段,或特定基因的部分或全部核苷酸序列;并且删除和插入可顺序发生或同时发生。在又一实例中,删除的核苷酸可为两个以上核苷酸的片段。此处,插入的核苷酸可为1个、2个、3个、4个或5个碱基对,核苷酸片段,或特定基因的部分或全部核苷酸序列;并且删除和插入可顺序发生或同时发生。此外,插入可发生在被删除的两个以上位点的部分或全部中。根据grna和crispr酶的类型,grna-crispr酶复合体可将多种人工操纵或修饰添加至重复基因的转录调节区域。在一个实例中,当crispr酶为spcas9蛋白时,经人工操纵或修饰的重复基因的转录调节区域可在位于临近靶区域中存在的5'-ngg-3'(n为a、t、g或c)pam序列的5'端和/或3'端的1bp-50bp、1bp-40bp、1bp-30bp、或优选1bp-25bp的连续核苷酸序列区域中包含以下修饰中的一个或多个:i)一个或多个核苷酸的删除;ii)将一个或多个核苷酸置换为不同于野生型基因的核苷酸;iii)一个或多个核苷酸的插入;或iv)选自于i)-iii)中的两种以上的组合。在另一实例中,当crispr酶为cjcas9蛋白时,经人工操纵或修饰的重复基因的转录调节区域可在位于临近靶序列中存在的5′-nnnnryac-3′(n各自独立地为a、t、c或g;r为a或g;y为c或t)pam序列的5'端和/或3'端的1bp-50bp、1bp-40bp、1bp-30bp、或优选1bp-25bp的连续核苷酸序列区域中包含以下修饰中的一种或多种:i)一个或多个核苷酸的删除;ii)将一个或多个核苷酸置换为不同于野生型基因的核苷酸;iii)一个或多个核苷酸的插入;或iv)选自于i)-iii)中的两种以上的组合。在又一实例中,当crispr酶为stcas9蛋白时,经人工操纵或修饰的重复基因的转录调节区域可在位于临近靶序列中存在的5'-nnagaaw-3'(n各自独立地为a、t、c或g;w为a或t)pam序列的5'端和/或3'端的1bp-50bp、1bp-40bp、1bp-30bp、或优选1bp-25bp的连续核苷酸序列区域中包含以下修饰中的一种或多种:i)一个或多个核苷酸的删除;ii)将一个或多个核苷酸置换为不同于野生型基因的核苷酸;iii)一个或多个核苷酸的插入;或iv)选自于i)-iii)中的两种以上的组合。在一个实例中,当crispr酶为nmcas9蛋白时,经人工操纵或修饰的重复基因的转录调节区域可在位于临近靶序列中存在的5'-nnnngatt-3'(n各自独立地为a、t、c或g)pam序列的5'端和/或3'端的1bp-50bp、1bp-40bp、1bp-30bp、或优选1bp-25bp的连续核苷酸序列区域中包含以下修饰中的一种或多种:i)一个或多个核苷酸的删除;ii)将一个或多个核苷酸置换为不同于野生型基因的核苷酸;iii)一个或多个核苷酸的插入;或iv)选自于i)-iii)中的两种以上的组合。在另一实例中,当crispr酶为sacas9蛋白时,经人工操纵或修饰的重复基因的转录调节区域可在位于临近靶序列中存在的5'-nngrr(t)-3'(n各自独立地为a、t、g或c;r为a或g;并且任选包含(t))pam序列的5'端和/或3'端的1bp-50bp、1bp-40bp、1bp-30bp、或优选1bp-25bp连续核苷酸序列区域中包含以下修饰中的一个或多个:i)一个或多个核苷酸的删除;ii)将一个或多个核苷酸置换为不同于野生型基因的核苷酸;iii)一个或多个核苷酸的插入;或iv)选自于i)-iii)中的两种以上的组合。在又一实例中,当crispr酶为cpf1蛋白时,经人工操纵或修饰的重复基因的转录调节区域可在位于临近靶序列中存在的5′-ttn-3'(n为a、t、c或g)pam序列的5'端和/或3'端的1bp-50bp、1bp-40bp、1bp-30bp、或优选1bp-25bp的连续核苷酸序列区域中包含以下修饰中一个或多个:i)一个或多个核苷酸的删除;ii)将一个或多个核苷酸置换为不同于野生型基因的核苷酸;iii)一个或多个核苷酸的插入;或iv)选自于i)-iii)中的两种以上的组合。通过grna-crispr酶复合体人工操纵重复基因的转录调节区域的效果可为敲除。可通过grna-crispr酶复合体抑制由重复基因编码的蛋白的表达。通过grna-crispr酶复合体人工操纵重复基因的转录调节区域的效果可为敲减。可通过grna-crispr酶复合体降低由重复基因编码的蛋白的表达。通过grna-crispr酶复合体人工操纵重复基因的转录调节区域的效果可为敲入。此处,可通过grna-crispr酶复合体和额外包含外源性核苷酸序列或基因的供体诱导敲入效果。通过表达由外源性核苷酸序列或基因编码的肽或蛋白,可诱导通过grna-crispr酶复合体和供体人工操纵重复基因的转录调节区域的效果。此处,可通过grna-crispr酶复合体以及包含期望插入的核苷酸序列的供体诱导敲入效果。本说明书中公开的一个方面涉及调控表达的方法。本说明书中公开的一个示例性实施方式涉及调控重复基因的表达的方法,所述方法可在体内、离体(exvivo)或体外实施。在一些实施方式中,所述方法可包括从人或非人动物对细胞或细胞集落进行取样,以及对所述细胞进行修饰。培养可在离体的任何步骤中进行。细胞甚至可重新导入非人动物或植物中。所述方法可为对真核细胞进行人工工程化的方法,所述方法包括将表达调控组合物导入具有重复基因的真核细胞中。表达调控组合物的描述如上所述。在一个实施方式中,表达调控组合物可包含以下:(a)引导核酸或者编码所述引导核酸的核酸序列,所述引导核酸能够靶向位于重复基因的转录调节区域中的靶序列;以及(b)编辑蛋白或者编码所述编辑蛋白的核酸序列,所述编辑蛋白包括选自于由以下蛋白所组成的组中的一种或多种蛋白:酿脓链球菌衍生而来的cas9蛋白、空肠弯曲杆菌衍生而来的cas9蛋白、嗜热链球菌衍生而来的cas9蛋白、金黄色葡萄球菌衍生而来的cas9蛋白、脑膜炎奈瑟菌衍生而来的cas9蛋白以及cpf1蛋白。此处,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。或者,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。转录调节区域的描述如上所述。引导核酸和编辑蛋白可以以单个核酸序列的形式存在于一个或多个载体中,或借助引导核酸和编辑蛋白的偶联形成复合体而存在。任选地,表达调控组合物可进一步包含供体或编码所述供体的核酸序列,所述供体包含期望插入的核酸序列。引导核酸、编辑蛋白和/或供体可以以单个核酸序列的形式存在于一个或多个载体中。导入步骤可在体内或离体进行。例如,可通过选自电穿孔、脂质体、质粒、病毒载体、纳米粒子和蛋白易位结构域(ptd)融合蛋白法中的一种或多种方法来实施导入步骤。例如,病毒载体可为选自于由逆转录病毒、慢病毒、腺病毒、腺相关病毒(aav)、痘苗病毒、痘病毒和疱疹病毒所组成的组中的一种或多种。本说明书中公开的一个方面涉及使用用于调控表达的组合物治疗基因重复疾病的方法,以治疗基因重复疾病。本说明书中公开的一个示例性实施方式涉及使用如下方法治疗基因重复疾病的用途:所述方法包括将用于对重复基因的转录调节区域进行人工操纵的表达调控组合物给予待治疗的受试者。此处,待治疗的受试者可包括哺乳动物,包括人、灵长类动物(例如猴)以及啮齿类动物(例如小鼠和大鼠)。基因重复疾病的描述如上所述。在一个示例性实施方式中,基因重复疾病可为由pmp22基因的重复产生的疾病。在一个实例中,由pmp22基因的重复产生的疾病可为charcot-marie-tooth1a型(cmt1a)、dejerine-sottas病(dejerine-sottas综合征,dss)、先天性髓鞘形成不良神经病(chn)或roussy-levy综合症(rls)。-charcot-marie-tooth病(cmt)cmt病是由人染色体中发生的基因重复引起的遗传性疾病,涉及手和足中的周围神经发育的基因通过突变被重复,从而导致变形(例如倒置的香槟瓶的形状)。cmt病是相对常见的神经学遗传疾病,在美国100,000人中有36人发生,并且全世界的患者数为280万,即使在韩国也估计有约17,000。根据遗传方面,cmt病大致分为总计5种类型:cmt1、cmt2、cmt3、cmt4和cmtx,cmt1,cmt2和cmt3为显性,且在儿童中遗传的概率为50%,而cmt4为隐性的,以25%的概率遗传。cmt1和cmt2在大多数韩国国内患者中显性遗传(分别为80%和20%-40%),而cmt3和cmt4极为罕见。cmtx是沿x染色体通过母系遗传的,但其频率为10%-20%。cmt1是由于神经轴突周围的髓鞘的蛋白形成中涉及的基因重复而无法进行正常的基因表达过程所引起的疾病。cmt1分为3种类型。cmt1a为常染色体显性遗传疾病,由位于17号染色体17p11.2-p12的pmp22基因的重复导致,其结果是由pmp22(其为髓鞘的重要成分)的过表达引起了髓鞘的结构和功能异常。cmt2与轴突异常有关,是一种神经传导速度接近正常状态但运动感觉神经的动作电位显著降低的神经病,cmt3作为极为罕见的常染色体隐性遗传疾病在儿童早期发生,是临床症状和神经传导速度的降低非常严重的类型。cmt4也是发病年龄早并且临床症状严重的类型,其为常染色体隐性遗传;而cmtx伴随x染色体发生,其症状在男性中比在女性中更严重。-dejerine-sottas病(dejerine-sottas综合征,dss)dss是发生在幼年的脱髓鞘运动感觉神经病,是通常由常染色体显性遗传但也由常染色体隐性遗传的疾病,表现出严重的脱髓鞘神经病并自婴儿时期表现出运动神经异常,其特征在于表现出非常缓慢的神经传导以及脑脊液中特定蛋白的增加。dejerine-sottas病具有非常快的进展速度,其特征在于步态障碍从早年开始,也是遗传性的但也会零星发生。与cmt1a类似,在一些dss患者中发现了pmp22重复,此外,确认了存在相应基因的错义突变。-先天性髓鞘形成不良神经病(chn)chn是一种神经系统疾病,其症状在出生后立即出现,并且其主要症状为出现呼吸衰竭、肌肉无力、肌肉运动失调,肌肉张力的下降、无反射、运动不协调(运动神经机能病;共济失调)、麻痹或感觉迟钝,并且其以相同的比例影响男性和女性。chn是一种遗传疾病,其中运动神经和感觉神经中发生紊乱,其特征为髓鞘形成减少,同时髓鞘反复脱髓鞘和髓鞘再生。-roussy-levy综合征(rls)rls是一种稀有类型的遗传性运动感觉神经病,最早于1926年由roussy和levy等描述,是一种肢体震颤、步态丧失等比其它遗传性运动感觉神经病更为严重的情况,但随后在多种遗传性运动感觉神经病亚型中发现了相同的症状,因此rls目前被认为是遗传性运动感觉神经病中出现的一种症状。对于rls,在首次报道患有rls的患者的基因检测中发现mpz基因(作为髓磷脂蛋白零基因)突变为,并且在其它患者中,已报道了存在pmp22基因(作为周围神经的髓鞘蛋白22的基因)的重复的情况。在一个示例性实施方式中,基因重复疾病可为由plp1基因的重复产生的疾病。在一个实例中,由plp1基因的重复产生的疾病可为pelizaeus-merzbacher病(pmd)。-pelizaeus-merzbacher病(pmd)pelizaeus-merzbacher病(pmd)是一种非常罕见的嗜苏丹性脑白质营养不良(sudanophilicleukodystrophy),由于中枢神经系统的白质的髓鞘形成障碍而表现出多种神经病症状。其患病率估计约为1/400,000。1885年,pelizaeus首次报道了一个患有发育性脑双侧瘫痪的家家族,其为x染色体依赖性遗传,特征在于眼球震颤、共济失调,僵硬和获得性小头畸形,在疾病初期即显现。pmd的临床体征出现在婴儿和儿童早期,并且pmd的特征性临床症状为摆动性眼球震颤、喘鸣、精神运动发育障碍或退化、共济失调、不规则运动、非自愿运动、口腔功能障碍以及智力低下。pmd是一种神经退行性疾病或脑白质营养不良,由于少突胶质细胞的减少和蛋白脂质蛋白(plp)的合成障碍引起的中枢神经系统的白质的髓鞘形成障碍而导致。蛋白脂质蛋白(plp)是中枢神经系统的髓鞘中最丰富的蛋白,其由于位于x染色体长臂上的plp1基因(xq22)的突变而异常表达或产生,从而引起中枢神经系统中的髓鞘形成障碍。pmd在脑组织病理学中对苏丹红具有亲和力,这是由于一些偶氮化合物与脂质反应而导致的,指示髓鞘的分解,在半卵圆中心、小脑以及脑干中观测到。但是,由于未发现分解产物,pmd的病因被认为是髓鞘形成障碍或髓鞘形成不良,而不是脱髓鞘。通常,pmd的先天形式的特征在于完全的髓鞘形成障碍,而pmd的典型形式的特征在于部分的髓鞘形成障碍。当部分的髓鞘形成障碍发生时,正常的髓质白质显示出虎斑状外观。具有髓鞘形成障碍的病灶的轴突和神经元通常得到良好的保留,少见的少突胶质细胞的数量减少,在白质中发现星形胶质细胞的增加以及纤维状神经胶质增生(fibrousgliosis),并在小脑的颗粒层和多小脑回中发现了萎缩。在80%以上的男性患者中,发现位于x染色体长臂上的plp1基因(xq22)的突变。在这些患者中,10%-30%具有该基因中的点突变,并且在此情况下已知表现出更严重的临床症状。在60%-70%以上pmd患者中更经常发现整个plp1基因重复的现象。目前,由于plp1基因位于x染色体上,pmd通常为x染色体依赖性遗传,具有家族史,并且多数发生在男性中。但是,pmd的发病机理可能不能仅用plp1基因解释,有时pmd的先天形式为常染色体隐性,pmd的成年形式为常染色体显性,或者pmd零星地以无家族史形式出现。据报道,即使在女性中罕见表现出pmd的症状。在一个示例性实施方式中,基因重复疾病可为由于mecp2基因的重复而发生的疾病。在一个实例中,由mecp2基因的重复引起的疾病可为mecp2重复综合征。-mecp2重复综合征称为mecp2重复综合征的脑疾病是由遗传物质的重复导致的,该重复发生在具有mecp2基因的x染色体的特定区域。此疾病伴有多种症状,包括低的肌肉张力、发育迟缓、呼吸道感染、言语异常、癫痫、自闭行为以及严重智力残疾等症状。该疾病为遗传性紊乱,但甚至在无家族史的情况下发生。mecp2重复综合征主要在男性中发生,而由于mecp2基因缺陷发生的rett综合征主要在女性中发生。在一个示例性实施方式中,基因重复疾病可为由rai1基因的重复导致的疾病。在一个实例中,由rai1基因重复导致的疾病可为potocki-lupski综合症(ptls)。-potocki-lupski综合征(ptls)ptls是具有17号染色体的短臂上的11.2区域(17p11.2)的微重复的邻近基因综合征(contiguousgenesyndrome),并在1996年报道了第一例ptls研究案例。已知ptls由于具有视黄酸诱导-1(rai1)基因的17p11.2处的1.3mb-3.7mb重复而发生。ptls被认为是罕见疾病,其发病率预计为20,000名新生儿中1例。ptls的特征为多种先天性异常和智力低下,并且80%的ptls病例患有自闭症谱系障碍。此外,ptls的其它独特特征包括睡眠呼吸暂停、结构性心血管异常、社交行为异常、学习障碍、注意力缺陷障碍、强迫行为以及身高矮小。在一个示例性实施方式中,基因重复疾病可为由eln基因的重复导致的疾病。在一个实例中,由eln基因重复导致的疾病可为williamsbeuren综合症(wbs)。-williamsbeuren综合征(wbs)wbs是具有特征性临床表现的近端基因综合征,其与7号染色体异常有关,wbs的发病率为20,000名新生儿中1例。作为7号染色体长臂的近端部分(7q11.23)的微删除的原因,多种基因(包括与形成弹性组织(例如血管壁)的弹性蛋白产生有关的弹性蛋白基因以及与认知能力有关的limk1基因)定位在该区域。由于此类基因的删除,显示出多种特征性外观和临床症状。7q11.23的微删除在多数情况下自然发生,并且很少显示微删除的家族史。患有wbs的儿童具有特征性的外观,例如略微抬头、鼻尖小、人中长、嘴宽、嘴唇丰满、面颊小(颧骨发育不全)、眼睛肿胀、指甲形成不良以及拇外翻。在一个示例性实施方式中,基因重复疾病可为由jagged1基因的重复导致的疾病。在一个实例中,由jagged1基因重复导致的疾病可为alagille综合症(as)。-alagille综合征(as)as是肝中的胆管数量显著减少的综合征,诱导胆汁淤积,并伴有心血管系统、骨骼系统、眼球、面部、胰腺和神经发育异常。根据国外报道,as的发病率为1/100,000,并且由于该疾病的特征,如果包括具有轻微症状的患者,其发病率预计会更高。as由于位于20号染色体的短臂上的jagged1基因的异常而发生。目前已知通过基因检测在50%-70%的病例中可发现致病的突变或重复。as的临床症状通常在出生后三个月内表现。as通常在新生儿时期由于连续性黄疸和胆汁淤积而被发现,并且在儿童期由于慢性肝疾病而被发现,甚至也有在晚年被发现的。由于as具有多种临床症状并且可不完全遗传,其可能难以被诊断。多数患者具有黄疸和胆汁淤积、由此引起的瘙痒以及婴儿期的进行性肝衰竭的症状。在多数患者中观察到黄疸,并且在超过一半的患者中持续至儿童晚期。发生了由胆汁淤积引起的瘙痒,并且一些儿童的皮下组织具有黄色瘤。尽管肝中的合成功能得到了良好的保留,大约20%的患者发展为肝硬化和肝衰竭。在一个示例性实施方式中,基因重复疾病可为由snca基因的重复导致的疾病。在一个实例中,由snca基因重复导致的疾病可为帕金森病。-帕金森病帕金森病是一种通常表现为震颤、肌肉僵硬和运动障碍(例如运动缓慢)的疾病。如果帕金森病得不到适当的治疗,运动障碍逐渐发展,导致行走和日常活动困难。帕金森病是一种主要发生在老年人中的疾病,并且随着年龄增长,疾病发作的风险可能逐渐增加。尽管韩国没有准确的统计数据,据估计帕金森病的发病率为1-2人/1000人。通过各种研究,已知多数发生在老年人中的帕金森病的病例受遗传因素的影响较小。但是,已知一些发生在40岁以下的年轻人中的帕金森病病例与遗传因素有关。帕金森病是由于随着黑质中存在的多巴胺神经元逐渐死亡引起的降低的多巴胺浓度引起的疾病。帕金森病的另一病理特征是在脑尸检中观测到的蛋白聚集体(称为lewy体)的形成。lewy体具有称为α-突触核蛋白的蛋白作为主要成分,并且lewy体和α-突触核蛋白也与lewy体痴呆和突触核蛋白病等其它疾病相关。α-突触核蛋白聚集开始于迷走神经和前嗅核而非中脑,然后在最后阶段经由中脑扩散至大脑皮层。α-突触核蛋白随帕金森病的进展广泛传播至大脑多个区域的假说得到了最近的报道的支持,其中α-突触核蛋白从一个细胞释放,然后传输至另一细胞。该报道首次提出了帕金森病的可遗传性,在该报道中lewy体的主要成分α-突触核蛋白的突变体(a53t和a30p)诱发帕金森病。此后,据报道α-突触核蛋白基因(snca)的重复和三重复是帕金森病的其它病因。这意味着除α-突触核蛋白的突变外,正常蛋白的过表达导致α-突触核蛋白在细胞中的积聚以及聚集体的形成,从而引起帕金森病的发作。在一个示例性实施方式中,基因重复疾病可为由app基因的重复导致的疾病。在一个实例中,由app基因重复导致的疾病可为阿尔茨海默病。-阿尔茨海默病阿尔茨海默病是一种因导致记忆力的进行性退化的脑异常引起的疾病。此外,阿尔茨海默病导致痴呆症,其带来智力功能(思维、记忆和推理)的严重丧失,足以干扰日常生活。在多数病例中,阿尔茨海默病在超过65岁的年龄发生,但在65岁前很少发生。在美国,约3%的65-74岁的人、大约19%的75-84岁的人以及大约50%的85岁以上的人患有阿尔茨海默病。在韩国,根据最近一项基于农村地区的研究,据报道约21%的60岁以上的人患有痴呆,而受影响的人中约63%患有阿尔茨海默痴呆。2006年,全世界有266,000人患有阿尔茨海默病。预计至2050年,阿尔茨海默病在85人中将影响1人。该疾病的特征因人而异,但其中一些在所有受影响的人中都常见。早期症状往往被误认为由衰老导致的简单症状或由压力导致的症状。在疾病的早期阶段,受影响的人经历常见的短期记忆丧失,其中姓名、日期和地点从记忆中消失。如果疾病恶化,显示出混淆、强化行为、双相障碍、言语障碍以及长期记忆丧失的症状。结果,身体功能丧失,导致死亡。由于每个个体的不同症状,难以预测疾病将如何影响人。当怀疑患有阿尔茨海默病时,通常会进行测试思维或行动能力的诊断,并在必要时进行脑测试。但是,为了准确的诊断,有必要检查颅神经。尽管发生了阿尔茨海默病,通常要花很长时间才完全诊断出疾病,因此,疾病可能进展数年而得不到诊断。发生该疾病时,平均预期寿命为7年,并且不到3%的受影响的人在诊断后生存14年。阿尔茨海默病被归类为神经退行性疾病。该疾病的病因尚未完全明晰,但据估计,淀粉样蛋白斑块修饰正常的阿尔茨海默病蛋白,以形成斑块团,从而导致固有功能丧失。阿尔茨海默病具有组织病理学特征,包括整体脑萎缩、心室扩大、神经原纤维缠结以及神经炎性斑。在一个示例性实施方式中,基因重复疾病可为由sox3基因、tbx1基因、nsd1基因、mmp23基因或lmb1基因的重复导致的疾病。在一实例中,基因重复疾病可为x连锁垂体机能减退(xlhp)、颚心面综合征(vcfs)、生长障碍综合征、颅缝早闭或常染色体显性脑白质营养不良(adld)。在一个示例性实施方式中,基因重复疾病可为由癌基因的重复产生的癌症。此处,癌症基因可为myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因或akt2基因。在一个实例中,癌症可为乳腺癌、宫颈癌、结直肠癌、食管癌、胃癌、胶质母细胞瘤、头颈癌、肝细胞癌、神经母细胞瘤、卵巢癌、肉瘤或小细胞肺癌。本说明书中公开的一个示例性实施方式提供了药物组合物,所述药物组合物包含可对重复基因的转录调节区域进行人工工程化的表达调控组合物。表达调控组合物的描述如上所述。在一个示例性实施方式中,表达调控组合物可包含以下:(a)引导核酸或者编码所述引导核酸的核酸序列,所述引导核酸能够靶向位于重复基因的转录调节区域中的靶序列;以及(b)编辑蛋白或者编码所述编辑蛋白的核酸序列,所述编辑蛋白包括选自于由以下蛋白所组成的组中的一种或多种蛋白:酿脓链球菌衍生而来的cas9蛋白、空肠弯曲杆菌衍生而来的cas9蛋白、嗜热链球菌衍生而来的cas9蛋白、金黄色葡萄球菌衍生而来的cas9蛋白、脑膜炎奈瑟菌衍生而来的cas9蛋白以及cpf1蛋白。此处,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。或者,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。转录调节区域的描述如上所述。引导核酸和编辑蛋白各自可以以核酸序列的形式存在于一个或多个载体中,或借助引导核酸和编辑蛋白的偶联形成复合体而存在。任选地,表达调控组合物可进一步包含供体或编码所述供体的核酸序列,所述供体包含期望插入的核酸序列。引导核酸、编辑蛋白和/或供体各自可以以核酸序列的形式存在于一个或多个载体中。药物组合物可进一步包含额外的要素。额外的要素可包括用于递送入受试者体内的合适的运载体。本说明书中公开的一个示例性实施方式提供了治疗基因重复疾病的方法,所述方法包括向患有基因重复疾病的生物体给予用于基因工程化的组合物以治疗基因重复疾病。治疗方法可为通过对活体内存在的重复基因的转录调节区域进行操作来调控重复基因的表达的治疗方法。此类方法可通过直接注射用于操纵活体内存在的重复基因的转录调节区域的表达调控组合物来进行。表达调控组合物的描述如上所述。在一个示例性实施方式中,表达调控组合物可包含以下:(a)引导核酸或者编码所述引导核酸的核酸序列,所述引导核酸能够靶向位于重复基因的转录调节区域中的靶序列;以及(b)编辑蛋白或者编码所述编辑蛋白的核酸序列,所述编辑蛋白包括选自于由以下蛋白所组成的组中的一种或多种蛋白:酿脓链球菌衍生而来的cas9蛋白、空肠弯曲杆菌衍生而来的cas9蛋白、嗜热链球菌衍生而来的cas9蛋白、金黄色葡萄球菌衍生而来的cas9蛋白、脑膜炎奈瑟菌衍生而来的cas9蛋白以及cpf1蛋白。此处,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:pmp22基因、plp1基因、mecp2基因、sox3基因、rai1基因、tbx1基因、eln基因、jagged1基因、nsd1基因、mmp23基因、lmb1基因、snca基因和app基因。或者,重复基因可为选自于由以下基因所组成的组中的一个或多个基因:myc基因、erbb2(her2)基因、ccnd1(细胞周期蛋白d1)基因、fgfr1基因、fgfr2基因、hras基因、kras基因、myb基因、mdm2基因、ccne(细胞周期蛋白e)基因、met基因、cdk4基因、erbb1基因、mycn基因和akt2基因。转录调节区域的描述如上所述。引导核酸和编辑蛋白各自可以以核酸序列的形式存在于一个或多个载体中,或借助引导核酸和编辑蛋白的偶联形成复合体而存在。任选地,表达调控组合物可进一步包含供体或编码所述供体的核酸序列,所述供体包含期望插入的核酸序列。引导核酸、编辑蛋白和/或供体各自可以以核酸序列的形式存在于一个或多个载体中。此处,载体可为质粒或病毒载体。此处,病毒载体可为选自于由以下所组成的组中的一种或多种:逆转录病毒、慢病毒、腺病毒、腺相关病毒(aav)、痘苗病毒、痘病毒和疱疹病毒。基因重复疾病的描述如上所述。基因重复疾病可为charcot-marie-tooth1a型(cmt1a)、dejerine-sottas病(dsd)、先天性髓鞘形成不良神经病(chn)、roussy-levy综合征(rls)、pelizaeus-merzbacher病(pmd)、mecp2重复综合征、x连锁垂体机能减退(xlhp)、potocki-lupski综合征(ptls)、腭心面综合征(vcfs)、williamsbeuren综合征(wbs)、alagille综合征(as)、生长障碍综合征、颅缝早闭、常染色体显性脑白质营养不良(adld)、帕金森病或阿尔茨海默病。此外,癌症可为乳腺癌、宫颈癌、结直肠癌、食管癌、胃癌,胶质母细胞瘤、头颈癌、肝细胞癌、神经母细胞瘤、卵巢癌、肉瘤或小细胞肺癌。可将表达调控组合物给予患有基因重复疾病的治疗受试者。治疗受试者可包括哺乳动物,包括人、灵长类动物(例如猴)以及啮齿类动物(例如小鼠和大鼠)。可将表达调控组合物给予治疗受试者。可通过注射、输注、植入或移植进行给予。可通过选自以下的给予途径进行:神经内、视网膜下、皮下、皮内、眼内、玻璃体内、瘤内、结内、髓内、肌内、静脉内、淋巴内和腹膜内途径。表达调控组合物的剂量(药学有效量,以获得预定的期望效果)为约104-109个细胞/kg(给药受试者的体重),例如约105-106个细胞/kg(体重),并且可选自数值范围内的所有整数,但本发明不限于此。可考虑给药的受试者的年龄、健康状况和体重,同时治疗的类型,(如果有的话)治疗频率以及期望效果来对组合物进行前档规定。当通过根据本说明书中公开的一些示例性实施方式的方法和组合物对重复基因的转录调节区域进行人工操纵时,可调控重复基因的mrna和/或蛋白的表达,因此,可获得使异常表达的重复基因的表达得到正常控制的效果。实施例下文将参考实施例进一步详细地描述本发明。提供这些实施例仅为进一步详细地描述本发明,并且对于本领域普通技术人员可能显而易见的是,本发明的范围不限于以下实施例。实验方法1.grna设计使用crisprrgen工具(www.rgenome.net)筛选人pmp22基因、人plp1基因和小鼠plp1基因的crispr/cas9靶区域。pmp22基因和plp1基因的靶区域可根据crispr酶的类型而改变。上表1中总结了针对spcas9的人pmp22基因的启动子区域(tata盒)和增强子区域(例如egr2、sox10或tead1结合区域)或远端增强子区域b或c的靶序列;表2中总结了针对cjcas9的人pmp22基因的启动子区域(tata盒)和增强子区域(例如egr2或sox10结合区域)的靶序列。此外,上表3中总结了针对spcas9的人plp1基因的启动子区域(tata盒区域)和增强子区域(例如wmn1增强子)的靶序列;上表4总结了针对cjcas9的人plp1基因的启动子区域(tata盒区域)和增强子区域(例如wmn1增强子)的靶序列。上表5中总结了针对spcas9的小鼠plp1基因的启动子区域(tata盒区域)和增强子区域(例如wmn1增强子)的靶序列;上表6中总结了针对cjcas9的小鼠plp1基因的启动子区域(tata盒区域)和增强子区域(例如wmn1增强子)的靶序列。所有grna均以嵌合单链rna(sgrna)的形式生成。除靶序列外,cj特异性sgrna和sp特异性sgrna的骨架序列为5′-guuuuagucccugaaaagggacuaaaauaaagaguuugcgggacucugcgggguuacaauccccuaaaaccgcuuuu-3′和5′-guuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc-3′。2.grna的构建与合成sgrna被包装至aav载体中或与rna合成。为了将sgrna插入病毒载体中,设计对应于sgrna的20-22个碱基序列的dna寡核苷酸并使其退火,使用bsmbi位点连接至prgen-cas9(内部开发的)载体中。分别通过cmv和u6启动子表达cas9和在5′端包含可变靶序列的sgrna。此外,对于借助rnp的递送系统,在通过对由phusiontaq介导的聚合产生的两个部分互补的寡核苷酸进行退火而产生模板后,通过t7rna聚合酶转录sgrna。转录的sgrna进行纯化以及使用光谱测定进行定量。3.cas9蛋白的纯化将包含nls和ha表位的经密码子优化的cas9dna序列亚克隆至pet28载体中,并在最佳培养条件下使用iptg在bl21(de3)中表达。表达的cas9蛋白使用ni-nta琼脂糖珠进行纯化,并用适当的缓冲液进行透析。通过使用众所周知的有效sgrna的体外切割测试确认cas9的活性。4.细胞培养根据制造商的手册培养人雪旺样细胞系(atcc)和人原代雪旺细胞(sciencell)。在补充有1×青霉素/链霉素(welgene)和10%胎牛血清(welgene)的含有高浓度葡萄糖的dulbecco改良eagle培养基(dmem)(welgene)中培养人雪旺样细胞。将人原代雪旺细胞维持在供应商提供的雪旺细胞培养溶液(sciencell)中。为了分化,将细胞在补充有1%胎牛血清(welgene)、100ng/mlnrg1(peprotech,用于髓鞘生成(髓鞘形成)信号)和100μmdbcamp(sigma-aldrich)的含有低浓度葡萄糖的dmem(welgene)中培养7天。5.转导(转染)为了转导(转染),将含有4μgcas9蛋白(toolgen)和1μgsgrna的rnp复合体在室温下孵育15分钟。此后,通过使用10μl电穿孔头和neon电穿孔仪(thermofisher)对rnp复合体进行电穿孔,并递送至2×105个细胞。对于靶向深度测序,在转导后72小时从转导的细胞收集基因组dna(gdna)。6.体外实时pcr(qrt-pcr)根据制造商的方案,使用rneasyminikit(qiagen)从人原代雪旺细胞提取mrna。此后,使用高容量cdna逆转录试剂盒(thermofisher)对100ngmrna进行逆转录。根据制造商的方案,使用quantstudio3(thermofisher)用100ngtaqman基因表达预混液进行qrt-pcr。使用ct值计算pmp22表达水平,并将gapdh用作内参。下表7中总结了本研究中使用的taqman探针(thermofisher)。[表7]靶基因taqman基因表达测试登记号pmp22hs00165556_m1nm_000304.3gapdhhs02786624_g1nm_001256799.27.靶向深度测序使用phusion聚合酶taq(newenglandbiolabs),通过pcr由从转导的细胞中提取的gdna扩增中靶(on-target)位点。此后,作为pcr扩增产物,使用mi-seq(illumina)进行双末端深度测序(paired-enddeepsequencing)。使用在线cas-analyzer工具(www.rgenome.net)分析深度测序结果。对是否通过由cas9引起的插入缺失引起pam序列上游3bp处发生突变进行确认。下表8中总结了本研究中使用的引物。[表8]8.脱靶位点的计算机设计使用在线工具(www.rgenome.net)对脱靶潜在位点进行了计算机设计。最多3bp的错配被认为是脱靶位点。9.digenome-seq使用dneasy血液和组织试剂盒(qiagen)根据供应商的方案对hela细胞的基因组dna进行纯化。在37℃下,将预先孵育的cas9蛋白(100nm)和sgrna(300nm)与基因组dna(10μg)在1ml反应溶液(100mmnacl、50mmtris-hcl、10mmmgcl2、100μg/mlbsa,ph7.9)中混合8小时。切割的基因组dna用rnasea(50μg/ml)处理,并使用dneasytissuekit(qiagen)再次纯化。使用covaris系统将1μg切割的基因组dna分裂成片段,并将用于产生文库的连接物连接至dna片段。此后,使用hiseqxtensequencer(illumina)以30-40×的测序深度(macrogen)对文库进行全基因组测序(wgs)。通过dna切割评分系统计算基因组中切割的每个碱基序列的位置处的体外切割评分。10.小鼠和神经内注射本研究中使用的c22小鼠品系(b6;cbaca-tg(pmp22)c22clh/h)购自mrcharwell(oxfordshire,uk)。用pmp22-tatarnp处理c22小鼠(4只雄性和7只雌性)。以与先前研究相同的方式进行神经内注射(daisukeino.,jvisexp.,(2016)115)。对6日龄的小鼠进行麻醉,并通过手术暴露小鼠坐骨神经。为了使神经损伤最小化,立即使用连接至微注射器的拉制玻璃微量移液器在坐骨切迹的末端处进行神经内注射。将每只小鼠11μgcas9蛋白和2.75μgsgrna的rnp复合体与lipofectamine3000(invitrogen,carlsbad,ca,usa)一起注入小鼠。本研究中使用的所有动物的管理、使用和处理均在三星动物管理和使用委员会(smc-20170206001)根据国际实验动物护理评估和认可委员会制定的准则下进行。11.旋转杆实验(旋转杆测试)使用旋转杆设备(b.s.technolabinc.,韩国)对运动协调进行评价。进行该实验以评价小鼠的平衡和运动协调。实验前,小鼠经历3天的训练期。在实验中,水平旋转杆(21rpm)用于旋转杆实验。测量小鼠在旋转杆上的停留时间,并允许小鼠在杆上停留多至300秒。12.电生理测试为了评价电生理状态,以与先前研究相同的方式进行了神经传导测试(ncs)(jinholee.,jbiomedsci.,(2015)22,43)。总的来说,将小鼠用二氧化碳气体麻醉,并在实验过程中使用鼻锥提供1.5%异氟烷来维持麻醉。从末端到后爪的毛发完全去除。使用nicoletvikingquest设备(natusmedical)进行ncs。对于坐骨神经的运动神经传导测试,通过以下过程分别确定远端部分和近端部分的响应:将活动记录针状电极放置在腓肠肌上,将参比电极连接至腱,并在靠近距离眶后部大腿的中心线以及臀部内侧的身体中心6mm处的记录电极的位置处放置刺激性负电极。测量远端潜伏期(dl)、运动神经传导速度(mncv)和复合肌肉动作电位(cmap)的幅度。在最大过度刺激时测量cmap。13.神经组织学和图像对小鼠的坐骨神经进行活检,并通过显微镜分析对受影响的样品进行病理学检查。分别使用含有2%戊二醛的25mm甲次胂酸盐缓冲液固定样品。用甲苯胺蓝对半薄切片进行染色。在1%oso4中孵育1小时后,将样品在乙醇中连续脱水,然后使其通过环氧丙烷,并包埋在环氧树脂(epon812,oken,nagano,日本)中。使用leica超薄切片机(leicamicrosystems)将细胞切成一定厚度(1μm),并用甲苯胺蓝染色30-45秒。通过使用zeisszen2程序(carlzeiss,oberkochen,德国)测量髓磷脂的内径和外径来计算g比(轴突直径/纤维直径)。14.统计学分析使用post-hoctukey多重比较通过单因素anova评价了与mrna表达水平相关的数据的统计学意义。使用mann-whitneyu检验(http://www.socscistatistics.com/tests/mannwhitney/default2.aspx)计算了呈现的数据的其它类型。使用graphpadprism对本研究产生的数据和图表进行了分析。显著性水平设定为0.05。15.用于plp1基因靶向的sgrna筛选根据制造商的手册培养小鼠成纤维细胞、nih-3t3(atcc,crl-1658)、成肌细胞(即c2c12系,atcc,crl-1772)和少突胶质细胞n20.1(cedarlanelaboratories,clu108-p)。将细胞在补充有1×青霉素/链霉素(welgene)和10%胎牛血清(welgene)的含有高浓度葡萄糖的dulbecco改良eagle培养基(dmem)中在37℃和5%co2下进行培养。为了转染crispr/cas9组合物,制备了由4μgcas9蛋白和1μgsgrna组成的rnp复合体(spcas9)或cjcas9质粒(图18)。此后,使用10μl电穿孔头和neon电穿孔仪(thermofisher)通过电穿孔将rnp复合体或cjcas9质粒递送至2×105个细胞。对于靶向深度测序,在转染后72小时,从转染的细胞中收集基因组dna(gdna)。16.plp1基因的下调测定根据制造商的方案,使用rneasymini试剂盒(qiagen)从n20.1细胞系中提取mrna。此后,使用高容量cdna逆转录试剂盒(thermofisher)对1μgmrna进行逆转录。根据制造商的方案,使用quantstudio3(thermofisher)用100ngtaqman基因表达预混液进行实时定量逆转录聚合酶链式反应(qrt-pcr)。使用ct值计算plp1表达水平,并将gapdh用作内参。下表9中总结了本研究中使用的taqman探针(thermofisher)。[表9]靶基因taqman基因表达测试登记号plp1mm01297210_m1nm_001290561.1gapdhmm99999915_g1nm_001289726.117.用于plp1基因靶向的sgrna筛选根据制造商的手册培养人淋巴母细胞jurkat细胞系(atcc,tib-152)和人上皮293t细胞系(atcc,crl-3216)。将细胞在补充有1×青霉素/链霉素(welgene)和10%胎牛血清(welgene)的含有高浓度葡萄糖的dulbecco改良eagle培养基(dmem)中在37℃和5%co2下培养。为了crispr/cas9组合物转染,制备了由4μgcas9蛋白和1μgsgrna组成的rnp复合体(spcas9)或cjcas9质粒(图18)。此后,使用10μl电穿孔头和neon电穿孔仪(thermofisher)通过电穿孔将rnp复合体或cjcas9质粒递送至2×105个细胞。对于靶向深度测序,在转染后72小时,从转染的细胞中收集基因组dna(gdna)。实施例1.针对pmp22基因的sgrna筛选为了筛选可将人pmp22表达降低至正常范围的治疗上有效的sgrna序列,用设计用于靶向pmp22基因的启动子(tata盒)和内在增强子结合位点的多种sgrna和cas9s对人细胞系进行转导。简而言之,将jurkat人t细胞用于spcas9筛选,将hek293t细胞用于cjcas9。从细胞收集gdna并进行靶向深度测序。通过nhej介导的插入缺失鉴别了由sgrna序列诱导的多种模式的突变。数种spcas9-sgrna在两个调节位点中强烈诱导了插入缺失(图1)。确认了在特定cjcas9-sgrna中诱导了30%-40%的插入缺失(图2)。实施例2.雪旺样细胞的基因操纵尽管在人细胞中鉴别出由sgrna导致的有效插入缺失突变,但尚不确定在雪旺细胞中是否也能够产生效果。因此,为了研究在雪旺细胞中pmp22表达抑制和基因操纵的效果,使用snf02.0细胞(雪旺样细胞)确认spcas9-sgrna的效果。在snf02.0细胞中重复测试了在jurkat细胞中鉴别出的有效spcas9-sgrna。转导后,通过深度测序分析确认了通过相同的sgrna获得了相同的高插入缺失频率。靶向启动子(tata盒)位点和增强子结合位点的单个sgrna的转导分别诱导了31%和59%的插入缺失(图3)。有趣的是,在经双sgrna处理的大量细胞中发现了40bp-50bp的小缺失,其中包含髓磷脂基因的主要调控因子(例如egr或sox10结合位点)或重要的tata盒(图5)。实施例3.通过基因操纵对pmp22的表达调控为了评价由有效sgrna引起的pmp22表达的变化,使雪旺样细胞进行分化,并进行qrt-pcr。结果,多数靶向pmp22的sgrna有效地抑制了pmp22的表达(图6)。当使用单个sgrna时,与仅经cas9处理的对照相比,pmp22的表达降低了约30%;当使用双sgrna时,与仅经cas9处理的对照相比,pmp22的表达降低了约50%。实施例4.雪旺细胞的基因操纵先前在雪旺样细胞中确认了pmp22的表达抑制和基因操纵效果后,对先前的结果在人原代雪旺细胞中是否表现出相似的效果进行了确认。使用spcas9-sgrna在人原代雪旺细胞中的人pmp22基因的每个靶位点处观测根据靶位点的插入缺失频率。结果,确认了对于靶向pmp22基因的编码序列、tata盒和增强子的多数sgrna,靶位点处的插入缺失频率高(图7a)。进一步地,即使当使用各自靶向tata盒和增强子的双sgrna时,显示出高的插入缺失频率。确认了使用靶向编码远端增强子位点b和c的序列的sgrna在靶位点处额外地发生插入缺失(图7c),并且在此情况下,将靶向apoc3的sgrna用作对照。此外,为了确认spcas9-sgrna在每个靶位点处是否引起pmp22基因表达的降低,进行了qrt-pcr分析。由于pmp22在雪旺细胞分化的最后阶段转录,用众所周知的分化信号因子(包括神经调节蛋白1(nrg1)和二丁酰环amp(dbcamp))将人原代雪旺细胞处理7天。结果,确认了与未经nrg1或dbcamp处理的细胞相比,在经nrg1和dbcamp处理的细胞中pmp22的表达增加了9倍。相反,当用每个靶位点处的spcas9-sgrna处理细胞时,确认了pmp22的表达被诱导了4-6倍。确定这是由于每个靶位点处的spcas9-sgrna对pmp22的每个靶位点的修饰而引起的pmp22的表达抑制(图7b)。实施例5.使用靶向人pmp22基因的tata盒位点的crispr/cas9减少pmp22的有效和特异性表达的效果通过选择sgrna_tata_sp#1(此后称为pmp22-tatasgrna,其在先前筛选的靶向tata盒位点的sgrna中显示出高的插入缺失效率并可靶向tata盒)在人原代雪旺细胞中进行实验。通过用包含sgrna和cas9蛋白的rnp复合体转导人原代雪旺细胞来诱导插入缺失(图8b),并通过靶向深度测序分析确认了在人pmp22的tata盒位点处生成了总插入缺失的89.54±1.39%(图8c)。此外,为了确认在pmp22的tata盒处形成的突变是否导致pmp22基因表达的降低,进行了qrt-pcr分析。由于pmp22在雪旺细胞分化的最后阶段转录,因此用众所周知的分化信号因子(包括神经调节蛋白1(nrg1)和二丁酰环amp(dbcamp))将人原代雪旺细胞处理7天。结果,确认了与未经nrg1或dbcamp处理的细胞相比,在经nrg1和dbcamp处理的细胞中pmp22的表达增加了9倍。相反,确认了当将细胞与pmp22-tatarnp一起处理时,pmp22的表达被诱导了6倍。确定这是由于crispr/cas9对pmp22的tata修饰而引起的pmp22的表达抑制(图8d)。在经分化信号因子和aavs1靶rnp处理的对照中,均确认pmp22基因表达无差异。为了确认pmp22-tatarnp的特异性,进行了基于计算机的脱靶分析。通过靶向深度测序,在通过计算机分析确认的脱靶位点处未确认超过测序错误率(平均0.1%)的插入缺失突变(图9)。由于基于计算机的脱靶分析可能是有偏倚的方法,还进行了digenome-seq(基于整个测序的脱靶分析,其无偏倚)。结果,能够确认9个被pmp22-tatarnp体外切割的脱靶位点(图10a,图10b)。然而,作为通过靶向深度测序的重新分析的结果,在脱靶位点处未发现异常的插入缺失突变(图10c)。这些结果表明,pmp22-tatarnp对pmp22的tata盒的有效且特异性的修饰可调控人原代雪旺细胞中pmp22的转录水平。实施例6.通过cmt1a小鼠中crispr/cas9介导的pmp22的表达抑制来缓解疾病表型的效果为了测试pmp22-tatarnp对pmp22的体内转录调控,将脂质体包裹的pmp22-tatarnp直接注入c22小鼠的坐骨神经中(图11)。在此情况下,靶向rosa26(mrosa26)的rnp复合体用作对照。将mrosa26rnp或pmp22-tatarnp神经内注射入并递送至6日龄(p6)小鼠的左坐骨神经(同侧),并将右坐骨神经用作内参(对侧)。注射后4周,通过从坐骨神经收集基因组dna,借助靶向深度测序确认rnp复合体的神经内递送效率。结果,分别经mrosa26rnp和pmp22-tatarnp处理的所有坐骨神经显示出约11%的插入缺失效率(图12a)。进一步地,在总体插入缺失测序读段中确认了98.48±0.15%的tata盒突变,其与体外结果一致(图12b)。此外,为了确认体内tata盒突变对pmp22的表达抑制,在经rnp处理的坐骨神经上进行了从整个坐骨神经提取的mrna的qrt-pcr分析。与体外结果相似,确认了与对照相比pmp22基因的表达降低了38%(图12c)。为了确认pmp22-tatarnp是否在坐骨神经中发生了脱靶突变,进行了基于计算机的脱靶分析。结果,从小鼠基因组确认了包含3bp以上错配的8处脱靶(图13a),并作为进行靶向深度测序的结果,从经pmp22-tatarnp处理的神经(同侧)未确认到超过测序错误率的插入缺失突变(图13b)。为了测试由pmp22-tatarna导致的pmp22转录的减少是否可防止脱髓鞘,获得了经pmp22-tatarnp或mrosa26rnp处理的c22小鼠的坐骨神经,并且用甲苯胺蓝对其半薄截面切片进行了染色(髓磷脂染色)。此外,为了测量g比,测量了轴突直径和纤维(包含髓磷脂的轴突)直径。结果,确认了在经pmp22-tatarnp处理的实验组中形成了较厚的髓磷脂片层(图14a,图14b)。此外,与经mrosa26rnp处理的对照相比,当用pmp22-tatarnp处理实验组时,发现具有大的直径的轴突的数目增加(图14a,图14b)。在经pmp22-tatarnp处理的实验组中测量具有6μm以上直径的大的有髓鞘纤维的数目的结果(16.5%)显示出比对照组更明显的治疗效果(2.6%,p<0.01)。考虑到髓鞘形成组织学分析的显著改善,研究了两个组的电生理学特征。结果,与经mrosa26rnp处理的对照相比,确认了在经pmp22-tatarnp处理的实验组的坐骨神经中远端潜伏期(dl)降低并且运动神经传导速度(ncv)增加(图15a,图15b),该结果对应于经pmp22-tatarnp处理的神经中髓磷脂厚度和轴突直径的增加。此外,确认了在经pmp22-tatarnp处理的神经中,复合肌肉动作电位(cmap)的幅度显著增加(图15c),这符合先前的结果。考虑到pmp22-tatarnp在组织学和电生理学上的改善作用,通过旋转杆实验分析了小鼠的运动行为。结果,确认了经pmp22-tatarnp处理的小鼠(11-16周龄)比经mrosa26rnp处理的小鼠(11-16周龄)在杆上保持更长的时间(图16a)。此外,确认了与经mrosa26rnp处理的小鼠相比,经pmp22-tatarnp处理的小鼠的肌肉增加(图16b)。这些结果显示出pmp22-tatarnp通过pmp22(例如cmt1a)的过表达缓解或治疗脱髓鞘的治疗作用。因此,上述结果显示了使用靶向pmp22的启动子位点的crispr/cas9对pmp22的表达抑制作用。此外,结果显示将pmp22-tatarnp直接非病毒递送至c22小鼠的坐骨神经可改善与pmp22的过表达引起的脱髓鞘相关的临床和神经病理学表型。因此,认为crispr/cas9介导的pmp22的转录调节区域的修饰可为治疗cmt1a和其它表现出脱髓鞘神经病的疾病的良好策略。实施例7.plp基因表达调节作用当重复plp1基因时,plp1基因过表达,这成为pmd疾病的主要原因。因此,为了调控plp1转录以治疗pmd疾病,使用crispr/cas9人工修饰plp基因的转录调节区域以确认其效果。为此,针对小鼠plp1的启动子序列的tata盒和增强子(wmn1)进行spcas9和cjcas9筛选,选择具有最高活性的sgrna,然后通过qrt-pcr确认plp1下调(图17)。此处,plp1的增强子可为ase(hamdan等,2015;meng等,2005;wight,2017)或wmn1(hamdan等,2018;tuason等,2008)区域。基于sgrna筛选结果,选择了具有高插入缺失率的针对spcas9和cjcas9的各sgrna(图19-图22和表10),当使用少突胶质细胞(即表达plp1基因的n20.1细胞系)靶向plp1的tata盒和wmn1增强子区域时,通过qrt-pcr进行了关于何种因素可导致plp1基因下调的研究。[表10]筛选的sgrna列表(mplp1-tata、mplp1-wmn1spcas9和cjcas9前导sgrna列表)结果,确认了使用spcas9和cjcas9靶向plp1的tata盒或wmn1增强子区域导致plp1的显著下调(图23)。此外,进行了针对人plp1基因的wmn1增强子区域的spcas9和cjcas9筛选,以确认插入缺失率(%)(图24和图25)。因此,认为crispr/cas9介导的plp1的转录调节区域的人工修饰可为pmd治疗的良好策略。[工业实用性]可使用用于调控重复基因表达的表达调控组合物获得用于基因重复疾病的治疗剂。例如,通过对重复基因的转录调节区域进行人工操纵和/或修饰来调控重复基因的表达,包含能够靶向重复基因的转录调节区域的引导核酸的表达调控组合物可用作用于由基因重复导致的疾病的治疗剂。[序列表自由文本]实施例中使用的引物序列以及重复基因的转录调节区域的靶序列<110>toolgenincorporated<120>用于调控基因表达的基因组编辑<130>opp17-074-pct<150>us62/564478<151>2017-09-28<150>us62/565868<151>2017-09-29<160>260<170>kopatentin3.0<210>1<211>20<212>dna<213>智人(homosapiens)<400>1ggaccagcccctgaataaac20<210>2<211>20<212>dna<213>智人(homosapiens)<400>2ggcgtctttccagtttattc20<210>3<211>20<212>dna<213>智人(homosapiens)<400>3gcgtctttccagtttattca20<210>4<211>20<212>dna<213>智人(homosapiens)<400>4cgtctttccagtttattcag20<210>5<211>20<212>dna<213>智人(homosapiens)<400>5ttcaggggctggtccaatgc20<210>6<211>20<212>dna<213>智人(homosapiens)<400>6tcaggggctggtccaatgct20<210>7<211>20<212>dna<213>智人(homosapiens)<400>7accatgacatatcccagcat20<210>8<211>20<212>dna<213>智人(homosapiens)<400>8tttccagtttattcaggggc20<210>9<211>20<212>dna<213>智人(homosapiens)<400>9cagttacagggagcaccacc20<210>10<211>20<212>dna<213>智人(homosapiens)<400>10ctggtctggcttcagttaca20<210>11<211>20<212>dna<213>智人(homosapiens)<400>11cctggtctggcttcagttac20<210>12<211>20<212>dna<213>智人(homosapiens)<400>12aactggaaagacgcctggtc20<210>13<211>20<212>dna<213>智人(homosapiens)<400>13gaataaactggaaagacgcc20<210>14<211>20<212>dna<213>智人(homosapiens)<400>14tccaatgctgggatatgtca20<210>15<211>20<212>dna<213>智人(homosapiens)<400>15aatgctgggatatgtcatgg20<210>16<211>20<212>dna<213>智人(homosapiens)<400>16atagaggctgagaacctctc20<210>17<211>20<212>dna<213>智人(homosapiens)<400>17ttgggcatgtttgagctggt20<210>18<211>20<212>dna<213>智人(homosapiens)<400>18tttgggcatgtttgagctgg20<210>19<211>20<212>dna<213>智人(homosapiens)<400>19gagctggtgggcgaagcata20<210>20<211>20<212>dna<213>智人(homosapiens)<400>20agctggtgggcgaagcatat20<210>21<211>20<212>dna<213>智人(homosapiens)<400>21tgggcgaagcatatgggcaa20<210>22<211>20<212>dna<213>智人(homosapiens)<400>22ggcctccatcctaaacaatg20<210>23<211>20<212>dna<213>智人(homosapiens)<400>23gggttgggaggtttgggcgt20<210>24<211>20<212>dna<213>智人(homosapiens)<400>24aggtttgggcgtgggagtcc20<210>25<211>19<212>dna<213>智人(homosapiens)<400>25ttcagagactcagctattt19<210>26<211>19<212>dna<213>智人(homosapiens)<400>26ggccacattgtttaggatg19<210>27<211>19<212>dna<213>智人(homosapiens)<400>27ggctttgggcatgtttgag19<210>28<211>19<212>dna<213>智人(homosapiens)<400>28aacatgcccaaagcccagc19<210>29<211>19<212>dna<213>智人(homosapiens)<400>29acatgcccaaagcccagcg19<210>30<211>20<212>dna<213>智人(homosapiens)<400>30cgatgatactcagcaacagg20<210>31<211>20<212>dna<213>智人(homosapiens)<400>31atggacacgcaactgatctc20<210>32<211>22<212>dna<213>智人(homosapiens)<400>32gccctctgaatctccagtcaat22<210>33<211>22<212>dna<213>智人(homosapiens)<400>33aatctccagtcaattccaacac22<210>34<211>22<212>dna<213>智人(homosapiens)<400>34aattaggcaattcttgtaaagc22<210>35<211>22<212>dna<213>智人(homosapiens)<400>35ttaggcaattcttgtaaagcat22<210>36<211>22<212>dna<213>智人(homosapiens)<400>36aaagcataggcacacatcaccc22<210>37<211>22<212>dna<213>智人(homosapiens)<400>37gcctggtctggcttcagttaca22<210>38<211>22<212>dna<213>智人(homosapiens)<400>38gtgtccaactttgtttgctttc22<210>39<211>22<212>dna<213>智人(homosapiens)<400>39gtattctggaaagcaaacaaag22<210>40<211>22<212>dna<213>智人(homosapiens)<400>40cagtcttggcatcacaggcttc22<210>41<211>22<212>dna<213>智人(homosapiens)<400>41ggacctcttggctattacacag22<210>42<211>22<212>dna<213>智人(homosapiens)<400>42ggagccagtgggacctcttggc22<210>43<211>22<212>dna<213>智人(homosapiens)<400>43taaatcacagaggcaaagagtt22<210>44<211>22<212>dna<213>智人(homosapiens)<400>44ttgcatagtgctagactgtttt22<210>45<211>22<212>dna<213>智人(homosapiens)<400>45gggtcatgtgttttgaaaacag22<210>46<211>22<212>dna<213>智人(homosapiens)<400>46cccaaacctcccaacccacaac22<210>47<211>22<212>dna<213>智人(homosapiens)<400>47actcagctatttctggaatgac22<210>48<211>22<212>dna<213>智人(homosapiens)<400>48tcatcgcctttgtgagctccat22<210>49<211>22<212>dna<213>智人(homosapiens)<400>49cagacacaggctttgctctagc22<210>50<211>22<212>dna<213>智人(homosapiens)<400>50caaagcctgtgtctggccacta22<210>51<211>22<212>dna<213>智人(homosapiens)<400>51agcagtttgtgcccactagtgg22<210>52<211>22<212>dna<213>智人(homosapiens)<400>52atgtcaaggtattccagctaac22<210>53<211>22<212>dna<213>智人(homosapiens)<400>53gaataactgtatcaaagttagc22<210>54<211>22<212>dna<213>智人(homosapiens)<400>54ttcctaattaagaggctttgtg22<210>55<211>22<212>dna<213>智人(homosapiens)<400>55gagctagtttgtcagggtctag22<210>56<211>23<212>dna<213>智人(homosapiens)<400>56gactttgggagctaatatctagg23<210>57<211>23<212>dna<213>智人(homosapiens)<400>57ccctttcatcttcccattcgtgg23<210>58<211>23<212>dna<213>智人(homosapiens)<400>58cctttcatcttcccattcgtggg23<210>59<211>23<212>dna<213>智人(homosapiens)<400>59cccacgaatgggaagatgaaagg23<210>60<211>23<212>dna<213>智人(homosapiens)<400>60catcttcccattcgtgggcaagg23<210>61<211>23<212>dna<213>智人(homosapiens)<400>61tctccaccttgcccacgaatggg23<210>62<211>23<212>dna<213>智人(homosapiens)<400>62gtctccaccttgcccacgaatgg23<210>63<211>23<212>dna<213>智人(homosapiens)<400>63cccaatgcttgcacataaattgg23<210>64<211>23<212>dna<213>智人(homosapiens)<400>64ccaatttatgtgcaagcattggg23<210>65<211>23<212>dna<213>智人(homosapiens)<400>65tccaatttatgtgcaagcattgg23<210>66<211>23<212>dna<213>智人(homosapiens)<400>66tgtgcgcgtctgaagaggagtgg23<210>67<211>23<212>dna<213>智人(homosapiens)<400>67gtgcgcgtctgaagaggagtggg23<210>68<211>23<212>dna<213>智人(homosapiens)<400>68tgcgcgtctgaagaggagtgggg23<210>69<211>23<212>dna<213>智人(homosapiens)<400>69tagtccagatgctgttgccgtgg23<210>70<211>23<212>dna<213>智人(homosapiens)<400>70attaccacggcaacagcatctgg23<210>71<211>23<212>dna<213>智人(homosapiens)<400>71gacacgatttagtattaccacgg23<210>72<211>23<212>dna<213>智人(homosapiens)<400>72ctaaatcgtgtccaaagaggagg23<210>73<211>23<212>dna<213>智人(homosapiens)<400>73aggaatctcagcctcctctttgg23<210>74<211>23<212>dna<213>智人(homosapiens)<400>74gtggacaaggttaactaaaaagg23<210>75<211>23<212>dna<213>智人(homosapiens)<400>75atagtcaaatcatgtggacaagg23<210>76<211>23<212>dna<213>智人(homosapiens)<400>76tgctggatagtcaaatcatgtgg23<210>77<211>23<212>dna<213>智人(homosapiens)<400>77acatgatttgactatccagcagg23<210>78<211>23<212>dna<213>智人(homosapiens)<400>78atttgactatccagcaggcttgg23<210>79<211>23<212>dna<213>智人(homosapiens)<400>79gtcccgaagtctctggggcctgg23<210>80<211>23<212>dna<213>智人(homosapiens)<400>80aaaacagtcccgaagtctctggg23<210>81<211>23<212>dna<213>智人(homosapiens)<400>81gaaaacagtcccgaagtctctgg23<210>82<211>23<212>dna<213>智人(homosapiens)<400>82tatataccacattcaagtgctgg23<210>83<211>23<212>dna<213>智人(homosapiens)<400>83tggatataacgaagttgtgtggg23<210>84<211>23<212>dna<213>智人(homosapiens)<400>84atggatataacgaagttgtgtgg23<210>85<211>23<212>dna<213>智人(homosapiens)<400>85atatgtttgttcaccccaacagg23<210>86<211>23<212>dna<213>智人(homosapiens)<400>86gaaaacttgaaatcctgttgggg23<210>87<211>23<212>dna<213>智人(homosapiens)<400>87tagacattaggagaaacagaagg23<210>88<211>23<212>dna<213>智人(homosapiens)<400>88ctagcagtgacatagacattagg23<210>89<211>23<212>dna<213>智人(homosapiens)<400>89agccacctgactttgatgaaagg23<210>90<211>23<212>dna<213>智人(homosapiens)<400>90tgagaaatgttattactatatgg23<210>91<211>23<212>dna<213>智人(homosapiens)<400>91agactgcgagatgagagagttgg23<210>92<211>23<212>dna<213>智人(homosapiens)<400>92ctcgcagtctgtacttagactgg23<210>93<211>23<212>dna<213>智人(homosapiens)<400>93aatgtctcttgagagagccaagg23<210>94<211>30<212>dna<213>智人(homosapiens)<400>94atgggaagatgaaagggaagtaactggtac30<210>95<211>30<212>dna<213>智人(homosapiens)<400>95actttgattgttaaaacttatccttggcac30<210>96<211>30<212>dna<213>智人(homosapiens)<400>96agtcctacctcagcttcccaatgcttgcac30<210>97<211>30<212>dna<213>智人(homosapiens)<400>97caatgcttgcacataaattggaatgtgtac30<210>98<211>30<212>dna<213>智人(homosapiens)<400>98acacagagagagacagaatgaatgatgtac30<210>99<211>30<212>dna<213>智人(homosapiens)<400>99tcctcttcagacgcgcacacacacacacac30<210>100<211>30<212>dna<213>智人(homosapiens)<400>100actcctcttcagacgcgcacacacacacac30<210>101<211>30<212>dna<213>智人(homosapiens)<400>101ccactcctcttcagacgcgcacacacacac30<210>102<211>30<212>dna<213>智人(homosapiens)<400>102ccccactcctcttcagacgcgcacacacac30<210>103<211>30<212>dna<213>智人(homosapiens)<400>103ctccccactcctcttcagacgcgcacacac30<210>104<211>30<212>dna<213>智人(homosapiens)<400>104tactccccactcctcttcagacgcgcacac30<210>105<211>30<212>dna<213>智人(homosapiens)<400>105tatactccccactcctcttcagacgcgcac30<210>106<211>30<212>dna<213>智人(homosapiens)<400>106acagcatctggactatcttgtttcctatac30<210>107<211>30<212>dna<213>智人(homosapiens)<400>107atagtccagatgctgttgccgtggtaatac30<210>108<211>30<212>dna<213>智人(homosapiens)<400>108aaaaggaatctcagcctcctctttggacac30<210>109<211>30<212>dna<213>智人(homosapiens)<400>109tgtcactgctagtgtgcttaattcttgtac30<210>110<211>30<212>dna<213>智人(homosapiens)<400>110atgtgaattcagtacaagaattaagcacac30<210>111<211>30<212>dna<213>智人(homosapiens)<400>111ttatgtgaattcagtacaagaattaagcac30<210>112<211>30<212>dna<213>智人(homosapiens)<400>112ctttcatttctgtttatgtgaattcagtac30<210>113<211>30<212>dna<213>智人(homosapiens)<400>113ttcacataaacagaaatgaaagaaaaacac30<210>114<211>30<212>dna<213>智人(homosapiens)<400>114atgccaactctctcatctcgcagtctgtac30<210>115<211>30<212>dna<213>智人(homosapiens)<400>115gagacattctcacatttccagtctaagtac30<210>116<211>23<212>dna<213>小家鼠(musmusculus)<400>116tgtttggtagtatagtaagtagg23<210>117<211>23<212>dna<213>小家鼠(musmusculus)<400>117ggtctagaaaagatcaagccagg23<210>118<211>23<212>dna<213>小家鼠(musmusculus)<400>118gccaggactgtgacctgataagg23<210>119<211>23<212>dna<213>小家鼠(musmusculus)<400>119tcaccttcacactttaaccaagg23<210>120<211>23<212>dna<213>小家鼠(musmusculus)<400>120caaggttgagacaatgttccagg23<210>121<211>23<212>dna<213>小家鼠(musmusculus)<400>121ccaattcatgtgcaaacatttgg23<210>122<211>23<212>dna<213>小家鼠(musmusculus)<400>122catcacagtttatacttagctgg23<210>123<211>23<212>dna<213>小家鼠(musmusculus)<400>123atcacagtttatacttagctggg23<210>124<211>23<212>dna<213>小家鼠(musmusculus)<400>124ggaatacctcaggctcaacaggg23<210>125<211>23<212>dna<213>小家鼠(musmusculus)<400>125tctctgtttcggaatacctcagg23<210>126<211>23<212>dna<213>小家鼠(musmusculus)<400>126ctgtcgactactttgatgaaagg23<210>127<211>23<212>dna<213>小家鼠(musmusculus)<400>127tgaaccaagatgattatttgtgg23<210>128<211>23<212>dna<213>小家鼠(musmusculus)<400>128atcttggttcatagaaatttggg23<210>129<211>23<212>dna<213>小家鼠(musmusculus)<400>129agccttgcatggcagagcttggg23<210>130<211>23<212>dna<213>小家鼠(musmusculus)<400>130acactttaaccaaggaaagaggg23<210>131<211>23<212>dna<213>小家鼠(musmusculus)<400>131taccagatcccctctttccttgg23<210>132<211>23<212>dna<213>小家鼠(musmusculus)<400>132catttggaggccaaaatacaagg23<210>133<211>23<212>dna<213>小家鼠(musmusculus)<400>133ccaaatgtttgcacatgaattgg23<210>134<211>23<212>dna<213>小家鼠(musmusculus)<400>134agtccagatgctgtccctgaagg23<210>135<211>23<212>dna<213>小家鼠(musmusculus)<400>135cgcaagccattcaaacacaaagg23<210>136<211>23<212>dna<213>小家鼠(musmusculus)<400>136tcaaaaccctgttgagcctgagg23<210>137<211>23<212>dna<213>小家鼠(musmusculus)<400>137cggaatacctcaggctcaacagg23<210>138<211>23<212>dna<213>小家鼠(musmusculus)<400>138gtcaaaatgtgaattctaacagg23<210>139<211>23<212>dna<213>小家鼠(musmusculus)<400>139ttatctattctattagagctcgg23<210>140<211>23<212>dna<213>小家鼠(musmusculus)<400>140atcaagtaatgaaatggacaagg23<210>141<211>23<212>dna<213>小家鼠(musmusculus)<400>141ctcccactgccttattaggcagg23<210>142<211>23<212>dna<213>小家鼠(musmusculus)<400>142agagctcaaatgggttctaaagg23<210>143<211>23<212>dna<213>小家鼠(musmusculus)<400>143accacattcaagagctcaaatgg23<210>144<211>23<212>dna<213>小家鼠(musmusculus)<400>144ttacagattggttacacttgggg23<210>145<211>23<212>dna<213>小家鼠(musmusculus)<400>145atcactgctgctactacttatgg23<210>146<211>23<212>dna<213>小家鼠(musmusculus)<400>146atacctgcctaataaggcagtgg23<210>147<211>23<212>dna<213>小家鼠(musmusculus)<400>147gatcaggagagtcagtgggatgg23<210>148<211>23<212>dna<213>小家鼠(musmusculus)<400>148ctattgtgagtctcagattaagg23<210>149<211>23<212>dna<213>小家鼠(musmusculus)<400>149tattacagattggttacacttgg23<210>150<211>23<212>dna<213>小家鼠(musmusculus)<400>150attacagattggttacacttggg23<210>151<211>23<212>dna<213>小家鼠(musmusculus)<400>151tacagattggttacacttggggg23<210>152<211>23<212>dna<213>小家鼠(musmusculus)<400>152acagattggttacacttgggggg23<210>153<211>30<212>dna<213>小家鼠(musmusculus)<400>153ctacttactatactaccaaacacaccgcac30<210>154<211>30<212>dna<213>小家鼠(musmusculus)<400>154aaagcctacttactatactaccaaacacac30<210>155<211>30<212>dna<213>小家鼠(musmusculus)<400>155caaaagcctacttactatactaccaaacac30<210>156<211>30<212>dna<213>小家鼠(musmusculus)<400>156gggtctgaatcaaaagcctacttactatac30<210>157<211>30<212>dna<213>小家鼠(musmusculus)<400>157agagtgggattctacaagtcaccttcacac30<210>158<211>30<212>dna<213>小家鼠(musmusculus)<400>158ggaaagaggggatctggtagcataaagtac30<210>159<211>30<212>dna<213>小家鼠(musmusculus)<400>159gggatctggtagcataaagtacagctacac30<210>160<211>30<212>dna<213>小家鼠(musmusculus)<400>160atctgtcactagcgacaagtgtagctgtac30<210>161<211>30<212>dna<213>小家鼠(musmusculus)<400>161tcatgtgcaaacatttggaggccaaaatac30<210>162<211>30<212>dna<213>小家鼠(musmusculus)<400>162gacatacagagagggggcggagagaaatac30<210>163<211>30<212>dna<213>小家鼠(musmusculus)<400>163atactgacgccatcacatcacagtttatac30<210>164<211>30<212>dna<213>小家鼠(musmusculus)<400>164taaaactataagctctctgtttcggaatac30<210>165<211>30<212>dna<213>小家鼠(musmusculus)<400>165tcatcaaagtagtcgacagtcaaagcatac30<210>166<211>30<212>dna<213>小家鼠(musmusculus)<400>166tgaattctaacaggaaaactcagaacatac30<210>167<211>30<212>dna<213>小家鼠(musmusculus)<400>167actgctgctactacttatggtgactagtac30<210>168<211>30<212>dna<213>小家鼠(musmusculus)<400>168agtcaccataagtagtagcagcagtgatac30<210>169<211>30<212>dna<213>小家鼠(musmusculus)<400>169cataagtagtagcagcagtgatactaatac30<210>170<211>30<212>dna<213>小家鼠(musmusculus)<400>170ttgaatggcttgcgaacaaagattaaacac30<210>171<211>30<212>dna<213>小家鼠(musmusculus)<400>171ttaatctttgttcgcaagccattcaaacac30<210>172<211>30<212>dna<213>小家鼠(musmusculus)<400>172ttgctgcatctctaacgtgaactctaacac30<210>173<211>30<212>dna<213>小家鼠(musmusculus)<400>173ttcacgttagagatgcagcaaagtctatac30<210>174<211>30<212>dna<213>小家鼠(musmusculus)<400>174tggaagcaactctaaatcaccacccgatac30<210>175<211>30<212>dna<213>小家鼠(musmusculus)<400>175ttccaaagttctgtcacccagtaaaaacac30<210>176<211>30<212>dna<213>小家鼠(musmusculus)<400>176ttcaagagctcaaatgggttctaaaggcac30<210>177<211>30<212>dna<213>小家鼠(musmusculus)<400>177ttgaatgtggtataagtgctaatatcatac30<210>178<211>30<212>dna<213>小家鼠(musmusculus)<400>178gtataagtgctaatatcatacaggaaacac30<210>179<211>30<212>dna<213>小家鼠(musmusculus)<400>179gtgtttcctgtatgatattagcacttatac30<210>180<211>30<212>dna<213>小家鼠(musmusculus)<400>180gactttgtgtttcctgtatgatattagcac30<210>181<211>30<212>dna<213>小家鼠(musmusculus)<400>181aaaacaattatcaggcagtgacagagacac30<210>182<211>30<212>dna<213>小家鼠(musmusculus)<400>182ccaagatactagagtagctgtgactggcac30<210>183<211>30<212>dna<213>小家鼠(musmusculus)<400>183ggcctatagccattcaaatggccaagatac30<210>184<211>30<212>dna<213>小家鼠(musmusculus)<400>184gtcccatctccctaagtctcgaatctgcac30<210>185<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>185cacagggcagtcagagaccc20<210>186<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>186gcaaacaaagttggacactg20<210>187<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>187agactcccgcccatcttctagaaa24<210>188<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>188aagtcgctctgagttgttatcagt24<210>189<211>19<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>189cagtgaaacgcaccagacg19<210>190<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>190aatctgcctaacaggaggtg20<210>191<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>191gagggaatggggaccaaaggcatt24<210>192<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>192tcatgtggggtgatgttcaggaag24<210>193<211>23<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>193agagcagctgacctgaggtccaa23<210>194<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>194cccaagggtagagtgcaagtaaac24<210>195<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>195gcatcctagctcatttggtctgct24<210>196<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>196gagaggattcctcatgaatgggat24<210>197<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>197accaaacactacacttggttactg24<210>198<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>198ctcccactagcaattttaaagtct24<210>199<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>199gaatgttcagcacaggtttccttg24<210>200<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>200ggtcaaaaggagctccatatttga24<210>201<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>201caggacacccatggccaaatccag24<210>202<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>202cagagcctcctgcagggatgtcaa24<210>203<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>203gcctgccaaggtgactctcatcta24<210>204<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>204tgcccaggctgatcttgaactcct24<210>205<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>205cccagagttaagaggttctttcct24<210>206<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>206gaagctactccagtgcaactagct24<210>207<211>23<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>207acgcagtctgttctgtgcagtgt23<210>208<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>208aggccttcccaaggaagaccctga24<210>209<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>209gctgatcactggccaaatccagct24<210>210<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>210gggaaacaatgggatcaagctgca24<210>211<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>211gcccctttgtaagttgaggagcat24<210>212<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>212ccctctacctctctcaatgggctt24<210>213<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>213cagacaagcaaatgctgagagatt24<210>214<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>214cctgtcattatgatgttcgctagt24<210>215<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>215ccagagttggcctcctacagagat24<210>216<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>216gtggatgccccactactgttcatt24<210>217<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>217tacccaatttgccagtctgtgtct24<210>218<211>23<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>218accaccaggcctgccctacaaga23<210>219<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>219tgtgaatttgatcctggcattatg24<210>220<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>220tacagacaagcagatgctgagaga24<210>221<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>221cagtcaacagagctctaacctcct24<210>222<211>23<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>222agcacctggttgcacatcaactt23<210>223<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>223catgtggtccctgaacgtgaatga24<210>224<211>23<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>224gtctgtcgcttgccctcttctct23<210>225<211>24<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>225atgcagggcctctagaccatttca24<210>226<211>23<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>226ctcagccctttgtgcactcacct23<210>227<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>227tgcacatcgcaaacatttcg20<210>228<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>228tgggtatcgcactgtgtcag20<210>229<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>229aggttcacatggcttgtggt20<210>230<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>230atatctgaaatgcccgcagg20<210>231<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>231tgcacatcgcaaacatttcg20<210>232<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>232tgggtatcgcactgtgtcag20<210>233<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>233tctttaaaggccttatctcc20<210>234<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>234ttctgcttgagaattcatcc20<210>235<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>235ctcctaatctttcacttagg20<210>236<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>236caaagcctggtataacatag20<210>237<211>19<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>237tcacttcgagcatctgtgg19<210>238<211>19<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>238ccaaatgacaggctgagct19<210>239<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>239agcaggaagtgaaggctaag20<210>240<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>240atgtaacgtggcaactctgg20<210>241<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>241gtgttgctctcgtcaattag20<210>242<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>242aggtgttgtacatggagaag20<210>243<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>243tgtgagccaccatacccagc20<210>244<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>244cctgcagtcctttgcggatc20<210>245<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>245tcgctgccagtataacatgc20<210>246<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>246aactccagtctctagactcg20<210>247<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>247aatagtttgacgttggagcc20<210>248<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>248actcccaacatgttctcctg20<210>249<211>19<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>249atcatcgctcacagagtcc19<210>250<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>250acgactgcaggatcttaatg20<210>251<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>251tggatggaggttgggaatcc20<210>252<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>252ttgaggcagcagcactctcc20<210>253<211>19<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>253agtctatcctagcagctcc19<210>254<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>254actgagaccagataatgcag20<210>255<211>19<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>255aagagatgcgagttgttcc19<210>256<211>19<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>256cctcttctactctgagtgg19<210>257<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>257acctggtttatcacaagcta20<210>258<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>258aacgtgaacagaaggatttc20<210>259<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>259atcactccatcagagtcagg20<210>260<211>20<212>dna<213>人工序列(artificialsequence)<220><223>引物<400>260tggctccttctattctctcc20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1