RNA水平检测PD-1和PD-L1表达量的试剂盒及方法与流程
本发明涉及生物医学
技术领域:
,具体而言,涉及一种rna水平检测pd-1和pd-l1表达量的试剂盒及方法。
背景技术:
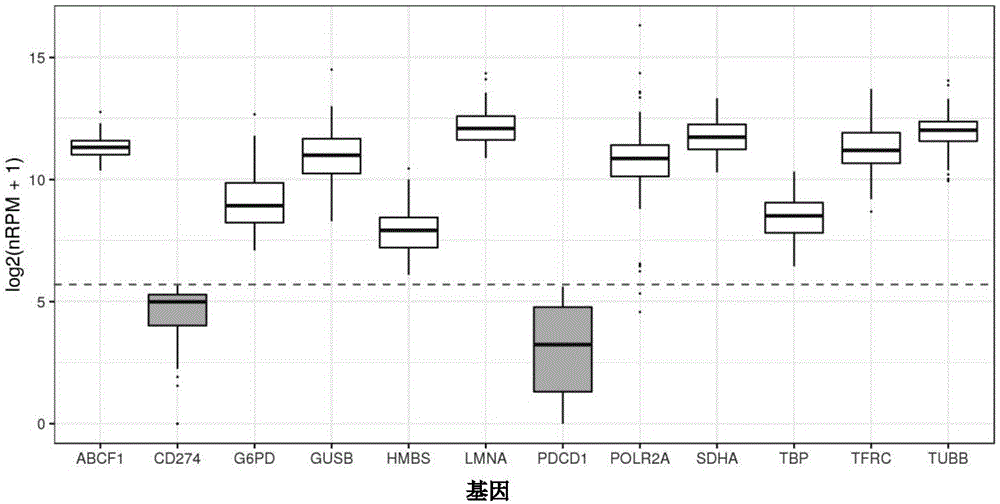
:随着国家食品药品监督总局(cfda)正式批准国内首个pd-1抑制剂纳武单抗的上市,预示着我国进入了免疫治疗新时代。肿瘤免疫治疗是通过调动机体自身的免疫系统,增强抗肿瘤免疫力,从而抑制和杀伤肿瘤细胞,它的出现改变了肿瘤传统的手术治疗、放化疗的格局,是当前肿瘤治疗领域中最具前景的研究方向之一。在肿瘤细胞和免疫效应细胞的相互作用中,肿瘤细胞表面的程序化死亡受体的配体(programmedcelldeath-ligand1,pd-l1)与免疫效应细胞(t淋巴细胞)表面的程序化死亡受体-1(programmedcelldeath-1,pd-1)的结合,传递免疫抑制信号,是肿瘤细胞逃避淋巴细胞杀伤的重要机制。研究表明,pd-1/pd-l1在多种肿瘤组织中发生明显表达上调,是一种广泛表达的生物标志物。以pd-1及pd-l1为靶点的免疫抑制剂,可以阻断pd-1与pd-l1的结合,阻断负向调控信号,使t细胞恢复活性,增强对抗肿瘤生长、转移的能力。因此,用合适的检测方法筛选出肿瘤组织或细胞高度表达pd-1/pd-l1的癌症患者,以便针对性的进行靶向药物的治疗,具有十分重要的临床意义。目前,pd-1/pd-l1的检测主要基于蛋白水平,临床实验中以免疫组化的方法(immunohistochemistry,ihc)为主,即对手术或穿刺后取得的肿瘤组织进行切片染色,抗体着色后,根据颜色深浅来判断表达情况。由此可知,除染色技术及条件外,抗体的特异性及病理评价标准也尤其重要,这些因素的存在导致现有检测试剂盒存在灵敏度低,特异性不高,稳定性差等缺点,使得我国pd-1/pd-l1的检测市场较为混乱,检测价值偏低。因此,临床上急需统一的可量化的检测方法筛选出高度表达pd-1/pd-l1的肿瘤患者,为个性化地免疫治疗提供指导和依据。技术实现要素:本发明旨在提供一种rna水平检测pd-1和pd-l1表达量的试剂盒及方法,以解决现有技术中免疫组化检测灵敏度低、特异性不高、稳定性差的技术问题。为了实现上述目的,根据本发明的一个方面,提供了一种rna水平检测pd-1和pd-l1表达量的试剂盒。该试剂盒包括:看家基因扩增引物、pd-1扩增引物和pd-l1扩增引物。进一步地,看家基因扩增引物扩增的看家基因包括gusb、hmbs、g6pd、polr2a、abcf1、tfrc、tbp、sdha、tubb和lmna。进一步地,看家基因扩增引物、pd-1扩增引物和pd-l1扩增引物均为按照跨外显子引物设计的原则设计的多重pcr引物。进一步地,pd-1扩增引物为seqidno:1~3和seqidno:37~39所示的核苷酸序列,pd-l1扩增引物为seqidno:4~6和seqidno:40~42所示的核苷酸序列,看家基因扩增引物为seqidno:7~36和seqidno:43~72所示的核苷酸序列。进一步地,试剂盒还包括反转录混合液、反转录酶、第一文库扩增混合液、第二文库扩增混合液、引物p5、引物p7、纯化磁珠和ph=8的idte缓冲液。进一步地,反转录混合液包括:80mmkcl、200mmph=8tris-hcl、20mmmgso4、每种dntp共10mm和20μm反转录随机引物;优选的,第一文库扩增混合液包括:80mmkcl、200mmph=8tris-hcl、20mmmgso4、每种dntp共2mm和酶活力50u的taqdna聚合酶;优选的,第二文库扩增混合液包括:80mmkcl、200mmph=8tris-hcl、20mmmgso4、每种dntp共2mm和酶活力200u的taqdna聚合酶;优选的,反转录随机引物为12bp的多聚胸腺嘧啶。根据本发明的另一方面,提供了一种rna水平检测pd-1和pd-l1表达量的方法。该方法采用上述任一种试剂盒进行检测。进一步地,包括以下步骤:s1,提取待测样本中的rna并反转录为cdna;s2,以cdna为模板特异性扩增看家基因、pd-1基因和pd-l1基因;s3,构建测序文库并进行测序;以及s4,对测序数据进行分析,得到pd-1和pd-l1表达量。进一步地,s4中对测序数据进行分析中,pd-1和pd-l1表达量是根据看家基因的测序数据量的倍数的中位数进行标准化,得到nrpm值。进一步地,s4中,若取待测样本的靶基因pd-l1的nrpm低于50则判为阴性样本,反之判为阳性样本。应用本发明的试剂盒或方法,可以从rna水平进行检测pd-1/pd-l1表达水平,对于无法用常规ihc(免疫组化,immunohistochemistry,ihc)检测的癌症病人的组织样本,特别是组织保存液浸泡过的组织样本或者是穿刺样本,本发明能够实现对这些样本pd-1/pd-l1表达水平的检测,主要是因为本发明的适用范围较广,所需样本量少,只要是能够提取rna的ffpe样本和组织样本均可进行检测。附图说明构成本申请的一部分的说明书附图用来提供对本发明的进一步理解,本发明的示意性实施例及其说明用于解释本发明,并不构成对本发明的不当限定。在附图中:图1示出了实施例1中12个基因在人群基线的nrpm分布图。具体实施方式需要说明的是,在不冲突的情况下,本申请中的实施例及实施例中的特征可以相互组合。下面将参考附图并结合实施例来详细说明本发明。为了克服现有ihc检测pd-1/pd-l1表达的不足,本发明提供一系列在rna水平检测pd-1和pd-l1表达量的试剂盒及方法。主要发明构思如下:与传统ihc在蛋白水平检测pd-1/pd-l1表达相比,rna水平检测pd-1/pd-l1表达量的原理是先将从癌症患者处取得的组织或ffpe样本进行rna提取,rna反转录成cdna后,用引物对其进行扩增,所得的扩增子用含有特异性序列的引物,进一步扩增后上机测序,测序数据与预设的基线对比,判定最终该患者pd-1/pd-l1表达量情况。根据本发明一种典型的实施方式,提供一种rna水平检测pd-1和pd-l1表达量的试剂盒。该试剂盒包括:看家基因扩增引物、pd-1扩增引物和pd-l1扩增引物。应用本发明的试剂盒,可以从rna水平进行检测pd-1/pd-l1表达水平,对于无法用常规ihc(免疫组化,immunohistochemistry,ihc)检测的癌症病人的组织样本,特别是组织保存液浸泡过的组织样本或者是穿刺样本,本发明能够实现对这些样本pd-1/pd-l1表达水平的检测,主要是因为本发明的适用范围较广,所需样本量少,只要是能够提取rna的ffpe样本和组织样本均可进行检测。优选的,看家基因扩增引物扩增的看家基因包括gusb、hmbs、g6pd、polr2a、abcf1、tfrc、tbp、sdha、tubb和lmna,这些看家基因表达稳定,可以在后续测序数据分析时作为参照进行pd-1/pd-l1表达水平的判定。优选的,看家基因扩增引物、pd-1扩增引物和pd-l1扩增引物均为按照跨外显子引物设计的原则设计的多重pcr引物,以排除dna干扰,可通过多重pcr扩增,优选的扩增得到约110bp的扩增子。更优选的,pd-1扩增引物为seqidno:1~3和seqidno:37~39所示的核苷酸序列,pd-l1扩增引物为seqidno:4~6和seqidno:40~42所示的核苷酸序列,看家基因扩增引物为seqidno:7~36和seqidno:43~72所示的核苷酸序列,这些引物的扩增特异性较好。根据本发明一种典型的实施方式,试剂盒还包括反转录混合液、反转录酶、第一文库扩增混合液、第二文库扩增混合液、引物p5(seqidno:73,aatgatacggcgaccaccgagatctacacnnnnnnnnacactctttccctacacgacgctcttccgatct)、引物p7(seqidno:74,gatcggaagagcacacgtctgaactccagtcacnnnnnnnnatctcgtatgccgtcttctgcttg)、纯化磁珠和ph=8的idte缓冲液(10mmtris,0.1mmedta)。优选的,反转录混合液包括:80mmkcl、200mmph=8tris-hcl、20mmmgso4、每种dntp共10mm和20μm反转录随机引物;优选的,第一文库扩增混合液包括:80mmkcl、200mmph=8tris-hcl、20mmmgso4、每种dntp共2mm和酶活力50u的taqdna聚合酶;优选的,第二文库扩增混合液包括:80mmkcl、200mmph=8tris-hcl、20mmmgso4、每种dntp共2mm和酶活力200u的taqdna聚合酶。优选的,反转录随机引物为12bp的多聚胸腺嘧啶。根据本发明一种典型的实施方式,提供一种rna水平检测pd-1和pd-l1表达量的方法。该方法采用上述任一种试剂盒进行检测。优选的,包括以下步骤:s1,提取待测样本中的rna并反转录为cdna;s2,以cdna为模板特异性扩增看家基因、pd-1基因和pd-l1基因;s3,构建测序文库并进行测序;以及s4,对测序数据进行分析,得到pd-1和pd-l1表达量。应用本发明的方法,可以从rna水平进行检测pd-1/pd-l1表达水平,对于无法用常规ihc(免疫组化,immunohistochemistry,ihc)检测的癌症病人的组织样本,特别是组织保存液浸泡过的组织样本或者是穿刺样本,本发明能够实现对这些样本pd-1/pd-l1表达水平的检测,主要是因为本发明的适用范围较广,所需样本量少,只要是能够提取rna的ffpe样本和组织样本均可进行检测。优选的,s4中对测序数据进行分析中,pd-1和pd-l1表达量是根据看家基因的测序数据量的倍数的中位数进行标准化,得到nrpm值。也就是说,pd-1和pd-l1表达量结果可以是根据10个看家基因(hk,house-keepinggenes)测序数据量的倍数(hk的rpm/hk标准值rpm)的中位数进行标准化,得到nrpm值。在hk表达稳定的基础上,根据靶基因的nrpm值判断样本pd-1/pd-l1表达情况。在本发明一种典型的实施方式,基于ihc检测平台,选取70例pd-1/pd-l1表达阴性的病人样本,对这些样本rna进行提取,建库,上机测序等,选取看家基因较稳定,且on_target_ratio>=0.6,mapped_reads>200k的69例样本,确定pd-l1在阴性样本中的cutoff值为nrpm=50。所以,优选的,s4中,若取待测样本的靶基因pd-l1的nrpm低于50则判为阴性样本,反之判为阳性样本。基于此,本发明的评判标准的准确性得到了进一步的提高。在本发明一种典型的实施方式,rna水平检测pd-1和pd-l1表达量的方法具体包括以下步骤:1.rna反转录为cdna1.1按照rna提取试剂盒(可为常规商购试剂盒)说明书要求进行rna提取,取10~100ngrna,若样本为ffperna时,需80℃孵育10分钟,室温冷却,按照如下表1格配制反应体系。表11.2将rna和反应体系充分混匀,放入pcr仪中,按照如表2所示的反应程序将rna反转成cdna。表2温度时间25℃10min42℃60min85℃5min4℃hold2.特异性扩增扩增子2.1冰上融化文库扩增mix1(第一文库扩增混合液),10×正向扩增引物和10×反向扩增引物,短暂涡旋并离心。2.2用idte(ph8.0)将10ngcdna稀释到11μl。2.3如下表3所示,在96孔板中准备pcr体系:表3pcr试剂体积文库扩增mix15μl10×正向扩增引物2μl10×反向扩增引物2μl样本cdna11μl总体系20μl2.4封闭96孔板,彻底混匀并离心。2.5将pcr板放到热循环仪中,进行如表4中所示的程序,热盖温度设为105℃。表4其中,扩增循环数优选为10,扩增温度优选为61℃。2.6继续进行磁珠纯化。3.扩增子纯化3.1使用之前,室温平衡磁珠30min,彻底涡旋混匀。3.2每个反应中加入30μl磁珠,移液器上下吹打混匀。3.3室温孵育10min。3.4在磁力架上放置5min收集磁珠,直到液体澄清,弃掉上清。3.5每个反应加入200μl80%乙醇,室温孵育1min。3.6弃上清。3.7再次加入200μl80%乙醇,进行第二次清洗。3.8第二次清洗后,弃掉所有乙醇残留,避免碰到磁珠。3.9室温干燥3min,避免过干。3.10将pcr板从磁力架上移开,加入15μlidte(ph8.0),移液器吹打混匀。3.11室温孵育3min,将样品从磁珠上洗脱下来。3.12将pcr板放到磁力架上3min,收集磁珠。3.13转移11μl样本到新的pcr板上,确保没有吸到磁珠。3.14继续进行pcr扩增。4.文库扩增4.1在新的pcr板或1.5ml管中,用idte(ph8.0)将引物p5和p7稀释成5μm,4℃存放5μm的稀释液以减少反复冻融,若长时间保存原液和稀释液,推荐使用-20℃。冰上融化文库扩增mix2(第二文库扩增混合液),短暂涡旋并离心。4.2如下表5所示,在96孔板中准备pcr体系。表5pcr试剂体积文库扩增mix25μl引物p5(5μm)2μl引物p7(5μm)2μl洗脱样品11μl总体系20μl4.3封闭96孔板,彻底混匀并离心。4.4将pcr板放到热循环仪中,进行如下表6所示的程序,热盖温度设为105℃。表64.5将pcr板从热循环仪中移开。4.6继续进行文库纯化。5.文库纯化5.1使用之前,室温平衡磁珠30min,彻底涡旋混匀。5.2每个反应中加入20μl磁珠,移液器上下吹打混匀。5.3室温孵育10min。5.4在磁力架上放置5min收集磁珠,直到液体澄清,弃掉上清。5.5每个反应加入200μl80%乙醇,室温孵育1min。5.6弃上清。5.7再次加入200μl80%乙醇,进行第二次清洗。5.8第二次清洗后,弃掉所有乙醇残留,避免碰到磁珠。5.9室温干燥3min,避免过干。5.10将pcr板从磁力架上移开,加入22μlidte(ph8.0),移液器吹打混匀。5.11室温孵育3min,将样品从磁珠上洗脱下来。5.12将pcr板放到磁力架上3min,收集磁珠。5.13转移20μl样本到新的低吸管或者pcr板中,确保没有吸到磁珠。5.14根据illumina平台的标准化流程对文库进行定量和测序。6.结果判定当hk>=6,on_target_ratio>=0.6,mapped_reads>200k时,样本测序结果通过质控,可进一步判定pd-1/pd-l1的表达情况。确定pd-l1在阴性样本中的cutoff值为nrpm=50,若所测样本靶基因pd-l1的nrpm低于50为阴性样本,反之为阳性样本。下面将结合实施例进一步说明本发明的有益效果。本发明中如果没有明确写出的方法或试剂可采用本领域常规的方法或试剂实现。实施例1一、panel的设计与制备1.panel设计原则本panel选择了12个基因,其中包括pd-1、pd-l1和10个housekeeping基因,具体基因如表7所示。在本实施例中,每个基因上设计3个pcr区域,取三个区域的平均值作为表达量,进行标准化,得到nrpm值。引物设计原则为跨外显子引物设计,以排除dna干扰,通过多重pcr扩增,得到扩增子长度约为110bp。pd-1和pd-l1结果是根据10个hk基因的倍数=(hk的rpm)/(hk标准值rpm)的中位数进行标准化,得到nrpm值。表72.引物重悬与合并2.1引物的稀释合成后的引物,每个孔中含有0.4nmol的引物,将其稀释成50μm(0.05nmol/μl),即加入8μlidteph7.5。2.2合并10×正向扩增引物和反向扩增引物例如,每个引物取1μl,将36个正向引物合并在一起,形成10×正向扩增引物。同理适用于反向扩增引物。10×正向扩增引物的浓度为:2μl×50μm/72μl=1.4μm,正向和反向引物各得72μl。直到扩增时,才将正反向引物合并到一起。正反向引物合并到一起使用即为专利中所使用的panel。二、基线的确定与结果判读1.rna反转录为cdna1.1选取70例ihc检测pd-1/pd-l1为阴性的癌症病人的ffpe样本,按照rna提取试剂盒(truxtracffpernamicrotubekit,520161)说明书要求进行rna提取,取10~100ngrna,若样本为ffperna时,需80℃孵育10分钟,室温冷却,按照表1配制反应体系。1.2将rna和反应体系充分混匀,放入pcr仪中,按照如表2所示的反应程序将rna反转成cdna。2.特异性扩增扩增子2.1冰上融化文库扩增mix1(第一文库扩增混合液),10×正向扩增引物和10×反向扩增引物,短暂涡旋并离心。2.2用idte(ph8.0)将10ngcdna稀释到11μl。2.3如下表3所示,在96孔板中准备pcr体系:2.4封闭96孔板,彻底混匀并离心。2.5将pcr板放到热循环仪中,进行如表4中所示的程序,热盖温度设为105℃。其中,扩增循环数为10,扩增温度为61℃。2.6继续进行磁珠纯化。3.扩增子纯化3.1使用之前,室温平衡磁珠30min,彻底涡旋混匀。3.2每个反应中加入30μl磁珠,移液器上下吹打混匀。3.3室温孵育10min。3.4在磁力架上放置5min收集磁珠,直到液体澄清,弃掉上清。3.5每个反应加入200μl80%乙醇,室温孵育1min。3.6弃上清。3.7再次加入200μl80%乙醇,进行第二次清洗。3.8第二次清洗后,弃掉所有乙醇残留,避免碰到磁珠。3.9室温干燥3min,避免过干。3.10将pcr板从磁力架上移开,加入15μlidte(ph8.0),移液器吹打混匀。3.11室温孵育3min,将样品从磁珠上洗脱下来。3.12将pcr板放到磁力架上3min,收集磁珠。3.13转移11μl样本到新的pcr板上,确保没有吸到磁珠。3.14继续进行pcr扩增。4.文库扩增4.1在新的pcr板或1.5ml管中,用idte(ph8.0)将引物p5和p7稀释成5μm,4℃存放5μm的稀释液以减少反复冻融,若长时间保存原液和稀释液,推荐使用-20℃。冰上融化文库扩增mix2(第二文库扩增混合液),短暂涡旋并离心。4.2如下表5所示,在96孔板中准备pcr体系。4.3封闭96孔板,彻底混匀并离心。4.4将pcr板放到热循环仪中,进行如下表6所示的程序,热盖温度设为105℃。4.5将pcr板从热循环仪中移开。4.5将pcr板从热循环仪中移开。4.6继续进行文库纯化。5.文库纯化5.1使用之前,室温平衡磁珠30min,彻底涡旋混匀。5.2每个反应中加入20μl磁珠,移液器上下吹打混匀。5.3室温孵育10min。5.4在磁力架上放置5min收集磁珠,直到液体澄清,弃掉上清。5.5每个反应加入200μl80%乙醇,室温孵育1min。5.6弃上清。5.7再次加入200μl80%乙醇,进行第二次清洗。5.8第二次清洗后,弃掉所有乙醇残留,避免碰到磁珠。5.9室温干燥3min,避免过干。5.10将pcr板从磁力架上移开,加入22μlidte(ph8.0),移液器吹打混匀。5.11室温孵育3min,将样品从磁珠上洗脱下来。5.12将pcr板放到磁力架上3min,收集磁珠。5.13转移20μl样本到新的低吸管或者pcr板中,确保没有吸到磁珠。5.14根据illumina平台的标准化流程对文库进行定量和测序。6.基线的确定从测序所得的70例样本中,筛选出了69例hk>=6,on_target_ratio>=0.6,mapped_reads>200k的样本,用于基线的确定。表7中的12个基因在人群基线的nrpm分布如图1所示,其中,图中黑色无填充箱线图代表10个hk基因的表达量,黑色填充箱线图代表2个靶基因的表达量,黑色虚线为cutoff值为50时,log2(nrpm+1)的值,可以看出pd-1和pd-l1在人群基线中的nrpm均低于50,故将cutoff值定为50。则样本阴阳性的判定标准为:当hk>=6,on_target_ratio>=0.6,mapped_reads>200k时,样本测序结果通过质控,可进一步判定pd-1/pd-l1的表达情况。确定pd-l1在阴性样本中的cutoff值为nrpm=50,若所测样本靶基因pd-l1的nrpm低于50为阴性样本,反之为阳性样本。实施例2取10例ngs检测pd-1或pd-l1为阳性的肿瘤患者的ffpe样本进行验证。选取10例ngs检测pd-1/pd-l1为样本的肿瘤患者的ffpe样本,依次进行rna反转录为cdna、特异性扩增扩增子、扩增子纯化、文库扩增、文库纯化和数据分析等,具体的操作步骤均与实施例1中“二、基线的确定与结果判读”中的步骤一致。结果判定10例ffpe样本的测序结果如下表8所示:表8质控标准:当hk>=6,on_target_ratio>=0.6,mapped_reads>200k时,样本测序结果通过质控,可进一步判定pd-1/pd-l1的表达情况。确定pd-l1在阴性样本中的cutoff值为nrpm=50,若所测样本靶基因pd-l1的nrpm低于50为阴性样本,反之为阳性样本。结论:从表8中可以看出,10例样本都通过质控,且均为pd-1/pd-l1表达阳性。说明本专利与ngs测序结果非常一致。实施例3取10例ngs为pd-1或pd-l1阳性的肿瘤患者的组织进行验证。选取10例ngs检测pd-1/pd-l1为样本的肿瘤患者的组织样本,依次进行rna反转录为cdna、特异性扩增扩增子、扩增子纯化、文库扩增、文库纯化和数据分析等,具体的操作步骤参见实施例1中“二、基线的确定与结果判读”中的步骤,与之不同的是本实施例中的组织样本不是ffpe样本,是新鲜组织样本,所以不需若样本为ffperna时,需80℃孵育10分钟,其他相同。结果判定10例组织样本的测序结果如下表9所示:表9质控标准:当hk>=6,on_target_ratio>=0.6,mapped_reads>200k时,样本测序结果通过质控,可进一步判定pd-1/pd-l1的表达情况。确定pd-l1在阴性样本中的cutoff值为nrpm=50,若所测样本靶基因pd-l1的nrpm低于50为阴性样本,反之为阳性样本。结论:从表9中可以看出,10例样本都通过质控,且均为pd-1/pd-l1表达阳性。说明本专利与ngs测序结果非常一致。比较例1取10例未知pd-1或pd-l1表达量的肿瘤患者的病理切片进行ihc和本专利的方法(同实施例2步骤)验证。ihc检测将10例样本在蛋白水平上进行pd-1和pd-l1ihc检测,并记录检测结果。结果判定与比较见表10。表10质控标准:当hk>=6,on_target_ratio>=0.6,mapped_reads>200k时,样本测序结果通过质控,可进一步判定pd-1/pd-l1的表达情况。pd-1/pd-l1阴阳性判定标准:确定pd-l1在阴性样本中的cutoff值为nrpm=50,若所测样本靶基因pd-l1的nrpm低于50为阴性样本,反之为阳性样本。结论:从表10中可以看出,10例样本都通过质控,且均为pd-1/pd-l1表达阳性,与ihc结果完全一致。从以上的描述中,可以看出,本发明上述的实施例实现了如下技术效果:本发明对pd-1/pd-l1的检测是在rna表达水平,检测结果较为准确,可量化,没有主观差异,不涉及染色技术,抗体的特异性及病理评价标准等不确定因素。以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。序列表<110>臻悦生物科技江苏有限公司<120>rna水平检测pd-1和pd-l1表达量的试剂盒及方法<130>pn103141zhkej<141>2019-01-18<160>74<170>siposequencelisting1.0<210>1<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>1agagtgccgccttctccact20<210>2<211>18<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(18)<223>引物<400>2ttcggtcaccacgagcag18<210>3<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>3tgcgcctggctcctattgt19<210>4<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>4agttctgcgcagcttcccga20<210>5<211>21<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(21)<223>引物<400>5ccgctgcatgatcagctatgg21<210>6<211>21<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(21)<223>引物<400>6cacttggtaattctgggagcc21<210>7<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>7ggtgtcgtgatgcgcctgaa20<210>8<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>8tcatgctctagcgtgtcga19<210>9<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>9gcaggttgaagtccttcacc20<210>10<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>10cccacacacagcctactttc20<210>11<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>11catgatgctgttgtctttca20<210>12<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>12gtctggagtctagacggctc20<210>13<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>13atagcccacgatgaaggtg19<210>14<211>21<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(21)<223>引物<400>14ggataacgcaggcgatgttgt21<210>15<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>15ggaccacattgttggcctg19<210>16<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>16gattgagcggactggccgct20<210>17<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>17gattgtcacagtgctgcctg20<210>18<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>18gacttgcgtttccaccccaa20<210>19<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>19ggctggggaagaagagaaag20<210>20<211>21<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(21)<223>引物<400>20ggcagatgatccctatgctca21<210>21<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>21gccaaccgagccctgagcat20<210>22<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>22gttatcgccatctacttgcc20<210>23<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>23ggtccatgtatatcaacac19<210>24<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>24ccatcctacatgtagagtct20<210>25<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>25cgcttccgctggcccatagt20<210>26<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>26cgccagcttcggagagttct20<210>27<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>27cagaatgatcaaacccagaa20<210>28<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>28ggaacccgaggttttcactt20<210>29<211>21<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(21)<223>引物<400>29ccaggatgccatccactacat21<210>30<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>30gcatagaggacgggtccat19<210>31<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>31gggaaatcgtgcacatcca19<210>32<211>21<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(21)<223>引物<400>32gcagctggaccgcatctctgt21<210>33<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>33ggccagatctttagaccaga20<210>34<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>34gctggagctgagcaaagtg19<210>35<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>35gctgagaacaggctgcaga19<210>36<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>36catccgcatcgacagcctct20<210>37<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>37ggcgcagatcaaagagagcc20<210>38<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>38cagcctggtgctgctagtct20<210>39<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>39gagtggctgggacaagggat20<210>40<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>40cagcaaatgccagtaggtc19<210>41<211>21<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(21)<223>引物<400>41gaggtgactggatccacaacc21<210>42<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>42gtttgtatcttggatgccac20<210>43<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>43gcgcacaagagtggtgctga20<210>44<211>21<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(21)<223>引物<400>44ggactgcagcggtctgtactt21<210>45<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>45gatgtggtctgtggccaac19<210>46<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>46ctggctcttgcgggtaccc19<210>47<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>47ctgcagctgggctgctctt19<210>48<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>48cagtgatgcctaccaactgt20<210>49<211>21<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(21)<223>引物<400>49ctacgaggccgtcaccaagaa21<210>50<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>50tctggtggccatggagaagc20<210>51<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>51catgttcttcaaccccgagg20<210>52<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>52caccaggaagcccacgtga19<210>53<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>53gatcttcacgatgtcagcca20<210>54<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>54gtgcagagttggctgccgg19<210>55<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>55ccacatccttcttccgcct19<210>56<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>56gtcattttcagctgcattgg20<210>57<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>57cgaagaacagcctggactgc20<210>58<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>58gttgtcggagaaactggaca20<210>59<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>59gcctaatatacctgtccaga20<210>60<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>60gtaggcacagctctcctatt20<210>61<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>61gcacactcttctcagcaac19<210>62<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>62cggcgtttcgggcacgaagt20<210>63<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>63gaatagggtagatgttttc19<210>64<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>64ctgcatcaaattcatgatcc20<210>65<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>65cgctgataaatcttcccatc20<210>66<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>66agtgctggtgtgggcagacg20<210>67<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>67tgtccccgtggtaggtgcc19<210>68<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>68cctgagcgaacagagtccat20<210>69<211>19<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(19)<223>引物<400>69ggacagaatcaaccagctc19<210>70<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>70cctccaggtccttcagccga20<210>71<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>71attgtcaatctccaccagtc20<210>72<211>20<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(20)<223>引物<400>72ctcacgggccagtgagtcct20<210>73<211>70<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(70)<223>引物<220><221>misc_feature<222>(30)..(30)<223>nisa,c,g,toru<220><221>misc_feature<222>(31)..(31)<223>nisa,c,g,toru<220><221>misc_feature<222>(32)..(32)<223>nisa,c,g,toru<220><221>misc_feature<222>(33)..(33)<223>nisa,c,g,toru<220><221>misc_feature<222>(34)..(34)<223>nisa,c,g,toru<220><221>misc_feature<222>(35)..(35)<223>nisa,c,g,toru<220><221>misc_feature<222>(36)..(36)<223>nisa,c,g,toru<220><221>misc_feature<222>(37)..(37)<223>nisa,c,g,toru<400>73aatgatacggcgaccaccgagatctacacnnnnnnnnacactctttccctacacgacgct60cttccgatct70<210>74<211>65<212>dna<213>artificialsequence<220><221>primer_bind<222>(1)..(65)<223>引物<220><221>misc_feature<222>(34)..(34)<223>nisa,c,g,toru<220><221>misc_feature<222>(35)..(35)<223>nisa,c,g,toru<220><221>misc_feature<222>(36)..(36)<223>nisa,c,g,toru<220><221>misc_feature<222>(37)..(37)<223>nisa,c,g,toru<220><221>misc_feature<222>(38)..(38)<223>nisa,c,g,toru<220><221>misc_feature<222>(39)..(39)<223>nisa,c,g,toru<220><221>misc_feature<222>(40)..(40)<223>nisa,c,g,toru<220><221>misc_feature<222>(41)..(41)<223>nisa,c,g,toru<400>74gatcggaagagcacacgtctgaactccagtcacnnnnnnnnatctcgtatgccgtcttct60gcttg65当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1