一种鉴别四种镰刀菌的方法与流程
本发明涉及多种真菌检测技术领域,具体地说涉及一种鉴别四种镰刀菌的方法,所述四种镰刀菌为层生镰刀菌(fusariumproliferatum),尖孢镰刀菌(fusariumoxysporumschl.),半裸镰刀菌(fusariumsemitectum),腐皮镰刀菌(fusariumsolani)。
背景技术:
镰刀菌属于有丝分裂孢子真菌,广泛存在于土壤中和植物上,属于条件致病菌。镰刀菌属中有许多危害经济植物的种,它们是植物维管束系统的寄生菌,在适宜的环境条件下,不但破坏作物的输导组织维管束,而且在菌体生长发育代谢过程中产生毒素危害作物,造成作物萎蔫、穗腐、腐烂、死亡,影响产量和品质,严重时可导致产量显著下降。致病性尖孢镰刀菌侵染植物引起植物枯萎病,导致维管束病害,造成植株枯死,其在植株的全生育期均可发生,对生产造成巨大损失。腐皮镰刀菌可引起多种经济作物根腐病,还可引起一些植物的果实腐烂,如甜辣椒果腐病。半裸镰刀菌侵染双孢蘑菇子实体后,一般表现为生长发育受阻、生长无力,子实体颜色淡黄,严重者变成长大的僵菇或萎缩菇。
镰刀菌产生的毒素种类繁多,其中主要是玉米赤霉烯酮、单端孢霉毒素、串珠镰刀菌素和伏马菌素等,对人体和家畜都具有巨大危害。层生镰刀菌可引起角膜炎、眼内炎、甲真菌病(甲癣)、浅表性化脓性血栓性静脉炎,免疫力低下患者可致严重的系统感染甚至危及生命。研究显示,我国中原地区真菌性角膜炎感染镰刀菌以腐皮镰刀菌复合体最多,尖孢镰刀菌复合体居第三位。人进食被镰刀菌侵染的麦子或玉米后半小时至一小时可出现中毒症状,即醉谷病。拟枝孢镰刀菌等侵染谷物后,产生的毒素可侵害人造血系统和其他器官,致使白血细胞和血小板大量减少,导致中毒性白细胞缺乏症;其侵染牛毛草也可使牛食后出现失重、驼背、烂蹄、牛尾坏死等症状。镰刀菌毒素难以分解、毒素活性检测困难、有效的脱毒过程较为复杂,故对食品和农作物真菌污染的早期鉴定显得尤为重要。
真菌核糖体基因转录间隔区(internaltranscribedspacer,its),又叫内转录间隔区,是位于真菌核糖体dna(rdna)上18s和28s基因之间的区域片段。rdna在种内由于基因的流动而经常表现出很高的同源性,在种间则保持着各种程度的变异。变异的多少能够反映生物进化上属内种间亲缘关系的远近。its1和its2作为非编码区,承受的进化选择压力较小,其保守性基本上表现为种内相对一致,种间差异比较明显。这种特点使its适合于真菌物种的分子鉴定以及属内物种间或种内差异较明显的菌群间的系统发育关系分析。
传统的镰刀菌分类鉴定方法主要依靠其形态特征,比较费时,常常需要2~3周才能得到最终结果。有时培养物不典型或不产孢,使得鉴定困难。除形态学培养鉴定外,常用通用引物its1、its4对rdnaits基因pcr扩增后测序,将测序结果在ncbi进行基本局部比对,根据比对结果判断菌种,但有时该方法难以对所有真菌菌株进行准确鉴定,存在无法扩增的现象。此外,还常用pcr-rflp鉴定镰刀菌种,即比较不同镰刀菌rdnaits序列的限制型内切酶位点,设计特异性引物进行pcr扩增,在pcr扩增后用该限制酶切割pcr产物,所得扩增片段具有高度多态性,这些不同长度的等位片段可用page分离,从而区分不同菌种。上述方法虽然普遍应用,但耗时较长,可能延误治疗,使患者病情加重,或者无法及时鉴定病原影响食品安全监测的时效性。
技术实现要素:
本发明的目的是为了弥补现有技术的不足,提供一种基于rt-pcr熔解曲线分析的鉴别四种镰刀菌的方法,特别是一种利用高分辨熔解曲线分析及时鉴别层生镰刀菌(fusariumproliferatum),尖孢镰刀菌(fusariumoxysporumschl.),半裸镰刀菌(fusariumsemitectum),腐皮镰刀菌(fusariumsolani)的方法。
本发明的另一目的在于提供一种能同时鉴别层生镰刀菌(fusariumproliferatum),尖孢镰刀菌(fusariumoxysporumschl.),半裸镰刀菌(fusariumsemitectum),腐皮镰刀菌(fusariumsolani)的一对特异性引物。
本发明应用遗传分析方法——高分辨熔解曲线(highresolutionmelting,hrm)分析技术。通过实时监测升温过程中双链dna荧光染料与pcr产物的结合情况,its序列不同会使双链dna的tm值发生变化,从而双链dna在升温过程中先后解链,形成不同的熔解曲线形状。荧光染料从局部解链的dna分子上释放,从荧光强度和时间曲线上就能判断是否存在有差异的its片段,且its序列的差异会影响熔解曲线的峰形,能有效区分不同菌种的its序列。
为了实现本发明的目的,本发明仔细研究了层生镰刀菌(fusariumproliferatum),尖孢镰刀菌(fusariumoxysporumschl.),半裸镰刀菌(fusariumsemitectum),腐皮镰刀菌(fusariumsolani)这四种镰刀菌的基因组序列,在its序列中寻找差异性靶序列,通过精心设计和筛选,设计一对特异性引物,使用该对引物通过hrm技术能够特异地鉴别上述四种镰刀菌。
本发明首先提供了能鉴别上述四种镰刀菌的特异性引物,其核苷酸序列如seqidno.1-2所示。
本发明提供了含有上述特异性引物的试剂盒或检测试剂。
本发明提供了所述特异性引物在鉴别层生镰刀菌(fusariumproliferatum),尖孢镰刀菌(fusariumoxysporumschl.),半裸镰刀菌(fusariumsemitectum),腐皮镰刀菌(fusariumsolani)中的应用。
本发明提供了上述的特异性引物在保障食品安全中的应用。
本发明提供一种鉴别层生镰刀菌(fusariumproliferatum),尖孢镰刀菌(fusariumoxysporumschl.),半裸镰刀菌(fusariumsemitectum),腐皮镰刀菌(fusariumsolani)的方法,是应用上述的特异性引物,以待测样品的dna为模板,使用rt-pcr方法进行检测,根据熔解峰值图中熔解曲线的峰形判定结果。
上述rt-pcr方法中30μlpcr反应体系为:2×mixtaqmanpcrmaster15μl、上下游引物各0.9μl、roxreferencedyeⅱ(100×)0.3μl、evagreen,20×inwater1.5μl、待测dna模板2μl,用灭菌纯水补足体系至30μl。
rt-pcr反应条件为:预变性:95℃10min;95℃15s,58℃25s,72℃25s,35个循环,升降温速度1.6℃/s。
本发明上述方法的熔解曲线制作条件为:95℃10s,60℃1min,(0.025℃/s升温至)95℃15s,60℃15s,以1.6℃/s速率升降温,同时连续检测荧光强度;以荧光信号对温度的一阶负导数为纵坐标,温度为横坐标,得到熔解峰值图。
具体地,本发明方法鉴别结果的判断原则是每一种特异性扩增产物tm值的熔解峰代表一种特定的镰刀菌,
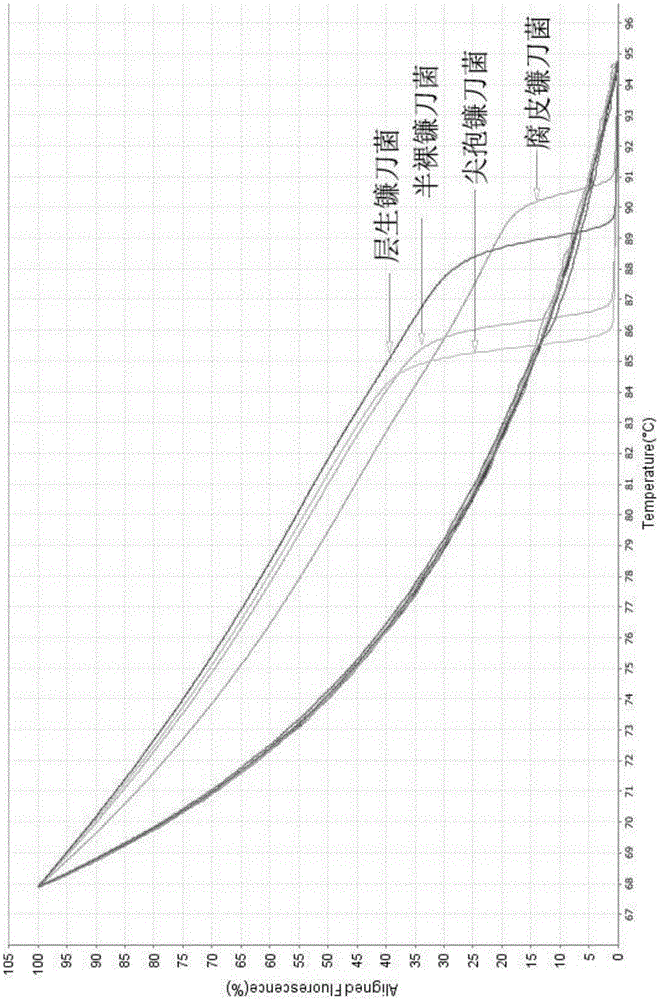
其中,tm值在87~89℃范围内出现熔解峰为层生镰刀菌(fusariumproliferatum)检出;tm值在84~85℃范围内出现熔解峰为尖孢镰刀菌(fusariumoxysporumschl.)检出;tm值在85~86℃范围内出现熔解峰为半裸镰刀菌(fusariumsemitectum)检出;tm值在89~90范围内出现熔解峰为腐皮镰刀菌(fusariumsolani)检出。
本发明提供了上述方法在农业生产、食品安全监测中的应用。
本发明的rt-pcr熔解曲线分析方法采用全密闭反应管检测与结果分析,无需进行后续pcr产物电泳检测与分析,简化操作流程、降低实验检测成本,提高检测效率、避免样品和环境的交叉污染。同时本发明的方法具有良好的特异性,保证该方法能够特异性的检出层生镰刀菌(fusariumproliferatum),尖孢镰刀菌(fusariumoxysporumschl.),半裸镰刀菌(fusariumsemitectum),腐皮镰刀菌(fusariumsolani),避免了假阳性。
本发明的hrm分析方法还具有较高的灵敏度,对上述四种镰刀菌检测下限为10-100拷贝,具有很高灵敏度,且标准峰形区分明显,能有效区分前述不同菌种的its序列。本发明采用hrm分析方法无需设计特异性探针,与其他鉴别菌种的方法相比,操作简单,具有灵敏度高、特异性好、成本低、快速、高通量检测等优点,pcr扩增后无需进行电泳,实现闭管操作,防止交叉污染。本发明方法可用于临床诊断、农业生产、食品安全等方面镰刀菌种的鉴别,从而为镰刀菌的感染治疗、农业生产和食品安全的监测提供确实可靠的依据。
附图说明
图1为本发明鉴别四种镰刀菌的高分辨熔解峰值图谱。
图2为本发明方法对四种镰刀菌dna模板的检测限结果图。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改或替换,均属于本发明的范围。
若未特别指明,实施例中所用的化学试剂均为常规市售试剂,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。
实施例1鉴别四种镰刀菌方法的建立和特性验证
本发明仔细研究了层生镰刀菌(fusariumproliferatum),尖孢镰刀菌(fusariumoxysporumschl.),半裸镰刀菌(fusariumsemitectum),腐皮镰刀菌(fusariumsolani)这四种镰刀菌的基因组序列,在its序列中寻找差异性靶序列,通过精心设计和筛选,设计一对特异性引物,其核苷酸序列如seqidno.1-2所示,使用该对引物通过hrm技术能够特异地鉴别上述四种镰刀菌。该引物的设计无法通过计算机软件自动得出,需要依据丰富的经验选取靶序列片段和长度,在考虑序列gc含量的同时依据以下公式计算tm值。合成引物后进行反复试验,得到一对能够区分上述四种镰刀菌的最佳引物。
利用高分辨熔解曲线分析技术鉴别四种镰刀菌,其步骤如下:
1.采用粗提法提取待测真菌样本的基因组dna,凝胶电泳成像法对dna的浓度和纯度作检测。
2.对比已鉴定出的四种镰刀菌(半裸镰刀菌,层生镰刀菌,腐皮镰刀菌,尖孢镰刀菌)its区的序列(序列分别见seqidno.3-6),利用seqidno.1-2所示的引物进行pcr扩增,pcr产物长度120-200bp,引物信息如下:
f:catgcctgttcgagcgtcatt,(seqidno.1)
r:cgaggtcaacattcagaagt;(seqidno.2)
3.配制反应体系。在每一个pcr反应孔中按照下表加入2×mixtaqmanpcrmaster15ul、引物f0.9μl、引物r0.9μl(引物浓度均为10μm)、roxreferencedyeⅱ(100×)0.3ul、evagreen,20×inwater1.5μl、待测dna模板2μl,用灭菌纯水补足体系至30μl。
4.在abiquantstudio6flex上进行反应,pcr反应程序如表1:
表1
5.利用abi软件进行结果分析,通过标准曲线基于曲线偏移和曲线形状变化展现不同镰刀菌种的its差异。
6.hrm灵敏度验证
以10倍系列稀释的四种镰刀菌(层生镰刀菌,尖孢镰刀菌,半裸镰刀菌,腐皮镰刀菌)标准dna为模板进行hrm扩增并分析其标准峰形。结果表明该方法对四种镰刀菌dna模板的检测下限10-100拷贝,具有很高的灵敏度,见图2。且其标准峰形区分明显,表明此方法适用范围较广。如图1所示。
7.hrm特异性验证
利用本发明对多种真菌菌株(fusariumproliferatum、fusariumoxysporumschl.、fusariumsemitectum、fusariumsolani、aspergillusversicolor、talaromyceschloroloma、kurtzmaniellacleridarum、cladosporiumsp、microascusmurinus、trichosporonasahii、curvulariaspicifera、alternariaalternata)同时进行种别鉴定。同时采用高分辨熔解曲线分析技术和传统pcr扩增后测序法,对20个来自不同地区的样品和菌株进行鉴定。经过菌株培养、dna提取等步骤后,用高分辨熔解曲线分析技术参照本实施例前述方法进行鉴定,同时使用传统pcr扩增后测序法,使用j.b.stielow等(stielowjb,levesqueca,seifertka,etal.onefungus,whichgenes?developmentandassessmentofuniversalprimersforpotentialsecondaryfungaldnabarcodes.persoonia.2015;35:242–63.)及laszloirinyi等(irinyil,serenac,garcia-hermosod,etal.2015b.internationalsocietyofhumanandanimalmycology(isham)-itsreferencednabarcodingdatabase–thequalitycontrolledstandardtoolforroutineidentificationofhumanandanimalpathogenicfungi.medicalmycology53:313–337.)所采用的多对引物对目标真菌菌株its区域进行准确扩增,得到pcr产物后进行测序,将测序结果在ncbi中进行比对,得到其菌种。将两种方法的检测结果进行鉴定比对。
结果显示,利用高分辨熔解曲线分析技术和传统pcr后扩增法鉴别出的四种镰刀菌均能一一对应;而非镰刀菌种均未成功扩增。表明本发明方法能有效地用于四种镰刀菌(层生镰刀菌,尖孢镰刀菌,半裸镰刀菌,腐皮镰刀菌)的区分鉴定。如图1所示。tm值在87~89℃范围内出现熔解峰为层生镰刀菌(fusariumproliferatum)检出;tm值在84~85℃范围内出现熔解峰为尖孢镰刀菌(fusariumoxysporumschl.)检出;tm值在85~86℃范围内出现熔解峰为半裸镰刀菌(fusariumsemitectum)检出;tm值在89~90℃范围内出现熔解峰为腐皮镰刀菌(fusariumsolani)检出。
实施例2本发明方法的应用
1.选取来自宁夏地区的发霉玉米粒标本30个,分别接种在sda培养基上,28℃培养并观察菌落生长情况。长出菌落后采用稀释涂布平板法挑选单克隆菌落,单独培养。
2.采用粗提法提取待测真菌样本的基因组dna,后续鉴定方法参照实施例1的鉴别方法进行。利用abi软件进行结果分析,通过标准曲线基于曲线偏移和曲线形状变化展现不同镰刀菌种的its差异。鉴定结果如下表2。
表2
对于采用实施例1方法鉴别出相应镰刀菌的样本,再采用与实施例1相同的传统pcr方法分别检测该样本,确定两种方法检测结果一致。可见本发明方法具有良好的准确性。
对于表2中检测结果显示未扩增的样本,采用与实施例1相同的传统pcr方法分别检测该样本,结果均不是层生镰刀菌(fusariumproliferatum),尖孢镰刀菌(fusariumoxysporumschl.),半裸镰刀菌(fusariumsemitectum),腐皮镰刀菌(fusariumsolani)四种镰刀菌中的任一种,可见本发明鉴别四种镰刀菌的方法准确性高,无假阴性、无假阳性。
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
序列表
<110>西安博睿康宁生物科技有限公司
<120>一种鉴别四种镰刀菌的方法
<130>khp191110387.1
<160>6
<170>siposequencelisting1.0
<210>1
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>1
catgcctgttcgagcgtcatt21
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
cgaggtcaacattcagaagt20
<210>3
<211>522
<212>dna
<213>人工序列(artificialsequence)
<400>3
ggggttccgagttacactcccaacccctgtgacatacctatacgttgcctcggcggatca60
gcccgcgccccgtaaaaagggacggcccgcccgaggacccctaaactctgtttttagtgg120
aacttctgagtaaaacaaacaaataaatcaaaactttcaacaacggatctcttggttctg180
gcatcgatgaagaacgcagcaaaatgcgataagtaatgtgaattgcagaattcagtgaat240
catcgaatctttgaacgcacattgcgcccgccagtattctggcgggcatgcctgttcgag300
cgtcatttcaaccctcaagctcagcttggtgttgggactcgcggtaacccgcgttcccca360
aatcgattggcggtcacgtcgagcttccatagcgtagtaatcatacacctcgttactggt420
aatcgtcgcggccacgccgtaaaaccccaacttctgaatgttgacctcggatcaggtagg480
aatacccgctgaacttaagcatatcaataagcggaggaacaa522
<210>4
<211>535
<212>dna
<213>人工序列(artificialsequence)
<400>4
aatccgagtttacactcccaacccctgtgaacataccaattgttgcctcggcggatcagc60
ccgctcccggtaaaacgggacggcccgccagaggacccctaaactctgtttctatatgta120
acttctgagtaaaaccataaataaatcaaaactttcaacaacggatctcttggttctggc180
atcgatgaagaacgcagcaaaatgcgataagtaatgtgaattgcagaattcagtgaatca240
tcgaatctttgaacgcacattgcgcccgccagtattctggcgggcatgcctgttcgagcg300
tcatttcaaccctcaagcccccgggtttggtgttggggatcggcgagcccttgcggcaag360
ccggccccgaaatctagtggcggtctcgctgcagcttccattgcgtagtagtaaaaccct420
cgcaactggtacgcggcgcggccaagccgttaaacccccaacttctgaatgttgacctcg480
gatcaggtaggaatacccgctgaacttaagcatatcaataaggccgggaggaaaa535
<210>5
<211>543
<212>dna
<213>人工序列(artificialsequence)
<400>5
ggaaactcgagtaacactcatcaccctgtgacatacctataacgttgcctcggcgggaac60
agacggccccgtaacacgggccgcccccgccagaggaccccctaactctgtttctataat120
gtttcttctgagtaaacaagcaaataaattaaaactttcaacaacggatctcttggctct180
ggcatcgatgaagaacgcagcgaaatgcgataagtaatgtgaattgcagaattcagtgaa240
tcatcgaatctttgaacgcacattgcgcccgccagtattctggcgggcatgcctgttcga300
gcgtcattacaaccctcaggcccccgggcctggcgttggggatcggcggaagccccctgc360
gggcacaacgccgtcccccaaatacagtggcggtcccgccgcagcttccattgcgtagta420
gctaacacctcgcaactggagagcggcgcggccacgccgtaaaacacccaacttctgaat480
gttgacctcgaatcaggtaggaatacccgctgaacttaagcatatcaataaggccggagg540
aaa543
<210>6
<211>521
<212>dna
<213>人工序列(artificialsequence)
<400>6
ggaatttcgagttaactcccaacccctgtgaacataccacttgttgcctcggcggatcag60
cccgctcccggtaaaacgggacggcccgccagaggacccctaaactctgtttctatatgt120
aacttctgagtaaaaccataaataaatcaaaactttcaacaacggatctcttggttctgg180
catcgatgaagaacgcagcaaaatgcgataagtaatgtgaattgcagaattcagtgaatc240
atcgaatctttgaacgcacattgcgcccgccagtattctggcgggcatgcctgttcgagc300
gtcatttcaaccctcaagcacagcttggtgttgggactcgcgttaattcgcgttcctcaa360
attgattggcggtcacgtcgagcttccatagcgtagtagtaaaaccctcgttactggtaa420
tcgtcgcggccacgccgttaaaccccaacttctgaatgttgacctcggatcaggtaggaa480
tacccgctgaacttaagcatatcaaaaggtgggaggaaaaa521
- 还没有人留言评论。精彩留言会获得点赞!