实现通用酶催化功能多样性的突变体菌株及其构建方法与流程
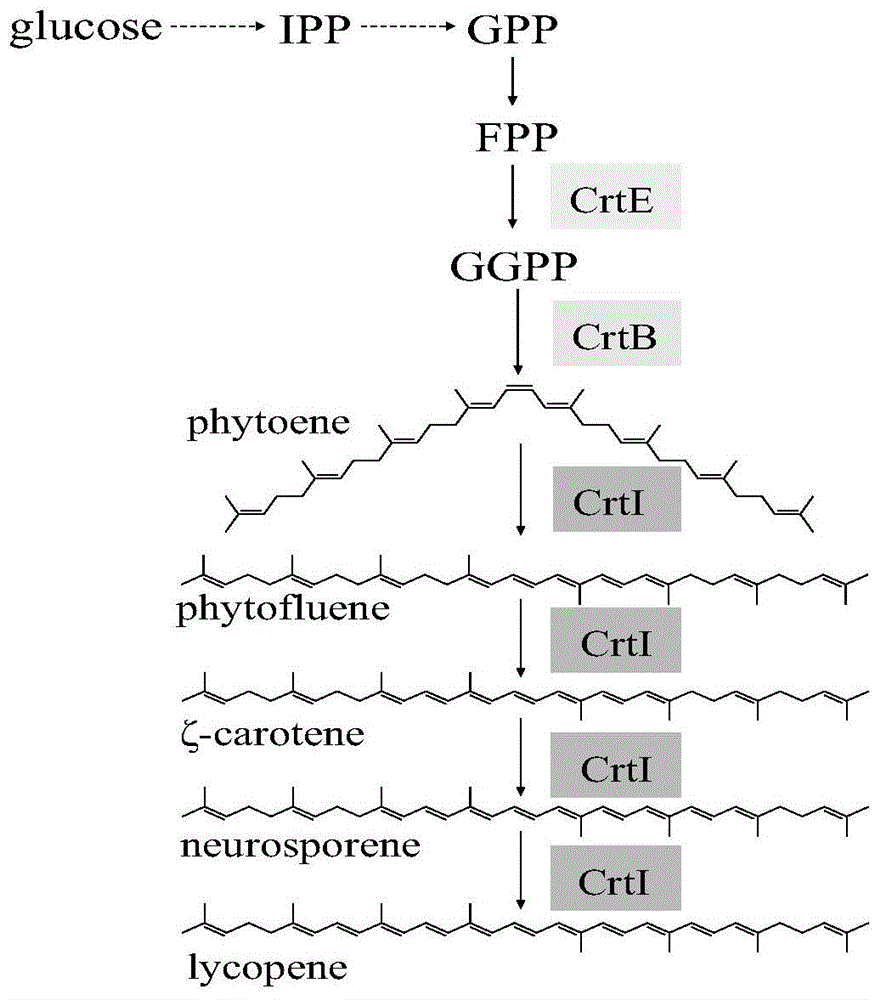
本发明属于微生物领域,具体涉及实现通用酶催化功能多样性的突变体菌株及其构建方法,尤其是涉及一种由btcrti催化二步脱氢反应产生zeta-胡萝卜素的重组酿酒酵母突变体菌株和一种催化四步脱氢产生的番茄红素与三步脱氢反应的链孢红素的比例为5:1的重组酿酒酵母突变体菌株及构建方法。
背景技术:
在生物代谢过程中存在一类可以催化多步连续反应的通用酶。据统计在生物代谢酶中约有20%可以归为通用酶,在生物的生命代谢过程中发挥着重要的作用,尤其是它们催化多步反应的功能特性可以有效地精简生物体内的酶系统。在低等生物中通用酶表现出更高的比重,如大肠杆菌的代谢酶中,通用酶的比例达到了37%左右。在生物体系中包含着多种类型的通用酶,其中植物和藻类来源的八氢番茄红素脱氢酶pds和ζ-胡萝卜素脱氢酶zds都可以催化2步连续脱氢反应,并且通过协同作用可催化八氢番茄红素发生4步连续脱氢反应。金黄色葡萄球菌来源的脱氢酶crtn在金黄色色素的生物合成通路中催化c30的多烯类底物最多可发生4步连续脱氢反应。在虾青素的合成路径中,羟化酶crtz和酮化酶crtw以β-胡萝卜素为底物发生2步连续羟化反应和2步连续羰基化反应。此外在水解酶和合成酶中也存在许多这类通用酶可以催化多步连续反应。
八氢番茄红素脱氢酶(crti)是催化多步连续反应通用酶中的典型代表,也是番茄红素下游类胡萝卜素合成途径中的首要限速酶。crti可以催化八氢番茄红素发生多步连续脱氢反应依次生成链孢红素、番茄红素等不同碳碳双键饱和度的多种脱氢产物。在研究脱氢酶crti的催化反应时,发现不同生物源的crti在催化多步脱氢反应时,会展现出不同的主导反应步数,发生3步、4步甚至5步的脱氢反应。如rrhodobacter来源的crti主要催化3步连续脱氢反应生成链孢红素,erwinia来源的crti主要催化4步连续脱氢反应生成番茄红素,neurospora来源的crti催化5步连续脱氢反应生成3,4-脱氢番茄红素。而对于特定生物来源的脱氢酶crti,其多步反应步数具有一定随机性,终产物组成中夹杂着不同脱氢步数的中间产物。
近年来,对于crti结构和功能多样性的研究并不多。2000年dannert和umeno等人通过点突变得到的欧文氏菌(p.ananatis)crti能够催化八氢番茄红素的四步脱氢反应转变为六步脱氢反应。2001年wang和liao研究团队利用酶定向进化策略,对三步脱氢球形红杆菌(rrhodobactersphaeroides)来源的crti进行突变,通过高通量筛选使得四步脱氢产物番茄红素的比例大幅提高。2010年sandmann和xiao的团队也获得了胶状红环菌(rubrivivaxgelatinosus)来源的crti突变体,使产物比例发生改变,由生产四步脱氢产物番茄红素转变为生产三步脱氢产物链孢红素。近年来,schaub和yu报道了欧文氏菌(pantoeaananatis)来源crti脱辅酶的晶体结构(pdb:4dgk),初步阐释了crti的基本结构信息,并推测了crti的反应机制。但该研究并没有结合酶的活性实验,缺乏结构与功能的相关性研究和验证,而事实上这对crti深入研究和探索是十分必要的。
crti属于黄素蛋白家族,黄素腺嘌呤二核苷酸(fad)作为辅基与脱辅酶组装形成全酶后才能发挥催化活性,目前由于八氢番茄红素脱氢酶全酶的体外组装效率低,酶的活性表现不理想,这为全酶的体外实验的可能性和准确性造成困难,增加了研究的难度和不确定性。酿酒酵母作为公认的安全模式微生物,遗传背景清楚,基因操作简单,在类胡萝卜素产业化方面具有很重要的地位,而目前在真核生物中研究crti功能多样性的报道很少。申请人在番茄红素生物合成的研究中,考察了agrobacteriumaurantiacum,pantoeaagglomerans,blakesleatrispora等多种生物来源的脱氢酶crti在酿酒酵母中的催化效果。其中blakesleatrispora来源的crti表现出了更好的底物转化效率,但是仍然夹杂着少量的三步脱氢产物链孢红素。因此,以blakesleatrispora来源的crti为研究基础,若能够探索调控crti催化步数及影响其功能多样性的关键位点信息,解明该位点的具体的调控作用,将不仅可以丰富crti的结构和功能信息,也为深入研究crti这类催化多步反应的通用酶提供重要参考,同时对于crti的改造、应用以及重要天然产物生物合成都具有重要意义。
技术实现要素:
本发明的目的是在酿酒酵母菌株中构建crti突变体菌株文库,通过筛选使突变体菌株的催化步数实现可控,即脱氢产物比例发生变化,并提供相应的酿酒酵母突变体菌株。
为实现本发明的目的,本发明采用如下技术方案:
本发明首先提供了一种实现通用酶催化功能多样性的突变体菌株的构建方法,其特征在于,构建通用酶的突变体文库,通过摇瓶发酵筛选催化功能多样性的突变体菌株。
进一步的,在一些实施方案中,所述构建方法对筛选获得的催化功能多样性的突变体菌株的通用酶基因进行测序分析,鉴定出影响通用酶功能多样化的关键氨基酸位点信息。
本发明所述构建方法中所述通用酶可以为生物体系中各种类型的通用酶包括但不限于八氢番茄红素脱氢酶crti,植物和藻类来源的八氢番茄红素脱氢酶pds、ζ-胡萝卜素脱氢酶zds,金黄色葡萄球菌来源的脱氢酶crtn,虾青素合成路径中的羟化酶crtz和酮化酶crtw。
本发明所述构建方法中所述菌株可以为酵母、藻类、霉菌、细菌。
在一些实施方案中,所述的构建方法中所述通用酶为blakesleatrispora来源的crti,所述菌株为酿酒酵母。
在一些具体实施例中,所述构建方法具体为以产生八氢番茄红素的重组酿酒酵母为底盘株,利用易错pcr构建编码btcrti基因的突变文库,通过在酵母体内同源重组将启动子gal7、终止子cyc1t和btcrti基因的突变体文库组装到质粒prs416上,直接在底盘中构建btcrti突变体菌株,通过摇瓶发酵筛选催化功能多样性的酿酒酵母菌株;
进一步的,在一些具体实施例中,所述构建方法还包括对btcrti突变体菌株的btcrti基因进行测序分析,鉴定出影响btcrti功能多样化的关键氨基酸位点信息的步骤。
通过本发明所述构建方法可以筛选获得通用酶催化功能多样性的突变体菌株。如在酿酒酵母菌株中构建blakesleatrispora来源的crti突变体菌株文库,通过筛选使突变体菌株的催化步数实现可控,获得相应的酿酒酵母突变体菌株。
本发明提供了一种由btcrti催化二步脱氢反应产生zeta-胡萝卜素的重组酿酒酵母突变体菌株和一种催化四步脱氢产生的番茄红素与三步脱氢反应的链孢红素的比例为5:1的重组酿酒酵母突变体菌株。
其中,所述由btcrti催化二步脱氢反应产生zeta-胡萝卜素的重组酿酒酵母突变体菌株其blakesleatrispora来源的btcrti基因的第453位组氨酸突变为精氨酸,命名为sybe_sc04020004(h453r)。
所述催化四步脱氢产生的番茄红素与三步脱氢反应的链孢红素的比例为5:1的重组酿酒酵母突变体菌株其blakesleatrispora来源的btcrti基因的第136位组氨酸突变为精氨酸,命名为sybe_sc04020003(h136r)。
野生型btcrti是四步脱氢的,番茄红素与链孢红素比例高达19:1sybe_sc04020004(h453r)和sybe_sc04020003(h136r)两菌株的脱氢比例和野生型相比产生了变化,为研究脱氢酶crti甚至是多步反应通用酶的多步脱氢功能提供了基础。研究crti脱氢功能为脱氢酶crti的改造和应用提供了指导,进而有利于类胡萝卜素的生产。根据这两株突变体菌株可以发掘btcrti的突变位点,从而有利于对多步脱氢功能的机制进行深入探索。此外还可以获得不同脱氢饱和度的类胡萝卜素,如由btcrti催化二步脱氢反应产生zeta-胡萝卜素的重组酿酒酵母突变体菌株sybe_sc04020004(h453r)可以用于生产zeta-胡萝卜素。
本发明还提供了所述sybe_sc04020004(h453r)和sybe_sc04020003(h136r)的构建方法。
其中,所述sybe_sc04020004(h453r)重组酿酒酵母突变体菌株的构建方法,包括:
步骤a、构建btcrti基因的第453位组氨酸突变为精氨酸的重组质粒prs416-btcrti453;
步骤b、将重组质粒转化酿酒酵母sybe_sc04020001中,筛选转化子,将筛得的转化子用ypd培养基培养获得重组酿酒酵母突变体菌株。
进一步的,步骤a所述重组质粒prs416-btcrti453的构建方法具体为以含有btcrti基因的prs416质粒为模版,扩增片段gal7-h453r和h453r-cyc1t,将两个片段通过oe-pcr方法拼接起来得到两端包含xbai和noti酶切位点的线性片段,将该线性片段与prs416质粒经xbai和noti内切酶处理后,使用t4连接酶连接。
所述sybe_sc04020003(h136r)重组酿酒酵母突变体菌株的构建方法,包括:
步骤a、构建btcrti基因的第136位组氨酸突变为精氨酸的重组质粒prs416-btcrti136;
步骤b、将重组质粒转化酿酒酵母sybe_sc04020001中,筛选转化子,将筛得的转化子用ypd培养基培养获得重组酿酒酵母突变体菌株。
进一步的,步骤a所述重组质粒prs416-btcrti136的构建方法具体为以含有btcrti基因的prs416质粒为模版,扩增片段gal7-h136r和h136r-cyc1t,将两个片段通过oe-pcr方法拼接起来,得到两端包含xbai和noti酶切位点的线性片段,将该线性片段与prs416质粒经xbai和noti内切酶处理后,使用t4连接酶连接。
上述构建方法中,所述筛选转化子的筛选的过程分为两轮。第一轮通过颜色筛选,不同脱氢步骤的脱氢产物的颜色深浅是不同的,从无色的底物八氢番茄红素到暗红色的四步脱氢产物番茄红素,颜色越来越深,因此第一轮时在含有d半乳糖的平板上挑选颜色变浅的菌株。第二轮筛选是通过发酵,hplc检测确定产物分配和比例,从而筛选出目标菌株。
由上述技术方案可知,本发明提供了一种实现通用酶催化功能多样性的突变体菌株的构建方法,构建通用酶的突变体文库,通过摇瓶发酵筛选催化功能多样性的突变体菌株。进一步在酿酒酵母菌株中构建blakesleatrispora来源的crti突变体菌株文库,通过筛选使突变体菌株的催化步数实现可控,获得相应的酿酒酵母突变体菌株。本发明筛选获得了一种由btcrti催化二步脱氢反应产生zeta-胡萝卜素的重组酿酒酵母突变体菌株和一种催化四步脱氢产生的番茄红素与三步脱氢反应的链孢红素的比例为5:1的重组酿酒酵母突变体菌株。本发明利用重组酿酒酵母菌株,通过随机突变的性质得到了不同催化功能的btcrti的突变体菌株,明确了关键氨基酸位点,实现了脱氢步骤的可控性,丰富了crti的功能信息,并为重要天然产物的生物合成打下基础。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍。
图1示crti催化的多步脱氢反应路径图;
图2示实施例1酿酒酵母底盘构建过程图;
图3示实施例2野生型crti菌株的构建过程;
图4示实施例3crti突变体菌株文库的构建过程图;
图5示实施例4野生型与不同突变体菌株脱氢产物的比例比较图,其中l为番茄红素(lycopene),n为链孢红素(neurosporene)。
具体实施方式
本发明公开了实现通用酶催化功能多样性的突变体菌株及其构建方法。本领域技术人员可以借鉴本文内容,适当改进工艺参数实现。特别需要指出的是,所有类似的替换和改动对本领域技术人员来说是显而易见的,它们都被视为包括在本发明。本发明的方法及产品已经通过较佳实施例进行了描述,相关人员明显能在不脱离本发明内容、精神和范围内对本文所述的方法进行改动或适当变更与组合,来实现和应用本发明技术。
为了进一步理解本发明,下面将结合本发明实施例,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
如无特殊说明,本发明实施例中所涉及的试剂均为市售产品,均可以通过商业渠道购买获得。
实施例1、高产八氢番茄红素酿酒酵母的获得
1、模块化整合质粒的构建
高产ggpp的酿酒酵母菌株,菌株编号为sybe_sc14c10(陈艳;高产番茄红素酿酒酵母的设计构建与发酵过程优化[d];天津大学;2017年)。
为了在酿酒酵母中实现八氢番茄红素的生产,通过pcr扩增获得酵母启动子gal1-10、基因crtb、终止子pgk1t、上游整合同源臂、下游整合同源臂等元件,所用的基因crtb按照酿酒酵母密码子进行优化并适当规避常用限制性酶切位点后由苏州金唯智公司合成,应用重叠延伸pcr(oe-pcr)将上述元件进行模块化组装,得到l-arm-pgal1-crtb-tpgk1-r-arm命名为module1(seqidno.1);将组装后的dna片段与平末端载体pjet1.2/blunt(购自thermo公司)进行连接,转化入大肠杆菌感受态dh5α中,菌落pcr筛选,提质粒进行单、双酶切验证以及测序验证,以确保目的片段连接正确且碱基序列未发生突变,从而构建正确的重组质粒(图2)。
2、模块化整合构建生产八氢番茄红素的重组酿酒酵母菌株
首先,将module1整合质粒用pmei酶切位点切割,获得module1整合片段l-arm-pgal1-crtb-tpgk1-r-arm,采用醋酸锂法将片段单独转化高产ggpp的酵母菌株,通过同源臂左、右同源序列与酵母基因组上序列发生重组而整合到基因组上。转化后采用sc-trp固体板(合成酵母氮源ynb6.7g/l,葡萄糖20g/l,缺色氨酸、亮氨酸、组氨酸和尿嘧啶的混合氨基酸粉末2g/l,2%的琼脂粉)进行筛选,得到的转化子进行划线分纯培养后提取酵母基因组进行pcr验证,测序结果完全正确则说明筛选到了正确的阳性克隆,对验证正确的重组菌株保存甘油菌并命名为sybe_sc04020001。
实施例2、在酿酒酵母中野生型btcrti的构建
1、crti催化八氢番茄红素产生番茄红素的酿酒酵母菌株构建(图3)
将gal7启动子、cyc1t终止子以及由苏州金唯智公司按照酿酒酵母密码子进行优化并适当规避常用限制性酶切位点后合成的基因btcrti通过oe-pcr方法拼接起来,得到两端包含xbai和noti酶切位点的线性片段gal7启动子-btcrti-cyc1t终止子,即pgal7-btcrti-tcyc1(seqidno.2);之后,将该线性片段与prs416质粒经xbai和noti内切酶处理后,使用t4连接酶连接,转化入大肠杆菌感受态dh5α中,菌落pcr筛选,提质粒进行单、双酶切验证以及测序验证,以确保目的片段连接正确且碱基序列未发生突变,从而构建正确的重组质粒。最后采用酵母醋酸锂转化法将构建的含有btcrti的prs416质粒(质粒prs416-btcrti),导入酿酒酵母sybe_sc04020001中,保存甘油菌并命名为sybe_sc04020002。
2、发酵测野生型菌株各脱氢产物的产量及比例
试验材料:菌株sybe_sc04020002
种子培养基:40g/l葡萄糖、20g/l蛋白胨、10g/l酵母浸粉;
发酵培养基:40g/l葡萄糖、20g/l蛋白胨、10g/l酵母浸粉,10g/ld-半乳糖。
挑取单菌落接种于5mlypd种子培养基里,30℃、250rpm培养大约18h;以初始od600=0.2转接至25ml种子培养基里,30℃、250rpm培养至od600=5~8;将二级种子接种于50mlypdg发酵培养基中,使得初始od600=0.1,30℃、250rpm培养48h。
取两份500μl发酵液,4000rpm离心1min收集菌体,并水洗一次;其中一份菌体80℃烘干至恒重,称重计算细胞干重;另一份菌体用以产物提取,具体步骤为:用1ml3mhcl重悬细胞,置于沸水浴中煮沸2min,然后立即冰浴3min,可以看到较为细腻的颗粒状。将细胞12000rpm离心1min弃上清,使用无菌水洗2次后加入少量石英砂以及1ml含1%(w/v)bht的丙酮,并涡旋5min,最后12000rpm离心5min,观察到菌体变为白色,然后收集丙酮相,用0.22μm有机滤膜过滤后上液相检测。值得注意的是,脂溶性类胡萝卜素性质不稳定,见光易分解,在提取过程中应尽量避光操作。
hplc检测:采用高效液相色谱hplc对类胡萝卜素进行测得。以bdshypersilc18柱为色谱柱,型号为:150mm×4.6mm,5μm;采用紫外检测器waterse2489uv/vis;泵为waterse2695;配制的流动相为乙腈:甲醇:二氯甲烷=21:21:8(v/v/v),流速1.0ml/min,柱温30℃。番茄红素、链孢红素、zeta-胡萝卜素、六氢番茄红素和八氢番茄红素在紫外检测器下的检测波长分别为471nm、440nm、401nm、349nm和287nm。
试验结果:发酵培养基中d-半乳糖为诱导剂,负责开启gal7启动子的转录。初始培养基中因有葡萄糖存在,gal启动子的转录受到葡萄糖抑制;随着发酵的进行,葡萄糖迅速被消耗,当葡萄糖耗尽后,葡萄糖抑制效应解除,d-半乳糖开启gal7启动子的转录,从而逐渐积累番茄红素。发酵72h,测得的各脱氢产物的产量为:番茄红素达到264.0mg/l,链孢红素达到13.89mg/l,未检测到二步脱氢产物zeta-胡萝卜素和一步脱氢产物六氢番茄红素。由此可知,在酿酒酵母中由野生型btcrti催化的脱氢反应使得四步脱氢产物与三步脱氢产物比例为19:1。
实施例3、在酿酒酵母中野生型btcrti的构建
1、易错pcr构建btcrti突变文库
试验材料:
菌株sybe_sc04020001与质粒prs416-btcrti
10×易错pcrbuffer:70mm六水合氯化镁、500mm氯化钾、100mm三羟甲基氨基甲烷、0.1w/v明胶
10×易错pcrdntps:10mmdctps与dttps、2mmdgtps与datps
10mm四水合氯化锰
将gal7启动子、cyc1t终止子通过pcr扩增,以含有btcrti的质粒为模版,通过易错pcr的随记突变扩增btcrti基因,其中易错pcr体系(100μl)配置方法为:加入10×易错pcrbuffer10μl,10×易错pcrdntps10μl,上下引物各4μl,模版2μl,氯化锰溶液5μl,fasttaq酶1μl,并补充水64μl。
需要注意的是,启动子、基因和终止子之间有40bp的同源序列,方便后续将三者组装。其中,prs416载体与启动子gal7之间的同源序列(68bp)(seqidno.3):tcgaggtcgacggtatcgataagcttgatatcgaattcctgcagcccgggggatccactagttctaga;启动子gal7与btcrti之间的同源序列(40bp)(seqidno.4):attccctcaaaaatgtctgatcagaagaagcacattgtcg;btcrti序列与终止子cyc1之间的同源序列(40bp)(seqidno.5):ctctaacgatattaggatataaggccgcatcatgtaatta;终止子cyc1与prs416载体之间的同源序列(73bp)(seqidno.6):gcggccgccaccgcggtggagctccagcttttgttccctttagtgagggttaattgcgcgcttggcgtaatca。
易错pcr的循环设置为:预变性为94℃3min,变性为94℃5s,退火为55℃15s,延伸为72℃50s,共30个循环,无彻底延伸阶段。胶回收后,得到启动子、btcrti基因突变文库和终止子三个线性片端。同时,将质粒prs416经xbai和noti内切酶酶切后获得线性载体;之后,采用酵母醋酸锂转化法将三个线性片段和酶切处理后的prs416线性载体导入酿酒酵母中,通过相互之间含有的同源序列组装和实现环状质粒形成,该质粒内连有不同突变点的btcrti基因,从而成功的构建了btcrti突变文库,转化后采用sc-ura+半乳糖固体板(合成酵母氮源ynb6.7g/l,葡萄糖20g/l,缺色氨酸、亮氨酸、组氨酸和尿嘧啶的混合氨基酸粉末2g/l,2%的琼脂粉)进行筛选,由于各脱氢产物的颜色不同,如zeta-胡萝卜素为黄色,链孢红素为粉色,番茄红素为暗红色,所以各个突变体的颜色也不同,由于实验目的是找到倾向于两步脱氢和三步脱氢的突变体,因此挑出颜色较浅的菌株,从而进行后续的验证分析。
2、发酵测突变体菌株各脱氢产物的产量及比例
试验材料:在平板上挑出的各个突变体菌株
试验方法:与实施例2完全相同。
试验结果:发酵培养基中d-半乳糖为诱导剂,负责开启gal7启动子的转录。初始培养基中因有葡萄糖存在,gal启动子的转录受到葡萄糖抑制;随着发酵的进行,葡萄糖迅速被消耗,当葡萄糖耗尽后,葡萄糖抑制效应解除,d-半乳糖开启gal启动子的转录,从而逐渐积累各脱氢产物。发酵72h,有一株突变体只检测到了二步脱氢产物zeta-胡萝卜素和一步脱氢产物六氢番茄红素,另一株菌检测到了少量的zeta-胡萝卜素和六氢番茄红素,四步脱氢产物番茄红素与三步脱氢产物链孢红素的比例为:5:1。
实施例4、突变体菌株的btcrti基因测序分析及验证
1、突变体菌株的btcrti基因测序
将两个突变菌株单菌落挑到sc-ura培养基中培养,30℃培养20h后提取酵母质粒,之后将酵母质粒转化入大肠杆菌感受态dh5α中,菌落pcr筛选,提质粒进行单、双酶切验证以及测序验证,以确保目的片段连接正确,检测碱基序列发生突变的位置,发现产生二步脱氢产物的菌株在453位氨基酸由组氨酸h突变成了精氨酸r,倾向于三步脱氢产物的菌株在136位由组氨酸h突变成了精氨酸r。
2、突变位点的表型验证
突变点的引入:分别在136位和453位设计引物,引入突变点使组氨酸h突变成了精氨酸r。以含有btcrti基因的prs416质粒为模版,扩增片段gal7-h136r(seqidno.7)、h136r-cyc1t(seqidno.8)和gal7-h453r(seqidno.9)、h453r-cyc1t(seqidno.10),将136位和453位的两个片段通过oe-pcr方法拼接起来,得到两端包含xbai和noti酶切位点。之后,将该线性片段与prs416质粒经xbai和noti内切酶处理后,使用t4连接酶连接,转化入大肠杆菌感受态dh5α中,菌落pcr筛选,提质粒进行单、双酶切验证以及测序验证,以确保目的片段连接正确且碱基序列未发生突变,从而构建正确的重组质粒。最后采用酵母醋酸锂转化法将构建的含有突变的btcrti的prs416质粒导入酿酒酵母sybe_sc04020001中,保存甘油菌并命名为sybe_sc04020003(h136r)和sybe_sc04020004(h453r)。
突变体的发酵验证:将菌株按照与实施例2完全相同的方法发酵。
结果:发酵培养基中d-半乳糖为诱导剂,负责开启gal7启动子的转录。初始培养基中因有葡萄糖存在,gal启动子的转录受到葡萄糖抑制;随着发酵的进行,葡萄糖迅速被消耗,当葡萄糖耗尽后,葡萄糖抑制效应解除,d-半乳糖开启gal启动子的转录,从而逐渐积累各脱氢产物。发酵72h,两株突变体菌株脱氢产物的比例与之前发酵的结果相同,h453r突变体只检测到了二步脱氢产物zeta-胡萝卜素和一步脱氢产物六氢番茄红素,h136r突变体检测到了少量的zeta-胡萝卜素和六氢番茄红素,四步脱氢产物番茄红素与三步脱氢产物链孢红素的比例为:5:1(图5)。
序列表
<110>天津大学
<120>实现通用酶催化功能多样性的突变体菌株及其构建方法
<130>mp1831161
<160>10
<170>siposequencelisting1.0
<210>1
<211>2466
<212>dna
<213>人工序列(artificialsequence)
<400>1
ggaagaggagtagggaatattactggctgaaaataagtcttgaatgaacgtatacgcgta60
tatttctaccaatctctcaacactgagtaatggtagttataagaaagagaccgagttagg120
gacagttagaggcggtggagatattccttatggcatgtctggcgatgataaaacttttca180
aacggcagccccgatctaaaagagctgacagggaaatggtcagaaaaagaaacgtgcacc240
cgcccgtctggacgcgccgctcacccgcacggcagagaccaatcagtaaaaatcaacggt300
taacgacattactatatatataatataggaagcatttaatagaacagcatcgtagaagta360
cggattagaagccgccgagcgggcgacagccctccgacggaagactctcctccgtgcgtc420
ctcgtcttcaccggtcgcgttcctgaaacgcagatgtgcctcgcgccgcactgctccgaa480
caataaagattctacaatactagcttttatggttatgaagaggaaaaattggcagtaacc540
tggccccacaaaccttcaaattaacgaatcaaattaacaaccataggatgataatgcgat600
tagttttttagccttatttctggggtaattaatcagcgaagcgatgatttttgatctatt660
aacagatatataaatggaaaagctgcataaccactttaactaatactttcaacattttca720
gtttgtattacttcttattcaaatgtcataaaagtatcaacaaaaaattgttaatatacc780
tctatactttaacgtcaaggagaaaaaactataatgtcacaaccaccattattggaccac840
gctacacaaactatggcaaacggttctaaatctttcgctactgctgctaaattattcgac900
ccagcaacaagaagatctgtattgatgttgtacacctggtgtagacattgcgatgacgtt960
atagatgaccaaactcacggttttgcttcagaagctgcagccgaagaagaagctacacaa1020
agattggcaagattaagaactttgacattagctgcattcgaaggtgccgaaatgcaagat1080
ccagcttttgccgctttccaagaagttgcattaacccatggtattactcctagaatggct1140
ttggatcacttagacggttttgcaatggatgtcgcccaaacaagatacgtaaccttcgaa1200
gacactttaagatattgttaccatgtcgccggtgttgtcggtttgatgatggctagagta1260
atgggtgttagagatgaaagagttttagatagagcatgtgacttgggtttagccttccaa1320
ttgacaaacatagctagagatataatagatgacgcagccatagacagatgctatttgcca1380
gctgaatggttacaagatgcaggtttgactcctgaaaattacgctgcaagagaaaacaga1440
gccgctttagccagagttgctgaaagattgatagatgcagccgaaccatattacatctct1500
tcacaagctggtttgcatgatttgccacctagatgcgcatgggccattgctaccgcaaga1560
tctgtttacagagaaatcggtattaaagtcaaggctgcaggtggttccgcatgggataga1620
agacaacacacttctaaaggtgaaaagatcgctatgttgatggccgctcctggtcaagtt1680
attagagcaaagaccaccagagtcaccccaagaccagccggtttatggcaaagacctgtt1740
taaattgaattgaattgaaatcgatagatcaatttttttcttttctctttccccatcctt1800
tacgctaaaataatagtttattttattttttgaatattttttatttatatacgtatatat1860
agactattatttatcttttaatgattattaagatttttattaaaaaaaaattcgctcctc1920
ttttaatgcctttatgcagtttttttttctcgatatttctatgttcgggttcagcgtatt1980
ttaagtttaataactcgaaaattctgcgttcgttatatatgtgtactttgcagttatgac2040
gccagatggcagtagtggaagatattctttattgaaaaatagcttgtcaccttacgtaca2100
atcttgatccggagcttttctttttttgccgattaagaattcggtcgaaaaaagaaaagg2160
agagggccaagagggagggcattggtgactattgagcacgtgagtatacgtgattaagca2220
cacaaaggcagcttggagtatgtctgttattaatttcacaggtagttctggtccattggt2280
gaaagtttgcggcttgcagagcacagaggccgcagaatgtgctctagattccgatgctga2340
cttgctgggtattatatgtgtgcccaatagaaagagaacaattgacccggttattgcaag2400
gaaaatttcaagtcttgtaaaagcatataaaaatagttcaggcactccgaaatacttggt2460
tggcgt2466
<210>2
<211>2729
<212>dna
<213>人工序列(artificialsequence)
<400>2
tttgccagcttactatccttcttgaaaatatgcactctatatcttttagttcttaattgc60
aacacatagatttgctgtataacgaattttatgctattttttaaatttggagttcagtga120
taaaagtgtcacagcgaatttcctcacatgtagggaccgaattgtttacaagttctctgt180
accaccatggagacatcaaagattgaaaatctatggaaagatatggacggtagcaacaag240
aatatagcacgagccgcgaagttcatttcgttacttttgatatcgctcacaactattgcg300
aagcgcttcagtgaaaaaatcataaggaaaagttgtaaatattattggtagtattcgttt360
ggtaaagtagagggggtaatttttcccctttattttgttcatacattcttaaattgcttt420
gcctctccttttggaaagctatacttcggagcactgttgagcgaaggctcattagatata480
ttttctgtcattttccttaacccaaaaataagggaaagggtccaaaaagcgctcggacaa540
ctgttgaccgtgatccgaaggactggctatacagtgttcacaaaatagccaagctgaaaa600
taatgtgtagctatgttcagttagtttggctagcaaagatataaaagcaggtcggaaata660
tttatgggcattattatgcagagcatcaacatgataaaaaaaaacagttgaatattccct720
caaaaatgtctgatcagaagaagcacattgtcgtcataggtgctggaataggaggtactg780
caacagcagcaaggttagcaagggagggtttcagagtcactgtcgtcgagaagaacgact840
tctctggaggaaggtgctctttcattcaccacgacggtcacaggttcgaccagggacctt900
cattgtacttgatgcctaagttgtttgaggacgctttcgctgacttagacgagaggatag960
gagaccacttggacttattaagatgtgacaacaattacaaagtccatttcgacgacggtg1020
acgctgtccaattgtcatcagacttaacaaagatgaagggtgagttggacaggattgagg1080
gacctttaggattcggtaggttcttagatttcatgaaagagacacacgtccactacgagc1140
agggtacattcattgctataaagagaaacttcgaaactatatgggacttaataaggttac1200
agtacgtcccagagatttttaggttgcacttattcggtaagatatacgacagagcatcaa1260
aatacttccaaacaaaaaagatgaggatggcttttacttttcaaacaatgtacatgggta1320
tgtcaccttacgacgcacctgcagtctactcattgttgcaatatacagagttcgcagagg1380
gaatttggtacccaaggggtggtttcaacatggtcgtccaaaagttggagtctatagctt1440
ctaagaagtacggagctgagttcaggtaccaatctcctgtcgctaagattaacactgtcg1500
ataaagacaagagggtcactggtgtcactttggagtctggagaagtcattgaggcagacg1560
ctgtcgtctgcaacgctgacttggtctacgcttaccaccacttgttgccaccttgcaact1620
ggacaaagaagactttggcatctaagaaattaacatcttcatcaatttctttttactggt1680
caatgtctactaaggtccctcaattggacgtccacaacattttcttggctgaggcttaca1740
aggagtcattcgacgagatttttaacgatttcggtttgccttctgaagcatctttctacg1800
tcaacgttccttcaaggatagacgagtctgcagcacctccaaataaggactcaattatag1860
ttttagttccaattggtcacatgaagtctaagacaggtaactcagcagaggagaactacc1920
cagagttggtcaacagggctagaaagatggtcttggaggtcatagagaggaggttgggag1980
tcaacaacttcgctaacttgatagaacacgaggaggtcaacgacccatcagtctggcaat2040
ctaagttcaacttgtggaggggatcaatattaggtttatcacatgatgtctttcaggttt2100
tgtggttcagaccttcaacaaaggactctactaacagatatgacaatttatttttcgtcg2160
gtgcatcaactcaccctggtacaggagtcccaatagtcttggcaggatctaaattaactt2220
ctgaccaggtctgtaagtcattcggacaaaaccctttgcctaggaagttacaggactctc2280
agaagaaatatgcacctgagcaaacaaggaagactgagtcacactggatttattactgct2340
tagcatgctactttgtcactttcttgttcttctatttctttcctagggacgacactacta2400
ctccagcatcttttattaatcagttgttgccaaacgtcttccaaggacagaactctaacg2460
atattaggatataaggccgcatcatgtaattagttatgtcacgcttacattcacgccctc2520
cccccacatccgctctaaccgaaaaggaaggagttagacaacctgaagtctaggtcccta2580
tttatttttttatagttatgttagtattaagaacgttatttatatttcaaatttttcttt2640
tttttctgtacaaacgcgtgtacgcatgtaacattatactgaaaaccttgcttgagaagg2700
ttttgggacgctcgaaggctttaatttgc2729
<210>3
<211>68
<212>dna
<213>人工序列(artificialsequence)
<400>3
tcgaggtcgacggtatcgataagcttgatatcgaattcctgcagcccgggggatccacta60
gttctaga68
<210>4
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>4
attccctcaaaaatgtctgatcagaagaagcacattgtcg40
<210>5
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>5
ctctaacgatattaggatataaggccgcatcatgtaatta40
<210>6
<211>73
<212>dna
<213>人工序列(artificialsequence)
<400>6
gcggccgccaccgcggtggagctccagcttttgttccctttagtgagggttaattgcgcg60
cttggcgtaatca73
<210>7
<211>1162
<212>dna
<213>人工序列(artificialsequence)
<400>7
ctagtctagatttgccagcttactatccttcttgaaaatatgcactctatatcttttagt60
tcttaattgcaacacatagatttgctgtataacgaattttatgctattttttaaatttgg120
agttcagtgataaaagtgtcacagcgaatttcctcacatgtagggaccgaattgtttaca180
agttctctgtaccaccatggagacatcaaagattgaaaatctatggaaagatatggacgg240
tagcaacaagaatatagcacgagccgcgaagttcatttcgttacttttgatatcgctcac300
aactattgcgaagcgcttcagtgaaaaaatcataaggaaaagttgtaaatattattggta360
gtattcgtttggtaaagtagagggggtaatttttcccctttattttgttcatacattctt420
aaattgctttgcctctccttttggaaagctatacttcggagcactgttgagcgaaggctc480
attagatatattttctgtcattttccttaacccaaaaataagggaaagggtccaaaaagc540
gctcggacaactgttgaccgtgatccgaaggactggctatacagtgttcacaaaatagcc600
aagctgaaaataatgtgtagctatgttcagttagtttggctagcaaagatataaaagcag660
gtcggaaatatttatgggcattattatgcagagcatcaacatgataaaaaaaaacagttg720
aatattccctcaaaaatgtctgatcagaagaagcacattgtcgtcataggtgctggaata780
ggaggtactgcaacagcagcaaggttagcaagggagggtttcagagtcactgtcgtcgag840
aagaacgacttctctggaggaaggtgctctttcattcaccacgacggtcacaggttcgac900
cagggaccttcattgtacttgatgcctaagttgtttgaggacgctttcgctgacttagac960
gagaggataggagaccacttggacttattaagatgtgacaacaattacaaagtccatttc1020
gacgacggtgacgctgtccaattgtcatcagacttaacaaagatgaagggtgagttggac1080
aggattgagggacctttaggattcggtaggttcttagatttcatgaaagagacacacgtc1140
aggtacgagcagggtacattca1162
<210>8
<211>1614
<212>dna
<213>人工序列(artificialsequence)
<400>8
tacgagcagggtacattcattgctataaagagaaacttcgaaactatatgggacttaata60
aggttacagtacgtcccagagatttttaggttgcacttattcggtaagatatacgacaga120
gcatcaaaatacttccaaacaaaaaagatgaggatggcttttacttttcaaacaatgtac180
atgggtatgtcaccttacgacgcacctgcagtctactcattgttgcaatatacagagttc240
gcagagggaatttggtacccaaggggtggtttcaacatggtcgtccaaaagttggagtct300
atagcttctaagaagtacggagctgagttcaggtaccaatctcctgtcgctaagattaac360
actgtcgataaagacaagagggtcactggtgtcactttggagtctggagaagtcattgag420
gcagacgctgtcgtctgcaacgctgacttggtctacgcttaccaccacttgttgccacct480
tgcaactggacaaagaagactttggcatctaagaaattaacatcttcatcaatttctttt540
tactggtcaatgtctactaaggtccctcaattggacgtccacaacattttcttggctgag600
gcttacaaggagtcattcgacgagatttttaacgatttcggtttgccttctgaagcatct660
ttctacgtcaacgttccttcaaggatagacgagtctgcagcacctccaaataaggactca720
attatagttttagttccaattggtcacatgaagtctaagacaggtaactcagcagaggag780
aactacccagagttggtcaacagggctagaaagatggtcttggaggtcatagagaggagg840
ttgggagtcaacaacttcgctaacttgatagaacacgaggaggtcaacgacccatcagtc900
tggcaatctaagttcaacttgtggaggggatcaatattaggtttatcacatgatgtcttt960
caggttttgtggttcagaccttcaacaaaggactctactaacagatatgacaatttattt1020
ttcgtcggtgcatcaactcaccctggtacaggagtcccaatagtcttggcaggatctaaa1080
ttaacttctgaccaggtctgtaagtcattcggacaaaaccctttgcctaggaagttacag1140
gactctcagaagaaatatgcacctgagcaaacaaggaagactgagtcacactggatttat1200
tactgcttagcatgctactttgtcactttcttgttcttctatttctttcctagggacgac1260
actactactccagcatcttttattaatcagttgttgccaaacgtcttccaaggacagaac1320
tctaacgatattaggatataaggccgcatcatgtaattagttatgtcacgcttacattca1380
cgccctccccccacatccgctctaaccgaaaaggaaggagttagacaacctgaagtctag1440
gtccctatttatttttttatagttatgttagtattaagaacgttatttatatttcaaatt1500
tttcttttttttctgtacaaacgcgtgtacgcatgtaacattatactgaaaaccttgctt1560
gagaaggttttgggacgctcaaggaaaaaagcggccgcgcaaattaaagccttc1614
<210>9
<211>2112
<212>dna
<213>人工序列(artificialsequence)
<400>9
ctagtctagatttgccagcttactatccttcttgaaaatatgcactctatatcttttagt60
tcttaattgcaacacatagatttgctgtataacgaattttatgctattttttaaatttgg120
agttcagtgataaaagtgtcacagcgaatttcctcacatgtagggaccgaattgtttaca180
agttctctgtaccaccatggagacatcaaagattgaaaatctatggaaagatatggacgg240
tagcaacaagaatatagcacgagccgcgaagttcatttcgttacttttgatatcgctcac300
aactattgcgaagcgcttcagtgaaaaaatcataaggaaaagttgtaaatattattggta360
gtattcgtttggtaaagtagagggggtaatttttcccctttattttgttcatacattctt420
aaattgctttgcctctccttttggaaagctatacttcggagcactgttgagcgaaggctc480
attagatatattttctgtcattttccttaacccaaaaataagggaaagggtccaaaaagc540
gctcggacaactgttgaccgtgatccgaaggactggctatacagtgttcacaaaatagcc600
aagctgaaaataatgtgtagctatgttcagttagtttggctagcaaagatataaaagcag660
gtcggaaatatttatgggcattattatgcagagcatcaacatgataaaaaaaaacagttg720
aatattccctcaaaaatgtctgatcagaagaagcacattgtcgtcataggtgctggaata780
ggaggtactgcaacagcagcaaggttagcaagggagggtttcagagtcactgtcgtcgag840
aagaacgacttctctggaggaaggtgctctttcattcaccacgacggtcacaggttcgac900
cagggaccttcattgtacttgatgcctaagttgtttgaggacgctttcgctgacttagac960
gagaggataggagaccacttggacttattaagatgtgacaacaattacaaagtccatttc1020
gacgacggtgacgctgtccaattgtcatcagacttaacaaagatgaagggtgagttggac1080
aggattgagggacctttaggattcggtaggttcttagatttcatgaaagagacacacgtc1140
aggtacgagcagggtacattcattgctataaagagaaacttcgaaactatatgggactta1200
ataaggttacagtacgtcccagagatttttaggttgcacttattcggtaagatatacgac1260
agagcatcaaaatacttccaaacaaaaaagatgaggatggcttttacttttcaaacaatg1320
tacatgggtatgtcaccttacgacgcacctgcagtctactcattgttgcaatatacagag1380
ttcgcagagggaatttggtacccaaggggtggtttcaacatggtcgtccaaaagttggag1440
tctatagcttctaagaagtacggagctgagttcaggtaccaatctcctgtcgctaagatt1500
aacactgtcgataaagacaagagggtcactggtgtcactttggagtctggagaagtcatt1560
gaggcagacgctgtcgtctgcaacgctgacttggtctacgcttaccaccacttgttgcca1620
ccttgcaactggacaaagaagactttggcatctaagaaattaacatcttcatcaatttct1680
ttttactggtcaatgtctactaaggtccctcaattggacgtccacaacattttcttggct1740
gaggcttacaaggagtcattcgacgagatttttaacgatttcggtttgccttctgaagca1800
tctttctacgtcaacgttccttcaaggatagacgagtctgcagcacctccaaataaggac1860
tcaattatagttttagttccaattggtcacatgaagtctaagacaggtaactcagcagag1920
gagaactacccagagttggtcaacagggctagaaagatggtcttggaggtcatagagagg1980
aggttgggagtcaacaacttcgctaacttgatagaacacgaggaggtcaacgacccatca2040
gtctggcaatctaagttcaacttgtggaggggatcaatattaggtttatcaagggatgtc2100
tttcaggttttg2112
<210>10
<211>663
<212>dna
<213>人工序列(artificialsequence)
<400>10
gatgtctttcaggttttgtggttcagaccttcaacaaaggactctactaacagatatgac60
aatttatttttcgtcggtgcatcaactcaccctggtacaggagtcccaatagtcttggca120
ggatctaaattaacttctgaccaggtctgtaagtcattcggacaaaaccctttgcctagg180
aagttacaggactctcagaagaaatatgcacctgagcaaacaaggaagactgagtcacac240
tggatttattactgcttagcatgctactttgtcactttcttgttcttctatttctttcct300
agggacgacactactactccagcatcttttattaatcagttgttgccaaacgtcttccaa360
ggacagaactctaacgatattaggatataaggccgcatcatgtaattagttatgtcacgc420
ttacattcacgccctccccccacatccgctctaaccgaaaaggaaggagttagacaacct480
gaagtctaggtccctatttatttttttatagttatgttagtattaagaacgttatttata540
tttcaaatttttcttttttttctgtacaaacgcgtgtacgcatgtaacattatactgaaa600
accttgcttgagaaggttttgggacgctcaaggaaaaaagcggccgcgcaaattaaagcc660
ttc663
- 还没有人留言评论。精彩留言会获得点赞!