一种基于比较基因组学的转座子插入多态TIP分子标记的挖掘方法与流程
本发明属于动物遗传育种领域,同时涉及生物信息学及分子生物学领域,具体涉及一种基于比较基因组学的转座子插入多态(Transposon insertion polymorphisims,TIP)分子标记的挖掘方法。
背景技术:
猪的基因组注释表明转座子占整个基因组的39.40%。转座子在猪等哺乳动物基因组上分布非常广泛,据预测转座子插入位点几乎覆盖了基因组上80%以上的基因。由于转座子能在其物种间和物种内转移,因此,像其它的突变源一样,转座子不仅促进了物种间基因组的分化,而且还产生了物种内丰富的遗传多样性,对物种、品种、亚种、品系形成发挥了重要作用。
相关研究表明转座子是驱动基因组结构变异的重要动力,转座子能够介导基因组序列的删除、扩增、倒位、移位、断裂等结构变异,改变宿主基因组大小,成为进化的种子序列,对基因组大小和进化发挥重要作用(H.H.Kazazian,“Mobile Elements:Drivers of Genome Evolution,”Science(80-.);H.L.Levin and J.V.Moran,“Dynamic interactions between transposable elements and their hosts,”Nat.Rev.Genet.;D.J.Finnegan,“Eukaryotic transposable elements and genome evolution,”Trends Genet.)。研究表明:转座能够重塑基因的结构,包括新序列引入、外显子改组、新基因产生、基因重组等;此外,转座插入能够引起宿主基因钝化或激活等多种遗传效应(D.J.Burgess,“Population genetics:Mobile elements across human populations.,”Nat.Rev.Genet.;D.J.Witherspoon et al.,“Mobile element scanning(ME-Scan)identifies thousands of novel Alu insertions in diverse human populations,”Genome Res.)。
TIP与SSR类似,揭示的是一个位点上的不同等位状态(即反转录转座子的插入和缺失),产生共显性标记。这种标记可以对特定位点有无转座子的插入进行检测,只需基因组为模板,不需要酶切、加接头等处理,利于自动化操作,特别适合大量样品的分析,TIP分子标记具有理想分子标记的诸多优点:如多态性高、共显性、基因组分布广泛、检测手段简单快速、重复性好和开发成本低等,同时检测结果带型特定、清楚便于判定,具有很高的应用价值。可应用于猪的分子辅助育种以及遗传进化分析等方面的研究。
技术实现要素:
本发明的目的是针对目前QTL精细定位缺乏多态性高、检测方便、重复性好且可靠性高的分子标记的背景下,提供一种基于比较基因组学的转座子插入多态(TIP)分子标记的挖掘方法。用于解决在目标基因或QTL定位后的特定区段没有良好分子标记的问题,该方法能够找到特定区段上参考序列和非参考序列中所有转座子的多态性插入位点,且开发出的标记检测方便、重复性好且可靠性高。能够很好解决QTL精细定位缺乏分子标记的问题。同时节约了大量时间和金钱。
本发明的目的通过以下方案来实现:一种基于比较基因组学的转座子插入多态(TIP)分子标记的挖掘方法,包括以下步骤:
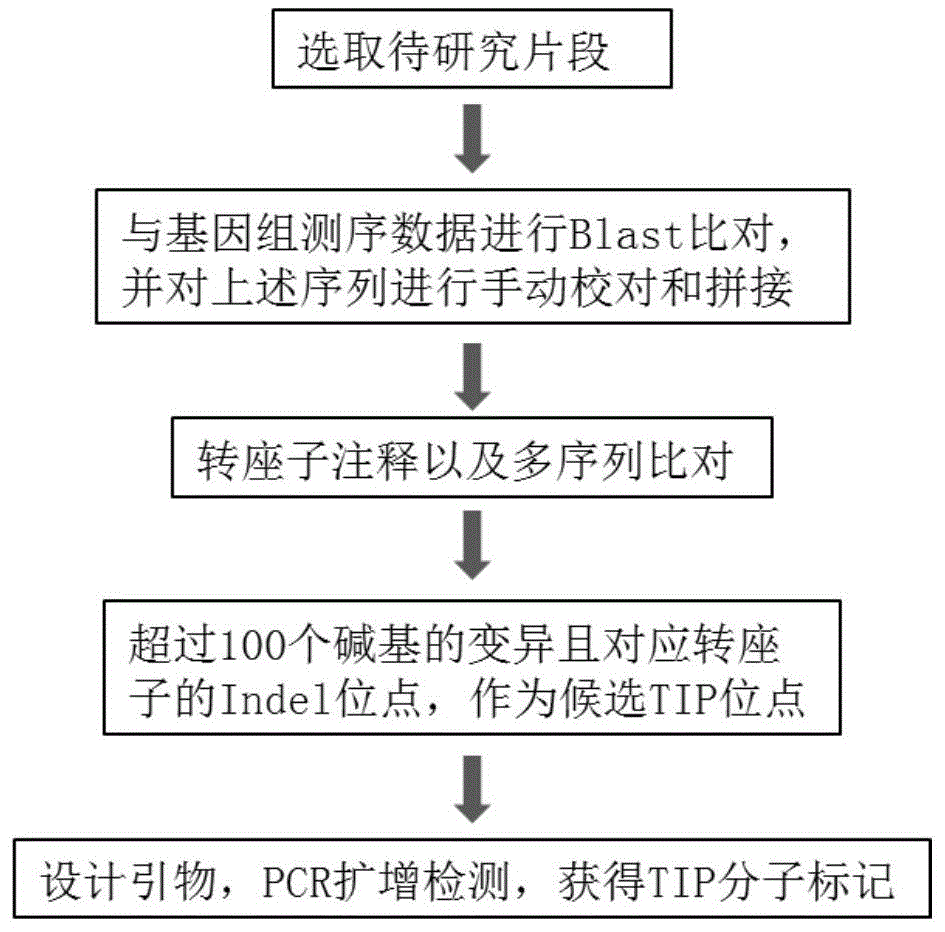
(1)选取目标基因或待研究的基因组片段为参考序列,一般为某一性状的主效基因或QTL定位区段。
(2)将(1)中的参考序列在NCBI中与WGS数据库中猪的基因组测序数据进行Blast比对,获取3个以上不同猪品种基因组中同源的序列,并对上述同源序列进行手动校对和拼接得到拼接序列。不同品种的选取可以根据研究目的进行,如研究脂肪沉积的基因,可以选用脂肪型猪和瘦肉型猪对比寻找。不少于3个品种,亲缘关系越远越好。
(3)根据猪转座子数据对(1)中的参考序列和(2)得到的拼接序列进行转座子注释以及多序列比对。
(4)根据(3)转座子注释结果和多序列比对结果挑选序列间存在的长度为0.1-11kb且对应转座子的Indel位点的结构变异位点(即长度为100-11000个碱基之间),作为候选TIP位点。
(5)根据(4)筛选出的候选TIP位点设计引物,并以不同品种的池DNA为模板进行PCR扩增检测,选取带型清楚且存在多态性的TIP位点,获得TIP分子标记。
本发明所述池DNA(即品种池DNA)由单一品种的若干个体的基因组DNA组成,每个池DNA为不少于2个个体的基因组DNA的等量混合物。针对不同品种,设置不同的池DNA。
所述步骤(2)将参考序列(目标基因或待研究的基因组片段)在NCBI的WGS数据库中进行Blast比对,获取3个以上不同品种基因组中同源的序列,并对上述序列进行手动校对和拼接。
所述步骤(3)中根据猪转座子数据对目标基因或基因组区段(即参考序列)及拼接序列使用RepeatMasker进行转座子标记。
所述步骤(3)中将由目标基因或待研究的基因组片段构成的参考序列及拼接序列使用ClustalX进行多序列比对。
所述步骤(4)中,多序列比对后,挑选不同序列间存在的长度为0.1-11kb且对应转座子的结构变异位点,作为候选TIP位点。
所述步骤(5)中,选取3个以上猪品种,每个品种选择不少于2个个体的基因组DNA制备成品种池DNA,然后以品种池DNA为模板进行PCR扩增检测,选取带型清楚且存在多态性的TIP位点,获得TIP分子标记。
PCR引物需要根据转座子的候选TIP位点两侧的序列进行设计,通过Primer3在线版进行设计,参数使用默认值。
所述参考序列为猪目标基因或猪待研究的基因组片段。
步骤(4)中,当多序列比对结果中长度为0.1-11kb的结构变异位点与步骤(3)猪转座子数据中的转座子注释位点相互对应且该结构变异位点的碱基序列中60%以上的长度能被转座子注解,则认为该结构变异位点是转座子插入引起的,即为候选TIP位点;长度为0.1-11kb的结构变异位点为位于某一条或多条序列(指拼接序列和/或参考序列)中部分区段的相对于其它一条或多条序列出现的插入或缺失的序列片段,这一插入或缺失的序列片段有60%以上的长度能被转座子注解。相互对应是指:多条序列在某一区域,如A-B位点之间,存在结构变异(即有的序列在A-B间有序列,有的序列没有序列),这个结构变异位点与对此片段(结构变异位点)注释出的某一转座子位点如C-D之间有超过60%的重合,即A-B与C-D之间有超过60%的重合。注释位点是猪基因组上存在的所有转座子(目前使用的是repbase数据库https://www.girinst.org/repbase/收录的数据及本实验室自己鉴定出的转座子数据,转座子序列发表在Mobile DNA(C.Chen et al.Retrotransposons Evolution and Impact on lncRNA and Protein Coding Genes in Pigs.Mobile DNA,2019)中)。
结构变异位点可以位于参考序列和/或拼接序列,本发明能够找到特定区段上参考序列和非参考序列(如拼接序列)中所有转座子的多态性插入位点(即对应转座子的结构变异)。
相对于现有技术,本发明具有如下优点:
(1)寻找TIP分子标记快速,高效。通过对数据库中的数据进行提取和比对,同时结合转座子注释信息,可以快速找到不同来源的目标序列中存在的不同转座子位点。并针对这些差异位点设计PCR引物进行检测。
(2)所需时间短,可以快速找到目标片段中存在的TIP分子标记以及完成后续的相关分析。
(3)成本低,标记检测方便,结果清楚。TIP分子标记只需要设计普通PCR引物进行常规PCR即可检测,检测结果清晰易判断。
附图说明
图1本发明实施例的猪GHR基因分子标记研发流程图;
图2GHR基因中转座子插入多态检测电泳图,1、巴马香猪2、五指山猪3、从江香猪4、江口萝卜猪5、藏猪6、梅山猪7、苏姜猪8、宁乡猪9、大围子猪10、杜洛克猪11、大白猪12、长白猪。
具体实施方式
在进一步描述本发明具体实施方式之前,应理解,本发明的保护范围不局限于下述特定的具体实施方案;还应当理解,本发明实施例中使用的术语是为了描述特定的具体实施方案,而不是为了限制本发明的保护范围。
以下结合猪GHR基因中TIP分子标记的研发进行详细说明。
一、序列获取
从NCBI数据库中(https://www.ncbi.nlm.nih.gov/)获取GHR基因(Gene ID:397488)的参考序列。然后利用参考序列与NCBI的WGS数据库中不同品种猪的基因组测序数据进行比对,从而获取到15个测序基因组中GHR基因的序列(含参考序列),其中部分品种的GHR序列由于数据库中基因组测序数据拼接长度不够,需要手动拼接。
二、转座子注释
通过使用RepeatMasker(版本:4.0.7,-cutoff 250-nolow)结合猪转座子数据库对GHR基因序列进行转录转座子注释。仅保留比对得分超过1000且标记长度超过100bp的位点进行后续分析。
三、多序列比对
将15个GHR基因序列使用Clustalx(2.0版)软件进行多序列比对以鉴定结构突变。
四、候选TIP位点筛选
当多序列比对结果中长度大于100bp的结构变异位点与二、转座子注释中转座子注释位点相互对应,更具体地,当结构变异位点中超过60%长度对应转座子(即能被该转座子注解或注释),则该结构变异位点被认定为转座子引起的结构变异,这些位点即为候选TIP位点。
五、引物设计
选取部分由年轻反转录转座子插入引起结构突变位点。根据候选TIP位点两侧的侧翼序列,使用primer3(http://bioinfo.ut.ee/primer3-0.4.0/)设计常规PCR检测引物。使上游引物在候选TIP位点的上游侧翼区上,下游引物在候选TIP位点下游的侧翼区。引物名称及序列如表1所示。
表1 GHR基因中12个候选TIP位点检测引物信息
六、TIP分子标记验证
1.池DNA准备
选取巴马香猪、五指山猪、从江香猪、江口萝卜猪、藏猪、梅山猪、苏姜猪、宁乡猪、大围子猪、杜洛克、大白、长白12个品种,每个品种6个个体,用MiniBEST Universal Genomic DNA Extraction Kit(Ver.5.0,TaKaRa,大连,中国)从耳组织中提取总DNA。主要步骤如下:(1)取2~25mg动物组织,用剪刀剪成碎块。或100μl抗凝全血加PBS补充至200μl。(2)加180μl Buffer GB、20μl蛋白酶K和10μl RNase A(10mg/ml),吸打混匀,于56℃水浴温浴至组织裂解。全血水浴10分钟。(3)向裂解液中加入200μl 100%乙醇,充分吸打混匀。(4)将Spin Column安置于Collection Tube上,将(3)中溶液移Spin Column中,12000rpm离心2分钟,弃滤液。(5)向Spin Column中加500μl的Buffer WA,12000rpm离心1分钟,弃滤液。(6)向Spin Column中加700μl的Buffer WB,12000rpm离心1分钟,弃滤液,重复操作步骤(6)一次。(7)将Spin Column安置于Collection Tube上,12000rpm离心2分钟。(8)将Spin Column安置于新的1.5ml的离心管上,在Spin Column膜的中央处加入100μl的Elution Buffer,室温静置5分钟。(9)12000rpm离心2分钟洗脱DNA。提取得到的基因组DNA通过紫外分光光度计和琼脂糖凝胶进行DNA浓度和质量检测。然后每个品种按等量混合的方式制备两个池DNA,每个池DNA样品含3个个体的DNA来源,共24个池DNA。
2.PCR扩增
设计以下PCR反应体系:
(1)在灭菌PCR管中配制20μl反应体系
表2 PCR反应体系
(2)将PCR管置于PCR仪中,进行如下反应程序
表3 PCR反应程序
注:1:南京诺唯赞生物科技有限公司
3、琼脂糖凝胶检测
(1)称取1.5g琼脂糖置于锥形瓶中,加入100ml 1*TAE电泳缓冲液,加热使之完全溶解。
(2)稍微冷却后倒入插有胶梳的制胶板中,并检查有无气泡存在。室温放置30min待凝胶完全凝固后,小心拔出胶梳,并浸没于加有1*TAE电泳缓冲液的电泳槽中。
(3)用微量上样枪吸取6μl PCR扩增产物加入凝胶孔,同时点5μl DNA标准分子量Maker作为参照。
(4)将电泳槽连接电泳仪,用120V的恒压电泳,根据指示剂的迁移位置,判断电泳情况。
(5)电泳结束后将凝胶置于盛有溴化乙锭溶液的塑料盒中,染色10分钟,将凝胶拿出放到紫外灯下观察并拍照记录。
七、结果分析
部分检测结果如图2所示。在12个品种猪中进行多态性检测,在初步筛选的12个检测位点中,有5个位点(GHR-TIP4、GHR-TIP9、GHR-TIP10、GHR-TIP11、GHR-TIP12)呈现多态现象,且检测结果非常清晰易判断(图2)。
八、结论
经过序列比对从而获得3个以上基因组中目标基因或待研究基因组片段的序列,然后进行转座子注释和多序列比对,从而找出由转座子引起的结构变异位点,获得候选TIP位点。在品种内或品种间进行PCR验证后获得分子标记,可以为品种鉴定提供鉴定方法,可以作为个体鉴定,分子辅助育种的良好标记。具有研发方便,快速,检测成本低、简单、迅速,重复性好,结果清晰等优点。
本发明主要鉴定基因组中特点片段(如某一基因)中存在的所有类型的转座子插入多态位点并开发分子标记。本发明需要使用不同品种的基因组测序数据,并借助比较基因组学中的方法进行多态位点查找,能找到非参基因组中的转座子插入多态位点并开发分析标记。本发明不需要转座子周边有微卫星序列。本发明适合QTL定位后针对性寻找所有转座子(不限种类)的所有多态插入位点(不限制于参考基因组)。
本发明所述的实例是对本发明的说明而不能限制本发明,在与本发明相当的含义和范围内的任何改变和调整,都应认为是在本发明的范围内。
序列表
<110> 扬州大学
<120> 一种基于比较基因组学的转座子插入多态TIP分子标记的挖掘方法
<130> xhx2019050702
<141> 2019-05-07
<160> 24
<170> SIPOSequenceListing 1.0
<210> 1
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
cttgtatccc catgacttgc cta 23
<210> 2
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
gcacctcaca aaatcaaata cctcg 25
<210> 3
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
cagcttttcc ttgcactctg t 21
<210> 4
<211> 17
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
aggttcacac gcggctc 17
<210> 5
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
agttcttggc atgtaagtcc t 21
<210> 6
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
gcctccccac tatccact 18
<210> 7
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
ttttcacaca tatgcttcat ggcta 25
<210> 8
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 8
tgccagaaca ctacattcta cact 24
<210> 9
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 9
ttgctgcatc ccataggttt 20
<210> 10
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 10
agcctccagc aaaaatactt cagac 25
<210> 11
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 11
aatgttggtt ctagctcact agga 24
<210> 12
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 12
tcaactggcc cagatctttc c 21
<210> 13
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 13
tcctcttagc tccaaatcac c 21
<210> 14
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 14
gggcaaaaca tagacaccct 20
<210> 15
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 15
tgccatgaca ggaactccc 19
<210> 16
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 16
tgacagatca tatgctggac cac 23
<210> 17
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 17
ctccatagtg gctgtaccag t 21
<210> 18
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 18
gcgacctaca ccacatgcaa g 21
<210> 19
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 19
catgaagccc cgtaacatcc g 21
<210> 20
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 20
tcctgtgcag cattaagcat 20
<210> 21
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 21
aactgggcac ttagataaat tccac 25
<210> 22
<211> 17
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 22
ccacgggtgc ctaccat 17
<210> 23
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 23
gaatttagga aagtagcaga acaca 25
<210> 24
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 24
tcagcatata agtctttcac ctcc 24
- 还没有人留言评论。精彩留言会获得点赞!