控制水稻开花时间的基因、蛋白质、基因表达盒、表达载体、宿主细胞、方法及应用与流程
本发明属于分子遗传学领域,具体是指一种控制水稻开花时间的基因、蛋白质、基因表达盒、表达载体、宿主细胞、方法及应用。
背景技术:
在高等植物中,开花代表着从营养生长到生殖生长的转换过程,其在植物体的整个生长发育阶段中具有重要的作用。而何时开花这一生物性状则受到植物体自身遗传因子及外界环境因素的双重作用。在这双重作用的影响下,一系列开花诱导过程在高等植物中具有普遍的规律,即植物体叶片通过感受外界生长条件(光照,温度,湿度等),在适宜的时间产生成花物质(或叫开花素florigen),成花物质经过输导组织由叶片运送到茎尖生长点,刺激顶端分生组织成花。
开花期是水稻进化和适应过程中的重要性状,理解水稻开花期性状的遗传基础、克隆候选基因可以提高水稻的环境适应能力和可塑性,这对培育适应不同生态区的优良水稻品种具有重要的意义,同时也将促进产量等与开花期密切相关的重要生产性状的遗传改良进程。此外,水稻的整体生育期主要决定于开花期的长短,适度缩短开花期能够减少整体的生育期,从而降低种植成本。
f-box蛋白是一类含有40-50个保守氨基酸f-box结构域的蛋白家族成员,在真核生物中广泛存在。在泛素-蛋白酶体途径(ubiquitin-proteasomepathway,upp)中,f-box蛋白因特异识别底物蛋白而参与细胞周期调控、转录调控、细胞凋亡、细胞信号转导等生命活动。在植物中,f-box蛋白质家族是最大的、也是进化最迅速的家族之一。水稻中的f-box蛋白编码基因多达687个(jainm,nijhawana,arorar,etal.f-boxproteinsinrice.genome-wideanalysis,classification,temporalandspatialgeneexpressionduringpanicleandseeddevelopment,andregulationbylightandabioticstress[j].plantphysiol,2007,143(4):1467-83.),他们对水稻的叶、根、花器官的发育、种子萌发、细胞分裂素信号均有调控作用(薛红卫,渠莉.调控叶倾角的f-box蛋白及其应用:中国专利,cn108727479[p],2018-11-02.;songs,daix,andzhangwh.aricef-boxgene,osfbx352,isinvolvedinglucose-delayedseedgerminationinrice[j].jexpbot,2012,63(15):5559-68.;kimhj,kieberjj,andschallerge.thericef-boxproteinkissmedeadly2functionsasanegativeregulatorofcytokininsignalling[j].plantsignalbehav,2013,8(12):e26434.;yanys,chenxy,yangk,etal.overexpressionofanf-boxproteingenereducesabioticstresstoleranceandpromotesrootgrowthinrice[j].molplant,2011,4(1):190-7.;lil,liy,songs,etal.anantherdevelopmentf-box(adf)proteinregulatedbytapetumdegenerationretardation(tdr)controlsriceantherdevelopment[j].planta,2015,241(1):157-66.)。由于水稻中的f-box蛋白基因众多,因此仍有很多成员的功能尚不清楚。
本发明通过利用rnai方法抑制水稻f-box蛋白编码基因osfbo14的表达量,发现了osfbo14基因对水稻开花时间的调控作用。利用osfbo14-rnai载体以及rnai方法,可以提早水稻的开花时间,从而可以缩短水稻生育期,减少种植成本,提高水稻的生态适应性。
技术实现要素:
为实现上述目的,本发明一方面提供了一种控制水稻开花时间的基因(osfbo14),其特征在于:包括seqidno.1或其互补序列、和/或seqidno.3或其互补序列。
本发明另一方面提供了一种蛋白质,其特征在于:所述蛋白质的氨基酸序列如seqidno.2所示;所述蛋白质的氨基酸序列为seqidno.2所示的序列经过取代和/或缺失和/或添加一个或多个氨基酸,且具有与seqidno.2所示的序列相同功能的序列。
本发明再另一方面提供了一种含有上述核酸序列的基因表达盒,其特征在于:所述表达盒含有权利要求1所述的核酸序列和其反向互补序列;所述核酸序列和其反向互补序列之间以水稻waxy-aintron1序列间隔;所述核酸序列、反向互补序列、水稻waxy-aintron1序列与ubiquitin启动子和camv35s终止子可操作地连接。
本发明再另一方面提供了一种含有上述基因表达盒的表达载体,其特征在于:所述载体还包含camv35s:hygromycin:camv35spolya表达盒和camv35s:gus:nos表达盒。
本发明再另一方面提供了一种包含上述表达载体的宿主细胞,其特征在于:所述宿主细胞为植物细胞或原核生物细胞;任选地,所述宿主细胞为大肠杆菌或农杆菌细胞。
本发明再另一方面提供了一种产生转基因作物的方法,其特征在于:所述方法为上述的表达载体或上述的宿主细胞转化作物,得到转基因作物。
本发明再另一方面提供了一种产生转基因种子的方法,其特征在于:利用上述方法产生的转基因作物产生转基因种子。
本发明再另一方面提供了一种上述的核酸序列、蛋白质、基因表达盒、表达载体、宿主细胞、产生转基因作物的方法在改变作物开花时间中的应用。
进一步地,所述作物为水稻;所述改变作物开花时间的方式为提早。
上述,本发明中的蛋白质,蛋白的氨基酸序列如seqidno.2所示;在一些实施方案中,蛋白质的氨基酸序列为seqidno.2所示的序列经过取代和/或缺失和/或添加一个或多个氨基酸,且具有与seqidno.2所示的序列相同功能的序列。
上述,本发明中的基因表达盒,表达盒含有上述的核酸序列和其反向互补序列;在一些实施方案中,核酸序列和其反向互补序列之间以水稻waxy-aintron1序列间隔;在一些实施方案中,核酸序列、反向互补序列、水稻waxy-aintron1序列与ubiquitin启动子和camv35s终止子可操作地连接。
上述,本发明中的表达载体,载体含有上述表达盒;在一些实施方案中,载体还包含camv35s:hygromycin:camv35spolya表达盒和camv35s:gus:nos表达盒。
上述,本发明中的宿主细胞,包含上述表达载体;在一些实施方案中,宿主细胞为植物细胞或原核生物细胞;在一些实施方案中,宿主细胞为大肠杆菌或农杆菌细胞。
上述,本发明中的产生转基因作物的方法,用上述表达载体或上述宿主细胞转化作物,得到转基因作物。
上述,本发明中的产生转基因种子的方法,从上述方法产生的转基因作物产生转基因种子。
上述,本发明中的上述核酸、蛋白、基因表达盒、表达载体、宿主细胞、方法在在改变作物开花时间中的应用;在一些实施方案中,作物为水稻;在一些实施方案中,改变作物开花时间的方式为提早。
本发明的优点及有益效果如下:osfbo14基因调控开花时间的功能是之前没有报道过的。本发明可以根据种植地点的气候特征通过增加或降低osfbo14基因的表达量,人为地调整水稻生育期,从而提高水稻的生态适应性。此外,水稻的整体生育期主要决定于开花期的长短,适度缩短开花期能够减少整体的生育期,从而降低种植成本。
附图说明
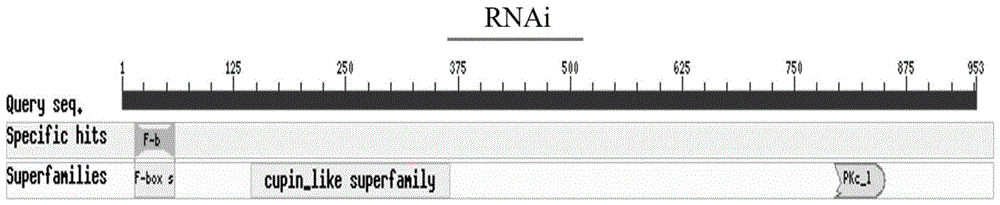
图1为本发明osfbo14基因编码蛋白的结构域分布和rnai干涉片段位置示意图。
图2为本发明osfbo14-pds1301载体图。其中,各元件英文及各缩写含义列举如下:
图3为本发明osfbo14-rnai材料中osfbo14基因表达检测。其中,zh11-1:第一份中花11野生型水稻;zh11-2:第二份中花11野生型水稻;r1-r13:不同的osfbo14基因rnai植株。
图4为本发明osfbo14-rnai材料抽穗期表现。其中,zh11:中花11野生型水稻;r3、r11:不同的osfbo14基因rnai植株。照片摄于播种后73天。
具体实施方式
提供以下定义和方法用以更好地界定本申请以及在本申请实践中指导本领域普通技术人员。除非另作说明,术语按照相关领域普通技术人员的常规用法理解。
如本文所用,所述“水稻”是指水稻(oryzasativa),包括所有可与水稻繁殖的植物品种,包括野生稻种以及那些属于稻属的允许物种间繁殖的植物。
除非另有所指,核酸以5’至3’方向从左向右书写;氨基酸序列以氨基至羧基方向从左向右书写。氨基酸在本文可以用其通常所知的三字母符号或iupac-iub生物化学命名委员会推荐的单字母符号来表示。同样地,可以用通常接受的单字母码表示核苷酸。数字范围包括限定该范围的数字。如本文所用,“核酸”包括涉及单链或双链形式的脱氧核糖核苷酸或核糖核苷酸多聚物,并且除非另有限制,包括具有天然核苷酸基本性质的已知类似物(例如,肽核酸),所述类似物以与天然存在的核苷酸类似的方式与单链核酸杂交。如本文所用,术语“编码”或“所编码的”用于特定核酸的上下文时,指该核酸包含指导该核苷酸序列翻译成特定蛋白的必需信息。使用密码子表示编码蛋白的信息。如本文所用,涉及特定多核苷酸或其所编码的蛋白的“全长序列”指具有天然(非合成)内源序列的整个核酸序列或整个氨基酸序列。全长多核苷酸编码该特定蛋白的全长、催化活性形式。本文可互换地使用术语“多肽”、“多肽”和“蛋白”,以指氨基酸残基的多聚物。该术语用于氨基酸多聚物,其中一个或多个氨基酸残基是相应天然存在的氨基酸的人工化学类似物。该术语还用于天然存在的氨基酸多聚物。本文可互换地使用术语“残基”或“氨基酸残基”或“氨基酸”,以指被并入蛋白、多肽或肽(统称“蛋白”)的氨基酸。氨基酸可以是天然存在的氨基酸,并且除非另有限制,可以包括天然氨基酸的已知类似物,所述类似物可以与天然存在的氨基酸相似的方式起作用。
术语“性状”是指植物或特定的植物材料或细胞的生理、形态、生化或物理特性。在一些情况下,此特性对人眼是可见的,诸如种子或植株大小,或者可通过生物化学技术测量,诸如检测种子或叶的蛋白质、淀粉或油含量,或者通过观察代谢或生理过程,例如通过测量对水剥夺或特定盐或糖或氮浓度的耐受性,或者通过观察一个或多个基因的表达水平,或者通过农艺观察结果诸如渗透胁迫耐受性或收率。
“转基因”是指其基因组因异源核酸(诸如重组dna构建体)的存在而发生改变的任何细胞、细胞系、愈伤组织、组织、植株部分或植株。本文所用的术语“转基因”包括那些最初的转基因事件以及从最初的转基因事件通过有性杂交或无性繁殖而产生的那些,并且不涵盖通过常规植物育种方法或通过自然发生的事件(诸如随机异花受精、非重组病毒感染、非重组细菌转化、非重组转座或自发突变)进行的基因组(染色体或染色体外)改变。
“植物”包括对整株植物、植物器官、植物组织、种子和植物细胞以及它们的子代的标引。植物细胞包括但不限于来自种子、悬浮培养物、胚芽、分生区域、愈伤组织、叶、根、苗、配子体、孢子体、花粉和小孢子的细胞。“子代”包含植物的任何后续世代。
在本申请中,将词语“包括”、“包含”或其变体应理解为除所描述的元素、数或步骤外,还包含其它元素、数或步骤。“受试植物”或“受试植物细胞”是指遗传改造已经生效的植物或植物细胞,或者如此改造的植物或细胞的子代细胞,该子代细胞包含所述改造。“对照”或“对照植物”或“对照植物细胞”提供用于测量受试植物或植物细胞表型改变的参考点。
阴性或对照植物可以包括,例如:(a)野生型植物或细胞,即与遗传改造起始材料具有相同基因型的植物或细胞,所述遗传改造产生受试植物或细胞;(b)与所述起始材料具有相同基因型但已用空构建体(即用对目的性状无已知效果的构建体,诸如包含标物基因的构建体)转化的植物或植物细胞;(c)是受试植物或植物细胞的非转化分离子的植物或植物细胞;(d)与所述受试植物或植物细胞在遗传上一致但未暴露于会诱导目的基因表达的条件或刺激物的植物或植物细胞;或(e)受试植物或植物细胞自身,其处于目的基因不被表达的条件下。
本领域技术人员会容易地认同,诸如位点特异性诱变和随机诱变、聚合酶链式反应方法和蛋白工程化技术的分子生物学领域的进步提供了广泛的适当的工具和操作步骤,以用于改造或者工程化农业上感兴趣的蛋白的氨基酸序列和潜在的基因序列。
在一些实施方案中,可以对本申请的核苷酸序列进行改变,以进行保守氨基酸替换。保守氨基酸替换的原则和实例在下文中进一步描述。在某些实施方案中,可以依照公开的单子叶密码子偏好性对本申请的核苷酸序列进行不改变氨基酸序列的替换,例如可以用单子叶植物偏好的密码子替换编码同一氨基酸序列的密码子,而不改变该核苷酸序列所编码的氨基酸序列。在一些实施方案中,以编码同一氨基酸序列的不同密码子替换本申请中的部分核苷酸序列,从而在改变核苷酸序列的同时不改变其编码的氨基酸序列。保守变体包括由于遗传密码子简并性而编码实施方案的蛋白中的一种的氨基酸序列的那些序列。在一些实施方案中,根据单子叶植物偏好密码子替换本申请中的部分核苷酸序列。本领域技术人员会认识到氨基酸添加和/或取代通常基于氨基酸侧链取代基的相对相似性,例如,所述取代基的疏水性、电荷、大小等等。具有各种前述所考虑性质的示例性氨基酸取代基团为本领域技术人员所公知,并且包括精氨酸与赖氨酸;谷氨酸和天门冬氨酸;丝氨酸和苏氨酸;谷氨酰胺和天冬酰胺;以及缬氨酸、亮氨酸和异亮氨酸。关于不影响目的蛋白生物学活性的适当氨基酸取代的指南可以在dayhoff等人(1978)atlasofproteinsequenceandstructure(蛋白序列和结构图集)(natl.biomed.res.found.,washington,d.c)(通过引用并入本文)的模型中找到。可以进行诸如将一个氨基酸换作具有相似性质的另一个氨基酸的保守性取代。序列一致性的鉴定包括杂交技术。例如,将已知核苷酸序列的全部或部分用作与其它相应核苷酸序列选择性杂交的探针,所述其它相应核苷酸序列存在于来自所选生物体的已克隆基因组dna片段或cdna片段群(即基因组文库或cdna文库)。所述杂交探针可以是基因组dna片段、cdna片段、rna片段或其它寡核苷酸,并且可以用诸如32p的可检测基团或其它可检测标志物来标记。因而,例如,可以通过标记基于实施方案序列的合成寡核苷酸制备杂交探针。制备杂交探针和构建cdna及基因组文库的方法通常为本领域已知。可以在严紧条件下进行所述序列的杂交。如本文所用,术语“严紧条件”或“严紧杂交条件”表示如下条件,即在该条件下,相对于与其它序列杂交,探针将以可检测的更大程度(例如,背景的至少2倍、5倍或10倍)与其靶序列杂交。严紧条件是序列依赖性的并且在不同环境中有所不同。通过控制杂交严紧性和/或控制清洗条件,可以鉴定与所述探针100%互补的靶序列(同源探针法)。可选择地,可以调节严紧条件,以允许一些序列错配,以便检测较低的相似度(异源探针法)。通常,探针长度少于约1000或500个核苷酸。通常,严紧条件是如下的条件,即在该条件中,盐浓度为ph7.0至8.3下,少于约1.5mna离子,通常约0.01m至1.0mna离子浓度(或其它盐),并且温度条件为:当用于短探针时(例如10到50个核苷酸),至少约30℃;当用于长探针时(例如大于50个核苷酸),至少约60℃。还可以通过添加诸如甲酰胺的去稳定剂来实现严紧条件。示例性的低严紧条件包括37℃下使用30%至35%的甲酰胺缓冲液、1mnacl、1%sds(十二烷基硫酸钠)杂交,50℃至55℃下在1×至2×ssc(20×ssc=3.0mnacl/0.3m柠檬酸三钠)中清洗。示例性的中度严紧条件包括37℃下在40%至45%甲酰胺、1.0mnacl、1%sds中杂交,55℃至60℃下在0.5×至1×ssc中清洗。示例性的高严紧条件包括37℃下在50%甲酰胺、1mnacl、1%sds中杂交,60℃至65℃下在0.1×ssc中最后清洗至少约20分钟。任选地,清洗缓冲液可以包含约0.1%至约1%sds。杂交持续时间通常少于约24小时,通常为约4小时至约12小时。特异性通常依赖杂交后的清洗,关键因素在于最后清洗溶液的离子强度和温度。dna-dna杂合体的tm(热力学熔点)可以近似自meinkothandwahl(1984)anal.biochem.138:267-284的公式:tm=81.5℃+16.6(logm)+0.41(%gc)-0.61(%甲酰胺)-500/l;其中m是一价阳离子的克分子浓度,%gc是dna中鸟苷和胞嘧啶核苷酸的百分数,“甲酰胺%”是杂交溶液的甲酰胺百分数,而l是杂合体的碱基对长度。tm是(确定的离子强度和ph下)50%的互补靶序列与完全匹配的探针杂交时的温度。通常将清洗至少进行至达到平衡,并且达到低的杂交背景水平,诸如进行2小时、1小时或30分钟。每1%的错配对应使tm降低约1℃;因而,可以调节tm、杂交和/或清洗条件,从而与所需一致性的序列杂交。例如,如果需要≥90%一致性的序列,可以将tm降低10℃。通常,将严紧条件选择为比确定离子强度和ph下的特异序列及其互补序列的tm低约5℃。然而,在非常严紧的条件下,可以在比所述tm低4℃下进行杂交和/或清洗;在中度严紧条件下,可以在比所述tm低6℃下进行杂交和/或清洗;在低严紧条件下,可以在比所述tm低11℃下进行杂交和/或清洗。
在一些实施方案中,还包括核苷酸序列及其编码的氨基酸序列的片段。如本文所用,术语“片段”指实施方案的多核苷酸的核苷酸序列的一部分或者多肽的氨基酸序列的一部分。核苷酸序列的片段可以编码蛋白片段,所述蛋白片段保留天然或相应全长蛋白的生物学活性,并因而具有蛋白活性。突变体蛋白包括天然蛋白的生物活性片段,其包含保留天然蛋白生物学活性的连续氨基酸残基。一些实施方案还包括转化的植物细胞或转基因植物,其包含至少一种实施方案的核苷酸序列。在一些实施方案中,使用表达载体转化植物,所述表达载体包含至少一种实施方案的核苷酸序列以及与其可操作地连接的在植物细胞中驱动表达的启动子。转化的植物细胞和转基因植物表示基因组内包含异源多核苷酸的植物细胞或植物。一般来说,所述异源多核苷酸在转化的植物细胞或转基因植物的基因组内稳定地整合,以致将所述多核苷酸传递给后代。可以将所述异源多核苷酸单独地或作为表达载体的一部分整合进基因组。在一些实施方案中,本申请涉及的植物包括植物细胞、植物原生质体、可以再生出植物的植物细胞组织培养物、植物愈伤组织、植物团块和植物细胞,其为完整的植物或者植物的部分,诸如胚胎,花粉,胚珠,种子,叶,花,枝,果实,果仁,穗,穗轴,壳,秸秆,根,根尖,花药等等。本申请还包括源于本申请的转基因植物或其子代、并因而至少部分地包含本申请的核苷酸序列的植物细胞、原生质体、组织、愈伤组织、胚胎以及花、茎、果实、叶以及根。
若无特别指明,实施例按照常规实验条件,如sambrook等人的分子克隆实验手册(sambrookj&russelldw,molecularcloning:alaboratorymanual,2001),或按照制造厂商说明书建议的条件。若未特别指明,实施例中所用的化学试剂均为常规市售试剂,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。
实施例一水稻osfbo14基因序列和干涉片段序列
水稻osfbo14(loc_os03g27250)基因的编码区序列见seqidno.1,其编码的蛋白包含f-box结构域(图1),序列见seqidno.2。尚无资料表明该基因在水稻中的功能以及其所控制的具体性状。为了明确基因在水稻中的功能,本发明拟采用rnai方法降低osfbo14基因在水稻中的表达量。本发明选取seqidno.1所示序列第1115-1529个核苷酸之间的区域作为构建rnai载体的干涉片段(序列见seqidno.3)。
实施例二水稻osfbo14基因rnai载体的构建。
根据osfbo14的全长cdna序列设计引物(seqidno.4和seqidno.5),pcr扩增得到带有酶切位点的osfbo14干涉片段。将干涉片段以及该片段的反向互补序列构建到表达载体上,具体步骤如下:
1、将osfbo14干涉片段用kpni和bamhi酶切,回收目标条带,与kpni和bamhi酶切的表达载体质粒pds1301(chuetal.,promotermutationsofanessentialgeneforpollendevelopmentresultindiseaseresistanceinrice.genesdev,2006,20:1250-1255.),连接(所使用的内切酶均购自takara公司,用法及用量按照该公司的产品说明书;连接酶为invitrogen公司产品,用法及用量按照该公司产品说明书);
2、连接产物通过电转化的方法(电转化仪为eppendorf公司产品,所用电压为1800v,操作方法见仪器说明书)导入dh10b(购自promega公司),在含有250ppm卡那霉素(roche公司产品)的la(la配方参见j.萨姆布鲁克,ef弗里奇,t曼尼阿蒂斯著,黄培堂,王嘉玺等译,分子克隆实验指南(第三版),科学出版社,2002版)抗性培养基上涂皿培养;
3、将la抗性培养基上长出的单菌落在超净工作台接种于灭菌的10ml离心管,管内预先加入3ml含250ppm卡那霉素的lb抗性培养基,然后在37℃摇床上培养16-18小时。按照(《分子克隆实验指南》,j.萨姆布鲁克和d.w.拉塞尔著,黄培堂等译,科学出版社,2002)所述抽提质粒,用kpni和bamhi酶切并电泳检测,根据插入片段的大小获得阳性质粒载体pds1301-fbo14-1;
4、将osfbo14干涉片段用spei和saci酶切,回收目标条带,与spei和saci酶切的质粒pds1301-fbo14-1连接,依据2)、3)步得到抑制表达载体:pds1301-hdt1-2;
5、把新构建的表达载体pds1301-fbo14-2通过电转化的方法(参考文献和使用的电压参数如上所述)导入农杆菌eha105(购于cambia公司)菌株中。
osfbo14-pds1301载体图谱见图2。
实施例三水稻osfbo14基因rnai转基因植株的制备
本发明采用农杆菌介导法将osfbo14基因表达载体转入水稻,所用农杆菌菌株为eha105。水稻种子消毒后诱导愈伤,然后与农杆菌共培养以感染愈伤,通过选择培养筛选被转化的愈伤,之后在选择培养基上进行植株再生。具体过程如下:
1、诱导水稻愈伤组织:取中花11(zh11)品种水稻成熟种子(中国农业科学院作物科学研究所选育并提供),人工或者机械脱壳,挑选饱满光洁无菌斑的种子,放入100ml无菌烧杯中,倒入70%酒精(15ml)消毒2min;倒去酒精,加入100ml30%次氯酸钠(naclo)溶液,浸泡30min;倒去次氯酸钠溶液,用无菌蒸馏水清洗种子4-5遍,最后一遍浸泡30min。将种子放在无菌滤纸上吸干,置入成熟胚诱导培养基中,每皿20-30颗;操作完毕用封口膜(microporetmsurgicaltape)封好培养皿,在28℃光照培养箱培养。在暗培养条件下诱导愈伤组织,需要7~10天;在超净工作台上打开培养皿,用镊子挑取自然分裂的胚性愈伤组织(淡黄色,致密呈球状),置入继代培养基中,在28℃暗培养1周继代。
2、农杆菌培养:挑取转化有目的表达载体的农杆菌单克隆于15mlyep培养液中(含相应的抗生素),28℃,250rpm振荡培养12~16h至菌液od600为0.8-1.0。
3、共培养和抗性愈伤组织的选择:将培养好的菌液于室温下,4000rpm离心10min,去上清。将长到一定大小的水稻愈伤组织挑出,放入农杆菌悬浮液,水平摇床上80rpm共培养30min;将愈伤组织取出,置于无菌的滤纸上沥干30-40min;将愈伤组织置于有一张无菌滤纸的共培养基上,25℃暗培养3天。
4、选择培养:愈伤组织取出,用无菌水清洗5-6次,其间需不停振荡。再用含300mg/l羧苄青霉素钠(carb)的无菌水清洗2遍,每次在水平摇床上摇晃30min,最后置于无菌滤纸上沥干2小时。将晾干的愈伤转入含300mg/l羧苄青霉素钠(carb)和相应筛选压力的选择培养基上,28℃暗培养14天,共进行两轮选择,直到颗粒性的抗性愈伤组织长出。
5、抗性愈伤组织的诱导分化和生根:挑取从不同愈伤来的颜色鲜黄的抗性愈伤3-5颗,移入装有分化培养基的塑料广口瓶中,用封口膜封好,放入恒温(25℃)培养室中(16h/8h),等待分化成苗(约40天)。待苗长至3cm左右,用剪刀从苗基部剪去老根和愈伤组织,放入生根培养基中壮苗(约1周)。
6、炼苗移栽:将苗根部和茎叶分化完好的试管挑出,打开封口膜,加入适量蒸馏水或无菌水(防止培养基长菌),炼苗2~3天,然后洗去琼脂,移栽苗到温室土钵中。
实施例四osfbo14基因在rnai水稻材料中的表达量检测。
采用qrt-pcr方法检测得到的13株rnai水稻材料(分别为r1-r13)中osfbo14基因的转录水平表达量。步骤包括:
1、rna提取。1)将25-50mg左右组织放入匀浆器内,加1mltrizol试剂匀浆,转入1.5ml的离心管中。2)2-8℃、12000g离心15min后,将上清液转入新的离心管中。3)上清液在室温温育5min后,然后以每1mltrizol试剂加入0.2ml比例加入氯仿,盖紧离心管,涡旋仪剧烈摇荡离心管15sec,室温下温育2-3min。3)2-8℃、12000g离心15min,混合物分层,下层为红色的酚-氯仿层,上层为无色的水相(rna在水相中)。4)用枪头将水相转移到新的离心管中,按1mltrizol液加0.5ml异丙醇的比例加入异丙醇沉淀rna室温温育10min。5)2-8℃、12000g离心10min。6)去除上清,按1mltrizol液加入至少1ml的比例加入75%乙醇洗涤,涡旋振荡样品,2-8℃、7500g离心5min。7)小心弃去上清液,然后空气或真空干燥5-10min,加30-40μl无rna酶的(rnase-free)水,用枪头吸几次后于55-60℃下温育10min,溶解rna。8)电泳检测:从提取的总rna中取2-5μl进行电泳检测。
2、反转录pcr。1)dna酶处理,按照invitrogen公司dnase1amplificationgradekit说明书操作。2)cdna第一条链的合成,按照罗氏transcriptorfirststrandcdnasynthesis试剂盒说明书操作。3)realtime-pcr。使用水稻内参基因actin1作为内参,设计引物seqidno.6和seqidno.7,并设计osfbo14基因的表达检测引物seqidno.8和seqidno.9,分别进行realtime-pcr,反应体系为:
注:引物稀释取10μl母液稀释至600μl。
pcr程序为:95℃,10min;(95℃,10sec;60℃,55sec)×40循环。
realtime-pcr反应完成后,根据仪器生成的内参基因和目标基因的平均ct(扩增循环数)值,依照公式rq=2^(-δδct),计算对应样本的相对表达量rq值,即内参基因平衡化后相对于对照表达量的倍数。
鉴定结果见图3。以zh11-1为对照参照,除r7材料外,其余rnai植株中osfbo14基因的表达量均有不同程度的降低。收获r1-r13植株的种子。
实施例五osfbo14基因rnai水稻材料的性状表现。
种植r3和r11种子,并对得到的转基因植株进行表型观察,发现r3和r11植株的开花(抽穗)时间比对照中花11(zh11)明显提早(生长环境:2017年和2018年武汉5中旬播种,6月12号插秧,于华中农业大学试验田),结果见图4。性状调查结果表明,zh11的开花时间为77.8天,r3和r11植株的开花时间为71.5天,结果见表1。osfbo14基因表达量降低后能够导致水稻开花提早,这表明osfbo14基因对水稻开花时间具有调控作用。而提早水稻开花时间有助于缩短水稻生育期,减少种植成本;同时,由于osfbo14基因对水稻开花时间具有调控作用,因此可以根据种植地点的气候特征通过增加或降低osfbo14基因的表达量,人为地调整水稻生育期,从而提高水稻的生态适应性。
表1osfbo14-rnai材料的开花时间
采用lsd法检验开花时间数据的差异显著性(α=0.05),*表示与zh11相比开花时间差异显著(p<0.05)。
序列表
<110>江汉大学
<120>控制水稻开花时间的基因、蛋白质、基因表达盒、表达载体、宿主细胞、方法及应用
<160>9
<170>siposequencelisting1.0
<210>1
<211>2862
<212>dna
<213>oryzasativa
<400>1
atggcggcggggaacggccggatggaggcggcgctgggctgcctcgcggcgctccccgac60
gaggtgctctgcgccgtcgtcgacctcctcccgcccaccgacgtcggccgcctcgcctgc120
gtcagcagtgtcatgtacatactttgcaatgaggagcctctctggatgagcaagtgtctt180
tcagttgggggtcttcttgtgtatagaggttcttggaagaaaacagcattgtctagactt240
aatctttgttcagaaaatgatgagatttaccagaagcctcgccattttgatgggttcaat300
tccatgcacttatacaggagatggtacagatgttttactaatttgagtagcttttccttt360
gataatgggcacgttgaaaggaaagatgacctttctctagaccaatttcgcgctcagtat420
gatggaaaatgtccagttttgcttactaaactggctgaaacctggccagcaaggactaaa480
tggacagcgcagcaactgacacatgattatggtgaagttccctttaggatatctcagaga540
agccctcaaaagataaaaatgaaactaaaagattatgttttttacatggaactccagcat600
gacgaagatccactttacatatttgatgataagtttggagaatcagcacctacactattg660
gaagattacagtgtccctcatctatttcaagaagatttctttgaaatcatggattacgac720
caacgaccagctttcagatggcttattattggaccagagagatcaggtgcttcttggcat780
gttgatccagggttgaccagtgcctggaatactcttctttgtggccgaaaaaggtgggca840
atgtaccctcctggaagagtaccaggtggtgtcacagtacatgtcagtgatgaagatggt900
gatgttgacattgaaactcctacatctttgcagtggtggctagatatctacccaaatctt960
gctgagcatgagaaaccactggaatgcacacaattaccaggagagaccatatttgttcct1020
agtgggtggtggcattgtgttttgaaccttgacatgacaattgctgtcacgcaaaatttt1080
gtcaaccaatcaaattttaagcatgtatgtttggacatggcacctggttactgtcacaaa1140
ggagtttgccgtgctggcttacttgctgctccagacaaatctattagagatattgaaaat1200
cttcctagtataacgagtagattgaaccactctgacatggcctgtaaggaaaaaagactg1260
aaaagttcagagcctataagaacttcaaataatgcaaatcagtgttctgcatttgagttc1320
tcagatgttcatgaaaacttgggggaccaagttttttcgtatgatatagatttcttatcc1380
caattccttgagaaagaaaaggatcactattcttctgtctggagccctactaattcaatt1440
ggccagagagaagcaagagaatggctacgtaggctatgggttcttaaacctgaattgaga1500
gaactaatatggaagggtgcatgtctagcaattaatgtagacaagtggtattcatgctta1560
gaggaaataagtgcatgccatagtttaccaccaccttctgaagatgagaagcttcctgtt1620
ggcacaggtagcaacccagtcttcattgtttctggcaatgtgatcaaaatttatgctgaa1680
ggagggttgggttattctatacatggtttgggcacagagcttgagttctatgatcttctg1740
caaaaacttggctcgccattgatcaaccatgtccctgagatcattgcaagtggctttctt1800
gtgtacctggatggtgtctacaagacagttccatgggatggaaacggaataccagatgtt1860
ctagctaaatactactctttggaggtgtcttatgcaaacggctcttttcctcttggatta1920
tggagcaagcaactgtttggattgagtaattcaactgatgctccagacagaccaatttgt1980
ccttacatggttaccagaaaatgcaaaggggatatttttgctcgcatacgtgataaattg2040
accaagactgatgttttgaatcttgcatcatccttgggagttcaaatgcgaaatattcat2100
caattaccccttccacatgtggaacacatatccaaatctgggaacgaagatatcaaagca2160
aaggaaaattcaatttctgatgtcactcatgttccgcctgaatggaaacaagtagtttct2220
actctagacaggagaaagaaaagtataaagaagcatctaagtaactggggtggttcaatt2280
ccacaggttctaattgagaaggctgaagaatatctccctgacgacatccgctttcttatc2340
aagtttgttaaggacgatgatggtgattcagtctatgtggtaccttcttggatacattca2400
gatataatggatgataacattctcattgaggggaccacagaaccaggaacttccactgat2460
tgcattgccgttgaagatctgaacaaaatggatgcaattcatatcattgatttcagtgat2520
ctgtccattggggatcctctatgtgacttaattccactgcacttggatgtattccgtggt2580
gatattgatcttctcaggcagtttttacgaagctatcagcttccttttctgagagcagaa2640
tcaaataaagatatatacaagtcaatacaaaattctaaattcagcagggcatcgtatcgt2700
gcgatgtgctactgcatacttcacgaggacaacgtcctgggagccatatttagcctgtgg2760
aaggatctgggcaccgcgacgtcatgggaagatgttgaacacttggtttggggagagctg2820
aatcaataccagcagtcatgcagcgtgggcgaaattaactga2862
<210>2
<211>953
<212>prt
<213>oryzasativa
<400>2
metalaalaglyasnglyargmetglualaalaleuglycysleuala
151015
alaleuproaspgluvalleucysalavalvalaspleuleupropro
202530
thraspvalglyargleualacysvalserservalmettyrileleu
354045
cysasnglugluproleutrpmetserlyscysleuservalglygly
505560
leuleuvaltyrargglysertrplyslysthralaleuserargleu
65707580
asnleucyssergluasnaspgluiletyrglnlysproarghisphe
859095
aspglypheasnsermethisleutyrargargtrptyrargcysphe
100105110
thrasnleuserserpheserpheaspasnglyhisvalgluarglys
115120125
aspaspleuserleuaspglnpheargalaglntyraspglylyscys
130135140
provalleuleuthrlysleualagluthrtrpproalaargthrlys
145150155160
trpthralaglnglnleuthrhisasptyrglygluvalprophearg
165170175
ileserglnargserproglnlysilelysmetlysleulysasptyr
180185190
valphetyrmetgluleuglnhisaspgluaspproleutyrilephe
195200205
aspasplyspheglygluseralaprothrleuleugluasptyrser
210215220
valprohisleupheglngluaspphephegluilemetasptyrasp
225230235240
glnargproalapheargtrpleuileileglyprogluargsergly
245250255
alasertrphisvalaspproglyleuthrseralatrpasnthrleu
260265270
leucysglyarglysargtrpalamettyrproproglyargvalpro
275280285
glyglyvalthrvalhisvalseraspgluaspglyaspvalaspile
290295300
gluthrprothrserleuglntrptrpleuaspiletyrproasnleu
305310315320
alagluhisglulysproleuglucysthrglnleuproglygluthr
325330335
ilephevalproserglytrptrphiscysvalleuasnleuaspmet
340345350
thrilealavalthrglnasnphevalasnglnserasnphelyshis
355360365
valcysleuaspmetalaproglytyrcyshislysglyvalcysarg
370375380
alaglyleuleualaalaproasplysserileargaspilegluasn
385390395400
leuproserilethrserargleuasnhisseraspmetalacyslys
405410415
glulysargleulyssersergluproileargthrserasnasnala
420425430
asnglncysseralapheglupheseraspvalhisgluasnleugly
435440445
aspglnvalphesertyraspileasppheleuserglnpheleuglu
450455460
lysglulysasphistyrserservaltrpserprothrasnserile
465470475480
glyglnargglualaargglutrpleuargargleutrpvalleulys
485490495
progluleuarggluleuiletrplysglyalacysleualaileasn
500505510
valasplystrptyrsercysleuglugluileseralacyshisser
515520525
leuproproprosergluaspglulysleuprovalglythrglyser
530535540
asnprovalpheilevalserglyasnvalilelysiletyralaglu
545550555560
glyglyleuglytyrserilehisglyleuglythrgluleugluphe
565570575
tyraspleuleuglnlysleuglyserproleuileasnhisvalpro
580585590
gluileilealaserglypheleuvaltyrleuaspglyvaltyrlys
595600605
thrvalprotrpaspglyasnglyileproaspvalleualalystyr
610615620
tyrserleugluvalsertyralaasnglyserpheproleuglyleu
625630635640
trpserlysglnleupheglyleuserasnserthraspalaproasp
645650655
argproilecysprotyrmetvalthrarglyscyslysglyaspile
660665670
phealaargileargasplysleuthrlysthraspvalleuasnleu
675680685
alaserserleuglyvalglnmetargasnilehisglnleuproleu
690695700
prohisvalgluhisileserlysserglyasngluaspilelysala
705710715720
lysgluasnserileseraspvalthrhisvalproproglutrplys
725730735
glnvalvalserthrleuaspargarglyslysserilelyslyshis
740745750
leuserasntrpglyglyserileproglnvalleuileglulysala
755760765
gluglutyrleuproaspaspileargpheleuilelysphevallys
770775780
aspaspaspglyaspservaltyrvalvalprosertrpilehisser
785790795800
aspilemetaspaspasnileleuilegluglythrthrgluprogly
805810815
thrserthraspcysilealavalgluaspleuasnlysmetaspala
820825830
ilehisileileasppheseraspleuserileglyaspproleucys
835840845
aspleuileproleuhisleuaspvalpheargglyaspileaspleu
850855860
leuargglnpheleuargsertyrglnleupropheleuargalaglu
865870875880
serasnlysaspiletyrlysserileglnasnserlyspheserarg
885890895
alasertyrargalametcystyrcysileleuhisgluaspasnval
900905910
leuglyalailepheserleutrplysaspleuglythralathrser
915920925
trpgluaspvalgluhisleuvaltrpglygluleuasnglntyrgln
930935940
glnsercysservalglygluileasn
945950
<210>3
<211>415
<212>dna
<213>人工序列(unknown)
<400>3
acatggcacctggttactgtcacaaaggagtttgccgtgctggcttacttgctgctccag60
acaaatctattagagatattgaaaatcttcctagtataacgagtagattgaaccactctg120
acatggcctgtaaggaaaaaagactgaaaagttcagagcctataagaacttcaaataatg180
caaatcagtgttctgcatttgagttctcagatgttcatgaaaacttgggggaccaagttt240
tttcgtatgatatagatttcttatcccaattccttgagaaagaaaaggatcactattctt300
ctgtctggagccctactaattcaattggccagagagaagcaagagaatggctacgtaggc360
tatgggttcttaaacctgaattgagagaactaatatggaagggtgcatgtctagc415
<210>4
<211>34
<212>dna
<213>人工序列(unknown)
<400>4
actagtggtaccacatggcacctggttactgtca34
<210>5
<211>33
<212>dna
<213>人工序列(unknown)
<400>5
gagctcggatccgctagacatgcacccttccat33
<210>6
<211>21
<212>dna
<213>人工序列(unknown)
<400>6
tgtatgccagtggtcgtacca21
<210>7
<211>19
<212>dna
<213>人工序列(unknown)
<400>7
ccagcaaggtcgagacgaa19
<210>8
<211>21
<212>dna
<213>人工序列(unknown)
<400>8
cgtgcgatgtgctactgcata21
<210>9
<211>20
<212>dna
<213>人工序列(unknown)
<400>9
gcgtcagttaatttcgccca20
- 还没有人留言评论。精彩留言会获得点赞!