表达人组织因子融合蛋白的重组痘苗病毒及其应用的制作方法
本发明涉及生物
技术领域:
,尤其涉及一种表达人组织因子融合蛋白的重组痘苗病毒及相关痘苗载体表达质粒及其应用。
背景技术:
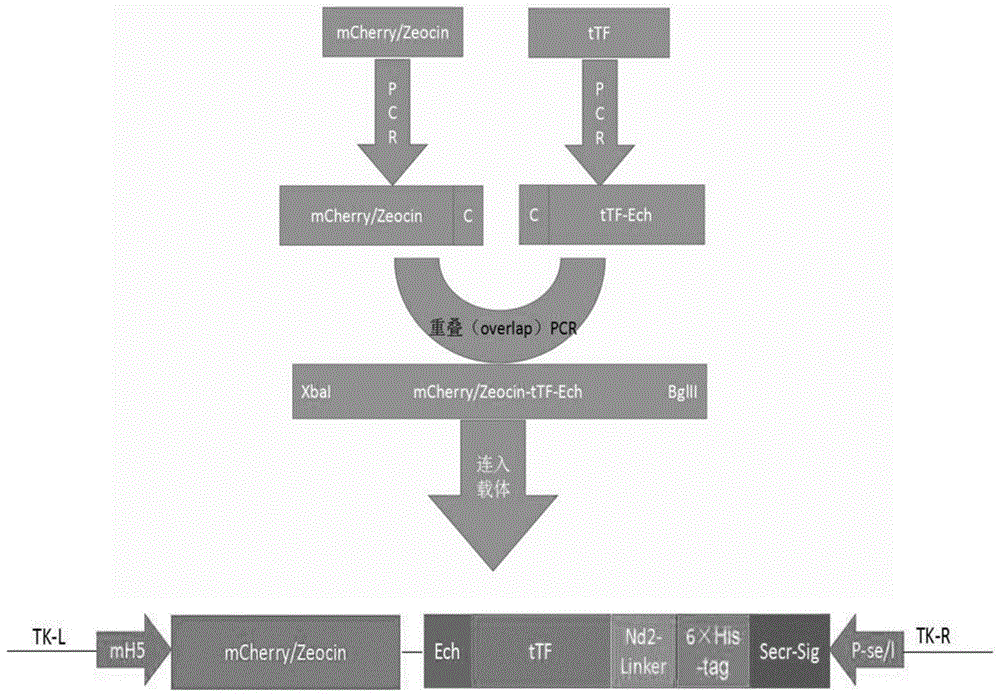
:当前,恶性肿瘤仍然是极大危害人类生命健康的严重疾病。随着分子生物学、免疫学及肿瘤生物学等基础研究的进步,肿瘤生物治疗异军突起,成为继外科手术,化学药物治疗(化疗)和放射治疗(放疗)等传统方法之后的肿瘤治疗“第四种模式”,日益引起重视、进展迅速,并酝酿突破。一些方案已获准临床应用,在某些类型的肿瘤(如黑色素瘤和肾细胞癌)已初见疗效。在肿瘤生物治疗策略中,肿瘤血管靶向治疗是一特别引人注目的方法。恶性肿瘤的发生发展涉及极为复杂的细胞信号传导通路和代谢途径的异常,针对任何单一环节的治疗干预都难以达到理想疗效,因为肿瘤细胞已进化出多种适应和代偿功能,往往足以抵消单一治疗干预,产生耐药性或放疗耐受性,最终使治疗归于失败。然而,任何类型的肿瘤,都有一共同的“软肋”或薄弱环节,那就是,相比于正常细胞,生长代谢更为旺盛的肿瘤细胞毫无例外都需要足够、通畅的血管供应丰富的血液营养物质和氧气,同时运走代谢废物。一旦破坏了肿瘤微血管或阻断了其血液供应,就等于扼住了肿瘤的“咽喉”,任何形态功能复杂的肿瘤细胞均无法继续生长,这就是肿瘤血管靶向治疗相比于其它治疗方法的独特之处。在肿瘤血管靶向治疗的策略中,迄今一个成功方案是设计促使肿瘤血管内凝血,造成肿瘤微血管内血栓形成和栓塞,从而达到切断肿瘤血管血流供应的目的,其原理是:组织因子(tissuefactor,tf)是生理性凝血系统的启动因子,采用原核系统表达融合有肿瘤靶向元件(抗体或多肽)的截短组织因子重组融合蛋白,该融合蛋白可特异性靶向结合肿瘤血管内皮细胞,并通过组织因子激活凝血系统,造成肿瘤血管内凝血和血栓形成,从而堵塞肿瘤血液供应,导致肿瘤的生长被抑制。迄今为止,诱导肿瘤血管内凝血的方案都是采用“注射原核表达的组织因子重组融合蛋白”的方式,然而,这种类型的肿瘤血管靶向治疗的方式存在一系列局限性。首先,生理状态下,组织因子系由哺乳动物体内真核细胞表达的蛋白产物,而原核表达系统(主要是大肠杆菌表达系统)表达蛋白时缺乏糖基化和翻译后修饰功能,现有研究证明,来自胎盘的天然组织因子其比活性高于原核重组表达的组织因子,并且,这种比活性的差异是由于天然组织因子具有糖基化等翻译后修饰,而原核重组表达的组织因子缺乏翻译后修饰所致。其次,不管是采用皮下注射还是静脉注射给药方式,组织因子融合蛋白进入机体循环系统后均遵循蛋白类药物代谢、排泄及衰减等药代动力学一般规律,其血药含量只会进行性减少而不可能自发再生,特别是进入系统循环的组织因子融合蛋白经体液“稀释”及血液内蛋白酶降解后,运抵肿瘤靶部位的含量将十分有限。最后,基于组织因子融合蛋白的肿瘤靶向治疗策略并不能破坏已有的肿瘤血管,理论上组织因子融合蛋白也不可能在所有肿瘤微血管内形成栓塞,这就为改进现有策略和如何基于不同作用机理达到协同增效目的提出了现实要求。有鉴于此,本发明另辟蹊径,设计采用重组痘苗病毒真核载体表达肿瘤血管靶向性组织因子融合蛋白。痘苗病毒(vacciniavirus)是迄今发现的最大、最复杂的病毒之一,它是预防天花的活疫苗病毒,具有大规模人群接种的历史而副反应发生率极低的良好生物安全记录。天花在全球根除后,痘苗病毒被用作基因工程真核表达载体,获得广泛研究应用。特别地,痘苗病毒载体具有天然的肿瘤靶向性。研究证明,将痘苗病毒静脉注射后,几乎所有的病毒都将聚集在肿瘤部位。如果将外源目的基因插入痘苗载体的tk基因区,导致tk基因的插入失活,则可进一步增强重组痘苗病毒肿瘤靶向性。痘苗病毒的这些特性使之成为肿瘤生物治疗的优秀载体。另一方面,业已证明,来源于锯鳞蝰蛇的毒液的多肽蛇毒锯鳞蝰素(echistatin,ech)具有高亲和力结合肿瘤血管内皮细胞表面受体整合素的能力,故在本发明中,ech多肽被融合进人组织因子融合蛋白中,用于将痘苗载体表达分泌的人组织因子融合蛋白靶向肿瘤血管,触发肿瘤血管内凝血以阻塞肿瘤血管内血流,切断肿瘤的血流供应,产生抗肿瘤治疗效能。技术实现要素:本发明的目的在于针对现有技术的不足,设计了一种属于真核表达系统的表达人组织因子融合蛋白(ttf-ech)的重组痘苗病毒及其应用。本发明的目的是通过以下技术方案来实现的:人组织因子融合蛋白基因(目的基因)被插入痘苗病毒tk基因区,由人工合成的痘苗病毒早/晚期启动子(p-se/l)驱动表达。进一步地,所述重组痘苗病毒表达的人组织因子融合蛋白依次包括以下元件:分泌信号肽、串联的组氨酸标签(6×his-tag)、nd2连接肽(nd2-linker)、截短的人组织因子(ttf1-218)、源于蛇毒锯鳞蝰素的血管靶向肽(ech),它们的核酸序列依次如seqidno.1、seqidno.2、seqidno.3、seqidno.4和seqidno.5所示。进一步地,所述人组织因子融合蛋白的氨基酸序列如seqidno.6所示。进一步地,所述表达人组织因子融合蛋白的重组痘苗病毒的母体病毒是痘苗病毒wr株。进一步地,所述由人工合成的痘苗病毒早/晚期启动子的核酸序列如seqidno.7所示。进一步地,还包括与人组织因子融合蛋白基因表达盒串联的可选择第一标志基因表达盒,所述可选择第一标志基因表达盒中,可选择标志基因为gpt基因,gpt基因由痘苗病毒p7.5早/晚期启动子驱动表达。p7.5启动子的核酸序列如seqidno.8所示,gpt基因的核酸序列如seqidno.9所示。进一步地,还包括与人组织因子融合蛋白基因表达盒串联的可选择第二标志基因表达盒,所述可选择第二标志基因表达盒中,可选择标志基因为mcherry红荧光蛋白与zeocin抗性蛋白融合基因mcherry-zeocin,具有红荧光和zeocin药物抗性双表型。可选择标志基因由痘苗病毒mh5启动子驱动表达。mh5启动子核酸序列如seqidno.10所示,mcherry-zeocin融合基因核酸序列如seqidno.11所示。进一步地,可选择第一标志基因表达盒的氨基酸序列如seqidno.12所示,可选择第二标志基因表达盒的氨基酸序列如seqidno.13所示。进一步地,所述重组痘苗病毒构建过程是基于重组痘苗载体表达质粒pcz-ttfech转染细胞后,再感染野型痘苗病毒wr株,通过细胞内同源重组机制而构建的。pcz-ttfech质粒的核酸全序列如seqidno.14所示。本发明还提供了一种上述任一重组痘苗病毒及相关元件在制备抗肿瘤药物中的应用。本发明的有益效果是:本发明重组痘苗病毒载体表达人组织因子融合蛋白,首先构建重组痘苗病毒载体表达质粒pcz-ttfech,采用该质粒转染vero细胞,然后用野型痘苗病毒wr株感染细胞,通过细胞内同源重组机制产生重组痘苗病毒。接着,通过与目的基因表达盒串联的可选择标志基因gpt的表达,以及霉酚酸、黄嘌呤和次黄嘌呤的药物选择性压力,配合红荧光表型和zeocin药物抗性,可方便地筛选出重组痘苗病毒。实验证明,无需如现有技术那样的耗时费力和复杂的原核表达及蛋白纯化过程,本发明重组痘苗病毒在感染细胞部位即可分泌表达人组织因子融合蛋白,且表达产物具有预期的生物活性,即该融合蛋白能够结合人脐静脉内皮细胞(该细胞如肿瘤血管内皮细胞那样,具有高水平表达整合素受体,可作为肿瘤血管内皮细胞的模拟物),并触发凝血反应。附图说明图1人组织因子融合蛋白基因的拼接图。图中,p-se/l为合成的痘苗病毒早/晚期启动子;secr-sig为分泌信号肽;6×his-tag为串联的组氨酸标签;nd2-linker为nd2连接肽;ttf为截短的人组织因子;mh5为修饰的痘苗病毒h5启动子;mcherry/zeocin为mcherry红荧光蛋白与zeocin抗性蛋白融合基因;图2重组痘苗载体表达质粒pcz-ttfech图谱。图例说明同图1。图3重组痘苗病毒表达ttf-ech融合蛋白的elisa检测结果柱状图。其中,1:细胞培养上清;2:细胞裂解物;3:总蛋白;4:野生型wr株感染vero细胞。图4重组痘苗病毒表达ttf-ech融合蛋白的westernblot检测结果图。采用兔抗人组织因子抗体,作westernblot检测,验证目的蛋白表达。其中,m:蛋白分子量标志;1:细胞裂解物;2:细胞裂解物与细胞培养上清混合物;3:细胞培养上清。图5重组痘苗病毒表达ttf-ech融合蛋白与脐静脉内皮细胞(huvec)结合试验—elisa检测结果图。图中,1:ttf-ech+huvec;2:ttf-ech+huvec未加一抗;3:ttf-ech+vero;4:ttf-ech+vero未加一抗。具体实施方式本发明产品表达人组织因子融合蛋白的重组痘苗病毒命名为vv-ttf-ech,其构成是以痘苗病毒wr株为母体,在该病毒的tk基因区插入人组织因子融合蛋白基因即ttf-ech融合基因,在该基因上游安置有驱动外源基因表达的人工合成的痘苗病毒早/晚期启动子即p-se/l启动子,作为优选,与目的基因表达盒串联的有用于重组痘苗病毒筛选的可选择第一标志基因,即:gpt基因,用于霉酚酸、黄嘌呤和次黄嘌呤药物压力筛选重组病毒,另外配合采用可选择第二标志基因,即:mcherry-zeocin融合基因,利用其红荧光表型和zeocin药物抗性表型辅助筛选重组痘苗病毒。本发明重组痘苗病毒载体表达质粒为pcz-ttfech,其结构图谱如图1和2所示。本发明采用经典的同源重组方法构建重组痘苗病毒。首先构建人组织因子融合基因,并克隆进痘苗表达载体质粒pcb,从而产生重组痘苗载体表达质粒pcz-ttfech,再以质粒pcz-ttfech转染vero细胞,然后用野型痘苗病毒wr株感染转染的细胞,通过标志基因gpt表达及霉酚酸、黄嘌呤和次黄嘌呤药物压力筛选和标志基因mcherry的红荧光表型,筛选出重组痘苗病毒。实施例1:人组织因子融合蛋白基因拼接和重组痘苗表达质粒pcz-ttfech的构建(1)pcr引物引物名称及序列h5-up(seqidno.15)tgtggagtctagaaaaaattgaaaataaatacaaagd8-dn(seqidno.16)gctacctgaatttaaattcagttttgtttttctcgcgaatatcgech(seqidno.17)acaaaactgaatttaaattcaggtagctggacccttgtgtgggtttctcgggcagsec(seqidno.18)agtcgacagatctgccaccatgtggtggcgcctgtggtggctgctgctgctgctgctg将截短的人组织因子(ttf1-218,序列如seqidno.4所示)构建至pcb质粒中,得到pcblac-ttf-ngr质粒,以pcblac-ttf-ngr质粒为模板,用引物ech和sec扩增出ttf-ech+bglii片段(seqidno.19)。两者都是由q5酶催化的pcr反应来扩增的,只是模板和引物不同。(2)一般pcr反应体系如下:成分加入量双蒸水补足至50μl5×q5反应缓冲液10μl10mmdntps1μl5×q5highgcenhancer10μl引物1(10μm)2.5μl引物2(10μm)2.5μl模板质粒1μlq5高保真dna聚合酶0.5μl反应过程:①98℃预变性30s,②98℃变性10s,③55℃退火30s,④72℃延伸50s,⑤再进入②步骤,重复30个循环,72℃延伸2min。取2μlpcr产物跑1%琼脂糖凝胶电泳,凝胶成像分析仪观察记录结果。(3)两条目的片段的融合用pcr柱式胶回收试剂盒对上述步骤中获得的两条目的片段进行纯化。切胶纯化过程如下:将剩余pcr产物全部跑1%琼脂糖凝胶电泳,紫外灯下将目的条带切下。①将带有目的片段的凝胶块转移至1.5ml离心管(离心管已称重)中,称重得出凝胶块的重量,加入胶块重量3-6倍的bufferb2,于50℃金属浴温浴至凝胶完全融化得到溶胶液。②将溶胶液转移至柱子中,8000×g离心30s.弃收集管中的滤液,将柱子套回2ml收集管内。③加500μlwashsolution(已用无水乙醇稀释)至柱子中,9000×g离心30min,倒掉收集管中的液体,将吸附柱放入同一个收集管中。④重复步骤3一次。⑤弃收集管中的滤液,将柱子套回2ml收集管内,9000×g离心1min以甩干柱子基质残余的液体。⑥把柱子装在一个干净的1.5ml离心管上,开盖金属浴中放置2min挥发残留酒精,在吸附膜中央加入15-40μl预热至60℃的elutionbuffer,放置1min,9000×g离心1min,洗脱dna。重叠(overlap)pcr及人组织因子融合蛋白基因的拼接流程见图1.第一步pcr反应体系如下:成分加入量双蒸水补足25μl5×q5反应缓冲液5μl10mmdntps0.5μl5×q5highgcenhancer5μlmcherry/zeocin片段(seqidno.11)1μlttf-ech+bglii片段1μlq5高保真dna聚合酶0.25μl反应过程:①98℃预变性30s,②98℃变性10s,③55℃退火30s,④72℃延伸1min15s,⑤再进入②步骤,重复10个循环,72℃延伸2min。收集pcr产物,第二步pcr反应体系如下:成分加入量双蒸水补足50μl5×q5反应缓冲液5μl10mmdntps0.5μl5×q5highgcenhancer5μlh5-up(10μm)2.5μlsec(10μm)2.5μl模板质粒(上述pcr产物)25μlq5高保真dna聚合酶0.25μl反应过程:①98℃预变性30s,②98℃变性10s,③55℃退火30s,④72℃延伸1min15s,⑤再进入②步骤,重复30个循环,72℃延伸2min。取2μlpcr产物跑1%琼脂糖凝胶电泳,凝胶成像分析仪观察记录结果。pcr产物切胶回收剩余pcr产物跑1%的琼脂糖凝胶电泳,在长波紫外灯(300-360nm)下切胶,参照生工切胶回收试剂盒说明书回收,步骤如下:①将带有目的片段的凝胶块转移至1.5ml离心管(离心管已称重)中,称重得出凝胶块的重量,加入胶块重量3-6倍的bufferb2,于50℃金属浴温浴至凝胶完全融化。②将溶胶液转移至柱子中,8000×g离心30s。弃收集管中的滤液,将柱子套回2ml收集管内。③用500μlwashsolution(已用无水乙醇稀释的)至柱子中,9000×g离心30min,倒掉收集管中的液体,将吸附柱放入同一个收集管中。④重复步骤3一次。⑤弃收集管中的滤液,将柱子套回2ml收集管内,9000×g离心1min以甩干柱子基质残余的液体。⑥把柱子装在一个干净的1.5ml离心管上,开盖金属浴中放置2min挥发残留酒精,在吸附膜中央加入15-40μl预热至60℃的elutionbuffer,放置1min,9000×g离心1min,洗脱dna,得到融合片段mcherry/zeocin-ttfech。(4)载体质粒与融合基因片段的连接利用限制性内切酶bglii、xbai酶切融合片段mcherry/zeocin-ttfech和质粒pcb。酶切融合片段mcherry/zeocin-ttfech产物反应如下:成分加入量双蒸水补足20μl10×缓冲液:2μlbglii酶1μlxbai酶1μl融合片段mcherry/zeocin-ttfech4μl质粒载体pcb的酶切反应体系:各组分混合均匀,低速离心,37℃酶切3h。通过t4连接酶将融合片段与质粒载体连接起来,反应体系如下:16℃反应3h。(5)融合片段mcherry-zeo-ttfech与质粒载体pcb连接后的转化反应①将感受态细胞ecolidh5α从-80℃冰箱取出置冰中融化。②超净台中取10μl连接产物加入100μl感受态细胞中,冰浴30min。③于42℃热激90s,然后冰浴2-3min。④加入900μl预热至37℃的lb培养基,放37℃摇床,200rpm,1h。⑤从摇床中取出转化物,3000rpm,离心5min,弃去900μl培养液,用剩余的100μl培养液重悬菌体,将之涂布在预热含氨苄青霉素100μg/ml和博来霉素50μg/ml的lbagar平板上,待干。⑥将lb平板倒置放入37℃恒温箱过夜,次日早晨观察结果。重组质粒pcz-ttfech经菌落pcr和dna测序验证确定序列正确无误。pcz-ttfech质粒图谱见图2,其中人组织因子融合蛋白依次包括以下元件:分泌信号肽、串联的组氨酸标签(6×his-tag)、nd2连接肽(nd2-linker)、截短的人组织因子(ttf1-218)、蛇毒锯鳞蝰素血管靶向肽(ech)。实施例2:表达人组织因子融合蛋白的重组痘苗病毒vv-ttfech的构建与筛选(1)重组痘苗病毒的包装在6孔板接种vero细胞,使其次日长至80%程度。取4μgpcz-ttfech质粒稀释于250μl无血清的dmem培养基中,作为a液,取10μl脂质体(biomiga产品)稀释于250μl无血清的dmem培养基中,作为b液,将a液和b液混匀后,室温下孵育20min,再加入300μl无血清dmem,混匀,混合液加入hanks液洗过2遍,37℃、5%co2培养箱中培养4h后,每孔加入滴度为2×106pfu/ml的野生型痘苗病毒wr株(购自atcc公司)10μl,37℃、5%co2培养箱中继续孵育2h后,每孔加入2ml含2%fcs的dmem培养基,37℃、5%co2培养箱中继续培养,48h后收集感染痘苗病毒的细胞,于-80℃和37℃反复冻融3次,保存于-80℃备用。(2)重组痘苗病毒盲筛在6孔板中接种vero细胞,待次日细胞生长至80%-90%成片时弃去培养基,用hanks液洗两遍,加入1ml上述步骤中产生的病毒液,37℃、5%co2培养箱中培养2h弃上清,用hanks液洗两遍,加入含1×gpt筛选液(初始浓度:400×次黄嘌呤10mg/ml,40×黄嘌呤10mg/ml,670×霉酚酸10mg/ml)及含200μg/mlzeocin的2%fcs的dmem培养基2ml。于荧光倒置显微镜下观察vero细胞红色荧光表达情况,待细胞完全病变后,收集病毒液。重复上述步骤一次。(3)重组痘苗病毒噬斑纯化在6孔板中接种vero细胞,待次日细胞生长至80-90%成片时弃去培养基,用hanks液洗两遍,加入经10倍系列稀释的病毒液200μl,37℃、5%co2培养箱中培养2h,期间每隔15min摇动6孔板以避免细胞变干,后弃上清,用hanks液洗两遍,加入含1×gpt筛选液及200μg/mlzeocin及2%fcs的dmem培养基2ml。于荧光倒置显微镜下观察vero细胞红色荧光表达情况,待有典型噬斑形成后用高贵级琼脂固定,预设45℃水浴备用。将浓度为2%的高贵级琼脂置微波炉融化后,与等体积含4%fcs的2×dmem培养液混合后,置45℃水浴待用。将上述配制好的高贵级琼脂/dmem混合物每孔2ml小心加入各孔,室温或4℃凝固后,挑取表达红色荧光处的单个噬斑于500μl2%fcs的dmem培养基中,于-80℃和37℃反复冻融3次。此后,每个噬斑感染一个孔,其余步骤同上,直至得到较为纯化的重组病毒,命名为vv-ttfech。实施例3:重组痘苗病毒表达目的蛋白的鉴定(1)重组痘苗病毒表达目的蛋白ttf-ech的elisa检测按照博士德公司产品人elisa检测试剂盒说明书操作如下:1)将1000pg/ml,500pg/ml,250pg/ml,125pg/ml,62.5pg/ml,31.3pg/ml,15.6pg/ml的标准品各100μl依次加入一排7孔中,还有1孔只加样品稀释液的作为零孔,做复孔。加待测样品原液100μl,做复孔。其中待测样品通过如下方法获得:在6孔板中接种vero细胞,待次日细胞生长至80%-90%时弃去培养基,用hanks液洗两遍,每孔加入滴度为2.65×107pfu/ml的重组病毒液10μl,48h后分别收集细胞培养上清、细胞裂解物、总蛋白作为待测样品。另外,野生型wr株感染vero细胞作为对照组。2)酶标板加上封板膜,37℃反应90分钟。3)反应后用自动洗板机吸去酶标板内的液体;或甩去酶标板内液体,再对着吸水纸拍几下。4)将准备好的生物素标记抗人组织因子抗体工作液按每孔100μl依次加入(tmb空白显色孔除外)。酶标板加上封板膜,37℃反应60分钟。5)1×洗涤缓冲液洗涤3次,每次浸泡1分钟左右(每孔洗液至少300μl)。6)将准备好的abc工作液按每孔100μl依次加入(tmb空白显色孔除外)。酶标板加上封板膜,37℃反应30分钟。7)1×洗涤缓冲液洗涤5次,每次浸泡1-2分钟左右(每孔洗液至少300μl)。8)按每孔90μl依次加入已在37℃平衡30分钟的tmb显色液,37℃避光反应25-30分钟。9)按每孔100μl依次加入tmb终止液,此时蓝色立转黄色。10)用酶标仪在450nm测定od值。实验证明,重组痘苗病毒感染细胞后可高水平表达ttf-ech融合蛋白,且分泌至细胞外的表达量大于滞留于细胞内表达量的一半(图3)。(2)重组痘苗病毒表达目的蛋白ttf-ech的westernblot检测在6孔板中接种vero细胞,待次日细胞生长至80%-90%时弃去培养基,用hanks液洗两遍,每孔加入滴度为2.65×107pfu/m的重组病毒液10μl,48h后分别收集细胞培养上清、细胞裂解物及二者混合物作为待测样品。westernblot检测实验按标准实验指南操作,将待测样品上样sds-page胶,常规电泳转膜后,5%脱脂牛奶4℃封闭过夜,用兔抗人组织因子抗体与5%脱脂牛奶1:100稀释后4℃摇床孵育6h。用tbst洗涤3次,每次常温摇床10min,接着用羊抗兔hrp酶标二抗与tbst1:10000稀释,常温摇床孵育1h。用tbst洗涤3次,每次常温摇床10min。最后,用显影液a、b各500μl加入暗盒,混合后将膜置于暗盒,避光反应5分钟。imagelab显影,结果如图4所示。实施例4:重组痘苗病毒表达目的蛋白的生物活性检测(1)ttf-ech融合蛋白与人脐静脉内皮细胞结合试验在6孔板中接种vero细胞,待次日细胞生长至80%-90%时弃去培养基,用hanks液洗两遍,每孔加入滴度为2.65×107pfu/ml的重组病毒液10μl,48h后收集细胞培养上清,takarabca蛋白定量检测试剂盒测定蛋白浓度,将蛋白作0、2、4、8、16、32、64、128倍稀释作为待测样品。96孔板接种整合素高表达的人脐静脉内皮细胞(huvec),作为肿瘤血管内皮细胞的模拟物,使其次日长至80-90%程度。4%福尔马林室温固定30min,3%过氧化氢消除内源性过氧化物酶,每步反应后0.05%pbst洗3次,每次2min,加入不同稀释度的待测样品100μl/孔,与人脐静脉内皮细胞及vero细胞共同孵育,每个稀释度设3个复孔。在4℃过夜,5%fcs室温封闭2h,1:100稀释抗人组织因子一抗37℃孵育2h,1:2500稀释hrp标记二抗37℃孵育1h,每步反应后0.05%pbst洗3次,每次2min,二抗孵育后0.05%pbst洗5次,每次3minel-abts显色试剂盒显色,405nm处测吸光值。以vero细胞及vero细胞未加一抗、huvec细胞未加一抗作为阴性对照(3个复孔)。实验证明,重组痘苗病毒感染细胞上清即使在稀释64倍后,其分泌至细胞上清中的表达产物仍能够显著结合huvec细胞,而阴性对照的vero细胞或不加一抗处理huvec细胞则未见结合反应(图5)(2)ttf-ech融合蛋白的凝血功能检测样本采集:静脉采血,置于含1/10体积0.109mol/l枸橼酸钠抗凝剂(1份抗凝剂+9份全血)的采血管中,轻轻颠倒摇匀,3000rpm(或2500g)离心15分钟,收集上层液(血浆)。每个ep管中加血浆50μl,分别加入系列浓度的培养上清来源的含融合蛋白ttf-ech(0、1.12、2.24、4.48、8.96μmol/l)的待检样品和终浓度为25mmol/l或0的氯化钙(cacl2)溶液,后按凝血仪要求操作。1)在测试杯内放入一颗磁珠,并用移液器吸取50ul样品到测试杯中;2)按照要求对样品进行预温3分钟;3)预温结束后,将测试杯移到测试通道,在测试杯内加入100ul已预温的pt试剂,加样时将按压帽压到底使仪器发出“嘀嘀”声启动测试;4)记录并整理结果。5)采用spss22.0统计软件进行统计分析,统计方法采用方差分析,当p<0.05认为有统计学差异。实验结果见表1,结果表明,重组痘苗病毒感染细胞上清处理能够显著缩短凝血时间,与阴性对照相比,差异有统计学显著性(p<0.05)。表1重组痘苗病毒表达ttf-ech融合蛋白的凝血活性测定结果将浓度为0.112、0.224、0.448、0.896μmol/l的融合蛋白ttf-ech与25mmol/l的cacl2混合,以0μmol/l的融合蛋白ttf-ech+25mmol/l的cacl2及0.896μmol/l的融合蛋白ttf-ech与0mmol/l的cacl2组作对照,共6个组,每组3个复孔,凝血仪测定凝血时间。a表示与1、2、6组相比差异有统计学意义(p<0.05)。序列表<110>杭州医学院<120>表达人组织因子融合蛋白的重组痘苗病毒及其应用<160>19<170>siposequencelisting1.0<210>1<211>63<212>dna<213>人工序列(artificialsequence)<400>1atgtggtggcgcctgtggtggctgctgctgctgctgctgctgctgtggcccatggtgtgg60gcc63<210>2<211>18<212>dna<213>人工序列(artificialsequence)<400>2caccatcatcatcatcat18<210>3<211>15<212>dna<213>人工序列(artificialsequence)<400>3ggatctaaaggacca15<210>4<211>654<212>dna<213>人工序列(artificialsequence)<400>4tcaggcactacaaatactgtggcagcatataatttaacttggaaatcaactaatttcaag60acaattttggagtgggaacccaaacccgtcaatcaagtctacactgttcaaataagcact120aagtcaggagattggaaaagcaaatgcttttacacaacagacacagagtgtgacctcacc180gacgagattgtgaaggatgtgaagcagacgtacttggcacgggtcttctcctacccggca240gggaatgtggagagcaccggttctgctggggagcctctgtatgagaactccccagagttc300acaccttacctggagacaaacctcggacagccaacaattcagagttttgaacaggtggga360acaaaagtgaatgtgaccgtagaagatgaacggactttagtcagaaggaacaacactttc420ctaagcctccgggatgtttttggcaaggacttaatttatacactttattattggaaatct480tcaagttcaggaaagaaaacagccaaaacaaacactaatgagtttttgattgatgtggat540aaaggagaaaactactgtttcagtgttcaagcagtgattccctcccgaacagttaaccgg600aagagtacagacagcccggtagagtgtatgggccaggagaaaggggaattcaga654<210>5<211>147<212>dna<213>人工序列(artificialsequence)<400>5gaatgcgaatcaggtccatgctgtcgtaactgcaagttccttaaggaaggtaccatctgt60aagcgcgcacgtggtgatgatctcgacgactactgcaacggtaagacctgtgactgcccg120agaaacccacacaagggtccagctacc147<210>6<211>300<212>prt<213>人工序列(artificialsequence)<400>6mettrptrpargleutrptrpleuleuleuleuleuleuleuleutrp151015prometvaltrpalaglyhishishishishishisglyserlysgly202530proserglythrthrasnthrvalalaalatyrasnleuthrtrplys354045serthrasnphelysthrileleuglutrpgluprolysprovalasn505560glnvaltyrthrvalglnileserthrlysserglyasptrplysser65707580lyscysphetyrthrthraspthrglucysaspleuthraspgluile859095vallysaspvallysglnthrtyrleualaargvalphesertyrpro100105110alaglyasnvalgluserthrglyseralaglygluproleutyrglu115120125asnserprogluphethrprotyrleugluthrasnleuglyglnpro130135140thrileglnserphegluglnvalglythrlysvalasnvalthrval145150155160gluaspgluargthrleuvalargargasnasnthrpheleuserleu165170175argaspvalpheglylysaspleuiletyrthrleutyrtyrtrplys180185190serserserserglylyslysthralalysthrasnthrasngluphe195200205leuileaspvalasplysglygluasntyrcyspheservalglnala210215220valileproserargthrvalasnarglysserthraspserproval225230235240glucysmetglyglnglulysglyglupheargglucysglusergly245250255procyscysargasncyslyspheleulysgluglythrilecyslys260265270argalaargglyaspaspleuaspasptyrcysasnglylysthrcys275280285aspcysproargasnprohislysglyproalathr290295300<210>7<211>53<212>dna<213>人工序列(artificialsequence)<400>7aagcttaaaaattgaaattttattttttttttttggaatataaataagctcga53<210>8<211>186<212>dna<213>人工序列(artificialsequence)<400>8cactaattccaaacccacccgctttttatagtaagtttttcacccataaataataaatac60aataattaatttctcgtaaaagtagaaaatatattctaatttattgcacggtaaggaagt120agatcataacgatctctataatctcgcgcaacctattttcccctcgaacactttttaagc180cgtaga186<210>9<211>459<212>dna<213>人工序列(artificialsequence)<400>9atgagcgaaaaatacatcgtcacctgggacatgttgcagatccatgcacgtaaactcgca60agccgactgatgccttctgaacaatggaaaggcattattgccgtaagccgtggcggtctg120gtaccgggtgcgttactggcgcgtgaactgggtattcgtcatgtcgataccgtttgtatt180tccagctacgatcacgacaaccagcgcgagcttaaagtgctgaaacgcgcagaaggcgat240ggcgaaggcttcatcgttattgatgacctggtggataccggtggtactgcggttgcgatt300cgtgaaatgtatccaaaagcgcactttgtcaccatcttcgcaaaaccggctggtcgtccg360ctggttgatgactatgttgttgatatcccgcaagatacctggattgaacagccgtgggat420atgggcgtcgtattcgtcccgccaatctccggtcgctaa459<210>10<211>71<212>dna<213>人工序列(artificialsequence)<400>10aaaaattgaaaataaatacaaaggttcttgagggttgtgttaaattgaaagcgagaaata60atcataaataa71<210>11<211>1122<212>dna<213>人工序列(artificialsequence)<400>11atggatgtagaagcttggttaggagctagagtaccattagtagaaactgtgagcaagggc60gaggaggataacatggccatcatcaaggagttcatgcgcttcaaggtgcacatggagggc120tccgtgaacggccacgagttcgagatcgagggcgagggcgagggccgcccctacgagggc180acccagaccgccaagctgaaggtgaccaagggtggccccctgcccttcgcctgggacatc240ctgtcccctcagttcatgtacggctccaaggcctacgtgaagcaccccgccgacatcccc300gactacttgaagctgtccttccccgagggcttcaagtgggagcgcgtgatgaacttcgag360gacggcggcgtggtgaccgtgacccaggactcctccctgcaggacggcgagttcatctac420aaggtgaagctgcgcggcaccaacttcccctccgacggccccgtaatgcagaagaagacc480atgggctgggaggcctcctccgagcggatgtaccccgaggacggcgccctgaagggcgag540atcaagcagaggctgaagctgaaggacggcggccactacgacgctgaggtcaagaccacc600tacaaggccaagaagcccgtgcagctgcccggcgcctacaacgtcaacatcaagttggac660atcacctcccacaacgaggactacaccatcgtggaacagtacgaacgcgccgagggccgc720cactccaccggcggcatggacgagctgtacaaggccaagttgaccagtgccgttccggtg780ctcaccgcgcgcgacgtcgccggagcggtcgagttctggaccgaccggctcgggttctcc840cgggacttcgtggaggacgacttcgccggtgtggtccgggacgacgtgaccctgttcatc900agcgcggtccaggaccaggtggtgccggacaacaccctggcctgggtgtgggtgcgcggc960ctggacgagctgtacgccgagtggtcggaggtcgtgtccacgaacttccgggacgcctcc1020gggccggccatgaccgagatcggcgagcagccgtgggggcgggagttcgccctgcgcgac1080ccggccggcaactgcgtgcacttcgtggccgaggagcaggac1122<210>12<211>152<212>prt<213>人工序列(artificialsequence)<400>12metserglulystyrilevalthrtrpaspmetleuglnilehisala151015arglysleualaserargleumetprosergluglntrplysglyile202530ilealavalserargglyglyleuvalproglyalaleuleualaarg354045gluleuglyilearghisvalaspthrvalcysilesersertyrasp505560hisaspasnglnarggluleulysvalleulysargalagluglyasp65707580glygluglypheilevalileaspaspleuvalaspthrglyglythr859095alavalalaileargglumettyrprolysalahisphevalthrile100105110phealalysproalaglyargproleuvalaspasptyrvalvalasp115120125ileproglnaspthrtrpilegluglnprotrpaspmetglyvalval130135140phevalproproileserglyarg145150<210>13<211>374<212>prt<213>人工序列(artificialsequence)<400>13metaspvalglualatrpleuglyalaargvalproleuvalgluthr151015valserlysglyglugluaspasnmetalaileilelysgluphemet202530argphelysvalhismetgluglyservalasnglyhisglupheglu354045ilegluglygluglygluglyargprotyrgluglythrglnthrala505560lysleulysvalthrlysglyglyproleuprophealatrpaspile65707580leuserproglnphemettyrglyserlysalatyrvallyshispro859095alaaspileproasptyrleulysleuserpheprogluglyphelys100105110trpgluargvalmetasnphegluaspglyglyvalvalthrvalthr115120125glnaspserserleuglnaspglyglupheiletyrlysvallysleu130135140argglythrasnpheproseraspglyprovalmetglnlyslysthr145150155160metglytrpglualasersergluargmettyrprogluaspglyala165170175leulysglygluilelysglnargleulysleulysaspglyglyhis180185190tyraspalagluvallysthrthrtyrlysalalyslysprovalgln195200205leuproglyalatyrasnvalasnilelysleuaspilethrserhis210215220asngluasptyrthrilevalgluglntyrgluargalagluglyarg225230235240hisserthrglyglymetaspgluleutyrlysalalysleuthrser245250255alavalprovalleuthralaargaspvalalaglyalavalgluphe260265270trpthraspargleuglypheserargaspphevalgluaspaspphe275280285alaglyvalvalargaspaspvalthrleupheileseralavalgln290295300aspglnvalvalproaspasnthrleualatrpvaltrpvalarggly305310315320leuaspgluleutyralaglutrpsergluvalvalserthrasnphe325330335argaspalaserglyproalametthrgluileglygluglnprotrp340345350glyarggluphealaleuargaspproalaglyasncysvalhisphe355360365valalaglugluglnasp370<210>14<211>7162<212>dna<213>人工序列(artificialsequence)<400>14cgcgcgtaatacgactcactatagggcgaattggagctctttttatctgcgcggttaacc60gcctttttatccatcaggtgatctgtttttattgtggagtctagaaaaaattgaaaataa120atacaaaggttcttgagggttgtgttaaattgaaagcgagaaataatcataaataagctg180cagcacgtgttgacaattaatcatcggcatagtatatcggcatagtataatacgactcac240tataggagggccaccccaccatggatgtagaagcttggttaggagctagagtaccattag300tagaaactgtgagcaagggcgaggaggataacatggccatcatcaaggagttcatgcgct360tcaaggtgcacatggagggctccgtgaacggccacgagttcgagatcgagggcgagggcg420agggccgcccctacgagggcacccagaccgccaagctgaaggtgaccaagggtggccccc480tgcccttcgcctgggacatcctgtcccctcagttcatgtacggctccaaggcctacgtga540agcaccccgccgacatccccgactacttgaagctgtccttccccgagggcttcaagtggg600agcgcgtgatgaacttcgaggacggcggcgtggtgaccgtgacccaggactcctccctgc660aggacggcgagttcatctacaaggtgaagctgcgcggcaccaacttcccctccgacggcc720ccgtaatgcagaagaagaccatgggctgggaggcctcctccgagcggatgtaccccgagg780acggcgccctgaagggcgagatcaagcagaggctgaagctgaaggacggcggccactacg840acgctgaggtcaagaccacctacaaggccaagaagcccgtgcagctgcccggcgcctaca900acgtcaacatcaagttggacatcacctcccacaacgaggactacaccatcgtggaacagt960acgaacgcgccgagggccgccactccaccggcggcatggacgagctgtacaaggccaagt1020tgaccagtgccgttccggtgctcaccgcgcgcgacgtcgccggagcggtcgagttctgga1080ccgaccggctcgggttctcccgggacttcgtggaggacgacttcgccggtgtggtccggg1140acgacgtgaccctgttcatcagcgcggtccaggaccaggtggtgccggacaacaccctgg1200cctgggtgtgggtgcgcggcctggacgagctgtacgccgagtggtcggaggtcgtgtcca1260cgaacttccgggacgcctccgggccggccatgaccgagatcggcgagcagccgtgggggc1320gggagttcgccctgcgcgacccggccggcaactgcgtgcacttcgtggccgaggagcagg1380accaaaaatatatcgaagagaataaaacattcgcaattattgccatagtattcgtgttta1440tacttaccgctattctcttttttatgagtcgacgatattcgcgagaaaaacaaaactgaa1500tttaaattcaggtagctggacccttgtgtgggtttctcgggcagtcacaggtcttaccgt1560tgcagtagtcgtcgagatcatcaccacgtgcgcgcttacagatggtaccttccttaagga1620acttgcagttacgacagcatggacctgattcgcattctctgaattcccctttctcctggc1680ccatacactctaccgggctgtctgtactcttccggttaactgttcgggagggaatcactg1740cttgaacactgaaacagtagttttctcctttatccacatcaatcaaaaactcattagtgt1800ttgttttggctgttttctttcctgaacttgaagatttccaataataaagtgtataaatta1860agtccttgccaaaaacatcccggaggcttaggaaagtgttgttccttctgactaaagtcc1920gttcatcttctacggtcacattcacttttgttcccacctgttcaaaactctgaattgttg1980gctgtccgaggtttgtctccaggtaaggtgtgaactctggggagttctcatacagaggct2040ccccagcagaaccggtgctctccacattccctgccgggtaggagaagacccgtgccaagt2100acgtctgcttcacatccttcacaatctcgtcggtgaggtcacactctgtgtctgttgtgt2160aaaagcatttgcttttccaatctcctgacttagtgcttatttgaacagtgtagacttgat2220tgacgggtttgggttcccactccaaaattgtcttgaaattagttgatttccaagttaaat2280tatatgctgccacagtatttgtagtgcctgatggtcctttagatccatgatgatgatgat2340ggtgtccggcccacaccatgggccacagcagcagcagcagcagcagcagccaccacaggc2400gccaccacatggtggcagatctgtcgacttcgagcttatttatattccaaaaaaaaaaaa2460taaaatttcaatttttaagctttcactaattccaaacccacccgctttttatagtaagtt2520tttcacccataaataataaatacaataattaatttctcgtaaaagtagaaaatatattct2580aatttattgcacggtaaggaagtagatcataacgatctctataatctcgcgcaacctatt2640ttcccctcgaacactttttaagccgtagataaacaggctgggacacttcacatgagcgaa2700aaatacatcgtcacctgggacatgttgcagatccatgcacgtaaactcgcaagccgactg2760atgccttctgaacaatggaaaggcattattgccgtaagccgtggcggtctggtaccgggt2820gcgttactggcgcgtgaactgggtattcgtcatgtcgataccgtttgtatttccagctac2880gatcacgacaaccagcgcgagcttaaagtgctgaaacgcgcagaaggcgatggcgaaggc2940ttcatcgttattgatgacctggtggataccggtggtactgcggttgcgattcgtgaaatg3000tatccaaaagcgcactttgtcaccatcttcgcaaaaccggctggtcgtccgctggttgat3060gactatgttgttgatatcccgcaagatacctggattgaacagccgtgggatatgggcgtc3120gtattcgtcccgccaatctccggtcgctaatcttttcaacgcctggcactgccgggcgtt3180gttctttttaacttcaggcgggttacaatagtttccagtaagtattctggaggctgcatc3240catgacacaggcaaacctgcggatcccagcttttgttccctttagtgagggttaattgcg3300cgcagttatagtagccgcactcgatgggacatttcaacgtaaaccgtttaataatatttt3360gaatcttattccattatctgaaatggtggtaaaactaactgctgtgtgtatgaaatgctt3420taaggaggcttccttttctaaacgattgggtgaggaaaccgagatagaaataataggagg3480taatgatatgtatcaatcggtgtgtagaaagtgttacatcgactcataatattatatttt3540ttatctaaaaaactaaaaataaacattgattaaattttaatataatacttaaaaatggat3600gttgtgtcgttagataaaccgtttatgtattttgaggaaattgataatgagttagattac3660gaaccagaaagtgcaaatgaggtcgcaaaaaaactgccgtatcaaggacagttaaaacta3720ttactaggagaattattttttcttagtaagttacagcgacacggtatattagatggtgcc3780accgtagtgtatataggatctgctcccggtacacatatacgttatttgagagatcatttc3840tataatttaggagtgatcatcaaatggatgctaattgacggccgccatcatgatcctatt3900ttaaatggattgcgtgatgtgactctagtgactcggttcgttgatgaggaatatctacga3960tccatcaaaaaacaactgcatccttctaagattattttaatttctgatgtgagatccaaa4020cgaggaggaaatgaacctagtacggcggatttactaagtaattacgctctacaaaatgtc4080atgattagtattttaaaccccgtggcgtctagtcttaaatggagatgcccgtttccagat4140caatggatcaaggacttttatatcccacacggtaataaaatgttacaaccttttgctcct4200tcatattcagctgaaatgagattattaagtatttataccggtgagaacatgagactgact4260cgggccgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaat4320cgacgctcaagtcagaggtggcgaaacccgacaggactataaagataccaggcgtttccc4380cctggaagctccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctgtcc4440gcctttctcccttcgggaagcgtggcgctttctcaatgctcacgctgtaggtatctcagt4500tcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgac4560cgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcg4620ccactggcagcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctaca4680gagttcttgaagtggtggcctaactacggctacactagaaggacagtatttggtatctgc4740gctctgctgaagccagttaccttcggaaaaagagttggtagctcttgatccggcaaacaa4800accaccgctggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaaaaa4860ggatctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaaac4920tcacgttaagggattttggtcatgagattatcaaaaaggatcttcacctagatcctttta4980aattaaaaatgaagttttaaatcaatctaaagtatatatgagtaaacttggtctgacagt5040taccaatgcttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccata5100gttgcctgactccccgtcgtgtagataactacgatacgggagggcttaccatctggcccc5160agtgctgcaatgataccgcgagacccacgctcaccggctccagatttatcagcaataaac5220cagccagccggaagggccgagcgcagaagtggtcctgcaactttatccgcctccatccag5280tctattaattgttgccgggaagctagagtaagtagttcgccagttaatagtttgcgcaac5340gttgttgccattgctgcaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattc5400agctccggttcccaacgatcaaggcgagttacatgatcccccatgttgtgcaaaaaagcg5460gttagctccttcggtcctccgatcgttgtcagaagtaagttggccgcagtgttatcactc5520atggttatggcagcactgcataattctcttactgtcatgccatccgtaagatgcttttct5580gtgactggtgagtactcaaccaagtcattctgagaatagtgtatgcggcgaccgagttgc5640tcttgcccggcgtcaacacgggataataccgcgccacatagcagaactttaaaagtgctc5700atcattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctgttgagatcc5760agttcgatgtaacccactcgtgcacccaactgatcttcagcatcttttactttcaccagc5820gtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataagggcgaca5880cggaaatgttgaatactcatactcttcctttttcaatattattgaagcatttatcagggt5940tattgtctcatgagcggatacatatttgaatgtatttagaaaaataaacaaataggggtt6000ccgcgcacatttccccgaaaagtgccacctgacgtctaagaaaccattattatcatgaca6060ttaacctataaaaataggcgtatcacgaggccctttcgtcttcgaataaatacctgtgac6120ggaagatcacttcgcagaataaataaatcctggtgtccctgttgataccgggaagccctg6180ggccaacttttggcgaaaatgagacgttgatcggcacgtaagaggttccaactttcacca6240taatgaaataagatcactaccgggcgtattttttgagttatcgagattttcaggagctaa6300ggaagctaaaatggagaaaaaaatcactggatataccaccgttgatatatcccaatggca6360tcgtaaagaacattttgaggcatttcagtcagttgctcaatgtacctataaccagaccgt6420tcagagcttttgcgatcaataaatggatcacaaccagtatctcttaacgatgttcttcgc6480agatgatgattcattttttaagtatttggctagtcaagatgatgaatcttcattatctga6540tatattgcaaatcactcaatatgtagctagactttctgttattattattgatccaatcaa6600aaaataaattagaagccgtgggtcattgttatgaatctctttcagaggaatacagacaat6660tgacaaaattcacagactttcaagattttaaaaaactgtttaacaaggtccctattgtta6720cagatggaagggtcaaacttaataaaggatatttgttcgactttgtgattagtttgatgc6780gattcaaaaaagaatcctctctagctaccaccgcaatagatcctgttagatacatagatc6840ctcgtcgcaatatcgcattttctaacgtgatggatatattaaagtcgaataaagtgaaca6900ataattaattctttattgtcatcatgaacggcggacatattcagttgataatcggcccca6960tgttttcaggtaaaagtacagaattaattagacgagttagacgttatcaaatagctcaat7020ataaatgcgtgactataaaatattctaacgataatagatacggaacgggactatggacgc7080atgataagaataattttgaagcattggaagcaactaaactatgtgatctcttggaatcaa7140ttacagatttctccgtgatagg7162<210>15<211>36<212>dna<213>人工序列(artificialsequence)<400>15tgtggagtctagaaaaaattgaaaataaatacaaag36<210>16<211>44<212>dna<213>人工序列(artificialsequence)<400>16gctacctgaatttaaattcagttttgtttttctcgcgaatatcg44<210>17<211>55<212>dna<213>人工序列(artificialsequence)<400>17acaaaactgaatttaaattcaggtagctggacccttgtgtgggtttctcgggcag55<210>18<211>58<212>dna<213>人工序列(artificialsequence)<400>18agtcgacagatctgccaccatgtggtggcgcctgtggtggctgctgctgctgctgctg58<210>19<211>801<212>dna<213>人工序列(artificialsequence)<400>19tcaggcactacaaatactgtggcagcatataatttaacttggaaatcaactaatttcaag60acaattttggagtgggaacccaaacccgtcaatcaagtctacactgttcaaataagcact120aagtcaggagattggaaaagcaaatgcttttacacaacagacacagagtgtgacctcacc180gacgagattgtgaaggatgtgaagcagacgtacttggcacgggtcttctcctacccggca240gggaatgtggagagcaccggttctgctggggagcctctgtatgagaactccccagagttc300acaccttacctggagacaaacctcggacagccaacaattcagagttttgaacaggtggga360acaaaagtgaatgtgaccgtagaagatgaacggactttagtcagaaggaacaacactttc420ctaagcctccgggatgtttttggcaaggacttaatttatacactttattattggaaatct480tcaagttcaggaaagaaaacagccaaaacaaacactaatgagtttttgattgatgtggat540aaaggagaaaactactgtttcagtgttcaagcagtgattccctcccgaacagttaaccgg600aagagtacagacagcccggtagagtgtatgggccaggagaaaggggaattcagagaatgc660gaatcaggtccatgctgtcgtaactgcaagttccttaaggaaggtaccatctgtaagcgc720gcacgtggtgatgatctcgacgactactgcaacggtaagacctgtgactgcccgagaaac780ccacacaagggtccagctacc801当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1