制备双特异性抗体的方法与流程
1.本发明涉及生物技术领域;具体地说,本发明涉及制备双特异性抗体的新方法及其应用。
背景技术:
2.重组单克隆抗体(mab)能与抗原高效、高特异性结合,为生物医学和生物技术领域提供了令人兴奋的机会。许多治疗性单克隆抗体已成功用于治疗多种疾病,包括癌症、免疫疾病和感染。此外,靶向一种以上抗原的双特异性抗体(bsabs)已显示出极大的潜力,将最大程度提高抗体治疗的益处。
3.抗体是识别并特异性结合抗原的蛋白质(通常称为免疫球蛋白,immunologlobulin,简写为ig)。在包括人和小鼠在内的大多数哺乳动物中,免疫球蛋白由两条相同的重链和两条相同的轻链组成。每条重链和轻链可分为两部分:恒定区(c)和可变区(v)。基于抗体重链恒定区结构的不同,免疫球蛋白可以分为五个类别:iga、igd、ige、igg和igm。每个类别都可以具有kappa(κ)或lambda(λ)轻链。基于重链结构,人igg可以分为四个亚类:igg1、igg2、igg3和igg4。全长igg包含两个相同的重链和两个相同的轻链。重链的氨基末端(n端)区域是可变区(vh),其余部分是重链恒定区(ch)。人igg的重链恒定区由三个结构域ch1、ch2和ch3组成。ch1和ch2结构域之间的区域称为铰链区。轻链在氨基末端的一半是可变区(vl)结构域,而轻链在羧基末端(c端)的一半是恒定区(cl)结构域。重链和轻链的可变区和恒定区都在结构上折叠成称为结构域的功能单元。每个重链通过二硫键和非共价相互作用与轻链结合形成异二聚体。两个轻链-重链异二聚体通过两条重链的铰链区中的二硫键连接,形成复杂的y形抗体(图1)。木瓜蛋白酶消化igg(分子量为150kda)产生保留抗原结合活性的两个相同片段,称为fab片段(分子量为45kda),以及一个可结晶片段(fc片段,分子量为50kda)。胃蛋白酶消化igg会产生一个称为f(ab’)2的片段(分子量为100kda),该片段由两个fab片段组成(图2)。y形抗体的两个臂也称为fab臂。fab臂由重链可变区和ch1结构域以及与其配对的轻链组成。
4.igg1、igg2、igg3和igg4在铰链区域内显示最大的序列多样性(图3),它们的链间二硫键结构,显示许多相似之处和不同之处。两条重链通过不同数量的链间二硫键在铰链区连接:igg1和igg4为2个,igg2为4个,igg3为11个链间二硫键。igg1的轻链通过kappa和lambda链的cl结构域c端的半胱氨酸(κ轻链中214位的半胱氨酸[eu编号],λ轻链中214位的半胱氨酸[kabat编号])和重链ch1结构域c端的半胱氨酸(重链中220位的半胱氨酸[eu编号])之间的链间二硫键与重链连接。与igg1不同,igg2、igg3或igg4的轻链通过kappa和lambda链中cl结构域c端的半胱氨酸与重链中ch1结构域n端的半胱氨酸(重链中131位的半胱氨酸[eu编号])之间的链间二硫键与重链连接(图1,a和b)。尽管在igg1和其他亚型(igg2、igg3、igg4)之间,半胱氨酸在氨基酸序列中的位置不同,但它们的空间位置相似以形成链间二硫键。轻链和重链之间的二硫键对于fab的稳定性很重要。关于免疫球蛋白的结构和功能综述可参考immunology(janis kuby,w.h.freeman and company,new york,
1997)。
[0005]
尽管抗体是具有两条相同重链和轻链的对称结构免疫球蛋白,但双特异性抗体可以是非对称的结构,具有两条不同的重链,每条重链均与一条不同的轻链结合。因此,抗体是二价和单特异性的,能同时与两个相同的抗原进行专一性结合,但是双特异性的抗体是单价和双特异性的,能结合两种不同的抗原或表位。
[0006]
为有效产生完整igg形式的双特异性抗体,两个主要的难点有待解决。一个是如何确保针对表位甲的重链甲仅与针对表位乙的重链乙配对(重链甲乙间的异二聚化),并且抗体两个相同重链间(甲与甲,乙与乙)的同二聚化被阻止。另一个难点是,如何确保重链甲只与自己的轻链甲配对,不与其他表位的轻链乙配对;同时重链乙只与自己的轻链乙配对,不与轻链甲配对。产生双特异性抗体的一些方法可参考综述文章,brinkmann u.&kontermann r.e.,the making of bispecific antibodies,mabs,9(2017),182-212)。
[0007]
通常使用所谓的“旋钮入孔”(knobs-into-holes,kih)方法来产生抗体重链异二聚化。在这种方法中,重链甲的ch3结构域中有t366w“旋钮”(knobs)突变,而重链乙的ch3结构域中有t366s/l368a/y407v“孔”(holes)突变[eu编号]。这些突变在甲、乙两条重链的ch3结构域产生不对称的空间互补结构,从而通过疏水相互作用促进重链异二聚化。
[0008]
另一种方法利用静电相互作用,通过静电吸引促进重链异二聚化,并通过静电排斥来避免相同重链间的同二聚化。在抗体两个重链的ch3-ch3结构域,k409和d399之间有电荷相互作用。在这种方法中,重链甲的ch3结构域中有k409d、k392d突变,重链乙的ch3结构域中有d399k、e356k突变[eu编号]。这些氨基酸突变通过静电相互作用能促进抗体重链甲、乙的异二聚化,并抑止相同重链间的同二聚化。
[0009]
虽然通过ch3结构域中的氨基酸突变可以促进抗体重链异二聚化,并抑止相同重链间的同二聚化,但这些方法无法避免轻链错配问题。当两个抗体重链形成异二聚体时,两条不同的轻链可以与重链产生四种不同的组合,其中只有一种是正确配对的双特异性抗体,其他三种是错配的重链和轻链。
[0010]
避免轻链错配问题的直接方法是使用共同轻链。这需要大量的筛选工作去寻找可与两条不同的重链配对并保持其对两种不同抗原高亲和力的共同轻链。但是,这种方法可能并非对所有抗体都可行。
[0011]
基于理性化设计,可能开发其他方法来实现重链和轻链的正确配对。其主要原理是改变一个fab臂中的重链和轻链结构域,同时使另一个fab臂保持野生型不变,这可以促进轻链与自己的重链配对。
[0012]
通过将抗体重链和轻链之间对应的结构域进行互换可以促进重链和轻链的正确配对(crossmab技术),通过这些互换可以促进野生型的轻链与野生型重链配对,互换的轻链与相应的互换重链配对。但是重链和轻链的结构域互换可能不利于双特异性抗体的结构稳定性。
[0013]
解决轻链错配问题的另一种方法是基于结构设计改变轻链和重链的结构域。通过改变一个fab臂的可变域(vh-vl)和恒定域(ch1-cl)之间的相互作用,促进轻链与其自身的重链相互结合。但是改变fab的可变域可能对其与抗原的结合有较大的影响。
[0014]
通过用工程改造的二硫键取代ch1-cl结构域中的天然链间二硫键,可以促进轻链与其自身重链的配对。结构建模在ch1-cl结构域中预测有三组位置,通过引入一对半胱氨
酸可能形成新的链间二硫键。比较这三组半胱氨酸突变表明,其中一组可以高效促进轻链与其自身重链的配对,其正确配对率达到98%。在这组突变中,重链(h)126位的苯丙氨酸和轻链(l)121位的丝氨酸[eu编号]被半胱氨酸取代(h:f126c/l:s121c;duetmab技术)。除了这组半胱氨酸突变,目前还不知道是否有其它半胱氨酸突变能形成新的链间二硫键,促进轻链与其自身重链的配对。
技术实现要素:
[0015]
本发明的目的在于提供一种全新的双特异性抗体的制备方法,采用本发明方法获得的双特异性抗体的重链和轻链正确配对率将显著提高。
[0016]
在第一方面,本发明提供了一种提高双特异性抗体制备过程中重链和轻链正确配对率的方法,所述方法包括以下步骤:
[0017]
在一个fab臂的ch1-cl结构域,通过氨基酸取代消除天然的链间二硫键,同时通过氨基酸取代形成工程改造的链间二硫键。
[0018]
在优选的实施方式中,所述双特异性抗体衍生自igg、iga、igd、ige和igm中的任一种;优选地,衍生自igg分子;更优选地,衍生自人或非人,例如灵长类动物或啮齿动物igg分子。
[0019]
在优选的实施方式中,所述双特异性抗体衍生自人igg1、igg2、igg3、igg4。
[0020]
在优选的实施方式中,利用非半胱氨酸的氨基酸取代半胱氨酸(例如半胱氨酸对突变为缬氨酸或丝氨酸,igg1重链c220v,igg2、igg3或igg4重链c131s,kappa轻链c214v[eu编号],lambda轻链c214v[kabat编号])以消除天然的链间二硫键,利用半胱氨酸对取代一对非半胱氨酸的氨基酸以形成工程改造的链间二硫键。
[0021]
在优选的实施方式中,形成工程改造的链间二硫键的氨基酸主要位于igg1重链结构域的以下三个区域:f126-t135、g166-t187、k218-s219;kappa cl结构域的以下四个区域:s114-s121、n158-t164、t172-t180、f209-e213。
[0022]
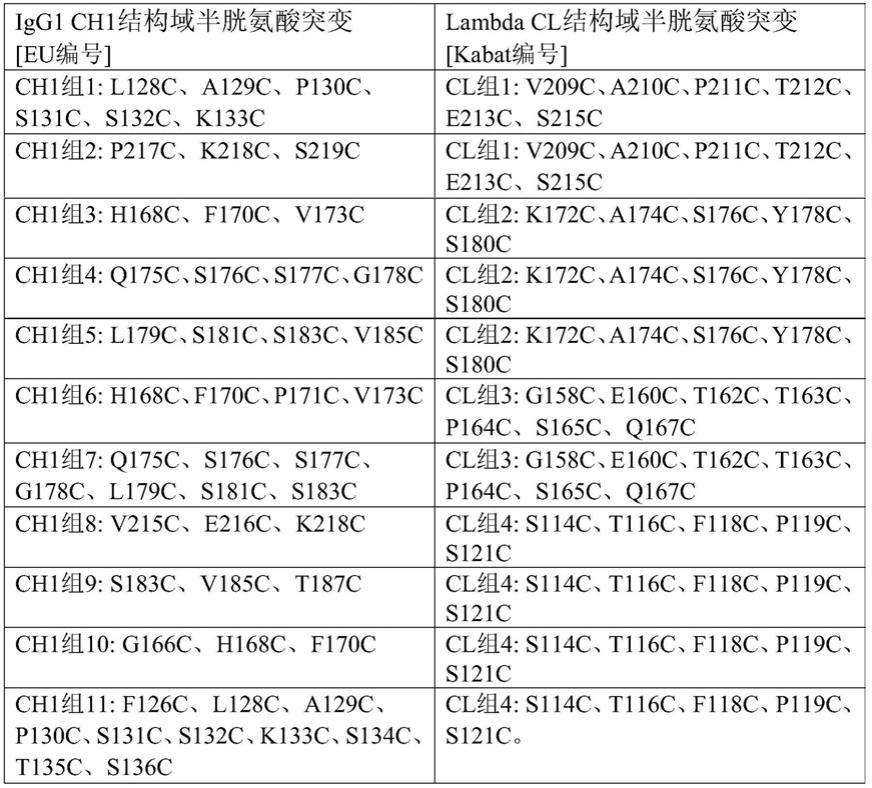
在具体的实施方式中,igg1的ch1-cl结构域中形成工程改造的链间二硫键的氨基酸选自下表:
[0023][0024]
在具体的实施方式中,igg1的ch1-cl结构域中形成工程改造的链间二硫键的氨基酸选自下组,其中每对半胱氨酸突变按以下方式列出:重链中选定的氨基酸[eu编号]突变为半胱氨酸/kappa轻链中选定的氨基酸[eu编号]突变为半胱氨酸:f126c/f118c、l128c/p120c、a129c/p120c、p130c/f118c、s132c/f118c、k133c/s121c、s134c/f118c、g166c/t172c、g166c/s174c、g166c/t180c、h168c/t180c、f170c/q160c、f170c/t164c、f170c/t172c、f170c/s174c、f170c/s176c、f170c/t180c、p171c/t172c、p171c/s174c、p171c/t180c、v173c/s174c、q175c/s174c、q175c/s176c、q175c/t180c、s176c/s162c、s181c/t172c、s181c/s176c、s181c/t180c、s183c/s114c、s183c/f118c、s183c/q160c、s183c/s174c、s183c/t178c、s183c/t180c、v185c/t172c、v185c/s174c、v185c/t178c、v185c/t180c、t187c/s114c、t187c/t172c、t187c/s174c、t187c/t178c、k218c/f118c、s219c/f116c、s219c/f118c、s219c/p120c。
[0025]
在优选的实施方式中,形成工程改造的链间二硫键的氨基酸主要位于igg1重链结构域的以下三个区域:f126-s136、g166-t187、v215-s219;lambda cl结构域的以下四个区域:s114-s121、g158-q167、k172-s180、v209-s215。
[0026]
在具体的实施方式中,igg1的ch1结构域以及相应的cl结构域中形成工程改造的链间二硫键的氨基酸残基选自下表:
[0027][0028][0029]
在具体的实施方式中,igg1的ch1结构域以及相应的cl结构域中形成工程改造的链间二硫键的氨基酸残基选自下组,其中每对半胱氨酸突变按以下方式列出:重链中选定的氨基酸[eu编号]突变为半胱氨酸/lambda轻链中选定的氨基酸[kabat编号]突变为半胱氨酸;
[0030]
s132c/s121c、k133c/t116c、k133c/p211c、s136c/s121c、f170c/g158c、p171c/t162c、p171c/p164c、s176c/t162c、l179c/g158c、s181c/p164c、v215c/t116c、e216c/f118c。
[0031]
在优选的实施方式中,形成工程改造的链间二硫键的氨基酸主要位于igg4重链结构域的以下二个区域:f126-e137、g166-p189;kappa cl结构域的以下四个区域:s114-s121、n158-e165、t172-s182、f209-e213。
[0032]
在具体的实施方式中,igg4的ch1-cl结构域中形成工程改造的链间二硫键的氨基酸选自下表:
[0033][0034]
在具体的实施方式中,igg4的ch1-cl结构域中形成工程改造的链间二硫键的氨基酸选自下组,其中每对半胱氨酸突变按以下方式列出:重链中选定的氨基酸[eu编号]突变为半胱氨酸/kappa轻链中选定的氨基酸[eu编号]突变为半胱氨酸;a129c/f209c、a129c/n210c、p130c/f116c、p130c/f118c、p130c/p119c、p130c/n210c、p130c/r211c、s132c/s114c、s132c/f116c、s132c/f118c、s132c/p120c、s132c/r211c、s132c/e213c、r133c/p119c、r133c/r211c、r133c/e213c、t135c/f116c、t135c/p120c、g166c/t178c、h168c/n158c、f170c/f182c、v173c/q160c、v173c/s162c、q175c/s162c、q175c/t180c、s181c/t172c、s181c/s176c、s183c/n158c、s183c/s176c、v185c/e165c、v185c/t178c。
[0035]
在优选的实施方式中,形成工程改造的链间二硫键的氨基酸主要位于igg4重链结构域的以下两个区域:f126-e137、h168-t187;lambda cl结构域的以下四个区域:s114-s121、g158-q167、k172-s180、v209-s215。
[0036]
在具体的实施方式中,igg4的ch1-cl结构域中形成工程改造的链间二硫键的氨基酸选自下表:
[0037][0038]
在具体的实施方式中,所述方法还包括以下步骤:通过氨基酸取代将fab臂的ch1-cl结构域的一对氨基酸电荷反转。
[0039]
在具体的实施方式中,重链213位的野生型带正电的赖氨酸被带负电的氨基酸取
代(例如,k213e、k213d);轻链第123位的野生型带负电荷的谷氨酸被正电荷的氨基酸取代(例如,e123k、e123r)。
[0040]
在优选的实施方式中,在igg1 kappa双特异性抗体的生产中,通过组合电荷反转(例如,k213d/e123k)和不同的半胱氨酸对,以便在fab臂的ch1-cl结构域形成工程改造的二硫键的方式来实现重链和轻链的正确配对:
[0041]
s132c/f116c、v173c/n158c、s131c/p120c、s132c/s114c、s132c/p120c、k133c/f116c、k133c/p120c、s134c/p120c、t135c/s114c、g166c/s176c、g166c/t178c、h168c/t172c、h168c/s174c、h168c/t178c、v173c/t180c、q175c/n158c、q175c/t172c、s176c/n158c、s176c/q160c、g178c/n158c、g178c/s162c、g178c/t164c、s181c/s174c、s183c/p120c、s183c/s162c、v185c/s114c、v185c/p120c、v185c/s176c、t187c/p120c、t187c/s176c、k218c/s114c、k218c/p120c,
[0042]
(每对半胱氨酸以以下方式列出:重链中野生型氨基酸[eu编号]突变为半胱氨酸/kappa链中野生型氨基酸[eu编号]突变为半胱氨酸)。
[0043]
在优选的实施方式中,在igg1 lambda双特异性抗体的生产中,通过组合电荷反转(例如k213d/e123k)与不同半胱氨酸对,以便在fab臂的ch1-cl结构域形成工程改造的二硫键来实现重链和轻链的正确配对,包括:
[0044]
l128c/t116c、a129c/p211c、a129c/t212c、p130c/a210c、p171c/t163c、e216c/t116c、p217c/s215c、k218c/f118c、k218c/p119c
[0045]
(每对半胱氨酸突变按以下方式列出:重链中野生型氨基酸[eu编号]突变为半胱氨酸/lambda链中野生型氨基酸[kabat编号]突变为半胱氨酸。
[0046]
在具体的实施方式中,也可以通过组合电荷反转(例如k213d/e123k)与上文列举的其它半胱氨酸对,以便在fab臂的ch1-cl结构域形成工程改造的二硫键来实现重链和轻链的正确配对。
[0047]
在具体的实施方式中,通过氨基酸取代形成工程改造的链间二硫键与通过氨基酸取代将fab臂的ch1-cl结构域的一对氨基酸残基电荷反转可以发生在同一个fab臂上,也可以发生在不同的fab臂上。
[0048]
在优选的实施方式中,所述方法还包括使得一个重链的ch3结构域中具有t366w“旋钮”突变,而在另一个重链的ch3结构域中具有t366s/l368a/y407v“孔”突变;和/或,使重链甲的ch3结构域中具有k409d、k392d突变(eu编号),在重链乙的ch3结构域中具有d399k、e356k突变。
[0049]
在第二方面,本发明提供一种提高双特异性抗体制备过程中重链和轻链正确配对率的方法,所述方法包括以下步骤:通过氨基酸取代将fab臂的ch1-cl结构域的一对氨基酸电荷反转,其中重链213位的野生型带正电的赖氨酸被带负电的氨基酸取代(例如,k213e、k213d);轻链123位的野生型带负电荷的谷氨酸被正电荷的氨基酸取代(例如,e123k、e123r)。
[0050]
在第三方面,本发明提供一种双特异性抗体的制备方法,所述方法包括在双特异性抗体的制备过程中采用第一方面或第二方面所述的方法以便提高双特异性抗体中重链和轻链正确配对率的步骤。
[0051]
在第四方面,本发明提供一种双特异性抗体,所述双特异性抗体是利用第三方面
所述的方法制备得到,或者采用第一方面或第二方面所述的方法提高了所述双特异性抗体中重链和轻链正确配对率。
[0052]
在优选的实施方式中,所述抗体是实施例中具体获得的双特异性抗体。
[0053]
应理解,在本发明范围内中,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。
附图说明
[0054]
这些附图示出了技术的各个方面,而不是限制性的。为了清楚和易于图示,未按比例绘制附图,并且在某些情况下,各个方面可能被夸大或放大以促进对特定方面的理解。
[0055]
图1.igg1和igg4抗体的结构域示意图。igg抗体是y型四聚体,具有两条重链(较长)和两条轻链(较短)。轻链通过cl和ch1结构域上的链间二硫键(-s-s-)与重链相连。两条重链在铰链区通过链间二硫键(-s-s-)连接在一起。vl:轻链可变结构域,cl:轻链恒定结构域,vh:重链可变结构域,ch1:重链恒定结构域1,ch2:重链恒定结构域2,ch3:重链恒定结构域3。
[0056]
图2.igg片段结构的示意图。木瓜蛋白酶消化igg产生两个相同的fab片段(保留抗原结合活性)和一个fc片段。胃蛋白酶消化igg产生f(ab’)2片段,该片段由两个fab样片段组成。
[0057]
图3.人igg1、igg2、igg3和igg4的铰链区的氨基酸序列。“_”表示在两个重链之间形成二硫键的半胱氨酸。序列是从imgt数据库(www.imgt.org)获得的。
[0058]
图4.人igg1、igg2、igg3和igg4的ch1结构域的氨基酸序列比较和eu编号。“*”表示序列同一性。“:”表示以下各组之一是完全保守的:sta、neqk、nhqk、ndeq、qhrk、milv、milf、hy或fyw。“.”表示以下各组之一是完全保守的:csa、atv、sag、stnk、stpa、sgnd、sndeqk、ndeqhk、neqhrk、fvlim或hfy。人igg1(p01857)、igg2(p01859)、igg3(p01860)和igg4(p01861)的ch1序列获自uniprot数据库(www.uniprot.org)。uniprot中的clustal omega程序用于氨基酸序列比较。
[0059]
图5.人抗体轻链kappa(κ)[eu编号]和lambda(λ)[kabat编号]的cl结构域的氨基酸序列比较。“*”表示序列同一性。“:”表示以下各组之一是完全保守的:sta、neqk、nhqk、ndeq、qhrk、milv、milf、hy或fyw。“.”表示以下各组之一是完全保守的:csa、atv、sag、stnk、stpa、sgnd、sndeqk、ndeqhk、neqhrk、fvlim或hfy。人抗体kappa轻链(p01834)和lambda轻链(p0doy3)的cl序列可从uniprot数据库(www.uniprot.org)获得。uniprot中的clustal omega程序用于氨基酸序列比较。
[0060]
图6.双特异性igg结构的示意图。(a)显示了抗体1fab臂中的突变型链间二硫键和抗体2fab臂中的野生型链间二硫键。抗体1重链中的“旋钮”突变和抗体2重链中的“孔”突变可促进重链异二聚化。抗体2重链中的rf突变用于在a蛋白纯化中去除抗体同二聚体。抗体2重链c末端的6xhis标签用于蛋白质印迹。(b)显示了抗体1fab臂中的突变型链间二硫键和电荷反转,以及抗体2fab臂中的野生型链间二硫键和残基电荷。
[0061]
图7.elisa鉴定双特异性igg1(kappa)半胱氨酸突变文库,其中在一个fab臂的ch1-cl结构域天然链间二硫键被工程改造的二硫键替代(每对半胱氨酸突变按以下方式列
出:重链野生氨基酸[eu编号]突变为半胱氨酸/kappa轻链野生氨基酸[eu编号]突变为半胱氨酸),wt代表两个fab臂中的两个天然链间二硫键。
[0062]
图8.elisa鉴定双特异性igg1(kappa)半胱氨酸和电荷突变文库,其中在一个fab臂的ch1-cl结构域k213/e123反转为相反电荷,天然链间二硫键被工程改造的二硫键替代(电荷反转按以下方式列出:重链中k213[eu编号]突变为d或e/轻链中e123[eu编号]突变为k或r(k213d/e123k、k213e/e123k、k213d/e123r、k213e/e123r)。每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/kappa轻链野生氨基酸[eu编号]突变为半胱氨酸,wt代表两个fab臂中的两个天然链间二硫键。
[0063]
图9.elisa鉴定双特异性igg1(lambda)半胱氨酸突变文库,其中在一个fab臂的ch1-cl结构域天然链间二硫键被工程改造的二硫键替代(每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/lambda轻链氨基酸[kabat编号]突变为半胱氨酸,wt代表两个fab臂中的两个天然链间二硫键。
[0064]
图10.elisa鉴定双特异性igg1(lambda)半胱氨酸和电荷突变文库,其中在一个fab臂的ch1-cl结构域k213/e123反转为相反电荷,并且天然链间二硫键被工程改造的二硫键替代(电荷反转按以下方式列出:重链中k213[eu编号]突变为d/轻链中e123[kabat编号]突变为k(k213d/e123k)。每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/lambda轻链野生氨基酸[kabat编号]突变为半胱氨酸,wt代表两个fab臂中的两个天然链间二硫键。
[0065]
图11.elisa鉴定双特异性igg1(kappa/lambda),其中在一个fab臂的ch1-cl结构域天然链间二硫键被工程改造的链间二硫键替代(每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/kappa轻链野生氨基酸[eu编号]突变为半胱氨酸,wt代表两个fab臂上的两个天然链间二硫键。
[0066]
图12.elisa鉴定双特异性igg1,其中在一个fab臂的ch1-cl结构域k213/e123反转为相反电荷,在另一个fab臂的ch1-cl结构域天然链间二硫键被工程改造的二硫键替代(电荷反转按以下方式列出:重链中k213突变为d/轻链中e123突变为k(k213d/e123k)。每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/轻链野生氨基酸[eu编号]突变为半胱氨酸,wt表示两个fab臂中均有天然链间二硫键,同时不存在电荷反转突变。
[0067]
图13.elisa鉴定双特异性igg4(kappa)半胱氨酸突变文库,其中在一个fab臂的ch1-cl结构域天然链间二硫键被工程改造的二硫键替代(每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/kappa轻链野生氨基酸[eu编号]突变为半胱氨酸。k147d/t129r代表在一个fab臂的ch1中147位赖氨酸突变为天冬氨酸(k147d),cl中129位苏氨酸突变为精氨酸(t129r)。
具体实施方式
[0068]
发明人经过广泛而深入的研究,提供了一种制备能够结合两种不同抗原的双特异性抗体的方法和相关组合物。具体地说,本发明提供了从两种已有抗体生产双特异性抗体的方法,包括在重链和轻链的界面处突变氨基酸。这样的突变包括用其它氨基酸替换半胱氨酸以消除天然链间二硫键,以及用半胱氨酸替换其它氨基酸形成工程改造的链间二硫
键。在这些突变的基础上,还可以包括通过氨基酸突变改变电荷相互作用。有这些突变存在下,可以产生双特异性抗体,其中重链优先与其自身的轻链配对,并且可以防止重链和轻链的错配。从而完成了本发明。
[0069]
术语定义
[0070]
除非另有定义,否则本发明中使用的科学和技术术语应具有本领域普通技术人员通常理解的含义。
[0071]
如本文所使用的,单数形式的术语在适当时可以以复数形式使用相应的术语,反之亦然。
[0072]
如本文所用,术语“氨基酸”或“氨基酸残基”包括天然氨基酸和非天然氨基酸。
[0073]
氨基酸在本文中通常以众所周知的三字母符号或iupac-iub生化命名委员会推荐的单字母符号来指代。同样,核苷酸通常用公认的单字母代码表示。
[0074]
本文的术语“氨基酸突变”包括多肽序列中的氨基酸取代、插入和/或缺失。本文中的“氨基酸取代”或“取代”是指用另一氨基酸替换多肽序列中特定位置的氨基酸。
[0075]
如本文所用,术语“天然”或“野生型”氨基酸是指天然存在于多肽的特定位置并且未被突变修饰的氨基酸残基。
[0076]
术语“多肽”、“寡肽”、“肽”和“蛋白”在本文可互换使用,是指任何长度的氨基酸链。可以通过本领域已知的用于蛋白合成的任何方法,特别是通过化学合成或通过重组表达技术来生产蛋白。
[0077]
如本文所用,蛋白表达中的术语“载体”和“质粒”可互换使用。
[0078]
如本文所用,蛋白表达中的“细胞”、“细胞系”和“细胞培养物”可互换使用。
[0079]“细胞转化”是指将外源dna引入细胞。通常是外源dna整合进基因组或引入自我复制质粒的结果。
[0080]
可以根据本领域技术人员众所周知的方法进行宿主细胞的转化和转染。合适的转化方法包括病毒感染、转染、接合、原生质体融合、电穿孔、粒子枪技术,磷酸钙沉淀、直接显微注射等。方法的选择通常取决于转化细胞的类型以及转化发生的环境。这些方法的一般性讨论可见于文献中(ausubel,et al.,short protocols in molecular biology,wiley&sons,1995)。
[0081]
可以采用不同的方法进行酵母转化,包括原生质球方法、电穿孔、聚乙二醇方法、碱金属阳离子方法等(gregg jm,pichia protocols,totowa,new jersey:humanna press,2010)。
[0082]
如本文所用,术语“抗原”是指与抗体特异性结合的任何物质。例如,抗原可以是蛋白、多肽、碳水化合物、多核苷酸、脂质或前述的组合。
[0083]
如本文所用,术语“表位”是指抗原上被特定抗体识别并结合的分子位点。
[0084]
如本文所用,术语“抗体”和“免疫球蛋白”可在最广义上互换使用。抗体是识别并特异性结合抗原的蛋白(免疫球蛋白)。抗体包括单克隆抗体、多克隆抗体,包含至少两个不同表位结合域的多特异性抗体(例如,双特异抗体)、人抗体、人源化抗体、骆驼抗体、嵌合抗体、抗体片段、包含抗体的抗原结合部分的融合蛋白以及包含抗原结合位点的任何其它经修饰的抗体分子。抗体可以源自任何哺乳动物,包括但不限于人、猴、山羊、马、兔、狗、猫、小鼠、鸡、骆驼、鲨鱼或其它动物。
[0085]
如本文所用,术语“重组抗体”旨在包括由被重组表达载体转染的宿主细胞,例如酵母或cho细胞,产生的所有抗体。
[0086]
免疫球蛋白由两条相同的重链和两条相同的轻链组成。基于其重链恒定区结构,免疫球蛋白可以分为五个类别,即iga、igd、ige、igg和igm。每个类别都可以具有kappa(κ)或lambda(λ)轻链。基于其重链结构,人igg可以分为四个亚类,即igg1、igg2、igg3、igg4。本发明的“抗体”可以是任何类别或亚类。优选地,本发明的抗体是人igg。
[0087]
在本文中,人igg(igg1、igg2、igg3和igg4)的ch1、ch2、ch3重链恒定区,其氨基酸序列编号采用“eu编号系统”(edelman gm et al.,proc natl acad sci usa,63(1):78-85(1969))。imgt数据库(国际immunogenetics)完整列出人igg1的ch1、铰链、ch2和ch3恒定区氨基酸序列及对应的编号。
[0088]
对于人igg kappa轻链恒定区,其氨基酸序列编号同样采用“eu编号系统”。imgt数据库完整列出人kappa轻链恒定区氨基酸序列及对应的编号。
[0089]
对于人igg lambda轻链恒定区,其氨基酸序列编号采用“kabat编号系统”(kabat ea et al,sequences of proteins of immunological interest.5th edition
–
us department of health and human services,nih publication,91-3242(1991))。imgt数据库完整列出人lambda轻链恒定区氨基酸序列及对应的编号。
[0090]
人igg1重链恒定区和铰链区边界在imgt数据库中有如下界定:ch1恒定区界定为氨基酸118至215,铰链区界定为氨基酸216至230,ch2恒定区界定为氨基酸231至340,ch3恒定区界定为氨基酸341至447。在igg1、igg2、igg3和igg4的ch1恒定区,其氨基酸序列高度保守(图4)。但是与imgt的铰链区界定不同,基于晶体学数据,在igg1空间结构上的铰链区界定为氨基酸221-237。因此在本发明中,igg1的ch1恒定区界定为氨基酸118至220,铰链区界定为氨基酸221-237,ch2恒定区界定为氨基酸238至340[eu编号]。人kappa轻链恒定区界定为氨基酸108至214[eu编号],人lambda轻链恒定区界定为氨基酸107a-215[kabat编号]次,如图5所示。
[0091]
kabat列出了抗体各亚型的许多氨基酸序列,并列出各亚型中每个位置最常见的氨基酸,从而列出保守序列。kabat对所列序列中的各氨基酸进行编号,并且这种编号方法已成为本领域的标准。应当理解,由于群体中存在同种异型和等位基因变异,这些位置上的野生型氨基酸残基可以不同于所列出的氨基酸,因此在所呈现的序列与现有技术序列之间可能存在个别氨基酸差异。
[0092]
在igg四种亚型中以及iga、igd、ige和igm之间,某个特定氨基酸的位置可以有变动。因此,某个特定氨基酸的位置并不是仅限于这个氨基酸在某个免疫球蛋白中这个特定位置,而是应该包括所有免疫球蛋白中对应的那些氨基酸位置。
[0093]
本领域技术人员对igg分子的各结构域相对应的氨基酸的界定可能有所不同。因此,上述结构域的n端或c端可以延伸或缩短1、2、3、4、5、6、7、8、9或甚至10个氨基酸。
[0094]
在本文中,氨基酸的突变用下列方法表示:野生氨基酸,氨基酸的位置,突变后的氨基酸。氨基酸用单字母代码表示,氨基酸的位置采用eu编号系统(igg重链恒定区和kappa轻链恒定区),和kabat编号系统(lambda轻链恒定区)。例如,h(c220v)表示抗体重链(h)中220位的野生半胱氨酸突变为缬氨酸。
[0095]
本发明的术语“全长igg”或“完整igg”是指结构完整的igg,但是它可以不具有igg
的所有功能。全长igg包含两个重链和两个轻链。每条重链通过链间二硫键和非共价相互作用与轻链结合,形成异二聚体。两条重链在铰链区通过链间二硫键连接。
[0096]
如本文所用,术语“二硫键”包括在两个半胱氨酸残基之间形成的共价键。半胱氨酸上有一个硫醇基,它可以与另一个半胱氨酸上的硫醇基形成二硫键。“链内二硫键”是指同一蛋白链内的两个半胱氨酸之间形成的二硫键。“链间二硫键”是指在同一蛋白的不同链上的两个半胱氨酸之间或在不同蛋白的两个半胱氨酸之间形成的二硫键。在本发明中,术语“半胱氨酸对”或“半胱氨酸配对”具有相同的含义,从而能互换使用,二者均是指能形成二硫键的两个半胱氨酸残基。
[0097]
本文所用,术语“蛋白结构域”是指蛋白的一部分,它可以空间折叠,有生物功能,可以不依赖于蛋白的其余部分而独立存在。类似地,本文所用的术语“ch1-cl结构域”是指抗体中重链ch1结构域与轻链cl结构域相互作用构成的蛋白结构。
[0098]
如本文所用,术语“界面”是指独立的蛋白结构域彼此接触的区域。
[0099]
术语“抗体突变体”包括自然界中不存在的抗体,以及其它非野生型抗体,这些抗体中至少有一个氨基酸或氨基酸侧链结构与野生型抗体的氨基酸不同。
[0100]
如本文所用,术语“抗体突变体”还包括非天然存在的其它形式的抗体,例如双特异性抗体和抗体片段(例如,fab、f(ab’)2等)。
[0101]
术语“fab部分”、“fab臂”、“fab”或“臂”在本文中可互换使用。
[0102]
在本文中,“电荷反转”是指带有某种电荷的氨基酸残基被带有相反电荷的氨基酸残基取代。例如,ch1结构域中213位野生型带正电荷的赖氨酸被带负电荷的氨基酸取代(k213e或k213d);cl结构域中123位野生型带负电荷的谷氨酸被带正电荷的氨基酸取代(e123k或e123r)。
[0103]
在一些方面,本文提供的抗体是双特异性的。如本文所用,“双特异性抗体”(bsabs)是对至少两种不同抗原或同一抗原内的至少两种不同表位具有结合特异性的抗体。抗体是具有两条相同的重链和轻链的对称结构免疫球蛋白,但双特异性抗体可以是非对称的,具有两条不同的重链,每条重链都与自己的轻链配对。因此,抗体是二价和单特异性的,这意味着它能同时与两个相同的抗原或表位进行专一性结合,但双特异性抗体是单价和双特异性的,每个fab臂都分别特异性结合不同的抗原或表位。
[0104]
本发明的双特异性抗体及其制备方法
[0105]
基于重链异二聚化的kih技术,我们构建了igg突变体文库,其第一个fab臂的ch1和cl结构域中的,包括以下取代:(a)将野生半胱氨酸突变为其它氨基酸以消除重链和轻链间的二硫键;(b)预测的野生氨基酸对突变为半胱氨酸对,用于形成工程改造的链间二硫键;(c)在ch1结构域的第213位的天然带正电荷的赖氨酸突变为带负电荷的氨基酸(k213e,k213d)取代;cl结构域中123位的天然带负电荷的谷氨酸突变位带正电荷的氨基酸(e123k,e123r)取代。igg第二个fab臂的ch1和cl结构域中对应的氨基酸未进行突变(野生型,wt)。像哺乳动物细胞一样,糖基工程改造的毕赤酵母可以正确进行蛋白质折叠、二硫键形成和糖基化修饰等细胞生物学机制(ellgaard l.and helenius a.,quality control in the endoplasmic reticulum,nat.rev.mol.cell biol.4(2003)181-191)。因此,可以将igg突变体文库在糖基工程改造的毕赤酵母(glycoengineered pichia pastoris)中表达,表达的igg突变体可以通过采用elisa方法进行筛选,用以确定那些突变引入的半胱氨酸对可形
成工程改造的链间二硫键的,促进双特异性抗体中重链和轻链的正确配对。
[0106]
本发明的双特异性抗体及其制备方法可应用于人igg各种亚型(igg1、igg2、igg3、igg4)和其它类别ig。在另一方面,本发明的双特异性抗体及其制备方法可应用于非人(例如灵长类动物或啮齿动物)不同亚型igg(例如鼠科igg1、igg2a、igg2b或igg3抗体)。
[0107]
在一些方面,本发明提供了产生双特异性抗体的方法。可以将双特异性抗体制成全长抗体或抗体片段,例如f(ab’)2。此外,本文提供的双特异性抗体易于表达,稳定且免疫原性低。本文所述的双特异性抗体结构为产生双特异性抗体提供了良好的平台,该双特异性抗体能实现双特异性抗体相关的优点,同时降低潜在的治疗风险。在某些方面,双特异性抗体可以特异性结合两种不同的抗原或同一抗原上的两个不同表位。双特异性抗体的两个fab臂通常包含两个不同的可变区。在一些方面,两个fab臂对两个独立抗原的结合亲和力可以大约相同。在一些方面,两个fab臂对两个独立抗原的结合亲和力可以不同。在一些方面,两个fab臂对同一抗原上的两个独立表位的结合亲和力可以大约相同。在一些方面,两个fab臂对同一抗原上的两个独立表位的结合亲和力可以不同。在其他方面,两个fab臂可以具有相同的特异性(例如,结合相同的或重叠的表位),但结合亲和力可以不同。在一些方面,可以将具有不同体内效能的两个抗体组合成双特异性抗体,其中一个fab臂具有高亲和力,另一个fab臂具有低亲和力,这样可能可以防止其中一个臂的给药过量或不足。
[0108]
在某些方面,本文提供了“半胱氨酸突变”多肽(例如,重链和轻链),其用于产生双特异性抗体并防止重链和轻链的错配。在一些方面,针对抗原(或表位)甲的抗体甲重链和轻链中野生半胱氨酸突变为其它氨基酸以消除天然链间二硫键,并且野生氨基酸突变为半胱氨酸以形成工程改造的链间二硫键。本文提供的此类突变位于ch1和cl结构域。针对抗原乙的抗体乙重链和轻链没有半胱氨酸突变。通过对抗体甲的重链和轻链进行半胱氨酸突变,可以将这四个多肽(重链和轻链)聚合在一起,以使抗体甲的重链与其轻链正确配对,而抗体乙的重链与其轻链正确配对,同时阻止抗体甲的重链与抗体乙的轻链错配,抗体乙的重链与抗体甲的轻链错配。如本文所用,术语“半胱氨酸未突变”多肽(例如,重链和轻链)是指重链和轻链含有野生半胱氨酸形成天然链间二硫键。此类“半胱氨酸突变”和“未突变”的重链和轻链可包含其它突变,例如,本文所述和/或本领域已知的fc区中的突变,以促进重链异二聚化。
[0109]
在一些方面,igg1、igg2、igg3或igg4重链中形成ch1和cl链间二硫键的野生半胱氨酸突变为其它氨基酸。将igg1重链220位[eu编号]野生半胱氨酸突变为其它氨基酸。将igg2、igg3和igg4重链131位[eu编号]野生半胱氨酸突变为其它氨基酸。在一些方面,igg kappa和lambda轻链中形成ch1和cl链间二硫键的野生半胱氨酸突变为其它氨基酸。igg kappa轻链214位[eu编号]野生半胱氨酸突变为其它氨基酸。igg lambda轻链214位[kabat编号]野生半胱氨酸突变为其它氨基酸。在一些方面,此类其它氨基酸包括天然存在的和/或非经典氨基酸。天然存在的其它氨基酸包括甘氨酸、丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、丝氨酸、苏氨酸、蛋氨酸、组氨酸、赖氨酸、精氨酸、谷氨酸、天冬氨酸、谷氨酰胺、天冬酰胺、苯丙氨酸、酪氨酸和色氨酸。非经典氨基酸包括但不限于鸟氨酸、二氨基丁酸、正亮氨酸、吡喃丙氨酸、噻吩丙氨酸、萘丙氨酸和苯基甘氨酸。优选的其它氨基酸是缬氨酸、丝氨酸或丙氨酸。
[0110]
在一些方面,igg1(kappa)的ch1和cl结构域中的野生氨基酸突变为不同的半胱氨
酸对。列表1总结了igg1(kappa)中野生氨基酸突变为不同的半胱氨酸对,它们可以形成工程改造的链间二硫键,在双特异抗体生产中促进重链和kappa轻链的正确配对。
[0111]
列表1:igg1(kappa)半胱氨酸对突变,形成工程改造的链间二硫键,促进重链和kappa轻链正确配对。
[0112]
f126c/f118c、l128c/p120c、a129c/p120c、p130c/f118c、s132c/f118c、k133c/s121c、s134c/f118c、g166c/t172c、g166c/s174c、g166c/t180c、h168c/t180c、f170c/q160c、f170c/t164c、f170c/t172c、f170c/s174c、f170c/s176c、f170c/t180c、p171c/t172c、p171c/s174c、p171c/t180c、v173c/s174c、q175c/s174c、q175c/s176c、q175c/t180c、s176c/s162c、s181c/t172c、s181c/s176c、s181c/t180c、s183c/s114c、s183c/f118c、s183c/q160c、s183c/s174c、s183c/t178c、s183c/t180c、v185c/t172c、v185c/s174c、v185c/t178c、v185c/t180c、t187c/s114c、t187c/t172c、t187c/s174c、t187c/t178c、k218c/f118c、s219c/f116c、s219c/f118c、s219c/p120c
[0113]
(每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/kappa链野生氨基酸[eu编号]突变为半胱氨酸,。例如:f126c/f118c表示重链126位f(苯丙氨酸)突变为c(半胱氨酸)/kappa轻链118位f(苯丙氨酸)突变为(半胱氨酸)。
[0114]
在一些方面,igg1(lambda)的ch1和cl结构域中的野生氨基酸突变为不同的半胱氨酸对。列表2总结了igg1(lambda)野生氨基酸突变为不同半胱氨酸对,从而能够形成工程改造的链间二硫键,在双特异抗体生产中促进重链和lambda轻链的正确配对。
[0115]
列表2:igg1(lambda)半胱氨酸对突变,形成工程改造的链间二硫键,促进重链和lambda轻链正确配对。
[0116]
s132c/s121c、k133c/t116c、k133c/p211c、s136c/s121c、f170c/g158c、p171c/t162c、p171c/p164c、s176c/t162c、l179c/g158c、s181c/p164c、v215c/t116c、e216c/f118c
[0117]
(每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/lambda链野生氨基酸[kabat编号]突变为半胱氨酸。例如:s132c/s121c表示重链132位s(丝氨酸)突变为c(半胱氨酸)/lambda轻链121位s(丝氨酸)突变为c(半胱氨酸)。
[0118]
本文提供在ch1和cl结构域中具有半胱氨酸突变的双特异性抗体,这些突变的抗体,可以进一步在下文描述的fc区中包含一个或多个突变。包含一个或多个突变的fc区在本文中称为“突变的fc区”。一对抗体fc之间的界面可以被突变以促进重链异二聚化,包括但不限于“kih”和静电相互作用突变。
[0119]
在一些方面,本文提供了在一个fab臂的ch1和cl结构域中具有工程改造的链间二硫键,而在另一个fab臂的ch1和cl结构域中具有天然链间二硫键的突变抗体。通常,一个fab臂的ch1和cl结构域有如下突变:野生半胱氨酸突变为其它氨基酸,野生氨基酸突变为半胱氨酸,从而在ch1和cl结构域形成新的二硫键替换天然二硫键。例如,表1和表2总结了将igg1野生氨基酸突变为一对半胱氨酸,在ch1和cl结构域形成工程改造的链间二硫键。
[0120]
在某些方面,本文提供了“电荷突变”多肽(例如重链和轻链),其用于产生双特异性抗体并防止重链和轻链的错误配对。在一些方面,针对抗原甲的抗体甲重链213位[eu编号]带正电荷的野生赖氨酸突变为带负电荷的氨基酸,例如天冬氨酸和谷氨酸(k213e,k213d)取代。抗体甲轻链123位带负电荷的野生谷氨酸[kappa中eu编号,lambda中kabat编号]突变为带正电荷的氨基酸,例如赖氨酸和精氨酸(e123k,e123r)。本文提供的改变电荷
极性的此类突变位于ch1和cl结构域中。针对抗原乙的抗体乙重链和轻链没有这些“电荷突变”。通过对抗体甲的重链和轻链进行“电荷突变”,可以将这四个多肽(重链和轻链)聚合在一起,以使抗体甲的重链与其轻链正确配对,而抗体乙的重链与其轻链正确配对,同时阻止抗体甲的重链与抗体乙的轻链错配,抗体乙的重链与抗体甲的轻链错配。如本文所用,术语“未突变”是指重链和轻链不包含如本文所述的用突变改变ch1和cl中野生氨基酸电荷极性。此类“电荷突变的”和“未突变的”重链和轻链可以包含其它突变,例如,本文所述和/或本领域已知的fc区中的突变,以有利于重链异二聚化。
[0121]
本文提供在ch1和cl结构域中具有电荷突变的双特异性抗体,这些突变的抗体可以进一步在下文描述的fc区包含一个或多个突变(突变的fc区),以促进重链异二聚化,包括但不限于“kih”和静电相互作用突变。
[0122]
在某些方面,本文提供了“半胱氨酸和电荷突变”多肽(例如重链和轻链),其用于产生双特异性抗体并防止重链和轻链的错配。在一些方面,针对抗原甲的抗体甲重链ch1结构域包含以下突变:(a)野生半胱氨酸突变为其它氨基酸(例如igg1c220v;igg2、igg3和igg4 c131s)以消除天然链间二硫键,野生氨基酸突变为半胱氨酸(如表1、2、3、4、5所列)以形成工程改造的链间二硫键;(b)213位带正电荷的野生赖氨酸突变为带负电荷的氨基酸(例如k213d,k213e)。针对抗原甲的抗体甲轻链的cl结构域包含以下突变:(a)野生半胱氨酸突变为其它氨基酸(例如kappa和lambda轻链c214v)以消除天然链间二硫键,野生氨基酸突变为半胱氨酸(如表1、2、3、4、5中所列)以形成新的链间二硫键;(b)123位带负电荷的野生谷氨酸突变为带正电荷的氨基酸(例如e123k,e123r)。针对抗原乙的抗体乙重链和轻链没有这些“半胱氨酸和电荷突变”。通过对抗体甲的重链和轻链进行“半胱氨酸和电荷突变”,可以将这四个多肽(重链和轻链)聚合在一起,以使抗体甲的重链与其轻链正确配对,而抗体乙的重链与其轻链正确配对,同时阻止抗体甲的重链与抗体乙的轻链错配,抗体乙的重链与抗体甲的轻链错配。如本文所用,术语“未突变”是指在本文所述的ch1和cl结构域中不包含半胱氨酸和电荷突变的重链和轻链。此类“半胱氨酸和电荷突变”和“未突变”的重链和轻链可以包含其它突变,例如本文所述和/或本领域已知的fc区中的突变,以促进重链异二聚化。列表3总结了igg1(kappa)野生氨基酸突变为不同半胱氨酸对,这些半胱氨酸对在“电荷突变”的协同作用下,能形成工程改造的链间二硫键,促进重链和kappa轻链的正确配对。
[0123]
列表3:igg1(kappa)野生氨基酸突变为不同半胱氨酸对,在“电荷突变”的协同作用下,能形成工程改造的链间二硫键,促进重链和轻链的正确配对。
[0124]
s132c/f116c、v173c/n158c、s131c/p120c、s132c/s114c、s132c/p120c、k133c/f116c、k133c/p120c、s134c/p120c、t135c/s114c、g166c/s176c、g166c/t178c、h168c/t172c、h168c/s174c、h168c/t178c、v173c/t180c、q175c/n158c、q175c/t172c、s176c/n158c、s176c/q160c、g178c/n158c、g178c/s162c、g178c/t164c、s181c/s174c、s183c/p120c、s183c/s162c、v185c/s114c、v185c/p120c、v185c/s176c、t187c/p120c、t187c/s176c、k218c/s114c、k218c/p120c
[0125]
(每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/kappa链野生氨基酸[eu编号]突变为半胱氨酸)。
[0126]
在一些方面,“半胱氨酸和电荷突变”可以应用于igg1(lambda)的ch1和cl结构域。
列表4总结了igg1(lambda)野生氨基酸突变为不同半胱氨酸对,这些半胱氨酸对在“电荷突变”的协同作用下,能形成工程改造的链间二硫键,促进重链和lambda轻链的正确配对。
[0127]
列表4:igg1(lambda)野生氨基酸突变为不同半胱氨酸对,在“电荷突变”(例如,k213d/e123k)的协同作用下,能形成工程改造的链间二硫键,促进重链和轻链的正确配对。
[0128]
l128c/t116c、a129c/p211c、a129c/t212c、p130c/a210c、p171c/t163c、e216c/t116c、p217c/s215c、k218c/f118c、k218c/p119c
[0129]
(每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/lambda链野生氨基酸[kabat编号]突变为半胱氨酸)。
[0130]
在一些方面,igg4(kappa)的ch1和cl结构域中的野生氨基酸突变为一对半胱氨酸。列表5总结了igg4(kappa)中野生氨基酸突变为不同的半胱氨酸对,它们可以形成工程改造的链间二硫键,促进igg4重链和kappa轻链的正确配对。
[0131]
列表5:igg4(kappa)半胱氨酸对突变,在ch1和cl结构域形成工程改造的链间二硫键,促进双特异性抗体生产中的重链和kappa轻链正确配对。
[0132]
a129c/f209c、a129c/n210c、p130c/f116c、p130c/f118c、p130c/p119c、p130c/n210c、p130c/r211c、s132c/s114c、s132c/f116c、s132c/f118c、s132c/p120c、s132c/r211c、s132c/e213c、r133c/p119c、r133c/r211c、r133c/e213c、t135c/f116c、t135c/p120c、g166c/t178c、h168c/n158c、f170c/f182c、v173c/q160c、v173c/s162c、q175c/s162c、q175c/t180c、s181c/t172c、s181c/s176c、s183c/n158c、s183c/s176c、v185c/e165c、v185c/t178c
[0133]
(每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/kappa轻链野生氨基酸[eu编号]突变为半胱氨酸)。
[0134]
在一些方面,如列表1、2和5中所列的半胱氨酸对突变也可以在“电荷突变”(例如,k213d/e123k)的协同作用下,形成工程改造的链间二硫键,促进双特异性抗体生产中的重链和轻链正确配对。
[0135]
在某些方面,本文提供的双特异抗体中,抗体甲的ch1和cl结构域中具有“半胱氨酸和电荷突变”的双特异性抗体,包括:(a)野生半胱氨酸突变为其它氨基酸以消除天然链间二硫键,野生氨基酸突变为不同的半胱氨酸对(如表1、2、3、4和5中所列)形成工程改造的链间二硫键;(b)ch1中213位带正电荷的野生赖氨酸突变为带负电荷的氨基酸(例如k213d,k213e);cl中123位带负电荷的野生谷氨酸突变为带正电荷的氨基酸(例如e123k,e123r)。抗体乙的ch1和cl结构域中没有这些“半胱氨酸和电荷突变”。抗体甲和抗体乙可以进一步在下文描述的fc区中包含一个或多个突变(突变的fc区),以促进重链异二聚化,包括但不限于“kih”和静电相互作用突变。
[0136]
在一些方面,本文提供的双特异抗体中,抗体甲的ch1和cl结构域中具有半胱氨酸突变,并且在抗体乙的ch1和cl结构域中具有电荷突变的双特异性抗体。抗体甲的ch1和cl结构域包含以下突变:野生半胱氨酸突变为其它氨基酸以消除天然链间二硫键,野生氨基酸突变为半胱氨酸对(如表1、2、3、4和5所示)形成新的链间二硫键。抗体乙的ch1和cl结构域包含以下突变:ch1中213位带正电荷的野生赖氨酸突变为带负电荷的氨基酸(例如k213d,k213e);cl中123位带负电荷的野生谷氨酸突变为带正电荷的氨基酸(例如e123k,e123r)。抗体甲和抗体乙可以进一步在下文描述的fc区中包含一个或多个突变(突变的fc
iggs in glycoengineered pichia pastoris.nat biotechnol 24:210-215)。
[0140]
本发明的优点:
[0141]
1.本发明的方法能够显著提高双特异性抗体生产中重链和轻链正确配对率;
[0142]
2.本发明的方法适用于各种抗体类型,包括但不限于igg、iga、igd、ige和igm;更优选igg1、igg2、igg3、igg4;
[0143]
3.本发明的方法可以直接将两个抗体组合成双特异性抗体。
[0144]
4.本发明的双特异性抗体其纯化方法与单克隆抗体相似。
[0145]
5.本发明的双特异性抗体具有完整的免疫球蛋白结构,稳定性好,免疫原性小。
[0146]
以下结合具体实施案例对本发明的技术方案进一步描述,但以下实施例不构成对本发明的限制,所有依据本发明的原理和技术手段采用的各种施用方法,均属于本发明范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件,或按照制造厂商所建议的条件。除非另外说明,否则百分比和份数按重量计算。
[0147]
材料
[0148]
用于创建、验证和应用文库的化学物质、酶、培养基和溶液是分子和细胞生物学领域技术人员普遍使用和众所周知的;它们可从许多公司获得,包括thermo fisher scientific、invitrogen、sigma、new england biolabs、takara biotechnology、toyobo、transgen biotech、vazyme biotech和generay biotechnology等。其中许多以试剂盒的形式提供。可以由其中一些公司化学合成dna。抗体序列数据主要来自uniprot数据库(www.uniprot.org)和imgt数据库(www.imgt.org)。
[0149]
方法
[0150]
除非另有说明,否则本发明中使用的方法,包括聚合酶链反应(pcr)、限制性酶克隆、dna纯化、细菌、酵母和真核细胞培养、转化、转染和蛋白质印迹均以分子和细胞生物学领域的技术人员众所周知的标准方法进行。本文所述的氨基酸突变(例如取代、缺失和插入)可以采用本领域已知的任何方法进行。这些方法包括但不限于pcr延伸重叠诱变、定点诱变或盒式诱变(通常参见sambrook j et al.(molecular cloning a laboratory manual,cold spring harbor laboratory press,cold spring harbor,n.y.,2001;ausubel f m et al.current protocols in molecular biology,wiley interscience,2010;and gregg jm(pichia protocols,totowa,new jersey:humanna press,2010)。
[0151]
根据制造商(vazyme biotech)操作说明,将dh5α感受态细胞用于质粒的构建和扩增。
[0152]
菌株在含有适当抗生素的luria-bertani(lb)培养基(10g/l胰蛋白、5g/l酵母提取物和5g/l氯化钠)或lb平板(10g/l胰蛋白、5g/l酵母提取物和5g/l氯化钠、20g/l琼脂)中生长。加入以下浓度的抗生素:100mg/l氨苄青霉素、50mg/l卡那霉素、25mg/l的zeocin和100mg/l的杀稻瘟素。
[0153]
按照制造商(biorad)的操作说明,通过micropulsertm电穿孔设备作电穿孔,以便进行毕赤酵母菌株的转化。
[0154]
毕赤酵母菌株在ypd培养基(10g/l酵母提取物、20g/l蛋白胨、20g/l葡萄糖)和ypd平板(10g/l酵母提取物、20g/l蛋白胨、20g/l葡萄糖、20g/l琼脂)中生长和选择。加入以下浓度的抗生素:250mg/l硫酸g-418、100mg/l的zeocin和300mg/l杀稻瘟素。
[0155]
在无氨基酸的ynb培养基(6.7g/l酵母氮基,20g/l葡萄糖)和无氨基酸的ynb板(6.7g/l酵母氮基,20g/l葡萄糖、20g/l琼脂)上培养并选择毕赤酵母营养型菌株,视需要适当补充氨基酸。
[0156]
毕赤酵母菌株在bmgy培养基(10g/l酵母提取物、20g/l蛋白胨、100mm磷酸钾,ph 6.0,13.4g/l酵母氮基、0.4mg/l生物素和10ml/l甘油)中生长,然后在bmmy培养基(10g/l酵母提取物,20g/l蛋白胨、100mm磷酸钾,ph 6.0,13.4g/l酵母氮基,0.4mg/l生物素和10ml/l甲醇)中诱导抗体表达。
[0157]
实施例1.设计igg1(kappa)ch1和cl结构域“半胱氨酸突变”文库
[0158]
igg1、igg2、igg3和igg4的ch1结构域序列高度保守。轻链在ch1-cl结构域通过链间二硫键与重链连接。在本发明中,我们希望通过对igg的ch1-cl结构域进行突变,使其天然链间二硫键被工程改造的链间二硫键替换。用来自蛋白质数据库的人igg1(kappa)的fab晶体结构(pdb代码:1vge)作为代表性结构,分析和设计了ch1和cl结构域中可能相互作用形成链间二硫键的半胱氨酸突变位点。其中形成天然链间二硫键的半胱氨酸对突变为缬氨酸,并在ch1-cl结构域的不同位置引入新的半胱氨酸对以形成工程改造的链间二硫键,由此,设计了igg1(kappa)突变体文库。表6列出在igg1(kappa)的ch1-cl结构域的不同氨基酸位置引入新的半胱氨酸对,这些半胱氨酸对可能形成工程改造的链间二硫键。
[0159]
表6.igg1(kappa)ch1-cl结构域的半胱氨酸突变文库。半胱氨酸突变聚集的区域称为一组。ch1结构域与cl结构域可能形成链间二硫键各组分别对应列出。
[0160][0161][0162]
实施例2.igg1(kappa)“半胱氨酸突变”文库的构建和表达
[0163]
包含t366s/l368a/y407v“孔”突变,h435r、y436f(rf)突变[eu编号]和c末端的6xhis标签的曲妥珠单抗(trastuzumab,erbb2 ab)重链(holerf-his),用作igg1(kappa)重链的代表(seq id no:1)。合成曲妥珠单抗重链(holerf-his)密码子优化的dna并用作pcr
扩增的模板(seq id no:2)。
[0164]
pcr 1,trah f(seq id no:3,该引物具有xho i限制酶位点)和trah r(seq id no:4,该引物具有not i限制酶位点)引物对用于曲妥珠单抗重链(holerf-his)的pcr扩增,使用合成的dna作为模板。将pcr产物用xho i和not i消化,并插入到ppic9(invitrogen)中相同的消化位点,以构建曲妥珠单抗重链(holerf-his)的表达载体,其命名为ppic9-trah(holerf-his)。
[0165]
野生型曲妥珠单抗kappa轻链用作igg1(kappa)轻链的代表(seq id no:5)。合成曲妥珠单抗kappa轻链密码子优化的dna并用作pcr扩增的模板(seq id no:6)。
[0166]
pcr 2,traκf(seq id no:7,该引物具有xho i限制酶位点)和traκr(seq id no:8,该引物具有not i限制酶位点)引物对用于曲妥珠单抗kappa轻链的pcr扩增,使用合成的曲妥珠单抗kappa轻链作为模板。将pcr产物用xho i和not i消化,并插入ppiczαa(invitrogen)的相同消化位点,以构建曲妥珠单抗kappa轻链的表达载体,命名为ppiczα-traκ。
[0167]
合成毕赤酵母arg2 3'和5'同源序列(带有afe i限制酶位点和adh1终止子序列)的arg2-adh1tt片段(seq id no:9),并将其用作pcr扩增的模板。
[0168]
pcr 3,arg2 f(seq id no:10,该引物具有bamh i限制酶位点)和adh1tt r(seq id no:11,该引物具有bamh i限制酶位点)引物对用于pcr扩增,合成的arg2-adh1tt片段作为模板。用bamh i限制性酶消化该pcr产物,并插入ppiczα-traκ中相同的位点,得到ppiczα-traκ-arg2表达载体,该载体用于整合在毕赤酵母arg2基因座上。
[0169]
pcr 4,picz f(seq id no:12)和picz r(seq id no:13)引物对用于pcr扩增无zeocin的ppiczα线性片段,ppiczα载体用作模板。
[0170]
pcr 5,kan f(seq id no:14)和kan r(seq id no:15)引物对用于pcr扩增卡那霉素片段,ppic9k载体(invitrogen)用作模板。
[0171]
用clonexpress ii一步克隆试剂盒(vazyme)将卡那霉素片段插入ppiczα线性片段,以产生peg载体,其中用卡那霉素代替ppiczα载体中的zeocin。
[0172]
pcr 6,eg f(seq id no:16)和eg r(seq id no:17)引物对用于pcr扩增peg线性片段,peg载体用作模板。
[0173]
包含t366w“旋钮”突变和c220v突变[eu编号]的clazakizumab(il6 ab)重链(knob)用作igg1(kappa)重链(seq id no:18)的一个代表。合成clazakizumab重链(knob)的密码子优化dna并用作pcr扩增的模板(seq id no:19)。
[0174]
pcr 7,重链n端引物clah-nt(seq id no:20)和c端引物clah-ct(seq id no:21)分别和表7中对应的反向引物(r),以及相应的正向引物(f)组成不同的引物对用于pcr扩增n端和c端重链,使用合成的clazakizumab重链(knob)作为模板。
[0175]
pcr 8,使用clah-nt和clah-ct引物对,通过重叠延伸pcr连接n端和c端重链pcr产物。以这种方式,产生了clazakizumab重链突变体,其包含野生氨基酸突变为半胱氨酸,t366w“旋钮”突变和c220v突变。
[0176]
表7.clazakizumab重链中野生氨基酸突变为半胱氨酸的pcr引物[eu编号]
[0177][0178][0179]
使用clonexpress ii一步克隆试剂盒(vazyme)将clazakizumab重链突变体插入peg线性片段中,生成表达载体文库,命名为peg-clah(x#c)(clah:clazakizumab重链,(x#c):编号#的野生氨基酸突变为半胱氨酸)。例如,peg-clah(l128c)表示重链128位的亮氨酸突变为半胱氨酸。各载体可表达clazakizumab重链,其含有t366w“旋钮”突变,c220v突变和表6所列的野生氨基酸突变为半胱氨酸。一种载体peg-clah可表达clazakizumab重链,其含有t366w“旋钮”突变和220位野生半胱氨酸,能形成天然链间二硫键。
[0180]
合成arg4-adh1tt片段(seq id no:53),其具有毕赤酵母arg4 3’和5’同源序列以及sma i限制酶位点和adh1终止子序列,并用作pcr扩增的模板。
[0181]
pcr 9,arg4 f(seq id no:54,该引物具有bamh i限制性酶切位点)和adh1tt r(seq id no:55,该引物具有bamh i限制性酶切位点)引物对用于pcr扩增arg4-adh1tt片段,使用合成的arg4-adh1tt片段作为模板。用bamh i限制性酶消化该pcr产物,并插入ppic6α(invitrogen)的相同位点,以产生ppic6α-arg4-adh1tt表达载体,该载体可以整合在毕赤酵母arg4基因座处。
[0182]
包含c214v突变的clazakizumab kappa轻链被用作igg1 kappa(κ)轻链的一个代表(seq id no:56)。合成带有c214v突变的clazakizumab kappa轻链的密码子优化dna,并用作pcr扩增的模板(seq id no:57)。
[0183]
pcr 10,轻链n端引物claκ-nt(seq id no:58,该引物具有xho i限制性酶切位点)
和轻链c端引物claκ-ct(seq id no:59,该引物具有not i限制酶位点)分别和表8中对应的反向引物(r)和正向引物(f)组成不同的引物对,用于pcr扩增n端和c端kappa轻链,合成的clazakizumab kappa链作为模板。
[0184]
pcr 11,使用claκ-nt和claκ-ct引物对,通过重叠延伸pcr连接n端和c端kappa轻链的pcr产物。它产生了clazakizumab kappa轻链突变体,其包含野生氨基酸突变为半胱氨酸和c214v突变。可以使用claκ-nt和表8中对应的反向引物(r),直接pcr扩增clazakizumab kappa轻链使c端野生氨基酸突变为半胱氨酸。可以使用claκ-nt和claκ-v214c反向引物(r)直接pcr扩增在214位具有野生半胱氨酸的clazakizumab kappa链。
[0185]
表8.clazakizumab kappa(κ)轻链中野生氨基酸突变为半胱氨酸的pcr引物[eu编号]
[0186][0187][0188]
clazakizumab kappa轻链突变体分别用xho i和not i消化,并插入到ppic6α-arg4-adh1tt的相同消化位点,以生成κ轻链突变表达载体文库,命名为ppic6-arg4-claκ(x#c)(claκ:clazakizumab kappa轻链,(x#c):编号#的野生氨基酸突变为半胱氨酸)。例如,ppic6-arg4-claκ(s114c)代表kappa轻链114位的野生丝氨酸突变为半胱氨酸。如表8所示,各载体可以表达包含c214v突变和野生氨基酸突变为半胱氨酸的clazakizumab kappa轻链,一种载体可以表达在214位具有野生半胱氨酸的clazakizumab kappa链。
[0189]
采用中国专利申请号201510220631.9、201710331370.7、201810108390.2中描述的方法构建了糖基工程改造的毕赤酵母菌株gs2-1(his4,ppbmt2-sfmns1::och1,scmnn10-atmns1::pno1)(ppbmt2-sfmns1是融合蛋白,包含毕赤酵母bmt2 n-端1-74氨基酸和草地贪夜蛾mnsi催化结构域164-670氨基酸;scmnn10-atmns1是融合蛋白,包含酿酒酵母mnn10 n
端1-116氨基酸和拟南芥mnsi催化结构域78-560氨基酸)。用gs2-1菌株表达的蛋白质n-聚糖的结构主要包含man5glcnac2。
[0190]
用限制酶afe i在arg2 3’和5’同源序列内将ppiczα-traκ-arg2的表达载体线性化,并电穿孔到gs2-1中。线性表达载体通过arg2 5'和3'同源序列重组整合到arg2基因座上。转化的细胞在补充了100mg/l zeocin的ypd平板上生长。这产生了新的表达菌株gs2-traκ,其可以表达曲妥珠单抗野生型kappa轻链。
[0191]
用限制酶sal i使ppic9-trah(holerf-his)表达载体线性化,电穿孔到gs2-traκ菌株中,并整合到his4基因座上。在ynb板上选择转化的细胞。这产生了新的表达菌株gs2-trahκ,以表达曲妥珠单抗野生型轻链和fc突变的重链,其中包含t366s/l368a/y407v“孔”突变,h435r、y436f(rf)突变和c末端的6xhis标签。
[0192]
用限制酶pme i线性化clazakizumab重链突变体文库peg-clah(x#c)的各表达载体,电穿孔入gs2-trahκ菌株,并整合在aox1基因座上。在补充了250mg/l g-418硫酸盐的ypd平板上选择转化的细胞。这产生了表达菌株的文库gs2-trahκ-clah(x#c)。这些菌株可以表达具有野生型轻链和fc突变重链的曲妥珠单抗,以及在ch1中具有半胱氨酸突变和在fc中具有“旋钮”突变的clazakizumab重链。
[0193]
用限制酶sma i将clazakizumab kappa轻链突变体文库ppic6-arg4-claκ(x#c)的各表达载体在arg4 3’和5’同源序列中线性化,然后电穿孔入表达菌株文库gs2-trahκ-clah(x#c)。线性表达载体通过arg4 3’和5’同源序列重组整合到arg4基因座上。转化的细胞在补充了300mg/l杀稻瘟菌素的ypd平板上生长。这样产生了表达菌株文库gs2-trahκ-clah(x#c)κ(x#c)。这些菌株可以表达不对称双特异性抗体。在不对称双特异性抗体中,双特异性抗体的一半是clazakizumab重链和轻链,其fab臂的ch1和cl结构域中具有工程改造的链间二硫键,在fc中具有“旋钮”突变。另一半是曲妥珠单抗重链和轻链,其fab臂的ch1和cl结构域中具有天然的链间二硫键,在fc中具有“孔”和rf突变,在重链的c端具有6xhis标签,
[0194]
gs2-trahκ-clah(x#c)κ(x#c)的表达菌株在bmgy培养基中以24℃和240rpm振荡培养72小时。然后通过3000g离心5分钟沉淀细胞,重悬于bmmy培养基中,以24℃和240rpm振荡培养,每天在培养基中加入甲醇(1%浓度),连续诱导表达72小时。随后,通过离心(3000g,10分钟)收获培养基上清液,并将上清液在-20℃冷冻直至下一步使用。
[0195]
实施例3.筛选鉴定igg1(kappa)“半胱氨酸突变”文库
[0196]
采用elisa测量培养上清液中双特异性抗体的浓度。简而言之,在96孔平板(maxisorp nunc-immuno,thermo scientific)加入100μl/孔5μg/ml agl蛋白(leeanntech),50mm碳酸钠缓冲液(ph 9.6),在4℃下包被过夜。用pbs-t(含0.05%tween-20的pbs)洗涤平板3次后,在37℃下用2%脱脂奶粉在pbs-t中封闭平板1小时。用pbs-t洗涤平板3次后,加入100μl/孔的人igg标准物(起始浓度2.5ug/ml)和在pbs-t中作1:2梯度稀释的培养上清液,在37℃下孵育1小时。将平板用pbs-t洗涤3次,加入100μl/孔在pbs-t和0.5%脱脂奶粉中作1:5000稀释的agl-hrp(leeanntech)。将平板在37℃下孵育1小时,用pbs-t洗涤3次,加入100μl/孔的tmb(leeanntech)。再加入100μl/孔的2m h2so4停止比色反应,并在450nm下测定每个孔的吸光度(a450nm)。
[0197]
根据制造商的说明,使用mabselect sure树脂(ge healthcare)通过a蛋白亲和层
gadre,martin jack borrok,xiang-qing yu,william dall’acqua,herren wu,and partha sarathi chowdhury,mabs 7,377
--
389;2015)。
[0202]
重链和轻链正确配对的双特异性抗体可以与抗原结合并在elisa中产生信号,但是重链和轻链错配的双特异性抗体不能与抗原结合,在elisa中不会产生信号。用横轴elisa吸光度值(a450nm)表示双特异性抗体的浓度,用纵轴elisa吸光度值(a450nm)表示双特异性抗体与抗原的结合量,用excel处理数据获得散点曲线图。在有效的elisa显色值范围内,曲线位置越高,即相同浓度条件下双特异性抗体与等量抗原的elisa反应显色值越高,则双特异抗体抗体的重链和轻链的正确配对率越高,错配率越低。曲线位置越低,则双特异抗体抗体的重链和轻链的正确配对率越低,错配率越高。如图7所示,在相同抗体浓度条件下(横轴elisa吸光度值),已报道的双特异性抗体的阳性对照f126c/s121c具有98%的重链和轻链正确配对,因此显示很高的抗体/抗原结合elisa吸光度值(纵轴),但是双特异性抗体的阴性对照wt预计只有25%重链和轻链正确配对,因此显示很低的抗体/抗原结合elisa吸光度值。在相同抗体浓度条件下(横轴elisa吸光度值),我们构建的许多双特异性抗体具有与阳性对照f126c/s121c相同或更高的抗体/抗原结合吸光度值。这表明我们的双特异性抗体具有98%或以上的重链和轻链正确配对。因此,在igg1 kappa双特异性抗体生产中,通过将天然的链间二硫键替换为不同的半胱氨酸配对,在一个fab臂的ch1-cl结构域形成工程改造的二硫键,可以实现重链和轻链的正确配对,包括:
[0203]
f126c/f118c、l128c/p120c、a129c/p120c、p130c/f118c、s132c/f118c、k133c/s121c、s134c/f118c、g166c/t172c、g166c/s174c、g166c/t180c、h168c/t180c、f170c/q160c、f170c/t164c、f170c/t172c、f170c/s174c、f170c/s176c、f170c/t180c、p171c/t172c、p171c/s174c、p171c/t180c、v173c/s174c、q175c/s174c、q175c/s176c、q175c/t180c、s176c/s162c、s181c/t172c、s181c/s176c、s181c/t180c、s183c/s114c、s183c/f118c、s183c/q160c、s183c/s174c、s183c/t178c、s183c/t180c、v185c/t172c、v185c/s174c、v185c/t178c、v185c/t180c、t187c/s114c、t187c/t172c、t187c/s174c、t187c/t178c、k218c/f118c、s219c/f116c、s219c/f118c、s219c/p120c、
[0204]
(每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/kappa轻链野生氨基酸[eu编号]突变为半胱氨酸)
[0205]
实施例4.igg1(kappa)“半胱氨酸和电荷突变”文库的构建和表达
[0206]
在igg1(kappa)ch1和cl结构域中,一对带电荷的氨基酸(重链中的k213和kappa轻链中的e123,eu编号)参与电荷-电荷相互作用。这对氨基酸在igg1、igg2、igg3和igg4中是保守性的,起同样作用。在中性ph(ph=7.0)下,天冬氨酸(d)和谷氨酸(e)带负电,赖氨酸(k)、精氨酸(r)和组氨酸(h)带正电。有相反电荷的氨基酸之间可以发生相互吸引作用,而有相似电荷的氨基酸之间则发生相互排斥作用。如表9所示,在抗体甲重链和轻链的ch1和cl结构域中突变这对氨基酸使其电荷极性发生反转(例如k213d和e123k),可能会导致错配的重链和轻链之间发生不利的相互排斥作用(例如,抗体甲重链k213d突变的负电荷和抗体乙轻链e123的负电荷相互排斥;抗体甲轻链e123k突变的正电荷和抗体乙重链k213的正电荷相互排斥)。在ch和cl结构域引入突变的链间二硫键或/和电荷反转可促进双特异性抗体中重链和轻链的正确配对。
[0207]
表9.重链和轻链中电荷极性反转的突变.
[0208][0209][0210]
pcr 1,eg f和eg r引物对用于以peg载体为模板pcr扩增peg线性片段(如实施例2中所述)。
[0211]
pcr 2,使用clah-nt和clah-k213d r引物对(seq id no:84),clah-k213d f(seq id no:85)和clah-ct引物分别进行n端和c端重链的pcr扩增,使用clazakizumab突变体文库peg-clah(x#c)的各表达载体作为模板。使用clah-nt和clah-ct引物对通过重叠延伸pcr连接n端和c端重链。这样产生了突变的clazakizumab重链,其包含t366w“旋钮”突变、c220v突变、半胱氨酸突变和k213d电荷突变。
[0212]
pcr 3,使用clah-nt和clah-k213d r引物对,clah-k213e f(seq id no:86)和clah-ct引物对分别进行n端和c端重链的pcr扩增,使用clazakizumab重链突变体文库peg-clah(x#c)的各表达载体作为模板。使用clah-nt和clah-ct引物对通过重叠延伸pcr连接n末端和c末端重链。这样产生了突变的clazakizumab重链,其包含t366w“旋钮”突变、c220v突变、半胱氨酸突变和k213e电荷突变。
[0213]
使用clonexpress ii一步克隆试剂盒(vazyme)将突变的clazakizumab重链插入peg线性片段,以构建clazakizumab突变表达载体文库,命名为peg-clah(x#cd)和peg-clah(x#ce)。各载体可以表达突变的clazakizumab重链,包含t366w“旋钮”突变、c220v突变、野生氨基酸突变为半胱氨酸,以及k213d或k213e突变。
[0214]
pcr 4,使用表10-1中对应的引物对分别进行n端和c端kappa轻链的pcr扩增,使用kappa轻链突变体文库的各表达载体ppic6α-claκ(x#c)作为模板。使用claκ-nt和claκ-ct引物对通过重叠延伸pcr连接n端和c端kappa链。这样产生了突变的clazakizumab kappa链,其包含c214v突变,半胱氨酸突变和e123k突变。
[0215]
表10-1 clazakizumab轻链n端和c端pcr扩增引物对
[0216][0217]
pcr 5,使用表10-2中对应的引物对分别进行n端和c-端kappa轻链的pcr扩增,使用kappa轻链突变体文库的各表达载体ppic6α-claκ(x#c)作为模板。使用claκ-nt和claκ-ct引物对通过重叠延伸pcr连接n端和c端kappa链。这样产生了突变的clazakizumab kappa
链,其包含c214v突变,半胱氨酸突变和e123r突变。
[0218]
表10-2 clazakizumab轻链n端和c端pcr扩增引物对
[0219][0220]
将突变的clazakizumab kappa链用xho i和not i消化,并插入ppic6α-arg4的相同消化位点,以生成kappa轻链突变表达载体文库,称为ppic6α-arg4-claκ(x#ck)和ppic6α-arg4-claκ(x#cr)。各载体可以表达突变的clazakizumab kappa轻链,其包含c214v突变、半胱氨酸突变和e123k或e123r突变。
[0221]
用限制性内切酶pme i线性化clazakizumab重链突变体文库的各表达载体peg-clah(x#cd)和peg-clah(x#ce),电穿孔入gs2-trahκ菌株,并整合在aox1基因座上。在补充了250mg/l g-418硫酸盐的ypd平板上选择转化的细胞。这样产生了表达菌株文库gs2-trahκ-clah(x#cd),gs2-trahκ-clah(x#ce)。
[0222]
用sma i线性化clazakizumab kappa轻链突变文库ppic6α-arg4-claκ(x#ck)和ppic6α-arg4-claκ(x#cr)的各表达载体,并电穿孔到表达菌株文库gs2-trahκ-clah(x#cd)和gs2-trahκ-clah(x#ce)。线性表达载体整合在arg4基因座处。转化的细胞在补充了300mg/l杀稻瘟菌素的ypd平板上生长。这样产生了表达菌株文库gs2-trahκ-clah(x#cd)κ(x#ck)、gs2-trahκ-clah(x#cd)κ(x#cr)、gs2-trahκ-clah(x#ce)κ(x#ck)、gs2-trahκ-clah(x#ce)κ(x#cr)。这些菌株可以表达不对称双特异性抗体。在不对称双特异性抗体中,双特异性抗体的一半是clazakizumab重链和轻链,其fab臂的ch1和cl结构域具有工程改造的链间二硫键和/或一对电荷突变,在fc中具有“旋钮”突变;另一半是曲妥珠单抗重链和轻链,其fab臂的ch1和cl结构域中具有天然的链间二硫键,在fc中具有“孔”和rf突变,在重链的c端具有6xhis标签。
[0223]
如实施例2中所述,表达菌株在bmgy培养基中培养,在bmmy培养基中诱导表达抗体。离心收获的上清液在-20℃冷冻直至下一步使用。
[0224]
实施例5.筛选鉴定igg1(kappa)“半胱氨酸和电荷突变”文库
[0225]
如实施例3中所述,通过a蛋白亲和层析纯化双特异性抗体,通过elisa测定纯化抗体的浓度。
[0226]
如实施例3中所述,采用elisa比较双特异性抗体中不同的链间二硫键和电荷突变是否能促进重链和轻链的正确配对,该双特异性抗体在ch1和cl结构域包含一对新的半胱氨酸和电荷反转突变(例如k213d和e123k)。如图8a所示,在相同抗体浓度条件下(横轴elisa吸光度值),双特异性抗体阳性对照f126c/s121c有98%的重链和轻链正确配对,具有
很高的抗体/抗原结合elisa吸光度值(纵轴),但双特异性抗体阴性对照wt只有25%的重链和轻链正确配对,有很低的抗体/抗原结合吸光度值(纵轴)。在相同的抗体浓度条件下(横轴elisa吸光度值),在fab臂的ch1-cl结构域包含电荷反转(k213d/e123k、k213e/e123k)的两个双特异性抗体具有高于阳性对照(f126c/s121c)的抗体/抗原结合吸光度值。但是,在fab臂的ch1-cl结构域包含电荷反转(k213d/e123r、k213e/e123r)的另外两个双特异性抗体的抗体/抗原结合吸光度值明显低于阳性对照f126c/s121c。因此,在igg双特异性抗体生产中,可以通过使用电荷反转突变(例如k213d/e123k、k213e/e123k)促进重链和轻链的正确配对(每对电荷反转突变按以下方式列出:重链213位赖氨酸k突变为带负电荷氨基酸d或e/轻链123位谷氨酸e突变为带正电荷的氨基酸k或r)。
[0227]
为了评估一个fab臂的ch1和cl结构域中一对新的半胱氨酸与电荷反转的组合对重链和轻链正确配对的影响,使用一个双特异性抗体trahκ-clah(s132c)κ(f116c),简记为s132/f116c和以下四个有电何突变的双特异性抗体为例:
[0228]
trahκ-clah(s132c,k213d)κ(f116c,e123k),简记为s132c,k213d/f116c,e123k;
[0229]
trahκ-clah(s132c,k213e)κ(f116c,e123k),简记为s132c,k213e/f116c,e123k;
[0230]
trahκ-clah(s132c,k213d)κ(f116c,e123r),简记为s132c,k213d/f116c,e123r,
[0231]
trahκ-clah(s132c,k213e)κ(f116c,e123r),简记为s132c,k213e/f116c,e123r。
[0232]
如图8b所示,在相同抗体浓度条件下(横轴elisa吸光度值),在一个fab臂的ch1-cl结构域含有一对新的半胱氨酸的双特异性抗体s132c/f116c具有与阳性对照f126c/s121c相似的抗体/抗原结合吸光度值。含有新的半胱氨酸配对和电荷反转的4个双特异性抗体s132c,k213d/f116c,e123k、s132c,k213e/f116c,e123k、s132c,k213d/f116c,e123r、s132c,k213e/f116c,e123r能进一步增加抗体/抗原结合吸光度值。
[0233]
为了评估一个fab臂的ch1和cl结构域中一对新的半胱氨酸与电荷反转的组合对重链和轻链正确配对的影响,使用一个双特异性抗体(trahκ-clah(v173c)κ(n158c),简记为v173c/n158c和以下四个双特异性抗体为另一例子:
[0234]
trahκ-clah(v173c,k213d)κ(n158c,e123k),简记为v173c,k213d/n158c,e123k;
[0235]
trahκ-clah(v173c,k213e)κ(n158c,e123k),简记为v173c,k213e/n158c,e123k;
[0236]
trahκ-clah(v173c,k213d)κ(n158c,e123r),简记为v173c,k213d/n158c,e123r;
[0237]
trahκ-clah(v173c,k213e)κ(n158c,e123r),简记为v173c,k213e/n158c,e123r。
[0238]
如图8c所示,在相同抗体浓度条件下(横轴elisa吸光度值),在一个fab臂的ch1-cl结构域含有一对新的半胱氨酸的双特异性抗体v173c/n158c具有比阳性对照f126c/s121c低的抗体/抗原结合吸光度值。两个含有新的半胱氨酸对和电荷反转的双特异性抗体v173c,k213d/n158c,e123k;v173c,k213e/n158c,e123k具有高于阳性对照f126c/s121c的抗体/抗原结合吸光度值。另两个含有新的半胱氨酸配对和电荷反转的双特异性抗体v173c,k213d/n158c,e123r;v173c,k213e/n158c,e123r)具有比双特异性抗体v173c/n158c的抗体/抗原结合吸光度值高,但比阳性对照f126c/s121c的抗体/抗原结合吸光度值低。这些实例表明,在一个fab臂ch1-cl结构域,重链k213和轻链e123的电荷反转突变可以进一步改善有半胱氨酸对突变的双特异性抗体的重链和轻链正确配对。看起来,k213d/e123k和k213e/e123k的电荷反转比k213d/e123r、k213e/e123r的电荷反转具有明显更大的改善效果。
[0239]
在本发明中,在一个fab臂的ch1-cl结构域,将k213d/e123k的电荷反转应用于改善其它有不同半胱氨酸突变的双特异性抗体的重链和轻链正确配对。如图8d所示,在相同抗体浓度条件下(横轴elisa吸光度值),在一个fab臂的ch1-cl结构域含有半胱氨酸突变的双特异性抗体具有与阳性对照f126c/s121c类似的或较低的抗体/抗原结合吸光度值。含有半胱氨酸和k213d/e123k电荷突变的相应双特异性抗体具有高于阳性对照f126c/s121c的抗体/抗原结合吸光度值。因此,在igg kappa双特异性抗体的生产中,为了进一步促进重链和轻链的正确配对形成工程改造的二硫键,可以在一个fab臂的ch1-cl结构域组合k213d/e123k的电荷突变和不同的半胱氨酸对突变,包括但不限于:
[0240]
s132c/f116c、v173c/n158c、s131c/p120c、s132c/s114c、s132c/p120c、k133c/f116c、k133c/p120c、s134c/p120c、t135c/s114c、g166c/s176c、g166c/t178c、h168c/t172c、h168c/s174c、h168c/t178c、v173c/t180c、q175c/n158c、q175c/t172c、s176c/n158c、s176c/q160c、g178c/n158c、g178c/s162c、g178c/t164c、s181c/s174c、s183c/p120c、s183c/s162c、v185c/s114c、v185c/p120c、v185c/s176c、t187c/p120c、t187c/s176c、k218c/s114c、k218c/p120c
[0241]
(每对半胱氨酸突变用以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/kappa轻链野生氨基酸[eu eu编号]突变为半胱氨酸,eu编号)。
[0242]
如有必要,为了进一步促进重链和轻链的正确配对形成工程改造的二硫键,可以在一个fab臂的ch1-cl结构域中将实施例3确定的不同半胱氨酸对突变和k213d/e123k电荷突变组合在一起。
[0243]
实施例6.设计igg1(lambda)ch1和cl结构域“半胱氨酸突变”文库
[0244]
igg lambda轻链通过ch1和cl结构域的链间二硫键连接至重链。在本发明中,我们对igg(lambda)的ch1-cl结构域进行突变,将形成天然链间二硫键的半胱氨酸对突变为缬氨酸,引入新的半胱氨酸对以形成工程改造的链间二硫键,取代天然链间二硫键。我们用来自蛋白质数据库的人igg1(lambda)的fab晶体结构(pdb代码:2fb4)作为代表性结构,分析和设计了ch1和cl结构域中可能相互作用形成链间二硫键的半胱氨酸突变位点。ch1和cl结构域中这些半胱氨酸突变位点列于表11,用于构建igg1(lambda)“半胱氨酸突变”文库。
[0245]
表11.igg1(lambda)ch1-cl结构域的半胱氨酸突变文库。半胱氨酸突变聚集的区域称为一组。ch1结构域与cl结构域可能形成链间二硫键各组分别对应列出。
[0246][0247][0248]
实施例7.igg1(lambda)半胱氨酸突变文库的构建和表达
[0249]
包含t366s/l368a/y407v“孔”突变、h435r、y436f(rf)突变和在c端的6xhis标签的fezakinumab(il22抗体)重链(holerf-his)用作igg1重链的代表(seq id no:102)。合成fezakinumab重链密码子优化dna(holerf-his),并用作pcr扩增的模板(seq id no:103)。
[0250]
pcr 1,fezh f(seq id no:104,引物具有xho i限制酶位点)和fezh r(seq id no:105,引物具有not i限制酶位点)引物对用于fezakinumab重链(holerf-his)的pcr扩增,使用合成的dna作为模板。用xho i和not i消化pcr产物,并插入ppic9(invitrogen)的相同消化位点,以构建fezakinumab重链(holerf-his)的表达载体,命名为ppic9-fezh(holerf-his)。
[0251]
fezakinumab轻链用作igg1 lambda(λ)轻链的代表(seq id no:106)。合成fezakinumab lambda轻链的密码子优化dna(seq id no:107),并将其用作pcr扩增的模板。
[0252]
pcr 2fezλf(seq id no:108,该引物具有xho i限制酶位点)和fezλr(seq id no:109,该引物具有not i限制酶位点)引物对用于fezakinumab lambda轻链的pcr扩增,利用合成的fezakinumab轻链作为模板。将pcr产物用xho i和not i消化,并插入ppiczα-traκ-arg2(如实施例2中所述)的相同消化位点,以构建fezakinumab轻链的表达载体,命名为ppiczα-fezλ-arg2。
[0253]
pcr 3,eg f和eg r引物对用于pcr扩增peg线性片段,以peg载体作为模板。
[0254]
将含有t366w“旋钮”突变和c220v突变的otelixizumab(cd3单抗)重链(旋钮)用作
igg1重链(seq id no:110)的另一个代表。合成otelixizumab重链(旋钮)的密码子优化dna,并用作pcr扩增的模板(seq id no:111)。
[0255]
pcr 4,重链n端引物oteh-nt(seq id no:112)和c端引物oteh-ct(seq id no:113)分别与表12中对应的反向引物(r)和正向引物(f)组成不同的引物对用于pcr扩增n端和c端重链,利用合成的otelixizumab重链(旋钮)为模板。
[0256]
pcr 5,使用oteh-nt和oteh-ct引物对,通过重叠延伸pcr连接n端和c端重链的pcr产物。以此方式产生了otelixizumab重链突变体,其包含野生氨基酸突变为半胱氨酸,t366w“旋钮”突变和c220v突变。
[0257]
使用clonexpress ii一步克隆试剂盒(vazyme)将otelixizumab重链突变体插入peg线性片段,生成表达载体文库,命名为peg-oteh(x#c)(oteh:otelixizumab重链,(x#c):在#位的野生氨基酸突变为半胱氨酸)。
[0258]
表12.otelixizumab重链野生氨基酸突变为半胱氨酸的pcr引物(eu编号).
[0259]
突变反向引物(r)正向引物(f)oteh-f126coteh-f126c r(seq id no:114)oteh-f126c f(seq id no:115)oteh-l128coteh-f126c r(seq id no:114)oteh-l128c f(seq id no:116)oteh-a129coteh-f126c r(seq id no:114)oteh-a129c f(seq id no:117)oteh-p130coteh-f126c r(seq id no:114)oteh-p130c f(seq id no:118)oteh-s131coteh-f126c r(seq id no:114)oteh-s131c f(seq id no:119)oteh-s132coteh-f126c r(seq id no:114)oteh-s132c f(seq id no:120)oteh-k133coteh-f126c r(seq id no:114)oteh-k133c f(seq id no:121)oteh-s134coteh-f126c r(seq id no:114)oteh-s134c f(seq id no:122)oteh-t135coteh-f126c r(seq id no:114)oteh-t135c f(seq id no:123)oteh-s136coteh-f126c r(seq id no:114)oteh-s136c f(seq id no:124)oteh-g166coteh-g166c r(seq id no:125)oteh-g166c f(seq id no:126)oteh-h168coteh-g166c r(seq id no:125)oteh-h168c f(seq id no:127)oteh-f170coteh-g166c r(seq id no:125)oteh-f170c f(seq id no:128)oteh-p171coteh-g166c r(seq id no:125)oteh-p171c f(seq id no:129)oteh-v173coteh-g166c r(seq id no:125)oteh-v173c f(seq id no:130)oteh-q175coteh-g166c r(seq id no:125)oteh-q175c f(seq id no:131)oteh-s176coteh-s176c r(seq id no:132)oteh-s176c f(seq id no:133)oteh-s177coteh-s176c r(seq id no:132)oteh-s177c f(seq id no:134)oteh-g178coteh-s176c r(seq id no:132)oteh-g178c f(seq id no:135)oteh-l179coteh-s176c r(seq id no:132)oteh-l179c f(seq id no:136)oteh-s181coteh-s181c r(seq id no:137)oteh-s181c f(seq id no:138)oteh-s183coteh-s181c r(seq id no:137)oteh-s183c f(seq id no:139)oteh-v185coteh-s181c r(seq id no:137)oteh-v185c f(seq id no:140)oteh-t187coteh-s181c r(seq id no:137)oteh-t187c f(seq id no:141)oteh-v215coteh-v215c r(seq id no:142)oteh-v215c f(seq id no:143)oteh-e216coteh-v215c r(seq id no:142)oteh-e216c f(seq id no:144)
oteλ-p211coteλ-p211c r(seq id no:175) oteλ-t212coteλ-t212c r(seq id no:176) oteλ-e213coteλ-e213c r(seq id no:177) oteλ-v214coteλ-v214c r(seq id no:178) oteλ-s215coteλ-s215c r(seq id no:179) [0265]
otelixizumab lambda轻链突变体用xho i和not i消化,并插入ppic6α-arg4的相同消化位点以生成lambda轻链突变体表达载体文库,命名为ppic6-arg4-oteλ(x#c)(oteλ:otelixizumab lambda轻链,(x#c):编号#位野生氨基酸突变为半胱氨酸,kabat编号)。例如,ppic6-arg4-otelλ-s114c表示otelixizumab lambda轻链114位的野生丝氨酸突变为半胱氨酸。如表13所示,各载体可表达包含c214v突变和野生氨基酸突变为半胱氨酸的otelixizumab lambda轻链,一个载体可表达在214位具有野生半胱氨酸的otelixizumab lambda链。
[0266]
如实施例3所述,用限制酶afe i消化arg2同源序列,将ppiczα-fezλ-arg2表达载体线性化,电穿孔入gs2-1,并通过arg2 5’和3’同源序列重组整合到arg2基因座上。转化的细胞在补充了100mg/l zeocin的ypd平板上生长。如此产生了新的表达菌株gs2-fezλ。
[0267]
用限制酶sal i将ppic9-fezh(holerf-his)表达载体线性化,电穿孔到gs2-fezλ菌株中,并整合到毕赤酵母基因组的his4基因座。在ynb板上选择转化的细胞。如此产生了新的表达菌株gs2-fezhλ。
[0268]
用限制酶pme i将otelixizumab重链突变体文库peg-oteh(x#c)的各表达载体线性化,电穿孔入gs2-fezhλ菌株中,并整合到毕赤酵母基因组的aox1基因座。在补充了250mg/l g-418硫酸盐的ypd平板上选择转化的细胞。如此产生了表达菌株文库gs2-fezhκ-oteh(x#c)。
[0269]
用限制酶sma i将otelixizumab lambda链突变体文库ppic6-arg4-oteλ(x#c)的各表达载体在arg4 3’和5’同源序列内线性化,电穿孔入表达菌株文库gs2-fezhλ-oteh(x#c)。该线性表达载体通过arg4 3’和5’同源序列重组整合在arg4基因座处。转化的细胞在补充了300mg/l杀稻瘟菌素的ypd平板上生长,如此产生了表达菌株文库gs2-fezhλ-oteh(x#c)λ(x#c)。它们可以表达不对称双特异性抗体,这些双特异性抗体的一半是otelixizumab重链和轻链,其fab臂的ch1和cl结构域中具有工程改造的链间二硫键,在fc中具有“旋钮”突变。另一半是fezakinumab重链和轻链,其fab臂的ch1和cl结构域中具有天然的链间二硫键,在fc中具有“孔”和rf突变,在重链的c末端具有6xhis标签。
[0270]
如实施例2中所述,表达菌株在bmgy培养基中培养,在bmmy培养基中诱导表达抗体。离心收获的上清液在-20℃冷冻直至下一步使用。
[0271]
实施例8.筛选鉴定igg1(lambda)“半胱氨酸突变”文库
[0272]
如实施例3中所述,双特异性抗体通过a蛋白亲和层析纯化,纯化的抗体浓度通过elisa测定。
[0273]
如实施例3所述,将a蛋白纯化的双特异性抗体用pbs作1:2梯度稀释后分为两部分,一部分用agl蛋白包被的平板通过elisa测定抗体浓度,另一部分用人白介素22(il22)(sino biological)包被的平板通过elisa测定抗体与抗原结合情况。
[0274]
根据报道,当otelixizumab重链ch1中126位苯丙氨酸突变为半胱氨酸(f126c,eu
编号),lambda轻链cl中121位丝氨酸突变为半胱氨酸(s121c,kabat编号)时,可以形成工程改造的链间二硫键,使重链和轻链正确配对。本文中将该不对称的双特异性抗体命名为fezhλ-oteh(f126c)λ(s121c),简记为f126c/s121c,并用作阳性对照。然而,当两个fab臂中均具有天然链间二硫键时,形成的双特异性抗体只有25%的重链和轻链正确配对。本文中将该双特异性抗体命名为fezhλ-otehλ,简记为wt,并用作阴性对照(yariv mazor,vaheh oganesyan,chunning yang,anna hansen,jihong wang,hongji liu,kris sachsenmeier,marcia carlson,dhanesh v gadre,martin jack borrok,xiang-qing yu,william dall’acqua,herren wu,and partha sarathi chowdhury,mabs 7,377
--
389;2015)。
[0275]
如实施例3中所述,采用elisa方法鉴定双特异性抗体中的工程改造的链间二硫键,该二硫键可以促进重链和轻链的正确配对。
[0276]
如图9所示,在相同的抗体浓度条件下(横轴elisa吸光度值),双特异性抗体阳性对照f126c/s121c显示很高的抗体/抗原结合elisa吸光度值(纵轴),但双特异性抗体阴性对照wt显示很低的抗体/抗原结合elisa吸光度值。在相同的抗体浓度条件下(横轴elisa吸光度值),我们构建的许多双特异性抗体与阳性对照f126c/s121c具有相同或更高的抗体/抗原结合吸光度值。这表明我们的双特异性抗体的重链和轻链能够正确配对。因此,在igg1(lambda)双特异性抗体生产中,通过将天然的链间二硫键替换为不同的半胱氨酸配对,在一个fab臂的ch1-cl结构域形成工程改造的二硫键,可以实现重链和轻链的正确配对,包括:
[0277]
s132c/s121c、k133c/t116c、k133c/p211c、s136c/s121c、f170c/g158c、p171c/t162c、p171c/p164c、s176c/t162c、l179c/g158c、s181c/p164c、v215c/t116c、e216c/f118c
[0278]
(每对半胱氨酸突变按以下方式列出:重链野生氨基酸[eu编号]突变为半胱氨酸/lambda轻链野生氨基酸[kabat编号]突变为半胱氨酸)。
[0279]
实施例9.igg1(lambda)“半胱氨酸和电荷突变”文库的构建和表达
[0280]
pcr 1,eg f和eg r引物对用于pcr扩增peg线性片段,用peg载体作为模板(如实施例2中所述)。“[0281]
pcr 2,使用表14-1中对应的引物对分别进行n端和c端重链的pcr扩增,用otelixizumab突变体文库peg-oteh(x#c)的各表达载体作为模板。用oteh-nt和oteh-ct引物对,通过重叠延伸pcr连接n端和c端重链,如此产生了突变的otelixizumab重链,其包含t366w“旋钮”突变、c220v突变、野生氨基酸突变为半胱氨酸以及k213d突变。
[0282]
表14-1 otelixizumab重链n端和c端pcr扩增引物对
[0283][0284]
pcr 3,使用表14-2中对应的引物对分别进行n端和c端重链的pcr扩增,用otelixizumab突变体文库peg-oteh(x#c)的各表达载体作为模板。用oteh-nt和oteh-ct引物对,通过重叠延伸pcr连接n端和c端重链,如此产生了突变的otelixizumab重链,其包含t366w“旋钮”突变、c220v突变、野生氨基酸突变为半胱氨酸以及k213e突变。
[0285]
表14-2 otelixizumab重链n端和c端pcr扩增引物对
[0286][0287]
使用clonexpress ii一步克隆试剂盒(vazyme)将突变的otelixizumab重链插入peg线性片段,以构建otelixizumab突变体表达载体文库,命名为peg-oteh(x#cd)和peg-oteh(x#ce)。
[0288]
pcr 4,使用表15中对应的引物对分别进行n端和c端lambda轻链的pcr扩增,用otelixizumab突变体文库ppic6α-arg4-otehλ(x#c)的各表达载体作为模板。用oteλ-nt和oteλ-ct引物对,通过重叠延伸pcr连接n端和c端lambda轻链,如此产生了突变的otelixizumab lambda轻链,其包含t366w“旋钮”突变、c220v突变、野生氨基酸突变为半胱氨酸以及e123k突变。
[0289]
表15 otelixizumab轻链n端和c端pcr扩增引物对
[0290][0291]
用xho i和not i消化突变的otelixizumab lambda轻链,并插入ppic6α-arg4的相同消化位点,以生成lambda轻链突变体表达载体文库,命名为ppic6α-arg4-oteλ(x#ck)。各载体可以表达突变的otelixizumab lambda轻链,包含c214v突变、野生氨基酸突变为半胱氨酸以及e123k。
[0292]
用限制酶pme i将otelixizumab重链突变体文库peg-oteh(x#cd)的各表达载体线性化,电穿孔到gs2-fezhλ菌株中,并整合在毕赤酵母基因组的aox1基因座。在补充了250mg/l g-418硫酸盐的ypd平板上选择转化的细胞,如此产生了表达菌株文库(gs2-fezhλ-oteh(x#cd)。
[0293]
用限制酶sma i将otelixizumab lambda轻链突变体文库ppic6α-arg4-oteλ(x#ck)的各表达载体在arg4 3’和5’同源序列中线性化,电穿孔入表达菌株文库gs2-fezhλ-oteh(x#cd),并整合到arg4基因座。转化的细胞在补充了300mg/l杀稻瘟菌素的ypd平板上生长,如此产生了表达菌株文库gs2-fezhλ-oteh(x#cd)λ(x#ck),用于表达不对称双特异性抗体。在不对称双特异性抗体中,一半是otelixizumab重链和轻链,其fab臂的ch1-cl结构域具有工程改造的链间二硫键和一对电荷反转突变,并在fc中具有“旋钮”突变;另一半是fezakinumab重链和轻链,其fab臂的ch1-cl结构域具有天然的链间二硫键,在fc中具有“孔”和rf突变,在重链c末端具有6xhis标签,如实施例2中所述,表达菌株在bmgy培养基中培养,在bmmy培养基中诱导表达抗体。离心收获的上清液在-20℃冷冻直至下一步使用。
[0294]
实施例10.筛选鉴定igg1(lambda)“半胱氨酸和电荷突变”文库
[0295]
如实施例3所述,将a蛋白纯化的双特异性抗体用pbs作1:2梯度稀释后分为两部分,一部分用agl蛋白包被的平板通过elisa测定抗体浓度,另一部分用人白介素22(il22)(sino biological)包被的平板通过elisa测定抗体与抗原结合情况。
[0296]
如图10所示,在相同的抗体浓度条件下(横轴elisa吸光度值),阳性对照f126c/s121c具有很高的抗体/抗原结合elisa吸光度值(纵轴),但阴性对照wt具有很低的抗体/抗原结合elisa吸光度值。在一个fab臂的ch1-cl结构域含有半胱氨酸突变的双特异性抗体具有与阳性对照f126c/s121c相似或更低的抗体/抗原结合吸光度值。含有半胱氨酸和k213d/e123k的电荷突变的相应双特异性抗体具有高于阳性对照f126c/s121c的抗体/抗原结合吸光度值。因此,在igg lambda双特异性抗体的生产中,为了进一步实现重链和轻链的正确配对,形成工程改造的二硫键,可以在一个fab臂的ch1-cl结构域组合k213d/e123k电荷突变
k213e r引物对以及clah-k213e f和trah r引物对进行n端和c端重链的pcr扩增。利用trah f和trah r引物,通过重叠延伸pcr连接n端和c端重链。
[0308]
pcr 1和2产生突变的曲妥珠单抗重链,其ch1结构域包含k213d或k213e突变,在fc域中包含t366s/l368a/y407v“孔”突变、h435r、y436f(rf)突变、在c端包含6xhis标签。
[0309]
用xho i和not i消化突变的曲妥珠单抗重链,插入ppic9(invitrogen)的相同消化位点,以构建曲妥珠单抗重链的表达载体,命名为ppic9-trah(d)和ppic9-trah(e)。两种载体均可表达突变的曲妥珠单抗重链,其在ch1结构域中包含k213d或k213e突变,在fc结构域中包含t366s/l368a/y407v“孔”突变、h435r、y436f(rf)突变和在c末端包含6xhis标签。
[0310]
pcr 3,用合成的曲妥珠单抗kappa链为模板,用traκf和claκ-e123k r引物对以及claκ-e123k f和traκr引物对pcr扩增n端和c端kappa链。利用traκf和traκr引物对,通过重叠延伸pcr连接n末端和c末端的kappa链。
[0311]
pcr 4,利用合成的曲妥珠单抗kappa链作为模板,用traκf和claκ-e123r r引物对以及claκ-e123r f和traκr引物对pcr扩增n末端和c末端kappa链。利用traκf和traκr引物对,通过重叠延伸pcr连接n末端和c末端的kappa链。
[0312]
用xho i和not i消化pcr 3和4产物,插入ppiczα-traκ-arg2的相同消化位点,以构建曲妥珠单抗kappa链的表达载体,命名为ppiczα-traκ(k)-arg2和ppiczα-traκ(r)-arg2。两种载体都可以表达突变的曲妥珠单抗kappa链,其在cl结构域中包含e123k或e123r突变。
[0313]
如实施例2所述,用afe i线性化ppiczα-traκ(k)-arg2和ppiczα-traκ(r)-arg2的表达载体,电穿孔入gs2-1中并整合在arg2基因座上。转化的细胞在补充了100mg/l zeocin的ypd平板上生长。如此产生了新的表达菌株gs2-traκ(k)和gs2-traκ(r),它们可以表达曲妥珠单抗kappa轻链。
[0314]
用sal i线性化ppic9-trah(d)和ppic9-trah(e)的表达载体,电穿孔入gs2-traκ(k)和gs2-traκ(r)菌株,并整合到his4基因座。在ynb板上选择转化的细胞。如此产生了gs2-trah(d)κ(k)、gs2-trah(d)κ(r)、gs2-trah(e)κ(k)和gs2-trah(e)κ(r)的菌株以表达突变的曲妥珠单抗。
[0315]
用pme i线性化clazakizumab重链突变体文库peg-clah(x#c)的某些表达载体,电穿孔入gs2-trah(d)κ(k)、gs2-trah(d)κ(r)、gs2-trah(e)κ(k)和gs2-trah(e)κ(r)的菌株,并整合在aox1基因座上。在补充了250mg/l g-418硫酸盐的ypd平板上选择转化的细胞。如此产生了表达菌株文库:
[0316]
gs2-trah(d)κ(k)-clah(x#c)、
[0317]
gs2-trah(d)κ(r)-clah(x#c)、
[0318]
gs2-trah(e)κ(k)-clah(x#c)、
[0319]
gs2-trah(e)κ(r)-clah(x#c)。
[0320]
用sma i线性化clazakizumab kappa链突变体文库ppic6-arg4-claκ(x#c)的表达载体,电穿孔到上述表达菌株文库中,并整合到arg4基因座。转化的菌株在补充了300mg/l杀稻瘟菌素的ypd平板上生长。如此这产生了表达菌株文库:
[0321]
gs2-trah(d)κ(k)-clah(x#c)κ(x#c)、
[0322]
gs2-trah(d)κ(r)-clah(x#c)κ(x#c)、
[0323]
gs2-trah(e)κ(k)-clah(x#c)κ(x#c)、
[0324]
gs2-trah(e)κ(r)-clah(x#c)κ(x#c)。
[0325]
如实施例2中所述,表达菌株在bmgy培养基中培养,在bmmy培养基中诱导表达抗体。离心收获的上清液在-20℃冷冻直至下一步使用。
[0326]
如实施例3所述,将a蛋白纯化的双特异性抗体用pbs作1:2梯度稀释后分为两部分,一部分用agl蛋白包被的平板通过elisa测定抗体浓度,另一部分用人her2/erbb2蛋白包被的平板通过elisa测定抗体与抗原结合情况。
[0327]
利用以下三组双特异性抗体为代表性示例:
[0328]
trah(d)κ(k)-clah(s219c)κ(p120c),简记为s219c/p120c,k213d/e123k;
[0329]
trah(e)κ(k)-clah(s219c)κ(p120c),简记为s219c/p120c,k213e/e123k;
[0330]
trah(d)κ(r)-clah(s219c)κ(p120c),简记为s219c/p120c,k213d/e123r;
[0331]
trah(e)κ(r)-clah(s219c)κ(p120c),简记为s219c/p120c,k213e/e123r;
[0332]
trah(d)κ(k)-clah(k218c)κ(f118c),简记为k218c/f118c,k213d/e123k;
[0333]
trah(e)κ(k)-clah(k218c)κ(f118c),简记为k218c/f118c,k213e/e123k;
[0334]
trah(d)κ(r)-clah(k218c)κ(f118c),简记为k218c/f118c,k213d/e123r;
[0335]
trah(e)κ(r)-clah(k218c)κ(f118c),简记为k218c/f118c,k213e/e123r,
[0336]
trah(d)κ(k)-clah(v173c)κ(n158c),简记为v173c/n158c,k213d/e123k;
[0337]
trah(e)κ(k)-clah(v173c)κ(n158c),简记为v173c/n158c,k213e/e123k;
[0338]
trah(d)κ(r)-clah(v173c)κ(n158c),简记为v173c/n158c,k213d/e123r;
[0339]
trah(e)κ(r)-clah(v173c)κ(n158c),简记为v173c/n158c,k213e/e123r。
[0340]
在这些不对称双特异性抗体中,一半是clazakizumab重链和kappa轻链,其在fab臂ch1-cl结构域中包含一对新的半胱氨酸(s219c/p120c、k218c/f118c或v173c/n158c),在“fc中包含“旋钮”突变;另一半是曲妥珠单抗重链和轻链,在其fab臂中保留了天然链间二硫键,但fab臂ch1包含k213d或k213e的电荷突变,在fab臂cl中包含e123k或e123r的电荷突变,在fc中包含“孔”和rf突变,在重链c末端包含6xhis标签,
[0341]
如图12所示,在相同抗体浓度条件下(横轴elisa吸光度值),所有这些双特异性抗体比阳性对照f126c/s121c具有更高的抗体/抗原结合吸光度值(纵轴elisa吸光度值)。
[0342]
这些实例证明,在igg双特异性抗体生产中,通过一个fab臂的ch1-cl结构域的电荷突变,在另一fab臂的ch1-cl结构域中半胱氨酸对突变形成工程改造的二硫键取代天然链间二硫键,可以实现重链和轻链的正确配对。
[0343]
实施例13.设计igg4(kappa)ch1和cl结构域“半胱氨酸突变”文库
[0344]
虽然igg1、igg2、igg3和igg4的ch1结构域氨基酸序列和空间结构非常相近,但是形成天然二硫键的半胱氨酸对位置却有所不同。igg1的轻链通过kappa和lambda链的cl结构域c端的半胱氨酸(κ轻链214位的半胱氨酸[eu编号];λ轻链214位的半胱氨酸[kabat编号])和重链ch1结构域c端的半胱氨酸(重链220位的半胱氨酸[eu编号])形成链间二硫键。相比之下,igg2、igg3或igg4的轻链通过kappa和lambda链中cl结构域c端的半胱氨酸与重链中ch1结构域n端的半胱氨酸(重链131位的半胱氨酸[eu编号])形成链间二硫键(图1,a和b)。尽管在igg1和其他亚型(igg2、igg3、igg4)之间,半胱氨酸在氨基酸序列中的位置不同,但它们的空间位置相似,以形成链间二硫键。为了了解igg2、igg3或igg4可能形成其它二硫
键的位置,我们用来自蛋白质数据库的人igg4(kappa)的fab晶体结构(pdb代码:5dk3)作为代表性结构,分析和设计了ch1和cl结构域中可能相互作用形成链间二硫键的半胱氨酸突变位点,设计了igg4(kappa)突变体文库,其中形成天然链间二硫键的重链和轻链半胱氨酸对分别突变为丝氨酸和缬氨酸,并在ch1-cl结构域的不同位置引入新的半胱氨酸对以形成工程改造的链间二硫键。表16列出在igg4(kappa)的ch1-cl结构域的不同氨基酸位置引入新的半胱氨酸对,这些半胱氨酸对可能形成工程改造的链间二硫键。
[0345]
表16.igg4(kappa)ch1-cl结构域的半胱氨酸突变文库。半胱氨酸突变聚集的区域称为一组。ch1结构域与cl结构域可能形成链间二硫键各组分别对应列出。
[0346][0347][0348]
实施例14.igg4(kappa)半胱氨酸突变文库的构建和表达
[0349]
包含t366s/l368a/y407v“孔”突变、h435r、y436f(rf)突变(eu编号)和在c端的6xhis标签的ixekizumab(il-17a抗体)重链(holerf-his)用作igg4重链的代表(seq id no:202)。合成ixekizumab重链密码子优化dna(holerf-his),并用作pcr扩增的模板(seq id no:203)。
[0350]
pcr 1,ixeh f(seq id no:204,引物具有xho i限制酶位点)和ixeh r(seq id no:205,引物具有not i限制酶位点)引物对用于ixekizumab重链(holerf-his)的pcr扩增,使用合成的dna作为模板。用xho i和not i消化pcr产物,并插入ppic9(invitrogen)的相同消化位点,以构建ixekizumab重链(holerf-his)的表达载体,命名为ppic9-ixeh(holerf-his)。
[0351]
ixekizumab轻链用作igg4 kappa(κ)轻链的代表(seq id no:206)。合成ixekizumab kappa轻链的密码子优化dna(seq id no:207),并将其用作pcr扩增的模板。
[0352]
pcr 2 ixeκf(seq id no:208,该引物具有xho i限制酶位点)和ixeκr(seq id no:209,该引物具有not i限制酶位点)引物对用于ixekizumab kappa轻链的pcr扩增,利用合成的ixekizumab轻链作为模板。将pcr产物用xho i和not i消化,并插入ppiczα-traκ-arg2(如实施例2中所述)的相同消化位点,以构建ixekizumab轻链的表达载体,命名为ppiczα-ixeκ-arg2。
[0353]
pcr 3,eg f和eg r引物对用于pcr扩增peg线性片段,以peg载体作为模板。
[0354]
将含有t366w“旋钮”突变和c131s突变[eu编号]的olokizumab(il6单抗)重链(旋
钮)用作igg4重链(seq id no:210)的另一个代表。合成olokizumab重链(旋钮)的密码子优化dna(seq id no:211),并用作pcr扩增的模板。
[0355]
pcr 4,重链n端引物oloh-nt(seq id no:212)和c端引物oloh-ct(seq id no:213)分别与表17中对应的反向引物(r)和正向引物(f)组成不同的引物对用于pcr扩增n端和c端重链,利用合成的olokizumab重链(旋钮)为模板。
[0356]
表17.clazakizumab重链中野生氨基酸突变为半胱氨酸的pcr引物(eu编号)
[0357][0358][0359]
pcr 5,使用oloh-nt和oloh-ct引物对,通过重叠延伸pcr连接n端和c端重链的pcr产物。以此方式产生了olokizumab重链突变体,其包含野生氨基酸突变为半胱氨酸,t366w“旋钮”突变和c131s突变。其中一种含有t366w“旋钮”突变和134位野生半胱氨酸,能形成天然链间二硫键。
[0360]
pcr 6,重链n端引物oloh-nt和oloh-k147d r(seq id no:246)引物对以及oloh-k147d f(seq id no:247)和c端引物oloh-ct引物对分别用于pcr扩增n端和c端重链,利用含有t366w“旋钮”突变和134位野生半胱氨酸的重链为模板。
[0361]
pcr 7,用oloh-nt和oloh-ct引物对,通过重叠延伸pcr连接n端和c端重链的pcr产物。以此方式产生了olokizumab重链突变体,其包含k147d突变,t366w“旋钮”突变和134位野生半胱氨酸,能形成天然链间二硫键
[0362]
使用clonexpress ii一步克隆试剂盒(vazyme)将olokizumab重链突变体插入peg线性片段,生成表达载体文库,命名为peg-oloh(x#c)(oloh:olokizumab重链,(x#c):在#位的野生氨基酸突变为半胱氨酸)。其中一种表达载体为peg-oloh(k147d)(oloh:olokizumab
重链,k147d:147位赖氨酸突变为天冬氨酸)。
[0363]
将含有c214v突变的olokizumab kappa(κ)轻链用作igg4 kappa轻链的另一个代表(seq id no:248)。合成olokizumab kappa轻链密码子优化dna(seq id no:249),并将其用作pcr扩增的模板。
[0364]
pcr 8,轻链n端引物oloκ-nt(seq id no:250,该引物具有xho i限制酶位点)和轻链c端引物oloκ-ct(seq id no:251,该引物具有not i限制酶位点)分别与表18中对应的反向引物(r)和正向引物(f)组成不同的引物对,用于pcr扩增n端和c端kappa轻链,用合成的olokizumab kappa链作为模板。
[0365]
表18.olokizumab kappa(κ)轻链中野生氨基酸突变为半胱氨酸的pcr引物[eu编号]
[0366]
突变反向引物(r)正向引物(f)oloκ-s114coloκ-s114c r(seq id no:252)oloκ-s114c f(seq id no:253)oloκ-f116coloκ-s114c r(seq id no:252)oloκ-f116c f(seq id no:254)oloκ-f118coloκ-s114c r(seq id no:252)oloκ-f118c f(seq id no:255)oloκ-p119coloκ-s114c r(seq id no:252)oloκ-p119c f(seq id no:256)oloκ-p120coloκ-s114c r(seq id no:252)oloκ-p120c f(seq id no:257)oloκ-s121coloκ-s114c r(seq id no:252)oloκ-s121c f(seq id no:258)oloκ-n158coloκ-n158c r(seq id no:259)oloκ-n158c f(seq id no:260)oloκ-q160coloκ-n158c r(seq id no:259)oloκ-q160c f(seq id no:261)oloκ-s162coloκ-n158c r(seq id no:259)oloκ-s162c f(seq id no:262)oloκ-v163coloκ-n158c r(seq id no:259)oloκ-v163c f(seq id no:263)oloκ-t164coloκ-n158c r(seq id no:259)oloκ-t164c f(seq id no:264)oloκ-e165coloκ-n158c r(seq id no:259)oloκ-e165c f(seq id no:265)oloκ-t172coloκ-t172c r(seq id no:266)oloκ-t172c f(seq id no:267)oloκ-s174coloκ-t172c r(seq id no:266)oloκ-s174c f(seq id no:268)oloκ-s176coloκ-t172c r(seq id no:266)oloκ-s176c f(seq id no:269)oloκ-t178coloκ-t172c r(seq id no:266)oloκ-t178c f(seq id no:270)oloκ-t180coloκ-t172c r(seq id no:266)oloκ-t180c f(seq id no:271)oloκ-s182coloκ-t172c r(seq id no:266)oloκ-s182c f(seq id no:272)oloκ-f209coloκ-f209c r(seq id no:273) oloκ-n210coloκ-n210c r(seq id no:274) oloκ-r211coloκ-r211c r(seq id no:275) oloκ-g212coloκ-g212c r(seq id no:276) oloκ-e213coloκ-e213c r(seq id no:277) oloκ-v214coloκ-v214c r(seq id no:278) [0367]
pcr 9,使用oloκ-nt和oloκ-ct引物对,通过重叠延伸pcr连接n端和c端轻链的pcr产物。以此方式,产生了包含野生氨基酸突变为半胱氨酸和c214v突变的olokizumab kappa轻链突变体。可以使用oloκ-nt和表18中对应的反向引物(r)直接pcr扩增olokizumab kappa轻链,使c端野生氨基酸突变为半胱氨酸。使用oloκ-nt和oloκ-v214c反向引物(r)直
接pcr扩增,生成在214位具有野生型半胱氨酸的olokizumab kappa轻链,能形成天然链间二硫键。
[0368]
pcr 10,轻链n端引物oloκ-nt和oloκ-t129r r(seq id no:279)引物对以及oloκ-t129r f(seq id no:280)和c端引物oloκ-ct引物对分别用于pcr扩增n端和c端轻链,用在214位具有野生型半胱氨酸的olokizumab kappa轻链作为模板。
[0369]
pcr 11,用oloκ-nt和oloκ-ct引物对,通过重叠延伸pcr连接n端和c端重链的pcr产物。以此方式产生了olokizumab轻链突变体,其包含t129r突变,和214位野生半胱氨酸,能形成天然链间二硫键
[0370]
olokizumab kappa轻链突变体用xho i和not i消化,并插入ppic6α-arg4的相同消化位点以生成轻链突变体表达载体文库,命名为ppic6-arg4-oloκ(x#c)(oloκ:olokizumab kappa轻链,(x#c):编号#位野生氨基酸突变为半胱氨酸)。
[0371]
如实施例2所述,用限制酶afe i消化arg2同源序列,将ppiczα-ixeκ-arg2表达载体线性化,电穿孔入gs2-1,并通过arg2 5’和3’同源序列重组整合到arg2基因座上。转化的细胞在补充了100mg/l zeocin的ypd平板上生长。如此产生了新的表达菌株gs2-ixeκ。
[0372]
用限制酶sal i将ppic9-ixeh(holerf-his)表达载体线性化,电穿孔到gs2-ixeκ菌株中,并整合到毕赤酵母基因组的his4基因座。在ynb板上选择转化的细胞。如此产生了新的表达菌株gs2-ixehκ。
[0373]
用限制酶pme i将olokizumab重链突变体文库peg-oloh(x#c)的各表达载体线性化,电穿孔入gs2-ixehκ菌株中,并整合到毕赤酵母基因组的aox1基因座。在补充了250mg/l g-418硫酸盐的ypd平板上选择转化的细胞。如此产生了表达菌株文库gs2-ixehκ-oloh(x#c)。
[0374]
用限制酶sma i将olokizumab kappa轻链突变体文库ppic6-arg4-oloκ(x#c)的各表达载体在arg4 3’和5’同源序列内线性化,电穿孔入表达菌株文库gs2-ixehκ-oloh(x#c)。该线性表达载体通过arg4 3’和5’同源序列重组整合在arg4基因座处。转化的细胞在补充了300mg/l杀稻瘟菌素的ypd平板上生长,如此产生了表达菌株文库gs2-ixehκ-oloh(x#c)κ(x#c)。它们可以表达不对称双特异性抗体,这些双特异性抗体的一半是olokizumab重链和轻链,其fab臂的ch1和cl结构域中具有工程改造的链间二硫键,在fc中具有“旋钮”突变。另一半是ixekizumab重链和轻链,其fab臂的ch1和cl结构域中具有天然的链间二硫键,在fc中具有“孔”和rf突变,在重链的c末端具有6xhis标签。
[0375]
如实施例2中所述,表达菌株在bmgy培养基中培养,在bmmy培养基中诱导表达抗体。离心收获的上清液在-20℃冷冻直至下一步使用。
[0376]
实施例15.筛选鉴定igg4(kappa)半胱氨酸突变文库
[0377]
如实施例3所述,将a蛋白纯化的双特异性抗体用pbs作1:2梯度稀释后分为两部分,一部分双特异性抗体直接包被平板,用抗fc-hrp(invitrogen)通过elisa测定抗体浓度,另一部分用人白介素17a(il17a)(sino biological)包被的平板通过elisa测定抗体与抗原结合情况。
[0378]
根据报道,当olokizumab重链ch1中147位赖氨酸突变为天冬氨酸(k147d),kappa轻链cl中129位苏氨酸突变为精氨酸(t129r)[eu编号],可以促进重链和轻链正确配对达到80-99%。本文中将该不对称的双特异性抗体命名为ixehκ-oloh(k147d)κ(t129r),简记为
k147d/t129r,并用作阳性对照。(maximilian carolin sellmann,daniel maresch,claudia halbig,stefan becker,lars toleikis,hock,and florian r
ü
ker,protein engineering,design&selection 30,685
–
696,2017)。我们发现文献报道的f126c/s121c半胱氨酸对突变在igg4(kappa)中未能有效地形成链间二硫键,无法用作正对照。
[0379]
如实施例3中所述,采用elisa方法鉴定双特异性抗体中的工程改造的链间二硫键,用于促进重链和轻链的正确配对。
[0380]
如图13所示,在相同的抗体浓度条件下(横轴elisa吸光度值),双特异性抗体阳性对照(k147d/t129r)显示很高的抗体/抗原结合elisa吸光度值(纵轴)。在相同的抗体浓度条件下(横轴elisa吸光度值),我们构建的许多双特异性抗体与阳性对照k147d/t129r具有相同或更高的抗体/抗原结合吸光度值。这表明我们的双特异性抗体的重链和轻链能够正确配对。因此,在igg4(kappa)双特异性抗体生产中,通过将天然的链间二硫键替换为不同的半胱氨酸配对,在一个fab臂的ch1-cl结构域形成工程改造的二硫键,可以实现重链和轻链的正确配对,包括:
[0381]
a129c/f209c、a129c/n210c、p130c/f116c、p130c/f118c、p130c/p119c、p130c/n210c、p130c/r211c、s132c/s114c、s132c/f116c、s132c/f118c、s132c/p120c、s132c/r211c、s132c/e213c、r133c/p119c、r133c/r211c、r133c/e213c、t135c/f116c、t135c/p120c、g166c/t178c、h168c/n158c、f170c/f182c、v173c/q160c、v173c/s162c、q175c/s162c、q175c/t180c、s181c/t172c、s181c/s176c、s183c/n158c、s183c/s176c、v185c/e165c、v185c/t178c
[0382]
(每对半胱氨酸突变按以下方式列出:igg4重链野生氨基酸[eu编号]突变为半胱氨酸/kappa轻链野生氨基酸[eu编号]突变为半胱氨酸)。
[0383]
实施例16.设计igg4(lambda)ch1和cl结构域链“半胱氨酸突变”文库
[0384]
我们用来自蛋白质数据库的人igg2(lambda)的fab晶体结构(pdb代码:5gks)作为代表性结构,分析和设计了ch1和cl结构域中可能相互作用形成链间二硫键的半胱氨酸突变位点,设计了igg4(kappa)突变体文库,其中形成天然链间二硫键的重链和轻链半胱氨酸对分别突变为丝氨酸和缬氨酸,并在ch1-cl结构域的不同位置引入新的半胱氨酸对以形成工程改造的链间二硫键。表19列出在igg4(lambda)的ch1-cl结构域的不同氨基酸位置引入新的半胱氨酸对,这些半胱氨酸对可能形成工程改造的链间二硫键。
[0385]
表19.igg4(lambda)ch1-cl结构域的半胱氨酸突变文库。半胱氨酸突变聚集的区域称为一组。ch1结构域与cl结构域可能形成链间二硫键各组分别对应列出。
[0386][0387]
本发明中所用的序列如下所示:
[0388]
seq id no:1
[0389]
evqlvesggglvqpggslrlscaasgfnikdtyihwvrqapgkglewvariyptngytryadsvkgrftisadtskntaylqmnslraedtavyycsrwggdgfyamdywgqgtlvtvssastkgpsvfplapsskstsggtaalgclvkdyfpepvtvswnsgaltsgvhtfpavlqssglyslssvvtvpssslgtqtyicnvnhkpsntkvdkkvepkscdkthtcppcpapellggpsvflfppkpkdtlmisrtpevtcvvvdvshedpevkfnwyvdgvevhnaktkpreeqynstyrvvsvltvlhqdwlngkeykckvsnkalpapiektiskakgqprepqvytlppsreemtknqvslscavkgfypsdiavewesngqpennykttppvldsdgsfflvskltvdksrwqqgnvfscsvmhealhnrftqkslslspghhhhhh
[0390]
seq id no:2
[0391]
gaggtccagttggtcgagtctggtggtggtttggttcagccaggtggatctttgagattgtcttgtgcagcatctggattcaacattaaagatacttacattcattgggttagacaggcaccaggtaaaggtttggagtgggttgctagaatttacccaactaacggttacactagatacgctgattctgtcaagggtagattcactatttctgctgatacatctaaaaacactgcttacttgcaaatgaactctttgagagctgaagatacagccgtttactattgctccagatggggaggagacggattttacgctatggattactggggacaaggtactttggttactgtttcttctgcttctactaagggaccatctgtctttccattggcaccatcttctaaatctacttctggaggaactgccgccttgggatgtttggtcaaggattatttccctgagcctgtcacagtttcttggaactctggtgcattgacatctggtgttcacacattcccagcagtcttgcaatcttctggtttgtactctttgtcttctgtcgttacagtcccttcttcttctttgggtactcaaacctacatctgtaatgttaatcataagccttctaacacaaaagttgataagaaagtcgagccaaaatcttgcgacaaaacacatacctgcccaccatgtccagctcctgagttgttgggaggtccttctgtttttttgttcccacctaagccaaaggatacattgatgatttccagaactcctgaagttacatgtgtcgttgtcgatgtttctcatgaagatcctgaggttaaattcaattggtacgttgatggtgtcgaagttcataacgctaagactaaaccaagagaagaacagtacaattctacttatagagtcgtctctgttttgaccgttttgcatcaagattggttgaacggaaaggagtataagtgcaaagtttctaataaggctttgcctgcccctattgagaagaccatttctaaggctaaaggtcagcctagagaacctcaagtttacactttgccaccttccagagaggaaatgactaagaaccaggtctctttgtcttgcgctgttaagggtttttacccttctgacatcgctgttgagtgggaatctaacggacagcctgaaaataactataaaacaacacctccagttttggattctgacggttctttctttttggtttctaagttgacagtcgataagtccagatggcaacaaggaaacgtcttttcttgt
tctgttatgcatgaagccttgcataatagatttacccagaaatctttgtctttgtctccaggtcatcatcatcatcatcat
[0392]
seq id no:3
[0393]
ccgctcgagaaaagagaggctgaagctgaggtccagttggtcgagtct
[0394]
seq id no:4
[0395]
ataagaatgcggccgctcaatgatgatgatgatgatgacctggagacaaagacaaaga
[0396]
seq id no:5
[0397]
diqmtqspsslsasvgdrvtitcrasqdvntavawyqqkpgkapklliysasflysgvpsrfsgsrsgtdftltisslqpedfatyycqqhyttpptfgqgtkveikrtvaapsvfifppsdeqlksgtasvvcllnnfypreakvqwkvdnalqsgnsqesvteqdskdstyslsstltlskadyekhkvyacevthqglsspvtksfnrgec
[0398]
seq id no:6
[0399]
gatattcaaatgactcaatctccttcttctttgtctgcttctgtcggagatagagtcactatcacttgtagagcttctcaagacgtcaacaccgctgttgcttggtatcagcaaaagccaggtaaggctccaaaattgttgatttactctgcatcttttttgtactctggtgtcccttccagattctctggttctaggtctggaaccgattttactttgactatctcttctttgcagcctgaagatttcgcaacttactactgtcaacagcattacactactccaccaacttttggtcaaggaactaaagttgagattaagagaactgtcgctgcaccttctgttttcatttttcctccatctgacgagcagttgaagtctggtacagcatctgttgtctgtttgttgaacaacttctacccaagagaagcaaaggttcaatggaaggttgacaacgccttgcaatctggtaactctcaggaatctgttactgaacaagattctaaggattctacttactctttgtcttctacattgacattgtctaaggctgattacgaaaagcataaggtttacgcttgtgaagttactcatcaaggattgtcttctcctgtcacaaaatcttttaacagaggtgagtgt
[0400]
seq id no:7
[0401]
ccgctcgagaaaagagaggctgaagctgatattcaaatgactcaatctccttc
[0402]
seq id no:8
[0403]
ataagaatgcggccgctcaacactcacctctgttaaaaga
[0404]
seq id no:9
[0405]
agacgagatcaattagaacctgttttggcaataaccgaggattaggaacaaagtccgggtaattatgcgactctctctttttcatatgcaggtcgagcaagaatcttgtttttcgtggtgggactgggcagcaattaacaaccaatcggcacttgcaataagtcgattagcgcatggtgggggacagtagatagctgctgaaatttttgggtgcggacaatttcaagagtctgaggccctgctctcactagcagtcagaatcatctgcctcacatacagtctcctgatgctgctacttacttggcaagcgatgatgtttacacaattgcagctttttagttgctgtcatggctctagaattccttggccttttatcagcatctcaggcaacaatgtgaaatgcgcaccttcagatttttatattggtggacggaaatggtaggaagtagtgaagaaaaggtagcgcgagggcattgtccccccatgcgaagaaaaattactagcataaaaaaatggaaacaagtgacctccgcagcatgtctgcccactttatactgaaactacattcttgctaatggatcgtaaaacactatcacctggtatgtcgtaatggacttgatggcgtgtactttgctttactgagttctagttgcctgctcagtgccgaagaacatggggctccatgtgtattgtattctcttatctagaacaatcgaagcgctactgacttgaaatccttgaatgatagacatagattggaccatgacaaggagaacttattcacaattgaaaagtacattttcattgacccattgggaggtatcccatctttggagaggtacaaaagtgcacatgtatatatcaatctattacaagagtatgaagacattgtgtcggagctttacatagggttcttaaagactggagaaagggaccagcatttgaagaacttaaacttgcttcagaaattgttgcaggtaaccacagatgcatcaggaatagttactactcctcaaatcgccatgttgaatcagaccgaccgattcaccaatccaataatttacaatgtc
ttaaccgataggccgacaatatcatcgtcattaccggttgatttgaaaaagacccctttgctaaacacttcaatcattaggagaggcgtaccggttgaagtttatgtggacgaatcatctgacaaaagtgggctgtgcctagactctcttctgaaacgaggagctttagacttagaaaagcttaagaatgtgatcgatttgtcgtttcgaaaggacttgaatatgaaaaagtacctagccagagtaaagaacaatgttgcagctatcttaatcgctggagattacgaaggcgtgatcatagttacttgggaggtaacggatgaagaaaagccgcagaaaatagcttatttagataagtttgcagtgtctcctaaggcccaaggatcgacaggggttgccgatgttcttttcaagtcattattgtccaattttgagaacgaattgttctggagatctcgatctaataatccagtgaacaaatggtactttgaacggagcaaaggttctcttactgttactggcacaaattggaaatgcttctacaccggcaagaactatccttcattggatagaatgaagggctatttcaacatctgtgagagaatccaaccttcctggaatggataagcgaatttcttatgatttatgatttttattattaaataagttataaaaaaaataagtgtatacaaattttaaagtgactcttaggttttaaaacgaaaattcttattcttgagtaactctttcctgtaggtcaggttgctttctcaggtatagcatgaggtcgctcttattgaccacacctctaccggcatgccgagcaaatgcctgcaaatcgctccccatttcacccaattgtagatatgctaactccagcaatgagttgatgaatctcggtgtgtattttatgtcctcagaggacaa
[0406]
seq id no:10
[0407]
gaccttcgtttgtgcggatccagacgagatcaattagaacctg
[0408]
seq id no:11
[0409]
gctatggtgtgtgggggatccttgtcctctgaggacataaaatac
[0410]
seq id no:12
[0411]
ggagaccaacatgtgagcaaaag
[0412]
seq id no:13
[0413]
ggatccgcacaaacgaaggtc
[0414]
seq id no:14
[0415]
cgtttgtgcggatccgacctgcagggggggggggg
[0416]
seq id no:15
[0417]
cacatgttggtctccgcgccagcaaccgcacctgtg
[0418]
seq id no:16
[0419]
catcatcatcatcatcattgagtttg
[0420]
seq id no:17
[0421]
agcttcagcctctcttttctcgag
[0422]
seq id no:18
[0423]
evqlvesggglvqpggslrlscaasgfslsnyyvtwvrqapgkglewvgiiygsdetayatsaigrftisrdnskntlylqmnslraedtavyycarddssdwdakfnlwgqgtlvtvssastkgpsvfplapsskstsggtaalgclvkdyfpepvtvswnsgaltsgvhtfpavlqssglyslssvvtvpssslgtqtyicnvnhkpsntkvdkkvepksvdkthtcppcpapellggpsvflfppkpkdtlmisrtpevtcvvvdvshedpevkfnwyvdgvevhnaktkpreeqynstyrvvsvltvlhqdwlngkeykckvsnkalpapiektiskakgqprepqvytlppsreemtknqvslwclvkgfypsdiavewesngqpennykttppvldsdgsfflyskltvdksrwqqgnvfscsvmhealhnhytqkslslspg
[0424]
seq id no:19
[0425]
gaggtccagttggtcgagtctggtggtggtttggttcagccaggtggatctttgagattgtcttgtgcagcatctggattctctttgtctaactactacgttacttgggttagacaggcaccaggtaaaggtttggagtgggtt
ggtattatttacggttctgatgaaactgcttacgctacttctgctattggtagattcactatttctagagataactctaaaaacactttgtacttgcaaatgaactctttgagagctgaagatacagccgtttactattgcgctagagatgattcttctgattgggatgctaagtttaacttgtggggacaaggtactttggttactgtttcttctgcttctactaagggaccatctgtctttccattggcaccatcttctaaatctacttctggaggaactgccgccttgggatgtttggtcaaggattatttccctgagcctgtcacagtttcttggaactctggtgcattgacatctggtgttcacacattcccagcagtcttgcaatcttctggtttgtactctttgtcttctgtcgttacagtcccttcttcttctttgggtactcaaacctacatctgtaatgttaatcataagccttctaacacaaaagttgataagaaagtcgagccaaaatctgttgacaaaacacatacctgcccaccatgtccagctcctgagttgttgggaggtccttctgtttttttgttcccacctaagccaaaggatacattgatgatttccagaactcctgaagttacatgtgtcgttgtcgatgtttctcatgaagatcctgaggttaaattcaattggtacgttgatggtgtcgaagttcataacgctaagactaaaccaagagaagaacagtacaattctacttatagagtcgtctctgttttgaccgttttgcatcaagattggttgaacggaaaggagtataagtgcaaagtttctaataaggctttgcctgcccctattgagaagaccatttctaaggctaaaggtcagcctagagaacctcaagtttacactttgccaccttccagagaggaaatgactaagaaccaggtctctttgtggtgcttggttaagggtttttacccttctgacatcgctgttgagtgggaatctaacggacagcctgaaaataactataaaacaacacctccagttttggattctgacggttctttctttttgtattctaagttgacagtcgataagtccagatggcaacaaggaaacgtcttttcttgttctgttatgcatgaagccttgcataatcattacacccagaaatctttgtctttgtctccaggt
[0426]
seq id no:20
[0427]
agagaggctgaagctgaggtccagttggtcgagtctggtg
[0428]
seq id no:21
[0429]
tcaatgatgatgatgatgatgtcaacctggagacaaagacaaagatttc
[0430]
seq id no:22
[0431]
gacagatggtcccttagtagaagc
[0432]
seq id no:23
[0433]
ctaagggaccatctgtctgtccattg
[0434]
seq id no:24
[0435]
ctaagggaccatctgtctttccatgtgcaccatcttc
[0436]
seq id no:25
[0437]
ctaagggaccatctgtctttccattgtgtccatcttctaaa
[0438]
seq id no:26
[0439]
ctaagggaccatctgtctttccattggcatgttcttctaaatctacttc
[0440]
seq id no:27
[0441]
ctaagggaccatctgtctttccattggcaccatgttct
[0442]
seq id no:28
[0443]
ctaagggaccatctgtctttccattggcaccatcttgtaaatc
[0444]
seq id no:29
[0445]
ctaagggaccatctgtctttccattggcaccatcttcttgttctact
[0446]
seq id no:30
[0447]
ctaagggaccatctgtctttccattggcaccatcttctaaatgtacttctggag
[0448]
seq id no:31
[0449]
ctaagggaccatctgtctttccattggcaccatcttctaaatcttgttctggaggaact
[0450]
seq id no:32
[0451]
agatgtcaatgcaccagagttcc
[0452]
seq id no:33
[0453]
ggtgcattgacatcttgtgttcac
[0454]
seq id no:34
[0455]
ggtgcattgacatctggtgtttgtacattc
[0456]
seq id no:35
[0457]
ggtgcattgacatctggtgttcacacatgtccagca
[0458]
seq id no:36
[0459]
ggtgcattgacatctggtgttcacacattctgtgcagtc
[0460]
seq id no:37
[0461]
ggtgcattgacatctggtgttcacacattcccagcatgtttgcaa
[0462]
seq id no:38
[0463]
ggtgcattgacatctggtgttcacacattcccagcagtcttgtgttcttct
[0464]
seq id no:39
[0465]
ttgcaagactgctgggaatgtgtg
[0466]
seq id no:40
[0467]
ccagcagtcttgcaatgttctggtttg
[0468]
seq id no:41
[0469]
ccagcagtcttgcaatcttgtggtttgtac
[0470]
seq id no:42
[0471]
ccagcagtcttgcaatcttcttgtttgtactctttgtc
[0472]
seq id no:43
[0473]
ccagcagtcttgcaatcttctggttgttactctttgtcttc
[0474]
seq id no:44
[0475]
gtacaaaccagaagattgcaagac
[0476]
seq id no:45
[0477]
caatcttctggtttgtactgtttgtc
[0478]
seq id no:46
[0479]
caatcttctggtttgtactctttgtgttctgtc
[0480]
seq id no:47
[0481]
caatcttctggtttgtactctttgtcttcttgtgttacagtcccttc
[0482]
seq id no:48
[0483]
caatcttctggtttgtactctttgtcttctgtcgtttgtgctcct
[0484]
seq id no:49
[0485]
tggctcgactctcttatcaac
[0486]
seq id no:50
[0487]
aagaaagtcgagccatgttctgttgac
[0488]
seq id no:51
[0489]
aagaaagtcgagccaaaatgtgttgac
[0490]
seq id no:52
[0491]
aagaaagtcgagccaaaatcttgcgac
[0492]
seq id no:53
[0493]
aggttttatactgagtttgttaatgatacaataaactgttatagtacatacaattgaaactctcttatctatactgggggaccttctcgcagaatggtataaatatctactaactgactgtcgtacggcctaggggtctcttcttcgattatttgcaggtcggaacatccttcgtctgatgcggatctcctgagacaaagttcacgggtatctagtattctatcagcataaatggaggacctttctaaactaaactttgaatcgtctccagcagcatcctcgcataatccttttgtcatttcctctatgtctattgtcactgtggttggcgcatcaagagtcgtccttctgtaaaccggtacagaattcctaccactagaagcttgaaatggggagggtttcagctttgtatcccgatactgtgctttaaaaagggagtccaaactgaaatctttttcggaatcattggatgatacctctgtattagatctcctatgtatcggtttcctcgggtagatagaactgtcgacagagtctctagcaaattgagaattggctttagacttcggagaagtaggtgacgatgtggtagatatatcagcagatgaaaagactgaattttttctctctggtgatgagttgattccctcttggtgttgatatcttgaaccgggctcatgttgaacctttgaattcaaaaaggtagcttgaagttgctcattgacagcatctgccacgtcgtatcttgcaacatctttgggaaactgatacgaagtgttcaacatctttgttgcggtatgacaagagcacttcgttgtacttttatcagagaaaagaaacacctcaattatggtatttaggtttatatattacgcaaattctattagaaaacccgggagctggagctttggctggtcatccttatggaattgatcgtgaatacattgctgagagattagggtttgattctgttattggtaattctttggccgctgtttcagacagagattttgtagtcgaaaccatgttctggtcttcgttgtttatgaatcatatttctcgattctcagaagatttgatcatttactccactggagagtttggatttatcaagttggcagatgcttattctactggatcttctctgatgcctacaaaaaaaaacccagactctttggagttattgaggggtaaatctggtagatgttttggggccttggctggtttcctcatgtctattaagtccattccgtcaacctataacaaagatatgcaagaggataaggagcctttatttgatactctaatcactgtagagcactcgattttgatagcatccggtgtagtttctaccttgaacattgatgccgaacgaatgaagaatgctctaactatggatatgctggctacagatcttgccgactatttagttagaaggggagttccattcagagaaactcaccacatttctggtgaatgtgtcagacaagccgaggagttgaacctttctggtattgatcagttgtccctcgaacaattgaaatccattgactcccgttttgaggctgatgtggcttcaacgtttgactttgaagccagtgttgaaaaaagaactgccaccggaggaacttctaagactgctgttttaaagcaattggatgcactgaatgaaaagctagagtcttgagcgaatttcttatgatttatgatttttattattaaataagttataaaaaaaataagtgtatacaaattttaaagtgactcttaggttttaaaacgaaaattcttattcttgagtaactctttcctgtaggtcaggttgctttctcaggtatagcatgaggtcgctcttattgaccacacctctaccggcatgccgagcaaatgcctgcaaatcgctccccatttcacccaattgtagatatgctaactccagcaatgagttgatgaatctcggtgtgtattttatgtcctcagaggacaa
[0494]
seq id no:54
[0495]
gaccttcgtttgtgcggatccaggttttatactgagtttgtta
[0496]
seq id no:55
[0497]
gctatggtgtgtgggggatccttgtcctctgaggacataaaatac
[0498]
seq id no:56
[0499]
aiqmtqspsslsasvgdrvtitcqasqsinnelswyqqkpgkapklliyrastlasgvpsrfsgsgsgtdftltisslqpddfatyycqqgyslrnidnafgggtkveikrtvaapsvfifppsdeqlksgtasvvcllnnfypreakvqwkvdnalqsgnsqesvteqdskdstyslsstltlskadyekhkvyacevthqglsspvtksfnrgev
[0500]
seq id no:57
[0501]
gctattcaaatgactcaatctccttcttctttgtctgcttctgtcggagatagagtcactatcacttgtcaagcttctcaatctattaacaacgaattgtcttggtatcagcaaaagccaggtaaggctccaaaattgttgatttacagagcatctactttggcttctggtgtcccttccagattctctggttctggttctggaaccgattttactttgactatctcttctttgcagcctgatgatttcgcaacttactactgtcaacagggttactctttgagaaacattgataacgcttttggtggtggaactaaagttgagattaagagaactgtcgctgcaccttctgttttcatttttcctccatctgacgagcagttgaagtctggtacagcatctgttgtctgtttgttgaacaacttctacccaagagaagcaaaggttcaatggaaggttgacaacgccttgcaatctggtaactctcaggaatctgttactgaacaagattctaaggattctacttactctttgtcttctacattgacattgtctaaggctgattacgaaaagcataaggtttacgcttgtgaagttactcatcaaggattgtcttctcctgtcacaaaatcttttaacagaggtgaggtt
[0502]
seq id no:58
[0503]
agagaggctgaagctgctattcaaatgactcaatctcc
[0504]
seq id no:59
[0505]
tcaatgatgatgatgatgatgtcaaacctcacctctgttaaaagattttg
[0506]
seq id no:60
[0507]
aggtgcagcgacagttctagtaatttg
[0508]
seq id no:61
[0509]
gaactgtcgctgcaccttgtgttttc
[0510]
seq id no:62
[0511]
gaactgtcgctgcaccttctgtttgtatttttcctccatc
[0512]
seq id no:63
[0513]
gaactgtcgctgcaccttctgttttcatttgtcctccatctgac
[0514]
seq id no:64
[0515]
gaactgtcgctgcaccttctgttttcattttttgtccatctgacgagcag
[0516]
seq id no:65
[0517]
gaactgtcgctgcaccttctgttttcatttttccttgttctgacgagcagttg
[0518]
seq id no:66
[0519]
gaactgtcgctgcaccttctgttttcatttttcctccatgtgacgagcagttg
[0520]
seq id no:67
[0521]
accagattgcaaggcgttgtcaac
[0522]
seq id no:68
[0523]
gccttgcaatctggttgttctcaggaatctg
[0524]
seq id no:69
[0525]
gccttgcaatctggtaactcttgtgaatctgttactg
[0526]
seq id no:70
[0527]
gccttgcaatctggtaactctcaggaatgtgttactgaac
[0528]
seq id no:71
[0529]
gccttgcaatctggtaactctcaggaatctgtttgtgaacaagattctaag
[0530]
seq id no:72
[0531]
agaatccttagaatcttgttcag
[0532]
seq id no:73
[0533]
gattctaaggattcttgttactctttgtc
[0534]
seq id no:74
[0535]
gattctaaggattctacttactgtttgtcttc
[0536]
seq id no:75
[0537]
gattctaaggattctacttactctttgtgttctaca
[0538]
seq id no:76
[0539]
gattctaaggattctacttactctttgtcttcttgtttgaca
[0540]
seq id no:77
[0541]
gattctaaggattctacttactctttgtcttctacattgtgtttgtctaaggctg
[0542]
seq id no:78
[0543]
tcaatgatgatgatgatgatgtcaaacctcacctctgttacaaga
[0544]
seq id no:79
[0545]
tcaatgatgatgatgatgatgtcaaacctcacctctacaaaaag
[0546]
seq id no:80
[0547]
tcaatgatgatgatgatgatgtcaaacctcaccacagttaaaag
[0548]
seq id no:81
[0549]
tcaatgatgatgatgatgatgtcaaacctcacatctgttaaaag
[0550]
seq id no:82
[0551]
tcaatgatgatgatgatgatgtcaaacacaacctctgttaaaag
[0552]
seq id no:83
[0553]
tcaatgatgatgatgatgatgtcaacactcacctctgttaaaag
[0554]
seq id no:84
[0555]
atcaacttttgtgttaga
[0556]
seq id no:85
[0557]
tctaacacaaaagttgatgataaagtcgagccaaaa
[0558]
seq id no:86
[0559]
tctaacacaaaagttgatgaaaaagtcgagccaaaa
[0560]
seq id no:87
[0561]
agatggaggaaaaat
[0562]
seq id no:88
[0563]
atttttcctccatctgacaagcagttgaagtctggt
[0564]
seq id no:89
[0565]
acatggaggaaaaatgaaaac
[0566]
seq id no:90
[0567]
atttttcctccatgtgacaagcagttgaagtctggt
[0568]
seq id no:91
[0569]
agaacaaggaaaaatgaaaac
[0570]
seq id no:92
[0571]
atttttccttgttctgacaagcagttgaagtctggt
[0572]
seq id no:93
[0573]
agatggacaaaaaatgaaaac
[0574]
seq id no:94
[0575]
attttttgtccatctgacaagcagttgaagtctggt
[0576]
seq id no:95
[0577]
agatggaggacaaatgaaaac
[0578]
seq id no:96
[0579]
atttgtcctccatctgacaagcagttgaagtctggt
[0580]
seq id no:97
[0581]
atttttcctccatctgacagacagttgaagtctggt
[0582]
seq id no:98
[0583]
atttttcctccatgtgacagacagttgaagtctggt
[0584]
seq id no:99
[0585]
atttttccttgttctgacagacagttgaagtctggt
[0586]
seq id no:100
[0587]
attttttgtccatctgacagacagttgaagtctggt
[0588]
seq id no:101
[0589]
atttgtcctccatctgacagacagttgaagtctggt
[0590]
seq id no:102
[0591]
qvqlvqsgaevkkpgasvkvsckasgytftnyymhwvrqapgqglewvgwinpytgsafyaqkfrgrvtmtrdtsistaymelsrlrsddtavyycarepekfdsddsdvwgrgtlvtvssastkgpsvfplapsskstsggtaalgclvkdyfpepvtvswnsgaltsgvhtfpavlqssglyslssvvtvpssslgtqtyicnvnhkpsntkvdkkvepkscdkthtcppcpapellggpsvflfppkpkdtlmisrtpevtcvvvdvshedpevkfnwyvdgvevhnaktkpreeqynstyrvvsvltvlhqdwlngkeykckvsnkalpapiektiskakgqprepqvytlppsreemtknqvslscavkgfypsdiavewesngqpennykttppvldsdgsfflvskltvdksrwqqgnvfscsvmhealhnrftqkslslspghhhhhh
[0592]
seq id no:103
[0593]
caagttcaattggttcaatcaggagctgaagttaagaagccaggtgcttccgttaaggtttcttgtaaggcttcaggatacacttttactaactactacatgcattgggttagacaagctccaggacaaggtttggaatgggttggatggattaacccatacactgggagtgctttttacgctcaaaagtttagaggtagagttactatgactagagatacttctatttccactgcttacatggaattgtcgcgtttgagatcagatgatactgctgtttactactgtgctagagaacctgaaaagtttgattccgatgattctgatgtttggggtagaggtactttggttactgtttcttctgcttctactaagggaccatctgtctttccattggcaccatcttctaaatctacttctggaggaactgccgccttgggatgtttggtcaaggattatttccctgagcctgtcacagtttcttggaactctggtgcattgacatctggtgttcacacattcccagcagtcttgcaatcttctggtttgtactctttgtcttctgtcgttacagtcccttcttcttctttgggtactcaaacctacatctgtaatgttaatcataagccttctaacacaaaagttgataagaaagtcgagccaaaatcttgcgacaaaacacatacctgcccaccatgtccagctcctgagttgttgggaggtccttctgtttttttgttcccacctaagcc
aaaggatacattgatgatttccagaactcctgaagttacatgtgtcgttgtcgatgtttctcatgaagatcctgaggttaaattcaattggtacgttgatggtgtcgaagttcataacgctaagactaaaccaagagaagaacagtacaattctacttatagagtcgtctctgttttgaccgttttgcatcaagattggttgaacggaaaggagtataagtgcaaagtttctaataaggctttgcctgcccctattgagaagaccatttctaaggctaaaggtcagcctagagaacctcaagtttacactttgccaccttccagagaggaaatgactaagaaccaggtctctttgtcttgcgctgttaagggtttttacccttctgacatcgctgttgagtgggaatctaacggacagcctgaaaataactataaaacaacacctccagttttggattctgacggttctttctttttggtttctaagttgacagtcgataagtccagatggcaacaaggaaacgtcttttcttgttctgttatgcatgaagccttgcataatagatttacccagaaatctttgtctttgtctccaggtcatcatcatcatcatcat
[0594]
seq id no:104
[0595]
ccgctcgagaaaagagaggctgaagctcaagttcaattggttcaatcag
[0596]
seq id no:105
[0597]
atagtttagcggccgctcaatgatgatgatgatgatgacctggagacaaaga
[0598]
seq id no:106
[0599]
qavltqppsvsgapgqrvtisctgsssnigagygvhwyqqlpgtapklliygdsnrpsgvpdrfsgsksgtsaslaitglqaedeadyycqsydnslsgyvfgggtqltvlgqpkaapsvtlfppsseelqankatlvclisdfypgavtvawkadsspvkagvetttpskqsnnkyaassylsltpeqwkshrsyscqvthegstvektvaptecs
[0600]
seq id no:107
[0601]
caagctgttttgactcaaccaccatccgtttcaggggcgcctggtcaaagagttactatttcatgtactgggtcctcatccaacattggtgctggatacggtgttcattggtatcaacaattgccaggaactgctcctaagttgttgatttacggtgattctaacagaccatctggtgttccagatagattttccggatctaagtccggaacttctgcttccttggctattactggtttgcaagctgaagatgaagctgattactactgtcaatcatacgataactccttgtccggatacgtttttggtggaggaactcaattgactgttttgggacaaccaaaggctgctccatccgttactttgtttccaccatcttcagaagaattgcaagctaacaaggctactttggtttgtttgatttccgatttttacccaggagctgttactgttgcttggaaggctgattcctcaccagttaaggctggtgttgaaactactactccatctaagcaatcaaacaacaagtacgctgcttcatcatacttgtccttgactccagaacaatggaagtcacatagatcatactcatgtcaagttactcatgaaggaagtactgttgaaaagactgttgctccaactgaatgttct
[0602]
seq id no:108
[0603]
ccgctcgagaaaagagaggctgaagctcaagctgttttgactcaacc
[0604]
seq id no:109
[0605]
aaggaaaaaagcggccgcttaagaacattcagttggagcaac
[0606]
seq id no:110
[0607]
evqllesggglvqpggslrlscaasgftfssfpmawvrqapgkglewvstistsggrtyyrdsvkgrftisrdnskntlylqmnslraedtavyycakfrqysggfdywgqgtlvtvssastkgpsvfplapsskstsggtaalgclvkdyfpepvtvswnsgaltsgvhtfpavlqssglyslssvvtvpssslgtqtyicnvnhkpsntkvdkkvepksvdkthtcppcpapellggpsvflfppkpkdtlmisrtpevtcvvvdvshedpevkfnwyvdgvevhnaktkpreeqynstyrvvsvltvlhqdwlngkeykckvsnkalpapiektiskakgqprepqvytlppsreemtknqvslwclvkgfypsdiavewesngqpennykttppvldsdgsfflyskltvdksrwqqgnvfscsvmhealhnhytqkslslspg
[0608]
seq id no:111
[0609]
gaggtccagttgttggagtctggtggtggtttggttcagccaggtggatctttgagattgtcttgtgcagcatctggattcactttttcttcttttccaatggcttgggttagacaggcaccaggtaaaggtttggagtgggtttctactatttctacttctggtggtagaacttactacagagattctgtcaagggtagattcactatttctagagataactctaaaaacactttgtacttgcaaatgaactctttgagagctgaagatacagccgtttactattgcgctaagtttagacaatactctggtggttttgattactggggacaaggtactttggttactgtttcttctgcttctactaagggaccatctgtctttccattggcaccatcttctaaatctacttctggaggaactgccgccttgggatgtttggtcaaggattatttccctgagcctgtcacagtttcttggaactctggtgcattgacatctggtgttcacacattcccagcagtcttgcaatcttctggtttgtactctttgtcttctgtcgttacagtcccttcttcttctttgggtactcaaacctacatctgtaatgttaatcataagccttctaacacaaaagttgataagaaagtcgagccaaaatctgttgacaaaacacatacctgcccaccatgtccagctcctgagttgttgggaggtccttctgtttttttgttcccacctaagccaaaggatacattgatgatttccagaactcctgaagttacatgtgtcgttgtcgatgtttctcatgaagatcctgaggttaaattcaattggtacgttgatggtgtcgaagttcataacgctaagactaaaccaagagaagaacagtacaattctacttatagagtcgtctctgttttgaccgttttgcatcaagattggttgaacggaaaggagtataagtgcaaagtttctaataaggctttgcctgcccctattgagaagaccatttctaaggctaaaggtcagcctagagaacctcaagtttacactttgccaccttccagagaggaaatgactaagaaccaggtctctttgtggtgcttggttaagggtttttacccttctgacatcgctgttgagtgggaatctaacggacagcctgaaaataactataaaacaacacctccagttttggattctgacggttctttctttttgtattctaagttgacagtcgataagtccagatggcaacaaggaaacgtcttttcttgttctgttatgcatgaagccttgcataatcattacacccagaaatctttgtctttgtctccaggt
[0610]
seq id no:112
[0611]
agagaggctgaagctgaggtccagttgttggagtctg
[0612]
seq id no:113
[0613]
tcaatgatgatgatgatgatgtcaacctggagacaaagacaaagatttc
[0614]
seq id no:114
[0615]
gacagatggtcccttagtagaagc
[0616]
seq id no:115
[0617]
ctaagggaccatctgtctgtccattg
[0618]
seq id no:116
[0619]
ctaagggaccatctgtctttccatgtgcaccatcttc
[0620]
seq id no:117
[0621]
ctaagggaccatctgtctttccattgtgtccatcttctaaatc
[0622]
seq id no:118
[0623]
ctaagggaccatctgtctttccattggcatgttcttctaaatctacttc
[0624]
seq id no:119
[0625]
ctaagggaccatctgtctttccattggcaccatgttctaaatctact
[0626]
seq id no:120
[0627]
ctaagggaccatctgtctttccattggcaccatcttgtaaatctact
[0628]
seq id no:121
[0629]
ctaagggaccatctgtctttccattggcaccatcttcttgttctacttctggag
[0630]
seq id no:122_
[0631]
ctaagggaccatctgtctttccattggcaccatcttctaaatgtacttctggag
[0632]
seq id no:123
[0633]
ctaagggaccatctgtctttccattggcaccatcttctaaatcttgttctggaggaact
[0634]
seq id no:124
[0635]
ctaagggaccatctgtctttccattggcaccatcttctaaatctacttgtggaggaact
[0636]
seq id no:125
[0637]
agatgtcaatgcaccagagttcc
[0638]
seq id no:126
[0639]
ggtgcattgacatcttgtgttcacacattccca
[0640]
seq id no:127
[0641]
ggtgcattgacatctggtgtttgtacattcccagc
[0642]
seq id no:128
[0643]
ggtgcattgacatctggtgttcacacatgtccagcagtcttgc
[0644]
seq id no:129
[0645]
ggtgcattgacatctggtgttcacacattctgtgcagtcttgcaa
[0646]
seq id no:130
[0647]
ggtgcattgacatctggtgttcacacattcccagcatgtttgcaatcttct
[0648]
seq id no:131
[0649]
ggtgcattgacatctggtgttcacacattcccagcagtcttgtgttcttctggtttg
[0650]
seq id no:132
[0651]
ttgcaagactgctgggaatgtgtg
[0652]
seq id no:133
[0653]
ccagcagtcttgcaatgttctggtttg
[0654]
seq id no:134
[0655]
ccagcagtcttgcaatcttgtggtttgtactctttg
[0656]
seq id no:135
[0657]
ccagcagtcttgcaatcttcttgtttgtactctttgtc
[0658]
seq id no:136
[0659]
ccagcagtcttgcaatcttctggttgttactctttgtcttc
[0660]
seq id no:137
[0661]
gtacaaaccagaagattgcaagac
[0662]
seq id no:138
[0663]
caatcttctggtttgtactgtttgtcttctgtc
[0664]
seq id no:139
[0665]
caatcttctggtttgtactctttgtgttctgtcgttacag
[0666]
seq id no:140
[0667]
caatcttctggtttgtactctttgtcttcttgtgttacagtcccttc
[0668]
seq id no:141_
[0669]
caatcttctggtttgtactctttgtcttctgtcgtttgtgtcccttcttcttc
[0670]
seq id no:142
[0671]
tttcttatcaacttttgtgttag
[0672]
seq id no:143
[0673]
caaaagttgataagaaatgtgagccaaaatctg
[0674]
seq id no:144
[0675]
caaaagttgataagaaagtctgtccaaaatctgttg
[0676]
seq id no:145
[0677]
caaaagttgataagaaagtcgagtgtaaatctgttgac
[0678]
seq id no:146
[0679]
aagaaagtcgagccatgttctgttgacaaaaca
[0680]
seq id no:147
[0681]
aagaaagtcgagccaaaatgtgttgacaaaaca
[0682]
seq id no:148
[0683]
aagaaagtcgagccaaaatcttgcgacaaaacacata
[0684]
seq id no:149.
[0685]
diqltqpnsvstslgstvklsctlssgniennyvhwyqlyegrspttmiydddkrpdgvpdrfsgsidrssnsafltihnvaiedeaiyfchsyvssfnvfgggtkltvlrqpkaapsvtlfppsseelqankatlvclisdfypgavtvawkadsspvkagvetttpskqsnnkyaassylsltpeqwkshrsyscqvthegstvektvaptevs
[0686]
seq id no:150
[0687]
gatattcaattgactcaacctaactcagtttccacttccttgggttcgactgttaagttgtcatgtactttgtcctctggtaacattgaaaacaactacgttcattggtatcaattgtacgaaggtagatcccctactactatgatttacgatgatgataagagacctgatggagttcctgatagattttccggttctattgatagaagctctaactccgcttttttgactattcataacgttgctattgaagatgaagctatttacttttgtcattcttacgtttcttcctttaacgtttttggaggtggaactaagttgactgttttgagacaaccaaaggctgctccatccgttactttgtttccaccatcttcagaagaattgcaagctaacaaggctactttggtttgtttgatttccgatttttacccaggagctgttactgttgcttggaaggctgattcctcaccagttaaggctggtgttgaaactactactccatctaagcaatcaaacaacaagtacgctgcttcatcatacttgtccttgactccagaacaatggaagtcacatagatcatactcatgtcaagttactcatgaaggaagtactgttgaaaagactgttgctccaactgaagtttct
[0688]
seq id no:151
[0689]
agagaggctgaagctgatattcaattgactcaac
[0690]
seq id no:152
[0691]
tcaatgatgatgatgatgatgtcaagaaacttcagttggagcaac
[0692]
seq id no:153
[0693]
tggagcagcctttggttgtctc
[0694]
seq id no:154
[0695]
ccaaaggctgctccatgtgttactttgtttccacc
[0696]
seq id no:155
[0697]
ccaaaggctgctccatccgtttgtttgtttccaccatcttc
[0698]
seq id no:156
[0699]
ccaaaggctgctccatccgttactttgtgtccaccatcttcag
[0700]
seq id no:157
[0701]
ccaaaggctgctccatccgttactttgttttgtccatcttcagaagaattg
[0702]
seq id no:158
[0703]
ccaaaggctgctccatccgttactttgtttccaccatgttcagaagaattg
[0704]
seq id no:159
[0705]
agccttaactggtgaggaatcagc
[0706]
seq id no:160
[0707]
tcaccagttaaggcttgtgttgaaactactactc
[0708]
seq id no:161
[0709]
tcaccagttaaggctggtgtttgtactactactccatctaag
[0710]
seq id no:162
[0711]
tcaccagttaaggctggtgttgaaacttgtactccatctaagcaatc
[0712]
seq id no:163
[0713]
tcaccagttaaggctggtgttgaaactacttgtccatctaagcaatca
[0714]
seq id no:164
[0715]
tcaccagttaaggctggtgttgaaactactacttgttctaagcaatcaaac
[0716]
seq id no:165
[0717]
tcaccagttaaggctggtgttgaaactactactccatgtaagcaatc
[0718]
seq id no:166
[0719]
tcaccagttaaggctggtgttgaaactactactccatctaagtgttcaaacaacaagta
[0720]
seq id no:167
[0721]
gttgtttgattgcttagatggag
[0722]
seq id no:168
[0723]
taagcaatcaaacaactgttacgctgcttcatcatac
[0724]
seq id no:169
[0725]
taagcaatcaaacaacaagtactgtgcttcatcatacttgtc
[0726]
seq id no:170
[0727]
taagcaatcaaacaacaagtacgctgcttgttcatacttgtccttgac
[0728]
seq id no:171
[0729]
taagcaatcaaacaacaagtacgctgcttcatcatgtttgtccttgactccag
[0730]
seq id no:172
[0731]
taagcaatcaaacaacaagtacgctgcttcatcatacttgtgtttgactccagaacaat
[0732]
seq id no:173
[0733]
caatgatgatgatgatgatgtcaagaaacttcagttggagcacaagtcttttcaacag
[0734]
seq id no:174
[0735]
caatgatgatgatgatgatgtcaagaaacttcagttggacaaacagtcttttcaaca
[0736]g[0737]
seq id no:175
[0738]
tcaatgatgatgatgatgatgtcaagaaacttcagtacaagcaacagtcttttcaac
[0739]
seq id no:176
[0740]
tcaatgatgatgatgatgatgtcaagaaacttcacatggagcaacagtcttttc
[0741]
seq id no:177
[0742]
tcaatgatgatgatgatgatgtcaagaaacacaagttggagcaacagtc
[0743]
seq id no:178
[0744]
tcaatgatgatgatgatgatgtcaagaacattcagttggagcaac
[0745]
seq id no:179
[0746]
tcaatgatgatgatgatgatgtcaacaaacttcagttggagcaac
[0747]
seq id no:180
[0748]
atcaacttttgtgttagaaggc
[0749]
seq id no:181
[0750]
taacacaaaagttgatgataaagtcgagccaaaatctg
[0751]
seq id no:182
[0752]
taacacaaaagttgatgataaatgtgagccaaaatctg
[0753]
seq id no:183
[0754]
taacacaaaagttgatgataaagtctgtccaaaatctg
[0755]
seq id no:184
[0756]
taacacaaaagttgatgataaagtcgagtgtaaatctg
[0757]
seq id no:185
[0758]
taacacaaaagttgatgataaagtcgagccatgttctg
[0759]
seq id no:186
[0760]
taacacaaaagttgatgataaagtcgagccaaaatgtg
[0761]
seq id no:187
[0762]
atcaacttttgtgttagaaggc
[0763]
seq id no:188
[0764]
taacacaaaagttgatgaaaaagtcgagccaaaatctg
[0765]
seq id no:189
[0766]
taacacaaaagttgatgaaaaatgtgagccaaaatctg
[0767]
seq id no:190
[0768]
taacacaaaagttgatgaaaaagtctgtccaaaatctg
[0769]
seq id no:191
[0770]
taacacaaaagttgatgaaaaagtcgagtgtaaatctg
[0771]
seq id no:192
[0772]
taacacaaaagttgatgaaaaagtcgagccatgttctg
[0773]
seq id no:193
[0774]
taacacaaaagttgatgaaaaagtcgagccaaaatgtg
[0775]
seq id no:194
[0776]
tgaagatggtggaaacaaagtaac
[0777]
seq id no:195
[0778]
gtttccaccatcttcaaaggaattgcaagctaacaaggctac
[0779]
seq id no:196
[0780]
tgaagatggtggaaacaaagtaacaca
[0781]
seq id no:197
[0782]
tgaagatggtggaaacaaacaaac
[0783]
seq id no:198
[0784]
gttagcttgcaattcctttgaacatggtggaaacaaag
[0785]
seq id no:199
[0786]
gaattgcaagctaacaaggctac
[0787]
seq id no:200
[0788]
gttagcttgcaattcctttgaagatggacaaaacaaag
[0789]
seq id no:201
[0790]
gttagcttgcaattcctttgaagatggtggacacaaag
[0791]
seq id no:202
[0792]
qvqlvqsgaevkkpgssvkvsckasgysftdyhihwvrqapgqglewmgvinpmygttdynqrfkgrvtitadeststaymelsslrsedtavyycarydyftgtgvywgqgtlvtvssastkgpsvfplapcsrstsestaalgclvkdyfpepvtvswnsgaltsgvhtfpavlqssglyslssvvtvpssslgtktytcnvdhkpsntkvdkrveskygppcppcpapeflggpsvflfppkpkdtlmisrtpevtcvvvdvsqedpevqfnwyvdgvevhnaktkpreeqfnstyrvvsvltvlhqdwlngkeykckvsnkglpssiektiskakgqprepqvytlppsqeemtknqvslscavkgfypsdiavewesngqpennykttppvldsdgsfflvsrltvdksrwqegnvfscsvmhealhnrftqkslslslghhhhhh
[0793]
seq id no:203
[0794]
caagttcaattggttcaatccggagctgaagttaagaagcctgggtcatccgttaaggtttcatgtaaggcttcaggatactcttttactgattaccatattcattgggttagacaagctccaggtcaaggtttggaatggatgggagttattaaccctatgtacggaactactgattacaaccaaagatttaagggtagagttactattactgctgatgaatccacttccactgcttacatggaattgtcctctttgagatcagaagatactgctgtttactactgtgctagatacgattactttactggaactggtgtttactggggacaaggaactttggttactgtttcttccgcttccactaagggaccatccgtttttccattggctccatgttctagatccacttccgaatcaactgctgctttgggatgtttggttaaggattactttccagaaccagttactgtttcatggaactcaggagctttgacttccggagttcatacttttccagctgttttgcaatcctcaggattgtactctttgtcttcagttgttactgttccatcttcttccttgggtactaagacttacacttgtaacgttgatcataagccatctaacactaaggttgataagagagttgaatctaagtacggacctccatgtccaccatgtccagctcctgaatttttgggtggaccatccgtttttttgtttccaccaaagccaaaggatactttgatgatttctagaactcctgaagttacttgtgttgttgttgatgtttcacaagaagatcctgaagttcaatttaactggtacgttgatggagttgaagttcataacgctaagactaagcctagagaagaacaatttaactccacttacagagttgtttccgttttgactgttttgcatcaagattggttgaacggtaaggaatacaagtgtaaggtttctaacaagggattgccatcctctattgaaaagactatttcaaaggctaagggtcaacctagagaacctcaagtttacactttgccaccttcacaagaagaaatgactaagaaccaagtttccttgtcttgtgctgttaagggattttacccatccgatattgctgttgaatgggaatcaaacggtcaacctgaaaacaactacaagactactccaccagttttggattctgatggatcattttttttggtttctagattgactgttgataagtctagatggcaagaaggtaacgttttttcatgttccgttatgcat
gaagctttgcataacagatttactcaaaagtccttgtccttgtcattgggacatcatcatcatcatcat
[0795]
seq id no:204
[0796]
ccgctcgagaaaagagaggctgaagctcaagttcaattggttcaatccg
[0797]
seq id no:205
[0798]
aaggaaaaaagcggccgcttaatgatgatgatgatgatgtcccaatgacaaggacaag
[0799]
seq id no:206.
[0800]
divmtqtplslsvtpgqpasiscrssrslvhsrgntylhwylqkpgqspqlliykvsnrfigvpdrfsgsgsgtdftlkisrveaedvgvyycsqsthlpftfgqgtkleikrtvaapsvfifppsdeqlksgtasvvcllnnfypreakvqwkvdnalqsgnsqesvteqdskdstyslsstltlskadyekhkvyacevthqglsspvtksfnrgec
[0801]
seq id no:207
[0802]
gatattgttatgactcaaactccattgtccttgtccgttactccaggtcaacctgcttctatttcatgtagatcatctaggtccttggttcattccagaggtaacacttacttgcattggtacttgcaaaagccaggacaatcaccacaattgttgatttacaaggtttctaacagatttattggtgttcctgatagattttccggatctggatcaggaactgattttactttgaagatttcgcgggttgaagctgaagatgttggagtttactactgttcccaatccactcatttgccttttacttttggtcaaggaactaagttggaaattaagagaactgttgctgcaccttctgttttcatttttcctccatctgacgagcagttgaagtctggtacagcatctgttgtctgtttgttgaacaacttctacccaagagaagcaaaggttcaatggaaggttgacaacgccttgcaatctggtaactctcaggaatctgttactgaacaagattctaaggattctacttactctttgtcttctacattgacattgtctaaggctgattacgaaaagcataaggtttacgcttgtgaagttactcatcaaggattgtcttctcctgtcacaaaatcttttaacagaggtgagtgt
[0803]
seq id no:208
[0804]
ccgctcgagaaaagagaggctgaagctgatattgttatgactcaaactc
[0805]
seq id no:209
[0806]
aaggaaaaaagcggccgcttaacactcacctctgttaaaag
[0807]
seq id no:210
[0808]
evqlvesggglvqpggslrlscaasgfnfndyfmnwvrqapgkglewvaqmrnknyqygtyyaeslegrftisrddsknslylqmnslktedtavyycaresyygftsywgqgtlvtvssastkgpsvfplapssrstsestaalgclvkdyfpepvtvswnsgaltsgvhtfpavlqssglyslssvvtvpssslgtktytcnvdhkpsntkvdkrveskygppcppcpapeflggpsvflfppkpkdtlmisrtpevtcvvvdvsqedpevqfnwyvdgvevhnaktkpreeqfnstyrvvsvltvlhqdwlngkeykckvsnkglpssiektiskakgqprepqvytlppsqeemtknqvslcclvkgfypsdiavewesngqpennykttppvldsdgsfflysrltvdksrwqegnvfscsvmhealhnhytqkslslslg
[0809]
seq id no:211.
[0810]
gaagttcaattggttgaatctggaggtggattggttcaacctggtggatcattgagattgtcatgtgctgcttctggatttaactttaacgattactttatgaactgggttagacaagctccaggaaagggattggaatgggttgctcaaatgagaaacaagaactaccaatacggtacttactacgctgaatccttggaaggtagatttactatttctagagatgattcaaagaactccttgtacttgcaaatgaactcattgaagactgaagatactgctgtttactactgtgctagagaatcatactacggatttacttcttactggggacaaggaactttggttactgtttcttccgcttccactaagggaccatccgtttttccattggctccatcttctagatccacttccgaatcaactgctgctttgggatgtttggttaaggattactttccagaaccagttactgtttcatggaactcaggagctttgacttccggagttcatacttttccagctgttttgcaatcctcaggattgtactctttgtcttcagttgttactgttccatcttcttccttgggtactaagact
tacacttgtaacgttgatcataagccatctaacactaaggttgataagagagttgaatctaagtacggacctccatgtccaccatgtccagctcctgaatttttgggtggaccatccgtttttttgtttccaccaaagccaaaggatactttgatgatttctagaactcctgaagttacttgtgttgttgttgatgtttcacaagaagatcctgaagttcaatttaactggtacgttgatggagttgaagttcataacgctaagactaagcctagagaagaacaatttaactccacttacagagttgtttccgttttgactgttttgcatcaagattggttgaacggtaaggaatacaagtgtaaggtttctaacaagggattgccatcctctattgaaaagactatttcaaaggctaagggtcaacctagagaacctcaagtttacactttgccaccttcacaagaagaaatgactaagaaccaagtttccttgtgttgtttggttaagggattttacccatccgatattgctgttgaatgggaa
[0811]
seq id no:212
[0812]
agagaggctgaagctgaagttcaattggttgaatctg
[0813]
seq id no:213
[0814]
tcaatgatgatgatgatgatgttatcccaatgacaaggac
[0815]
seq id no:214
[0816]
aacggatggtcccttagtggaagc
[0817]
seq id no:215
[0818]
taagggaccatccgtttgtccattggctcca
[0819]
seq id no:216
[0820]
taagggaccatccgtttttccatgtgctccatcttctag
[0821]
seq id no:217
[0822]
taagggaccatccgtttttccattgtgtccatcttctagatc
[0823]
seq id no:218
[0824]
taagggaccatccgtttttccattggcttgttcttctagatccac
[0825]
seq id no:219
[0826]
taagggaccatccgtttttccattggctccatgttctagatccac
[0827]
seq id no:220
[0828]
taagggaccatccgtttttccattggctccatcttgtagatccacttc
[0829]
seq id no:221
[0830]
agaagatggagccaatggaaaaac
[0831]
seq id no:222
[0832]
ttggctccatcttcttgttccacttccgaatc
[0833]
seq id no:223
[0834]
ttggctccatcttctagatgtacttccgaatc
[0835]
seq id no:224
[0836]
ttggctccatcttctagatcctgttccgaatcaactgc
[0837]
seq id no:225
[0838]
ttggctccatcttctagatccacttgtgaatcaactgctgc
[0839]
seq id no:226
[0840]
ttggctccatcttctagatccacttcctgttcaactgctgctttg
[0841]
seq id no:227
[0842]
ggaagtcaaagctcctgagttcca
[0843]
seq id no:228
[0844]
ggagctttgacttcctgtgttcatacttttc
[0845]
seq id no:229
[0846]
ggagctttgacttccggagtttgtacttttccagctg
[0847]
seq id no:230
[0848]
ggagctttgacttccggagttcatacttgtccagctgttttg
[0849]
seq id no:231
[0850]
ggagctttgacttccggagttcatactttttgtgctgttttgcaatcctc
[0851]
seq id no:232
[0852]
ggagctttgacttccggagttcatacttttccagcttgtttgcaatcctcagg
[0853]
seq id no:233
[0854]
aacagctggaaaagtatgaactc
[0855]
seq id no:234
[0856]
acttttccagctgtttgtcaatcctcaggattg
[0857]
seq id no:235
[0858]
acttttccagctgttttgtgttcctcaggattgt
[0859]
seq id no:236
[0860]
acttttccagctgttttgcaatgttcaggattgtactc
[0861]
seq id no:237
[0862]
acttttccagctgttttgcaatcctgtggattgtactctttg
[0863]
seq id no:238
[0864]
acttttccagctgttttgcaatcctcatgtttgtactctttgtc
[0865]
seq id no:239
[0866]
acttttccagctgttttgcaatcctcaggatgttactctttgtcttc
[0867]
seq id no:240
[0868]
gtacaatcctgaggattgcaaaac
[0869]
seq id no:241
[0870]
tcctcaggattgtactgtttgtcttcag
[0871]
seq id no:242
[0872]
tcctcaggattgtactctttgtgttcagttgttac
[0873]
seq id no:243
[0874]
tcctcaggattgtactctttgtcttcatgtgttactgttccatcttc
[0875]
seq id no:244
[0876]
tcctcaggattgtactctttgtcttcagttgtttgtgttccatcttcttc
[0877]
seq id no:245
[0878]
tcctcaggattgtactctttgtcttcagttgttactgtttgttcttcttccttgggtac
[0879]
seq id no:246
[0880]
aaccaaacatcccaaagcagcag
[0881]
seq id no:247
[0882]
ctttgggatgtttggttgatgattactttccagaa
[0883]
seq id no:248
[0884]
diqmtqspsslsasvgdrvtitcqasqdigislswyqqkpgkapklliynannladgvpsrfsgsgsgtdftltisslqpedfatyyclqhnsapytfgqgtkleikrtvaapsvfifppsdeqlksgtasvvcllnnfypreakvqwkvdnalqsgnsqesvteqdskdstyslsstltlskadyekhkvyacevthqglsspvtksfnrgev
[0885]
seq id no:249
[0886]
gatattcaaatgactcaatcaccatcctccttgtctgcttccgttggagatagagttactattacttgtcaagcttcccaagatattggaatttcattgtcatggtatcaacaaaagccaggtaaggctcctaagttgttgatttacaacgctaacaacttggctgatggtgttccatctagattttctgggagtggttctggaactgattttactttgactatttcttcattgcaacctgaagattttgctacttactactgtttgcaacataactcagctccatacacttttggtcaaggaactaagttggagattaagagaactgtcgctgcaccttctgttttcatttttcctccatctgacgagcagttgaagtctggtacagcatctgttgtctgtttgttgaacaacttctacccaagagaagcaaaggttcaatggaaggttgacaacgccttgcaatctggtaactctcaggaatctgttactgaacaagattctaaggattctacttactctttgtcttctacattgacattgtctaaggctgattacgaaaagcataaggtttacgcttgtgaagttactcatcaaggattgtcttctcctgtcacaaaatcttttaacagaggtgaggtt
[0887]
seq id no:250
[0888]
agagaggctgaagctgatattcaaatgactcaatc
[0889]
seq id no:251
[0890]
tcaatgatgatgatgatgatgttaaacctcacctctgttaaaagattttg
[0891]
seq id no:252
[0892]
aggtgcagcgacagttctcttaatc
[0893]
seq id no:253
[0894]
gaactgtcgctgcaccttgtgttttc
[0895]
seq id no:254
[0896]
gaactgtcgctgcaccttctgtttgtatttttcctccatc
[0897]
seq id no:255
[0898]
gaactgtcgctgcaccttctgttttcatttgtcctccatctgac
[0899]
seq id no:256
[0900]
gaactgtcgctgcaccttctgttttcattttttgtccatctgacgagcag
[0901]
seq id no:257
[0902]
gaactgtcgctgcaccttctgttttcatttttccttgttctgacgagcagttg
[0903]
seq id no:258
[0904]
gaactgtcgctgcaccttctgttttcatttttcctccatgtgacgagcagttg
[0905]
seq id no:259
[0906]
accagattgcaaggcgttgtcaac
[0907]
seq id no:260
[0908]
gccttgcaatctggttgttctcaggaatctg
[0909]
seq id no:261
[0910]
gccttgcaatctggtaactcttgtgaatctgttactg
[0911]
seq id no:262
[0912]
gccttgcaatctggtaactctcaggaatgtgttactgaac
[0913]
seq id no:263
[0914]
gccttgcaatctggtaactctcaggaatcttgtactgaacaagattc
[0915]
seq id no:264
[0916]
gccttgcaatctggtaactctcaggaatctgtttgtgaacaagattctaag
[0917]
seq id no:265
[0918]
gccttgcaatctggtaactctcaggaatctgttacttgtcaagattctaag
[0919]
seq id no:266
[0920]
agaatccttagaatcttgttcag
[0921]
seq id no:267
[0922]
gattctaaggattcttgttactctttgtc
[0923]
seq id no:268
[0924]
gattctaaggattctacttactgtttgtcttc
[0925]
seq id no:269
[0926]
gattctaaggattctacttactctttgtgttctaca
[0927]
seq id no:270
[0928]
gattctaaggattctacttactctttgtcttcttgtttgaca
[0929]
seq id no:271
[0930]
gattctaaggattctacttactctttgtcttctacattgtgtttgtctaaggctg
[0931]
seq id no:272
[0932]
gattctaaggattctacttactctttgtcttctacattgacattgtgtaaggctg
[0933]
seq id no:273
[0934]
tcaatgatgatgatgatgatgtcaaacctcacctctgttacaagattttgt
[0935]
seq id no:274
[0936]
tcaatgatgatgatgatgatgtcaaacctcacctctacaaaaagattttgt
[0937]
seq id no:275
[0938]
tcaatgatgatgatgatgatgtcaaacctcaccacagttaaaagattttgt
[0939]
seq id no:276
[0940]
tcaatgatgatgatgatgatgtcaaacctcacatctgttaaaag
[0941]
seq id no:277
[0942]
tcaatgatgatgatgatgatgtcaaacacaacctctgttaaaag
[0943]
seq id no:278
[0944]
tcaatgatgatgatgatgatgtcaacactcacctctgttaaaag
[0945]
seq id no:279
[0946]
accagacttcaactgctcgtcaga
[0947]
seq id no:280
[0948]
gcagttgaagtctggtagagcatctgttgtctg
[0949]
在本发明提及的所有文献都在本技术中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本技术所附权利要求书所限定的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1