基于KASP技术开发的用于茶树品种鉴定的核心SNP标记
基于kasp技术开发的用于茶树品种鉴定的核心snp标记
技术领域
1.本发明属于分子标记技术领域,具体涉及基于kasp技术开发的用于茶树品种鉴定的核心snp标记。
背景技术:
2.茶树(camellia sinensis(l.)o.ktze)是山茶科、山茶属灌木或小乔木。我国作为茶树起源地,有着悠久的种植历史。近年来,随着茶树育种工作的迅速开展,大批优良茶树品种在我国得到广泛推广和种植。然而,由于骨干亲本遗传基础狭窄等导致的不同茶树品种亲缘关系比较接近,在茶树良种推广过程中,市场上同名异物或异物同名所致的品种真实性问题时有发生。而依据形态特征和生化成分去鉴别品种真实性,易受到环境与主观因素的影响。因此,迫切需要建立一套稳定可靠、易于操作的茶树品种鉴定方法。
3.伴随着基因组学和分子生物学的发展,分子标记技术已经广泛的应用于种质资源遗传多样性、品种鉴定、种质资源遗多样性、遗传图谱构建、重要性状的q tl定位、基因挖掘和辅助育种中。由于茶树基因组较大、结构复杂、遗传基础狭窄,已开发的分子标记如ssr标记,遗传多态性较低,数量还太少,限制了茶树功能基因挖掘的进程和分子标记技术在育种中的应用。
4.随着高通量测序技术的快速发展,第三代单核苷酸多态性(snp)逐渐成为应用广泛的分子标记,在植物遗传育种研究中具有广阔的应用前景。植物基因组中包含着丰富的单碱基的插入、缺失转换和颠倒等变异,利用这些变异开发的sn p分子标记位点,具备准确性高、灵活性强和通量大等特征。
5.kasp基因分型技术是一项独特的竞争性等位基因特异性pcr,可对各种基因组dna样本,基于引物末端碱基的特异匹配来对指定的snps(单核苷酸多态性)进行高精度双等位基因分型。lcg intelliqube平台通过超高的单日检测反应数(>4万个数据点)和极小的反应体系(低至1.6μl),极大降低了检测成本,通过结合全自动一体化dna抽提、荧光定量pcr工作平台,快速检测大量 snp位点,在医学、农学检测方面lgc kasp都有非常好的应用。
6.目前,利用茶树基因组信息开发的snp分子标记位点的数量还十分有限,能够鉴定的茶树品种也并不多。例如周琳等运用est
‑
snp基因分型技术得到了 60个snp标记,但仅能鉴别40个茶树品种(周琳.est
‑
snp分子标记技术在茶和咖啡中的开发与应用[d].南京农业大学,2017.),并不能满足其他茶树品种的鉴定需求。本发明利用kasp基因分型技术,通过茶树基因组信息,鉴定其中的单碱基变异,开发易于检测的snp分子标记位点,不仅丰富了茶树分子标记,还可以广泛的应用于茶树品种进行基因分型、鉴别或辅助鉴别茶树品种、检测任意两个茶树的相似性等方面的应用。
技术实现要素:
[0007]
本发明目的是为了克服现有技术的不足,提供了一种基于kasp技术开发的用于茶树鉴定的核心snp分子标记位点。本发明利用kasp基因分型技术,通过茶树基因组信息,鉴
定其中的单碱基变异,开发易于检测的snp分子标记位点,不仅丰富了茶树分子标记,还可以广泛的应用于茶树品种进行基因分型、鉴别或辅助鉴别茶树品种、检测任意两个茶树的相似性等方面的应用。
[0008]
本发明的第一个目的是提供一种检测茶树基因组中的30个snp位点的物质的应用。
[0009]
本发明的第二个目的是提供一种检测茶树30个snp位点的物质、或一种对茶树品种进行基因分型的物质、或鉴别或辅助鉴别茶树品种的物质。
[0010]
本发明的第三个目的是提供一种对茶树品种进行基因分型和/或鉴别或辅助鉴别茶树品种的成套pcr试剂。
[0011]
本发明的第四个目的是提供含有上述物质或上述pcr试剂的试剂盒。
[0012]
本发明的第五个目的是提供一种对茶树品种进行基因分型的方法。
[0013]
本发明的第六个目的是提供一种鉴别或辅助鉴别多个待测茶树品种的方法。
[0014]
为了实现上述目的,本发明是通过以下方案予以实现的:
[0015]
通过对251份茶树资源进行简化基因组测序,获得96401个snp位点,基于 (1)mis(缺失率)<0.15;(2)maf(次等位基因频率)>0.2;(3)het(杂合率)<0.2这三个筛选标准,获得2528个snp位点。利用lociscan_v1.0位点筛选工具对2528个位点进行分层筛选。获得30个snp核心snp。利用30个核心snp 位点设计引物,对72份经过国家、省级审定或其他重要茶树品种/系进行kasp 检测。
[0016]
一种检测茶树基因组的30个snp位点的物质,所述30个snp位点如下:
[0017]
所述snp位点1位于茶树基因组scaffold30:5540851,其碱基为t或c;
[0018]
所述snp位点2位于茶树基因组scaffold154:3234153,其碱基为g或a;
[0019]
所述snp位点3位于茶树基因组scaffold204:616376,其碱基为a或g;
[0020]
所述snp位点4位于茶树基因组scaffold271:731317,其碱基为t或c;
[0021]
所述snp位点5位于茶树基因组scaffold447:471126,其碱基为t或c;
[0022]
所述snp位点6位于茶树基因组scaffold465:113375,其碱基为g或a;
[0023]
所述snp位点7位于茶树基因组scaffold524:138847,其碱基为c或g;
[0024]
所述snp位点8位于茶树基因组scaffold712:1923571,其碱基为a或c;
[0025]
所述snp位点9位于茶树基因组scaffold730:2485125,其碱基为a或g;
[0026]
所述snp位点10位于茶树基因组scaffold751:785736,其碱基为g或t;
[0027]
所述snp位点11位于茶树基因组scaffold966:875493,其碱基为g或a;
[0028]
所述snp位点12位于茶树基因组scaffold968:331490,其碱基为t或c;
[0029]
所述snp位点13位于茶树基因组scaffold1154:3443945,其碱基为t或c;
[0030]
所述snp位点14位于茶树基因组scaffold1496:1222981,其碱基为g或a;
[0031]
所述snp位点15位于茶树基因组scaffold1609:330430,其碱基为c或a;
[0032]
所述snp位点16位于茶树基因组scaffold1630:523864,其碱基为g或a;
[0033]
所述snp位点17位于茶树基因组scaffold1749:443540,其碱基为g或t;
[0034]
所述snp位点18位于茶树基因组scaffold1752:204591,其碱基为a或g;
[0035]
所述snp位点19位于茶树基因组scaffold2500:575332,其碱基为g或a;
[0036]
所述snp位点20位于茶树基因组scaffold2547:285704,其碱基为c或t;
[0037]
所述snp位点21位于茶树基因组scaffold2840:1074974,其碱基为a或c;
[0038]
所述snp位点22位于茶树基因组scaffold2936:133561,其碱基为a或g;
[0039]
所述snp位点23位于茶树基因组scaffold3102:1137721,其碱基为g或a;
[0040]
所述snp位点24位于茶树基因组scaffold3135:796917,其碱基为g或a;
[0041]
所述snp位点25位于茶树基因组scaffold3315:745861,其碱基为a或g;
[0042]
所述snp位点26位于茶树基因组scaffold3538:461720,其碱基为c或t;
[0043]
所述snp位点27位于茶树基因组scaffold4372:528796,其碱基为t或c;
[0044]
所述snp位点28位于茶树基因组scaffold5112:422952,其碱基为a或g;
[0045]
所述snp位点29位于茶树基因组scaffold7623:70503,其碱基为c或t;
[0046]
所述snp位点30位于茶树基因组scaffold21357:219587,其碱基为c或g;
[0047]
所述茶树基因组为茶树基因组数据库的“舒茶早”css栽培种茶树基因组。
[0048]
检测所述30个snp位点的物质在如下任何一种中的应用:
[0049]
茶树品种进行基因分型;
[0050]
鉴别或辅助鉴别茶树品种;
[0051]
检测任意两个茶树的相似性
[0052]
一种检测所述30个snp位点的物质、或一种对茶树品种进行基因分型的物质、或鉴别或辅助鉴别茶树品种的物质,所述物质包含核苷酸序列如seq idno:1~90所示的引物组合的任意一条或几条的组合。
[0053]
一种对茶树品种进行基因分型和/或鉴别或辅助鉴别茶树品种的成套pcr试剂,由pcr试剂1、pcr试剂2、pcr试剂3、pcr试剂4、pcr试剂5、pcr试剂6、pcr试剂7、pcr试剂8、pcr试剂9、pcr试剂10、pcr试剂11、pcr试剂12、pcr试剂13、pcr试剂14、pcr试剂15、pcr试剂16、pcr试剂17、p cr试剂18、pcr试剂19、pcr试剂20、pcr试剂21、pcr试剂22、pcr试剂2 3、pcr试剂24、pcr试剂25、pcr试剂26、pcr试剂27、pcr试剂28、pcr 试剂29和pcr试剂30组成;
[0054]
所述pcr试剂1包括核苷酸序列如seq id no:1~3所示的引物组合;
[0055]
所述pcr试剂2包括核苷酸序列如seq id no:4~6所示的引物组合;
[0056]
所述pcr试剂3包括核苷酸序列如seq id no:7~9所示的引物组合;
[0057]
所述pcr试剂4包括核苷酸序列如seq id no:10~12所示的引物组合;
[0058]
所述pcr试剂5包括核苷酸序列如seq id no:13~15所示的引物组合;
[0059]
所述pcr试剂6包括核苷酸序列如seq id no:16~18所示的引物组合;
[0060]
所述pcr试剂7包括核苷酸序列如seq id no:19~21所示的引物组合;
[0061]
所述pcr试剂8包括核苷酸序列如seq id no:22~24所示的引物组合;
[0062]
所述pcr试剂9包括核苷酸序列如seq id no:25~27所示的引物组合;
[0063]
所述pcr试剂10包括核苷酸序列如seq id no:28~30所示的引物组合;
[0064]
所述pcr试剂11包括核苷酸序列如seq id no:31~33所示的引物组合;
[0065]
所述pcr试剂12包括核苷酸序列如seq id no:34~36所示的引物组合;
[0066]
所述pcr试剂13包括核苷酸序列如seq id no:37~39所示的引物组合;
[0067]
所述pcr试剂14包括核苷酸序列如seq id no:40~42所示的引物组合;
[0068]
所述pcr试剂15包括核苷酸序列如seq id no:43~45所示的引物组合;
[0069]
所述pcr试剂16包括核苷酸序列如seq id no:46~48所示的引物组合;
[0070]
所述pcr试剂17包括核苷酸序列如seq id no:49~51所示的引物组合;
[0071]
所述pcr试剂18包括核苷酸序列如seq id no:52~54所示的引物组合;
[0072]
所述pcr试剂19包括核苷酸序列如seq id no:55~57所示的引物组合;
[0073]
所述pcr试剂20包括核苷酸序列如seq id no:58~60所示的引物组合;
[0074]
所述pcr试剂21包括核苷酸序列如seq id no:61~63所示的引物组合;
[0075]
所述pcr试剂22包括核苷酸序列如seq id no:64~66所示的引物组合;
[0076]
所述pcr试剂23包括核苷酸序列如seq id no:67~69所示的引物组合;
[0077]
所述pcr试剂24包括核苷酸序列如seq id no:70~72所示的引物组合;
[0078]
所述pcr试剂25包括核苷酸序列如seq id no:73~75所示的引物组合;
[0079]
所述pcr试剂26包括核苷酸序列如seq id no:76~78所示的引物组合;
[0080]
所述pcr试剂27包括核苷酸序列如seq id no:79~81所示的引物组合;
[0081]
所述pcr试剂28包括核苷酸序列如seq id no:82~84所示的引物组合;
[0082]
所述pcr试剂29包括核苷酸序列如seq id no:85~87所示的引物组合;
[0083]
所述pcr试剂30包括核苷酸序列如seq id no:88~90所示的引物组合;
[0084]
含有上述物质或含有上述pcr试剂的试剂盒。
[0085]
上述物质或含有上述pcr试剂或上述试剂盒在对茶树品种进行基因分型和/ 或鉴别或辅助鉴别茶树品种中的应用。
[0086]
一种对茶树品种进行基因分型的方法,包括如下步骤:用核苷酸序列如se q id no:1~90所示的引物组合对待测茶树基因组dna进行pcr扩增,得到待测茶树的pcr扩增产物;对所述pcr扩增产物进行分析,确定所述待测茶树的 30个snp位点的基因型;
[0087]
所述30个snp位点如下:
[0088]
所述snp位点1位于茶树基因组scaffold30:5540851,其碱基为t或c;
[0089]
所述snp位点2位于茶树基因组scaffold154:3234153,其碱基为g或a;
[0090]
所述snp位点3位于茶树基因组scaffold204:616376,其碱基为a或g;
[0091]
所述snp位点4位于茶树基因组scaffold271:731317,其碱基为t或c;
[0092]
所述snp位点5位于茶树基因组scaffold447:471126,其碱基为t或c;
[0093]
所述snp位点6位于茶树基因组scaffold465:113375,其碱基为g或a;
[0094]
所述snp位点7位于茶树基因组scaffold524:138847,其碱基为c或g;
[0095]
所述snp位点8位于茶树基因组scaffold712:1923571,其碱基为a或c;
[0096]
所述snp位点9位于茶树基因组scaffold730:2485125,其碱基为a或g;
[0097]
所述snp位点10位于茶树基因组scaffold751:785736,其碱基为g或t;
[0098]
所述snp位点11位于茶树基因组scaffold966:875493,其碱基为g或a;
[0099]
所述snp位点12位于茶树基因组scaffold968:331490,其碱基为t或c;
[0100]
所述snp位点13位于茶树基因组scaffold1154:3443945,其碱基为t或c;
[0101]
所述snp位点14位于茶树基因组scaffold1496:1222981,其碱基为g或a;
[0102]
所述snp位点15位于茶树基因组scaffold1609:330430,其碱基为c或a;
[0103]
所述snp位点16位于茶树基因组scaffold1630:523864,其碱基为g或a;
[0104]
所述snp位点17位于茶树基因组scaffold1749:443540,其碱基为g或t;
[0105]
所述snp位点18位于茶树基因组scaffold1752:204591,其碱基为a或g;
[0106]
所述snp位点19位于茶树基因组scaffold2500:575332,其碱基为g或a;
[0107]
所述snp位点20位于茶树基因组scaffold2547:285704,其碱基为c或t;
[0108]
所述snp位点21位于茶树基因组scaffold2840:1074974,其碱基为a或c;
[0109]
所述snp位点22位于茶树基因组scaffold2936:133561,其碱基为a或g;
[0110]
所述snp位点23位于茶树基因组scaffold3102:1137721,其碱基为g或a;
[0111]
所述snp位点24位于茶树基因组scaffold3135:796917,其碱基为g或a;
[0112]
所述snp位点25位于茶树基因组scaffold3315:745861,其碱基为a或g;
[0113]
所述snp位点26位于茶树基因组scaffold3538:461720,其碱基为c或t;
[0114]
所述snp位点27位于茶树基因组scaffold4372:528796,其碱基为t或c;
[0115]
所述snp位点28位于茶树基因组scaffold5112:422952,其碱基为a或g;
[0116]
所述snp位点29位于茶树基因组scaffold7623:70503,其碱基为c或t;
[0117]
所述snp位点30位于茶树基因组scaffold21357:219587,其碱基为c或g;
[0118]
所述茶树基因组为茶树基因组数据库的“舒茶早”css栽培种茶树基因组。
[0119]
优选地,所述引物组合含有正向引物1、正向引物2和反向引物。
[0120]
优选地,所述引物组合的正向引物1的5’端加上gaaggtgaccaagttcat gct接头序列;正向引物2的引物5’端加上gaaggtcggagtcaacggatt接头序列。
[0121]
优选地,所述引物组合的正向引物1、正向引物2和反向引物带有不同的荧光基团。
[0122]
优选地,所述荧光基团分别为fam、hex和rev。
[0123]
用于扩增所述snp位点1~30对应的引物组合的核苷酸序列如表1所示:
[0124]
表1用于扩增snp位点1~30对应的引物组合的核苷酸序列编号
[0125]
snp位点编号正向引物1编号正向引物2编号反向引物编号1seq id no:1seq id no:2seq id no:32seq id no:4seq id no:5seq id no:63seq id no:7seq id no:8seq id no:94seq id no:10seq id no:11seq id no:125seq id no:13seq id no:14seq id no:156seq id no:16seq id no:17seq id no:187seq id no:19seq id no:20seq id no:218seq id no:22seq id no:23seq id no:249seq id no:25seq id no:26seq id no:2710seq id no:28seq id no:29seq id no:3011seq id no:31seq id no:32seq id no:3312seq id no:34seq id no:35seq id no:3613seq id no:37seq id no:38seq id no:3914seq id no:40seq id no:41seq id no:4215seq id no:43seq id no:44seq id no:4516seq id no:46seq id no:47seq id no:4817seq id no:49seq id no:50seq id no:5118seq id no:52seq id no:53seq id no:54
19seq id no:55seq id no:56seq id no:5720seq id no:58seq id no:59seq id no:6021seq id no:61seq id no:62seq id no:6322seq id no:64seq id no:65seq id no:6623seq id no:67seq id no:68seq id no:6924seq id no:70seq id no:71seq id no:7225seq id no:73seq id no:74seq id no:7526seq id no:76seq id no:77seq id no:7827seq id no:79seq id no:80seq id no:8128seq id no:82seq id no:83seq id no:8429seq id no:85seq id no:86seq id no:8730seq id no:88seq id no:89seq id no:90
[0126]
上述方法在鉴别或辅助鉴别多个待测茶树品种中的应用。
[0127]
一种鉴别或辅助鉴别多个待测茶树品种的方法,包括如下步骤:根据权利要求7所述的方法确定所有待测茶树品种在所述30个snp位点的基因型,根据所得基因型数据,鉴别多个待测茶树品种。
[0128]
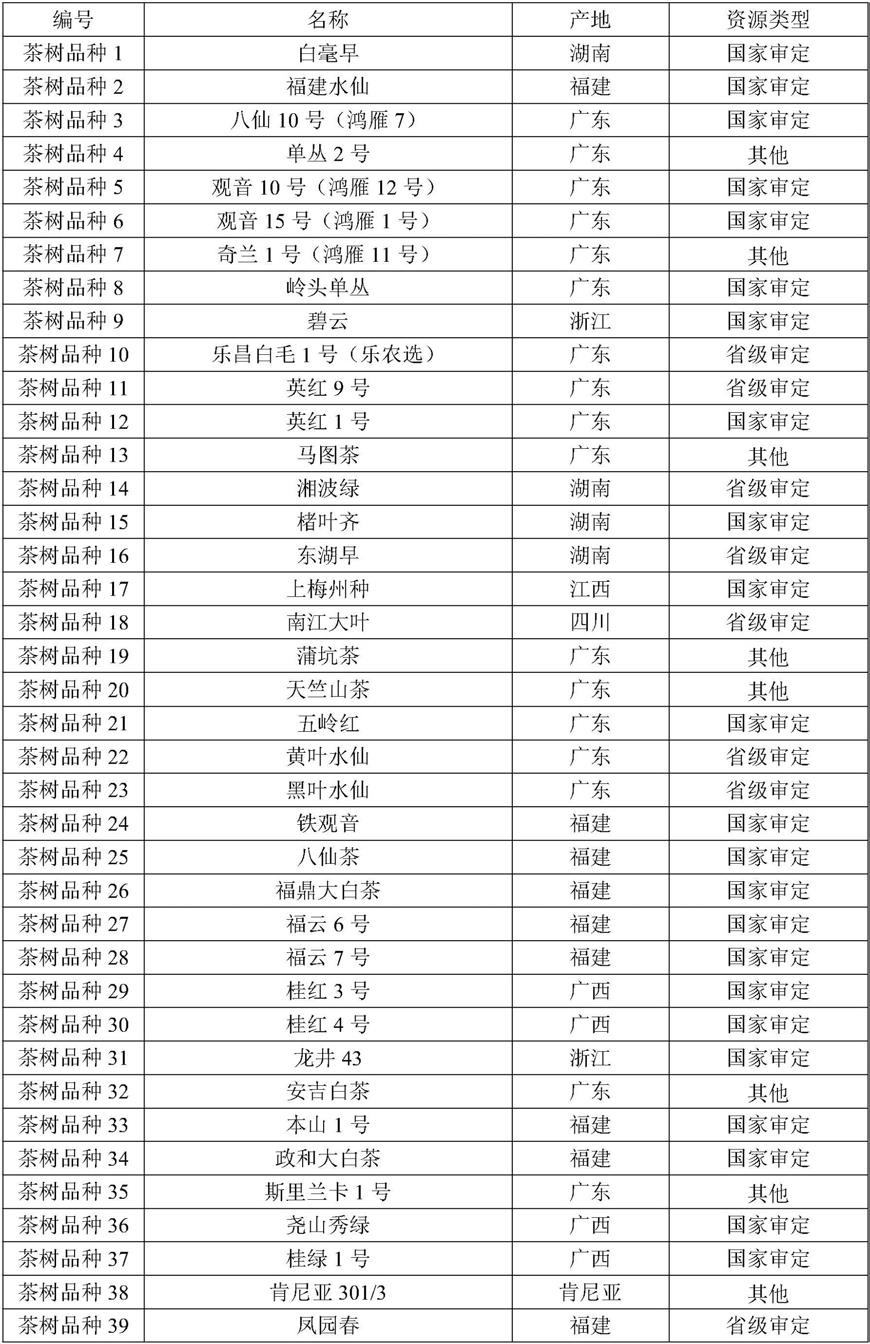
上述72份茶树品种信息如表2所示:
[0129]
表2 72份茶树品种信息
[0130]
[0131][0132]
与现有技术相比,本发明具有以下有益效果:
[0133]
本发明提供了一种基于kasp技术开发的用于茶树鉴定的核心snp分子标记位点。本发明利用kasp基因分型技术,通过茶树基因组信息,鉴定其中的单碱基变异,开发易于检测的snp分子标记位点。通过实验证明:本发明的30组 snp核心引物分布于茶树整个基因组,具有较高的多态性信息量(pic值)、最小等位基因频率(maf值)以及较低的期望杂合度,而且绝大部分位点位于基因的编码区,能够准确进行品种鉴别,并且基因分型结果稳定,有利于进行茶树品种鉴定等方面的研究,且本发明提供的snp位点和检测方法可实现高通量、低成本、标准化和稳定可靠品种真实性检测,实际操作过程还可获得实验数据的全程记录和清晰的可视化效果,为建立标准化、高通量、低成本的分子检测技术平台奠定了良好基
础,从而高效、准确的进行茶树品种的真实性和特异性鉴定。
附图说明
[0134]
图1为利用所述snp位点7对应的引物,对72份经过国家、省级审定或其他重要茶树品种进行kasp基因分型技术的检测图谱。
[0135]
图2为利用所述snp位点13对应的引物,对72份经过国家、省级审定或其他重要茶树品种进行kasp基因分型技术的检测图谱。
具体实施方式
[0136]
下面结合说明书附图和具体实施例对本发明作出进一步地详细阐述,所述实施例只用于解释本发明,并非用于限定本发明的范围。下述实施例中所使用的试验方法如无特殊说明,均为常规方法;所使用的材料、试剂等,如无特殊说明,为可从商业途径得到的试剂和材料。
[0137]
实施例1茶树snp位点的鉴定和标记开发
[0138]
1、茶树基因组数据的获取
[0139]
从茶树基因组数据库(http://tpia.teaplant.org/index.html)中下载“舒茶早
”ꢀ
css栽培种茶树基因组及其他251份茶树基因组。
[0140]
2、茶树基因组中snp标记的鉴定
[0141]
通过对251份茶树资源进行简化基因组测序,在群体水平进行snp检测和过滤,按照单个样本测序深度≥3(深度不足的位点判断为缺失),缺失率<20%, maf<5%的标准,获得96401个snp位点。
[0142]
基于mis(缺失率)<0.15、mar(次等位基因频率)>0.2和het(杂合率) <0.2这三个筛选标准,获得2528个snp位点,这2528个snp位点在染色体上分布均匀,2528个snp位点对应的识别率见表3。
[0143]
利用lociscan_v1.0位点筛选工具对2528个位点进行分层筛选,获得30个 snp核心snp,30个snp的识别率达到96.8%,并与上述2528个snp相关性极显著(也就是说这30个snp和2528个snp效果一致)。最终确定这30个 snp可以用于后续基于pcr引物的开发、茶树品种进行基因分型、鉴别或辅助鉴别茶树品种、检测任意两个茶树的相似性等。snp位点1~30在基因组上的位置信息、差异碱基及其对应引物的核苷酸序列编号见表4。
[0144]
表3 2528个snp位点对应的识别率
[0145]
snp(个)识别率(%)snp(个)识别率(%)snp(个)识别率(%)511.66096.880096.81055.68096.8100096.82092.410096.8150096.83096.820096.8200096.84096.860096.8252896.8
[0146]
表4 snp位点1~30在基因组上的位置信息及其对应引物的核苷酸序列编号
[0147]
[0148][0149]
实施例2利用茶树snp分子标记位点进行种质分析
[0150]
利用实施例1中得到的30个核心snp位点设计引物,所述snp位点1~30 在基因组上的位置信息及对应的引物的核苷酸序列如表4所示,对表5所示的 72份经过国家、省级审定或其他重要茶树品种/系进行kasp检测。snp转换成kasp标记后在72份茶树中进行验证或应用,证明这些标记确实可以用来进行品种区分/鉴定。
[0151]
主要实验材料为lgc品牌,货号为kbs
‑
1016
‑
012的kasp v4.0 2x mastermix;捷
尼斯品牌,货号为n/a的kbd引物。主要仪器为lgc品牌的intelliqube 自动点样仪和lgc品牌的hydrocycler pcr水浴系统。
[0152]
dna质检步骤为:取2μldna用于1.2%琼脂糖凝胶电泳检测,取2μldna 用于nanodrop分光光度计测浓度。
[0153]
合成引物时,所有正向引物1的5’端加上gaaggtgaccaagttcatgct 接头序列;正向引物2的5’端加上gaaggtcggagtcaacggatt接头序列,正向引物1、正向引物2和反向引物带有不同的荧光基团,所述荧光基团分别为 fam、hex和rev。
[0154]
kasp检测步骤如下:
[0155]
(1)384板每孔pcr反应体系为:2
×
kaspv4.0mastermix 0.8μl,72
×
kasp assaymix 0.022μl,dna template 0.8μl,
[0156]
根据要检测的样本数和检测的snp位点,配置相应的mastermix和assaymix 混匀,转到96孔的2ml深孔板中;
[0157]
(2)设置好相应条件,运用lgc的intelliqube仪器,让仪器将混合mix 和dna模板依次加入到384孔的pcr板上的小孔中,将pcr板进行密封;
[0158]
(3)kasp基因分型pcr扩增反应的反应程序为:
[0159]
阶段1:94℃预变性15min;阶段2:94℃20s,65~57℃(每个循环降 0.8℃)1min,共循环10次;阶段3:94℃20s,59℃ 1min,共循环27次;
[0160]
(4)pcr扩增产物的荧光扫描
[0161]
采用lgc的intelliqube仪器对pcr扩增产物进行荧光扫描,读取荧光信号。
[0162]
基因分型结果显示:每一组引物对待测茶树的分型效果均很好。其中图1和图2,分别为snp位点7和13对应的引物,对72份经过国家、省级审定或其他重要茶树品种进行kasp基因分型技术的检测图谱。对intelliqube扫描数据分析,根据分析结果按照如下确定基因的具体基因型:聚合在接近x轴的显示红色的样本的基因型为连接fam荧光标签序列的等位基因型,聚合在接近y轴上的显示蓝色的样本的基因型为连接hex荧光标签序列的等位基因型,中间显示紫色的样本的基因型为两种等位基因的杂合型,显示黑色drop out的样本可能由于dna质量不好或浓度过低或者为阴性对照,扩增产物没有被明确分型。灰色的样本indeterminate分群不明显不便判断。
[0163]
荧光信号分析结果为表6所示,为30个茶树snp位点在72份经过国家、省级审定或其他重要茶树品种的kasp基因型,其中“.”表示缺失或不确定的基因型。从图中可以看出,本发明的30组snp引物可以应用于茶树品种dna 品种的真实性检测。
[0164]
表5茶树品种信息
[0165]
编号名称产地资源类型茶树品种1白毫早湖南国家审定茶树品种2福建水仙福建国家审定茶树品种3八仙10号(鸿雁7)广东国家审定茶树品种4单丛2号广东其他茶树品种5观音10号(鸿雁12号)广东国家审定茶树品种6观音15号(鸿雁1号)广东国家审定茶树品种7奇兰1号(鸿雁11号)广东其他茶树品种8岭头单丛广东国家审定
茶树品种9碧云浙江国家审定茶树品种10乐昌白毛1号(乐农选)广东省级审定茶树品种11英红9号广东省级审定茶树品种12英红1号广东国家审定茶树品种13马图茶广东其他茶树品种14湘波绿湖南省级审定茶树品种15楮叶齐湖南国家审定茶树品种16东湖早湖南省级审定茶树品种17上梅州种江西国家审定茶树品种18南江大叶四川省级审定茶树品种19蒲坑茶广东其他茶树品种20天竺山茶广东其他茶树品种21五岭红广东国家审定茶树品种22黄叶水仙广东省级审定茶树品种23黑叶水仙广东省级审定茶树品种24铁观音福建国家审定茶树品种25八仙茶福建国家审定茶树品种26福鼎大白茶福建国家审定茶树品种27福云6号福建国家审定茶树品种28福云7号福建国家审定茶树品种29桂红3号广西国家审定
[0166]
[0167][0168]
[0169]
[0170]
[0171]
[0172][0173]
实施例3一种检测茶树snp位点的试剂盒
[0174]
利用实施例1中得到的30个核心snp位点设计引物,所述snp位点1~30 在基因组
上的位置信息及对应的引物的核苷酸序列如表4所示,对茶树品种/系进行kasp(kompetitive allele
‑
specific pcr),即竞争性等位基因特异性pcr检测。
[0175]
主要实验材料为lgc品牌,货号为kbs
‑
1016
‑
012的kasp v4.0 2x mastermix;捷尼斯品牌,货号为n/a的kbd引物。主要仪器为lgc品牌的intelliqube 自动点样仪和lgc品牌的hydrocycler pcr水浴系统。
[0176]
dna质检步骤为:取2μldna用于1.2%琼脂糖凝胶电泳检测,取2μldna 用于nanodrop分光光度计测浓度。
[0177]
合成引物时,所有正向引物1的5’端加上gaaggtgaccaagttcatgct 接头序列;正向引物2的5’端加上gaaggtcggagtcaacggatt接头序列,正向引物1、正向引物2和反向引物带有不同的荧光基团,所述荧光基团分别为 fam、hex和rev。
[0178]
kasp检测步骤如下:
[0179]
(1)384板每孔pcr反应体系为:2
×
kaspv4.0mastermix 0.8μl,72
×
kasp assaymix 0.022μl,dna template 0.8μl
[0180]
根据要检测的样本数和检测的snp位点,配置相应的mastermix和assaymix 混匀,转到96孔的2ml深孔板中。
[0181]
(2)设置好相应条件,运用lgc的intelliqube仪器,让仪器将混合mix 和dna模板依次加入到384孔的pcr板上的小孔中,将pcr板进行密封。
[0182]
(3)kasp基因分型pcr扩增反应的反应程序为:
[0183]
阶段1:94℃预变性15min;阶段2:94℃20s,65~57℃(每个循环降 0.8℃)1min,共循环10次;阶段3:94℃20s,59℃1min,共循环27次。
[0184]
(4)pcr扩增产物的荧光扫描
[0185]
采用lgc的intelliqube仪器对pcr扩增产物进行荧光扫描,读取荧光信号。
[0186]
查看基因分型结果,对intelliqube扫描数据分析,根据分析结果按照如下确定基因的具体基因型:聚合在接近x轴的显示红色的样本的基因型为连接fam 荧光标签序列的等位基因型,聚合在接近y轴上的显示蓝色的样本的基因型为连接hex荧光标签序列的等位基因型,中间显示紫色的样本的基因型为两种等位基因的杂合型,显示黑色drop out的样本可能由于dna质量不好或浓度过低或者为阴性对照,扩增产物没有被明确分型。灰色的样本indeterminate分群不明显不便判断。
[0187]
整理实验数据,进而得到了茶树snp分子标记位点在不同茶树品种的基因型。
[0188]
最后所应当说明的是,以上实施例仅用以说明本发明的技术方案而非对本发明保护范围的限制,对于本领域的普通技术人员来说,在上述说明及思路的基础上还可以做出其它不同形式的变化或变动,这里无需也无法对所有的实施方式予以穷举。凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明权利要求的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1