一种日本对虾耐低温的分子标记A257及其应用的制作方法
一种日本对虾耐低温的分子标记a257及其应用
技术领域
1.本发明属于水产动物分子标记辅助育种领域,具体涉及一种日本对虾耐低温的分子标记a257及其应用。
背景技术:
2.日本对虾(marsupenaeus japonicus)原称日本对虾,隶属十足目、对虾科、囊对虾属,分布在印度
‑
西太平洋热带、非洲东海岸、马来西亚、日本、朝鲜、中国东南沿海等海域,是中国主要对虾养殖品种之一,日本对虾生长迅速、耐干露、色泽艳丽和经济价值高,深受水产品市场和养殖者欢迎,从我国南方到北方沿海各省市均有养殖,并形成一定的规模。良种繁育对日本对虾养殖业具有重要意义。耐低温是日本对虾的重要抗逆性状。一方面,由于日本对虾具有较高的市场需求,该种对虾主要在中国北方水域进行养殖,其中包括山东、河北、江苏等北方省份的海水水域,耐低温成为中国北方日本对虾养殖业追求的重要抗逆性状;另一方面,在中国的春节时期,鲜活日本对虾的市场价格是平常季节的3倍以上,北方等地的对虾养殖农户经常冒险的将上市时间拖到冬季,又往往因冬季寒潮造成水温骤降引起养殖对虾大规模死亡,耐低温品种的选育也是日本对虾养殖业的迫切需求。
3.分子标记是以个体间遗传物质核苷酸序列变异为基础的遗传标记,是dna水平遗传多态性的直接的反映。分子标记具有显著的优越性:大多数分子标记为共显性,对隐性性状的选择十分便利;基因组变异极其丰富,分子标记的数量几乎是无限的;生物发育不同阶段、不同组织的dna都可用于标记分析;分子标记检测手段简单、迅速。目前,有关日本对虾耐低温的分子标记开发的研究较少,并且产业中缺乏可应用于分子标记辅助育种的标记。因此,开发耐低温性状相关分子标记对日本对虾的健康养殖及加速育种进程具有重要的意义。
技术实现要素:
4.本发明提供了一种日本对虾耐低温的分子标记a257及其应用。本发明的分子标记a257可以用于日本对虾低温耐受品种的筛选,且鉴定效率及准确率高,有助于日本对虾耐低温性状的培育及养殖业发展。
5.为实现上述发明目的,本发明采用以下技术方案予以实现:本发明提供了一种日本对虾耐低温的分子标记a257,所述分子标记a257的核苷酸序列如seq id no.1所示。
6.进一步的,所述分子标记a257中第510位碱基为a。
7.进一步的,所述分子标记a257的低温耐受基因型为aa纯合插入基因型。
8.本发明还提供了所述的分子标记a257的扩增引物,所述扩增引物的核苷酸序列如seq id no.2和seq id no.3所示。
9.本发明还提供了所述的分子标记a257或所述的扩增引物在筛选日本对虾耐低温品种中的应用。
10.进一步的,所述日本对虾耐低温品种的筛选步骤为:(1)合成核苷酸序列如seqidno.2和seqidno.3所示的扩增引物;(2)提取待测日本对虾的基因组dna;(3)利用扩增引物对提取的基因组dna进行pcr扩增,对扩增产物进行测序;(4)测序结果中若出现基因纯合插入突变,则该日本对虾为耐低温品种。
11.进一步的,所述步骤(4)中测序结果中出现aa基因纯合插入突变,即分子标记a257第510位碱基为a,则该日本对虾为耐低温品种。
12.进一步的,所述pcr扩增的条件为:预变性94℃,5min;变性94℃,30s,退火55℃,30s,延伸72℃,30s,重复32个循环;循环外延伸72℃,7min。
13.本发明还提供了所述的分子标记a257或所述的扩增引物在日本对虾遗传多样性分析、种质鉴定和遗传图谱构建中的应用。
14.本发明与现有技术相比,具有以下优点和有益效果:1、本发明提供的日本对虾耐低温的分子标记a257可以不受日本对虾生长阶段的限制,能够用于日本对虾早期虾苗及亲本的选育,进而明显加快日本对虾的选育进程以及快速选育出具备低温耐受优良性状的日本对虾新品种。2、利用本发明提供的分子标记a257及其扩增产物检测日本对虾耐低温的性状,方法准确可靠、操作简单,能够有效、快速的筛选出符合要求的性状,辅助早期实现短时间、低成本的选育出耐受低温的日本对虾的品种,增加优良品质的日本对虾数量,提高日本对虾的养成率及养殖周期,进而提高日本对虾产量,促进其健康繁殖,因此具有广阔的应用前景。
附图说明
图1:本发明中低温耐受群体和低温不耐受群体两组对应位置测序结果差异峰图。
具体实施方式
15.以下结合具体实施例对本发明的技术方案做进一步详细的说明。
16.本发明所用的日本对虾均来自青岛瑞兹海珍品有限公司实验基地。体重10
±
3g的日本对虾300只,放置于4个养殖池(500cm
×
300cm
×
150cm)中暂养7d,暂养期间水温保持在28
±
1℃,加水至20cm,ph8.2
±
0.5,持续供氧,每天早上8点更换新鲜海水,下午5点投喂新鲜蛤蜊肉,喂食量约为对虾体重的10%。7d后挑选活力好、体表完整的日本对虾进行后续实验。
17.以10
±
0.5℃作为应激条件,对300尾日本对虾进行低温耐受性实验,统计个体的耐受时间,将首先死亡的20尾虾作为低温敏感组(ut),将最后仍然存活的20尾虾作为低温耐受组(t)。利用天根海洋动物组织基因组提取试剂盒(tiangen,天根生化科技有限公司)分别提取低温不耐受组和低温耐受组的日本对虾的基因组dna。
18.实施例1一、耐低温相关候选分子标记筛选1、测序数据过滤和比对提取日本对虾的基因组dna,低温耐受组和不耐受组各20个个体。利用可以特异性结合dna的离心吸附柱和独特的缓冲液系统提取日本对虾基因组dna。首先,将大约30mg的
组织样品放到1.5 ml 的无菌酶离心管,加入200 μl ga缓冲液和20 μl proteinase k溶液,漩涡混匀,56℃孵育至组织完全裂解。然后加入200 μl gb缓冲液,充分颠倒混匀,70℃放置10min,溶液变清亮,剪短离心出去内壁水珠。加入200 μl 无水乙醇,充分颠倒混匀,此时可能会出现絮状沉淀,简短离心,然后将所得溶液和絮状沉淀都加入一个吸附柱cb3中(吸附柱放入收集管中),12000 rpm离心30s,倒掉废液,将吸附柱cb3放回收集管中。向吸附柱cb3中加入500 μl缓冲液gd,12000 rpm离心30s,倒掉废液,将吸附柱cb3放入收集管中。向吸附柱cb3中加入600 μl 漂洗液pw,12000 rpm离心30s,倒掉废液,将吸附柱cb3放入收集管中。弃废液(重复一次),再加入700 μl漂洗液pw,12000 rpm离心30s,弃废液。12000 rpm空离2min,以彻底除去残留的试剂。将吸附柱cb3置于干净的离心管中,在柱的中心加入40 μl水,室温静置15min,12000 rpm离心2min,洗脱dna。利用nanodrop检测dna的浓度和纯度。
19.将检验合格的dna样品包括低温耐受组和低温不耐受组单个dna样品分别通过covaris破碎机随机打断成长度为350bp的片段,采用truseq library construction kit进行建库,dna片段经末端修复、加ploya尾、加测序接头、纯化、pcr扩增等步骤完成整个文库制备。构建好的文库通过illumina hiseq pe150进行测序。
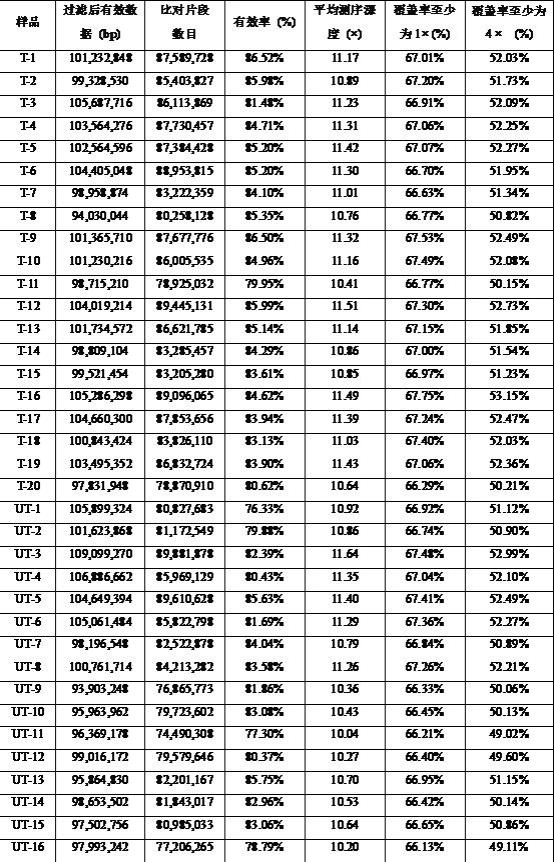
20.经过对测序数据的严格过滤,得到高质量的clean data。对40个样本产出数据进行统计,包括测序过滤后有效数据、比对片段数目等。总的测序数据量为611.10 gb,高质量的clean data数据量为605.67 gb,测序数据结果见表1。
21.表1 :测序数据质量汇总
备注:t代表低温耐受,ut代表低温不耐受。
22.过滤后的有效数据通过burrows
‑
wheeler alignment tool(bwa)软件进行比对,
比对结果经samtools去除重复。比对结果表明所有样本的比对率在85%以上,平均测序深度在25
×
以上,可用于后续分析。
23.2、标记检测及注释indel检测采用samtools软件进行。利用贝叶斯模型检测群体中的多态性位点,通过以下过滤和筛选得到高质量的indels:1)q20质量控制;2)indel位点彼此间至少相隔5 bp;3)indel的支持数(覆盖深度)在平均深度的[1/ 3, 5]倍之间。
[0024]
然后利用annovar对indel检测结果进行注释。检测结果见表2,该indel结果是按照深度(dp)大于4,缺失率0.2,最小等位基因频率0.05过滤后进行的统计。
[0025]
表2:indel检测结果统计分组indel数量上游27,922外显子87内含子392,013剪接体224下游33,304基因间1,132,068total1,585,6183、indel差异分析按照如下方法对低温耐受组和低温不耐受组的每个位点的indel
‑
index进行分析统计: var_t = 发生变异个体 (杂合突变0/1,纯合突变1/1) /耐受组个体数目;var_ut = 发生变异个体 (杂合突变0/1,纯合突变1/1)/非耐受组个体数目。
[0026]
同时计算indel频率差异分布,方向如下:δ(index) = var_ut (低温耐组)
‑ꢀ
var_t (低温不耐受组),过滤掉δindex绝对值小于0.55的位点,最后得到组间差异的indel共51个,然后对这些标记进行验证。
[0027]
二、耐低温相关分子标记验证采用pcr产物测序的方法在低温耐受群体和低温不耐受群体里对耐低温性状相关候选分子标记进行验证:(1)首先在标记位点侧翼序列设计引物,其中至少有一条引物距离标记位点70 bp以上;(2)利用设计好的引物分别以低温耐受群体和低温不耐受群体混合dna材料为模板进行pcr扩增,并将成功扩增的pcr产物进行测序,测序引物选择离标记位点较远的引物;(3)利用bio
‑
edit软件分析测序峰图,挑选低温耐受群体和低温不耐受群体在对应位置测序峰图有较大差异的标记继续进行个体dna模板的pcr扩增和测序分析;(4)根据测序结果统计每个个体的基因型,并通过spss软件分析标记与耐低温性状是否相关。
[0028]
具体的操作步骤如下:
1、dna混合池构建每个个体分别取1 μg dna进行混合,制备低温耐受组和低温不耐受组dna混合池,终浓度稀释至为200 ng/μl,每个混合池中包含20个个体,用于后续实验。2、引物设计通过比较两个群体中基因组测序和生物信息学分析方法筛选到indel突变位点,然后筛选耐低温分子标记。利用primer premier 5.0软件,在耐低温候选indel位点两端设计引物。
[0029]
引物的设计标准为:(1)引物退火温度55
‑
60℃;(2)尽量避免引物本身、引物之间形成稳定的二聚体和发夹结构;(3)片段大小大于200 bp。
[0030]
设计得到的引物信息见表3:表3:引物信息3、混合模板pcr扩增及电泳用混合gdna为模板,利用康为世纪公司酶进行pcr扩增,设置反应程序为:预变性94℃,5min;变性94℃,30s,退火55℃,30s,延伸72℃,30s,重复32个循环;循环外延伸72℃,7min。pcr体系如下:2
×
matermix10μl正向引物0.5μl反向引物0.5μlgdna2μlddh2o7μl总体积20μl将所得到的pcr扩增产物先进行琼脂糖凝胶电泳。剩余产物进行一代测序;若测序结果峰图在510位碱基处出现a纯合插入,则该日本对虾为耐低温个体,若测序峰图在在510位碱基处没有出现a纯合插入,则该日本对虾为低温不耐受个体。
[0031]
再根据测序结果统计每个个体的基因型,并将基因型信息导入spss软件,计算p值,选出p<0.01标记,最后选择出1个indel标记,编号为a257。
[0032]
分子标记a257的低温耐受基因型为aa基因纯合插入型,其核苷酸序列如seq id no.1所示,其中第510位碱基为a,开发该分子标记的扩增引物为表3中所示的a257
‑
f和a257
‑
r。
[0033]
4、低温耐受个体和低温不耐受个体pcr扩增及电泳提取40个日本对虾个体的组织dna作为模板进行pcr,pcr引物为:a257
‑
f: atgcaagcatctgcacagg;
a257
‑
r: ccaggcgtacactttaggaat。
[0034]
pcr体系和扩增条件均与上述相同。
[0035]
扩增后,将得到的个体模板pcr产物进行1%的琼脂糖凝胶电泳。然后将剩余pcr产物进行测序,测序结果见图1,低温耐受的日本对虾的碱基突变为a基因纯合插入。
[0036]
综上,本发明的分子标记a257能够准确鉴定日本对虾中具有耐低温性状的个体,应用步骤可概括为:提取日本对虾样本中的基因组dna,以提取的基因组dna为模板,使用分子标记的扩增引物a257
‑
f和a257
‑
r进行pcr扩增反应;对pcr扩增产物进行琼脂糖凝胶电泳并测序,如果测序结果中a257的基因型为aa,显示有纯合插入突变,即在分子标记a257第510位碱基为a,则该日本对虾样本具有耐低温性状。分子标记a257还能够用鉴定日本对虾耐低温亲本和苗种,有助于日本对虾的养殖发展。
[0037]
以上实施例仅用以说明本发明的技术方案,而非对其进行限制;尽管参照前述实施例对本发明进行了详细的说明,对于本领域的普通技术人员来说,依然可以对前述实施例所记载的技术方案进行修改,或者对其中部分技术特征进行等同替换;而这些修改或替换,并不使相应技术方案的本质脱离本发明所要求保护的技术方案的精神和范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1