生物标志物在诊断脑卒中中的应用的制作方法
1.本发明属于生物医药领域,具体涉及生物标志物在诊断脑卒中中的应用。
背景技术:
2.脑卒中已成为全球第二大死因。根据2013年全球疾病负担研究的估算,2013年全球共有2570万脑卒中患者,其中缺血性脑卒中1830万,出血性脑卒中740 万;新发病例脑卒中有1030万(参见feigin. update on the global burden of ischemic and hemorrhagic stroke in 1990-2013: the gbd 2013 study (vol 45, pg 161, 2015)[j]. neuroepidemiology, 2017.)。2013年脑卒中导致的死亡占全球所有死亡的12%。据估计,约有485万例脑卒中死亡发生在中低收入国家,而160万例发生在高收入国家。脑卒中不仅给患者带来身体和心理上的痛苦,同时给家庭和社会造成沉重的经济负担。
[0003]
脑卒中病后常见的不良结局包括功能残疾、死亡、卒中再发和心血管事件。脑卒中是一种由多种因素(包括基因与环境因素)所导致的复杂性状疾病。长期以来,脑卒中的诊断主要依赖于临床查体和各种神经成像技术,虽然脑电子计算机x射线断层扫描技术(ct)可能有效排除脑卒中,但对于脑卒中的诊断特异性并不强,而磁共振成像(mri)虽然在脑卒中诊断中具有重要的参考价值,但由于中国的普及率低及费用高等现实问题,亦难以被广泛使用。因此,开发快速诊断生物标志物具有显著的临床应用潜力。
技术实现要素:
[0004]
本发明提供了脑卒中的诊断方法。更具体地,本发明提供了可用于诊断脑卒中的生物标志物。
[0005]
基于发明人的发现:与正常人相比,脑卒中患者血液样本中的生物标志物水平发生显著变化,所述的的生物标志物包括ceacam6、folr3、tpst1。
[0006]
第一方面,本发明提供了一种检测样本中生物标志物水平的试剂,所述的生物标志物包括ceacam6、folr3和/或tpst1。
[0007]
如本文中在诸如
ꢀ“
a和/或b
”ꢀ
的短语中使用的术语
ꢀ“
和/或
”ꢀ
旨在包括a和b两者;a或b;a(单独) ;以及b(单独)。同样地,在诸如
ꢀ“
a、b和/或c
ꢀ”ꢀ
的短语中使用的术语
ꢀ“
和/或
”ꢀ
旨在涵盖以下实施方案的每一个:a、b和c;a、b或c;a或c;a或b;b或c;a和c;a和b;b和c;a(单独) ;b(单独);以及c(单独)。
[0008]
术语
ꢀ“
生物标志物
”ꢀ
是指可用于预测个体的疾病状态的不同浓度存在于个体中的生物分子。生物标志物可包括,但不限于,核酸、蛋白质及其变体和片段。生物标志物可以是包含编码该生物标志物的全部或部分核酸序列或这类序列的互补体的dna。可用于本发明的生物标志物核酸被认为包括包含任何目的核酸序列的全部或部分序列的dna和rna。
[0009]
在本发明中,生物标志物例如ceacam6(geneid:4680)、folr3(2352)、tpst1(geneid:8460),包括gene及其编码的蛋白及其同源物,突变,和同等型。该术语涵盖全长,未加工的生物标志物,以及源自细胞中加工的任何形式的生物标志物。该术语涵盖生物标
志物的天然发生变体(例如剪接变体或等位变体)。
[0010]
进一步,所述的试剂包括:特异性识别ceacam6、folr3和/或tpst1基因的寡核苷酸探针;特异性扩增ceacam6、folr3和/或tpst1基因的引物;或特异性结合ceacam6、folr3和/或tpst1编码的蛋白的结合剂。
[0011]
术语“探针”指能与另一分子的特定序列或亚序列或其它部分结合的分子。除非另有指出,术语“寡核苷酸探针”通常指能通过互补碱基配对与另一多核苷酸(往往称为“靶多核苷酸”)结合的多核苷酸探针。根据杂交条件的严谨性,探针能和与该探针缺乏完全序列互补性的靶多核苷酸结合。
[0012]
本发明中的示例性寡核苷酸探针包括pcr引物以及基因特异性dna寡核苷酸探针,例如固定于微阵列基底上的微阵列探针、定量核酸酶保护检验探针、与分子条形码连接的探针、以及固定于珠上的探针。
[0013]
这些探针具有与靶点基因的特定的碱基序列互补的碱基序列。这里,所谓“互补”,只要是杂交即可,可以不是完全互补。这些多核苷酸通常相对于该特定的碱基序列具有80%以上、优选90%以上、更优选95%以上、特别优选100%的同源性。这些探针可以是dna,也可以是rna,另外,可以为在其一部分或全部中核苷酸通过pna(polyamide nucleicacid,肽核酸)、lna(注册商标,locked nucleic acid,bridged nucleic acid,交联化核酸)、ena(注册商标,2
′‑
o,4
′‑
c-ethylene-bridged nucleic acids)、gna(glycerolnucleic acid,甘油核酸)、tna(threose nucleic acid,苏糖核酸)等人工核酸置换得到的多核苷酸。
[0014]“引物”是指寡核苷酸,它与靶核酸中的序列(“引物结合位点”)杂交并且能够用作在适用于合成的条件下沿着核酸的互补链启动该合成的点。
[0015]
蛋白的结合剂是例如蛋白质的受体、结合蛋白质的凝集素、针对蛋白质的抗体、针对蛋白质的肽抗体(peptidebody)、双特异性双重结合剂或双特异性抗体形式。
[0016]
进一步,所述的样本包括血液、鼻上皮细胞、组织、尿液、唾液、精液、乳汁、脑脊髓液、泪液、痰、粘液、淋巴、胞液、腹水、胸膜积液、羊水、膀胱冲洗液、或支气管肺泡灌洗液。在本发明的具体实施例中,所述的样本为血液。
[0017]
生物标志物水平的测定可以是对患者的一个或多个样品进行测定。患者样品可以用本领域常用方法采集。
[0018]
第二方面,本发明提供了前面所述试剂在制备诊断脑卒中的产品中的应用。
[0019]
进一步,所述的脑卒中包括缺血性脑卒中、出血性脑卒中。
[0020]
进一步,所述的脑卒中为缺血性脑卒中。
[0021]
进一步,所述的缺血性脑卒中包括大动脉粥样硬化性卒中、心源性脑栓塞、小动脉闭塞性卒中、或其他原因引发的缺血性卒中。在本发明的具体实施例中,所述的缺血性脑卒中为心源性脑栓塞。
[0022]
进一步,所述的产品包括核酸膜条、制剂、芯片、试剂盒。
[0023]
在本发明中芯片包括基因芯片、蛋白芯片;所述基因芯片包括固相载体;以及有序固定在所述固相载体上的寡核苷酸探针,所述的寡核苷酸探针特异性地对应于前面所述生物标志物所示的部分或全部序列。所述蛋白芯片包括固相载体,以及固定在固相载体上的前面所述生物标志物编码的蛋白的特异性抗体或配体。
[0024]
在本发明中,核酸膜条包括基底和固定于所述基底上的寡核苷酸探针;所述基底可以是任何适于固定寡核苷酸探针的基底,例如尼龙膜、硝酸纤维素膜、聚丙烯膜、玻璃片、硅胶晶片、微缩磁珠等。其中示例性探针包括pcr引物以及基因特异性dna寡核苷酸探针,例如固定于微阵列基底上的微阵列探针、定量核酸酶保护检验探针、与分子条形码连接的探针、以及固定于珠上的探针。
[0025]
本发明提供了一种试剂盒,所述试剂盒可用于检测前面所述生物标志物的水平。所述试剂盒包括用于扩增前面所述生物标志物的特异性引物对;标准dna模板;pcr反应液。
[0026]
在一个优选的实施方案中,所述特异性引物对包括上游引物和下游引物。
[0027]
诊断疾病的准确性最好是由其受试者工作特征 (receiver-operating characteristics,roc)来描述(zweig ,m .h .和campbell ,g.,临床化学39(1993)561-577)。roc曲线图是所有灵敏度/特异性的图,所述灵敏度/特异性对是通过在观察到的整个数据范围内不断变化判定阈值而得到的。roc曲线图通过绘制完整判定阈值范围内的灵敏度与1-特异性曲线示出了两个分布间的重叠部分。y轴是灵敏度,或是定义为[(真阳性测试结果数)/(真阳性+假阴性测试结果数)]的真阳性率。在疾病或病症情况中,这也被称为阳性。它仅由受影响的子群计算得到。x轴是假阳性率,或是1-特异性[定义为:(假阳性结果数)/(真阴性+假阳性结果数)]。这是特异性的一个指标,并完全由未受影响的子群计算得到。因为真、假阳性率是通过两个不同子群的测试结果而完全独立地计算地,roc曲线图与样品中的患病率无关。roc曲线图中的每一个点表示与一个特定判定阈值对应的一个灵敏度/特异性对。一个具有优秀鉴别力的测试(两个结果的分布没有重叠)的roc曲线图穿过左上角,其中真阳性率为1.0或100%(完美的灵敏度),并且假阳性率为0(完美的特异性)。一个没有鉴别力的测试的理论图(两个群的结果分布相同)是一条从左下角到右上角的45
°
斜线。大多数曲线图落在这两个极端之间。定性地,曲线图离左上角越近,测试的整体准确性越高。
[0028]
将实验室测试的诊断准确性量化的一个目的是用单个数字来表示实验测试的表现。最常见的计量是roc曲线图中的曲线下面积(auc)。roc曲线下面积是一种概率的度量,使病症的正确鉴别成为可能。按照惯例,这个面积通常大于0.5。数值范围在1.0(两个群的测试数值完全分离)和0.5(两组测试数值之间没有明显的分布差异)之间。此面积不仅依赖于曲线图的某一特定部分,比如最接近斜线的点或特异性为90%的灵敏度,而取决于整个曲线图。这定量地、描述性地表达了roc曲线图与完美曲线(面积=1.0)的相近程度。本发明人进一步发现,检测两种或以上的生物标志物组合水平的变化可以用于诊断脑卒中,所述的两种或以上的生物标志物选自由ceacam6、folr3、tpst1组成的组。在本发明的背景下,表2列出了两种或以上的生物标志物组合的auc值,所述生物标志物组合的auc值大于任何一个单独的生物标志物的auc值。
[0029]
第三方面,本发明提供了一种筛选治疗脑卒中的候选化合物的方法,所述的方法包括:(1)在测试组中,向待测对象施用测试化合物,检测测试组中来源于所述对象的样品中生物标志物的水平v1;在对照组中,向待测对象施用空白对照,检测对照组中来源于所述对象的样品中生物标志物的水平v2;(2)比较上一步骤检测得到的水平v1和水平v2,从而确定所述测试化合物是否是
治疗脑卒中的候选化合物;所述的生物标志物包括ceacam6、folr3和/或tpst1。
[0030]
进一步,所述的样本包括血液、鼻上皮细胞、组织、尿液、唾液、精液、乳汁、脑脊髓液、泪液、痰、粘液、淋巴、胞液、腹水、胸膜积液、羊水、膀胱冲洗液、或支气管肺泡灌洗液,优选为血液。
[0031]
进一步,所述的脑卒中包括缺血性脑卒中、出血性脑卒中,优选的,所述的脑卒中为缺血性脑卒中。
[0032]
进一步,所述的缺血性脑卒中包括大动脉粥样硬化性卒中、心源性脑栓塞、小动脉闭塞性卒中、或其他原因引发的缺血性卒中。
[0033]
进一步,所述的缺血性脑卒中为心源性脑栓塞。
[0034]
第四方面,本发明提供了一种诊断对象是否患有脑卒中或预测对象是否存在患脑卒中的风险的系统或装置,所述的系统/装置包括:分析单元,所述单元适于测量对象样本中生物标志物的量;和评估单元,其包含存储的参考和数据处理器,所述数据处理器已经实现了用于比较分析单元测量的生物标志物的量与存储的参考的算法,由此诊断对象是否患有脑卒中或预测对象是否存在患脑卒中的风险;所述的生物标志物包括ceacam6、folr3和/或tpst1。
[0035]
进一步,所述的脑卒中包括缺血性脑卒中、出血性脑卒中。
[0036]
进一步,所述的脑卒中为缺血性脑卒中。
[0037]
进一步,所述的缺血性脑卒中包括大动脉粥样硬化性卒中、心源性脑栓塞、小动脉闭塞性卒中、或其他原因引发的缺血性卒中。
[0038]
进一步,所述的缺血性脑卒中为心源性脑栓塞。
[0039]
第五方面,本发明提供了一种计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现前面所述的系统/装置。
[0040]
第六方面,本发明提供了生物标志物在筛选治疗脑卒中的候选化合物中的应用,所述的生物标志物包括ceacam6、folr3和/或tpst1。
[0041]
本发明的优点和有益效果:本发明公开了一种用于诊断脑卒中的生物标志物,所述的生物标志物包括ceacam6、folr3和/或tpst1。
[0042]
本发明提供了一种筛选治疗脑卒中的候选化合物的方法。
[0043]
本发明还提供了一种诊断对象是否患有脑卒中或预测对象是否存在患脑卒中的风险的系统或装置。
[0044]
附图说明
[0045]
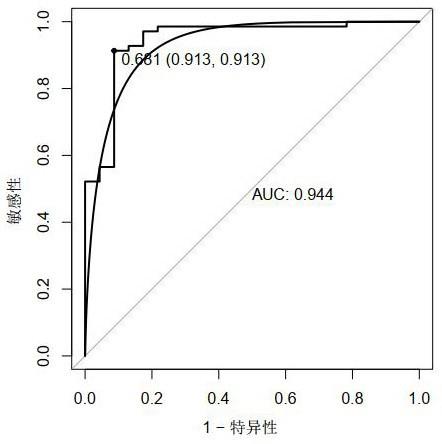
图1为ceacam6差异表达的箱线图;图2为folr3差异表达的箱线图;图3为tpst1差异表达的箱线图;图4为ceacam6诊断心源性脑栓塞的roc曲线图;
图5为folr3诊断心源性脑栓塞的roc曲线图;图6为tpst1诊断心源性脑栓塞的roc曲线图;图7为ceacam6和folr3联合诊断心源性脑栓塞的roc曲线图;图8为ceacam6和tpst1联合诊断心源性脑栓塞的roc曲线图;图9为folr3和tpst1联合诊断心源性脑栓塞的roc曲线图;图10为ceacam6+folr3+tpst1联合诊断心源性脑栓塞的roc曲线图。
具体实施方式
[0046]
以下结合附图对本技术的具体实施方式进行详细说明。应当理解的是, 此处所描述的具体实施方式仅用于说明和解释本技术,并不用于限制本技术。
[0047]
实施例1 高通量测序分析1、样本采集收集4例脑卒中患者和4例健康对照者的血液样本。
[0048]
1.1脑卒中患者纳入标准:(1)诊断为脑卒中;(2)尚未接受药物方式或手术等任何针对脑卒中的治疗;(3)无感染性、自身免疫性或严重慢性疾病。
[0049]
1.2健康对照者的纳入标准:(1)男性或女性志愿者,年龄在20到90岁之间;(2)身体健康,没有任何慢性、感染性或遗传学疾病;(3)近期没有服用常规处方药;(4)没有献血、怀孕,在研究开始前的3个月内没有接种过疫苗;(5)非过敏体质;(6)经常规体检确认身体健康。
[0050]
2、实验方法2.1 血液总rna的提取使用paxgene血液rna提取试剂盒(paxgene blood rna kit)提取外周血总rna。
[0051]
2.2样品检测使用agilent 2100 bioanalyzer (agilent rna 6000 nano kit)检测 total rna 的浓度、rin值、28s/18s和片段大小。
[0052]
2.3文库的构建与转录组测序1)dnase 消化去除dna:利用dnase i 消化total rna 样品中存在的dna 片段,磁珠纯化回收反应产物,最后溶解于depc 水;2)去除 rrna:取消化好的total rna 样品,使用试剂盒去除rrna,去除之后进行agilent 2100 检测,验证rrna 去除效果;3) rna 打断:取上一步样品,加入打断buffer,置于pcr 仪中进行热打断,打断到130-160nt;4)反转录一链的合成:向打断后的样品中加入适量引物,充分混匀后在 thermomixer 适温反应一定时间,使之打开二级结构并与引物结合,再加入提前配制的一
链合成反应体系mix,在pcr仪上按相应程序合成一链cdna;5)反转录二链的合成:配制二链合成反应体系,在thermomixer上适温反应一定时间,合成二链cdna,反应产物用磁珠进行纯化回收。产物用磁珠进行纯化回收;6)末端修复:配制末端修复反应体系,在thermomixer中适温反应一定时间,在酶的作用下,对反转录得到的cdna双链的粘性末端进行修复。末端修复产物用磁珠进行纯化回收,最后将样品溶于ebsolution;7)cdna末端加“a”:配制加“a”反应体系,在thermomixer中适温反应一定时间,在酶的作用下,使末端修复的产物cdna的3’末端加上a碱基;8)cdnaadapter的连接:配制接头连接反应体系,在thermomixer中适温反应一定时间,在酶的作用下,使接头与a碱基连接,产物用磁珠进行纯化回收;9)pcr反应及产物回收:配制pcr反应体系,选择适当的pcr反应程序,对上步骤得到的产物进行扩增。对pcr产物进行磁珠纯化回收。回收产物溶于eb溶液中。贴上标签,文库制备至此完成;10)文库质量检测:使用agilent2100bioanalyzer(agilentdna1000reagents)检测文库的片段大小及浓度;11)pcr产物环化:将pcr产物变性为单链后,配制环化反应体系,充分混匀适温反应一定时间,得到单链环形产物,消化掉未被环化的线性dna分子后,即得到最终的文库;12)上机测序:单链环状dna分子通过滚环复制,形成一个包含200多个拷贝的dna纳米球(dnb)。将得到的dnbs采用高密度dna纳米芯片技术,加到芯片上的网状小孔内。通过边合成边测序的方法,得到50bp/100bp的测序读长。
[0053]
2.4测序数据质量控制对原始测序数据进行过滤,从而得到高质量的测序数据(cleandata),步骤如下:去除reads中的adapter序列;剪切前去除5’端含有非agct的碱基;修剪测序质量较低的reads末端(测序质量值小于q20);去除含n的比例达到10%的reads;舍弃去adapter及质量修剪后长度小于25bp的小片段。
[0054]
2.5与参考基因组比对使用hisat2分析软件将测序数据比对到参考基因组上。参考基因组来自于ensembl数据库。
[0055]
2.6基因表达水平分析通过比对到参考基因组区域的序列(cleanreads)的数量来计算基因的表达水平。使用stringtie根据hisat2软件的比对结果,计算每个基因/转录本在样本中的fpkm值,以该值作为基因/转录本在样本中的表达量。
[0056]
2.7mrna差异表达分析使用deseq2比较对照组跟疾病组mrna的表达差异,差异分析步骤如下:首先对原始的readcount进行标准化(normalization),主要是对测序深度的校正;通过统计学模型进行假设检验概率(p-value)的计算,进行多重假设检验校正(bh),得到padj值(错误发现率),差异表达基因的筛选标准为:pvalue《0.05和|log2foldchange|》1。
[0057]
3、结果与分析
与对照组相比,本发明所涉及的生物标志物ceacam6、folr3、tpst1在脑卒中患者中表达显著上调,差异具有统计学意义。
[0058]
实施例2 生物标志物表达情况以来源于数据库的患有心源性脑栓塞受试者(n=23,女性11例,男性12例)、无症状性血管疾病史的受试者(n=23;11名女性,12名男性)的血液作为样本(数据集为gse58294),对基因表达数据进行差异分析。把对应到多个基因的探针去掉,然后有多个探针对应的基因只保留平均表达量最大的一个探针。对数据集scale标准化。差异分析使用工具为r-4.0.5,采用metama、limma包分析,meta分析中p值合并所用方法为inverse normal method。metama包对p、fdr值计算的描述(calculates differential expression p-values and effect sizes from data either from classical or moderated t-tests (limma, smvar) for study and combines these p-values by the inverse normal method,fdr值计算:benjamini hochberg threshold. by default, the false discovery rate is controlled at 5%)。
[0059]
差异表达基因筛选标准:adj.p.val 《0.05&|logfc|》1,得到208个差异表达基因,本发明所涉及的生物标志物在心源性脑栓塞受试者中的表达情况如表1、图1-3所示,所述生物标志物在疾病组中均是表达上调的,差异具有统计学意义。
[0060]
表1 生物标志物在心源性脑栓塞患者血液样本中的表达情况实施例3 诊断效能受试者操作特征曲线(receiver operating characteristic, roc)能够直观地鉴别各诊断指标的诊断效能,roc曲线越靠近左上角,曲线下面积(area under curve, auc)越大,诊断价值越高(参考the meaning and use of the area under a receiver operating characteristic (roc) curve.[j]. radiology, 1982)。本发明使用r语言proc对上述基因进行roc诊断分析,以筛选具有诊断价值的生物标志物。
[0061]
结果:auc>0.5的情况下,auc值越接近1,表明诊断标志物的诊断效果越好。基于geo数据集,本发明利用r语言绘制了本发明所涉及的基因的roc曲线。结果如图4-6所示,本发明所述的基因的auc值基本位于0.65~0.85,其中,ceacam6(auc=0.689),folr3(auc=0.826),tpst1(auc=0.841),进一步分析生物标志物组合的诊断效能,如图7-10,表2所示。
[0062]
表2 生物标志物组合诊断效能
与单个生物标志物相比,生物标志物组合具有更好的诊断效能。上述结果证明,本发明所涉及的生物标志物可用于诊断心源性脑栓塞。
[0063]
以上结合附图详细描述了本技术的优选实施方式,但是,本技术并不限于上述实施方式中的具体细节,在本技术的技术构思范围内,可以对本技术的技术方案进行多种简单变型,这些简单变型均属于本技术的保护范围。另外需要说明的是,在上述具体实施方式中所描述的各个具体技术特征,在不矛盾的情况下,可以通过任何合适的方式进行组合,为了避免不必要的重复,本技术对各种可能的组合方式不再另行说明。此外,本技术的各种不同的实施方式之间也可以进行任意组合,只要其不违背本技术的思想, 其同样应当视为本技术所公开的内容。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1