三角褐指藻ZEP1基因、蛋白及应用
三角褐指藻zep1基因、蛋白及应用
技术领域
1.本发明属于生物技术领域,具体涉及三角褐指藻zep1基因、蛋白及应用。
背景技术:
2.类胡萝卜素广泛存在于光合生物中,具有抗氧化与捕捉光能两重特性。由于结构的多样性,不同的类胡萝卜素具有不同的应用价值。多种类胡萝卜素,例如岩藻黄素、虾青素等具有较高的市场价值。近年来,除了抗氧化,岩藻黄素还被报道具有抗癌症的功能。
3.此外,岩藻黄素在光合作用光能捕捉方面具有重要应用。蓝绿光在海水中穿透能力强,因而海洋藻类大多含有岩藻黄素,用于捕捉蓝绿波段可见光。岩藻黄素如被转移到其他光合生物中,可用于拓宽捕光光谱,提高光合作用效率。
4.目前,岩藻黄素的合成通路尚未被完全解析。更没有关于合成岩藻黄素的基因的相关报道。
技术实现要素:
5.本发明的目的之一在于提供一种三角褐指藻(phaeodactylum tricornutum)zep1基因。
6.本发明的第二个目的是提供一种上述基因编码的蛋白。
7.本发明的最主要的一个目的在于提供该zep1基因或蛋白的应用。
8.为了实现上述目的,本发明采用的技术方案概述如下:
9.一种zep1基因,其核苷酸序列如seq id no.1所示,所述的核苷酸序列由1986个碱基组成,或在严格条件下与seq id no.1限定的dna序列杂交的dna分子。
10.上述基因编码的蛋白质(1),其氨基酸序列如seq id no.2所示。所述的序列由661个氨基酸残基组成。
11.上述zep1基因编码的蛋白还可以包括将seq id no.2氨基酸序列经过一个或多个((如1-30个;较佳地1-20个;更佳地1-10个;如5个,3个))氨基酸残基的取代、缺失或添加而形成的,且具有(1)蛋白功能的由(1)衍生的蛋白;或与(1)限定的蛋白序列有80%((较佳地90%以上,如95%,98%,99%或更高))以上同源性且具有(1)蛋白功能的由(1)衍生的蛋白。
12.含有zep1基因的重组微生物,基因编辑载体均属于本发明的保护范围,其中所述重组微生物包括藻类、真菌或细菌。
13.本发明最重要的一个发明点在于公开了zep1基因的一个新功能,本发明通过基因编辑获得zep1突变体,通过突变体和野生型的三角褐指藻相比,突变体的岩藻黄素含量比野生型低,但是积累了一种野生型中不存在的类胡萝卜素,根据其与已知化合物甲藻黄素的相似性,命名为7
′
,8
′‑
双脱氢去环氧甲藻黄素。可见,zep1基因或蛋白参与了岩藻黄素的合成,也就是说zep1基因具有提高三角褐指藻中岩藻黄素含量的功能,可以用于提高微生物(藻类、真菌、细菌)生产岩藻黄素的产量;并且,用于在光合生物(植物、藻类、光合细菌)
中能提高光合生物体内岩藻黄素含量,继而提高捕光效率,对光合作用效率和生物量积累有利,上述方式可以通过转基因的方式来实现。
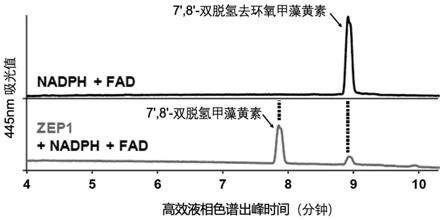
14.另外,通过对zep1蛋白的酶学功能的鉴定,结果发现,只有在zep1蛋白存在的前提下,7
′
,8
′‑
双脱氢去环氧甲藻黄素才会被转化为岩藻黄素的前体7
′
,8
′‑
双脱氢甲藻黄素,因此,zep1基因所编码蛋白还可用于岩藻黄素前体的体外合成。
15.这里需要说明的是,本发明所保护的基因的功能,不仅包括上述zep1基因,还包括与seq id no.1具有较高同源性(如同源性高于40%;较佳地高于50%;较佳地高于60%;更佳地高于70%;更佳地高于80%;更佳地高于90%;更佳地高于95%;更佳地高于98%)的同源基因。
16.本发明的优点:
17.在三角褐指藻中我们首次找到一个zep1基因,该基因所编码蛋白负责岩藻黄素合成的倒数第二步,也就是说本发明首次公开了zep1基因的一个新功能,其能够提高三角褐指藻中岩藻黄素含量。并且,用于在光合生物(植物、藻类、光合细菌)中能提高光合生物体内岩藻黄素含量,继而提高捕光效率,对光合作用效率和生物量积累有利,并且该基因所编码蛋白能够在生物体外用于岩藻黄素前体(7
′
,8
′‑
双脱氢甲藻黄素)的合成。
附图说明
18.图1是zep1突变体中zep1基因被编辑的证据;
19.图2是zep1突变体岩藻黄素含量降低的证据;
20.图中,右图为细胞外观拍照,左图为高效液相色谱(hplc)色素分析;
21.图3是zepl突变体中所积累色素,即zep1蛋白底物结构的核磁共振鉴定结果,也就是7
′
,8
′‑
双脱氢去环氧甲藻黄素分子结构;
22.图4是用于在大肠杆菌中生产zep1蛋白所用载体;
23.图5是实施例中采用zep1蛋白体外合成岩藻黄素前体的结果;
24.图6是7
′
,8
′‑
双脱氢甲藻黄素的分子结构;
25.图7是是用于在大肠杆菌中生产crtiso5蛋白所用载体;
26.图8是实施例中采用crtiso5蛋白体外合成岩藻黄素的结果。
具体实施方式
27.下面将通过具体实施例对本发明进行详细的描述。提供这些实施例是为了能够更透彻地理解本发明,并且能够将本发明的范围完整的传达给本领域的技术人员。
28.若未特别指明,实施例中所用技术手段为本领域技术人员所熟知的常规手段。下述实施例中的试验方法,如无特别说明,均为常规方法。如无特殊说明,所采用的试剂及材料,均可以通过商业途径获得。
29.除非另行定义,文中所使用的所有专业与科学用语与本领域熟练人员所熟悉的意义相同。此外,任何与所记载内容相似或均等的方法及材料皆可应用于本发明中。文中所述的较佳实施方法与材料仅作示范之用。
30.除非另有说明,本发明的实施将使用本领域技术人员显而易见的植物学常规技术、微生物、组织培养、分子生物学、化学、生物化学、dna重组及生物信息学技术。这些技术
均在已经公开的文献中进行了充分解释,另外,本发明所采用的dna提取、系统发育树的构建、基因编辑方法、基因编辑载体的构建、基因编辑植物获得等方法,除了下述实施例采用的方法外,采用现有文献中已经公开的方法均能实现。
31.此处使用的“核酸”、“核酸序列”、“核苷酸”、“核酸分子”或“多聚核苷酸”术语意思是指包括分离的dna分子(例如,cdna或者基因组dna),rna分子(例如,信使rna),自然类型,突变类型,合成的dna或rna分子,核苷酸类似物组成的dna或rna分子,单链或是双链结构。这些核酸或多聚核苷酸包括基因编码序列、反义序列及非编码区的调控序列,但不仅限于此。这些术语包括一个基因。“基因”或“基因序列”广泛用来指一有功能的dna核酸序列。因此,基因可能包括基因组序列中的内含子和外显子,和/或包括cdna中的编码序列,和/或包括cdna及其调控序列。在特殊实施方案中,例如有关分离的核酸序列,优先默认其为cdna。
32.实施例
33.一、zep1基因的获取
34.利用转录组和代谢组等多种技术手段筛选得到一个三角褐指藻zep1基因,该基因全长编码框核苷酸序列长度为1698bp,由565个氨基酸组成,其核苷酸序列如序列seq id no.1所示,其蛋白序列如seq id no.2所示。
35.二.crtiso5突变体的表型分析
36.此前,还没有关于三角褐指藻中zep1基因功能报道,申请人进行了如下研究,首次发现了zep1基因在三角褐指藻岩藻黄素合成中起着重要的作用。
37.1.zep1基因编辑载体的构建
38.使用crispor网站(http://crispor.tefor.net/)选取针对crtiso5基因编辑的导向序列(sgrna),根据所选sgrna设计两条引物(表1),在退火后获得含粘性末端的双链dna,克隆进含杀稻瘟素-s脱氨酶基因的目的载体,由三角褐指藻u6启动子负责sgrna的表达。cas9基因由γ微管蛋白启动子控制。
39.表1.crtiso5 sgrna引物
[0040][0041]
2.三角褐指藻接合转化与zep1突变体的获得
[0042]
将含有上述载体与接合转化所需pta-mob载体的大肠杆菌与野生型三角褐指藻以1000∶1的比例混合,浓缩后涂f/2培养基平板。两天后刮取细胞,涂于含5μg/ml杀稻瘟素的f/2培养基平板。
[0043]
两周后挑取克隆,用液体培养基重悬,重新涂板获得亚克隆。挑取多个亚克隆,进行菌落pcr与测序。所获得的zepl突变体中的crtiso5基因比野生型缺少15个碱基对(图1)。
[0044]
3.zep1突变体的岩藻黄素表型分析与积累的类胡萝卜素结构鉴定
[0045]
在恒定光(80μmol
·
m-2
·
s-1
)条件下培养野生型与突变体。离心收集107细胞,加入250μl 90%丙酮,在黑暗条件下超声混匀。离心后取上清,用高效液相色谱(hplc)进行色素分析。
[0046]
hplc参数如下:
[0047]
仪器:thermo ultimate3000uhplc;
[0048]
色谱柱:c18柱(5μm,250
×
4.6mm,waters,usa);
[0049]
温度:20℃;
[0050]
流速:每分钟1ml;
[0051]
进样体积:10μl;
[0052]
梯度流动相:由溶剂a(甲醇∶水=90∶10)与溶剂b(乙酸乙酯)组成。0时:100%a,0%b;20-22分钟:0%a,100%b;23-28分钟:100%a;0%b。
[0053]
hplc结果表明zep1突变体的岩藻黄素含量比野生型低,可以看出zep1基因或蛋白与岩藻黄素的合成有关,zep1突变体由于岩藻黄素含量低呈现绿色,但是积累了一种野生型中不存在的类胡萝卜素(图2)。
[0054]
在hplc过程中对所积累的类胡萝卜素进行收集,利用bruker avance neo 600mhz核磁共振仪进行结构分析,解析出的分子结构见图3,根据其与已知化合物甲藻黄素的相似性,命名为7
′
,8
′‑
双脱氢去环氧甲藻黄素,以下作为zep1蛋白的底物在体外体系中对zep1蛋白的酶学功能进行进一步的鉴定。
[0055]
三.表达载体的构建及蛋白的获得
[0056]
1.三角褐指藻zep1基因cdna(seq id no.1)的克隆
[0057]
采用rneasy plant minikit试剂盒提取野生型三角褐指藻rna。采用superscript
tm iii reverse transcriptase试剂盒进行反转录。以所获得总cdna为模板pcr克隆zep1基因cdna。
[0058]
pcr体系(表2):
[0059]
表2. 20μl扩增体系
[0060][0061]
pcr循环:
[0062]
1)94℃:5min;
[0063]
2)94℃:30s;
[0064]
3)55℃:30s;
[0065]
4)72℃:2min;
[0066]
步骤2)-4)循环35次;
[0067]
6)72℃:5min。
[0068]
pcr引物(表3)不仅包括与crtiso5 cdna序列重合序列(大写),也包括与目标载体同源序列(小写)。
[0069]
表3.zep1 cdna扩增引物
[0070][0071]
通过infusion同源重组法将pcr产物克隆进pmal-c5x载体。该载体中zep1基因在n端与mbp融合,c端具有多聚组氨酸标签(his-tag),外源基因表达受异丙基-β-d-硫代半乳糖苷(iptg)诱导。将构建完成质粒转化进表达bl21(de3)菌株,通过pcr筛选阳性克隆。所构建成功的载体见图4。
[0072]
2.三角褐指藻zepl基因在大肠杆菌中的表达
[0073]
采用含有100mg/l氨苄青霉素的lb培养基在37℃培养上述菌株,至od600为0.6-0.8。加入异丙基-β-d-硫代半乳糖苷(iptg)至终浓度为0.4mm,在16℃继续培养12小时。
[0074]
3.三角褐指藻zep1蛋白的纯化
[0075]
高压破碎后以13000g在4℃离心15分钟。所获得上清采用akta系统,采用source q离子交换柱进行蛋白纯化。采用30kdamwco超滤管进行蛋白浓缩。采用牛血清白蛋白(bsa)配置标准品,采用二辛可宁酸法测定蛋白浓度。分装至每管50μg后液氮速冻,保存于-80℃。
[0076]
四、crtiso5蛋白的酶学功能鉴定
[0077]
在体外体系中加入7
′
,8
′‑
双脱氢去环氧甲藻黄素与纯化zep1蛋白,验证岩藻黄素的产生。
[0078]
具体如下:
[0079]
1.酶学研究体系的建立
[0080]
将200μl缓冲液(0.1%tritonx-100,100mm tris,10mm mgcl2,1mm dtt,ph7.4)加入纯化干燥后的7
′
,8
′‑
双脱氢去环氧甲藻黄素,通过超声混匀来溶剂该前体。
[0081]
继而加入三种组分至括号内终浓度:
[0082]
黄素腺嘌呤二核苷酸(fad,100μm),氧化型;
[0083]
烟酰胺腺嘌呤二核苷酸磷酸(nadph;1mm);
[0084]
50μg纯化去环氧蛋白。
[0085]
在对照反应中省略zep1蛋白。两小时后,加入200μl丙酮与200μl乙酸乙酯,混匀以终止反应。
[0086]
2.zep1蛋白产物的分析
[0087]
在反应终止后通过离心对类胡萝卜素产物进行萃取:吸取上清有机层,氮气吹干,用50μl甲醇水溶液溶解。
[0088]
利用高效液相色谱(hplc)分析溶解物,以分析岩藻黄素前体7
′
,8
′‑
双脱氢甲藻黄素的产生与7
′
,8
′‑
双脱氢去环氧甲藻黄素的残留。hplc参数如下:
[0089]
仪器:waters acquity uplc;
[0090]
色谱柱:acquityuplc hss t31.8μm(3μm,2.1
×
150mm);
[0091]
温度:45℃;
[0092]
流速:每分钟0.3ml;
[0093]
进样体积:3μl;
[0094]
梯度流动相:由溶剂a(乙腈:甲醇:甲基叔丁基醚=70:20:10)与溶剂b(10mm醋酸铵)组成。0时:60%a,40%b;4分钟:75%a,25%b;12分钟:100%a。
[0095]
图5可以看出,通过和对照相比,加入zep1蛋白后,7
′
,8
′‑
双脱氢去环氧甲藻黄素被转化为岩藻黄素前体7
′
,8
′‑
双脱氢甲藻黄素(该物质是利用bruker avance neo 600mhz核磁共振仪进行结构分析,解析出的分子结构见图6,根据其与已知化合物甲藻黄素的相似性,命名为7
′
,8
′‑
双脱氢甲藻黄素),也就是说,zep1蛋白具有生物体外合成岩藻黄素前体的功能。
[0096]
五.7
′
,8
′‑
双脱氢甲藻黄素为合成岩藻黄素前体的证据
[0097]
1.三角褐指藻crtiso5基因edna(seq id no.3)的克隆
[0098]
采用rneasy plant minikit试剂盒提取野生型三角褐指藻rna。采用superscript
tm iii reverse transcriptase试剂盒进行反转录。以所获得总cdna为模板pcr克隆crtiso5基因cdna。
[0099]
pcr体系(表4):
[0100]
表4. 20μl扩增体系
[0101][0102]
pcr循环:
[0103]
1)94℃:5min;
[0104]
2)94℃:30s;
[0105]
3)55℃:30s;
[0106]
4)72℃:2min;
[0107]
步骤2)-4)循环35次;
[0108]
6)72℃:5min。
[0109]
pcr引物(表5)不仅包括与crtiso5 cdna序列重合序列(大写),也包括与目标载体同源序列(小写)。
[0110]
表5.crtiso5 cdna扩增引物
[0111][0112]
通过infusion同源重组法将pcr产物克隆进pmal-c5x载体。该载体中crtiso5基因在n端与mbp融合,c端具有多聚组氨酸标签(his-tag),外源基因表达受异丙基-β-d-硫代半乳糖苷(iptg)诱导。将构建完成质粒转化进表达bl21(de3)菌株,通过pcr筛选阳性克隆。所构建成功的载体见图7。
[0113]
2.三角褐指藻crtiso5基因在大肠杆菌中的表达
[0114]
采用含有100mg/l氨苄青霉素的lb培养基在37℃培养上述菌株,至od600为0.6-0.8。加入异丙基-β-d-硫代半乳糖苷(iptg)至终浓度为0.4mm,在16℃继续培养12小时。
[0115]
3.三角褐指藻crtiso5蛋白的纯化
[0116]
高压破碎后以13000g在4℃离心15分钟。所获得上清采用akta系统,采用source q离子交换柱进行蛋白纯化。采用30kdamwco超滤管进行蛋白浓缩。采用牛血清白蛋白(bsa)配置标准品,采用二辛可宁酸法测定蛋白浓度。分装至每管50μg后液氮速冻,保存于-80℃。
[0117]
4.crtiso5蛋白的酶学功能鉴定
[0118]
在体外体系中加入7
′
,8
′‑
双脱氢甲藻黄素与纯化crtiso5蛋白,验证岩藻黄素的产生。具体如下:
[0119]
4.1酶学研究体系的建立
[0120]
将200μl缓冲液(0.1%tritonx-100,100mm tris,10mm mgcl2,1mm dtt,ph 7.4)加入纯化干燥后的7
′
,8
′‑
双脱氢甲藻黄素,通过超声混匀来溶剂该前体。
[0121]
继而加入三种组分至括号内终浓度:
[0122]
黄素腺嘌呤二核苷酸(fad,100μm),氧化型;
[0123]
na2s2o4(1mm),以使fad转化为还原型;
[0124]
50μg纯化crtiso5蛋白。
[0125]
在对照反应中省略crtiso5蛋白。两小时后,加入200μl丙酮与200μl乙酸乙酯,混匀以终止反应。
[0126]
4.2 crtiso5蛋白产物的分析
[0127]
在反应终止后通过离心对类胡萝卜素产物进行萃取:吸取上清有机层,氮气吹干,用50μl甲醇水溶液溶解。
[0128]
利用高效液相色谱(hplc)分析溶解物,以分析岩藻黄素的产生与7
′
,8
′‑
双脱氢甲藻黄素的残留。hplc参数如下:
[0129]
仪器:waters acquity uplc;
[0130]
色谱柱:acquity uplc hss t3 1.8μm(3μm,2.1
×
150mm);
[0131]
温度:45℃;
[0132]
流速:每分钟0.3ml;
[0133]
进样体积:3μl;
[0134]
梯度流动相:由溶剂a(乙腈∶甲醇∶甲基叔丁基醚=70∶20∶10)与溶剂b(10mm醋酸铵)组成。0时:60%a,40%b;4分钟:75%a,25%b;12分钟:100%a。
[0135]
图8可以看出,通过和对照相比,加入crtiso5蛋白后,7
′
,8
′‑
双脱氢甲藻黄素被转化为岩藻黄素,也就是说,7
′
,8
′‑
双脱氢甲藻黄素是合成岩藻黄素的前体。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1