一种ADAR蛋白细胞内高效结合底物及应用
一种adar蛋白细胞内高效结合底物及应用
1.本技术为申请号202010158889.1、申请日2020-03-09、发明名称“一种获取adar蛋白细胞内高效结合的底物序列的方法及底物序列和应用”的中国发明专利申请的分案申请。
技术领域
2.本发明涉及分子生物学技术领域,具体地,涉及蛋白质与rna互作技术领域,更具体地,涉及一种adar蛋白细胞内高效结合的底物及应用。
背景技术:
3.基因突变是许多疾病及癌症发生的关键诱因,目前已经有3000多个基因特定位点的突变与疾病的发生有关联,随着二代测序技术发展,不论是遗传获得性的基因突变还是后天生长发育过程中某些体细胞的基因突变都可以通过二代测序技术获得突变位点信息,并且通过关联分析找到与疾病发生直接相关的突变位点。但目前各种肿瘤在不同的人群中会有不同的突变位点及突变类型,这样开发不同的靶向药物具有一定的难度,而基因疗法可直接对基因的突变位点进行修复。crispr(clustered regularly interspaced short palindromic repeats)最早发现于细菌免疫系统中,结合cas蛋白抵抗外源病毒的入侵。目前crispr/cas技术已经广泛应用到生物医学及农业学研究中。crispr/cas技术可以在真核细胞基因组水平引入正确编码的全长基因拷贝,也可以沉默产生突变的基因的表达、对突变位点进行定点修复。在2014年已经有第一例报道成功利用crispr技术对地中海贫血症进行临床治疗。地中海贫血症由hbb基因突变引起的血红蛋白水平降低造成。该研究团队将患者皮肤成纤维细胞诱导分化成多能干细胞并用crispr/cas9技术修复突变位点,并将修复后的干细胞诱导成红细胞前体细胞并重新移植到患者体内达到治疗目的。但crispr/cas系统也有其不可避免的缺点,该系统需要同时表达cas蛋白及导向rna(grna)及提供同源修复dna序列,且在多篇研究报道中都检测出其在dna水平或rna水平有明显的脱靶效应,crispr/cas蛋白切割基因组dna造成dsb(double-strand break),脱靶会造成基因组的不稳定性以及不可预测的细胞命运。此外,crispr/cas系统还可能会引发细胞固有免疫反应,例如,金黄色葡萄球菌来源的cas9蛋白会诱导血液中的t细胞产生炎症反应进而引起组织坏死。crispr/cas系统的严重缺陷造成其临床应用的局限性,故开发更加安全有效的基因疗法是目前具有巨大临床价值的研究。
4.a-to-i(腺苷到肌苷)rna编辑是adar蛋白家族介导的且在后生动物中广泛存在的一种rna修饰方式。a碱基发生脱氨基反应转变成i碱基,继而在细胞内被识别成g碱基,故adar蛋白可以在rna水平逆转基因组上的g到a的突变。目前已知的报道中约20,000种g-to-a的突变都与疾病的发生发展有关联。相比crispr/cas系统的基因组编辑方法,adar介导的rna水平的可逆的编辑不会影响基因组序列,且可通过改变rna序列使得突变的蛋白序列得到修复。最新的研究表明利用adar2的一段双链rna底物序列作为adar1蛋白招募序列,结合一段特定位点的互补靶向序列构建出adrna(adar guide rna)可对內源基因特定位点的a
进行编辑。该方法的优势在于其不需表达其他外源蛋白,仅需要将adrna片段导入到细胞内便可招募细胞內源的adar1蛋白发挥编辑功能,该方法不会引起脱靶效应也不会引起细胞固有免疫反应。但该方法目前的局限性编辑效率不够高。为了提高adrna介导的a-to-i编辑效率,最终达到临床治疗的目的,则需要在全基因组水平找出adar的高亲和力底物,从而合成更高效的adrna。但由于adar作用的双链rna底物研究的空缺,使得利用adar蛋白结合adrna修复与疾病相关的g-to-a突变方法难以得到广泛的临床应用。同时,目前用于研究蛋白与rna相互作用的方法,例如hiclip和clash等,获取的嵌合体比例少且实验耗时长,均无法有效用于adar蛋白作用底物信息的获取。
技术实现要素:
5.本发明的目的在于克服现有技术中存在的上述缺陷和不足,提供一种获取adar蛋白细胞内高效结合的底物序列的方法。
6.本发明的另一目的在于提供上述方法获得的底物序列。
7.本发明的再一目的在于提供上述方法和底物序列的应用。
8.本发明的上述目的是通过以下技术方案给予实现的:
9.一种获取adar蛋白细胞内高效结合的底物序列的方法,包括如下步骤:
10.s1.在细胞中过表达adar蛋白,再交联蛋白质和rna;
11.s2.裂解细胞,免疫共沉淀蛋白与rna交联复合体,消化未被蛋白保护的rna,用tsap酶对消化后的rna进行去磷酸化处理,接着用t4 pnk对rna的5’末端做磷酸化修饰,进行rna分子间连接,然后对连接后的rna末端加接头,纯化120kb~200kb大小范围的蛋白及rna复合体并提取rna后进行逆转录反应得cdna;
12.s3.以步骤s2获得的cdna为模板做环化反应,纯化后再线性化并进行两轮pcr扩增,第二轮pcr时在扩增产物两端分别接上测序文库3'接头和5'接头,对构建好的文库进行高通量测序;
13.s4.将高通量测序获取的序列信息去接头、质控、基因组对比,将读长比对到转录组,丢弃比对上的读长,保留未比对上的读长并记录为u1;把u1读长比对到基因组并丢弃连续比对上的读长,保留未比对上的读长并记录为u2;把u2读长比对到基因组,把唯一且不连续的比对上的读长记录为嵌合体读长并记录为u3;把u3读长比对到基因组上,并且根据比对结果对u3进行重新组合;把重组之后的读长比对到基因组上,并提取嵌合体读长;再对上述结果进行整合,获得完整的嵌合体读长集;再以嵌合体两条臂读长为中心取交集后再分别与非嵌合体读长取交集,重组后的双臂读长再进行结构预测截取出双链rna底物序列,分别以左臂l和右臂r为标记注释其序列信息组装出adar蛋白的作用底物序列。
14.本发明开发了一种获取adar蛋白细胞内高效结合的底物序列的方法,又称为irclash(infrared cross-linking,ligation,and sequencing of hybrids),本发明从实验及生物信息学的角度填补现有adar蛋白结合底物研究存在的空缺;通过实验技术及后续的双链rna底物的生物信息学组装方法,获取高亲和力的adar双链rna底物。主要为在步骤s2实验上,通过tsap酶和t4pnk的组合使用,提高rna分子间的连接效率,进而提高后续嵌合体读长的得率;同时在步骤s4数据处理时考虑双链rna在连接的过程中会发生两种连接情况:即第一条链的3’端连接到第二条链的5’端和第一条链的5’端连接到第二条链的3’端,
在比对到基因组步骤中增加了一轮数据处理,从而可以从未比对到基因组的序列中回收一定量的嵌合体读长,显著提高嵌合体读长的得率;再通过自行开发的底物拼接方法,最终获得了adar蛋白在细胞内高效结合的底物序列。同时步骤s3选择对cdna进行环化代替直接在cdna5’末端加接头,此方法显著降低了实验操作的复杂度及实验耗时。
15.优选地,本发明上述方法还包括后续的高效结合底物的评估,为利用生物信息学软件bprna结合转录组的rna编辑位点编辑水平高低解析利用上述方法获取的adar对底物结合及编辑的偏好性,将有助于后续对靶向位点编辑的序列设计。
16.优选地,步骤s1为在细胞中过表达携带外源蛋白标签的adar蛋白;从而更易被抗体识别,提高抗体在免疫共沉淀rna时的特异性。
17.更优选地,所述外源蛋白标签为3
×
flag标签。以提高抗体在免疫共沉淀rna时的特异性,很大程度上也提高了后续组装的adar底物的特异性及准确性。
18.具体地,所述adar蛋白为adar1、adar2或adar3蛋白;
19.优选地,所述adar蛋白为adar1。
20.优选地,步骤s1所述交联为紫外交联。
21.优选地,步骤s2所述消化为利用rna酶进行消化。
22.优选地,步骤s2所述rna分子间连接为采用t4rnaligasei连接。
23.优选地,步骤s2所述接头为ir800染料标记的接头,免除了放射性标记接头可能引起的信号衰减问题及降低对实验环境的要求。
24.本发明还提供由上述任一所述方法获得的adar蛋白高效结合的底物序列,每段底物序列包括左臂(l)和右臂(r),其核苷酸序列依次如seq id no:1~seq id no:100所示;所述底物核苷酸序列在细胞内可形成能够招募adar蛋白的双链rna。
25.本发明上述方法可获得adar蛋白在细胞内高效结合的底物序列,而所述底物核苷酸序列能够在细胞内可形成能够招募adar蛋白的双链rna,同时再结合一段特定位点的互补靶向序列便可构建出adrna,可对特定位点的a进行编辑,实现定点靶向rna编辑。
26.因此,本发明请求保护上述任一所述方法或上述任一所述adar蛋白高效结合的底物序列在定点靶向rna编辑中的应用,具体是在adar介导的定点靶向rna编辑中的应用。
27.以及上述任一所述方法或上述任一所述adar蛋白高效结合的底物序列在制备rna基因治疗药物中的应用,具体是在adar介导的rna基因治疗药物中的应用。
28.与现有技术相比,本发明具有以下有益效果:
29.(1)本发明首次开发了一种获取adar蛋白细胞内高效结合的底物序列的方法irclash,本发明从实验及生物信息学的角度填补现有adar结合底物研究存在的空缺,可获得大量的adar蛋白高效结合底物序列。
30.(2)本发明方法与现有其他研究蛋白与rna相互作用的方法相比,可显著提高嵌合体读长的得率,可最大限度提高了adar蛋白作用底物的构建数量,同时缩短实验耗时、简化了实验操作。
31.(3)本发明获得了大量的高亲和力的adar蛋白双链rna底物,且具有与目前已报道的其他底物相当的定点编辑能力,为adar介导的靶向rna编辑提供更多可供选择的adar蛋白招募序列。
附图说明
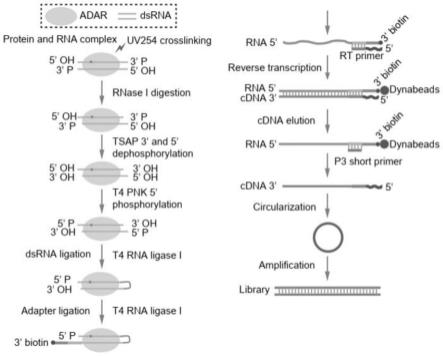
32.图1为本发明开发的irclash实验方法整体流程。
33.图2为本发明开发的irclash实验测序文库数据分析流程。
34.图3为本发明开发的irclash实验耗时与其他发表的方法对比,结果显示本发明的方法大大的缩短了实验耗时。
35.图4为本发明开发的irclash实验及数据分析方法得到的嵌合体读长比例与目前发表的其他蛋白的同类方法的比较,结果表明本发明的方法可显著提高嵌合体读长的得率,这有助于充分解析dsrbp底物的特性。
36.图5为发明在数据处理上提高在嵌合体读长比例的方法,即通过此步的数据处理可回收一定比例的嵌合体读长。目前已发表的同类方法都忽略了此部分数据。
37.图6为本发明开发的adar1双链rna底物构建方法。该方法的核心是以嵌合体两条臂读长为中心取交集后再分别与非嵌合体读长取交集,重组后的双臂读长再进行结构预测截取出双链rna底物序列,分别以左臂(l)和右臂(r)为标记注释其序列信息。
38.图7为从本发明构建的adar1底物图谱中抽取的随机高亲和力的底物,截取其中的部分序列构建adar1介导的靶向定点rna编辑。
39.图8为本发明开发的底物应用到adar1介导的靶向定点rna编辑,结果表明,本发明构建的底物能够达到与目前已报道的其他底物相当的定点编辑能力。
具体实施方式
40.以下结合说明书附图和具体实施例来进一步说明本发明,但实施例并不对本发明做任何形式的限定。除非特别说明,本发明采用的试剂、方法和设备为本技术领域常规试剂、方法和设备。
41.除非特别说明,以下实施例所用试剂和材料均为市购。
42.实施例1在hek293细胞系中构建adar1蛋白高亲和力底物序列及rna水平定点靶向编辑
43.本发明实验方法整体流程如图1所示,实验测序文库数据分析流程如图2所示,与其他发表的方法对比,结果显示本发明的方法大大的缩短了实验耗时(如图3);本实施例以adar蛋白家族adar1蛋白为例,进一步说明本发明;具体的,所述方法包括如下步骤:
44.1、细胞准备及蛋白与rna交联反应
45.在一个15cm的细胞培养皿中培养hek293细胞并过表达flag标签的adar1,48小时后用pbs清洗细胞并在254nm的紫外交联仪上进行蛋白与rna的交联反应。
46.2、免疫共沉淀及双链rna消化,连接和反转
47.裂解细胞后用flag抗体免疫共沉淀adar1及其交联上的rna,并用4u/ml浓度rnasei消化rna,用tsap酶对消化后的rna在37℃下孵育1小时进行去磷酸化处理,反应组分如下表1所示:
48.表1
[0049][0050]
在做分子间连接之前需要对rna进行5’端磷酸化处理,用t4pnk在37℃条件处理1小时,反应组分如下表2所示;本发明通过先用tsap酶去除rna的磷酸化末端,接着用t4 pnk对rna的5’末端做磷酸化修饰,通过这两步提高分子间连接的效率,再经过后续数据处理,得到的嵌合体读长比例与目前发表的其他蛋白的同类方法的比较,可显著提高嵌合体读长的得率(如图4所示);
[0051]
表2
[0052][0053][0054]
经过此两步处理后的rna便可进一步做分子间连接,用t4rnaligasei在16℃条件下孵育12小时,反应组分如下表3所示:
[0055]
表3
[0056][0057]
经过第一轮rna分子间连接后加入2μl10um的接头继续孵育12小时。加入接头后需对adar1与底物rna复合体进行聚丙烯酰胺凝胶电泳并转膜切取120kb~200kb的复合体并消化蛋白,纯化结合的rna片段。因为是双链rna底物,其结构及序列上不同于普通的细胞总rna,故本发明中用superscript iv反转录酶进行逆转录反应,反应组分如下表4所示:
[0058]
表4
[0059][0060]
混合均匀后65℃条件孵育5分钟,立即置于冰上5分钟再加入如下表5所示组分:
[0061]
表5
[0062][0063]
混合均匀后55℃孵育10分钟。
[0064]
3、cdna环化、建库和高通量测序
[0065]
对cdna进行环化处理可以简化实验操作与耗时,本发明中用lucigen公司的circligase ii ssdna ligase试剂盒环化cdna,纯化后进行常规的pcr建库并在illumina测序平台进行测序。
[0066]
4、adar底物拼装
[0067]
得到irclash文库测序结果后通过生物信息学的方法对测序读长进行去接头、质控、基因组对比等一系列处理,与目前发表的研究不同之处在于本发明在比对到基因组步骤中增加了一轮数据处理,由于双链rna在连接的过程中会发生两种连接情况,即第一条链的3’端连接到第二条链的5’端或第一条链的5’端连接到第二条链的3’端,在之前的研究中都只考虑了第一种情况,本发明在开发数据处理方法时考虑了两种情况,通过此步骤处理可增加一定数量的嵌合体读长(如图5所示),具体地,是在对测序读长去接头、质控后,在进行基因组对比时,通过用bowtie软件将读长比对到转录组,丢弃比对上的读长,保留未比对上的读长并记录为u1;用bowtie软件把u1读长比对到基因组并丢弃连续比对上的读长,保留未比对上的读长并记录为u2;用star软件把u2读长比对到基因组,把唯一且不连续的比对上的读长记录为嵌合体读长并记录为u3;把u3读长比对到基因组上,并且根据比对结果对u3进行重新组合;利用star把重组之后的读长比对到基因组上,并提取嵌合体读长;对上述结果进行整合,获得完整的嵌合体读长集。基于此,我们又开发了一种双链rna结合蛋白的底物拼接方法(如图6所示),该方法的核心是以嵌合体两条臂读长为中心取交集后再分别与非嵌合体读长取交集,重组后的双臂读长再进行结构预测截取出双链rna底物序列,分别以左臂(l)和右臂(r)为标记注释其序列信息,获得的adar1蛋白高亲和力底物序列如表6所示:
[0068]
表6
[0069]
[0070]
[0071]
[0072]
[0073]
[0074]
[0075]
[0076]
[0077]
[0078]
[0079]
[0080]
[0081]
[0082]
[0083][0084]
5、高效结合底物的评估
[0085]
为了更加具体的解析adar1对底物结合及编辑的特性,我们把带有a-to-i编辑位点的底物进行分析,通过比较底物上的编辑位点及编辑水平分布发现被编辑的底物5’端相比于3’端有更高的编辑活性,该结果将有助于对设计adar1介导的rna靶向编辑招募序列提供设计基础。此外,本发明还进一步利用bprna软件解析编辑位点处的结构特性,通过这两方面的分析与评估将为设计更加高效的adrna提供帮助。
[0086]
6、基于高效结合底物的靶向定点rna编辑
[0087]
为了评估本发明开发的irclash实验及分析方法的可靠性,本发明随机从底物中截取部分序列设计出几条adrna序列(如图7所示),并构建到psilencer 2.1-u6 hygro载体中,并在hek293细胞中与adar1质粒同时过表达,通过内源基因靶向位点的编辑水平判断其有效性,结果如图8所示,结果表明本发明开发出的adrna同样可以达到细胞内靶向定点编辑rna的能力,能够达到与目前已报道的其他底物相当的定点编辑能力,表明本发明开发的irclash实验及分析方法可获得adar1蛋白细胞内高效结合的底物序列,进行后续的靶向定点编辑rna。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1