用于检测甲状腺结节良恶性的甲基化标志物和用途的制作方法
1.本发明属于分子生物医学技术领域,特别涉及用于检测甲状腺结节良恶性的甲基化标志物和用途。
背景技术:
2.甲状腺癌是最常见的内分泌系统恶性肿瘤,近年来甲状腺癌的发病率呈现快速增长趋势,已位列恶性肿瘤前10位。甲状腺癌主要包括乳头状癌(ptc)、滤泡状癌(ftc)、未分化癌(atc)和髓样癌(mtc)四种病理类型,其中ptc是最常见的病理亚型,占全部甲状腺癌的75%~80%。目前甲状腺癌的临床标准诊疗流程是通过超声影像学检测和细针穿刺细胞学检测对甲状腺结节进行术前良恶性诊断,其中约30%是无法确定良恶性的。这类患者由于缺少明确的治疗方案,易导致治疗延误或过度诊疗。为了解决甲状腺癌临床诊断上的难题,近年来,以基因变异检测为代表的辅助分子诊断方法开始投入临床应用。借助基因变异检测,临床诊断的准确性虽然有所提高,但由于部分肿瘤缺少明确的驱动基因变异,使得仍有很多患者无法确诊。因此需要寻找能够进一步提高甲状腺癌诊断准确性的方法。
3.越来越多的研究表明,异常的dna甲基化是甲状腺癌的一个重要特征。特定基因的异常甲基化在甲状腺癌中已被发现。有研究报道,抑癌基因(tshr、pten、rassf1a、cdkn2a、dapk1、timp3、ecad和rap1gap)在甲状腺癌发生过程中发生高甲基化,而map17、cxcl12、hormad2和pfkfb2的低甲基化与侵袭性甲状腺癌有关。另有研究发现,rassf1a的高甲基化可作为侵袭性肿瘤的生物标志物,rarb2的异常甲基化可作为鉴别未分化癌的标志物。这些研究揭示了基因的异常甲基化在甲状腺癌发生发展中起到的重要作用,也提示基因甲基化具有作为甲状腺癌诊断标志物的潜力。
4.北美市场的主流产品thyroseq和afirma都是基于基因组变异和rna表达差异的产品,其在灵敏度和阴性预测值方面表现较好,然而其特异性和阳性判断值性能还有较大提升空间。美国希望城的一个实验室报道了,一组可以用于区分甲状腺结节良恶性的甲基化分子标记物,但其构建的预测模型对于某些样本仍然会无法判断,仍具有优化提升空间。
5.虽然国外有研究和专利报道了,基因甲基化作为甲状腺癌的诊断标记物,但由于表观遗传容易受到环境影响,在不同人群间可能存在一定差异性,到目前为止,基于中国人群甲基化标记物作为甲状腺癌诊断的研究较少,更没有相应的产品在临床中应用。
6.因此,开发针对中国人群的、基于甲基化标志物的甲状腺结节良恶性判别,有助于提高中国人群甲状腺癌临床诊断的准确性,在临床实践中具有重要的应用价值。
技术实现要素:
7.本发明提供了用于检测甲状腺结节良恶性的甲基化标志物和用途,用以解决现有技术中存在的问题。
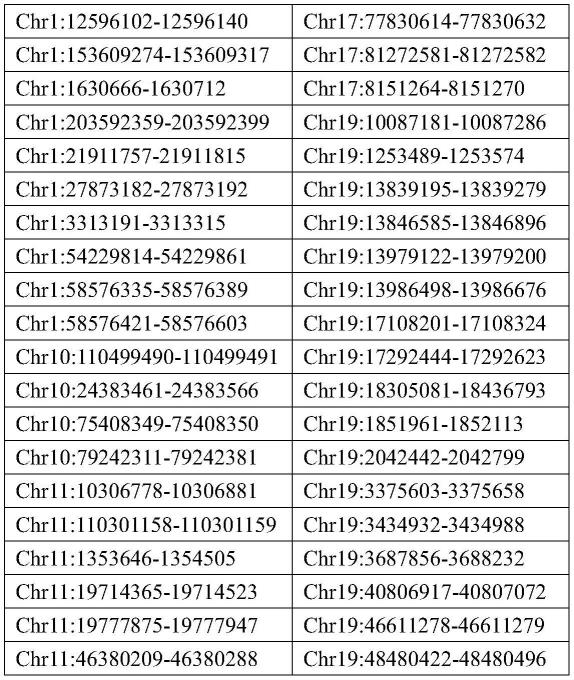
8.本发明的目的之一在于提供,一种用于检测甲状腺结节良恶性的甲基化标志物,所述甲基化标志物包括以下甲基化区域中的一种或多种:chr1:12596102-12596140区域、
chr17:77830614-77830632区域、chr1:153609274-153609317区域、chr17:81272581-81272582区域、chr1:1630666-1630712区域、chr17:8151264-8151270区域、chr1:203592359-203592399区域、chr19:10087181-10087286区域、chr1:21911757-21911815区域、chr19:1253489-1253574区域、chr1:27873182-27873192区域、chr19:13839195-13839279区域、chr1:3313191-3313315区域、chr19:13846585-13846896区域、chr1:54229814-54229861区域、chr19:13979122-13979200区域、chr1:58576335-58576389区域、chr19:13986498-13986676区域、chr1:58576421-58576603区域、chr19:17108201-17108324区域、chr10:110499490-110499491区域、chr19:17292444-17292623区域、chr10:24383461-24383566区域、chr19:18305081-18436793区域、chr10:75408349-75408350区域、chr19:1851961-1852113区域、chr10:79242311-79242381区域、chr19:2042442-2042799区域、chr11:10306778-10306881、chr19:3375603-3375658区域、chr11:110301158-110301159区域、chr19:3434932-3434988区域、chr11:1353646-1354505区域、chr19:3687856-3688232区域、chr11:19714365-19714523区域、chr19:40806917-40807072区域、chr11:19777875-19777947区域、chr19:46611278-46611279区域、chr11:46380209-46380288区域、chr19:48480422-48480496区域、chr11:57426966-57427173区域、chr19:51098902-51099067区域、chr11:64851310-64851314区域、chr19:51601422-51601624、chr11:66336997-66337092区域、chr19:52536666-52536686区域、chr11:70073960-70074053区域、chr19:55087135-55087394区域、chr11:70742594-70742814区域、chr2:101152526-101152688区域、chr12:119804311-119804352区域、chr2:237202186-237202187区域、chr12:130980102-130980198区域、chr2:240598627-240598628区域、chr12:47881830-47881926区域、chr2:25277096-25277319区域、chr12:94644417-94644501区域、chr2:80302861-85584386区域、chr13:98476650-98476740区域、chr20:22582417-22582620区域、chr14:102928211-102928212区域、chr20:2752143-2752688区域、chr14:105087674-105088536区域、chr20:57212578-57212700区域、chr15:100115182-100115183区域、chr22:50577975-50578304区域、chr15:40769136-40769213区域、chr3:65597378-65597606区域、chr16:15143862-15144467区域、chr4:112131117-112131180区域、chr16:19114827-19115044区域、chr4:153486942-153486953区域、chr16:2124308-2176703区域、chr4:1752763-1752832区域、chr16:27769896-27769905区域、chr5:139482856-139482885区域、chr16:4943479-4943543区域、chr5:139697245-139697591区域、chr16:88634971-88634991区域、chr5:141514157-141514273区域、chr15:99123755-99123765区域、chr6:152302126-152302178区域、chr16:56635666-56635695、chr6:165854274-165854683区域、chr16:56648042-56648061区域、chr6:41470838-41470918区域、chr17:17700407-17700520区域、chr7:1405246-1405342区域、chr17:21464252-21464279区域、chr7:1940287-1940414区域、chr17:28227417-28227453区域、chr7:256963-256964区域、chr17:38868437-38868530区域、chr7:74093943-74094754区域、chr17:3963840-3963844区域、chr8:22554216-22554275区域、chr17:42673716-42673823区域、chr8:27667002-27667143区域、chr17:45143922-45144232区域、chr9:127121856-127122337区域、chr17:47695358-47695495区域、chr9:131733731-131733759区域、chr17:57875502-57875627、chr9:136050771-136050820区域、chr17:58487622-58487664区域、
chr9:136299368-136299372区域、chr17:58487747-58488275区域、chr9:136688112-136688211区域、chr17:63438487-63446755区域、chr9:137461863-137461864区域、chr17:7174295-7174472区域、chr9:37954187-37954325区域。
9.根据本发明的技术方案,所述甲状腺为甲状腺乳头状癌(ptc)。
10.根据本发明的技术方案,所述甲基化区域包括如下甲基化位点中的一种或多种:chr1:1041251、chr5:141514157、chr11:1353646、chr16:15144467、chr19:3688090、chr1:1630666、chr5:141514263、chr11:1354505、chr16:19114827、chr19:3688100、chr1:1630712、chr5:141514264、chr11:8699310、chr16:19115044、chr19:3688141、chr1:2230562、chr5:141514273、chr11:10306778、chr16:25368575、chr19:3688179、chr1:3290597、chr5:150297782、chr11:10306781、chr16:27769896、chr19:3688232、chr1:3313191、chr5:150640873、chr11:10306784、chr16:27769904、chr19:3695513、chr1:3313201、chr6:30076148、chr11:10306824、chr16:27769905、chr19:4474315、chr1:3313216、chr6:30682948、chr11:10306864、chr16:28322373、chr19:5048327、chr1:3313227、chr6:41470838、chr11:10306865、chr16:29821869、chr19:5111340、chr1:3313250、chr6:41470852、chr11:10306881、chr16:30359428、chr19:5325651、chr1:3313252、chr6:41470918、chr11:19714365、chr16:55331495、chr19:5686513、chr1:3313269、chr6:152302126、chr11:19714401、chr16:56635666、chr19:7483269、chr1:3313271、chr6:152302127、chr11:19714402、chr16:56635667、chr19:10087181、chr1:3313289、chr6:152302145、chr11:19714445、chr16:56635695、chr19:10087286、chr1:3313292、chr6:152302146、chr11:19714446、chr16:56648042、chr19:13839195、chr1:3313297、chr6:152302157、chr11:19714514、chr16:56648043、chr19:13839223、chr1:3313308、chr6:152302158、chr11:19714523、chr16:56648061、chr19:13839279、chr1:3313311、chr6:152302159、chr11:19723747、chr16:56662918、chr19:13846585、chr1:3313315、chr6:152302160、chr11:19777875、chr16:66845951、chr19:13846896、chr1:3505198、chr6:152302167、chr11:19777883、chr16:66936068、chr19:13881524、chr1:3543255、chr6:152302168、chr11:19777947、chr16:67150229、chr19:13979122、chr1:3594910、chr6:152302169、chr11:46380209、chr16:68371421、chr19:13979200、chr1:9717812、chr6:152302170、chr11:46380288、chr16:72013968、chr19:13986498、chr1:10635787、chr6:152302171、chr11:57426966、chr16:81568574、chr19:13986533、chr1:12596102、chr6:152302172、chr11:57427099、chr16:81752390、chr19:13986590、chr1:12596119、chr6:152302178、chr11:57427153、chr16:85436695、chr19:13986591、chr1:12596120、chr6:165854274、chr11:57427172、chr16:88634971、chr19:13986594、chr1:12596140、chr6:165854364、chr11:57427173、chr16:88634991、chr19:13986595、chr1:15786745、chr6:165854456、chr11:64851310、chr16:88637438、chr19:13986598、chr1:15944130、chr6:165854655、chr11:64851314、chr17:1937395、chr19:13986599、chr1:16181978、chr6:165854683、chr11:64858680、chr17:3963840、chr19:13986676、chr1:17540993、chr7:256963、chr11:65233327、chr17:3963844、chr19:14480374、chr1:17688909、chr7:256964、chr11:66012259、chr17:6566846、chr19:17108201、chr1:18927289、chr7:1081092、chr11:66336997、chr17:7174295、chr19:17108324、chr1:
21911757、chr7:1323349、chr11:66337010、chr17:7174309、chr19:17292444、chr1:21911758、chr7:1405246、chr11:66337014、chr17:7174322、chr19:17292460、chr1:21911815、chr7:1405342、chr11:66337015、chr17:7174352、chr19:17292467、chr1:22318703、chr7:1474325、chr11:66337092、chr17:7174391、chr19:17292510、chr1:27873182、chr7:1940287、chr11:68455664、chr17:7174426、chr19:17292547、chr1:27873192、chr7:1940414、chr11:68750637、chr17:7174472、chr19:17292548、chr1:41358067、chr7:2075109、chr11:68840785、chr17:7384019、chr19:17292564、chr1:54229814、chr7:2418150、chr11:70073960、chr17:8151264、chr19:17292569、chr1:54229815、chr7:5931371、chr11:70074002、chr17:8151270、chr19:17292605、chr1:54229860、chr7:73904895、chr11:70074053、chr17:17700407、chr19:17292623、chr1:54229861、chr7:74093943、chr11:70486699、chr17:17700408、chr19:18305081、chr1:58576335、chr7:74093947、chr11:70663065、chr17:17700410、chr19:18436793、chr1:58576389、chr7:74093981、chr11:70742594、chr17:17700415、chr19:30594807、chr1:58576421、chr7:74094104、chr11:70742652、chr17:17700418、chr19:31351829、chr1:58576422、chr7:74094105、chr11:70742653、chr17:17700425、chr19:34577534、chr1:58576423、chr7:74094163、chr11:70742654、chr17:17700432、chr19:35806723、chr1:58576603、chr7:74094245、chr11:70742697、chr17:17700436、chr19:40806917、chr1:112722970、chr7:74094640、chr11:70742698、chr17:17700457、chr19:40807049、chr1:151838359、chr7:74094664、chr11:70742738、chr17:17700468、chr19:40807072、chr1:151992616、chr7:74094665、chr11:70742739、chr17:17700476、chr19:46611278、chr1:153609274、chr7:74094687、chr11:70742755、chr17:17700516、chr19:46611279、chr1:153609317、chr7:74094688、chr11:70742756、chr17:17700520、chr19:48480422、chr1:156066660、chr7:74094725、chr11:70742776、chr17:21464252、chr19:48480496、chr1:203592359、chr7:74094726、chr11:70742786、chr17:21464269、chr19:48630748、chr1:203592399、chr7:74094732、chr11:70742814、chr17:21464279、chr19:49519137、chr1:228990544、chr7:74094733、chr11:74683980、chr17:28227417、chr19:51098902、chr1:234963449、chr7:74094754、chr11:110301158、chr17:28227423、chr19:51098906、chr2:3613345、chr7:155392408、chr11:110301159、chr17:28227453、chr19:51098914、chr2:9235477、chr8:21790103、chr11:117444044、chr17:31596788、chr19:51098924、chr2:9801337、chr8:21811216、chr11:119709773、chr17:38868437、chr19:51098928、chr2:10029534、chr8:22554216、chr11:125953540、chr17:38868530、chr19:51098930、chr2:10498150、chr8:22554275、chr11:130156804、chr17:42673716、chr19:51098945、chr2:10947098、chr8:23164199、chr11:134536759、chr17:42673728、chr19:51098973、chr2:25251595、chr8:27667002、chr12:1661591、chr17:42673757、chr19:51098997、chr2:25277096、chr8:27667143、chr12:7108181、chr17:42673759、chr19:51099012、chr2:25277109、chr8:33565861、chr12:47881830、chr17:42673823、chr19:51099022、chr2:25277110、chr8:38651209、chr12:47881926、chr17:44279942、chr19:51099032、chr2:25277116、chr8:38788172、chr12:58027065、chr17:44558988、chr19:51099041、chr2:25277124、chr8:38936737、chr12:64222777、chr17:45143922、chr19:51099067、chr2:
25277130、chr8:140310313、chr12:94150630、chr17:45143923、chr19:51601422、chr2:25277131、chr8:141308720、chr12:94644417、chr17:45144227、chr19:51601423、chr2:25277135、chr9:35117297、chr12:94644488、chr17:45144232、chr19:51601435、chr2:25277136、chr9:37954187、chr12:94644501、chr17:47695358、chr19:51601436、chr2:25277145、chr9:37954267、chr12:111443740、chr17:47695395、chr19:51601468、chr2:25277319、chr9:37954268、chr12:117155549、chr17:47695495、chr19:51601469、chr2:30230790、chr9:37954291、chr12:119804311、chr17:50469031、chr19:51601476、chr2:33903795、chr9:37954325、chr12:119804352、chr17:57868238、chr19:51601477、chr2:46983376、chr9:90729572、chr12:122959902、chr17:57875456、chr19:51601483、chr2:64831509、chr9:92189571、chr12:124326125、chr17:57875502、chr19:51601484、chr2:68529849、chr9:101724394、chr12:124928495、chr17:57875544、chr19:51601507、chr2:74556187、chr9:111851651、chr12:130980102、chr17:57875573、chr19:51601508、chr2:80302861、chr9:113548848、chr12:130980198、chr17:57875612、chr19:51601509、chr2:85584290、chr9:114108496、chr13:34750527、chr17:57875627、chr19:51601510、chr2:85584348、chr9:121282620、chr13:80490728、chr17:58487622、chr19:51601580、chr2:85584364、chr9:124811105、chr13:98476650、chr17:58487643、chr19:51601581、chr2:85584373、chr9:125577538、chr13:98476685、chr17:58487664、chr19:51601597、chr2:85584379、chr9:127121856、chr13:98476703、chr17:58487747、chr19:51601603、chr2:85584386、chr9:127121880、chr13:98476740、chr17:58487814、chr19:51601624、chr2:101152526、chr9:127121963、chr14:23115298、chr17:58488080、chr19:52536666、chr2:101152638、chr9:127121964、chr14:38256108、chr17:58488116、chr19:52536686、chr2:101152643、chr9:127121994、chr14:64743251、chr17:58488275、chr19:55087135、chr2:101152644、chr9:127122016、chr14:68695352、chr17:59246476、chr19:55087177、chr2:101152688、chr9:127122021、chr14:73718571、chr17:63438487、chr19:55087394、chr2:113251274、chr9:127122092、chr14:91375612、chr17:63446755、chr20:2752143、chr2:127064916、chr9:127122093、chr14:94291257、chr17:74852370、chr20:2752154、chr2:219252952、chr9:127122165、chr14:101621513、chr17:76140257、chr20:2752233、chr2:236643030、chr9:127122179、chr14:102928211、chr17:77409611、chr20:2752646、chr2:237202186、chr9:127122257、chr14:102928212、chr17:77830614、chr20:2752655、chr2:237202187、chr9:127122258、chr14:103135447、chr17:77830632、chr20:2752673、chr2:240598627、chr9:127122266、chr14:103932682、chr17:78130182、chr20:2752688、chr2:240598628、chr9:127122267、chr14:104790584、chr17:78884051、chr20:20887989、chr3:19234357、chr9:127122297、chr14:104978811、chr17:80892292、chr20:22582417、chr3:43994917、chr9:127122337、chr14:105087674、chr17:81272581、chr20:22582495、chr3:47542858、chr9:131380762、chr14:105087732、chr17:81272582、chr20:22582508、chr3:49685862、chr9:131733731、chr14:105087733、chr17:81401923、chr20:22582551、chr3:50322471、chr9:131733759、chr14:105087752、chr17:82807710、chr20:22582552、chr3:65597378、chr9:133772391、chr14:105087766、chr18:2898307、chr20:22582556、chr3:65597401、chr9:136050771、chr14:105087773、chr18:10454023、chr20:22582560、chr3:
65597443、chr9:136050820、chr14:105087774、chr18:47531839、chr20:22582620、chr3:65597606、chr9:136299368、chr14:105087820、chr18:75089977、chr20:23656139、chr3:94044263、chr9:136299372、chr14:105087821、chr19:1016182、chr20:33644652、chr3:100399111、chr9:136588605、chr14:105087825、chr19:1046455、chr20:36808863、chr3:107430527、chr9:136688112、chr14:105087826、chr19:1136348、chr20:40690738、chr3:138071317、chr9:136688211、chr14:105087827、chr19:1253489、chr20:45373105、chr3:146497401、chr9:136997998、chr14:105087828、chr19:1253490、chr20:50865778、chr3:180829739、chr9:137461863、chr14:105087829、chr19:1253574、chr20:51487679、chr3:194277184、chr9:137461864、chr14:105087841、chr19:1257191、chr20:57212578、chr3:194397453、chr10:1516171、chr14:105087842、chr19:1387756、chr20:57212681、chr3:194487586、chr10:3780554、chr14:105087845、chr19:1851961、chr20:57212686、chr4:1023408、chr10:17287619、chr14:105088536、chr19:1852113、chr20:57212687、chr4:1752763、chr10:24383461、chr14:105489404、chr19:1860626、chr20:57212700、chr4:1752824、chr10:24383504、chr15:31393344、chr19:2042442、chr20:58613587、chr4:1752832、chr10:24383566、chr15:40769136、chr19:2042495、chr20:61698174、chr4:2270694、chr10:44293658、chr15:40769163、chr19:2042544、chr20:62259203、chr4:2801347、chr10:71260292、chr15:40769213、chr19:2042594、chr20:62416735、chr4:7740007、chr10:71715990、chr15:43799969、chr19:2042637、chr20:63562476、chr4:8156798、chr10:75399202、chr15:55492672、chr19:2042799、chr20:63978593、chr4:8229022、chr10:75408349、chr15:70349077、chr19:2207249、chr21:31539560、chr4:112131117、chr10:75408350、chr15:72227398、chr19:3208942、chr21:43194186、chr4:112131180、chr10:77636464、chr15:77887216、chr19:3375603、chr21:44653329、chr4:121711736、chr10:79080072、chr15:88639608、chr19:3375604、chr21:45430879、chr4:153486942、chr10:79113409、chr15:99123755、chr19:3375607、chr22:23457136、chr4:153486953、chr10:79242311、chr15:99123765、chr19:3375617、chr22:30224646、chr4:185741527、chr10:79242326、chr15:100115182、chr19:3375627、chr22:36577478、chr5:5495050、chr10:79242354、chr15:100115183、chr19:3375658、chr22:37676630、chr5:14577348、chr10:79242360、chr16:628678、chr19:3434932、chr22:38294213、chr5:55221002、chr10:79242372、chr16:785820、chr19:3434965、chr22:39292621、chr5:139482856、chr10:79242373、chr16:1303808、chr19:3434987、chr22:43244984、chr5:139482885、chr10:79242377、chr16:1379904、chr19:3434988、chr22:44625144、chr5:139697245、chr10:79242380、chr16:1964269、chr19:3458219、chr22:44841705、chr5:139697251、chr10:79242381、chr16:2124308、chr19:3687856、chr22:45537390、chr5:139697256、chr10:86224885、chr16:2124309、chr19:3687881、chr22:45922257、chr5:139697265、chr10:97922461、chr16:2124359、chr19:3687882、chr22:45996819、chr5:139697304、chr10:100371395、chr16:2176703、chr19:3687924、chr22:46006251、chr5:139697306、chr10:110499490、chr16:2929683、chr19:3687984、chr22:46535376、chr5:139697317、chr10:110499491、chr16:4943479、chr19:3688013、chr22:49600835、chr5:139697446、chr10:114631841、chr16:4943481、chr19:3688023、chr22:49959739、chr5:
139697484、chr10:120456377、chr16:4943543、chr19:3688033、chr22:50197602、chr5:139697498、chr10:124094248、chr16:10877393、chr19:3688041、chr22:50577975、chr5:139697550、chr10:127907972 chr16:11182276、chr19:3688042、chr22:50578304、chr5:139697582、chr10:132699462、chr16:11669773、chr19:3688069、chr22:50704114、chr5:139697591、chr11:627379、chr16:15143862、chr19:3688070、chrx:132420212。
11.根据本发明的技术方案,所述甲基化标志物包括如下甲基化位点中的一种或多种:chr20:22582560、chr14:64743251、chr9:133772391、chr22:50704114、chr7:2418150、chr10:79080072、chr2:85584290、chr20:62259203、chr11:70486699、chr22:36577478、chr16:1964269、chr22:49600835、chr17:76140257、chr1:1041251、chr20:33644652、chr9:127122179、chr16:66845951、chr7:256964、chr16:66936068、chr20:61698174、chr14:103932682、chr19:3375607、chr19:10087286、chr20:50865778、chr20:22582508、chr3:146497401、chr5:139697251、chr20:62416735、chr11:70742739、chr4:8156798。
12.根据本发明的技术方案,所述甲基化标志物包括如下甲基化位点中的一种或多种:chr20:22582560、chr17:76140257、chr14:64743251、chr1:1041251、chr7:2418150、chr16:66845951、chr10:79080072、chr7:256964、chr11:70486699、chr14:103932682、chr22:36577478、chr19:3375607。
13.本发明的目的之二在于提供,检测如上文所述的甲基化标志物的物质在制备检测甲状腺结节良恶性的产品中的用途。
14.根据本发明的技术方案,所述产品包括检测如上文所述的甲基化标志物的甲基化水平的物质。
15.优选地,所述检测甲基化标志物的物质选自引物、探针、pcr缓冲液、聚合酶、dntps、重亚硫酸盐及其衍生物、甲基化敏感或不敏感的限制性内切酶、酶切缓冲液、荧光染料、荧光淬灭剂、荧光报告剂、外切核酸酶、碱性磷酸酶、内标和对照物中的一种或多种。其中,所述引物和探针用于检测所述甲基化标志物的甲基化水平。
16.优选地,所述甲基化水平通过简化甲基化测序(rrbs)、甲基化特异性pcr(msp)、荧光定量pcr(msre-qpcr)、全基因组甲基化测序(wgbs)和甲基化芯片中的一种或多种获得。在本技术的某个优选实施方式中,通过简化甲基化测序(rrbs)获得。
17.本发明的目的之三在于提供,一种检测甲状腺结节良恶性的产品,所述产品包含检测如上文所述的甲基化标志物的物质。
18.根据本发明的技术方案,所述产品包括试剂盒、芯片、探针或膜条中的至少一种。
19.本发明的目的之四在于提供,一种甲状腺结节良恶性评估模型的构建方法,所述构建方法包括如下:
20.1)基于受试者生物样本中如上文所述的甲基化标志物的甲基化水平、样本信息及良恶性信息构建样本数据集,其中所述受试者包括已确诊甲状腺良性结节患者和甲状腺恶性结节患者,赋予恶性样本及良性样本不同的分类编码,并基于所述分类编码设置分类阈值;
21.2)将所述样本数据集随机分割为测试集和训练集,使用机器学习方法学习所述训练集的数据,得到所述的甲状腺结节良恶性评估模型。
22.根据本发明的技术方案,所述生物样品本选自血浆、血清、全血、尿液、羊水和组织
中的至少一项。
23.根据本发明的技术方案,所述机器学习方法选自支持向量机、随机森林算法和神经网络算法中的至少一种。
24.根据本发明的技术方案,所述测试集和训练集的分割比为(5~7)(3~5)。
25.根据本发明的技术方案,所述甲状腺结节良恶性评估模型用于输出预测值,当预测值高于所述分类阈值时,则评估为甲状腺恶性结节;当预测值低于所述分类阈值时,则评估为甲状腺良性结节。
26.根据本发明的技术方案,还包括步骤3)利用测试集对所述甲状腺结节良恶性评估模型进行验证。
27.本发明的目的之五在于提供,一种甲状腺结节良恶性评估模型的构建装置,所述装置包括:
28.数据收集模块,用于基于受试者生物样品本中如上文所述的甲基化标志物的甲基化水平构建样本数据集,其中所述受试者包括已确诊甲状腺良性结节患者和甲状腺恶性结节患者,赋予恶性样本及良性样本不同的分类编码,并基于所述分类编码设置分类阈值;
29.模型构建模块,用于将所述样本数据集随机分割为训练集和验证集,并使用机器学习方法学习所述训练集,得到所述的甲状腺结节良恶性评估模型。
30.根据本发明的技术方案,所述生物样品本选自血浆、血清、全血、尿液、羊水和组织中的至少一项。
31.根据本发明的技术方案,所述机器学习方法选自支持向量机、随机森林算法和神经网络算法中的至少一种。在本技术的某些优选实施方式中,为随机森林模型。
32.根据本发明的技术方案,所述测试集和训练集的分割比为(5~7)(3~5)。
33.根据本发明的技术方案,所述甲状腺结节良恶性评估模型用于输出预测值,当预测值高于所述分类阈值时,则评估为甲状腺恶性结节;当预测值低于所述分类阈值时,则评估为甲状腺良性结节。
34.优选地,所述分类阈值为0.5。
35.优选地,所述恶性样本的编码为1,所述良性样本的编码为0。
36.本发明的目的之六在于提供,一种用于评估甲状腺结节良恶性的装置,所述装置包括:
37.输入模块,用于获取待测者的生物样品本中如上文所述的甲基化标志物的检出量数据;
38.良恶性评估模块,应用如上文所述的构建方法得到的甲状腺结节良恶性评估模型对所述输入模块的检出量数据进行计算,得到预测值,并与分类阈值进行比较获得甲状腺结节良恶性结果;
39.输出模块,用于输出甲状腺结节良恶性结果。
40.根据本发明的技术方案,所述甲状腺结节良恶性评估模型用于输出预测值,当预测值高于所述分类阈值时,则评估为甲状腺恶性结节;当预测值低于所述分类阈值时,则评估为甲状腺良性结节。
41.优选地,所述分类阈值为0.5。
42.本发明的目的之七在于提供,一种检测甲状腺结节良恶性风险的方法,包括如下
步骤:
43.s1、获取待测者的生物样本中如上文所述的甲基化标志物的检出量数据;
44.s2、应用如上文所述的构建方法得到的甲状腺结节良恶性评估模型对所述检出量数据进行计算,得到预测值,并与分阈值进行比较获得甲状腺结节良恶性结果;
45.s3、根据计算比较结果,鉴别输出甲状腺结节的良恶性结果。
46.根据本发明的技术方案,在步骤s1中,所述甲基化标志物的检出量为标志物的甲基化水平,通过对所述生物样本中的基因组dna进行定量分析确定。
47.优选地,所述甲基化水平通过简化甲基化测序(rrbs)、甲基化特异性pcr(msp)、荧光定量pcr(msre-qpcr)、全基因组甲基化测序(wgbs)和甲基化芯片中的一种或多种获得。在本技术的某个优选实施方式中,通过简化甲基化测序(rrbs)获得。
48.根据本发明的技术方案,在步骤s1中,所述生物样本选自血浆、血清、全血、尿液、羊水和组织中的至少一项。作为优选地的实施方案,所述组织选自受试者的甲状腺结节新鲜组织样本、细针穿刺样本或福尔马林固定石蜡包埋(ffpe)样本。在本技术的某个优选实施方式中,为甲状腺结节新鲜组织样本。
49.本发明的目的之八在于提供,一种设备,包括处理器和存储器,所述存储器用于存储计算机程序,所述处理器用于执行所述存储器存储的计算机程序,以使所述设备执行如上文所述的构建方法或如上文所述的检测方法。
50.本发明的目的之九在于提供,一种计算机可读存储介质,其上存储有计算机程序,所述程序被处理器执行时实现如上文所述的构建方法或如上文所述的检测方法。
51.本发明的有益效果:
52.1)本发明的用于检测甲状腺结节良恶性的甲基化标志物是基于甲状腺恶性结节和良性结节的数据筛选得到,能够有效体现良恶性结节中的基因甲基化水平差异,对良恶性结节具有良好的区分效果,与其他研究中常使用的甲状腺恶性结节和癌旁组织相比,更符合临床诊断实际,具有更高的临床实践价值。
53.2)本发明进行了高通量筛选,并鉴定了780个全新的甲基化位点,关联273个肿瘤发生、发展相关基因,涉及吞噬作用信号通路(fc gamma r-mediated phagocytosis),提供的用于检测甲状腺结节良恶性的甲基化标志物包含多个基因和多个甲基化位点,通过多基因多位点的甲基化水平检测,克服单基因或单位点的甲基化信号低、不稳定的问题,从而提高了检测的灵敏度和特异性。
54.3)本发明提供的甲基化标志物及甲状腺结节良恶性评估模型,能用于甲状腺结节的良恶性辅助诊断,该模型的准确性为95%,auc为100%,敏感性为100%,特异性为91%,对甲状腺结节的良恶性具有良好的预测效果,能提高甲状腺结节良恶性临床诊断的准确性。
55.4)本发明的甲状腺良恶性评估模型不仅有极高的敏感性,有助于减少漏诊,利于早期治疗,提高患者生存率;而且其具有较高的阴性预测值,可避免患者不必要的手术,减轻社会医疗和经济负担,提高甲状腺结节患者整体生存质量。
56.5)本发明提供的甲状腺良恶性检测用模型可以通过分析待检生物样品中甲基化标志物的甲基化水平,直接给出良恶性预测结果,避免了超声影像学检测和细胞病理学检测中存在的结果判读的主观性,提高了结果判读的一致性和准确性,便于临床推广应用。
57.6)在临床实践中,本发明的方法可作为辅助传统诊断手段,与细针穿刺细胞学检测联合使用,无需额外取样,简化了诊断流程,提高了检测效率,减轻了患者的痛苦。
58.7)本发明是基于中国人群的临床数据进行开发的,所鉴定出的甲基化标志物和构建甲状腺良恶性评估模型更加适用于中国人群的甲状腺结节的良恶性评估。
附图说明
59.图1显示为本发明的实施例1中的甲状腺结节良恶性评估模型的重要特征。
60.图2显示为本发明的实施例1中的甲状腺结节良恶性评估模型的roc曲线。
具体实施方式
61.以下通过特定的具体实例说明本发明的实施方式,本领域技术人员可由本说明书所揭露的内容轻易地了解本发明的其他优点与功效。本发明还可以通过另外不同的具体实施方式加以实施或应用,本说明书中的各项细节也可以基于不同观点与应用,在没有背离本发明的精神下进行各种修饰或改变。
62.在进一步描述本发明具体实施方式之前,应理解,本发明的保护范围不局限于下述特定的具体实施方案;还应当理解,本发明实施例中使用的术语是为了描述特定的具体实施方案,而不是为了限制本发明的保护范围;在本发明说明书和权利要求书中,除非文中另外明确指出,单数形式“一个”、“一”和“这个”包括复数形式。
63.当实施例给出数值范围时,应理解,除非本发明另有说明,每个数值范围的两个端点以及两个端点之间任何一个数值均可选用。除非另外定义,本发明中使用的所有技术和科学术语与本技术领域技术人员通常理解的意义相同。除实施例中使用的具体方法、设备、材料外,根据本技术领域的技术人员对现有技术的掌握及本发明的记载,还可以使用与本发明实施例中所述的方法、设备、材料相似或等同的现有技术的任何方法、设备和材料来实现本发明。
64.本发明的第一方面保护,一种用于检测甲状腺结节良恶性的甲基化标志物,所述甲基化标志物包括以下甲基化区域中的一种或多种,甲基化区域见表1。
65.本发明中,所述甲状腺优选为甲状腺乳头状癌(ptc)。
66.表1
67.[0068][0069]
本发明中,所述甲基化区域包括如下甲基化位点中的一种或多种,甲基化位点见表2。
[0070]
表2
[0071]
[0072]
[0073]
[0074]
[0075][0076]
本发明中,作为优选地的实施方案,所述甲基化标志物包括如下甲基化位点中的一项或多项。甲基化位点见表3。
[0077]
表3
[0078]
chr20:22582560chr14:64743251chr9:133772391chr22:50704114chr7:2418150chr10:79080072chr2:85584290chr20:62259203chr11:70486699chr22:36577478chr16:1964269chr22:49600835chr17:76140257chr1:1041251chr20:33644652chr9:127122179chr16:66845951chr7:256964chr16:66936068chr20:61698174chr14:103932682chr19:3375607chr19:10087286chr20:50865778chr20:22582508chr3:146497401chr5:139697251 chr20:62416735chr11:70742739chr4:8156798 [0079]
本发明中,作为优选地的实施方案,所述甲基化标志物包括如下甲基化位点中的一项或多项。甲基化位点见表4。
[0080]
表4
[0081]
chr20:22582560chr17:76140257chr14:64743251chr1:1041251chr7:2418150chr16:66845951chr10:79080072chr7:256964chr11:70486699chr14:103932682chr22:36577478chr19:3375607
[0082]
本发明另一方面保护,一种与上文所述的甲基化标志物相关的基因,所述基因选
自如下中的至少一种,所述基因见表5。
[0083]
表5
[0084]
[0085][0086]
本发明另一方面保护,检测如上文所述的甲基化标志物的物质在制备检测甲状腺结节良恶性的产品中的用途。
[0087]
本发明中,所述检测甲基化标志物的物质选自引物、探针、pcr缓冲液、聚合酶、dntps、重亚硫酸盐及其衍生物、甲基化敏感或不敏感的限制性内切酶、酶切缓冲液、荧光染料、荧光淬灭剂、荧光报告剂、外切核酸酶、碱性磷酸酶、内标和对照物中的一种或多种。其中,所述引物和探针用于检测所述甲基化标志物的甲基化水平。
[0088]
本发明中,所述产品包括检测如上文所述的甲基化标志物的甲基化水平的物质。
[0089]
本发明中,所述甲基化标志物的甲基化水平的检测方法选自简化甲基化测序(rrbs)、甲基化特异性pcr(msp)、荧光定量pcr(msre-qpcr)、全基因组甲基化测序(wgbs)和甲基化芯片中的一种或多种。
[0090]
本发明的另一方面保护,一种检测甲状腺结节良恶性的产品,所述产品包含检测如上文所述的甲基化标志物的物质。
[0091]
本发明中,所述产品包括试剂盒、芯片、探针或膜条中的至少一种。
[0092]
本发明的另一方面保护,一种甲状腺结节良恶性评估模型的构建方法,所述构建方法包括如下:
[0093]
1)基于受试者生物样本中如上文所述的甲基化标志物的甲基化水平、样本信息及良恶性信息构建样本数据集,其中所述受试者包括已确诊甲状腺良性结节患者和甲状腺恶性结节患者,赋予恶性样本及良性样本不同的分类编码,并基于所述分类编码设置分类阈值;
[0094]
2)将所述样本数据集随机分割为测试集和训练集,使用机器学习方法学习所述训练集的数据,得到所述的甲状腺结节良恶性评估模型。
[0095]
本发明中,所述机器学习方法选自支持向量机、随机森林算法和神经网络算法中的至少一种。在本技术的某些优选实施方式中,为随机森林模型。
[0096]
本发明中,所述测试集和训练集的分割比为(5~7)(3~5)。在本技术的某些优选
实施方式中,所述测试集和训练集的分割比位6:4。
[0097]
本发明中,所述甲状腺结节良恶性评估模型用于输出预测值,当预测值高于所述分类阈值时,则评估为甲状腺恶性结节;当预测值低于所述分类阈值时,则评估为甲状腺良性结节。
[0098]
本发明中,所述分类阈值为0.5。
[0099]
本发明中,所述恶性样本的分类编码为1,所述良性样本的分类编码为0。
[0100]
本发明中,所述分类阈值根据诊断数据进行设置,例如建模时,恶性样本的分类编码为1,良性样本的分类编码为0,由r软件和随机森林训练模型的过程中,默认将分类阈值设置为0.5。构建出的模型最终也以0.5为分类阈值,对受试者进行良性和恶性区分。
[0101]
本发明中,还包括步骤3)利用测试集对所述甲状腺结节良恶性评估模型进行验证。
[0102]
本发明的另一方面保护,一种甲状腺结节良恶性评估模型的构建装置,所述装置包括:
[0103]
数据收集模块,用于基于受试者生物样品本中如上文所述的甲基化标志物的甲基化水平构建样本数据集,其中所述受试者包括已确诊甲状腺良性结节患者和甲状腺恶性结节患者,赋予恶性样本及良性样本不同的分类编码,并基于所述分类编码设置分类阈值;
[0104]
模型构建模块,用于将所述样本数据集随机分割为训练集和测试集,并使用机器学习方法学习所述训练集,得到所述的甲状腺结节良恶性评估模型。
[0105]
本发明中,所述机器学习方法选自支持向量机、随机森林算法和神经网络算法中的至少一种。在本技术的某些优选实施方式中,为随机森林模型。
[0106]
本发明中,所述测试集和训练集的分割比为(5~7)(3~5)。在本技术的某些优选实施方式中,测试集和训练集的分割比位6:4。
[0107]
本发明中,所述甲状腺结节良恶性评估模型用于输出预测值,当预测值高于所述分类阈值时,则评估为甲状腺恶性结节;当预测值低于所述分类阈值时,则评估为甲状腺良性结节。
[0108]
本发明中,所述分类阈值为0.5。
[0109]
本发明中,所述恶性样本的分类编码为1,所述良性样本的分类编码为0。
[0110]
本发明中,所述分类阈值根据诊断数据进行设置,例如建模时,恶性样本的分类编码为1,良性样本的分类编码为0,由r软件和随机森林训练模型的过程中,默认将分类阈值设置为0.5。构建出的模型最终也以0.5为分类阈值,对受试者进行良性和恶性区分。
[0111]
本发明的另一方面保护,一种用于评估甲状腺结节良恶性的装置,所述装置包括:
[0112]
输入模块,用于获取待测者的生物样品本中如上文所述的甲基化标志物的检出量数据;
[0113]
良恶性评估模块,应用如上文所述的构建方法得到的甲状腺结节良恶性评估模型对所述输入模块的检出量数据进行计算,得到预测值,并与分类阈值进行比较获得甲状腺结节良恶性结果;
[0114]
输出模块,用于输出甲状腺结节良恶性结果。
[0115]
本发明中,当预测值高于所述分类阈值时,则评估为甲状腺恶性结节;当预测值低于所述分类阈值时,则评估为甲状腺良性结节。
[0116]
本发明中,所述分类阈值为0.5。
[0117]
本发明的另一方面保护,一种检测甲状腺结节良恶性风险的方法,包括如下步骤:
[0118]
s1、获取待测者的生物样本中如上文所述的甲基化标志物的检出量数据;
[0119]
s2、应用如上文所述的构建方法得到的甲状腺结节良恶性评估模型对所述检出量数据进行计算,得到预测值,并与分类阈值进行比较获得甲状腺结节良恶性结果;
[0120]
s3、根据计算比较结果,鉴别输出甲状腺结节的良恶性结果。
[0121]
本发明中,在步骤s1中,所述检出量为甲基化标志物的甲基化水平,通过对所述生物样本中的基因组dna进行定量分析确定。
[0122]
本发明中,在步骤s2中,当预测值高于所述分类阈值时,则评估为甲状腺恶性结节;当预测值低于所述分类阈值时,则评估为甲状腺良性结节。
[0123]
本发明的另一方面保护,一种设备,包括处理器和存储器,所述存储器用于存储计算机程序,所述处理器用于执行所述存储器存储的计算机程序,以使所述设备执行如上文所述的构建方法或如上文所述的检测方法。
[0124]
本发明的另一方面保护,一种计算机可读存储介质,其上存储有计算机程序,所述程序被处理器执行时实现如上文所述的构建方法或如上文所述的检测方法。
[0125]
本发明中,所述生物样本选自血浆、血清、全血、尿液、羊水和组织中的至少一项。作为优选地的实施方案,所述组织选自受试者的甲状腺结节新鲜组织样本、细针穿刺样本或福尔马林固定石蜡包埋(ffpe)样本。在本技术的某个优选实施方式中,为甲状腺结节新鲜组织样本。
[0126]
本发明中,所述甲基化标志物的甲基化水平的检测方法选自简化甲基化测序(rrbs)、甲基化特异性pcr(msp)、荧光定量pcr(msre-qpcr)、全基因组甲基化测序(wgbs)和甲基化芯片中的一种或多种。
[0127]
优选地,简化甲基化测序(rrbs)是使用甲基化不敏感限制性内切酶mspi对dna进行酶切处理;对酶切后的dna片段进行末端修复、加a尾,并连接上所有胞嘧啶均经过甲基化修饰的测序接头;通过凝胶电泳切胶回收插入片段长度在40-220bp的dna片段;然后进行重亚硫酸盐(bisulfite)处理(使用ez dna methylation gold kit,zymo research),处理后,未发生甲基化的c变成u(经pcr扩增后变为t),而甲基化的c保持不变,最后进行pcr扩增,得到最终的dna文库并上机测序,然后对下机数据进行统计分析,从而得到基因组dna的甲基化水平。
[0128]
优选地,甲基化特异性pcr(msp)是基于用亚硫酸氢钠处理基因组dna,未甲基化的胞嘧啶变成尿嘧啶,而甲基化的胞嘧啶不变,通过针对性设计不同的特异性引物,对感兴趣的dna甲基化区域进行pcr扩增,将产物进行sanger一代测序或者琼脂糖凝胶电泳,从而确定与引物互补的dna序列的甲基化状态。优选地,甲基化敏感的限制性内切酶依赖的荧光定量pcr(msre-qpcr)是基于用甲基化敏感的限制性内切酶处理基因组dna,未甲基化的dna序列被切断,而甲基化的dna序列仍然完整,通过特异性引物探针,对感兴趣的目标区域进行扩增,甲基化修饰的完整dna片段被成功扩增,未被甲基化的区域由于被限制性内切酶切割成小片段而不能扩增,最后根据仪器捕捉到的荧光信号强度,通过软件分析确定dna序列的甲基化状态。
[0129]
优选地,全基因组甲基化测序(wgbs),首先将基因组片段化,再利用亚硫酸氢钠
(sodium bisulfite)处理dna导致未甲基化的c变成u并在后续pcr和测序过程成为t,而甲基化的c不受影响,然后进行dna修复,加接头并进行pcr扩增,得到最终的dna文库并上机测序,然后对下机数据进行统计分析,从而得到基因组dna的甲基化水平。
[0130]
优选地,甲基化芯片技术,是基于dna目标区域设计探针,通过两种不同探针的信号来区分甲基化的cpg位点和非甲基化的cpg位点的比例,并根据两者荧光信号的值来计算目的位点的甲基化比例,目前市面上用的较多的是850k芯片。
[0131]
实施例1甲状腺结节dna甲基化水平的分析
[0132]
本实施例中,以恶性甲状腺结节患者和良性甲状腺结节患者为研究对象,提取基因组dna,并对提取的基因组dna采用rrbs进行测序,得到恶性甲状腺结节患者和良性甲状腺结节患者的基因组dna的各个c位点的甲基化水平。包括如下步骤:
[0133]
1.1研究对象
[0134]
恶性甲状腺结节患者30人和良性甲状腺结节患者19人,恶性甲状腺结节患者患有乳头状癌(ptc)。
[0135]
2.2 dna提取
[0136]
获取步骤1.1中49例患者的甲状腺结节组织样本,使用qiaamp dna mini kit提取基因组dna。包括如下:
[0137]
(1)切取小块组织放入1.5ml离心管,加入裂解缓冲液和蛋白酶k,充分混匀,置于56℃孵育至组织充分裂解。
[0138]
(2)加入rna酶rnase a,混匀,室温温育2分钟。
[0139]
(3)加入终止缓冲液,混匀,70℃温育10分钟。
[0140]
(4)加入无水乙醇,混匀,短暂离心,转移至收集柱中,离心1分钟,弃废液。
[0141]
(5)加入洗涤缓冲液,离心1分钟,弃废液。
[0142]
(6)再加入洗涤缓冲液,离心3分钟,弃废液。
[0143]
(7)将收集柱置于最高转速空离1分钟,弃残留废液。
[0144]
(8)向收集柱的膜中央加入洗脱缓冲液,室温孵育1分钟,离心1分钟,洗脱dna。
[0145]
(9)使用琼脂糖凝胶电泳分析dna降解程度以及是否有rna污染,dna应无明显降解和污染;使用qubit荧光定量仪测定dna浓度,dna总量应不少于1.5μg,为合格的dna。
[0146]
1.3甲基化标志物的筛选
[0147]
1)简化甲基化测序(rrbs)文库的构建
[0148]
(1)在步骤1.2得到的合格dna中加入1%的阴性对照(lambda dna)。
[0149]
(2)使用甲基化不敏感的限制性内切酶mspi对dna进行酶切处理。
[0150]
(3)对酶切后的dna片段进行末端修复并加a尾,得到加a尾的dna片段。
[0151]
(4)在步骤(3)的加a尾的dna片段两端连接上所有胞嘧啶均经过甲基化修饰的测序接头。
[0152]
(5)将步骤(4)中加上测序接头的dna片段进行琼脂糖凝胶电泳,切胶回收长度在40-220bp的dna片段。
[0153]
(6)使用ez dna methylation gold kit(zymo research)对步骤(5)的dna片段进行重亚硫酸盐(bisulfite)处理。处理后,未发生甲基化的胞嘧啶(c)转换成尿嘧啶(u)(之后经pcr扩增变为胸腺嘧啶(t),而甲基化的胞嘧啶(c)保持不变。
[0154]
(7)将步骤(6)中经处理后的dna进行pcr扩增,得到最终的dna文库。
[0155]
2)文库质检
[0156]
(1)使用qubit荧光定量仪对步骤1)的dna文库进行初步定量,并将文库稀释至1ng/μl。
[0157]
(2)使用agilent 2100生物分析仪对文库的插入片段长度进行检测,主峰在250-350bp。
[0158]
(3)使用qpcr对文库的有效浓度进行准确定量,合格文库的有效浓度应大于2nm。
[0159]
3)文库测序
[0160]
质检合格的文库按照有效浓度和目标下机数据量进行混库后,在illumina测序平台上进行文库测序,得到测序结果。
[0161]
4)甲基化位点分析
[0162]
(1)将步骤3)得到的测序结果采用fastqc进行质量评估,然后用fastp进行质控过滤。
[0163]
(2)采用bismark进行参考基因组的比对分析:首先bismark将质控过滤后的测序reads和参考基因组序列都进行c到t和g到a(反向互补序列)的转化,然后将转化后的测序reads和转化后的基因组序列分别进行两两比对,将四种平行比对结果中唯一最佳的比对结果作为最终的比对结果。统计所有c位点的甲基化状态。
[0164]
另外通过lambda dna参考序列中c位点测序得到c的百分比,计算重亚硫酸盐未转换率(r)。
[0165]
(3)计算甲基化水平:对于每个c位点,统计支持该位点发生甲基化的reads数(mc)和支持该位点未发生甲基化的reads数(umc)。
[0166]
基于c位点的甲基化reads数(mc)、总reads数(mc+umc)和未转换率(r)进行二项分布检验,fdr矫正后的p值《0.05的位点定义为甲基化位点。
[0167]
每个位点的甲基化水平,统计覆盖每个c位点的甲基化和非甲基化的reads数,计算每个c位点的甲基化水平(ml),计算公式如下:
[0168]
ml=mc/(mc+umc)。
[0169]
(4)获得恶性和良性甲状腺结节的基因组dna的各个c位点的甲基化水平。
[0170]
实施例2甲基化标志物的筛选和甲状腺良恶性评估模型的构建
[0171]
本实施例中,对实施例1得到的恶性甲状腺结节患者和良性甲状腺结节患者的基因组dna的各个c位点的甲基化水平进行筛选,得到用于检测甲状腺结节良恶性的甲基化标志物,并根据筛选得到的甲基化标志物构建得到甲状腺良恶性评估模型。包括如下:
[0172]
2.1分析差异甲基化位点,得到用于检测甲状腺结节良恶性的甲基化标志物
[0173]
使用r(v3.6.2)程序包中的methylkit(v1.12.0)进行差异甲基化分析,包括如下步骤:
[0174]
1)首先过滤掉覆盖度小于10的位点和覆盖度大于99.9%覆盖度的位点。
[0175]
2)然后采用中位数法对每个样本的覆盖度进行均一化处理。
[0176]
3)接下来应用逻辑回归模型计算恶性和良性甲状腺结节样本中的差异甲基化水平,同时对患者年龄和性别进行协变量校正,计算的p值通过slim方法进行校正。
[0177]
4)最后将恶性和良性甲状腺结节样本中,甲基化水平差异绝对值大于阈值(如阈
值设为15%)且具有显著性差异(p值《0.001)的位点定义为差异甲基化位点。
[0178]
5)筛选得到的差异甲基化位点可作为甲基化标志物,总共获得了780个甲基化位点,780个甲基化位点以及与甲基化位点相关的273个基因见表1。
[0179]
表1
[0180]
[0181]
[0182]
[0183]
[0184]
[0185]
[0186]
[0187]
[0188]
[0189][0190]
2.构建甲状腺结节良恶性评估模型
[0191]
1)将30例恶性和19例良性样本共49例,其中49例中的60%作为训练集,剩余40%作为测试集。
[0192]
2)比对训练集中恶性甲状腺结节样本和良性甲状腺结节样本的780个甲基化标志物的甲基化水平上的差异,使用训练集对随机森林模型进行训练(50000trees,mtry=5)。
[0193]
甲状腺结节良恶性评估模型构建:将30例恶性甲状腺结节患者和19例良性甲状腺结节患者的甲基化标志物的甲基化水平形成数据矩阵(markers),49例样本的分类信息存储为第一列为样本名,第二列为分类信息甲状腺癌样本名为p.ca,良性样本名为(n.normal)的矩阵(pheno)。
[0194]
具体训练过程如下:
[0195]
a)打开r(v3.6.2)程序包,导入49例样本的分类信息矩阵(pheno)和相应的甲基化水平矩阵(markers)
[0196]
pheno=read.delim(“分类信息矩阵的存储路径”,sep="\t",as.is=t,header=t,check.names=false)
[0197]
markers=ead.delim(“甲基化水平矩阵的存储路径”,sep="\t",as.is=t,header=t,check.names=false)
[0198]
b)将甲基化水平矩阵转置为行名为样本名称,列名为甲基化标志物名称的矩阵
[0199]
imput=t(markers)
[0200]
c)用临近值补充法补上na值
[0201]
library(dmwr)
[0202]
imputed=knnimputation(imput)
[0203]
d)设置建模参数
[0204]
library(randomforest)
[0205]
mtry《-tunerf(imputed,pheno,ntreetry=50000,mtrystart=3,
[0206]
stepfactor=1,improve=0.001,trace=true,plot=true)
[0207]
e)构建随机森林训练模型
[0208]
mod_rf《-train(imputed,pheno,method='rf',mtry=mtry)
[0209]
在构建模型的过程中,可赋予恶性类型及良性类型不同的编码,并设置分类阈值,由r软件和随机森林方法训练模型,最终训练获得可基于分类阈值区分样本良性和恶性的甲状腺结节良恶性评估模型。
[0210]
在一个具体实施方式中,将恶性类型的分类编码为1,良性类型的分类编码为0,由r软件和随机森林训练模型的过程中,默认将分类阈值设置为0.5。
[0211]
构建出的模型最终也以0.5为分类阈值对样本进良性和恶性区分,最终训练获得的模型即为甲状腺结节良恶性评估模型。
[0212]
然后使用测试集的数据验证随机森林训练模型的分类效果。也即将测试集的数据经过甲状腺结节良恶性评估模型计算得到预测值。
[0213]
当预测值≥0.5时,则判定为恶性;当模型的预测值<0.5时,则判定为良性。
[0214]
图1为本实施例中构建的甲状腺结节良恶性评估模型的重要特征。
[0215]
图2为本实施例中构建的甲状腺结节良恶性评估模型的roc曲线。
[0216]
从图1和图2可知,构建的甲状腺结节良恶性评估模型的roc曲线线下面积auc为100%,敏感性为100%,特异性为91%,阴性预测值为100%,阳性预测值为94%,表明本发明构建的甲状腺结节良恶性评估模型具有良好的预测效果。
[0217]
实施例3甲状腺结节良恶性评估模型的测试
[0218]
本实施例中,对临床样本进行dna甲基化测序,根据测序结果中的甲基化标志物的甲基化水平,采用实施例2构建的甲状腺良恶性评估模型进行分析。包括如下步骤:
[0219]
3.1.临床样本
[0220]
14例恶性甲状腺结节患者和16例良性甲状腺结节患者的甲状腺结节组织,采用实施例1的方法提取基因组dna后,采用rrbs文库对提取的基因组dna进行测序,得到测序结果。
[0221]
3.2.将本实施例中步骤3.1得到的测序结果经过质控过滤后,采用bismark进行参考基因组的比对分析,然后计算各临床样本中甲基化标志物的甲基化水平,形成30个待评估样品的样本-标志物的数值矩阵。
[0222]
3.3.将本实施例中步骤3.2得到的30个待评估样品的样本-标志物的数值矩阵代
入实施例2中构建的甲状腺良恶性评估模型中,计算并输出临床样本属于恶性的概率。
[0223]
具体过程如下:
[0224]
a)打开r程序包,导入30例待评估样本-标志物的数值矩阵
[0225]
valdata=read.delim(“样本-标志物矩阵的存储路径”,sep="\t",as.is=t,
[0226]
row.names=1,header=t,check.names=f)
[0227]
b)将矩阵转置为行名为样本名称,列名为甲基化标志物名称的矩阵
[0228]
imput=t(valdata)
[0229]
c)并用临近值补充法补上na值
[0230]
library(dmwr)
[0231]
imputed=knnimputation(imput)
[0232]
d)再导入实施例2构建的甲状腺良恶性评估模型(rfmodeltc)
[0233]
model=readrds("rfmodeltc存储路径")
[0234]
e)之后开始进行模型评估,并计算输出每例样本的预测值,根据预测值判定每一例样本属于甲状腺结节恶性或甲状腺结节良性的风险。
[0235]
library(randomforest)
[0236]
class=predict(model,imputed,type="response")
[0237]
probs=predict(model,imputed,type="prob")
[0238]
当预测值>0.5时,即判断为甲状腺结节恶性,反之为甲状腺结节良性。
[0239]
将甲状腺结节良恶性评估模型的预测结果与临床病理结果进行比较,结果见表2。
[0240]
表2
[0241]
[0242][0243]
从表2可知,预测结果的准确性为93%(28/30),敏感性为93%(13/14),特异性为94%(16/17),阴性预测值为94%,阳性预测值为93%,表明本发明的甲状腺结节良恶性评估模型具有良好的分类效果。
[0244]
本发明通过对甲状腺组织的dna进行了甲基化水平测序,经对测序结果采用fastqc进行质量评估,然后用fastp进行质控过滤,采用bismark进行参考基因组的比对分析,得到甲基化水平;进一步应用逻辑回归模型计算恶性和良性甲状腺结节样本中的差异甲基化水平,甲基化水平差异绝对值大于阈值(如阈值设为15%)且具有显著性差异(p值《0.001)的位点定义为差异甲基化位点,筛选得到780个全新的甲基化位点,关联273个肿瘤发生、发展相关基因,涉及吞噬作用信号通路(fc gamma r-mediated phagocytosis),通过多基因多位点的甲基化水平检测,克服单基因或单位点的甲基化信号低、不稳定的问题,从而提高了检测的灵敏度和特异性。本发明提供的甲基化标志物及甲状腺结节良恶性评估模型,能用于甲状腺结节的良恶性辅助诊断,该模型的准确性为95%,auc为100%,敏感性为100%,特异性为91%,对甲状腺结节的良恶性具有良好的预测效果,能提高甲状腺结节良恶性临床诊断的准确性。采用本发明的模型进行临床诊断时,预测结果的的准确性为93%(28/30),敏感性为93%(13/14),特异性为94%(16/17),阴性预测值为94%,阳性预测值为93%,不仅有极高的敏感性,有助于减少漏诊,利于早期治疗,提高患者生存率;而且其具有较高的阴性预测值,可避免患者不必要的手术,减轻社会医疗和经济负担,提高甲状腺结节患者整体生存质量。此外,本发明提供的甲状腺良恶性检测用模型可以通过分析待检生物样品中甲基化标志物的甲基化水平,直接给出良恶性预测结果,避免了超声影像学检测和细胞病理学检测中存在的结果判读的主观性,提高了结果判读的一致性和准确性,具有临床推广应用价值。
[0245]
以上所述,仅为本发明的较佳实施例,并非对本发明任何形式上和实质上的限制,应当指出,对于本技术领域的普通技术人员,在不脱离本发明方法的前提下,还将可以做出若干改进和补充,这些改进和补充也应视为本发明的保护范围。凡熟悉本专业的技术人员,在不脱离本发明的精神和范围的情况下,当可利用以上所揭示的技术内容而做出的些许更动、修饰与演变的等同变化,均为本发明的等效实施例;同时,凡依据本发明的实质技术对上述实施例所作的任何等同变化的更动、修饰与演变,均仍属于本发明的技术方案的范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1