肝细胞肝癌预后生物标志物及应用
1.本发明实施例涉及生物医学技术领域,具体涉及一种肝细胞肝癌预后生物标志物及应用。
背景技术:
2.在全球范围内,肝癌的死亡率居恶性肿瘤死亡的第三位,其发病率位于第六,其中肝细胞肝癌(hepatocellular carcinoma,hcc)是肝癌的主要类型。在中国,新发肝癌占全球55%。在我国的所有癌症中,肝癌发病率居第四位,死亡率居第二位,五年生存率只有14%。尽管最近在肝癌诊断和治疗方面有所改善,但多数肝癌患者在确诊时均为中晚期癌,手术切除的病人只有20~30%,术后5年转移/复发率达60%-70%。尽管对肝癌发病和演进机制有着多年研究,但仍未完全明确肝癌早期诊断、肿瘤复发、转移等导致预后差相关因素的机制。对肝癌患者早诊早治、抗转移复发的研究是提高肝癌患者总体生存率的关键因素。其中,作为癌症生物标志物的基因、突变和蛋白等在癌症早期诊断中发挥重要作用。截至目前,有超过2万个人类基因被找出,然而,癌症中有价值的生物标志物却寥寥无几。因此,新的癌症标志物亟待发掘。
3.近年来,高通量测序技术的出现大大加快了许多人类肿瘤分子谱研究的速度。迄今为止,大规模的基因组和转录组学研究已经探索了hcc的遗传和表观遗传异质性。由于全面的基因组分析和生物信息学技术的发展,已经发现了更多的分子特征来研究hcc的新生物标志物和治疗靶标。
4.但大多是研究专注于单一层面的研究。癌症是多个基因突变、转录后修饰及其之间复杂调控关系的紊乱,以及与环境因素等共同作用的结果。传统的基于单一因素的识别方法不再满足当前研究的需要。癌症生物标志物识别算法逐渐从单基因单组学向多基因多组学转变,从基于静态关系向动态关系转变,从单一层面到系统生物学转变。系统识别基因和蛋白质等功能的变化,以及彼此间复杂调控关系的转变是当前研究的重点。因此,迫切需要从不同角度更加全面筛选用于hcc诊断和预后的更有效的预后生物标志物,以进一步探索关键基因促进肿瘤转移复发的相关机制。
技术实现要素:
5.为此,本发明实施例提供一种肝细胞肝癌预后生物标志物及应用。
6.为了实现上述目的,本发明实施例提供如下技术方案:
7.根据本发明实施例的第一方面,提供了生物标志物在评估肝细胞肝癌预后的产品中的应用,所述生物标志物为abat、acads、adh1a、adh1c、glyatl1、igf2bp2和sfn的组合。
8.根据本发明实施例的第二方面,提供了一种评估肝细胞肝癌预后的产品,所述产品包括检测生物标志物的试剂,所述生物标志物为abat、acads、adh1a、adh1c、glyatl1、igf2bp2和sfn的组合。
9.进一步地,所述产品还包括检测abat、acads、adh1a、adh1c、glyatl1、igf2bp2和
free survival,dss)、无病生存期(disease free surviva,dfs)、无进展生存期(progression free survival,pfs),发现基因模型在与肿瘤复发及进展过程中均有良好的临床意义。同时为验证我们构建模型的是否具有普适性,在hcc患者的临床队列和icgc生信队列中对模型进行独立验证,也得到了较好的效果。总之在单细胞数据中,通过cnv水平分析将肿瘤细胞分成2簇,对比分析得出不同肿瘤细胞簇中的差异基因;进一步结合转录组学和蛋白组学数据,得出一个由7个基因构成的预后模型,该模型在生信数据集和临床数据集中均得到了很好的验证,该模型有助于更好地预测肝癌患者的进展和预后,并可能成为判断肝癌预后的重要生物标记物。
21.本发明实施例具有如下优点:
22.本发明考虑到肝细胞肝癌的表型异质性,采用转录组学、蛋白组学等多组学数据集结合单细胞测序数据构建7基因的预后模型,该模型在临床数据集和icgc数据集进行了验证,拥有较好的灵敏度和特异性,可系统地评估肝癌的临床进展并预测患者的预后。结果表明,本发明提供的abat、acads、adh1a、adh1c、glyatl1、igf2bp2和sfn作为生物标志物能够有效评估肝细胞肝癌预后,对于肝癌患者的分子分型、早期诊断、个性化治疗和预后评估至关重要。
附图说明
23.为了更清楚地说明本发明的实施方式或现有技术中的技术方案,下面将对实施方式或现有技术描述中所需要使用的附图作简单地介绍。显而易见地,下面描述中的附图仅仅是示例性的,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据提供的附图引伸获得其它的实施附图。
24.图1为本发明实施例提供的生物信息学筛选肝细胞肝癌与预后效果恶性差异基因;其中,(a)tcga数据库差异基因;(b)tcga数据库预后相关基因;(c)cptac数据库差异基因;(d)cptac数据库预后相关基因;(e-f)gse125449肝细胞癌中不同细胞亚群;(g)共同差异基因;
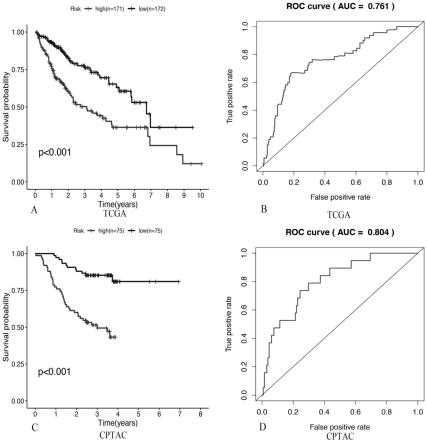
25.图2为本发明实施例提供的模型在tcga和cptac的预后分析;其中,(a-b)tcga数据集高危和低危人群的kaplan-meier曲线分析和预后模型的roc曲线分析;(c-d)cptac数据集高危和低危人群的kaplan-meier曲线分析和预后模型的roc曲线分析;
26.图3为本发明实施例提供的模型在tcga中疾病特异性生存期、无病生存期和无进展生存期分析;
27.图4为本发明实施例提供的模型在临床数据集和icgc的预后验证;其中,(a-b)icgc数据集高危和低危人群的kaplan-meier曲线分析和预后模型的roc曲线分析;(c-d)临床数据集高危和低危人群的kaplan-meier曲线分析和预后模型的roc曲线分析;
28.图5为本发明实施例提供的预后模型富集分析和免疫细胞的组成;其中,(a)根据tcga、cptac和icgc数据库风险评分,高风险组中共同激活的通路,nom p《0.05;(b)根据tcga、cptac和icgc数据库风险评分,高低风险组中免疫细胞浸润情况,蓝色代表低风险组,红色代表高风险组。
具体实施方式
29.以下由特定的具体实施例说明本发明的实施方式,熟悉此技术的人士可由本说明书所揭露的内容轻易地了解本发明的其他优点及功效,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
30.实施例1
31.1.研究对象
32.1)基因表达芯片数据库:从geo(基因表达综合)数据库下载gse125449数据,gse125449为单细胞测序数据,包含九名肝细胞癌患者的肝癌生物标本,这些肝癌患者都接受了标准根治手术,并且没有接受新辅助治疗,患者样本处理过程以及相关数据均在geo数据库呈现。从tcga(the cancer genome atlas,癌症基因组图谱)获得了374例肝细胞肝癌患者的mrna测序表达谱和预后相关数据;从临床蛋白质组学肿瘤分析联合会(the national cancer institute’s clinical proteomic tumor analysis consortium,cptac)下载了zhou-live数据集165例肝癌蛋白质表达数据及临床数据;从国际癌症基因组协会(international cancer genome consortium,icgc)获得liri-jp数据集150例hcc患者的基因表达文件和预后随访数据。纳入标准为:诊断为肝细胞肝癌,有完整的表达谱数据,具有完整的预后随访数据。
33.2)临床样本:本研究回顾性分析了2012年1月-2018年1月在石河子大学第一附属医院肝细胞癌患者的临床病理资料,选取了石河子大学第一附属医院病理科保存的肝癌组织105例,所有的肝癌样本均经病理医师确诊,并按2012年第四版who标准分级,入选标准如下:1)单发肝癌;2)根治性手术切除;3)术后病理证实为肝细胞肝癌;4)有完整预后资料。先前的研究表明,血管浸润是肝癌患者发生转移的主要途径,而且脉管浸润是hcc患者复发和不良预后的重要因素,因此将随访时确诊的转移患者和存在脉管癌栓定义为转移组织。该研究得到石河子大学第一附属医院伦理委员会的批准。
34.2.实验方法
35.数据库数据处理
36.1)单细胞rna-seq数据处理
37.针对gse125449单细胞rna-seq基因表达数据,使用seurat r程序包将scrna-seq数据转换为seurat对象。通过计算基因数、细胞计数和线粒体测序计数的百分比,对数据预处理:排除了细胞数少于3个的基因及基因数少于200个的细胞,线粒体的比例被限制在5%以下。进一步利用使用monocle2软件包的t-sne算法对细胞样本进行聚类分析。通过gse125449注释文件,结合singler软件包对细胞类别进行注释。根据基因在cnv水平对肿瘤细胞恶性进行分类。筛选出|log2-差异倍数|》1以及满足错误发现率(false discoveryrate,fdr)《0.05的高恶性肿瘤细胞与低恶性肿瘤细胞间差异基因(differentially expressed genes,degs)。
38.2)基因表达数据处理
39.将tcga肝癌队列转录组测序数据、icgc和cptac蛋白组数据导入r软件中,使用“limma”包来计算肿瘤与正常组织的差异表达基因。筛选标准为:|log2-差异倍数|》1以及满足fdr《0.05。之后通过单因素cox风险模型在tcga和cptac数据集中筛选具有预后价值的
degs。
40.3)构建基因预后模型并验证
41.通过整合gse125449、tcga和cptac数据,使用r软件“survival”和“glmnet”包通过cox回归和lasso分析选择与总生存期(overall survival,os)相关的基因并构建风险模型(p《0.05)。根据每个基因的标准化表达水平(ci)及其相应的回归系数(vi)计算患者的风险评分。所有患者的风险评分计算公式如下:
[0042][0043]
以中位风险评分作为临界值,将所有肝癌患者分为两组:低风险组和高风险组。log-rank检验和kaplan-meier生存曲线用于评估预后价值。接受者工作特征曲线(receiver operating characteristic,roc)分析用于评估预后预测模型的准确性。
[0044]
4)基因富集分析
[0045]
运用基因集富集分析(gene set enrichment analysis,gsea)软件(4.0.3)“c2.cp.kegg.v7.0.symbols.gmt”数据集识别高危和低危人群之间显著的通路,使用r包(包括plyr,ggplot2,grid和gridextra)以多重gsea形式显示了结果。nom p值《0.05被认为具有统计学意义。
[0046]
3.结果
[0047]
1)肝细胞肝癌预后相关差异基因的筛选
[0048]
通过对tcga转录组学、cptac蛋白组学数据进行差异分析及单变量cox回归分析,进而筛选出与预后相关的差异基因,其中tcga共筛选出3787个预后相关差异基因,cptac共筛选出215个基因与预后相关(图1a-d,p《0.05)。通过单细胞测序数据中cnv水平肿瘤细胞分成恶性肿瘤细胞和良性肿瘤细胞,通过对比分析,共得出2532个差异基因(图1e和f,p《0.05);对基因组学、蛋白组学和单细胞测序筛选的基因取交集,共得出37个基因(图1g)。
[0049]
2)构建多组学预后模型
[0050]
通过对tcga数据中37个多组学预后相关基因采用cox回归分析和lasso回归分析,确定了一个7基因(abat、acads、adh1a、adh1c、glyatl1、igf2bp2和sfn)模型,模型依照基因的表达水平和lasso系数计算所有患者的预后风险评分(表1)。我们使用风险评分的中位数将hcc患者分为低风险和高风险组。在tcga和cptac中,低、高风险组患者的生存率显显著不同,高风险组的患者生存率均明显低于低风险患者(p《0.01,图2a和c)。除了预测存活率外,我们的基因模型同时具有较好的诊断准确性:在两组队列中,较高的auc值证明了模型出色性能(tcga auc=0.761,cptac auc=0.804,图2b和d)。
[0051]
表1预后风险评分
[0052][0053][0054]
为了进一步分析模型在肝癌进展与预后中的作用,我们分析了模型与疾病特异性生存期(disease free survival,dss)、无病生存期(disease free surviva,dfs)、无进展生存期(progression free survival,pfs)的关系(图3)。其中dss代表患者死亡是由肝癌导致的,dfs代表活着疾病复发或死亡,pfs代表患者发生恶化或死亡。从这三项指标中可以得出,我们的模型就有很好的临床意义(p均《0.05)。
[0055]
3)验证并评估多组学预后模型对肝癌预后的作用价值
[0056]
为了验证多组学预后模型的稳定性,随后在临床数据集和icgc数据集分别选取50例和232例肝细胞肝癌患者,进行了进一步的验证。与tcga数据集分析相似,高危患者的os低于低危患者(p《0.05,图4)。并且具有良好的灵敏性和特异性(icgc auc=760,clinical data auc=0.787)。综上所述,这些结果证明并突出了我们的遗传模型在预测hcc患者的不良和准确诊断方面的稳健性,提示我们构建的模型可以较好适用于大多数肝细胞癌患者。
[0057]
4)预后模型与免疫密切相关
[0058]
为了揭示模型背后的分子机制,使用gsea软件分析了tcga、cptac和icgc等数据集高风险组的相关通路。如图5a所示,白细胞穿透迁移、b细胞受体信号通路和趋化因子信号通路等免疫相关效果途径在三个数据集高风险组的肿瘤中均显著富集。为了更好地研究免疫特征与hcc的关系,我们采用cibersort算法探究在cptac、tcga和icgc数据集中两个(低/高)风险组间的22种免疫细胞浸润情况。在cptac、tcga和icgc中,共有55例低风险样本和108例高风险样本符合筛选标准(cibersort p《0.05)。结果表明,与低风险样本相比,高风险hcc组织包含更多的未活化的nk细胞,静息树突状细胞,m0巨噬细胞和记忆b细胞,而未活化的b细胞、cd8 t、激活的nk细胞和静息肥大细胞减少(p《0.05,图5b)。提示我们筛选和鉴定的肝癌预后模型中的关键基因可能参与了肿瘤免疫微环境免疫细胞的调控。
[0059]
4.结论
[0060]
由单细胞转录组学分析、蛋白组学和转录组学分析构建的7基因(abat、acads、adh1a、adh1c、glyatl1、igf2bp2和sfn)模型可以较好的预测肝癌患者的进展及预后。
[0061]
虽然,上文中已经用一般性说明及具体实施例对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1