一种与凡纳滨对虾耐高氨氮性状相关的SNP标记、应用及引物的制作方法
一种与凡纳滨对虾耐高氨氮性状相关的snp标记、应用及引物
技术领域
1.本发明属于水产动物遗传及分子标记辅助选择育种技术领域,具体涉及一种与凡纳滨对虾耐高氨氮性状相关的snp标记及其应用、用于检测该snp标记的引物。
背景技术:
2.凡纳滨对虾(litopenaeus vannamei)是原产于太平洋东岸的对虾品种,俗称南美白对虾,1988年从美国引进我国,不久突破了人工繁育技术,但养殖规模一直较小。直至1999年我国再次引进无特定病原(spf)亲虾后,其养殖规模开始逐年增加。因其经济价值较高且具有生活力强、适应环境范围广、抗病力强等特点,目前是我国乃至世界养殖最广泛的甲壳类动物。目前凡纳滨对虾养殖常采用半集约化和集约化养殖方式,此类大规模养殖模式通常需要往水体中投喂大量饲料。这种情况下,残留饵料及对虾产生的粪便经过长期积累会导致水质恶化及水体氨氮浓度增加。非离子氨(nh3)和离子氨(nh
4+
)是水产养殖水体中氨氮的两种主要存在形式。其中非离子氨具有细胞膜透性,可通过水生动物细胞膜扩散并在其器官内累积,导致器官损伤并破坏生物的氧化/抗氧化平衡,使得出现氧化应激和对虾蜕皮频率增加的现象,最终导致膜完整性丧失,对虾免疫能力降低甚至出现死亡。
3.目前凡纳滨对虾选育通常采用群体和家系选育,但此类选育方法存在周期长、成本高、对遗传信息利用有限等缺点,特别是对无法直接测量且遗传力低的性状选育效率较低。因此,有效改善凡纳滨对虾的种质资源,培育对高氨氮具有较强耐受性的凡纳滨对虾新品种对对虾养殖业可持续发展尤为重要。
4.新一代的标记辅助育种(mas)可借助与性状紧密关联的遗传标记直接选择个体作为亲本进行育种。遗传标记是指用来区分不同的个体或群体并且能稳定遗传的物质或性状,常用的分子标记包括第一代分子标记rflp、rapd,第二代ssr、aflp和第三代分子标记snp等,其中第三代分子标记snp是指基因组特定位置上单个碱基存在的可遗传变异,且任一种等位基因在种群中的发生频率高于1%,变异的形式包括转换、颠换、插入/缺失等。snp标记因其具有数量巨大、分布广泛、共显性、高度稳定、易于自动化规模分析、能显示其他技术无法检测的隐藏多态性、可能与基因功能相关的众多优点,在分子标记辅助育种中得到广泛应用。如今,snp分子标记技术已广泛应用于凡纳滨对虾的遗传育种中,且多集中于凡纳滨对虾一些重要的功能基因的研究,包括生长、繁殖、耐低溶氧、耐高亚硝酸盐、抗副溶血弧菌等性状。此外,亦有关于对凡纳滨对虾的免疫相关基因进行snp位点分析的报道。但未见有与凡纳滨对虾耐高氨氮相关的snp分子标记的相关报道。
技术实现要素:
5.针对上述背景技术中的技术问题,本发明的目的是提供一种与凡纳滨对虾耐高氨氮性状相关的snp标记及其应用、检测所述的snp标记的引物对,为进行耐高氨氮性状关联研究提供有用信息,有利于凡纳滨对虾耐高氨氮性状的遗传改良。
6.本发明通过以下技术方案实现:
7.第一方面,本发明提供一种与凡纳滨对虾耐高氨氮性状相关的snp标记,从ozf基因中扩增得到一个snp分子标记,所述snp标记为如序列表中seq idno.1所示核苷酸序列,其中自5’端起第364位碱基为t或c。
8.第二方面,本发明提供一种用于检测所述与凡纳滨对虾高氨氮耐受性相关的snp标记的引物对,所述引物对具体是引物ozf-fp和引物ozf-rp,是以凡纳滨对虾锌指蛋白(ozf)基因m rna(gen bank accession:no.xm_027370330.1)序列为模板,利用primier 5.0软件设计所得。
9.所述引物对的核苷酸序列如seq id no.2和seq id no.3所示:
10.(1)seq id no.2:ccgattggatgcctttgga;
11.(2)seq id no.3:tcgtgtgaaagtattgttac。
12.第三方面,本发明提供包含上述引物对的试剂盒,用来检测上述的与凡纳滨对虾高氨氮耐受性相关的snp分子标记。
13.第四方面,本发明提供一种与凡纳滨对虾耐高氨氮性状相关的snp标记的应用,所述与凡纳滨对虾耐高氨氮性状相关的snp标记在筛选耐高氨氮抗性的凡纳滨对虾亲虾中的应用:
14.第五方面,本发明提供与凡纳滨对虾耐高氨氮性状相关的snp分子标记的筛选方法,包括如下步骤:
15.(1)以经高氨氮胁迫实验后凡纳滨对虾个体的基因组dna为模板,利用上述的引物对ozf-fp和ozf-rp进行pcr扩增,获得扩增片段,所述扩增片段的长度为710bp;
16.(2)将步骤(1)获得的扩增片段进行测序,得到ozf基因部分序列,将序列结果进行blast比对,筛选出碱基突变位点,即snp位点;
17.(3)用spss 24.0软件的卡方检验对凡纳滨对虾高氨氮耐受性与基因型进行相关性分析。
18.上述步骤(1)所述基因组dna的提取方法不受特别限制,可以采用任何已知的基因组dna提取方法或试剂盒进行;对基因组dna进行pcr扩增的条件也不受特别限制,pcr扩增条件可以由本领域技术人员进行优化选择。
19.直接测序是一种准确性最高、灵活性强,通量大、检测周期短的检测技术。该方法只需在snp位点的两侧设计一对引物,扩增得到的产物,通过测序即可直接检测到snp位点。因此,本发明优选采用直接测序的方法进行snp标记的检测。
20.本发明的有益效果为:
21.(1)本发明提供一个新发现的高氨氮胁迫抗性相关snp标记,该snp标记是在前期全基因组关联分析(gwas)结果的基础上通过候选snp关联分析鉴定得到的。经统计发现,本发明所述snp标记的位点处基因型为tt或tc的凡纳滨对虾的高氨氮耐受性显著高于基因型为纯合cc的凡纳滨对虾,通过检测凡纳滨对虾的上述snp位点,能够有效地确定其高氨氮的耐受性能;本发明的snp标记与凡纳滨对虾的耐高氨氮性状紧密相关,能够有效用于凡纳滨对虾的分子标记辅助育种。
22.(2)采用本发明技术方案可根据实际育种需求对凡纳滨育种材料进行早期选择,有效提高育种的效率和准确性,提高凡纳滨对虾繁殖群体的遗传水平,从而能够准确、高效地选育出抗逆性强的凡纳滨对虾品种。
23.(3)方法实用性强、基因组dna提取、测序方法没有特定要求,适用性广。
具体实施方式
24.通过具体的实施例,对本说明技术方案的具体实施方式进行进一步描述,这些实施例是为了对本技术方案的详细描述,而不是为了限制本技术方案。基于本说明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本说明保护的范围。
25.本发明提供的凡纳滨对虾耐高氨氮性状相关的snp位点可应用于凡纳滨对虾耐高氨氮性状的选育过程,具体是在凡纳滨对虾的选择育种过程中,对凡纳滨对虾育种候选群体进行snp位点分型,再结合其他与抗逆性状相关位点的分型信息,优先选择snp位点为tt基因型或tc基因型的个体作为耐高氨氮凡纳滨对虾选育亲本进行规模化养殖,避免选择snp位点为cc基因型的凡纳滨对虾个体作为选育亲本进行规模化养殖。
26.实施例1
27.本实施例通过对凡纳滨对虾进行全基因组关联分析,挖掘到了1个与高氨氮性状相关位点,位于基因锌指蛋白ozf内,发生了c/t突变,且t/c基因型凡纳滨对虾耐高氨氮能力略高于t/t基因型个体,并且显著高于c/c基因型个体。
28.与凡纳滨对虾耐高氨氮性状相关的snp分子标记的筛选方法,包括如下步骤:
29.1)将经高氨氮胁迫实验后凡纳滨对虾个体的基因组dna为模板,利用引物对ozf-fp和ozf-rp进行pcr扩增。所述引物ozf-fp和引物ozf-rp是以凡纳滨对虾锌指蛋白(ozf)基因m rna(gen bank accession:no.xm_027370330.1)序列为模板,利用primier 5.0软件设计所得。获得扩增片段,所述扩增片段的长度为710bp。
30.所述引物对的核苷酸序列如seq id no.2和seq id no.3所示:
31.(1)seq id no.2:ccgattggatgcctttgga;
32.(2)seq id no.3:tcgtgtgaaagtattgttac。
33.2)将步骤(1)获得的扩增片段进行测序,得到ozf基因部分序列,将序列结果进行blast比对,筛选出碱基突变位点,即snp位点。所述snp标记为如序列表中seq id no.1所示核苷酸序列,其中自5’端起第364位碱基为t或c。
34.3)用spss 24.0软件的卡方检验对凡纳滨对虾高氨氮耐受性与基因型进行相关性分析。
35.凡纳滨对虾ozf基因中snp位点364c》t与高氨氮耐受性的相关性分析,按照下列步骤进行:
36.(a)实验样品制备
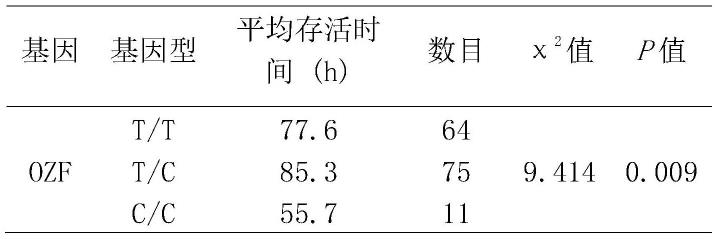
37.从不同家系中挑选150尾对虾放入3
×
3m的试验池,经过暂养后,以48小时半致死浓度进行高氨氮胁迫实验。在整个实验过程中,每天更换胁迫用水并调整好浓度。每小时观察1次死亡情况,记录每尾对虾的存活时间,并剪取每尾虾的肌肉组织保存在无水乙醇中,用于后续dna提取实验。对虾死亡的判断标准为对虾如果发生侧躺且没有触觉反应,则认为已死亡,高氨氮胁迫实验一直持续至所有对虾死亡后结束。
38.(b)凡纳滨对虾基因组dna提取
39.剪取25mg保存的对虾肌肉组织,放入灭菌的离心管中,加入200μl ga缓冲液和2粒
灭菌的钢珠,使用组织震荡研磨仪研磨2分钟。在研磨完成后样本中加入20μl蛋白酶k(proteinase k,20mg/ml)溶液,56℃水浴1h,每隔20分钟对样品进行振荡混合,每次振荡时间为15s。加入200μl缓冲液gb,充分颠倒混匀,70℃水浴10min。加入20μl rnase a(10mg/ml),室温静置5min。加入200μl无水乙醇,充分颠倒混匀,将溶液吸入吸附柱中,12,000rpm离心30sec,弃收集管中的废液,将吸附柱重新放回收集管中。向吸附柱中加入500μl缓冲液gd,12000rpm离心30s,弃收集管中的废液,将吸附柱重新放回收集管中。向吸附柱中加入600μl漂洗液pw,12000rpm离心30s,弃收集管中的废液,将吸附柱重新放回收集管中。此步骤需重复1次。将吸附柱放回收集管中,12000rpm离心2min,弃收集管中的废液。将吸附柱打开管盖,置于室温放置5分钟。将吸附柱放入灭菌离心管中,向吸附膜的中间部位加入60μl洗脱缓冲液te,室温放置5min,12000rpm离心2min。吸取离心管中的溶液重新吸入吸附柱中,室温放置2min,12000rpm离心2min,丢弃吸附柱,保留离心管及其中溶液,即为提取得到的dna。
40.采用1%琼脂糖凝胶电泳分析dna的完整性。使用nano drop nd-2000核酸定量仪检测dna溶液的纯度和浓度。将检测合格的dna样品储存在-20℃环境下,以备后续使用。合格dna的标准为:电泳检测条带完整单一、且没有拖尾现象,样品浓度大于50ng/ul,样品纯度(od 260/280)介于1.6~2.0范围。
41.(c)凡纳滨对虾ozf基因snp位点筛查及序列位点364c》t分型;
42.在ncbi网站中得到凡纳滨对虾锌指蛋白ozf基因(gen bank accessionno.xm_027370330.1)序列,通过premier 5.0软件设计得到特异性引物对ozf-fp和ozf-rp。以经高氨氮胁迫后对虾基因组dna为模板,利用上述引物在pcr条件下进行扩增。pcr反应体系为50μl:25μlpremix taqtm(lataqtm version2.0,takara)、17.5μl去离子水、2.5μl正向引物、2.5μl反向引物以及2.5μl基因组dna模板。pcr扩增反应程序如下:95℃预变性5min;95℃变性30s,54℃退火45s,72℃延伸1min,上述程序循环33次;72℃延伸10min后将pcr产物冷藏运输至广州生工生物科技有限公司进行测序。
43.(d)c》t基因型与耐高氨氮性状的相关性分析
44.据测序结果,统计高氨氮胁迫实验364c》t位点各基因型凡纳滨对虾数目和平均存活时间,利用spss19.0软件的卡方检验方法分析位点364c》t与凡纳滨对虾高氨氮耐受性的相关性,统计结果见表1。
45.结果表明:在位点364c》t中,tt、tc和cc这3种不同基因型的分布与高氨氮耐受性有显著关联性(χ2=9.414,p=0.009)。说明该位点的基因型多态性对凡纳滨对虾高氨氮耐受性有极显著的影响,tt和tc基因型个体耐高氨氮性能优于cc基因型。在选育及养殖过程中,优先选择位点364c》t为tt型或tc型的个体,尽量避免选择364c》t位点为cc型的个体。
46.表1基因ozf单核苷酸多态性的关联分析结果
[0047][0048]
尽管已经示出和描述了本说明的实施例,对于本领域的普通技术人员而言,可以理解在不脱离本说明的原理和精神的情况下可以对这些实施例进行多种变化、修改、替换和变型,本说明的范围由所附权利要求及其等同物限定。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1