一种萨福克羊体重相关SNP标记及其筛选方法与应用与流程
本发明涉及生物技术与动物遗传育种领域,具体涉及一种萨福克羊体重相关snp标记及其筛选方法与应用。
背景技术:
1、萨福克是世界公认的优质绵羊品种,体型壮硕,肉质好、瘦肉率高,具有良好的肉用饲养价值。世界上多个国家和地区引进萨福克羊,不仅作为纯种繁育进行饲养生产,同时还用作杂交育种的优质父本。
2、随着单核苷酸多态性(single nucleotide polymorphism snp)第三代分子标记的不断完善,利用分子育种提高绵羊的育种效率成了热点。而全基因组关联分析凭借其高效、快速的优点,迅速成为了分子育种的有效手段。现有技术中缺少与萨福克羊体重相关的snp标记。
技术实现思路
1、本发明提供一种萨福克羊体重相关snp标记及其筛选方法与应用,为今后优质萨福克羊的选育提供支撑。
2、本发明提供与绵羊体重相关的snp标记,包括以下分子标记中一种或多种:
3、1)chr7:76314472位点的snp标记:位于绵羊第7号染色体第76314472位,该处碱基为a或g;
4、2)chr7:76314515位点的snp标记:位于绵羊第7号染色体第76314515位,该处碱基为t或c。
5、根据所述与绵羊体重相关的snp标记,所述chr7:76314472位点的snp标记含有如seq id no.1所示的核苷酸序列;多态性位点位于如seq id no.1所示序列的第31位。
6、或,所述chr7:76314472位点的snp标记含有如seq id no.2所示的核苷酸序列;多态性位点位于如seq id no.2所示序列的第40位。
7、和/或,所述chr7:76314472位点的snp标记含有如seq id no.3所示的核苷酸序列;多态性位点位于如seq id no.3所示序列的第60位。
8、和/或,所述chr7:76314472位点的snp标记含有如seq id no.4所示的核苷酸序列;多态性位点位于如seq id no.4所示序列的第80位。
9、和/或,所述chr7:76314472位点的snp标记含有如seq id no.5所示的核苷酸序列;多态性位点位于如seq id no.5所示序列的第100位。
10、和/或,所述chr7:76314515位点的snp标记含有如seq id no.6所示的核苷酸序列;多态性位点位于如seq id no.6所示序列的第34位。
11、和/或,所述chr7:76314515位点的snp标记含有如seq id no.7所示的核苷酸序列;多态性位点位于如seq id no.7所示序列的第40位。
12、和/或,所述chr7:76314515位点的snp标记含有如seq id no.8所示的核苷酸序列;多态性位点位于如seq id no.8所示序列的第60位。
13、和/或,所述chr7:76314515位点的snp标记含有如seq id no.9所示的核苷酸序列;多态性位点位于如seq id no.9所示序列的第78位。
14、和/或,所述chr7:76314515位点的snp标记含有如seq id no.10所示的核苷酸序列;多态性位点位于如seq id no.10所示序列的第100位。
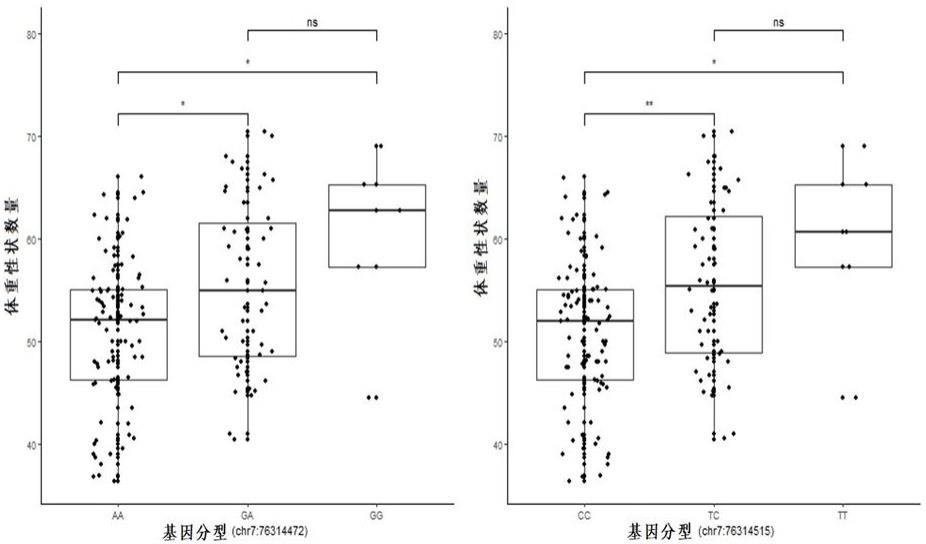
15、根据所述与绵羊体重相关的snp标记, chr7:76314472位点的snp标记,gg基因型对应的绵羊具有更高的体重,ga基因型对应的绵羊具有较高的体重,aa基因型对应的绵羊具有较低的体重;
16、和/或,chr7:76314515位点的snp标记,tt基因型对应的绵羊具有更高的体重,tc基因型对应的绵羊具有较高的体重,cc基因型对应的绵羊具有较低的体重。
17、本发明还提供一种引物,用于扩增所述snp标记。
18、本发明还提供含有所述引物的检测产品,所述检测产品为检测试剂、试剂盒或基因芯片。
19、本发明还提供所述snp标记、所述引物、或所述检测产品在以下任一项中的应用:
20、(1)在检测或辅助检测绵羊体重中的应用;
21、(2)在绵羊体重的早期预测中的应用;
22、(3)在绵羊分子标记辅助育种中的应用;
23、(4)在绵羊种质资源改良中的应用;
24、(5)在筛选高体重绵羊中的应用;
25、(6)在绵羊体重基因分型中的应用;
26、(7)在鉴定和/或筛选绵羊体重性状中的应用。
27、根据所述应用,鉴定和/或筛选高体重绵羊;
28、和/或,所述绵羊为萨福克羊;
29、和/或,chr7:76314472位点的snp标记,gg基因型对应的绵羊具有更高的体重,ga基因型对应的绵羊具有较高的体重,aa基因型对应的绵羊具有较低的体重;
30、和/或,chr7:76314515位点的snp标记,tt基因型对应的绵羊具有更高的体重,tc基因型对应的绵羊具有较高的体重,cc基因型对应的绵羊具有较低的体重;
31、和/或,所述snp位点参考基因组版本号为ovis aries (assembly ars-ui_ramb_v2.0);
32、和/或,包括以下步骤:
33、1)提取待测绵羊的基因组dna;
34、2)以待测绵羊的基因组dna为模板,利用所述引物进行pcr扩增反应;
35、3)分析pcr扩增产物。
36、本发明还提供了一种分子标记辅助选育大体重的萨福克羊的方法:利用chr7:76314472分子标记的多态性,不同基因型对体重的影响为gg>ga>aa,故保留g/g基因型的萨福克羊个体;利用chr7:76314515分子标记的多态性,不同基因型对体重的影响为tt>tc>cc,故保留t/t基因型的萨福克羊个体。
37、本发明还提供一种基于全基因组关联分析筛选绵羊体重性状相关候选标记方法,所述方法包含以下步骤:
38、采集多头绵羊的dna样品,进行全基因组重测序进行基因分型检测,并统计绵羊不同个体的体重性状,将得到的数据基于gmat全基因组关联分析方法进行分析,筛选与体重性状显著关联的单核苷酸多态snps及候选基因,整合基因功能注释筛选绵羊体重性状相关的snp分子标记。
39、根据所述基于全基因组关联分析筛选绵羊体重性状相关候选标记方法,全基因组关联测序数据经过质控后,利用gmat软件基于lmm模型进行关联分析,挖掘出与绵羊体重性状显著相关的snps,其中lmm模型为,式中y为表型向量,为固定效应或协变量,为的关联矩阵,为待检验标记效应,为的关联矩阵,为多基因效应,e为残差效应;
40、和/或,基于注释获得全基因组关联分析的显著snps后,下载该位点上下游500bp的序列后,通过ncbi和ensembl数据库进行比对,对显著snps进行候选基因注释;
41、和/或,用琼脂糖凝胶电泳检测dna的质量,用nanodrop检测dna的纯度,用qubit精确定量dna浓度。
42、根据所述基于全基因组关联分析筛选绵羊体重性状相关候选标记方法,所述绵羊为萨福克羊;
43、和/或,采集绵羊的耳组织提取dna;
44、和/或,记录绵羊的体重;
45、和/或,群体分层对绵羊体重性状的指标绘制q-q图,来判断关联分析中是否出现偏差和样本群体的分层现象;
46、和/或,适用r语言绘制qq plot图以及曼哈顿图;
47、和/或,只保留常染色上的位点,剔除缺失率大于10%的位点,剔除次等位基因品类小于0.05的位点,剔除哈代-温伯格平衡检验p值小于10-6的位点;
48、和/或,将质量合格的dna稀释至45-55ng/ul;进一步优选为48-52ng/ul;更优选为50ng/ul。
49、本发明还提供一种萨福克羊体重性状候选标记的筛选方法,主要目的是为其在培育优质萨福克羊的分子育种中的应用。
50、开展分子育种的前提是筛选某一性状的有效遗传标记,方可对其进行进一步的功能试验及群体验证。因此本发明通过对萨福克羊体重性状的全基因组关联分析,以其获得与体重性状显著相关的重要snps位点及候选基因。
51、本发明提供一种基于全基因组关联分析筛选萨福克羊体重性状相关候选标记的方法,所述方法包含以下步骤:
52、步骤1、实验动物的养殖及样本采集
53、实验选用的动物是萨福克羊,来自新疆康普森生物技术有限公司羊场,从该群体中根据系谱挑选300只作为全基因组测序对象并采集耳组织提取全基因组dna。
54、步骤3、生长性能数据记录
55、对300只羊的体重数据进行收集记录。
56、步骤4、基因组dna的提取及检测
57、基因组dna的提取采用dna试剂盒,按照说明说方法提取基因组dna,并对其进行完整性、纯度以及浓度的检测,符合要求的样本保留,不符合的重新提取,仍不合格的样本则淘汰。
58、步骤5、测序数据质控及参考基因组比对
59、实验按照华大基因提供的标准protocol执行,对检测合格的基因组dna样品,通过凝胶电泳的方法选择合适的大小片段,通过pcr扩增建库,再对建好的文库进行质量检测,合格的文库再用mgiseq-t7测序,该测序由北京康普森生物技术有限公司完成。为保证信息分析质量,在测序时,要进行碱基测序质量分布分析、碱基含量分布分析及将高通量测序得到的原始图像数据(raw reads)文件进行过滤。
60、步骤6、数据处理和统计分析,具体包括
61、(1)使用excel对体重数据进行分析,分析的结果为有效记录数、极大值、极小值、平均值、方差及标准差。
62、(2)全基因组关联分析:测序数据经过质控后,使用gmat软件基于lmm模型进行关联分析,挖掘与萨福克羊体重性状相关的snps,其中lmm模型为,式中y为表型向量,为固定效应或协变量,为的关联矩阵,为待检验标记效应,为的关联矩阵,为多基因效应,e为残差效应。
63、(3)在进行关联分析时,运用置换检验计算全基因组关联分析结果p值,使用1000次置换检验,提取千分之一的位置为每一次置换检验的p值,最后提取5%的位置作为最终阈值,来检测到有效snp是否达到显著水平。
64、(4)群体分层:群体分层是指由于祖先不同导致的等位基因频率差异,已被证实是一个混杂因素,可能会导致假阳性结果,因此对萨福克羊的体重指标绘制q-q图,来判断关联分析中是否出现偏差样本和本群体的群体分层现象。
65、(5)显著snps基因注释:在获得全基因组关联分析的显著snps位点后,下载该位点上下游500kb的碱基序列,通过ncbi和ensembl等数据库进行序列比对,确定显著snps附近的基因,并筛选出与该位点距离最近的基因。
66、进一步,步骤3中,关联分析了萨福克羊体重性状。
67、进一步,步骤5中,将测序获得的最终的序列重新比对到参考基因组上,再进行后续分析。参考基因物种为绵羊,参考基因组链接为:ovis aries genome assembly ars-ui_ramb_v2.0 - ncbi - nlm (nih.gov)。
68、有益效果
69、本发明提供的分子标记不受萨福克羊的年龄等限制,可用于具有更优质萨福克羊的早期育种,甚至刚出生即可进行准确地进行体重筛选,能大大加快萨福克羊的育种进程。
70、萨福克羊作为肉用绵羊,对其生长、肉质等重要形状的遗传改良进展直接影响企业的经济效益,而gwas在鉴定这些重要性状的遗传标记或主效基因上具有强大优势,可显著促进遗传育种进展,本研究通过gwas技术筛选与萨福克羊体重性状相关主效基因及snp,本发明方法方便快捷,准确性高,为后续选育优质萨福克羊提供理论支撑。
- 还没有人留言评论。精彩留言会获得点赞!