肿瘤标志物、以及将肿瘤细胞与夹杂细胞相区分来回收及检测的方法与流程
本发明涉及肿瘤标志物以及试样中包含的肿瘤细胞的回收及检测方法。特别地,本发明涉及使用试样中包含的肿瘤细胞所表达的蛋白质或基因将上述肿瘤细胞与上述试样中包含的夹杂细胞相区分、并回收及检测上述肿瘤细胞的方法。
背景技术:
肿瘤细胞一旦离开原发灶,则渗透到血管内或淋巴管内,在血液中或淋巴液中进行循环后最终入侵其它脏器、组织,从而形成转移灶。这里,在血液中进行循环的肿瘤细胞也被称为ctc(circulatingtumorcell,循环肿瘤细胞),进行了大量的临床试验、研究。例如,测定了从患者采集的血液中所含的ctc的数量(专利文献1);或者,通过调查上述ctc中的蛋白质表达、基因突变或易位来提供与上述患者的预后预测、癌症复发预测有关的信息。但是,血液中所含的ctc的数量非常少且血液中包含红细胞、白细胞等多种多样的夹杂细胞,因此在ctc检测/分析时需要从大量的细胞中仅区分并回收及检测少量的细胞的技术。
以往,在ctc的检测中,使用细胞角蛋白、epcam(epithelialcelladhesionmolecule,上皮细胞粘附分子)(专利文献1)等在ctc中表达的蛋白质(肿瘤标志物)来进行。但是,细胞角蛋白存在与血液所含的夹杂细胞的交叉(非特异检测)问题,epcam存在仅能回收及检测上皮性来源的部分ctc的问题。
现有技术文献
专利文献
专利文献1:日本特表2008-533487号公报
技术实现要素:
发明要解决的问题
本发明的课题在于,提供肿瘤标志物、以及将试样中包含的肿瘤细胞与该试样中包含的夹杂细胞相区分、并回收及检测试样中包含的肿瘤细胞的方法。
用于解决问题的方案
为了解决上述课题,本发明人们进行了10种癌细胞株与作为健康人试样的白细胞的、使用下一代测序仪的比较表达分析,结果发现了能够将试样中包含的肿瘤细胞与该试样中包含的夹杂细胞相区分、并回收及检测试样中包含的肿瘤细胞的肿瘤标志物,从而完成了本发明。
即,本发明可以例示如下。
[1]
一种将试样中包含的肿瘤细胞与夹杂细胞相区分来检测的方法,其特征在于,检测试样中存在的选自由以下的(i)至(iii)组成的组中的1种以上多肽。
(i)至少包含序列号1至6中的任一者记载的氨基酸序列的多肽;
(ii)包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的氨基酸序列的多肽;
(iii)至少包含序列号1至6中的任一者记载的氨基酸序列的剪接变体的多肽、或至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的多肽的剪接变体的多肽。
[2]
根据[1]所述的方法,其中,使用特异性地识别选自由(i)至(iii)组成的组中的1种以上多肽的抗体或适配体进行检测。
[3]
一种将试样中包含的肿瘤细胞与夹杂细胞相区分来检测的方法,其特征在于,检测试样中存在的编码以下的(i)至(iii)的任一多肽的基因。
(i)至少包含序列号1至6中的任一者记载的氨基酸序列的多肽;
(ii)包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的氨基酸序列的多肽;
(iii)至少包含序列号1至6中的任一者记载的氨基酸序列的剪接变体的多肽、或至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的多肽的剪接变体的多肽。
[4]
一种肿瘤细胞的回收方法,其中,将试样中包含的肿瘤细胞与夹杂细胞相区分来检测,并使用回收单元回收该检测出的肿瘤细胞,
通过检测试样中存在的选自由以下的(i)至(iii)组成的组中的1种以上多肽来进行肿瘤细胞的检测。
(i)至少包含序列号1至6中的任一者记载的氨基酸序列的多肽;
(ii)至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的氨基酸序列的多肽;
(iii)至少包含序列号1至6中的任一者记载的氨基酸序列的剪接变体的多肽、或至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的多肽的剪接变体的多肽。
[5]
根据[4]所述的方法,其中,使用特异性地识别选自由(i)至(iii)组成的组中的1种以上多肽的抗体或适配体进行肿瘤细胞的检测。
[6]
根据[1]至[5]中任一项所述的方法,其中,试样为血液,试样中包含的夹杂细胞为白细胞。
[7]
一种肿瘤标志物,其能够将试样中包含的肿瘤细胞与该试样中包含的白细胞相区分来检测,所述肿瘤标志物由以下的(i)至(iii)的任一多肽构成。
(i)至少包含序列号1至6中的任一者记载的氨基酸序列的多肽;
(ii)至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的氨基酸序列的多肽;
(iii)至少包含序列号1至6中的任一者记载的氨基酸序列的剪接变体的多肽、或至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的多肽的剪接变体的多肽。
[8]
一种肿瘤标志物,其能够将试样中包含的肿瘤细胞与该试样中包含的白细胞相区分来检测,所述肿瘤标志物由编码以下的(i)至(iii)的任一多肽的基因构成。
(i)至少包含序列号1至6中的任一者记载的氨基酸序列的多肽;
(ii)至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的氨基酸序列的多肽;
(iii)至少包含序列号1至6中的任一者记载的氨基酸序列的剪接变体的多肽、或至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的多肽的剪接变体的多肽。
[9]
一种肿瘤标志物,其能够将试样中包含的肿瘤细胞与该试样中包含的白细胞相区分来回收,所述肿瘤标志物由以下的(i)至(iii)的任一多肽构成。
(i)至少包含序列号1至6中的任一者记载的氨基酸序列的多肽;
(ii)至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的氨基酸序列的多肽;
(iii)至少包含序列号1至6中的任一者记载的氨基酸序列的剪接变体的多肽、或至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的多肽的剪接变体的多肽。
以下详细说明本发明。
本发明中,试样不限于从患者等采集的组织、全血、腹水等,也包括:将该组织、全血、腹水等用合适的缓冲液稀释而得的试样;血清、血浆、脐带血、成分采血液等来自全血的成分;将一片肝脏、肺、脾脏、肾脏、肿瘤、淋巴节等包含血液的组织用合适的缓冲液悬浮而成的悬浮液。进一步地,本发明中的试样还包括通过离心分离等对这些试样、悬浮液进行分离回收而得到的、包含肿瘤细胞的级分。
本发明中,试样中包含的夹杂细胞是指肿瘤细胞以外的细胞,在试样为全血、全血稀释液、源自全血的成分、包含血液的组织的悬浮液、以及由这些试样或悬浮液得到的包含肿瘤细胞的级分之类的血液试样的情况下,可列举红细胞、白细胞、血小板等。其中,本发明的肿瘤细胞的检测方法为与通过以往的肿瘤细胞的检测方法极有可能被判断为假阳性的、血液试样中包含的白细胞的分辨性优异的方法。另外,本发明的肿瘤细胞的回收方法为在通过以往的肿瘤细胞的回收方法极有可能被判断为假阴性的、血液试样中包含的epcam(epithelialcelladhesionmolecule,上皮细胞粘附分子)阴性的肿瘤细胞的回收方面优异的方法。
本发明中的肿瘤细胞的检测中,可以基于该细胞中存在的源自该细胞的上述(i)至(iii)的任一多肽或编码该多肽的基因利用染色、探针杂交法等直接检测试样中包含的肿瘤细胞,另外,也可以通过检测试样中包含的肿瘤细胞所分泌的源自该细胞的上述(i)至(iii)的任一多肽或编码该多肽的基因间接地检测肿瘤细胞。
本发明基于源自肿瘤细胞的上述(i)至(iii)的任一多肽或编码该多肽的基因的表达量来检测上述肿瘤细胞,可以定性地、即基于肿瘤细胞的有无来进行该检测,也可以定量地、即基于肿瘤细胞数、检测强度来进行该检测。另外,检测的判断基准也没有特别限定,例如,可以是只要检测信号存在、则即使少也判断为肿瘤细胞,也可以是若存在一定阈值以上的检测信号则判断为肿瘤细胞,还可以是将相对于试样中包含的夹杂细胞(例如在血液试样的情况下为红细胞、白细胞等)所具有的信号具有一定值以上或该信号的平均值的2sd以上、3sd以上或4sd以上的信号的细胞判断为肿瘤细胞。
本发明中的肿瘤细胞的回收可以如下进行:在通过检测该细胞中存在的来自该细胞的上述(i)至(iii)的任一多肽而将试样中包含的肿瘤细胞与夹杂细胞相区分并检测试样中包含的肿瘤细胞后,使用回收单元回收该检测出的细胞。需要说明的是,上述(i)至(iii)的任一多肽均为跨膜型的多肽。
本发明的特征在于,在试样中包含的肿瘤细胞的检测和/或回收中,使用作为该肿瘤细胞所表达的蛋白质之一的上述(i)至(iii)的任一多肽。
由序列号1记载的氨基酸序列构成的多肽是被称为tm4sf1(transmembrane4lsixfamilymember1,四次跨膜蛋白l6超家族成员1)、l6、m3s1、taal6、或h-l6的蛋白质,其氨基酸序列如genbankno.np_055035.1所示。
由序列号2记载的氨基酸序列构成的多肽是被称为tnfrsf12a(tnfreceptorsuperfamilymember12a,tnf受体超家族成员12a)、fn14、tweakr、或cd266的蛋白质,其氨基酸序列如genbankno.np_057723.1所示。
由序列号3记载的氨基酸序列构成的多肽是被称为sdc1(syndecan1,黏结蛋白聚糖1)、sdc、synd1、黏结蛋白聚糖、或cd138的蛋白质,其氨基酸序列如genbankno.np_002988.3所示。
由序列号4记载的氨基酸序列构成的多肽是被称为f3(凝血因子iii(coagulationfactoriii),组织因子(tissuefactor))、tf、tfa、或cd142的蛋白质,其氨基酸序列如genbankno.np_001984.1所示。
由序列号5记载的氨基酸序列构成的多肽是被称为epha2(ephreceptora2,肝配蛋白a2受体)、ctpa、ctpp1、ctrct6、eck、或arcc2的蛋白质,其氨基酸序列如genbankno.np_004422.2所示。
由序列号6记载的氨基酸序列构成的多肽是被称为itga2(integrinsubunitalpha2,整合蛋白α2亚基)、cd49b、gpia、hpa-5、vla-2、vlaa2、或br的蛋白质,其氨基酸序列如genbankno.np_002194.2所示。
其中,如后述的实施例所示,tm4sf1(序列号1)及tnfrsf12a(序列号2)在肺腺癌细胞、乳腺癌细胞、乳癌细胞、前列腺癌细胞、肝癌细胞、胰腺癌细胞中均高表达,不受癌症种类限制,与以往的检测中使用的作为肿瘤标志物的epcam相比可以广泛地检测试样中包含的肿瘤细胞。另外,tm4sf1(序列号1)及tnfrsf12a(序列号2)为跨膜型蛋白,因此与以往的回收中使用的作为肿瘤标志物的epcam相比可以大范围地回收试样中包含的肿瘤细胞。
另外,在想要特异性地检测胰腺癌细胞时,后述的实施例所示的基因表达分析的结果表明优选使用itga2(序列号6)。需要说明的是,itga2(序列号6)为跨膜型蛋白,因此表明在想要特异性地回收胰腺癌细胞时优选使用该蛋白质。
需要说明的是,本发明中,只要与由序列号1至6中的任一者记载的氨基酸序列构成的蛋白质具有相同的活性,则也可以为将序列号1至6中的任一者记载的氨基酸序列的1至数个氨基酸残基置换为其他氨基酸残基或使其缺失的形态,另外也可以为在序列号1至6中的任一者记载的氨基酸序列中插入1至数个氨基酸残基而成的形态。这里,本说明书中,“数个”是指20、19、18、17、16、15、14、13、12、11、10、9、8、7、6、5、4、3或2个的整数。1至数个氨基酸残基是指:优选氨基酸残基为1个以上且10个以下,更优选为1个以上且5个以下,进一步优选为1个以上且3个以下,特别优选为2个以下。
另外,只要同样具有蛋白质功能,则也可以为在这些多肽的n末端和/或c末端添加有一个以上氨基酸残基的形态。另外,本发明的方法中使用的标志物只要与由序列号1至6中的任一者记载的氨基酸序列构成的蛋白质具有相同的活性,则可以为相对于序列号1至6中的任一者记载的氨基酸序列整体具有70%以上的同源性的蛋白质,优选为具有80%以上、更优选为具有90%以上、进一步更优选为具有95%以上的同源性的蛋白质。
进一步地,在真核细胞中由编码蛋白质的基因表达该蛋白质时,会发生除去基因(rna)前体中的内含子、将前后的外显子重新结合的反应(剪接),由于残留的外显子存在多样性而生产出各种成熟mrna,因此有时会表达出活性不同的蛋白质(剪接变体)。本发明中能够用于检测的多肽还包括:
至少包含序列号1至6中的任一者记载的氨基酸序列的剪接变体的多肽、
至少包含将序列号1至6中的任一者记载的氨基酸序列的1至数个氨基酸残基置换为其他氨基酸残基或使其缺失的多肽的剪接变体的多肽、
至少包含在序列号1至6中的任一者记载的氨基酸序列中插入了1至数个氨基酸残基的多肽的剪接变体的多肽、
至少包含与序列号1至6中的任一者记载的氨基酸序列具有70%以上的同源性的多肽的剪接变体的多肽。
在进行本发明的检测时,可以添加能够特异性地标记上述(i)至(iii)记载的任一多肽的色素(例如化学发光色素、荧光色素等)而将其染色,基于其染色图像或染色强度(例如发光强度、荧光强度)进行检测,从通用性的角度出发,使用针对上述多肽的抗体(以下也简单表述为抗多肽抗体)或适配体进行检测的方法为常规方法,并且能够用于直接检测/间接检测两者,因此可以说是优选方式。
另外,上述(i)至(iii)记载的多肽为跨膜型蛋白,因此也能成为将试样中包含的肿瘤细胞与该试样中包含的白细胞相区分、并回收试样中包含的肿瘤细胞的肿瘤标志物。
在使用抗多肽抗体检测试样中包含的肿瘤细胞时,优选通过任意方法将标记物修饰到上述抗体上,基于该标记物的有无或量进行检测。标记物可以从利用抗原抗体反应的测定领域通常所使用的物质中适当选择,作为一例,可列举荧光素等荧光物质、碱性磷酸酶等酶、放射性物质。上述抗体与标记物的结合方式没有限定,可以为上述抗体通过化学键等与标记物直接结合的方式,也可以为上述抗体通过针对标记物所结合的上述抗体的抗体(标记二抗)而进行间接结合的方式。使用了抗多肽抗体的、对于试样中所包含的肿瘤细胞的检测格式没有特别限定,可以通过惯用方法利用elisa(enzyme-linkedimmunosorbentassay,酶联免疫)法、蛋白质印迹法等进行检测,也可以使用aia-900、aia-cl2400(均为东曹株式会社制)等酶联免疫分析装置自动进行检测。
这样,上述(i)至(iii)记载的多肽也能成为能够将试样中包含的肿瘤细胞与该试样中包含的白细胞相区分、并检测试样中包含的肿瘤细胞的肿瘤标志物。
在利用本发明将肿瘤细胞与夹杂细胞相区分、并回收肿瘤细胞时,在使用上述(i)至(iii)中的任一者记载的多肽将该肿瘤细胞与夹杂细胞相区分并检测该肿瘤细胞后,使用回收单元回收该检测出的肿瘤细胞即可。作为回收单元的一例,可列举具备设置有能够保持肿瘤细胞的保持部的基板、和通过利用喷嘴的抽吸、吐出来回收该肿瘤细胞的回收部的单元,作为具体例,可列举日本特开2016-142616号中公开的回收装置。
另一方面,本发明中,编码上述的(i)至(iii)的任一多肽的基因只要是使用密码子将上述(i)至(iii)记载的任一多肽转换而得到的多核苷酸就没有特别限定,特别优选使用人类密码子转换而成的多核苷酸。
作为编码至少包含由序列号1至6中的任一者记载的氨基酸序列构成的多肽的多肽的基因的一例,可列举以下序列。
对由序列号1记载的序列构成的多肽进行转换而成的、由序列号7记载的序列中第235位至第840位为止的核苷酸构成的多核苷酸(genbankno.nm_014220.2)、
对由序列号2记载的序列构成的多肽进行转换而成的、由序列号8记载的序列中第87位至第473位为止的核苷酸构成的多核苷酸(genbankno.nm_016639.2)、
对由序列号3记载的序列构成的多肽进行转换而成的、由序列号9记载的序列中第392位至第1321位为止的核苷酸构成的多核苷酸(genbankno.nm_001006946.1)、
对由序列号4记载的序列构成的多肽进行转换而成的、由序列号10记载的序列中第222位至第1106位为止的核苷酸构成的多核苷酸(genbankno.nm_001993.4)、
对由序列号5记载的序列构成的多肽进行转换而成的、由序列号11记载的序列中第156位至第3083位为止的核苷酸构成的多核苷酸(genbankno.nm_004431.3)、
对由序列号6记载的序列构成的多肽进行转换而成的、由序列号12记载的序列中第144位至第3686位为止的核苷酸构成的多核苷酸(genbankno.nm_002203.3)。
上述的基因检测方法没有特别限定,例如,可以对上述多核苷酸中的合适位置设计探针、在此基础上通过杂交法进行检测,也可以对上述多核苷酸中的合适位置设计引物组并在此基础上使用pcr法、rt-pcr法、trc(transcriptionreversetranscriptionconcerted,转录-逆转录协同)法、nasba(nucleicacidsequence-basedamplification,基于核酸序列扩增)法、tma(transcription-mediatedamplification,转录介导的扩增)法等扩增、检测上述多核苷酸,还可以将包含上述多核苷酸的试样供于直接测序仪来进行检测。
这样,编码上述的(i)至(iii)记载的多肽的基因可成为能够将试样中包含的肿瘤细胞与该试样中包含的白细胞相区分、并检测试样中包含的肿瘤细胞的肿瘤标志物。
发明的效果
根据本发明,通过检测下述任一者记载的多肽或编码这些多肽的基因,可以实施试样中包含的肿瘤细胞的检测,所述多肽为:(i)至少包含包括tm4sf1(genbankno.np_055035.1)及tnfrsf12a(genbankno.np_057723.1)在内的6种氨基酸序列中的任一者的多肽、(ii)包含与上述(i)的氨基酸序列具有70%以上的同源性的氨基酸序列的多肽、(iii)至少包含上述(i)或(ii)的氨基酸序列的剪接变体的多肽。通过上述检测方法,可以将试样中仅少量地包含的肿瘤细胞与该试样中包含的夹杂细胞相区分,并检测试样中仅少量地包含的肿瘤细胞。
另外,通过使用上述(i)至(iii)中的任一者记载的多肽检测肿瘤细胞,使用回收单元回收该检测出的肿瘤细胞,从而可以实施试样中包含的肿瘤细胞的回收。通过上述回收方法,可以将试样中仅少量地包含的肿瘤细胞与该试样中包含的夹杂细胞相区分,并回收试样中仅少量地包含的肿瘤细胞。
附图说明
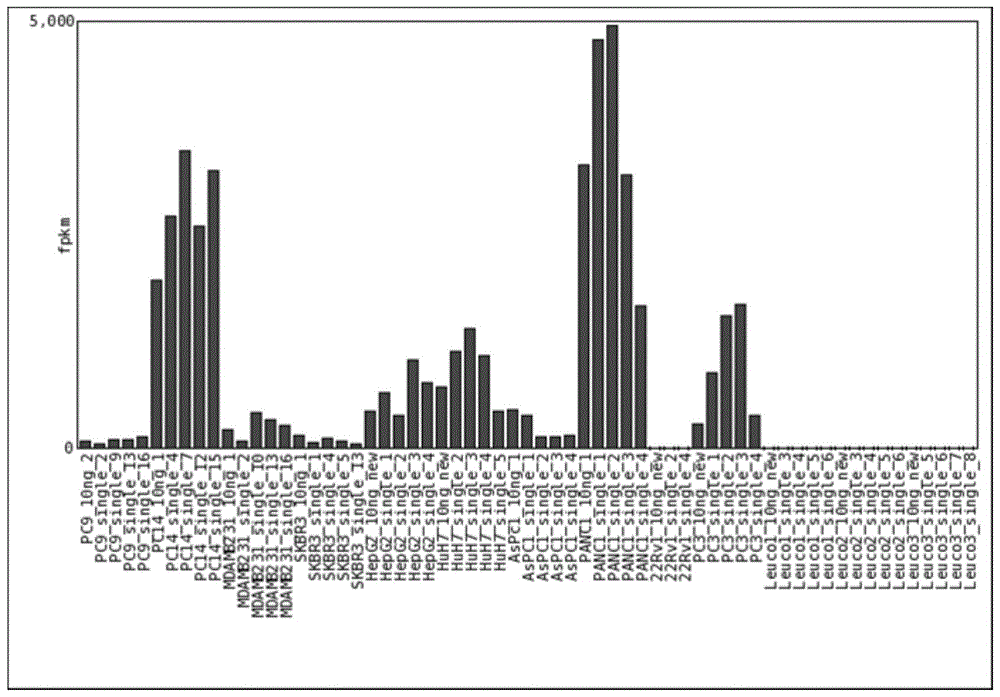
图1为示出健康人白细胞及各种癌细胞的tm4sf1基因表达分析结果的图。
图2为示出健康人白细胞及各种癌细胞的tnfrsf12a基因表达分析结果的图。
图3为示出用(a)抗tm4sf1抗体及(b)抗cd45抗体对掺加(spike)了肺腺癌细胞(pc14)的血液试样进行免疫染色的结果的图。
图4为示出用(a)抗tm4sf1抗体及(b)抗cd45抗体对掺加了胰腺癌细胞(panc1)的血液试样进行免疫染色的结果的图。
图5为示出用(a)抗tnfrsf12a(cd266)抗体及(b)抗cd45抗体对掺加了肺腺癌细胞(pc9)的血液试样进行免疫染色的结果的图。
图6为示出用(a)抗tnfrsf12a(cd266)抗体及(b)抗cd45抗体对掺加了乳腺癌细胞(mdamb231)的血液试样进行免疫染色的结果的图。
图7为示出用(a)抗cd142(f3)抗体及(b)抗cd45抗体对掺加了肺腺癌细胞(pc9)的血液试样进行免疫染色的结果的图。
图8为示出用(a)抗cd142(f3)抗体及(b)抗cd45抗体对掺加了乳腺癌细胞(mdamb231)的血液试样进行免疫染色的结果的图。
图9为示出用(a)抗epha2抗体及(b)抗cd45抗体对掺加了前列腺癌细胞(pc3)的血液试样进行免疫染色的结果的图。
图10为示出用(a)抗itga2抗体及(b)抗cd45抗体对掺加了胰腺癌细胞(panc1)的血液试样进行免疫染色的结果的图。
图11为示出用(a)抗itga2抗体及(b)抗cd45抗体对掺加了胰腺癌细胞(aspc-1)的血液试样进行免疫染色的结果的图。
图12为示出用(a)抗sdc1抗体及(b)抗cd45抗体对掺加了乳癌细胞(skbr3)的血液试样进行免疫染色的结果的图。
图13为示出用(a)抗sdc1抗体及(b)抗cd45抗体对掺加了胰腺癌细胞(aspc-1)的血液试样进行免疫染色的结果的图。
具体实施方式
实施例
以下,使用试样为血液试样时的实施例进一步详细说明本发明,但本发明不受该例子限定。
实施例1健康人白细胞及各种癌细胞的基因表达分析
作为癌细胞株,选择人肺腺癌细胞(pc9及pc14)、人乳腺癌细胞(mdamb231)、人乳癌细胞(skbr3)、人前列腺癌细胞(22rv1及pc3)、人肝癌细胞(hepg2及huh-7)、人胰腺癌细胞(panc1及aspc-1)这10种,使用下一代测序仪,用以下的方法分析这些癌细胞株与健康人白细胞的基因表达量的差异。
(1)将癌细胞株分别用以下所示的培养基在5%co2环境下在37℃下培养后,使用0.25%胰蛋白酶/1mmedta从培养基剥离细胞,从而以单细胞单位回收癌细胞(n=4),由该单细胞的rna利用smart-seqv4ultralowinputrnakitforsequencing(clontech)进行cdna合成和扩增。另外,用从3名健康人的血液回收的白细胞,也同样地以单细胞单位(n=4)由rna进行cdna的合成和扩增。
包含pc9、pc14及panc1:10%(v/v)fbs(胎牛血清)的dmem(杜尔贝科改良伊格尔培养基)/ham’sf-12培养基
aspc-1:包含10%(v/v)fbs和1mm丙酮酸的rpmi-1640培养基
mdamb231:包含10%(v/v)fbs的、加入了l-谷氨酰胺的leibovitz’sl-15培养基的培养基
skbr3:包含10%(v/v)fbs的mccoy’s5a培养基
hepg2及huh-7:包含10%(v/v)fbs的dmem/高葡萄糖培养基
22rv1:包含10%(v/v)fbs的rpmi0211-1640培养基
pc3:在10%(v/v)fbs中包含卡那霉素/链霉素的ham’sf-12k培养基。
(2)将多个上述(1)中得到的细胞集中在一起,用rneasyminikit(qiagen)回收总rna后,由10ng的rna利用smart-seqv4ultralowinputrnakitforsequencing(clontech)进行cdna合成和扩增。另外,对于由3名健康人的血液回收的白细胞,也同样地由多个细胞回收rna,由10ng分的rna进行cdna合成和扩增。
(3)使用上述(1)及(2)中得到的cdna1ng分,利用nexteraxtdnalibrarypreparationkit(illumina)及nexteraxtv2indexkitseta(illumina)进行文库制备,使用next-seq500(illumina)在读长75bp、单端测序的条件下进行测序分析,从而对每一试样解读1000万读取以上的序列。
(4)将上述(3)中解读的核苷酸序列(测序数据)用tophat2(johnshopkins大学)及bowtie2(johnshopkins大学)映射于人基因组序列。需要说明的是,人基因组序列及人基因信息使用了ncbi(nationalcenterforbiologicalinformation)公开的buildgrch38。对于进行了映射的核苷酸序列,通过cufflinks(washington大学)由解读而得的各基因的读取数求出以fpkm(fragmentsperkilobaseofexonpermillionreadsmapped)为单位的每基因的表达值。
(5)对于健康人白细胞的3个被检体的15个样品和10种癌细胞株的48个样品进行表达比较。
癌细胞株(10种、48个样品)的表达值(fpkm值)的平均为健康人白细胞(3个被检体、15个样品)的表达值(fpkm值)的平均的10.00倍以上,并且将编码跨膜型蛋白的基因示于表1。另外,针对上述基因中的编码tm4sf1(序列号1)及tnfrsf12a(序列号2)的基因,将各样品每一个的分析结果分别示于图1及图2。图中,分别地,pc9及pc14为人肺腺癌细胞的结果,mdamb231为人乳腺癌细胞的结果,skbr3为人乳癌细胞的结果,hepg2及huh-7为人肝癌细胞的结果,aspc-1及panc1为人胰腺癌细胞的结果,22rv1及pc3为人前列腺癌细胞的结果,leuco1至leuco3为健康人白细胞的结果,single为单细胞(n=4)中的rna的结果,10ng为多个细胞中的rna(10ng分)的结果。
[表1]
tm4sf1基因(序列号7)的、癌细胞株(10种、48个样品)的表达值(fpkm值)的平均为白细胞(3个被检体、15个样品)的fpkm值的平均的300倍以上,可知tm4sf1基因在癌细胞(肿瘤细胞)中特异性表达且高表达(表1)。另外,tm4sf1基因在肺腺癌细胞、乳腺癌细胞、乳癌细胞、肝癌细胞、胰腺癌细胞、前列腺癌细胞中均高表达,不受癌症种类限制(图1),因此可知tm4sf1蛋白(序列号1)及基因(序列号7)与以往的检测中使用的作为肿瘤标志物的epcam相比可以广泛地检测试样中包含的肿瘤细胞。另外可知,tm4sf1蛋白(序列号1)为跨膜型蛋白,因此与以往的回收中使用的作为肿瘤标志物的epcam相比可以广泛地回收试样中包含的肿瘤细胞。
同样地,tnfrsf12a基因(序列号8)的、癌细胞株(10种、48个样品)的表达值(fpkm值)的平均为白细胞(3个被检体、15个样品)的fpkm值的平均的50倍以上,可知tnfrsf12a基因在癌细胞(肿瘤细胞)中特异性表达且高表达(表1)。另外,tnfrsf12a基因在肺腺癌细胞、乳腺癌细胞、乳癌细胞、肝癌细胞、胰腺癌细胞、前列腺癌细胞中均高表达,不受癌症种类限制(图2),因此可知tnfrsf12a蛋白(序列号2)及基因(序列号8)与以往的检测中使用的作为肿瘤标志物的epcam相比可以广泛地检测试样中包含的肿瘤细胞。另外可知,tnfrsf12a蛋白(序列号2)为跨膜型蛋白,因此与以往的回收中使用的作为肿瘤标志物的epcam相比可以广泛地回收试样中包含的肿瘤细胞。
肺腺癌细胞株(2种、10个样品)的表达值(fpkm值)的平均为健康人白细胞(3个被检体、15个样品)的表达值(fpkm值)的平均的10.00倍以上,并且将编码跨膜型蛋白的基因示于表2。
[表2]
乳腺癌或乳癌细胞株(2种、10个样品)的表达值(fpkm值)的平均为健康人白细胞(3个被检体、15个样品)的表达值(fpkm值)的平均的10.00倍以上,并且将编码跨膜型蛋白的基因示于表3。
[表3]
前列腺癌细胞株(2种、8个样品)的表达值(fpkm值)的平均为健康人白细胞(3个被检体、15个样品)的表达值(fpkm值)的平均的10.00倍以上,并且将编码跨膜型蛋白的基因示于表4。
[表4]
肝癌细胞株(2种、10个样品)的表达值(fpkm值)的平均为健康人白细胞(3个被检体、15个样品)的表达值(fpkm值)的平均的10.00倍以上,并且将编码跨膜型蛋白的基因示于表5。
[表5]
胰腺癌细胞株(2种、10个样品)的表达值(fpkm值)的平均为健康人白细胞(3个被检体、15个样品)的表达值(fpkm值)的平均的10.00倍以上,并且将编码跨膜型蛋白的基因示于表6。
[表6]
将表1所示的、癌细胞株(10种、48个样品)的表达值(fpkm值)的平均为健康人白细胞(3个被检体、15个样品)的表达值(fpkm值)的平均的10.00倍以上的基因中的、在全部癌症种类(肺腺癌、乳腺癌或乳癌、前列腺癌、肝癌、胰癌)的细胞中达到健康人白细胞(3个被检体、15个样品)的表达值(fpkm值)的平均的10.00倍以上的基因示于表7。如上所述,表明该表所示的蛋白质及基因与以往的检测中使用的作为肿瘤标志物的epcam相比可以广泛地检测试样中包含的肿瘤细胞。另外,由于该表所示的蛋白质为跨膜型蛋白,因此,如上所述还表明与以往的回收中使用的作为肿瘤标志物的epcam相比可以广泛地回收试样中包含的肿瘤细胞。
[表7]
将表6所示的、胰腺癌细胞株(2种、10个样品)的表达值(fpkm值)的平均为健康人白细胞(3个被检体、15个样品)的表达值(fpkm值)的平均的10.00倍以上的基因中的、在胰癌以外(肺腺癌、乳腺癌或乳癌、前列腺癌、肝癌)的细胞株中的表达值(fpkm值)的平均低于全部健康人白细胞(3个被检体、15个样品)的表达值(fpkm值)的平均的10.00倍的基因示于表8。表明该表所示的蛋白质及基因可以特异性地检测胰腺癌细胞。另外,该表所示的蛋白质为跨膜型蛋白,因此表明特异性地回收胰腺癌细胞。
[表8]
实施例2使用抗tm4sf1抗体的癌细胞和白细胞的染色
实施例1中已经明确了:与健康人白细胞相比,tm4sf1基因在癌细胞中高表达,因此研究了tm4sf1能否用于血液试样中包含的肿瘤细胞的特异性检测。
(1)将使用比重差分离法从健康人血液中除去红细胞而成的试样作为本实施例中使用的血液试样,向该试样中分别掺加人肺腺癌细胞(pc14)或人胰腺癌细胞(panc1)。
(2)向掺加了癌细胞株的血液试样100μl中加入fcr封闭剂(miltenyibiotec)10μl,在室温下进行10分钟的封闭处理。
(3)封闭处理后,加入针对tm4sf1的抗体(humantm4sf1phycoerythrinmab、r&dsystems)溶液10μl及用于检测白细胞的针对cd45的抗体(cd45-fitc,beckmancoulter)溶液10μl,在室温下孵育30分钟。
(4)孵育后,用包含1%bsa和5mmedta的hbss(hank’s平衡盐溶液)洗涤细胞3次,用permountfisher(fisherscientific)封入载玻片,进行荧光显微镜观察。
分别地,将人肺腺癌细胞(pc14)的染色结果示于图3,将人胰腺癌细胞(panc1)的染色结果示于图4。图中,箭头所示的细胞为掺加的癌细胞。图3及图4的结果均为:被针对tm4sf1的抗体(抗tm4sf1抗体)染色的细胞(图3的(a)及图4的(a)中箭头所示的细胞)未被针对cd45的抗体染色(图3的(b)及图4的(b))。由该结果可知:tm4sf1(序列号1)可以作为将血液试样中包含的肿瘤细胞与该试样中包含的白细胞相区分、并检测血液试样中包含的肿瘤细胞的肿瘤标志物使用。
实施例3使用抗tnfrsf12a抗体的癌细胞和白细胞的染色
实施例1中已经明确了:与健康人白细胞相比,tnfrsf12a基因在癌细胞中高表达,因此研究了tnfrsf12a能否用于血液试样中包含的肿瘤细胞的特异性检测。
(1)将使用比重差分离法从健康人血液中除去红细胞而成的试样作为本实施例中使用的血液试样,向该试样中分别掺加人肺腺癌细胞(pc9)或人乳腺癌细胞(mdamb231)。
(2)向掺加了癌细胞株的血液试样100μl中加入fcr封闭剂(miltenyibiotec)10μl,在室温下进行10分钟的封闭处理。
(3)封闭处理后,加入针对tnfrsf12a的抗体(pe抗人cd266(fn14、tweakr)抗体、biolegend)溶液2.5μl及用于检测白细胞的针对cd45的抗体(cd45-fitc,beckmancoulter)溶液10μl,在室温下孵育30分钟。
(4)孵育后,用包含1%bsa和5mmedta的hbss(hank’s平衡盐溶液)洗涤细胞3次,用permountfisher(fisherscientific)封入载玻片,进行荧光显微镜观察。
分别地,将人肺腺癌细胞(pc9)的染色结果示于图5,将人乳腺癌细胞(mdamb231)的染色结果示于图6。图中,箭头所示的细胞为掺加的癌细胞。图5及图6的结果均为:被针对tnfrsf12a的抗体(抗tnfrsf12a抗体)染色的细胞(图5的(a)及图6的(a)中箭头所示的细胞)未被针对cd45的抗体染色(图5的(b)及图6的(b))。由该结果可知:tnfrsf12a(序列号2)可以作为将血液试样中包含的肿瘤细胞与该试样中包含的白细胞相区分、并检测血液试样中包含的肿瘤细胞的肿瘤标志物使用。
实施例4使用抗f3抗体的癌细胞和白细胞的染色
实施例1中已经明确了:与健康人白细胞相比,f3基因在癌细胞中高表达,因此研究了f3能否用于血液试样中包含的肿瘤细胞的特异性检测。
(1)将使用比重差分离法从健康人血液中除去红细胞而成的试样作为本实施例中使用的血液试样,向该试样中分别掺加人肺腺癌细胞(pc9)或人乳腺癌细胞(mdamb231)。
(2)向掺加了癌细胞株的血液试样100μl中加入fcr封闭剂(miltenyibiotec)10μl,在室温下进行10分钟的封闭处理。
(3)封闭处理后,加入针对f3的抗体(cd142-fitc、miltenyibiotec)溶液10μl及用于检测白细胞的针对cd45的抗体(cd45-pe,beckmancoulter)溶液7μl,在室温下孵育30分钟。
(4)孵育后,用包含1%bsa和5mmedta的hbss(hank’s平衡盐溶液)洗涤细胞3次,用permountfisher(fisherscientific)封入载玻片,进行荧光显微镜观察。
将人肺腺癌细胞(pc9)的染色结果示于图7,将人乳腺癌细胞(mdamb231)的染色结果示于图8。图中,箭头所示的细胞为掺加的癌细胞。图7及图8的结果均为:被针对f3的抗体(抗f3抗体)染色的细胞(图7的(a)及图8的(a)中箭头所示的细胞)未被针对cd45的抗体染色(图7的(b)及图8的(b))。由该结果可知:f3(序列号4)可以作为将血液试样中包含的肿瘤细胞与该试样中包含的白细胞相区分、并检测血液试样中包含的肿瘤细胞的肿瘤标志物使用。
实施例5使用抗epha2抗体的癌细胞和白细胞的染色
实施例1中已经明确了:与健康人白细胞相比,epha2基因在前列腺癌细胞中高表达,因此研究了epha2能够用于血液试样中包含的前列腺癌细胞的特异性检测。
(1)将使用比重差分离法从健康人血液中除去红细胞而成的试样作为本实施例中使用的血液试样,向该试样中掺加人前列腺癌细胞(pc3)。
(2)向掺加了癌细胞株的血液试样100μl中加入fcr封闭剂(miltenyibiotec)10μl,在室温下进行10分钟的封闭处理。
(3)封闭处理后,加入针对epha2的抗体(pe抗人epha2、biolegend)溶液5μl及用于检测白细胞的针对cd45的抗体(cd45-fitc、beckmancoulter)溶液10μl,在室温下孵育30分钟。
(4)孵育后,用包含1%bsa和5mmedta的hbss(hank’s平衡盐溶液)洗涤细胞3次,用permountfisher(fisherscientific)封入载玻片,进行荧光显微镜观察。
将人前列腺癌细胞(pc3)的染色结果示于图9。图中,箭头所示的细胞为掺加的前列腺癌细胞。图9的结果为被针对epha2的抗体(抗epha2抗体)染色的细胞(图9的(a)中箭头所示的细胞)未被针对cd45的抗体染色(图9的(b))。由该结果可知:epha2(序列号5)可以作为将血液试样中包含的前列腺癌细胞与该试样中包含的白细胞相区分、并检测血液试样中包含的前列腺癌细胞的肿瘤标志物使用。
实施例6使用抗itga2抗体的癌细胞和白细胞的染色
实施例1中已经明确了:与健康人白细胞相比,itga2基因在胰腺癌细胞中高表达,因此研究了itga2能否用于血液试样中包含的胰腺癌细胞的特异性检测。
(1)将使用比重差分离法从健康人血液中除去红细胞而成的试样作为本实施例中使用的血液试样,向该试样中分别掺加人胰腺癌细胞(panc1或aspc-1)。
(2)向掺加了癌细胞株的血液试样100μl中加入fcr封闭剂(miltenyibiotec)10μl,在室温下进行10分钟的封闭处理。
(3)封闭处理后,加入针对itga2的抗体(fitc-抗人itga2抗体、biolegend)溶液5μl及用于检测白细胞的针对cd45的抗体(cd45-pe、beckmancoulter)溶液20μl,在室温下孵育40分钟。
(4)孵育后,用包含1%bsa、5mmedta和6μg/ml替罗非班的hbss(hank’s平衡盐溶液)洗涤细胞2次,接种到载玻片上,进行荧光显微镜观察。
分别地,将人胰腺癌细胞panc1的染色结果示于图10,将人胰腺癌细胞aspc-1的染色结果示于图11。图中,箭头所示的细胞为掺加的胰腺癌细胞。图10及图11的结果均为:被针对itga2的抗体(抗itga2抗体)染色的细胞(图10的(a)及图11的(a)中箭头所示的细胞)未被针对cd45的抗体染色(图10的(b)及图11的(b))。由该结果可知:itga2(序列号6)可以作为将血液试样中包含的胰腺癌细胞与该试样中包含的白细胞相区分、并检测血液试样中包含的胰腺癌细胞的肿瘤标志物使用。
实施例7使用抗tm4sf1抗体、抗tnfrsf12a抗体及抗epcam抗体的癌细胞染色
实施例1中已经明确了:与健康人白细胞相比,tm4sf1基因及tnfrsf12a基因在癌细胞中高表达,因此使用识别tm4sf1、tnfrsf12a及作为现有的肿瘤标志物的epcam的抗体研究了tm4sf1蛋白及tnfrsf12a蛋白能否用于癌细胞的检测。需要说明的是,在本实施例中,作为癌细胞,使用了实施例1所用的癌细胞株(10株)。
(1)向2.5×103个/100μl(pc-9或pc-14)或1×104个/100μl(mdamb231、skbr3、pc-3、22rv1、hepg2、huh-7、panc-1或aspc-1)的癌细胞株悬浮液100μl中加入fcr封闭剂(miltenyibiotec)10μl,在室温下进行10分钟的封闭处理。
(2)封闭处理后,加入抗tm4sf1抗体10μl(pe-抗人tm4sf1抗体、r&dsystems)、抗tnfrsf12a抗体2.5μl(pe-抗人tnfrsf12a抗体、biolegend)或抗epcam抗体3μl(alexafluoro488-抗人epcam抗体、biolegend),在室温下孵育60分钟。
(3)孵育后,用包含1%bsa、5mmedta和6μg/ml替罗非班的hbss(hank’s平衡盐溶液)洗涤细胞2次,接种到载玻片上,进行荧光显微镜观察。
将使用抗tm4sf1抗体、抗tnfrsf12a抗体及抗epcam抗体的癌细胞染色结果示于表9。需要说明的是,在上述表中,用源于各荧光标记抗体的荧光信号的强度(+至+++++的5级)表示染色结果,该信号的强度越大(+的数越多)越表示高表达(信号强度(表达量):+<++<+++<++++<+++++)。结果是,tm4sf1蛋白(序列号1)及tnfrsf12a蛋白(序列号2)不受癌症种类限制地在肺腺癌细胞、乳腺癌细胞、乳癌细胞、前列腺癌细胞、肝癌细胞、胰腺癌细胞中表达,可知可以广泛地检测试样中包含的肿瘤细胞。另外,该结果表明,tm4sf1蛋白及tnfrsf12a蛋白可以不受癌症种类(肺癌、乳腺癌、乳癌、前列腺癌、肝癌、胰癌)限制地广泛地回收试样中包含的肿瘤细胞。进一步地,对于现有的肿瘤标志物epcam为低表达(+仅为1个)的细胞株(pc-14、mdamb231、pc-3、panc1)而言,tm4sf1和/或tnfrsf12a也高表达(+为4个以上),因此暗示了通过将tm4sf1和/或tnfrsf12a与epcam组合可以高精度地检测及回收试样中包含的肿瘤细胞。
[表9]
实施例8使用抗tm4sf1抗体、抗tnfrsf12a抗体及抗epcam抗体的癌细胞回收
实施例7中已经明确了:对于现有的肿瘤标志物epcam蛋白为低表达的细胞株(pc-14、mdamb231、pc-3、panc1)而言,tm4sf1蛋白(序列号1)及tnfrsf12a蛋白(序列号2)也高表达,因此研究了使用分别识别tm4sf1蛋白、tnfrsf12a蛋白及作为现有的肿瘤标志物的epcam蛋白的抗体能否回收试样中包含的癌细胞。需要说明的是,在本实施例中,作为癌细胞,使用了低表达epcam蛋白的人胰腺癌细胞株panc1。另外,以下的步骤中使用的“缓冲液”为包含0.5%bsa和2mmedta的d-pbs(-)(杜尔贝科磷酸缓冲生理盐水,不含mg2+和ca2+)。
(1)向健康人血液1ml中掺加人胰腺癌细胞(panc1)100μl(100个细胞)。
(2)向(1)的掺加样品中加入1×bdpharmlyse(bd)10ml,在室温下孵育10分钟后,用缓冲液洗涤细胞,进行悬浮。
(3)向细胞悬浮液100μl中加入fcr封闭剂(miltenyibiotec)50μl,在室温下进行10分钟的封闭处理。
(4)封闭处理后,加入抗tm4sf1抗体50μl(pe-抗人tm4sf1抗体、r&dsystems)、抗tnfrsf12a抗体2.5μl(pe-抗人tnfrsf12a抗体、biolegend)及抗epcam抗体10μl(pe-抗人epcam抗体、miltenyibiotec)中的任意一种或全部3种,在冷藏下孵育10分钟后,用缓冲液洗涤细胞,进行悬浮。
(5)向细胞悬浮液80μl中加入anti-pemicrobeadsultrapure(miltenyibiotec)20μl,在冷蔵下孵育15分钟后,用缓冲液洗涤细胞,进行悬浮。
(6)将ms色谱柱(miltenyibiotec)设置于macsseparator(miltenyibiotec),用缓冲液洗涤。
(7)将细胞悬浮液500μl添加到(6)的色谱柱中,用缓冲液500μl洗涤色谱柱3次。
(8)将洗涤后的色谱柱从macsseparator中拆下后,向色谱柱中添加缓冲液1ml,将柱塞压入色谱柱中而回收细胞。
(9)将回收的细胞接种到载玻片上,在显微镜下进行细胞数的测定。
将使用抗tm4sf1抗体、抗tnfrsf12a抗体及抗epcam抗体中的任一者或全部时的癌细胞的回收结果示于表10。需要说明的是,上述表中,示出回收细胞数相对于掺加的细胞数的比例来作为细胞回收率[%]。结果为:单独使用抗tm4sf1抗体或抗tnfrsf12a抗体的情况下,显示出高于单独使用抗epcam抗体时的细胞回收率。进一步地,在同时使用抗tm4sf1抗体、抗tnfrsf12a抗体及抗epcam抗体这3种时,与分别单独使用时相比显示出更高的细胞回收率。由该结果可知,tm4sf1蛋白及tnfrsf12a蛋白可以从血液试样中高效率地回收在用现有标志物epcam蛋白的情况下回收率低的癌细胞。另外可知,通过将tm4sf1蛋白及tnfrsf12a蛋白与epcam蛋白组合,可以更高效率地回收用epcam蛋白时回收率低的癌细胞。
[表10]
实施例9使用抗sdc1抗体的癌细胞染色
实施例1中已经明确了:与健康人白细胞相比,sdc1基因在癌细胞中高表达,因此研究了能否使用识别sdc1的抗体将sdc1用于癌细胞的检测。需要说明的是,在本实施例中,作为癌细胞,使用了实施例1所用的癌细胞株(10株)。
(1)向各癌细胞株悬浮液(1×104个/100μl)100μl中加入fcr封闭剂(miltenyibiotec)10μl,在室温下进行10分钟的封闭处理。
(2)封闭处理后,加入抗sdc1抗体10μl(pe-抗人sdc1抗体、miltenyibiotec),在室温下孵育60分钟。
(3)孵育后,用包含1%bsa、5mmedta和6μg/ml替罗非班的hbss(hank’s平衡盐溶液)洗涤细胞2次,接种到载玻片上,进行荧光显微镜观察。
将使用抗sdc1抗体的癌细胞染色结果示于表11。需要说明的是,在上述表中,用源于各荧光标记抗体的荧光信号的强度(+至+++++的5级)表示染色结果,该信号的强度越大(+的数越多)越表示高表达(信号强度(表达量):+<++<+++<++++<+++++)。结果是,sdc1蛋白(序列号3)不受癌症种类限制地在肺腺癌细胞、乳腺癌细胞、乳癌细胞、前列腺癌细胞、肝癌细胞、胰腺癌细胞中表达,可知可以广泛地检测试样中包含的肿瘤细胞。另外,该结果表明,sdc1蛋白可以不受癌症种类(肺癌、乳腺癌、乳癌、前列腺癌、肝癌、胰癌)限制地广泛地回收试样中包含的肿瘤细胞。进一步地,对于现有的肿瘤标志物epcam为低表达(+仅为1个,参照表9)的细胞株(pc-14、mdamb231、pc-3)而言,sdc1也高表达(+为4个以上),因此暗示了通过将sdc1和epcam组合可以高精度地检测及回收试样中包含的肿瘤细胞。
[表11]
实施例10使用抗sdc1抗体的癌细胞和白细胞的染色
实施例1中已经明确了:与健康人白细胞相比,sdc1基因在乳癌或乳腺癌细胞、及胰腺癌细胞中高表达,因此研究了sdc1能否用于血液试样中包含的乳癌细胞及胰腺癌细胞的特异性检测。
(1)将使用比重差分离法从健康人血液中除去红细胞而成的试样作为本实施例中使用的血液试样,向该试样中分别掺加人乳癌细胞(skbr3)或人胰腺癌细胞(aspc-1)。
(2)向掺加了癌细胞株的血液试样100μl中加入fcr封闭剂(miltenyibiotec)10μl,在室温下进行10分钟的封闭处理。
(3)封闭处理后,加入针对sdc1的抗体(pe-抗人sdc1抗体、miltenyibiotec)溶液10μl及用于检测白细胞的针对cd45的抗体(cd45-fitc、biolegend)溶液10μl,在室温下孵育40分钟。
(4)孵育后,用包含1%bsa、5mmedta和6μg/ml替罗非班的hbss(hank’s平衡盐溶液)洗涤细胞2次,接种到载玻片上,进行荧光显微镜观察。
分别地,将人乳癌细胞skbr3的染色结果示于图12,将人胰腺癌细胞aspc-1的染色结果示于图13。图中,箭头所示的细胞为掺加的乳癌细胞及胰腺癌细胞。图12及图13的结果均为:被针对sdc1的抗体(抗sdc1抗体)染色的细胞(图12的(a)及图13的(a)中箭头所示的细胞)未被针对cd45的抗体染色(图12的(b)及图13的(b))。由该结果可知:sdc1(序列号3)可以作为将血液试样中包含的乳癌细胞及胰腺癌细胞与该试样中包含的白细胞相区分、并检测血液试样中包含的乳癌细胞及胰腺癌细胞的肿瘤标志物使用。
实施例11使用抗tm4sf1抗体、抗tnfrsf12a抗体、抗sdc1抗体及抗epcam抗体的癌细胞回收
实施例7中已经明确了:对于低表达现有的肿瘤标志物epcam蛋白的细胞株(pc-14、mdamb231、pc-3)而言,tm4sf1蛋白(序列号1)及tnfrsf12a蛋白(序列号2)高表达,实施例9中已经明确了:对于低表达现有的肿瘤标志物epcam蛋白的细胞株(pc-14、mdamb231、pc-3)而言,sdc1蛋白(序列号3)高表达,因此研究了使用分别识别tm4sf1蛋白、tnfrsf12a蛋白、sdc1蛋白及作为现有的肿瘤标志物的epcam蛋白的抗体能否回收试样中包含的癌细胞。需要说明的是,在本实施例中,作为癌细胞,使用了低表达epcam蛋白的人前列腺癌细胞株pc-3。另外,以下的步骤中使用的“缓冲液”为包含0.5%bsa和2mmedta的d-pbs(-)(杜尔贝科磷酸缓冲生理盐水,不含mg2+和ca2+)。
(1)向健康人血液1ml中掺加人前列腺癌细胞株(pc-3)100μl(100细胞)。
(2)向(1)的掺加样品中加入1×bdpharmlyse(bd)10ml,在室温下孵育10分钟后,用缓冲液洗涤细胞,进行悬浮。
(3)向细胞悬浮液100μl中加入fcr封闭剂(miltenyibiotec)50μl,在室温下进行10分钟的封闭处理。
(4)封闭处理后,加入抗tm4sf1抗体50μl(pe-抗人tm4sf1抗体、r&dsystems)、抗tnfrsf12a抗体2.5μl(pe-抗人tnfrsf12a抗体、biolegend)、抗sdc1抗体5μl(pe-抗人sdc1抗体、biolegend)及抗epcam抗体10μl(pe-抗人epcam抗体、miltenyibiotec)中的任意一种、或除抗epcam抗体之外的3种、或全部4种,在冷蔵下孵育10分钟后,用缓冲液洗涤细胞,进行悬浮。
(5)向细胞悬浮液80μl中加入anti-pemicrobeadsultrapure(miltenyibiotec)20μl,在冷蔵下孵育15分钟后,用缓冲液洗涤细胞,进行悬浮。
(6)将ms色谱柱(miltenyibiotec)设置于macsseparator(miltenyibiotec),用缓冲液洗涤。
(7)向(6)的色谱柱中添加细胞悬浮液500μl,用缓冲液500μl将色谱柱洗涤3次。
(8)将洗涤后的色谱柱从macsseparator拆下后,向色谱柱添加缓冲液1ml,将柱塞压入色谱柱而回收细胞。
(9)将回收的细胞接种到载玻片上,在显微镜下进行细胞数的测定。
将使用抗tm4sf1抗体、抗tnfrsf12a抗体、抗sdc1抗体及抗epcam抗体中的任一者、以及使用除抗epcam抗体以外的3种或使用全部4种时的癌细胞回收结果示于表12。需要说明的是,上述表中,示出回收细胞数相对于掺加的细胞数的比例作为细胞回收率[%]。结果是:单独使用抗tm4sf1抗体、抗tnfrsf12a抗体或抗sdc1抗体时,显示出高于单独使用抗epcam抗体时的细胞回收率。另外,使用除抗epcam抗体以外的3种或全部4种时,与分别单独使用时相比,显示出更高的细胞回收率。进一步地,使用全部4种时,与使用除抗epcam抗体以外的3种时相比,显示出更高的细胞回收率。由该结果可知,tm4sf1蛋白、tnfrsf12a蛋白及sdc1蛋白可以从血液试样中高效率地回收用现有标志物epcam蛋白时回收率低的癌细胞。另外可知,通过将tm4sf1蛋白、tnfrsf12a蛋白及sdc1蛋白与epcam蛋白组合,可以更高效率地回收用epcam蛋白时回收率低的癌细胞。
[表12]
虽然详细地、另外参照特定的实施方式说明了本发明,但是可以在不脱离本发明的本质和范围的条件下加以各种变更、改进,这对于本领域技术人员而言是显而易见的。
需要说明的是,将2018年3月9日提出的日本专利申请2018-042710号、2018年6月1日提出的日本专利申请2018-105966号、及2018年10月12日提出的日本专利申请2018-193273号的说明书、序列表、权利要求书、附图及摘要的全部内容援引于此,并且作为本发明的说明书的公开而引入。
序列表
<110>东曹株式会社(tosohcorporation)
<120>肿瘤标志物、以及将肿瘤细胞与夹杂细胞相区分来回收及检测的方法
<130>217-0238wo
<150>jp2018-042710
<151>2018-03-09
<150>jp2018-105966
<151>2018-06-01
<150>jp2018-193273
<151>2018-10-12
<160>12
<170>patentinversion3.5
<210>1
<211>202
<212>prt
<213>智人(homosapiens)
<300>
<308>genbank/np_055035.1
<309>2016-10-07
<313>(1)..(202)
<400>1
metcystyrglylyscysalaargcysileglyhisserleuvalgly
151015
leualaleuleucysilealaalaasnileleuleutyrpheproasn
202530
glygluthrlystyralasergluasnhisleuserargphevaltrp
354045
phepheserglyilevalglyglyglyleuleumetleuleuproala
505560
phevalpheileglyleugluglnaspaspcyscysglycyscysgly
65707580
hisgluasncysglylysargcysalametleuserservalleuala
859095
alaleuileglyilealaglyserglytyrcysvalilevalalaala
100105110
leuglyleualagluglyproleucysleuaspserleuglyglntrp
115120125
asntyrthrphealaserthrgluglyglntyrleuleuaspthrser
130135140
thrtrpserglucysthrgluprolyshisilevalglutrpasnval
145150155160
serleupheserileleuleualaleuglyglyileglupheileleu
165170175
cysleuileglnvalileasnglyvalleuglyglyilecysglyphe
180185190
cyscysserhisglnglnglntyraspcys
195200
<210>2
<211>129
<212>prt
<213>智人(homosapiens)
<300>
<308>genbank/np_057723.1
<309>2016-09-09
<313>(1)..(129)
<400>2
metalaargglyserleuargargleuleuargleuleuvalleugly
151015
leutrpleualaleuleuargservalalaglygluglnalaprogly
202530
thralaprocysserargglysersertrpseralaaspleuasplys
354045
cysmetaspcysalasercysargalaargprohisseraspphecys
505560
leuglycysalaalaalaproproalapropheargleuleutrppro
65707580
ileleuglyglyalaleuserleuthrphevalleuglyleuleuser
859095
glypheleuvaltrpargargcysargargargglulysphethrthr
100105110
proileglugluthrglyglygluglycysproalavalalaleuile
115120125
gln
<210>3
<211>310
<212>prt
<213>智人(homosapiens)
<300>
<308>genbank/np_002988.3
<309>2016-09-09
<313>(1)..(310)
<400>3
metargargalaalaleutrpleutrpleucysalaleualaleuser
151015
leuglnproalaleuproglnilevalalathrasnleuproproglu
202530
aspglnaspglyserglyaspaspseraspasnpheserglysergly
354045
alaglyalaleuglnaspilethrleuserglnglnthrproserthr
505560
trplysaspthrglnleuleuthralaileprothrserproglupro
65707580
thrglyleuglualathralaalaserthrserthrleuproalagly
859095
gluglyprolysgluglyglualavalvalleuprogluvalglupro
100105110
glyleuthralaarggluglnglualathrproargproarggluthr
115120125
thrglnleuprothrthrhisglnalaserthrthrthralathrthr
130135140
alaglngluproalathrserhisprohisargaspmetglnprogly
145150155160
hishisgluthrserthrproalaglyproserglnalaaspleuhis
165170175
thrprohisthrgluaspglyglyproseralathrgluargalaala
180185190
gluaspglyalaserserglnleuproalaalagluglyserglyglu
195200205
glnaspphethrphegluthrserglygluasnthralavalvalala
210215220
valgluproaspargargasnglnserprovalaspglnglyalathr
225230235240
glyalaserglnglyleuleuasparglysgluvalleuglyglyval
245250255
ilealaglyglyleuvalglyleuilephealavalcysleuvalgly
260265270
phemetleutyrargmetlyslyslysaspgluglysertyrserleu
275280285
glugluprolysglnalaasnglyglyalatyrglnlysprothrlys
290295300
glnglugluphetyrala
305310
<210>4
<211>295
<212>prt
<213>智人(homosapiens)
<300>
<308>genbank/np_001984.1
<309>2016-10-07
<313>(1)..(295)
<400>4
metgluthrproalatrpproargvalproargprogluthralaval
151015
alaargthrleuleuleuglytrpvalphealaglnvalalaglyala
202530
serglythrthrasnthrvalalaalatyrasnleuthrtrplysser
354045
thrasnphelysthrileleuglutrpgluprolysprovalasngln
505560
valtyrthrvalglnileserthrlysserglyasptrplysserlys
65707580
cysphetyrthrthraspthrglucysaspleuthraspgluileval
859095
lysaspvallysglnthrtyrleualaargvalphesertyrproala
100105110
glyasnvalgluserthrglyseralaglygluproleutyrgluasn
115120125
serprogluphethrprotyrleugluthrasnleuglyglnprothr
130135140
ileglnserphegluglnvalglythrlysvalasnvalthrvalglu
145150155160
aspgluargthrleuvalargargasnasnthrpheleuserleuarg
165170175
aspvalpheglylysaspleuiletyrthrleutyrtyrtrplysser
180185190
serserserglylyslysthralalysthrasnthrasnglupheleu
195200205
ileaspvalasplysglygluasntyrcyspheservalglnalaval
210215220
ileproserargthrvalasnarglysserthraspserprovalglu
225230235240
cysmetglyglnglulysglygluphearggluilephetyrileile
245250255
glyalavalvalphevalvalileileleuvalileileleualaile
260265270
serleuhislyscysarglysalaglyvalglyglnsertrplysglu
275280285
asnserproleuasnvalser
290295
<210>5
<211>976
<212>prt
<213>智人(homosapiens)
<300>
<308>genbank/np_004422.2
<309>2016-10-09
<313>(1)..(976)
<400>5
metgluleuglnalaalaargalacysphealaleuleutrpglycys
151015
alaleualaalaalaalaalaalaglnglylysgluvalvalleuleu
202530
aspphealaalaalaglyglygluleuglytrpleuthrhisprotyr
354045
glylysglytrpaspleumetglnasnilemetasnaspmetproile
505560
tyrmettyrservalcysasnvalmetserglyaspglnaspasntrp
65707580
leuargthrasntrpvaltyrargglyglualagluargilepheile
859095
gluleulysphethrvalargaspcysasnserpheproglyglyala
100105110
sersercyslysgluthrpheasnleutyrtyralagluseraspleu
115120125
asptyrglythrasnpheglnlysargleuphethrlysileaspthr
130135140
ilealaproaspgluilethrvalserserasppheglualaarghis
145150155160
vallysleuasnvalglugluargservalglyproleuthrarglys
165170175
glyphetyrleualapheglnaspileglyalacysvalalaleuleu
180185190
servalargvaltyrtyrlyslyscysprogluleuleuglnglyleu
195200205
alahispheprogluthrilealaglyseraspalaproserleuala
210215220
thrvalalaglythrcysvalasphisalavalvalproproglygly
225230235240
glugluproargmethiscysalavalaspglyglutrpleuvalpro
245250255
ileglyglncysleucysglnalaglytyrglulysvalgluaspala
260265270
cysglnalacysserproglyphephelyspheglualasergluser
275280285
procysleuglucysprogluhisthrleuproserprogluglyala
290295300
thrsercysglucysglugluglyphepheargalaproglnasppro
305310315320
alasermetprocysthrargproproseralaprohistyrleuthr
325330335
alavalglymetglyalalysvalgluleuargtrpthrproprogln
340345350
aspserglyglyarggluaspilevaltyrservalthrcysglugln
355360365
cystrpprogluserglyglucysglyprocysglualaservalarg
370375380
tyrsergluproprohisglyleuthrargthrservalthrvalser
385390395400
aspleugluprohismetasntyrthrphethrvalglualaargasn
405410415
glyvalserglyleuvalthrserargserpheargthralaserval
420425430
serileasnglnthrgluproprolysvalargleugluglyargser
435440445
thrthrserleuservalsertrpserileproproproglnglnser
450455460
argvaltrplystyrgluvalthrtyrarglyslysglyaspserasn
465470475480
sertyrasnvalargargthrgluglypheservalthrleuaspasp
485490495
leualaproaspthrthrtyrleuvalglnvalglnalaleuthrgln
500505510
gluglyglnglyalaglyserlysvalhisglupheglnthrleuser
515520525
progluglyserglyasnleualavalileglyglyvalalavalgly
530535540
valvalleuleuleuvalleualaglyvalglyphepheilehisarg
545550555560
argarglysasnglnargalaargglnserprogluaspvaltyrphe
565570575
serlyssergluglnleulysproleulysthrtyrvalaspprohis
580585590
thrtyrgluaspproasnglnalavalleulysphethrthrgluile
595600605
hisprosercysvalthrargglnlysvalileglyalaglygluphe
610615620
glygluvaltyrlysglymetleulysthrserserglylyslysglu
625630635640
valprovalalailelysthrleulysalaglytyrthrglulysgln
645650655
argvalasppheleuglyglualaglyilemetglyglnpheserhis
660665670
hisasnileileargleugluglyvalileserlystyrlyspromet
675680685
metileilethrglutyrmetgluasnglyalaleuasplyspheleu
690695700
argglulysaspglyglupheservalleuglnleuvalglymetleu
705710715720
argglyilealaalaglymetlystyrleualaasnmetasntyrval
725730735
hisargaspleualaalaargasnileleuvalasnserasnleuval
740745750
cyslysvalserasppheglyleuserargvalleugluaspasppro
755760765
glualathrtyrthrthrserglyglylysileproileargtrpthr
770775780
alaproglualailesertyrarglysphethrseralaseraspval
785790795800
trpserpheglyilevalmettrpgluvalmetthrtyrglygluarg
805810815
protyrtrpgluleuserasnhisgluvalmetlysalaileasnasp
820825830
glypheargleuprothrprometaspcysproseralailetyrgln
835840845
leumetmetglncystrpglnglngluargalaargargprolysphe
850855860
alaaspilevalserileleuasplysleuileargalaproaspser
865870875880
leulysthrleualaasppheaspproargvalserileargleupro
885890895
serthrserglysergluglyvalpropheargthrvalserglutrp
900905910
leugluserilelysmetglnglntyrthrgluhisphemetalaala
915920925
glytyrthralaileglulysvalvalglnmetthrasnaspaspile
930935940
lysargileglyvalargleuproglyhisglnlysargilealatyr
945950955960
serleuleuglyleulysaspglnvalasnthrvalglyileproile
965970975
<210>6
<211>1181
<212>prt
<213>智人(homosapiens)
<300>
<308>genbank/np_002194.2
<309>2016-02-21
<313>(1)..(1181)
<400>6
metglyprogluargthrglyalaalaproleuproleuleuleuval
151015
leualaleuserglnglyileleuasncyscysleualatyrasnval
202530
glyleuproglualalysilepheserglyprosersergluglnphe
354045
glytyralavalglnglnpheileasnprolysglyasntrpleuleu
505560
valglyserprotrpserglypheprogluasnargmetglyaspval
65707580
tyrlyscysprovalaspleuserthralathrcysglulysleuasn
859095
leuglnthrserthrserileproasnvalthrglumetlysthrasn
100105110
metserleuglyleuileleuthrargasnmetglythrglyglyphe
115120125
leuthrcysglyproleutrpalaglnglncysglyasnglntyrtyr
130135140
thrthrglyvalcysseraspileserproasppheglnleuserala
145150155160
serpheserproalathrglnprocysproserleuileaspvalval
165170175
valvalcysaspgluserasnseriletyrprotrpaspalavallys
180185190
asnpheleuglulysphevalglnglyleuaspileglyprothrlys
195200205
thrglnvalglyleuileglntyralaasnasnproargvalvalphe
210215220
asnleuasnthrtyrlysthrlysgluglumetilevalalathrser
225230235240
glnthrserglntyrglyglyaspleuthrasnthrpheglyalaile
245250255
glntyralaarglystyralatyrseralaalaserglyglyargarg
260265270
seralathrlysvalmetvalvalvalthraspglygluserhisasp
275280285
glysermetleulysalavalileaspglncysasnhisaspasnile
290295300
leuargpheglyilealavalleuglytyrleuasnargasnalaleu
305310315320
aspthrlysasnleuilelysgluilelysalailealaserilepro
325330335
thrgluargtyrphepheasnvalseraspglualaalaleuleuglu
340345350
lysalaglythrleuglygluglnilepheserilegluglythrval
355360365
glnglyglyaspasnpheglnmetglumetserglnvalglypheser
370375380
alaasptyrserserglnasnaspileleumetleuglyalavalgly
385390395400
alapheglytrpserglythrilevalglnlysthrserhisglyhis
405410415
leuilepheprolysglnalapheaspglnileleuglnaspargasn
420425430
hissersertyrleuglytyrservalalaalaileserthrglyglu
435440445
serthrhisphevalalaglyalaproargalaasntyrthrglygln
450455460
ilevalleutyrservalasngluasnglyasnilethrvalilegln
465470475480
alahisargglyaspglnileglysertyrpheglyservalleucys
485490495
servalaspvalasplysaspthrilethraspvalleuleuvalgly
500505510
alapromettyrmetseraspleulyslysglugluglyargvaltyr
515520525
leuphethrilelysgluglyileleuglyglnhisglnpheleuglu
530535540
glyprogluglyilegluasnthrargpheglyseralailealaala
545550555560
leuseraspileasnmetaspglypheasnaspvalilevalglyser
565570575
proleugluasnglnasnserglyalavaltyriletyrasnglyhis
580585590
glnglythrileargthrlystyrserglnlysileleuglyserasp
595600605
glyalapheargserhisleuglntyrpheglyargserleuaspgly
610615620
tyrglyaspleuasnglyaspserilethraspvalserileglyala
625630635640
pheglyglnvalvalglnleutrpserglnserilealaaspvalala
645650655
ileglualaserphethrproglulysilethrleuvalasnlysasn
660665670
alaglnileileleulysleucyspheseralalyspheargprothr
675680685
lysglnasnasnglnvalalailevaltyrasnilethrleuaspala
690695700
aspglypheserserargvalthrserargglyleuphelysgluasn
705710715720
asngluargcysleuglnlysasnmetvalvalasnglnalaglnser
725730735
cysprogluhisileiletyrileglngluproseraspvalvalasn
740745750
serleuaspleuargvalaspileserleugluasnproglythrser
755760765
proalaleuglualatyrsergluthralalysvalpheserilepro
770775780
phehislysaspcysglygluaspglyleucysileseraspleuval
785790795800
leuaspvalargglnileproalaalaglngluglnpropheileval
805810815
serasnglnasnlysargleuthrpheservalthrleulysasnlys
820825830
arggluseralatyrasnthrglyilevalvalaspphesergluasn
835840845
leuphephealaserpheserleuprovalaspglythrgluvalthr
850855860
cysglnvalalaalaserglnlysservalalacysaspvalglytyr
865870875880
proalaleulysarggluglnglnvalthrphethrileasnpheasp
885890895
pheasnleuglnasnleuglnasnglnalaserleuserpheglnala
900905910
leusergluserglnglugluasnlysalaaspasnleuvalasnleu
915920925
lysileproleuleutyraspalagluilehisleuthrargserthr
930935940
asnileasnphetyrgluileserseraspglyasnvalproserile
945950955960
valhisserphegluaspvalglyprolyspheilepheserleulys
965970975
valthrthrglyservalprovalsermetalathrvalileilehis
980985990
ileproglntyrthrlysglulysasnproleumettyrleuthrgly
99510001005
valglnthrasplysalaglyaspilesercysasnalaaspileasn
101010151020
proleulysileglyglnthrserserservalserphelysserglu
1025103010351040
asnphearghisthrlysgluleuasncysargthralasercysser
104510501055
asnvalthrcystrpleulysaspvalhismetlysglyglutyrphe
106010651070
valasnvalthrthrargiletrpasnglythrphealaserserthr
107510801085
pheglnthrvalglnleuthralaalaalagluileasnthrtyrasn
109010951100
progluiletyrvalilegluaspasnthrvalthrileproleumet
1105111011151120
ilemetlysproaspglulysalagluvalprothrglyvalileile
112511301135
glyserileilealaglyileleuleuleuleualaleuvalalaile
114011451150
leutrplysleuglyphephelysarglystyrglulysmetthrlys
115511601165
asnproaspgluileaspgluthrthrgluleuserser
117011751180
<210>7
<211>1712
<212>dna
<213>智人(homosapiens)
<300>
<308>genbank/nm_014220.2
<309>2016-10-07
<313>(1)..(1712)
<400>7
aagggcgggacattccccctgcctcttcgcaccacagccagagcctgccattaggaccaa60
tgaaagcaaagtacctcatcccctcagtgactaagaatcgcagtatttaagaggtagcag120
gaatgggctgagagtggtgtttgctttctccaccagaagggcacactttcatctaatttg180
gggtatcactgagctgaagacaaagagaagggggagaaaacctagcagaccaccatgtgc240
tatgggaagtgtgcacgatgcatcggacattctctggtggggctcgccctcctgtgcatc300
gcggctaatattttgctttactttcccaatggggaaacaaagtatgcctccgaaaaccac360
ctcagccgcttcgtgtggttcttttctggcatcgtaggaggtggcctgctgatgctcctg420
ccagcatttgtcttcattgggctggaacaggatgactgctgtggctgctgtggccatgaa480
aactgtggcaaacgatgtgcgatgctttcttctgtattggctgctctcattggaattgca540
ggatctggctactgtgtcattgtggcagcccttggcttagcagaaggaccactatgtctt600
gattccctcggccagtggaactacacctttgccagcactgagggccagtaccttctggat660
acctccacatggtccgagtgcactgaacccaagcacattgtggaatggaatgtatctctg720
ttttctatcctcttggctcttggtggaattgaattcatcttgtgtcttattcaagtaata780
aatggagtgcttggaggcatatgtggcttttgctgctctcaccaacagcaatatgactgc840
taaaagaaccaacccaggacagagccacaatcttcctctatttcattgtaatttatatat900
ttcacttgtattcatttgtaaaactttgtattagtgtaacatactccccacagtctactt960
ttacaaacgcctgtaaagactggcatcttcacaggatgtcagtgtttaaatttagtaaac1020
ttcttttttgtttgtttatttgtttttgtttttttttaaggaatgaggaaacaaaccacc1080
ctctgggggtaatttacagactgagtgacagtactcagtatatctgagataaactctata1140
atgttttggataaaaataacattccaatcactattgtatatatgtgcatgtattttttaa1200
attaaagatgtctagttgctttttataagaccaagaaggagaaaatccgacaacctggaa1260
agatttttgttttcactgcttgtatgatgtttcccattcatacacctataaatctctaac1320
aagaggccctttgaactgccttgtgttctgtgagaaacaaatatttacttagagtggaag1380
gactgattgagaatgttccaatccaaatgaatgcatcacaacttacaatgctgctcattg1440
ttgtgagtactatgagattcaaatttttctaacatatggaaagccttttgtcctccaaag1500
atgagtactagggatcatgtgtttaaaaaaagaaaggctacgatgactgggcaagaagaa1560
agatgggaaactgaataaagcagttgatcagcatcattggaacatggggacgagtgacgg1620
caggaggaccacgaggaaataccctcaaaactaacttgtttacaacaaaataaagtattc1680
actaccatgttaaaaaaaaaaaaaaaaaaaaa1712
<210>8
<211>1048
<212>dna
<213>智人(homosapiens)
<300>
<308>genbank/nm_016639.2
<309>2016-09-09
<313>(1)..(1048)
<400>8
aaggcgggggcgggggcggggcggcggccgtgggtccctgccggccggcggcgggcgcag60
acagcggcgggcgcaggacgtgcactatggctcggggctcgctgcgccggttgctgcggc120
tcctcgtgctggggctctggctggcgttgctgcgctccgtggccggggagcaagcgccag180
gcaccgccccctgctcccgcggcagctcctggagcgcggacctggacaagtgcatggact240
gcgcgtcttgcagggcgcgaccgcacagcgacttctgcctgggctgcgctgcagcacctc300
ctgcccccttccggctgctttggcccatccttgggggcgctctgagcctgaccttcgtgc360
tggggctgctttctggctttttggtctggagacgatgccgcaggagagagaagttcacca420
cccccatagaggagaccggcggagagggctgcccagctgtggcgctgatccagtgacaat480
gtgccccctgccagccggggctcgcccactcatcattcattcatccattctagagccagt540
ctctgcctcccagacgcggcgggagccaagctcctccaaccacaaggggggtggggggcg600
gtgaatcacctctgaggcctgggcccagggttcaggggaaccttccaaggtgtctggttg660
ccctgcctctggctccagaacagaaagggagcctcacgctggctcacacaaaacagctga720
cactgactaaggaactgcagcatttgcacaggggaggggggtgccctccttcctagaggc780
cctgggggccaggctgacttggggggcagacttgacactaggccccactcactcagatgt840
cctgaaattccaccacgggggtcaccctggggggttagggacctatttttaacactaggg900
ggctggcccactaggagggctggccctaagatacagacccccccaactccccaaagcggg960
gaggagatatttattttggggagagtttggaggggagggagaatttattaataaaagaat1020
ctttaactttaaaaaaaaaaaaaaaaaa1048
<210>9
<211>3309
<212>dna
<213>智人(homosapiens)
<300>
<308>genbank/nm_001006946.1
<309>2016-09-09
<313>(1)..(3309)
<400>9
ttcagcccctctcccgggctgcgcctccgcactccgggcccgggcagaagggggtgcgcc60
tcggccccaccacccagggagcagccgagctgaaaggccgggaaccgcggcttgcgggga120
ccacagctcccgaaagcgacgttcggccaccggaggagcgggagccaagcaggcggagct180
cggcgggagaggtgcgggccgaatccgagccgagcggagaggaatccggcagtagagagc240
ggactccagccggcggaccctgcagccctcgcctgggacagcggcgcgctgggcaggcgc300
ccaagagagcatcgagcagcggaacccgcgaagccggcccgcagccgcgacccgcgcagc360
ctgccgctctcccgccgccggtccgggcagcatgaggcgcgcggcgctctggctctggct420
gtgcgcgctggcgctgagcctgcagccggccctgccgcaaattgtggctactaatttgcc480
ccctgaagatcaagatggctctggggatgactctgacaacttctccggctcaggtgcagg540
tgctttgcaagatatcaccttgtcacagcagaccccctccacttggaaggacacgcagct600
cctgacggctattcccacgtctccagaacccaccggcctggaggctacagctgcctccac660
ctccaccctgccggctggagaggggcccaaggagggagaggctgtagtcctgccagaagt720
ggagcctggcctcaccgcccgggagcaggaggccaccccccgacccagggagaccacaca780
gctcccgaccactcatcaggcctcaacgaccacagccaccacggcccaggagcccgccac840
ctcccacccccacagggacatgcagcctggccaccatgagacctcaacccctgcaggacc900
cagccaagctgaccttcacactccccacacagaggatggaggtccttctgccaccgagag960
ggctgctgaggatggagcctccagtcagctcccagcagcagagggctctggggagcagga1020
cttcacctttgaaacctcgggggagaatacggctgtagtggccgtggagcctgaccgccg1080
gaaccagtccccagtggatcagggggccacgggggcctcacagggcctcctggacaggaa1140
agaggtgctgggaggggtcattgccggaggcctcgtggggctcatctttgctgtgtgcct1200
ggtgggtttcatgctgtaccgcatgaagaagaaggacgaaggcagctactccttggagga1260
gccgaaacaagccaacggcggggcctaccagaagcccaccaaacaggaggaattctatgc1320
ctgacgcgggagccatgcgccccctccgccctgccactcactaggcccccacttgcctct1380
tccttgaagaactgcaggccctggcctcccctgccaccaggccacctccccagcattcca1440
gcccctctggtcgctcctgcccacggagtcgtggggtgtgctgggagctccactctgctt1500
ctctgacttctgcctggagacttagggcaccaggggtttctcgcataggacctttccacc1560
acagccagcacctggcatcgcaccattctgactcggtttctccaaactgaagcagcctct1620
ccccaggtccagctctggaggggagggggatccgactgctttggacctaaatggcctcat1680
gtggctggaagatcctgcgggtggggcttggggctcacacacctgtagcacttactggta1740
ggaccaagcatcttgggggggtggccgctgagtggcaggggacaggagtccactttgttt1800
cgtggggaggtctaatctagatatcgacttgtttttgcacatgtttcctctagttctttg1860
ttcatagcccagtagaccttgttacttctgaggtaagttaagtaagttgattcggtatcc1920
ccccatcttgcttccctaatctatggtcgggagacagcatcagggttaagaagacttttt1980
ttttttttttttaaactaggagaaccaaatctggaagccaaaatgtaggcttagtttgtg2040
tgttgtctcttgagtttgtcgctcatgtgtgcaacagggtatggactatctgtctggtgg2100
ccccgtttctggtggtctgttggcaggctggccagtccaggctgccgtggggccgccgcc2160
tctttcaagcagtcgtgcctgtgtccatgcgctcagggccatgctgaggcctgggccgct2220
gccacgttggagaagcccgtgtgagaagtgaatgctgggactcagccttcagacagagag2280
gactgtagggagggcggcaggggcctggagatcctcctgcagaccacgcccgtcctgcct2340
gtggcgccgtctccaggggctgcttcctcctggaaattgacgaggggtgtcttgggcaga2400
gctggctctgagcgcctccatccaaggccaggttctccgttagctcctgtggccccaccc2460
tgggccctgggctggaatcaggaatattttccaaagagtgatagtcttttgcttttggca2520
aaactctacttaatccaatgggtttttccctgtacagtagattttccaaatgtaataaac2580
tttaatataaagtagtcctgtgaatgccactgccttcgcttcttgcctctgtgctgtgtg2640
tgacgtgaccggacttttctgcaaacaccaacatgttgggaaacttggctcgaatctctg2700
tgccttcgtctttcccatggggagggattctggttccagggtccctctgtgtatttgctt2760
ttttgttttggctgaaattctcctggaggtcggtaggttcagccaaggttttataaggct2820
gatgtcaatttctgtgttgccaagctccaagccccatcttctaaatggcaaaggaaggtg2880
gatggccccagcacagcttgacctgaggctgtggtcacagcggaggtgtggagccgaggc2940
ctaccccgcagacaccttggacatcctcctcccacccggctgcagaggccagaggccccc3000
agcccagggctcctgcacttacttgcttatttgacaacgtttcagcgactccgttggcca3060
ctccgagaggtgggccagtctgtggatcagagatgcaccaccaagccaagggaacctgtg3120
tccggtattcgatactgcgactttctgcctggagtgtatgactgcacatgactcgggggt3180
ggggaaaggggtcggctgaccatgctcatctgctggtccgtgggacggtgcccaagccag3240
aggctgggttcatttgtgtaacgacaataaacggtacttgtcatttcgggcaaaaaaaaa3300
aaaaaaaaa3309
<210>10
<211>2393
<212>dna
<213>智人(homosapiens)
<300>
<308>genbank/nm_001993.4
<309>2016-10-07
<313>(1)..(2393)
<400>10
ggagtcgggaggagcggcgggggcgggcgccgggggcgggcagaggcgcgggagagcgcg60
ccgccggccctttatagcgcgcggggcaccggctccccaagactgcgagctccccgcacc120
ccctcgcactccctctggccggcccagggcgccttcagcccaacctccccagccccacgg180
gcgccacggaacccgctcgatctcgccgccaactggtagacatggagacccctgcctggc240
cccgggtcccgcgccccgagaccgccgtcgctcggacgctcctgctcggctgggtcttcg300
cccaggtggccggcgcttcaggcactacaaatactgtggcagcatataatttaacttgga360
aatcaactaatttcaagacaattttggagtgggaacccaaacccgtcaatcaagtctaca420
ctgttcaaataagcactaagtcaggagattggaaaagcaaatgcttttacacaacagaca480
cagagtgtgacctcaccgacgagattgtgaaggatgtgaagcagacgtacttggcacggg540
tcttctcctacccggcagggaatgtggagagcaccggttctgctggggagcctctgtatg600
agaactccccagagttcacaccttacctggagacaaacctcggacagccaacaattcaga660
gttttgaacaggtgggaacaaaagtgaatgtgaccgtagaagatgaacggactttagtca720
gaaggaacaacactttcctaagcctccgggatgtttttggcaaggacttaatttatacac780
tttattattggaaatcttcaagttcaggaaagaaaacagccaaaacaaacactaatgagt840
ttttgattgatgtggataaaggagaaaactactgtttcagtgttcaagcagtgattccct900
cccgaacagttaaccggaagagtacagacagcccggtagagtgtatgggccaggagaaag960
gggaattcagagaaatattctacatcattggagctgtggtatttgtggtcatcatccttg1020
tcatcatcctggctatatctctacacaagtgtagaaaggcaggagtggggcagagctgga1080
aggagaactccccactgaatgtttcataaaggaagcactgttggagctactgcaaatgct1140
atattgcactgtgaccgagaacttttaagaggatagaatacatggaaacgcaaatgagta1200
tttcggagcatgaagaccctggagttcaaaaaactcttgatatgacctgttattaccatt1260
agcattctggttttgacatcagcattagtcactttgaaatgtaacgaatggtactacaac1320
caattccaagttttaatttttaacaccatggcaccttttgcacataacatgctttagatt1380
atatattccgcactcaaggagtaaccaggtcgtccaagcaaaaacaaatgggaaaatgtc1440
ttaaaaaatcctgggtggacttttgaaaagcttttttttttttttttttttttttgagac1500
ggagtcttgctctgttgcccaggctggagtgcagtagcacgatctcggctcactgcaccc1560
tccgtctctcgggttcaagcaattgtctgcctcagcctcccgagtagctgggattacagg1620
tgcgcactaccacgccaagctaatttttgtattttttagtagagatggggtttcaccatc1680
ttggccaggctggtcttgaattcctgacctcaggtgatccacccaccttggcctcccaaa1740
gtgctagtattatgggcgtgaaccaccatgcccagccgaaaagcttttgaggggctgact1800
tcaatccatgtaggaaagtaaaatggaaggaaattgggtgcatttctaggacttttctaa1860
catatgtctataatatagtgtttaggttcttttttttttcaggaatacatttggaaattc1920
aaaacaattggcaaactttgtattaatgtgttaagtgcaggagacattggtattctgggc1980
accttcctaatatgctttacaatctgcactttaactgacttaagtggcattaaacatttg2040
agagctaactatatttttataagactactatacaaactacagagtttatgatttaaggta2100
cttaaagcttctatggttgacattgtatatataattttttaaaaaggttttctatatggg2160
gattttctatttatgtaggtaatattgttctatttgtatatattgagataatttatttaa2220
tatactttaaataaaggtgactgggaattgttactgttgtacttattctatcttccattt2280
attatttatgtacaatttggtgtttgtattagctctactacagtaaatgactgtaaaatt2340
gtcagtggcttacaacaacgtatctttttcgcttataatacattttggtgact2393
<210>11
<211>3970
<212>dna
<213>智人(homosapiens)
<300>
<308>genbank/nm_004431.3
<309>2015-03-15
<313>(1)..(3970)
<400>11
ggttctcacccaacttccattaaggactcggggcaggaggggcagaagttgcgcgcaggc60
cggcgggcgggagcggacaccgaggccggcgtgcaggcgtgcgggtgtgcgggagccggg120
ctcggggggatcggaccgagagcgagaagcgcggcatggagctccaggcagcccgcgcct180
gcttcgccctgctgtggggctgtgcgctggccgcggccgcggcggcgcagggcaaggaag240
tggtactgctggactttgctgcagctggaggggagctcggctggctcacacacccgtatg300
gcaaagggtgggacctgatgcagaacatcatgaatgacatgccgatctacatgtactccg360
tgtgcaacgtgatgtctggcgaccaggacaactggctccgcaccaactgggtgtaccgag420
gagaggctgagcgtatcttcattgagctcaagtttactgtacgtgactgcaacagcttcc480
ctggtggcgccagctcctgcaaggagactttcaacctctactatgccgagtcggacctgg540
actacggcaccaacttccagaagcgcctgttcaccaagattgacaccattgcgcccgatg600
agatcaccgtcagcagcgacttcgaggcacgccacgtgaagctgaacgtggaggagcgct660
ccgtggggccgctcacccgcaaaggcttctacctggccttccaggatatcggtgcctgtg720
tggcgctgctctccgtccgtgtctactacaagaagtgccccgagctgctgcagggcctgg780
cccacttccctgagaccatcgccggctctgatgcaccttccctggccactgtggccggca840
cctgtgtggaccatgccgtggtgccaccggggggtgaagagccccgtatgcactgtgcag900
tggatggcgagtggctggtgcccattgggcagtgcctgtgccaggcaggctacgagaagg960
tggaggatgcctgccaggcctgctcgcctggattttttaagtttgaggcatctgagagcc1020
cctgcttggagtgccctgagcacacgctgccatcccctgagggtgccacctcctgcgagt1080
gtgaggaaggcttcttccgggcacctcaggacccagcgtcgatgccttgcacacgacccc1140
cctccgccccacactacctcacagccgtgggcatgggtgccaaggtggagctgcgctgga1200
cgccccctcaggacagcgggggccgcgaggacattgtctacagcgtcacctgcgaacagt1260
gctggcccgagtctggggaatgcgggccgtgtgaggccagtgtgcgctactcggagcctc1320
ctcacggactgacccgcaccagtgtgacagtgagcgacctggagccccacatgaactaca1380
ccttcaccgtggaggcccgcaatggcgtctcaggcctggtaaccagccgcagcttccgta1440
ctgccagtgtcagcatcaaccagacagagccccccaaggtgaggctggagggccgcagca1500
ccacctcgcttagcgtctcctggagcatccccccgccgcagcagagccgagtgtggaagt1560
acgaggtcacttaccgcaagaagggagactccaacagctacaatgtgcgccgcaccgagg1620
gtttctccgtgaccctggacgacctggccccagacaccacctacctggtccaggtgcagg1680
cactgacgcaggagggccagggggccggcagcaaggtgcacgaattccagacgctgtccc1740
cggagggatctggcaacttggcggtgattggcggcgtggctgtcggtgtggtcctgcttc1800
tggtgctggcaggagttggcttctttatccaccgcaggaggaagaaccagcgtgcccgcc1860
agtccccggaggacgtttacttctccaagtcagaacaactgaagcccctgaagacatacg1920
tggacccccacacatatgaggaccccaaccaggctgtgttgaagttcactaccgagatcc1980
atccatcctgtgtcactcggcagaaggtgatcggagcaggagagtttggggaggtgtaca2040
agggcatgctgaagacatcctcggggaagaaggaggtgccggtggccatcaagacgctga2100
aagccggctacacagagaagcagcgagtggacttcctcggcgaggccggcatcatgggcc2160
agttcagccaccacaacatcatccgcctagagggcgtcatctccaaatacaagcccatga2220
tgatcatcactgagtacatggagaatggggccctggacaagttccttcgggagaaggatg2280
gcgagttcagcgtgctgcagctggtgggcatgctgcggggcatcgcagctggcatgaagt2340
acctggccaacatgaactatgtgcaccgtgacctggctgcccgcaacatcctcgtcaaca2400
gcaacctggtctgcaaggtgtctgactttggcctgtcccgcgtgctggaggacgaccccg2460
aggccacctacaccaccagtggcggcaagatccccatccgctggaccgccccggaggcca2520
tttcctaccggaagttcacctctgccagcgacgtgtggagctttggcattgtcatgtggg2580
aggtgatgacctatggcgagcggccctactgggagttgtccaaccacgaggtgatgaaag2640
ccatcaatgatggcttccggctccccacacccatggactgcccctccgccatctaccagc2700
tcatgatgcagtgctggcagcaggagcgtgcccgccgccccaagttcgctgacatcgtca2760
gcatcctggacaagctcattcgtgcccctgactccctcaagaccctggctgactttgacc2820
cccgcgtgtctatccggctccccagcacgagcggctcggagggggtgcccttccgcacgg2880
tgtccgagtggctggagtccatcaagatgcagcagtatacggagcacttcatggcggccg2940
gctacactgccatcgagaaggtggtgcagatgaccaacgacgacatcaagaggattgggg3000
tgcggctgcccggccaccagaagcgcatcgcctacagcctgctgggactcaaggaccagg3060
tgaacactgtggggatccccatctgagcctcgacagggcctggagccccatcggccaaga3120
atacttgaagaaacagagtggcctccctgctgtgccatgctgggccactggggactttat3180
ttatttctagttctttcctccccctgcaacttccgctgaggggtctcggatgacaccctg3240
gcctgaactgaggagatgaccagggatgctgggctgggccctctttccctgcgagacgca3300
cacagctgagcacttagcaggcaccgccacgtcccagcatccctggagcaggagccccgc3360
cacagccttcggacagacatatgggatattcccaagccgaccttccctccgccttctccc3420
acatgaggccatctcaggagatggagggcttggcccagcgccaagtaaacagggtacctc3480
aagccccatttcctcacactaagagggcagactgtgaacttgactgggtgagacccaaag3540
cggtccctgtccctctagtgccttctttagaccctcgggccccatcctcatccctgactg3600
gccaaacccttgctttcctgggcctttgcaagatgcttggttgtgttgaggtttttaaat3660
atatattttgtactttgtggagagaatgtgtgtgtgtggcagggggccccgccagggctg3720
gggacagagggtgtcaaacattcgtgagctggggactcagggaccggtgctgcaggagtg3780
tcctgcccatgccccagtcggccccatctctcatccttttggataagtttctattctgtc3840
agtgttaaagattttgttttgttggacatttttttcgaatcttaatttattatttttttt3900
atatttattgttagaaaatgacttatttctgctctggaataaagttgcagatgattcaaa3960
ccgaaaaaaa3970
<210>12
<211>7878
<212>dna
<213>智人(homosapiens)
<300>
<308>genbank/nm_002203.3
<309>2016-02-21
<313>(1)..(7878)
<400>12
ttttccctgctctcaccgggcgggggagagaagccctctggacagcttctagagtgtgca60
ggttctcgtatccctcggccaagggtatcctctgcaaacctctgcaaacccagcgcaact120
acggtcccccggtcagacccaggatggggccagaacggacaggggccgcgccgctgccgc180
tgctgctggtgttagcgctcagtcaaggcattttaaattgttgtttggcctacaatgttg240
gtctcccagaagcaaaaatattttccggtccttcaagtgaacagtttggctatgcagtgc300
agcagtttataaatccaaaaggcaactggttactggttggttcaccctggagtggctttc360
ctgagaaccgaatgggagatgtgtataaatgtcctgttgacctatccactgccacatgtg420
aaaaactaaatttgcaaacttcaacaagcattccaaatgttactgagatgaaaaccaaca480
tgagcctcggcttgatcctcaccaggaacatgggaactggaggttttctcacatgtggtc540
ctctgtgggcacagcaatgtgggaatcagtattacacaacgggtgtgtgttctgacatca600
gtcctgattttcagctctcagccagcttctcacctgcaactcagccctgcccttccctca660
tagatgttgtggttgtgtgtgatgaatcaaatagtatttatccttgggatgcagtaaaga720
attttttggaaaaatttgtacaaggcctggatataggccccacaaagacacaggtggggt780
taattcagtatgccaataatccaagagttgtgtttaacttgaacacatataaaaccaaag840
aagaaatgattgtagcaacatcccagacatcccaatatggtggggacctcacaaacacat900
tcggagcaattcaatatgcaagaaaatatgcttattcagcagcttctggtgggcgacgaa960
gtgctacgaaagtaatggtagttgtaactgacggtgaatcacatgatggttcaatgttga1020
aagctgtgattgatcaatgcaaccatgacaatatactgaggtttggcatagcagttcttg1080
ggtacttaaacagaaacgcccttgatactaaaaatttaataaaagaaataaaagcaatcg1140
ctagtattccaacagaaagatactttttcaatgtgtctgatgaagcagctctactagaaa1200
aggctgggacattaggagaacaaattttcagcattgaaggtactgttcaaggaggagaca1260
actttcagatggaaatgtcacaagtgggattcagtgcagattactcttctcaaaatgata1320
ttctgatgctgggtgcagtgggagcttttggctggagtgggaccattgtccagaagacat1380
ctcatggccatttgatctttcctaaacaagcctttgaccaaattctgcaggacagaaatc1440
acagttcatatttaggttactctgtggctgcaatttctactggagaaagcactcactttg1500
ttgctggtgctcctcgggcaaattataccggccagatagtgctatatagtgtgaatgaga1560
atggcaatatcacggttattcaggctcaccgaggtgaccagattggctcctattttggta1620
gtgtgctgtgttcagttgatgtggataaagacaccattacagacgtgctcttggtaggtg1680
caccaatgtacatgagtgacctaaagaaagaggaaggaagagtctacctgtttactatca1740
aagagggcattttgggtcagcaccaatttcttgaaggccccgagggcattgaaaacactc1800
gatttggttcagcaattgcagctctttcagacatcaacatggatggctttaatgatgtga1860
ttgttggttcaccactagaaaatcagaattctggagctgtatacatttacaatggtcatc1920
agggcactatccgcacaaagtattcccagaaaatcttgggatccgatggagcctttagga1980
gccatctccagtactttgggaggtccttggatggctatggagatttaaatggggattcca2040
tcaccgatgtgtctattggtgcctttggacaagtggttcaactctggtcacaaagtattg2100
ctgatgtagctatagaagcttcattcacaccagaaaaaatcactttggtcaacaagaatg2160
ctcagataattctcaaactctgcttcagtgcaaagttcagacctactaagcaaaacaatc2220
aagtggccattgtatataacatcacacttgatgcagatggattttcatccagagtaacct2280
ccagggggttatttaaagaaaacaatgaaaggtgcctgcagaagaatatggtagtaaatc2340
aagcacagagttgccccgagcacatcatttatatacaggagccctctgatgttgtcaact2400
ctttggatttgcgtgtggacatcagtctggaaaaccctggcactagccctgcccttgaag2460
cctattctgagactgccaaggtcttcagtattcctttccacaaagactgtggtgaggacg2520
gactttgcatttctgatctagtcctagatgtccgacaaataccagctgctcaagaacaac2580
cctttattgtcagcaaccaaaacaaaaggttaacattttcagtaacgctgaaaaataaaa2640
gggaaagtgcatacaacactggaattgttgttgatttttcagaaaacttgttttttgcat2700
cattctccctgccggttgatgggacagaagtaacatgccaggtggctgcatctcagaagt2760
ctgttgcctgcgatgtaggctaccctgctttaaagagagaacaacaggtgacttttacta2820
ttaactttgacttcaatcttcaaaaccttcagaatcaggcgtctctcagtttccaagcct2880
taagtgaaagccaagaagaaaacaaggctgataatttggtcaacctcaaaattcctctcc2940
tgtatgatgctgaaattcacttaacaagatctaccaacataaatttttatgaaatctctt3000
cggatgggaatgttccttcaatcgtgcacagttttgaagatgttggtccaaaattcatct3060
tctccctgaaggtaacaacaggaagtgttccagtaagcatggcaactgtaatcatccaca3120
tccctcagtataccaaagaaaagaacccactgatgtacctaactggggtgcaaacagaca3180
aggctggtgacatcagttgtaatgcagatatcaatccactgaaaataggacaaacatctt3240
cttctgtatctttcaaaagtgaaaatttcaggcacaccaaagaattgaactgcagaactg3300
cttcctgtagtaatgttacctgctggttgaaagacgttcacatgaaaggagaatactttg3360
ttaatgtgactaccagaatttggaacgggactttcgcatcatcaacgttccagacagtac3420
agctaacggcagctgcagaaatcaacacctataaccctgagatatatgtgattgaagata3480
acactgttacgattcccctgatgataatgaaacctgatgagaaagccgaagtaccaacag3540
gagttataataggaagtataattgctggaatccttttgctgttagctctggttgcaattt3600
tatggaagctcggcttcttcaaaagaaaatatgaaaagatgaccaaaaatccagatgaga3660
ttgatgagaccacagagctcagtagctgaaccagcagacctacctgcagtgggaaccggc3720
agcatcccagccagggtttgctgtttgcgtgaatggatttctttttaaatcccatatttt3780
ttttatcatgtcgtaggtaaactaacctggtattttaagagaaaactgcaggtcagtttg3840
gaatgaagaaattgtggggggtgggggaggtgcggggggcaggtagggaaataataggga3900
aaatacctattttatatgatgggggaaaaaaagtaatctttaaactggctggcccagagt3960
ttacattctaatttgcattgtgtcagaaacatgaaatgcttccaagcatgacaactttta4020
aagaaaaatatgatactctcagattttaagggggaaaactgttctctttaaaatatttgt4080
ctttaaacagcaactacagaagtggaagtgcttgatatgtaagtacttccacttgtgtat4140
attttaatgaatattgatgttaacaagaggggaaaacaaaacacaggttttttcaattta4200
tgctgctcatccaaagttgccacagatgatacttccaagtgataattttatttataaact4260
aggtaaaatttgttgttggttccttttagaccacggctgccccttccacaccccatcttg4320
ctctaatgatcaaaacatgcttgaataactgagcttagagtatacctcctatatgtccat4380
ttaagttaggagagggggcgatatagagaataaggcacaaaattttgtttaaaactcaga4440
atataacatgtaaaatcccatctgctagaagcccatcctgtgccagaggaaggaaaagga4500
ggaaatttcctttctcttttaggaggcacaacagttctcttctaggatttgtttggctga4560
ctggcagtaacctagtgaatttctgaaagatgagtaatttctttggcaaccttcctcctc4620
ccttactgaaccactctcccacctcctggtggtaccattattatagaagccctctacagc4680
ctgactttctctccagcggtccaaagttatcccctcctttacccctcatccaaagttccc4740
actccttcaggacagctgctgtgcattagatattaggggggaaagtcatctgtttaattt4800
acacacttgcatgaattactgtatataaactccttaacttcagggagctattttcattta4860
gtgctaaacaagtaagaaaaataagctcgagtgaatttctaaatgttggaatgttatggg4920
atgtaaacaatgtaaagtaagacatctcaggatttcaccagaagttacagatgaggcact4980
ggaagccaccaaattagcaggtgcaccttctgtggctgtcttgtttctgaagtacttaaa5040
cttccacaagagtgaatttgacctaggcaagtttgttcaaaaggtagatcctgagatgat5100
ttggtcagattgggataaggcccagcaatctgcattttaacaagcaccccagtcactagg5160
atgcagatggaccacactttgagaaacaccacccatttctactttttgcaccttattttc5220
tctgttcctgagcccccacattctctaggagaaacttagaggaaaagggcacagacacta5280
catatctaaagctttggacaagtccttgacctctataaacttcagagtcctcattataaa5340
atgggaagactgagctggagttcagcagtgatgcttttagttttaaaagtctatgatctg5400
gacttcctataatacaaatacacaatcctccaagaatttgacttggaaaaaaatgtcaaa5460
ggaaaacaggttatctgcccatgtgcatatggacaaccttgactaccctggcctggcccg5520
tggtggcagtccagggctatctgtactgtttacagaattactttgtagttgacaacacaa5580
aacaaacaaaaaaggcataaaatgccagcggtttatagaaaaaacagcatggtattctcc5640
agttaggtatgccagagtccaattcttttaacagctgtgagaatttgctgcttcattcca5700
acaaaattttatttaaaaaaaaaaaaaaaagactggagaaactagtcattagcttgataa5760
agaatatttaacagctagtggtgctggtgtgtacctgaagctccagctacttgagagact5820
gagacaggaagatcgcttgagcccaggagttcaagtccagcctaagcaacatagcaagac5880
cctgtctcaaaaaaatgactatttaaaaagacaatgtggccaggcacggtggctcacacc5940
tgtaatcccaacactttgggaggctgaggccggtggatcacgaggtcaggagtttgagac6000
tagcctggccaacatggtgaaaccccatctctaataatataaaaattagctgggcgtagt6060
agcaggtgcctgtaatcccagttactcgggaagctgaggcaggagaatcacttgaacccg6120
ggaggcagaggtttcagtgagccgagatcgcgccactgcactccagcctgggtgacaggg6180
caagactctgtctcaaacaaacaaacaaaaaaaaagttagtactgtatatgtaaatacta6240
gcttttcaatgtgctatacaaacaattatagcacatccttccttttactctgtctcacct6300
cctttaggtgagtacttccttaaataagtgctaaacatacatatacggaacttgaaagct6360
ttggttagccttgccttaggtaatcagcctagtttacactgtttccagggagtagttgaa6420
ttactataaaccattagccacttgtctctgcaccatttatcacaccaggacagggtctct6480
caacctgggcgctactgtcatttggggccaggtgattcttccttgcaggggctgtcctgt6540
accttgtaggacagcagccctgtcctagaaggtatgtttagcagcattcctggcctctag6600
ctacccgatgccagagcatgctccccccgcagtcatgacaatcaaaaaatgtctccagac6660
attgtcaaatgcctcctggggggcagtatttctcaagcacttttaagcaaaggtaagtat6720
tcatacaagaaatttagggggaaaaaacattgtttaaataaaagctatgtgttcctattc6780
aacaatatttttgctttaaaagtaagtagagggcataaaagatgtcatattcaaatttcc6840
atttcataaatggtgtacagacaaggtctatagaatgtggtaaaaacttgactgcaacac6900
aaggcttataaaatagtaagatagtaaaatagcttatgaagaaactacagagatttaaaa6960
ttgtgcatgactcatttcagcagcaaaataagaactcctaactgaacagaaatttttcta7020
cctagcaatgttattcttgtaaaatagttacctattaaaactgtgaagagtaaaactaaa7080
gccaatttattatagtcacacaagtgattatactaaaaattattataaaggttataattt7140
tataatgtatttacctgtcctgatatatagctataacccaatatatgaaaatctcaaaaa7200
ttaagacatcatcatacagaaggcaggattccttaaactgagatccctgatccatcttta7260
atatttcaatttgcacacataaaacaatgcccttttgtgtacattcaggcatacccattt7320
taatcaatttgaaaggttaatttaaacctctagaggtgaatgagaaacatgggggaaaag7380
tatgaaataggtgaaaatcttaactatttctttgaactctaaagactgaaactgtagcca7440
ttatgtaaataaagtttcatatgtacctgtttattttggcagattaagtcaaaatatgaa7500
tgtatatattgcataactatgttagaattgtatatattttaaagaaattgtcttggatat7560
tttcctttatacataatagataagtcttttttcaaatgtggtgtttgatgtttttgatta7620
aatgtgttttgcctctttccacaaaaactgtaaaaataaatgcatgtttgtacaaaaagt7680
tgcagaattcatttgatttatgagaaacaaaaattaaattgtagtcaacagttagtagtt7740
tttctcatatccaagtataacaaacagaaaagtttcattattgtaacccacttttttcat7800
accacattattgaatattgttacaattgttttgaaaataaagccattttctttgggcttt7860
tataagttaaaaaaaaaa7878
- 还没有人留言评论。精彩留言会获得点赞!