一种基于菌群拉曼特征快速识别样品类别的方法
1.本发明涉及一种基于菌群拉曼特征快速识别样品类别的方法,属于微生物与生态学应用领域。
背景技术:
2.拉曼光谱(raman spectra),是一种散射光谱。拉曼光谱分析法是基于印度科学家c.v.拉曼(raman)所发现的拉曼散射效应,对与入射光频率不同的散射光谱进行分析以得到分子振动、转动方面信息,并应用于分子结构研究的一种分析方法。一般而言,拉曼光谱是特定分子或材料独有的化学指纹。近年来发现,拉曼光谱可应用于生物领域,应用拉曼光谱对单个细胞分析,单个细胞的拉曼光谱是其胞内组分分子振动模式的叠加,由分别对应于一类化学键的拉曼谱峰组成,反映的是单个细胞内代谢物的组成及相对含量的多维信息。单个细胞样品往往是液状菌悬液或滴于拉曼芯片的菌悬液风干样品类型,然后利用显微镜镜下对菌悬液或菌悬液滴于拉曼芯片的风干样品中单个细胞聚焦,针对单个细胞一个一个进行拉曼光谱采集。该技术已在检测单个细胞“胞内组分”、“底物代谢”等领域均取得了一些重要成果。但该技术较少涉及到微生物群落领域。对于来自同一环境样品往往代表着一个微生物群落,针对此复杂微生物样品的拉曼检测需要成千上万个单细胞采集量,才能充分地反映该样品中微生物群落多样性,采集量越大越能完整的表征样品的细胞多样性信息。但这种拉曼光谱检测单细胞的方法耗时耗力,分析数据复杂,大量单细胞数据建立数学建模鉴定分析微生物群落样品类型不易。
3.微生物以群落形式广泛存在于自然环境中,环境中存在着庞大而复杂的微生物群落,微生物群落中的微生物细胞对环境变化敏感,外界环境变化通常会导致微生物群落的多样性和群落结构的变化。因此环境变化或人为活动引起的微生物群落组成了不同类型的环境样品,评估环境类型的微生物检测尤为重要,常规对微生物分析多采用高通量16srrna和its rrna基因测序得到微生物群落结构,但该方法往往需要较长时间才能得到分析结果,过程复杂,对测试样品要求高,测序成本高,在整体水平、快速表征群落环境样品微生物细胞变化仍然是一大挑战。
4.生物大分子多是处在水溶液环境中,研究它们在水溶液中的结构对于了解微生物的结构与性能的关系非常重要。由于水的红外吸收很强,因此用红外光谱研究生物体系有很大局限性,而水的拉曼散射很弱,此外,水分子的拉曼光谱也非常简单,只有为数不多的几个拉曼峰,对于溶解物质的拉曼峰干扰甚小,拉曼光谱是研究水溶液中的生物样品和化学化合物的理想工具,对于细胞内容物如核酸、蛋白质、脂质、糖类、碳水化合物和微量元素等均可生成特定的拉曼光谱。微生物群落是有大量细胞组成,将细胞内容物释放,对细胞主要成分的结构与功能特性的变化测定上拉曼光谱技术比传统化学方法具有更强的优势。此外,通过拉曼谱图不仅可以定性分析被测微生物群落所含成分的组成,还可以定量检测某些成分的含量变化。
技术实现要素:
5.针对常规分析环境微生物样品的测序成本高、测序速度慢,利用拉曼光谱对环境中微生物单细胞采集信号,收集群落单细胞信息耗时费力等缺点,本发明对环境样品的微生物细胞前处理后进行速度更加快速、信息更加完整的拉曼检测,结合机器学习对环境样品类型快速鉴别。
6.本发明提供了一种基于拉曼光谱的分析环境中微生物群落细胞内代谢以及快速检测鉴定环境样本的方法,其目的在于提供一种快速、可靠、操作简单且能同时获得多种微生物群落特征的方法。其依赖于整个微生物群落细胞内容物的光谱测量,而非微生物群落中单个细胞内容物的组成,以及涉及整合其他基于光测量方法(例如傅里叶红外拉曼光谱)的手段和机器学习方法。该方法可适用于食品、环境地质、海洋的微生物样品分析。
7.本发明通过对来自环境的微生物样品进行前处理,利用外力破坏细胞膜和细胞壁,使细胞内容物释放出来,以服务于后续的拉曼信号采集;相对于无破坏的环境微生物单细胞拉曼光谱采集方案,本发明大大提高针对复杂微生物群落环境样品拉曼光谱的检测时间;同时,本发明显著提高了细胞拉曼图谱的信息量,代表了该样品更加丰富的信息。对细胞破坏后的细胞释放内容物,拉曼光谱对微生物群落样品内容物样品检测表现出空间均匀性,拉曼光谱通过机器学习训练足以识别不同样品,将拉曼光谱技术与细胞粉碎技术结合增加了拉曼光谱技术的实用性。
8.本发明提供了一种用于快速识别样品类别的模型,所述模型是按照下述步骤建立的:
9.(1)标准样品的收集:
10.收集环境样品,将样品离心后收集沉淀,再将沉淀悬浮在无菌水或等渗溶液中,得到悬浮液,将悬浮液离心后,取沉淀,再使用无菌水或等渗溶液重悬,重复至少2次,得到菌悬液;
11.(2)菌悬液的预处理
12.将步骤(1)得到的菌悬液进行细胞破碎,并过滤除去细胞碎片及固体颗粒,得到细胞破碎液;
13.(3)拉曼光谱检测:
14.将步骤(2)得到的细胞破碎液盛于无荧光背景信号的容器中,或直接将步骤(2)得到的细胞破碎液点样于拉曼检测芯片上并风干10~30分钟;使用拉曼平台对无荧光背景信号的容器中的细胞破碎液或拉曼检测芯片上的细胞破碎液进行拉曼光谱的采集,其中,光谱采集条件为:使用532nm激光,扫描光谱范围为500~3750cm-1
,激光强度为1~300mw,采集时间为1~20s/次,累积次数1次,不同类别样品分别采集50~1000个光谱;
15.(4)拉曼光谱数据的处理:
16.将步骤(3)得到的拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理;
17.(5)构建模型:
18.分别使用k最邻近法(knn)机器学习算法、支持向量机(svm)机器学习算法、线性判别降维(lda)机器学习算法、极致梯度提升(xgboost)机器学习算法将步骤(4)得到的不同样品类别的拉曼光谱数据进行机器学习,设置训练数据集和检测数据集,其中,训练数据集
为收集数据的70%,检测数据集为收集数据的30%,分类效果最好作为最佳分类器;
19.所述k最邻近法(knn)机器学习算法的参数为:n_neighbors为样品类别数量,algorithm为auto,其他参数为默认值;
20.所述支持向量机(svm)机器学习算法的参数为:核函数为linear,目标函数的惩罚系数c为1000,其他参数为默认值;
21.所述线性判别降维(lda)机器学习算法的参数为:n_components为样品类别数量,其他参数为默认值;
22.所述致梯度提升(xgboost)机器学习算法的参数为:目标函数为multi:softmax,评价指标为merror,其他参数为默认值。
23.(6)模型的选择
24.使用不同模型k最邻近法(knn)、支持向量机(svm)、线性判别降维(lda)、极致梯度提升(xgboost)模型,训练后对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型,采用最佳模型即可实现对于未知样品的快速鉴别。其中准确率的计算是模型预测正确数量所占总量的比例。
25.在本发明的一种实施方式中,环境样品包括微生物群落的任何类型,不仅限于液体,也包含固体。固体样品也可将直接加入无菌水得到包含固体颗粒的菌悬液。在一些情况下,生物样品包括单一类型微生物细胞。
26.在本发明的一种实施方式中,步骤(1)中所述离心的条件为,离心力可为0~100000
×
g,其不包括0,时间可为0~60min,其不包括0。
27.在本发明的一种实施方式中,步骤(2)中的细胞破碎方式包括但不限于:超声、研磨、反复冻融、高压均质中的一种;破坏程度、时间为测量菌悬液od600不发生变化为止。
28.在本发明的一种实施方式中,拉曼检测芯片为括镀铝拉曼芯片、镀纳米颗粒拉曼芯片、滤纸、棉签、薄膜。
29.在本发明的一种实施方式中,拉曼信号采集的拉曼平台包括自发拉曼系统、激光拉曼系统、受激拉曼系统、傅里叶红外拉曼光谱、便携式拉曼系统、原子力拉曼系统、表面增强拉曼系统和针尖增强拉曼系统中的一种。
30.本发明还提供了一种用于快速识别样品类别的方法,所述方法包括最佳模型的判定和样品的识别:
31.1、最佳模型的判定
32.(1)标准样品的收集:
33.收集要检测的环境样品,将样品离心后收集沉淀,再将沉淀悬浮在无菌水或等渗溶液中,得到悬浮液,将悬浮液离心后,取沉淀,再使用无菌水或等渗溶液重悬,重复至少2次,得到菌悬液;
34.(2)菌悬液的预处理
35.将步骤(1)得到的菌悬液进行细胞破碎,并过滤除去细胞碎片及固体颗粒,得到细胞破碎液;
36.(3)拉曼光谱检测:
37.将步骤(2)得到的细胞破碎液盛于无荧光背景信号的容器中,或直接将步骤(2)得到的细胞破碎液点样于拉曼检测芯片上并风干10~30分钟;使用拉曼平台对无荧光背景信
号的容器中的细胞破碎液或拉曼检测芯片上的细胞破碎液进行拉曼光谱的采集,其中,光谱采集条件为:使用532nm激光,扫描光谱范围为500~3750cm-1
,激光强度为1~300mw,采集时间为1~20s/次,累积次数1次,不同类别样品分别采集50~1000个光谱;
38.(4)拉曼光谱数据的处理:
39.将步骤(3)得到的拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理;
40.(5)构建模型:
41.分别使用k最邻近法(knn)机器学习算法、支持向量机(svm)机器学习算法、线性判别降维(lda)机器学习算法、极致梯度提升(xgboost)机器学习算法,将步骤(4)得到的不同样品类别的拉曼光谱数据进行机器学习,设置训练数据集和检测数据集,其中,训练数据集为收集数据的70%,检测数据集为收集数据的30%;
42.所述k最邻近法(knn)机器学习算法的参数为:n_neighbors为样品类别数量,algorithm为auto,其他参数为默认值;
43.所述支持向量机(svm)机器学习算法的参数为:核函数为linear,目标函数的惩罚系数c为1000,其他参数为默认值;
44.所述线性判别降维(lda)机器学习算法的参数为:n_components为样品类别数量,其他参数为默认值;
45.所述致梯度提升(xgboost)机器学习算法的参数为:目标函数为multi:softmax,评价指标为merror,其他参数为默认值;
46.(6)模型的选择
47.使用不同模型:k最邻近法(knn)、支持向量机(svm)、线性判别降维(lda)、极致梯度提升(xgboost)模型,训练后对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型。其中准确率的计算是模型预测正确数量所占总量的比例。
48.2、样品的识别
49.(1)样品的收集:
50.收集未知环境样品,将样品离心后收集沉淀,再将沉淀悬浮在无菌水或等渗溶液中,得到悬浮液,将悬浮液离心后,取沉淀,再使用无菌水或等渗溶液重悬,重复至少2次,得到菌悬液;
51.(2)菌悬液的预处理
52.将步骤(1)得到的菌悬液进行细胞破碎,并过滤除去细胞碎片及固体颗粒,得到细胞破碎液;
53.(3)拉曼光谱检测:
54.将步骤(2)得到的细胞破碎液盛于无荧光背景信号的容器中,或直接将步骤(2)得到的细胞破碎液点样于拉曼检测芯片上并风干10~30分钟;使用拉曼平台对无荧光背景信号的容器中的细胞破碎液或拉曼检测芯片上的细胞破碎液进行拉曼光谱的采集,其中,光谱采集条件为:使用532nm激光,扫描光谱范围为500~3750cm-1
,激光强度为1~300mw,采集时间为1~20s/次,累积次数1次,不同类别样品分别采集50~1000个光谱;
55.(4)拉曼光谱数据的处理:
56.将步骤(3)得到的拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理;
57.(5)样品的鉴定:
58.将步骤(4)得到输入拉曼光谱数据至所述的最佳分类模型中,输出对不同样品类别预测概率即得分,按照判定的标准可鉴定得到该样品的类型,其中判定标准为:若得分大于或等于0.9,其中得分最高的样品类别鉴定为该样品;若得分低于0.9鉴定为不属于所收集的样品类别,属于其他类型样品。
59.在本发明的一种实施方式中,所述样品包括但不限于盐碱地土壤样品、海洋样品、酒曲样品。
60.在本发明的一种实施方式中,所述拉曼信号采集的拉曼平台所使用的测定条件均为本领域中常规条件。
61.在本发明的一种实施方式中,步骤(3)所述拉曼检测芯片,包括镀铝拉曼芯片、镀纳米颗粒拉曼芯片、滤纸、棉签、薄膜。
62.在本发明的一种实施方式中,步骤(3)所述拉曼信号采集的拉曼平台包括自发拉曼系统、激光拉曼系统、受激拉曼系统、傅里叶红外拉曼光谱、便携式拉曼系统、原子力拉曼系统、表面增强拉曼系统和针尖增强拉曼系统中的一种。
63.有益效果
64.(1)本发明通过对来自环境的微生物细胞样品进行简单处理,相对于微生物群落大量单细胞单点采集的方案,本发明大大减少微生物群落拉曼光谱收集的时间,例如,酒曲样品微生物群落减少了90min,本发明大大提高了细胞拉曼图谱的信息量,代表了该样品微生物群落更加丰富的信息,提高了检测精确度;
65.(2)本发明将每个样品或其部分细胞释放内容物经拉曼显微光谱法以便确定样品类型,利用拉曼光谱反映出来的化学键信息对样品的化学物质、生物物质进行相对定量,并结合机器学习为快速鉴别未知环境样品的类型提供了一种方案。
66.(3)本发明具有简单可行、适用范围广、可扩展性强等优点,易于自动化实施,为来自环境微生物群落样品拉曼信号采集的自动化奠定了基础。
附图说明
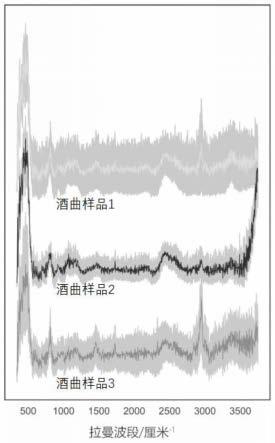
67.图1为本发明不同类型酒曲微生物群落的拉曼图谱。
68.图2为本发明机器学习训练获得的不同类型酒曲菌群的混淆矩阵。
69.图3为本发明中不同小鼠粪便微生物群落的拉曼图谱。
70.图4为本发明中机器学习训练获得的模型检验不同粪便菌群的混淆矩阵。
71.图5为本发明中不同类型土壤微生物群落的拉曼图谱。
72.图6为本发明中机器学习训练获得的模型检验不同类型土壤菌群的混淆矩阵。
73.图7为本发明中不同来源海水微生物群落的拉曼图谱。
74.图8为本发明中机器学习训练获得的模型检验不同类型海水菌群的混淆矩阵。
具体实施方式
75.以下结合具体实施例和附图对本发明作进一步说明。应当理解的是,此处所描述
的具体实施方式仅用于说明和解释本发明,并不用于限制本发明。
76.还需要说明的是,术语“包括”、“包含”或者其任何其他变体意在涵盖非排他性的包含,从而使得包括一系列要素的过程、方法、商品或者设备不仅包括那些要素,而且还包括没有明确列出的其他要素,或者是还包括为这种过程、方法、商品或者设备所固有的要素。在没有更多限制的情况下,由语句“包括一个
……”
限定的要素,并不排除在包括所述要素的过程、方法、商品或者设备中还存在另外的相同要素。
77.下述实施例中所使用的实验方法如无特殊说明,均为常规方法;下述实施例中所用的试剂、材料等,如无特殊说明,均可从商业途径得到。
78.实施例1:用于快速识别样品类别的模型的构建
79.一种用于快速识别样品类别的模型,是按照下述步骤建立的:
80.(1)标准样品的收集:
81.收集环境样品,通过离心收集微生物菌体,得到悬浮液,将悬浮液离心后,取沉淀,再使用无菌水或等渗溶液重悬,重复3次,得到菌悬液;
82.(2)菌悬液的预处理
83.将步骤(1)得到的菌悬液进行细胞破碎,并过滤除去细胞碎片及固体颗粒,得到细胞破碎液;
84.(3)拉曼光谱检测:
85.将步骤(2)得到的细胞破碎液盛于无荧光背景信号的容器中,或直接将步骤(2)得到的细胞破碎液点样于拉曼检测芯片上并风干数分钟;使用拉曼平台对无荧光背景信号的容器中的细胞破碎液或拉曼检测芯片上的细胞破碎液进行拉曼光谱的采集,其中,光谱采集条件为:使用532nm激光,扫描光谱范围为500~3750cm-1
,激光强度为3mw,采集时间为5s/次,累积次数1次,不同类别样品分别采集50个光谱;
86.(4)拉曼光谱数据的处理:
87.将步骤(3)得到的拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理;
88.(5)构建模型:
89.使用机器学习对建立不同样品类别拉曼光谱的神经网络判别分析分类模型:使用机器学习分类器模型包括k最邻近法(knn)、支持向量机(svm)、线性判别降维(lda)、极致梯度提升(xgboost)模型,将步骤(4)得到的不同样品类别的拉曼光谱数据进行机器学习,设置训练数据集和检测数据集,其中,训练数据集为收集数据的70%,检测数据集为收集数据的30%;
90.所述k最邻近法(knn)机器学习算法的参数为:n_neighbors为样品类别数量,algorithm为auto,其他参数为默认值;
91.所述支持向量机(svm)机器学习算法的参数为:核函数为linear,目标函数的惩罚系数c为1000,其他参数为默认值;
92.所述线性判别降维(lda)机器学习算法的参数为:n_components为样品类别数量,其他参数为默认值;
93.所述致梯度提升(xgboost)机器学习算法的参数为:目标函数为multi:softmax,评价指标为merror,其他参数为默认值。
94.(6)模型的选择
95.使用不同模型k最邻近法(knn)、支持向量机(svm)、线性判别降维(lda)、极致梯度提升(xgboost)模型,训练后对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型,采用最佳模型即可实现对于未知样品的快速鉴别,其中准确率的计算是模型预测正确数量所占总量的比例。
96.实施例2:不同类型盐碱地土壤菌群鉴别
97.(1)最佳模型的判定
98.采用实施例1中的不同模型:k最邻近法(knn)、支持向量机(svm)、线性判别降维(lda)、极致梯度提升(xgboost)模型,对采集到的该已知环境中的样品进行训练后,对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型,具体如下:
99.1)取5g盐碱地不同区域样品2个,分别编号为盐碱地1~2,加20ml无菌水充分震荡混匀,静置5min,吸出洗脱液6ml,随后7000g,离心2min。弃上清液,沉淀加6ml无菌水,吹打混匀。7000g,离心2min弃上清,沉淀加6ml无菌水,吹打混匀。使用水反复冲洗并离心6次。
100.2)菌悬液使用高压均质机破碎细胞,工作5s,间隙5s,次数200次,功率300w。样品置于冰中以便于热量散发。
101.3)通过0.22μm滤膜过滤样品。
102.4)取2.5μl步骤3处理后样品滴于拉曼芯片,静置10min风干。使用共聚焦拉曼光谱测定拉曼光谱,参数532nm激光,光栅600g/mm,镜下功率3mw,采集时间5s。每个海水采集50个拉曼光谱。拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理。
103.5)对拉曼光谱数据进行机器学习,使用knn、lda、svm、xgboost机器学习算法,训练数据集(收集数据的70%)和检测数据集(收集数据的30%)。训练后对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型。其中准确率的计算是模型预测正确数量所占总量的比例。
104.结果显示,knn算法的准确率为96.95%,lda算法的准确率为96.95%,svm算法的准确率为91.75%,xgboost算法的准确率为91.15%,因此选择svm作为最优模型分类器svm对盐碱地土壤类别鉴定。
105.(2)采用最佳模型对未知样品的鉴定方法
106.1)采集未知区域盐碱地的土壤,加20ml无菌水充分震荡混匀,静置5min,吸出洗脱液6ml,随后7000g,离心2min。弃上清液,沉淀加6ml无菌水,吹打混匀。7000g,离心2min弃上清,沉淀加6ml无菌水,吹打混匀。使用水反复冲洗并离心6次。
107.2)菌悬液使用高压均质机破碎细胞,工作5s,间隙5s,次数200次,功率300w。样品置于冰中以便于热量散发。
108.3)通过0.22μm滤膜过滤样品。
109.4)取2.5μl步骤3)处理后样品滴于拉曼芯片,静置10min风干。使用共聚焦拉曼光谱测定拉曼光谱,参数532nm激光,光栅600g/mm,镜下功率3mw,采集时间5s。每个样品采集50个拉曼光谱。拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理。
110.5)对未知区域盐碱地土壤的拉曼数据输入到svm机器学习算法(参数同实施例1),输出得到不同类别盐碱地土壤的预测概率得分,其中盐碱地1的概率得分是0.93,盐碱地2的概率得分是0.41,按照判定标准盐碱地1的得分大于0.9,可输出得到该样品类别为盐碱地1。
111.对比例1:
112.具体步骤如下:
113.(1)采集未知区域盐碱地的土壤,加20ml无菌水充分震荡混匀,静置5min,吸出洗脱液6ml,随后7000g,离心2min。弃上清液,沉淀加6ml无菌水,吹打混匀。7000g,离心2min弃上清,沉淀加6ml无菌水,吹打混匀。使用水反复冲洗并离心6次,
114.(2)取2.5μl处理后样品滴于拉曼芯片,静置10min风干,使用共聚焦拉曼光谱测定芯片上单细胞中心处拉曼光谱,参数532nm激光,光栅600g/mm,镜下功率3mw,采集时间5s。每个样品采集50个单细胞的拉曼光谱。
115.(3)拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理。对未知区域盐碱地土壤的拉曼数据输入到svm机器学习算法(参数同实施例1)。
116.使用svm训练后对检测数据集进行预测评估无破坏的环境菌群单细胞拉曼光谱采集(对比例)和破坏的环境菌群拉曼光谱采集(实施例)两种方案好坏,根据真实结果和预测结果得到准确率、精确率、召回率、f1因子,准确率、精确率、召回率、f1因子越高,说明该方案采集的光谱越能准确地鉴定样品。其中准确率是指预测正确的结果占总样本的百分比,精确率是指分类正确的正样本个数占分类器判定为正样本的样本个数的比例,召回率是指在实际为正的样本中被预测为正样本的概率,f1因子是精确率和召回率的调和平均值。
117.对比无破坏的环境菌群单细胞拉曼光谱采集方案和破坏的环境菌群拉曼光谱采集方案,结果表明在使用相同采集时间内使用破坏的环境微生物细胞拉曼光谱采集方案的准确率、召回率、f1因子更高。
118.表1:不同方法采集不同盐碱地土壤微生物群落光谱数据svm算法评价比对
[0119][0120]
实施例3:不同酒曲菌群快速鉴别
[0121]
(1)最佳模型的判定
[0122]
采用实施例1中的不同模型:k最邻近法(knn)、支持向量机(svm)、线性判别降维(lda)、极致梯度提升(xgboost)模型,对采集到的该已知环境中的样品进行训练后,对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型,具体如下:
[0123]
1)采集不同厂家洋河、茅台镇、泸州酒曲,分别编号为酒曲1~3,在曲皮到曲心之间的3个不同的位置取样,随后将三个位置的样品充分混匀后作为一个样品进行拉曼检测。取5g大曲固体粉末,加20ml无菌水充分震荡混匀,静置5min,吸出洗脱液6ml,随后7000g,离心2min。弃上清液,沉淀加6ml无菌水,吹打混匀。7000g,离心2min弃上清,沉淀加6ml无菌水,吹打混匀。使用水反复冲洗并离心3次。
[0124]
2)菌悬液使用超声破碎细胞,选用6mm超声探头,超声5s,间隙5s,次数70次(总时间为10min)。功率300w,样品置于冰中以便于热量散发。
[0125]
3)通过0.22μm滤膜过滤步骤2)样品。
[0126]
4)使用内径0.3mm、管长100mm玻璃毛细管,吸取适量步骤3)液体。
[0127]
5)使用拉曼光谱测定找到毛细管平面,测定拉曼光谱,参数532nm激光,光栅600g/mm,镜下功率3mw,采集时间2s。每个酒曲采集50个拉曼光谱。拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理(图1)。
[0128]
6)对三种酒曲拉曼光谱数据进行机器学习,使用knn、lda、svm、xgboost机器学习算法,训练数据集(收集数据的70%)和检测数据集(收集数据的30%)。训练后对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型。其中准确率的计算是模型预测正确数量所占总量的比例。混淆矩阵是机器学习中总结分类模型预测结果的情形分析表,以矩阵形式将数据集中的记录按照真实的类别与分类模型预测的类别判断两个标准进行汇总。其中矩阵的行表示真实值,矩阵的列表示预测值,结果如图2所示,为不同模型对三种酒曲的预测结果百分数。
[0129]
结果显示,knn算法的准确率为96.95%,lda算法的准确率为96.95%,svm算法的准确率为91.75%,xgboost算法的准确率为91.15%,因此选择knn作为最优模型分类器对酒曲类别鉴定(图2)。
[0130]
(2)采用最佳模型对未知样品的鉴定方法
[0131]
1)采集未知厂家的酒曲,在曲皮到曲心之间的3个不同的位置取样,随后将三个位置的样品充分混匀后作为一个样品进行拉曼检测。
[0132]
取5g大曲固体粉末,加20ml无菌水充分震荡混匀,静置5min,吸出洗脱液6ml,随后7000g,离心2min。弃上清液,沉淀加6ml无菌水,吹打混匀。7000g,离心2min弃上清,沉淀加6ml无菌水,吹打混匀。使用水反复冲洗并离心3次。
[0133]
2)菌悬液使用超声破碎细胞,选用6mm超声探头,超声5s,间隙5s,次数70次(总时间为10min)。功率300w,样品置于冰中以便于热量散发。
[0134]
3)通过0.22μm滤膜过滤步骤2)样品。
[0135]
4)使用内径0.3mm、管长100mm玻璃毛细管,吸取适量步骤3)液体。
[0136]
5)使用拉曼光谱测定找到毛细管平面,测定拉曼光谱,参数532nm激光,光栅600g/mm,镜下功率3mw,采集时间2s。酒曲采集50个拉曼光谱。拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理。
[0137]
6)对未知酒曲的拉曼数据输入到knn机器学习算法(参数同实施例1),输出得到不同类别酒曲的预测概率得分,其中酒曲1的概率得分是0.78,酒曲2的概率得分是0.9,酒曲3的概率得分是0.94,按照判定标准酒曲2和酒曲3的得分大于0.9,两者相比酒曲3得分更高,可输出得到该酒曲类别为酒曲3泸州酒曲。
[0138]
实施例4:不同肠道菌群快速鉴别
[0139]
(1)最佳模型的判定
[0140]
采用实施例1中的不同模型:k最邻近法(knn)、支持向量机(svm)、线性判别降维
(lda)、极致梯度提升(xgboost)模型,对采集到的该已知环境中的样品进行训练后,对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型,具体如下:
[0141]
1)乳酸菌、双歧杆菌和小球菌等多种益生菌对小鼠肠道菌群具有调节和对抑郁症状的缓解作用,采集正常小鼠、应激小鼠、应激加益生菌治疗的小鼠组粪便样本,分别编号为肠道菌群1、肠道菌群2、肠道菌群3。
[0142]
分别取5g粪便样品,加20ml无菌水充分震荡混匀,静置5min,吸出洗脱液6ml,随后7000g,离心2min。弃上清液,沉淀加6ml无菌水,吹打混匀。7000g,离心2min弃上清,沉淀加6ml无菌水,吹打混匀。使用水反复冲洗并离心6次。
[0143]
2)菌悬液使用高压均质机破碎细胞,工作5s,间隙5s,次数200次,功率300w。样品置于冰中以便于热量散发。
[0144]
3)通过0.22μm滤膜过滤步骤2)样品。
[0145]
4)使用内径0.3mm、管长100mm玻璃毛细管,吸取步骤3)适量液体。
[0146]
5)使用受激拉曼光谱仪测定找到毛细管平面,测定拉曼光谱,参数532nm激光,光栅600g/mm,镜下功率3mw,采集时间5s。每个样品采集50个拉曼光谱。拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理(图3)。
[0147]
6)对拉曼光谱数据进行机器学习,使用knn、lda、svm、xgboost机器学习算法,训练数据集(收集数据的70%)和检测数据集(收集数据的30%)。训练后对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型。其中准确率的计算是模型预测正确数量所占总量的比例。混淆矩阵是机器学习中总结分类模型预测结果的情形分析表,以矩阵形式将数据集中的记录按照真实的类别与分类模型预测的类别判断两个标准进行汇总。其中矩阵的行表示真实值,矩阵的列表示预测值,结果如图4所示,为为不同模型对三种肠道菌群的预测结果百分数。
[0148]
结果显示,knn算法的准确率为90.95%,lda算法的准确率为89.15%,svm算法的准确率为91.5%,xgboost算法的准确率为97.15%,因此选择xgboost作为最优模型分类器对不同肠道样品鉴定。
[0149]
(2)采用最佳模型对未知样品的鉴定方法
[0150]
1)采集未知小鼠粪便样本,取5g粪便样本,加20ml无菌水充分震荡混匀,静置5min,吸出洗脱液6ml,随后7000g,离心2min。弃上清液,沉淀加6ml无菌水,吹打混匀。7000g,离心2min弃上清,沉淀加6ml无菌水,吹打混匀。使用水反复冲洗并离心3次。
[0151]
2)菌悬液使用高压均质机破碎细胞,工作5s,间隙5s,次数200次,功率300w。样品置于冰中以便于热量散发。
[0152]
3)通过0.22μm滤膜过滤步骤2)样品。
[0153]
4)使用内径0.3mm、管长100mm玻璃毛细管,吸取步骤3)适量液体。
[0154]
5)使用受激拉曼光谱仪测定找到毛细管平面,测定拉曼光谱,参数532nm激光,光栅600g/mm,镜下功率3mw,采集时间5s。样品采集50个拉曼光谱。拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理。
[0155]
6)对未知小鼠粪便样本的拉曼数据输入到xgboost机器学习算法,其中肠道菌群1的概率得分是0.78,肠道菌群2的概率得分是0.9,肠道菌群3的概率得分是0.94,按照判定标准肠道菌群2和肠道菌群3的得分大于0.9,两者相比肠道菌群3的得分更高,可输出得到该肠道菌群类别为肠道菌群3。
[0156]
实施例5:不同类型土壤菌群鉴别
[0157]
1)采集汉江上游、中游、下游的三份土壤样品,分别为汉江土壤1~3。
[0158]
取5g固体粉末,加20ml无菌水充分震荡混匀,静置5min,吸出洗脱液6ml,随后7000g,离心2min。弃上清液,沉淀加6ml无菌水,吹打混匀。7000g,离心2min弃上清,沉淀加6ml无菌水,吹打混匀。使用水反复冲洗并离心3次。
[0159]
2)菌悬液使用超声破碎细胞,选用6mm超声探头,超声5s,间隙5s,次数70次(总时间为10min)。功率300w。样品置于冰中以便于热量散发。
[0160]
3)通过0.22μm滤膜过滤步骤2)样品。
[0161]
4)取2.5μl步骤3)处理后样品滴于镀纳米金膜定性滤纸,静置10min风干。
[0162]
5)使用共聚焦拉曼光谱仪测定找到毛细管平面,测定拉曼光谱,参数785nm激光,光栅600g/mm,镜下功率3mw,采集时间5s。每个土壤样品采集50个拉曼光谱。拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理(图5)。
[0163]
6)对拉曼光谱数据进行机器学习,使用knn、lda、svm、xgboost机器学习算法,训练数据集(收集数据的70%)和检测数据集(收集数据的30%)。训练后对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型。其中准确率的计算是模型预测正确数量所占总量的比例。混淆矩阵是机器学习中总结分类模型预测结果的情形分析表,以矩阵形式将数据集中的记录按照真实的类别与分类模型预测的类别判断两个标准进行汇总。其中矩阵的行表示真实值,矩阵的列表示预测值,结果如图6所示,为不同模型对三种土壤样品的预测结果百分数。
[0164]
结果显示,knn算法的准确率为80.55%,lda算法的准确率为89.56%,svm算法的准确率为98.52%,xgboost算法的准确率为79.35%,因此选择svm作为最优模型分类器对不同土壤样品鉴定。
[0165]
(2)采用最佳模型对未知样品的鉴定方法
[0166]
1)采集未知汉江上游、中游、下游的土壤样品。
[0167]
分别取5g固体粉末,加20ml无菌水充分震荡混匀,静置5min,吸出洗脱液6ml,随后7000g,离心2min。弃上清液,沉淀加6ml无菌水,吹打混匀。7000g,离心2min弃上清,沉淀加6ml无菌水,吹打混匀。使用水反复冲洗并离心3次。
[0168]
2)菌悬液使用超声破碎细胞,选用6mm超声探头,超声5s,间隙5s,次数70次(总时间为10min)。功率300w。样品置于冰中以便于热量散发。
[0169]
3)通过0.22μm滤膜过滤步骤2)样品。
[0170]
4)取2.5μl步骤3)处理后样品滴于镀纳米金膜定性滤纸,静置10min风干。
[0171]
5)使用共聚焦拉曼光谱仪测定找到毛细管平面,测定拉曼光谱,参数785nm激光,光栅600g/mm,镜下功率3mw,采集时间5s。样品采集50个拉曼光谱。拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据
进行归一化处理。
[0172]
6)对未知土壤样品的拉曼数据输入到svm机器学习算法,输出得到不同类别土壤的预测概率得分,其中土壤1的概率得分是0.95,土壤2的概率得分是0.71,土壤3的概率得分是0.44,按照判定标准土壤1的得分大于0.9,可输出得到该样品类别为汉江上游的土壤样品。
[0173]
实施例6:不同海水菌群快速鉴别
[0174]
1)海洋中存在着大量浮游生物、藻类,人类活动的微塑料等成分。对于不同类型的海水快速识别会开始帮助确定是否存在污染问题。采集不同海水样品3个,所述样品是连云港3个不同区域入海河口上游、中游下游海水样品,分别为编号为海水1~3。每个样品分别为3ml。
[0175]
2)使用反复冻融方法释放样品中微生物细胞内容物,将液体放在低温下冷冻(约-20℃),然后在室温中融化,反复5次而达到破壁作用。
[0176]
3)通过0.22μm滤膜过滤步骤2)样品。
[0177]
4)取2.5μl步骤3)处理后样品滴于镀金纳米拉曼芯片,静置10min风干。使用共聚焦拉曼光谱测定拉曼光谱,参数532nm激光,光栅600g/mm,镜下功率3mw,采集时间5s。每个海水采集50个拉曼光谱。拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理(图7)。
[0178]
5)对拉曼光谱数据进行机器学习,使用knn、lda、svm、xgboost机器学习算法,训练数据集(收集数据的70%)和检测数据集(收集数据的30%)。训练后对检测数据集进行预测,根据真实结果和预测结果得到准确率,准确率最高的模型即为最佳模型。其中准确率的计算是模型预测正确数量所占总量的比例。混淆矩阵是机器学习中总结分类模型预测结果的情形分析表,以矩阵形式将数据集中的记录按照真实的类别与分类模型预测的类别判断两个标准进行汇总。其中矩阵的行表示真实值,矩阵的列表示预测值,结果如图8所示,为不同模型对三种海水的预测结果百分数。
[0179]
结果显示,knn算法的准确率为90.25%,lda算法的准确率为90.25%,svm算法的准确率为90.25%,xgboost算法的准确率为90.25%,选择xgboost作为模型分类器对不同海水样品鉴定。
[0180]
(2)采用最佳模型对未知样品的鉴定方法
[0181]
1)采集未知连云港区域入海河口海水样品,样品采集为3ml。
[0182]
2)使用反复冻融方法释放样品中微生物细胞内容物,将液体放在低温下冷冻(约-20℃),然后在室温中融化,反复5次而达到破壁作用。
[0183]
3)通过0.22μm滤膜过滤步骤2)样品。
[0184]
4)取2.5μl步骤3)处理后样品滴于镀金纳米拉曼芯片,静置10min风干。使用共聚焦拉曼光谱测定拉曼光谱,参数532nm激光,光栅600g/mm,镜下功率3mw,采集时间5s。每个海水采集50个拉曼光谱。拉曼光谱数据进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、savitzky-golay平滑并对所有的数据进行归一化处理。
[0185]
5)对未知连云港区域入海河口海水样品的拉曼数据输入到xgboost机器学习算法,输出得到不同类别海水的预测概率得分,其中海水1的概率得分是0.98,海水2的概率得分是0.71,海水3的概率得分是0.57,按照判定标准海水1的得分大于0.9,可输出得到该海
水样品为入连云港海河口上游海水1。
[0186]
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1