基于在线顺序极限学习机的样品成分含量测定方法与流程
本发明涉及一种基于在线顺序极限学习机的样品成分含量测定方法,属于样品成分含量测定技术领域。
背景技术:
近红外光谱技术是一种快速无损低成本的间接分析技术,利用红外光谱仪可以快速地测得样本的近红外光谱,再结合化学计量学的方法,建立起样本的近红外光谱与有效组分含量之间的多元标定模型,进而可以对未知样本的响应组分进行预测。然而,在实际使用过程中近红外光谱数据并不是一次产生的,而是流式产生的。如果已经在已有的数据样本上建立了模型,而随着时间的变化可能会有新的数据样本产生,为了提高模型的泛化性能和预测精度,必然需要将新产生的数据和以前的数据一起进行建模。最简单直接的方法就是将现有的所有数据重新运行一遍原来的算法,但是这种方法在数据量很小的时候是可以接受的,而如果数据是以gb为单位衡量的,新到来的数据样本可能会多达数mb,那么这样将原有的数据加之新来的数据一起建立模型,是十分费时耗力的,有时候可能前面新到来的数据还没有处理完成,又有更新的数据到来,显然在这种情况下,完全重新建模是不可能办到的。在线流式算法也越来越适用于具有径向基函数(radialbasisfunction,rbf)节点的前馈神经网络。在处理在线流式学习算法发展过程中出现了许多算法,其中比较典型的算法有gap-pbf算法和ggap-rbf算法。人们希望这些算法能够简化学习流程并提高学习速度,这些算法需要输入样本的分布或者输入样本顺序的信息。但是这些算法的建模速度依旧很慢,且泛化性能也一般。并且这些算法处理新来的数据时只能是一个一个(onebyone)的而不能是一块一块的(chunkbychunk)。
另外,在近红外光谱的测量过程中,由于测量仪器的不同或者测量条件的改变,会导致原有的多元标定模型失去效果,而重新建立模型又是耗时耗力的一件事,甚至有的时候重新建模不具有可行性。更可接受的方式是做标定迁移,用来纠正主仪器和另一个仪器(子仪器)的光谱数据。本质上,就是将子仪器的光谱转换,使之看起来更像主光谱仪的数据,然后就可以使用主光谱仪的模型对之进行处理了。在过去的几年里,不同的标定迁移技术得到发展,常用的标定迁移方法包括:多元散射校正法(简写为msc)、直接标准化方法(简写为ds)、间接标准化方法(简写为pds)、典型相关性分析法(简写为cca)等。但是现有的上述标定迁移方法仍然存在成分含量预测精度和稳定性较差的问题;而且所述的多元散射校正法,需要测定一个待测样本的理想光谱,然后利用该理想光谱对测得的其他光谱进行修正,但是在实际应用中是很难得到所谓的理想光谱的。
技术实现要素:
本发明的目的在于,提供一种基于在线顺序极限学习机的样品成分含量测定方法,它可以有效解决现有技术中存在的问题,尤其是现有的算法建模速度慢,泛化性能一般,而且只能逐个地处理新来的数据而不能逐块地进行处理的问题。
为解决上述技术问题,本发明采用如下的技术方案:一种基于在线顺序极限学习机的样品成分含量测定方法,采集样品的光谱数据样本,并利用在线顺序极限学习机算法进行建模;利用所建立的模型对样品的成分含量进行测定。
前述的基于在线顺序极限学习机的样品成分含量测定方法,具体包括以下步骤:
s1,根据初始主光谱spmaster(0)及相应的样品成分含量y0和隐层节点个数l,计算出隐层到输出层的初始权重矩阵α(0),其中,spmaster(0)和y0包含m0个样本;
s2,当有新的主光谱spmaster(k+1)及相应的样品成分含量yk+1到来时,根据在线顺序极限学习机算法计算出隐层到输出层的权重矩阵α(k+1);其中,第k+1次到来的数据spmaster(k+1)和yk+1包含mk+1个样本;k≥0;
s3,如果还存在新的主光谱spmaster(k+1)和相应的样品成分含量yk+1,则令k=k+1,并转至s2,否则转至s4;
s4,根据以下公式计算获得样品的光谱数据spmasterpre所对应的成分含量预测值pre_y:
pre_y=hpreα(new);
其中,
α(new)为目前最新的隐层到输出层的权重矩阵,spmasterpre包含n个样本;w和b分别为随机生成的正交的输入权重矩阵和偏置;g(w,spmasterpre,b)为激活函数。
优选的,还包括:利用在线顺序极限学习机算法,对主光谱仪及从光谱仪上采集的样品的光谱数据样本进行建模,实现将从光谱仪的光谱数据迁移至主光谱仪的光谱数据空间;然后利用主光谱仪建立的含量预测模型进行样品成分含量的测定。
更优选的,所述的利用在线顺序极限学习机算法,对主光谱仪及从光谱仪上采集的样品的光谱数据样本进行建模,实现将从光谱仪的光谱数据迁移至主光谱仪的光谱数据空间包括以下步骤:
s01,根据初始主光谱spmaster0和从光谱spslave0及隐层节点数l,生成隐层到输出层的权重矩阵β(0),其中spmaster0中包含的样本个数为m0;
s02,当有新的包含mk+1个样本的spmaster(k+1)和spslave(k+1)到来时,根据在线顺序极限学习机算法计算出隐层到输出层的权重矩阵β(k+1);其中,k≥0;
s03,如果还有新的样本的spmaster(k+1)和spslave(k+1)到来,则令k=k+1,并转至s02,否则转至s04;s04,根据以下公式对包含n个样本的测试数据spslavetest进行迁移:
spslavetomaster=h'testβnew
其中,spslavetomaster表示迁移后的光谱数据;βnew表示最新的隐层到输出层间的权重矩阵;
前述的基于在线顺序极限学习机的样品成分含量测定方法中,所述的隐层节点个数l小于等于初始样本个数m0。从而使得基于oselm的样品成分含量预测模型具有更快的计算速度,且对系统资源的耗费更少。
优选的,步骤s1中,
α(0)=(h0th0)-1h0ty0;
其中,
优选的,步骤s2中,
其中,
优选的,步骤s01中,
β(0)=(h'0th'0)-1h'0tspmaster0;
其中,
优选的,步骤s02中,
其中,
本发明中,通过k折交叉验证的方法确定最佳隐层节点数l;所述的激活函数采用sigmoid函数,从而可以提高样品成分含量的预测精度。
本发明所述的样品成分含量测定方法,适用于在线检测中所有种类的光谱样本,尤其是对于药片和玉米的成分含量测定具有更好的效果。
优选的,将从光谱仪的光谱数据迁移至主光谱仪的光谱数据空间作为新的主光谱spmaster(k+1),利用主光谱仪建立的含量预测模型进行样品的成分含量测定后获得相应的样品成分含量yk+1,令k=k+1,并转至s2。从而可以进一步提高模型的预测精度。
与现有技术相比,本发明通过利用在线顺序极限学习机算法进行建模,从而无需保留前面使用过的数据而只将前面学习到的知识保留下来以备后用;每次有新的光谱数据到来时,只需要计算新到数据的隐层输出,然后利用前面学习到的知识动态更新中间隐层到输出层之间的输出权重,即可进行快速建模。本发明相较于传统的建模方法,提高了建模速度,减少了不必要的重复计算量以及对数据存储空间的消耗,提高了模型的精度和泛化性能,而且既可以处理每次一个一个到来的数据也可以处理一块一块到来的数据。另外,本发明通过利用在线顺序极限学习机算法,对主光谱仪及从光谱仪上采集的样品的光谱数据样本进行建模,实现将从光谱仪的光谱数据迁移至主光谱仪的光谱数据空间;然后利用主光谱仪建立的含量预测模型进行样品成分含量的测定,从而提高了样品成分含量预测的精度和稳定性。实验结果表明:本发明的基于在线顺序极限学习机算法的标定迁移方法,在药片数据集和玉米数据集上表现出相较于pds和基于cca的光谱迁移算法更好的性能。
附图说明
图1为玉米数据集中的光谱(a)为mp5,(b)为m5,(c)为mp6光谱;
图2为药片数据集主光谱(a)从光谱(b),二者偏差光谱(c);
图3为rmsep随隐层节点数的变化示意图;
图4是玉米数据集迁移后的m5与mp5偏差(a)和未经迁移的m5与mp5偏差(b);
图5是建模样本数对rmsep的影响示意图;
图6是理化性征的rmsep与隐层节点数的关系示意图;
图7是各类光谱关于水的预测值示意图;
图8是关于蛋白质(a)、淀粉(b)、油(c)、水(d)的预测值情况示意图;
图9是基于elm的迁移算法与基于oselm的迁移算法建模运行时间对比示意图;
图10是药片数据集中隐层节点与光谱rmsep的关系示意图;
图11是基于药片数据(a)光谱迁移残差(b)未迁移残差的示意图;
图12是药片中第一种活性成分的预测值与真实值的比较示意图;
图13是药片中(a)第二种活性成分与(b)第三种活性成分预测值与真实值的比较示意图;
图14是pds、cca、tloselm基于玉米数据含水量的预测结果对比示意图;
图15是pds、cca、tloselm基于药片第三种活性成分的预测结果对比示意图;
图16是本发明的方法流程示意图。
下面结合附图和具体实施方式对本发明作进一步的说明。
具体实施方式
本发明的实施例1:一种基于在线顺序极限学习机的样品成分含量测定方法,如图16所示,采集样品的光谱数据样本,并利用在线顺序极限学习机算法进行建模;利用所建立的模型对样品的成分含量进行测定。
具体可包括以下步骤:
s1,根据初始主光谱spmaster(0)及相应的样品成分含量y0和隐层节点个数l,计算出隐层到输出层的初始权重矩阵α(0),其中,spmaster(0)和y0包含m0个样本;
s2,当有新的主光谱spmaster(k+1)及相应的样品成分含量yk+1到来时,根据在线顺序极限学习机算法计算出隐层到输出层的权重矩阵α(k+1);其中,第k+1次到来的数据spmaster(k+1)和yk+1包含mk+1个样本;k≥0;
s3,如果还存在新的主光谱spmaster(k+1)和相应的样品成分含量yk+1,则令k=k+1,并转至s2,否则转至s4;
s4,根据以下公式计算获得样品的光谱数据spmasterpre所对应的成分含量预测值pre_y:
pre_y=hpreα(new)
其中,
α(new)为目前最新的隐层到输出层的权重矩阵,spmasterpre包含n个样本;w和b分别为随机生成的正交的输入权重矩阵和偏置;g(w,spmasterpre,b)为激活函数。
为了使得基于oselm的样品成分含量预测模型具有更快的计算速度,且对系统资源的耗费更少,所述的隐层节点个数l小于等于初始样本个数m0。
步骤s1中,
α(0)=(h0th0)-1h0ty0;
其中,
步骤s2中,
其中,
为了对从光谱仪所采集的光谱数据进行准确的含量预测,还包括:利用在线顺序极限学习机算法,对主光谱仪及从光谱仪上采集的样品的光谱数据样本进行建模,实现将从光谱仪的光谱数据迁移至主光谱仪的光谱数据空间;然后利用主光谱仪建立的含量预测模型进行样品成分含量的测定。
其中,所述的利用在线顺序极限学习机算法,对主光谱仪及从光谱仪上采集的样品的光谱数据样本进行建模,实现将从光谱仪的光谱数据迁移至主光谱仪的光谱数据空间可包括以下步骤:
s01,根据初始主光谱spmaster0和从光谱spslave0及隐层节点数l,生成隐层到输出层的权重矩阵β(0),其中spmaster0中包含的样本个数为m0;
s02,当有新的包含mk+1个样本的spmaster(k+1)和spslave(k+1)到来时,根据在线顺序极限学习机算法计算出隐层到输出层的权重矩阵β(k+1);其中,k≥0;
s03,如果还有新的样本的spmaster(k+1)和spslave(k+1)到来,则令k=k+1,并转至s02,否则转至s04;s04,根据以下公式对包含n个样本的测试数据spslavetest进行迁移:
spslavetomaster=h'testβnew
其中,spslavetomaster表示迁移后的光谱数据;βnew表示最新的隐层到输出层间的权重矩阵;
为了使得基于oselm的样品成分含量预测模型具有更快的计算速度,且对系统资源的耗费更少,所述的隐层节点个数l小于等于初始样本个数m0。
具体的,步骤s01中,
β(0)=(h'0th'0)-1h'0tspmaster0;
其中,
步骤s02中,
其中,
以上方法中,通过k折交叉验证的方法确定最佳隐层节点数l;所述的激活函数采用sigmoid函数。
实施例2:一种基于在线顺序极限学习机的样品成分含量测定方法,采集样品的光谱数据样本,并利用在线顺序极限学习机算法进行建模;利用所建立的模型对样品的成分含量进行测定。
具体可包括以下步骤:
s1,根据初始主光谱spmaster(0)及相应的样品成分含量y0和隐层节点个数l,计算出隐层到输出层的初始权重矩阵α(0),其中,spmaster(0)和y0包含m0个样本;
s2,当有新的主光谱spmaster(k+1)及相应的样品成分含量yk+1到来时,根据在线顺序极限学习机算法计算出隐层到输出层的权重矩阵α(k+1);其中,第k+1次到来的数据spmaster(k+1)和yk+1包含mk+1个样本;k≥0;
s3,如果还存在新的主光谱spmaster(k+1)和相应的样品成分含量yk+1,则令k=k+1,并转至s2,否则转至s4;
s4,根据以下公式计算获得样品的光谱数据spmasterpre所对应的成分含量预测值pre_y:
pre_y=hpreα(new)
其中,
α(new)为目前最新的隐层到输出层的权重矩阵,spmasterpre包含n个样本;w和b分别为随机生成的正交的输入权重矩阵和偏置;g(w,spmasterpre,b)为激活函数。
为了使得基于oselm的样品成分含量预测模型具有更快的计算速度,且对系统资源的耗费更少,所述的隐层节点个数l小于等于初始样本个数m0。
具体的,步骤s1中,
α(0)=(h0th0)-1h0ty0;
其中,
具体的,步骤s2中,
其中,
为了验证本发明的效果,发明人利用pds算法和基于cca的迁移算法与文发明中的基于oselm(即在线顺序极限学习机)的迁移算法进行了对比。在试验过程中,需要将slave光谱利用pds、cca和基于oselm的算法模型迁移至master空间。再将迁移后的光谱数据带入在master光谱上建立的光谱与成分含量对应的预测模型中进行成分含量预测。
1.1实验环境
本实验是基于python2.7完成的。电脑的操作系统为win8.1,64位操作系统,cpu为amd的a84500,内存为8gb。实验用到了python一些常用的包如:numpy、matplotpy和sklearn包。实验中用到的程序都是在集成开发环境eclipse上开发完成的。
1.2实验数据
在本实验中用到了玉米nir光谱数据集,这个nir光谱数据集包含有八十个不同的nir数据样本。数据集中包含m5,mp5,mp6三张nir光谱数据表(这三张光谱数据表是来自不同光谱仪对同一物质即玉米的nir光谱测量),这个数据集中还有与这八十个光谱数据样本所对应的理化性征表,如:含水量(water)、油(soil)、蛋白质含量(protein)、淀粉含量(starch)。光谱数据的波长范围从1100nm至2498nm,间隔为2nm(包含700个通道)。mp5这张光谱数据表来自作为主(master)仪器的fossnirsystem5000,而m5、mp6则是来自作为从属(slave)光谱分别测量自fossnirsystem6000和fossnirsystem5000。
图1中(a)子图是mp5的光谱图,(b)子图是m5的光谱图,(c)子图是mp6的光谱图,从图1中可以看出mp5和mp6的图像十分相似,因为它们都是用同一型号的光谱仪测量出来的。而m5则与mp5图像差异较大,m5相较于mp5有明显的上移趋势。
本实验中还用到了药片光谱数据集,该数据集是2002年,idrc发表的一份nir光谱数据,其中包括来自两台光谱仪的654种药片。这两台光谱仪分别为fossnirsystems和silver-spring。这份两份来自不同光谱仪的nir光谱数据被划分为校准集(calibrationset包含155个光谱数据样本)和测试集(testset包含460个光谱数据样本)还有验证集(validationset包含40个光谱数据样本)。数据集中的光谱波长集中在1100nm~1750nm之间。
图2中子图(a)和子图(b)分别画出的是药片数据集中的验证集在两台光谱仪spec1和光谱仪spec2上对同一物质的采集结果。图2中子图(c)表示的是spec1上测得的光谱结果与spce2上测得结果的差值。从子图(c)的结果可以看出在两台光谱仪上得出的光谱差异性在大部分波段的差异不是很明显。子图(c)的结果甚至可以说两者测得的结果极为相似,因为差值基本趋于0,只有在1700nm之后的波段变化才开始比较大。
1.3实验设计
1.3.1基于玉米的实验
将玉米的nir光谱数据划分为三份,将数据划分校准集(calibrationset),验证集(validationset)和测试集(testset)。校准集包含56个nir光谱数据,验证集包含8个nir光谱数据,测试集也包含16个nir光谱数据。其中校准集是用来建立在线迁移模型,而验证集则是用来选择最佳的隐层节点数。测试集是用来表现算法泛化性能的。
1、对参数最佳隐层节点数l的选择
为了选取对迁移模型来说最佳的隐层节点数,发明人使用了8折交叉验证的方式选取。将不同隐层节点数经过交叉验证得到的均方根误差用图画出。其中迁移后的光谱均方根误差的计算公式如下所示:
公式(1)就是计算均方根误差的公式,其中numsample表示这批要迁移的nir光谱的样本数目。从图3中可以看出随着隐层节点数目的增加,rmsep先下降后缓慢上升,当隐层节点到达一定数目时,rmsep的增长速度明显加快。从图中可以得出:就光谱迁移的均方根误差来说,隐层节点设置为19最为合适。
因此,模型的隐层节点l设置为最优节点数19。初始时给算法32个spmasternir光谱数据样本和32个spslavenir光谱数据样本。分别作为模型的输入和输出,其实质也就是将spslave向spmaster迁移。建立初始的迁移模型便可得出起始的输出权重矩阵β0,然后假设后续每次有一个nir光谱数据样本到来,基于oselm的思想计算更新后的输出权重矩阵β(k)。当有需要迁移的slave光谱时代入建立的迁移模型即可。nir光谱数据这样便实现了基于oselm算法向master光谱空间迁移的目的。
图4中上面的子图(a)是对spslavetest迁移后得到的结果减去对应的spmastertest的结果,下面的子图(b)是spslavetest直接减去spmastertest的结果。从上图(a)可以看出通过oselm实现了迁移学习的建模,上面的子图(a)中大多spslavetest都有较好的迁移效果,迁移后的spslavetest与spmastertest之差基本都在零附近有轻微的波动。而未经迁移的slave光谱与master光谱之差在0.04~0.06之间波动。
2、对光谱进行标定迁移时初始初入的样本数对算法精度的影响
该模型是基于oselm建立的迁移学习模型。在oselm算法中有一个初始化的问题就是使用多少个样本作为初始时所拥有的样本来生成基础模型。很有可能初始时样本不同对建立的迁移模型会有影响,下面的这张表格1就会展现出初始样本数对模型的影响。
表1中initnum表示初始化时所拥有的nir光谱样本数目,rmsep依旧表示预测的均方根误差。此次试验隐层节点都设置为15个且样本是一个一个到来的。从表1中的数据得出这样的结论:基于oselm的迁移模型的初始化时nir光谱数据样本的数目一定要大于等于隐层节点数。一旦初始时的样本数大于等于隐层节点数,那么预测的误差就保持不变。其实上面的结果也符合oselm算法的内涵,在线流式到来的nir光谱数据集在线建模过程中最终利用上了所有到来的数据样本,oselm与elm的本质其实没有什么不同,oselm作为在线建模的算法只是减少了不必要的重复计算量。但是如果初始时所拥有的nir样本数少于隐层节点数,那么在求解输出矩阵β(0)时可能会涉及到求解违逆矩阵的问题,这样迁移处理的结果均方根误差就会比较大。
表1初始化样本数与rmsep的关系
3、每次顺序到来的数据块大小对算法精度的影响
在以上的实验中都是假设样本一个一个到来的,下面看看nir光谱数据到来的方式是否会对模型的精度有影响。实验的默认设置为隐层节点为15个,初始时样本数为32个。表2中step表示每次来的nir光谱样本数目。从表中的内容可以看出不论nir光谱数据样本是一块一块来的还是一个一个来的都对算法的性能没有影响。到来的nir样本都最终被利用起来了,所以对于测试时,只要它们建模时用到了相同数目的nir样本和隐层节点数l,那么关于预测的均方根误差就是一样的。
表2流式数据样本大小对rmsep的影响
4、基于oselm迁移模型中样本个数对于预测精度的影响
图5中表示的是随着nir光谱数据样本顺序到来,通过oselm迁移后的nir光谱数据与spmastertest均方根误差大小。可以看出随着在线学习的进行累积起来的nir数据样本越来越多,对测试集迁移后的均方根误差大体趋势是越来越小。在图中可以发现大约在第55,56个样本到来的时候出现了预测误差增大的情况,这可能是出现了比较不正常的数据样本,导致情况变差。但就预测的总体趋势来说,在线学习的时间越长累积的关于玉米光谱数据的知识也就越多进而迁移学习的精确度也就有所提升。从图中的数据可以得出结论:到来的样本数越多建立的模型也就越精确越稳定泛化能力也就越强。这个结论相当于回应了在线建模的必要性,如果数据每次不是全部一次性到来而是一块一块或者一个一个的到来而且在这期间还有预测的需要,那么就必须基于现有的样本数据建立模型,当下一次有样本到来时只需更新模型即可而不需要再从头重新建构模型,这便是oselm的内涵所在。
在前面的实验中都是探讨关于迁移后的光谱与主光谱的均方根误差,然而该迁移模型的终极目的便是使在specslave上采集到的spslave可以利用在specmaster上建立的在线更新模型,即最终希望可以通过specmaster上建立的模型预测出关于spslave的理化性征。因此在后面的试验中将会从理化性征的高度看待迁移的效果。由于spslave向spmaster迁移的过程是在线进行的,因此对于spmaster与对应理化性征y之间的预测模型也需要动态更新。以下的实验中首先将在slave光谱仪上采集到的spslave在线的迁移到spmaster数据所在的空间,再将迁移后的光谱代入基于spmaster与y之间的在线预测模型。在玉米数据集中需要通过spslave数据进行关于含水量、蛋白质含量、含油量、淀粉含量的预测。
5、在理化指标的高度看待迁移模型的性能
首先通过k折交叉验证选取主光谱到理化性征预测模型的最佳隐层节点数。下面的实验中将迁移后的光谱数据代入最新建立的理化性征预测模型。从表3中可以看出,l表示迁移模型隐层节点数,rmsep表示关于理化性征预测的均方根误差。可以看出如果隐层节点数设置的过小,那么预测结果的均方根误差会比较大,而如果隐层节点数大于某一个点时,预测误差也会开始增加。在表中极为明显的是隐层节点数大于初始样本数时,计算出来的均方根误差会急剧变大。
表3隐层节点数对理化指标的rmsep的影响
为了更为清楚的将结果展示出来,可以将rmsep和隐层节点数l的关系,用折线图画出来。图6可以更为直观的看出其趋势来。
图7中横坐标表示watertest的值而纵坐标表示对应的预测值pre_water。三角表示直接将spslvaetest带入理化性征的预测模型得到的预测值,五角星表示将spslavetest进行迁移后带入理化性征的预测模型的预测值。加号则表示spmastertest带入模型的预测值。图7中可以看出经过迁移后的光谱数据预测出来的理化属性与真实值是很接近的,如果specslave上采集到的光谱不经迁移就直接使用specmaster上建立的理化性征预测模型,那么预测的误差是非常大的。从图7中可以看出基于oselm的迁移模型在玉米数据集上取得了很大的成功。
如图8所示,子图中的三角、星星和加号均与上图表达的是一样的意义。其中子图(a)、(b)、(c)、(d)的横坐标依次为蛋白质、淀粉、含油量和含水量,每幅子图的纵坐标依次为相应横坐标的预测值。第一幅子图描述的是基于玉米理化性征为蛋白质建立的预测模型。第二幅子图描述的是基于玉米数据集中理化性征为淀粉(starch)建立的预测模型。第三幅子图描述的是基于玉米中含油量建立的预测模型。第四幅图就是上图7。图8中的结果可以清晰的看出光谱数据迁移的效果。在(a)、(b)、(c)、(d)四幅子图中,三角形与星星之间有着明显的分解间隔。
6、oselm的运行效率
截图9第一行表现的是传统elm运行一次需要的时间。第二行表示oselm在线迁移11次所需要的时间。第三行表示如果用elm算法建模11次需要的时间。从图9中可以看出就玉米数据集这个实验运行一次elm迁移算法的时间大约为0.02秒,而oselm迁移算法建模了11次大约运行了0.04秒。通过简单的计算不难得出基于oselm的迁移模型要比仅仅基于elm的迁移模型块很多。oselm之所以比传统elm反复建模快是因为oselm减少了重复计算量。oselm保留了上一次建模时的知识,如果有新的数据到来它只需要对新知识建模。
7、基于玉米数据集实验的总结:
从上面的实验结果可以看出,基于oselm的nir光谱数据迁移模型在玉米数据集上表现出比较理想的性能。迁移后的光谱与未经迁移的光谱代入关于理化性征的预测模型结果有明显的差别。该算法相对与传统的在线学习算法具有更快,适应性更强的优点。在玉米数据集上不论数据是每次一个一个的到来还是两个、三个或多个,该算法模型均能解决。且算法在开始运行前无需指定下一次带来的数据量的大小。该算法使salvenir光谱无需重复建模而直接利用master仪器上的模型来预测对应的含水量、油脂含量、蛋白质含量、淀粉含量成为可能。
1.3.2基于药片的实验
药片数据集中的数据是在两台不同的光谱仪上采集得到的。为了后面表述方便这里将光谱仪1记为spec1,光谱仪2记为spec2。spec1采集到的nir光谱数据记为sp1,spec2采集得到的nir光谱数据记为sp2。在上面数据介绍时数据集中自带了9张不同的表分别为:校准集(calibrate_1,calibrate_2,calibrate_y)、验证集(validate_1,validate_2,validate_y)、测试集(test_1,test_2,test_y)。其中的标号1,2分别表示nir光谱数据来自光谱仪spec1和spec2。在机器学习算法中calibrarteset一般是用来建立模型而calibrateset则是用来选择算法模型的超参数。testset一般会用来检验模型的性能,将其带入建立好的模型得出结果。通过该结果可以对模型泛化能力、精确度及是否出现过拟合现象的一样判断方式。但在这个数据集calibrateset只有155个样本而test却多达460个样本。这样的情形并不符合一般机器学习算法中测试集的设定,因此在本实验中将实验数据重新划分。在本实验总暂时不要validateset,算法中的超参数会通过多次试验的结果加以展现其变化导致算法性能的变化。本次实验中假设用于测试的数据为40个nir光谱数据样本。
1、基于光谱迁移选取最佳隐层节点数
对于不同的数据集,最佳隐层节点数的选取必然是不一样的。药片数据集拥有相较于玉米数据集更多的nir光谱样本个数。因此在建立初试迁移模型求β(0)时,初始样本数设置为100。在这样的背景下,希望探究基于光谱迁移的均方根误差与隐层节点数l的选取。
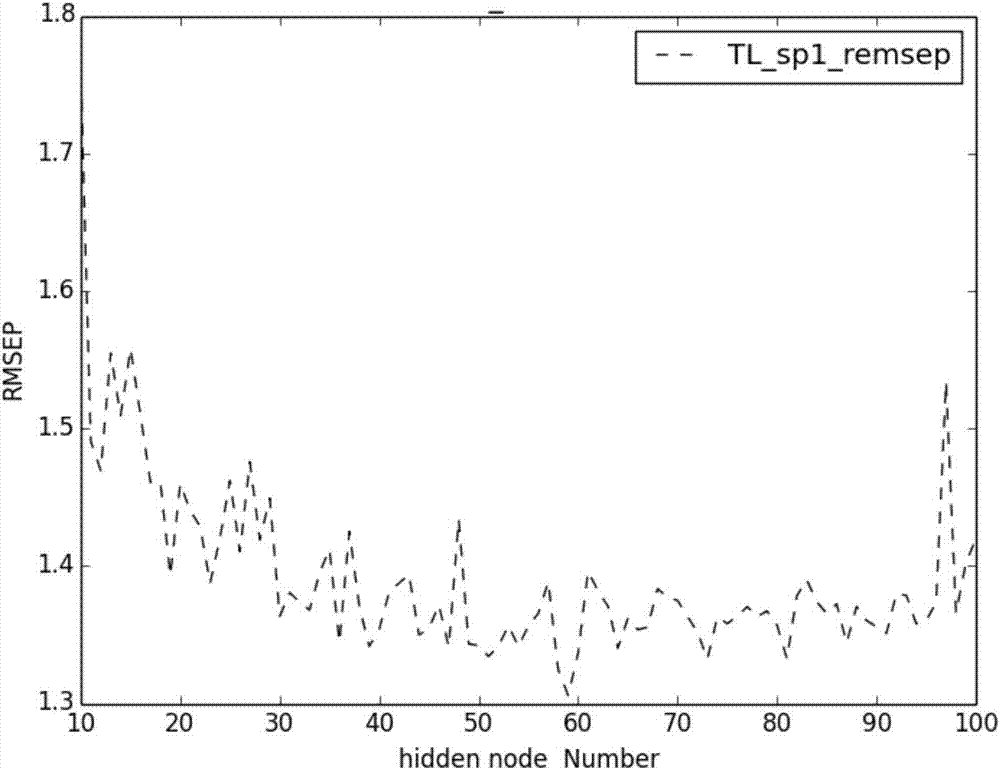
下图10中的横坐标hiddennum表示迁移算法中的隐层节点数目相应的纵坐标rmsep表示迁移后的光谱与对应主光谱的均方根误差。图10中可以看出rmsep随着隐层节点数目的增加呈现出上下波动的不稳定状态,但就大体趋势来说依旧是呈现下降趋势的,当hiddennum大约为59时,rmsep的值最小。随着hiddennum继续增大,rmsep开始呈现出缓慢上升的趋势。当隐层节点数超过初始时赋予的样本数时可以看到图中rmsep会急剧增加。
从图11中看出经过oselm算法迁移后的nir光谱数据大体集中在0附近波动且大体范围为[-0.02,0.02]。但是有少数样本的迁移效果并不是很好。这可能是因为原来数据本身的差异就很小。如果仔细观察图可以发现在spec1上采集的nir数据和在spec2上采集的nir数据差异在[-0.1,0.1]之间而且从图中可以看出在大部分波段,光谱之间的差异基本趋近于0。
2、激活函数的选择对算法精度的影响
有时候激活函数的选取对算法性能有着直接的影响,例如tanh函数将最终值映射到[-1,1]之间,而sigmoid函数则将最终值映射到[0,1]之间,进而得出的结果差异也就比较大。因此在本部分实验准备探究激活函数对算法精度的影响。这里使用常用的sigmoid,tanh,tribas,hardlim函数。下面的实验便从选择最佳激活函数入手。表4中rmsep表示关于药片数据集中第一理化性征预测值的均方根误差大小,n表示参与建模的样本个数,本次实验中初始输入20个nir样本,设置隐层节点l为15。从实验结果可以发现不论选取哪种激活函数,随着模型学习到的样本数增加rmsep都是呈现下降趋势。对比表格中的数据可以获知sigmoid函数在药片数据集上表现出的性能要好于tanh、tribas、hardlim函数。
表4激活函数对算法精度的影响
3、初始化输入样本数与隐层节点的关系
下面实验探究当迁移模型迁移药片nir光谱数据时,初始化时输入的nir光谱数据样本数与隐层节点数l的设置会对算法的性能产生怎样的影响。表5中横轴表示隐层节点数l,sn表示初始化时输入的nir样本数,表格中间的数字表示rmsep。表5中空下的地方表示此处误差很大。从表格中的数字变化个明显看出一旦隐层节点数设置超过初始时的样本数那么rmsep就会急剧增大,有时误差可以达到几千。因此又一次证实了隐层节点数不能超过初始时的样本数。表格中的数字还能看出在隐层节点不变时,初始化输入的nir光谱样本数目的变化基本对rmsep没有影响(但样本数需要大于等于隐层节点数)。
表5药片数据集中初始化输入样本数与隐层节点的关系
4、每次到来的数据块大小对算法精度的影响
下面的表格用来表示流式到来的数据块大小是否对算法的精度有影响。在本次实验中,隐层节点设置为90,初始化的nir样本数为100。
从表6中的数据可以看出,用oselm算法处理流式数据,每次数据到来的大小并不会影响算法的精度。oselm算法在不需要知道下一次到来的数据块大小。且oselm算法具有大多数其他算法所不具有的优点就是既可以处理一个一个到来的数据样本也可以处理大小变化的数据块。
表6流式数据快样本数大小与rmsep的关系
5、从理化指标的高度看待基于oselm迁移模型的性能
使用药片nir数据集,先将从仪器spec2上测得的光谱数据在线标定迁移至主仪器spec2的光谱空间,然后利用迁移后的光谱数据代入关于药片中第一活性成分的预测模型得到预测值。关于对从仪器上采集到的光谱预测出的第一活性成分效果可以通过图12观察到。图12中sp1_predict表示主仪器的测试数据代入药片第一活性成分预测模型得到的预测值。tlsp2_predict表示使用oselm迁移模型后基于第一活性成分预测模型的预测值。sp2_predict表示sp2不经迁移直接代入关于药片中第一活性成分预测模型得到预测值。图中可以看到三角与加号和星星之间有明显的分解线,因此对于从光谱的标定迁移效果还是比较显著的。
图13中绘制的是sp1_test、tlsp2_test、sp2关于药片中第二活性成分和第三活性成分的预测值。第一幅子图虽然五角星和加号没有回归到直线,这只能说明参数没有选好或者该数据集在理化性征预测模型上的效果不好,但是仍然可以看出加号和五角星的预测值基本一样,而三角很明显与五角星和加号之间有明显的分割。第二幅子图表现出的性能与第一幅子图差不多,此处便不再论述。
1.3.3对比试验
在上面建立的模型是否优于其他算法,需要通过比较方能知晓。在对比试验中用到了背景技术中介绍过的pds算法和cca算法。其中pds算法中有超参数为窗口大小,不同的窗口大小会对算法的效果产生一定影响。下面这张表中通过选取不同的窗口值大小来展现pds在不同窗口下的性能。将pds、cca、tloselm(即本发明中提出的基于oselm的迁移算法)对salve光谱数据进行迁移,然后将迁移后的模型代入在线建立的关于理化性征的预测模型(该预测模型的超参数通过交叉验证选出),便可以预测出迁移后的光谱对应的理化特征值。在玉米数据集中只对含水量和蛋白质这两种理化性征进行均方根误差大小进行对比。在药片数据集中则进行了对三种活性成分的预测值的均方根误差进行了对比。
表7和表8表示分布表示三种算法在玉米含水量和蛋白质含量上预测误差的对比。表9、表10、11分布表示pds、cca、tloselm在药片数据集上分布对第一、二、三活性成分的预测值误差。以上诸表中的字母n表示用于构建迁移模型的样本数,rmsep表示关于理化性征的均方根误差。从表7和图9中的数据可以得出在玉米数据集中,当m5光谱向mp5光谱迁移时基于cca算法迁移模型要优于pds算法,但是mp6向mp5光谱迁移时pds的效果要更好一些。就整个玉米nir光谱数据集来说,tloselm算法(即本发明中的基于在线顺序极限学习机的迁移算法)在这三种算法中表现性能最优。三种算法都是随着建模样本数的增加,tloselm算法模型和cca算法模型的rmsep不断减小,但是pds算法则表现出不稳定的一面,刚开始随着样本的增加,rmsep开始下降,随着样本数继续增加,rmsep又开始呈现出增长的趋势。表9、10、11可得出,在药片数据集中tloselm算法依旧是三种算法中性能最优的。除了对于第一活性成分的预测cca要好于pds,在关于第二、三活性成分的预测中pds则要优于cca算法。图14是三种算法对玉米含水量的预测效果,图15是三种算法对药片数据第三活性成分的预测结果。
表7玉米数据集在不同算法下对水预测误差
表8玉米数据集在不同算法下对蛋白质的预测误差
表9药片数据集第一种活性成分的预测误差
表10药片数据集第二种活性成分的预测误差
表11药片数据集第三种活性成分的预测误差
从上面的实验中得出结论:在玉米和药片数据集上,本发明的tloselm算法相较于pds算法和cca算法在从光谱向主光谱迁移过程中拥有更好的性能。在玉米数据集中当使用mp6作为从光谱而mp5作为主光谱时,虽然依旧是tloselm最佳,但是可以发现cca算法并没有比pds算法优太多。而在药片集或者m5作为从光谱mp5为主光谱时,cca和tloselm相较于pds有很大的优势,且本发明的tloselm算法性能最优。
- 还没有人留言评论。精彩留言会获得点赞!