一种基于生物医学知识图谱推理的药物识别方法与流程
本发明涉及数据挖掘方法领域,尤其是一种基于生物医学知识图谱推理的药物识别方法。
背景技术:
药物发现(drugdiscovery)是医药产业发展的核心驱动力,也是社会发展的重要需求。目前主要存在两类药物发现的方法,分别是高通量筛选(high-throughputscreening,hts)和计算机辅助药物发现方法(computer-aideddrugdiscovery/design,cadd)。然而,尽管药物研发模式和技术有了巨大革新,药物发现依旧是一个十分漫长且耗资巨大的过程,开发一款新药平均需要14年时间,耗资约18亿美元。因此,如何提高药物发现的效率具有重大的理论价值和实用价值。
从已发表的生物医学文献中发现新的药物是一种经济安全的药物发现方法。已发表的生物医学文献中隐含着无法治愈疾病的潜在治疗方法,比如雷诺士病(raynauddisease)在1986年以前是一种无法治愈的疾病,donr.swanson通过阅读一部分医学文献发现雷诺士病的患者都伴随血粘稠度升高、血脂升高等医学特征;swanson又通过阅读另一部分医学文献发现食用鱼油(fishoil)可以降低血脂、降低血粘稠度等现象。因此swanson作出了食用鱼油可以治疗雷诺士病的假设,这个结论在两年后被临床试验所验证。因此,使用文本挖掘(literaturemining)进行药物发现是一个可行的方法。现有的文本挖掘相关的药物发现方法主要分以下几种:
一、基于共现的方法:该方法主要通过与药物和疾病都相关的中间物质来推断药物和疾病可能存在的关系。
二、基于语义的方法:该方法首先通过关系抽取等技术有选择性的从文献中抽取出药物-实体、疾病-实体关系,再利用已抽取的关系作出药物-疾病关系预测。
三、基于图结构的方法:该方法首先通过抽取得到的实体关系构造一个网络,然后在该网络上使用聚类、分类等机器学习算法进行药物-疾病关系预测。
然而,近年来随着生物医学领域的快速发展,生物医学文献数量呈指数性增加,海量的文献和信息为现有的文本挖掘方法带来了难题。
技术实现要素:
本发明的目的是提供一种能够充分利用现有的海量医学文献进行药物识别,并可辅助药物研发工作的基于生物医学知识图谱推理的药物识别方法。
本发明解决现有技术问题所采用的技术方案:一种基于生物医学知识图谱推理的药物识别方法,包括以下步骤:
s1、下载生物医学文本数据:在医学文献检索系统中下载生物医学文献,并将下载得到的生物医学文献全文以字符串的形式存储在本地,得到生物医学文献库;
s2、构造生物医学知识图谱:包括以下步骤:
a1、抽取实体间关系:利用关系抽取工具semrep从所述生物医学文献库中抽取得到生物实体间关系,并将抽取得到的生物实体间关系以字符串的形式存储在本地;
a2、基于频率的候选实体关系过滤:预设最小频率阈值,并将在步骤a1中得到的生物实体间关系中出现次数小于预设最小频率阈值的生物实体间关系过滤掉,得到用于构造生物医学知识图谱的实体间关系数据集s;
a3、构造生物医学知识图谱:利用步骤a2得到的实体间关系数据集s构造知识图谱;在知识图谱中以实体间关系数据集s中的各个生物实体作为节点,知识图谱中的边为实体间关系数据集s中生物实体间关系,从而得到生物医学知识图谱;
s3、构造药物-靶标-疾病关系数据集:在生物医学知识图谱中,建立药物-靶标-疾病三元关系路径e0r0e1r1e2r2...el-1rl-1el,其中e0,e1,e2,...,el-1,el为生物医学知识图谱中的节点,e0为药物,e1,e2,...,el-1中至少一个为药物e0的靶标,el为疾病,r0,r1,r2,...,rl-1分别为e0,e1,e2,...,el-1,el中相邻节点间的生物实体间关系,l为实体e0到实体el的路径长度,l≥2;
以正例路径数据和负例路径数据构成药物-靶标-疾病关系数据集;所述正例路径的构造方法:对于一个已知的药物-靶标-疾病三元关系,首先通过路径搜索算法构造路径长度为l的训练集πl=ρ(药物→疾病;靶标,l),其中,ρ()为广度优先搜索算法,l≥2,πl为在生物医学知识图谱中以该已知药物为起点,以已知的药物-靶标-疾病三元关系中的疾病为终点且穿过已知的药物-靶标-疾病三元关系中的靶标且长度为l的所有路径;然后使用相同的路径搜索算法构造出长度为2到l的所有路径数据的集合p={π2,π3...πl}作为训练药物发现模型的正例路径数据;所述负例路径的构造方法:对于所述已知的药物-靶标-疾病三元关系,首先通过随机替换的方式将已知的药物-靶标-疾病三元关系中的药物、靶标、疾病分别替换成therapeutictargetdatabase数据库中的已知药物、靶标和疾病构造出随机药物-靶标-疾病三元关系:药物’-靶标’-疾病’,并保证该随机药物-靶标-疾病三元关系在therapeutictargetdatabase数据库中不存在;然后使用路径搜索算法构造数据集p'={π'2,π'3...π'l}作为训练药物发现模型的负例路径数据;
s4、使用图嵌入的方法对图进行表示学习:利用图嵌入方法将表示形式为图结构的数据转化成低维空间向量表示的数据,具体方法为:在步骤a3构造的生物医学知识图谱中,使用(s,r,t)表示该生物医学知识图谱中一条边的头结点s、尾节点t及头节点与尾节点之间的关系r,利用图嵌入方法将头节点s、尾节点t及头节点与尾节点之间的关系r分别转化为头节点向量vs、尾节点向量vt及头节点与尾节点之间的关系向量vr,vs、vt及vr的向量长度均为m,m≥2;图嵌入方法的目标函数为
s5、训练基于长短记忆神经网络的药物发现模型步骤:利用长短记忆神经网络对步骤s3构造好的药物-靶标-疾病关系数据集进行有监督学习建模,其具体过程如下:
b1、将药物疾病关系数据集表示为向量形式:使用步骤s4中得到的生物医学知识图谱中节点和边的向量表示对步骤s3构造的药物-靶标-疾病三元关系路径e0r0e1r1e2r2...rl-1rl中的每一个实体e0,e1,e2,...,el-1,el和生物实体间关系r0,r1,r2,...,rl-1进行向量表示,使该药物-靶标-疾病三元关系路径转化为m×l维矩阵pmatrix=e0r0e1r1e2r2…rl-1el,得到药物-靶标-疾病三元关系矩阵;
b2、构造及训练长短记忆神经网络:以药物-靶标-疾病三元关系矩阵pmatrix=e0r0e1r1e2r2…rl-1el为输入训练长短记忆神经网络:
长短记忆神经网络的构造如下:
ct=f⊙ct-1+i⊙g
ht=o⊙tanh(ct)
其中i为输入门向量,f为忘记门向量,o为输出门向量,g为临时状态向量,ct为t时刻细胞激活向量;xt是t时刻输入的实体向量,h为隐层向量,ht为t时刻的隐层向量,⊙是位乘操作,σ为sigmod函数;初始输入时h0=e0,
训练方法:对于给定疾病时候选药物的概率:p(y|pmatrix)=σ([whzht+b]),其中whz为t时刻隐层h与输出层z间的向量,ht为t时刻隐层向量,b为与隐层向量维度相同的向量;定义目标函数为l(θ)=-σlog(p(y|pmatrix)),其中pmatrix为药物-靶标-疾病关系路径的矩阵,σ为sigmod函数;并在训练数据上迭代地进行优化,直到所有参数收敛或达到预设的终止条件为止:即损失函数差小于10e-5;输出训练好的参数集合θ,其中θ包括
s6、使用训练好的模型进行药物识别步骤:对于给定的疾病,评价潜在药物drugpotential治疗该疾病的可能性的步骤为:首先构造所有起点为drugpotential终点为diseasepotential且穿过targetpotential的路径集ppotential={ρ(drugpotential→disease;targetpotential)},其中targetpotential为therapeutictargetdatabase数据库中的所有药物靶标;然后使用打分函数
医学文献检索系统以时间检索的方式下载生物医学文献。
所述医学文献检索系统为pubmed检索系统。。
本发明的有益效果在于:本发明具有以下特点:
(1)本发明可通用的在各类生物医学文献中进行药物发现,该方法不局限于某类(些)疾病的药物发现。
(2)本发明可给出药物的作用机制(mechanismofaction),从而可辅助医学研究人员进一步理解、研究药物-疾病关系,进而辅助药物不良反应预测、精确医疗等领域。
附图说明
图1为本发明总体流程图。
图2为本发明构造的生物医学知识图谱的结构示意图。
图3为本发明训练药物发现模型的逻辑结构示意图。
具体实施方式
以下结合附图及具体实施方式对本发明进行说明:
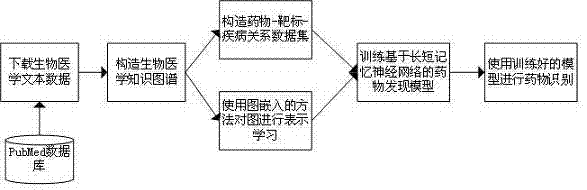
图1为本发明一种基于生物医学知识图谱推理的药物识别方法总体流程图。一种基于生物医学知识图谱推理的药物识别方法,包括以下步骤:
s1、下载生物医学文本数据:利用医学文献检索系统pubmed以时间检索的方式下载生物医学文献,并将下载得到的生物医学文献全文以字符串的形式存储在本地,得到生物医学文献库;
s2、构造生物医学知识图谱:包括以下步骤:
a1、抽取实体间关系:利用关系抽取工具semrep从所述生物医学文献库中抽取生物实体间关系,并将抽取得到的生物实体间关系以字符串的形式存储在本地;如表1所示,semrep从表中所示的文本中抽取出4个实体间关系。其中“|”左边为实体名称,右边为实体的类型。
表1实体间关系抽取示例
a2、基于频率的候选实体关系过滤:预设最小频率阈值,并将在步骤a1中得到的生物实体间关系中出现次数小于预设最小频率阈值的生物实体间关系过滤掉,得到用于构造生物医学知识图谱的实体间关系数据集s;
a3、构造生物医学知识图谱:利用步骤a2得到的实体关系数据集s构造知识图谱;如图2所示,在知识图谱中以实体间关系数据集s中的各个生物实体作为节点,其属性包括该实体的语义类型、该语义类型被抽取出的次数及抽取出该实体关系的文档号(pmid号)。知识图谱中的边为实体间关系数据集s中生物实体间关系,其属性包括该关系被抽取出的次数及抽取出该关系的文档号(pmid号),得到生物医学知识图谱;
s3、构造药物-靶标-疾病关系数据集:在生物医学知识图谱中,建立药物-靶标-疾病三元关系路径e0r0e1r1e2r2...el-1rl-1el,其中e0,e1,e2,...,el-1,el为生物医学知识图谱中的节点,e0为药物,e1,e2,...,el-1中至少一个为药物e0的靶标,el为疾病,r0,r1,r2,...,rl-1分别为e0,e1,e2,...,el-1,el中相邻节点间的生物实体间关系,l为实体e0到实体el的路径长度,l≥2;
以正例路径数据和负例路径数据构成药物-靶标-疾病关系数据集;所述正例路径的构造方法:对于一个已知的药物-靶标-疾病三元关系,首先通过路径搜索算法构造路径长度为l的训练集πl=ρ(药物→疾病;靶标,l),其中,ρ()为广度优先搜索算法,l≥2,πl为在生物医学知识图谱中以该已知药物为起点,以已知的药物-靶标-疾病三元关系中的疾病为终点且穿过已知的药物-靶标-疾病三元关系中的靶标且长度为l的所有路径;然后使用相同的路径搜索算法构造出长度为2到l的所有路径数据的集合p={π2,π3...πl}作为训练药物发现模型的正例路径数据;所述负例路径的构造方法:对于所述已知的药物-靶标-疾病三元关系,首先通过随机替换的方式将已知的药物-靶标-疾病三元关系中的药物、靶标、疾病分别替换成therapeutictargetdatabase数据库中的已知药物、靶标和疾病构造出随机药物-靶标-疾病三元关系:药物’-靶标’-疾病’,并保证该随机药物-靶标-疾病三元关系在therapeutictargetdatabase数据库中不存在;然后使用路径搜索算法构造数据集p'={π'2,π'3...π'l}作为训练药物发现模型的负例路径数据。
s4、使用图嵌入的方法对图进行表示学习:利用图嵌入方法(graphembeddingmethod)将表示形式为图结构的数据转化成低维空间向量表示的数据,具体方法为:在步骤a3构造的生物医学知识图谱中,使用(s,r,t)表示该生物医学知识图谱中一条边的头结点s、尾节点t及头节点与尾节点之间的关系r,利用图嵌入方法将头节点s、尾节点t及头节点与尾节点之间的关系r分别转化为头节点向量vs、尾节点向量vt及头节点与尾节点之间的关系向量vr,vs、vt及vr的向量长度均为m,m≥2;图嵌入方法的目标函数为
s5、训练基于长短记忆神经网络的药物发现模型步骤:利用长短记忆神经网络(lstm)对步骤s3构造好的药物-靶标-疾病关系数据集进行有监督学习建模,其具体过程如下:
b1、将药物疾病关系数据集表示为向量形式:使用步骤s4中得到的生物医学知识图谱中节点和边的向量表示对步骤s3构造的药物-靶标-疾病三元关系路径e0r0e1r1e2r2...rl-1el中的每一个实体e0,e1,e2,...,el-1,el和生物实体间关系r0,r1,r2,...,rl-1进行向量表示,使该药物-靶标-疾病三元关系路径转化为m×l维矩阵pmatrix=e0r0e1r1e2r2...rl-1el,得到药物-靶标-疾病三元关系矩阵;
b2、构造及训练长短记忆神经网络:以药物-靶标-疾病三元关系矩阵pmatrix=e0r0e1r1e2r2...rl-1el为输入训练长短记忆神经网络:
长短记忆神经网络的构造如下:
ct=f⊙ct-1+i⊙g
ht=o⊙tanh(ct)
其中i为输入门(inputgate)向量,f为忘记门(forgetgate)向量,o为输出门(outputgate)向量,g为临时状态向量,ct为t时刻细胞(cell)激活向量。xt是t时刻输入的实体向量,h为隐层向量,ht为t时刻的隐层向量,⊙是位乘操作,σ为sigmod函数。初始输入时h0=e0,
训练方法:对于给定疾病时候选药物的概率:p(y|pmatrix)=σ([whzht+b]),其中whz为t时刻隐层h与输出层z间的向量,ht为t时刻隐层向量,b为与隐层向量维度相同的向量;定义目标函数为l(θ)=-σlog(p(y|pmatrix)),其中pmatrix为药物-靶标-疾病关系路径的矩阵,σ为sigmod函数;并在训练数据上迭代地进行优化,直到所有参数收敛或达到预设的终止条件为止:即损失函数差小于10e-5;输出训练好的参数集合θ,其中θ包括
s6、使用训练好的模型进行药物识别步骤:对于给定的疾病,评价潜在药物drugpotential治疗该疾病的可能性的步骤为:首先构造所有起点为drugpotential终点为diseasepotential且穿过targetpotential的路径集ppotential={ρ(drugpotential→disease;targetpotential)},其中targetpotential为therapeutictargetdatabase数据库中的所有药物靶标;然后使用打分函数
如表2所示,对于给定疾病冠心病(cardiovasculardisease),本发明的方法发现碘克酸(ioxaglate)可以治疗冠心病(cardiovasculardisease),该候选药物的分数为0.57分,在所有候选药物中排名第1。经ttd(therapeutictargetdatabase)数据库证实碘克酸(ioxaglate)确实可以治疗冠心病,但其作用机制尚不明确。我们的方法给出其作用机制如表2所示。
表2药物发现示例
以上内容是结合具体的优选技术方案对本发明所作的进一步详细说明,不能认定本发明的具体实施只局限于这些说明。对于本发明所属技术领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干简单推演或替换,都应当视为属于本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!