基于多尺度蒸馏的脑肿瘤MRI图像分割方法
基于多尺度蒸馏的脑肿瘤mri图像分割方法
技术领域
1.本发明涉及数字医学图像识别技术领域,具体是基于多尺度蒸馏的脑肿瘤mri图像分割方法。
背景技术:
2.脑胶质瘤是最常见的原发性中枢神经系统肿瘤之一,具有极高的发病率和致死率。核磁共振成像(magnetic resonance imaging,mri)能对大脑软组织进行高空间分辨率和高对比度成像,是医生进行脑结构分析的最佳选择,因此在临床上被广泛使用。图像分割是脑肿瘤研究的关键步骤,其为后续诊断和治疗提供指导依据。然而手动分割是一项十分费时费力的工作,且严重依赖于医生的经验。由于脑肿瘤形状多变、结构复杂以及类别极不平衡等特点,传统的图像分割算法如区域生长和像素分类等往往很难获得令人满意的分割效果。因此自动脑肿瘤分割算法的研究仍是一项十分具有挑战性的任务。
3.目前,专家手工分割是临床中最常用的分割方法。专家手动分割脑肿瘤即在图像上手动标注脑肿瘤的边界,标注时采用不同的标签表示不同的解剖结构区域。因此,脑肿瘤手工分割专家需要掌握解剖学等知识。这种脑肿瘤分割方式存在一些缺点,包括:
4.(1)随着脑肿瘤影像的增加,人工分割脑肿瘤的不同区域任务变得不仅耗时且容易出错,并且分割效果也逐渐下降,进而影响临床医生对脑肿瘤患者的诊断、治疗方案的制定以及患者术后恢复效果评估。
5.(2)人工脑肿瘤分割方法具有主观性,无法复现。主观性体现在,对于相同的脑肿瘤核磁共振影像,不同专家手工分割的结果未必相同。同一个专家在不同时间对相同影像进行标注,得到的结果也可能不同,即该方法是不能复现的。
6.为了减轻临床医生或脑肿瘤专家的工作量,需要研究一种全自动的、可复现的脑肿瘤分割方法。
技术实现要素:
7.本发明要解决的技术问题是提供基于多尺度蒸馏的脑肿瘤mri图像分割方法,用以对多模态核磁共振图像上的脑胶质母细胞瘤区域能够准确定位和更加准确精细分割。
8.为了解决上述技术问题,本发明提供基于多尺度蒸馏的脑肿瘤mri图像分割方法,包括采集多模态核磁共振图像,在上位机中对多模态核磁共振图像进行预处理,然后输入到多尺度蒸馏训练操作后的deeplabv3+网络,输出多模态mri图像的分割结果图;
9.所述预处理包括对多模态mri图像x轴的图像矩阵依次人工加入黑色切片、z-score标准化处理、归一化处理、裁剪处理和图像切片化处理;
10.所述分割结果图采用不同灰度进行区域描述,包括背景区域、坏疽区域、浮肿区域和增强肿瘤区域。
11.作为本发明的基于多尺度蒸馏的脑肿瘤mri图像分割方法的改进:
12.所述人工加入黑色切片为所述多模态核磁共振图像的x轴前后分别插入3张和2张
全0的图像矩阵;
13.所述z-score标准化处理为:
[0014][0015]
其中x表示待处理的图像矩阵,η代表均值,σ代表标准差,z代表处理后的图像矩阵;
[0016]
所述归一化处理为:
[0017][0018]
z代表标准化处理后的图像矩阵,y代表归一化后的图像矩阵
[0019]
所述裁剪处理为:
[0020]
x
crop
=x
old
[i1:i2,j1:j2]
ꢀꢀ
(3)
[0021]
其中,x
old
为归一化处理后的图像矩阵,x
crop
为裁剪后的图像矩阵,i1,i2为行方向的裁剪范围的始末值,j1,j2为列方向的裁剪范围的始末值;
[0022]
所述图像切片化处理为将所述裁剪处理后的图像进行切片处理,获得的每个切片图像高为h和宽为w。
[0023]
作为本发明的基于多尺度蒸馏的脑肿瘤mri图像分割方法的进一步改进:
[0024]
所述deeplabv3+网络的训练过程为:
[0025]
1)、将训练集分成3个数据子集cn(n=1,2,3),分别为坏疽子集c1、浮肿子集c2和增强肿瘤子集c3,每个数据子集cn由一组对(i
t
,s
t
)组成,其中i
t
表示大小为w
×
h的输入图像,s
t
表示相应的地面真相分割掩码,t=0,1,2;
[0026]
2)、设置训练的超参数和损失函数并使用损失函数来计算模型预测值和真实值的差异;然后将将坏疽子集c1输入到deeplabv3+网络进行训练,g为deeplabv3+网络的编码器,f为deeplabv3+网络的解码器,获得可以识别分割背景区域与坏疽区域的模型g0·
f0;
[0027]
3)、将坏疽子集c1和浮肿子集c2作为输入数据集,对模型g0·
f0进行多尺度蒸馏训练操作和伪标签处理,获得可以识别分割背景区域、坏疽区域和浮肿区域的模型g1·
f1;
[0028]
4)、将浮肿子集c2和增强肿瘤子集c3别作为输入数据集,对模型g1·
f1进行多尺度蒸馏训练操作和伪标签处理,获得可以识别分割背景区域、坏疽区域、浮肿区域和增强肿瘤区域的模型g2·
f2;
[0029]
然后在测试集上测试验证模型g2·
f2,获得所述多尺度蒸馏训练操作后的deeplabv3+网络。
[0030]
作为本发明的基于多尺度蒸馏的脑肿瘤mri图像分割方法的进一步改进:
[0031]
所述多尺度蒸馏训练操作具体为:
[0032][0033]
其中,x为特征张量,h,w,c分别为张量的长、宽和通道数,h和ω分别为长和宽的索引,ω(x)为特征张量的合并切片;
[0034]
[0035]
其中,f为特征提取器,t为分类类别,i为当前提取的特征层数,l为特征层总数,l
pod
为嵌入特征层之间的最小l2距离;
[0036]
ψ(x)=[[ψ1(x)||
…
||ψs(x)]∈r
(h+w)
×c×sꢀꢀ
(7)
[0037]
其中,ψs(x)为蒸馏特征的最终嵌入特征,
[0038][0039]
其中是嵌入张量x的一个子区域,大小为w/s
×
h/s。
[0040]
作为本发明的基于多尺度蒸馏的脑肿瘤mri图像分割方法的进一步改进:
[0041]
所述伪标签处理具体为:
[0042][0043]
其中u表示像素(ω,h)的不确定性,τc是特定于类别的阈值,ω,h为当前张量的长度和高度,c
′
为当前学习的类别,c
bg
为当前图片中的背景类;p
t
(ω,h,c
bg
)为步骤t时模型对先前类别的分割图,p
t
(ω,h,c
′
)为步骤t时模型对当前学习类别的分割图,为步骤t-1时模型对当前学习类别的分割图,为步骤t时模型对目前学习的总类别的分割图。
[0044]
作为本发明的基于多尺度蒸馏的脑肿瘤mri图像分割方法的进一步改进:
[0045]
所述损失函数为:
[0046][0047]
其中λ是超参数;
[0048][0049]
其中,v是可接受的旧类像素与此类像素总数的比率;
[0050][0051]
作为本发明的基于多尺度蒸馏的脑肿瘤mri图像分割方法的进一步改进:
[0052]
将brats2020比赛开源数据集中的多模态mri图像的x轴的图像矩阵进行所述预处理,然后将预处理获得的切片图像打乱后按8:2的比例划分为所述训练集和测试集。
[0053]
本发明的有益效果主要体现在:
[0054]
1、本发明采用了gbm(脑胶质母细胞瘤)患者t1、t1c、t2和flair四种模态的核磁共振图像进行肿瘤分割,增加了样本的多样性,使得模型能够更好的进行特征提取。
[0055]
2、本发明采用了deeplabv3+网络作为主体模型,改进了蒸馏损失方法,通过保持
长程和短程空间关系,能够更好的保留学习的知识,提高模型精度。
[0056]
3、本发明采用旧模型的输出作为标签,用以识别当前图像背景中的旧类,防止背景像素对新类学习产生影响。
[0057]
4、本发明将整个肿瘤图像分割为背景区域、坏疽区域、浮肿区域、增强肿瘤区域,对辅助医生诊断有着积极的作用。
附图说明
[0058]
下面结合附图对本发明的具体实施方式作进一步详细说明。
[0059]
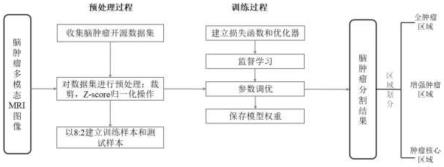
图1为本发明的基于多尺度蒸馏的脑肿瘤mri图像分割方法的流程示意图;
[0060]
图2为本发明的数据预处理后的结构示意图;
[0061]
图3为本发明的deeplabv3+网络的结构示意图;
[0062]
图4为本发明的训练过程原理图;
[0063]
图5为本发明的多尺度蒸馏的原理图;
[0064]
图6为测试集中的一个原始图像(上图)及对应的分割结果图(下图)。
具体实施方式
[0065]
下面结合具体实施例对本发明进行进一步描述,但本发明的保护范围并不仅限于此:
[0066]
实施例1、基于多尺度蒸馏的脑肿瘤mri图像分割方法,使用编码器-解码器结构的deeplabv3+网络,将脑部的多模态核磁共振图像(核磁共振图像简称为mri图像)分割为背景区域、坏疽(net)区域、浮肿区域(ed)、增强肿瘤区域(et)。以上4个分割区域不存在交叉区域,多模态核磁共振图像包括t1、t1ce、t2和flair四种模态图像信息。同时使用改进后的蒸馏方案,对模型进行多尺度蒸馏操作,蒸馏过程中使用旧模型的输出对图片背景进行处理,减少了先前知识对学习新类别的影响。由于本发明在对多模态mri图像进行分割时,对除背景外的每一个类别都进行了单独分割,将多模态数据样本输出模型训练,可将整个肿瘤图像得到最全面的分割,最后本发明将flair模态图像进行分割预测,并使用itk-snap软件展示预测结果,如图1-6所示。具体过程为:
[0067]
1构建数据集
[0068]
1.1训练数据来源
[0069]
本实施例中所使用的多模态mri图像来自于brats2020比赛开源数据集,brats 2020是miccai脑肿瘤分割比赛的数据集,训练集包含高级胶质瘤和低级胶质瘤患者共369个病例,每个病例包含四个模态图像(多模态图像)和一张对应的标签图像,四种模态图像分别为液体衰减反转恢复(flair)图像,t1加权(t1)图像,t1加权对比增强(t1gd)图像和t2加权(t2)图像;标签图像包括4个标签,分别是背景区域、坏疽(net)区域、浮肿区域(ed)、增强肿瘤区域(et),这四个标签能够共同反映whole tumor(wt),enhance tumor(et),and tumor core(tc)。
[0070]
1.2数据预处理
[0071]
依次预处理每个病例的每个多模态mri图像,获取对应的切片图像,以及每个切片图像中所有像素信息,具体包括以下步骤:
[0072]
1.2.1人工加入黑色切片
[0073]
多模态mri图像为3d图像,计算机读入后为3维图像矩阵,大小为[155,240,240],分别对应x轴,y轴,z轴方向,在3d数据的x轴前后分别插入3张和2张全0大小为[240,240]的矩阵,使得多模态mri图像的x轴的图像矩阵大小变为[160,240,240],方便后续裁剪以及图像的输出尺寸的统一。
[0074]
1.2.2标准化处理
[0075]
由于brats2020比赛开源数据集中的四个序列是不同模态的图像,因此图像对比度也不一样,且mri图像的像素值数值过大且灰度分布不均,不利于模型的参数收敛,所以采用z-score方式来对每个模态图像进行标准化处理,即将每个模态的数据标准化为零均值和单位标准差,计算公式如下:
[0076][0077]
其中x表示待处理的图像矩阵(即步骤1.2.1获得的x轴的图像矩阵),η代表均值,σ代表标准差,z代表处理后的图像矩阵。
[0078]
1.2.3归一化处理
[0079]
将标准化处理后的图像矩阵进行归一化操作,使得矩阵数值分布在0-255之间,便于模型计算处理,计算公式如下:
[0080][0081]
其中,z代表标准化处理后的图像矩阵,y代表归一化后的图像矩阵。
[0082]
1.2.4裁剪处理
[0083]
mri图像大部分都是黑色背景,不利于特征提取和模型的计算,对于模型计算收敛无帮助,需要对该三维图像进行裁剪处理和对模型推理出的结果图像进行填充,即将每一张归一化后的图像矩阵周围的黑色部分裁剪掉,并将输出结果进行填充,使得模型能够专注于脑肿瘤部分,裁剪后的大小为[160,160,160]。
[0084]
x
crop
=x
old
[i1:i2,j1:j2]
ꢀꢀ
(3)
[0085]
其中,x
old
为裁剪前的图像矩阵(即归一化处理后的图像矩阵),x
crop
为裁剪后的图像矩阵,i,j分别代表行方向与列方向的裁剪大小的范围,其中i1,i2为行方向的裁剪范围的始末值,j1,j2为列方向的裁剪范围的始末值。
[0086]
1.2.5图像切片化处理
[0087]
mri图像为3d图像,而本发明使用的deeplabv3+网络是二维分割模型,故对步骤1.2.4裁剪处理后的图像矩阵从x轴方向进行切片处理,每个切片图像高h和宽w的大小均为160。
[0088]
1.3划分为训练集和测试集
[0089]
将步骤1.2.5获得的切片图像打乱后按8:2的比例划分训练集和测试集,然后将训练集按照分类类别分成3个数据子集cn(n=1,2,3),其中n代表数据子集的类别,分别为坏疽子集c1、浮肿子集c2和增强肿瘤子集c3,测试集不变;每个数据子集cn由一组对(i
t
,s
t
)组成,其中i
t
表示大小为w
×
h的输入图像,s
t
表示相应的地面真相分割掩码,t=0,1,2。设c
t
表示类别,c
bg
表示背景类。最终得到:由x轴方向的切片组成的训练集(包括3个数据子集cn(n
=0,1,2))和测试集。
[0090]
2多尺度蒸馏
[0091]
2.1多尺度蒸馏
[0092]
该蒸馏损失应该在强约束(太强的约束,导致无法学习新类)和弱约束(松散的约束,导致灾难性地忘记旧类)之间进行适当的权衡,来实现可持续性学习。多尺度蒸馏包括在旧模型和当前模型之间的不同特征水平上匹配全局统计数据。设x表示大小为h
×w×
c的嵌入张量。提取蒸馏嵌入ω包括连接x的h
×
c宽度合并切片和w
×
c高度合并切片:
[0093][0094]
其中,h,ω为张量索引;[
·
||
·
]表示通道轴上的串联。对于旧模型和当前模型,这种嵌入是在几个特征层上计算的。然后,pod损耗包括在当前网络参数ω
t
上最小化两组嵌入之间的l2距离:
[0095][0096]
其中,t为分类类别,i为当前提取的特征层数,l为特征层总数;
[0097]
该方法能够约束空间统计信息,因此在分类的持续学习环境中,该方法可以产生最先进的结果,多尺度蒸馏的过程如图5所示。由于语义分割需要更高的空间精度,对整个宽度或高度的统计信息进行建模会导致对较小对象非常重要的局部统计信息变得模糊,所以蒸馏方案应保持长程和短程空间关系,因此局部蒸馏特征提取方案,包括计算在不同尺度{1/2s}
s=0
…s下提取的多个区域上的宽度和高度合并切片。对于尺寸为h
×w×
c的嵌入张量x,在比例为1/2s时,将比例处的局部蒸馏特征嵌入ψs(x)计算为嵌入的串联:
[0098][0099]
其中是嵌入张量x的一个子区域,大小为w/s
×
h/s。然后(沿通道轴)连接每个标度s的局部蒸馏特征嵌入ψs(x),形成最终嵌入:
[0100]
ψ(x)=[ψ1(x)||
…
||ψs(x)]∈r
(h+w)
×c×sꢀꢀ
(7)
[0101]
将旧模型和当前模型的几层局部蒸馏特征嵌入。最终局部蒸馏损失为:
[0102][0103]
综上所述,该蒸馏方案不仅能约束空间统计信息,还可以对长期相关性进行建模,而且局部蒸馏允许保留长程和短程空间关系,提高了空间精度。
[0104]
2.2使用旧模型的输出作为标记
[0105]
在训练时,被标记为背景的像素可以属于旧类(包括背景类c
bg
)或新类。因此,将它们作为背景会导致模型遗忘已学习的知识。相反,使用背景像素的标记策略来处理背景偏移。旧模型输出通常用于语义分段的域适配,其中模型根据源数据集的真实标签和分配给未标记目标数据集的伪标签的并集进行训练。
[0106]
2.2.1旧模型标签
[0107]
形式上,让c
t
=card(c
t
)-1表示当前类(不包括后台类)的基数。让表示当前模型的预测(包括真实背景类、所有旧类以及当前类)。让将目标作为步骤step,使用步骤step中的one-hot ground-truth分割图以及使用旧模型预测提取的标签进行计算,如下所示:
[0108][0109]
其中,ω,h为当前张量的长度和高度,c
′
为当前学习的类别,c
bg
为当前图片中的背景类;p
t
(ω,h,c
bg
)为步骤t时模型对先前类别的分割图,p
t
(ω,h,c
′
)为步骤t时模型对当前学习类别的分割图,为步骤t-1时模型对当前学习类别的分割图,为步骤t时模型对目前学习的总类别的分割图。
[0110]
2.2.2标签计算
[0111]
在非背景像素的情况下,复制ground truth标签。否则,将使用旧模型g
t-1
(f
t-1
(
·
))预测的类别。这种标签策略允许将标记为背景的每个像素指定为其真正的语义标签(如果该像素属于任何旧类)。如果所有背景像素可能是无效的,那么在旧模型上可能存在失败的不确定像素。因此,只保留旧模型足够“自信”的标签。考虑到这种不确定性,将式(9)修改为:
[0112][0113]
其中u表示像素(ω,h)的不确定性,τc是特定于类别的阈值。因此,丢弃旧模型不确定的所有像素(u≥τc)并将归一化因子wh减小1,这样可以避免背景偏移导致知识遗忘。
[0114]
3训练和测试模型
[0115]
3.1模型结构描述
[0116]
本发明对脑肿瘤mri图像分割的模型采用deeplabv3+网络,如图3所示,该网络包含编码器g和解码器f,编码器g可以逐步减少特征图并提取更高语义信息,解码器f用来逐步恢复空间信息,将两部分的特征提取信息合并可以获得更清晰的分割结果,deeplabv3+模型为公共开源模型。
[0117]
3.2损失函数
[0118]
使用熵作为测量不确定度u。在学习任务t之前,计算旧模型的中值熵,该熵覆盖所有先前类别c预测为c的d
t
的所有像素∈c
1:t-1
,提供阈值τc∈c
1:t-1
。旧类的伪标记交叉熵损失可以写成:
[0119]
[0120]
其中,v是可接受的旧类像素与此类像素总数的比率,在总损失中自适应地加权伪标记的重要性。
[0121]
综上所述,本发明的总损失为:
[0122][0123]
其中λ是一个超参数,l
localpod
为局部蒸馏损失。
[0124]
3.3训练和测试过程
[0125]
3.3.1训练过程
[0126]
deeplabv3+网络可以表示为g
·
f,g为编码器,f为解码器,训练过程如图4所示,训练集分为3个子集,即坏疽子集c1、浮肿子集c2和增强肿瘤子集c3;设置超参数:类别数class为4,epoch为60,学习率lr为0.0875,动量为0.9,采用adam优化器进行参数优化,式(12)的总损失为损失函数来计算模型预测值和真实值的差异。
[0127]
1)、将坏疽子集c1输入到deeplabv3+网络进行训练,获得可以识别分割背景区域与坏疽区域的模型g0·
f0;
[0128]
2)、将坏疽子集c1和浮肿子集c2作为输入数据集,对模型g0·
f0按步骤2.1进行多尺度蒸馏训练操作和按步骤2.2进行伪标签处理,从而获得可以识别分割背景区域、坏疽区域和浮肿区域的模型g1·
f1。
[0129]
步骤2.1多尺度蒸馏训练操作的目的是将模型g0·
f0从坏疽子集c1上学习到的参数通过知识蒸馏保存到模型g1·
f1,避免了因坏疽子集c1和浮肿子集c2数据量的不平衡而导致的分割精度不足。
[0130]
步骤2.2伪标签处理的目的是因为浮肿子集c2中也有坏疽区域,将g0·
f0对浮肿子集c2中坏疽区域的分割结果作为伪标记,避免了因学习浮肿类别而对坏疽类别的遗忘。
[0131]
3)、将浮肿子集c2和增强肿瘤子集c3别作为输入数据集,对模型g1·
f1按步骤2.1进行多尺度蒸馏训练操作和按步骤2.2进行伪标签处理,从而获得可以识别分割背景区域、坏疽区域、浮肿区域和增强肿瘤区域的模型g2·
f2,即为完整模型。
[0132]
步骤2.1多尺度蒸馏训练操作的目的是将模型g1·
f1从坏疽子集c1和浮肿子集c2上学习到的参数通过知识蒸馏保存到模型g2·
f2,避免了因坏疽子集c1、浮肿子集c2和增强肿瘤子集c3数据量的不平衡而导致的分割精度不足。
[0133]
步骤2.2伪标签处理的目的是因为增强肿瘤子集c3中也坏疽区域和浮肿区域,将g1·
f1对增强肿瘤子集c3中坏疽区域和浮肿区域的分割结果作为伪标记,避免了因学习浮肿类别而对坏疽类别的遗忘。
[0134]
3.3.2模型的测试过程:
[0135]
将步骤1.3获得的测试集数据输入步骤3.3.1经过多尺度蒸馏训练后获得的模型g2·
f2(即多尺度蒸馏训练后的deeplabv3+网络)中,预测输出为大小[160,160]的矩阵,其中背景区域数值为0,坏疽区域数值为1、浮肿区域数值为2、增强肿瘤区域数值为4。将预测结果与对应的标签图像计算准确率,经计算的评价指标结果如下:
[0136]
(1)、dice系数为0.71,0.83,0.73;dice相似系数计算公式为:
[0137]
[0138]
其中tp为真正例的个数,fp为假正例的个数,fn为假反例的个数,tn为真反例的个数。
[0139]
(2)、灵敏度sensitivity为0.76,0.88,0.81;灵敏度sensitivity可用于评估真阳性和假阴性的数量,用于测量模型对分段区域的灵敏度,定义为:
[0140][0141]
(3)、特异性specificity为0.99,0.99,0.99;特异性specificity可以用来评估真阴性和假阳性的数量,它用来衡量模型预测背景区域的能力,定义为:
[0142][0143]
(4)、hausdorff95距离为17.3,5.3,6.9;hausdorff95是距离测量实际区域表面和预测区域之间的距离,预测区域对分段边界更敏感,定义为:
[0144][0145]
其中sup表示上确界,inf表示内确界,t和p表示地面真值区域表面t上的点和预测区域表面p上的点,d(
·
,
·
)是点t和p之间距离的函数。
[0146]
统计评价指标已到达训练目标,获得可在线使用的deeplabv3+模型。
[0147]
最后将预测数据生成.nii.gz文件,使用itk-snap软件展示预测效果,其中一个测试样例如图6所示。
[0148]
4在线使用
[0149]
在实际生产过程中,采集脑肿瘤病人的多模态mri图像,多模态mri图像的输入格式为[c,h,w],c代表通道数,h代表图像矩阵的高,w代表图像矩阵的宽;将多模态mri图像按照步骤1.2进行预处理,将预处理后的图像输入到步骤3.3.2测试验证好的deeplabv3+模型,预测输出分割结果,包括采用不同灰度进行区域描述,分别为背景区域、坏疽(net)区域、浮肿区域(ed)和增强肿瘤区域(et)。
[0150]
实验:
[0151]
以实施例1的brats2020数据集的作为实验数据,采用deeplabv3+、unet网路(ronneberger o,fischer p,brox t.u-net:convolutional networks for biomedical image segmentation[c]//international conference on medical image computing and computer-assisted intervention.springer,cham,2015:234-241.)、vnet网络(milletari f,navab n,ahmadi s a.v-net:fully convolutional neural networks for volumetric medical image segmentation[c]//2016fourth international conference on 3d vision(3dv).ieee,2016:565-571.)和mib网络(cermelli f,mancini m,bulo s r,et al.modeling the background for incremental learning in semantic segmentation[c]//proceedings of the ieee/cvf conference on computer vision and pattern recognition.2020:9233-9242.)作为实验对比网络,实验统计评判指标采用dice系数、灵敏度sensitivity、特异性specificity和hausdorff95距离,分别进行以下3项对比实验:
[0152]
1)、deeplabv3+分别采用常规的训练方法和本发明的多尺度蒸馏的训练方法,比较两者之间的评价指标,实验设置参数如下
[0153]
模型(mdoel)训练方案数据集(dataset)学习率(lr)轮次(epoch)动量(p)deeplabv3+常规brats20200.0041470.9deeplabv3+多尺度蒸馏brats20200.0875300.9
[0154]
说明:以上是模型经过训练调优后的最佳超参数。
[0155]
实验对比如下
[0156][0157]
其中,ed代表浮肿区域、et代表增强肿瘤区域、net代表坏疽区域。由上表可知,deeplabv3+采用多尺度蒸馏方案比常规训练方案后准确率和性能有明显的提升。
[0158]
2)、使用deeplabv3+、unet、vnet、mib均采用多尺度蒸馏的训练方案,比较这4个模型的评判指标;实验设置参数如下
[0159][0160]
说明:由于各模型采用不同的结构,其最佳超参数的数值往往不相同,以上是各模型经过训练调优后的最佳超参数。
[0161]
实验结果如下
[0162][0163][0164]
由上表可知,模型均采用多尺度蒸馏方案后的前提下,deeplabv3+取得了较好的结果,相比于医学图像中分割效果最好的unet网络,在少部分指标上效果追平,其余指标上
有明显的优势。
[0165]
3)、deeplabv3+采用多尺度蒸馏的训练手段,unet、vnet、mib均采用常规训练手段,实验设置参数如下
[0166][0167]
说明:由于各模型采用不同的结构,其最佳超参数的数值往往不相同,以上是各模型经过训练调优后的最佳超参数。
[0168]
实验结果如下:
[0169][0170]
由上表可知,deeplabv3+采用多尺度蒸馏的训练手段,unet、vnet、mib均采用常规训练手段的前提下,deeplabv3+取得了明显的结果。
[0171]
综上所述,本发明仅采用了脑肿瘤患者t1,t1c,t2和flair四种模态的核磁共振图像进行肿瘤分割,并采用.npy格式进行存贮,方便压缩降低存储成本能;此外,本发明将整个脑肿瘤分割为背景区域、坏疽(net)区域、浮肿区域(ed)和增强肿瘤区域(et),能够获取对gbm具有重要预测作用的高层次影像特征。
[0172]
最后,还需要注意的是,以上列举的仅是本发明的若干个具体实施例。显然,本发明不限于以上实施例,还可以有许多变形。本领域的普通技术人员能从本发明公开的内容直接导出或联想到的所有变形,均应认为是本发明的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1