一种基于深度学习的医学高光谱图像微流控单细胞识别方法
1.本发明提供一种基于深度学习的医学高光谱图像微流控单细胞识别方法,属于细胞检测技术领域。
背景技术:
2.细胞是生物学中构成生物体的最基本单位,单细胞识别技术是医疗辅助诊断中的重要方法,可以定性定量的诊断病情,在医学临床中:血液中红、白细胞分析、癌细胞识别、淋巴细胞识别、免疫细胞识别等等对疾病的预防、指导治疗方面起到了重要的作用。
3.病毒的检测成为人们对抗疫情不可或缺的一部分,其中通过免疫学检测方法可以检测出人体内是否被新冠病毒入侵,即检测人体内是否含有由于对抗病毒而产生特异性抗体免疫球蛋白g和免疫球蛋白m的细胞。常用的检测细胞内抗体的方法是化学发光免疫分析法,即使用特定的酶或者化学发光剂来标记细胞内的特定蛋白,即抗原或抗体形成免疫复合物,其中免疫复合物产生的发光信号被化学发光仪检测出来。
4.传统依赖于荧光信号探测的细胞检测技术价格昂贵,操作过程复杂,探测过程中存在信号猝灭、信噪比不理想、实验环境干扰的问题;其余方法如质谱法、pcr、基因芯片、基因探针等检测技术需要裂解细胞且无法实现单细胞的分析鉴定;单细胞测序实现了单细胞的dna和rna序列测定,但该方法成本较高,限制样本制备数量。同时这些基于细胞形态特征、血清学反应特征、生理生化反应特征的细胞识别检测技术都需要人工标记或操作,判断时也非常依赖医生的主观经验,不但耗时耗力,还容易造成误判。
技术实现要素:
5.针对上述技术问题,本发明通过数字图像处理技术,提供一种智能化程度高、快速、无损、免标记、高通量的单细胞精准识别检测技术,结合微流控技术下的细胞显微图像,其具有以下特点:
6.1)先验信息丰富:结合微流控技术拍摄的显微高光谱图像具有很多先验信息,如显微镜的放大倍数固定,拍摄距离固定,不同图像视野中的微流控板凹槽大小、凹槽间隔基本固定;凹槽为正方形,拍摄时保证凹槽的边缘与图像边缘平行。
7.2)细胞分散:微流控板将细胞分散到多个不同的凹槽内,为单细胞识别提供可能。
8.3)目标占比小:细胞在整张显微高光谱图像中所占的比例非常小,一张图片平均会出现十几到二十几个凹槽中包含有效细胞,一个细胞的直径大约在十几个像素左右,而获取的显微高光谱图像大小为696
×
520,细胞占比面积约为图片的2%左右,如果逐像素分类来定位、区分细胞会造成大量计算资源的浪费。
9.具体的技术方案为:
10.一种基于深度学习的医学高光谱图像微流控单细胞识别方法
11.s1、数据采集
12.细胞捕获过程由微流控芯片实现,通过电脉冲来实现捕单细胞捕获;制片后,将芯
片安放至显微高光谱图像采集系统,进行数据采集,凹槽边缘与视野边缘平行。
13.s2、数据标记
14.用envi软件对在凹槽内部、形状规则,轮廓鲜明的细胞数据进行标记,输出分类标签图像,作为后续训练卷积神经网络分类模型的训练数据标签。
15.s3、细胞定位
16.s3.1、数据预处理
17.首先进行波段选择:soc710高光谱成像获取光谱范围在400nm-1000nm之间波段的光谱图像去除无法提供足够有效信息的噪声大波段,选取清晰的波段作为使用波段。然后根据高光谱图像波段的方差图曲线,使用pca主特征提取法将图像降维至3个通道,通过高斯滤波去除图像中白噪声。
18.s3.2、模板匹配
19.随机选取预处理后的高光谱图像,截取模板,选用标准相关性系数匹配方法对经过pca的三个主要波段分别进行模板匹配,进行多目标识别,阈值设置为0.55,叠加并去除重复识别结果。储存所有识别出方框位置,方框内部为感兴趣区域,为1或0,分别对应细胞像素或凹槽内背景像素;外部为100为非感兴趣区域,输出mask,灰色部分为灰度值为100的非感兴趣区域,黑色部分为凹槽内感兴趣区域,包含灰度值为0的凹槽内背景和灰度值为1的细胞像素点。
20.对于不理想的识别结果,进一步结合边缘投影定位法改进识别结果,补全模板匹配后未识别出的凹槽。同时感兴趣区域变为规则的正方形,编号、保存并输出每个正方形左上角顶点坐标位置,以上工作定位出显微高光谱图像中的凹槽位置。
21.s3.3、凹槽中背景、细胞分离
22.定位出凹槽后,选取特殊波段分离二者像素。分别计算凹槽内部背景和细胞的类内间距和类间间距:
23.类内距离(intra-class):同一类各模式样本点间的均方距离:
[0024][0025]
其中,为类中的样本,为类别中的样本数类间距离(inter-class):类与之间的距离表示为:
[0026][0027]
计算每一个波段的类内距离和类间距离,然后做比,即:
[0028][0029]
根据k值给波段排序,排序后越靠前的波段类内距离越小、和其他类别类间距离最大,综合类别1、类别2、两类k值求和结果以及信噪比,选择波段为分离细胞和凹槽内背景的中心波段,输出二者像素直方图,确定灰度分割阈值,采取闭运算去除噪声,同时给不同凹槽内部的细胞赋予不同编号,输出mask。
[0030]
s4、细胞分类
[0031]
s4.1、数据准备
[0032]
取图片中人工标记的细胞像素位置对模型进行训练,测试集定位出的细胞像素;
[0033]
按照像素点分类,在图像上裁剪patch,裁剪出图像类别标签与中心像素点图像类别相同,选取patch的方法:选取16
×
16大小的patch再分别向四周做镜像;
[0034]
训练集根据人工标记的细胞位置,取两类细胞的像素点,测试集根据自动定位的细胞位置,裁切patch,然后进行拼接,随机排序得到训练、测试数据。
[0035]
s4.2、卷积神经网络搭建
[0036]
利用深度学习方法对细胞进行分类,搭建卷积神经网络模型,模型将50层的32
×
8组的resnext网络与注意力模块相结合:最初的嵌入层内核大小为3
×
3,步幅为1,接着是三个连续的残差块,以提取显微高光谱图像的空间和光谱特征。在残差块后面加入一个注意力模块;注意力模块的输出后由一个3d自适应平均池化层生成4096
×1×
1特征向量,4096个节点通过全连接层到每种样本类别可能性输出。
[0037]
s4.3、模型训练与测试
[0038]
对细胞像素进行二分类,训练后得到测试集分类结果;
[0039]
s4.4、实现细胞层面分类:
[0040]
卷积神经网络分类器训练完毕并进行相应权重的保存,完成细胞自动定位后,将自动测试集数据输入进训练好的神经网络模型进行分类。统计每一个凹槽内部所有像素的分类结果,取相比较多的分类结果作为编号所对应的微流控芯片中细胞的种类。
[0041]
本发明具有的技术效果:
[0042]
1、细胞定位阶段:首先对细胞的显微高光谱图像进行预处理,去除噪声大的波段,然后应用pca提取主特征波段;充分利用微流控芯片的形态学先验信息:芯片上凹槽等大、等间距、与图像边缘平行,通过模板识别和边缘投影定位的形态学方法定位住微流控芯片上的凹槽;通过分析细胞和凹槽内背景的类内间距、类间间距筛选出两类差异大的光谱波段,波长范围约在460nm-465nm之间,用灰度值阈值分割实现快速分离细胞、背景,识别精度可以达到95%。
[0043]
该方法区别于传统方法的优势是:由于细胞像素在整张图片上的占比很小,用传统语义分割方法或按像素分类方法会耗费大量的时间去训练分类模型,在计算时也会耗费大量计算资源,而本发明使用的细胞、背景分离方法是一种免人工标记、免训练的无监督分割方法,利用微流控芯片的形态学优势定位凹槽,利用高光谱图像包含丰富的光谱信息筛选特殊波段,可以实现迅速细胞、背景分离。
[0044]
2、细胞分类阶段:根据细胞定位的基础,结合训练的深度学习网络,可以实现在细胞层面上的分类。在数据预处理过程中,考虑细胞显微高光谱图像自身特点,充分利用上下文信息,裁剪patch并对patch做镜像输入网络;在网络搭建过程中将嵌入层卷积核从5
×
5改成3
×
3,更好地适应32
×
32大小的图像,使用rrelu函数来增加模型的非线性,采用了resnext50网络结构并加宽网络,结合注意力模块提取更深层次的特征;训练过程中,采用kaiming正态分布初始化方法,使用mix-up数据增强方法提升模型分类精度,最终像素分类精度可以达到97.5%。通过前期的细胞定位工作,为不同凹槽内部的细胞赋予不同标签,进而在像素分类的基础上进一步实现细胞层面的分类,分类精度达98.3%,跟像素分类结果
相比有所提升,且随着样本量的增大这种优势会更加明显,表1展示了测试集细胞识别的最终结果。
[0045]
表1测试集细胞识别结果
[0046][0047]
该方法区别于传统方法,根据医学图像的自身特点调整深度学习网络结构和训练策略,提升基于卷积神经网络的深度学习方法对医学图像像素分类的分类精度;同时,从实际应用角度出发,最终给出的结果是在细胞层面给出分类结果,提高模型的容错率,且理论上可以达到100%对细胞的正确分类。
[0048]
结合微流控技术实现单细胞捕获后,通过细胞定位和细胞分类两个阶段完成了自动化细胞识别工作,初步实现了无接触、免标记、自动化的迅速单细胞高精度识别。
附图说明
[0049]
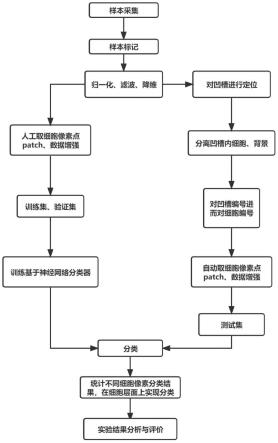
图1为本发明的单细胞识别的整体实验流程;
[0050]
图2为实施例的微流控芯片凹槽模板;
[0051]
图3为实施例的模板识别输出mask;
[0052]
图4为实施例的不理想的识别结果;
[0053]
图5(a)为实施例的边缘投影定位原图;
[0054]
图5(b)为实施例的横向投影效果;
[0055]
图5(c)为实施例的纵向投影排列效果;
[0056]
图5(d)为实施例的边缘投影定位后排列效果;
[0057]
图6为实施例的波段15两类像素点直方图;
[0058]
图7(a)为实施例的原图;
[0059]
图7(b)为实施例的阈值分类结果;
[0060]
图7(c)为实施例的开运算后结果;
[0061]
图8为实施例的patch方法结果;
[0062]
图9为实施例的卷积神经网络结构;
[0063]
图10为实施例的残差层结构。
具体实施方式
[0064]
结合实施例说明本发明的具体技术方案。
[0065]
一种基于深度学习的医学高光谱图像微流控单细胞识别方法,如图1所示的流程:
[0066]
s1、数据采集
[0067]
细胞捕获过程由微流控芯片技术实现,其通过电脉冲来实现捕单细胞捕获,具有
高通量、集成性、操作便利、检测快速等优势。制片后,将芯片安放显微高光谱图像采集系统,调整显微镜光源和成像仪曝光时间,将细胞内光强稳定在2500-3000之间以尽可能保留细胞图像光谱信息,移动芯片和调节显微镜焦距来选择视野清晰明亮,且包含细胞数目多的区域进行数据采集,尽量保持凹槽边缘与视野边缘平行。
[0068]
s2、数据标记
[0069]
如图2所示,用envi软件对在凹槽内部、形状规则,轮廓鲜明的细胞数据进行标记,输出分类标签图像,作为后续训练卷积神经网络分类模型的训练数据标签。
[0070]
s3、细胞定位
[0071]
s3.1、数据预处理
[0072]
首先进行波段选择:soc710高光谱成像获取光谱范围在400nm-1000nm之间128个波段的光谱图像去除无法提供足够有效信息的噪声大波段,选取较为清晰的15-80共66个波段作为使用波段。然后根据高光谱图像波段的方差图曲线,使用pca主特征提取法将图像降维至3个通道,其中包含了图像绝大部分信息,然后通过高斯滤波去除图像中白噪声。
[0073]
s3.2、模板匹配
[0074]
随机选取预处理后的高光谱图像,截取模板,选用标准相关性系数匹配方法对经过pca的三个主要波段分别进行模板匹配,进行多目标识别,阈值设置为0.55,叠加并去除重复识别结果。储存所有识别出方框位置,方框内部为感兴趣区域,为1或0,分别对应细胞像素或凹槽内背景像素;外部为100为非感兴趣区域,输出mask如图3所示,灰色部分为灰度值为100的非感兴趣区域,黑色部分为凹槽内感兴趣区域,包含灰度值为0的凹槽内背景和灰度值为1的细胞像素点(由于灰度值相差太小,此处肉眼不便识别)。
[0075]
对于如图4所示不理想的识别结果,进一步结合边缘投影定位法改进识别结果。由于微流控芯片中凹槽有固定位置,且凹槽边缘也与图像边缘垂直,有较为清晰的间距,通过模板匹配得到的mask也具有较为固定的位置和间距,同时与边缘垂直,故采用边缘投影定位方法补全模板匹配后未识别出的凹槽。如图5(a)到图5(d)所示,经过进一步处理后的mask补全了之前未识别凹槽,同时感兴趣区域变为规则的正方形,编号、保存并输出每个正方形左上角顶点坐标位置,以上工作定位出显微高光谱图像中的凹槽位置。
[0076]
s3.3、凹槽中背景、细胞分离
[0077]
定位出凹槽后,要进一步明确细胞的位置,需分离凹槽中的背景与细胞。由于凹槽内背景和细胞成分差异较大,易于区分,所以选取特殊波段分离二者像素。分别计算凹槽内部背景和细胞的类内间距和类间间距:
[0078]
类内距离(intra-class):同一类各模式样本点间的均方距离:
[0079][0080]
其中,为类中的样本,为类别中的样本数类间距离(inter-class):类与之间的距离可以表示为:
[0081][0082]
计算每一个波段的类内距离和类间距离,然后做比,即:
[0083][0084]
根据k值给128个波段排序,排序后越靠前的波段类内距离越小、和其他类别类间距离最大,综合类别1、类别2、两类k值求和结果以及信噪比,最终选择波段15为分离细胞和凹槽内背景的中心波段,即在460nm-465nm之间,细胞与背景的光透射率差异较大。输出二者像素直方图如图6所示,确定灰度分割阈值,图7(a)为原图,单波段灰度分割结果如图7(b)所示,采取闭运算去除噪声,如图7(c),同时给不同凹槽内部的细胞赋予不同编号,输出mask。
[0085]
s4、细胞分类
[0086]
s4.1、数据准备
[0087]
共采集3张cd45细胞图片、4张sp20细胞图片。划分情况如表2所示:为了更好地模拟实际情况,即在模型设计、训练完成后,输入模型的应为全新数据,拍摄环境与训练集存在微小差异,所以将训练集、验证集分为一组,样本数量占比分别占该组0.9、0.1,测试集则与他们完全分离,最终效果以测试集的分类效果为准。由于测试集的部分已经完成细胞定位,所以只需要取图片中人工标记的细胞像素位置对模型进行训练,经统计,训练集共5091个样本,验证集900个样本,测试集定位出的细胞像素共5079个样本。
[0088]
表2训练集、验证集、测试集划分情况
[0089][0090]
按照像素点分类,需要在图像上裁剪patch,裁剪出图像类别标签与中心像素点图像类别相同,考虑单个细胞所占像素大小,采用如图8所示,框内为16
×
16大小patch,经过镜像翻转得到32
×
32patch大小,在充分结合上下文信息的同时也突出了中心细胞像素点所占整张patch的比例,有利于分类效果提升。
[0091]
训练集根据人工标记的细胞位置,只取两类细胞的像素点,测试集根据自动定位的细胞位置,裁切patch,cd45细胞设置标签为0,sp20细胞设置标签为1,然后进行拼接,随机排序得到训练、测试数据。
[0092]
s4.2、卷积神经网络搭建
[0093]
利用深度学习方法对细胞进行分类,搭建卷积神经网络模型,模型将50层的32
×
8组的resnext网络与注意力模块相结合,具有注意力网络的架构如图9所示:最初的嵌入层内核大小为3
×
3,步幅为1,紧接着是三个连续的残差块,每个残差块构成的层结构如图10所示以提取显微高光谱图像的空间和光谱特征。在残差块后面加入一个注意力模块以提高模型的表示能力。注意力模块的输出然后由一个3d自适应平均池化层生成4096
×1×
1特征向量,4096个节点通过全连接层到每种样本类别可能性输出。
[0094]
s4.3、模型训练与测试
[0095]
卷积神经网络模型训练的实验软硬件环境如表3所示:
[0096]
表3实验软硬件环境
[0097][0098]
其中,模型训练过程中的超参数如表4所示:
[0099]
表4实验训练超参数
[0100][0101]
对cd45,sp20两类细胞像素进行了二分类,训练过程中,测试集两类共有两千多个像素点,训练100轮次进行测试,共训练了3次,得到测试集分类结果如表5所示。
[0102]
表5分类结果统计
[0103][0104]
s4.4、实现细胞层面分类:
[0105]
卷积神经网络分类器训练完毕并进行相应权重的保存,完成细胞自动定位后,将自动测试集数据输入进训练好的神经网络模型进行分类。统计每一个凹槽内部所有像素的分类结果,取较多的分类结果作为编号所对应的微流控芯片中细胞的种类。在细胞层面上分类而非在像素层面上分类可以提高整体模型的容错率,即使会分错个别像素,但是取分类结果的众数还是可以正确地对凹槽内的细胞分类。
[0106]
本发明充分利用显微高光谱图像包含丰富的光谱信息,利用模板识别、阈值分割、深度学习中基于卷积神经网络的分类算法完成了单细胞高精度、免标记、自动化快速识别;本发明针对细胞的显微高光谱图像的特点及存在问题,做了以下工作:
[0107]
1)128个通道的显微高光谱图像有些波段噪声大,冗余信息多,本发明选取信噪比高的15-80波段,在模板识别微流控芯片凹槽时用pca特征提取、高斯滤波的方法处理图像,
降低噪声的影响并去除冗余信息,提高识别效果和计算速度。
[0108]
2)芯片上凹槽内部的细胞为实验感兴趣区域,在细胞的定位过程中充分利用先验信息,利用正方形凹槽、固定间距在形态学上的优势,采取模板识别方法对含有细胞的凹槽编号,再进一步定位出细胞,加速细胞定位的过程;在分离细胞和凹槽内背景时,计算细胞和凹槽内背景类内间距和类间间距,选取两类样本差异较大的敏感波段,大约在670nm左右,并根据输出的样本灰度值直方图确定阈值,直接根据单波段图像灰度值进行细胞和背景的快速分离,提高细胞定位过程的效率;实现在细胞水平上分类,即在定位细胞后统计一个凹槽内部细胞所有的像素分类结果,选取占比最大的分类结果作为细胞层面的分类结果,提高模型的容错性。
[0109]
3)训练高精度的细胞分类器过程中,由于整张医学图像过大,包含的信息过于复杂,细胞的所占的像素点数量较少,故选择细胞像素点作为中心像素点,裁剪16
×
16像素块patch后做镜像处理,充分结合上下文信息,以32
×
32大小的patch输入神经网络进行分类,选取mix-up数据增强方法来提高模型的泛化能力和鲁棒性;根据5000个样本点左右的数据量搭建合适深度的神经网络,本发明最终采用resnext50网络,8
×
32的分组卷积,网络首层卷积核大小为3
×
3,不进行下采样,增加bam注意力模块的神经网络,调节神经网络参数使神经网络更好的适应医学图像数据,最终达到98.3%的细胞分类精度。
[0110]
本发明根据获取的细胞显微高光谱图像数据特点对图像进行处理,将深度学习算法迁移到医学高光谱图像不同的应用背景中,为医学提供一种快速、精准、免标记的单细胞识别新手段。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1