用于组表征的方法和系统与流程
本申请是于2017年5月26日提交的美国申请序列号15/606,743(其是2015年10月21日提交的序列号为14/919,614的美国申请的接续)的部分接续申请,其要求于2014年10月21日提交的序列号为62/066,369的美国临时申请、于2014年12月04日提交的序列号为62/087,551的美国临时申请、于2014年12月17日提交的序列号为62/092,999的美国临时申请、于2015年4月14日提交的序列号62/147,376的美国临时申请、于2015年4月14日提交的序列号为62/147,212的美国临时申请、于2015年4月14日提交的序列号为62/147,362的美国临时申请、于2015年4月13日提交的序列号为62/146,855的美国临时申请以及于2015年8月18日提交的序列号为62/206,654的美国临时申请的权益,这些申请各自通过引用整体并入本文。本申请还要求于2017年9月16日提交的美国临时申请序列号62/395,939,2017年6月15日提交的美国临时申请序列号62/520,058,和2017年6月27日提交的美国临时申请序列号62/525,379的权益,这些申请各自通过引用整体并入本文。
本发明总体上涉及微生物学领域,并更具体地涉及微生物学领域中用于表征病症组的新的且有用的方法和系统。
附图说明
图1a-1b是用于表征病症组的方法的实施方案的变型的流程图表示;
图2是用于表征病症组的方法的实施方案的变型的流程图表示;
图3是系统的实施方案的示意图表示;
图4是方法的实施方案的变型的示意图表示;
图5是用于表征病症组的方法的变型中的过程的示意图表示;
图6是用于确定目标分类单位的优化参数的实例的图表表示;
图7是表征过程的验证的实例的图形表示;
图8是健康参考相对丰度范围的实例的图表表示;
图9a-9b是目标分类单位的实例;
图10是选择用于表征的益生菌的实例;
图11-12是益生菌和相关分类单位组的实例;
图13a-13b是与益生菌有关的分类单位组关联的相对丰度的实例;和
图14-15是界面的实例。
具体实施方式
以下对本发明实施方案的描述无意于将本发明限于这些实施方案,而是旨在使本领域技术人员能够进行和使用本发明。
1、概述
如图3所示,用于表征与微生物有关的分类单位集合相关的病症(例如,与消化道(gut)相关病症)组(例如,多个)的系统200的实施方案能包括分类数据库205,其包括与病症组相关的分类单位集合的参考微生物群系特征(例如,微生物群系组成多样性特征;微生物群系功能多样性特征;微生物群系药物基因组学特征;等);处理系统210(例如,样品处理系统等),其能够操作以从用户(例如,人类受试者、患者、动物受试者、环境生态系统、护理提供者等)收集包括生物材料(例如,核酸材料等)的容器,处理系统210包括能够操作以从生物材料确定用户的微生物序列数据集的测序仪系统;组表征系统220,其能够操作以:基于微生物序列数据集确定用户的分类单位集合的用户微生物群系特征(例如,相对丰度范围),生成用户微生物群系特征与参考微生物群系特征(例如,参考相对丰度范围等)之间的比较,并基于比较确定用户病症组的组表征;和治疗系统230能够操作以基于所述组表征来促进针对所述病症组的病症的治疗(例如,所述治疗能够操作以调节用户微生物群系组成以改善所述病症的状态等)。
方法100和/或系统200能用于基于用户的生物样品,为用户表征跨多个分类单位(例如,跨多个种和属的微生物)的微生物群系组成和/或微生物群系功能多样性,以表征与多个分类单位相关的多个病症。在变型中,方法100和/或系统200能用于基于源自多个用户的多个生物样品,以多路复用的方式基本上同时生成对多个用户的表征。然而,方法100和/或系统200能以与各自通过引用整体并入本文的2015年1月9日提交的美国申请no.14/593,424和2015年10月21日提交的美国申请no.14/919,614中描述的方式类似的任何方式起作用,和/或能以任何合适的方式起作用。方法100和/或系统200能另外或可替代用于促进(例如,提供)治疗(therapies)(例如,治疗(treatments)等)(诸如用于治疗病症组的病症的对用户的治疗措施(例如,基于组表征))和/或执行任何合适的功能。系统200和/或方法100的变型能进一步促进监测和/或调整提供给受试者的此类疗法,例如,通过在整个治疗过程中接受、处理和分析来自受试者的其他样品(例如,用于评估和/或改善来自组的多个病症)。
在实例中,方法100和/或系统200能生成和/或促进对病症组的表征和/或治疗,其包括以下项中的一个或更多个:症状、病因、疾病、失调、微生物群系药物基因组学谱(例如,描述对抗生素的抗性和/或敏感性)和/或与该组病症相关的任何其他合适的方面。该病症组优选包括消化道相关病症组,包括以下项中的任何一种或更多种:肠胃胀气、腹胀、腹泻、胃肠炎、消化不良、腹痛、腹部压痛、便秘、感染、癌症、生态失调、肠易激综合征(ibs)、炎症性肠病(ibd)、溃疡性结肠炎、克罗恩病、腹腔疾病、肠道控制问题(如大便失禁)、乳糖不耐症、憩室病、憩室炎、胃酸反流(如ger、gerd等)、先天性巨结肠症、腹膜粘连、阑尾炎、结肠息肉、食源性疾病、胆结石、胃炎、胃轻瘫、胃肠道出血、痔疮、胰腺炎、溃疡、惠普尔病、佐林格-埃利森综合征、相关病症和/或任何其他合适的消化道相关病症。另外或可替代地,该病症组可包括以下项中的一种或更多种:益生菌有关病症(例如,与益生菌中包含的分类单位组相关,受其影响的和/或另外的与益生菌中包含的分类单位组有关;可用一种或更多种益生菌治疗;等);阴道有关病症(例如、人乳头瘤病毒感染、梅毒、宫颈癌、高级别和低级别的鳞状上皮内病变、性传播感染、宫颈炎、盆腔炎、细菌性阴道病、好氧性阴道炎、先天性不育等);精神和行为病症(例如心理障碍;抑郁症;精神错乱;焦虑症等);与沟通有关病症(例如、表达性语言障碍;口吃;语音障碍;自闭症;声音病症;听力病症;眼睛病症等;);与睡眠有关病症(例如、失眠、睡眠呼吸暂停等);心血管有关病症(例如冠状动脉疾病;高血压等);代谢有关病症(例如糖尿病等)、类风湿病有关病症(例如关节炎等);体重有关病症(如肥胖等);疼痛有关病症;内分泌有关病症;遗传有关病症;慢性病;和/或任何其他合适类型的病症。然而,方法100和/或系统200能以任何合适的方式配置。
2、益处
微生物群系分析能实现对由微生物引起和/或以其他方式与微生物相关的病症组的准确和有效表征和/或治疗供应。该技术能克服常规方法在对病症的表征和/或促进治疗所面临的若干挑战。首先,传统方法需要患者访问一个或多个护理提供者以接收针对病症的表征和/或治疗建议,这相当于与诊断/或治疗之前流逝的时间量相关的低效率和健康风险。其次,传统方法需要执行许多不同的诊断测试来表征病症组,这还能导致效率低下和健康风险。再次,用于人类基因组测序的常规基因测序和分析技术在应用于微生物群系时(例如,其中人类微生物群系能包括比人类细胞多超过10倍的微生物细胞;其中优化样品处理技术能不同;其中表征病症组的缩放样品处理程序能不同;其中病症类型可能不同;其中序列参考数据库能不同;其中微生物群系能在用户的不同身体区域之间变化等)可能是不相容的和/或低效的。第四,测序技术(例如,下一代测序)的出现引发了若非与遗传物质测序相关的速度和数据生成的前所未有的发展将不会存在的技术问题(例如,数据处理问题、以多路复用方式处理的问题、信息显示问题、微生物群系分析问题、治疗预测问题、治疗供应问题等)。系统200和方法100的实例能为至少上述挑战提供技术起源的解决方案。
第一,该技术能通过促进先前不可执行的功能的计算机性能来赋予对计算机相关技术(例如,与病症组的表征和/或促进治疗相关的建模;改善存储、检索和/或处理针对病症组的微生物相关数据的计算效率;与生物样品处理相关的计算处理等)的改进。例如,该技术能基于由于样品处理技术和测序技术的进步而最近可行的技术(例如,利用微生物分类数据库等)计算地生成组表征和/或相关的推荐疗法。
第二,该技术可赋予对处理速度、组表征准确度、微生物群系相关治疗的确定和促进,和/或与病症组有关的其他合适方面的改进。例如,该技术能生成和应用特征选择规则(例如,针对比较、功能、药物基因组学等的微生物群系药物多样性特征选择规则),以从大量潜在的特征(例如,可从大量的微生物群系数据(诸如序列数据)中提取)池中选择优化的特征子集(例如,微生物群系组成多样性特征诸如表示与病症组相关的分类单位组的健康范围的参考相对丰度特征;可与参考相对丰度特征进行比较的用户相对丰度特征;等)用于生成和应用表征模型和/或治疗模型。微生物群系(例如,人类微生物群系、动物微生物群系等)的潜在大小能转化成大量数据,产生如下问题:如何处理和分析大量数据阵列以生成与病症组有关的可行的微生物群系见解。然而,特征选择规则和/或其他合适的计算机可实现的规则能实现更短的生成和执行时间(例如,用于生成和/或应用分类数据库;用于确定组表征和/或相关治疗;等)、模型简化有利于对结果的有效解释、减少过度拟合、在数据源中(例如,生成分类数据库,等)的改进,鉴定和呈现与微生物群系相关的组病症见解(例如,通过收集和处理与越来越多的用户相关的越来越多的数据以提高技术的预测能力)的改进、数据存储和检索(例如,存储与用户和/或用户集合相关的特定模型、微生物序列、特征和/或其他合适的数据以改善针对病症组的个性化表征和/或治疗的递送等)的改进,和其他合适的以促进表征和/或治疗的快速确定的改进。
第三,该技术能将实体(例如,用户、生物样品、包括医疗设备的治疗系统等)转化成不同的状态或事物。例如,该技术能将生物样品转化为多种病症的组表征。在另一个实例中,系统200和/或方法100能确定治疗以促进患者改变微生物群系组成、微生物功能多样性、微生物群系药物基因组学谱和/或其它微生物群系相关方面从而预防和/或改善病症组的一种或更多种病症,从而改变患者的微生物群系和/或健康。在另一个实例中,该技术能将从患者接收的生物样品转化为(例如,通过片段化、多重扩增、测序等)微生物群系数据集,其随后可将其转化为与病症组相关的特征。以用于生成组表征模型和/或治疗模型。在另一个实施例中,该技术能控制治疗系统以促进治疗(例如,通过生成用于治疗系统进行执行的控制指令),由此转化治疗系统。在另一个实例中,计算机相关技术的改进能推动生物样品处理方法(诸如选择与病症组相关的遗传靶标相容的引物子集)的转变。
第四,该技术能实现在包括分类数据库、样品处理系统、组表征系统和多个用户在内的网络中功能性的创造性分布,其中样品处理系统能同时处理来自多个用户的生物样品(例如,以多路复用的方式),其能与分类数据库一起在对于病症组生成个性化表征和/或治疗(例如,针对用户的微生物群系(诸如用户的饮食行为、益生菌相关行为、病史、人口统计学、其它行为、偏好等)定制)中由组表征系统利用。
第五,该技术能改善至少与微生物群系数字医学、一般数字用药(digitalmedicinegenerally)、基因测序和/或其他相关领域有关的病症组的计算建模的技术领域。第六,该技术能利用专用计算设备(例如,与样品处理系统(诸如测序仪系统;组表征系统;治疗系统等)相关的设备)来确定和处理用于对病症组的表征和/或确定治疗的微生物群系数据集。然而,在使用用于组表征和/或微生物群系调节的非通用计算机系统的情况下,该技术能提供任何其他合适的益处。
3.1、系统-分类数据库
系统200的分类数据库205能用于提供与病症组关联的标志物信息,并且适于在生成一个或更多个组表征时与用户微生物群系特征进行比较。例如,分类数据库205能存储与相应的多个分类单位相关的微生物遗传序列,其能与一个或更多个相应的病症相关地存储。在另一个实例中,分类数据库205能存储与病症组相关的微生物分类单位组的参考相对丰度范围(例如,与一种或更多种病症的健康状态相关,与不健康状态相关等)和/或其他合适的微生物群系特征,其中能基于来自用户群体的生物样品集合提取参考微生物群系特征(例如,展示病症组的一种或更多种病症;不展示病症;等等)。在另一个实例中,分类数据库205能存储用户相对丰度范围(例如,对于具有与病症组有关的未知微生物群系谱的用户;等)和/或其他合适的用户微生物群系特征。
分类数据库205优选地存储包括以下项中任何一种或更多种的标志物:遗传序列(例如,鉴定分类单位组的序列;微生物序列;人序列;表示来自病症组的病症的序列;在微生物分类单位组集合和/或用户中不变的序列;保守序列;包括突变的序列;包括多态性的序列;等等);肽序列;靶标;特征(例如,微生物群系组成多样性特征、微生物群系功能多样性特征、微生物群系药物基因组学特征等);蛋白质类型(例如,血清蛋白质、抗体等);碳水化合物类型;脂质类型;全细胞标志物;代谢标志物;天然产物标志物;遗传易感生物标志物;诊断生物标志物;预后生物标志物;预测生物标志物;其他分子生物标志物;基因表达标志物;成像生物标志物;对应于与微生物相关的功能、结构、进化和/或其它合适特征的标记物;和/或与微生物(例如,分类单位)和/或相关病症相关的其他合适标志物。由分类数据库205存储的遗传序列优选包括rrna(例如,rrna基因序列的可变区)的一个或更多个基因序列,其可包括下项中任何一种或更多种:16s、18s、30s、40s、50s、60s、5s、23s、5.8s、28s、70s、80s和/或任何其他合适的rrna。另外或可替代地,遗传序列可包括以下项和/或以其他方式与以下项相关:其他rna基因、蛋白质基因、其他rna序列、dna序列和/或任何其他合适的遗传方面。由分类数据库205存储的不同标志物优选地共享标志物特征,其能包括以下项中的一种或更多种:跨多个分类单位的保守遗传序列(例如,包括可变区的半保守遗传序列;能通过用于靶向与病症组相关的多个分类单位族的引物靶向的保守序列;等)、保守肽序列、共享生物标志物和/或任何其他合适的标志物相关信息。
经存储的标志物优选地与多个分类单位相关,以便能够基于与经存储的标志物的比较将用户微生物序列(例如,源自用户的收集的生物样品等)映射到特定的分类单位(例如,比较用户微生物序列与经存储标记以找到满足预定条件的匹配;鉴定与经匹配标志物相关的分类单位;以及将分类单位与用户微生物序列相关;等)。与分类数据库205、病症组(例如,消化道相关病症)、其他系统组件和/或系统200和方法100的任何部分有关的分类单位组能包括以下项中的一项或更多项:梭菌(clostridium)(属)、艰难梭菌(clostridiumdifficile)(种)、另枝菌(alistipes)(属)、拟普雷沃菌(alloprevotella)(属)、厌氧细杆菌(anaerofilum)(属)、拟杆菌(bacteroides)(属)、barnesiella(属)、双歧杆菌(bifidobacterium)(属)、blautia(属)、丁酸弧菌(butyricimonas)(属)、弯曲杆菌(campylobacter)(属)、catenibacterium(属)、christensenella(属)、collinsella(属)、粪球菌(coprococcus)(属)、小杆菌(dialister)(属)、伊格尔兹氏菌(eggerthella)(属)、埃希氏志贺杆菌(escherichia-shigella)(属)、粪杆菌(faecalibacterium)(属)、flavonifractor(属)、梭杆菌(fusobacterium)(属)、gelria(属)、嗜血杆菌(haemophilus)(属)、holdemania(属)、乳杆菌(lactobacillus)(属)、odoribacter(属)、颤杆菌(oscillibacter)(属)、颤螺菌(oscillospira)(属)、parabacteroides(属)、paraprevotella(属)、peptoclostridium(属)、考拉杆菌(phascolarctobacterium)(属)、普氏菌(prevotella)(属)、pseudoflavonifractor(属)、罗斯氏菌(roseburia)(属)、瘤胃球菌(ruminococcus)(属)、沙门氏菌(salmonella)(属)、链球菌(streptococcus)(属)、turicibacter(属)、tyzzerella(属)、韦荣球菌(veillonella)(属)、acetobacternitrogenifigens(种)、鲍曼不动杆菌(acinetobacterbaumannii)(种)、阿克曼氏菌(akkermansiamuciniphila)(种)、anaerotruncuscolihominis(种)、巴西固氮螺菌(azospirillumbrasilense)(种)、蜡状芽孢杆菌(bacilluscereus)(种)、凝结芽孢杆菌(bacilluscoagulans)(种)、地衣芽孢杆菌(bacilluslicheniformis)(种)、脆弱拟杆菌(bacteroidesfragilis)(种)、普通拟杆菌(bacteroidesvulgatus)(种)、长双歧杆菌(bifidobacteriumlongum)(种)、动物双歧杆菌(bifidobacteriumanimalis)(种)、两歧双歧杆菌(bifidobacteriumbifidum)(种)、侧孢芽孢杆菌(brevibacilluslaterosporus)(种)、穗状丁酸弧菌(butyrivibriocrossotus)(种)、空肠弯曲杆菌(campylobacterjejuni)(种)、结肠弯曲杆菌(campylobactercoli)(种)、红嘴鸥弯曲杆菌(campylobacterlari)(种)、密执安棍状杆菌(clavibactermichiganensis)(种)、丁酸梭菌(clostridiumbutyricum)(种)、产气柯林斯菌(collinsellaaerofaciens)(种)、规则粪球菌(coprococcuseutactus)(种)、desulfovibriopiger(种)、dialisterinvisus(种)、斜体肠球菌(enterococcusitalicus)(种)、大肠杆菌(escherichiacoli)(种)、大肠杆菌o157(escherichiacolio157)(种)、普拉梭菌(faecalibacteriumprausnitzii)(种)、产琥珀酸丝状杆菌(fibrobactersuccinogenes)(种)、嗜根考克氏菌(kocuriarhizophila)(种)、短乳杆菌(lactobacillusbrevis)(种)、棒状乳杆菌(lactobacilluscoryniformis)(种)、戴耳布吕克氏乳杆菌(lactobacillusdelbrueckii)(种)、发酵乳杆菌(lactobacillusfermentum)(种)、瑞士乳杆菌(lactobacillushelveticus)(种)、开菲尔基质乳杆菌(lactobacilluskefiranofaciens)(种)、lactobacilluskunkeei(种)、鼠李糖乳杆菌(lactobacillusrhamnosus)(种)、唾液乳杆菌(lactobacillussalivarius)(种)、斐济乳球菌(lactococcusfujiensis)(种)、格氏乳球菌(lactococcusgarvieae)(种)、乳酸乳球菌(lactococcuslactis)(种)、leptotrichiahofstadii(种)、leuconostocfallax(种)、泡菜明串珠菌(leuconostockimchii)(种)、史密斯甲烷杆菌(methanobrevibactersmithii)(种)、酒类酒球菌(oenococcusoeni)(种)、产甲酸草酸杆菌(oxalobacterformigenes)(种)、蜂似芽孢杆菌(paenibacillusapiarius)(种)、戊糖片球菌(pediococcuspentosaceus)(种)、peptoclostridiumdifficile(种)、费氏丙酸杆菌(propionibacteriumfreudenreichii)(种)、苍黄假棍状杆菌(pseudoclavibacterhelvolus)(种)、鲑鱼肾杆菌(renibacteriumsalmoninarum)(种)、白色瘤胃球菌(ruminococcusalbus)(种)、生黄瘤胃球菌(ruminococcusflavefaciens)(种)、布氏瘤胃球菌(ruminococcusbromii)(种)、活泼瘤胃球菌(ruminococcusgnavus)(种)、邦戈尔沙门氏菌(salmonellabongori)(种)、肠道沙门氏菌(salmonellaenterica)(种)、波伊德氏志贺氏菌(shigellaboydii)(种)、宋内志贺氏菌(shigellasonnei)(种)、福氏志贺氏菌(shigellaflexneri)(种)、痢疾志贺氏菌(shigelladysenteriae)(种)、松鼠葡萄球菌(staphylococcussciuri)(种)、血链球菌(streptococcussanguinis)(种)、嗜热链球菌(streptococcusthermophilus)(种)、霍乱弧菌(vibriocholerae)(种)、韩国魏斯氏菌(weissellakoreensis)(种)、小肠结肠炎耶尔森菌(yersiniaenterocolitica)(种),和/或任何其他合适的标志物相关信息(例如,分类单位)。另外或可替代地,分类单位组能包括2015年10月21日提交的美国申请no.14/919,614中描述的任何内容。例如,与上述多个分类单位中的一个或更多个相关地储存的标志物能包括与多个分类单位相关的16srrna遗传序列。标志物和/或多个分类单位能与以下项中一种或更多种相关(例如,正相关、负相关等):病症、病原体、共生细菌、益生细菌和/或任何其他标志物相关信息。
在变型中,分类数据库205能存储标志物(例如,微生物序列、丰度特征(诸如相对丰度范围)、微生物群系组成多样性特征、微生物群系功能多样性特征,其他特征等)、相关的分类单位组和/或其他合适的与益生菌有关的数据(和/或其他合适的微生物相关疗法)。照此,分类数据库205能改善益生菌相关数据的存储和/或检索,用于表征与益生菌有关的微生物(例如益生菌中存在的分类组)和/或相关病症(例如,消化道相关病症组和/或其他合适的病症等)相关的用户微生物群系。益生菌的食物来源能包括:牛奶(例如生牛奶)、开菲尔、奶酪(例如,羊奶酪)、可可、泡菜、酸奶、康普茶、德国泡菜、蜂产品、酱菜、纳豆、酱菜、发酵食品(例如,发酵香肠)、其他益生菌食品、益生菌补充剂(例如,益生菌丸、商业益生菌等)和/或其他合适类型的益生菌。
如图11-12和13a-13b所示,与益生菌、病症、其他系统组分和/或系统200和方法100的任何部分相关的分类单位组能包括以下项中一项或更多项:凝结芽孢杆菌(bacilluscoagulans)(种)、动物双歧杆菌(bifidobacteriumanimalis)(种),丁酸梭菌(clostridiumbutyricum)(种)、短乳杆菌(lactobacillusbrevis)(种)、棒状乳杆菌(lactobacilluscoryniformis)(种)、发酵乳杆菌(lactobacillusfermentum)(种)、瑞士乳杆菌(lactobacillushelveticus)、鼠李糖乳杆菌(lactobacillusrhamnosus)(种)、唾液链球菌(streptococcussalivarius)(种)、acetobacternitrogenifigens(种)、巴西固氮螺菌(azospirillumbrasilense)(种)、地衣芽孢杆菌(bacilluslicheniformis)(种)、两歧双歧杆菌(bifidobacteriumbifidum)(种)、侧孢芽孢杆菌(brevibacilluslaterosporus)(种)、密执安棍状杆菌(clavibactermichiganensis)(种)、斜体肠球菌(enterococcusitalicus)(种)、嗜根考克氏菌(kocuriarhizophila)(种)、戴耳布吕克氏乳杆菌(lactobacillusdelbrueckii)(种)、开菲尔基质乳杆菌(lactobacilluskefiranofaciens)(种)、lactobacilluskunkeei(种)、唾液乳杆菌(lactobacillussalivarius)(种)、格氏乳球菌(lactococcusgarvieae)(种)、乳酸乳球菌(lactococcuslactis)(种)、leptotrichiahofstadii(种)、leuconostocfallax(种)、泡菜明串珠菌(leuconostockimchii)(种)、酒类酒球菌(oenococcusoeni)(种)、蜂似芽孢杆菌(paenibacillusapiarius)(种)、戊糖片球菌(pediococcuspentosaceus)(种)、费氏丙酸杆菌(propionibacteriumfreudenreichii)(种)、苍黄假棍状杆菌(pseudoclavibacterhelvolus)(种)、鲑鱼肾杆菌(renibacteriumsalmoninarum)(种)、生黄瘤胃球菌(ruminococcusflavefaciens)(种)、松鼠葡萄球菌(staphylococcussciuri)(种)、停乳链球菌(streptococcusdysgalactiae)(种)、streptococcusparauberis(种)和韩国魏斯氏菌(weissellakoreensis)(种)。在一个具体的实例中,分类数据库205能包括用于特定的分类单位组集合的标志物,所述分类单位包括凝结芽孢杆菌(bacilluscoagulans)(种)、动物双歧杆菌(bifidobacteriumanimalis)(种),丁酸梭菌(clostridiumbutyricum)(种)、短乳杆菌(lactobacillusbrevis)(种)、棒状乳杆菌(lactobacilluscoryniformis)(种)、发酵乳杆菌(lactobacillusfermentum)(种)、瑞士乳杆菌(lactobacillushelveticus)、鼠李糖乳杆菌(lactobacillusrhamnosus)(种)、唾液链球菌(streptococcussalivarius)(种),其中标志物(例如,用于特定的分类单位组集合、用于任何合适的分类单位组集合等)能在生成与对应的益生菌有关的益生菌有关微生物的组表征(例如,组成表征、功能多样性表征)中利用(例如,如图14-15所示)。在与益生菌相关的表征的分类单位组的特定实例中,能包括,对于戊糖片球菌(pediococcuspentosaceus)(种)的分类单位组:在生牛奶、泡菜(kimchi)、德国泡菜(sauerkraut)、酱菜中发现;球形;尺寸0.5-1.0微米;无孢子形成;不运动;无鞭毛;g+;乳酸生产者;用作不同发酵的起始培养物;和/或其他合适的特征。在另一具体实例中,能利用分类数据库来表征与病症集合有关的特定的分类单位组集合和/或其他合适的分类单位组集合,诸如基于与ibs的反相关、与2型糖尿病的反相关、与肥胖的反相关、与ibd的反相关、呼吸感染持续时间的反相关、与体重减轻相关,和/或与任何合适的病症的任何合适的相关(例如,反相关、正相关等)。然而,分类数据库205能以任何合适的方式应用于益生菌。
能通过执行方法100的各部分(例如,框s110)来生成用于存储、检索、确定和/或以其他方式应用的分类数据库205。例如,分类数据库205能包括参考相对丰度范围集合(和/或其他合适的参考微生物群系特征),其来源于:确定与病症组(例如,消化道相关病症等)相关的分类单位的靶标集合、确定参考标志物集合;并且基于参考标志物集合和分类单位的靶标集合之间的比较确定所选择的分类单位集合的参考相对丰度范围的集合。确定参考标志物集合(和/或其他参考微生物群系特征)能包括基于源自基于跨多个分类单位组共享的标志物特征选择的引物集合的预测读数确定参考标志物集合(例如,能提高样品处理的效率以促进组表征,其中相同或相似类型的引物可用于靶向与病症组相关的多个分类单位组的标志物等),其中参考标志物集合和分类单位的靶标集合之间的比较能包括预测的读数和与分类单位的靶标集合相关的参考微生物序列之间的序列相似性。
3.2、系统-处理系统
系统200的处理系统210可用于接收和处理(例如,片段化、扩增、测序等)生物样品。处理系统210能另外或可替代地用于(诸如通过邮件递送系统和/或其他合适的过程)为多个用户(例如,对于样品试剂盒250的购买订单的响应)提供和/或收集样品试剂盒250(例如,包括配置用于接收生物材料的容器、对用户的指导自我取样过程的说明等)。在一些实例中,样品试剂盒250能包括针对用户的材料和相关说明以从一个或更多个收集部位收集样品(例如,通过棉签拭抹;流体抽吸;活组织检查等)。收集部位能与以下一种或更多种相关:女性生殖器、男性生殖器、直肠、消化道(gut)、皮肤、口腔、鼻子、任何粘膜,和/或任何其他合适的样品提供部位(例如,血液、汗液、尿液、粪便、精液、阴道分泌物、眼泪、组织样品、间质液、其他体液等),其中任何个别部位或部位组合可与本文所述的任何合适的分类单位组和/或相关病症相关。处理系统210能另外或可替代地包括文库制备系统和/或任何合适的组件,所述文库制备系统能够操作以自动制备生物样品(例如,使用与抗生素相关病症相关的核酸序列相容的引物进行片段化和/或扩增(诸如以多重方式等))以由测序系统(例如,下一代测序平台)测序。在另一个实例中,处理系统210能够操作以基于使用引物组的引物扩增来自生物材料的核酸来确定微生物序列数据集(例如,通过执行框s110和/或方法100的其他合适部分等的选择),其中引物靶向对应于与病症组(和/或一种或更多种益生菌)的一种或更多种病症相关的分类单位的微生物序列。在变型中,处理系统210能以任何方式配置和/或包括与2015年10月21日提交的美国申请no.14/919,614类似的任何方式描述的组件(例如,测序系统)。然而,处理系统210和相关组件能以任何合适的方式配置。
3.3、系统-组表征系统
系统200的组表征系统220能用于确定和/或分析用于病症组的表征和/或确定治疗的微生物群系数据集和/或补充数据集(例如,通过执行方法100的各部分等)。在一个变型中,组表征系统220能获得和/或应用计算机执行的规则(例如,分类数据库205生成规则;特征选择规则;模型生成规则;用户偏好规则;数据存储、检索和/或显示规则;微生物序列生成规则;序列比对规则;和/或任何其他合适的规则)。然而,微生物群系表征系统220能以任何合适的方式配置。
3.4、系统-治疗系统
系统200的治疗系统230用于促进对用户(例如,人类受试者;利于治疗提供的护理提供者等)的一种或更多种治疗,用以治疗病症组的一种或更多种病症(例如,降低病症的风险;改善病症状态;改善症状和/或病症的其它合适方面;将用户的微生物群系药物基因组学谱改变为易受用病症治疗的状态,等)。治疗系统230能包括以下项中任何一种或更多种:通信系统(例如,通过通知护理提供者以传达治疗建议(诸如通过界面240),以推荐和/或提供治疗;能够远程医疗;等)、可在用户设备上执行的应用(例如,用于促进消化道相关病症治疗的肠病症组应用;药物提醒应用;能够操作地以与自动药物分配器通信的应用程序等)、消耗品治疗(诸如补充益生菌(如包括,类型、剂量、治疗计划、分类单位的数量和类型等)、益生菌食品、抗生素(如,类型、剂量、用药计划等))、辅助医疗设备(例如,药物分配器;含抗生素的药物设备等)、用户设备(例如,包括生物度量传感器),和/或任何其他合适的组件。在一个实例中,治疗系统230能够操作以基于组表征促进消耗品治疗的供应,其中消耗品治疗能够操作以影响用户与该病症(例如,消化道相关病症等)相关的微生物群系组成和微生物群系功能中的至少一个,而促进病情状态的改善。在一个具体实例中,治疗能包括针对该病症的益生菌有关治疗,其中益生菌有关治疗与分类单位集合(例如,包括本文所述的分类单位组等)相关,并且其中治疗系统230包括用于促进来自该分类单位集合的分类单位组与益生菌有关疗法相关的界面240。一个或更多个治疗系统230优选地可由组表征系统220控制。例如,群表征系统220能生成控制指令和/或通知以发送到治疗系统230以激活和/或以其他方式操作治疗系统230以促进治疗。然而,治疗系统230能以任何其他方式配置。
3.5、系统-界面
如图14-15所示,系统200能另外或可替代地包括界面240,界面240能用于改善与例如组表征、相关治疗建议、与其他用户的比较、基于人口统计学和/或其它用户特征的比较、微生物群系组成多样性、微生物群系功能多样性、微生物群系药物基因组学和/或其它合适方面有关的组表征信息、益生菌有关信息,和/或其它合适微生物群系相关信息的显示。在另一个实例中,界面240能呈现组表征信息,包括微生物群系组成(例如,分类单位组的相对丰度)、功能多样性(例如,基因的相对丰度和/或其它与功能相关的表征等),和/或对于病症组的其它合适信息(例如,与病症组有关的组成)。在另一个实例中,组表征信息、益生菌有关信息,和/或其它合适信息能呈现为一个共享特征(例如,相似饮食行为、相似人口统计学特征、共享病症的患者、吸烟者、锻炼者、不同饮食方案的用户、益生菌的消费者、抗生素用户、正在进行特殊疗法的群体等)的用户子群组。
在另一个实例中,界面240能够操作以呈现抗生素有关信息,包括与治疗和抗生素相关病症有关的微生物群系药物基因组学谱(和/或微生物群系组成、微生物群系功能多样性等)随时间的变化。在一个具体实例中,界面240能够操作以改善与抗生素能治疗病症相关的抗生素相关信息,并基于用户相对于共享人口统计特征的用户群体的用户微生物群系药物基因组学谱之间的比较而得出的相关信息的显示。在另一个具体实例中,界面240能促进(例如,呈现、提供通知等)与来自该分类单位集合(例如,推荐包括与病症组的病症相关的分类单位的微生物的益生菌等)的分类单位组相关地治疗(例如,益生菌有关疗法)。在另一个具体实例中,能通过选择(例如,基于满足阈值条件的组表征的组件;与超过阈值相似性的参考谱匹配的用户的微生物群系谱;超过阈值的病症组的风险;其他触发事件等)和微生物群系相关信息子集合的呈现(例如,突出显示和/或以其他方式强调信息的子集合)来改善微生物群系相关信息的界面显示。然而,界面240能显示任何合适的信息并且能以任何合适的方式配置。
系统200和/或系统200的组件能完全或部分地由以下项执行、托管(hoston)、与以下项通信和/或包括以下项:远程计算系统(例如,服务器、至少一个联网计算系统、无状态的(stateless)、有状态的(stateful))、本地计算系统、数据库(例如,分类数据库205、用户数据库、微生物群系数据集数据库、病症组数据库、治疗数据库等)、用户设备(例如,用户智能电话、计算机、笔记本电脑、辅助医疗设备、可穿戴医疗设备、护理提供者设备等)和/或任何合适的组件。例如,系统200能包括计算系统,其能够操作以与处理系统210(例如,处理系统210的下一代测序平台)通信以执行方法100的合适部分(诸如确定微生物群系药物基因组学数据)。虽然系统200的组件一般被描述为不同的组件,但是它们能以任何方式物理地和/或逻辑地集合成。例如,智能手机应用能部分或完全地执行组表征系统220(例如,应用组表征模型以诸如实时生成病症组的组表征;测序生物样品;处理微生物序列;从微生物群系数据集合中提取特征等)和治疗系统230(例如,与智能电话的日历应用程序通信以通知用户根据由益生菌治疗模型确定的参数来服用益生菌等)。另外或可替代地,系统200的功能性也能在任何合适的系统组件之间以任何合适的方式分布。然而,系统200的组件能以任何合适的方式配置。
4、方法
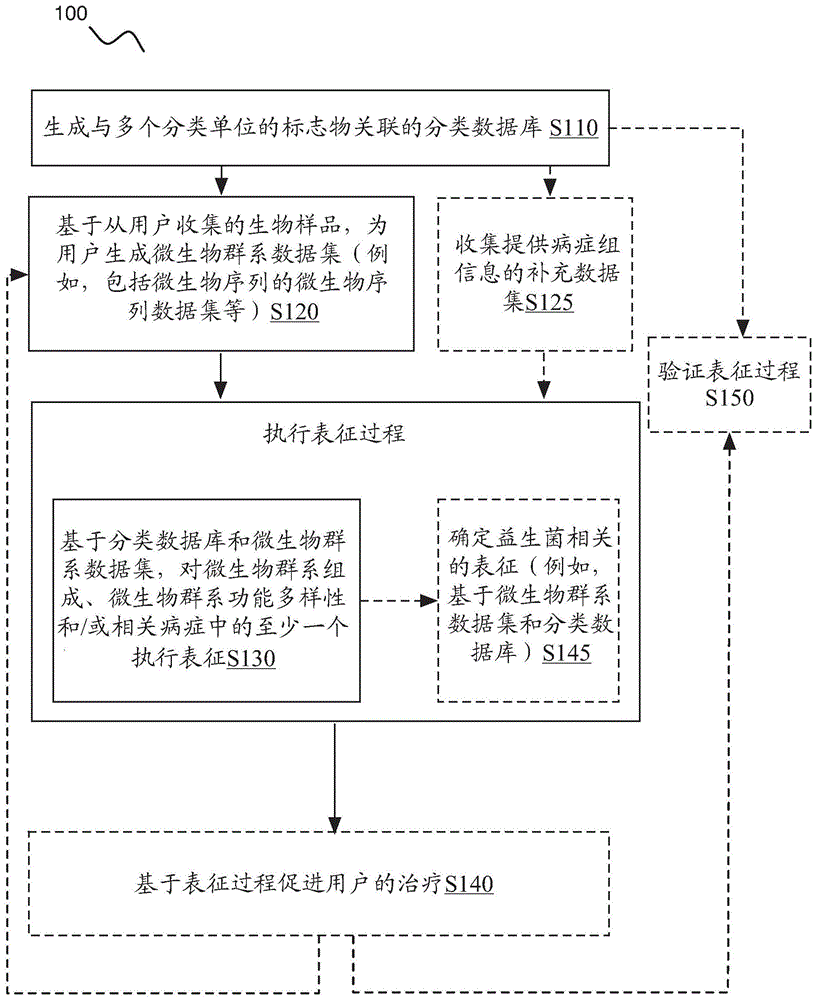
如图1a-1b和2所示,用于基于处理生物样品来表征病症组(例如,消化道相关病症)的方法100的实施方案能包括:生成与多个分类单位的标志物相关的分类数据库s110;基于从用户收集的生物样品,为用户生成微生物群系数据集(例如,包括微生物序列的微生物序列数据集等)s120;和/或,基于分类数据库和微生物群系数据集(和/或补充数据集和/或其他合适的数据),对微生物群系组成、微生物群系功能多样性和/或相关病症(例如,确定病症组的组表征)中的至少一个执行表征过程s130。方法100能另外或可替代地包括:收集病症组的补充数据集s125;基于表征过程促进用户的治疗s140;确定益生菌有关的表征s145;验证表征过程s150;和/或任何其他合适的过程。
在变型中,方法100的框能以任何合适的顺序重复执行以实现分类数据库的精炼(例如,通过鉴定与不同的分类单位和/或病症相关的新标志物等),精炼表征过程(例如,通过更新参考丰度来比较用户目标的相对丰度,以鉴定临床相关结果;通过生成和更新表征模型;通过增加能使用单一生物样品表征的病症数量;等)、治疗过程(例如,通过随时间的治疗监测和调节微生物群系组成,诸如通过随时间迭代地执行框s120和s130,其中能基于具有灵敏性、特异性、精确性和阴性预测值的表征结果来选择治疗;等)和/或其他合适的过程。
本文描述的方法100和/或过程的一个或更多个示例能通过和/或使用本文描述的系统(例如,包括样品处理网络,组表征系统、治疗系统、样品试剂盒等)、元件和/或实体的一个或更多个示例不同时地(例如,顺序地)、同时(例如,并行地;多路复用以实现并行处理多个生物样品;在用于并行计算的不同线程上同时计算地表征不同病症以改善系统处理能力;等)、与触发事件的时间关系,和/或在任何合适的时间和频率以任何其他合适的顺序执行。
另外或可替代地,本文描述的数据(例如,微生物序列数据、微生物群系特征、诸如组表征和/或益生菌有关表征的表征、群体水平数据;用户水平数据;治疗相关数据;等)能是与任何合适的时间指示符(例如,秒、分、小时、天、周等;时间指示符指示何时收集、确定和/或以其他方式处理数据;时间指示符提供由数据描述的内容的上下文,诸如作为时间指示符指示收集生物样品时的病症组的状态;等)和/或时间指示符的变化(例如,微生物群系特征随时间的变化;微生物群系组成多样性、功能多样性和/或其他合适的方面随着时间的变化;数据的变化;数据模式;数据趋势;数据外推和/或其他预测;等)相关。然而,该方法能以任何合适的方式执行。
4.1、方法-生成分类数据库
框s110记载:生成与多个分类单位的标志物相关的一个或更多个分类数据库,其可用于创建数据库,该数据库包括适于在生成一个或更多个表征时与用户微生物序列进行比较的标志物信息。
生成分类数据库s110优选地包括确定用于分类数据库的参考标志物集合(例如,基于源自基于多个分类单位的共享标志物特征选择的引物的预测读数;等);确定分类单位的靶标列表(例如,与消化道相关病症相关);基于与参考标志物的比较(例如,序列比较)过滤分类单位的靶标列表(例如,在使用优化参数的同时);以及在分类数据库中,存储与相应的参考标志物相关的过滤的分类单位(例如,如图9a-9b所示)。
关于框s110,优选基于一种或更多种引物(例如,用于扩增来自生物样品的遗传物质的引物,如框s120中所述等)确定参考标志物集合。例如,框s110能包括:基于引物预测扩增子(例如,用于正向的v4引物gtgccagcmgccgcggtaa和用于反向的ggactachvgggtwtctaat等),使得能够满足阈值条件的退火(例如,在整个序列上最多2个错配)用于比较来自参考数据库的序列(例如,silva数据库);基于简并性过滤扩增子(例如,过滤掉扩增至超过20种可能的非简并序列的简并扩增子);修饰经过滤的扩增子以表示正向读数(例如,包括正向引物和至正向引物的3'末端的125bp等)和反向读数(例如,包括反向引物和至反向引物的3'末端的124bp等);处理经修饰的扩增子(例如,除去引物);并且存储作为参考标志物的处理过的扩增子(例如,正向读取后的125bp加上反向读取后的124bp;连接形式;等等)。另外或可替代地,扩增子预测、处理和/或相关操作能基于任何合适的引物,和/或能以任何合适的方式配置以确定参考标志物。
关于框s110,确定分类单位的靶标列表(例如,与病症集合相关的属集合和种集合等)优选地包括处理病症有关的信息源(例如,第三方信息源,诸如科学文献、临床试验等;包括关于病症、相关微生物和/或相关标志物等的信息的来源)。在变型中,框s110能包括手动处理病症相关信息源(例如,标志物和/或相关信息的人工管理等)以生成分类单位的靶标列表。在另一个变型中,框s110能包括自动处理病症相关的信息源。例如,框s110能包括:生成在线信息源列表;基于列表获取在线信息源;处理在线信息源以提取用于生成分类单位的靶标列表的分类单位集合,相关病症和/或其他相关数据(例如,通过应用自然语言处理技术等)。
确定分类单位的靶标列表优选地包括基于与参考标志物集合的比较来过滤分类单位的靶标列表。例如,框s110能包括将来自参考标志物集合的参考标志物与来自分类单位的靶标列表的分类单位相关,诸如基于相对于该参考标志物集合使用在来自多个分类单位(例如,分类单位的16srrna基因v4区域)的一个或多个分类单位的相关基因序列的长度的100%的100%同一性,进行序列相似性搜索。然而,任何合适的同一性参数、长度参数和/或其他合适的参数能应用于序列相似性搜索,并且能以任何合适的方式将参考标记与分类单位相关。优选地,根据优化参数(例如,优化灵敏性、特异性、精确性、阴性预测值和/或其他度量(诸如通过使用混淆矩阵)等)来过滤初步靶标列表的不同类群的参考标志物。在一个实例中,如图6和9a-9b所示,能基于优化参数阈值(例如,要求每个优化参数超过90%;要求精度超过95%;等等)来过滤来自初步靶标列表的分类单位。在另一实例中,框s120能包括:生成将给定分类单位与不同数量的参考标志物(例如,序列)相关的多个子数据库,从而产生不同的优化参数谱。在一个具体实例中,框s110能包括:接受明确对应于分类单位的第一参考标志物子集;基于dt/ti的商对来自参考标志物的第二子集的参考标志物进行排序,其中“ti”表示对感兴趣的分类单位的序列的注释,且“dt”表示对不同的分类单位的序列的注释;基于不同的商条件为分类单位生成子数据库集合(例如,基于0的商条件优化特异性的子数据库;基于商条件100优化用于鉴定真阳性的子数据库);确定该子数据库集合的优化参数集合;基于对应于满足优化参数阈值的优化参数的分类单位的子数据库,过滤分类单位的初步靶标列表;并且将与相应参考标志物相关的经过滤的分类单位存储在分类数据库中。另外或可替代地,确定分类单位的靶标列表能以任何合适的方式执行。
关于框s110,另外或可替代地,生成分类数据库能包括基于处理与接收自用户群体的补充数据集相关的从用户群体接收的生物样品来鉴定参考标志物和相关分类单位(例如,基于源自从用户收集的生物样品的微生物群系组成特征和/或微生物群系功能多样性特征,确定与用户自身报告的病症的相关性),但是确定对应于靶标分类单位的参考标志物能以任何合适的方式执行。然而,生成分类数据库能以任何合适的方式执行。
4.2、方法-生成微生物群系数据集
框s120记载:基于从多个用户收集的生物样品,为一个或更多个用户(例如,用于确定组表征的当前受试者;用于生成分类数据库的受试者群体;等)生成一个或更多个微生物群系数据集(例如,包括微生物序列的微生物序列数据集等)。框s120用于处理从用户收集的生物样品,以便基于分类数据库确定能随后处理的微生物序列(例如,执行微生物序列和存储在分类数据库中的遗传序列之间的序列比较)以确定用户表征。
框s120能包括以下项中的任意一种或更多种:裂解生物样品(例如结合使用稳定缓冲液等)、破坏生物样品的细胞膜、从生物样品中分离非期望的组分(例如,rna、蛋白质)(例如,使用液体处理机器人利用基于柱的方法提取微生物dna等)、纯化生物样品中的核酸(例如,dna)、扩增(例如,利用文库制备系统)来自生物样品的核酸、进一步纯化生物样品的扩增的核酸、生物样品的扩增的核酸进行测序(例如,在nextseq平台上以对末端模式生成2×150bp一对末端序列;等等)和/或任何其他合适的样品处理操作,诸如关于2016年12月9日提交的美国申请no.15/374,890所述的那些操作,其全部内容通过引用并入本文。
在框s120的变型中,经纯化的核酸的扩增能包括进行以下项中的一种或更多种:基于聚合酶链式反应(pcr)的技术(例如,固相pcr、rt-pcr、qpcr、多重pcr、降落式pcr、纳米pcr、巢式pcr、热启动pcr等)、解旋酶依赖性扩增(hda)、环介导的等温扩增(lamp)、自主序列复制(3sr)、基于核酸序列的扩增(nasba)、链置换扩增(sda)、滚环扩增(rca)、连接酶链式反应(lcr)和/或任何其它合适的扩增技术。在扩增经纯化的核酸时,所用的引物优选地被选择为防止或最小化扩增偏差,和/或配置为扩增与分类数据库中存储的标志物相关的核酸区域/序列(例如,16s区域、18s区域、its区域等)(例如,在框s130中,扩增可与生物分类数据库中的标志物进行比较的基因序列;扩增对应于标志物特征的遗传序列;扩增用于诊断、用于制剂(诸如用于益生菌制剂)的在分类学上,系统发育上多信息的遗传序列;等),和/或配置为用于任何其他合适的目的。
在与框s120相关的实例中,配置为避免扩增偏差的通用引物(例如,用于16srna的f27-r338引物组,用于16srna的f515-r806引物组等)用于扩增。在一个具体实例中,框s120可包括用通用v4引物(例如,515f:gtgccagcmgccgcggtaa和806r:ggactachvgggtwtctaat)和与可变(例如,半保守高可变区)区(例如,v1-v8区域)相关的其他合适的引物扩增16s基因(例如,编码16srrna的基因),和/或rna基因的任何其他合适部分。在另一个实例中,方框s120能包括选择与蛋白质基因相关的引物(例如,编码跨多个分类单位的保守蛋白质基因序列等)。在另一个实例中,在框s120的变型中使用的引物能另外或可替代地包括对每个生物样品特异的掺入条形码序列,其能促进扩增后生物样品的鉴定。选择的引物能另外或可替代地与病症、微生物群系组成特征(例如,与遗传靶标相对应的鉴定的引物,其对应于与肠胃胀气相关的分类单位组相关的微生物群系组成特征;源自相对丰度特征来源的遗传序列等)、功能多样性特征、补充特征和/或其他合适的特征相关。引物能另外或可替代地包括适配区,其配置成与涉及互补适配体的测序技术(例如,illumina测序)配合。引物(和/或本文所述的其他合适的分子、标志物和/或生物材料)可具有任何合适的大小(例如,序列长度、碱基对数、保守序列长度、可变区长度等)。另外或可替代地,能在样品处理中使用任何合适数量的引物来进行表征(例如,组表征、益生菌有关表征等),其中引物能与任何合适数量的目标、序列、分类单位、病症和/或其他合适的方面相关。能通过框s120和/或方法100的任何其他合适的部分中描述的方法(例如,基于用于产生分类数据库的参数的引物选择)来选择框s120中使用的引物和/或方法100的其他合适的部分。另外或可替代地,引物(和/或与引物相关的过程)能包括和/或类似于2015年10月21日提交的美国申请no.14/919,614中描述的那些。然而,引物的鉴定和/或使用能以任何合适的方式配置。
在变型中,与序列读数相关,框s120能包括以下项中的一种或更多种:过滤、修剪、附加、聚类、标记(例如,作为实际遗传序列;作为错误;等)。在一个具体实例中,框s120能包括基于16s基因的扩增产生读数集合;使用平均q分数>30过滤读数;从读数中修剪引物和前导碱基;附加正向和反向读取;使用1个核苷酸的距离进行聚类(例如,使用swarm算法);将每簇最丰富的读取序列标记为实际的遗传序列;对于每个簇,分配最丰富的读数序列,其具有对应于簇中的读数的计数;并且,对于每个簇,对最丰富的读取序列执行嵌合体去除(例如,使用vsearch算法等)。然而,能以任何合适的方式进行测序。
对于任何合适数量的生物样品,能以多路复用方式执行框s120中描述的任何合适的过程。在一个实例中,框s120能包括用正向和反向索引(例如,唯一组合)对多个样品进行条形码编码、以多路复用方式对多个样品进行测序;并且,在测序之后,分成多路处理对应于不同用户的样品(例如,利用bcl2fastq算法等)。另外或可替代地,能在任何合适的时间和频率执行框s120的各部分的任何数量的示例。然而,框s120能以类似于全部内容通过引用并入本文的2016年12月9日提交的美国申请no.15/374,890的任何合适的方式执行,和/或能以任何合适的方式执行。
4.3、方法-收集补充数据集
框s125记载:接收提供病症组和/或益生菌有关信息的补充数据集。框s125能用于获取与用户集合的一个或更多个用户相关的附加数据,其能用于训练和/或验证在框s130中生成的表征过程(例如,表征模型)、框s140中的治疗过程(例如,治疗模型)和/或任何其他合适的过程。补充数据集优选地包括调查得到的数据,但是能另外或可替代地包括以下项中的任何一种或更多种:诊断相关数据(例如,乳糜泻测试、结肠镜检查、乙状结肠镜检查,下胃肠道摄影、上胃肠道内窥镜检查,上胃肠道摄影、模拟结肠镜检查等)、来自传感器和/或任何其他合适组件(例如,系统200的组件,其可包括治疗设备、诸如智能电话、可穿戴医疗设备等用户设备)的全方位数据、医疗数据(例如,当前和历史医学数据,诸如抗生素病史)、组的一个或更多个病症的多信息数据(例如,病症存在或缺乏的指示、相关诊断、相关治疗、随时间的进展等)和/或任何其他合适类型的数据。在框s125的变型中,调查得到的数据能提供与受试者相关的生理、人口统计和行为信息。另外或可替代地,框s125能以与全部内容通过引用并入本文的2015年10月21日提交的美国申请no.14/919,614类似的任何方式执行。然而,处理补充数据集框s125能以任何合适的方式执行。
4.4、方法-执行表征过程
框s130记载:基于分类数据库和微生物群系数据集,对微生物群系组成,微生物群系功能多样性和/或相关病症中的至少一个执行表征过程。框s130能用于处理与分类数据库(例如,在框s110中生成)有关的微生物群系数据集(例如,在框s120中生成)以为用户生成一个或更多个表征。用户的表征能包括与全部内容通过引用并入本文的2016年12月9日提交的美国申请no.15/374,890中描述的那些类似的任何表征(例如,涉及用户的不同人口统计特征的不同类群的微生物群系组成的相对丰度;病症风险;随时间变化的相关趋势;等)。
框s130能包括以下项中的一种或更多种:确定参考微生物群系参数范围(例如,健康参考相对丰度范围,诸如图8中所示,其中范围能与一个或更多个病症的缺乏相关;与一种或更多种病症的存在和/或风险相关的风险参考丰度范围;一个或更多个分类单位的丰度的微生物组成范围;与一种或更多种分类单位相关的功能特征的微生物功能多样性范围;等);确定用户的用户微生物群系参数;基于用户微生物群系参数和参考微生物群系参数范围之间的比较来生成用户的表征(例如,如处理与普氏菌属相关的不健康的微生物群系组成而表征用户,所述普氏菌属基于指示在普氏菌属健康参考范围之外的普氏菌属丰度的用户微生物群系参数;等)和/或任何其他合适的操作。参考微生物群系参数范围能具有任何合适的下限和上限(例如,对于瘤胃球菌属的相对丰度,下限高于0%)。参考微生物群系参数范围能包括任何合适的置信区间代表的范围(例如,跨用户群体的99%置信区间)。在一个实例中,能针对任何合适的分类单位(例如,来自分类单位的靶标列表)计算参考相对丰度范围,诸如基于将对应于该分类单位的读数计数除以总读数(例如,经聚合和过滤的读数的总数);然而,参考相对丰度范围能以任何合适的方式计算。
框s130优选地包括确定一个或更多个病症组(例如,消化道相关病症组等)的一种或更多种组表征。对于该组的一种或更多种病症,组表征能包括以下项中一种或更多种:病症的存在、病症的缺乏、病症的风险、病症的严重性、与病症相关的推荐、与病症相关的微生物群系组成(例如,微生物群系组成多样性,其包括与病症相关的分类单位的相对丰度)、与病症相关的微生物群系功能多样性、与病症相关的微生物群系药物基因组学(例如,针对病症的不同抗生素潜在功效的用户药物基因组学谱)、与病症相关的益生菌(例如,来源、相关的分类组、相关性等),和/或与病症组相关的任何其他合适的方面。
在框s130的变型中,能凭经验执行确定参考微生物群系参数范围。例如,框s130能包括从用户群体收集生物样品和补充数据集。该用户群体能包括与微生物群系组成、微生物群系功能多样性、病症和/或其他合适特征的任何合适的状态相关的用户,其中补充数据集(例如,在与用户相关的移动设备上执行的应用程序处的数字管理调查)能提供多信息的特征。在一个实例中,补充数据集能告知包括以下项病症中的一种或更多种:癌症、感染、肥胖、慢性健康问题、精神健康障碍和/或任何其他合适的病症。在具体实例中,方法100能包括:处理来自健康用户群体的生物样品(例如,从未被诊断为患有高血糖和/或糖尿病、消化道相关症状和/或其他病症的用户等);处理生物样品(例如,如框s120)以确定微生物序列;确定每个用户的每个分类单位的相对丰度(例如,来自分类单位的靶标列表);并根据健康用户群体的相对丰度为每个分类单位生成健康范围。在另一具体实例中,方法100能包括:确定该分类单位集合的参考相对丰度范围集合包括:为用户群体收集补充生物样品集合和补充数据集的集合;使用与微生物相关病症组相关的引物组处理该补充生物样品集合以产生补充微生物序列数据集;并且基于补充微生物序列数据集和补充数据集的集合确定参考相对丰度范围集合。然而,凭经验确定参考微生物群系参数范围能以任何合适的方式执行。在框s130的另一变型中,确定参考微生物群系参数范围能非经验地执行,诸如基于手动和/或自动处理病症相关信息源。然而,确定参考微生物群系参数范围能以任何合适的方式进行。
关于框s130,确定用户的用户微生物群系参数优选地基于源自用户的生物样品而产生的微生物序列(例如,如框s120中;经聚类和过滤的读数;等)。例如,确定用户微生物群系参数能包括确定不同分类单位(例如,在分类单位的靶标列表中鉴定的)的相对丰度。在进一步的实例中,确定用户微生物群系参数能包括提取与组相关的特征(例如,如图4所示),其能包括以下项中的一种或更多种:与组的一种或更多种病症相关的微生物群系组成特征、微生物群系功能特征、微生物群系药物基因组学特征和/或其他合适的特征,诸如以类似于2016年12月9日提交的美国申请no.15/374,890的方式,其全部内容通过引用并入本文。在一个实例中,方法100能包括:基于微生物序列数据集为用户提取组相关特征集合;确定参考特征与用户的组相关特征集合之间的比较;基于比较确定用户针对微生物有关病症组的组特征。在一个具体实例中,方法100能包括:提取组相关的特征集合,其包括基于微生物序列数据集提取该组相关特征集合的微生物群系组成多样性特征和微生物群系功能多样性特征,并且其中确定比较包括确定具有微生物群系组成多样性特征的参考特征和具有微生物群系功能多样性特征的参考特征的比较。在一个具体实例中,方法100能包括:从微生物群系组成特征和/或微生物群系功能多样性特征的值(例如,从健康用户的生物样品中获得等)确定参考微生物群系参数范围;并且将用户微生物群系组成特征值和/或用户微生物群系功能多样性特征值与参考微生物群系参数范围进行比较,以确定用户的表征(例如,对于与参考微生物群系参数范围正相关和/或负相关的病症)。
关于框s130,将一个或更多个用户微生物群系参数与一个或更多个与特征(例如,分类单位、病症等)相关的一个或更多个参考微生物群系参数范围进行比较能包括基于用户微生物群系参数值是否落在参考微生物群系参数范围之内或之外将用户表征为拥有或不拥有该特征。例如,框s130:能包括得到史密斯甲烷杆菌(methanobrevibactersmithii)的健康参考相对丰度范围;并且响应于用户具有超过健康参考相对丰度范围的史密斯甲烷杆菌(methanobrevibactersmithii)的相对丰度,表征用户处于肠易激综合症的风险中。在另一个实例中,确定参考特征和组相关的特征集合之间的比较能包括确定组相关的特征集合与以下项中的至少一个相关:微生物群系组成特征的存在、微生物群系组成特征的缺失、分类单位集合的各分类单位组的相对丰度、与分类单位集合相关的至少两个特征之间的比率、分类单位组之间的相互作用,以及分类单位组之间的系统发育距离。在另一个实例中,产生分类数据库能包括确定该分类单位集合的参考相对丰度范围集合,其中该参考相对丰度范围集合与微生物有关病症组相关;基于微生物序列数据集为用户提取该分类单位集合的用户相对丰度范围集合;并确定该参考相对丰度范围集合与该用户相对丰度范围集合之间的比较。在另一个实例中,确定参考特征与组相关特征集合之间的比较能包括执行以下项中的至少一个:预测分析、多假设检验、随机森林检验,和主成分分析。然而,比较一个或更多个用户微生物群系参数能以任何合适的方式执行。
另外或可替代地,对于框s130,执行表征过程能基于阈值(例如,基于涉及与该病症相关的阈值组的分类单位集合的相对丰度来确定病症组的风险,等)、权重(例如,第一分类单位的相对丰度的加权比第二分类单位的相对丰度的加权更重,诸如当第一分类单位与感兴趣的病症具有更大的相关性时等)、机器学习模型(例如,分类数据库中存储的微生物群系特征的分类模型训练和分类单位的相应标签;等)、计算机实现的规则(例如,用于提取微生物群系特征的特征设计规则;模型生成规则;用户偏好规则;微生物序列生成规则;序列比对规则;等)和/或任何其他合适的方面。
另外或可替代地,对于框s130,执行表征过程能被配置为测量以下项中的至少一个:风险分数,和/或显著性指数以将分类单元或分类单位集合与感兴趣的病症(或病症组)以与2017年9月14日提交的美国临时申请序列号62/558,489中描述的方式类似的任何方式相关,其通过引用整体并入本文。然而,框s130能以任何合适的方式执行。
在变型中,框s130和/或方法100的其他合适部分能包括应用一个或更多个模型(例如,组表征模型;益生菌表征模型;治疗模型;等),其包括以下项中的一个或更多个:概率属性、启发式属性、确定性属性,和/或任何其他合适的属性。每个模型都能运行或更新:一次;以预定的频率;每次执行方法和/或子过程的实施方案的示例;每次满足触发条件(例如,检测音频数据集中的音频活动;检测语音活动;检测到未预料到的测量;等),和/或以任何其他合适的时间频率。模块能与一个或更多个其他模型同时地、连续地、以变化的频率,和/或在任何其他合适的时间运行或更新。能根据新接收的最新数据;历史数据或基于任何其他合适的数据进行更新的数据验证、查实、加强、校准或以其他方式更新每个模型。另外或可替代地,模型和/或相关方面(例如,方法、算法等)能以与通过引用整体并入本文的2016年12月9日提交的美国申请no.15/374,890中描述的方式类似的方式配置。然而,框s130能以任何合适的方式执行。
4.5、方法-促进治疗
方法100能另外或可替代地包括框s140,其记载:基于表征过程(例如,基于组表征、基于益生菌有关的特征、基于特征;等)促进治疗。框s140能用于向用户确定、推荐和/或提供个性化治疗,以便将用户的微生物群系组成和/或功能特征调节到期望的平衡状态,和/或改善一种或更多种病症。例如,框s140能包括基于组表征(和/或益生菌有关表征)向用户推广益生菌消耗品,其中益生菌消耗品能够操作以改善微生物相关病症组的多个微生物相关病症。在另一个实例中,方法100能包括收集与用户的饮食行为相关的饮食相关的补充数据集,其中推广益生菌消耗品包括基于饮食相关的补充数据集和组表征(和/或益生菌表征)向用户推广益生菌消耗品。
疗法可包括以下任何一种或更多种:益生菌、消耗品(例如各类食品、各类饮料等)、局部疗法(例如,乳液、软膏、防腐剂等)、营养补充剂(例如,维生素、矿物质、纤维、脂肪酸、氨基酸、益生元等)、药物、抗生素、噬菌体和任何其他合适的治疗措施。在框s130中生成的表征能用于确定和/或促进给用户的定制疗法,诸如包括制剂和方案(例如,剂量、使用说明)。例如,方法100能包括:确定分类单位的健康参考相对丰度范围之外的分类单位的用户相对丰度;以及促进益生菌和/或其他合适的疗法调节用户的微生物群系组成,以使用户相对丰度在健康参考相对丰度范围内。因此,框s140能包括确定和/或提供被配置为校正生态失调特征(例如,基于在框s130中确定的表征来鉴定,等)的治疗。
在变型中,框s140能包括确定和/或提供具有一个或更多个治疗系统的治疗,所述治疗系统能包括以下项中的任何一个或更多个:通信系统(例如,用于传达治疗建议;用于启用远程医疗;等等)、可在用户设备上执行的应用程序(例如,用于促进对消化道的适当护理的消化道相关病症应用等)、辅助医疗设备(例如,用于消化道相关病症的治疗设备和/或诊断设备、药物分配器、益生菌分配器等)、用户设备(例如,包括生物鉴定传感器)和/或任何其他合适的组件。照此,框s140能另外或可替代地包括生成用于治疗系统的控制指令和/或通知,所述治疗系统是用于激活和/或以其他方式操作与促进治疗相关的治疗系统。然而,使用治疗系统执行框s140能以任何合适的方式执行。
在另一个变型中,框s140能包括向用户生成和/或提供关于治疗、在框s130中生成的特征,和/或任何其他合适的信息的通知(例如,患者的微生物群系报告,如图5所示)。通知的类型和提供通知的方式能类似于全部内容通过引用并入本文的2016年12月9日提交的美国申请no.15/374,890中所描述的。然而,框s140能以任何合适的方式执行。
4.6、方法-确定益生菌有关表征
方法100能另外或可替代地包括框s145:确定益生菌有关表征。框s145能用于处理与分类数据库(例如,包括在分类数据库中的益生菌有关信息等)相关的微生物群系数据集(例如,在框s120中生成),以为用户生成一个或更多个益生菌相关的表征。另外或可替代地,框s145能用于促进益生菌有关疗法能基于(例如,确定和/或促进)的组表征的确定。如图10中所示,框s145能包括以下项中的任何一个或更多个:确定益生菌来源、确定与益生菌有关的分类单位组、确定与益生菌有关的病症(例如,病症组)、产生描述本文所述的益生菌有关信息的表征和/或其他合适的信息、确定益生菌有关特征(例如,表征和/或治疗可基于的那些;等),和/或任何其他合适的过程。在一个具体实例中,框s145能包括:鉴定潜在的益生菌;基于益生菌的表征与益生菌相关的性能度量的比较,过滤潜在的益生菌;鉴定益生菌有关病症(例如,健康益处、益生菌来源、与益生菌相关的分类单位组);并且基于与益生菌有关病症的比较进行益生菌的第二次过滤。在另一具体实例中,方法100能包括:为益生菌菌株确定范围(例如,相对丰度范围;健康范围等)(例如,可通过分析性能度量可靠地鉴定,诸如通过执行一个或更多个在本文中描述的过程);将范围(例如,参考范围)与一个或更多个病症相关;确定用户的用户范围;将用户范围与参考范围进行比较;和/或基于比较确定治疗。与益生菌相关的分类单位组能包括本文所述的任何合适的分类单位组(例如,与分类数据库有关等)。然而,框s145能以任何合适的方式执行。
4.7、方法-验证表征过程
方法100能另外或可替代地包括框s150,其记载:验证表征过程。框s150能用于基于微生物群系数据集和分类数据库验证用于为用户生成一个或更多个表征(例如,如框s130中)所使用的过程,以便促进用户微生物群系参数和/或参考微生物群系参数范围(例如,靶标分类单位的相对丰度)的准确确定。验证表征过程优选地包括执行与参考样品相关的框s110-s140中的一个或更多个(例如,具有已知的微生物群系组成和/或微生物群系功能多样性,诸如涉及分类单位的靶标列表等)。在一个变型中,框s150能包括基于稀释与目标分类单位相关的遗传物质(例如,合成的遗传物质,诸如代表不同靶标分类单位的16srrna基因的v4区域的合成双链dna,如图7中的“sdna”所示等)(例如,以任何合适的比例)产生参考样品;并通过执行框s110-s140中的一个或更多个来处理参考样品,以验证与参考样品相关的靶标分类单位的检测。在另一个变型中,框s150能包括处理来自真实或合成生物样品(例如,具有已知组成的活的或重组材料的粪便样品,如图7中的“验证样品”所示;等)的参考样品以验证与参考样品相关的靶标分类单位的检测。另外或可替代地,框s150能包括基于验证表征过程的结果来修改与框s110-s140中的一个或更多个相关的一个或更多个参数。然而,框s150能以任何合适的方式执行。
将方法100和/或实施方案的系统至少部分地体现为和/或实施为被配置为接收存储计算机可读指令的计算机可读介质的机器。这些指令能由与应用、小程序、主机、服务器、网络、网站、通信服务、通信界面、患者计算机或移动设备的硬件/固件/软件元件或其任何合适的组合等集合成的计算机可执行组件执行。实施方案的其它系统和方法能至少部分地体现为和/或被实施为被配置为接收存储计算机可读指令的计算机可读介质的机器。这些指令能由与上述类型的设备和网络集合成的计算机可执行组件来执行。计算机可读介质能被存储在诸如ram、rom、闪存、eeprom、光学设备(cd或dvd)、硬盘驱动器、软盘驱动器或任何合适设备的任何合适的计算机可读介质上。计算机可执行组件能是处理器,但是任何合适的专用硬件设备能(可替代地或另外)执行指令。
这些图说明了根据优选的实施方案、实例性构造及其变型,组成、方法和系统的可能的实现的结构、功能和操作。还应当注意,在一些替代实施方式中,提及的功能可不按照图中指出的顺序发生。例如,基于所涉及的功能,连续示出的方面实际上能基本上同时执行,或者方面有时可以相反的顺序执行。实施方案包括各种系统组件和各种方法过程的每个组合和置换,包括任何变型、实施例和特定实施例。如本领域技术人员将从先前的详细描述以及从附图和权利要求中认识到的,能对实施方案进行修改和改变而不脱离范围。
- 还没有人留言评论。精彩留言会获得点赞!