一种基于卷积神经网络的细胞反卷积预测方法
1.本发明主要涉及基于单细胞rna测序数据的下游分析领域,主要是关于一种细胞反卷积方法,特别是涉及到一种基于卷积神经网络的对单细胞rna测序数据进行细胞反卷积方法。
背景技术:
2.随着高通量测序技术被广泛的运用在生物和医学领域,近年来开发的单细胞rna测序技术可以对单个细胞进行无偏、可重复、高分辨率和高通量的转录分析。传统的测序技术是基于群体细胞进行测序,反映出的是一群细胞的平均表达值,并不能揭示不同细胞之间的异质性。而单细胞rna测序技术可以研究单个细胞表达谱,从而避免单个细胞的基因表达值被群体的平均值所掩盖,以揭示复杂细胞群体的异质性。单细胞rna测序技术对单个细胞的全部rna进行提取、逆转录、扩增和测序得到单细胞rna测序数据,对测序数据的分析可以揭示生物组织的细胞构成、发现罕见的细胞群、探究细胞组分的变化等。
3.细胞反卷积是单细胞rna测序数据下游分析的一个方面,细胞反卷积即从组织样本的单细胞rna测序数据中推断出该组织存在的细胞类型及比例,这可用于发现新的细胞亚型、讨论癌组织的免疫浸润情况、探究疾病的发病机理等。而传统反卷积算法存在一些弊端,如使用的数学模型需要添加各种约束来规范模型,模型不够直观难以理解。需要进行繁琐的数据预处理,并对特定细胞类型的基因表达矩阵的准确度及组织的基因表达矩阵的准确度要求都较高。目前机器学习技术在细胞反卷积领域应用的还不够广泛,在使用机器学习技术提升细胞反卷积性能上仍具有较大探索空间。针对这些问题,我们急需发展出一种新的细胞反卷积方案来应对较高的生物医学的数据处理及分析需求。
技术实现要素:
4.针对现有细胞反卷积算法的缺陷,本发明提供了一种基于卷积神经网络的细胞反卷积预测方法cbccon。cbccon通过使用深度学习技术即卷积神经网络来进行组织细胞比例预测,cbccon模型的隐藏节点可以有效的挖掘出基因之间的内部联系,节点可以学习到对噪音和偏差具有鲁棒性的特征,具有更好的反卷积性能。建立cbccon模型的目的在于解决当前细胞反卷积算法受到噪音和偏差的影响从而导致精度不高,并且需要添加各种约束来规范模型等问题。
5.为实现上述目的,本发明提供如下技术方案:一种基于卷积神经网络的细胞反卷积方法,包括以下步骤:(1)使用单细胞rna测序数据模拟人工组织,确定一个模拟人工组织中的细胞总数k和需要生成的人工组织个数q;从单细胞rna测序数据中抽取k个细胞,合并抽取细胞的基因表达矩阵形成模拟人工组织的基因表达矩阵为模拟组织的特征,并记录该组织各个类型细胞所占比例作为该
组织的标记信息,为某细胞类型占该组织的细胞比例数;t为该组织细胞类型数目;所述的k为大于1的正整数,q为大于1的正整数;(2)对步骤(1)所得的模拟人工组织进行特征筛选,并对每个特征执行转化到对数空间和归一化操作,通过以上处理,得到数据集;(3)对步骤(2)中得到的数据集,数据集若来源于s个不同数据集,将其分为训练集和测试集进行s折交叉验证,训练集由s-1个来源不同的数据组成,测试集由剩下的1个来源的部分数据组成,确定batch size的大小,在训练集中随机抽取batch size个数据作为一次训练的输入数据;(4)从步骤(3)中输入数据中获得组织的细胞类型数目t作为该卷积神经网络的全连接模块中最后一层神经元的个数,构建卷积神经网络模型cbccon,确定模型的学习率learning rate,模型训练测次数step,模型的优化算法optimized algorithm;将步骤(3)中的作为一次训练的数据输入cbccon模型中进行模型训练,得到预测的组织细胞比例(1≤i≤t)为训练集预测的某细胞类型占该组织的细胞比例数。通过公式计算细胞比例预测值和真实值之间计算损失函数,是该组织真实的细胞分数标签,是训练集该组织细预测的细胞比例,利用优化算法对损失函数进行优化;依据步骤(3)中再随机抽取step-1次的继续训练,训练完毕后,保存训练好的cbccon模型中参数;(5)使用步骤(4)中训练好的cbccon模型进行数据预测,将输入训练好的模型中,得到预测结果即预测的测试集的组织细胞类型比例(1≤i≤t)为测试集数据中预测的某细胞类型占该组织的细胞比例数。
6.对步骤(4-5)中的得到的模型构建评价指标对模型性能评价,分别通过公式,公式,公式,和公式评价cbccon模型的性能并与cpm、cibersort(ci)、cibersortx(cix)、music方法进行比较。 是预测的细胞比例, 是实际的细胞比例,分别代表预测和实际细胞比例的标准差,分别代表预测和实际细胞比例的均值。通过比较模型评价指标,可以得出cbccon模型与其他算法相比,rmse值更低且变化
幅度更小,relate值更高。这可以表明cbccon方法与其他算法相比具有更好的反卷积性能。cbccon对细胞反卷积预测精度的提升主要是由于模型使用的卷积层可以从单细胞rna测序数据中充分挖掘各个基因之间的内在联系,从而提取出数据的隐藏特征,并且cbccon的网络节点对数据的噪音和偏差具有很高的鲁棒性,因此对细胞比例预测的精度更高。并且cbccon解决了传统算法需要特定细胞类型的基因表达矩阵才能进行细胞反卷积问题,或需要添加各种约束来规范模型等问题,并且模型结构直观易于理解具有高度的扩展性。
7.优选的,步骤(1)中,所述的k为100-5000,所述的q为1000-100000。
8.优选的,步骤(1)中所述的使用单细胞rna测序数据进行模拟包括以下步骤:(1-1)通过公式(1≤i≤t)确定单个模拟的细胞组织中各个细胞类型的比例,即确定模拟组织的标记信息为某细胞类型占该模拟组织的细胞比例数;是为单个细胞类型的创建的随机数,取值在[0,1]之间,是为所有细胞类型创建的随机数的总和,;(1-2)通过公式(1≤i≤t)确定单个模拟的细胞组织实际应抽取的各个细胞类型的细胞数目,即确定为单个模拟细胞组织的各个细胞类型抽取的细胞个数为某模拟组织单个细胞类型应抽取的细胞数,是为某细胞类型占该模拟组织的细胞比例数,k是设定的一个模拟人工组织中的细胞总数,为单个模拟的细胞组织实际应抽取的各个细胞类型的细胞数目,。
[0009]
优选的,步骤(2)中所述的对模拟人工组织x进行数据预处理包括以下步骤:(2-1)通过公式将数据转换到对数空间得到;(2-2)通过公式(1≤i≤n,1≤j≤m)线性归一化得到。
[0010]
优选的,步骤(3)中的batch size的取值为128。
[0011]
优选的,步骤(4)中所述的cbccon模型结构为卷积神经网络由为多个卷积层池化层和一个全连接层组成,使用了两个64个提取特征的filter卷积层,使用一层最大池化层减少特征数,使用了两个32个filter卷积层,使用一层最大池化减少特征数,使用了两个16个filter卷积层,使用一层最大池化减少特征数,使用了两个8个filter卷积层,使用一层最大池化减少特征数,使用了两个4个filter卷积层,使用一层最大池化减少特征数,再将数据输入压平层中,将数据转化为一维数据;最后使用三个全连接层,节点数分别是128,64,细胞种类的数;所有的卷积层均为一维,且卷积层的激活函数统一设置为relu函数,步长均为1,前两个全连接层使用relu激活函数,最后一个全连接层使用softmax层进行组织细胞比例预测。
[0012]
优选的,步骤(4)中所述的cbccon模型学习率learning rate的取值为0.0001,模型训练测次数step取值为5000,模型的optimized algorithm(优化算法)设置为rmsprop算
法。
[0013]
与现有技术方法相比,本发明的有益效果是:本专利提出了新的细胞反卷积预测算法的方案,能够更加准确的预测组织的细胞比例。该算法基于单细胞rna测序数据模拟异质组织的基因表达矩阵,在一定程度上解决了单细胞rna测序数据获取昂贵的问题,并且该方法基于卷积神经网络,模型结构清晰易于理解,无需进行繁琐的数据预处理,且不需要特定的细胞表达矩阵建立复杂的数学模型。
附图说明
[0014]
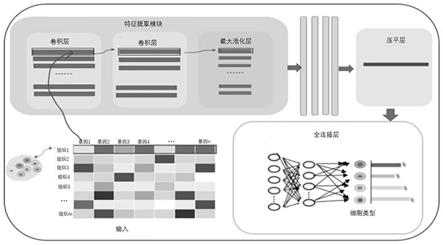
图1为cbccon的模型结构示意图;图2为cbccon模型具体参数;图3为cbccon测试集的部分预测结果;图4为cbccon模型与cpm、cibersort(ci)、cibersortx(cix)、music反卷积模型的各评价指标对比图;图5为cbccon模型与cpm、cibersort(ci)、cibersortx(cix)、music反卷积模型的rmse评价指标对比图;图6为cbccon模型与cpm、cibersort(ci)、cibersortx(cix)、music反卷积模型的relate评价指标对比图。
具体实施方式
[0015]
为了清楚的阐明本发明的技术方案,下面结合附图1-6以及实例对本发明进行阐述,此处的实例仅用于解释本发明,并不限定本发明。
[0016]
应该指出,以下详细说明都是示例性的,旨在对本发明提供进一步的说明。除非另有指明,本文使用的所有技术和科学术语具有与本发明所属技术领域的普通技术人员通常理解的相同含义。
[0017]
图1展示了使用单细胞rna测序数据进行组织细胞反卷积的cbccon模型的简要图示,首先将预处理后的模拟组织的基因表达矩输入卷积神经网络中。每一行是一个模拟组织的各个基因的表达量,该行标签是对应的模拟组织的细胞类型比例。cbccon模型分为将数据输入特征提取层中,将两个卷积层和一个最大池化层作为特征提取层,共进行了五次特征提取,再将得到的数据输入压平层中,将数据格式转化为一个一维向量。最后将一维向量输入一个三层的全连接神经网络中,经过训练可以得到预测的组织细胞比例。
[0018]
图2展示了卷积神经网络中的参数设置,第一个特征提取层使用了两个64个提取特征的filter卷积层,使用一层最大池化层减少特征数。使用了两个32个filter卷积层,使用一层最大池化减少特征数,使用了两个16个filter卷积层,使用一层最大池化减少特征数,使用了两个8个filter卷积层,使用一层最大池化减少特征数,使用了两个4个filter卷积层,使用一层最大池化减少特征数,再将数据输入压平层中,将数据转化为一维数据。最后使用三个全连接层,节点数分别是128,64,细胞种类的数。所有的卷积层均为一维,且卷积层的激活函数统一设置为relu函数,步长均为1,前两个全连接层使用relu激活函数,最后一个全连接层使用softmax层进行组织细胞比例预测;数据选定来自人外周血单个核细胞(pbmc)数据的单细胞rna测序数据,分别来自
四个数据集,本文对以上数据将以data6k、data8k、donora和donorc引用。cbccon的输入文件包含两个txt文件,count.txt中为pbmc数据的单个细胞基因表达矩阵,celltype.txt中pbmc组织中包含细胞的种类。cbccon的输出文件包括以一个pb文件、个txt文件、一个csv文件。savemodel.pb文件中保存的是训练完毕后模型中参数,prediction.txt预测组织的各细胞类型比例,compare.csv文件为cbccon模型与cpm、ci、cix、music方法的各项评价指标rmse、relate、hrelate、uniform的得分结果比较,用来对比模型的性能。设定一个模拟人工组织中的细胞总数k=500和需要生成的人工组织个数q=32000,一次训练的数据个数即批大小batch size=128,模型的学习率learning rate=0.0001,模型训练测次数step=5000,模型的optimized algorithm设置为rmsprop算法。以下是进行细胞反卷积算法的具体步骤:1. 用单细胞rna测序数据模拟人工组织使用pbmc的data6k、data8k、donora、donorc的单细胞rna测序数据模拟人工组织,确定一个模拟人工组织中的细胞总数k=500和需要生成的人工组织个数q=32000。从单细胞rna 测序数据中抽取500个细胞,合并抽取细胞的基因表达矩阵形成模拟人工组织的基因表达矩阵为模拟组织的特征,并记录该组织各个类型细胞所占比例作为该组织的标记信息,为某细胞类型占该模拟组织的细胞比例数,包括如下步骤:(1-1)通过公式确定单个模拟的细胞组织中各个细胞类型的比例,即确定模拟组织的标记信息为某细胞类型占该模拟组织的细胞比例数。是为单个细胞类型的创建的随机数,取值在[0,1]之间,是为所有细胞类型创建的随机数的总和,;(1-2)通过公式(1≤i≤6),k=500确定单个模拟的细胞组织实际应抽取的各个细胞类型的细胞数目,即确定为单个模拟细胞组织的各个细胞类型抽取的细胞个数为某模拟组织单个细胞类型应抽取的细胞数,是为某细胞类型占该模拟组织的细胞比例数,k是设定的一个模拟人工组织中的细胞总数,为单个模拟的细胞组织实际应抽取的各个细胞类型的细胞数目,; 2. 数据预处理对步骤1所得的模拟人工组织进行数据预处理,对数据集x中每个特征执行筛选去除了21410个特征项,剩下11328个特征,再将x转化到对数空间和归一化操作,通过以上数据预处理,得到数据集,包括如下步骤:(2-1)通过公式将数据转换到对数空间得到;以
为例,即a1bg特征的特征值从[105.2,83.5,55.8,....]转化为[6.73,6.4,5.82,...];(2-2)通过公式(1≤i≤n,1≤j≤m)线性归一化,将的的值缩放到[0,1]之间,得到。以为例,即a1bg特征的的最大值为10.54,最小值为0.53。
[0019]
3. 划分数据集 对步骤2中得到的数据集,数据集来源于4个不同数据集data6k、data8k、donora、donorc,数据集中有六种细胞类型分别为monocytes、unknown 、cd4tcells、bcells、nk、cd8tcells,其中unknown代表未知的细胞类型。将数据集分为训练集和测试集进行4折交叉验证,训练集由3个来源不同的数据组成,测试集由剩下的1个来源的部分数据组成。我们从选取来自data6k、data8k、donorc的数据作为训练集,使用donora中的数据作为测试集,为了方便测试,我们仅从donora中抽取500个数据作为测试集,确定batch size的大小为128。在训练集中随机抽取128个数据作为一次训练的输入数据;4. 训练cbccon模型从步骤3中输入数据中获得组织的细胞类型数目t=6作为该卷积神经网络的全连接模块中最后一层神经元的个数,构建卷积神经网络模型cbccon,确定模型的学习率learning rate=0.0001,模型训练测次数step=5000,模型的优化算法optimized algorithm为rmsprop算案发。将步骤3中的作为一次训练的数据输入cbccon模型中进行模型训练,得到训练集的预测的组织细胞比例(1≤i≤6)为训练集预测的某细胞类型占该组织的细胞比例数。通过公式计算细胞比例预测值和真实值之间计算损失函数,是该组织真实的细胞分数标签,是该组织细预测的细胞比例,利用优化算法rmsprop对损失函数进行优化。依据步骤3中再随机抽取4999次的继续训练,训练完毕后,保存训练好的cbccon模型中参数; 5. 使用训练好的模型进行预测使用步骤4中训练好的cbccon模型进行数据预测,将测试集数据即即donora中500条测试数据输入训练好的模型中,得到预测结果即测试集上预测的组织细胞类型比例(1≤i≤t)为测试集数据中预测的某细胞类型占该组织的细胞比例数。以测试集中一个名为v241模拟组织为例,v241的组织细胞比例预测结果为:monocytes类型的细胞比例0.171;unknown类型的细胞比例为0.027;cd4tcells类型的细胞比例为0.428;bcells类型的细胞比例为0.102;nk类型的细胞比例为0.086;cd8tcells类型的细胞比例为0.185。500个模拟组织的细胞类型比例部分预测结果如图4所示。
[0020]
6. 模型评估对步骤4-5中的得到的模型构建评价对模型性能评价,分别通过公式分别通过公式,公式,公式,和公式评价cbccon模型的性能并与cpm、cibersort(ci)、cibersortx(cix)、music方法进行比较, 是预测的细胞比例, 是是实际的细胞比例,分别代表预测和实际细胞比例的标准差,分别代表预测和实际细胞比例的均值,通过比较模型评价指标,可以得出cbccon模型与其他算法相比,rmse值更低且变化幅度小,relate值更高。这可以表明cbccon方法与其他算法相比具有更好的反卷积性能。cbccon对细胞反卷积预测精度的提升主要是由于模型使用的卷积层可以从单细胞rna测序数据中充分挖掘各个基因之间的内在联系,从而提取出数据的隐藏特征,并且cbccon的网络节点对数据的噪音和偏差具有很高的鲁棒性,因此对细胞比例预测的精度更高。并且cbccon解决了传统算法需要特定细胞类型的基因表达矩阵才能进行细胞反卷积、需要添加各种约束来规范模型等问题,并且模型结构直观易于理解具有高度的扩展性。对比结果如图4、图5和图6所示。
[0021]
在步骤4中使用训练数据对模型拟合完毕后,cbccon达到的数据覆盖率统计如下:(1)细胞比例预测值和真实值误差在10%以内的数据;覆盖率:99.8%;(2)细胞比例预测值和真实值误差在5%以内的数据;覆盖率:85%;(3)细胞比例预测值和真实值误差在1%以内的数据;覆盖率:30%;图4、图5和图6中比较结果可以看出cbccon的rmse相对来说更低,并且变化幅度更小,relate相关性与其他方法相比也更高,达到了0.900,这表明cbccon模型在组织比例预测上,模型具有较好的精确性并对噪声的抗干扰能力更强。
[0022]
最后应说明的是:以上所述仅为本发明的优选实施例而已,并不用于限制本发明,尽管参照前述实施案例对本发明进行了详细的说明,对于本领域的技术人员来说,其依然可以对前述各实施案例所记载的技术方案进行修改,或者对其中部分技术特征进行等同替换。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1