头颈部癌症的生物标志物及其使用方法与流程
相关申请的交叉引用本申请要求于2014年10月17日提交的申请号为62/065122的美国临时专利申请的优先权,其通过引用以其整体并入本申请,并且用于所有目的。本公开涉及检测和治疗癌症。更具体地说,其涉及生物标志物用于检测、治疗和监测头颈部癌症的用途。
背景技术:
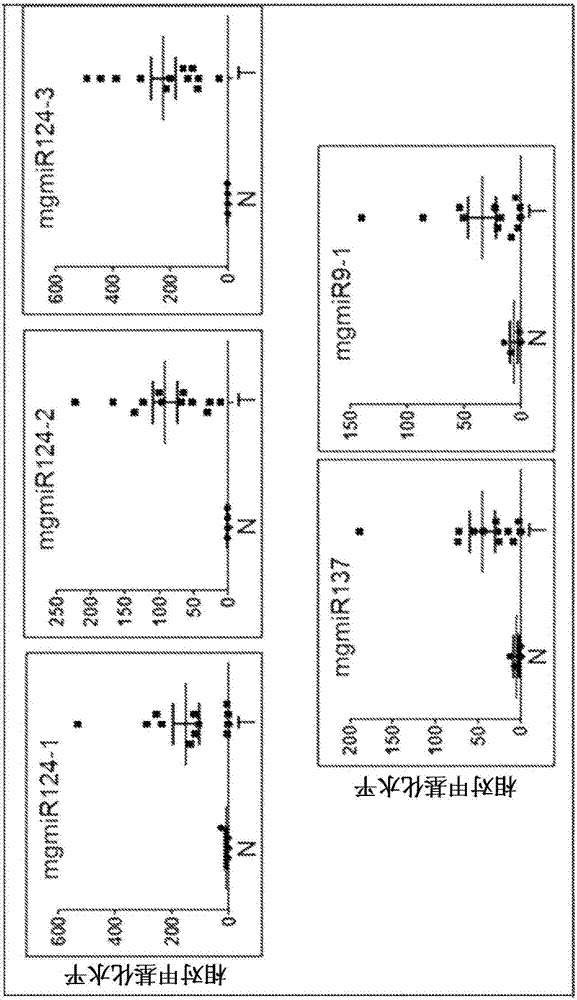
:头颈部鳞状细胞癌(hnscc)表示大约90%的所有头颈部癌症以及5%的所有恶性肿瘤。参见jemal等人(2009)和worshem等人(2013年)。口腔和咽部癌症各自是2008年美国男性中第八最常见的癌症,并且美国2009年发现超过48000名hnscc的新患者(jemal等人)。已经发现hnscc在过去30年以增长的速度流行起来。参见worsham等人(2013年)。尽管医学和癌症治疗在进步,但是患有hnscc的患者的存活率却相当停滞。参见siegel等人(2013年)。这种低存活率与许多其它癌症的存活率的增加成鲜明对比。对于hnscc不良预后的一个主要原因是超过半数的hnscc患者在诊断时已经发展为局部或转移阶段。因此,早期检测可能是在将来改善存活率的关键。对于头颈部癌症的早期检测,还未报道有效的方法。技术实现要素:本发明通过提供用于早期检测某些类型的癌症的方法使本领域前进。更具体地,公开了一组诊断性编码微rna的甲基化基因组基因座(mgmir)标志物,其在检测头颈部鳞状细胞癌(hnscc)中显示出了90%的敏感性和100%的特异性。这些结果表示首次采用定量ms-pcr用于利用mgmir标志物来检测甲基化水平。此外,该组微rna已经证实检测癌症患者旁黏膜中超甲基化的能力,表明其在早期检测上的效用。该组还可以通过采用唾液、血液和细针抽吸(fna)组织样品等等用于检测癌症。在一个实施方式中,公开了用于检测受试者体内癌症的方法,其可以包括(a)测定选自由mir124-1、124-2、124-3、137和9-1所组成的组的编码至少一种微rna的至少一个基因组基因座的甲基化水平,和(b)将从受试者获得的所述甲基化水平与来自已知无癌症的个体的编码相同微rna的相应基因组基因座的甲基化水平(后者在本公开中还被称为“基础甲基化水平”或“基础水平”)进行比较。所公开的方法可以进一步包括提供诊断,其中受试者体内dna片段与基础甲基化水平相比显著更高的甲基化水平指示癌症或癌前病变。为了本公开的目的,术语“显著更高”可以是指至少20%、40%、50%、80%、100%、150%、200%或甚至更高。在另一个实施方式中,用于检测癌症的方法可以包括:(a)从受试者的组织或体液制备dna提取物,其中该dna提取物含有至少一种第一dna片段,所述至少一种第一dna片段包含选自由mir124-1、124-2、124-3、137和9-1所组成的组的编码至少一种微rna的至少一个基因组基因座,(b)使用第一dna片段作为模板且使用特异于甲基化dna的寡核苷酸作为引物,通过聚合酶链反应(pcr)产生第二dna片段,(c)测定步骤(b)中产生的第二dna片段的水平,和(d)将第二dna片段的水平与基础水平进行比较,其中第二dna片段与基础水平相比更高的水平指示癌症或癌前病变。在一个方面,基础水平是从已知无癌症的个体的相同组织或体液中以相同的方式产生的相应dna片段的水平。在另一个实施方式中,用于qms-pcr的引物是选自由seqidno.1-10组成的组的引物对。“f”表示正向引物,“r”表示反向引物。在一个方面,所公开的方法可以在修改或不修改的情况下合适于癌症(或癌前病变)的早期检测。癌症的实例可以包括但不限于头颈部鳞状细胞癌(hnscc)、食道癌、肺癌、宫颈癌、乳腺癌、结肠癌、直肠癌、胃癌、胰腺癌、肝癌、胆囊癌、胆管癌、小肠癌、泌尿道癌、女性生殖道癌、男性生殖道癌、内分泌腺癌、皮肤癌、血管瘤、黑色素瘤、肉瘤、脑瘤、神经癌、眼肿瘤、脑膜癌或来自造血系统恶性肿瘤的实体瘤,等等。在另一个实施方式中,该至少一种微rna或者编码该至少一种微rna的dna片段可以分离自选自由头部组织、颈部组织、口交换物、鼻交换物、唾液、痰、血液、血清、脑脊液(csf)、尿液、fna组织、其它体液或它们的组合所组成的组的组织或体液。该编码微rna的五个基因组基因座(也被称为“mgmir”标志物)也可以作为组使用,其中在做出癌症或癌前病变的阳性判断(positivecall)之前,需要该受试者体内至少两个、三个、四个或所有五个与基础水平相比显示出显著更高的甲基化水平。在另一个实施方式中,该基础水平可以是通过对从两个以上已知无癌症的个体获得的水平取平均值而设立的预设值。通过举例的方式,预设值用于本公开的实施例中,但是其可以被本领域的技术人员修改。在另一个实施方式中,公开了用于检测受试者体内癌症的方法,其可以包括(a)测定分离自受试者的组织或体液中的至少一种微rna的水平,其中至少一种微rna选自由mir124-1、124-2、124-3、137和9-1所组成的组,和(b)将该至少一种微rna的水平与基础水平进行比较,所述基础水平是来自已知无癌症的个体的相同组织或体液的相同微rna的水平,其中受试者体内至少一种微rna与基础水平相比显著更低的水平指示癌症或癌前病变。附图说明图1示出了全基因组搜索hnscc中的mgmir的流程图。图2示出了由qms-pcr在12个hnscc细胞系(t)和4个头颈部对照细胞系(n)中所检查的mgmir的相对甲基化水平。图3示出了患者队列1中由各个mgmir生物标志物测定的相对甲基化水平。n-来自无癌症个体的正常颊粘膜,m-来自hnscc患者的旁粘膜,t-来自hnscc患者的肿瘤组织。图4a示出了对于标志物mgmir124-1所揭示的对照和受试者样品两者的总体相对甲基化水平以及每个患者的唾液(下部)和肿瘤样品(上部)的甲基化水平的qms-pcr结果。图4b示出了对于标志物mgmir124-2所揭示的对照和受试者样品两者的总体相对甲基化水平以及每个患者的唾液(下部)和肿瘤样品(上部)的甲基化水平的qms-pcr结果。图4c示出了对于标志物mgmir124-3所揭示的对照和受试者样品两者的总体相对甲基化水平以及每个患者的唾液(下部)和肿瘤样品(上部)的甲基化水平的qms-pcr结果。图4d示出了对于标志物mgmir137所揭示的对照和受试者样品两者的总体相对甲基化水平以及每个患者的唾液(下部)和肿瘤样品(上部)的甲基化水平的qms-pcr结果。图4e示出了对于标志物mgmir9-1所揭示的对照和受试者样品两者的总体相对甲基化水平以及每个患者的唾液(下部)和肿瘤样品(上部)的甲基化水平的qms-pcr结果。图5示出了由hnscc的旁粘膜中的mgmir检查出的阳性病例。图6示出了手术切除肿瘤后患者唾液dna中的mg-mir124s和mg-mir137)的甲基化水平降低。b(手术前),a(手术后)。图7示出了mg-mir124s(a,上部小图)或mgmir137(b,上部小图)的甲基化水平与mir124(a,下部小图)和mir137(b,下部小图)的表达水平之间的相关性。图8示出了在5-氮杂胞苷(5-氮杂)治疗后的mir124或mir137表达的恢复图9示出了mir124和mir137模拟物的恢复(a)抑制了细胞增殖(b)和sp尺寸(c)。图10示出了由targetscan对mir124和mir137两者预测出的共同靶标。图11示出了ezh2作为mir124和mir137二者的下游共同靶标的验证。在mir124和mir137模拟物恢复后的ezh2mrna水平(a)和蛋白质水平(b)。图12示出了用于qms-pcr的引物的序列(seqidno.1-10)以及编码mir124-1、124-2、124-3、137和9-1的基因组基因座的序列(seqidno.11-15)。具体实施方式微rna(mirna)的发现是分子生物学的重大事件。参见lee等人(1993)。mirna是长约18-25个核苷酸的非编码rna。这些分子似乎是进化保守的,并且它们的主要作用是基因调控,而不是被翻译成蛋白质。在过去二十年内,多项研究已经证实,mirna充当重要角色,并且影响基本的细胞过程,如细胞发育、分化、增殖、生存和死亡。ambros等人(2004年)。随着研究证实一个mirna家族平均影响约500个基因,已经在人类基因组中发现了超过1000种mirna。参见lewis等人(2003);krek等人(2005),betel等人(2008)和friedman等人(2009)。因为这种在细胞基因组学上的突出的调节作用,研究人员已经探索了改变的mirna是否在肿瘤发生中发挥作用。有趣的是,研究已经表明,mirna可以在具有明显不同的mirna表达信号的人类肿瘤的肿瘤发生中发挥作用。此外,mirna在肿瘤发生中似乎具有抑癌和致癌两种作用。利用这个发现,深入的研究已经集中于mirna作为癌症诊断和治疗的潜在靶标的可能性。参见例如,garzon等人(2010)。基因研究中的另一个显著发展是越来越理解表观遗传学。表观遗传学是不由实际dna的改变所造成的基因活性的可遗传变化的研究。大概最有名的和科学证实的表观遗传学方法是dna甲基化。将甲基基团添加至dna启动子区域,特别是特定基因的富含cpg的序列的dna启动子区域,已被证实强烈地阻抑转录,其与基因的实际突变或缺失等效。在过去15年左右,表观遗传学已被证实在癌症发展中发挥显著作用。具体地,在包括hnscc的许多癌症中已经揭露了关键的肿瘤抑制基因的超甲基化。参见worsham等人(2013)。多数的hnscc影响上呼吸消化道的粘膜表面,其中还包括食道癌和肺癌。迄今为止,对于特定的表观遗传学变化是否可以作为hnscc的诊断工具,仍然没有答案。本公开提供了使用qms-pcr在hnscc中检查编码微rna的基因组基因座的甲基化水平(mgmir“)的方法。经过全基因组搜索1881种微rna的1kb5utr中的cpg岛并筛选编码微rna标志物的超过五十个基因组基因座后,证实了mir124(124-1,124-2和124-3)、mir137和mir9(9-1)是用于检测hnscc的最好标志物。该5种标志物的集合,当以组合作为组使用时,以100%的特异性和90%的敏感性检测出hnscc。这些标志物可以在不同的位点或通过不同的机制起作用。在一个实施方式中,在旁粘膜组中的相对甲基化水平显著低于肿瘤组中的相对甲基化水平。然而,与正常对照组相比,旁粘膜中有相对更高的甲基化水平,且是统计上显著的。由于该组具有检查周围组织中癌前变化的潜力,这一发现具有早期诊断意义。roh等人在2011年的研究检查了组织印迹的可行性以及利用qms-pcr评估头颈部癌样本的边缘中四个基因的甲基化模式。我们的研究检查了旁粘膜中微rna甲基化水平的存在,并且还发现,这是以足够的敏感性和特异性检测肿瘤周围的看上去非常正常的组织中的癌症的技术。在另一个实施方式中,与具有客观数字结果的任何测试一样,可以将阈值水平设定高于所认为测试是“阳性”的水平。除了mgmir9-1之外的所有标志物表现出无癌症的对照组内一定水平的基线甲基化表达。为了定义和开发我们的用于潜在临床应用的小组,对每个标志物选择阈值甲基化水平。为此,将加权约登指数(youdenindex)用于导出每个mgmir标志物的最优截止值。术语“癌症”是指涉及可能侵入或扩散到机体的其它部位的异常细胞生长的一类疾病。术语“癌前病变”是指可能已经形成恶性肿瘤、或增生/发育不良但还没有开始侵入或扩散到机体的其它部位或者获得侵入或扩散到机体的其它部位的能力的状态。对本领域的技术人员显而易见的是,对本文所描述的方法的其它合适的修改和适应是明显的,并且在不偏离本文所公开的实施方式的范围的情况下可以利用合适的等价物来进行。现在已经详细地描述了某些实施方式,通过引用所包括的仅仅用于举例说明的目的而非意图是限制性的以下实施例,将会更清楚地理解其。实施例实施例1鉴定hnscc细胞系中编码mir124、mir137和mir9基因组基因座的dna甲基化。为了鉴定hnscc中编码mirna的甲基化基因组基因座,利用ucscgenomebrowser获得mir类数据库中的1881种智人主要(pri-)mirna的1kb5'-utr的基因组序列。然后利用cpg岛预测软件(methprimer)来鉴定具有cpg岛的约90个基因组基因座(定义为岛大小>200个碱基,gc含量>50%,观察到的/预期>0.6)。通过设计甲基化特异性引物以及运行定量甲基化特异性pcr(qms-pcr),我们筛选了这些基因组基因座,并且发现了编码pri-mir的5个基因座,即124-1、124-2、124-3、137和9-1,其相比于正常的头颈部细胞系,在人类hnscc细胞系中具有增加的甲基化(图1和表1)。这些编码mirna的甲基化基因组基因座被称为mgmir。如表1所示,我们检查了12种hnscc细胞系中的相对甲基化水平,包括源自年龄在22岁到70岁的范围内、男性和女性、hpv阳性和阴性hnscc、来自范可尼贫血(fanconianemia)患者的hnscc以及头颈部区域的不同解剖位点的细胞系。包括四个头颈部正常细胞系作为对照。lscc:喉部scc,lnmets:淋巴结转移,hpscc:下咽部scc,osccrec.:口腔scc复发,hpv:人乳头状瘤病毒,pscc:咽scc,fa-a:范可尼贫血a实施例2一组诊断性用于患者队列1中头颈部癌症的mgmir生物标志物的开发。本研究遵照由相关机构的协议认可的伦理审查委员会(irb)在人类hnscc手术样品上实施。使用了总共64个不同的组织样本(n=64)。表2示出了研究群体的分类。三十个样品是来自手术切除时的hnscc样本(n=30个肿瘤样本)。该群组组成我们的“肿瘤”组。这些患者中的26例还具有在手术时从邻近于原发肿瘤的区域中收集的非常正常的组织。这些样本构成我们的“旁黏膜”组(n=26),并表示来自患有已知的头颈部癌症的患者的看上去非常正常的组织,其在显微镜下具有增生/发育不良。将来自8名无恶性肿瘤史的健康患者的口腔组织用作为正常对照群体(n=8)。(表2)。表2-患者队列1中的研究群体组.将所有新鲜的肿瘤库组织保存在液氮中直至dna提取时。使用dneasy血液和组织试剂盒(qiagen)从每个组织样品提取基因组dna,并且使用nanovue分光光度计(gehealthcare)进行定量。然后使用ezdna甲基化金试剂盒(zymo)并按照制造商的说明,用硫酸氢盐处理基因组dna。使经硫酸氢盐转化的dna进行基于sybr-绿的定量甲基化特异性pcr(qms-pcr),用于五个mgmir标志物。对于每个单独的标志物,为了鉴定适当的退火温度和最大化结果以获得典型的s型结果曲线,在开始运行样品之前,对qms-pcr方案进行优化。将熔解曲线与跑胶一起用于确定每个标志物的特异性。调整的变量包括温度、循环次数和每次循环的长度。将β-肌动蛋白用作为内部对照。在cfxconnecttm实时检测系统(biorad)上以一式三份运行qms-pcr。每个板包括患者dna样品、阳性对照(体外甲基化的dna)、阴性对照(来自已知未甲基化的细胞系的dna)和水空白。对于在每个标志物内的每个样品,通过标准的2-δδct方法,使用ct值的差异来计算相对甲基化水平。qms-pcr结果的统计分析。为了允许将所有qms-pcr结果进行比较,将一个对照患者设立为阴性对照并且将该患者的甲基化水平任意地定义为用于校准所有其它患者的结果的甲基化水平。针对每次qms-pcr实验,由每次qms-pcr实验中每个样品的一式三份产生三个数据点(δδct值,+sd,-sd)。将接受者操作特性(roc)曲线用于评估每个生物标志物的性能。roc曲线是图解在其鉴别阈(待鉴定的割点)变化时的二元分类系统(在该项目中,所感兴趣的生物标志物)的性能的图表。在此报道了每个绘制的roc曲线的曲线下面积(auc)。auc值越大,通常表示性能更好的生物标志物。完美的诊断性测试将具有1的auc值。使用约登指数以导出割点。最佳的割点应将约登指数最大化。未加权的约登指数定义为j=敏感性+特异性-1。加权的约登指数定义为j=w*敏感性+(1-w)*特异性,其中w表示给定的权重。当然,未加权的约登指数同等地重视敏感性和特异性。为了增强特异性,我们分配了较高的权重至特异性而非敏感性。分别鉴定了将两种约登指数最大化的最佳割点。对于加权的约登指数,任意地将20%的权重分配至敏感性以及将80%的权重分配至特异性(在未加权约登中,可以认为敏感性和特异性均具有50%的权重)。然后使用具有不等方差的双侧t检验计算正常对照、旁粘膜和肿瘤组之间的差异的显著性。单独地对每个标志物,并且对作为整体的标志物组,检验显著性。p<0.05被认为是统计学上显著。以下示出了通过qms-pcr测定的每个甲基化的微rna标志物的相对甲基化水平,并且还总结于表3中。表3.来自hnscc患者的肿瘤组织和旁粘膜以及来自对照患者的正常组织中阳性甲基化的微rna案例的百分数肿瘤旁粘膜正常粘膜mgmir124-170%(21/30)15.4%(4/26)0%(0/8)mgmir124-270%(21/30)3.8%(1/26)0%(0/8)mgmir124-363.3%(19/30)23.1%(6/26)0%(0/8)mgmir13760%(18/30)3.8%(1/26)0%(0/8)mgmir9-156.7%(17/30)11.5%(3/26)0%(0/8)组合90%(27/30)38.5%(10/26)0%(0/8)mgmir124-1:在正常对照群体中,mgmir124-1的相对甲基化水平为6.60。在肿瘤组内,平均相对甲基化水平是128.08(se30.62),并且在旁粘膜组内,平均相对甲基化水平是18.14(se6.13)(图3a)。mgmir124-2:在正常对照群体中,mgmir124-2的相对甲基化水平为6.29。在肿瘤组内,平均相对甲基化水平是40.21(se6.24),并且在旁粘膜组内,平均相对甲基化水平是5.73(se1.23)(图3b)。mgmir124-3:在正常对照群体中,mgmir124-3的相对甲基化水平为4.37。在肿瘤组内,平均相对甲基化水平是57.66(se16.29),并且在旁粘膜组内,平均相对甲基化水平是18.22(se7.57)(图3c)。mgmir137:在正常对照群体中,mgmir137的相对甲基化水平为3.19。在肿瘤组内,平均相对甲基化水平是109.30(se25.31),并且在旁粘膜组内,平均相对甲基化水平是8.08(se0.82)(图3d)。mgmir9-1:mgmir9-1在该组标记物中是唯一一个在对照群体内不显示基线甲基化水平而仅在癌症样本中具有甲基化水平的标志物。在肿瘤组内,平均相对甲基化水平是25.14(标准误差(se)7.44),并且在旁粘膜组内,平均相对甲基化水平是1.80(se0.79)(图3e)。5个生物标志物的组合:当比较作为整体的所有5个mgmir标志物的相对甲基化水平时,正常对照群体的相对甲基化信号是4.09,而肿瘤群体的相对甲基化信号是72.08。这种差异是统计学显著的(p<0.001)。在旁粘膜中的相对甲基化水平是10.39。当与正常对照的甲基化水平相比时,这种差异也表现出统计学显著性(p=0.005)。在已经测定了该五个mgmir生物标志物的相对甲基化水平后,采用接收者操作特性曲线和加权约登指数以导出截止值。有关更多的详情,参见以上的材料和方法部分。对于每个标志物和每个样品由qpcr结果完成这些计算。以组合成组的方式使用这5个标志物,这些结果示出了,在30个肿瘤样本内,在检测鳞状细胞癌上敏感性为90%且特异性为100%(表3和4)。有趣的是,在旁粘膜样本内,对于检测鳞状细胞癌,敏感性为38.5%且特异度为100%(表3和4)。表4来自患者队列1的hnscc组织和对照组织的敏感性和特异性实施例3针对hnscc患者队列2鉴定并采用唾液mgmir作为新的非侵入性生物标志物。本研究在人类hnscc组织样品以及来自患者的唾液样品中实施。这项研究由伦理审查委员会(irb)认可并且在使用样品之前获得患者的同意。从作为队列的36名患者收集组织和/或唾液样品。将组织和唾液样品标记为受试者(肿瘤)或对照。受试者组由诊断为hnscc的患者组成。组织和唾液从24名hnscc患者获得。基于临床检查和/或先前的活检确定来自这些患者的组织是hnscc组织。当患者经受手术切除或活检时从手术室获得肿瘤组织。经受手术切除之前从患者收集唾液。对照组由无癌症的患者组成。从12名经受扁桃体切除术的对照患者获得组织和唾液。扁桃体切除术的适应症是阻塞性睡眠呼吸暂停和/或慢性扁桃体炎。在手术室中收集组织并且该组织由来自前扁桃体支柱(anteriortonsillarpillar)的正常粘膜和/或扁桃体组织组成。经受手术切除之前从患者收集唾液。唾液还收集自五名无疾病的健康志愿者。该研究中还收集患者的人口统计信息,以及人乳头状瘤病毒(hpv)状态、烟草产品和酒精的使用、任何既往化疗和/或放疗、癌症病史和家族癌症史。在受试者组中,一旦最后定下组织样品的病理,还将回顾和编辑关于临床阶段和肿瘤组织的等级以及病理特征和分子标志物的信息。将所有收集的信息置于加密的数据库中并且在不带患者标识符的情况下列出样品。在收集组织后后,立刻将组织送到实验室冷冻并储存在液氮中,直到随后dna提取。在唾液收集前30分钟,患者/志愿者需要停止进食、饮水、嚼口香糖和吸烟。在唾液收集时,患者用正常的盐水冲洗他们的嘴两次,每次间隔2分钟。然后指示患者/志愿者将他们的唾液吐到falcon50ml的收集管中2-3次,该2-3次间的间隔为2分钟。从多数患者获得大约4-5ml的唾液,一些样品由于口腔干燥而具有有限的体积。一旦收集,将样品送到实验室,将其储存在-20摄氏度的冰箱中。基因组dna的分离和亚硫酸氢盐转化。根据制造商的说明,使用dneasy血液和组织试剂盒(qiagen),从每个组织样品中提取基因组dna。对于唾液基因组dna提取,将冷冻的唾液样品在室温下缓慢熔化,并且加入额外的5ml唾液制备缓冲液至每个唾液样品,用于在室温下稳定基因组dna。然后以5000rpm将唾液样品离心15分钟。测试了五种不同的基因组dna提取试剂盒。qiaampdnamini试剂盒给出了来自唾液的基因组dna的最佳收率和质量。然后,使用nanovue分光光度计(gehealthcare)对基因组dna定量。然后使用ezdna甲基化金试剂盒(zymo),并按照制造商的说明,用亚硫酸氢盐处理基因组dna。定量甲基化特异性pcr(qms-pcr)。使经硫酸氢盐转化的dna进行基于sybr-绿的qms-pcr,用于五个mgmir标志物(用于qms-pcr的引物序列示于seqidno.1-10中)。对于每个单独的标志物,为了鉴定适当的退火温度和最大化结果以获得典型的s型结果曲线,在开始运行样品之前,对qms-pcr方案进行优化。将熔解曲线与跑胶一起用于确定每个标志物的特异性。使用sybrgreenmix(biorad)按照以下热条件运行qms-pcr:40次循环的95c3min,95c30sec,55-60c30sec。将β-肌动蛋白用作为内部对照。在cfxconnecttm实时检测系统(biorad)上以一式三份运行qms-pcr。对于在每个标志物内的每个样品,通过标准的2-δδct方法,使用ct值的差异来计算相对甲基化水平。qms-pcr结果的统计分析。为了允许将所有qms-pcr结果进行比较,将一个对照患者设立为阴性对照并且将该患者的甲基化水平任意地定义为用于校准所有其它患者的结果的甲基化水平。针对每次qms-pcr实验,由每次qms-pcr实验的每个样品的一式三份产生三个数据点(δδct值,+sd,-sd)。将接受者操作特性(roc)曲线用于评估每个生物标志物的性能。roc曲线是图解在其鉴别阈(待鉴定的割点)变化时的二元分类系统(在该项目中,所感兴趣的生物标志物)的性能的图表。在此报道了每个绘制的roc曲线的曲线下面积(auc)。auc值越大,通常表示性能更好的生物标志物。完美的诊断性测试将具有1的auc值。使用约登指数以导出割点。最佳的割点应将约登指数最大化。未加权的约登指数定义为j=敏感性+特异性-1。加权的约登指数定义为j=w*敏感性+(1-w)*特异性,其中w表示给定的权重。当然,未加权的约登指数同等地重视敏感性和特异性。为了增强特异性,将较高的权重分配至特异性而非敏感性。分别鉴定了将两种约登指数最大化的最佳割点。对于加权的约登指数,任意地将20%的权重分配至敏感性以及将80%的权重分配至特异性(在未加权约登中,可以认为敏感性和特异性均具有50%的权重)。然后使用具有不等方差的双侧t检验计算正常对照、旁粘膜和肿瘤组之间的差异的显著性。单独地对每个标志物,并且对作为整体的标志物组,检验显著性。p<0.05被认为是统计学上显著。以下分析了五个mgmir:mgmir124-1、2、3,mgmir137和mgmir9-1。图4(小图a-e)中所示的是,揭示了对于每个标志物的对照和受试者样品两者的总体相对甲基化水平以及每个患者的唾液和肿瘤样品的甲基化水平的qms-pcr结果。获得了比较这些mgmir生物标志物的热图(heatmap),并且利用约登指数计算唾液和组织样品中每个mirna标志物的敏感性和特异性。另外,当所有的mirna标志物一起使用时,测定整体的敏感性和特异性。这些结果列于表5和表6中。表5hnscc患者组织中的敏感性和特异性表6hnscc患者唾液中的敏感性和特异性实施例4生物标志物作为用于评估hnscc治疗效果以及用于治疗后监测的手段将这两个患者队列组合在一起,并对总共51个hnscc组织进行了评估,包括来自口腔的23个,来自口咽的9个,来自下咽部的2个,来自喉部的14个,以及3个皮肤hnscc病例。来自患者队列2的21个唾液样品包括10例口腔,5例口咽,1例下咽部和5个喉部hnscc病例。阳性病例和解剖部位或t阶段之间没有观察到显著差异。如表7中所示,当肿瘤是处于t1阶段时,从组织检测到88%的阳性病例以并且唾液样品检测到75%的阳性病例。这些结果表明,当肿瘤都还小时,这些mgmir可以用作早期检测标志物。在患者队列1中相同的截止点被用于二分旁粘膜组(图3)。在26个旁粘膜样品中鉴定了10例(38.5%)阳性病例。如图5所示,组织学检查显示了在7例发育不良病例中的5例(71.4%)阳性病例,以及在7例增生病例中的4例(57.1%)阳性病例。该百分数比12个正常旁黏膜样品中的1例(9.1%)阳性病例高,这表明使用五个mgmir生物标志物用于早期检测hnscc的可能性。表7由5个mgmir检查的阳性病例组织唾液t17/8(88%)3/4(75%)t214/17(82%)4/5(80%)t310/11(91%)5/6(83%)t414/15(93%)5/6(83%)另外,如图6所示,在三名手术切除其肿瘤后的hnscc患者中,患者的唾液dna中的mgmir124-1、124-2、124-3和137的甲基化水平显著降低。因此,患者唾液dna中的mgmir的甲基化水平与肿瘤负荷相关。这一结果支持了使用mgmir与患者的唾液dna用于手术后的hnscc监控的可行性。这项研究的一个目的是调查患有hnscc的患者对于可能具有肿瘤抑制作用的特定mirna(mir124-1,124-2,124-3,137,9-1)在5个基因组基因座上是否具有更高的甲基化比率。假定不仅可以在组织样品中也可以从患者唾液中检测到这种差异。所公开的数据揭示了在受试者和对照样品之间的甲基化的不同。所有对照和受试者组织样品的比较证实了对于所有五个mgmirna在甲基化上的差异显著。类似地,当将对照和组织唾液样品进行编辑和比较时,观察到所有5个mgmirna的甲基化水平更高,其中124-1、124-2和137在对照和受试者样品之间显示出显著差异。受试者群体中的甲基化水平更高可以突出它们相应编码的mirna的下调并且强调它们在肿瘤发生中的可能的抑制作用。将甲基化数据进一步分解成每个单独的对照和受试者样品,进一步地显示了受试者样品比对照样品具有更高的甲基化水平的趋势。敏感性和特异性值证实了,在唾液样品中mgmir124-2是最敏感的mirna标志物,随后是137、124-3、9-1和124-1。在组织样品中,mgmir124-2也是最敏感的mirna标志物,随后是137、124-1、124-3和9-1。组织样品整体上比唾液样品更敏感,其中组织样品中所有五个mirna标志物具有大于50%的敏感性。组织和唾液样品两者中的mgmir标志物的特异性较高,全都具有大于80%的特异性。在十个mirna标志物(5个唾液和5个组织)中,六个具有100%的特异性。总体而言,相比于唾液样品,更多的组织样品是阳性的。由于组织应该具有稳健的hnsccdna而唾液样品可能含有一些来自肿瘤剥离的hnsccdna,因此该结果不是出乎意外的。有趣的是,超过80%的阳性唾液样品在它们相关的组织样品中也是阳性的,这存在强关联。来自这些数据的最有效的结果是当将所有五个mirna一起使用时的整体敏感性和特异性的计算结果。该结果表明这些发现在dna微阵列上的潜在应用。在将其完成时,分别地,唾液样品的敏感性和特异性是81%和100%,并且组织样品的敏感性和特异性是90.5%和100%(表5和6)。采用临床和病理信息,基于多个不同的变量,独立地分析受试者样品。表8示出了在每个阳性和阴性mgmir生物标志物之间的年龄差异。表8.hnscc患者中阳性和阴性mgmir的平均年龄阳性阴性mgmir13762.2岁63.2岁表9按性别划分的hnscc患者中的阳性mgmir病例表10按位置划分的阳性mgmirhnscc病例osccopscclsccmgmir124-170.6%(12/17)77.8%(7/9)73.3%(11/15)mgmir124-235.3%(6/17)66.7%(6/9)46.7%(7/15)mgmir124-358.8%(10/17)55.6%(5/9)53.3%(8/13)mgmir13755%(11/20)45.5%(5/11)56.3%(9/16)mgmir9-160%(12/20)54.5%(6/11)25%(4/16)表11按hpv状态划分的阳性mgmirhnscc病例hpv+hpv-mgmir124-171.4%(15/21)79.2%(19/24)mgmir124-276.2%(16/21)*25%(6/24)mgmir124-352.4%(11/21)50%(12/24)mgmir13756%(14/25)46.4%(13/28)mgmir9-144%(11/25)48.1%(13/27)*p<0.01表9示出了每个阳性mgmir生物标志物的性别差异。表10示出了每个mgmir生物标志物与解剖位点之间的相关性。在每个阳性mgmir生物标志物中的解剖位置。表11示出了每个mgmir生物标志物与人乳头状瘤病毒(hpv)状态之间的相关性。总体而言,在每个mgmir阳性和阴性病例之间没有年龄差异。在每个mgmir病例之间没有性别差异,除了mgmir124-2,其中女性中的阳性病例的百分数比男性中的阳性比例显著更高。在每个mgmir生物标志物与解剖位置之间没有显著差异。除了mgmir124-2,mgmir生物标志物与hpv状态之间不存在显著相关性,其中hpv+hnscc患者中mgmir124-2阳性病例的百分数显著更高。在本研究中,与对照患者相比,患有hnscc的患者中具有假定的肿瘤抑制作用的特定肿瘤抑制mirna具有更高比例的dna甲基化。在患者的组织和唾液二者中都可以检测到这种甲基化的差异(discrepancy),并可能强调这些mirna在肿瘤发生中的作用。此外,利用来自该研究的数据,其证实组织和唾液样品中超过90%的整体敏感性和特异性,可以将这种表观遗传差异用于诊断测试。实施例5在hnscc中,编码mir124和mir137的基因组基因座的甲基化使mir124和mir137的表达沉默。检查了十二个人类hnscc细胞系和四个正常人类头颈部细胞系,以确定dna甲基化水平和表达水平之间的相关性。观察到mir124和mir137的dna甲基化水平与表达水平之间负相关。一个实例示于图7中。mir124的所有这三个基因组基因座在hnscc细胞系(fadu)中比在人永生化头颈部细胞系(okf6)中以更高的水平甲基化(图7a,上部小图)。相应地,mir124在fadu细胞系中的表达水平比在okf6细胞系中的表达水平更低(图7a,下部小图)。在相同细胞系中的mgmir137的甲基化水平和mir137的表达水平之间观察到类似的负相关。(图7b,上部小图和下部小图)。在用脱甲基化试剂,5-氮杂胞苷(5-氮杂)处理后,进一步地证实了mir124和mir137的表达由于dna甲基化而被沉默。如图8所示,在5-氮杂处理后,mir124和mir137的表达水平恢复。实施例6评估hnscc中mir124和mir137的肿瘤抑制功能。为了确定mir124和mir137在hnscc肿瘤发生中的功能性作用,通过转染将mir124或mir137的mirna模拟物引入到fadu细胞系中(图9a)。在转染后72小时进行mtt细胞增殖试验。通过恢复mir124或mir137表达,细胞增殖受到显著抑制(图9b)。进一步地评估了mir124或mir137对调节hnscc的肿瘤起始细胞(tic)的影响。使用两种功能上相关的试验测定了mir124和mir137在tic形成和膨胀上的作用:球形成能力和侧群细胞(sp)。球体形成试验已经被验证为在评估hnscc中tic的自我更新和形成的能力是可靠的。另外,sp试验,一种依赖于干细胞外排hoechst染料的能力的功能性分选方法,对于测定hnscc中的tic膨胀是一种可靠的标志物。当将mir124或mir137模拟物引入至fadu细胞中时,显著地破坏tic的球形成能力并且sp的百分数降低。这些结果表明,mir124和mir137二者都抑制hnscc中的ict形成和膨胀(图9c)。实施例7鉴定hnscc中mir124和mir137的下游共同靶标假定mir124和mir137的下游共同靶标(一个或多个)可以显性地介导mir124和mir137在hnscc肿瘤发生中的功能。通过搜索targetscan微mirna数据库(6.2版本),发现了3个潜在mir124和mir137的共同靶标,即ezh2、cdk6、akt2和e2f6。对于脊椎动物中保守的mir124和mir137两者,这些潜在的共同靶标中的每一个在它们的3'-utr内具有>7-mer结合位点(图10)。鉴于它维持角化细胞和tic中的正常干细胞的功能相关性,ezh2是主要的焦点。如图11所示,mir124或mir137的恢复显著降低ezh2在mrna(a)和蛋白质(b)水平两者上的表达。参考文献:可以在整个本申请中所引用的或以下列出的所有引用的参考文献(包括文献参考、专利、专利申请和网站)的内容通过引用以其全部内容在此明确地并入本公开以用于任何目的。除非另有规定,本公开可以使用在本领域公知的免疫学、分子生物学和细胞生物学的常规技术。本公开还通过引用以其全部内容并入了在分子生物学的领域熟知的技术和方法。这些技术包括,但不限于,在下面出版物中描述的技术。1.watsonjd,crickfh.molecularstructureofnucleicacids;astructurefordeoxyribosenucleicacid.nature.1953;171(4356):737-8.2.leerc,feinbaumrl,ambrosv.thec.elegansheterochronicgenelin-4encodessmallrnaswithantisensecomplementaritytolin-14.cell.1993;75(5):843-54.3.ambrosv.thefunctionsofanimalmicrornas.nature.2004;431(7006):350-5.4.lewis,b.,shih,i.,jones-rhoades,m.,bartel,d.&burge,c.predictionofmammalianmicrornatargets.cell115,787–798(2003).5.krek,d.等人.combinatorialmicrornatargetpredictions.naturegenet.37,495–500(2005).6.betel,d.,wilson,m.,gabow,a.,marks,d.s.&sander,c.themicrorna.orgresource:targetsandexpression.nucleicacidsres.36,d149–d153(2008).7.friedman,r.c.,farh,k.k.,burge,c.b.&bartel,d.p.mostmammalianmrnasareconservedtargetsofmicrornas.genomeres.19,92–105(2009).8.garzonr,marcuccig,crocecm.targetingmicrornasincancer:rationale,strategiesandchallenges.natrevdrugdiscov.2010;9(10):775-89.9.worshammj,alih,dragovicj,schweitzervp.molecularcharacterizationofheadandneckcancer:howclosetopersonalizedtargetedtherapy?.moldiagnther.2012;16(4):209-22.10.minorj,wangx,zhangf,等人.methylationofmicrorna-9isaspecificandsensitivebiomarkerfororalandoropharyngealsquamouscellcarcinomas.oraloncol.2012;48(1):73-8.11.boylejo,maol,brennanja,等人.genemutationsinsalivaasmolecularmarkersforheadandnecksquamouscellcarcinomas.amjsurg.1994;168(5):429-32.12.worshammj,chenkm,ghanemt,stephenjk,divineg.epigeneticmodulationofsignaltransductionpathwaysinhpv-associatedhnscc.otolaryngolheadnecksurg.2013;149(3):409-16.13.jemal,a.;siegel,r.;ward,e.;hao,y.;xu,j.;thun,m.j.,cancerstatistics,2009.ca:acancerjournalforclinicians2009,59(4),225-49.14.siegel,r.;naishadham,d.;jemal,a.,cancerstatistics,2013.ca:acancerjournalforclinicians2013,63(1),11-30.15.jemal,a.;siegel,r.;ward,e.;hao,y.;xu,j.;thun,m.j.,cancerstatistics,2009.ca:acancerjournalforclinicians2009,59(4),225-49.16.siegel,r.;naishadham,d.;jemal,a.,cancerstatistics,2013.ca:acancerjournalforclinicians2013,63(1),11-30.17.iorio,m.v.;croce,c.m.,micrornadysregulationincancer:diagnostics,monitoringandtherapeutics.acomprehensivereview.embomolmed2012,4(3),143-59.18.croce,c.m.,causesandconsequencesofmicrornadysregulationincancer.natrevgenet2009,10(10),704-14.19.lujambio,a.;calin,g.a.;villanueva,a.;ropero,s.;sanchez-cespedes,m.;blanco,d.;montuenga,l.m.;rossi,s.;nicoloso,m.s.;faller,w.j.;gallagher,w.m.;eccles,s.a.;croce,c.m.;esteller,m.,amicrornadnamethylationsignatureforhumancancermetastasis.procnatlacadsciusa2008,105(36),13556-61.20.(a)gangaraju,v.k.;lin,h.,micrornas:keyregulatorsofstemcells.natrevmolcellbiol2009,10(2),116-25;(b)rosenfeld,n.;aharonov,r.;meiri,e.;rosenwald,s.;spector,y.;zepeniuk,m.;benjamin,h.;shabes,n.;tabak,s.;levy,a.;lebanony,d.;goren,y.;silberschein,e.;targan,n.;ben-ari,a.;gilad,s.;sion-vardy,n.;tobar,a.;feinmesser,m.;kharenko,o.;nativ,o.;nass,d.;perelman,m.;yosepovich,a.;shalmon,b.;polak-charcon,s.;fridman,e.;avniel,a.;bentwich,i.;bentwich,z.;cohen,d.;chajut,a.;barshack,i.,micrornasaccuratelyidentifycancertissueorigin.natbiotechnol2008,26(4),462-9.21.iorio,m.v.;piovan,c.;croce,c.m.,interplaybetweenmicrornasandtheepigeneticmachinery:anintricatenetwork.biochimbiophysacta2010,1799(10-12),694-701.22.saito,y.;liang,g.;egger,g.;friedman,j.m.;chuang,j.c.;coetzee,g.a.;jones,p.a.,specificactivationofmicrorna-127withdownregulationoftheproto-oncogenebcl6bychromatin-modifyingdrugsinhumancancercells.cancercell2006,9(6),435-43.23.hildebrandt,m.a.;gu,j.;lin,j.;ye,y.;tan,w.;tamboli,p.;wood,c.g.;wu,x.,hsa-mir-9methylationstatusisassociatedwithcancerdevelopmentandmetastaticrecurrenceinpatientswithclearcellrenalcellcarcinoma.oncogene29(42),5724-8.24.saito,y.;jones,p.a.,epigeneticactivationoftumorsuppressormicrornasinhumancancercells.cellcycle2006,5(19),2220-2.25.minor,j.;wang,x.;zhang,f.;song,j.;jimeno,a.;wang,x.j.;lu,x.;gross,n.;kulesz-martin,m.;wang,d.;lu,s.l.,methylationofmicrorna-9isaspecificandsensitivebiomarkerfororalandoropharyngealsquamouscellcarcinomas.oraloncol2012,48(1),73-8.26.jones,p.a.;baylin,s.b.,theepigenomicsofcancer.cell2007,128(4),683-92.27.ha,p.k.;califano,j.a.,promotermethylationandinactivationoftumour-suppressorgenesinoralsquamous-cellcarcinoma.lancetoncol2006,7(1),77-82.28.babu,j.m.;prathibha,r.;jijith,v.s.;hariharan,r.;pillai,m.r.,amir-centricviewofheadandneckcancers.biochimbiophysacta2011,1816(1),67-72.29.roh,j.l.;westra,w.h.;califano,j.a.;sidransky,d.;koch,w.m.,tissueimprintformolecularmappingofdeepsurgicalmarginsinpatientswithheadandnecksquamouscellcarcinoma.headneck2012,34(11),1529-36.当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1