一种切割效率增强的HRV3C蛋白酶底物突变体及其应用的制作方法
本发明属于生物工程技术领域,具体而言,涉及一种切割效率增强的hrv3c蛋白酶底物序列突变体及其应用。
背景技术:
人鼻病毒(hrv)是单股正链小rna病毒,其编码的3c蛋白酶属于半胱氨酸蛋白酶家族,可以识别多肽序列levlfq↓gp,并在gln和gly之间进行切割。hrv3c蛋白酶具有特异性强,酶活高等特性,即使在4℃低温条件下依然有较高的酶活。目前,hrv3c蛋白酶已广泛商品化,特别是用来在蛋白纯化过程中移除纯化标签。然而,作为一种高价值的商业酶,hrv3c蛋白酶的底物特异性还没有相关报道。
另外,蛋白酶底物切割位点特异性的研究是全面了解和应用蛋白酶的一个必要的过程,而传统的研究方法是定点突变后,表达、纯化得到相应底物突变体,然后进行体外反应,检测、定量分析。该方法工作量大,操作复杂,效率低,操作误差大,不利于实验的高效开展,因此很难筛选得到切割效率增强的hrv3c蛋白酶底物序列突变体。
技术实现要素:
鉴于现有技术的不足,本发明开发了一种利用酵母内质网滞留筛选系统(yess)来快速解析蛋白酶底物切割位点特异性的新方法--yess-pssc。
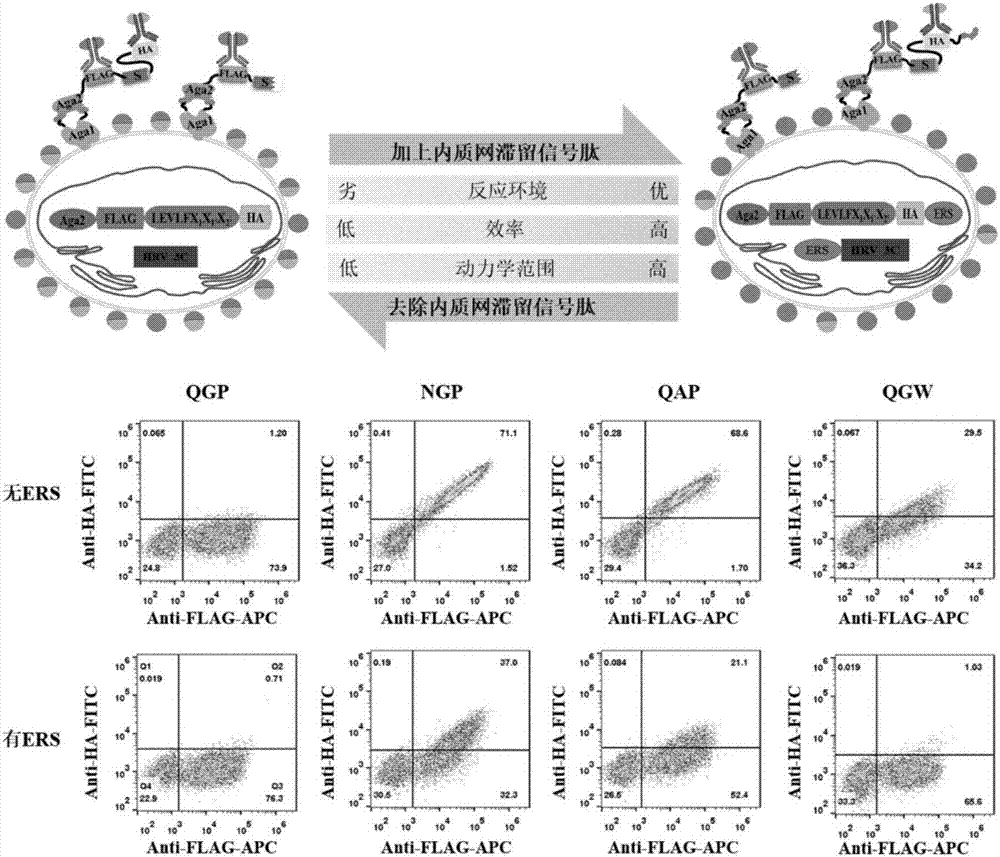
酵母内质网滞留信号肽(ers,ersequestrationsignal)是存在于蛋白羧基端的一段6个氨基酸长度的多肽(fehdel),它可以与内质网内膜上的蛋白相互作用,使携带滞留信号肽的蛋白往返穿梭于内质网与高尔基体中以延长蛋白在内质网与高尔基体中的滞留时间,增加酶动力学范围。aga2为酿酒酵母表面的α凝集素成分,外源蛋白可借助与aga2的融合表达而在酵母细胞表面被展示;所使用的荧光标签分别是flag和ha,对应的氨基酸序列分别是dykddddk和ypydvpdya,当各自对应的荧光抗体anti-flag-apc和anti-ha-fitc与之相结合时,根据底物的不同切割情况,可通过流式细胞仪检测到相应的荧光信号。
在带内质网滞留信号肽和不带内质网滞留信号肽两种情况下,都得到hrv3c-p底物p1、p1’、p2’3个位点各3种突变体底物。其中,特异性最高的p1位点包括氨基酸为q、n、f的3种底物,特异性较高的p1’位点包括氨基酸为g、a、d的3种底物,特异性较低的p2’位点包括氨基酸为p、f、w的3种底物,然后让酶和底物复合体在内质网中发生反应,不同切割情况的底物会展示在细胞表面。当发明人用对应的荧光抗体anti-flag-apc和anti-ha-fitc孵育细胞时,不被切割的底物复合体可以同时被两种荧光抗体anti-flag-apc和anti-ha-fitc标记;而被切割的底物复合体只能被荧光抗体anti-flag-apc标记。所以,根据细胞表面展示底物的不同切割情况,流式细胞仪可以检测到不同的信号,取一定量的细胞分析即可得到该底物突变体的切割效率。即说明可以利用酵母内质网滞留筛选系统(yess)来解析蛋白酶底物切割位点特异性。结果表明:对于特异性高的p1和p1’位点底物突变体,展示时,酶和底物端都需要加内质网滞留信号肽(fehdel);对于特异性低的p2’位点底物突变体展示时,酶和底物端都不需要加内质网滞留信号肽;则可以通过酵母内质网滞留信号肽的不同应用来解析hrv3c蛋白酶针对其底物切割位点p1(q),p1’(g),p2’(p)的特异性(参见图2)。
因此,本发明利用酵母内质网滞留筛选系统(yess)快速解析蛋白酶底物切割位点特异性,最终提供了一种切割效率增强的hrv3c蛋白酶底物序列突变体及其应用,具体的技术方案概况如下:
本发明提供一种hrv3c蛋白酶底物变体,其包含序列表中seqidno.1所示的氨基酸序列。进一步优选地,所述hrv3c蛋白酶底物变体,其为序列表中seqidno.1所示的氨基酸序列。
在本发明的一个具体的实施例中,上述的hrv3c蛋白酶底物变体,其为mbp-substrare-6xhis-gst,其中substrare为序列表中seqidno.1所示的氨基酸序列。
另一方面,本发明还可以提供编码上述hrv3c蛋白酶底物变体的核苷酸序列;以及含有该核苷酸序列的重组表达载体。
另一方面,本发明还可以提供一种遗传工程化的宿主细胞,所述的细胞含有上述的重组表达载体。进一步优选地,所述的宿主细胞为大肠杆菌。
最后一方面,本发明还可以提供上述hrv3c蛋白酶底物变体在蛋白纯化过程中移除纯化标签中的应用。
与现有技术相比,本发明具有如下优点和显著进步:
(1)为了解析hrv3c蛋白酶底物切割位点p1(q),p1’(g),p2’(p)特异性,发明人对hrv3c蛋白酶底物切割位点p1(q),p1’(g),p2’(p)分别进行饱和突变,然后利用酵母内质网滞留筛选系统(yess)来解析蛋白酶底物切割位点特异性。我们的研究表明,hrv3c蛋白酶在p1和p1’位点分别偏好gln和gly,然而在p2’位点相对原始的pro氨基酸,更偏好met。yess-pssc对hrv3c-p底物特异性的成功解析,证明了该方法广泛用于快速解析蛋白酶底物特异性的可行性和优越性;并找到了一种切割效率优于原始hrv3c-p的底物序列的新底物序列-levlfq↓gm。新底物序列-levlfq↓gm-切割效率高,且切割残基中的met可以作为蛋白质的第一个氨基酸,即只有一个gly残留,对一些药用蛋白在去除融合标签后的应用中,引起免疫反应的可能更低。所以,底物序列levlfqgm作为hrv3c-p的新型底物具有更高的价值。
(2)针对特异性高的p1和p1’位点底物突变体(levlfxgp和levlfqxp),试验中需要一个高效的反应体系,因此酶和底物复合体形式分别为hrv3c-ers和aga2-flag-substrare-ha-ers;但对于特异性低的p2’位点底物突变体(levlfqgx),只有在一个低效的反应体系中才能呈现不同突变体的差异,即酶和底物复合体形式为hrv3c和aga2-flag-substrare-ha。本发明通过酵母内质网滞留信号肽的不同应用扩展了这一方法的动力学范围,快速、准确的解析了hrv3c蛋白酶针对其底物切割位点p1(q),p1’(g),p2’(p)的特异性,在开发了一种简单易行的方法的同时,发现了一种hrv3c蛋白酶可以更高效切割的底物,从而有利于hrv3c蛋白酶的进一步应用和研究。
(3)本发明获得的hrv3c蛋白酶底物突变体序列levlfqgm在实际应用中具有更为高效的切割优势。将含有hrv3c蛋白酶与底物编码序列的质粒转化到大肠杆菌表达菌株bl21(de3)感受态细胞中,诱导蛋白表达后纯化,并用sds-page检测纯化结果。hrv3c蛋白酶的底物由融合蛋白mbp和gst融合表达,其中mbp大小为42kda,gst大小为26kda,底物融合蛋白mbp-levlfqgp/m-6xhis-gst在70kda左右。纯化得到的hrv3c蛋白酶和底物融合蛋白按照摩尔比为1:10比例混合,分别在4℃、16℃、22℃、30℃反应,sds-page检测酶切结果。从图4可知,hrv3c蛋白酶可以高效切割mbp-levlfqgp/m-6xhis-gst融合蛋白,将底物融合蛋白切割成42kda的mbp和26kda的gst,而且切割底物levlfqgm的效率明显高于levlfqgp。
附图说明
图1:本发明方法质粒构建形式示意图;
图2:利用酵母内质网滞留筛选系统(yess)来快速、灵敏的解析蛋白酶底物切割位点特异性的原理图;
图3:hrv3c蛋白酶底物切割位点p1(q),p1’(g),p2’(p)各位点饱和突变体底物切割效率结果;
图4:hrv3c蛋白酶原始底物序列和突变体序列levlfqgm在4℃、16℃、22℃、30℃的体外反应的sds-page胶图。
具体实施方式
以下结合附图对本发明的原理和特征进行描述,所举实例只用于解释本发明,并非用于限定本发明的范围。下述实施例中所使用的实验方法如无特殊说明,均为常规方法。下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
实施例1构建hrv3c蛋白酶及其底物突变体在酿酒酵母中诱导表达的质粒
(1)通过三轮聚合酶链式反应(pcr)扩增得到底物突变体基因片段。其中,第一轮pcr扩增进行定点突变,得到hrv3c蛋白酶底物切割位点p1、p1’、p2’的突变体底物基因;第二轮pcr扩增确定基因片段是否带滞留信号肽基因;第三轮pcr扩增带上限制性内切酶酶切基因序列,得到完整基因。其中,p1位点带有滞留信号肽基因的有20种氨基酸饱和突变体形式,不带滞留信号肽基因的有氨基酸为q、n、f共3种形式的突变体基因;p1’位点带有滞留信号肽基因的有20种氨基酸饱和突变体形式,不带滞留信号肽基因的有氨基酸为g、a、d共3种形式的突变体基因;p2’位点不带滞留信号肽基因的有20种氨基酸饱和突变体形式,带有滞留信号肽基因的有氨基酸为p、f、w共3种形式的突变体基因。
pcr反应体系:10×pfubuffer,5μl;dntp(2.5mm),5μl;引物f(10μm),2μl;引物r(10μm),2μl;pfu聚合酶,0.5μl;模板(载体pesd),1μl;加ddh2o至反应体系为50μl。
pcr扩增体系:94℃,3min;94℃,30s,58℃,20s,72℃,30s,25个循环;72℃,5min;4℃,∞。
设计的引物列表如下:
(2)将步骤(1)获得的pcr产物回收后,用限制性内切酶pstⅰ和ecorⅰ双酶切,再把经过限制性核酸内切酶pstⅰ和ecorⅰ双酶切后回收的载体pesd和片段以t4dnaligase酶连;酶连产物转化到大肠杆菌感受态细胞中,挑选转化子进行测序验证。
最终载体注释:ers-hrv3c-gal1-gal10-aga2-flag-levlfx1x1’x2’-ha-ers(fehdel)、hrv3c-gal1-gal10-aga2-flag-levlfx1x1’x2’-ha(参见图1)。
实施例2含有hrv3c蛋白酶及其底物突变体的质粒在酿酒酵母中的诱导表达
将最终载体转化于酿酒酵母eby100,转化所用的试剂为:eby100感受态细胞,20μl;重组质粒,1.5μl;singlestrandcarrierdna,25μl;1mliac,36μl;peg4000(50%w/v),240μl;混合好体系后,30℃水浴30min,转移至42℃热激20min,离心收集菌体,加1mlypd液体培养基于30℃摇床培养1.5hr,用超纯水洗细胞一次后,涂布于ynb-caa-glucose固体培养基,30℃培养箱培养2-3days。ynb-caa-glucose固体培养基成分为20g/l葡萄糖,6.7g/lynb,5.4g/lna2hpo4,8.6g/lnah2po4·h2oand5g/lcasaminoacids,ph7.4,15g/lagar。
接种单菌落于液体培养基ynb-caa-glucose中,30℃,230rpm摇床中培养至od600=3左右,转移至ynb-caa-galactose液体培养基于30℃、230rpm摇床中培养,并使起始od600=0.6,诱导8hr后取样,ynb-caa-galactose固体培养基成分为20g/l半乳糖,6.7g/lynb,5.4g/lna2hpo4,8.6g/lnah2po4·h2oand5g/lcasaminoacids,ph7.4,15g/lagar。
实施例3应用流式细胞仪检测hrv3c蛋白酶及其底物突变体的反应结果
取106个细胞,加200μlbuffera液混匀后离心沉淀细胞,3000rpm,4℃,1min。再用bufferb液洗细胞,去上清,然后每个样品加20μlbufferb液,0.15μlfitc,0.15μlapc,4℃避光标记15min,转移至室温避光标记30min。离心去上清,再用150μlbufferb洗一次,继续用1xpbs洗一次后,并用300μl重悬。重悬的细胞用于cytoflex流式细胞仪分析,检测的荧光信号通道是fitc(525/40nm)和apc(660/20nm)。从流式细胞仪中给出的结果分析得出hrv3c蛋白酶对其底物突变体的切割效率(参见图3)。
实施例4hrv3c蛋白酶底物突变体质粒的构建及其在大肠杆菌中诱导表达
(1)通过聚合酶链式反应(pcr)扩增得到hrv3c蛋白酶的底物突变体基因片段。
pcr反应体系:10×pfubuffer,5μl;dntp(2.5mm),5μl;引物f(10μm),2μl;引物r(10μm),2μl;pfu聚合酶,0.5μl;模板(载体ly105:即表达原始底物的质粒),1μl;加ddh2o至反应体系为50μl。
pcr扩增体系:94℃,3min;94℃,30s,58℃,15s,72℃,30s,25个循环;72℃,5min;4℃,∞。
设计的引物列表如下:
(2)将步骤(1)获得的pcr产物回收后,用限制性核酸内切酶ncoⅰ和hindⅲ双酶切,再与经过限制性核酸内切酶ncoⅰ和hindⅲ双酶切后的载体ly105酶连;酶连产物直接转化到大肠杆菌感受态细胞中,挑选转化子经测序验证,得到重组质粒。最终载体注释:mbp-substrate-6xhis-gst(substrate为levlfqgp或levlfqgm)。
(3)把上述步骤(2)得到的质粒及已有的原始的底物和酶的表达质粒转化入大肠杆菌感受态bl21(de3)中,涂平板后,在37℃培养。
(4)从平板上挑取含有质粒的大肠杆菌表达菌株接种到2mllb培养液中,37℃摇床培养10hr;转接到200mllb培养液(加有相应抗生素)中,37℃摇床培养3hr左右,测其od600在0.5-0.6,加入1miptg诱导蛋白表达,使其终浓度为0.5mm,18℃摇床诱导20hr;
(5)4℃、5000rpm离心10min收集菌体,去掉上清,用无菌水重悬,再离心来洗涤细胞;
(6)用lysisbuffer重悬细胞,加入lysozyme混匀后4℃搅拌1hr;
(7)超声波破菌,超3s停6s,35%功率破菌10min;
(8)4℃、17000rpm离心30min离心破菌液,使破菌碎片与上清液完全分离;
(9)取1mlni-nta6xhis树脂填充到蛋白纯化柱中,用washbuffer洗涤树脂2次;
(10)将破菌上清加入到蛋白纯化柱中,静置后待树脂沉降下来,将杂蛋白释放出来;
(11)用washbuffer洗涤树脂,去掉未结合的杂蛋白;
(12)用elutionbuffer洗脱目标蛋白,用过的树脂回收;
(13)纯化得到的hrv3c蛋白酶和底物融合蛋白按照摩尔比为1:10比例混合,分别在4℃、16℃、22℃、30℃反应1hr,2hr和4hr,sds-page检测酶切结果。
结果显示hrv3c-p突变底物序列levlfqgm的切割效率比序列levlfqgp的高(参见图4)。按反应时间为1hr计算,在4℃、16℃、22℃、30℃反应条件下,hrv3c蛋白酶变体底物序列levlfqgm的切割效率分别是序列levlfqgp的1.03倍、1.16倍、1.39倍、1.73倍。
需要说明的是,虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
序列表
<110>湖北大学
<120>一种切割效率增强的hrv3c蛋白酶底物突变体及其应用
<160>1
<170>siposequencelisting1.0
<210>1
<211>8
<212>prt
<213>人工序列(artificialsequence)
<400>1
leugluvalleupheglnglymet
15
- 还没有人留言评论。精彩留言会获得点赞!