转座酶组合物、制备方法和筛选方法与流程
相关申请的交叉引用
本申请要求2017年8月30日提交的临时申请ussn62/552,214的权益,其内容通过引用整体并入本文。
序列表的并入
命名为“rmsi-010_001woseqlisting_st25.txt”的文本文件(其创建于2018年8月28日且大小为60.4kb)的内容特此通过引用以其整体并入。
本公开内容涉及分子生物学的领域;且更具体地涉及能够实现突变的和诱变处理的转座酶的快速且有效高通量筛选的分子工具。
背景
在本领域中存在长期感觉到但是未得到满足的对分子工具的需要,所述分子工具能够实现突变的和诱变处理的转座酶的快速且有效高通量筛选以鉴别为转座酶作为分子工具的应用带来期望特征的罕见突变。本公开内容提供了系统和方法来解决这些长期感觉到但是未得到满足的需要。
技术实现要素:
本公开内容提供了筛选多个转座酶的方法,所述方法包括:(a)在足以诱导包含第一末端序列的第一寡核苷酸的转座的条件下使第一转座酶与第一核酸样品接触,由此产生具有所述第一末端序列的第一多个插入位点的第一转座的核酸样品;(b)在足以诱导包含第二末端序列的第二寡核苷酸的转座的条件下使第二转座酶与第二核酸样品接触,由此产生具有第二多个插入位点的第二转座的核酸样品,第二转座酶具有与第一转座酶相差至少一个氨基酸的氨基酸序列;(c)对第一转座的核酸样品的第一多个插入位点的至少一部分测序,由此产生第一组测序读出,所述第一组测序读出中的每一个包含所述第一末端序列的插入位点之一;(d)对所述第二转座的核酸样品的第二多个插入位点的至少一部分测序,由此产生第二组测序读出,所述第二组测序读出中的每一个包含所述第二末端序列的插入位点之一;(e)将所述第一组测序读出与所述第二组测序读出对比;和(f)基于对比的步骤(e),指定所述第二转座酶显著不同于所述第一转座酶的概率。
在本公开内容的筛选多个转座酶的方法的某些实施方案中,对比的步骤(e)包括:为所述第一组测序读出计算在每个核苷酸位置处的每种可能核苷酸碱基的频率,由此产生第一组频率值;为所述第二组测序读出计算在每个核苷酸位置处的每种可能核苷酸碱基的频率,由此产生第二组频率值;为在每个核苷酸位置处的每种可能核苷酸碱基计算所述第一组频率值和所述第二组频率值之间的绝对差异,由此产生绝对差异值的组;和将所述绝对差异值中的每一个平均化,由此确定基序间距离。
在本公开内容的筛选多个转座酶的方法的某些实施方案中,对比的步骤(e)包括:为所述第一组测序读出测量或确定在每个核苷酸位置处的每种可能核苷酸碱基的频率,由此产生第一组频率值;为所述第二组测序读出测量或确定在每个核苷酸位置处的每种可能核苷酸碱基的频率,由此产生第二组频率值;为在每个核苷酸位置处的每种可能核苷酸碱基测量或确定所述第一组频率值和所述第二组频率值之间的绝对差异,由此产生绝对差异值的组;和将所述绝对差异值中的每一个平均化,由此确定基序间距离。
在本公开内容的筛选多个转座酶的方法的某些实施方案中,指定的步骤(f)包括:产生由模拟的随机序列读出定义的基序间距离概率图;和基于在步骤(e)中确定的基序间距离中的每一个和所述基序间距离概率图,指定所述第二转座酶显著不同于所述第一转座酶的概率值。
在本公开内容的筛选多个转座酶的方法的某些实施方案中,对比的步骤(e)包括:在与第一转座的核酸样品中的第一多个插入位点对应的位置处,计算在第一参照核酸样品内的确定长度的区段处的覆盖的第一测序深度;在与第二转座的核酸样品中的第二多个插入位点对应的位置处,计算在第一参照核酸样品内的确定长度的区段处的覆盖的第二测序深度;和将所述覆盖的第一测序深度与所述覆盖的第二测序深度对比。
在本公开内容的筛选多个转座酶的方法的某些实施方案中,对比的步骤(e)包括:在与第一转座的核酸样品中的第一多个插入位点对应的位置处,测量或确定在第一参照核酸样品内的确定长度的区段处的覆盖的第一测序深度;在与第二转座的核酸样品中的第二多个插入位点对应的位置处,测量或确定在第一参照核酸样品内的确定长度的区段处的覆盖的第二测序深度;和将所述覆盖的第一测序深度与所述覆盖的第二测序深度对比。
在本公开内容的筛选多个转座酶的方法的某些实施方案中,指定的步骤(f)包括:执行mann-whitney检验(关于平均值差异)、kolmogorov-smirnoff检验(关于不同的分布形状)、参数检验、非参数检验、形状差异的目检和基于百分位数的度量计算中的至少一种。
在本公开内容的筛选多个转座酶的方法的某些实施方案中,对比的步骤(e)包括:在与第一转座的核酸样品中的第一多个插入位点对应的位置处,计算在第一参照核酸样品中的确定长度的核酸区段的第一gc含量分数;在与第二转座的核酸样品中的第二插入位点对应的位置处,计算在第一参照核酸样品中的确定长度的核酸区段的第二gc含量分数;和鉴别所述第一gc含量分数和所述第二gc含量分数之间的差异。
在本公开内容的筛选多个转座酶的方法的某些实施方案中,对比的步骤(e)包括:在与第一转座的核酸样品中的第一多个插入位点对应的位置处,测量或确定在第一参照核酸样品中的确定长度的核酸区段的第一gc含量分数;在与第二转座的核酸样品中的第二插入位点对应的位置处,测量或确定在第一参照核酸样品中的确定长度的核酸区段的第二gc含量分数;和鉴别所述第一gc含量分数和所述第二gc含量分数之间的差异。
在本公开内容的筛选多个转座酶的方法的某些实施方案中,指定的步骤(f)包括:执行mann-whitney检验(关于平均值差异)、kolmogorov-smirnoff检验(关于不同的分布形状)、参数检验、非参数检验、形状差异的目检和基于百分位数的度量计算中的至少一种。
本公开内容提供了一种包含核酸和编码转座酶的独特核酸序列的组合物,所述核酸从5'至3'包含:(a)第一转座子末端序列,(b)唯一标识符(uid)条形码,和(c)第二转座子末端序列,其中所述核酸能够转座。在某些实施方案中,所述核酸从5'至3'进一步包含位于所述唯一标识符(uid)条形码和所述第二转座子末端序列之间的选择标记。在某些实施方案中,所述uid条形码与所述编码转座酶的独特核酸序列有关。
在本公开内容的组合物的某些实施方案中,包含元件(a)至(c)的核酸不包含所述编码转座酶的独特核酸序列。在某些实施方案中,第一载体包含含有元件(a)至(c)的核酸,且第二载体包含所述编码转座酶的独特核酸序列。
在本公开内容的组合物的某些实施方案中,包含元件(a)至(c)的核酸进一步包含所述编码转座酶的独特核酸序列。在某些实施方案中,所述编码转座酶的独特核酸序列位于所述第一转座子末端序列的5'。在某些实施方案中,载体包含含有元件(a)至(c)的核酸和所述编码转座酶的独特核酸序列。
在本公开内容的组合物的某些实施方案中,所述uid条形码包含5-200个碱基对,包括端点。在某些实施方案中,所述uid条形码包含10-100个碱基对,包括端点。在某些实施方案中,所述uid条形码包含10-50个碱基对,包括端点。在某些实施方案中,所述uid条形码包含15-25个碱基对,包括端点。
在本公开内容的组合物的某些实施方案中,所述uid条形码与所述编码转座酶的独特核酸序列相关。本文中使用的术语“相关”意在描述uid条形码的核酸序列与编码转座酶的独特核酸序列匹配的数据库中的记录。在本公开内容的方法的某些实施方案中,在开始所述方法之前,可以将所述uid条形码和所述编码转座酶的独特核酸序列测序。此外,在本公开内容的方法的某些实施方案中,在开始所述方法之前,所述uid条形码和所述编码转座酶的独特核酸序列可以是相关的。
在本公开内容的组合物的某些实施方案中,所述转座酶是野生型转座酶。在某些实施方案中,所述野生型转座酶分离或衍生自任何物种。
在本公开内容的组合物的某些实施方案中,所述转座酶是野生型转座酶。在某些实施方案中,所述野生型转座酶是野生型tnaa-转座酶。在某些实施方案中,所述野生型tnaa-转座酶包含seqidno:2的氨基酸序列。
在本公开内容的组合物的某些实施方案中,所述转座酶是野生型转座酶。在某些实施方案中,所述野生型转座酶是野生型tn5-转座酶。在某些实施方案中,所述野生型tn5-转座酶包含seqidno:17的氨基酸序列。
在本公开内容的组合物的某些实施方案中,所述转座酶是突变型转座酶。在某些实施方案中,所述突变型转座酶具有与野生型转座酶相比增加的转座酶活性。在某些实施方案中,所述突变型转座酶具有与野生型转座酶相比减少的插入位点偏倚。在某些实施方案中,所述突变型转座酶包含至少一个已知的或天然存在的突变。
在本公开内容的组合物的某些实施方案中,所述转座酶是突变型转座酶。在某些实施方案中,所述突变型转座酶包含至少一个已知的或天然存在的突变。在某些实施方案中,所述突变型转座酶是突变型tnaa-转座酶。在某些实施方案中,所述突变型转座酶是突变型tn5-转座酶。
在本公开内容的组合物的某些实施方案中,所述转座酶是突变型转座酶。在某些实施方案中,所述突变型转座酶是突变型tnaa-转座酶。在某些实施方案中,所述突变型tnaa-转座酶包含p47k或m50a。在某些实施方案中,所述突变型tnaa-转座酶包含p47k。在某些实施方案中,包括其中所述突变型tnaa-转座酶包含p47k的那些,所述突变型tnaa-转座酶包含seqidno:5的氨基酸序列。在某些实施方案中,所述突变型tnaa-转座酶包含m50a。在某些实施方案中,包括其中所述突变型tnaa-转座酶包含m50a的那些,所述突变型tnaa-转座酶包含seqidno:4的氨基酸序列。在某些实施方案中,所述突变型tnaa-转座酶包含p47k和m50a。在某些实施方案中,包括其中所述突变型tnaa-转座酶包含p47k和m50a的那些,所述突变型tnaa-转座酶包含seqidno:3的氨基酸序列。
在本公开内容的组合物的某些实施方案中,所述转座酶是突变型转座酶。在某些实施方案中,所述突变型转座酶包含在特定位置处的突变,其在功能上等同于在根据seqidno:17的序列的位置30、40、41、47、54、56、62、97、110、188、212、319、322、326、330、333、342、344、345、348、372、438、445、462或466处在tn5-转座酶中的突变。
在某些实施方案中,所述突变型转座酶是突变型tn5-转座酶。本公开内容的突变型tn5-转座酶可以包括、但不限于在例如uniprot.org/uniprot/q46731处提供的突变。在某些实施方案中,所述突变型tn5-转座酶包含在根据seqidno:17的序列的位置30、40、41、47、54、56、62、97、110、188、212、319、322、326、330、333、342、344、345、348、372、438、445、462或466处的突变。在某些实施方案中,所述突变型tn5-转座酶包含根据seqidno:17的序列的r30q、k40q、y41h、t47p、e54k、e54v、m56a、r62q、d97a、e110k、d188a、k212m、y319a、r322a、r322k、e326a、k330a、k330r、k333a、k333r、r342a、r344a、e345k、n348a、l372p、s438a、k438a、s445a、g462d或a466d。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比降低转座酶活性的突变,包括、但不限于,根据seqidno:17的序列的r30q、k40q、r62q、d97a、e326a、k330a和s445a。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比增加转座酶活性的突变,包括、但不限于,根据seqidno:17的序列的r62q、d97a、e110k、d188a和l372p。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比降低dna裂解活性的突变,包括、但不限于,根据seqidno:17的序列的k333a和k333r。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比降低链转移活性的突变,包括、但不限于,根据seqidno:17的序列的y319a、r322a、r322k、k333a和k333r。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比增加转座频率的突变,包括、但不限于,根据seqidno:17的序列的y41h、t47p、e54k和e54v。在某些实施方案中,所述突变型tn5-转座酶包含消除转座酶抑制剂的表达的突变,包括、但不限于,根据seqidno:17的序列的m56a。在某些实施方案中,所述突变型tn5-转座酶包含根据seqidno:17的序列的e54k、m56a或l372p。在某些实施方案中,所述突变型tn5-转座酶包含根据seqidno:17的序列的e54k、m56a和l372p(在本文中也被称作“超活跃的tn5-转座酶)。在某些实施方案中,包括其中突变型tn5-转座酶包含根据seqidno:17的序列的e54k、m56a和l372p的那些,突变型tn5-转座酶包含seqidno:1的氨基酸序列。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比降低靶标特异性的突变,包括、但不限于根据seqidno:17的序列的k212m。
在本公开内容的组合物的某些实施方案中,所述转座酶是诱变处理过的转座酶。在某些实施方案中,编码诱变处理过的转座酶的独特核酸序列或编码诱变处理过的转座酶的序列已经(a)暴露于诱变剂或(b)进行随机诱变、定位诱变或它们的组合。在某些实施方案中,所述诱变剂是物理诱变剂。在某些实施方案中,所述物理诱变剂是电离辐射。在某些实施方案中,所述物理诱变剂是紫外辐射。在某些实施方案中,所述诱变剂是化学诱变剂。在某些实施方案中,所述化学诱变剂是活性氧、金属、脱氨剂或烷化剂。
在本公开内容的组合物的某些实施方案中,所述转座酶是诱变处理过的转座酶。在某些实施方案中,编码诱变处理过的转座酶的独特核酸序列或编码诱变处理过的转座酶的序列已经(a)暴露于诱变剂或(b)进行随机诱变、定位诱变、或它们的组合。在某些实施方案中,所述随机诱变包含(a)使编码诱变处理过的转座酶的序列与物理诱变剂和/或化学诱变剂接触,(b)对编码诱变处理过的转座酶的序列进行易出错的聚合酶链式反应(pcr),或(c)(a)和(b)的组合。在某些实施方案中,所述定位诱变包含丙氨酸扫描。在某些实施方案中,所述物理诱变剂是紫外辐射。在某些实施方案中,所述化学诱变剂包含烷化剂。在某些实施方案中,所述烷化剂包含n-乙基-n-亚硝基脲(enu)。在某些实施方案中,所述化学诱变剂包含甲磺酸乙酯(ems)。
在本公开内容的组合物的某些实施方案中,所述转座酶是诱变处理过的转座酶。在某些实施方案中,已经诱变处理过的,编码诱变处理过的转座酶的独特核酸序列或编码诱变处理过的转座酶的序列是编码野生型转座酶的序列。在某些实施方案中,所述编码野生型转座酶的序列或所述野生型转座酶分离或衍生自任何物种。
在本公开内容的组合物的某些实施方案中,所述转座酶是诱变处理过的转座酶。在某些实施方案中,已经诱变处理过的,编码诱变处理过的转座酶的独特核酸序列或编码诱变处理过的转座酶的序列是编码野生型转座酶的序列。在某些实施方案中,所述编码野生型转座酶的序列或所述野生型转座酶分离或衍生自任何物种。在某些实施方案中,所述野生型转座酶是野生型tnaa-转座酶。在某些实施方案中,包括其中野生型转座酶是野生型tnaa-转座酶的那些,所述野生型tnaa-转座酶包含seqidno:2的氨基酸序列。
在本公开内容的组合物的某些实施方案中,所述转座酶是诱变处理过的转座酶。在某些实施方案中,已经诱变处理过的,编码诱变处理过的转座酶的独特核酸序列或编码诱变处理过的转座酶的序列是编码野生型转座酶的序列。在某些实施方案中,所述编码野生型转座酶的序列或所述野生型转座酶分离或衍生自任何物种。在某些实施方案中,所述野生型转座酶是野生型tn5-转座酶。在某些实施方案中,包括其中野生型转座酶是野生型tn5-转座酶的那些,所述野生型tn5-转座酶包含seqidno:17的氨基酸序列。
在本公开内容的组合物的某些实施方案中,所述转座酶是诱变处理过的转座酶。在某些实施方案中,已经诱变处理过的,编码诱变处理过的转座酶的独特核酸序列或编码诱变处理过的转座酶的序列是编码突变型转座酶的序列。在某些实施方案中,所述编码突变型转座酶的序列或所述突变型转座酶分离或衍生自任何物种。在某些实施方案中,所述突变型转座酶包含至少一个非天然存在的突变。
在本公开内容的组合物的某些实施方案中,所述转座酶是诱变处理过的转座酶。在某些实施方案中,已经诱变处理过的,编码诱变处理过的转座酶的独特核酸序列或编码诱变处理过的转座酶的序列是编码突变型转座酶的序列。在某些实施方案中,所述编码突变型转座酶的序列或所述突变型转座酶分离或衍生自任何物种。在某些实施方案中,在诱变之前,所述突变型转座酶具有与野生型转座酶相比增加的转座酶活性。在某些实施方案中,在诱变之前,所述突变型转座酶具有与野生型转座酶相比减少的插入位点偏倚。在某些实施方案中,在诱变之前,所述突变型转座酶包含至少一个已知的或天然存在的突变。在某些实施方案中,在诱变之前,所述突变型转座酶是突变型tnaa-转座酶。在某些实施方案中,在诱变之前,所述突变型转座酶是突变型tn5-转座酶。
在本公开内容的组合物的某些实施方案中,所述转座酶是诱变处理过的转座酶。在某些实施方案中,已经诱变处理过的,编码诱变处理过的转座酶的独特核酸序列或编码诱变处理过的转座酶的序列是编码突变型转座酶的序列。在某些实施方案中,所述编码突变型转座酶的序列或所述突变型转座酶分离或衍生自任何物种。在某些实施方案中,所述突变型转座酶是突变型tnaa-转座酶。在某些实施方案中,所述突变型tnaa-转座酶包含p47k或m50a。在某些实施方案中,所述突变型tnaa-转座酶包含p47k。在某些实施方案中,包括其中所述突变型tnaa-转座酶包含p47k的那些,所述突变型tnaa-转座酶包含seqidno:5的氨基酸序列。在某些实施方案中,所述突变型tnaa-转座酶包含m50a。在某些实施方案中,包括其中所述突变型tnaa-转座酶包含m50a的那些,所述突变型tnaa-转座酶包含seqidno:4的氨基酸序列。在某些实施方案中,所述突变型tnaa-转座酶包含p47k和m50a。在某些实施方案中,包括其中所述突变型tnaa-转座酶包含p47k和m50a的那些,所述突变型tnaa-转座酶包含seqidno:3的氨基酸序列。
在本公开内容的组合物的某些实施方案中,所述转座酶是诱变处理过的转座酶。在某些实施方案中,已经诱变处理过的,编码诱变处理过的转座酶的独特核酸序列或编码诱变处理过的转座酶的序列是编码突变型转座酶的序列。在某些实施方案中,所述编码突变型转座酶的序列或所述突变型转座酶分离或衍生自任何物种。在某些实施方案中,所述突变型转座酶包含在特定位置处的突变,其在功能上等同于在根据seqidno:17的序列的位置30、40、41、47、54、56、62、97、110、188、212、319、322、326、330、333、342、344、345、348、372、438、445、462或466处在tn5-转座酶中的突变。
在本公开内容的组合物的某些实施方案中,所述转座酶是诱变处理过的转座酶。在某些实施方案中,已经诱变处理过的,编码诱变处理过的转座酶的独特核酸序列或编码诱变处理过的转座酶的序列是编码突变型转座酶的序列。在某些实施方案中,所述编码突变型转座酶的序列或所述突变型转座酶分离或衍生自任何物种。在某些实施方案中,所述突变型转座酶是突变型tn5-转座酶。本公开内容的突变型tn5-转座酶可以包括、但不限于在例如uniprot.org/uniprot/q46731处提供的突变。在某些实施方案中,所述突变型tn5-转座酶包含在根据seqidno:17的序列的位置30、40、41、47、54、56、62、97、110、188、212、319、322、326、330、333、342、344、345、348、372、438、445、462或466处的突变。在某些实施方案中,所述突变型tn5-转座酶包含根据seqidno:17的序列的r30q、k40q、y41h、t47p、e54k、e54v、m56a、r62q、d97a、e110k、d188a、k212m、y319a、r322a、r322k、e326a、k330a、k330r、k333a、k333r、r342a、r344a、e345k、n348a、l372p、s438a、k438a、s445a、g462d或a466d。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比降低转座酶活性的突变、包括、但不限于,根据seqidno:17的序列的r30q、k40q、r62q、d97a、e326a、k330a和s445a。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比增加转座酶活性的突变,包括、但不限于,根据seqidno:17的序列的r62q、d97a、e110k、d188a和l372p。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比降低dna裂解活性的突变,包括、但不限于,根据seqidno:17的序列的k333a和k333r。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比降低链转移活性的突变,包括,但不限于,根据seqidno:17的序列的y319a、r322a、r322k、k333a和k333r。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比增加转座频率的突变,包括、但不限于,根据seqidno:17的序列的y41h、t47p、e54k和e54v。在某些实施方案中,所述突变型tn5-转座酶包含消除转座酶抑制剂的表达的突变,包括、但不限于,根据seqidno:17的序列的m56a。在某些实施方案中,所述突变型tn5-转座酶包含根据seqidno:17的序列的e54k、m56a或l372p。在某些实施方案中,所述突变型tn5-转座酶包含根据seqidno:17的序列的e54k、m56a和l372p(在本文中也被称作“超活跃的tn5-转座酶)。在某些实施方案中,包括其中突变型tn5-转座酶包含根据seqidno:17的序列的e54k、m56a和l372p的那些,所述突变型tn5-转座酶包含seqidno:1的氨基酸序列。在某些实施方案中,所述突变型tn5-转座酶包含与野生型转座酶相比降低靶标特异性的突变,包括、但不限于根据seqidno:17的序列的k212m。
在本公开内容的组合物的某些实施方案中,所述选择标记是抗生素抗性基因。本公开内容的示例性抗生素抗性基因赋予抗生素抗性,所述抗生素包括、但不限于卡那霉素、大观霉素、链霉素、氨苄西林、羧苄西林、博来霉素、红霉素、多粘菌素b、四环素和新霉素。在例如ardb.cbcb.umd.edu/browsegene.shtml)可以找到本公开内容的另外的抗生素抗性基因。
本公开内容提供了一种载体,其包含本公开内容的组合物。
本公开内容提供了一种细胞,其包含本公开内容的组合物。本公开内容提供了包含本公开内容的载体的细胞,所述载体包含本公开内容的组合物。在某些实施方案中,所述细胞是细菌细胞。在某些实施方案中,所述细胞是酵母细胞。
本公开内容提供了一种筛选多个转座酶的方法,所述方法包括:(a)在适合多个细胞中的至少一个细胞被多种组合物中的至少一种组合物转化的条件下,将本公开内容的多种组合物引入多个细胞中,其中所述多个转座酶包含至少一个转座酶的野生型、突变型或诱变处理过的形式;(b)在足以诱导包含第一末端序列、uid条形码、选择标记和第二转座子末端序列的核酸的转座的条件下,表达所述多个转座酶的至少一个转座酶;(c)在(b)中的转座的核酸的插入位点处对包含插入位点重复、第一末端序列和uid条形码的核酸序列测序;(d)为所述多个转座酶的每个转座酶产生插入位点共有序列,和(e)选择第一转座酶,其具有的插入位点共有序列不同于第二转座酶的插入位点共有序列。
在本公开内容的方法的某些实施方案中,(e)的第一转座酶是诱变处理过的转座酶且(e)的第二转座酶是相同转座酶的野生型形式。在某些实施方案中,(e)的第一转座酶是诱变处理过的转座酶且(e)的第二转座酶是相同转座酶的突变形式。在某些实施方案中,(e)的第一转座酶是诱变处理过的转座酶且(e)的第二转座酶是相同转座酶的诱变处理过的形式。
在本公开内容的方法的某些实施方案中,(e)的第一转座酶是野生型转座酶且(e)的第二转座酶是野生型转座酶。
在本公开内容的方法的某些实施方案中,所述表达步骤(b)包括在足以诱导包含第一末端序列、uid条形码、选择标记和第二转座子末端序列的核酸的转座的条件下表达所述多个转座酶的每个转座酶。
在本公开内容的方法的某些实施方案中,所述表达步骤(b)包括在足以诱导包含第一末端序列、uid条形码、选择标记和第二转座子末端序列的核酸的转座的条件下短暂地表达所述多个转座酶的至少一个转座酶。在某些实施方案中,所述表达步骤(b)包括在足以诱导包含第一末端序列、uid条形码、选择标记和第二转座子末端序列的核酸的转座的条件下短暂地表达所述多个转座酶的每个转座酶。
在本公开内容的方法的某些实施方案中,所述多个细胞包含多个细菌细胞。
在本公开内容的方法的某些实施方案中,所述多个转座酶包含至少100个转座酶且其中所述多个转座酶的每个转座酶是由独特核酸序列编码。在某些实施方案中,所述多个转座酶包含至少500个转座酶且其中所述多个转座酶的每个转座酶是由独特核酸序列编码。在某些实施方案中,所述多个转座酶包含至少1000个转座酶且其中所述多个转座酶的每个转座酶是由独特核酸序列编码。在某些实施方案中,所述多个转座酶包含至少5000个转座酶且其中所述多个转座酶的每个转座酶是由独特核酸序列编码。在某些实施方案中,所述多个转座酶包含至少10,000个转座酶且其中所述多个转座酶的每个转座酶是由独特核酸序列编码。
在本公开内容的方法的某些实施方案中,载体包含所述多种组合物的每种组合物。在某些实施方案中,所述载体包含质粒、表达载体或病毒载体。在某些实施方案中,所述载体不在所述细胞内复制。在某些实施方案中,所述载体包含组成型启动子且所述组合物是在所述组成型启动子的控制下。
在本公开内容的方法的某些实施方案中,所述多个转座酶包含两个或更多个野生型转座酶。
在本公开内容的方法的某些实施方案中,所述多个转座酶包含相同转座酶的野生型、突变型和诱变处理形式中的两个或更多个。在某些实施方案中,所述多个转座酶包含相同转座酶的野生型和诱变处理形式。在某些实施方案中,所述多个转座酶包含相同转座酶的野生型、突变型和诱变处理形式。
在本公开内容的方法的某些实施方案中,所述测序是下一代测序(ngs)。
在本公开内容的方法的某些实施方案中,所述方法还包括分析(e)的选定第一转座酶的至少一个特征的步骤。
在本公开内容的方法的某些实施方案中,所述方法还包括分析(e)的选定第一转座酶的至少一个特征的步骤。在某些实施方案中,所述分析包括:(a)诱导包含第一末端序列、uid条形码和第二转座子末端序列的核酸的转座,其中所述转座由(e)的选定诱变处理过的转座酶介导且所述uid条形码与(e)的选定第一转座酶有关,(b)诱导包含第一末端序列、uid条形码和第二转座子末端序列的核酸的转座,其中所述转座由(e)的选定诱变处理过的转座酶的野生型形式介导且所述uid条形码与所述第二转座酶有关,(c)测量(e)的选定第一转座酶和所述第二转座酶中的每一种的转座酶活性或转座频率,和(d)将(e)的选定第一转座酶鉴别为具有与所述第二转座酶相比增加的转座酶活性和/或增加的转座频率,或(e)将(e)的选定第一转座酶鉴别为具有与所述第二转座酶相比降低的转座酶活性和/或降低的转座频率。在某些实施方案中,所述选定第一转座酶是超活跃的转座酶。
在本公开内容的方法的某些实施方案中,所述方法还包括分析(e)的选定第一转座酶的至少一个特征的步骤。在某些实施方案中,所述分析包括:(a)将(e)的选定第一转座酶的插入位点共有序列与(e)的第二转座酶的插入位点共有序列比对,和(b)当选定第一转座酶的插入位点共有序列含有更大数目的可变位置时,将(e)的选定第一转座酶鉴别为具有与所述第二转座酶相比降低的插入位点偏倚,或(c)当选定第一转座酶的插入位点共有序列含有更小数目的可变位置时,将(e)的选定第一转座酶鉴别为具有与所述第二转座酶相比增加的插入位点偏倚。
在本公开内容的方法的某些实施方案中,所述方法还包括分析(e)的选定第一转座酶的至少一个特征的步骤。在某些实施方案中,所述分析包括:(a)将(e)的选定第一转座酶的插入位点共有序列与(e)的第二转座酶的插入位点共有序列比对,和(b)当选定第一转座酶的插入位点共有序列在一个或多个位置处含有增加的序列变异时,将(e)的选定第一转座酶鉴别为具有与所述第二转座酶相比降低的插入位点偏倚,或(c)当选定第一转座酶的插入位点共有序列在一个或多个位置处含有减少的序列变异时,将(e)的选定第一转座酶鉴别为具有与所述第二转座酶相比增加的插入位点偏倚。
在本公开内容的方法的某些实施方案中,所述方法还包括分析(e)的选定第一转座酶的至少一个特征的步骤。在某些实施方案中,所述分析包括:(a)将(e)的选定第一转座酶的插入位点共有序列与(e)的第二转座酶的插入位点共有序列比对,和(b)当选定第一转座酶的插入位点共有序列在一个或多个位置处含有减少的序列变异时,将(e)的选定第一转座酶鉴别为具有与所述第二转座酶相比增加的插入位点偏倚,或(c)当选定第一转座酶的插入位点共有序列在一个或多个位置处含有增加的序列变异时,将(e)的选定第一转座酶鉴别为具有与所述第二转座酶相比降低的插入位点偏倚。
在本公开内容的方法的某些实施方案中,包括其中所述方法还包括分析(e)的选定第一转座酶的至少一个特征的步骤的那些,所述选定第一转座酶是诱变处理过的转座酶且所述第二转座酶是诱变处理过的转座酶的野生型形式。
在本公开内容的方法的某些实施方案中,包括其中所述方法还包括分析(e)的选定第一转座酶的至少一个特征的步骤的那些,(e)的选定第一转座酶具有与所述第二转座酶相比降低的插入位点偏倚。
在本公开内容的方法的某些实施方案中,包括其中所述方法还包括分析(e)的选定第一转座酶的至少一个特征的步骤的那些,(e)的选定第一转座酶具有在所述第二转座酶中不存在的期望特征。
在本公开内容的方法的某些实施方案中,包括其中所述方法还包括分析(e)的选定第一转座酶的至少一个特征的步骤的那些,其中所述选定第一转座酶是诱变处理过的转座酶,所述方法还包括鉴别在(e)的选定第一转座酶或其序列内的至少一个突变。在某些实施方案中,所述方法还包括鉴别在(e)的选定第一转座酶或其序列内的每个突变。在某些实施方案中,所述序列是(e)的选定第一转座酶的氨基酸序列。在某些实施方案中,所述序列是编码(e)的选定第一转座酶的核酸序列。在某些实施方案中,所述鉴别包括对编码(e)的选定第一转座酶的核酸序列测序。
附图说明
专利申请文件含有至少一幅彩色绘制的图。在请求并支付必要的费用后,含有彩图的该专利或专利申请公开文本的复制件将由官方提供。
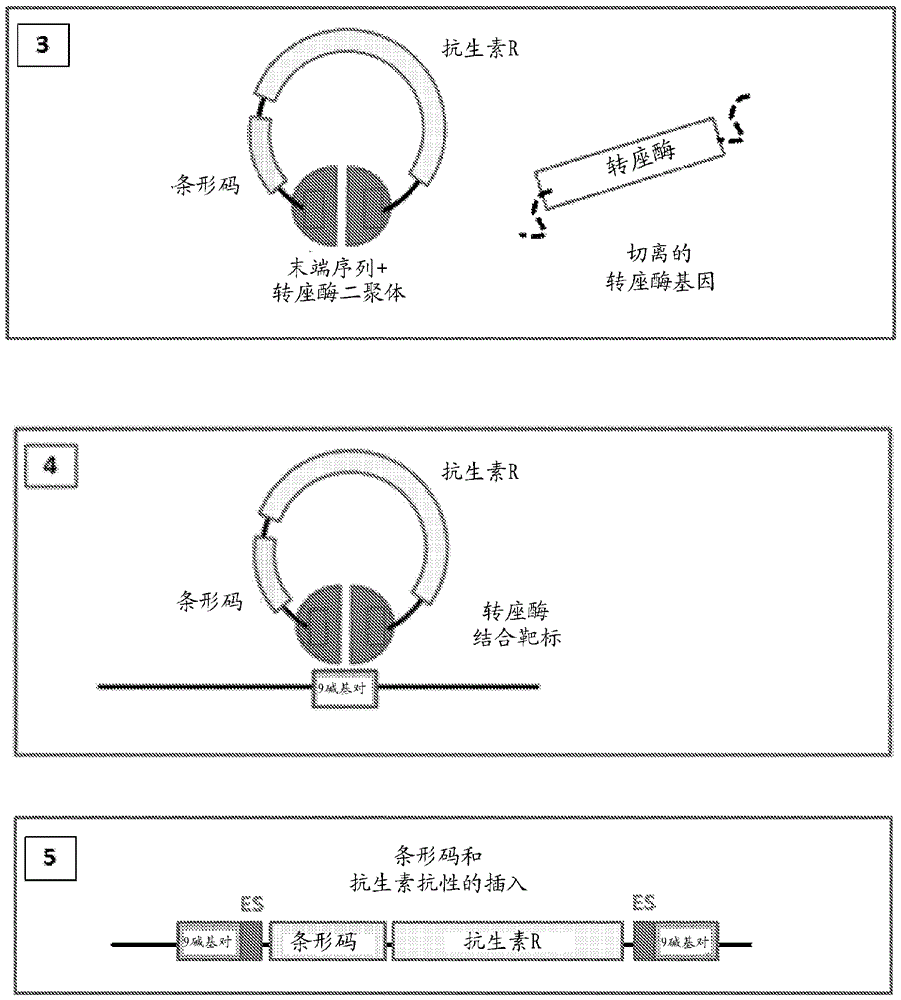
图1是一系列描绘tn5转座酶的转座的剪切和粘贴机制的示意图。(1)转座子最初位于供体dna(蓝色)中;它包含被两个反向重复末端序列(黑色)包围的转座酶基因(绿色)。转座酶的表达启动转座。(2)转座酶分子(橙色椭圆形)在末端序列处结合转座子的每个末端,且这些二聚化以形成复合物,其中转座子形成环结构。(3)将复合物从供体dna切离以形成游离的转座体,其携带整个转座子环。(4)转座体结合靶dna(红色),覆盖最终将被复制的9-碱基对。(5)在具有9-碱基对突出端的相对链上切割靶标,且末端序列与靶位点末端连接以将转座子插入在新位置;填充突出端以在插入的任一侧上建立9-碱基对重复靶区域。
图2是描绘标记化(tagmentation)的过程的示意图。转座体将2个dna“臂”插入dna靶标,剪切它并同时标记它。
图3a-b是一对图,其证实了取决于产生的方法,下一代测序(ngs)文库末端的序列偏倚。将每种碱基(a、g、c或t)的百分比相对于该碱基与文库片段的末端位置的相对位置绘图。“0”表示发生剪切或插入的位置。(a)通过机械剪切和末端修复制备的文库,(b)通过tn5标记化(tagmentation)制备的文库。
图4a-b是一对示意图,其描绘了tn5插入和tn5插入位点的共有序列。(a)二聚体的两个转座酶分子各自侵入靶标的相对链上的不同位置(位置+1和+9)。偏倚剪切和填充最终产生在转座子的任一个末端处的靶标(黄色)的重复(一旦它已经插入)。(b)结果,tn5插入位点的共有序列是回文的(来自两个重叠的倒置的转座酶偏好),具有在中心的重复区域。显示了tn5的共有插入位点。
图5的图提供了tnaa-tpn和t5-tpn插入位点共有序列的对比。tnaa-tpn[p47k]和tn5-tpn[超]的插入位点的共有序列,其中每个已经被用于产生ngs文库。基本上如在图3中所述衍生出共有序列。
图6的示意图描绘了发酵海鲜异希瓦氏菌(alishewanellajeotgali)中的4个tnaa插入位点。显示了在发酵海鲜异希瓦氏菌kctc22429中发现的4个tnaa转座子的插入位点,转座子倒置末端序列以灰色突出显示,且直接地重复的插入位点以黄色突出显示。绿色高亮显示了插入位点重复中的缺陷。插入物1具有10-碱基完美重复。插入物2具有tn5-型9-碱基完美重复。插入物3具有不完美的tn5-型9-碱基重复。插入物4具有8-碱基完美重复。
图7是可活动的和可选择的条形码区域的概述的示意图(不是按比例)。所述可活动的和可选择的条形码包含3个组分。活动单元包括2个转座子末端序列(红色),其包围一个唯一标识符条形码(uid,粉红色)和一个选择标记,诸如一个抗生素抗性基因(黄色)。该单元可以通过转座酶基因的表达而活动。转座酶基因本身不需要紧密靠近可活动单元。
图8是一系列示意图,其描绘了转座酶突变体条形码转座和筛选的概述(不是按比例)。(1)建立了数千载体的文库。每个载体不同于其它载体,因为每个携带特定单个突变型转座酶和特定单个可活动的和可选择的条形码区域。一旦制成,将文库克隆以制备载体的储备物。将文库在包括条形码和突变型转座酶的区域上测序。因为每个条形码不同于其它条形码且它连接至特定突变体,所以它可以随后用于每个突变体的鉴别。(2)用所述文库转化宿主细菌,且在细胞内短暂地表达每个突变型转座酶(启动子是组成型,但是载体不复制)。(3)如果突变型转座酶是功能性的,两个分子连接至两个末端序列,形成二聚体复合物,且将可活动的、可选择的条形码区域切离以形成游离转座体。(4)在每个细胞中仅存在单个转座体,因为仅存在单个原始载体分子来供给可活动的区域,且载体不可复制。该转座体可以结合染色体上的靶区域,且靶偏好取决于转座酶突变体的性质。(5)插入发生,且插入的效率取决于转座酶突变体的性质。插入导致条形码和选择标记掺入染色体中。细菌变成抗生素抗性的,且可以通过适当抗生素的应用进行选择。可以选择许多克隆(可能数百万)。这些代表由数千不同的突变型转座酶驱动的转座事件,每个突变体有许多不同的转座事件(在不同的位点中)。(6)然后通过ngs将每个插入位点和条形码测序。因为每个条形码可以与特定突变型转座酶相关联,且因为针对每个条形码插入对许多插入位点测序,可能确定插入位点的共有序列和将它与特定突变型转座酶关联。以此方式,可以鉴别表现出不同插入位点偏倚的转座酶。也参见图9a-c。(7)通过鉴别哪些突变型转座酶具有特定插入偏倚或活性(或其它)特征和鉴别它们共同具有的突变和突变位点,可以描述关键功能位置、特定突变和有用组合。
图9a-c是一系列示意图,其描绘了用于下一代测序(ngs)的插入位点的分离和制备的示例性方法。(a)反转pcr以测序插入位点的一个末端。(1)显示了插入靶染色体中的活动的标志物和条形码。分离了所有不同克隆的染色体dna,每个具有不同的插入。(2)使用一旦靠近条形码就切割的限制性酶消化dna。这会释放插入位点的一侧、条形码和短附近区域。(3)通过分子内连接将片段环化。(4)然后使用面向外引物进行反转pcr。(5)因为靶分子是圆形,pcr产物包含染色体的反向段,其携带插入位点的一个末端和条形码。然后可以使用这些片段来建立ngs文库,并对扩增子的一个末端测序以揭示一个插入位点末端和转座酶的uid条形码。(b)反转pcr以测序插入位点的两个末端。(1)显示了插入靶染色体中的活动的标志物和条形码。分离了所有不同克隆的染色体dna,每个具有不同的插入。(2)使用不在末端序列之间的区域内切割的限制性酶消化dna。这会释放整个插入位点、条形码和抗生素选择片段。(3)通过分子内连接将片段环化。(4)然后使用面向外引物进行反转pcr。(5)因为靶分子是圆形,pcr产物包含染色体的反向段,且扩增子的末端携带插入位点和条形码。然后可以使用这些片段来建立ngs文库,并对扩增子的两个末端测序以揭示插入位点和转座酶的uid条形码。(c)捕获以测序插入位点的一个末端。(1)显示了插入靶染色体中的活动的标志物和条形码。分离了所有不同克隆的染色体dna,每个具有不同的插入。(2)使用一旦靠近条形码就切割的限制性酶消化dna。另外,随机地剪切dna。这会释放插入位点的一侧、条形码和短附近区域。(3)将dna按大小分级分离,并连接测序衔接子。可以应用扩增。(4)将链分离,并应用杂交捕获以分离插入位点的仅一个末端。(5)将文库扩增和准备好用于测序。
图10的示意图提供了最小试验载体的概述(不是按比例)。显示了完整最小试验载体的总布局。它包含两个邻接的区域;第一个是转座酶表达单元,第二个是可活动的可选择的单元。这两个区域中的每一个被短无功能段包围,所述短无功能段可以用作pcr的引发位点。转座酶单元由转座酶基因(多种)和表达信号组成,其表达由tet启动子组成性地驱动。可活动的可选择的单元基本上如在图7中所述,但是尚未添加条形码(但是限制克隆位点存在)且选择标记是从它的天然启动子组成性地表达的卡那霉素抗性基因。
图11是用于纯化的最小试验载体扩增子的显影的琼脂糖凝胶的照片。将pcr扩增的最小试验载体纯化。在进一步使用之前,将该pcr扩增的最小试验载体在7.5%tbe琼脂糖凝胶上分离,用溴化乙锭染色,并在紫外线下显影。预期tn5产物(2734碱基对)稍微大于tnaa产物(2674碱基对);所有条带都在正确位置。随后显示tnaa双突变产物(泳道4)携带单个碱基删除且在这里不进一步考虑。
图12a-b是用于生产环化的最小试验载体的琼脂糖凝胶的一对照片。制备pcr扩增的最小试验载体用于环化以后的转化。在不同的步骤,将试验样品在7.5%tbe琼脂糖凝胶上分离,用溴化乙锭染色,并在紫外线下显影。(a)在平端建立和磷酸化以后的线性载体。(b)连接以后的载体。泳道1-3显示了用磷酸化的载体的连接。泳道4-6显示了用没有磷酸化且因此预期不会连接或环化的载体的连接。箭头指示环化的分子。
图13是用于tnaa-tpn[p47k]的插入位点的反转pcr的显影的琼脂糖凝胶的照片。来自自我连接的、环化的插入位点的反转pcr的分离产物的tbe琼脂糖凝胶。
图14a-c是使用tnaa-tpn[p47k]最小试验载体描绘9个插入位点的序列和共有序列的一系列示意图。对9个插入位点的两侧测序。重新建立原始位点(在插入之前),并鉴别重复区域(以灰色高亮显示)。(a)显示了在左手侧插入位点处对齐的位点。(b)显示了在左-和右-手侧插入位点处对齐的位点,具有中央间隔,它是补偿重复长度差异所需要的。包括原始插入位点和它们的反向互补体。(c)显示了从(b)衍生出的weblogo共有序列,不包括中央碱基(n),因为在该位置不存在有意义的充分表示。
图15a-f的一系列示意图描绘了克隆载体组分和制备最终构建体的方法。(a)原始最小试验载体。(b)将其分部分克隆到3个单独质粒上并测序。(c)将转座酶诱变处理和扩增,将条形码uid克隆进可活动的区域。(d)将突变型转座酶克隆进携带带条形码的可活动的区域的质粒。(e)通过限制酶切消化将表达片段和转座酶可活动的片段dna从2个输入质粒切出并纯化。(f)将2个片段连接并通过组装pcr进行扩增,且然后准备好环化和转化。
图16a-b的一对示意图描绘了制备定位饱和诱变构建体的方法。(a)将野生型转座酶基因克隆在适当的位置,邻近带条形码的可活动的区域。将在转座酶基因内的面向外的引物用于扩增整个质粒。因为引物之一具有随机序列的核苷酸三联体,所以将转座酶内的单个位置诱变处理成所有可能的密码子。将质粒通过连接重新环化并用于转化宿主。(b)用于诱变处理转座酶tn5的位置k212、h213和p214的引物的一个例子,根据tn5超核酸酶转座酶最小试验载体的序列(seqidno:6)编号。
图17是作为读出中位置的函数的,以“c”开始的读出的份数的图,描绘了2种转座酶构建体参照物tn5-e54k和突变型p214n的起始位点偏倚。所述图指示了对于大肠杆菌基因组中的插入位点的序列读出的前20个位置中的每一个,以“c”开始的读出的份数。垂直箭头指示对于读出中的每个位置在两幅图之间的绝对差异。
图18的图证实了对于基序a和b中给定的序列数目,基序间距离的统计上显著的距离评分的概率之间的关联。
图19的一系列图解释了各种tn5突变体和参照物tn5e54k的起始位点偏倚图。x-轴指示在读出中的位置,且y-轴指示在该位置具有“t”、“c”、“g”或“a”的读出的份数。
图20的序列标志解释了各种tn5突变体和参照物超活跃的tn5突变体(seqidno:1)的起始位点偏倚。基于每个转座酶的读出的比对,使用weblogo3产生了所述标志。
图21的图描绘了在差覆盖区域具有提高的覆盖的突变型。背景基因型b是超活跃的tn5突变体(实线),其为在bats实验中突变出突变型的实际背景基因型。
图22的图描绘了在差覆盖区域具有更低覆盖的突变型。背景基因型b是超活跃的tn5突变体(实线),其为在bats实验中突变出突变型的实际背景基因型。
图23的图描绘了通过参照或两种突变型转座酶,催化可活动元件插入基因座中的概率,取决于基因座的gc含量。
图24的图描绘了通过参照或三种突变型转座酶,催化可活动元件插入基因座中的概率,取决于基因座的gc含量。
图25:具有超活跃的tn5-转座酶的整个最小试验载体的注解序列(seqidno:6)。tn5-tpn[超]基因(虚下划线)跨位置168-1622,其具有来自位置165-167的起始密码子和来自位置1623-1625的终止密码子。e54k突变跨位置351-353。m56a突变跨位置357-359。l372p突变跨位置1305-1307。tet-35位点跨位置53-58。tet-10位点跨位置66-71。bglii限制位点跨位置1-6。xbai限制位点跨位置124-129。bamhi限制位点跨位置1647-1652。跨位置1679-1702的条形码克隆位点包括三个限制位点spei(1679-1684)、stui(1688-1693)和asuii(1697-1702)。lhses位点跨1658-1676。核糖体结合位点(rbs)位点跨位置154-158。卡那霉素抗性基因(点下划线)跨位置1854-2653,具有在位置1851-1853处的起始密码子和在位置2654-2656处的终止密码子。rbs跨位置1841-1843。mfei限制位点跨2678-2683。avrii限制位点跨位置2729-2734。另外,在图25中显示的引物序列列出在表d中。
图26:具有p47k-突变型tnaa-转座酶的整个最小试验载体的注解序列(seqidno:7)。tnaa-tpn[p47k]基因(虚下划线)跨位置168-1562,其具有来自位置165-167的起始密码子和来自位置1563-1565的终止密码子。p47k突变跨位置330-332。tet-35位点跨位置53-58。tet-10位点跨位置66-71。bglii限制位点跨位置1-6。xbai限制位点跨位置124-129。ncoi限制位点跨位置163-168。mlui限制位点跨位置258-263。bamhi限制位点跨位置1587-1592。跨位置1679-1702的条形码克隆位点包括三个限制位点spei(1619-1624)、stui(1628-1633)和asuii(1637-1642)。lhses位点跨1598-1616。核糖体结合位点(rbs)位点跨位置154-158。卡那霉素抗性基因(点下划线)跨位置1794-2603,其具有在位置1791-1793的起始密码子和在位置2604-2606的终止密码子。rbs跨位置1781-1783。mfei限制位点跨2618-2623。avrii限制位点跨位置2636-2641。rhses位点跨位置2626-2644。另外,在图26中显示的引物序列列出在表d中。
具体实施方式
本公开内容提供了用于平行地筛选多个转座酶以快速地和有效地鉴别赋予或增强期望的转座酶功能的罕见突变的组合物和高通量方法,所述转座酶作为分子工具用于用在例如下一代测序(ngs)中。本公开内容的组合物将唯一标识符(uid)条形码掺入可转座的核酸中,其在插入后将uid条形码放置成紧密靠近插入位点重复序列。通过将uid条形码与移动含有uid条形码的可转座核酸的转座酶的核酸序列关联和通过使uid条形码紧密靠近插入位点重复序列,必须得到序列的最小长度以确定移动uid条形码的多个转座酶中的一个转座酶的身份和该转座酶的插入位点偏好。本公开内容的方法意图用于筛选数百万种诱变处理的转座酶,这造成潜在测序数十亿插入位点的负担。将本公开内容的组合物和方法设计成使测序负担最小化,同时使从单个实验可以得到的信息最大化。
条形码辅助的转座酶筛选(bats)
本公开内容提供了诱变和筛选转座酶的方法,所述转座酶在转座过程中表现出与野生型转座酶或已知突变型转座酶相比降低的偏倚的靶标选择。通过本公开内容的方法鉴别出的转座酶可以用于下一代测序(ngs)应用以及分子生物学领域中的其它应用。
进行本公开内容的方法的转座酶可以包括任何转座酶。在某些实施方案中,所述转座酶源自潮汐异希瓦氏菌(alishewanellaaestuarii)。在某些实施方案中,所述转座酶是野生型tnaa-转座酶(例如具有seqidno:2的氨基酸序列的转座酶)或突变型tnaa-转座酶(例如具有seqidno:3-5中的任一个的氨基酸序列的转座酶)。在某些实施方案中,所述转座酶是野生型tn5-转座酶。在某些实施方案中,所述转座酶是与野生型tn5-转座酶相比具有增加的转座活性的突变型tn5-转座酶。在某些实施方案中,所述转座酶是包含e54k、m56a和l372p(具有根据seqidno:6的编号的突变位置)中的一个或几个的突变型tn5-转座酶。在某些实施方案中,所述转座酶是包含e54k、m56a和l372p的突变型tn5-转座酶(例如具有seqidno:1的氨基酸序列的转座酶)。在某些实施方案中,所述转座酶是与野生型tn5-转座酶相比具有减少的靶标特异性的突变型tn5-转座酶。在某些实施方案中,所述转座酶是包含k212m且与野生型tn5-转座酶相比具有减少的靶标特异性的突变型tn5-转座酶。
下文提供了示例性转座酶的序列(突变显示为粗体且带有下划线)。
tn5-转座酶突变体w125g(seqidno:18)
tn5-转座酶突变体e146a(seqidno:19).
tn5-转座酶突变体e146c(seqidno:20).
tn5-转座酶突变体e146n(seqidno:21).
tn5-转座酶突变体e146s(seqidno:22).
tn5-转座酶突变体p214s(seqidno:23).
tn5-转座酶突变体g251a(seqidno:24).
“超活跃的”tn5-转座酶(e54km56al372p)(seqidno:1).
野生型tnaa-转座酶(无突变)(seqidno:2).
双突变tnaa-转座酶(p47km50a)(seqidno:3).
单突变tnaa-转座酶(m50a)(seqidno:4).
单突变tnaa-转座酶(p47k)(seqidno:5).
携带“超活跃的”tn5-转座酶(e54km56al372p)的最小试验载体(seqidno:6).
携带单个突变型tnaa-转座酶(p47k)的最小试验载体(seqidno:7).
野生型tn5-转座酶(seqidno:17)。
一种鉴别与野生型转座酶相比具有改变的插入偏倚的突变型转座酶的现有方法可能包括以下步骤:(1)产生多个突变型转座酶;(2)将所述多个突变型转座酶的第一个突变型转座酶插入宿主生物细胞;(3)诱导由所述第一个突变型转座酶介导的至少10个转座;(4)鉴别所述第一个突变型转座酶的插入偏倚;和(5)用第二个和后续突变型转座酶重复步骤(2)至(4),直到鉴别出具有来自第一个突变型转座酶的不同插入偏倚的突变型转座酶。随后通过测序突变型转座酶中的每一个来表征第一个、第二个和后续突变型转座酶包含的突变。执行步骤(1)至(4)是没有问题的,且可以以多种方式实现;步骤1是标准的基因诱变方法,步骤2-4是标准的基于转座子的插入诱变(基因敲除)方法。诱变技术是充分确定的。诱变可以是随机的或可以针对转座酶基因中的特定位置。诱变可以包括例如点突变、缺失和/或插入的建立。典型地将转座酶掺入转座子中,且这通常放在载体(例如质粒或病毒)内。所述载体可以具有或不具有将在靶宿主(例如,大肠杆菌菌株)中工作的复制起点。然后使用携带转座子的载体转化宿主(例如,通过转染或通过使用电或化学感受态细胞)。一旦在宿主中,典型地从天然的或克隆的人工表达信号表达转座酶。转座酶蛋白然后与转座子末端序列结合并开始转座。试验转座子典型地携带选择标记(诸如抗生素抗性基因)。在载体供体dna不可复制的情况下,仅其中转座子和它的标志物已经通过转座插入复制熟练靶标(不同的复制活性质粒或染色体)的克隆将在选择条件(诸如适当抗生素的存在)下是可存活的。然后关于转座子插入偏倚研究这些活克隆。这可以如下完成:通过杂交捕获、锚定多重pcr(2014,zheng等人.naturemedicine,20,p1479-1484)或反转pcr来鉴别插入位点,随后测序。如果表征了足够的插入位点,那么可以衍生出插入位点共有序列且可以建立插入偏倚(和来自原始野生型的可能变异)。
实际限制存在于步骤(5)中。非常罕见的克隆是其中插入偏倚已经按期望改变的克隆(其中许多是未受影响的或无活性的)。为了找到这些非常罕见的克隆,必须筛选显著更大数目的克隆,例如,如在上文步骤1-4中所述。实现不仅针对插入偏倚的差异,而且针对插入偏倚的期望变化的筛选大数目的克隆的当前方法(即步骤5)在性质上是劳动密集的和低处理量的,且因此,不可能导致许多有用克隆的鉴别,即使耗费了大量时间和许多资源。
本公开内容的方法提供了长期感觉到且未得到满足的对筛选大数目的克隆以鉴别转座酶中的罕见突变的方法的需要的解决方案。具体地,本公开内容提供了一种平行地筛选大数目的突变型转座酶的方法,使得足够数目的克隆和足够数目的插入事件被筛选,以鉴别与野生型相比表现出不同转座酶活性和不同插入位点偏倚的那些突变型转座酶(和它们携带的具体突变)。此外,本公开内容的方法可以用于鉴别不仅与野生型相比表现出不同转座酶活性和/或不同插入位点偏倚、而且与野生型相比表现出期望的转座酶活性(例如活动过度)和期望的插入位点偏倚(例如减少的插入位点偏倚)的那些突变型转座酶(和它们携带的具体突变)。
通过鉴别和测序足够数目的转座子插入位点,可以衍生出野生型转座酶的插入偏倚共有序列。通过测序足够来自由突变体转座子驱动的转座的插入位点,可以鉴别该突变体的插入偏倚并与转座子的野生型形式对比。插入位点共有序列的衍生和与该转座酶的野生型形式相比任何给定突变型转座酶的插入偏倚的鉴别可以是同时的或依次的。如果这些操作同时且在相同筛选实验中进行,必须阻止样品之间的交叉污染。如果这些操作同时且在相同筛选实验中进行,那么还重要的是,在每个插入位点,对整个转座酶和插入位点测序以鉴别哪个野生型或突变型转座酶驱动在该位点处的插入。该平行筛选方法可以用数千(或甚至数百万)不同突变体转座子的混合物执行,只要测序能力会表征每个插入位点(可以是数十亿)和在每个处插入的突变型转座酶中的每一个(可以是数十亿)。不幸的是,缺乏这样的能力;在每个插入位点处测序整个转座酶基因(约1.5kb)的要求限制了该方案。
为了解决该问题,可以减小由转座酶的大小造成的测序负担。为此目的,可以产生突变型转座酶的文库,使得每个突变型转座酶基因被短的(15-25个碱基对)唯一标识符(uid)序列或条形码标记。如果在用于转座实验之前对标记的突变体文库测序,使得与每个突变体有关的uid条形码是已知的,那么仅必须对条形码测序以鉴别突变型转座酶。如果将条形码定位使得它在转座后出现在插入位点附近,那么单个短测序读出可以覆盖条形码和插入位点。因此,当前的ngs方法可以递送关于数亿插入以及驱动它们中的每一个的突变型转座酶的身份和所携带的突变的信息。
通过使用uid条形码,转座酶本身甚至不必携带至插入位点,仅需要插入条形码。转座酶可以从位于转座子末端序列支托的区域之外的基因表达。转座酶蛋白形成es-转座酶复合物并造成插入区域被切离和插在别处。如果插入区域携带uid条形码,它会转座到新位点。
需要鉴别和测序含有uid的插入位点。第一步是鉴别和分离在其中以及发生转座的克隆。大体而言,所述方法与在上文“现有”的步骤2-4中所述相同。选择标记可以位于末端序列之间,在环区域内,使得选择标记也将与uid条形码一起转座至新插入位点。包含uid条形码和选择标记的该dna-构建体(其在每个末端被末端序列支托)在本文中被称作可活动的和可选择的条形码区域。它需要表达单独的转座酶基因才能发挥功能(图7)。
转座酶突变体条形码转座和筛选的基本方法概述在图8中。首先,制备并测序突变型转座酶的文库,每种突变型转座酶连接至可活动的和可选择的uid。将宿主细菌用文库转化,并短暂地表达每个突变型转座酶,形成二聚体复合物,并将可活动的、可选择的uid切离以插入染色体。细菌因此变成抗生素抗性的且可以选择。然后通过ngs将每个插入位点和条形码测序。对于每个条形码(换而言之,对于每个突变体),将许多插入位点测序,所以可能确定插入位点的共有序列并将其与特定突变型转座酶关联。
在图8(小图6)所示的实施例中,对插入位点的仅一个末端测序。其实现方法显示在图9(a)和9(c)中;这些包括切离插入位点的一个末端,然后通过反转pcr或杂交捕获分离要求的区域。对于tn5的转座酶,这将足以产生共有序列,因为已知插入会产生9碱基对重复。但是,tnaa-tpn似乎会产生交错切口,在它们之间存在多个间隔。在该情况下,必须对插入的两个末端测序,使得可以做出正确比对。
其实现方法显示在图9(b)中。在该情况下,将整个插入位点切离和环化,然后应用反转pcr以扩增一个扩增子上的两个插入末端,然后将其测序。
转座机制和标记化(tagmentation)
由于现代分子遗传学的出现,插入序列(is)和转座子(tn,is的一种复杂形式)已经被广泛地用作研究工具,主要用于产生基因敲除。近年来,这些敲除系统已经变得复杂(例如wetmore等人(2015).mbio6:e00306-e00315),且它们的应用已经变得更多样(例如reznikoff(2006).biochem.soc.trans.34:320-323)。转座子和它们的组分诸如转座酶(tpn)和转座子dna末端序列(es)甚至已经用在快速进展的下一代测序(ngs)领域中,并且转座酶用于制备ngs文库的用途现在是非常确定的技术。该方法典型地使用通过“剪切-粘贴机制”运行的类型的转座酶(图1),诸如tn5-转座酶(tn5-tpn),如reznikoff(2008)所描述和评论的(ann.rev.genet.42:269-286;其内容通过引用整体并入本文)。
通过“剪切和粘贴”实现的转座包括结合支托转座子或is的末端序列的转座酶蛋白,然后形成二聚体复合物,插入isdna环出。然后将该复合物从供体位点切离,以形成游离转座体(tsome),其携带isdna的环。转座体然后侵入dna靶标位点,其被剪切,且插入is环。在该过程中,如果切口具有突出末端,可以在插入位点处建立重复的靶dna的短区域;在tn5的情况下,制备9-碱基对突出端,且插入位点的9-碱基对重复支托转座子。
可以如下改进该机制以制备ngs文库:首先,给纯化的转座酶加载dna“臂”,它们是转座子末端序列的基本上截短的形式。最终的活性复合物是二聚体转座体,其包含2个转座酶和2个dna末端序列,所述dna末端序列(即dna“臂”,每个具有金属离子辅因子(例如mg2+))在两个活性部位中的每一个中。
当使转座体与靶dna接触时,二者象正常的转座事件那样相互作用。但是,在该情况下,臂并非通过环连接,所以效果是剪切靶dna;该切口将被臂支托,其中的每一个必须融合至剪切位点的边缘之一(图2)。靶dna因而被片段化和在剪切的末端处被标记(因此称作“标记化(tagmented)”),随后使该dna进入ngs文库。
上述方法可以用于替代更传统的文库制备方法且比后者更简单,后者包括机械剪切dna,随后修复片段末端并给其添加标记。标记化(tagmentation)的主要缺点是,通过转座体实现的剪切偏向优先发生在某些序列(图3)。该偏倚导致当使用转座酶文库制备系统时测序数据的不均匀散布。相反,机械剪切方法几乎没有偏倚,且剪切在靶标中随机地发生。
甚至亲本转座子(以它的天然方式运行)在它所插入的靶标的序列中表现出偏倚。天然的转座子转座和人工的转座体标记化(tagmentation)表现出类似的偏倚,因为它们利用相同的转座酶蛋白和dna末端序列。所述偏好至少在某种水平依赖于在靶标处的紧密局部基本序列(可能在转座酶结合脚印内)。
因为转座体是二聚体结构,插入位点的共有序列通常在某种程度上是回文的。因为每个dna臂的剪切位点和插入位置在插入位点内偏移,回文对称的中心含有在每个剪切末端处重复的序列(图4a和b)。
突变型tn5转座酶
tn5转座酶的已知突变和功能位置的总结可以参见:uniprot.org/uniprot/q46731。
在tn5转座酶的这些已知突变和功能位置中,可能最重要的(含义是,当将tn5-转座酶用作分子工具时最常利用它们)包括、但不限于e54k、m56a和l372p。组合起来,e54k、m56a和l372p可以产生“超活跃的”-tn5-转座酶(参见,例如,us7,083,980;其内容通过引用整体并入本文)。每个突变在它们使转座酶成为分子工具的优点上是根本不同的,但是,e54k、m56a和l372p以相同的原理运行。e54k、m56a和l372p会抵消自我调节并解除转座酶活性的固有抑制。转座酶活性的抑制通常是转座子在它的天然场合中的适合性的重要要求,以便防止致命水平的转座。但是如果要将转座酶用作分子工具,转座酶活性的抑制是不利的。
e54k被用在原始“标准”以及随后的超活跃的突变体中。e54k会改善转座酶对转座子末端序列的识别。
m56a不会影响转座酶亚单位的活性,但是相反,甲硫氨酸残基的丢失会阻止在表达过程中产生来自内部翻译起始位点的转座酶的n-端-截短形式。在天然的表达系统中,转座酶的n-端-截短形式结合转座酶的全长形式以形成无活性的异二聚体。
l372p通过减少c-和n-末端的相互作用(遏制二聚化和末端序列结合的相互作用)来促进更有效的二聚化和末端序列结合。
本公开内容使用含有e54k、m56a和l372p的tn5-转座酶的“超活跃的”形式(“tn5-tpn[超]”)。
转座子aa
本公开内容提供了其它转座酶,包括、但不限于,与tn5-转座酶有关的转座酶,在本文命名为“tnaa-转座酶”或tnaa-tpn”。tnaa-tpn源自潮汐异希瓦氏菌且可以用于例如制备ngs文库。tnaa-tpn转座酶在氨基酸水平与野生型tn5-转座酶具有42%同一性。
本公开内容提供了携带单突变或双突变的突变型tnaa-转座酶。本公开内容的突变型tnaa-转座酶的单突变或双突变可以在功能上对应于tn5-转座酶的e54k和m56a超活跃突变(根据seqidno:1的tn5-转座酶的e54k和m56a超活跃突变的编号)。这些突变分别是tnaa-转座酶p47k和m50a突变(根据seqidno:2-5中的任一项,p47k和m50atnaa-转座酶突变的编号)。在功能上与tn5-转座酶的l372p突变对应的突变不可在tnaa-转座酶中产生,因为tnaa-转座酶不含有与在tn5-转座酶中发现该突变的结构域对应的结构域。
包含p47k的单突变型tnaa-转座酶(“tnaa-tpn[p47k]”)已经被用于制备和ngs文库,但是与tn5-转座酶的那些特征相比,它表现出一些独特特征。最令人注目的是,对于突变体和野生型tnaa-转座酶,插入位点偏倚不仅是独特的,而且难以确定明显的共有序列。
当为tnaa-转座酶驱动的插入衍生出共有序列时,共有序列的前几个碱基被明确定义(从-8至+2,图5),但是该序列此后缺失定义。此外,在共有序列中没有明显的回文对称。
差定义和清楚回文对称在共有序列中的缺失可能是因为tnaa-转座酶具有比tn5-转座酶更少隆起的靶标结合要求。而tn5-转座酶似乎总是攻击具有9-碱基交错裂缝的靶标以在插入后产生9-碱基重复,数据指示tnaa-转座酶可能不限于在它的裂缝中的9-碱基偏移。
在图6中,显示了在发酵海鲜异希瓦氏菌的完整测序基因组中的tnaa的4个天然插入位点(登录号ahth01000009、ahth01000020、ahth01000026和ahth01000041)。在4个天然插入位点中,仅1个插入位点产生了完美的tn5-样9-碱基同向重复。其它3个插入位点是不同长度的或不完美的。这样的变化如果大规模重复的话会产生在插入点(在该处锚定共有序列-分析)以外不精确的共有序列。
条形码辅助的转座酶筛选(bats)。
一种新颖的条形码辅助的转座酶筛选(bats)的高通量平行方法可以如在本文中所述执行,且在某些实施方案中,如在实施例8和12中所述执行。超活跃的tn5(seqidno:1)可以用作参照转座酶。可以产生几种构建体,其具有除了在超活跃的tn5中的那些突变以外的突变。可以构建包含突变型转座酶区域和带条形码的可活动的区域的扩增子,环化并用于转化大肠杆菌。活性转座酶可以催化可活动的区域向大肠杆菌基因组中的“跳跃”,从而产生卡那霉素抗性的菌落。可以将基因组dna分离,且在某些示例性实施方案中,可以制备含有uid(条形码)和基因组插入位点(跳跃位点)的基因组dna的illumina即用测序文库。在转化大肠杆菌之前,可以使用pacbio或其它长读出测序技术将文库测序以建立条形码和它们的有关转座酶序列之间的联系。还可以单独测序在转化大肠杆菌之前存在于文库中的条形码,例如使用illumina测序。
基因型向条形码的有效映射
在bats实验的分析的一个实证中的总信息流分三步工作:
1.处理来自长读出pacbio测序运行的基因型-条形码数据g,以同时从含有已知序列的构建体内提取条形码和基因型。该过程被称作基因型分段。我们在python中使用常规表达以识别已知序列,并从而分离可变的基因型和条形码。该过程是迭代优化,其中通过改变在基因型分段的常规表达中允许的错配、插入或缺失的数目来优化在g和j(来自跳跃位点分段,参见下文)之间的重叠条形码的数目。
2.处理来自短读出illumina测序运行的跳跃位点数据j,以同时从含有已知序列的构建体内提取条形码和跳跃位点(插入位点)。这也是一个迭代分段过程。在该情况下,我们使用与第三数据集b的重叠,其仅由从已知序列的构建体内分段的条形码组成。在转化大肠杆菌之前,通过测序来自所述文库的切离的条形码来产生数据集b。从r2得到的大肠杆菌dna插入物在长度上在1个碱基对至60个碱基对之间变化。对于基序分析,仅可以使用至少20个碱基对的dna插入物,而对于通过基因座方案实现的覆盖子采样(cssl),可以使用更短的dna插入物,另外,全长r1也可以用在后一种方法中(以后讨论)。
3.处理基因型数据。一个限制是,长读出测序(诸如在pacbio上)是非常易出错的。我们使用专门的抛光步骤来除去长读出测序特有的插入和缺失的丰度。为此,我们使用了转座酶序列与预期的tn5序列的逐对比对,其中使用程序clustal。随后,将所有插入删除,它们典型地是多核苷酸重复,而所有缺失作为‘n’被填入。将条形码的长度控制为20个碱基对。
4.经由条形码使基因型-条形码和跳跃数据集交叉。存在一个限制,因为条形码中的测序误差可以使得条形码在一个或多个数据集中是不可识别的。即使编辑距离或leuvenstein距离方案稍微改善了条形码的重叠,条形码的精确重叠对于数据集g和j之间的映射是足够的,而考虑到大读出数目,基于距离的方法是在计算上不可行的。在条形码-基因型计数矩阵bg中捕获条形码-基因型关联。将基因型定义为接头密码子基因型,该一般术语用于表示与背景基因型相比可能具有任意数目的突变的基因型,即使在描述的数据集中仅靶向单突变。在bg中的行代表条形码,而列代表基因型。为了定义基因型的基序,在bg的列中的每个非零输入(代表基因型)用于提取相关的条形码(行标识符)。对于每个条形码,将携带那些条形码的跳跃数据j中的所有读出收获,建立经比对的dna序列堆积(20个碱基对),并产生基序。对于覆盖子采样方案,从具有带条形码的读出标识符的分段的r2跳跃数据的dna插入物产生的经比对的bam文件中的数据反而是横向的。然后在矩阵bg中跟踪条形码以得到基因型。
5.存在一个限制,因为单个条形码可能映射至多个基因型,这是由于原始条形码多样性中的低复杂性。方便地使用矩阵bg仅提取映射至单个基因型的条形码(纯条形码)。然后可以仅在纯条形码上容易地完成相同的交叉步骤(步骤4)。
基序间距离的分析
本公开内容的条形码辅助的转座酶筛选(bats)的方法同时产生关于突变体基因型和它的优选插入基序的丰富信息。但是,几个方面限制了用于分析剪切偏倚的传统的基于基序的方法。对于这些限制中的每一种,必须开发在分析中的革新作为这些方法的部分,且在本文中描述。
每个基因型的低跳跃数目要求基序间距离的统计解释
在包含剪切dna或rna的突变酶的进化和选择的研究中,重点典型地是分析在dna剪切位点的5'-末端处的组合的序列基序。以位置权重矩阵或位置频率矩阵的形式对作为基序的此类位点的分析,使用该5'-偏倚作为基因组覆盖偏倚的替代。以与kia等人描述的方式类似的方式(kia等人.2017.bmcbiotechnology.17:6),这些矩阵又可以作为偏倚图而显示。第一个限制是,由于bats实验的巨大地平行性质,高突变体文库多样性可能限制每个突变体得到的dna插入位点(跳跃位点)的数目。结果是,插入位点作为5'偏倚基序的典型分析对于人眼区分而言变得太困难,这归因于在基因组中太少的跳跃,即太少的采样事件。一个不同的方案是使用基序熵,其并入读出的数目。但是,在这里为bats实验提出了一个不同的方案,它是使用基序之间的网络距离方案,具有适当的统计解释,如下所述。
从转座酶突变体在基因组中的跳跃的序列读出的位置频率矩阵计算在我们的研究中2个转座酶的基序之间的距离。首先,计算在参照和试验转座酶之间在位置1处具有“c”的读出的份数的绝对差异(图17)。对位置2-20重复该计算,并将读出中跨所有20个位置的绝对差异求和。对核苷酸“t”、“a”和“g”执行类似的计算。将在所有20个位置处的所有4种核苷酸的绝对差异求和,并除以20以得到每个位置的平均差异。该差异被称作距离或基序间距离。在本文中,术语距离评分和基序间距离可以互换使用。
不可直接解释基序间距离,因为它们依赖于对比的2个基序中的每一个内的序列的数目。我们开发了引导方法,其与模拟数据集的内插偶联以提供p-值的平滑查找(给定计算的距离)和2个基序中的每一个内的读出的数目(在下文描述)。引导结果显示在图18中,这证实了2个基序中的序列的数目对距离的影响。
用于查找p-值的该累积分布函数得自概率密度函数,其通过随机背景采样经验地产生。关于实验,将背景基因组随机地采样为k-聚体(在本申请中为20个碱基对),每次编译2个堆积(pileups),具有a和b序列,并将该过程重复许多次,与a和b一起保存每次的距离。随后,将距离分箱进箱d,将数据转换成具有值a、b和d和c的表格,其中d是距离箱,且c是已经观察到箱d中的距离的次数。此后,将计数转化成概率p并随后转化成累积概率。例如,可以将大于0.95的距离评分值解释为显著的。
该方案的主要限制是将模拟数据转化成稠密采样的数据集,具有方便的查找功能性,其还可以做出任何给定的距离测量。由于得到的距离的大动态范围,采样向较低序列计数a和b越来越稠密。但是,足够稠密以允许近似p-值查找的采样在计算上是棘手的。使用python脚本库中的内插包来产生真实采样值之间的更多数据点,我们替代性地在给定的a和b处内插距离-概率结构域。这给我们提供了准确地得到有关p-值所需要的要求的采样密度。接着,再次使用内插来拟合模拟数据,用a、b和d作为输入,从而允许容易地接近p-值p,这足够快以允许包含数千基序的全部对全部对比统计。
另一种解释跳跃位点偏倚的方法是通过绘制序列标志。为了确定每个转座酶的跳跃位点核苷酸序列偏倚,使用网站“weblogo3”产生了变体序列标志(http://weblogo.threeplusone.com/;crooks等人(2004)genomeresearch,14:1188-1190;schneider等人(1990),nucleicacidsresearch.18:6097-6100)。为此目的,比对了多个60碱基对序列,其含有每种变体的各个跳跃位点。序列标志显示在图20中。含有核苷酸字母的堆叠的总高度指示在该位置处的序列保守,而在堆叠内的核苷酸字母的高度指示在该位置处的各个核苷酸的相对频率。该分析突显了某些核苷酸位置的重要性和对于转座酶变体-dna相互作用重要的组成。但是,为了将重要的核苷酸位置与促成背景噪音的那些分离,要求检查足够的跳跃序列。这通过如下清楚地说明:在从变体e146c的86个序列产生的基序中观察到低总背景信号,与此相比,在从更多(超过177个)序列构建的所有其它变体中不存在该背景信号(参见表f)。
通过基因座进行的覆盖子采样,作为来自bats实验的数据的基序间距离分析的替代
另一个限制是,5'-基序可能不足以捕获覆盖偏倚的本质,正如在以经纯化的dna的标记化(tagmentation)形式的文库制备的情景下。文库制备方案的一个重要目标是得到靶向的基因组或转录组的均匀且完整的覆盖,且5'-偏倚可以视作与基因组覆盖仅部分相关联,或甚至仅一个外观特征。并且,基序作为单个位置权重矩阵或作为单个位置频率矩阵的表示基本上捕获平均的结合强度相关的特性,且没有充分利用在潜在可变距离处的邻近碱基的连接可能性。使用更复杂的模型诸如markov模型或神经网络基本上要求更多的读出,与模型的次序强烈成比例,使它们不太可用于检测有限数据中的差异。理想的是,能够以在基因组上的覆盖的方式直接解释结果。事实上,在基因组上的覆盖在非常多路的实验如bats(其中可以潜在地平行地筛选数千突变体)中是不足够的,这有效地排除了从低序列读出数目对覆盖的直接靶向,从而导致基序分析的替代应用。但是,本公开内容显示,通过使用来自bats实验的跳跃的基因组基因座,比较基因组覆盖实际上是可接近的。
通过基因座的覆盖子采样(cssl)如下起作用:首先将转座酶跳跃位点的测序基因组dna插入物映射至适当的参照基因组,并将基因组基因座与具有足够覆盖以充当参照覆盖分布的参照数据集r的预期覆盖关联。使用相同的基因组坐标从分布r有效地采样感兴趣的样品数据集s。基因组基因座有效地建立合理连接用于将来自参照基因型r的数据映射至感兴趣的样品基因型s。参照基因型r可以例如是在正常文库制备实验的标记化(tagmentation)中应用的tn5转座酶的野生型形式,或使用以在市场上可得到的酶的标记化的文库制备,而样品基因型s可能表示起源于bats或有关实验中的定位诱变或随机诱变的突变体基因型。对于样品基因型中的每一个,经由基因组基因座从参照分布r采样样品分布s,并随后使用统计检验对比2个分布r和s,诸如1)mann-whitney检验(关于平均值差异),2)kolmogorov-smirnoff检验(关于不同的分布形状),3)其它参数或非参数检验,4)形状差异的目检,5)基于百分位数的度量诸如在亲本分布的平均覆盖的小于25%采样的基因座的百分比,或检测形状的差异的任意其它方法。以此方式,可以选择突变型,其可以比参照r或背景b转座酶可以接近更好地接近那些基因座。
所述方法中的另一个灵活性是,样品s可以彼此对比。例如,突变型样品s1可以与野生型或背景基因型b(最初从其突变出s1,也使用参照r通过cssl得到其分布)对比。
作为一个例子,对来自与实施例12相同的bats实验的数据执行cssl分析,并将结果指示在图21和22中。在该实验中,参照基因型b是超活跃的tn5转座酶,产生参照数据集b。样品数据集s源自tn5突变体,其各自具有除了超活跃的tn5中的那些以外的突变。对s与b的对比进行统计检验,并将关于mann-whitney和kolmogorov-smirnoff检验的得到的p-值指示在表f中。在图21中的数据显示了两种突变体w125g和g251a以及参照物超活跃的tn5的数据。用超活跃的tn5产生了插入基因组内的基因座中的与两种转座酶突变体有关的可活动元件,其倾向于显示与参照数据集b的更低覆盖。在与参照转座酶r或背景转座酶b具有低覆盖的区域中提高的覆盖将是有益的,例如在变体呼叫中,如下所述。通过计算和绘制3个数据点的平均值,将图21和22中的数据平滑化,仅为了可视化目的。
以类似的方式,突变e146a、e146c、e146n和e146s的数据的cssl分析的结果描绘在图22中。从图22显而易见,在tn5的位置e146处的特定突变导致在被参照转座酶较好覆盖的基因组的基因座处向基因组中的优先插入。这样,在位置e146处的这些突变具有偏倚的增加。这进一步解释了该方法在确定突变体之间的偏倚差异中的实用性和这如何涉及转座酶突变体向基因组的低或高覆盖区域中插入的偏好。
由于变体调用者的证据的缺乏,低覆盖区域典型地是假变体呼叫诸如snp和插入缺失的原因。相反,读出与具有相等映射质量的多个位置的不正确比对(由于基因组中的重复)也会导致假变体呼叫,这是由于比对产生的误差的引入,从而与这样的区域中过度覆盖的区域关联。在这样的情形下,覆盖子采样将自身提供给在高覆盖区域中缺乏覆盖的突变体的选择。子采样也可能例如限于最有关的区域,诸如目标区域,包括生物编码区域,用于变体呼叫的靶基因座的列表,或任意其它基因座特异性的标准。
因此,即使对于在用高数目的基因型的巨大地平行的bats实验中得到的较低读出数目,可以在不需要基序分析的情况下接近基因组覆盖。在cssl的另一种形式中,可以将分布r、s和b转化成选定的描述特征,诸如gc含量、较高维的k-聚体频率或已知的dna修饰模式(作为基因座的函数)。在基因组水平以它们的gc-偏倚的方式对比文库制备技术是常见的实践。cssl提供了一种有效方法来在bats实验过程中选择gc-无偏倚酶。
为了确定tn5转座酶突变体是否表现出改变的gc-偏倚,分析了来自以上bats实验的数据。将参照转座酶(超活跃的tn5)和突变体的跳跃位点(插入位点)映射至参照基因组,并计算100碱基对窗口的gc含量。图23和24是作为靶dna的gc含量的函数的,给定转座酶的插入概率的图。图23显示了两种突变体和参照物超活跃的tn5的跳跃位点数据。对于较低gc含量,两种突变体p214s(seqidno:23)和g251a(seqidno:24)具有比参照转座酶显著增加的偏好(关于p-值,参见表g)。tn5突变体g251a对低gc-基因座具有增加的插入偏好,这可能是相同突变体在作为低覆盖基因座的基因座处具有增加的插入概率的机制,如在图21中所示。
图24显示了来自三种tn5突变体的跳跃位点数据。与参照转座酶的插入概率相比,这三种突变体e146n、e146a和e145s具有在高-gc基因座中更高的插入概率。执行了分别关于平均值差异和曲线形状的mann-whitney和kolmogorov试验,并将p-值指示在表g中。
定义
如在本公开内容中使用的,单数形式“一个”、“一种”和“所述”包括复数指示物,除非上下文另外清楚地指明。因而,例如,对“一种方法”的提及包括多个这样的方法,且对“一种转座酶”的提及包括对一种或多种转座酶以及本领域技术人员已知的其等同物的提及,诸如此类。
本公开内容提供了分离的或基本上纯化的多核苷酸或蛋白组合物。“分离的”或“纯化的”多核苷酸或蛋白或其生物活性部分实质上或基本上不含有在它的天然存在的环境中发现的通常伴随所述多核苷酸或蛋白或与所述多核苷酸或蛋白相互作用的组分。因而,分离的或纯化的多核苷酸或蛋白当通过重组技术来生产时基本上不含有其它细胞物质或培养基,或当化学合成时基本上不含有化学前体或其它化学物质。最佳地,“分离的”多核苷酸不含有在所述多核苷酸的来源生物的基因组dna中天然地侧接所述多核苷酸的序列(最佳地,蛋白编码序列)(即,位于所述多核苷酸的5'和3'末端处的序列)。例如,在不同的实施方案中,所述分离的多核苷酸可以含有小于约5kb、4kb、3kb、2kb、1kb、0.5kb或0.1kb的核苷酸序列,该序列在所述多核苷酸的来源细胞的基因组dna中天然地侧接所述多核苷酸。基本上不含有细胞物质的蛋白包括具有小于约30%、20%、10%、5%或1%(按干重计)污染蛋白的蛋白制品。当重组地生产本公开内容的蛋白或其生物活性蛋白时,最佳地培养基代表小于约30%、20%、10%、5%或1%(按干重计)的化学前体或非目标蛋白化学物质。
本公开内容提供了公开的dna序列的片段、变体、突变体(突变)和由这些dna序列编码的蛋白。如在本公开内容中使用的,术语“片段”表示dna序列的部分或氨基酸序列的部分和因而由其编码的蛋白。包含编码序列的dna序列的片段可以编码保留天然蛋白的生物活性和因此保留对本文所述的靶dna序列的dna识别或结合活性的蛋白片段。可替换地,可用作杂交探针的dna序列的片段通常不会编码保留生物活性或不保留启动子活性的蛋白。因而,dna序列的片段可以在从至少约20个核苷酸、约50个核苷酸、约100个核苷酸且直到本公开内容的全长多核苷酸的范围内。
通过模块方案可以构建本公开内容的核酸或蛋白,所述模块方案包括在靶载体中预组装单体单元和/或重复单元,随后可以将所述靶载体组装进最终的目标载体。本公开内容的多肽可以包含本公开内容的重复单体,且可以通过模块方案来构建,所述模块方案包括在靶载体中预组装重复单元,随后可以将所述靶载体组装进最终的目标载体。本公开内容提供了通过该方法生产的多肽以及编码这些多肽的核酸序列。本公开内容提供了宿主生物和细胞,其包含通过该模块方案生产的编码多肽的核酸序列。
“结合”表示大分子之间(例如,蛋白和核酸之间,或两个蛋白之间)的特异性非共价相互作用。这样的特异性结合通常是基于特定结构基序之间的特异性相互作用,所述特定结构基序通常、但并非总是反映在天然生物场合中存在的那些。
“序列特异性结合”表示大分子之间(例如,在蛋白和核酸之间)的序列特异性的非共价相互作用。不需要结合相互作用的所有组分都是序列特异性的(例如,与dna主链中的磷酸酯残基接触),只要所述相互作用作为整体是序列特异性的即可。术语“序列特异性结合”不限于强烈的、狭窄的序列偏好,但是也包括由这样的分子表现出的弱偏好:所述分子可以结合多种多核苷酸靶标,但是对某些的偏好胜过其它。这样的结合也可能被称作“半随机序列结合”或“偏倚序列结合”。
术语“优先结合”表示转座酶或转座体(有活性的或无活性的)与靶dna(例如基因组dna)内的序列的分层结合次序。本公开内容的转座酶或转座体(有活性的或无活性的)将优先结合某个位点,所以这些优选的序列比替代序列更容易被占据。随着这些优选的序列被占据,转座酶或转座体(有活性的或无活性的)具有更多的自由来结合替代性的且不太优选的序列。在饱和浓度,转座酶或转座体(有活性的或无活性的)将结合所以可得到的序列;但是,优选的位点将倾向于首先被占据。因而,在本公开内容的转座酶或转座体(有活性的或无活性的)的低浓度,首先被占据的序列被“优先结合”。
术语“包括”意指组合物和方法包括所列举的要素,但是并不排除其它要素。当用于定义组合物和方法时,“基本上由……组成”应当意在排除当用于预期目的时对于所述组合具有任何基本重要性的其它要素。因而,基本上由如本文中定义的要素组成的组合物不会排除痕量污染物或惰性载体。“由……组成”应当是指排除超过痕量的其它成分和实质方法步骤。由这些过渡术语中的每一个定义的实施方案是在本公开内容的范围内。
本文中使用的“表达”表示将多核苷酸转录成mrna的过程和/或随后将转录的mrna翻译成肽、多肽或蛋白的过程。如果多核苷酸源自基因组dna,表达可以包括在真核细胞中的mrna的剪接。
“基因表达”表示在基因中所含的信息向基因产物的转化。基因产物可以是基因的直接转录产物(例如,mrna、trna、rrna、反义rna、核酶、shrna、微rna、结构rna或任意其它类型的rna)或通过mrna的翻译产生的蛋白。基因产物也包括通过诸如加帽、多腺苷酸化、甲基化和编辑等过程修饰的rna,和通过例如甲基化、乙酰化、磷酸化、泛素化、adp-核糖基化、豆蔻基化和糖基化修饰的蛋白。
公开了非共价地连接的组分以及制备和使用非共价地连接的组分的方法。各种组分可以呈如本文中所述的多种不同的形式。例如,非共价地连接的(即,可操作地连接的)蛋白可以用于允许暂时相互作用,其避免了本领域中的一个或多个问题。非共价地连接的组分(诸如蛋白)结合和解离的能力仅或主要在期望的活性需要这种结合的情况下实现功能结合。所述连接可以持续足够长以允许期望的作用。
“结合位点”或“结合序列”是限定转座酶、dna衔接子和/或转座体将结合(只要存在对于结合而言足够的条件)的核酸的部分的靶核酸序列,。
“共有序列”是限定转座酶、dna衔接子和/或转座体将结合(只要存在对于结合而言足够的条件)的核酸的部分的靶核酸序列,所述部分存在于结合序列或结合位点的超过一种变体中。尽管本公开内容的转座酶、dna衔接子和/或转座体可能优先结合第一序列,包含该序列的所有位点将被占据,本公开内容的转座酶、dna衔接子和/或转座体可能结合第二序列,所述第一和第二序列包含共有序列。例如,在比对第一和第二序列后,尽管一个或多个碱基可能变化,但是剩余的不变碱基可能包含共有序列。
术语“靶标”和“输入”dna在本公开内容中可以互换使用。
术语“核酸”或“寡核苷酸”或“多核苷酸”表示共价地连接在一起的至少两个核苷酸。单链的描述也定义了互补链的序列。因而,核酸也可以包括描述的单链的互补链。本公开内容的核酸也包括保留相同结构或编码相同蛋白的基本上相同的核酸及其互补体。
本公开内容的核酸可以是单链的或双链的。本公开内容的核酸可以含有双链序列,甚至当大多数分子是单链时。本公开内容的核酸可以含有单链序列,甚至当大多数分子是双链时。本公开内容的核酸可以包括基因组dna、cdna、rna或其杂合物。本公开内容的核酸可以含有脱氧核糖核苷酸和核糖核苷酸的组合。本公开内容的核酸可以含有碱基的组合,所述碱基包括尿嘧啶、腺嘌呤、胸腺嘧啶、胞嘧啶、鸟嘌呤、肌苷、黄嘌呤、次黄嘌呤、异胞嘧啶和异鸟嘌呤。可以合成本公开内容的核酸以包含非天然的氨基酸修饰。通过化学合成方法或通过重组方法,可以得到本公开内容的核酸。
本公开内容的核酸(它们的整个序列或其任何部分)可以是非天然存在的。本公开内容的核酸可以含有一个或多个天然地不存在的突变、取代、删除或插入,使得整个核酸序列是非天然存在的。本公开内容的核酸可以含有一个或多个复制的、倒置的或重复的序列,得到的其序列天然地不存在,使得整个核酸序列是非天然存在的。本公开内容的核酸可以含有天然地不存在的经修饰的、人工的或合成的核苷酸,使得整个核酸序列是非天然存在的。
鉴于遗传密码中的冗余,多个核苷酸序列可以编码任何特定蛋白。本文涵盖所有这样的核苷酸序列。
如在本公开内容中使用的,术语“基本上互补的”表示在8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、180、270、360、450、540个或更多个核苷酸或氨基酸的区域上与第二序列的互补体具有至少60%、65%、70%、75%、80%、85%、90%、95%、97%、98%或99%同一性的第一序列,或两个序列在严谨杂交条件下杂交。
如在本公开内容中使用的,术语“基本上相同的”表示在8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、180、270、360、450、540个或更多个核苷酸或氨基酸的区域上具有至少60%、65%、70%、75%、80%、85%、90%、95%、97%、98%或99%同一性的第一和第二序列,或关于核酸,如果第一序列与第二序列的互补体是基本上互补的。
如在本公开内容中使用的,术语“完美互补性”表示彼此杂交的第一和第二序列,沿着核酸双链体的长度没有碱基的间隙或错配。例如,第一和第二序列可以根据沃森-克里克碱基配对规则以完美互补性彼此杂交。
如在本公开内容中使用的,术语“不完美互补性”表示彼此杂交的第一和第二序列,沿着核酸双链体的长度没有一个或多个间隙、或一个或多个碱基的一个或多个错配。例如,第一和第二序列可以彼此杂交,沿着核酸双链体的长度有70%、75%、80%、85%、90%、95%、99%或之间的任何百分比的碱基彼此杂交。
如在本公开内容中使用的,术语“变体”当用于描述核酸时,表示(i)提及的核苷酸序列的部分或片段;(ii)提及的核苷酸序列或其部分的互补体;(iii)与提及的核酸或其互补体基本上相同的核酸;或(iv)在严谨条件下与所述核酸、其互补体或与其基本上相同的序列杂交的核酸。
如在本公开内容中使用的,术语“变体”当用于描述肽或多肽时,表示在氨基酸序列中相差氨基酸的插入、缺失或保守取代、但是保留至少一种生物活性的肽或多肽。变体还可以是指这样的蛋白:其氨基酸序列与提及的具有保留至少一种生物活性的氨基酸序列的蛋白基本上相同。
氨基酸的保守取代,即,用具有类似性能(例如,亲水性、带电荷区域的程度和分布)的不同氨基酸替换一个氨基酸,在本领域中被公认为通常涉及微小变化。如本领域所理解的,可以部分地通过考虑氨基酸的亲水指数而鉴定这些微小变化.kyte等人,j.mol.biol.157:105-132(1982)。氨基酸的亲水指数是基于对其疏水性和电荷的考虑。可以取代具有类似亲水指数的氨基酸且仍然保留蛋白功能。在一个方面,具有±2的亲水指数的氨基酸被取代。氨基酸的亲水性还可以用于揭示会产生保留生物学功能的蛋白的取代。在肽的上下文中,对氨基酸的亲水性的考虑允许对该肽的最大局部平均亲水性的计算,这是据报道与抗原性和免疫原性良好关联的有用的量度。美国专利号4,554,101,通过引用完整地并入本文。
具有类似亲水性值的氨基酸的取代可以产生保留生物活性(例如免疫原性)的肽。用具有在彼此的±2内的亲水性值的氨基酸可以执行取代。氨基酸的疏水性指数和亲水性值受该氨基酸的特定侧链影响。与该观察一致,将与生物学功能相容的氨基酸取代理解为取决于氨基酸(尤其是那些氨基酸的侧链)的相对相似性,如通过疏水性、亲水性、电荷、大小和其它特性所揭示的。
本文中使用的“保守的”氨基酸取代可以如下表a、b或c中所示来定义。在某些实施方案中,融合多肽和/或编码这样的融合多肽的核酸包括通过编码本公开内容的多肽的多核苷酸的修饰已经引入的保守取代。根据物理性能以及对二级和三级蛋白结构的贡献,可以将氨基酸分类。保守取代是将一个氨基酸取代为另一个具有类似性能的氨基酸。在表a中展示了示例性的保守取代。
表a--保守取代i
可替代地,可以如在lehninger(biochemistry,第2版;worthpublishers,inc.ny,n.y.(1975),第71-77页)中所述将保守氨基酸分组,如在表b中所述。
表b--保守取代ii
可替代地,示例性的保守取代如在表c中所述。
表c--保守取代iii
应当理解,本公开内容的多肽意图包括带有一个或多个氨基酸残基的插入、缺失或取代或它们的任意组合以及除了氨基酸残基的插入、缺失或取代以外的修饰的多肽。本公开内容的多肽或核酸可以含有一个或多个保守取代。
如在本公开内容中使用的,术语“超过一个”前述氨基酸取代表示2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19或20个或更多个列举的氨基酸取代。术语“超过一个”可以表示2、3、4或5个列举的氨基酸取代。
本公开内容的多肽和蛋白(它们的整个序列或其任何部分)可以是非天然存在的。本公开内容的多肽和蛋白可以含有一个或多个天然地不存在的突变、取代、删除或插入,使得整个氨基酸序列是非天然存在的。本公开内容的多肽和蛋白可以含有一个或多个复制的、倒置的或重复的序列,得到的其序列天然地不存在,使得整个氨基酸序列是非天然存在的。本公开内容的多肽和蛋白可以含有天然地不存在的经修饰的、人工的或合成的氨基酸,使得整个氨基酸序列是非天然存在的。
如在本公开内容中使用的,使用默认参数(tatusova和madden,femsmicrobiollett.,1999,174,247-250;其通过引用整体并入本文),可以使用独立的可执行的用于blast2个序列的blast引擎程序(bl2seq)确定“序列同一性”,该程序可以得自国家生物技术信息中心(nationalcenterforbiotechnologyinformation,ncbi)ftp站点。术语“相同的”或“同一性”当在两个或更多个核酸或多肽序列的背景下使用时,表示在每个序列的指定区域中指定百分比的相同残基。所述百分比可以如下计算:将2个序列进行最佳比对,在指定区域上对比所述2个序列,确定在2个序列中出现同一残基的位置的数目以产生匹配位置的数目,将匹配位置的数目除以指定区域中的位置的总数,并将结果乘以100以产生序列同一性的百分比。在2个序列具有不同长度或者所述比对产生一个或多个交错末端且指定的对比区域仅包括单个序列的情况下,单个序列的残基被包括在分母中,但不是计算的分子。当对比dna和rna时,可以认为胸腺嘧啶(t)和尿嘧啶(u)相同。可以手工地或通过使用计算机序列算法诸如blast或blast2.0确定同一性。
如在本公开内容中使用的,术语“内源性”表示与引入它的靶基因或宿主细胞天然地相关的核酸或蛋白序列。
除非另外指出,否则按重量计算所有百分比和比率。
除非另外指出,否则基于总组合物计算所有百分比和比率。
在本公开内容中给出的每个最大数字限制包括每个更低的数字限制,如同这样的更低数字限制明确地写在本文中。在本公开内容中给出的每个最小数字限制将包括每个更高的数字限制,如同这样的更高数字限制明确地写在本文中。在本公开内容中给出的每个数字范围将包括落在这样的更宽数字范围内的每个更狭窄的数字范围,如同这样的更狭窄的数字范围都明确地写在本文中。
本文中公开的值不应当理解为严格限于列举的确切数值。相反,除非另外指出,否则每个这样的值意图指列举的值和围绕该值的在功能上等同的范围。例如,公开为“20μm”的值意图指“约20μm”。
在本文中引用的每个文件(包括任何交叉引用的或有关的专利或应用)特此通过引用整体并入本文,除非明确地排除或以其它方式限制。任何文件的引用并非承认:它是关于本文中公开或要求保护的任何发明的现有技术,或它单独地或与任何其它参考文献以任何组合教导、提示或公开任何这样的发明。进一步,如果在本文件中的术语的任何含义或定义与通过引用并入的文件中的相同术语的任何含义或定义冲突,以在本文件中指定给该术语的含义或定义为准。
尽管已经解释和描述了本公开内容的特定实施方案,但是在不背离本公开内容的精神和范围的情况下可以做出多种其它变化和修改。所附权利要求的范围包括在本公开内容的范围内的所有这样的变化和修改。
实施例
为了可以更有效地理解本文中公开的发明,下文提供了实施例。应当理解,这些实施例仅仅用于例证目的,不应解释为以任何方式限制本发明。在这些实施例中,除非另外指出,否则根据以下描述的方法进行标准的重组dna或其它分子生物学技术:(1)green和sambrook,molecularcloning:alaboratorymanual,第4版,coldspringharborpress(2012).(2)市售试剂盒和试剂的供应商.(3)基于网络的方案汇编,诸如protocol-on-line(protocol-online.org/)。在这些实施例中,除非另外指出,否则根据市售试剂盒和试剂的供应商所推荐的方法进行蛋白表达、纯化、测定和显影以及其它标准蛋白生产技术。
实施例1:转座-试验-扩增子的设计和构建
要在最终的平行条形码转座和筛选实验中使用的最终构建体所要求的基本组分显示在图8的部分1和2中。但是,为了试验和开发基本方法,最初的dna构建体不需要携带突变型转座酶或条形码。相反,初步实验用野生型或有限数目的超活跃的转座酶各自在单独基础上进行,所以uid是不必要的。重组dna构建体的这些简单初始形式被称作“最小试验载体”。为了进一步简化,所述构建体不需要克隆和纯化;使用仅通过pcr产生和维持的dna试验材料进行初步试验。
因此,将最初的不同最小试验载体作为线性扩增子产生和维持。构建了5种不同的最小试验载体,每种具有不同的转座酶。所述构建体之一携带tn5-转座酶的超活跃形式(突变e54km56al372p,seqidno:1)。其它4种构建体携带tnaa-转座酶的不同形式。第一种是野生型转座酶(seqidno:2);第二种携带2个突变(p47k和m50a,seqidno:3)。剩余两种转座酶各自仅携带单突变(m50a,序列id4或p47k,seqidno:5)。5种构建体的总体结构显示在图10中。
具有超活跃的tn5-转座酶的整个最小试验载体的注解序列(seqidno:6)显示在图25中。tn5-tpn[超]基因跨位置168-1622,其具有来自位置165-167的起始密码子和来自位置1623-1625的终止密码子。e54k突变跨位置351-353。m56a突变跨位置357-359。l372p突变跨位置1305-1307。tet-35位点跨位置53-58。tet-10位点跨位置66-71。bglii限制位点跨位置1-6。xbai限制位点跨位置124-129。ncoi限制位点跨位置163-168。bamhi限制位点跨位置1647-1652。跨位置1679-1702的条形码克隆位点包括三个限制位点spei(1679-1684)、stui(1688-1693)和asuii(1697-1702)。lhses位点跨1658-1676。核糖体结合位点(rbs)位点跨位置154-158。卡那霉素抗性基因跨位置1854-2653,具有在位置1851-1853处的起始密码子和在位置2654-2656处的终止密码子。rbs跨位置1841-1843。mfei限制位点跨2678-2683。avrii限制位点跨位置2729-2734。rhses位点跨位置2686-2704。另外,在图25中显示的引物序列列出在表d中。
具有p47k-突变型tnaa-转座酶的整个最小试验载体的注解序列(seqidno:7)显示在图26中。tnaa-tpn[p47k]基因跨位置168-1562,其具有来自位置165-167的起始密码子和来自位置1563-1565的终止密码子。p47k突变跨位置330-332。tet-35位点跨位置53-58。tet-10位点跨位置66-71。bglii限制位点跨位置1-6。xbai限制位点跨位置124-129。ncoi限制位点跨位置163-168。mlui限制位点跨位置258-263。bamhi限制位点跨位置1587-1592。跨位置1679-1702的条形码克隆位点包括三个限制位点spei(1619-1624)、stui(1628-1633)和asuii(1637-1642)。lhses位点跨1598-1616。核糖体结合位点(rbs)位点跨位置154-158。卡那霉素抗性基因跨位置1794-2603,其具有在位置1791-1793的起始密码子和在位置2604-2606的终止密码子。rbs跨位置1781-1783。mfei限制位点跨2618-2623。avrii限制位点跨位置2636-2641。rhses位点跨位置2626-2644。另外,在图26中显示的引物序列列出在表d中。
通过标准技术产生最小试验载体的组成部分,并通过pcr组装全长载体。使用引物对tpcr-2-f和tpcr-1-r通过pcr产生载体的储备物(参见下表d以及上文seqidno:6和7)。通过sanger测序来检查序列。
表d
在最初的构建努力中,携带具有2个突变(p47k和m50a)的tnaa-转座酶的最小试验载体也携带单碱基删除突变。以后制备正确载体,但是来自早期试验的结果(下文)因此不包括该构建体。
实施例2:使用线性扩增子的初步转化和转座试验
通过pcr制备了5种不同的最小试验载体扩增子。使用引物对tpcr-2-f和tpcr-1-r(参见上文的表d以及seqidno:6和7)和下述循环方案,用kapabiosystemshifi聚合酶进行扩增:4℃,保持/95℃,2min/(98℃,30秒/58℃,30秒/72℃,2min)x20/4℃,保持。
将pcr产物使用qiagenpcr纯化试剂盒纯化,通过光谱测定法定量,并在-20℃保存。
为了制备用于电穿孔进大肠杆菌dh10b中的最小试验载体扩增子,使用zymodna清洁试剂盒(#5)进一步纯化样品。在每种情况下,加载1μg样品并最终将其洗脱在30μl超纯水中。通过在琼脂糖凝胶上分离5μl(大约170ng),证实样品完整性,如在图11中所示。
在1mm-通道电穿孔比色皿中,将20μl电感受态的大肠杆菌dh10b细胞加给dna(33ng最小试验载体或1ng对照pet29质粒),并在20kv、200欧姆、25μf脉冲处理。时间常数在5.5-5.7变化。加入500μlsoc以后,将细胞在37℃温育50min。然后将未稀释的样品(100μl)在含有30μg/ml的卡那霉素的luria琼脂上铺板。将培养皿在37℃温育过夜,此后关于菌落数目对平板评分。
仅在pet29质粒对照的情况下观察到任何菌落。
实施例3:用环化扩增子的最初转化和转座试验
如上所述,最小试验载体没有将可活动的、可选择的区域有效地递送至大肠杆菌的染色体,可能是由于扩增子在进入细胞后受到损伤的事实。这样的损伤可能通过外切核酸酶攻击较多地发生在扩增子的末端。可替换地,或另外,线性dna不可能像环状dna一样有效地转化大肠杆菌。为了解决这些可能性和更接近地模仿包含质粒dna的传统转化,在转化之前通过连接试验了将最小试验扩增子环化的影响。
再次通过pcr制备了携带tn5-转座酶[超]和tnaa-转座酶[m50a]的最小试验载体。使用前述引物对tpcr-2-f和tpcr-1-r(表d以及seqidno:6和7)执行扩增,但是这次使用kapabiosystems2grobustreadymix和下述循环方案:4℃,保持/95℃,2min/(98℃,30秒/58℃,30秒/72℃,2min)x15/4℃,保持。
另外,产生了最小试验载体的更短形式,即缺少转座酶组分且仅包含可活动的卡那霉素区域(1117碱基对片段,在上文seqidno:6中的核苷酸1617-2734)的形式。这也使用2grobustreadymix和如上全长模板通过pcr扩增完成,但是用tpcr-3-f替代tpcr-2-f引物(表d,上文seqidno:6)。
在所有情况下,将扩增子在tbe琼脂糖凝胶上分离并使用qiagen凝胶纯化试剂盒纯化,且然后通过光谱测定法定量。2grobust可以留下3'-a-突出端,所以使用kapabiosystemshifi聚合酶(0.5uhifi,0.3mmdntp,25μl中,在72℃保持5分钟)将突出末端转化成平头末端。使用qiagenpcr纯化试剂盒纯化平端pcr产物。在tbe琼脂糖凝胶上检查完整性(显示在图12a中),并通过密度测定法定量样品。样品浓度是在40-100ng/μl之间。
为了能够通过连接来环化扩增子,需要将5’-末端磷酸化(pcr引物没有磷酸化)。这使用10μl(400-1000ng)的扩增子dna在20μl反应中用kapabiosystems多核苷酸激酶完成。此后,使用kapabiosystems连接酶在标准的(非快速的)连接酶缓冲液中在20μl反应中自我连接10μl(200-500ng)的磷酸化的扩增子。在16℃连接过夜。同样连接等量(5μl,200-500ng)的相同扩增子,但是没有磷酸化处理。将5μl连接样品(50-125ngdna)在琼脂糖凝胶上分离(显示在图12b中),这证实一些扩增子已经环化且一些已经连接以形成多聚体复合物。在大肠杆菌dh10b的转化准备中,将剩余的连接混合物使用zymodna清洁试剂盒(#5)进一步纯化并洗脱在12μl超纯水中。
转化基本上如前面所述(实施例3),例外是,使用3μl纯化的连接混合物(由于在纯化过程中可能的损失,终浓度未知),并将转化的细胞在37℃温育3小时,然后将150μl等分试样铺板,以便使在其中可发生可活动的区域的转座的时间段最大化。
在携带tnaa-转座酶[m50a]的最小试验载体和截短的可活动的仅卡那霉素区域的载体的情况下,仅看到几个菌落(平均5个/板)。这些可能代表不正常的重组驱动的插入事件。相反,使用携带tn5-转座酶[超]的最小试验载体的转化产生了多到难以计数的菌落,从而指示转座已经发生。应当指出,菌落的高数目可能部分地由于可在铺板之前延长的3小时温育中已经发生的细胞分裂。
携带tnaa-转座酶[m50a]的最小试验载体没有产生可辨别数目的转座事件并不意外,因为已知m50a-型突变在活性二聚体的表达水平在细菌中起作用,且表达在该情况下可能不是限制性的。相反,可能需要增强活性的突变p47k。
为了试验如果使用p47k突变体形式,tnaa-转座酶驱动的转座是否增加至可检测的水平,用携带转座酶的该形式的最小试验载体重复实验。在该实验中,对比了三种最小试验载体;它们携带来自tn5的超活跃的转座酶,来自tnaa的野生型转座酶,和来自tnaa的p47k突变型转座酶。
实验基本上如前,具有以下具体细节:制备最小试验载体模板dna的新工作储备物。扩增是使用前述的kapabiosystemshifi,将扩增的dna在0.75%tbe琼脂糖凝胶上分离,并使用qiagen凝胶纯化试剂盒分离适当的扩增子。将dna通过光谱法定量,并在10mmtris-cl(ph8.0)中在-20℃保存。
如前在25μl反应中使用工作储备物作为模板(100-200ng)用kapabiosystemshifi聚合酶扩增实验样品。将样品使用qiagenpcr纯化试剂盒纯化和如前定量。在该情况下,不需要钝末端化,所以将每个dna样品以50ng/μl的终浓度立即磷酸化。然后将10μl(500ng)它在20μl终体积中自我连接,且然后纯化,如前所述。
转化如上所述,例外是,在铺板之前将转化的细胞在37℃温育1小时。减小的表达时间是为了最小化在铺板之前由细胞分裂引起的菌落数目增加的可能性。将100μl未稀释的和50μl1:10稀释的样品铺板。
在携带野生型tnaa-转座酶的最小试验载体的情况下,仅观察到几个菌落(对于未稀释的样品,平均47个/板)。相反,用携带超活跃的tn5-转座酶和p47k突变型tnaa转座酶的最小试验载体的转化分别从1:10稀释样品产生了112和63个菌落/板的平均计数。由此我们计算出,在这两种情况下,在总转化混合物中捕获1.2x104和0.6x104个转座事件。
实施例4:改善样品制备、转化和转座的方法
理想地,本文所述的插入位点筛选实验要求捕获大数目的转座事件。此外,这些中的每一个应当由仅与特定uid-标记的可活动区域联合起作用的单突变型转座酶驱动。应当尽可能避免无关的转座酶和带uid条形码的区域之间的串扰。如果两种不同的构建体同时转化同一个细菌宿主,这样的交叉反应性可能发生。这可以视作小可能性,除非两种不同的构建体已经在转化之前变得连接,例如通过在pcr或连接步骤中形成异质多聚体。
改善细菌的转化或随后转座的数目和效率的方法中的任何变化将是有用的改善。同样,减小交叉反应的机会的任何改善将是有益的。
对所述过程做出的改善如下所述:
为了减小包括pcr-衍生的有裂缝的构建体的机会,在线性载体的初步扩增以后立即执行凝胶纯化步骤,这使得能够实现用于后续步骤的正确大小的选择性dna取回。
为了改善在环化时的连接效率,已经修饰两个扩增引物的步骤以包括相同的限制位点(例如,产生突出末端的限制位点)。在扩增和凝胶纯化以后,将pcr产物用限制性酶消化,并然后纯化。此后,环化是更有效的,因为突出的相容末端促进连接。这样的引物对的一个例子是引物tpcr-2reeco-f和tpcr-1reeco-r(tpcr-2-f和tpcr-1-r的修饰形式,显示在表1中)。
为了消除连接以后的剩余线性分子,已经完成了一个步骤,其中用外切核酸酶iii和外切核酸酶vii消化样品。
为了减小包括连接衍生的多聚体构建体的机会,在连接以后立即完成凝胶纯化步骤。结果,仅环形单体分子被分离,且将纯化的和/或富集的环形单体分子用于后续转化。
这些额外步骤中的一个或多个可以以任意组合用于优化本公开内容的方法。
实施例5:插入位点分离的初步试验和初步序列.
为了进行插入位点分离的初步试验,合并了有限数目的其中已经发生可活动区域的转座的克隆,并将插入位点扩增,且在几个情况下,克隆和测序。试验了2个库;在一种情况下,该库含有5个单个克隆,在第二种情况下存在80个克隆。在两种情况下,转座由tnaa-tpn[p47k]驱动,且如在实施例3和4中所述产生克隆。
为了产生小库,将5个菌落用牙签挑入单个luria液体培养基(卡那霉素30ug/ml)中,培养过夜,在离心机中沉淀,并然后用于基因组dna分离。对于较大库,从携带80个菌落的培养皿的琼脂表面直接收获菌落。将这些刮入已经加入培养皿的1mlluria液体培养基中,并制备细胞的均匀混合物。通过离心来沉淀细胞,并分离dna。在两种情况下,使用sigmagenelute细菌基因组试剂盒从大肠杆菌细胞分离基因组dna。
然后通过能够测序插入位点的两个末端的反转pcr方法分离插入位点,基本上如在图9(b)中概述的。
在细节上:在相互相容的newenglandbiolabscutsmart缓冲液中,用5种限制性酶bamhi、ecori、ncoi、ndei、xhoi的混合物消化1μg基因组dna。消化是在80μl具有20u每种酶的终体积中,并在37℃温育过夜。这些限制性酶都没有在可活动的区域序列内剪切,且因此仅在包围插入位点的基因组dna内消化。将消化的dna使用zymodna清洁试剂盒(#5)纯化,且然后将突出末端使用kapabiosystemshifi聚合酶填充和钝化(0.5uhifi,0.3mmdntp,25μl中,在72℃保持5分钟)。将dna如前用zymo试剂盒纯化,并然后通过光谱测定法定量。5-和80-菌落库的浓度分别是10.3ng/μl和15.3ng/μl。
此后,在标准连接酶缓冲液中使用kapabiosystems连接酶在10μl反应中自我连接不同体积(0.1-5μl)的dna。在16℃连接过夜。然后使用kapabiosystemslongrangepcr试剂盒进行反转pcr,将所有10μl连接混合物直接放入最终的125μl反应体积(0.625ulongrange酶,1.75mmmgcl2,0.3mm每种dntp,0.3μm每种引物)。使用的引物是kan-af和ipcr-r(表d和seqidno6和7)。扩增循环如下:4℃,保持/94℃,3min/(94℃,15秒/57℃,20秒/72℃,3min)x30/4℃,保持。
反转pcr以后,将10μl产物在琼脂糖凝胶上显影(图13)。可以看出,得到了多个产物条带,并且用较高的开始菌落数目得到较多的类型。在初步连接中的小量dna在凝胶上产生了干净条带和极少的“污迹”。具有较低循环数目的较高最初dna水平可产生类似的结果。
将反转pcr的产物使用zymodna清洁试剂盒(#5)纯化,并然后使用promegapgem-teasy载体和克隆试剂盒进行克隆。为此使用的反转pcr产物来自这样的扩增:对于5-和80-菌落库,分别利用在连接混合物中的0.5μl和0.1μl经消化的靶dna(图13,泳道3和8)。
从培养皿挑选各个菌落,并使用kapabiosystems2g聚合酶和用于原始反转pcr的相同引物(kan-af和ipcr-r,表d,seqidno:6和7)对这些进行菌落pcr。将扩增产物在琼脂糖凝胶上分离和显影,从而允许我们区分几种不同大小的产物。将质粒dna从这些克隆分离,并使用引物ipcr-f和ipcr-r(表d,seqidno:6和7)对dna进行sanger测序。由此,确立了9个(分别来自5-和80-菌落库的3和6个)完整插入位点的性质。插入位点显示在图14中。
在9个中,仅4个插入事件产生了9-碱基重复。3个为10-碱基重复,并且也存在8-和11-碱基重复各一个。为了产生共有插入位点,如下解释差异:通过锚定在右-和左-手侧剪切位点处的比对,并通过在中央插入空间来补偿。将数据库衍生的序列和它们的反向互补体用于产生共有序列,这是为了抵消由链选择引起的任何明显偏倚。当分析大数目的插入位点时,这不应当是必要的。结果表明插入位点是高度偏倚的,特别是在相对于重复区域边界的位置-2、-3和-4(回文对称的左侧)。在该实验中,对于ccc而言在那些位置处的偏倚是100%,与此相比,标记化(tagmentation)反应在每个位置为大致40%c,如在图5中所示。更显著的偏倚可能是由于以下事实:所述转座酶在它的天然环境(在其中它已经进化至严格限制活性)中起作用,且在包含缓冲液、温度和辅因子(设计成促进杂乱转座)的人工环境中不起作用。此外,低浓度(单个转座体/细胞),和可能地转座体在破坏之前可能存在的短阶段,也可能促进极端偏倚。
实施例6:经验证的开始材料的构建.
上述的早期概念验证(proof-of-concept)实验利用了通过pcr组装和维持的最小试验载体。这样,在工作储备物和试验样品内可能存在在pcr过程中已经获得错误(突变)的子群体。对于筛选实验,优选的是,应当尽可能仅包括有意地靶向转座酶的突变。为了实现该目的,必须克隆开始材料和证实它不含有非故意突变。
关于用于筛选实验的克隆和载体生产的实验方案概述在图15中;在该情况下,仍然在pcr中组装载体,但是,从已经克隆和测序的开始材料组装。开始材料本身包含2个单独部分;将转座酶表达信号(包括tet-启动子和核糖体结合位点)与其余载体分开维持。这是为了确保所述载体是稳定的且不存在转座酶驱动的重排或不稳定性。两个不同的载体组分具有小程度的序列重叠;这允许它们通过pcr进行组装和扩增。pcr包括适当量的开始模板、高保真度聚合酶和几个扩增循环,以便减小出现不希望的突变的可能性。
为了制备经验证的开始材料,使用不同的最初试验载体的工作储备物(图15a)来分离和克隆制备图15b中所示的构建体所需的区域。1个质粒携带bglii-ndei启动子片段(来自seqidno:6和7中的位置1-194)。2个质粒携带ncoi-xhoi转座酶片段;克隆了tn5-tpn[超]和tnaa-tpn[p47k](分别来自seqidno:6中的位置163-1622和seqidno:7中的位置163-1562)。1个质粒携带xhoi-avrii可活动片段(来自seqidno:6的位置1617-2734)。
将克隆的区域测序并显示是正确的。
实施例7:输入载体的构建和实验载体的生产.
最终可能需要两种输入载体(图15d向前);如上所述(实验6)制备第一种,其携带启动子区域。
携带突变的转座酶(包括tn5-tpn[超]和tnaa-tpn[p47k])和整个可活动的可选择的区域(包括uid条形码)的第二种输入载体的制备概述在图15(b-d)中。
在第一步中,将包含20碱基对随机序列的uid条形码插入携带可活动区域的以前验证过序列的质粒中(在图15中,在spei和asuii位点之间)。因此,制备了107-108个克隆之间的“带条形码的、可活动的、可选择的文库”;每个克隆携带用不同条形码标记的可活动区域(图15c)。
在下一步中,对转座酶(以前验证过序列,上文)进行诱变和克隆(图15d)。诱变是通过易错pcr、定位诱变、其组合和/或其它方法。诱变可以包括产生点突变和/或缺失和/或插入的和/或不同转座酶之间的重组。随机诱变方法特别适合用于鉴别转座酶内的重要位置和发现转座酶内的新突变。定位诱变方法适合用于详细研究特定位置。筛选特定位置的方法的一个例子显示在图16中。
诱变以后,通过测序dna片段的子集可以确定突变率和/或类型。然后将诱变处理过的转座酶克隆(在图15中,作为ncoi-xhoi片段)进带条形码的、可活动的、可选择的文库的适当位点。克隆受到严格控制以产生已知数目的不同克隆的有限大小的库。产生了103-108的库。将这些克隆库培养并然后作为原代储备物储存。
然后制备细胞的工作储备物,并在进一步培养以后,从培养物分离质粒dna。
在代表将在以后用于筛选的突变体库的质粒的情况下,制备测序文库,使得可以表征uid条形码和有关的突变型转座酶。一种实现这一点的方法是分离有关的dna片段(在图15中,在ndei-asuii限制性片断上),并然后连接smrtbell测序衔接子,并然后使用pacbio系统测序。其它测序方法同样起作用。
在最后的步骤中,制备转化载体。首先,为了制备完整线性载体,必须分离2个组成部分,如在图15e中所示。简而言之,将质粒dna用限制性酶剪切以释放表达区域片段和转座酶可活动的区域片段(在图15中,分别作为bglii-ndei和nco-avrii片段),然后将它们通过凝胶纯化进行分离。为后者选择的特定库大小将决定在筛选中最终包括的突变体的数目。
此后,将两个区域通过组装pcr连接和扩增,如在图15f中所示。为此,使用两种片段(作为混合的模板)和仅针对外末端的引物(例如引物对tpcr-2reeco-f和tpcr-1reeco-r,表d)进行pcr。
在已经制备线性载体以后,制备用于将突变型转座酶和可活动的区域递送进宿主中的环形载体,如在实施例3和4中所述。
实施例8:制备和筛选突变型转座酶.
为了证实所述方法整体适合预期的用途,对在试验转座子内的有限数目的以前鉴别出的重要位置各自进行定位饱和诱变,如在图16中所述。然后以在最终的文库中包括代表性和已知数目的克隆的方式,将突变的转座酶样品合并。除此以外,操作基本上如在实验7、3、4和5中所述,但是在有限的规模,转变成有限数目的突变体和样品,并在最终的步骤中对合并的插入位点使用足够的ngs能力,以鉴别和计数位点和有关的条形码。为此,在分离已经在其中发生转座的克隆以后,使用以前描述的方法之一将染色体dna纯化并将插入位点分离和扩增。然后通过添加illumina衔接子制备用于测序的插入位点片段,并然后测序。
测序以后,将读出通过uid条形码分选,并将插入位点与大肠杆菌参照序列比对。由此,可以计数每个条形码的所有不同插入位点,并然后彼此比对,锚定在插入位点。如果对插入的两个末端测序,可以确定重复长度,且可以应用任一种适当间距,或可以根据长度将插入位点分箱(binned)并分别分析。比对后,确定四种碱基中的每一种在每个位置(相对于插入位点)处的份数,并确定偏倚,可以衍生出共有序列,并然后将突变体基因型与偏倚特性关联。在可行时,鉴别相同的突变(但是具有不同的条形码),并检查这些以确定是否发现了类似的偏倚特性。类似地分析了在相同位置的不同突变。
除了检查偏倚以外,检查了转座酶的活性与插入位点数目(其反映了转座可能性)的关联。容易地鉴别出无效和低活性突变体,因为这些由存在于原始文库中的条形码呈现,但是不呈现在插入位点集合中。在没有发生活性变化的情况下,插入的数目类似于用非突变的亲本转座酶得到的那些。但是,在插入位点集合中过度呈现的那些条形码代表表现出更高活性的转座酶突变体,至少在该实验的条件下。再次,通过将条形码与突变体文库交叉参考,鉴别造成它的突变。
实施例9:新颖突变体的鉴别.
新颖突变体和重要位置的鉴别要求:要筛选的突变体文库通过随机或半随机诱变产生,并且筛选大数目的突变体。除此以外,操作基本上如在实施例7、3、4、5和8中所述,但是在大得多的规模,并在最终的步骤中使用深ngs以鉴别插入位点和有关的条形码。
典型的筛选实验因此包括利用1x105或更多突变型转座酶的文库,且针对1x107或更多转座事件。平均而言,这将在每个检查的突变体中产生超过100个转座,因为原始突变体库的一部分将是无活性的。
测序以后,将读出通过uid条形码分选,并将插入位点与大肠杆菌参照序列比对,并如在实施例8中所述分析结果。由于涉及的大数目,针对信息含量预筛选插入位点,以便鉴别可能的偏倚型变体,然后进行更详细分析。
实施例10:重组和饱和诱变.
从实施例9中所述的分析,得到有用新颖突变的位置和类型。然后进行另一个实验,其中对这些新鉴别的位置进行定位饱和诱变和筛选,基本上如在实施例8中所述。以此方式,检查了在每个目标位置处的每种可能突变。个别地试验这些突变体,并且也在特异性地或随机地重组时试验。
实施例11:文库产生.
将选择的突变型转座酶克隆、表达和纯化。然后将这些突变型转座酶用于通过标记化(tagmentation)制备ngs文库。然后评估这样的文库的插入偏倚。
实施例12:条形码辅助的转座酶筛选(bats).
基本上如在实施例8中所述,执行使用上述条形码辅助的转座酶筛选(bats)的新颖巨大平行方法的实验。将超活跃的tn5(seqidno:1)用作参照转座酶。制备几种构建体,其具有除了在超活跃的tn5中的那些以外的突变。构建包含突变型转座酶区域和带条形码的可活动的区域的扩增子,环化并用于转化大肠杆菌。活性转座酶催化可活动的区域向大肠杆菌基因组中的“跳跃”,从而产生卡那霉素抗性的菌落。将基因组dna分离,并制备含有uid(条形码)和基因组插入位点(跳跃位点)的基因组dna的illumina即用测序文库。将在转化大肠杆菌之前的文库使用pacbio测序以建立条形码和它们的有关转座酶序列之间的联系。还单独测序在转化大肠杆菌之前在文库中的条形码,例如使用illumina测序。
实施例13:基因型向条形码的有效映射.
在bats实验的分析的一个实证中的总信息流分三步工作:
1.处理来自长读出pacbio测序运行的基因型-条形码数据g,以同时从含有已知序列的构建体内提取条形码和基因型。该过程被称作基因型分段。我们在python中使用常规表达以识别已知序列,并从而分离可变的基因型和条形码。该过程是迭代优化,其中通过改变在基因型分段的常规表达中允许的错配、插入或缺失的数目来优化在g和j(来自跳跃位点分段,参见下文)之间的重叠条形码的数目。
2.处理来自短读出illumina测序运行的跳跃位点数据j,以同时从含有已知序列的构建体内提取条形码和跳跃位点(插入位点)。这也是一个迭代分段过程。在该情况下,我们使用与第三数据集b的重叠,其仅由从已知序列的构建体内分段的条形码组成。在转化大肠杆菌之前,通过测序来自所述文库的切离的条形码来产生数据集b。从r2得到的大肠杆菌dna插入物在长度上在1个碱基对至60个碱基对之间变化。对于基序分析,仅可以使用至少20个碱基对的dna插入物,而对于通过基因座方案实现的覆盖子采样(cssl),可以使用更短的dna插入物,另外,全长r1也可以用在后一种方法中(以后讨论)。
3.处理基因型数据。一个限制是,长读出测序(诸如在pacbio上)是非常易出错的。我们使用专门的抛光步骤来除去长读出测序特有的插入和缺失的丰度。为此,我们使用了转座酶序列与预期的tn5序列的逐对比对,其中使用程序clustal。随后,将所有插入删除,它们典型地是多核苷酸重复,而所有缺失作为‘n’被填入。将条形码的长度控制为20个碱基对。
4.经由条形码使基因型-条形码和跳跃数据集交叉。存在一个限制,因为条形码中的测序误差可以使得条形码在一个或多个数据集中是不可识别的。即使编辑距离或leuvenstein距离方案稍微改善了条形码的重叠,条形码的精确重叠对于数据集g和j之间的映射是足够的,而考虑到大读出数目,基于距离的方法是在计算上不可行的。在条形码-基因型计数矩阵bg中捕获条形码-基因型关联。将基因型定义为接头密码子基因型,该一般术语用于表示与背景基因型相比可能具有任意数目的突变的基因型,即使在描述的数据集中仅靶向单突变。在bg中的行代表条形码,而列代表基因型。为了定义基因型的基序,在bg的列中的每个非零输入(代表基因型)用于提取相关的条形码(行标识符)。对于每个条形码,将携带那些条形码的跳跃数据j中的所有读出收获,建立经比对的dna序列堆积(20个碱基对),并产生基序。对于覆盖子采样方案,从具有带条形码的读出标识符的分段的r2跳跃数据的dna插入物产生的经比对的bam文件中的数据反而是横向的。然后在矩阵bg中跟踪条形码以得到基因型。
5.存在一个限制,因为单个条形码可能映射至多个基因型,这是由于原始条形码多样性中的低复杂性。方便地使用矩阵bg仅提取映射至单个基因型的条形码(纯条形码)。然后可以仅在纯条形码上容易地完成相同的交叉步骤(步骤4)。
实施例14:基序间距离的分析
条形码辅助的转座酶筛选(bats)的新颖巨大平行方法同时产生关于突变体基因型和它的优选插入基序的丰富信息。但是,几个方面限制了用于分析剪切偏倚的传统的基于基序的方法。对于这些限制中的每一种,必须开发在下文描述的在分析中的革新。
每个基因型低跳跃数目要求基序间距离的统计解释
在包含剪切dna或rna的突变酶的进化和选择的研究中,重点典型地是分析在dna剪切位点的5'-末端处的组合的序列基序。以位置权重矩阵或位置频率矩阵的形式对作为基序的此类位点的分析,使用该5'-偏倚作为基因组覆盖偏倚的替代。以与kia等人描述的方式类似的方式(kia等人.2017.bmcbiotechnology.17:6),这些矩阵又可以作为偏倚图而显示。第一个限制是,由于bats实验的巨大地平行性质,高突变体文库多样性可能限制每个突变体得到的dna插入位点(跳跃位点)的数目。结果是,插入位点作为5'偏倚基序的典型分析对于人眼区分而言变得太困难,这归因于在基因组中太少的跳跃,即太少的采样事件。一个不同的方案是使用基序熵,其并入读出的数目。但是,在这里为bats实验提出了一个不同的方案,它是使用基序之间的网络距离方案,具有适当的统计解释,如下所述。
从转座酶突变体在基因组中的跳跃的序列读出的位置频率矩阵计算在我们的研究中2个转座酶的基序之间的距离。首先,计算在参照和试验转座酶之间在位置1处具有“c”的读出的份数的绝对差异(图17)。对位置2-20重复该计算,并将读出中跨所有20个位置的绝对差异求和。对核苷酸“t”、“a”和“g”执行类似的计算。将在所有20个位置处的所有4种核苷酸的绝对差异求和,并除以20以得到每个位置的平均差异。该差异被称作距离或基序间距离。
不可直接解释基序间距离,因为它们依赖于对比的2个基序中的每一个内的序列的数目。我们开发了引导方法,其与模拟数据集的内插偶联以提供p-值的平滑查找(给定计算的距离)和2个基序中的每一个内的读出的数目(在下文描述)。引导结果(在本文中定义为基序间距离概率图)显示在图18中,这证实了2个基序中的序列的数目对距离的影响。
用于查找p-值的该累积分布函数得自概率密度函数,其通过随机背景采样经验地产生。关于实验,将背景基因组随机地采样为k-聚体(在本申请中为20个碱基对),每次编译2个堆积(pileups),具有a和b序列,并将该过程重复许多次,与a和b一起保存每次的距离。随后,将距离分箱进箱d,将数据转换成具有值a、b和d和c的表格,其中d是距离箱,且c是已经观察到箱d中的距离的次数。此后,将计数转化成概率p并随后转化成累积概率。例如,可以将大于0.95的距离评分值解释为显著的。
该方案的主要限制是将模拟数据转化成稠密采样的数据集,具有方便的查找功能性,其还可以做出任何给定的距离测量。由于得到的距离的大动态范围,采样向较低序列计数a和b越来越稠密。但是,足够稠密以允许近似p-值查找的采样在计算上是棘手的。使用python脚本库中的内插包来产生真实采样值之间的更多数据点,我们替代性地在给定的a和b处内插距离-概率结构域。这给我们提供了准确地得到有关p-值所需要的要求的采样密度。接着,再次使用内插来拟合模拟数据,用a、b和d作为输入,从而允许容易地接近p-值p,这足够快以允许包含数千基序的全部对全部对比统计。
以位置频率矩阵的方式计算在实施例12中描述的bats实验的起始位点偏倚,并如在图19中所图示产生偏倚图。计算基序之间的距离,并将结果显示在表e中。
这些数据显示,在位置e146、w125和g251处的突变产生了显著不同于参照转座酶超活跃的tn5的插入位点基序。
表e:对于选择的突变体,到插入位点参照的距离。考虑用于计算基序的序列的数目,具有与大于0.95的p-值对应的距离的一对基序可以视作可显著辨别的。
*用于参照的序列的数目:3490个读出。
另一种解释跳跃位点偏倚的方法是通过绘制序列标志。为了确定每个转座酶的跳跃位点核苷酸序列偏倚,使用网站“weblogo3”产生了变体序列标志(http://weblogo.threeplusone.com/;crooks等人(2004)genomeresearch,14:1188-1190;schneider等人(1990),nucleicacidsresearch.18:6097-6100)。为此目的,比对了多个60碱基对序列,其含有每种变体的各个跳跃位点。序列标志显示在图20中。含有核苷酸字母的堆叠的总高度指示在该位置处的序列保守,而在堆叠内的核苷酸字母的高度指示在该位置处的各个核苷酸的相对频率。该分析突显了某些核苷酸位置的重要性和对于转座酶变体-dna相互作用重要的组成。但是,为了将重要的核苷酸位置与促成背景噪音的那些分离,要求检查足够的跳跃序列。这通过如下清楚地说明:在从变体e146c的86个序列产生的基序中观察到低总背景信号,与此相比,在从更多(超过177个)序列构建的所有其它变体中不存在该背景信号(参见表f)。
实施例15:通过基因座的覆盖子采样,作为来自bats实验的数据的基序间距离分析的替代
另一个限制是,5'-基序可能不足以捕获覆盖偏倚的本质,正如在以经纯化的dna的标记化(tagmentation)形式的文库制备的情景下。文库制备方案的一个重要目标是得到靶向的基因组或转录组的均匀且完整的覆盖,且5'-偏倚可以视作与基因组覆盖仅部分相关联,或甚至仅一个外观特征。并且,基序作为单个位置权重矩阵或作为单个位置频率矩阵的表示基本上捕获平均的结合强度相关的特性,且没有充分利用在潜在可变距离处的邻近碱基的连接可能性。使用更复杂的模型诸如markov模型或神经网络基本上要求更多的读出,与模型的次序强烈成比例,使它们不太可用于检测有限数据中的差异。理想的是,能够以在基因组上的覆盖的方式直接解释结果。事实上,在基因组上的覆盖在非常多路的实验如bats(其中可以潜在地平行地筛选数千突变体)中是不足够的,这有效地排除了从低序列读出数目对覆盖的直接靶向,从而导致基序分析的替代应用。但是,在该方法公开中,我们显示,通过使用来自bats实验的跳跃的基因组基因座,比较基因组覆盖实际上是可接近的。
通过基因座的覆盖子采样(cssl)如下起作用:首先将转座酶跳跃位点的测序基因组dna插入物映射至适当的参照基因组,并将基因组基因座与具有足够覆盖以充当参照覆盖分布的参照数据集r的预期覆盖关联。我们使用相同的基因组坐标从分布r有效地采样感兴趣的样品数据集s。基因组基因座有效地建立合理连接用于将来自参照基因型r的数据映射至感兴趣的样品基因型s。参照基因型r可以例如是在正常文库制备实验的标记化(tagmentation)中应用的tn5转座酶的野生型形式,或使用以在市场上可得到的酶的标记化的文库制备,而样品基因型s可能表示起源于bats或有关实验中的定位诱变或随机诱变的突变体基因型。对于样品基因型中的每一个,经由基因组基因座从参照分布r采样样品分布s,并随后使用统计检验对比2个分布r和s,诸如1)mann-whitney检验(关于平均值差异),2)kolmogorov-smirnoff检验(关于不同的分布形状),3)其它参数或非参数检验,4)形状差异的目检,5)基于百分位数的度量诸如在亲本分布的平均覆盖的小于25%采样的基因座的百分比,或检测形状的差异的任意其它方法。以此方式,可以选择突变型,其可以比参照r或背景b转座酶可以接近更好地接近那些基因座。
所述方法中的另一个灵活性是,样品s可以彼此对比。例如,突变型样品s1可以与野生型或背景基因型b(最初从其突变出s1,也使用参照r通过cssl得到其分布)对比。
作为一个例子,对来自与实施例12相同的bats实验的数据执行cssl分析,并将结果指示在图21和22中。在该实验中,参照基因型b是超活跃的tn5转座酶,产生参照数据集b。样品数据集s源自tn5突变体,其各自具有除了超活跃的tn5中的那些以外的突变。对s与b的对比进行统计检验,并将关于mann-whitney和kolmogorov-smirnoff检验的得到的p-值指示在表f中。在图21中的数据显示了两种突变体w125g和g251a以及参照物超活跃的tn5的数据。用超活跃的tn5产生了插入基因组内的基因座中的与两种转座酶突变体有关的可活动元件,其倾向于显示与参照数据集b的更低覆盖。在与参照转座酶r或背景转座酶b具有低覆盖的区域中提高的覆盖将是有益的,例如在变体呼叫中,如下所述。通过计算和绘制3个数据点的平均值,将图21和22中的数据平滑化,仅为了可视化目的。
以类似的方式,突变e146a、e146c、e146n和e146s的数据的cssl分析的结果描绘在图22中。从图22显而易见,在tn5的位置e146处的特定突变导致在被参照转座酶较好覆盖的基因组的基因座处向基因组中的优先插入。这样,在位置e146处的这些突变具有偏倚的增加。这进一步解释了该方法在确定突变体之间的偏倚差异中的实用性和这如何涉及转座酶突变体向基因组的低或高覆盖区域中插入的偏好。
表f:选择的突变体的参照数据分布的统计分析。对于kolmogorov-smirnoff和mann-whitney分析,<0.15和<0.05的p-值分别被视作显著的。
*用于参照的序列的数目:2005个读出。
由于变体调用者的证据的缺乏,低覆盖区域典型地是假变体呼叫诸如snp和插入缺失的原因。相反,读出与具有相等映射质量的多个位置的不正确比对(由于基因组中的重复)也会导致假变体呼叫,这是由于比对产生的误差的引入,从而与这样的区域中过度覆盖的区域关联。在这样的情形下,覆盖子采样将自身提供给在高覆盖区域中缺乏覆盖的突变体的选择。子采样也可能例如限于最有关的区域,诸如目标区域,包括生物编码区域,用于变体呼叫的靶基因座的列表,或任意其它基因座特异性的标准。
因此,即使对于在用高数目的基因型的巨大地平行的bats实验中得到的较低读出数目,可以在不需要基序分析的情况下接近基因组覆盖。在cssl的另一种形式中,可以将分布r、s和b转化成选定的描述特征,诸如gc含量、较高维的k-聚体频率或已知的dna修饰模式(作为基因座的函数)。在基因组水平以它们的gc-偏倚的方式对比文库制备技术是常见的实践。cssl提供了一种有效方法来在bats实验过程中选择gc-无偏倚酶。
为了确定tn5转座酶突变体是否表现出改变的gc-偏倚,分析了来自以上bats实验的数据。将参照转座酶(超活跃的tn5)和突变体的跳跃位点(插入位点)映射至参照基因组,并计算100碱基对窗口的gc含量。图23和24是作为靶dna的gc含量的函数的,给定转座酶的插入概率的图。图23显示了两种突变体和参照物超活跃的tn5的跳跃位点数据。对于较低gc含量,两种突变体p214s(seqidno:23)和g251a(seqidno:24)具有比参照转座酶显著增加的偏好(关于p-值,参见表g)。tn5突变体g251a对低gc-基因座具有增加的插入偏好,这可能是相同突变体在作为低覆盖基因座的基因座处具有增加的插入概率的机制,如在图21中所示。
图24显示了来自三种tn5突变体的跳跃位点数据。与参照转座酶的插入概率相比,这三种突变体e146n、e146a和e145s具有在高-gc基因座中更高的插入概率。执行了分别关于平均值差异和曲线形状的mann-whitney和kolmogorov检验,并将p-值指示在表g中。
表g:选择的突变体的参照数据分布的统计分析。对于kolmogorov-smirnoff和mann-whitney分析,<0.15和<0.05的p-值分别被视作显著的。
*用于参照的序列的数目:2005个读出。
- 还没有人留言评论。精彩留言会获得点赞!
- 一种新的多肽——人支链转酰酶31和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人支链alpha-酮酸脱氢酶(BCKDH)E1-beta亚基11和编码这种多肽的 ...的制作方法
- 一种新的多肽——人遍在蛋白c端水解酶8.8和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人支链alpha-酮酸脱氢酶(BCKDH)E1-beta亚基19.14和编码这种多 ...的制作方法
- 一种新的多肽——人缬氨酰tRNA合成酶33和编码这种多肽的多核苷酸的制作方法
- 利用柚子皮粉生产低温果胶酶及低温澄清苹果汁的方法
- 一种新的多肽——人遍在蛋白c端水解酶13和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人分支alpha-酮酸脱氢酶E1-beta亚单位9和编码这种多肽的多核苷酸的制作方法