一种游动球菌耐盐碱基因mceT及其鉴定方法与流程
本发明属于基因工程技术领域,尤其涉及一种游动球菌耐盐碱基因mcet及其鉴定方法。
背景技术:
目前,业内常用的现有技术是这样的:
目前认为实现盐碱地土壤改良需要多学科共同攻关才能得以完成。从生物学角度,主要的方法则是从微生物和植物同时入手,高效耐盐微生物与植物协同作用。现有表明固氮菌、根瘤菌等微生物肥料的施用与化学肥料相比,在促进高效耐盐碱植物改良盐碱地土壤方面,效果更加明显。但很多植物无法直接获得盐碱地贫瘠的碳氮源,且只施用化学肥料则会造成更严重的环境灾难。
可用于土壤改良的微生物肥料和植物却不具有抵抗高盐碱的能力,这成了目前技术的难题。因此,除了发掘高效耐盐碱的微生物和植物资源以外,规模化地发掘高效耐盐碱的新功能基因用以构建相应基因工程菌株微生物肥料和转基因植物显得尤为重要。
na+/h+逆向转运蛋白是能够利用跨膜质子梯度势将细胞内na+排出到细胞外的一类次级转运蛋白,是生物维持体内na+和ph稳态的主要蛋白。由于na+/h+逆向转运蛋白同时具有li+/h+逆向转运蛋白活性,它们也被称为na+(li+)/h+逆向转运蛋白。其中有些报道显示出同时具有k+/h+逆向转运蛋白。根据其蛋白结构的不同,na+/h+逆向转运蛋白分为nhaa、nhab、nhac、nhad、nhae、cpa1、cpa2、cpa3和dha1这9大家族。其中除cpa3家族由多个基因组成以外,其余都是单个基因编码的蛋白。na+/h+逆向转运蛋白是微生物以及植物抵抗盐碱胁迫的一类重要蛋白,但是目前已知的的na+/h+逆向转运蛋白种类有限,功能单一,且应用较少。
综上所述,现有技术存在的问题是:
现有技术中很多根际促生微生物和植物无法直接在盐碱地土壤上正常生长,且物理化学方法对土壤改造存在着环境污染的潜在问题。
现有技术中na+/h+逆向转运蛋白是微生物以及植物抵抗盐碱胁迫的一类重要蛋白,目前已知的的na+/h+逆向转运蛋白种类有限,功能单一,且应用较少。
技术实现要素:
针对现有技术存在的问题,本发明提供了一种游动球菌耐盐碱基因mcet及其鉴定方法。
本发明是这样实现的,一种游动球菌耐盐碱基因mcet,所述的游动球菌耐盐碱基因mcet,含一个921bp的完整开放阅读框orf,orf的核苷酸序列为seqidno.1。
本发明的另一目的在于提供一种利用所述游动球菌耐盐碱基因mcet编码的蛋白,所述的蛋白氨基酸序列为seqidno.2。
进一步,所述蛋白具有na+(li+、k+)/h+逆向转运蛋白活性,且na+(li+、k+)/h+逆向转运蛋白活性具有ph依赖性。
进一步,所述蛋白具有促进zn2+吸收转运能力。
本发明的另一目的在于提供一种所述游动球菌耐盐碱基因mcet的鉴定方法包括以下步骤:
(1)通过基因文库与基因功能互补相结合的筛选技术,从游动球菌中获得含有耐盐碱相关的基因片段,所述的耐盐碱相关的基因片段中含有一个长921bp的mcet基因的完整开放阅读框orf,orf的核苷酸序列如seqidno.1。
(2)构建含有游动球菌耐盐碱基因mcet基因的原核表达载体;
(3)通过化转异源宿主escherichiacoliknabc,将携带基因mcet的质粒电击转化的方法导入至knabc宿主中,对基因mcet生理功能及编码的蛋白活性进行鉴定。
本发明的另一目的在于提供一种利用所述游动球菌耐盐碱基因mcet构建的原核表达载体,所述原核表达载体为ptrchisb-mcet。
本发明的另一目的在于提供一种原核表达载体的构建方法包括:
根据mcet基因序列,首先设计引物,并在基因的5’和3’端分别引入bamhi和hindiii两个酶切位点,然后进行mcet基因的pcr扩增,对mcet基因进行亚克隆;
将pcr扩增产物酶切纯化后连接到表达载体ptrchisb上,化转到knabc感受态细胞中,将转化产物进行蓝白斑筛选;对得到的转化子进行测序。
本发明的另一目的在于提供一种利用所述游动球菌耐盐碱基因mcet制备的开发微生物肥料。
本发明的另一目的在于提供一种利用所述游动球菌耐盐碱基因mcet表达的失去耐盐功能突变体,所述失去耐盐功能突变体只具有促进zn2+吸收能力,氨基酸定点突变体为q150a、s158a。
本发明的另一目的在于提供一种利用所述游动球菌耐盐碱基因mcet表达的失去促进zn2+吸收能力突变体,所述失去促进zn2+吸收能力突变体只具有耐盐功能的氨基酸定点突变体d41a、d154a。
综上所述,本发明的优点及积极效果为:
本发明提供了一种德昌游动球菌新型na+/h+逆向转运蛋白基因mcet及其鉴定方法。mcet蛋白根据其序列和结构相似性属于阳离子扩散促进家族(cationdiffusionfacilitatorfamily),所有已鉴定的这一家族蛋白都是只具有二价阳离子(me2+)外排转运功能,而发明发现的mcet蛋白具有单价阳离子(me+)外排转运功能,能够有效的提高一株盐碱敏感型大肠杆菌突变株knabc的盐碱抗性;mcet蛋白同时具有促进zn2+向细胞内扩散的功能,能够抑制一株zn2+敏感型大肠杆菌突变株在含0.75mmzncl2的培养基中的生长。并且构建了mcet蛋白的多种突变体,使其只能行使耐盐碱或促进zn2+向细胞内扩散的其中一种功能。类似松嫩平原盐碱地的碳酸盐型盐碱地具有高盐浓度,高ph值,低zn2+浓度的特点。本发明的功能将有效提高有机体的耐盐碱能力和zn2+摄取能力。
附图说明
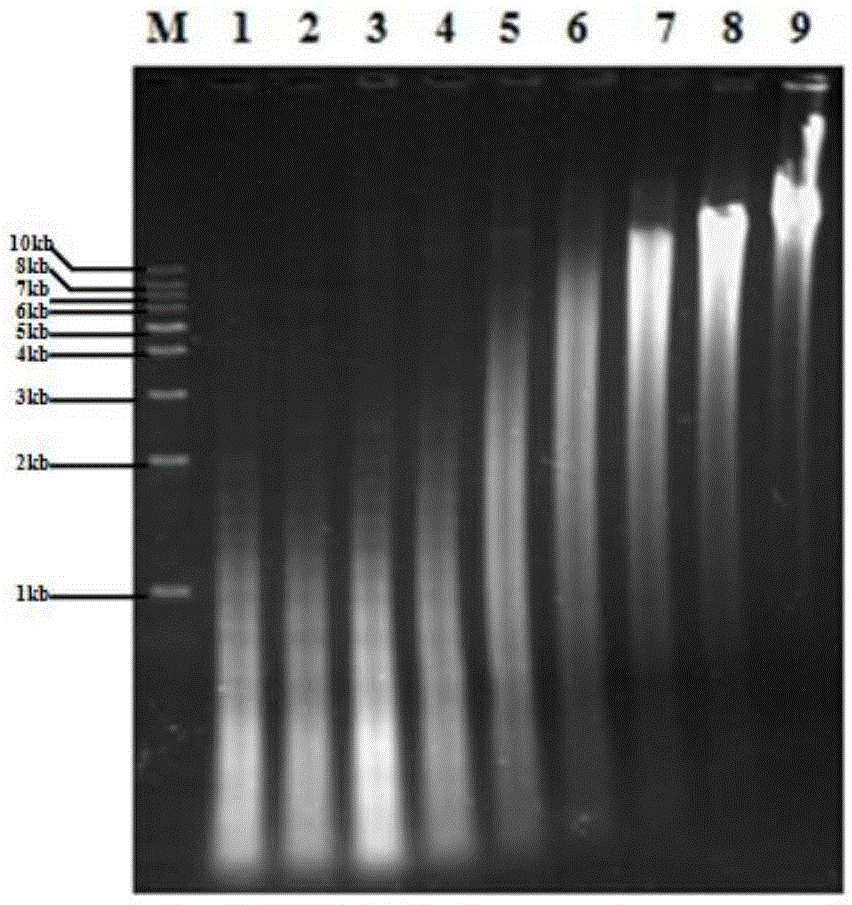
图1是本发明实施例提供的neau-st10-9总基因组部分酶切结果电泳图。
图中:第1-9泳道使用的酶量依次为:4.950u,2.475u,1.2375u,0.61875u,0.30937u,0.15468u,0.07734u,0.03867u,0u。m:1kbdnaladde。
图2是本发明实施例提供的载体puc18的完全酶切结果电泳图。
图中:m:trans2kplusiidnamarker;1:puc18质粒;2:puc18质粒bamhi完全酶切产物。
图3是本发明实施例提供的puc-s5目的片段序列分析图。
图4是本发明实施例提供的mcet蛋白耐盐碱生理活性测定图。
图中:黑色柱为knabc/ptrchisb-mcet,白色柱为knabc/ptrchisb。
图5是本发明实施例提供的mcet蛋白na+(li+、k+)/h+逆向转运活性的测定图。
图中:(a)、(b)、(c)为在ph8.0条件下反转膜活性测试图,图左为knabc/ptrchisb的反转膜活性测试,图右为knabc/ptrchisb的反转膜活性;(d)图为knabc/ptrchisb-mcet在不同ph缓冲液中计算后反转膜活性值。
图6是本发明实施例提供的mcet蛋白促进zn2+吸收生理活性测定图。
图7是本发明实施例提供的mcet蛋白促进zn2+吸收原子吸收实验测定图。
图8是本发明实施例提供的mcet的拓扑学分析图。
图中:灰线代表靠近周质空间一侧的亲水性环(loop);黑色线组成的长方形代表疏水性跨膜区;粗线代表靠近细胞质一侧的亲水性环(loop)。
图9是本发明实施例提供的mcet蛋白的表达和亚细胞定位检测图。
图中:(a)图为knabc/ptrchisb-mcet和knabc/ptrchisb各组分蛋白sds-page胶;(b)图为(a)图对应的western-blot检测。
图10是本发明实施例提供的mcet蛋白突变体功能分析图。
具体实施方式
为了使本发明的目的、技术方案及优点更加清楚明白,以下结合实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
现有技术中很多植物无法直接获得盐碱地贫瘠的碳氮源,且只施用化学肥料则会造成更严重的环境污染。
现有技术中na+/h+逆向转运蛋白是微生物以及植物抵抗盐碱胁迫的一类重要蛋白,目前已知的的na+/h+逆向转运蛋白种类有限,且应用较少。
为解决上述问题,下面结合具体方案对本发明做详细描述。
本发明实施例提供的德昌游动球菌耐盐碱基因mcet,所述德昌游动球菌耐盐碱基因mcet,含一个921bp的完整开放阅读框orf,orf的核苷酸序列为seqidno.1。
本发明实施例提供的一种利用所述德昌游动球菌耐盐碱基因mcet编码的蛋白,氨基酸序列为seqidno.2。
在本发明实施例中,所述蛋白在荧光猝灭实验中具有na+(li+、k+)/h+逆向转运蛋白活性,且na+(li+、k+)/h+逆向转运蛋白活性具有ph依赖性。
本发明实施例提供的一种德昌游动球菌耐盐碱基因mcet的鉴定方法,所述德昌游动球菌耐盐碱基因mcet的鉴定方法包括以下步骤:
(1)通过基因文库与基因功能互补相结合的筛选技术,从德昌游动球菌中获得含有耐盐碱相关的基因片段,所述的耐盐碱相关的基因片段中含有一个长921bp的mcet基因的完整开放阅读框orf,orf的核苷酸序列如seqidno.1;
(2)构建含有德昌游动球菌耐盐碱基因mcet基因的原核表达载体。
(3)通过化转异源宿主escherichiacoliknabc,将携带基因mcet的质粒电击转化的方法导入至knabc宿主中,对基因mcet生理功能及编码的蛋白活性进行鉴定。
进一步,构建含有mcet基因的原核表达载体,包括原核表达载体ptrchisb-mcet;所述原核表达载体ptrchisb-mcet的构建方法包括:
根据mcet基因序列,首先设计引物,并在基因的5’和3’端分别引入bamhi和hindiii两个酶切位点,然后进行mcet基因的pcr扩增,对mcet基因进行亚克隆。
将pcr扩增产物酶切纯化后连接到表达载体ptrchisb上,化转到knabc感受态细胞中,将转化产物进行蓝白斑筛选;为进一步确定序列的准确性,对得到的转化子进行测序。
本发明实施例中,提供一种利用所述德昌游动球菌耐盐碱基因mcet表达的具有na+(li+)/h+逆向转运蛋白。
本发明实施例中,提供一种利用所述德昌游动球菌耐盐碱基因mcet构建的耐盐碱工程菌。
本发明实施例中,提供一种利用所述德昌游动球菌耐盐碱基因mcet制备的开发微生物肥料。
本发明实施例中,提供一种利用所述德昌游动球菌耐盐碱基因mcet制备的耐盐碱性的转基因植物。
本发明实施例中,提供一种利用所述德昌游动球菌耐盐碱基因mcet表达的具有促进zn2+吸收能力的转运蛋白。
本发明实施例中,提供一种利用所述德昌游动球菌耐盐碱基因mcet表达的失去耐盐功能只具有促进zn2+吸收能力的氨基酸定点突变体q150a、s158a。
本发明实施例中,提供一种利用所述德昌游动球菌耐盐碱基因mcet表达的失去促进zn2+吸收能力只具有耐盐功能的氨基酸定点突变体d41a、d154a。
本发明实施例中,提供一种利用德昌游动球菌耐盐碱基因mcet构建的zn2+富集工程菌。
本发明实施例中,一种利用所述德昌游动球菌耐盐碱基因mcet构建的zn2+富集转基因植物。
本发明实施例中,原核表达载体ptrchisb-mcet的dna序列seqidno.3。
mcet蛋白的氨基酸序列seqidno.2。
mcet蛋白q150a突变体的氨基酸序列seqidno.4。
mcet蛋白s158a突变体的氨基酸序列seqidno.5。
mcet蛋白d41a突变体的氨基酸序列seqidno.6。
mcet蛋白d154a突变体的氨基酸序列seqidno.7。
下面结合具体实施例对本发明作进一步描述。
本发明实施例提供的mcet基因的筛选分离方法包括:
1)利用限制性内切酶sau3ai对planococcusdechangensis(cgmcc1.12151)总基因组进行不完全酶切。由于酶量的不同,相同量的总基因组dna酶切后形成不同酶切程度的片段。如图1所示,为酶切后片段的凝胶电泳图。由电泳图得出,如要获得4-10kb范围的dna片段,最适酶量为每50μl体系加0.1547usau3ai。用最适酶量一半的活性单位数对neau-st10-9总基因组进行大量酶切。酶切后,利用0.7%琼脂糖凝胶电泳回收4-10kb的酶切片段,用于与载体的连接。
2)通过碱裂解法提取puc18质粒,将质粒溶于适量te中,-20℃保存备用。用bamhi对提取的puc18质粒完全酶切,酶切产物用ciap进行去磷酸化处理。如图2所示,为puc18质粒酶切后的凝胶电泳图。由电泳图得出,puc18质粒用bamhi酶切,产生线性的2686bp的条带。
3)为了从planococcusdechangensis中筛选具有na+/h+逆向转运蛋白功能的基因,本发明分别回收经过sau3ai部分酶切的总基因组dna、经过bamhi酶切并去磷酸化的线性puc18,然后将片段与puc18载体连接。连接产物电转化大肠杆菌(e.coli)knabc。通过功能互补实验,在含有0.2mnacl的平板上筛选得到了1个重组质粒,将该质粒命名为puc-s5。
4)通过序列分析,puc-s5插入的目的基因片段如图3所示。mcet为起到耐盐碱功能的基因。mcet的核酸序列如seqidno.1所示,mcet的氨基酸序列如seqno.2所示。
本发明实施例中,mcet基因的表达载体构建方法包括:
通过pcr方法克隆mcet并在前后加入bamhi和hindiii酶切位点,通过酶切,连接到ptrchisb载体中,在n末端融合表达一个6xhis标签,以用于western-blot表达检测。连接产物化转到knabc中,筛选得到阳性克隆knabc/ptrchisb-mcet。
本发明实施例中,mcet蛋白的耐盐碱功能鉴定方法包括:
1)为检测mcet蛋白的耐盐功能,将重组子knabc/ptrchisb-mcet和负对照knabc/ptrchisb分别接种于含有0-0.4mnacl或含0-10mmlicl的ph7.0的lbk培养基中,进行耐盐性测试。结果表明,在ph7.0,0mnacl的lbk液体培养基中,重组子knabc/ptrchisb-mcet和对照组knabc/ptrchisb均能正常生长;当nacl浓度升高到0.2m时,knabc/ptrchisb无法生长,而knabc/ptrchisb-mcet将knabc的nacl耐受性提高到0.3m(图4a)。knabc/ptrchisb在5mmlicl时无法生长,而在knabc/ptrchisb-mcet将licl耐受性提高到7.5-10mm(图4b)。
2)为检测mcet蛋白的耐碱功能,将重组子knabc/ptrchisb-mcet和负对照knabc/ptrchisb分别接种于ph7.0-8.5的lbk培养基。knabc/ptrchisb-mcet在ph7.5时明显比knabc/ptrchisb长势较好,并且将knabc耐碱性提高到ph8.0,而此时knabc/ptrchisb无法生长(图4c)。
mcet蛋白na+(li+、k+)/h+逆向转运活性的测定
3)为了鉴定mcet蛋白的功能,本发明测试了mcet的阳离子/氢离子逆向转运蛋白活性。具体操作为:利用knabc/ptrchisb-mcet和knabc/ptrchisb使用frenchpress法式细胞破碎仪,在1000psi的压强下,破碎细胞,10000g离心去除细胞碎片,再将上清液100000g超速离心收集细胞膜组分,用缓冲液重悬沉淀,即为反转膜。在总膜蛋白含量相同的条件下,分别测定这两种反转膜表现出的na+(li+,k+,)/h+逆向转运蛋白活性。向2ml的反应缓冲液中加入一定量的反转膜和吖啶橙混匀,当向反应混合物中加入乳酸后,会发生呼吸链产生的膜两侧质子浓度变化,从而使吖啶橙的荧光值产生稳定猝灭。这时向两种反应体系中加入等量盐溶液后,反应体系出现荧光值恢复。说明此种反转膜可以利用跨膜ph梯度转运盐离子,反之,说明此种反转膜不能利用跨膜ph梯度转运离子。如图5所示,knabc/ptrchisb-mcet具有na+(li+,k+)/h+逆向转运蛋白活性,而负对照knabc/ptrchisb不具有na+(li+,k+)/h+逆向转运蛋白活性,这表明mcet蛋白具有na+/h+逆向转运蛋白功能。并且mcet蛋白在ph8.0的条件下活性最高。
本发明实施例中,mcet蛋白促进zn2+吸收功能的鉴定方法包括:
1)为检测mcet蛋白的zn2+的转运能力,将表达载体ptrchisb-mcet及副对照ptrchisb转化到一株大肠杆菌zn2+敏感突变株kzab04中,构建得到重组子kzab04/ptrchisb-mcet和kzab04/ptrchisb。并分别按1%接种于含有(0-0.75mmzncl2)的lb培养基中,37℃,150rpm培养24h,其结果如图6所示:表达了mcet蛋白的kzab04/ptrchisb-mcet相比副对照kzab04/ptrchisb在含有0.75mmzncl2的情况下生长受到明显的抑制,几乎不生长,这是因为mcet蛋白促进了zn2+的向细胞内的转运,增加了细胞内zn2+的胁迫作用。
2)为进一步证明mcet蛋白促进了zn2+的吸收,本发明利用原子吸收分光光度计测定菌株kzab04/ptrchisb-mcet和kzab04/ptrchisb了在0.75mmzncl2的缓冲液中不同时间(0-30min)的zn2+积累浓度,以及在20min条件下,在含有0-1.5mmzncl2的缓冲液中的zn2+积累浓度(图7)。由图可见,表达mcet蛋白的菌株的zn2+积累浓度明显高于对照组,再次证明mcet蛋白对zn2+吸收的促进作用。
下面结合具体分析方法对本发明作进一步描述。
本发明的mcet基因的生物信息学分析方法包括:
将mcet基因的氨基酸序列通过ncbi网站进行blast分析,mcet属于cationdiffusionfacilitator(cdf)家族蛋白,与一个bacillusokhensis中的推测的cdf家族蛋白的同源性为63%。而所有已经鉴定的cdf家族蛋白都是二价阳离子/质子逆向转运蛋白,如(zn2+、cd2+、co2+、mn2+、fe2+/h+antiporter),而mcet蛋白是na+(li+,k+)/h+逆向转运蛋白。并且mcet蛋白与所有已鉴定的cdf家族蛋白相比,同源性最高不到30%。因此mcet是属于cdf家族的一个全新的蛋白,不仅仅是序列上是新的,而且功能也是全新的。
本发明的mcet蛋白的拓扑学结构分析方法包括:
使用tmhmm软件对mcet进行拓扑学分析,发现mcet具有6个跨膜螺旋(图8),这与所有已知的na+/h+逆向转运蛋白的结构都不一致,再次证明mcet蛋白的创新性。
本发明的mcet蛋白的亚细胞定位方法包括:
利用法式细胞破碎法,分离knabc/ptrchisb-mcet的总蛋白、膜组分和胞质蛋白,同过sds-page和western-blot检测mcet蛋白的表达位置。从图9可见,mcet蛋白只存在于细胞膜中,而不存在于细胞质中,说明mcet是一个膜蛋白,具有na+(li+、k+)/h+逆向转运活性。
本发明的mcet蛋白氨基酸定点突变体功能分析方法包括:
利用氨基酸定点突变技术,构建了mcet蛋白的17个氨基酸突变体,分别是e8a、s15a、s35a、f40a、d41a、y44a、s45a、e78a、c127a、e147a、q150a、s158a、f165a、y182a、d184a(数字表述氨基酸残基在mcet蛋白中的位置,数字前为突变前的氨基酸残基,数字后表示突变成的氨基酸残基),并通过生理实验分别比较了突变体与野生型在耐盐与促进zn2+吸收能力上的差异性(图10a、b)。图10a表示的是突变体耐盐能力的差异性wt是野生型,emptyvector为空对照,横坐标为相对于野生型的生长比率,越长说明耐盐性越好;图10b表示的是突变体zn2+吸收能力的生理实验,wt是野生型,emptyvector为空对照,横坐标为相对于副对照的生长比率,越长表示促进zn2+吸收能力越弱。图10c为各个突变体蛋白的western-blot结果,以检测突变体是否正常表达。
由图10a、b分析可见,d150a和s158a突变体失去了耐盐能力,而继续保留了促进zn2+吸收的能力;d41a和d154a突变体失去了促进zn2+吸收能力,而保留了耐盐能力;d184a突变体同时失去了耐盐能力和促进zn2+吸收能力。
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明的保护范围之内。
<110>东北农业大学
<120>一种游动球菌耐盐碱基因mcet及其鉴定方法
<160>7
<210>1
<211>921
<212>dna
<213>人工序列(artificialsequence)
<400>1
atgtttggaaaatcaattaacgagaaaaaattattatggatatcggtgattgctgcattg
atttttgcgttagtcggcattgtttgggggattgccatctcttctcaaatcattttgttc
gacggcgcatattctttcatcagcgtattgctgtcgctaatgtcattgatcgtcgcgcgc
tatattcaacaaagtgatgccgcgcgctttccatacggaaaagaaatgttggagcccttg
gtcattatcgtgaaatacaccatcatcttggtgctctgtattgcagccatctctgctgct
atagagtcgctggcgtctggaggtcgcgaagtcagcattgggcacgcactcgtattcgca
gccatcagcacgattggatgcgccgctgtctactggatgctgaaccgccacaaaaagaat
tcgggttttatccgcgcagaagcgaaccagtggaaaatggatactttgctcagcgcggcc
gtgttgttcggattcgccgccgcctgggtggtttccttgacgccttatagatatttgatg
ccgtatgtcgatccgataatggttttgctggtagttgggtatttcctgaaaacacctatc
cgcgaaataaccaagtctttcaaggaagtgcttgaaatgacacctgaccgggatatcgaa
accgagtttaaaacagctgtccaagccattgaatcacgttatcaaataccggagtccatc
gttagggtcgccaaggtaggaaataaattattcatcgatgtagacttcattctagggccg
caatctaaaattgtcacgacggtggatcaggatgaggtaagggcggaaattacccgcaac
acgtctgcagtcggctataagaaatggctgacggtgtcatttacccacgatgcaaaatgg
gcaaagaaaactcatttataa
<210>2
<211>306
<212>氨基酸
<213>人工序列(artificialsequence)
<400>2
metpheglylysserileasnglulyslysleuleutrpileserval
ilealaalaleuilephealaleuvalglyilevaltrpglyileala
ileserserglnileileleupheaspglyalatyrserpheileser
valleuleuserleumetserleuilevalalaargtyrileglngln
seraspalaalaargpheprotyrglylysglumetleugluproleu
valileilevallystyrthrileileleuvalleucysilealaala
ileseralaalailegluserleualaserglyglyarggluvalser
ileglyhisalaleuvalphealaalaileserthrileglycysala
alavaltyrtrpmetleuasnarghislyslysasnserglypheile
argalaglualaasnglntrplysmetaspthrleuleuseralaala
valleupheglyphealaalaalatrpvalvalserleuthrprotyr
argtyrleumetprotyrvalaspproilemetvalleuleuvalval
glytyrpheleulysthrproilearggluilethrlysserphelys
gluvalleuglumetthrproaspargaspilegluthrgluphelys
thralavalglnalailegluserargtyrglnileprogluserile
valargvalalalysvalglyasnlysleupheileaspvalaspphe
ileleuglyproglnserlysilevalthrthrvalaspglnaspglu
valargalagluilethrargasnthrseralavalglytyrlyslys
trpleuthrvalserphethrhisaspalalystrpalalyslysthr
hisleu
305
<210>3
<211>5286
<212>dna
<213>人工序列(artificialsequence)
<400>核苷酸序列3
gtttgacagcttatcatcgactgcacggtgcaccaatgcttctggcgtcaggcagccatcggaagctgtggtatggctgtgcaggtcgtaaatcactgcataattcgtgtcgctcaaggcgcactcccgttctggataatgttttttgcgccgacatcataacggttctggcaaatattctgaaatgagctgttgacaattaatcatccggctcgtataatgtgtggaattgtgagcggataacaatttcacacaggaaacagcgccgctgagaaaaagcgaagcggcactgctctttaacaatttatcagacaatctgtgtgggcactcgaccggaattatcgattaactttattattaaaaattaaagaggtatatattaatgtatcgattaaataaggaggaataaaccatggggggttctcatcatcatcatcatcatggtatggctagcatgactggtggacagcaaatgggtcgggatctgtacgacgatgacgataaggatccgatgtttggaaaatcaattaacgagaaaaaattattatggatatcggtgattgctgcattgatttttgcgttagtcggcattgtttgggggattgccatctcttctcaaatcattttgttcgacggcgcatattctttcatcagcgtattgctgtcgctaatgtcattgatcgtcgcgcgctatattcaacaaagtgatgccgcgcgctttccatacggaaaagaaatgttggagcccttggtcattatcgtgaaatacaccatcatcttggtgctctgtattgcagccatctctgctgctatagagtcgctggcgtctggaggtcgcgaagtcagcattgggcacgcactcgtattcgcagccatcagcacgattggatgcgccgctgtctactggatgctgaaccgccacaaaaagaattcgggttttatccgcgcagaagcgaaccagtggaaaatggatactttgctcagcgcggccgtgttgttcggattcgccgccgcctgggtggtttccttgacgccttatagatatttgatgccgtatgtcgatccgataatggttttgctggtagttgggtatttcctgaaaacacctatccgcgaaataaccaagtctttcaaggaagtgcttgaaatgacacctgaccgggatatcgaaaccgagtttaaaacagctgtccaagccattgaatcacgttatcaaataccggagtccatcgttagggtcgccaaggtaggaaataaattattcatcgatgtagacttcattctagggccgcaatctaaaattgtcacgacggtggatcaggatgaggtaagggcggaaattacccgcaacacgtctgcagtcggctataagaaatggctgacggtgtcatttacccacgatgcaaaatgggcaaagaaaactcatttataagcttggctgttttggcggatgagagaagattttcagcctgatacagattaaatcagaacgcagaagcggtctgataaaacagaatttgcctggcggcagtagcgcggtggtcccacctgaccccatgccgaactcagaagtgaaacgccgtagcgccgatggtagtgtggggtctccccatgcgagagtagggaactgccaggcatcaaataaaacgaaaggctcagtcgaaagactgggcctttcgttttatctgttgtttgtcggtgaacgctctcctgagtaggacaaatccgccgggagcggatttgaacgttgcgaagcaacggcccggagggtggcgggcaggacgcccgccataaactgccaggcatcaaattaagcagaaggccatcctgacggatggcctttttgcgtttctacaaactctttttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataaccctgataaatgcttcaataatattgaaaaaggaagagtatgagtattcaacatttccgtgtcgcccttattcccttttttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaaagtaaaagatgctgaagatcagttgggtgcacgagtgggttacatcgaactggatctcaacagcggtaagatccttgagagttttcgccccgaagaacgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgtgttgacgccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggttgagtactcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgccataaccatgagtgataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgcttttttgcacaacatgggggatcatgtaactcgccttgatcgttgggaaccggagctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgtagcaatggcaacaacgttgcgcaaactattaactggcgaactacttactctagcttcccggcaacaattaatagactggatggaggcggataaagttgcaggaccacttctgcgctcggcccttccggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgcggtatcattgcagcactggggccagatggtaagccctcccgtatcgtagttatctacacgacggggagtcaggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactgattaagcattggtaactgtcagaccaagtttactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgcctgatgcggtattttctccttacgcatctgtgcggtatttcacaccgcatatggtgcactctcagtacaatctgctctgatgccgcatagttaagccagtatacactccgctatcgctacgtgactgggtcatggctgcgccccgacacccgccaacacccgctgacgcgccctgacgggcttgtctgctcccggcatccgcttacagacaagctgtgaccgtctccgggagctgcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcgaggcagcagatcaattcgcgcgcgaaggcgaagcggcatgcatttacgttgacaccatcgaatggtgcaaaacctttcgcggtatggcatgatagcgcccggaagagagtcaattcagggtggtgaatgtgaaaccagtaacgttatacgatgtcgcagagtatgccggtgtctcttatcagaccgtttcccgcgtggtgaaccaggccagccacgtttctgcgaaaacgcgggaaaaagtggaagcggcgatggcggagctgaattacattcccaaccgcgtggcacaacaactggcgggcaaacagtcgttgctgattggcgttgccacctccagtctggccctgcacgcgccgtcgcaaattgtcgcggcgattaaatctcgcgccgatcaactgggtgccagcgtggtggtgtcgatggtagaacgaagcggcgtcgaagcctgtaaagcggcggtgcacaatcttctcgcgcaacgcgtcagtgggctgatcattaactatccgctggatgaccaggatgccattgctgtggaagctgcctgcactaatgttccggcgttatttcttgatgtctctgaccagacacccatcaacagtattattttctcccatgaagacggtacgcgactgggcgtggagcatctggtcgcattgggtcaccagcaaatcgcgctgttagcgggcccattaagttctgtctcggcgcgtctgcgtctggctggctggcataaatatctcactcgcaatcaaattcagccgatagcggaacgggaaggcgactggagtgccatgtccggttttcaacaaaccatgcaaatgctgaatgagggcatcgttcccactgcgatgctggttgccaacgatcagatggcgctgggcgcaatgcgcgccattaccgagtccgggctgcgcgttggtgcggatatctcggtagtgggatacgacgataccgaagacagctcatgttatatcccgccgttaaccaccatcaaacaggattttcgcctgctggggcaaaccagcgtggaccgcttgctgcaactctctcagggccaggcggtgaagggcaatcagctgttgcccgtctcactggtgaaaagaaaaaccaccctggcgcccaatacgcaaaccgcctctccccgcgcgttggccgattcattaatgcagctggcacgacaggtttcccgactggaaagcgggcagtgagcgcaacgcaattaatgtgagttagcgcgaattgatctg
<210>4
<211>306
<212>氨基酸
<213>人工序列(artificialsequence)
<400>4
metpheglylysserileasnglulyslysleuleutrpileserval
ilealaalaleuilephealaleuvalglyilevaltrpglyileala
ileserserglnileileleupheaspglyalatyrserpheileservalleuleuserleumetserleuilevalalaargtyrileglnglnseraspalaalaargpheprotyrglylysglumetleugluproleu
valileilevallystyrthrileileleuvalleucysilealaala
ileseralaalailegluserleualaserglyglyarggluvalser
ileglyhisalaleuvalphealaalaileserthrileglycysala
alavaltyrtrpmetleuasnarghislyslysasnserglypheile
argalaglualaasnalatrplysmetaspthrleuleuseralaala
valleupheglyphealaalaalatrpvalvalserleuthrprotyr
argtyrleumetprotyrvalaspproilemetvalleuleuvalval
glytyrpheleulysthrproilearggluilethrlysserphelys
gluvalleuglumetthrproaspargaspilegluthrgluphelys
thralavalglnalailegluserargtyrglnileprogluserile
valargvalalalysvalglyasnlysleupheileaspvalaspphe
ileleuglyproglnserlysilevalthrthrvalaspglnaspglu
valargalagluilethrargasnthrseralavalglytyrlyslys
trpleuthrvalserphethrhisaspalalystrpalalyslysthr
hisleu
<210>5
<211>306
<212>氨基酸
<213>人工序列(artificialsequence)
<400>5
metpheglylysserileasnglulyslysleuleutrpileserval
ilealaalaleuilephealaleuvalglyilevaltrpglyileala
ileserserglnileileleupheaspglyalatyrserpheileser
valleuleuserleumetserleuilevalalaargtyrileglngln
seraspalaalaargpheprotyrglylysglumetleugluproleu
valileilevallystyrthrileileleuvalleucysilealaala
ileseralaalailegluserleualaserglyglyarggluvalser
ileglyhisalaleuvalphealaalaileserthrileglycysala
alavaltyrtrpmetleuasnarghislyslysasnserglypheile
argalaglualaasnglntrplysmetaspthrleuleualaalaala
valleupheglyphealaalaalatrpvalvalserleuthrprotyr
argtyrleumetprotyrvalaspproilemetvalleuleuvalval
glytyrpheleulysthrproilearggluilethrlysserphelys
gluvalleuglumetthrproaspargaspilegluthrgluphelys
thralavalglnalailegluserargtyrglnileprogluserile
valargvalalalysvalglyasnlysleupheileaspvalaspphe
ileleuglyproglnserlysilevalthrthrvalaspglnaspglu
valargalagluilethrargasnthrseralavalglytyrlyslys
trpleuthrvalserphethrhisaspalalystrpalalyslysthr
hisleu
<210>6
<211>306
<212>氨基酸
<213>人工序列(artificialsequence)
<400>6
metpheglylysserileasnglulyslysleuleutrpileserval
ilealaalaleuilephealaleuvalglyilevaltrpglyileala
ileserserglnileileleuphealaglyalatyrserpheileser
valleuleuserleumetserleuilevalalaargtyrileglngln
seraspalaalaargpheprotyrglylysglumetleugluproleu
valileilevallystyrthrileileleuvalleucysilealaala
ileseralaalailegluserleualaserglyglyarggluvalser
ileglyhisalaleuvalphealaalaileserthrileglycysala
alavaltyrtrpmetleuasnarghislyslysasnserglypheile
argalaglualaasnglntrplysmetaspthrleuleuseralaala
valleupheglyphealaalaalatrpvalvalserleuthrprotyr
argtyrleumetprotyrvalaspproilemetvalleuleuvalval
glytyrpheleulysthrproilearggluilethrlysserphelys
gluvalleuglumetthrproaspargaspilegluthrgluphelys
thralavalglnalailegluserargtyrglnileprogluserile
valargvalalalysvalglyasnlysleupheileaspvalaspphe
ileleuglyproglnserlysilevalthrthrvalaspglnaspglu
valargalagluilethrargasnthrseralavalglytyrlyslys
trpleuthrvalserphethrhisaspalalystrpalalyslysthr
hisleu
<210>7
<211>306
<212>氨基酸
<213>人工序列(artificialsequence)
<400>7
metpheglylysserileasnglulyslysleuleutrpileserval
ilealaalaleuilephealaleuvalglyilevaltrpglyileala
ileserserglnileileleupheaspglyalatyrserpheileser
valleuleuserleumetserleuilevalalaargtyrileglngln
seraspalaalaargpheprotyrglylysglumetleugluproleu
valileilevallystyrthrileileleuvalleucysilealaala
ileseralaalailegluserleualaserglyglyarggluvalser
ileglyhisalaleuvalphealaalaileserthrileglycysala
alavaltyrtrpmetleuasnarghislyslysasnserglypheile
argalaglualaasnglntrplysmetalathrleuleuseralaala
valleupheglyphealaalaalatrpvalvalserleuthrprotyr
argtyrleumetprotyrvalaspproilemetvalleuleuvalval
glytyrpheleulysthrproilearggluilethrlysserphelys
gluvalleuglumetthrproaspargaspilegluthrgluphelys
thralavalglnalailegluserargtyrglnileprogluserile
valargvalalalysvalglyasnlysleupheileaspvalaspphe
ileleuglyproglnserlysilevalthrthrvalaspglnaspglu
valargalagluilethrargasnthrseralavalglytyrlyslys
trpleuthrvalserphethrhisaspalalystrpalalyslysthr
hisleu
- 还没有人留言评论。精彩留言会获得点赞!