用于鉴定毛白杨单倍体或纯合二倍体的引物组及其应用的制作方法
本发明涉及用于鉴定毛白杨单倍体或纯合二倍体的引物组,还涉及所述引物组在鉴定毛白杨单倍体或纯合二倍体中的应用,属于毛白杨单倍体或纯合二倍体的分子生物学鉴定领域。
背景技术:
正常的二倍体含有两套染色体,分别来自父本和母本。而单倍体则只含有一套染色体,来自父本或母本。花药或花粉培养是获得单倍体的主要手段。植株单倍体在基因组学研究方面,尤其在基因组测序、基因功能的鉴定和构建分子遗传图谱等领域具有突出作用。自从1964年guha等人利用花药离体培养成功获得曼陀罗花粉植株以来,世界各国相继开展了杨树花药培养诱导单倍体植株的研究。
鉴定单倍体植株和二倍体植株的方法有许多,包括形态学特征观察、细胞学水平观察、流式细胞仪技术等。随着分子生物技术的快速发展,以dna分子为基础的倍性测定方法如dna分子标记技术已经在多种植物中得以成功应用。插入缺失序列indel分子标记,具有数量丰富、共显性遗传、信息含量高、分析方法简单、结果重现性好等优点。开发一种利用普通pcr和indel分子标记相结合鉴定毛白杨单倍体或纯合二倍体的方法,将对于毛白杨在遗传育种、分子和生物信息学等方面的研究及应用奠定重要基础。
技术实现要素:
本发明所要解决的第一个技术问题是提供用于鉴定毛白杨单倍体或纯合二倍体的引物组;
本发明所要解决的第二个技术问题是提供所述的引物组在鉴定毛白杨单倍体或纯合二倍体中的应用。
为解决上述技术问题,本发明所采取的技术方案是:
本发明基于毛白杨(lm50)基因组数据的信息,设计了位于毛白杨19条染色体的多对pcr引物,通过以毛白杨lm50的基因组dna为模板分别对所设计的pcr引物进行筛选,结果筛选出了分别位于19条染色体上的19对特异引物。
本发明所筛选的用于鉴定毛白杨单倍体或纯合二倍体的pcr引物,包括:由核苷酸序列为seqidno.1所示的上游引物和核苷酸序列为seqidno.2所示的下游引物组成的引物对1;或者,
由核苷酸序列为seqidno.3所示的上游引物和核苷酸序列为seqidno.4所示的下游引物组成的引物对2;或者,
由核苷酸序列为seqidno.5所示的上游引物和核苷酸序列为seqidno.6所示的下游引物组成的引物对3;或者,
由核苷酸序列为seqidno.7所示的上游引物和核苷酸序列为seqidno.8所示的下游引物组成的引物对4;或者,
由核苷酸序列为seqidno.9所示的上游引物和核苷酸序列为seqidno.10所示的下游引物组成的引物对5;或者,
由核苷酸序列为seqidno.11所示的上游引物和核苷酸序列为seqidno.12所示的下游引物组成的引物对6;或者,
由核苷酸序列为seqidno.13所示的上游引物和核苷酸序列为seqidno.14所示的下游引物组成的引物对7;或者,
由核苷酸序列为seqidno.15所示的上游引物和核苷酸序列为seqidno.16所示的下游引物组成的引物对8;或者,
由核苷酸序列为seqidno.17所示的上游引物和核苷酸序列为seqidno.18所示的下游引物组成的引物对9;或者,
由核苷酸序列为seqidno.19所示的上游引物和核苷酸序列为seqidno.20所示的下游引物组成的引物对10;或者,
由核苷酸序列为seqidno.21所示的上游引物和核苷酸序列为seqidno.22所示的下游引物组成的引物对11;或者,
由核苷酸序列为seqidno.23所示的上游引物和核苷酸序列为seqidno.24所示的下游引物组成的引物对12;或者,
由核苷酸序列为seqidno.25所示的上游引物和核苷酸序列为seqidno.26所示的下游引物组成的引物对13;或者,
由核苷酸序列为seqidno.27所示的上游引物和核苷酸序列为seqidno.28所示的下游引物组成的引物对14;或者,
由核苷酸序列为seqidno.29所示的上游引物和核苷酸序列为seqidno.30所示的下游引物组成的引物对15;或者,
由核苷酸序列为seqidno.31所示的上游引物和核苷酸序列为seqidno.32所示的下游引物组成的引物对16;或者,
由核苷酸序列为seqidno.33所示的上游引物和核苷酸序列为seqidno.34所示的下游引物组成的引物对17;或者,
由核苷酸序列为seqidno.35所示的上游引物和核苷酸序列为seqidno.36所示的下游引物组成的引物对18;或者,
由核苷酸序列为seqidno.37所示的上游引物和核苷酸序列为seqidno.38所示的下游引物组成的引物对19中的任意一对或多对。
本发明进一步将所筛选出的19对pcr引物设计indel分子标记引物(荧光标记选用m13)。
进一步的,本发明公开了用于鉴定毛白杨单倍体或纯合二倍体的indel引物,包括:由核苷酸序列为seqidno.39所示的上游引物和核苷酸序列为seqidno.2所示的下游引物组成的引物对20;或者,
由核苷酸序列为seqidno.40所示的上游引物和核苷酸序列为seqidno.4所示的下游引物组成的引物对21;或者,
由核苷酸序列为seqidno.41所示的上游引物和核苷酸序列为seqidno.6所示的下游引物组成的引物对22;或者,
由核苷酸序列为seqidno.42所示的上游引物和核苷酸序列为seqidno.8所示的下游引物组成的引物对23;或者,
由核苷酸序列为seqidno.43所示的上游引物和核苷酸序列为seqidno.10所示的下游引物组成的引物对24;或者,
由核苷酸序列为seqidno.44所示的上游引物和核苷酸序列为seqidno.12所示的下游引物组成的引物对25;或者,
由核苷酸序列为seqidno.45所示的上游引物和核苷酸序列为seqidno.14所示的下游引物组成的引物对26;或者,
由核苷酸序列为seqidno.46所示的上游引物和核苷酸序列为seqidno.16所示的下游引物组成的引物对27;或者,
由核苷酸序列为seqidno.47所示的上游引物和核苷酸序列为seqidno.18所示的下游引物组成的引物对28;或者,
由核苷酸序列为seqidno.48所示的上游引物和核苷酸序列为seqidno.20所示的下游引物组成的引物对29;或者,
由核苷酸序列为seqidno.49所示的上游引物和核苷酸序列为seqidno.22所示的下游引物组成的引物对30;或者,
由核苷酸序列为seqidno.50所示的上游引物和核苷酸序列为seqidno.24所示的下游引物组成的引物对31;或者,
由核苷酸序列为seqidno.51所示的上游引物和核苷酸序列为seqidno.26所示的下游引物组成的引物对32;或者,
由核苷酸序列为seqidno.52所示的上游引物和核苷酸序列为seqidno.28所示的下游引物组成的引物对33;或者,
由核苷酸序列为seqidno.53所示的上游引物和核苷酸序列为seqidno.30所示的下游引物组成的引物对34;或者,
由核苷酸序列为seqidno.54所示的上游引物和核苷酸序列为seqidno.32所示的下游引物组成的引物对35;或者,
由核苷酸序列为seqidno.55所示的上游引物和核苷酸序列为seqidno.34所示的下游引物组成的引物对36;或者,
由核苷酸序列为seqidno.56所示的上游引物和核苷酸序列为seqidno.36所示的下游引物组成的引物对37;或者,
由核苷酸序列为seqidno.57所示的上游引物和核苷酸序列为seqidno.38所示的下游引物组成的引物对38中的任意一对或多对。
本发明进一步公开了所述的pcr引物或者indel引物在鉴定毛白杨单倍体或纯合二倍体中的应用。
本发明所述毛白杨单倍体为由毛白杨lm50花药诱导得到的单倍体植株;所述毛白杨纯合二倍体为所述单倍体植株自然加倍形成的纯合二倍体。进一步优选的,本发明所述毛白杨单倍体为由毛白杨lm50花药诱导得到的单倍体植株gm107;所述毛白杨纯合二倍体为所述单倍体植株gm107自然加倍形成的纯合二倍体。
本发明还公开了一种鉴定毛白杨单倍体或纯合二倍体的pcr方法,包括以下步骤:(1)提取待测毛白杨的基因组dna;(2)以提取的dna为模板,以本发明所述pcr引物建立pcr反应体系,进行pcr扩增;(3)对扩增产物进行琼脂糖凝胶电泳,若电泳结果显示为单一条带,则待测毛白杨是单倍体或纯合二倍体。
其中,所述pcr反应体系包括:反应体系的总体积为20μl,其中,2μl10×pcrbuffer,3.2μldntp,上游引物和下游引物各0.4μl,2μldna模板,0.2μl用lataq酶,11.8μlddh2o。所述pcr扩增的程序包括:94℃预变性3min;94℃变性30s,60℃退火30s,72℃延伸90s,进行34个循环;72℃延伸5min。
本发明还公开了一种鉴定毛白杨单倍体或纯合二倍体的indel方法,包括以下步骤:(1)提取待测毛白杨的基因组dna;(2)以提取的dna为模板,以本发明所述indel引物建立pcr反应体系,进行pcr扩增;(3)对扩增产物进行毛细管电泳,若电泳结果显示为单一峰,则待测毛白杨是单倍体或纯合二倍体。
其中,所述pcr反应体系包括:反应体系的总体积为25μl,其中,2μl10×pcrbuffer,3.2μldntp,上游引物和下游引物各0.4μl,5μlm13,2μldna模板,0.2μllataq酶,11.8μlddh2o。所述pcr扩增的程序包括:94℃预变性3min;94℃变性30s,60℃退火30s,72℃延伸1min,进行34个循环;72℃延伸5min。
本发明优选将上述pcr方法和indel分子标记的方法相结合,用于鉴定毛白杨单倍体或纯合二倍体。
本发明利用筛选出的分别位于毛白杨19条染色体上的19对pcr引物对毛白杨lm50和花药诱导的单倍体植株进行鉴定,以毛白杨lm50和花药诱导的单倍体植株叶片基因组dna为模板进行pcr扩增,采用琼脂糖凝胶电泳检测pcr产物扩增情况,pcr结果显示lm50均为两条带,而由花药诱导的单倍体gm107则为单一条带。本发明进一步将筛选出的19对引物设计indel分子标记引物进行毛细管电泳检测,检测结果与上述pcr结果相符。本发明通过pcr方法和indel分子标记的方法相结合鉴定出毛白杨lm50通过花药诱导得到单倍体植株。
本发明所设计的位于毛白杨9条染色体的9对引物chr2-1、chr4-1、chr6-1、chr7-1、chr9-1、chr13-1、chr16-1、chr18-1、chr19-1均不能鉴定由毛白杨lm50花药诱导的单倍体植株。
本发明技术方案与现有技术相比,具有以下有益效果:
本发明基于毛白杨(lm50)基因组数据的信息,设计并筛选出了分别位于毛白杨19条染色体上的19对pcr引物。本发明进一步将所筛选出的19对pcr引物设计indel分子标记引物。本发明通过普通pcr和indel分子标记的方法鉴定毛白杨单倍体或纯合二倍体,为之后在毛白杨遗传育种、分子和生物信息学方面研究等奠定了基础。
附图说明
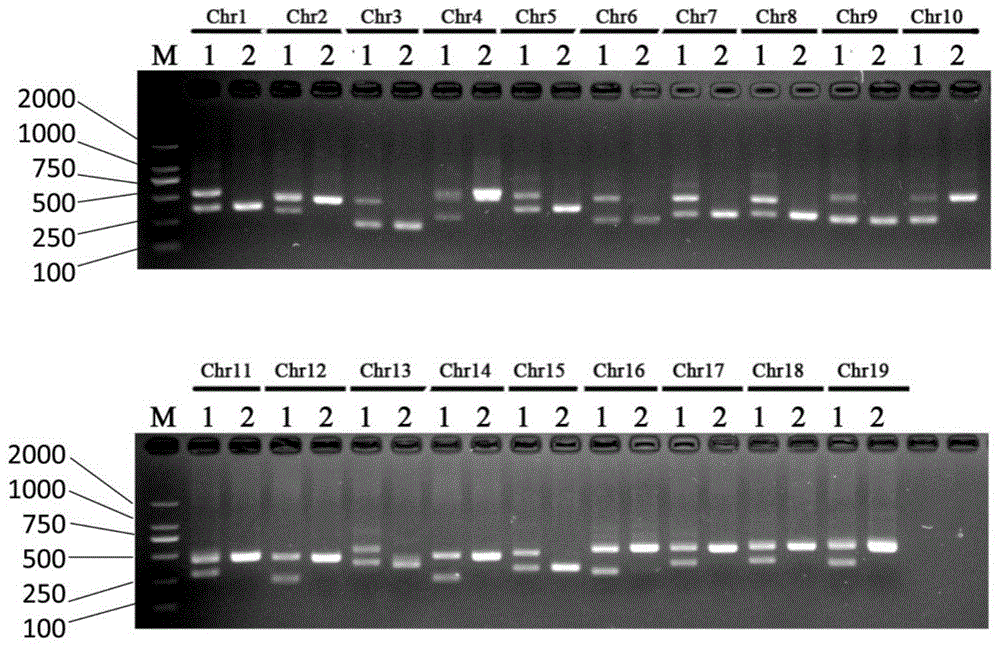
图1为19对毛白杨目的基因引物扩增产物;其中,m:dl2000marker;1:lm50;2:花药诱导的单倍体;
图2为毛白杨目的基因分子标记引物chr1、chr2扩增产物;其中,+:lm50;-:花药诱导的单倍体;
图3为毛白杨目的基因分子标记引物chr3、chr4扩增产物;其中,+:lm50;-:花药诱导的单倍体;
图4为毛白杨目的基因分子标记引物chr5、chr6扩增产物;其中,+:lm50;-:花药诱导的单倍体;
图5为毛白杨目的基因分子标记引物chr7、chr8扩增产物;其中,+:lm50;-:花药诱导的单倍体;
图6为毛白杨目的基因分子标记引物chr9、chr10扩增产物;其中,+:lm50;-:花药诱导的单倍体;
图7为毛白杨目的基因分子标记引物chr11、chr12扩增产物;其中,+:lm50;-:花药诱导的单倍体;
图8为毛白杨目的基因分子标记引物chr13、chr14扩增产物;其中,+:lm50;-:花药诱导的单倍体;
图9为毛白杨目的基因分子标记引物chr15、chr16扩增产物;其中,+:lm50;-:花药诱导的单倍体;
图10为毛白杨目的基因分子标记引物chr17、chr18扩增产物;其中,+:lm50;-:花药诱导的单倍体;
图11为毛白杨目的基因分子标记引物chr19扩增产物;其中,+:lm50;-:花药诱导的单倍体;
图12为19对毛白杨目的基因引物扩增gm00、gm15、gm93植株产物,以上三种植株属于花药诱导的杂合植株;其中,1-19分别代表表1中位于19条染色体上的引物信息;
图13为9对毛白杨目的基因引物扩增产物;其中,chr2-1、chr4-1、chr6-1、chr7-1、chr9-1、chr13-1、chr16-1、chr18-1、chr19-1分别代表位于2、4、6、7、9、13、16、18、19号染色体的引物片段;1、2、3、4、5分别代表lm50、gm107、gm00、gm15、gm93植株;
图2-图11中,横坐标为扩增片段大小;纵坐标为荧光信号强度。
具体实施方式
下面结合具体实施例来进一步描述本发明,本发明的优点和特点将会随着描述而更为清楚。但是应理解所述实施例仅是范例性的,不对本发明的范围构成任何限制。本领域技术人员应该理解的是,在不偏离本发明的精神和范围下可以对本发明技术方案的细节和形式进行修改或替换,但这些修改或替换均落入本发明的保护范围。
实施例1鉴定毛白杨单倍体或纯合二倍体的引物组设计及筛选
1、材料
毛白杨lm50(雄株)为杂合二倍体,定植于北京林业大学科研基地山东冠县国有苗圃,采集其花枝,按照li等(liying,lihao,chenzhong,jile-xiang,yemei-xia,wangjia,wanglina,anxin-min*haploidplantsfromantherculturesofpoplar(populus×beijingensis)plantcelltissueorganculture.2013,114(1):39-48.)的方法,通过花药诱导获得花药诱导的植株;
gm00、gm15、gm93、gm107植株是由毛白杨lm50花药诱导的部分植株,其中,gm107植株经流式细胞仪检测、染色体计数、分子标记鉴定后为单倍体,自然加倍成为双单倍体,即纯合的二倍体,其余为杂合的二倍体。
2、试验方法
(1)参照dna提取试剂盒(天根dp320-03)方法提取毛白杨lm50和花药诱导单倍体植株的叶片基因组dna,并将其作为pcr模板。
(2)基于毛白杨(lm50)基因组数据的信息,提取两套染色体间基因位置比较一致的共线性同源基因对,一共17209对;提取每对基因的基因区序列,含外显子和内含子,用muscle进行比对,检测indel,对大于100bp的indel(7404个基因对)设计引物,产物长度100-500bp,合成120对引物,通过pcr产物琼脂糖凝胶电泳进行检测,筛选出对应于毛白杨19条染色体的19对特异的indelpcr引物。进一步将所筛选出的19对引物合成荧光标记引物(荧光标记选用m13)进行毛细管电泳检测分析;所有引物均在上海生工生物工程股份有限公司合成。
(3)通过毛白杨lm50为模板分别对所设计的位于19条染色体的pcr引物进行筛选,本发明的筛选过程选用lataq酶,pcr反应体为20μl:2μl10×pcrbuffer,3.2μldntp,上、下游引物各0.4μl,2μldna模板,0.2μllataq酶,11.8μlddh2o;pcr反应程序:94℃预变性3min;94℃变性30s,60℃退火30s,72℃延伸90s,进行34个循环;72℃延伸5min,最后在4℃保存;然后用琼脂糖凝胶电泳对pcr结果进行检测;然后将筛选出的分别位于19条染色体上的19对引物对lm50和花药诱导的单倍体植株进行鉴定;进一步将筛选出的19对引物设计indel分子标记引物,indel分子标记检测的pcr反应体系为25μl:2μl10×pcrbuffer,3.2μldntp,上、下游引物各0.4μl,m13为5μl,2μldna模板,0.2μllataq酶,11.8μlddh2o;pcr反应程序:94℃预变性3min;94℃变性30s,60℃退火30s,72℃延伸1min,进行34个循环;72℃延伸5min,避光保存,并取10μl送至瑞博兴科生物技术有限公司进行毛细管电泳检测。
3、试验结果
(1)通过基因组信息,设计并筛选出的位于19条染色体的目的基因引物见表1,以lm50和花药诱导的单倍体叶片基因组dna为模板进行pcr扩增,采用1.7%琼脂糖凝胶电泳检测pcr产物扩增情况,pcr结果显示lm50均为两条带,而由花药诱导的单倍体gm107则为单一条带(图1)。
(2)将筛选出的19对引物设计其indel分子标记引物(表2),通过用毛细管电泳检测同上述pcr结果相符,进一步验证了这一结果(图2-11)。
表1位于毛白杨19条染色体的19对引物信息
表2位于毛白杨19条染色体的19对分子标记引物信息
(3)本发明所筛选的19对毛白杨目的基因引物(表1)扩增gm00、gm15、gm93植株产物见图12。通过pcr扩增检测发现gm00、gm15、gm93均为两条带,说明以上三种植株不是单倍体,属于杂合的二倍体。
(4)本发明通过基因组信息设计的位于毛白杨9条染色体的9对引物信息见表3。
表3位于毛白杨9条染色体的9对引物信息
以毛白杨lm50、gm107、gm00、gm15、gm93植株的基因组dna为模板进行pcr扩增,采用琼脂糖凝胶电泳检测pcr扩增情况。9对毛白杨目的基因引物扩增产物见图13。pcr结果显示,上述9对引物扩增毛白杨lm50、gm107、gm00、gm15、gm93植株产物均为两条带,说明该9对引物均不能鉴定由毛白杨lm50花药诱导的单倍体植株。
sequencelisting
<110>北京林业大学
<120>用于鉴定毛白杨单倍体或纯合二倍体的引物组及其应用
<130>bj-1006-181002a
<160>57
<170>patentinversion3.5
<210>1
<211>20
<212>dna
<213>artificalsequence
<400>1
tatggctttggcatccgtgt20
<210>2
<211>20
<212>dna
<213>artificalsequence
<400>2
gtgcaaacgacgaccattca20
<210>3
<211>20
<212>dna
<213>artificalsequence
<400>3
gcagcatggaaggttttgca20
<210>4
<211>20
<212>dna
<213>artificalsequence
<400>4
agcaacctagcttggtcaca20
<210>5
<211>18
<212>dna
<213>artificalsequence
<400>5
ggcgtgctccgtcttgtc18
<210>6
<211>20
<212>dna
<213>artificalsequence
<400>6
gtaggctagcatgtcgcagt20
<210>7
<211>20
<212>dna
<213>artificalsequence
<400>7
acgcatctttgtccctcctg20
<210>8
<211>20
<212>dna
<213>artificalsequence
<400>8
cacagttctcagggaggcag20
<210>9
<211>20
<212>dna
<213>artificalsequence
<400>9
caagcgttgaccctttgcac20
<210>10
<211>20
<212>dna
<213>artificalsequence
<400>10
acactgtgggcaaaagcaac20
<210>11
<211>20
<212>dna
<213>artificalsequence
<400>11
cgcagcgtgatgatttggag20
<210>12
<211>20
<212>dna
<213>artificalsequence
<400>12
tctcagctgcagtggacaag20
<210>13
<211>20
<212>dna
<213>artificalsequence
<400>13
gagctcggagtaggcaacaa20
<210>14
<211>20
<212>dna
<213>artificalsequence
<400>14
catgttgatggcagcaaccc20
<210>15
<211>20
<212>dna
<213>artificalsequence
<400>15
agggttggaggaaaggacct20
<210>16
<211>20
<212>dna
<213>artificalsequence
<400>16
cctacacagccagccaaaga20
<210>17
<211>21
<212>dna
<213>artificalsequence
<400>17
ggctaaggcttggtatgtcca21
<210>18
<211>21
<212>dna
<213>artificalsequence
<400>18
tccctcacaaggtgataccca21
<210>19
<211>20
<212>dna
<213>artificalsequence
<400>19
gctggcctattttgctctgc20
<210>20
<211>20
<212>dna
<213>artificalsequence
<400>20
acatgctctgctctgcgtta20
<210>21
<211>20
<212>dna
<213>artificalsequence
<400>21
gctgtggatggcaagcattt20
<210>22
<211>20
<212>dna
<213>artificalsequence
<400>22
gtgttccaccgatcatccca20
<210>23
<211>20
<212>dna
<213>artificalsequence
<400>23
ttcagaatggcctgcaaggt20
<210>24
<211>23
<212>dna
<213>artificalsequence
<400>24
agtcagctactccactctcttct23
<210>25
<211>20
<212>dna
<213>artificalsequence
<400>25
cgttggttgcaaggaagacg20
<210>26
<211>20
<212>dna
<213>artificalsequence
<400>26
agtaccgggaattgagctgc20
<210>27
<211>21
<212>dna
<213>artificalsequence
<400>27
tgcgttaggaatggatcgaga21
<210>28
<211>21
<212>dna
<213>artificalsequence
<400>28
ccctttttgccaagccaaaga21
<210>29
<211>20
<212>dna
<213>artificalsequence
<400>29
tcatgtgtccgactcttggc20
<210>30
<211>20
<212>dna
<213>artificalsequence
<400>30
caccacggctgtctctcaat20
<210>31
<211>20
<212>dna
<213>artificalsequence
<400>31
gtgcctctgatgcctctgtt20
<210>32
<211>20
<212>dna
<213>artificalsequence
<400>32
gctcctcatcgttgaaccca20
<210>33
<211>20
<212>dna
<213>artificalsequence
<400>33
tctgtttgtgctttgctgca20
<210>34
<211>20
<212>dna
<213>artificalsequence
<400>34
atgttgcgccacttaaggga20
<210>35
<211>20
<212>dna
<213>artificalsequence
<400>35
agaggggtgggtgattgtct20
<210>36
<211>20
<212>dna
<213>artificalsequence
<400>36
ttggatcagcaacccttccc20
<210>37
<211>20
<212>dna
<213>artificalsequence
<400>37
aatctgcgttgacgtgcaag20
<210>38
<211>20
<212>dna
<213>artificalsequence
<400>38
gggcacaagtttgaagctgg20
<210>39
<211>38
<212>dna
<213>artificalsequence
<400>39
tgtaaaacgacggccagttatggctttggcatccgtgt38
<210>40
<211>38
<212>dna
<213>artificalsequence
<400>40
tgtaaaacgacggccagtgcagcatggaaggttttgca38
<210>41
<211>36
<212>dna
<213>artificalsequence
<400>41
tgtaaaacgacggccagtggcgtgctccgtcttgtc36
<210>42
<211>38
<212>dna
<213>artificalsequence
<400>42
tgtaaaacgacggccagtacgcatctttgtccctcctg38
<210>43
<211>38
<212>dna
<213>artificalsequence
<400>43
tgtaaaacgacggccagtcaagcgttgaccctttgcac38
<210>44
<211>38
<212>dna
<213>artificalsequence
<400>44
tgtaaaacgacggccagtcgcagcgtgatgatttggag38
<210>45
<211>38
<212>dna
<213>artificalsequence
<400>45
tgtaaaacgacggccagtgagctcggagtaggcaacaa38
<210>46
<211>38
<212>dna
<213>artificalsequence
<400>46
tgtaaaacgacggccagtagggttggaggaaaggacct38
<210>47
<211>39
<212>dna
<213>artificalsequence
<400>47
tgtaaaacgacggccagtggctaaggcttggtatgtcca39
<210>48
<211>38
<212>dna
<213>artificalsequence
<400>48
tgtaaaacgacggccagtgctggcctattttgctctgc38
<210>49
<211>38
<212>dna
<213>artificalsequence
<400>49
tgtaaaacgacggccagtgctgtggatggcaagcattt38
<210>50
<211>38
<212>dna
<213>artificalsequence
<400>50
tgtaaaacgacggccagtttcagaatggcctgcaaggt38
<210>51
<211>38
<212>dna
<213>artificalsequence
<400>51
tgtaaaacgacggccagtcgttggttgcaaggaagacg38
<210>52
<211>39
<212>dna
<213>artificalsequence
<400>52
tgtaaaacgacggccagttgcgttaggaatggatcgaga39
<210>53
<211>38
<212>dna
<213>artificalsequence
<400>53
tgtaaaacgacggccagttcatgtgtccgactcttggc38
<210>54
<211>38
<212>dna
<213>artificalsequence
<400>54
tgtaaaacgacggccagtgtgcctctgatgcctctgtt38
<210>55
<211>38
<212>dna
<213>artificalsequence
<400>55
tgtaaaacgacggccagttctgtttgtgctttgctgca38
<210>56
<211>38
<212>dna
<213>artificalsequence
<400>56
tgtaaaacgacggccagtagaggggtgggtgattgtct38
<210>57
<211>38
<212>dna
<213>artificalsequence
<400>57
tgtaaaacgacggccagtaatctgcgttgacgtgcaag38
- 还没有人留言评论。精彩留言会获得点赞!