核酸文库、肽文库及它们的用途的制作方法
描述
本发明涉及编码多于一种肽的核酸文库,所述肽代表天然存在的蛋白质的片段。每种肽可以选自物种或生物体的蛋白质组,并且多于一种肽可以共同代表物种或生物体的蛋白质组,或者此类肽可以选自一个以上的蛋白质组,并且因此多于一种肽可以共同代表宏蛋白质组。肽也可以选自在细胞或组织类型之间差异表达的蛋白质。本发明还涉及此类肽的文库,涉及牵涉核酸和/或肽的此类文库的方法,和/或涉及包含与此类文库相关的信息的计算机可读介质或数据处理系统。
鉴定新的治疗靶是药物发现的关键起点。药物发现工作传统上集中于鉴定经典的可成药靶(druggabletarget),例如激酶、g蛋白偶联受体(gpcr)和离子通道。然而,此类化学上容易获得的靶并不总是代表生物学上最重要的用于治疗干预的靶。将蛋白质:蛋白质相互作用(ppi)成药(drugging)特别令人感兴趣,因为它们代表了参与癌细胞利用的缺陷信号传导途径中的主要靶类型,以及人类疾病中一大组潜在可作用的接口。不幸的是,对将ppi和其他“不可成药”的靶成药的系统尝试受到技术的限制,这在很大程度上是由于当前基于高通量dna和rna的基因组学技术对于能够在蛋白质组水平上鉴定新的可成药空间的限制。
可以使用无偏的“表型”测定来鉴定与疾病生物学相关的候选药物靶的目前的基于基因组学的技术,通常使用基因敲除(例如crispr)来执行,或者在转录物组水平上使用rnai来执行。这些方法产生了关于哪些靶可以代表疾病进展和疾病治疗干预中的重要节点的重要信息,但是受到严重限制:因为它们是在基因水平而不是蛋白质水平上进行筛选,所以它们无法确定如何将那些靶成药,也无法作为过程中固有的一部分来确定那些靶是否代表可成药的候选物。这是因为这样的基因筛选去除了靶蛋白而不是抑制它们。为了获得此类重要的关于可成药性的另外信息,将需要使用新的高通量蛋白质组水平筛选技术;一种可以处理比基因功能(~30,000个基因及其剪接变体)更高复杂度的筛选蛋白质功能(>300,000个独特的蛋白质转录本和数百万个独特的ppi)的筛选技术。
最近,随着dna编码的、蛋白片段表达文库的引入,直接在人类蛋白质组中系统鉴定新的药物靶位点获得了一定程度的可操作性和关注,这些文库可以在表型测定中以高通量筛选(诸如wo2013/116903中所描述的);常被称为“蛋白质干扰”(protein-i)。此类蛋白质片段文库(通常来源于不同的细菌基因组),由形成更大蛋白质的进化构建模块的小的自折叠子结构域组成。当组装为用于哺乳动物细胞中细胞内表达的文库时,它们代表用于对接靶蛋白和探索跨越人蛋白质组的候选的新的可成药位点的三维形状的高度多样化集合。至关重要的是,这些蛋白质片段小到足以描述靶蛋白质中离散的空间位点(discretespatialsites),并且因此可以用随后设计成与该形状匹配的小分子药物重现。此外,由于蛋白质片段文库描述了比目前的小分子文库多得多的形状,这为指导对于新的经验证的靶的未来小分子药物的合理设计提供了更加可靠的方法。
尽管由于细菌基因组主要由编码序列组成,细菌衍生的蛋白质片段文库已显示在protein-i筛选中是成功的,并且通过片段化和克隆到表达文库中而直接地产生,然而,与使用哺乳动物或人类蛋白质组本身的片段相比,它们可能在拥有大比例的能与哺乳动物(例如人类)蛋白功能性地相互作用的蛋白片段方面力不从心。
然而,直接从哺乳动物(例如人类)的基因组创建蛋白质片段文库的复杂性在于,高等生物的dna主要含有非编码序列(估计>95%的人类dna是非编码的)以及绝对数量要大得多的编码序列,并且因此通常需要较大程度的人工定制克隆来将其片段组装为用于表型筛选的表达文库。
迄今为止描述的那些细菌衍生的蛋白质片段文库(例如在w02013/116903中)是通过机械剪切(mechanicallyshearing)基因组并将片段随机插入载体中获得的。这产生对于细菌中的原始基因为符合读框(1:6的机会)或不符合读框(5:6的机会)的许多随机大小的片段。对于真核生物来说,同样的策略是行不通的,因为它们的dna大部分是非编码的。此外,细菌衍生的蛋白质片段文库诸如这些没有“清单”,即,因为序列是随机克隆的,所以除了通过非常深度的测序之外,不可能准确说出给定文库中包含的序列。
这些实际的限制导致了在人类细胞的靶鉴定和验证筛选中,挖掘直接相关蛋白质折叠结构多样性的潜在丰富的替代矿脉时存在显著的惰性。
其他筛选方法描述于例如wo2001/86297中。这里产生随机短肽(40-mer和20-mer)噬菌体展示文库,并将该文库用于寻找结合到预先选择的靶或已知的、预先鉴定的共有基序的肽。这依赖于已知/已识别的现有的疾病靶,并且不协助鉴定新的靶。wo2007/097923公开了肽结构的文库和产生此类文库的方法,所述肽结构代表了自然界中存在的全部蛋白质结构。然而,选择此类文库以包含那些不依赖于人工支架或它们所来源的蛋白质中的侧翼序列就能够折叠或呈现其天然构象的肽。
wo2010/129310描述了编码来自蛋白质的肽的核酸文库,所述蛋白质包含完整的天然蛋白质组(或已知的生物活性肽),所述蛋白质在每种情况下被表达并分泌到细胞外。其中描述了使用这样的文库来分离生物活性分泌肽(“basp”),以及如何从高通量寡核苷酸合成开始构建这样的文库,但未公开合成的序列或在这样的文库中编码的肽。事实上,其中很少描述关于所编码的肽的氨基酸序列或其他特定(例如有利)特征或编码此类肽的核酸的序列或其他特定(例如有利)特征的信息,也没有描述关于选择此类肽以包含在(或排除于)此类文库中的方法,或选择为此类文库合成的核酸的设计(和特征,例如序列的特征)的信息。在该技术的相应科学出版物(natarajan等人,2014;pnas111:e474)中提供了很少的关于文库设计的此类重要事项(例如计算机模拟构建)的信息。
还存在若干已知的噬菌体展示文库。wo2015/095355涉及检测针对病原体的抗体。它描述了包含病毒蛋白质序列的噬菌体展示文库。一篇相关论文:xu等人,2015;science348描述了virscan技术,并且据称它将dna微阵列合成和噬菌体展示相结合,创建了组成人类病毒组(virome)的肽表位的统一、合成展示。同一研究组的更早出版物,larman等人,2011,natbiotechnol29:535描述了类似的方法,但涉及t7“肽组(peptidome)”噬菌体展示文库,该文库包含来自人类基因组的肽(即,来自人类基因组的大约24,000个独特orf的36个氨基酸的肽)。
因此,本发明的一个目的是提供编码蛋白质片段/肽的文库,其中此类文库可用于筛选方法,包括但不限于ppi筛选。在其他目的中,本发明提供了解决这些或其他问题中的一个或更多个的替代的、改进的、更简单的、更便宜的和/或集成的手段或方法。本发明的一个目的通过本文任何地方公开或定义的主题,例如通过所附权利要求的主题来解决。
附图显示了:
图1:描绘筛选在hupex文库中表达的能够克服6-硫鸟嘌呤毒性的sep。将携带表达sep的插入物的文库的细胞用500nm6-硫鸟嘌呤处理6天。显示了6-硫鸟嘌呤处理(n=3)和dmso对照(n=3)之间的富集。
图2:描绘在所有三个文库(hupex、bugpex、omepex)中筛选能够选择性地杀伤缺乏pten肿瘤抑制基因的细胞的sep。
图3:描绘与空载体和阳性对照(针对nlk的shrna)相比,从实施例4(2)中描述的筛选鉴定的由pmost25表达的肽(7-924)对mcf10aptenko细胞的作用。
图4:描绘实施例9和实施例10的mnng诱导的parthanatos表型筛选的实验原理。
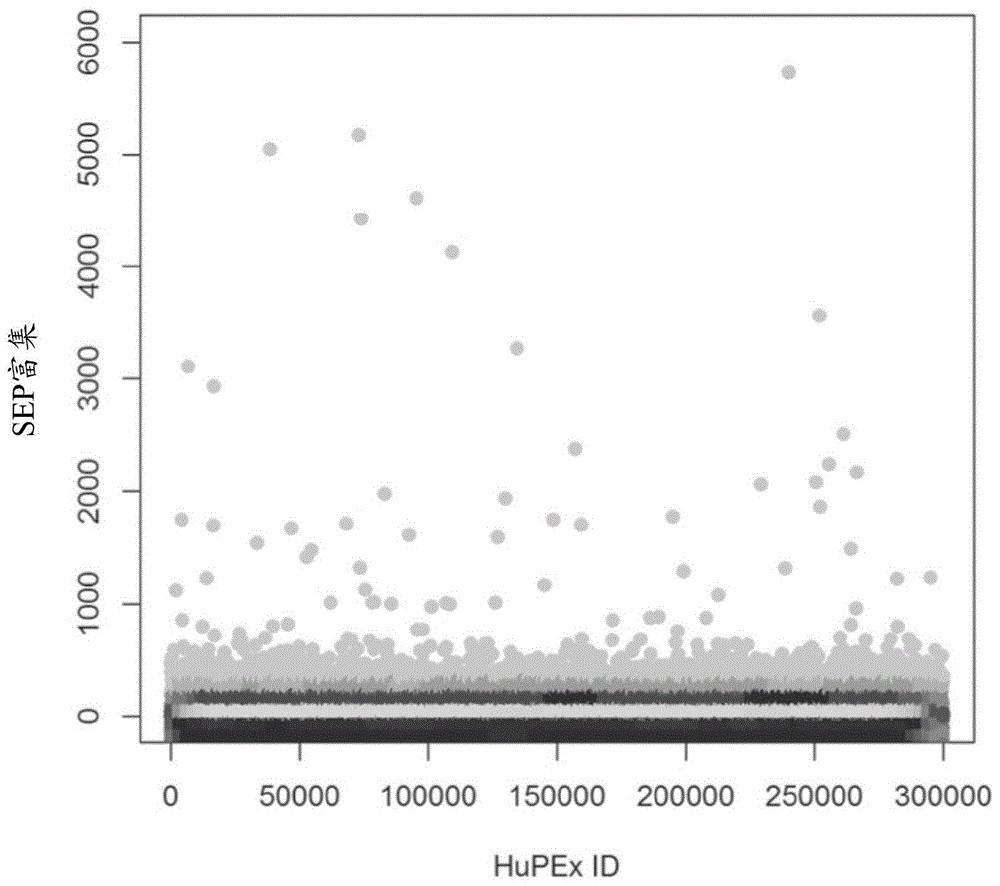
图5:描绘在用6.7ummnng处理之前(d0)在表达hupex的hela细胞的对照等分试样中,以及在此类mnng处理8天之后(d8)在此类表达hupex的hela细胞的处理等分试样中,存在的编码来自hupex文库的sep的dna序列的相对丰度。在d8显示相对丰度显著增加的肽用三角形标记。
图6:描绘在用6.7ummnng处理之前(d0)在表达bugpex的hela细胞的对照等分试样中,以及在此类mnng处理8天之后(d8)在此类表达bugpex的hela细胞的处理等分试样中,存在的编码来自bugpex文库的sep的dna序列的相对丰度。轴如图5所示,并且在d8显示相对丰度显著增加的肽用三角形标记。
图7:描绘在用6.7ummnng处理之前(d0)在表达omepex的hela细胞的对照等分试样中,以及在此类mnng处理8天之后(d8)在此类表达omepex的hela细胞的处理等分试样中,存在的编码来自omepex文库的sep的dna序列的相对丰度。轴如图5所示,并且在d8显示相对丰度显著增加的肽用三角形标记。
图8和图9:用于自噬诱导的pex文库的表型筛选
图8(a-c):将hek293ft细胞工程化成稳定表达gfp-lc3/rfp-lc3dg自噬报告基因(kaizuka等人molecularcell2016)。随后,将自噬报告细胞用汇集的hupex(hpx)、bugpex(bpx)和omepex(opx)文库感染,并在嘌呤霉素上选择。选择后,与未分选的对照相比,在低gfp-lc3门中富集的细胞被流式分选,并且肽序列被扩增并送至ngs分析,如前所述。图(图8a(hpx)、图8b(bpx)和图8c(opx))显示与对照相比,标记区域中选定命中(hit)(自噬诱导物)的群体。
图9:在用携带对照序列或推定的命中的慢病毒感染后,在流式细胞术实验中单独重新运行命中。显示了一系列候选物,其中bpx-497507代表强有力的命中,能够诱导自噬,如通过gfp-lc3的减少来测量的。torin1(250nm)显示为阳性对照。
本发明及其特定的非限制性方面和/或实施方案可以更详细地描述如下:
在第一个方面,本发明提供了一种核酸文库,每种核酸包含编码肽的确定的核酸序列的编码区,所述肽具有25和110个氨基酸之间的长度并且具有为选自一种或更多种生物体的天然存在的蛋白质的氨基酸序列中的序列区域的氨基酸序列;其中该文库包含编码至少约10,000种(或5,000种)不同的此类肽中的多于一种肽的核酸,并且其中至少50种(或至少25种)此类肽中每一种肽的氨基酸序列是多于一种不同的此类天然存在的蛋白质中的不同蛋白质的氨基酸序列中的序列区域(或者,为了清楚起见,其中对于所述不同的天然存在的蛋白质中的每一种蛋白质,该文库包含一种或更多种编码具有为此类天然存在的蛋白质的氨基酸序列中的序列区域的氨基酸序列的肽的核酸)。
合适地,根据本发明的任何方面或实施方案的文库包含编码至少约20,000、50,000、100,000、200,000、250,000、300,000、475,000或500,000种不同的此类肽中的多于一种肽的核酸。该文库还可以包含编码超过300,000或500,000种不同的此类肽的核酸。例如,在某些实施方案中,文库可以包含编码50,000种不同的此类肽中的多于一种肽的核酸,并且其中至少100种此类肽中的每一种肽的氨基酸序列是至少100种不同的天然存在的蛋白质的氨基酸序列中的序列区域(或者,为了清楚起见,其中对于至少100,000种不同的天然存在的蛋白质中的每一种蛋白质,该文库包含一种或更多种编码肽的核酸,该肽具有为此类天然存在的蛋白质的氨基酸序列中的序列区域的氨基酸序列);特别地,在这样的实施方案中,该文库可以包含编码至少100,000种不同的此类肽中的多于一种肽的核酸,并且其中至少150种此类肽中的每一种肽的氨基酸序列是至少150种不同的天然存在的蛋白质的氨基酸序列中的序列区域(或者其中,对于至少150种不同的天然存在的蛋白质中的每一种蛋白质,该文库包含一种或更多种编码肽的核酸,该肽具有为此类天然存在的蛋白质的氨基酸序列中的序列区域的氨基酸序列)。在另一种实施方案中,文库可以包含编码至少10,000种不同的此类肽中的多于一种肽的核酸,并且其中至少1,000种此类肽的每一种肽的氨基酸序列是此类多于一种不同的天然存在的蛋白质的不同蛋白质的氨基酸序列中的序列区域。
在一种实施方案中,文库可以包含编码至少200,000种不同的此类肽中的多于一种肽的核酸,并且其中至少20,000种此类肽的每一种肽的氨基酸序列是至少20,000种不同的天然存在的蛋白质的氨基酸序列中的序列区域;特别地,在这样的实施方案中,文库可以包含编码至少300,000种不同的此类肽中的多于一种肽的核酸,并且其中至少25,000种此类肽的每一种肽的氨基酸序列是至少25,000种不同的天然存在的蛋白质的氨基酸序列中的序列区域。在一种实施方案中,混合物中存在的核酸的量与感兴趣的生物体的基因组或转录组的复杂度和大小成正比。在其他实施方案中,根据本发明的文库中不同核酸的数目可取决于所需的筛选应用和在特定应用中可行的序列数目。例如,这可包括考虑文库是用作主要筛选还是次要筛选。
如本文使用的,在由本发明的文库中的核酸编码的肽的上下文中,术语“不同”是指任何一种肽与文库中编码的任何其他肽相比具有至少一个氨基酸差异。换句话说,文库中的每种核酸编码一种独特的肽。
在一种实施方案中,“天然存在的蛋白质”是具有在参考蛋白质组中发现的序列的蛋白质。本文描述了参考蛋白质组以及如何使用来自参考蛋白质组的信息的实例。如本文使用的,在天然存在的蛋白质的上下文中,术语“不同”是指任何一种这样的蛋白质与任何其他这样的蛋白质相比具有至少一个氨基酸差异。换句话说,文库中的每种核酸编码一种独特的肽。合适地,“不同的”天然存在的蛋白质具有小于约98%、95%或92%序列同一性,诸如小于约95%或90%序列同一性的氨基酸序列同一性。在一种合适的实施方案中,“不同的”天然存在的蛋白质具有数据库的不同条目编号(或其他标识符),诸如具有不同的uniprot标识符。例如,在这样的实施方案中,具有uniprot(www.uniprot.org)标识符p24941和p11802(分别为人类cdk2和cdk4)的细胞周期蛋白依赖性激酶是“不同的”天然存在的蛋白质。
有利地,每种核酸包含编码肽的确定的(或已知的)核酸序列的编码区。“确定的”(或“已知的”)核酸序列是指,文库中基本上所有(例如每种)核酸序列的序列是确定的(或已知的)。特别地,文库是非随机的,即它不代表随机基因组序列(其可以表达或不表达肽序列)的集合,即使基因组序列作为一个整体可能已经被确定(例如已知),但是该文库已经被设计(从蛋白质序列开始)并且任选地被过滤以生成编码具有特定预测特征、特别地具有特定和确定的氨基酸序列的肽的核酸子集。因此,有利地,文库中基本上所有(例如每种)序列的身份将是确定的(或已知的),使得根据本发明的文库可以具有清单(即,例如,包括确定的(或已知的)序列的单个成员的预先指定或预先设计的集合或由确定的(或已知的)序列的单个成员的预先指定或预先设计的集合组成),即使可能不知道哪个特定序列在文库的哪个特定成员中。这允许易于鉴定其中的序列。如本文所述,此类文库可被设计成具有所需的复杂度和/或过滤掉不需要的序列。
在一些实施方案中,文库中的核酸是合成的(例如,它们至少最初是通过化学过程而不是生物过程生成的)。因此,合适地,文库提供合成的或非天然的核酸(和/或包含非天然的核酸序列)。合适地,此类核酸根据本文所述的任何一种方法设计并根据本领域技术人员可获得的方法,特别地包括本文别处所述的那些方法的高体积/高通量方法合成。重要的是,此类合成的核酸包含使它们区别于天然存在的核酸的设计特征。这样的设计特征包括,例如,使用密码子频率表来生成核酸,使得在编码肽的核酸中构成密码子的核苷酸序列不代表在天然存在的蛋白质的氨基酸序列中的那个位置发现的那些密码子。此外,用于根据本发明的文库的核酸可以包含天然存在的核酸序列中不存在的限制位点,并且将生成包含天然存在的蛋白质序列中不存在的另外的氨基酸的肽序列。合适地,用于根据本发明的文库中的核酸(例如,其序列)可以使用本文所述方法中阐述的设计原则生成。
在根据本发明的文库的一种实施方案中,该文库提供了编码源自任何一种蛋白质的多于一种肽的多于一种核酸序列。也就是说,就每种不同的天然存在的蛋白质而言,该文库包含编码具有是此类天然存在的蛋白质的氨基酸序列中的序列区域的氨基酸序列的肽的多于一种(即一种以上)核酸。因此,以此类实施方案的这种含义,本发明的第一方面可以可选地被陈述为(例如,为了清楚起见,如上所述)涉及核酸文库,每种核酸包含编码肽的确定的(或已知的)核酸序列的编码区,所述肽具有在25和110个氨基酸之间的长度,并且具有是选自一种或更多种生物体的天然存在的蛋白质的氨基酸序列中的序列区域的氨基酸序列;其中该文库包含编码至少10,000种不同的此类肽中的多于一种肽的核酸,并且其中对于每种不同的天然存在的蛋白质,该文库包含一种或更多种编码具有是此类天然存在的蛋白质的氨基酸序列中的序列区域的氨基酸序列的肽的核酸。
例如,在一种此类实施方案中,就至少约1%的天然存在的蛋白质而言,多于一种核酸编码来自此类天然存在的蛋白质的氨基酸序列的不同肽。合适地,就至少约5%、10%、25%或50%的天然存在的蛋白质而言,多于一种核酸编码来自此类天然存在的蛋白质的氨基酸序列的不同肽。在其他实施方案中,多于一种核酸编码来自至少约30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%或100%的天然存在的蛋白质的不同肽。在特定实施方案中,多于一种核酸编码来自约90%和100%之间的此类天然存在的蛋白质的氨基酸序列的不同肽。合适地,“不同肽”是具有不同氨基酸,诸如相差一个或更多个(例如,在约2个和10个之间、5个和20个之间、15个和40个之间或30个和50个之间或50个以上)氨基酸的肽。
在另一种实施方案中,本发明提供了核酸(例如合成核酸)文库,其中多于一种核酸编码不同的肽,其氨基酸序列是沿着天然存在的蛋白质的氨基酸序列间隔的序列区域。适当地,该间隔被选择为生成编码可行数目的肽的文库,并且可以根据文库所代表的肽的期望数目而变化。“可行数目”是指在提供用于所选的筛选应用或方法中的合适数目的同时,能够经济地生成的合适数目。例如,对于初级的基于细胞的选择筛选,“可行数目”可以比用于二级筛选或基于阵列或下拉筛选的数目(使用例如,数万至约250,000)更大(例如,300,000、500,000或甚至数百万)。合适地,本发明的核酸表达文库可以合适地以约250,000和500,000个之间的不同肽(诸如约300,000个)的复杂度使用;并且由其表达的肽文库可以用于复杂度大于500,000,诸如大于750,000、1,000,000、1,500,000或2,000,000(或更大)的固相筛选。在其他实施方案中,“可行数目”可以小于肽的此类数目(诸如,在针对下文描述的天然存在的蛋白质的“聚焦”部分的那些实施方案中)。合适地,在这样的实施方案中,本发明的核酸表达文库可以以约5,000和250,000个之间的不同肽(诸如至少约10,000个)的复杂度使用,例如约10,000和25,000个之间、20,000和50,000个之间、40,000和100,000个之间或100,000和200,000个之间的不同肽。
因此,在一种实施方案中,序列区域沿着天然存在的蛋白质的氨基酸序列被一个氨基酸窗口或多于一个此类窗口分隔开,其中窗口在1、5、10、15、20、25、30、35、40或45个氨基酸和约55个氨基酸之间;特别地,其中窗口在约2和40个氨基酸之间,更特别地,其中窗口在约5和约20个氨基酸之间;最特别地,其中间隔窗口是约8、10、12或15个氨基酸。在合适的实施方案中,在沿着天然存在的蛋白质(并且任选地,对于用于形成由文库的核酸编码的肽序列的其他天然存在的蛋白质中的每一个)的氨基酸序列的每个序列区域之间,用于分隔序列区域的窗口是相同数目的氨基酸(或其倍数)。在另一种实施方案中,文库可以包含由多于一个这样的窗口分隔开的序列区域,因为一个或更多个间插肽/氨基酸序列可能由于不符合一个或更多个不同的过滤标准而从选择过程中去除。
合适地,文库被设计成复杂文库。“复杂文库(complexlibrary)”是指其中代表了大量不同蛋白质和肽的文库,特别地包含序列(或结构上)不同的肽的文库,诸如由演化的不同物种定义的那些。因此,“复杂度”是根据蛋白质和肽的数目来考虑的。有利的是,本发明的文库提供了比现有技术中可用的那些文库高得多的复杂度。在一种实施方案中,文库包含编码来自至少5,000种不同蛋白质的至少5,000种不同肽的(合成)核酸。在另一种实施方案中,根据本发明的(合成)核酸的文库包含编码来自至少10,000(或分别来自2,000或5,000)种不同的天然存在的蛋白质的至少100,000(或20,000或50,000)种不同肽的核酸。应当理解,由根据本发明的文库编码的不同肽的数目可以是由至少50、75、100或15010,000种不同的天然存在的蛋白质编码的超过至少10,000、20,000、50,000或100,000种不同肽;例如,文库的大小可以超过至少50,000、100,000或250,000种肽。不同肽的数目和它们所来源的不同的天然存在蛋白质的数目可以根据具体应用而变化。因此,并且如上所述,文库可以编码至少大约5,000、10,000、50,000、100,000、200,000、250,000、300,000、475,000或500,000种不同的此类肽,超过300,000或500,000种不同的此类肽(或者,例如,适合于聚焦的文库,超过10,000或50,000种不同的此类肽);合适地,其中平均两种或更多种(诸如约5、8、10或15种)肽来源于相同的天然存在的蛋白质的氨基酸序列。
考虑到本发明文库的某些实施方案的复杂度——例如,编码(或包含)来自至少1,000或10,000(或分别20,000或25,000种)不同的天然存在的蛋白质的至少10,000(或200,000或300,000)种肽的那些——例如,在这些实施方案中,编码的肽通常代表来源于不同的天然存在的蛋白质的不同选集的那些。实际上,典型地,不同的天然存在的蛋白质的不同选集将(例如还,或任选地仅)包含为非分泌性蛋白质和/或非胞外蛋白质的蛋白质,包括来自多于一个物种的那些蛋白质。例如,不同的天然存在的蛋白质可以包括(例如还,或任选地仅)一组蛋白质,该组蛋白质包括除了(例如,人类和小鼠)细胞因子、趋化因子、生长因子及其受体之外的其他蛋白质(任选地,这些其他蛋白质以及这些细胞因子、趋化因子、生长因子及其受体)。在特定的实施方案中,不同的天然存在的蛋白质可以包含一组蛋白质,所述一组蛋白质包括为细胞质蛋白质的蛋白质(任选地,细胞质蛋白质以及包括非细胞质蛋白质,诸如还包括分泌蛋白质和/或细胞外蛋白质)。
在另外的或替代的实施方案中,本发明的文库编码(或包含)不是(先前)已知的——例如,不是预先选择的——生物活性肽,和/或推测可以通过与细胞表面受体相互作用来调节细胞响应的肽。
在本发明文库的其他实施方案中——例如,编码(或包含)至少50,000(或10,000或100,000)种来自至少100种(或分别50或150种)不同的天然存在的蛋白质的肽的那些,——例如,在这些实施方案中,编码的肽通常代表来源于不同的天然存在的蛋白质的聚焦选集的那些。在这样的实施方案中,不同的天然存在的蛋白质可以基于一个或更多个(例如预定的)标准从一组更大的天然存在的蛋白质(诸如来自一个或更多个参考蛋白质组的那些蛋白质)预先选择或者是其子集。特别地,在这样的实施方案中,所有不同的天然存在的蛋白质可以满足这样的标准,并且这样的文库的每一个(例如,所有的)(编码的)肽旨在具有是满足这样的(例如,预定的)标准的天然存在的蛋白质的氨基酸序列中的序列区域的序列。此类标准的一个非限制性实例可以是,天然存在的蛋白质是分泌蛋白质和/或细胞外蛋白质,诸如(例如所有的)天然存在的蛋白质可以是细胞因子、趋化因子、生长因子及其受体。可选地,这样的标准可以包括,天然存在的蛋白质不是分泌蛋白质和/或不是细胞外蛋白质,诸如(例如所有的)天然存在的蛋白质可以不是细胞因子、趋化因子、生长因子及其受体。
在替代实施方案中,由文库编码(或包含在文库中)的肽所来源的(例如,每种和/或所有)天然存在的蛋白质的标准可以包括以下标准中的一种或更多种或由以下标准中的一种或更多种组成;这些蛋白质是:
·来自亚细胞区室,例如细胞质、细胞核、线粒体、细胞骨架或核糖体
·一种或更多种给定的酶类。例如,激酶、蛋白酶、酯酶或磷酸酶;
·一种或更多种给定受体类型。例如,g-偶联蛋白受体或核激素受体;
·膜转运蛋白和/或离子通道蛋白;
·结构蛋白;
·转录因子或dna结合蛋白;
·dna修复蛋白。例如,错配修复途径的蛋白质;
·参与一种或更多种(例如相关或内在相关的)细胞信号传导/信号转导途径。例如,mapk/erk途径、pi3k/akt信号传导、erbb/her信号传导、mtor信号传导、nf-κb信号传导或jak/stat(il-6)受体信号传导;
·与给定蛋白质或一类蛋白质(例如功能性蛋白质)中的至少一种蛋白质相互作用(例如在体内,或由实验室程序诸如酵母双杂交或亲和纯化/质谱法确定)。例如,与kras或与激酶(例如,abl、bcl-abl、src、kit、plk、cdk、plk、aurora、mapk、jak、flt或egfr)相互作用;
·与给定疾病(例如来自全基因组关联研究gwas的命中),诸如癌症相关。例如,如brca1和/或brca2与乳腺癌相关,或如jak2与骨髓增生性肿瘤相关(stadler等人2010,jclinoncol28:4255);
·来自功能筛选,例如crispr、rnai、基因捕获、诱变、cdna筛选、proteini筛选的命中。
在另一种实施方案中,本发明提供了根据本发明的(例如合成的)核酸文库,其中每种核酸编码不同的肽。合适地,编码来自天然存在的蛋白质的(例如不同的)肽的核酸的平均数目大于1;特别地每种这样的蛋白质约1.01和1.5个之间这样的核酸(肽)(诸如约1.02、1.05、1.1、1.2、1.3或1.4),或每种这样的蛋白质至少约10个(或2或5个)核酸(肽),特别地其中平均值是每种这样的蛋白质约5和约2,000个之间的这样的核酸(肽),或是每种这样的蛋白质约5和约1,000个之间的这样的核酸(肽)(或每种这样的蛋白质约100和约1,500个之间、或约250和约1,000个之间、特别地约5和约100之间或约5和约50之间的这样的核酸(肽))。在一些实施方案中,核酸的平均数目可以高达至少500个核酸,尽管可以理解,编码任何特定蛋白质的不同肽的核酸的数目将取决于蛋白质的大小以及氨基酸序列间隔的窗口的大小。在相关的实施方案中,本发明提供了根据本发明的(例如合成的)核酸文库,其中所述文库中代表的天然存在的蛋白质的95%由约2和约20种之间(诸如约3和约35种之间或40种)肽序列表示。
在另一种实施方案中,提供了根据本发明的(例如合成的)核酸文库,其中天然存在的蛋白质的氨基酸序列是选自参考蛋白质组中包含的蛋白质的氨基酸序列的组的氨基酸序列;特别地其中天然存在的蛋白质的氨基酸序列是选自参考蛋白质组中包含的(例如,非冗余的)蛋白质的氨基酸序列的组的氨基酸序列。
在另一种实施方案中,参考蛋白质组是选自本文表a和/或表b的任何一个中列出的参考蛋白质组的组的一个或更多个参考蛋白质组,或其中列出的蛋白质组的任何更新版本。
在另一种实施方案中,多于一种编码肽的氨基酸序列是选自具有已知三维结构的天然存在的蛋白质(或其多肽链或结构域)的氨基酸序列中的序列区域;特别地其中天然存在的蛋白质(或其多肽链或结构域)被包含在蛋白质数据库(https://www.wwpdb.org)中,并且任选地具有pfam注释(http://pfam.xfam.org)。在另一种实施方案中,选自蛋白质的氨基酸序列中的序列区域不包括包含在参考蛋白质组或蛋白质数据库中的此类氨基酸序列中的不明确氨基酸。
合适地,从其选择天然存在的蛋白质的氨基酸序列来生成根据本发明的(合成)核酸文库的生物体或物种是智人(homosapiens)。
在另一种实施方案中,用于生成根据本发明的(例如合成的)核酸文库的不同的天然存在的蛋白质是多于一种不同生物体或物种的天然存在的蛋白质。合适地,多于一种不同的物种选自表a中所列的生物体(微生物)物种的组;特别地,其中多于一种不同的物种包括至少2种、3种或5种生物体(微生物)物种(诸如至少约10种、20种、25种或50种),跨越表a中所列的门的至少2种(诸如来自至少约3种或5种)。
在另一种实施方案中,多于一种不同的物种选自表b中所列的物种的组,特别地其中多于一种不同的物种包括表b中所列的至少2、3、5或10种(诸如至少约20、50、100、200、300或超过400种)物种,跨越本文描述的表的至少约3、5或5个部分(特别地跨越至少5或6个部分),诸如:古细菌、细菌、真菌、无脊椎动物、植物、原生动物、哺乳动物和非哺乳类脊椎动物。
合适地,多于一种不同的生物体或物种是至少10种、20种、50种、100种(特别地)或250种不同的生物体或物种。在一些实施方案中,多于一种不同的生物体或物种可包括多达约20、50、100、250或500种不同的生物体或物种。因此,可以实现文库内的高度多样性。
在另一种实施方案中,用于生成根据本发明的(例如合成的)核酸文库的不同的天然存在的蛋白质是在两种或更多种不同细胞群体(例如细胞类型)或组织类型之间差异表达的天然存在的蛋白质。例如,不同的天然存在的蛋白质可以在患病和正常的细胞或组织类型之间差异表达;特别地其中不同的天然存在的蛋白质在例如人类癌细胞和非癌人类细胞之间差异表达。在另外的实施方案中,不同的天然存在的蛋白质是疾病特异性的;特别地其中不同的天然存在的蛋白质由人类癌细胞表达,但不由非癌人类细胞表达。在另一种实施方案中,不同的天然存在的蛋白质可以在已被病原体感染或用物质(诸如致病物质或药物)处理的一个细胞群体和未被如此处理(或不同地处理)的相应细胞类型的第二细胞群体之间差异表达。例如,可以通过用促炎物质诸如肉豆蔻酸佛波酯(pma)处理在细胞系中诱导炎性表型,并且考虑在此类处理的细胞群体中与未处理的细胞相比差异表达的天然存在的蛋白质。在另一个实例中,可以通过比较已经变得衰竭的免疫细胞(t细胞衰竭)与成熟的免疫细胞(例如t细胞)之间的蛋白质表达来鉴定差异表达的天然存在的蛋白质。
在另一种实施方案中,根据本发明的(例如合成的)核酸文库是其中多于一种编码肽具有不同序列的文库;特别地其中多于一种编码肽的氨基酸序列彼此相差至少2或3个氨基酸;特别地其中多于一种编码肽的氨基酸序列彼此相差至少约5、8或10个氨基酸的文库。合适地,(合成的)核酸文库是这样的文库,其中多于一种编码肽的多样性具有小于约90%或80%的序列相似性;特别地具有小于约70%、60%或50%的序列相似性。此类序列相似性可以用以cd-hit的聚类分析来确定(fu等人2012,bioinformatics28:3150;http://weizhongli-lab.org/cd-hit/)。本文的实施例部分描述了用于此的合适的参数。
在另一种实施方案中,由根据本发明的文库编码的肽被预测不包含无序区域;特别地其中预测由slider(具有长的内在无序区域的蛋白质的超快速预测物(super-fastpredictorofproteinswithlongintrinsicallydisorderedregions);peng等人,2014;proteins:structure,functionandbioinformatics82:145;http://biomine.cs.vcu.edu/servers/slider)和/或disembl(linding等人2003,structure11:1453;http://dis.embl.de)确定。本文的实施例部分描述了用于此的合适的参数。
在另一种实施方案中,由根据本发明的文库编码的肽被预测具有小于约6和大于约8的等电点(pi)。在本文的实施例部分描述了使用“r肽包”的“pi”函数的合适实例。
在另一种实施方案中,由根据本发明的文库编码的肽的氨基酸序列的至少约30%、40%或50%与不同生物体中的肽相同;特别地,由根据本发明的文库编码的肽的氨基酸序列的约30%和70%之间与不同生物体中的肽相同。
在另外的实施方案中,根据本发明的文库中的每种核酸编码相同长度的肽。本领域技术人员应该理解,这可包括一些有限的可变性。在其他实施方案中,编码的肽具有约25个氨基酸和约100、90、85、80、75、70、65、60、55、50或45个氨基酸之间;约30个氨基酸和110个氨基酸之间;约30个氨基酸和约100、90、85、80、75、70、65、60、55或50个氨基酸之间;约30个氨基酸和约100、90、85、80、75、70、65、60或55个氨基酸之间的长度;特别地其中编码的肽具有在30个氨基酸和约75个氨基酸之间的长度;更具体地,其中编码的肽具有约35个氨基酸和约70个氨基酸之间或约35个氨基酸和约50个氨基酸之间的长度;并且最具体地,其中编码的肽具有35和60个氨基酸之间的长度,诸如42、43、44、45、46、47或48个氨基酸的长度。合适地,由根据本发明的文库编码的肽的长度将由实际考虑来确定,诸如所使用的寡核苷酸合成技术的最大限度(并考虑所添加的另外特征,例如如本文所述)。合适地,在根据本发明的(合成)核酸文库中,编码肽的编码区使用表1.1中列出的人类密码子频率表。然而,可以使用替代的人类密码子频率表,或者,根据意图表达核酸文库的表达系统的物种,可以使用其他物种的密码子频率表。在一种实施方案中,最常见的人类密码子用于编码肽的氨基酸。因此,至少一部分核酸序列将不是天然基因组序列。这些序列也可以被进一步修饰。在另外的实施方案中,替代的人类密码子用于编码肽的一个或更多个氨基酸。
合适地,“不需要的序列或子序列”被排除在根据本发明的文库之外。此类“不需要的序列或子序列”可以包括,例如,由密码子的组合制成的那些序列。此类不需要的子序列的具体实例包括内部kozak序列和/或意图用于克隆所得文库的限制酶的限制酶位点,在每种情况下从密码子的组合生成。通过使用第二最常用的(或另一个)密码子代替组合中的一个或另一个密码子,可以避免此类不需要的序列或子序列。因此,合适地,在根据本发明的(合成)核酸文库中,编码区不包含形成内部kozak序列的密码子的组合。普通技术人员将理解术语“kozak序列”,并且这样的含义可以包括由符号“(gcc)gccrccaugg”标识的核苷酸碱基序列,其中:(i)小写字母表示在碱基仍可变化的位置上最常见的碱基;(ii)大写字母表示高度保守的碱基,即“augg”序列是恒定的或很少变化(如果变化的话),例外是iupac歧义代码“r”,它表示嘌呤(腺嘌呤或鸟嘌呤)总是在这个位置被观察到(腺嘌呤被认为更频繁);和(iii)括号中的序列“(gcc)”具有不确定的意义。在特定实施方案中,kozak序列是“ccatgg”。
在本发明核酸文库的一些实施方案中,下列序列也是不需要的序列或子序列:“ggatcc”、“ctcgag”、“gggggg”、“aaaaaa”、“tttttt”、“cccccc”、引起寡核苷酸合成或测序或pcr扩增问题的序列、发夹序列、框内终止密码子(在末端时除外)。
在另一种实施方案中,根据本发明的文库中的每种核酸还包含编码区5’和/或3’的一个或更多个核酸序列,其包含至少一个限制酶识别序列;任选地在编码区和限制酶识别序列之间具有接头核酸序列。合适地,编码区不包含形成包含在5’和/或3’核酸序列中的限制酶识别序列的密码子的组合。如上所述,这可以通过使用第二最常用的密码子代替组合中的一个或另一个密码子来避免。
本发明的核酸文库(还包括其(例如,最初)被包含在合成核酸文库中的那些实施方案)可以通过非化学方法进行扩增、繁殖或以其他方式保持。例如,此类文库可以通过体外酶促(例如生物)过程诸如pcr或体外转录/翻译来扩增;或者可以通过体内过程复制(和/或繁殖/维持),诸如克隆到在宿主细胞(例如细菌或哺乳动物宿主细胞)中复制的载体中。
在另一种实施方案中,根据本发明的文库中的每种核酸还包含能够扩增核酸序列的另外序列(合适地,在每种情况下不在编码肽的编码区内,诸如编码区的5’和/或3’以及任选的限制酶识别序列);特别地其中此类扩增通过pcr扩增进行。
在另一种实施方案中,将根据本发明的文库中的每种核酸克隆到载体中。术语“载体”是本领域公认的,并包括可用于在/向细胞中繁殖、产生、维持或引入包含在其中的核酸的核酸的含义,诸如用于表达由包含所述核酸的序列编码的肽或多肽。一种类型的载体是质粒,其是指可以将另外的核酸区段连接到其中的线性或环状双链dna分子。另一种类型的载体是病毒载体(例如,复制缺陷型逆转录病毒、腺病毒和腺病毒相关病毒),其中另外的dna区段可以被引入到病毒基因组中。某些载体能够在引入了它们的细胞诸如宿主细胞中自主复制(例如,包含细菌复制起点的细菌载体和附加型哺乳动物载体(episomalmammalianvector))。其他载体(例如,非附加型哺乳动物载体)在引入到细胞中并在选择压力下培养后整合进入细胞的基因组中,并且从而与基因组一起复制。载体可用于指导选择的肽或多核苷酸在细胞中表达;特别地由包含在本发明文库中的核酸编码的肽的表达。合适地,当用于在真核细胞(诸如哺乳动物细胞)中表达本发明的肽时,载体是慢病毒载体,或者是逆转录病毒载体。
合适地,每种核酸还包含起始密码子、kozak序列、终止密码子和/或编码肽标签的核酸序列,或者其中载体包含与核酸可操作地连接的起始密码子、kozak序列、终止密码子和/或编码肽标签的核酸序列。合适地,肽标签(如果包括的话)是选自以下组成的组的肽标签:v4、flag、strep/ha和gfp;特别地其中标签是v4标签或flag标签。
在一种实施方案中,每种核酸适合于或能够表达包含编码肽的多肽。
在另一种实施方案中,多肽包含编码的肽,并且还包含n-末端甲硫氨酸、由一个或更多个限制酶识别序列编码的一个或更多个另外的氨基酸和/或一个或更多个肽标签。
在另一种实施方案中,根据本发明的(例如合成的)核酸文库的单个成员(或成员子集)呈汇集的格式(或形式)。合适地,本发明的“汇集格式”(或“汇集形式”)的文库包括其中其单个成员(或成员子集)与其他成员(或子集)混合的那些;例如,包含在单个器皿中的这些成员的溶液(或其干燥沉淀物),或包含本发明的重组载体的宿主细胞的群体。
合适地,根据本发明的文库中的单个成员(或成员子集)是空间上分开的。“空间上分开的”文库可以被认为是这样的文库,其中文库的多于一个成员(或成员子集)适当地以有序的方式彼此物理上分开。空间上分开的文库的实例包括其中单个成员(或成员子集)包含在一个或更多个微滴定板的单个孔中、排列在固体表面上或(以有序方式)结合到硅片的那些文库。在另一种实施方案中,根据本发明的文库中的单个成员(或成员子集)各自是可单独寻址的;也就是说,可以从文库中检索它们(例如,无需过度搜索或筛选)。用于寻址或询问根据本发明的文库的合适方法可以包括下一代测序(ngs)、pcr等。此外,当文库以空间上分开的格式(或形式)存在时,通过知晓适用的单个成员(或子集)的空间位置,单个成员(或成员子集)可以是“可单独寻址的”。在这些实施方案的任一个中,使用计算机程序、数据文件或数据库(诸如利用本发明的计算机可读介质或数据处理系统的那些)可以有助于检索包含在本发明的可单独寻址的文库中的单个成员(或成员子集)。
合适地,本发明的文库不是从cdna生成的。
在另一方面,本发明提供了肽文库,所述肽文库由根据本发明的(例如合成的)核酸文库编码。合适地,肽是合成的或重组的。在一种实施方案中,其单个成员(或成员子集)呈汇集的格式(或形式)。在另一种实施方案中,其单个成员(或成员子集)是空间上分开的和/或可单独寻址的。
在某些实施方案中,本发明的任何(合成)核酸文库可以被包含在编码具有约25个和110个氨基酸之间的长度的肽的另一个核酸文库的混合物中,可以是该另一个核酸文库的附加物(或以其他方式与之组合),或者可以与该另一个核酸文库一起使用。例如,此类其他文库可以是在共同待决申请pct/gb2016/054038(其内容通过引用并入本文)中描述的文库;特别地,这样的文库编码小的开放阅读框架(sorf),诸如编码至少500、1000、1500或约2000个人类sorf。
因此:(1)本发明的任何肽文库也可以被包含在由此类其他核酸文库编码的肽的另一个文库的混合物中,可以是该另一个文库的附加物(或以其他方式与之组合)或可以与该另一个文库一起使用;和(2)涉及本发明的核酸或肽文库的用途、方法和过程,也可以包括其与此类其他核酸或肽文库混合使用、作为其附加物(或以其他方式与之组合)或与此类其他核酸或肽文库一起使用(分别地)。例如,本文的实施例4描述了使用本发明的“hupex”文库和任选的pct/gb2016/054038中描述的人类sorf文库二者的筛选。
在一些实施方案中,肽文库不是t7展示文库,或者不是噬菌体展示文库,或者不是展示文库。在一些实施方案中,文库不是来源于多于一种(例如人类)病原体的多于一种肽,诸如来自多于一种病毒、细菌或真菌,例如对人类致病的病毒、细菌或真菌。
在另一方面,提供了一种容器或载体,所述容器或载体包含根据本发明的(例如合成的)核酸文库和/或包含根据本发明的肽文库。合适的容器包括器皿(诸如eppendorf管)、微量滴定板或硅载体。
在另一方面,本发明还涉及一种鉴定本发明文库中包含的肽与靶(特别地蛋白质靶)之间的结合相互作用的至少一种结合配偶体的方法,该方法包括以下步骤:
·在允许靶与文库的至少一种肽结合的条件下将本发明的肽文库暴露于靶;和
·鉴定结合肽或结合的靶。
在某些实施方案中,通过以某种形式(例如在宿主细胞中)提供本发明的核酸文库,并且在使得肽文库由核酸文库表达的条件下,将肽文库暴露于靶。
在另一方面(或上述实施方案),本发明提供了一种鉴定靶和/或肽的方法;包括例如洗脱肽/核酸,选择表达肽的细胞,随后例如pcr和测序鉴定。靶可以通过例如下拉质谱法来鉴定。此类方法描述于本文的其他地方、pct/gb2016/054038和/或wo2013/116903中。
在一个特定方面,本发明还涉及一种鉴定调节哺乳动物细胞的表型的靶蛋白的方法,所述方法包括将能够展示所述表型的体外培养的哺乳动物细胞群体暴露于本发明的核酸文库(或本发明的肽文库),鉴定在所述暴露后所述细胞群体中所述表型的改变,选择经历表型改变的所述细胞,并鉴定由此类文库编码的改变细胞表型的肽(或此类文库中改变细胞表型的肽),提供所述肽并鉴定与所述肽结合的细胞蛋白,所述细胞蛋白是调节哺乳动物细胞的表型的靶蛋白。合适的方法和表型筛选在例如pct/gb2016/054038中描述,并且在其中描述的这些方法和筛选的技术特征,但使用了本发明的文库,通过引用并入本文。
在某些实施方案中,此类方法包括鉴定结合所述靶蛋白并置换或阻断所述肽的结合的化合物的另外步骤。此类另外的步骤,从而鉴定出结合靶蛋白并置换或阻断所述肽的结合的化合物,其中该化合物调节哺乳动物细胞的表型。
因此,在另一个特定方面,本发明还涉及一种鉴定结合靶蛋白并置换或阻断肽的结合的化合物的方法,其中该化合物调节哺乳动物细胞的表型,所述方法包括以下步骤:
i.将能够展示所述表型的体外培养的哺乳动物细胞群体暴露于本发明的核酸文库或本发明的肽文库;
ii.鉴定在所述暴露后群体中显示所述表型的改变的细胞;
iii.鉴定由改变细胞的所述表型的此类文库编码的肽或此类文库中的肽;
iv.鉴定结合所述肽的细胞蛋白,所述细胞蛋白是调节哺乳动物细胞的所述表型的靶蛋白;
v.鉴定结合所述靶蛋白并置换或阻断所述肽的结合的化合物。
在一个其他方面,本发明涉及:(a)根据本发明的核酸文库;和/或(b)根据本发明的肽文库,用于鉴定结合靶(特别地蛋白质靶)的肽的用途。在某些实施方案中,鉴定的肽调节哺乳动物细胞的表型。
在另一个其他方面,本发明涉及:(a)根据本发明的核酸文库;和/或(b)根据本发明的肽文库,用于鉴定调节哺乳动物细胞的表型的靶(特别地蛋白质靶)的用途。
在又另一个其他方面,本发明涉及:(a)根据本发明的核酸文库;和/或(b)根据本发明的肽文库,用于鉴定结合靶(特别地蛋白质靶),并且任选地置换或阻断肽与靶的结合的化合物的用途。在某些实施方案中,肽和/或化合物调节哺乳动物细胞的表型。
合适地,在这些方面,使用本发明的文库(或肽)进行表型筛选的方法可以选自:(1)途径特异性读出,其使用异源报告物(例如gfp或萤光素酶)记录活细胞中的总蛋白质水平、蛋白质定位或在基因转录水平的最终途径活性;(2)在固定的“非活”细胞中使用抗体或其他亲和试剂记录内源性蛋白质水平或其定位,或使用qpcr或rna测序记录途径特异性转录输出;(3)活细胞中基于高含量或“整体”的读出,其能够记录治疗重要的特定“目的地”表型读出,例如分化、衰老和细胞死亡,所有这些都是协调的,并且可以通过多种细胞途径的复杂相互作用被特异地调节。在本发明的一些实施方案中,测定读出方法使用gfp报告物,例如在kaizuka等人molecularcell2016中描述的。
在本发明涵盖“整体”表型测定的特定方面,合成致死性(syntheticlethality)筛选特别重要。合成致死性筛选是寻找靶(例如癌症靶)和候选治疗剂的方法,候选治疗剂可通过利用不可预测的次级弱点,与正常细胞相比,选择性地影响肿瘤细胞,由于肿瘤细胞猛烈地重接信号传导通路以支持无限制的细胞增殖,所以所述次级弱点可发生在肿瘤细胞中。因此,此类筛选必须在活细胞中并且以无偏的方式进行,通过抑制或调节细胞中的基因(使用crispr)、mrna(使用rnai)或蛋白质或蛋白质构象(使用protein-i),然后确定是否发生对肿瘤细胞类型的整体生长或存活的一致的负面影响;优选的是,与正常细胞类型相比,具有发生在肿瘤情况下的特定遗传改变的细胞。这些基于直接的“整体的”细胞存活力输出的筛选是这样进行的:使用大组(largepanel)遗传表征的肿瘤细胞和正常细胞来获取关于肿瘤基因型依赖性响应的相关性信息,或更有效地使用特定工程化的细胞系来进行,所述细胞系对于分别存在于癌细胞与正常细胞中的选择的突变体与正常基因型是等基因的。
适当地,在某些实施方案中,牵涉本文描述的创造性文库的本发明的用途和方法涉及与细胞信号传导途径的调节相关的表型;特别地涉及鉴定调节细胞信号传导途径的肽(例如,来自本发明的文库)和鉴定参与信号转导并且可用作调节细胞信号传导途径,特别地在癌细胞中有活性的途径的药物靶的此类蛋白质上的蛋白质靶和表面位点的用途或方法。
合适地,细胞信号传导途径是细胞中的一系列相互作用因子,它们在细胞表面处响应细胞外刺激而在细胞内传递细胞内信号,并导致细胞表型的改变。信号沿着细胞信号传导途径的传递通常导致一种或更多种转录因子的激活,这改变基因表达。本发明涉及的用途、方法或筛选的优选细胞信号传导途径在疾病模型中显示异常活性,例如在病变细胞(诸如癌细胞)中激活、上调或错调。例如,途径可以在癌细胞中组成性激活(即永久地开启),或被细胞外配体不适当地激活,例如在类风湿性关节炎的炎性细胞中。
功能性细胞信号传导途径通常被认为是完整且如果该途径被开启或激活,例如通过适当的细胞外刺激,能够传递信号的途径。活跃的细胞信号传导途径通常被认为是例如通过适当的细胞外刺激而被开启,并且活跃地传递信号的途径。
合适的细胞信号传导途径包括响应于细胞接收的信号而导致转录事件的任何信号传导途径。
本文所述的用于研究的细胞信号传导途径可包括可在癌细胞中激活或改变的细胞信号传导途径,例如ras/raf、20hedgehog、fas、wnt、akt、erk、tgfβ、egf、pdgf、met、pi3k和notch信号传导途径。
在又另一方面,本发明涉及一种其上存储有信息的计算机可读介质(例如,一种用于——例如,一种特别适用于——本文所述的筛选方法的计算机可读介质),包括:(a)包含在本发明核酸文库中的核酸序列;和/或(b)由所述核酸编码的肽的氨基酸序列。
在相关方面,本发明涉及一种存储和/或处理信息的数据处理系统(例如,一种用于——例如,一种特别适用于——本文描述的筛选方法的数据处理系统),所述信息包括:(a)包含在本发明核酸文库中的核酸序列;和/或(b)由所述核酸编码的肽的氨基酸序列。
鉴于以上所述,将理解,本发明还涉及以下项目:
项目1:如本发明第一方面所述的核酸文库,其中生物体的物种是智人。
项目2:根据项目1的核酸文库,其中不同的天然存在的蛋白质是不同物种的多于一种生物体的天然存在的蛋白质。
项目3:根据项目2的核酸文库,其中多于一种不同物种选自表a中所列的生物体(微生物)物种的组;特别地其中多于一种不同物种包含跨越表a中所列的至少5个门的至少20种生物体(微生物)物种。
项目4:根据项目2的核酸文库,其中多于一种不同物种选自表b中所列的物种的组,特别地其中多于一种不同物种包括表b中所列的跨越本文所述的表b的至少5个部分的至少50种物种:古细菌、细菌、真菌、无脊椎动物、植物、原生动物、哺乳动物和非哺乳类脊椎动物。
项目5:根据项目2至4中任一项的核酸文库,其中多于一种不同生物体是至少100种不同生物体。
项目6:根据项目1至4中任一项的核酸文库,其中不同的天然存在的蛋白质是在两种或更多种不同细胞群体或组织类型之间差异表达的蛋白质。
项目7:根据项目6的核酸文库,其中不同的天然存在的蛋白质在患病和正常细胞或组织类型之间差异表达;特别地其中不同的天然存在的蛋白质在人类癌细胞和非癌人类细胞之间差异表达。
项目8:根据项目6或7的核酸文库,其中不同的天然存在的蛋白质是疾病特异性的;特别地其中不同的天然存在的蛋白质由人类癌细胞表达,但不由非癌人类细胞表达。
项目9:根据项目1至8中任一项的核酸文库,其中多于一种编码肽具有不同的序列;特别地其中多于一种编码肽的氨基酸序列彼此相差至少2或3个氨基酸;特别地其中多于一种编码肽的氨基酸序列彼此相差至少约5、8或10个氨基酸。
项目10:根据项目9的核酸文库,其中多于一种编码肽的序列相似性小于约80%。
项目11:根据项目1至10中任一项的核酸文库,其中所述肽被预测不包含无序区域。
项目12:根据项目1至11中任一项的核酸文库,其中所述肽被预测具有小于约6和大于约8的等电点(pi)。
项目13:根据项目1至12中任一项的核酸文库,其中所述肽的氨基酸序列的至少约40%与不同生物体中的肽相同。
项目14:根据项目1至13中任一项的核酸文库,其中每种核酸编码相同长度的肽。
项目15:根据项目1至14中任一项的核酸文库,其中所述编码肽具有35和60个氨基酸之间的长度,诸如42、43、44、45、46、47或48个氨基酸的长度。
项目16:根据项目1至15中任一项的核酸文库,其中编码肽的编码区使用表1.1中列出的人类密码子频率表。
项目17:项目16的核酸文库,其中最常见的人类密码子用于编码肽的氨基酸。
项目18:项目16或17的核酸文库,其中使用替代的人类密码子来编码肽的一个或更多个氨基酸。
项目19:根据项目1至18中任一项的核酸文库,其中所述编码区不包含形成内部kozak序列的密码子的组合;特别地其中kozak序列是ccatgg。
项目20:根据项目1至19中任一项的核酸文库,其中每种核酸还包含编码区5’和/或3’的一个或更多个核酸序列,其包含至少一个限制酶识别序列;任选地在编码区和限制酶识别序列之间具有接头核酸序列。
项目21:根据项目20的核酸文库,其中编码区不包含形成包含在5’和/或3’核酸序列中的限制酶识别序列的密码子的组合。
项目22:根据项目1至21中任一项的核酸文库,其中每种核酸还包含能够扩增核酸序列的另外序列;特别地其中此类扩增通过pcr扩增进行。
项目23:根据项目1至22中任一项的核酸文库,其中每种核酸被克隆到载体中。
项目24:根据项目1至23中任一项的核酸文库,其中每种核酸还包含起始密码子、kozak序列、终止密码子和/或编码肽标签的核酸序列,或者其中载体包含与核酸可操作地连接的起始密码子、kozak序列、终止密码子和/或编码肽标签的核酸序列。
项目25:项目24的核酸文库,其中每种核酸适合于或能够表达包含编码肽的多肽。
项目26:项目25的核酸文库,其中多肽包含编码的肽,并且还包含n-末端甲硫氨酸、由一个或更多个限制酶识别序列编码的一个或更多个另外的氨基酸和/或一个或更多个肽标签。
项目27:根据项目1至26中任一项的核酸文库,其中其单个成员呈汇集的格式。
项目28:根据项目1至27中任一项的核酸文库,其中其单个成员是空间上分离的。
项目29:一种肽文库,由如本发明另一方面所述的核酸文库,合适地根据项目1至28中任一项的核酸文库编码。
项目30:项目29的肽文库,其中肽是合成的或重组的。
项目31:项目29的肽文库,其中其单个成员呈汇集的格式。
项目32:项目29的肽文库,其中其单个成员是空间上分离的。
项目33:一种容器或载体,其包含如本发明另一方面所述的核酸文库,合适地根据项目1至33中任一项的核酸文库。
项目33a:一种容器或载体,其包含如本发明另一方面所述的肽文库,合适地根据项目30至32中任一项的肽文库。
项目34:项目33或33a的容器或载体,所述容器或载体是微量滴定板或硅载体。
项目35:一种鉴定肽与蛋白质靶之间的结合相互作用的至少一种结合配偶体的方法,所述肽:(a)被包含在如本发明另一方面所述的文库,合适地根据项目29至32的文库中,或(b)由如本发明另一方面所述的核酸文库,合适地根据项目1至28中的任一项的核酸文库表达;该方法包括以下步骤:
·在允许靶与文库中至少一种肽或由文库表达的至少一种肽结合的条件下将本发明暴露于蛋白质靶;和
·鉴定结合肽或结合的靶。
项目36:如本发明另一方面所述的核酸文库,适当地根据项目1至28中任一项的核酸文库,鉴定结合靶蛋白的肽的用途;特别地其中肽调节哺乳动物细胞的表型。
项目37:如本发明另一方面所述的核酸文库,适当地根据项目1至28中任一项的核酸文库,鉴定调节哺乳动物细胞的表型的靶蛋白的用途。
项目38:一种在其上存储有信息的计算机可读介质,所述信息包括:(a)如本发明另一方面所述的核酸文库,合适地根据项目1至28中任一项的核酸文库中包含的核酸序列;和/或(b)由所述核酸编码的肽的氨基酸序列。
项目39:一种存储和/或加工信息的数据处理系统,所述信息包括:(a)如本发明另一方面所述的核酸文库,合适地根据项目1至28中任一项的核酸文库中包含的核酸序列;和/或(b)由所述核酸编码的肽的氨基酸序列。
鉴于以上所述,将理解,本发明还涉及以下条款:
条款1:一种核酸文库,每种核酸包含编码肽的确定的核酸序列的编码区,所述肽具有25和110个氨基酸之间的长度,并且具有为选自一种或更多种生物体的天然存在的蛋白质的氨基酸序列中的序列区域的氨基酸序列;其中该文库包含编码至少10,000种不同的此类肽中的多于一种肽的核酸,并且其中至少50种此类肽中每一种肽的氨基酸序列是多于一种不同的此类天然存在的蛋白质中不同蛋白质的氨基酸序列中的序列区域。
条款2:条款1的核酸文库,其中多于一种不同的天然存在的蛋白质中的每一种蛋白质满足一个或更多个预定标准。
条款3:条款2的核酸文库,其中多于一种天然存在的蛋白质中的每一种蛋白质与给定的疾病诸如癌症相关。
条款4:条款3的核酸文库,其中所述疾病是乳腺癌。
条款5:条款2的核酸文库,其中多于一种天然存在的蛋白质中的每一种蛋白质是细胞质蛋白质。
条款6:条款5的核酸文库,其中多于一种天然存在的蛋白质中的每一种蛋白质是细胞质激酶。
条款7:条款2的核酸文库,其中多于一种天然存在的蛋白质中的每一种蛋白质与给定的蛋白质或来自蛋白质(功能)类的至少一种蛋白质相互作用。
条款8:条款7的核酸文库,其中多于一种天然存在的蛋白质中的每一种与kras相互作用。
条款9:条款1至8中任一项的核酸文库,其中所述文库包含编码至少50,000种不同的此类肽中的多于一种肽的核酸,并且其中至少100种此类肽的每一种肽的氨基酸序列是至少100种不同的天然存在的蛋白质的氨基酸序列中的序列区域;特别地,其中所述文库包含编码至少100,000种不同的此类肽中的多于一种肽的核酸,并且其中至少150种此类肽的每一种肽的氨基酸序列是至少150种不同的天然存在的蛋白质的氨基酸序列中的序列区域。
条款10:条款1至9中任一项的核酸文库,其中所述文库包含编码至少10,000种不同的此类肽中的多于一种肽的核酸,并且其中至少1,000种此类肽的每一种肽的氨基酸序列是此类多于一种不同的天然存在的蛋白质的不同蛋白质的氨基酸序列中的序列区域。
条款11:条款1至10中任一项的核酸文库,其中所述文库包含编码至少200,000种不同的此类肽中的多于一种肽的核酸,并且其中至少20,000种此类肽的每一种肽的氨基酸序列是至少20,000种不同的天然存在的蛋白质的氨基酸序列中的序列区域;特别地,其中所述文库包含编码至少300,000种不同的此类肽中的多于一种肽的核酸,并且其中至少25,000种此类肽的每一种的氨基酸序列是至少25,000种不同的天然存在的蛋白质的氨基酸序列中的序列区域。
条款12:条款1或11中任一项的核酸文库,其中对于至少约1%的天然存在的蛋白质,多于一种所述核酸编码来自此类天然存在的蛋白质的氨基酸序列的不同肽。
条款13:条款12的核酸文库,其中对于至少约50%的天然存在的蛋白质,多于一种所述核酸编码来自此类天然存在的蛋白质的氨基酸序列的不同肽。
条款14:条款13的核酸文库,其中所述多于一种核酸编码不同的肽,并且其氨基酸序列是沿着所述天然存在的蛋白质的氨基酸序列被间隔开的序列区域。
条款15:根据条款14所述的核酸文库,其中所述序列区域沿着天然存在的蛋白质的氨基酸序列被一个氨基酸窗口或多于一个此类窗口分隔开,其中所述窗口在1和约55个氨基酸之间;特别地其中该窗口在约5和约20个氨基酸之间;最特别地,其中间隔窗口是约8、10、12或15个氨基酸。
条款16:条款1至14任一项的核酸文库,其包含编码来自至少10,000种不同的天然存在的蛋白质的至少100,000种不同肽的核酸。
条款17:条款1至16中任一项的核酸文库,其中每种核酸编码不同的肽。
条款18:条款1至17中任一项的核酸文库,其中编码来自天然存在的蛋白质的不同肽的核酸的平均数目大于1;特别地每种此类蛋白质约1.01和1.5个之间这样的核酸(肽)。
条款19:条款18的核酸文库,其中编码来自天然存在的蛋白质的不同肽的核酸的平均数目为每种此类蛋白质至少约5个这样的核酸(肽),特别地其中平均数目为每种此类蛋白质约5和约2,000个之间的这样的核酸(肽)或每种此类蛋白质约5和约1,000个之间的核酸(肽)。
条款20:条款19的核酸文库,其中编码来自天然存在的蛋白质的不同肽的核酸的平均数目为每种此类蛋白质约100和约1,500个之间的这样的核酸(肽),或每种此类蛋白质约250和约1,000个之间的这样的核酸(肽)。
条款21:条款20的核酸文库,其中编码来自天然存在的蛋白质的不同肽的核酸的平均数目为每种此类蛋白质约5和约100个之间的这样的核酸(肽),或每种此类蛋白质约5和约50个之间的这样的核酸(肽)。
条款22:条款1至21中任一项的核酸文库,其中天然存在的蛋白质的氨基酸序列是选自参考蛋白质组中包含的非冗余蛋白质的氨基酸序列的组的氨基酸序列,合适地,参考蛋白质组是选自表a和/或表b中列出的参考蛋白质组的组的一种或更多种参考蛋白质组、或此类参考蛋白质组的更新版本。
条款23:条款1至22中任一项的核酸文库,其中所述多于一种编码肽的氨基酸序列是选自具有已知三维结构的天然存在的蛋白质(或其多肽链或结构域)的氨基酸序列中的序列区域;特别地其中所述天然存在的蛋白质(或其多肽链或结构域)被包含在蛋白质数据库中,并且任选地具有pfam注释。
条款24:条款22至23中任一项的核酸文库,其中选自所述蛋白质的氨基酸序列中的序列区域不包括包含在所述参考蛋白质组或蛋白质数据库中的此类氨基酸序列的不明确氨基酸。
条款25:一种肽文库,所述肽文库由条款1至24中任一项的核酸文库编码。
合适地,本发明是核酸文库,每种核酸包含编码肽的确定的核酸序列的编码区,所述肽具有25和110个氨基酸之间的长度,并且具有这样的氨基酸序列:选自一种或更多种生物体的天然存在的蛋白质的氨基酸序列的序列区;其中所述文库包含编码至少10,000种不同的此类肽中多于一种肽的核酸,并且其中至少50种此类肽中每一种肽的氨基酸序列是多于一种不同的此类天然存在的蛋白质中不同蛋白质的氨基酸序列的序列区,并且其中每种编码的氨基酸序列的等电点(pi)大于7.4或小于6.0,和/或其中所述核酸序列不包含序列“ggatcc”和/或“ctcgag”,和/或其中所述核酸序列不包含序列“gggggg”和/或“aaaaaa”和/或“tttttt”和/或“cccccc”,和/或其中所述核酸序列不包含引起寡核苷酸合成或测序或pcr扩增问题的序列,和/或其中所述核酸序列不包含发夹序列,和/或其中所述核酸序列不包含框内终止密码子(除了在末端),和/或其中所述核酸序列不包含kozak序列(除了在起点),即不包含内部kozak序列。
本文使用的术语“本发明的”、“根据本发明的”、“根据本发明”等意图指本文描述和/或要求保护的本发明的所有方面和实施方案。
如本文使用的,术语“包括”应被解释为涵盖“包含”和“由......组成”二者,这两个含义都是特别意图的,并因此与本发明单独公开的实施方案一致。在本文中使用时,“和/或”应被视为两个指定的特征或部件中的每一个在有或没有另一个的情况下的具体公开。例如“a和/或b”被认为是(i)a、(ii)b和(iii)a和b中的每一个的具体公开,如同每一个在本文中单独列出一样。在本发明的上下文中,术语“约”和“大约”表示本领域技术人员将理解的仍然确保所讨论的特征的技术效果的准确度间隔。该术语通常表示与指示的数值偏离±20%、±15%、±10%,和例如±5%。如普通技术人员所理解的,给定技术效果的数值的具体此类偏差将取决于技术效果的性质。例如,自然或生物技术效果通常可比人为或工程技术效果具有更大的此类偏差。如普通技术人员所理解的,给定技术效果的数值的具体此类偏差将取决于技术效果的性质。例如,自然或生物技术效果通常可比人为或工程技术效果具有更大的此类偏差。当使用不定冠词或定冠词指代单数名词时,例如“一(a)”、“一(an)”或“该(the)”,这包括该名词的复数形式,除非另有特别说明。
应当理解,根据本文包含的教导,将本发明的教导应用于特定的问题或环境,以及包括本发明的变化形式或另外特征(诸如另外的方面和实施方案),将在本领域普通技术人员的能力范围内。
除非上下文另有说明,否则上述特征的描述和定义不限于本发明的任何特定方面或实施方案,并且同样适用于所描述的所有方面和实施方案。
本文引用的所有参考文献、专利和出版物通过引用以其整体特此并入。
表a:微生物的演化多样集的参考蛋白质组的数据库来源
表b:物种的演化多样集的参考蛋白质组的数据库来源
实施例表明:
实施例1:本发明的核酸文库的设计,其基于在人类蛋白质组(“hupex”)中包含的蛋白质的氨基酸序列编码短肽。
首先,通过串联智人参考蛋白质组中包含的多于一种单独蛋白质(在这种情况下,所有21,018种这样的蛋白质)的氨基酸序列,生成单个“巨型蛋白质”氨基酸序列。因此,此类氨基酸序列可以被认为是天然存在的蛋白质的氨基酸序列;即它们天然存在于人类中。使用的参考蛋白质组(up000005640_9606.fasta)从基于embl-ebi网络的资源获得:“referenceproteomes-primaryproteomesetsforthequestfororthologs”(http://www.ebi.ac.uk/reference_proteomes,2017年2月5日访问),2017_01版,基于uniprot2017_01版。
通过使用间隔符号“_”来标记两个串联蛋白质的氨基酸序列之间的每个连接,使得(例如)在人类参考蛋白质组中以fasta格式列出的两个示例蛋白质:
>tr|a0a024r161|a0a024r161_humanguaninenucleotide-bindingproteinsubunitgammaos=homosapiensgn=dnajc25-gng1ope=3sv=1
mgapllspgwgagaagrrwwmllapllpalllvrpagalveglycgtrdcyevlgvsrsagkaeiarayrqlarryhpdryrpqpgdegpgrtpqsaeeafllvatayetlkvsqaaaelqqycmqnackdallvgvpagsnpfreprscall
>tr|a0a075b6f4|a0a075b6f4_humant-cellreceptorbetavariable21/or9-2(pseudogene)(fragment)os=homosapiensgn=trbv21or9-2pe=4sv=1
xrflseptrclrllccvalsfwgaasmdtkvtqrprflvkaneqkakmdcvpikrhsyvywyhktleeelkffiyfqneeiiqkaeiinerfsaqcpqnspctleiqstesgdtaryfcansk
将被串联形成单个氨基酸序列,连接周围的区域如下所示:
[...]lvgvpagsnpfreprscall_xrflseptrclrllccvals[...]
第二,计算机程序从代表多于一种蛋白质(在这种情况下,所有21,018种)的“巨大蛋白质”氨基酸序列选择了都为46个氨基酸长的区域,这些区域由10个氨基酸的窗口隔开。然而,在其他实施方案中,可以指示计算机程序选择具有可选的(预定)长度的氨基酸区域,例如在25个氨基酸和110个氨基酸之间的任何这样的长度;和/或可以指示计算机程序将这些区域用可选的(预定)窗口,例如约2和40个氨基酸之间的窗口隔开。例如,从上述第一个蛋白质的串联间隔10个氨基酸窗口的46个氨基酸的区域的归档平铺(filingtiling),将生成代表短肽的下列氨基酸序列(只显示了前三个这样的肽序列):
肽1:mgapllspgwgagaagrrwwmllapllpalllvrpagalveglycg
肽2:gagaagrrwwmllapllpalllvrpagalveglycgtrdcyevlgv
肽2:mllapllpalllvrpagalveglycgtrdcyevlg[...]
第三,所得的46个氨基酸长的肽序列的集合,通过从集合中去除具有以下任何一个(或更多个)特征的任何序列来过滤:
(a)长度不等于46个氨基酸的任何序列(既作为计算机程序的质量控制步骤,又去除由蛋白质序列末端产生的任何短序列);
(b)包含代表两种蛋白质的氨基酸序列之间的连接的间隔符号(在本例中为“_”)的任何序列;
(c)包含不明确氨基酸符号的任何序列(例如,“b”、“j”、“x”或“z”;在某些数据库中,此类模糊代码可能具有以下含义:b=d或n,j=i或l,x=未知,z=e或q;例如,上面显示的第二种蛋白质的前46个氨基酸长的肽序列——因为它以“x”开头——因此将从集合中去除);和/或
(d)任何不独特的序列(即与任何其他序列100%相同的任何序列)。
第四,使用普遍可用的pi预测软件来预测集合中剩余的每种所得肽序列的等电点(pi)(在这种情况下,“r肽包”的“pi”函数;osorio等人2015,therjournal.7:4;https://cran.r-project.org/web/packages/peptides/index.html;使用参数(argument)pkscale=“emboss”)。具有被预测为具有在6和8之间的pi的氨基酸序列的肽也被从该集合中去除。肽的等电点是肽没有净电荷时的ph,并且通常可以从此类ph的溶液中沉淀出来。因此,在一些实施方案中,具有大约生理ph(例如6至7.4或6至8)的预测pi的肽被排除在该集合之外,因为它们可能是在此类条件下更可能具有不利性质的那些(例如,通过在表达时沉淀)。
第五,对所得的氨基酸序列集合(在第三和第二步中描述的过滤后)进行最终过滤,以仅阳性选择与小鼠(小家鼠;up000000589_10090.fasta,如上所述获得)的参考蛋白质组中存在的氨基酸序列显示100%同一性的那些46个氨基酸的序列。
如普通技术人员现在将理解的,(46-长)氨基酸序列的最终集合将沿着天然存在的蛋白质以一个(在本例中)10氨基酸窗口或多于一个10氨基酸窗口间隔开。这是因为一个或更多个间插的氨基酸序列可能已经从最终集合中省略,因为它不符合各个过滤标准中的一个或另一个。
在本实施例中,(46-长)氨基酸序列的最终集合由约300,000个单独序列组成,至少一个这样的序列代表人类蛋白质组中超过21,000种不同的蛋白质,并且每种蛋白质平均约14.2个这样的序列,并且标准偏差为3.8个序列(95%的蛋白质将由7.6和20.7个之间的肽序列代表)。
现在对普通技术人员来说明显的是,代表用于选择的蛋白质组的肽的氨基酸序列的最终集合可以多于或少于这个数目,并且用于每种蛋白质的肽数目的分布也可以不同。氨基酸序列的最终集合的数量和性质不仅取决于首先串联的多于一种蛋白质的序列(例如,使用的单个物种的蛋白质组,或多于一个物种的蛋白质组),而且其他因素也会影响最终集合。例如,最初选择的序列区域可以比本文使用的46个氨基酸更短(或更长),和/或间隔窗口可以比本文使用的10个氨基酸更短(或更长)。此外,可以省略上述一个或更多个过滤标准,或者可以替代地或还应用不同的过滤标准(其他过滤步骤的实例在本文的其他实施例中描述)。实际上,取决于本发明的文库所需的特定性质(诸如大小、多样性、覆盖度和/或溶解度等)和/或其物理制备方法(诸如以下所述),并且特别地用于其物理制备的方法在大小、复杂度或成本上的限制,普通技术人员现在将能够只选择用于形成适合其需要的任何特定文库的肽的那些氨基酸序列。
第六,使用每种氨基酸的最常用密码子对46个氨基酸的序列中的每一个进行反向翻译,这些密码子见下文表1.1中所示的人密码子频率表(密码子使用数据库;nakamura等人,2000,nar28:292;http://www.kazusa.or.jp/codon/cgi-bin/showcodon.cgi?species=9606&aa=1&style=n,accessed04-jun-2017)。
然而,对于普通技术人员来说明显的是,可以使用替代的人类密码子频率表,或者,根据意图表达核酸文库的表达系统的物种,可以使用其他物种的密码子频率表。
表1.1:人类密码子频率表(密码子|aa|每个密码子的分数/aa)。
第七,然后分析编码肽的所得核酸序列,寻找由密码子的组合产生的不需要的子序列。此类不需要的子序列的具体实例包括内部kozak序列和/或意图用于克隆所得文库的限制酶的限制酶位点,在每种情况下从密码子的组合生成。如果核酸序列中存在不需要的子序列,则使用第二最常用的密码子来代替组合中的一个或另一个密码子,使得核酸序列中不再存在不需要的子序列。
在此实施例中,kozak序列“ccatgg”被认为是不需要的,并且形成此类子序列的核酸序列中的任何密码子组合适于使用不太常见的密码子,使得此类子序列不再存在。
由于本实施例的文库将使用某些限制酶克隆到表达载体中,因此核酸序列中作为此类限制酶识别位点的任何子序列也被认为是不需要的。限制酶bamhi和xhoi被设想为可以在此类克隆过程中使用的限制酶,并且因此任何下列子序列(此类限制酶的各自识别位点)被认为是不期望的:5'ggatcc和5’ctcgag。核酸序列中形成任何这样的子序列的任何密码子组合是这样的(预定的)限制酶识别位点,这些密码子组合适于使用不太常用的密码子,使得这样的子序列不再存在。现在对普通技术人员来说明显的是,根据计划使用的任何限制酶,由此类限制酶的识别位点组成的适用子序列可以通过适当使用替代密码子来去除。
对于本领域普通技术人员来说同样明显的是,由于核酸序列是通过(天然存在的)氨基酸序列的反向翻译生成的,因此所得核酸序列文库将包含本身是非天然的核酸序列,因为将使用天然存在的基因组序列所不使用的密码子(和/或密码子的组合)来编码蛋白质中该位置的一个或更多个特定氨基酸。因此,本发明的核酸文库将包含多于一种非天然核酸序列。
实施例2:本发明的hupex核酸文库的合成。
首先,在实施例1中设计的编码天然存在的人类蛋白质的独特的46个氨基酸长的肽“片段”的文库中的每种核酸序列用5’和3’序列进行适配,所述5’和3’序列适于提供使得能够克隆核酸文库和/或使得能够表达核酸文库中编码的肽的寡核苷酸序列。
在该实施例中,此类寡核苷酸(具有5’和3’区域)的每个所得核酸序列的一般结构是:
forward-amp_bamhi_kozac_gly_variable-region_stop_xhoi_reverse-amp,
其中“forward-amp”和“reverse-amp”代表选择的核酸序列,使得产生的寡核苷酸可以通过使用合适引物的pcr扩增;“bamhi”和“xhoi”代表各自的限制酶的识别位点的核酸序列;“kozak”代表kozak序列(包括起始密码子);“gly”代表单个甘氨酸氨基酸接头的密码子;“stop”代表终止密码子;并且“variable-region”代表来自实施例1中设计的核酸文库的编码给定46个氨基酸长的肽(存在于集合中)的单个138bp核酸。
在该实施例中,所用的正向和反向扩增序列、kozak序列和终止密码子列于下表2.1。
表2.1:hupex文库中使用的常见序列。
因此,由此类设计产生并编码集合中存在的下列指示性46个氨基酸的序列(seqidno.13)
seqidno.3:指示性寡核苷酸的核酸序列
正如现在对普通技术人员来说明显的,最终寡核苷酸的核酸序列可以,例如,替代性地设计成不包含kozak/起始或终止密码子。在这种情况下,kozak/起始和终止密码子可以由宿主载体提供。通过将文库克隆到载体中、使得载体编码的flag标签位于所编码的肽的n-或c-末端,此类设计(如下文其他实施例所示)可以允许产生加标签的格式的文库。
在替代实施方案中,可以构建本发明文库的两个版本:一个有起始密码子,一个没有起始密码子。这样的实施方案可用作使用文库进行筛选的数据分析的内部参考,因为它将允许区分肽的作用和载体/构建体本身的作用(载体/构建体本身的作用应被鉴定为假阳性)。例如,为了区分通过pcr更好地扩增特定序列的情况,引起(attract)一些细胞机制并引起间接效应的dna序列等。
第二,所有大约300,000个寡核苷酸的核酸序列的集合用于通过常规方法化学合成每个寡核苷酸。
在这种情况下,twistbioscience(sanfrancisco,california)的基于半汇集的10,000孔硅芯片的寡核苷酸合成方法用于合成所有大约300,000个寡核苷酸,并且因此寡核苷酸可在各自具有大约2,000个寡核苷酸的复杂度的子池中获得。这里应该注意的是,如果每个子池合成的寡核苷酸是用引物对的不同组合合成的(例如,所有组合中使用的45个不同的引物对将提供45*45=2025个不同的组合),那么有可能通过仅使用适用的特异性引物对组合进行pcr来回收*单独*序列。然而,可以使用其他合成寡核苷酸方法,诸如基于阵列的合成(例如,affymetrix)或原位合成印刷(agilent)。
实施例3:将本发明的hupex核酸文库克隆到表达载体中。
将大约300,000个寡核苷酸的集合(以汇集或亚汇集的格式)克隆到慢病毒表达系统中,简述如下。
首先,对寡核苷酸的池进行pcr扩增(通过标准程序并使用具有序列5'-tgccacctgacgtctaagaa-3'(seqidno.4)和5'-attaccgcctttgagtgagc-3'(seqidno.5)的引物,分别对应于寡核苷酸中的正向和反向扩增序列)。第二,所得产物用适用的限制酶(在本例中为bamhi和xhoi)消化,以提供用于克隆的粘性末端构建体。
第三,将粘性末端构建体连接到bamhi/xhoi消化的慢病毒载体(例如pmost25)的样品中。pmost25的克隆位点的六十(60)个碱基对在以下由seqidno.6示出,并且具有实施例2所示的指示性寡核苷酸的bamhi/xhoi消化的扩增产物的所得重组构建体,将具有由seqidno.7所示的序列。bamhi和xhoi限制位点加框;kozak序列加双下划线;起始/终止密码子的第一个碱基上方标有“*”;并且编码46个氨基酸的序列的138bp区域加粗。
seqidno.6:pmost25慢病毒载体的克隆位点的60bp。
seqidno.7:指示性重组构建体。
将重组载体转化/转染入宿主细胞将使得能够繁殖、表达和/或筛选本发明的核酸文库。对普通技术人员来说明显的是,核酸文库在宿主细胞中扩增和/或繁殖/保持后,可以被认为不再是“合成的”,因为届时核酸分子将被酶促产生(在体外或在体内)。尽管如此,这样的核酸文库仍然被认为是本发明的核酸文库。
seqidno.7中所示的构建体的表达将产生具有seqidno.8所示的序列的48个氨基酸的肽,其中最初编码的46个氨基酸的指示性肽的序列以粗体显示在起始的甲硫氨酸和连接甘氨酸之后。
seqidno.8:指示性表达肽。
作为本领域技术人员或普通技术人员,我们注意到,此类表达的肽包括对应于5’和3’限制酶识别位点的n-和c-末端氨基酸(和在这种情况下,单个连接val)。因此,虽然最初的46个氨基酸的序列是天然存在的蛋白质的片段,得到的52个氨基酸的表达的肽是非天然的。因此,本发明的文库将编码多于一种非天然肽。
文库的克隆可以以汇集方式或以半汇集方式进行。此外,可以使用自动化细胞分选技术,诸如qpix(moleculardevices)、facs或单细胞分配(例如solentimltd,uk的vipstmcelldispenser,或cytenagmbh,germany的singlecellprinter–scp),单独挑选/排列所得的汇集/亚汇集克隆。
实施例4:用慢病毒克隆的hupex文库进行表型筛选。
使用hupex文库(诸如以上设计和构建的hupex文库)用以上描述的测定格式(无论是汇集的还是排列的文库)筛选表型改变。在进行此类筛选之前,可选地,可以将hupex文库与一个或更多个表达短肽的其他类似文库汇集。例如,hupex文库可以与表达人类小型开放阅读框(sorf)的文库,诸如在pct/gb2016/054038中(特别地在pct/gb2016/054038的实施例a)中)描述的sorf文库一起在池中筛选。
(1)汇集的6-硫鸟嘌呤抗性筛选
对化疗药物6-硫鸟嘌呤的抗性先前已被证明是一种相当严格的选择系统;一组狭窄的蛋白质能够介导该表型(参见wang等人2014,science343:80)。发明人试图使用该系统来证明本发明的文库如何在如此严格的条件下也可用于鉴定表型调节蛋白。
用汇集的hupex核酸文库转染hek293细胞,克隆到如上所述的慢病毒载体中,所述慢病毒载体被设计成表达多于一种短表达肽(sep)。收获病毒,滴定,并用表达sep的病毒感染一批kbm7细胞。随后将病毒转导的kbm7细胞文库暴露于一定浓度的6-硫鸟嘌呤,该浓度通过实验被确定为杀死99.999%的kbm7细胞。将携带表达诱导抗性的sep的插入物的幸存者从池中分离,扩增并收获基因组dna。使用pcr扩增表达诱导抗性的sep的插入物,并进行下一代测序。在对数据进行生物信息学分析后,鉴定出介导对6-硫鸟嘌呤的抗性并可能对错配修复过程起作用的sep(图1)。
(2)汇集的pten合成致死性筛选
为了发现能够选择性地抑制增殖或杀死缺乏pten肿瘤抑制因子的细胞的sep,在同基因细胞模型对(mcf10awt和mcf10apten敲除)中筛选sep。
如以上(1)所述,用表达sep的hupex文库感染细胞池。还以类似的方式用bugpex和omepex文库感染细胞。在所有条件下,平行地感染靶细胞系mcf10a和mcf10aptenko。将细胞以低密度铺板,以允许细胞在五天的时间段生长。然后将来自任一细胞群体的样品提交给ngs,如(1)中所述。比较野生型对照对照组(mcf10a)和pten敲除组(mcf10aptenko)中sep序列的相对丰度,并鉴定敲除细胞中耗尽的sep(图2)。
然后在相同的模型中,通过使用另外的对照重复初级测定来验证鉴定的命中。如果在三次生物重复中,命中在mcf10aptenko细胞中显示与mcf10a细胞相比细胞生长显著减少,则认为命中是有效的。图3显示示例性经验证的命中为hupex序列#30-325,一个来自人类四次穿膜蛋白-3的46个氨基酸的序列(其序列连同两个前导的两个氨基酸“mg”一起显示为seqidno.15,如由pmost25表达的),与对照细胞系相比,其在pten敲除细胞中抑制>60%的生长,达到与阳性对照(针对nlk的shrna,先前描述为对pten敲除的合成致死性。mendes-pereira等人,plosone2012)相当的量。合成的表达此类肽的寡核苷酸的核酸序列显示在seqidno.16中。
实施例5:本发明的核酸文库的设计和构建,所述文库基于在演化多样性微生物群的蛋白质组(“bugpex”)中包含的蛋白质的氨基酸序列编码短肽。
设计并构建了本发明的文库,其中来源蛋白质的氨基酸序列是来自多于一种不同物种的天然存在的蛋白质的氨基酸序列;在这个实施例中,是微生物演化多样性集合的参考蛋白质组中包含的蛋白质序列。
首先,如实施例1中所述,但通过使用表a中列出的参考蛋白质组的集合中包含的所有蛋白质序列,生成了巨型蛋白质氨基酸序列。实施例1的第二、第三和第四步骤通过类比进行,以生成数十万个独特的46个氨基酸长的序列的过滤的集合。
然而,在本例中,实施例1的第五步骤被替换为另一个过滤步骤,以从这数十万个氨基酸序列的列表中选择500,000个被预测为最不可能具有无序区段的此类序列。例如,程序disembl(linding等人2003,structure11:1453;http://dis.embl.de)可以用来考虑和分选环/卷曲、热环(hot-loops)和remark-465的三个内在无序的蛋白质参数。对给定肽中无序链段(stretch)中存在的氨基酸的所得数目进行计数,并将此类计数对这三个参数中的每一个进行排序。然后,通过所有三个排序参数的平均值对肽序列进行排序,使得被预测为高度无序的肽被排列在列表的底部。可选地,具有长无序区段的肽可以使用slider(具有长内在无序区域的蛋白质的超快速预测器;peng等人,2014;proteins:structure,functionandbioinformatics82:145;http://biomine.cs.vcu.edu/servers/slider)使用默认参数预测。
过滤的集合中所得的600,000个氨基酸序列如实施例1第六步中所述被反向翻译成核酸序列,如实施例1第七步所述使用替代密码子来避免不期望的密码子组合。估计有80,000种天然存在的蛋白质由这600,000种肽所代表,这表明平均覆盖度为每种天然存在的蛋白质约7.5个核酸(肽)。
对于包含这600,000个核酸序列的寡核苷酸的合成,按照在实施例2中描述的程序,除了在本例中所得的寡核苷酸(具有5’和3’区域)的一般结构为:
forward-amp_bamhi_val_variable-region_xhoi_reverse-amp,
其中“val”代表单个缬氨酸氨基酸接头的密码子,并且其他特征如实施例2所述,使得指示性的46个氨基酸的序列(seqidno.14)为:
所得的寡核苷酸序列具有如下所示的完整核酸序列(seqidno.9),其中下列特征标记如下:编码上述46个氨基酸的序列的138bp区域加粗;正向和反向扩增序列为小写;限制酶位点加框。
seqidno.9:指示性寡核苷酸的核酸序列
注意,在本例中,寡核苷酸不包含kozak/起始或终止密码子,在该实施例中,kozak/起始或终止密码子由寡核苷酸克隆到的表达载体提供,如下所述。
如实施例3的第一步和第二步所述,扩增和消化寡核苷酸。然而,在本例中,将所得的消化产物克隆到包含kozak/起始和终止密码子的bamhi/xhoi消化的慢病毒载体(例如pmost25a)的样品中。pmost25a的克隆位点的六十(60)个碱基对在以下由seqidno.10示出,并且带有本实施例5所示的指示性寡核苷酸的bamhi/xhoi消化的扩增产物的所得重组构建体,将具有由seqidno.11所示的序列。bamhi和xhoi限制位点加框;起始/终止密码子的第一个碱基上方标有“*”;并且编码46个氨基酸的序列的138bp区域加粗。
seqidno.10:pmost25a慢病毒载体的克隆位点的60bp。
seqidno.11:指示性重组构建体。
seqidno.11中所示的构建体的表达将产生具有seqidno.12所示的序列的52个氨基酸的肽,其中最初编码的46个氨基酸的指示性肽的序列以粗体显示在最初的甲硫氨酸、由bamhi位点编码的两个氨基酸和连接缬氨酸之后,随后是由xhoi位点编码的两个氨基酸。
seqidno.12:指示性表达肽。
实施例6:本发明的核酸文库的设计和构建,所述文库基于在演化多样性生物体的蛋白质组(“omepex”)中包含的蛋白质的氨基酸序列编码短肽。
设计和构建本发明的另一个文库,其中来源蛋白质的氨基酸序列是在更演化多样的物种集合的参考蛋白质组中包含的天然存在的蛋白质的氨基酸序列,所述更演化多样的物种集合包括来自这些的每一种的许多物种:古细菌;细菌、真菌、无脊椎动物、植物、原生动物、哺乳动物和非哺乳类脊椎动物。
首先,如实施例1中所述,但通过使用表b中列出的467个参考蛋白质组中包含的所有蛋白质序列,生成了巨型蛋白质氨基酸序列。实施例1的第二和第三步骤通过类比进行,以生成超过100万个独特的46个氨基酸长的序列的预过滤的集合。
这个预过滤的序列的集合使用cd-hit(fu等人2012,bioinformatics28:3150;http://weizhongli-lab.org/cd-hit/)的迭代运行用更严格的阈值进行分级聚类,以获得所得肽的最大多样性,简述如下:进行了三轮聚类;第一轮使用80%序列相似性阈值参数(“-c0.8”;示例命令:cdhit-ipeptides.faa-opeptides_0.8.faa-c0.8-t0-m32000-n5);第二轮使用第一轮的输出,使用60%的序列相似性阈值(“-c0.6”);并且第三次也是最后一次运行使用第二轮的输出,使用50%的序列相似性阈值(“-c0.5”)。
如实施例1的步骤4所述,预测剩余的每个所得肽序列的等电点(pi),并且还从该集合去除具有预测pi在6和8之间的任何肽序列。
类似于实施例2中所述,进一步过滤所得的氨基酸序列集合,以去除那些被预测具有内在无序区域的氨基酸序列。然而,在本例中,选择前475,000个序列(即那些被最少预测包含内在无序区域的序列)来形成独特的46个氨基酸的序列的第一集合。
另外,除了首先串联的天然存在的蛋白质的氨基酸序列是那些具有已知三维结构的蛋白质之外,25,000个独特的46个氨基酸的序列的第二集合使用实施例6中上述描述的程序生成。在本例中,约10,150个多肽链存在于蛋白质数据库(https://www.wwpdb.org,2017年2月5日)中,并且具有pfam注释(http://pfam.xfam.org,30.0版)。
将氨基酸序列的第一集合和第二集合合并,并如实施例5所述设计并合成500,000个寡核苷酸(具有相同的一般结构),每个寡核苷酸编码单独的氨基酸序列。如实施例5所述,将所得的合成的寡核苷酸进行pcr扩增,bamhi/xhoi消化并克隆到pmost25a中,以形成本发明的表达文库。
实施例7[预见性]:本发明的核酸文库的设计和构建,所述文库编码基于差异表达蛋白质(“diffpex”)的短肽。
在这个实施例中,进行基因表达数据库(例如embl-ebi表达图谱;geenhttp://www.ebi.ac.uk/gxa/home/)的查询来鉴定蛋白质组中在两种组织类型之间差异表达的基因的子集。例如,在人类黑色素瘤细胞系(或患者样品)和可比较但非癌性的人类细胞系之间的5000个差异表达最大的蛋白质通过这样的查询来鉴定。
这组5,000个蛋白质的参考氨基酸序列用于生成超过20,000个独特的46个氨基酸的序列的过滤的集合,并且如实施例5所述地,设计、合成超过20,000个寡核苷酸(具有相同的一般结构)并克隆到pmost25a中,每个寡核苷酸编码单独的氨基酸序列;除了在这种情况下:(1)沿每个蛋白质氨基酸序列的窗口间距小于10个氨基酸,以增加氨基酸序列跨这些天然存在的蛋白质序列的平铺密度;(2)不使用slider程序(用于预测和过滤掉非结构化区域)。
实施例8[预见性]:由本发明的核酸文库编码的肽文库的生成。
如下生成本发明的肽文库(例如,由本发明的核酸库编码的肽文库)。
首先,代替地,将实施例7的扩增和bamhi/xhoi消化的寡核苷酸克隆到顺式展示构建体(odegrip等人2003;pnas101:2806)中,具有以下一般设计:
promoter_nucleic_acidlibrary_repa_cis_ori
顺式展示利用了dna复制起始蛋白(repa)与表达它的模板dna排他性地结合的能力,此类特性被称为顺式活性。肽文库通过将本发明的核酸连接到编码repa的dna片段而产生。在体外转录和翻译后,形成了蛋白质-dna复合物的池,其中每个蛋白与编码它的dna稳定地缔合。这些复合物适合于配体对感兴趣的靶的亲和选择。
顺式展示利用了一组细菌质粒dna复制起始蛋白所表现出的高保真顺式活性,所述细菌质粒dna复制起始蛋白以r1质粒的repa为代表(nikoletti等人1988,j.bacteriol.170:1311)。在这种情况下,顺式活性指的是repa家族蛋白质排他性地与表达它们的模板dna结合的特性。r1质粒复制通过repa与质粒复制起点(ori)的结合而启动。ori通过被称为cis的dna元件与repa编码序列分开。此类元件被认为对于控制repa顺式活性是至关重要的(masai&arai1988,nucleicacidsres.16:6493)。顺式活性的共识模型是,包含依赖rho的转录终止子的cis元件导致宿主rna聚合酶停滞。此类延迟允许正在进行翻译的核糖体产生的新生repa多肽瞬时结合到cis,继而指导蛋白质结合到邻近的ori位点(praszkier&pittard1999,j.bacteriol.181:2765)。
通过将肽文库遗传融合到repa蛋白的n-末端,我们可以实现肽与编码肽的dna分子的直接连接;因此,基因型与表型之间的联系(这是展示技术的共同特征)得以确立。
如odegrip等人(2003)所描述的,肽文库通过使用大肠杆菌(e.coli)裂解物系统的体外转录和翻译生成,并且还如odegrip等人(2003)所描述的,可以对与固定的靶结合的肽(以及因此编码它们的dna序列)进行固相选择(例如,通过一轮或更多轮选择)。
实施例9:用慢病毒克隆的hupex文库进行表型筛选,以及结合sep的靶的鉴定。
利用表型筛选,本发明人能够从上文描述的hupex文库鉴定增加了用甲基硝基亚硝基胍(mnng)处理的hela细胞的存活的短表达肽(sep),甲基硝基亚硝基胍(mnng)是通过parthanatos引起细胞死亡的诱导物。parthanatos是一种依赖parp-1的程序性细胞死亡形式(yu等人,2006;pnas103:2653),其在神经元细胞死亡中起作用,并与包括帕金森氏病、中风、心脏病发作和糖尿病的疾病相关。
用汇集的hupex核酸文库转染hek293细胞,克隆到如上所述的慢病毒载体中,所述慢病毒载体被设计成表达多于一种sep。收获病毒,滴定,并用表达sep的病毒感染一批hela细胞并持续8天进行选择。然后将病毒转导的hela细胞文库暴露(“d0”时间点)于接近致死剂量(6.7um)的mnng。该剂量被确定为与parp-1抑制剂olarparib共同孵育仍能挽救hela细胞免于parthanatos/细胞死亡的最大剂量。
从在d0没有进行mnng处理的此类hela细胞的对照等分试样提取基因组dna,并且进行大规模扩增子dna测序以确定hupexsep编码核酸的相对丰度,并且从在6.7ummnng的存在下培养8天后(“d8”)的此类hela细胞的第二等分试样再次提取基因组dna(图4),以便从hupex文库鉴定那些在d8显示出与d0相比增加的相对丰度的表达的sep,并且因此增加了在mnng的存在下的hela细胞的存活。
在hupex文库中代表的预测的300,000个不同的编码sep的插入物中,几乎288,000个在d0展示至少一个成员(如通过扩增子dna测序发现的);并且通过在d8显示相对丰度的显著增加,从hupex文库鉴定出增加了在mnng的存在下的hela细胞的存活的至少72个sep(图5)。
在这些sep中,许多sep被确定为与解毒机制相关的天然存在的蛋白质的片段。例如,kegg途径分析(http://www.kegg.jp/kegg/pathway.html)确定了某些此类sep是参与“化学致癌作用”和“细胞色素p450对异生物质的代谢”的天然存在的蛋白质的片段。
在酵母双杂交筛选技术中,某些已鉴定的sep被用作“诱饵”(针对作为猎物的人类cdna文库),以鉴定hela细胞中hupexsep将结合的蛋白质靶。
实施例10:用慢病毒克隆的bugpex和omepex文库进行表型筛选。
与实施例9中描述的类似,在parthanatos的表型筛选中筛选bugpex和omepex文库,以鉴定从这些文库中的每一个文库表达的sep,与d0相比,sep在d8显示出增加的相对丰度,并且因此能够增加在诱导parthanatos的mnng的存在下的hela细胞的存活。
在bugpex文库中代表的600,000个不同的sep编码插入物中,几乎510,000个sep在d0代表至少一个成员(如通过扩增子dna测序发现的);并且通过在d8显示其相对丰度的显著增加,从bugpex文库鉴定出增加了在mnng的存在下的hela细胞的存活的至少58个sep(图6)。
在omepex文库中代表的500,000个不同的sep编码插入物中,几乎490,000个在d0代表至少一个成员(如通过扩增子dna测序发现的);并且通过在d8显示其相对丰度的显著增加,从omepex文库鉴定出增加了在mnng的存在下的hela细胞的存活的至少64个sep(图7)。
实施例11:用慢病毒克隆的-pex文库进行表型筛选。
利用表型筛选,本发明人能够从组合的上文描述的hupex(hpx)、bugpex(bpx)和omepex(opx)文库鉴定出短表达肽(sep),所述短表达肽(sep)降低了经工程化以表达gfp-lc3/rfp-lc3dg自噬通量报告子的hek293ft细胞(afr细胞,kaizuka等人.molecularcell2016)中的gfp-lc3。
将hek293ft细胞用汇集的hupex、bugpex和omepex核酸文库转染,克隆到本文所述的慢病毒载体中,例如seqidno.10所示的被设计为表达多于一种sep的pmost25a。收获病毒,滴定,并用表达sep的病毒感染一批hek293ft-afr细胞并持续4天进行选择,并且然后将表达sep的细胞再扩增2天而不进行选择。然后通过流式细胞术评估病毒转导的hek293ft-afr细胞的文库,与未分选的对照相比,富集在低gfp-lc3门的sep转导的hek293ft-afr细胞被流式分选,并且如前述实施例所述,肽序列被扩增并送至ngs分析,即扩增子dna测序。图8a-图8c显示了与对照相比的所选命中(标记区域)的群体。
将被鉴定为富集在低gfp-lc3门的sep序列克隆到合适的慢病毒表达载体和在hek293ft细胞中生成的表达sep的病毒中。在上述条件中,通过流式细胞术单独评估每个sep表达病毒群体。将每一个表达sep的细胞群体与对照的表达sep的细胞群体或用torin1(一种自噬通量的诱导剂)处理的未感染的hek293ft-afr细胞进行评估比较(图9)。显示了一系列候选物,其中bpx-497507代表一种强有力的命中,能够诱导自噬,如通过gfp-lc3减少来测量的。torin1(250nm)显示为阳性对照。
序列表
<110>福慕斯特有限公司
<120>核酸文库、肽文库及它们的用途
<130>p9328wo2
<160>16
<170>patentinversion3.5
<210>1
<210>1
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>实施例1中使用的正向扩增序列
<400>1
tgccacctgacgtctaagaa20
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>实施例1中使用的反向扩增序列
<400>2
gctcactcaaaggcggtaat20
<210>3
<211>199
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>实施例2中所示的指示性寡核苷酸
<220>
<221>misc_feature
<222>(1)..(20)
<223>正向扩增序列
<220>
<221>misc_feature
<222>(21)..(26)
<223>限制酶位点
<220>
<221>misc_feature
<222>(25)..(30)
<223>kozak序列
<220>
<221>misc_feature
<222>(33)..(170)
<223>编码区
<220>
<221>misc_feature
<222>(174)..(179)
<223>限制酶位点
<220>
<221>misc_feature
<222>(180)..(199)
<223>反向扩增序列
<400>3
tgccacctgacgtctaagaaggatccatgggactcgcccagaccgcctgtgtggtgggca60
ggcccggcccccaccccacccagttcctcgccgccaaggaaaggaccaagagccacgtgc120
ccagcctcctcgacgccgacgtggaaggccagagcagggactacaccgtgtaactcgagg180
ctcactcaaaggcggtaat199
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>实施例3中使用的正向引物
<400>4
tgccacctgacgtctaagaa20
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>实施例3中使用的反向引物
<400>5
attaccgcctttgagtgagc20
<210>6
<211>60
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>pmost25慢病毒载体的克隆位点
<220>
<221>misc_feature
<222>(20)..(25)
<223>限制酶位点
<220>
<221>misc_feature
<222>(38)..(43)
<223>限制酶位点
<400>6
agtagcatcgcattagccgggatccagcgctgctaccctcgagtaagtgactaggcaatc60
<210>7
<211>195
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>实施例3中所示的指示性重组构建体
<220>
<221>misc_feature
<222>(20)..(25)
<223>限制酶位点
<220>
<221>misc_feature
<222>(24)..(29)
<223>kozak序列
<220>
<221>misc_feature
<222>(32)..(169)
<223>编码区
<220>
<221>misc_feature
<222>(173)..(178)
<223>限制酶位点
<400>7
agtagcatcgcattagccgggatccatgggactcgcccagaccgcctgtgtggtgggcag60
gcccggcccccaccccacccagttcctcgccgccaaggaaaggaccaagagccacgtgcc120
cagcctcctcgacgccgacgtggaaggccagagcagggactacaccgtgtaactcgagta180
agtgactaggcaatc195
<210>8
<211>48
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>实施例3中所示的指示性表达肽
<400>8
metglyleualaglnthralacysvalvalglyargproglyprohis
151015
prothrglnpheleualaalalysgluargthrlysserhisvalpro
202530
serleuleuaspalaaspvalgluglyglnserargasptyrthrval
354045
<210>9
<211>193
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>实施例5中所示的指示性寡核苷酸
<220>
<221>misc_feature
<222>(1)..(20)
<223>正向扩增序列
<220>
<221>misc_feature
<222>(21)..(26)
<223>限制酶位点
<220>
<221>misc_feature
<222>(30)..(167)
<223>编码区
<220>
<221>misc_feature
<222>(168)..(173)
<223>限制酶位点
<220>
<221>misc_feature
<222>(174)..(193)
<223>反向扩增序列
<400>9
tgccacctgacgtctaagaaggatccgtgcctaggtacctgaagggctggctgaaggacg60
tggtgcagctgagcctgaggaggcctagcttcagggccagcaggcagaggcctatcatca120
gcctgaacgagaggattctggagttcaacaagaggaacatcacagccctcgaggctcact180
caaaggcggtaat193
<210>10
<211>60
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>pmost25a慢病毒载体的克隆位点
<220>
<221>misc_feature
<222>(7)..(12)
<223>kozak序列
<220>
<221>misc_feature
<222>(12)..(17)
<223>限制酶位点
<220>
<221>misc_feature
<222>(30)..(35)
<223>限制酶位点
<400>10
cattagccatgggatccagcgctgctaccctcgagtaagtgactaggcaatctaatctat60
<210>11
<211>187
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>实施例5中所示的指示性重组构建体
<220>
<221>misc_feature
<222>(7)..(12)
<223>kozak序列
<220>
<221>misc_feature
<222>(12)..(17)
<223>限制酶位点
<220>
<221>misc_feature
<222>(21)..(158)
<223>编码区
<220>
<221>misc_feature
<222>(159)..(164)
<223>限制酶位点
<400>11
cattagccatgggatccgtgcctaggtacctgaagggctggctgaaggacgtggtgcagc60
tgagcctgaggaggcctagcttcagggccagcaggcagaggcctatcatcagcctgaacg120
agaggattctggagttcaacaagaggaacatcacagccctcgagtaagtgactaggcaat180
ctaatct187
<210>12
<211>52
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>实施例5中所示的指示性表达肽
<400>12
metglyservalproargtyrleulysglytrpleulysaspvalval
151015
glnleuserleuargargproserpheargalaserargglnargpro
202530
ileileserleuasngluargileleuglupheasnlysargasnile
354045
thralaleuglu
50
<210>13
<211>46
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>实施例2中描述的指示性肽
<400>13
leualaglnthralacysvalvalglyargproglyprohisprothr
151015
glnpheleualaalalysgluargthrlysserhisvalproserleu
202530
leuaspalaaspvalgluglyglnserargasptyrthrval
354045
<210>14
<211>46
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>实施例5中描述的指示性肽
<400>14
proargtyrleulysglytrpleulysaspvalvalglnleuserleu
151015
argargproserpheargalaserargglnargproileileserleu
202530
asngluargileleuglupheasnlysargasnilethrala
354045
<210>15
<211>48
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>来自实施例4pten合成致死性筛选的#30-325命中肽
<400>15
metglyvalvalileilealavalglyalaleuleupheileilegly
151015
leuileglycyscysalathrilearggluserargcysglyleuala
202530
thrphevalileileleuleuleuvalphevalthrgluvalvalval
354045
<210>16
<211>199
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>编码#30-325命中肽的寡核苷酸
<400>16
tgccacctgacgtctaagaaggatccatgggagtggtgattattgccgtgggcgccctcc60
tcttcattattggcctcattggctgttgtgccaccattagggaaagcaggtgtggcctcg120
ccaccttcgtgattattctcctcctcgtgttcgtgaccgaagtggtggtgtaactcgagg180
ctcactcaaaggcggtaat199
- 还没有人留言评论。精彩留言会获得点赞!