控制玉米株高和花期的基因及其应用的制作方法
本发明属于分子遗传学领域。具体涉及控制玉米株高和花期的基因及其在改变玉米株高或花期性状方面的应用。本发明提供了一个控制玉米株高和花期基因的序列,且公开了利用基因工程手段突变基因以降低玉米株高或延迟花期的方法。
背景技术:
矮化性状带来了作物产量的突破,降低作物株高的方法具有极大的应用潜力。在玉米的主要农艺性状中,株高影响到玉米的抗倒性、光合效能、收获指数,与玉米产量密切相关。因此,在玉米育种实践与种质资源改良工作中,株高性状具有重要的价值。
由于株高受主效基因和微效多基因的共同控制,表现为典型的数量性状遗传。尽管有一些调控玉米株高的基因被定位与克隆(华南农业大学.玉米zmpif3s突变型蛋白、其编码基因及其在育种上的应用:cn201910273522.1[p].2019-08-02.;杭州瑞丰生物科技有限公司.cyp78a基因在增加玉米株高和增强植株长势中的应用:cn201510230547.5[p].2016-12-07.;中国农业大学.与玉米株高相关基因及其编码蛋白与应用:cn200410037404.4[p].2005-01-26.;中国农业科学院作物科学研究所.利用基因编辑技术创制玉米矮化材料的方法:cn201910371358.8[p].2019-08-16.),更多的株高性状相关基因有待进一步克隆。
在高等植物中,开花代表着从营养生长到生殖生长的转换过程,其在植物体的整个生长发育阶段中具有重要的作用。而何时开花这一生物性状则受到植物体自身遗传因子及外界环境因素的双重作用。在这双重作用的影响下,一系列开花诱导过程在高等植物中具有普遍的规律,即植物体叶片通过感受外界生长条件(光照,温度,湿度等),在适宜的时间产生成花物质(或叫开花素florigen),成花物质经过输导组织由叶片运送到茎尖生长点,刺激顶端分生组织成花。
开花期是作物进化和适应过程中的重要性状,理解作物开花期性状的遗传基础、克隆候选基因可以提高作物的环境适应能力和可塑性,这对培育适应不同生态区的优良作物品种具有重要的意义,同时也将促进产量等与开花期密切相关的重要生产性状的遗传改良进程。此外,作物的整体生育期主要决定于开花期的长短,适度缩短开花期能够减少整体的生育期,从而降低种植成本。
技术实现要素:
本发明的目的之一在于提供一个控制玉米株高和花期性状的基因的序列。
本发明的目的之二在于公开一种降低玉米株高或延迟花期性状的方法。
为实现上述目的,本发明采用如下技术方案:
本发明提供了一种控制玉米株高和花期的基因及其在改变玉米株高或花期性状中的应用,其特征在于:上述基因的核酸序列如seqidno.1-seqidno.4任意一个所示。其中,seqidno.1序列为玉米自交系b73中的基因组序列,seqidno.2序列为玉米自交系kn5585中的基因组序列,seqidno.3序列为玉米自交系b73中的基因编码区序列,seqidno.4序列为玉米自交系kn5585中的基因编码区序列。上述基因的核酸序列经过取代和/或缺失和/或添加一个或多个氨基酸,且具有相同功能的序列也属于本发明的内容。
在另一方面,本发明还提供了一种降低玉米株高或延迟花期的方法,其特征在于:抑制玉米中上述基因编码蛋白的表达和/或活性,选择玉米株高降低或花期延迟的植株。
在一些实施方案中,上述蛋白的氨基酸序列如seqidno.5或seqidno.6所示。其中,seqidno.5序列为玉米自交系b73中的基因编码蛋白序列,seqidno.6序列为玉米自交系kn5585中的基因编码蛋白序列。
在一些实施方案中,上述抑制蛋白表达和/或活性的方法包括基因编辑、rna干扰、t-dna插入、物理或化学诱变中任一种。
在一些实施方案中,上述基因编辑采用crispr/cas9方法。
在一些实施方案中,上述crispr/cas9方法在玉米中的基因组靶标区域的dna序列如seqidno.7或seqidno.8所示。
在另一方面,本发明还提供了一种用于降低玉米株高和花期的试剂盒,其特征在于:包括如下任一种:
(1)sgrna分子,其序列如seqidno.9或seqidno.10所示;
(2)所述sgrna的编码dna分子;
(3)表达所述sgrna的载体。
在另一方面,本发明还提供了用上述试剂盒得到的一种玉米突变基因型,其特征在于:所述突变基因型序列如seqidno.11-seqidno.16任意一个所示。
本发明的优点及有益效果如下:本发明利用多亲本高世代自交系群体定位到控制玉米株高性状的基因组区域,该区域并不存在已知的影响株高的基因,只存在一个未知功能的注释基因grmzm2g331652。利用crispr/cas9方法突变grmzm2g331652基因,证明该基因能够控制玉米株高性状,并意外发现该基因还能够控制玉米花期性状。利用crispr/cas9基因编辑的方法和编辑后的突变基因型序列,可以降低玉米株高或延迟花期,创制矮化或花期延迟玉米品种,从而提高玉米产量,降低倒伏率,提高种植效率,方便机械化收获,提高玉米的生态适应性。
附图说明
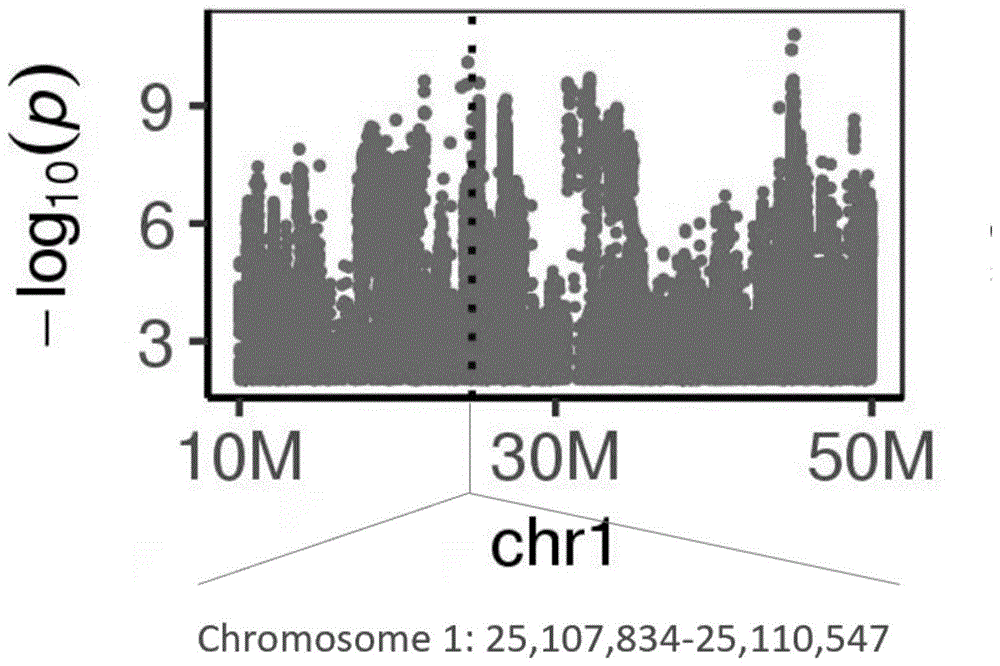
图1株高qtl定位结果。纵轴表示每个标记关联分析检验的p-value,取-log10,横轴表示染色体的位置。
图2基因编辑载体图。各元件英文及缩写含义列举如下:
图3利用crispr-cas9技术突变玉米基因后的株高和花期表现。左:突变前;右:突变后。
具体实施方式
提供以下定义和方法用以更好地界定本申请以及在本申请实践中指导本领域普通技术人员。除非另作说明,术语按照相关领域普通技术人员的常规用法理解。本文所引用的所有专利文献、学术论文、行业标准及其他公开出版物等,其中的全部内容整体并入本文作为参考。
如本文所用,“玉米”是任何玉米植物并包括可以与玉米育种的所有植物品种,包括整株植物、植物细胞、植物器官、植物原生质体、植物可以从中再生的植物细胞组织培养物、植物愈伤组织、植物或植物部分中完整的植物细胞,所述植物部分例如胚、花粉、胚珠、种子、叶、花、枝、果实、茎杆、根、根尖、花药等。除非另有所指,核酸以5’至3’方向从左向右书写;氨基酸序列以氨基至羧基方向从左向右书写。氨基酸在本文可以用其通常所知的三字母符号或iupac-iub生物化学命名委员会推荐的单字母符号来表示。同样地,可以用通常接受的单字母码表示核苷酸。数字范围包括限定该范围的数字。如本文所用,“核酸”包括涉及单链或双链形式的脱氧核糖核苷酸或核糖核苷酸多聚物,并且除非另有限制,包括具有天然核苷酸基本性质的已知类似物(例如,肽核酸),所述类似物以与天然存在的核苷酸类似的方式与单链核酸杂交。如本文所用,术语“编码”或“所编码的”用于特定核酸的上下文时,指该核酸包含指导该核苷酸序列翻译成特定蛋白的必需信息。使用密码子表示编码蛋白的信息。如本文所用,涉及特定多核苷酸或其所编码的蛋白的“全长序列”指具有天然(非合成)内源序列的整个核酸序列或整个氨基酸序列。全长多核苷酸编码该特定蛋白的全长、催化活性形式。本文可互换地使用术语“多肽”和“蛋白”,以指氨基酸残基的多聚物。该术语用于氨基酸多聚物,其中一个或多个氨基酸残基是相应天然存在的氨基酸的人工化学类似物。该术语还用于天然存在的氨基酸多聚物。本文可互换地使用术语“残基”或“氨基酸残基”或“氨基酸”,以指被并入蛋白、多肽或肽(统称“蛋白”)的氨基酸。氨基酸可以是天然存在的氨基酸,并且除非另有限制,可以包括天然氨基酸的已知类似物,所述类似物可以与天然存在的氨基酸相似的方式起作用。
如本文所用,可以互换地使用术语“分离的”和“纯化的”,以涉及核酸或多肽或其生物学活性部分,其基本上或本质上不含如在其天然存在的环境中所发现的通常伴随或反应于该核酸或多肽的组分。因而,用重组技术产生分离的或纯化的核酸或多肽时,分离的或纯化的核酸或多肽基本上不含其它细胞物质或培养基,或者化学合成分离的或纯化的核酸或多肽时,基本上不含化学前体或其它化学品。“分离的”核酸通常不含在该核酸所衍生自的生物体的基因组dna中天然侧翼于该核酸(即位于该核酸5’和3’端的序列)的序列(诸如,编码蛋白的序列)。例如,在各种实施方案中,所分离的核酸可以包含在该核酸所衍生自的细胞的基因组dna中天然侧翼于该核酸的少于约0.5kb的核苷酸序列。
在本申请中,将词语“包括”、“包含”或其变体应理解为除所描述的元素、数或步骤外,还包含其它元素、数或步骤。“受试植物”或“受试植物细胞”是指遗传改造已经生效的植物或植物细胞,或者如此改造的植物或细胞的子代细胞,该子代细胞包含所述改造。“对照”或“对照植物”或“对照植物细胞”提供用于测量受试植物或植物细胞表型改变的参考点。对照植物或植物细胞可以包括,例如:(a)野生型植物或细胞,即与遗传改造起始材料具有相同基因型的植物或细胞,所述遗传改造产生受试植物或细胞;(b)与所述起始材料具有相同基因型但已用空构建体(即用对目的性状无已知效果的构建体,诸如包含标物基因的构建体)转化的植物或植物细胞;(c)是受试植物或植物细胞的非转化分离子的植物或植物细胞;(d)与所述受试植物或植物细胞在遗传上一致但未暴露于会诱导目的基因表达的条件或刺激物的植物或植物细胞;或(e)受试植物或植物细胞自身,其处于目的基因不被表达的条件下。
本领域技术人员会容易地认同,诸如位点特异性诱变和随机诱变、聚合酶链式反应方法和蛋白工程化技术的分子生物学领域的进步提供了广泛的适当的工具和操作步骤,以用于改造或者工程化农业上感兴趣的蛋白的氨基酸序列和潜在的基因序列。
在一些实施方案中,可以对本申请的核苷酸序列进行改变,以进行保守氨基酸替换。保守氨基酸替换的原则和实例在下文中进一步描述。在某些实施方案中,可以依照公开的单子叶密码子偏好性对本申请的核苷酸序列进行不改变氨基酸序列的替换,例如可以用单子叶植物偏好的密码子替换编码同一氨基酸序列的密码子,而不改变该核苷酸序列所编码的氨基酸序列。在一些实施方案中,以编码同一氨基酸序列的不同密码子替换本申请中的部分核苷酸序列,从而在改变核苷酸序列的同时不改变其编码的氨基酸序列。保守变体包括由于遗传密码子简并性而编码实施方案的蛋白中的一种的氨基酸序列的那些序列。在一些实施方案中,根据单子叶植物偏好密码子替换本申请中的部分核苷酸序列。本领域技术人员会认识到氨基酸添加和/或取代通常基于氨基酸侧链取代基的相对相似性,例如,所述取代基的疏水性、电荷、大小等等。具有各种前述所考虑性质的示例性氨基酸取代基团为本领域技术人员所公知,并且包括精氨酸与赖氨酸;谷氨酸和天门冬氨酸;丝氨酸和苏氨酸;谷氨酰胺和天冬酰胺;以及缬氨酸、亮氨酸和异亮氨酸。关于不影响目的蛋白生物学活性的适当氨基酸取代的指南可以在dayhoff等人(1978)atlasofproteinsequenceandstructure(蛋白序列和结构图集)(natl.biomed.res.found.,washington,d.c)(通过引用并入本文)的模型中找到。可以进行诸如将一个氨基酸换作具有相似性质的另一个氨基酸的保守性取代。序列一致性的鉴定包括杂交技术。例如,将已知核苷酸序列的全部或部分用作与其它相应核苷酸序列选择性杂交的探针,所述其它相应核苷酸序列存在于来自所选生物体的已克隆基因组dna片段或cdna片段群(即基因组文库或cdna文库)。
在一些实施方案中,还包括核苷酸序列及其编码的氨基酸序列的片段。如本文所用,术语“片段”指实施方案的多核苷酸的核苷酸序列的一部分或者多肽的氨基酸序列的一部分。核苷酸序列的片段可以编码蛋白片段,所述蛋白片段保留天然或相应全长蛋白的生物学活性,并因而具有蛋白活性。突变体蛋白包括天然蛋白的生物活性片段,其包含保留天然蛋白生物学活性的连续氨基酸残基。一些实施方案还包括转化的植物细胞或转基因植物,其包含至少一种实施方案的核苷酸序列。在一些实施方案中,使用表达载体转化植物,所述表达载体包含至少一种实施方案的核苷酸序列以及与其可操作地连接的在植物细胞中驱动表达的启动子。转化的植物细胞和转基因植物表示基因组内包含异源多核苷酸的植物细胞或植物。一般来说,所述异源多核苷酸在转化的植物细胞或转基因植物的基因组内稳定地整合,以致将所述多核苷酸传递给后代。可以将所述异源多核苷酸单独地或作为表达载体的一部分整合进基因组。在一些实施方案中,本申请涉及的植物包括植物细胞、植物原生质体、可以再生出植物的植物细胞组织培养物、植物愈伤组织、植物团块和植物细胞,其为完整的植物或者植物的部分,诸如胚胎,花粉,胚珠,种子,叶,花,枝,果实,果仁,穗,穗轴,壳,秸秆,根,根尖,花药等等。本申请还包括源于本申请的转基因植物或其子代、并因而至少部分地包含本申请的核苷酸序列的植物细胞、原生质体、组织、愈伤组织、胚胎以及花、茎、果实、叶以及根。
以下实施例用于说明本发明,但不用来限制本发明的范围。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改或替换,均属于本申请的范围。若无特别指明,实施例按照常规实验条件,如sambrook等人的分子克隆实验手册(sambrookj&russelldw,molecularcloning:alaboratorymanual,2001),或按照制造厂商说明书建议的条件。若未特别指明,实施例中所用的化学试剂均为常规市售试剂,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。
实施例
实施例1玉米株高qtl的定位过程
1、群体构建
本发明将24个中国的优良玉米自交系,通过两代双列杂交、六代开放授粉及六代自交的方式获得了1404个子代组成的多亲本高世代自交系群体(cubic)。这24个中国优良玉米自交系包括lv28(旅28)、e28、dan340(丹340)、f349、zi330(自330)、zong3(综3)、zong31(综31)、huangc(黄c)、hzs(黄早四)、hys(黄野四)、ty4(天涯4)、yuangfh(原辅黄)、chang7-2(昌7-2)、k12、xi502(西502)、lx9801、h21、shuang741(双741)、q1261、ji853(吉853)、ji53(冀53)、5237、81515、nx110(农系110),该群体的24个亲本及1404子代自交系全部进行基因组测序,获得了超过14m的snp和indel变异。
2、表型分析
所有24个亲本和1404子代自交系在黄淮海及东北5个地点种植,表型变异丰富。所考查农艺表型包括植株高度、花期、开花期、穗重等产量性状等。将所有环境重复计算出每个自交系的最佳线性无偏预测因子(blup)值用于后续分析,包括基本表型统计、相关性分析和gwas。通过线性回归估算由给定性状的qtl解释的表型方差或遗传力,并联合估算解释的总方差。
3、基于单变量的gwas分析(sgwas)
根据最小等位基因频率(maf)过滤后,共计1180万个高质量snp用于下游分析。前十个主成分(pc)共解释了8.76%的遗传方差,同时整合了亲缘关系k矩阵作为随机效应一起用于sgwas分析。关联的显着性阈值设置为1.23e-8,等于0.05/ne,其中ne是根据所有变异计算的独立检验的有效数量。确定qtl区间的标准为:每个性状的显著snp被首先提取,只保留至少两个连续snp小于20kb的位点同时合并作为基本区间单元,20kb以内只有单独显著位点的snp在下一步分析中被排除;如果相邻区间单元之间的任何显著snp有较高连锁不平衡关系(ld,r2≥0.2),则将相邻区间单元进一步合并为qtl候选区间。为了减少由较小的qtl效应而获得的qtl间隔较小而未包含功能基因的可能性,对于那些间隔小于50kb且最显著snp的显著性低于阈值100倍的qtl(即1.23e-10),将对qtl区间向两侧继续延伸25kb获得最终qtl区间。将区间内最显著的snp作为sqtl的显著性,将区间间隔内所有基因作为候选基因。
4、获得qtl区间
利用上述的定位方法,本发明找到了玉米株高性状的1个最显著snp,位于第1染色体25107834-25110547区间内(图1)。该区间内经maizegdb数据库(https://www.maizegdb.org/)的查询包含1个有注释的基因,编号为grmzm2g331652,数据库中并未明确该基因的功能。该基因的基因组序列如seqidno.1所示,全长2714bp,编码区序列如seqidno.3所示,编码的氨基酸序列如seqidno.5所示。本发明同时测定了未米生物科技(江苏)有限公司选育的玉米自交系kn5585中该区间的序列,如seqidno.2所示。编码区序列如seqidno.4所示,编码的氨基酸序列如seqidno.6所示。seqidno.1和seqidno.2序列间的相似性超过99%。
实施例2基因编辑敲除候选基因分析基因功能
本发明利用crispr-cas9基因编辑技术对上述区间内的基因进行定点突变。实施方式包括基因编辑载体的构建、玉米的遗传转化以及编辑效果的功能验证。
具体如下:
1.基因编辑载体的构建
本发明的基因编辑载体为g01652-cpb-zmubi-hspcas9,其载体图如图2所示。该载体的基础载体为cpb-zmubi-hspcas9。本发明通过overlappcr获得双靶u6-sgrna进而通过同源重组克隆到基础载体中,具体的构建流程如下:
(1)u6启动子的克隆。从b73中克隆u6启动子。
(2)靶标grna的设计。将受体材料kn5585基因组序列输入http://cbi.hzau.edu.cn/crispr/进行靶标设计。本发明优选的两个靶标区域的dna序列如seqidno.7或seqidno.8所示。本发明的sgrna骨架序列通过人工合成得到,其序列信息如seqidno.9和seqidno.10所示。
(3)通过overlappcr获得u6-sgrna。引物对pu6f1/pu6r用于扩增第一个靶标的u6启动子,产物长度为515bp;引物对p1652-1f(3f)/pgrr1用于扩增第一个靶标的sgrna,产物长度为127bp;引物对pu6f1/grr1用于进行overlappcr第2步扩增(u6-sgrna),产物长度为634bp。overlappcr第1步分别扩增u6和sgrna,pcr产物分别稀释50倍后混合作为模板进行overlappcr第2步扩增。扩增产物电泳切胶回收并测序确认序列。overlappcr体系和条件如下:overlappcr第1步的15μl的反应体系如下,模板dna(u6或sgrna,≥30ng/μl):0.5μl,primerf/r:各1.2μl,灭菌ddh2o:3.7μl,2×phantamaxbuffer:7.5μl,dntpmix:0.6μl,phanta酶(产品编号:p505-d1/d2/d3):0.3μl。overlappcr第2步的反应体系为30μl体系。u6吸1μl,加49μlddh2o稀释;sgrna吸1μl,加49μlddh2o稀释各吸10μl,混匀。具体如下:混合模板dna(u6+sgrna):1.5μl,primerf/r:各2.4μl,灭菌ddh2o:6.9μl,2×phantamaxbuffer:15μl,dntpmix:1.2μl,phanta酶:0.6μl。overlappcr程序如下:(1)94℃5分钟,(2)94℃30秒,(3)62℃35秒,(4)72℃30秒,第(5)步是从(2)步-(4)步循环32次,(6)72℃10分钟,(7)25℃5分钟。载体构建所需的引物序列见表1。
表1载体构建所需的引物序列
(4)通过重组克隆构建到骨架载体。将cpb-ubi-hspcas9载体用hindiii消化,回收。通过同源重组将u6-grna和载体二者连接。配反应液前保证各overlap产物浓度接近一致,20μl的同源重组体系如下:cashindiii:3μl,t-1foverlap:1μl,灭菌ddh2o:10μl,5×cemultisbuffer:4μl,exnasemultis(产品编号:c113-01/02):2μl。
2.玉米遗传转化
将载体通过电击法转入农杆菌eha105中,pcr进行鉴定。以新鲜剥离的1mm左右的玉米自交系kn5585(未米生物科技(江苏)有限公司选育的自交系)的幼胚为材料,将剥取的玉米胚放入含有1.8ml悬浮液的2ml塑料离心管中,30min内大约处理未成熟幼胚150个;吸去悬浮液,余下玉米胚在管中然后加入1.0ml农杆菌悬浮液,放置5min。将离心管中的幼胚悬浮后倒入共培养基上,并用移液器吸去表面多余的农杆菌菌液,于23℃黑暗共培养3天。共培养后,将幼胚转移到休息培养基中,于28℃黑暗培养6天后,放至含5mg/lbialaphos的筛选培养基上,开始筛选培养2周,然后转到含8mg/lbialaphos筛选培养基上筛选培养2周。将抗性愈伤组织转移至分化培养基1中,25℃,5000lx,光照培养1周。再将愈伤转移至分化培养基2中,光照培养2周;将分化生出的小苗转移至生根培养基上,25℃,5000lx,光照培养直到生根;将小苗转入小盆中生长,一定生长阶段后移栽于温室中,3-4个月后收获后代种子。
3.基因编辑植株的性状鉴定
对t1代材料提取苗期dna检测基因编辑情况,设计引物(引物序列为hr2-2f:5’-agcgcgtcttcgcacat-3’,hr2-1r:5’-cttgtagaactgcccactgc-3’)扩增靶标编辑区段,扩增体系为:dna:3μl,双向引物各1μl,2×taqmix:7.5μl,ddh2o:2.5μl,总体积10μl。pcr反应条件如下:(1)94℃5分钟,(2)94℃40秒,(3)57℃30秒,(4)72℃60秒,(5)从(2)步-(4)步循环35次,(6)72℃7分钟,(7)4℃保存。pcr产物交由武汉擎科生物科技有限公司进行sanger测序。将转化株测序结果与野生型kn5585的基因组比对,发生碱基替换、插入或缺失的材料即为阳性编辑材料,否则为阴性材料。
pcr扩增靶标编辑区段并对pcr产物进行测序。将pcr产物序列与原始的kn5585中的序列(seqidno.2)进行比对,发现有2个独立转化事件a1和a2的扩增产物(序列如seqidno.15和seqidno.16所示)在基因靶点处发生了编辑,为阳性植株,突变后的靶点处基因型序列如seqidno.11-seqidno.14所示。调查阳性转化事件t1代材料授粉后期的株高,发现阳性突变体和受体kn5585之间的株高具有显著差异(表2),证明该基因控制株高性状,基因突变后株高降低。
此外,本发明意外发现阳性突变体和受体kn5585之间的花期性状也存在显著差异(表2),表明该基因也控制花期性状。基因突变后花期延迟。
表2基因编辑玉米材料的株高和花期性状数据
数据以“平均值±标准差”表示,不同字母表示各材料在p=0.05水平的显著性差异。“-”表示碱基缺失;粗体表示碱基插入或变异,方框表示pam序列。
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
序列表
<110>未米生物科技(江苏)有限公司,华中农业大学
<120>控制玉米株高和花期的基因及其应用
<130>1
<160>16
<170>siposequencelisting1.0
<210>1
<211>2714
<212>dna
<213>zeamaysl.
<400>1
atgcgagcattcaacggccgccgcctccacgatccgaagcccccctcattcctccccctc60
cccttagccccacgcaccacaacgccgccaccgccgcccccctcctcgccgccgcacgcc120
atcccgccgctcaacttccgctacctcaccccctgtccgcgctggtctgcctgggtcgcc180
accgcactcagcgaccccgttttcgctcccatcctcgcctcgtccgccatctccggcgcc240
gtcgccgcctccaccgccgtcgtctcccccgaccgtgcggccctctccgctctgctctcg300
ctctgggacccggatacccacgcgttccgtctcccagcgggccccgccaccttcacactc360
gaggatgcgctcgtcctcgctggtctaccgcccgcaggcgcgccgctggaccggaccctc420
acgccagaggaggatgacctccgcgttcgcctcgttgtcgagagggagaagatcaaggag480
ctccacccctgtgcccgcgccgcgcgccgcgtgtctgcggaggtgtggctcgagtggttc540
gacggcggtggcatacgccccggcgaggacgacgaactgcggtggcttgggttcctcgct600
tactggctcgctttttttgttacgccaaggctccggtcaaggggtggcgagctgccggag660
cgcgtcttcgcacatgctgcgaggcttagtcttggcgagcgcattgcccttggccagggg720
atggtggcaaatctatatgcggaaatggataagattgttacttgcactgtggccgatggt780
gtgtgtggccgtctagatgtctgcgtgccagtctggatgctgcaggtgtggatgtgggag840
cgctacaagaggctgtgcccgccggagctgaaggcaccgcagttccctgtctccaatgtc900
agggtattgtattggagccggaggaagaagaagagcacatctgaagaggctctaaagatt960
ttgctggacgaagtatgctttgagtggaggccctatcggcacaactccctcaactggatg1020
gaacccaagtggttcaacaaggacaccattttggtgacctgtcatggtaaagataagcca1080
gagtggttgttggattacattgctgttatcagtcaaacaatgttgaccgggtttcatagt1140
gatgacaccgacaattcagtactgtacaatccacagcttgttgcgagacagtttggttat1200
gatcaggctgctcctgtgtccattgtcagggaaatccactttgagggaatcgagttgtgg1260
ataccaagtatcggtagatatgggatgcctggtgaagattatgttgcatggtgtagcagc1320
agtgggcagttctacaagcatcaaaatgatgtccaatatggttgctcggtattacgaaat1380
catgaaaatggggctatttcgtcgcaactgaatgtaaatgagaaatgtgttgtagtgcca1440
acacttgatcaatttatcacccaagtcactaggagagaccacatcaattacattgtagaa1500
gggcagccggagaaaatggataatggaagtcatgaggatgaaacagaggtgattgtctgt1560
ggccttgaagcttgtgtcaaggatagcagaactacttcagtgaaacagaatgtgcagaaa1620
caaagagataaatttgctgaagatggaggtgatagtaagaagaaaagaaaagtagaaagt1680
aacacgaaaaggagttcgttgcaattggaaggtcagaagtattcttcactccagaaagct1740
ctaaattctgattctaagaaatgtgaggagttggctcaagttgacagtgatgatgaatgc1800
attgttctcgaacagcccaagaataaatgtgaagtaataaacctggatgatgatgaggaa1860
cagagtgtttctaatcgagaacatcataacatgcaacttgtcctagaactggaagagttt1920
gtccgctctggtcttctctcgcaatgggaggaaagctcagatgaagatgatgtaagtgga1980
agcaagcaagaaacccagaagaaaagcaacaatgacccctatgctgaagcagccatgagg2040
gagtaccctctgttctttgagttcattcctcagaaaccacattacagaggcttcgtgaac2100
tatgatgaaactttaggagatctaccttacagtggattatggtttttgctaattggcttg2160
gctaaggaggtgctcaagacgtcatgtgacacagatgcttctgaaattgcatatctgatg2220
aaaaaagcccagcatttagagcaactgggtttcaacgtgacacatctcattgcccgctta2280
aaggagccacaaattaggcttaagaagcttcaagattctagagcaaggcttgatcatgcc2340
cgtgagaaagaggagggcaacgtagtcgagtctctttcaagccatctgaataaactgaag2400
ggcaacataaggatgatggatagtcatctggatggaacgaagcaagcttttatttcaaat2460
atacaagataaattgaatgaaggaattaatcttgccagtttggaaaaggaagtagaaact2520
gctgaaaaatgttgtcaggcaatgaaggatgaagtagctgcaatgagaatgaggtactcg2580
gaccgtggaatttagattgttccagtgtccgctatcttcatttaagaagttactttcttc2640
ctgacatagagcttgcttgatatctgtctgtatcatcaagagtctgatcaatagaatata2700
accatcttactttc2714
<210>2
<211>2712
<212>dna
<213>zeamaysl.
<400>2
atgcgagcattcaacggccgccgcctccacgatccgaagcccccctcattcctccccctc60
cccttagccccacgcaccacaacgccgccaccgccgcccccctcctcgccgccgcacgcc120
atcccgccgctcaacttccgctacctcaccccctgtccgcgctggtctgcctgggtcgcc180
accgcactcagcgaccccgttttcgctcccatcctcgcctcgtccgccatctccggcgcc240
gtcgccgcctccaccgccgtcgtctcccccgaccgtgcggccctctccgctctgctctcg300
ctctgggacccggatacccacgcgttccgtctcccagcgggccccgccaccttcacactc360
gaggacgcgctcgtcctcgctggtctaccgcccgcaggcgcgccgctggaccggaccctc420
acgccagaggaggatgacctccgcgttcgcctcgtcgtcgagagggagaagatcaaggag480
ctccacccctgtgcccgcgccgcgcgccgcgtgtctgcggaggtgtggctcgagtggttc540
gacggcggtggcatacgccccggcgaggacgacgaactgcggtggcttgggttcctcgct600
tactggctcgctttttttgtcacgccaaggctccggtcaaggggtggcgagctgccggag660
cgcgtcttcgcacatgctgcgaggcttagtcttggcgagcgcattgcccttggccagggg720
atggtggcaaatctatatgcggaaatggataagattgttacttgcactgtggccgatggt780
gtgtgtggccgtctagatctctgcgtgccagtctggatgctgcaggtgtggatgtgggag840
cgctacaagaggctgtgcccgccggagctgaaggcgccgcagttccctgtctccaatgtc900
agggtattgtattggagccggaggaagaagaagagcacatctgaggaggctctaaagatt960
ttgctggacgaagtatgctttgagtggaggccctatcggcacaactccctcaactggatg1020
gaacccaagtggttcaacaaggacaccattttggtgacctgtcatggtaaagataagcca1080
gagtggttgttggattacattgctgttatcagtcaaacaatgttgactgggtttcatagt1140
gatgacaccgacaattcagtactgtacaatccacagcttgttgcgagacaatttggttat1200
gatcaggctgctcctgtgtccattgtcagggaaatccactttgagggaatcgagttgtgg1260
ataccaagtatcggtagatatgggatgcctggtgaagattatgttgcatggtgcagcagc1320
agtgggcagttctacaagcatcaaaatgatgtccaatacggttgctcggtattacgagat1380
cacgaaaatggggctatttcgtcgcaactgaatgtaagtgagaaatgtgttgtagtgcca1440
acacttgatcaatttatcacccaagtcactaggagagaccacatcaattacattgtagaa1500
gggcagctggagaaaatggataatggaagtcatgaggatgaaacagaggtgattgtctgt1560
ggccttgaagcttgtgtcaaggatagcagaactacttcagtgaaacagaatgtgaagaaa1620
caaagagataaatttgctgaagatggaggtaatagtaagaagaaaagaaaagtagaaagt1680
aacacggaaaggagttcgttgcaattggaaggtcagaagtattcttcactccagaaagct1740
ctaaattctgattctaaaaaatgtgaggagttggctcaagttgacagtgatgatgaatgc1800
attgttctcgaacagcccaagaataaatgtgaagtaataaacctggatgatgatgaggaa1860
cagagtgtttctaatcgagaacatcataacatgcaacttgtcctagaactggaagagttt1920
gttcgctctggtcttctctcgcaatgggaggaaagctcagatgaagatgatgtaagtgga1980
agcaagcaagaaacccagaagaaaagcaacaatgacccctatgctgaagcagccatgagg2040
gagtaccctctgttctttgagttcattcctcagaaaccacattacagaggcttcatgaac2100
tatgatgaaactttaggagatctaccttacagtggattatggtttttgctaattggcttg2160
gctaaggaggtgctcaagacgtcatgtgacacagatgcttctgaaattgcatatctgatg2220
aaaaaagcacagcatttagagcaactgggtttcaacgtgacacatctcattgcccgctta2280
aaggagccacaaattaggcttaagaagcttcaagattctagagcaaggcttgatcatgcc2340
cgtgagaaagaggagggcaacgtagtcgagtctctttcaagccatctgaataaactgaaa2400
ggcaacataaggatgatggatagtcatctggatggaacgaagcaagcttttatttcaaat2460
atacaagataaattgaatgaaggaattaatcttgccagtttggaaaaggaagtagaaact2520
gctgaaaaatgttgtcaggcaatgaaggatgaagtagctgcaatgagaatgaggtactcg2580
gaccgtggaatttagattgttccagtgtccgctatcttcatttaagaagttactttcttc2640
ctgacatagagcttgcttgatatctgtctgtcatcaagagtctgatcaatagaatataac2700
catcttactttc2712
<210>3
<211>2595
<212>dna
<213>人工合成(unknown)
<400>3
atgcgagcattcaacggccgccgcctccacgatccgaagcccccctcattcctccccctc60
cccttagccccacgcaccacaacgccgccaccgccgcccccctcctcgccgccgcacgcc120
atcccgccgctcaacttccgctacctcaccccctgtccgcgctggtctgcctgggtcgcc180
accgcactcagcgaccccgttttcgctcccatcctcgcctcgtccgccatctccggcgcc240
gtcgccgcctccaccgccgtcgtctcccccgaccgtgcggccctctccgctctgctctcg300
ctctgggacccggatacccacgcgttccgtctcccagcgggccccgccaccttcacactc360
gaggatgcgctcgtcctcgctggtctaccgcccgcaggcgcgccgctggaccggaccctc420
acgccagaggaggatgacctccgcgttcgcctcgttgtcgagagggagaagatcaaggag480
ctccacccctgtgcccgcgccgcgcgccgcgtgtctgcggaggtgtggctcgagtggttc540
gacggcggtggcatacgccccggcgaggacgacgaactgcggtggcttgggttcctcgct600
tactggctcgctttttttgttacgccaaggctccggtcaaggggtggcgagctgccggag660
cgcgtcttcgcacatgctgcgaggcttagtcttggcgagcgcattgcccttggccagggg720
atggtggcaaatctatatgcggaaatggataagattgttacttgcactgtggccgatggt780
gtgtgtggccgtctagatgtctgcgtgccagtctggatgctgcaggtgtggatgtgggag840
cgctacaagaggctgtgcccgccggagctgaaggcaccgcagttccctgtctccaatgtc900
agggtattgtattggagccggaggaagaagaagagcacatctgaagaggctctaaagatt960
ttgctggacgaagtatgctttgagtggaggccctatcggcacaactccctcaactggatg1020
gaacccaagtggttcaacaaggacaccattttggtgacctgtcatggtaaagataagcca1080
gagtggttgttggattacattgctgttatcagtcaaacaatgttgaccgggtttcatagt1140
gatgacaccgacaattcagtactgtacaatccacagcttgttgcgagacagtttggttat1200
gatcaggctgctcctgtgtccattgtcagggaaatccactttgagggaatcgagttgtgg1260
ataccaagtatcggtagatatgggatgcctggtgaagattatgttgcatggtgtagcagc1320
agtgggcagttctacaagcatcaaaatgatgtccaatatggttgctcggtattacgaaat1380
catgaaaatggggctatttcgtcgcaactgaatgtaaatgagaaatgtgttgtagtgcca1440
acacttgatcaatttatcacccaagtcactaggagagaccacatcaattacattgtagaa1500
gggcagccggagaaaatggataatggaagtcatgaggatgaaacagaggtgattgtctgt1560
ggccttgaagcttgtgtcaaggatagcagaactacttcagtgaaacagaatgtgcagaaa1620
caaagagataaatttgctgaagatggaggtgatagtaagaagaaaagaaaagtagaaagt1680
aacacgaaaaggagttcgttgcaattggaaggtcagaagtattcttcactccagaaagct1740
ctaaattctgattctaagaaatgtgaggagttggctcaagttgacagtgatgatgaatgc1800
attgttctcgaacagcccaagaataaatgtgaagtaataaacctggatgatgatgaggaa1860
cagagtgtttctaatcgagaacatcataacatgcaacttgtcctagaactggaagagttt1920
gtccgctctggtcttctctcgcaatgggaggaaagctcagatgaagatgatgtaagtgga1980
agcaagcaagaaacccagaagaaaagcaacaatgacccctatgctgaagcagccatgagg2040
gagtaccctctgttctttgagttcattcctcagaaaccacattacagaggcttcgtgaac2100
tatgatgaaactttaggagatctaccttacagtggattatggtttttgctaattggcttg2160
gctaaggaggtgctcaagacgtcatgtgacacagatgcttctgaaattgcatatctgatg2220
aaaaaagcccagcatttagagcaactgggtttcaacgtgacacatctcattgcccgctta2280
aaggagccacaaattaggcttaagaagcttcaagattctagagcaaggcttgatcatgcc2340
cgtgagaaagaggagggcaacgtagtcgagtctctttcaagccatctgaataaactgaag2400
ggcaacataaggatgatggatagtcatctggatggaacgaagcaagcttttatttcaaat2460
atacaagataaattgaatgaaggaattaatcttgccagtttggaaaaggaagtagaaact2520
gctgaaaaatgttgtcaggcaatgaaggatgaagtagctgcaatgagaatgaggtactcg2580
gaccgtggaatttag2595
<210>4
<211>2595
<212>dna
<213>人工合成(unknown)
<400>4
atgcgagcattcaacggccgccgcctccacgatccgaagcccccctcattcctccccctc60
cccttagccccacgcaccacaacgccgccaccgccgcccccctcctcgccgccgcacgcc120
atcccgccgctcaacttccgctacctcaccccctgtccgcgctggtctgcctgggtcgcc180
accgcactcagcgaccccgttttcgctcccatcctcgcctcgtccgccatctccggcgcc240
gtcgccgcctccaccgccgtcgtctcccccgaccgtgcggccctctccgctctgctctcg300
ctctgggacccggatacccacgcgttccgtctcccagcgggccccgccaccttcacactc360
gaggacgcgctcgtcctcgctggtctaccgcccgcaggcgcgccgctggaccggaccctc420
acgccagaggaggatgacctccgcgttcgcctcgtcgtcgagagggagaagatcaaggag480
ctccacccctgtgcccgcgccgcgcgccgcgtgtctgcggaggtgtggctcgagtggttc540
gacggcggtggcatacgccccggcgaggacgacgaactgcggtggcttgggttcctcgct600
tactggctcgctttttttgtcacgccaaggctccggtcaaggggtggcgagctgccggag660
cgcgtcttcgcacatgctgcgaggcttagtcttggcgagcgcattgcccttggccagggg720
atggtggcaaatctatatgcggaaatggataagattgttacttgcactgtggccgatggt780
gtgtgtggccgtctagatctctgcgtgccagtctggatgctgcaggtgtggatgtgggag840
cgctacaagaggctgtgcccgccggagctgaaggcgccgcagttccctgtctccaatgtc900
agggtattgtattggagccggaggaagaagaagagcacatctgaggaggctctaaagatt960
ttgctggacgaagtatgctttgagtggaggccctatcggcacaactccctcaactggatg1020
gaacccaagtggttcaacaaggacaccattttggtgacctgtcatggtaaagataagcca1080
gagtggttgttggattacattgctgttatcagtcaaacaatgttgactgggtttcatagt1140
gatgacaccgacaattcagtactgtacaatccacagcttgttgcgagacaatttggttat1200
gatcaggctgctcctgtgtccattgtcagggaaatccactttgagggaatcgagttgtgg1260
ataccaagtatcggtagatatgggatgcctggtgaagattatgttgcatggtgcagcagc1320
agtgggcagttctacaagcatcaaaatgatgtccaatacggttgctcggtattacgagat1380
cacgaaaatggggctatttcgtcgcaactgaatgtaagtgagaaatgtgttgtagtgcca1440
acacttgatcaatttatcacccaagtcactaggagagaccacatcaattacattgtagaa1500
gggcagctggagaaaatggataatggaagtcatgaggatgaaacagaggtgattgtctgt1560
ggccttgaagcttgtgtcaaggatagcagaactacttcagtgaaacagaatgtgaagaaa1620
caaagagataaatttgctgaagatggaggtaatagtaagaagaaaagaaaagtagaaagt1680
aacacggaaaggagttcgttgcaattggaaggtcagaagtattcttcactccagaaagct1740
ctaaattctgattctaaaaaatgtgaggagttggctcaagttgacagtgatgatgaatgc1800
attgttctcgaacagcccaagaataaatgtgaagtaataaacctggatgatgatgaggaa1860
cagagtgtttctaatcgagaacatcataacatgcaacttgtcctagaactggaagagttt1920
gttcgctctggtcttctctcgcaatgggaggaaagctcagatgaagatgatgtaagtgga1980
agcaagcaagaaacccagaagaaaagcaacaatgacccctatgctgaagcagccatgagg2040
gagtaccctctgttctttgagttcattcctcagaaaccacattacagaggcttcatgaac2100
tatgatgaaactttaggagatctaccttacagtggattatggtttttgctaattggcttg2160
gctaaggaggtgctcaagacgtcatgtgacacagatgcttctgaaattgcatatctgatg2220
aaaaaagcacagcatttagagcaactgggtttcaacgtgacacatctcattgcccgctta2280
aaggagccacaaattaggcttaagaagcttcaagattctagagcaaggcttgatcatgcc2340
cgtgagaaagaggagggcaacgtagtcgagtctctttcaagccatctgaataaactgaaa2400
ggcaacataaggatgatggatagtcatctggatggaacgaagcaagcttttatttcaaat2460
atacaagataaattgaatgaaggaattaatcttgccagtttggaaaaggaagtagaaact2520
gctgaaaaatgttgtcaggcaatgaaggatgaagtagctgcaatgagaatgaggtactcg2580
gaccgtggaatttag2595
<210>5
<211>864
<212>prt
<213>zeamaysl.
<400>5
metargalapheasnglyargargleuhisaspprolysproproser
151015
pheleuproleuproleualaproargthrthrthrpropropropro
202530
proproserserproprohisalaileproproleuasnpheargtyr
354045
leuthrprocysproargtrpseralatrpvalalathralaleuser
505560
aspprovalphealaproileleualaserseralaileserglyala
65707580
valalaalaserthralavalvalserproaspargalaalaleuser
859095
alaleuleuserleutrpaspproaspthrhisalapheargleupro
100105110
alaglyproalathrphethrleugluaspalaleuvalleualagly
115120125
leuproproalaglyalaproleuaspargthrleuthrprogluglu
130135140
aspaspleuargvalargleuvalvalgluargglulysilelysglu
145150155160
leuhisprocysalaargalaalaargargvalseralagluvaltrp
165170175
leuglutrppheaspglyglyglyileargproglygluaspaspglu
180185190
leuargtrpleuglypheleualatyrtrpleualaphephevalthr
195200205
proargleuargserargglyglygluleuprogluargvalpheala
210215220
hisalaalaargleuserleuglygluargilealaleuglyglngly
225230235240
metvalalaasnleutyralaglumetasplysilevalthrcysthr
245250255
valalaaspglyvalcysglyargleuaspvalcysvalprovaltrp
260265270
metleuglnvaltrpmettrpgluargtyrlysargleucyspropro
275280285
gluleulysalaproglnpheprovalserasnvalargvalleutyr
290295300
trpserargarglyslyslysserthrsergluglualaleulysile
305310315320
leuleuaspgluvalcyspheglutrpargprotyrarghisasnser
325330335
leuasntrpmetgluprolystrppheasnlysaspthrileleuval
340345350
thrcyshisglylysasplysproglutrpleuleuasptyrileala
355360365
valileserglnthrmetleuthrglyphehisseraspaspthrasp
370375380
asnservalleutyrasnproglnleuvalalaargglnpheglytyr
385390395400
aspglnalaalaprovalserilevalarggluilehispheglugly
405410415
ilegluleutrpileproserileglyargtyrglymetproglyglu
420425430
asptyrvalalatrpcysserserserglyglnphetyrlyshisgln
435440445
asnaspvalglntyrglycysservalleuargasnhisgluasngly
450455460
alaileserserglnleuasnvalasnglulyscysvalvalvalpro
465470475480
thrleuaspglnpheilethrglnvalthrargargasphisileasn
485490495
tyrilevalgluglyglnproglulysmetaspasnglyserhisglu
500505510
aspgluthrgluvalilevalcysglyleuglualacysvallysasp
515520525
serargthrthrservallysglnasnvalglnlysglnargasplys
530535540
phealagluaspglyglyaspserlyslyslysarglysvalgluser
545550555560
asnthrlysargserserleuglnleugluglyglnlystyrserser
565570575
leuglnlysalaleuasnseraspserlyslyscysglugluleuala
580585590
glnvalaspseraspaspglucysilevalleugluglnprolysasn
595600605
lyscysgluvalileasnleuaspaspaspglugluglnservalser
610615620
asnarggluhishisasnmetglnleuvalleugluleuglugluphe
625630635640
valargserglyleuleuserglntrpglugluserseraspgluasp
645650655
aspvalserglyserlysglngluthrglnlyslysserasnasnasp
660665670
protyralaglualaalametargglutyrproleuphephegluphe
675680685
ileproglnlysprohistyrargglyphevalasntyraspgluthr
690695700
leuglyaspleuprotyrserglyleutrppheleuleuileglyleu
705710715720
alalysgluvalleulysthrsercysaspthraspalasergluile
725730735
alatyrleumetlyslysalaglnhisleugluglnleuglypheasn
740745750
valthrhisleuilealaargleulysgluproglnileargleulys
755760765
lysleuglnaspserargalaargleuasphisalaargglulysglu
770775780
gluglyasnvalvalgluserleuserserhisleuasnlysleulys
785790795800
glyasnileargmetmetaspserhisleuaspglythrlysglnala
805810815
pheileserasnileglnasplysleuasngluglyileasnleuala
820825830
serleuglulysgluvalgluthralaglulyscyscysglnalamet
835840845
lysaspgluvalalaalametargmetargtyrseraspargglyile
850855860
<210>6
<211>864
<212>prt
<213>zeamaysl.
<400>6
metargalapheasnglyargargleuhisaspprolysproproser
151015
pheleuproleuproleualaproargthrthrthrpropropropro
202530
proproserserproprohisalaileproproleuasnpheargtyr
354045
leuthrprocysproargtrpseralatrpvalalathralaleuser
505560
aspprovalphealaproileleualaserseralaileserglyala
65707580
valalaalaserthralavalvalserproaspargalaalaleuser
859095
alaleuleuserleutrpaspproaspthrhisalapheargleupro
100105110
alaglyproalathrphethrleugluaspalaleuvalleualagly
115120125
leuproproalaglyalaproleuaspargthrleuthrprogluglu
130135140
aspaspleuargvalargleuvalvalgluargglulysilelysglu
145150155160
leuhisprocysalaargalaalaargargvalseralagluvaltrp
165170175
leuglutrppheaspglyglyglyileargproglygluaspaspglu
180185190
leuargtrpleuglypheleualatyrtrpleualaphephevalthr
195200205
proargleuargserargglyglygluleuprogluargvalpheala
210215220
hisalaalaargleuserleuglygluargilealaleuglyglngly
225230235240
metvalalaasnleutyralaglumetasplysilevalthrcysthr
245250255
valalaaspglyvalcysglyargleuaspleucysvalprovaltrp
260265270
metleuglnvaltrpmettrpgluargtyrlysargleucyspropro
275280285
gluleulysalaproglnpheprovalserasnvalargvalleutyr
290295300
trpserargarglyslyslysserthrsergluglualaleulysile
305310315320
leuleuaspgluvalcyspheglutrpargprotyrarghisasnser
325330335
leuasntrpmetgluprolystrppheasnlysaspthrileleuval
340345350
thrcyshisglylysasplysproglutrpleuleuasptyrileala
355360365
valileserglnthrmetleuthrglyphehisseraspaspthrasp
370375380
asnservalleutyrasnproglnleuvalalaargglnpheglytyr
385390395400
aspglnalaalaprovalserilevalarggluilehispheglugly
405410415
ilegluleutrpileproserileglyargtyrglymetproglyglu
420425430
asptyrvalalatrpcysserserserglyglnphetyrlyshisgln
435440445
asnaspvalglntyrglycysservalleuargasphisgluasngly
450455460
alaileserserglnleuasnvalserglulyscysvalvalvalpro
465470475480
thrleuaspglnpheilethrglnvalthrargargasphisileasn
485490495
tyrilevalgluglyglnleuglulysmetaspasnglyserhisglu
500505510
aspgluthrgluvalilevalcysglyleuglualacysvallysasp
515520525
serargthrthrservallysglnasnvallyslysglnargasplys
530535540
phealagluaspglyglyasnserlyslyslysarglysvalgluser
545550555560
asnthrgluargserserleuglnleugluglyglnlystyrserser
565570575
leuglnlysalaleuasnseraspserlyslyscysglugluleuala
580585590
glnvalaspseraspaspglucysilevalleugluglnprolysasn
595600605
lyscysgluvalileasnleuaspaspaspglugluglnservalser
610615620
asnarggluhishisasnmetglnleuvalleugluleuglugluphe
625630635640
valargserglyleuleuserglntrpglugluserseraspgluasp
645650655
aspvalserglyserlysglngluthrglnlyslysserasnasnasp
660665670
protyralaglualaalametargglutyrproleuphephegluphe
675680685
ileproglnlysprohistyrargglyphemetasntyraspgluthr
690695700
leuglyaspleuprotyrserglyleutrppheleuleuileglyleu
705710715720
alalysgluvalleulysthrsercysaspthraspalasergluile
725730735
alatyrleumetlyslysalaglnhisleugluglnleuglypheasn
740745750
valthrhisleuilealaargleulysgluproglnileargleulys
755760765
lysleuglnaspserargalaargleuasphisalaargglulysglu
770775780
gluglyasnvalvalgluserleuserserhisleuasnlysleulys
785790795800
glyasnileargmetmetaspserhisleuaspglythrlysglnala
805810815
pheileserasnileglnasplysleuasngluglyileasnleuala
820825830
serleuglulysgluvalgluthralaglulyscyscysglnalamet
835840845
lysaspgluvalalaalametargmetargtyrseraspargglyile
850855860
<210>7
<211>20
<212>dna
<213>zeamaysl.
<400>7
gtcagggaaatccactttga20
<210>8
<211>20
<212>dna
<213>zeamaysl.
<400>8
gttacttgcactgtggccga20
<210>9
<211>103
<212>rna
<213>人工合成(unknown)
<400>9
gucagggaaauccacuuugaguuuuagagcuagaaauagcaaguuaaaauaaggcuaguc60
cguuaucaacuugaaaaaguggcaccgagucggugcuuuuuuu103
<210>10
<211>103
<212>rna
<213>人工合成(unknown)
<400>10
guuacuugcacuguggccgaguuuuagagcuagaaauagcaaguuaaaauaaggcuaguc60
cguuaucaacuugaaaaaguggcaccgagucggugcuuuuuuu103
<210>11
<211>21
<212>dna
<213>人工合成(unknown)
<400>11
gttacttgcactgtggcgcga21
<210>12
<211>19
<212>dna
<213>人工合成(unknown)
<400>12
gtcagggaaatccacttga19
<210>13
<211>19
<212>dna
<213>人工合成(unknown)
<400>13
gttacttgcactgcgatgg19
<210>14
<211>21
<212>dna
<213>人工合成(unknown)
<400>14
tccattgtcaggggagggaat21
<210>15
<211>680
<212>dna
<213>人工合成(unknown)
<400>15
agcgcgtcttcgcacatgctgcgaggcttagtcttggcgagcgcattgcccttggccagg60
ggatggtggcaaatctatatgcggaaatggataagattgttacttgcactgtggcgcgat120
ggtgtgtgtggccgtctagatctctgcgtgccagtctggatgctgcaggtgtggatgtgg180
gagcgctacaagaggctgtgcccgccggagctgaaggcgccgcagttccctgtctccaat240
gtcagggtattgtattggagccggaggaagaagaagagcacatctgaggaggctctaaag300
attttgctggacgaagtatgctttgagtggaggccctatcggcacaactccctcaactgg360
atggaacccaagtggttcaacaaggacaccattttggtgacctgtcatggtaaagataag420
ccagagtggttgttggattacattgctgttatcagtcaaacaatgttgactgggtttcat480
agtgatgacaccgacaattcagtactgtacaatccacagcttgttgcgagacaatttggt540
tatgatcaggctgctcctgtgtccattgtcagggaaatccacttgagggaatcgagttgt600
ggataccaagtatcggtagatatgggatgcctggtgaagattatgttgcatggtgcagca660
gcagtgggcagttctacaag680
<210>16
<211>671
<212>dna
<213>人工合成(unknown)
<400>16
agcgcgtcttcgcacatgctgcgaggcttagtcttggcgagcgcattgcccttggccagg60
ggatggtggcaaatctatatgcggaaatggataagattgttacttgcactgcgatggtgt120
gtgtggccgtctagatctctgcgtgccagtctggatgctgcaggtgtggatgtgggagcg180
ctacaagaggctgtgcccgccggagctgaaggcgccgcagttccctgtctccaatgtcag240
ggtattgtattggagccggaggaagaagaagagcacatctgaggaggctctaaagatttt300
gctggacgaagtatgctttgagtggaggccctatcggcacaactccctcaactggatgga360
acccaagtggttcaacaaggacaccattttggtgacctgtcatggtaaagataagccaga420
gtggttgttggattacattgctgttatcagtcaaacaatgttgactgggtttcatagtga480
tgacaccgacaattcagtactgtacaatccacagcttgttgcgagacaatttggttatga540
tcaggctgctcctgtgtccattgtcaggggagggaatgggaatcgagttgtggataccaa600
gtatcggtagatatgggatgcctggtgaagattatgttgcatggtgcagcagcagtgggc660
agttctacaag671
- 还没有人留言评论。精彩留言会获得点赞!