基于牡丹叶绿体基因组序列开发的cpSSR标记引物及应用
基于牡丹叶绿体基因组序列开发的cpssr标记引物及应用
技术领域
1.本发明属于植物分子育种技术领域,涉及一种微卫星分子标记的开发,具体涉及基于植物叶绿体全基因组序列的叶绿体微卫星分子标记。
背景技术:
2.微卫星标记(或ssr)是对不同等位基因间重复序列两侧的相对保守序列设计特异性引物,以此扩增植物基因组dna的方法。由于该标记具有共显性、扩增稳定、信息量大、价格低廉且高效的特点,作为植物遗传多样性分析研究的一种理想标记选择而得到了广泛应用(李珊珊等,2014;杨海平等,2017;ebrahimi et al,2017)。叶绿体基因组独立于细胞核基因组,是细胞器基因组的一种,其在序列和结构上呈现出高度的保守性。叶绿体基因组因其基因组结构简单,具有更小的分子量和拷贝数,与核基因组相比,其基因结构顺序及基因含量具有高度保守性、基因替换率较低等特点,更有利于物理图谱的构建、特定基因的分离与鉴定、序列测定、进化研究等。
3.首次获得叶绿体基因组的完整序列是在地钱与烟草(1986年)的研究中,叶绿体基因组是细胞器基因组的一种,它独立于细胞核基因组,在序列和结构上呈现出高度的保守性,这为开发叶绿体微卫星标记提供了可行性。植物叶绿体基因组在结构上一般为四段式:一个大单拷贝区域(large single copy, lsc)、一个小单拷贝区域(small single copy, ssc)和两个反向重复区域(inverted repeats, irs)。植物的叶绿体基因组结构高度保守,一般编码约110
‑
130个基因。这些基因根据其功能主要分为叶绿体自我复制相关基因、光合作用相关基因、其他基因三类。
4.叶绿体微卫星标记(简称cpssr)位点通常分布在非编码区域,相比于编码区,其表现出了更高的变异性,而且具有低进化速率和重组率几乎为零的优势。此外,cpssr基因座两侧的区域是高度保守的,在近缘物种中具有较高的通用性。
技术实现要素:
5.针对核基因标记的不足,本发明的目的在于提供基于植物叶绿体全基因组序列的叶绿体微卫星标记,不仅可以弥补核基因组标记数量的不足,还可以研究植物母系遗传的特点,较好地排除父本基因组的干扰,cpssr标记的检测开发能为群体遗传学研究,特别是近缘物种的识别与鉴定提供一种简便且可靠的方法。
6.为了实现上述目的,本发明采用的具体方案为:基于牡丹叶绿体基因组开发的cpssr标记引物组,所述引物组包括以下引物:cpssr
‑
1标记引物:正向序列如seq id no:1,所示,反向序列如seq id no:2所示;cpssr
‑
4标记引物:正向序列如seq id no:3,所示,反向序列如seq id no:4所示;cpssr
‑
5标记引物:正向序列如seq id no:5,所示,反向序列如seq id no:6所示;cpssr
‑
6标记引物:正向序列如seq id no:7,所示,反向序列如seq id no:8所示;cpssr
‑
7标记引物:正向序列如seq id no:9,所示,反向序列如seq id no:10所
示;cpssr
‑
8标记引物:正向序列如seq id no:11,所示,反向序列如seq id no:12所示;cpssr
‑
9标记引物:正向序列如seq id no:13,所示,反向序列如seq id no:14所示;cpssr
‑
11标记引物:正向序列如seq id no:15,所示,反向序列如seq id no:16所示;cpssr
‑
12标记引物:正向序列如seq id no:17,所示,反向序列如seq id no:18所示;cpssr
‑
15标记引物:正向序列如seq id no:19,所示,反向序列如seq id no:20所示;cpssr
‑
17标记引物:正向序列如seq id no:21,所示,反向序列如seq id no:22所示;cpssr
‑
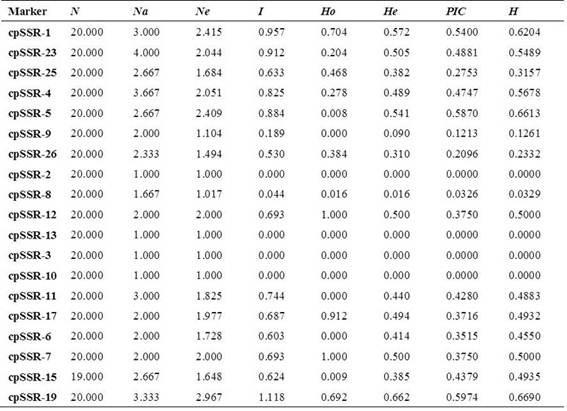
19标记引物:正向序列如seq id no:23,所示,反向序列如seq id no:24所示;cpssr
‑
21标记引物:正向序列如seq id no:25,所示,反向序列如seq id no:26所示;cpssr
‑
23标记引物:正向序列如seq id no:27,所示,反向序列如seq id no:28所示;cpssr
‑
25标记引物:正向序列如seq id no:29,所示,反向序列如seq id no:30所示;cpssr
‑
26标记引物:正向序列如seq id no:31,所示,反向序列如seq id no:32所示。
7.进一步地,所述cpssr标记引物组,通过以下方法筛选获得:步骤一、植物组织样本的总dna提取;步骤二、总dna完整性、纯度及浓度的检测;步骤三、将步骤二得到的植物总dna片段化,用二代测序平台illumina xten对文库片段进行高通量测序,以获取植物样品的原始测序数据;步骤四、对步骤三中得到的植物叶绿体基因组数据进行序列拼接和验证;步骤五、对步骤四中拼接好的序列数据批量检测潜在的微卫星重复序列,并通过制定的筛选标准获得cpssr引物序列,具体的筛选条件为重复单位为5个及以上,重复片段的大小范围为2~6bp,在初步得到引物信息后,再用以下的约束条件进一步筛选ssr位点:
①
扩增产物大小为150
‑
400bp;
②
退火温度60℃,且正反向引物退火温度接近;
③
侧翼区的长度≤2000bp;
④
删除重复引物序列。
8.步骤六、对步骤五中筛选得到的cpssr引物进行过滤,删除重复的引物序列;步骤七、对步骤六中过滤得到的cpssr引物进行引物合成,并进行稀释、保存备用;步骤八、采集植物表型和来源差异大的多株植物样本的无病害嫩叶,植物样本数量一般不超过80,提取植物叶片的总dna,检测dna的完整性、纯度及浓度,并稀释、保存备用;
步骤九、从步骤八中随机挑选表型和来源差异大的8~10株植物的dna等量混合后,采用pcr扩增程序对步骤七中得到的cpssr引物进行dna筛选;步骤十、用1%琼脂糖凝胶电泳初步检测产物的扩增效率;步骤十一、将经步骤十中挑选出的cpss引物,用步骤八中的植物样本的dna,通过毛细管电泳技术对cpssr引物进行二次筛选,保留扩增效率和多态性高、稳定性好的引物。
9.本发明还提供所述cpssr标记引物组在牡丹指纹图谱构建和品种亲缘关系分析中的应用。
10.有益效果:本发明通过筛选叶绿体基因组序列中大量的ssr位点,获得一组多态性高、通用性好的cpssr标记位点,开发的cpssr标记不仅可以弥补核基因组标记的不足,还可以研究植物母系遗传的特点,较好地排除父本基因组的干扰,cpssr标记的检测开发能为群体遗传学研究,特别是近缘物种的识别与鉴定提供一种简便且可靠的方法。
附图说明
11.图1是cpssr
‑
6引物在部分牡丹品种样本中的毛细管电泳图。
具体实施方式
12.一种基于植物叶绿体全基因组技术快速有效开发叶绿体微卫星标记的方法,包括以下步骤:一、植物组织样本的总dna提取,采用改进的ctab法,具体操作流程如下:1、取适量的牡丹嫩叶片于预冷且装有适量液氮的研钵中,迅速研磨成粉末状(在此过程中要始终保持研钵中的液氮充足,以保证提取dna的质量),取适量粉末快速转移至液氮预冷的2ml离心管中;2、向离心管中加入1500μl 65℃预热的ctab和100μl β
‑
巯基乙醇,将离心管盖紧后剧烈震荡混匀,并保证离心管底部的粉末在ctab缓冲液中均匀地分散开来,将离心管置于65℃水浴锅中50分钟。在此期间,每隔5min取出离心管剧烈震荡30s;3、水浴结束后,取出离心管,待其降低到室温,放入高速冷冻离心机中,4℃ 12000rpm离心12min;4、吸取上清液于另一支2ml的离心管中,加入等体积的24:1三氯甲烷/异戊醇,上下颠倒剧烈震荡10min;5、高速冷冻离心机 4℃ 12 000rpm 离心15min后,重复步骤4;6、重复步骤4和5两次,以充分去除蛋白质和多酚类物质;7、吸取上清液于另一支2ml离心管中,加入等体积
‑
20℃预冷的无水乙醇,上下轻轻颠倒混匀,然后置于
‑
20℃冰箱30min,使dna充分沉淀;8、用200μl抢头挑出离心管中的絮状dna沉淀于另一1.5ml离心管中,加入1000μl 70%无水乙醇漂洗沉淀,手指轻弹使沉淀离开离心管底部,上下轻轻颠倒数次,重复清洗3次;9、将装有漂洗干净的dna离心管放于超净工作台中晾干至半透明状,然后加入200~300μl无菌双蒸水溶解,加入2μl rnase 去除总dna中的rna,37℃水浴1h后,
‑
20℃保存备
用。
13.二、总dna完整性、纯度及浓度的检测:使用1%琼脂糖凝胶电泳检测dna的完整性,使用nanodrop 2000微量分光光度计进行纯度及浓度测定。
14.三、采用vahtstm universal dna library prep kit for illumina
ꢀ®ꢀ
v2 试剂盒将步骤二得到的植物总dna片段化,用二代测序平台illumina xten对文库片段进行高通量测序,以获取植物样品的原始测序数据。
15.四、对步骤三中得到的植物叶绿体基因组数据进行序列拼接和验证。
16.五、对步骤四中拼接好的序列数据批量检测潜在的微卫星重复序列,并制定严格的筛选标准,以获得cpssr引物序列。
17.六、对步骤五中筛选得到的cpssr引物进行过滤,删除重复的引物序列。
18.七、对步骤六中过滤得到的cpssr引物在北京瑞博兴科生物技术有限公司进行引物合成,按引物离心管上的标准,进行稀释,并立即储存于
‑
20℃保存备用。
19.八、采集植物表型和来源差异大的多株植物样本的嫩叶(植物样本数量一般不超过80),采集的嫩叶要求叶片完整且无病害,用改进的ctab法提取植物叶片的总dna。
20.九、使用步骤二中描述的方法检测dna的完整性、纯度及浓度,以满足实验需求。
21.十、将总dna用洗脱缓冲液tb(tiangen)稀释等份(20ng/μl),并立即储存于
‑
20℃保存备用。
22.十一、从步骤八中随机挑选表型和来源差异大的8~10株植物的dna等量混合后,采用pcr程序对步骤七中得到的cpssr引物通过待测样本的dna进行筛选。
23.十二、用1%琼脂糖凝胶电泳初步检测步骤十一中产物的扩增效率。
24.十三、将步骤十二中挑选出的cpss引物,用步骤八中的植物样本的dna,通过毛细管电泳技术对cpssr引物进行二次筛选,保留多态性高、稳定性好的引物。
25.下面将结合本发明实施例,对本发明实施例中的技术方案进行清楚、完整地描述。
26.①
针对高通量测序数据获得的海量ssr标记序列,从这些序列中快速获得有效的多个标记用于植物群体遗传学、近缘物种的识别与鉴定等基础研究显得十分必要。本发明中提供的方法省时省力,且标记的扩增效率高,不仅适合植物群体差异性和群体结构的研究,也适用于植物群体的核心种质构建等相关研究。
27.②
需要说明的是,本技术适用于植物微卫星标记的开发。本发明中所用植物材料以牡丹为例阐述具体实施方法。在不冲突的情况下,本技术中的实施方法也可用其他植物。
28.③
对构建叶绿体全基因组文库用的植物组织的dna样本,在材料收集时用液氮保存带回实验,并立即储存于
‑
80℃保存备用。
29.④
在实验地对60株牡丹植物样本的叶片取样时,将其放入自封袋中,并用锡纸包裹后放入液氮中速冻,将其带回实验室,
‑
80℃保存备用。
30.⑤
利用改进的ctab法提取各植物样品的总dna,操作步骤按照实施步骤中的步骤一进行。
31.⑥
对测序得到的fa.格式文件序列用gmata v2.2程序(https://github.com/xuewenwanguga/gmata)对测序得到的序列进行cpssr位点挖掘。筛选条件为重复单位为5个及以上,重复片段的大小范围为2~6bp,在初步得到引物信息后,再用下面的约束条件进一步筛选ssr位点:
①
扩增产物大小为150
‑
400bp;
②
退火温度60℃,且正反向引物退火温度接
近;
③
侧翼区的长度≤2000bp;
④
删除重复引物序列。
32.⑦
本发明中用1%的琼脂糖凝胶电泳对cpssr引物进行初筛扩增的体系为:10μl,包括2μl基因组dna(20 ng /μl),5μl 2
×
taq plus master mix
ꢀⅱ
(dye plus),0.2μl(10μm)正向引物和0.2μl(10μm)反向引物,用dd h2o 补齐。
33.⑧
本发明中涉及毛细管电泳pcr扩增体系为10μl,包括2μl基因组dna(20 ng /μl),5μl 2
×
taq plus master mix
ꢀⅱ
(dye plus),0.6μl 荧光染料(fam,hex,rox,tamer)的m13引物(10μm; 5'
‑
tgtaaaacgacggccagt
‑
3'),0.2μl(10μm)正向引物和0.6μl(10μm)反向引物,并用dd h2o 补齐。
34.⑨
采用pcr程序引物对待测样本的dna进行扩增,扩增程序为见下表1。
35.表1:pcr扩增程序。
36.⑩
、经
⑥
中获得的cpssr标记在60个牡丹样本中的毛细管电泳检测图1所示。
37.通过
⑦
和
⑧
两次筛选,并借助genalex v6.502和powermarker v3.25软件计算各标记的遗传参数(见表2)。
38.表2. cpssr标记在60个牡丹品种中的遗传多样性分析。
39.注:n:变异位点数;na:等位位点数;ne:有效等位位点数;i:香浓信息指数;ho:观察杂合度;he:期望杂合度;pic:多态性信息含量; h:基因多态性。
40.经综合分析表2后,删除扩增效率低和单态性的引物后,最终获得多态性高、通用性好的cpssr引物标记可用于群体遗传学、近缘物种的识别与鉴定等研究,以牡丹叶绿体基因组开发的cpssr引物信息如下表3和seq id no:1~ seq id no:32所示。
41.表3 cpssr引物信息。
42.根据遗传参数计算结果,进一步对这16对标记的扩增效率排序,其中,cpssr
‑
19的
扩增效率表现尤为突出,因此,该标记可以作为指纹图谱构建、亲缘关系分析的首选标记。
43.需要说明的是,以上所述的实施方案应理解为说明性的,而非限制本发明的保护范围,本发明的保护范围以权利要求书为准。对于本领域技术人员而言,在不背离本发明实质和范围的前提下,对本发明作出的一些非本质的改进和调整仍属于本发明的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1