一种催化活性提高的羧酸还原酶突变体及其编码基因、基因工程菌以及应用
1.本发明涉及蛋白质工程技术领域,具体涉及一种催化活性提高的羧酸还原酶突变体及其应用。
背景技术:
2.醛是药物化学中的关键中间体。醛类的高反应性合成化学家期望的性质,但是也为它们的合成带来挑战。合成醛的经典方法多通过在特定反应中心氧化实现,例如:可以使用naclo/tempo/co(oac)2系统或ibx氧化苄基位置;在wacker条件下末端双键可被o2有效地氧化;臭氧化反应可以高原子经济的方式将烯烃转化为醛,但需对生成的臭氧化物进行还原后处理;ru催化剂对末端三键的水合作用也可以良好的产量生成产物醛;从羧酸到醛的化学转化通常需要多步操作,因为羧酸的直接还原会导致过度还原而生成醇类产物。为了解决这个问题,通常采用将羧酸或其衍生物完全还原为醇并随后进行再氧化的方法,典型的程序先使用dibal
‑
h或lialh4作为还原剂,再使用铬(vi)试剂、高价碘化合物、tpap/nmo或tempo/naocl系统进行选择性氧化。虽然dibal
‑
h可以直接还原酯和腈生成相应的醛,但该反应不光收率低,还需要严格的温度控制。
3.羧酸还原酶可以实现羧酸到醛的选择性还原而避免过度还原产物醇的生成,但需要atp预先将羧酸底物激活再以nadph提供还原力而实现其还原。因此,羧酸还原酶已经用于合成香精香料香兰素,胡椒醛和其他醛或相关的醇类香料。目前已有报道展示了羧酸还原酶的几种应用,这些应用的目标都是醛的后续产品,其中包括醇和烷烃(用于润滑剂或燃料等应用)、胺、二胺、手性胺等,从简单的小分子化合物到复杂的手性砌块都有所涉及。虽然羧酸还原酶可以选择性的催化羧酸到醛的选择性还原,但是在实际应用中发现羧酸还原酶对香草酸等一系列有机酸的催化活力较低,导致催化过程时空产率偏低,因此距离实现工业化生产还有一定的距离。
技术实现要素:
4.本发明的目的是提供一种催化活性提高的羧酸还原酶突变体及其编码基因、基因工程菌以及应用,从而解决现有技术中羧酸还原酶对香草酸等一系列有机酸的催化活力较低,导致催化过程时空产率偏低的问题。
5.为了解决上述技术问题,本发明采用以下技术方案:
6.根据本发明的第一方面,提供一种催化活力提高的羧酸还原酶突变体,为seq id no.1所示野生型羧酸还原酶mscar的第505位氨基酸发生如下突变后得到的突变株中的一种:第505位的精氨酸r突变为谷氨酸e的羧酸还原酶突变体r505e,氨基酸序列如seq id no.2;第505位的精氨酸r突变为异亮氨酸i的羧酸还原酶突变体r505i,氨基酸序列如seq id no.3;第505位的精氨酸r突变为甲硫氨酸m的羧酸还原酶突变体r505m,氨基酸序列如seq id no.4;第505位的精氨酸r突变为谷氨酰胺q的羧酸还原酶突变体r505q,氨基酸序列
如seq id no.5。
7.还提供一种催化活性提高的羧酸还原酶突变体,包括以seq id no.1所示野生型羧酸还原酶mscar的第506位氨基酸残基发生如下突变后所得突变体:第506位的天冬酰胺n突变为赖氨酸k的羧酸还原酶突变体n506k,氨基酸序列如seq id no.6。
8.还提供一种催化活性提高的羧酸还原酶突变体,包括以seq id no.1所示野生型羧酸还原酶mscar的第505位氨基酸残基和第506位氨基酸残基发生如下突变后所得突变体的一种:第505位的精氨酸r突变为异亮氨酸i,第506位的天门冬酰胺n突变为赖氨酸k的羧酸还原酶突变体r505i/n506k,氨基酸序列如seq id no.7;第505位的精氨酸r突变为谷氨酸f,第506位的天门冬酰胺n突变为甘氨酸g的羧酸还原酶突变体r505f/n506g,氨基酸序列如seq id no.8;第505位的精氨酸r突变为谷氨酸f,第506位的天门冬酰胺n突变为丙氨酸a的羧酸还原酶突变体r505f/n506a,氨基酸序列如seq id no.9;第505位的精氨酸r突变为谷氨酸f,第506位的天门冬酰胺n突变为半胱氨酸c的羧酸还原酶突变体r505f/n506c,氨基酸序列如seq id no.10;第505位的精氨酸r突变为谷氨酸f,第506位的天门冬酰胺n突变为缬氨酸v的羧酸还原酶突变体r505f/n506v,氨基酸序列如seq id no.11;第505位的精氨酸r突变为异亮氨酸i,第506位的天门冬酰胺n突变为精氨酸r的羧酸还原酶突变体r505i/n506r,氨基酸序列如seq id no.12;第505位的精氨酸r突变为赖氨酸k,第506位的天门冬酰胺n突变为甲硫氨酸m的羧酸还原酶突变体r505k/n506m,氨基酸序列如seq id no.13;第505位的精氨酸r突变为苏氨酸t,第506位的天门冬酰胺n突变为丝氨酸s的羧酸还原酶突变体r505t/n506s,氨基酸序列如seq id no.14;第505位的精氨酸r突变为酪氨酸y,第506位的天门冬酰胺n突变为谷氨酸e的羧酸还原酶突变体r505y/n506e,氨基酸序列如seq id no.15。
9.根据本发明的第二方面,提供一种如上面所述的羧酸还原酶突变体的编码基因。
10.优选地,所述羧酸还原酶突变体r505i的编码基因的核苷酸序列如seq id no.17所示。
11.特别优选的,羧酸还原酶突变体n506k的编码基因的核苷酸序列如seq id no.18所示。
12.特别优选的,羧酸还原酶突变体r505f/n506g的编码基因的核苷酸序列如seq id no.19所示。
13.根据本发明的第三方面,提供一种包含上面所述编码基因的基因工程菌。
14.根据本发明的第四方面,提供一种如上面所述的羧酸还原酶突变体或如上面所述的基因工程菌在催化羧酸到醛的选择性还原中的应用。
15.特别优选地,所述应用还包括所述羧酸还原酶突变体或基因工程菌在催化香草酸制备香草醛中的应用。但是应当理解的是,本发明包括但不仅限于催化香草酸的还原生成相应的醛。
16.应当理解的是,本发明所使用的野生型羧酸还原酶mscar同其他种类的羧酸还原酶一样可以选择性的催化羧酸到醛的选择性还原,但是在实际应用中同样存在催化效率低的问题,距离实现工业化生产醛还有一定的距离。面对这种缺陷,本发明的发明人想到采用蛋白质工程手段来解决,主要涉及定向进化以及理性设计两种技术。其中,定向进化技术模拟自然进化过程,首先向酶分子的编码基因序列中随机引入突变,再对这些产生的突变体
进行筛选而得到具有目标性质的酶分子突变株,但是这种随机突变方法应用于羧酸还原酶的改造时就会遭遇棘手的处境,因为单个羧酸还原酶的编码基因长度逾3.6kb,向序列中随机引入突变后就会增大筛选环节的压力而对整个改造过程产生不利影响。因此,理性设计技术被选择成为改造羧酸还原酶的优势技术。
17.本发明选择了从菌株mycobacterium smegmatis mc2 155中克隆的羧酸还原酶mscar基因;该羧酸还原酶mscar的氨基酸序列如seq id no.1所示,编码基因序列如seq id no.16所示,在结构上,mscar包含n
‑
端的腺苷化结构域、c
‑
端的还原结构域以及连接二者的pcp结构域,同时腺苷化结构域又包含前端的a core和a sub两个亚结构域。研究发现,结构域间的铰链区对酶分子的催化作用具有重要的调节作用,通过与羧酸还原酶及同家族的其它anl酶进行同源性分析,确定了对505位点进行饱和突变以及对506位点进行定点突变的策略,对两点联合的最佳突变株进行分子动力学模拟发现了其内在的调控机制并获得了酶活关联参数d1;遂对这两点的其它突变可能进行虚拟突变和筛选得到活力最高的突变株。
18.实验证明,本发明得到的突变体r505e、r505i、r505m、r505q、n506k、r505i/n506k、r505f/n506g、r505f/n506a、r505f/n506c、r505f/n506v、r505i/n506r、r505k/n506m、r505t/n506s及r505y/n506e的催化效率相对野生型均有所提高,特别的,突变株r505f/n506g的催化效率达到30.08s
‑1mm
‑1,达到野生型的8倍(野生型羧酸还原酶mscar的催化效率约为3.64s
‑1mm
‑1)。因此,本发明获得的羧酸还原酶突变体相对于野生型羧酸还原酶mscar的催化效率具有较大提升。
19.综上所述,由于野生型羧酸还原酶mscar对香草酸等一系列有机酸的催化活力较低,导致催化过程时空产率偏低,本发明通过对羧酸还原酶mscar腺苷化结构域的铰链区进行理性设计,经突变后该羧酸还原酶的活性得到了提高,并可用于催化香草酸等有机酸制备香草醛等香精香料,其中突变株mscar
‑
r505f/n506g对香草酸的催化活力最高,相对野生型提高了8倍,对实现从酸到醛的高效生物催化具有重要意义。
附图说明
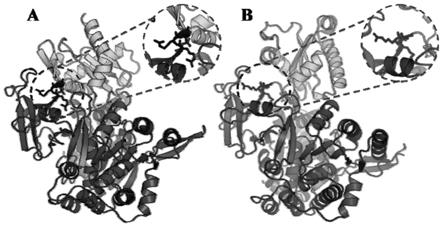
20.图1是羧酸还原酶mscar腺苷化结构域两种状态的空间结构;
21.图2是通过序列保守性分析构建突变体库并进行活力筛选所得的结果;
22.图3是通过虚拟突变所得突变体库并进行活力筛选所得的结果;
23.图4是羧酸还原酶mscar各突变株的蛋白表达sds
‑
page图,所有的分析均基于这些突变株的细胞破碎液上清;其中,泳道m是蛋白质marker;泳道1
‑
11为突变株r505y/n506e、r505t/n506s、r505f/n506a、r505v/n506r、r505l/n506p、r505p/n506v、r505k/n506m、r505i/n506r、r505f/n506v、r505f/n506c以及r505f/n506g的蛋白质电泳条带;
24.图5是羧酸还原酶mscar突变株r505f/n506g蛋白纯化sds
‑
page图;其中,泳道m是蛋白质marker;泳道1是该突变株的胞破碎液上清;泳道2是将破碎后所得的细胞碎片复溶后所得;泳道3是该突变纯化时的穿出液;泳道4是纯化过程中以含50mm咪唑的缓冲液洗脱而得的洗脱液;泳道5是纯化过程中以含100mm咪唑的缓冲液洗脱而得的洗脱液。
具体实施方式
25.以下结合具体实施例,进一步说明本发明。应理解,以下实施例仅用于说明本发明
而非用于限制本发明的范围。如非特殊说明,实施例中所用的技术手段为本领域常规操作。
26.实施例1底物对接与分子动力学模拟
27.为获得羧酸还原酶mscar腺苷化结构域的空间结构,遂对该结构域进行同源建模。利用swiss
‑
model在线服务器(http://www.swissmodel.expasy.org/)进行建模,该结构域腺苷化状态的3d模型以来自nocardia iowensis的羧酸还原酶(pdb编号:5msc)的结构为模板获得,硫酯化状态的3d模型以来自mycobacterium marinum的羧酸还原酶(pdb编号:5mss)的结构为模板获得。然后将经过结构优化的香草酰
‑
amp复合物对接进mscar腺苷化结构域的腺苷化状态的结构中。以羧酸还原酶晶体结构5msc中amp的位置对接结果进行筛查,并结合对接评分选择最佳对接姿态作为初始结构进行后续的分子动力学模拟。
28.分子对接利用discovery studio 2020软件,相关操作按照软件官方操作指南进行。本实施例中所有分子动力学模拟使用amber 18软件进行,相关操作同样按照软件官方操作指南进行。对香草酰
‑
amp中间体首先使用amber 18中antechamber模块的bcc方法计算力场参数和电荷分布,随后使用leap模块给对接后的蛋白质进行结构补充,然后将其沉浸在tip3p水盒子环境中并用抗衡离子钠原子维持系统的电中性。并用amber ff14sb力场文件处理整个系统并产生拓扑文件和坐标文件。系统首先进行5000步的最速下降法和15000步的共轭梯度能量最小化。然后,将系统用nvt方法从0k逐渐加热到300k。系统上升到300k后,采用npt方法将系统平衡2ns,再进行一个100ns的动力学模拟。本实施例中的全原子分子动力学模是为了探查羧酸还原酶mscar进行铰链区改造之后催化活力提高的机理。
29.其中,羧酸还原酶mscar腺苷化结构域两种状态的空间结构如图1所示。结果表明,羧酸还原酶腺苷化结构域在两种状态下具有较大的构象差异,n端的acore亚结构域构象基本不变,而c端的asub结构域却发生较大规模的构象旋转,连接二者的铰链区构象发生明显变化,因此选择该区域作为突变热点。通过突变体的构建和筛选发现突变株r505i/n506k具有最高的催化效率,通过分子动力学模拟发现,与野生型酶相比该突变株中残基k610侧链氨基氮原子到香草酰
‑
amp复合物糖环氧的距离较小。
30.实施例2羧酸还原酶mscar铰链区的虚拟突变及其筛选
31.以羧酸还原酶mscar腺苷化结构域的腺苷化状态结构与香草酰
‑
amp复合物结构为基础,在discovery studio 2020软件中的macromolecules模块下的build and edit protein程序进行蛋白质的点突变,即在结构中选中需要进行突变的位点,在choose amino acid操作栏中选择目标氨基酸即完成了氨基酸的突变,随后在该模块下调用minimize and refine protein程序对突变后的蛋白质进行结构优化,待优化完成后即可认为获得了较为合理的蛋白质
‑
香草酰
‑
amp复合物结合构象。再调用discovery studio 2020软件中的原子间距离测量功能计算残基k610中侧链氨基氮原子到香草酰
‑
amp复合物糖环氧之间的距离,待全部虚拟突变结果获得后比较该距离选择合适的突变株按照实施例3、4中的操作对突变体进行质粒构建和活性检测。
32.结果表明,突变株r505f/n506a、r505f/n506c、r505f/n506g、r505f/n506v、r505i/n506r、r505k/n506m、r505t/n506s及r505y/n506e这8个突变株的催化关键距离进一步缩短,因此构建这8个突变株进行活力测试。
33.实施例3mscar突变体的构建与筛选
34.将羧酸还原酶mscar生产菌株在大试管中培养10
‑
12h,抽提其质粒作为后续pcr的
模板。利用日本东洋纺公司的kod
‑
plus点突变试剂盒进行突变库构建。具体操作为:1、根据产品说明书以目标序列为模板进行引物设计;2、采用高保真的kod
–
plus
‑
酶,通过反向pcr法进行点突变引入;3、用dpn i对模板质粒dna进行消化;4、使用试剂盒中的t4 polynucleotide kinase和ligation high,对pcr产物进行自环化;5、转化,将所得的环化产物导入大肠杆菌bl21(de3)感受态细胞;6、挑取单菌落并接种于5ml lb培养基,37℃培养过夜后将菌种测序;7、确保序列正确后,将各突变体进行诱导表达,收获菌体后进行冻干。然后,参见实施例4的操作对各突变体进行酶活检测。
35.本实施例中使用的引物如下表1所示。
36.表1引物的核苷酸序列
37.其中,相同位点的突变体使用相同的上游引物。
38.实施例4酶活检测与生物还原反应
39.以香草酸底物,用含有辅酶循环系统的生物还原反应体系确定羧酸还原酶mscar及其突变株对香草酸的相对活性大小。羧酸还原酶相对活力的检测步骤为:向2ml ep管中加入5mg羧酸还原酶冻干粉和5mg葡萄糖脱氢酶(gdh)冻干粉,再加入50μl辅酶nadph(5mm)、50μl辅底物atp(100mm)、50μl dmso、50μl葡萄糖(125mm)、及50μl的底物香草酸(100mm,溶于dmso),最后用磷酸盐缓冲液(20mm,ph 8.0)补充至0.5ml。将ep管放入35℃恒温摇床中在
200rpm的条件下进行12h。反应结束后,用50μl 1m的盐酸终止反应。将混合液放入离心机中以14000rpm转速离心5
‑
10min使反应体系中的固相和液相分离。将上清液用移液器吸出后经滤膜过滤并放入液相样品瓶内测试反应的转化率。检测条件为:搭载美国安捷伦公司的infinitylab poroshell120ec
‑
c18 column(4.6mm
×
100mm,2.7μm)色谱柱的液相色谱,按照5μl的进样量、0.5ml/min的流速、283nm的紫外检测器检测波长的分析条件,采用乙腈:0.1%的甲酸溶液=8:2的流动相进行10min的液相检测,香草酸的保留时间为3.62min,香草醛的保留时间为5.77min。
40.其中,通过序列保守性分析构建突变体库并进行活力筛选所得的结果如图2所示;通过虚拟突变所得突变体库并进行活力筛选所得的结果如图3所示。在第一步根据序列保守性对铰链区进行突变研究时发现单突变株r505e、r505i、r505m、r505q、n506k以及双突变株r505i/n506k的催化活力均有所提高;在第二步对虚拟突变的结果进行测试时,发现8个突变株的催化活力均高于野生型酶,且突变株r505f/n506g具有最高的催化活力。
41.实施例5羧酸还原酶mscar及其突变体的纯化
42.羧酸还原酶纯化的操作过程如下:1、菌体收集与破碎,离心收集lb培养基中的菌体,经洗涤后用ph 8.0的tris
‑
hcl缓冲液将其重悬(浓度为50g/l)破碎,弃沉淀将上清用0.45μm膜进行过滤;2、柱平衡,用含20mm咪唑的tris
‑
hcl缓冲液对镍柱进行平衡,冲洗至检测器示数稳定;3、蛋白上样,将过滤后的上清液以1ml/min的流速通过镍柱进行蛋白吸附;4、用10倍体积的含20mm及50mm咪唑的tris
‑
hcl缓冲液以2ml/min的流速进行杂蛋白洗脱,至检测器示数稳定;5、用含200mm咪唑的tris
‑
hcl缓冲液洗脱镍柱中的目的蛋白并收集流出液;5、用含500mm咪唑的tris
‑
hcl缓冲液清洗镍柱,将全部蛋白洗脱干净;6、用20%乙醇水溶液冲洗镍柱后保存以防止杂菌滋生;7、将收集洗脱液进行sds
‑
page电泳检测;8、经电泳检测正确后,将样品转移至超滤管,将超滤管以4000rpm在4℃条件下离心20min,弃滤出液,用tris
‑
hcl缓冲液进行缓冲液置换以除去咪唑;9、向纯酶中加入甘油(终浓度25%)保存于
‑
40℃冰箱备用。
43.其中,羧酸还原酶mscar各突变株的蛋白表达sds
‑
page图如图4所示;羧酸还原酶mscar突变株r505f/n506g的蛋白纯化sds
‑
page图如图5所示。
44.以上所述的,仅为本发明的较佳实施例,并非用以限定本发明的范围,本发明的上述实施例还可以做出各种变化。即凡是依据本发明申请的权利要求书及说明书内容所作的简单、等效变化与修饰,皆落入本发明的权利要求保护范围。本发明未详尽描述的均为常规技术内容。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1