肿瘤相关科学文献和科学数据的非结构化整合分析方法与流程
本发明涉及肿瘤医学领域,具体涉及一种肿瘤相关科学文献和科学数据的非结构化整合分析方法。
背景技术:
随着计算机、网络、大数据等技术的发展,生物医学的实验手段和研究方法均发生了巨大的变革,由此带来了领域内实验数据的“指数性”增长,得到了各行业专家学者的广泛关注并取得了大量研究成果,积累了海量的科学文献。其数目之大,增长速度之快远远超过了其他学科领域。这些文献资源以规范化的方式存储在pubmedcentral(文中其它地方以pmc简称)、sinomed等数据库中,为肿瘤的转化医学研究提供了一定的基础。
另一方面,随着高通量测序技术的发展及肿瘤相关科研项目的不断开展,积累了很多跟肿瘤基因组、表型组、临床等相关的数据信息(比如样本属性、临床因素、患者预后、dna序列信息、表达谱芯片信息、甲基化状况等),越来越多的平台开始对这些数据进行结构化存储、整理与分析。例如,截至到2015年1月份,tcga(thecancergenomeatlas)(http://cancergenome.nih.gov/)存储了33种肿瘤的不同类型的组学数据。同时,科学家开始倡导开展全球数据共享机制,海量的科学数据逐渐成为可开放获取的,为肿瘤研究提供了非常宝贵的资源。例如,研究者可以对这些可开放获取的数据重新进行整合分析,探索新的科学假设;或者提取这些数据中的信息来验证自己的研究成果。这种科学数据的开放获取与重利用在一定程度上促进了科研成果的产生。
然而,在实现了对肿瘤高通量转化医学科研数据的存储和管理基础上,需要进一步从用户的角度来考虑信息的获取方式。比如用户输入查询词,如何准确地理解用户的需求、提供全面的科学文献和科学数据的关联关系的检索结果以及便捷的数据下载方式,是需要解决的关键问题。加强肿瘤相关科学数据资源的开发和利用,构建转化医学研究中肿瘤相关开放科学数据与科学文献整合平台,支持科研人员及时跟进肿瘤基因组学的最新研究成果,具有重要的科学意义。
对于实际检索过程中,从用户的角度来说,我们发现科学数据与科学文献的关联检索是一个非常大的需求。而现有的数据库/平台只能基于特定的关键词单独对科学文献或者科学数据进行检索。而不能针对特定的数据集找到相关的文献,或者从科学文献链接到相关的科学数据。比如,我们可以通过关键词或者ftp获取相关的科学数据或者科学文献列表。但是,对于特定文献研究中涉及到的数据集,我们无法进行准确地定位或者关联。此外,对于数据库中存储的科学数据的共享和利用情况,不能很好地进行检索与分析。
技术实现要素:
本发明旨在至少解决上述技术问题之一。
为此,本发明的目的在于提出一种肿瘤相关科学文献和科学数据的非结构化整合分析方法。
为了实现上述目的,本发明的实施例公开了一种肿瘤相关科学文献和科学数据的非结构化整合分析方法,包括以下步骤:从开放平台获取肿瘤科学数据和肿瘤科学文献;对所述肿瘤科学数据和所述肿瘤科学文献进行结构化存储和归类,并建立索引;利用自然语言处理技术对所述肿瘤科学文献进行处理,提取文献所研究的肿瘤类型、采用的肿瘤科学数据类型信息,其中,所述肿瘤科学数据类型的相关信息包括基于原数据集发表的肿瘤科学文献和基于数据重利用发表的肿瘤科学文献;对所述肿瘤科学数据的重利用情况进行统计性分析,得到热门研究的科学数据类型、不同数据类型之间的相关性和汇总整合分析策略;或对所述肿瘤科学文献的进行统计性分析,提取所述肿瘤科学文献对应的期刊信息、作者信息和所在国家信息,进一步将信息汇总。
根据本发明实施例的肿瘤相关科学文献和科学数据的非结构化整合分析方法,能够将主流的肿瘤科学数据存储平台与可开放获取的科学文献数据关联起来,使用户能够查询到现有的肿瘤研究趋势(通过对肿瘤科学数据的重利用情况的分析,发现不同肿瘤类型及高通量数据类型、临床数据类型等研究热度);基于特定科学数据集,检索到与该数据集相关的科学文献;基于特定的科学文献,检索到跟该文献相关的数据集。
另外,根据本发明上述实施例的肿瘤相关科学文献和科学数据的非结构化整合分析方法,还可以具有如下附加的技术特征:
进一步地,所述肿瘤科学数据的获取来源包括国际癌症基因组图谱计划和国际癌症基因组联盟。
进一步地,所述肿瘤科学数据类型的来源包括美国国家生物技术信息中心,收集所述美国国家生物技术信息中心中的肿瘤科学文献的表达方式和所述表达方式的同义词总结肿瘤科学文献目录。
进一步地,所述肿瘤类型来源包括所述国际癌症基因组图谱计划,根据所述美国国家生物技术信息中心中的肿瘤类型信息、肿瘤畸变信息和疾病本体信息总结肿瘤类型目录。
进一步地,所述利用自然语言处理技术对所述肿瘤科学文献进行处理的方式包括结构分析、关键词查找和语句分析。
进一步地,所述对所述肿瘤科学数据的共享和重利用情况进行统计性分析,得到相关联的肿瘤科学文献进一步包括:辨识所述肿瘤科学文献中不同的肿瘤类别、组学数据类型;对所述相关的肿瘤科学文献进行趋势统计和预测,构建肿瘤相关科学数据与科学文献的智能化查询界面,通过渐次锁定科学文献与科学数据的关系将使用户能够从所关心的肿瘤类型或者数据类型出发,找到相应的跟特定肿瘤数据库相关的科学文献。
本发明的附加方面和优点将在下面的描述中部分给出,部分将从下面的描述中变得明显,或通过本发明的实践了解到。
附图说明
本发明的上述和/或附加的方面和优点从结合下面附图对实施例的描述中将变得明显和容易理解,其中:
图1是本发明一个实施例的肿瘤相关科学文献和科学数据的非结构化整合分析方法的流程图;
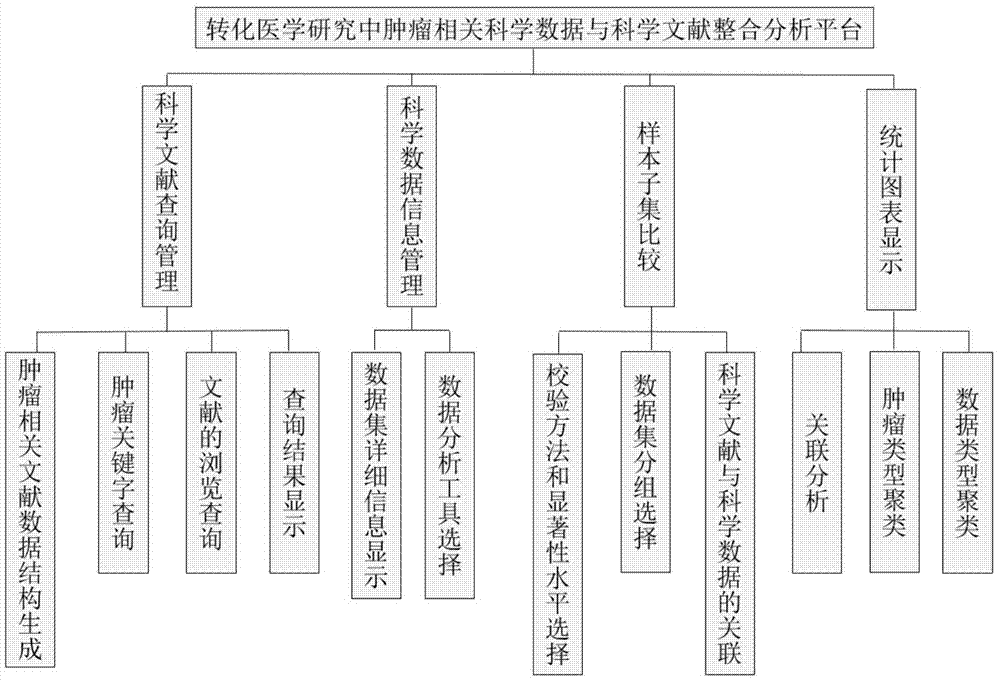
图2是本发明一个实施例的肿瘤相关科学文献和科学数据的非结构化整合分析系统的结构示意图。
具体实施方式
下面详细描述本发明的实施例,所述实施例的示例在附图中示出,其中自始至终相同或类似的标号表示相同或类似的元件或具有相同或类似功能的元件。下面通过参考附图描述的实施例是示例性的,仅用于解释本发明,而不能理解为对本发明的限制。
在本发明的描述中,需要理解的是,术语“中心”、“纵向”、“横向”、“上”、“下”、“前”、“后”、“左”、“右”、“竖直”、“水平”、“顶”、“底”、“内”、“外”等指示的方位或位置关系为基于附图所示的方位或位置关系,仅是为了便于描述本发明和简化描述,而不是指示或暗示所指的装置或元件必须具有特定的方位、以特定的方位构造和操作,因此不能理解为对本发明的限制。此外,术语“第一”、“第二”仅用于描述目的,而不能理解为指示或暗示相对重要性。
在本发明的描述中,需要说明的是,除非另有明确的规定和限定,术语“安装”、“相连”、“连接”应做广义理解,例如,可以是固定连接,也可以是可拆卸连接,或一体地连接;可以是机械连接,也可以是电连接;可以是直接相连,也可以通过中间媒介间接相连,可以是两个元件内部的连通。对于本领域的普通技术人员而言,可以具体情况理解上述术语在本发明中的具体含义。
参照下面的描述和附图,将清楚本发明的实施例的这些和其他方面。在这些描述和附图中,具体公开了本发明的实施例中的一些特定实施方式,来表示实施本发明的实施例的原理的一些方式,但是应当理解,本发明的实施例的范围不受此限制。相反,本发明的实施例包括落入所附加权利要求书的精神和内涵范围内的所有变化、修改和等同物。
以下结合附图描述根据本发明实施例的肿瘤相关科学文献和科学数据的非结构化整合分析方法。
图1是本发明一个实施例的肿瘤相关科学文献和科学数据的非结构化整合分析方法的流程图。图2是本发明一个实施例的肿瘤相关科学文献和科学数据的非结构化整合分析系统的结构示意图。请参考图1和图2,本发明实施例的肿瘤相关科学文献和科学数据的非结构化整合分析方法,包括以下步骤:
步骤1:从开放平台获取肿瘤科学数据和肿瘤科学文献。
在本发明的一个实施例中,肿瘤科学数据的获取来源包括国际癌症基因组图谱计划(thecancergenomeatlas(tcga))、国际癌症基因组联盟(theinternationalcancergenomeconsortium(icgc))和(thecatalogueofsomaticmutationsincancer(cosmic))等。
在本发明的一个示例中,国际肿瘤基因组图谱计划tcga中存储了大量不同肿瘤不同类型的数据及相关的研究成果,其数据被广泛应用于肿瘤基因组的研究之中。首先从科学文献数据库pubmed中采集从2008年开始到目前为止的跟tcga相关的文献。通过关键词查询及条件限定,共下载到了5372条跟tcga相关的xml格式的科学文献的数据信息。经过对xml数据的初步解析发现,其中一些文献是在参考文献中引用了tcga或者其全称cancergenomeatlas相关的文章,对进一步过滤掉这部分的文献信息,剩余5千篇左右的文献。同时,从tcga官方网站(http://cancergenome.nih.gov/publications)上随机下载了25篇可开放获取的文章,这些文献是被证实的对tcga中的数据集进行了重利用的文章,作为标准的对照。
在本发明的一个实施例中,肿瘤科学文献的来源包括美国国家生物技术信息中心(pubmedcentral)等。
步骤2:对肿瘤科学数据和所述科学文献进行结构化存储和归类,并建立索引。
在本发明的一个实施例中,肿瘤科学数据的获取来源包括:国际癌症基因组图谱计划tcga,通过收集tcgadataportal里面的terms,尽量考虑到不同terms的同义词存在的情况,总结出一套数据类型list,作为参考;肿瘤类型来源包括:tcga中的cancertype全称及abberation。为了更系统地囊括作者可能使用的关于肿瘤类型的同义词,综合肿瘤diseaseontology(http://disease-ontology.org/)的信息,总结肿瘤类型list。
步骤3:利用自然语言处理技术对肿瘤科学文献进行处理,提取文献所研究的肿瘤类型、采用的肿瘤科学数据类型的相关信息。其中,肿瘤科学数据数据类型的相关信息包括基于原数据集发表的肿瘤科学文献和基于数据重利用发表的肿瘤科学文献。
在本发明的一个实施例中,利用自然语言处理技术对所述肿瘤科学文献进行处理的方式包括结构分析、关键词查找和语句分析等。步骤4:对肿瘤科学数据的共享和重利用情况进行统计性分析,得到相关联的肿瘤科学文献;或对肿瘤科学文献的进行统计性分析,找到相关联的肿瘤科学数据。
在本发明的一个示例中,对过滤后的tcga相关文献的发表时间、国家、期刊等信息通过高通量测序技术进行统计分析。例如,自2010年威斯康辛大学的科研人员第一次成功地将基因组测序技术应用于疾病的诊断和治疗当中,人们更加关注从基因组、转录组合表观组等分子水平来探索人类复杂疾病的致病机理,并探索将研究结果推广至临床应用。从文献来源国来分析,tcga相关文献来源于37个国家,而美国对肿瘤基因组研究贡献的文章数是最多的,其次是中国、加拿大、澳大利亚等。面对日益增长的肿瘤基因组文献,需要采用精细的方法深入分析。进一步对这部分科学文献的出版期刊的信息,发现了749个期刊,而且其中大部分是可开放获取的。发表tcga相关文献数最多的期刊有plosone,ontotarget,nucleicacidsresearch等。而其中很大一部分比例的期刊是可开放获取的,这为肿瘤基因组研究人员提供了很大的便利。
在本发明的另一个示例中,从pmc上获取的文献信息与从tcga网站上下载的重利用了tcga数据的文献信息进行了比较分析。前者可以认为是包含了真正使用了tcga数据或者与tcga无关而仅仅是在文章中提到了这个项目,而后者则是被证实的真正使用了tcga数据的文章,因此,这两个数据集可以进行比较分析。具体而言,首先对文献的xml格式进行解析,将文献具体内容分成以下几个部分:title,abstract,keywords,introduction/background,method/material,result,discussion/conclusion,other。其次,用自然语言处理技术分析关键词tcga或者其全称cancergenomeatlas在文献的不同位置的分布情况。对于pmc中的5千多篇tcga相关文献,采用随机抽样的方法,每次抽取24篇文献,一共抽取1000次,并对多次抽样的结果取均值作为最终结果。对比分析发现在先验数据集(即从tcga官网上下载的真正使用了tcga数据的文献)中,关键词tcga或其全称cancergenomeatlas在文献的result或者method/material中出现的频率是最高的,几乎能达到90%,后续将这部分的信息作为进一步解析tcga相关文献的依据。
在本发明的一个实施例中,对肿瘤科学数据的共享和重利用情况进行统计性分析,得到相关联的肿瘤科学文献进一步包括:按照不同的肿瘤类别、组学数据类型解析识别相关的肿瘤科学文献。对相关的肿瘤科学文献进行趋势统计和预测,构建肿瘤相关科学数据与科学文献的智能化查询界面,使用户能够从所关心的肿瘤类型或者数据类型出发,找到相应的跟特定肿瘤数据库相关的科学文献。其中,智能化查询界面包括:1.后台数据存储管理;2.基于自然语言处理技术(提取文献中关于肿瘤类型、科学数据信息,通过限定关键词在语段中的聚类,推断相互关系)解析科学文献中的科学数据信息;3.将科学数据与科学文献建立关联,构建搜索页面,供查询;4.统计分析模块包括科学文献的统计分析(所在期刊、是否可开放获取、作者、国家)、科学文献中的科学数据统计分析(数据类型汇总、不同数据类型之间的相关性)。具体呈现框架模块参考图2。
另外,本发明实施例的肿瘤相关科学文献和科学数据的非结构化整合分析方法的其它构成以及作用对于本领域的技术人
员而言都是已知的,为了减少冗余,不做赘述。
在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不一定指的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任何的一个或多个实施例或示例中以合适的方式结合。
尽管已经示出和描述了本发明的实施例,本领域的普通技术人员可以理解:在不脱离本发明的原理和宗旨的情况下可以对这些实施例进行多种变化、修改、替换和变型,本发明的范围由权利要求及其等同限定。
- 还没有人留言评论。精彩留言会获得点赞!