一种自适应变异粒子群优化算法的制作方法
本算法属于数据挖掘领域,涉及到SVM分类器和参数寻优,特别涉及到其变异操作,使得能够最大限度的搜索到全局最优解。
背景技术:
网格划分法是SVM分类器中常用的一种参数寻优方法,原因如下:一是网格划分法是全局搜索,我们能得到全局最优解,从心理上我们会感到更安全相比于使用避免穷举参数搜索的启发式方法;第二是通过网格划分法找到最优参数值所花费的时间并不比那些先进的算法多,由于只有两个参数。此外,网格划分容易并行处理因为每对(C,g)是相互独立的。但是有时在实际应用问题中,我们要在广度十分庞大的区间上搜寻最佳参数C和g,这时再利用网格划分法会很浪费时间,因此采用粒子群算法可以避免遍历所有的样本点,也有可能找到全局最优解,并且节省了时间。
但是基本的粒子群优化算法(PSO)与其他进化计算算法类似,它也是基于种群迭代的算法。因此,该算法将低效率地计算并通过所需的评估函数的数量来衡量。此外,虽然粒子群优化算法最主要的优势之一就是其快速的收敛性,但是这也可能是个问题,因为如果一个早熟的解是局部最优解,这个种群在没有任何外在的压力下将很容易陷入搜索停滞(即停止继续搜索),我们也称之为早熟收敛。特别当解决复杂的多峰问题时,基本的PSO算法很容易陷入局部最优。这些缺点限制了PSO更广泛的应用。
技术实现要素:
为了避免网格划分进行全局搜索耗时、基本粒子群算法容易陷入早熟收敛(即局部最优)等问题,提出了自适应变异粒子群优化算法。它能够在不进行全局搜索的前提下,最大限度的找到全局最优解,提高分类器性能。
本发明从遗传算法的变异思想中得到启发,在PSO中引入变异操作,即对整个种群位置引入变异概率因子,这种变异操作通常是在每代或在前缀间隔或在自适应策略控制下用于停滞代数(早熟收敛)的一个触发器。它能够跳出当 前搜索到的局部最优位置,在更大的解空间中再次搜索,在扩大解空间搜索范围的同时,又保持了种群的多样性,使得算法能够有效地进行全局搜索,从而提高种群搜索全局最优解的能力。
本发明的技术方案是:
一种自适应变异粒子群优化算法,步骤如下:
设粒子群的粒子数目为N,fi为第i个粒子的适应度,favg为粒子目前的平均适应度,σ2为粒子群的群体适应度方差,则σ2可以定义为:
上式中f是归一化定标因子,它是用来限制σ2的大小。这里群体适应度方差σ2反映的是粒子群中所有粒子的收敛程度。σ2越小,说明粒子越趋于收敛;反之,粒子群则处于随机搜索阶段。当σ2等于0时,则说明粒子群优化算法陷入早熟收敛或者达到全局收敛。为了避免陷入早熟收敛,我们要对粒子群进行位置变异操作,具体形式如下:
xi(k+1)=C·rand()·xi(k) (2)
C为变异因子,服从Gauss(0,1)分布的随机变量;k为当前迭代次数,rand()为(0,1)之间的随机数。考虑到粒子在当前的gbest的作用下可能寻找到更好的位置,因此新算法将变异操作引入一个随机算子,即满足变异条件的xi(k+1)按一定概率pk变异。pk可以在[0.1,0.5]之间取值,随机产生。当粒子进行位置变异后,它将改变原有的搜索方向,使其进入其它区域进行搜索,可能发现新的pbest和gbest。如此循环迭代,变异算法增大了发现全局最优解的概率。
自适应变异粒子群优化算法流程图如图1所示。
本发明算法的有益效果是,相比于网格划分法收敛快,耗时少;相比于基本粒子群算法,它有跳出局部最优的能力,达到全局最优,提高分类器准确率。
附图说明
下面结合附图和实施例对本发明进一步说明。
图1是自适应变异粒子群优化算法过程图。
图2是基本粒子群算法训练样本的适应度曲线图。
图3是自适应变异粒子群算法训练样本的适应度曲线图。
图4是(网格划分)SVM训练集预测结果分类图。
图5是(网格划分)SVM测试集预测结果分类图。
图6是(基本粒子群算法)SVM训练集预测结果分类图。
图7是(基本粒子群算法)SVM测试集预测结果分类图。
图8是(自适应变异粒子群算法)SVM训练集预测结果分类图。
图9是(自适应变异粒子群算法)SVM测试集预测结果分类图。
具体实施方式
在图1中,与基本粒子群优化算法不同的是,在自适应变异粒子群优化算法的流程中我们加入了早熟收敛判断和粒子位置变异操作。即当速度更新和位置更新之后,我们计算群体适应度方差σ2和随机算子pk,然后产生随机数r(r∈[0,1]),判断r是否小于pk,如果小于则进行位置变异操作,判断是否收敛,如果是则得出全局最优解,否则再次更新速度和位置,进行正常迭代;如果r大于等于pk则直接判断是否收敛,步骤同上。
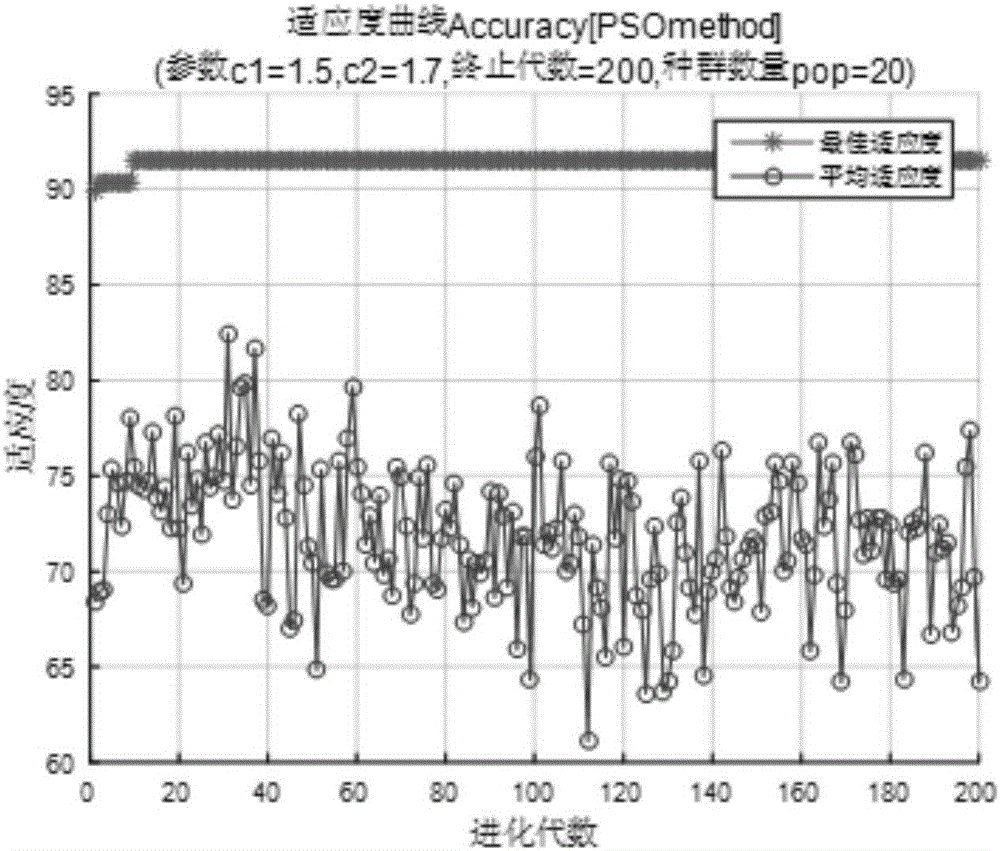
在图2所示,适应度函数在第10代附近达到最大值,此时best c=5.17713,best g=3.12506,bestacc=91.4729%。与网格划分相比,最优准确率从94.186%下降到91.4729%,这符合基本PSO算法的特点,一旦陷入局部最优值后,它就很难再有突破,即出现早熟现象。
在图3所示,适应度函数在第95代附近达到最大值,此时best c=100,best g=0.178551,bestacc=94.186%。自适应变异PSO与网格划分法相比,最优准确率相同都为94.186%,收敛速度与基本的PSO大体相当,但是有很强的突破局部收敛的能力,从适应度值明显的呈阶梯状上升可以看出,最终能够达到全局最优。
在图4所示,258例训练样本中,预测错误总计9例,其中肝癌141例中错误7例,肝病117例中错误2例,因此分类准确率为96.5116%。
在图5所示,172例测试样本中,预测错误总计10例,其中肝癌94例中错误5例,肝病78例中错误5例,因此分类准确率为94.186%。
在图6所示,258例训练样本中,预测错误总计6例,其中肝癌141例中错误5例,肝病117例中错误1例,因此分类准确率为97.6744%。
在图7所示,172例测试样本中,预测错误总计12例,其中肝癌94例中错误7例,肝病78例中错误5例,因此分类准确率为93.0233%。与网格划分法分类器的预测准确率相比,准确率并没有进一步提高,这与取到的参数c和g陷入局部最优有关。
在图8所示,258例训练样本中,预测错误总计5例,其中肝癌141例中错误4例,肝病117例中错误1例,因此分类准确率为98.062%。
在图9所示,172例测试样本中,预测错误总计8例,其中肝癌94例中错误5例,肝病78例中错误3例,因此分类准确率为95.3488%。与网格划分法分类器的预测准。
实验例:
在matlab中,我们首先利用基本的粒子群算法对构建的分类器中的参数进行优化。这里,我们先要对普通PSO函数的参数进行初始化,设置参数局部搜索能力c1=1.5,参数全局搜索能力c2=1.7;设置速度更新公式中速度前面的弹性系数ω=1;设置最大进化数量maxgen=200,种群最大数量sizepop=20;设置交叉验证的折叠次数v=5;最后设置参数c的变化范围在[0.1,100]之间,参数g的变化范围在[0.01,1000]之间。
首先利用基本的粒子群优化函数对SVM分类器的最佳参数进行寻找,适应度曲线如图2所示:
在利用自适应变异粒子群算法对SVM分类器的最佳参数进行寻找,适应度曲线如图3所示:
仿真例:
(1)利用Grid-RBF核函数构建的分类模型(c=90.5097,g=0.25)分别对训练集和测试集进行仿真实验。训练集样本数258例,其中141例为肝癌,117例为肝病,仿真结果如图4所示:
测试集样本数172例,其中94例为肝癌,78例为肝病,仿真结果如图5所示:
(2)利用基本的粒子群算法寻找的最佳参数best c=5.17713和best g=3.12506构建的分类模型对训练集和测试集进行仿真实验。训练集样本数258例,其中141例为肝癌,117例为肝病,仿真结果如图6所示:
测试集样本数172例,其中94例为肝癌,78例为肝病,仿真结果如图7所示:
(3)再利用自适应变异粒子群算法寻找的最佳参数best c=100和best g=0.178551构建的分类模型对训练集和测试集进行仿真实验。仿真结果如图8所示:
测试集样本数172例,其中94例为肝癌,78例为肝病,仿真结果如图9所示:
最终可以看出提出的自适应变异粒子群优化算法解决了上面提到的两个问题,并提高了分类器性能。
- 还没有人留言评论。精彩留言会获得点赞!