增加营养物利用效率的方法与流程
发明领域
本发明涉及用于增加植物中的氮摄取、氮同化、氮利用效率以及产量而不影响植物高度的方法,该方法包括增加生长调节因子(grf)的表达或水平。还描述了以上述表型表征的遗传改变的植物以及产生此类植物的方法。
背景技术:
1960年代的农业“绿色革命”提高了谷物产量,为不断增长的世界人口提供了食物,并且由半矮化绿色革命品种(grv)1-3的快速采用所推动。大多数现代小麦和稻优良品种保留了grv的半矮化基因特征4-6。但是,半矮化grv土壤氮(n)利用效率低7,高产量严重依赖于氮肥投入,所述氮肥投入不仅是主要的投入成本,而且对环境造成不可持续的破坏。因此,开发高产且氮施肥减少的新品种是全球迫切重要性的战略性可持续农业目标1,8。
因此,需要增加商业上重要的谷类作物如稻和小麦中的氮摄取以及产量,但更重要的是,在半矮化绿色革命品种中增加氮摄取以及产量,而不损失半矮态的产量益处。本发明解决了这种需要。
发明概述
为了提高grv的营养物利用效率,必须了解生长和代谢之间的调节关系。迄今为止,这种关系的分子机制仍然很大程度上未知。在这里,我们显示稻生长调节因子4(osgrf4)与della生长抑制剂直接相互作用,并且这种相互作用赋予碳(c)-氮(n)平衡的稳态共调节。osgrf4促进和整合c固定、n同化和细胞增殖,而della抑制它们。grv的della积累使平衡倾斜,以有利于具有降低的n同化的半矮态。相反,我们显示grvosgrf4丰度的增加改变了osgrf4-della平衡,有利于c(碳)和n(氮)同化的增加,而不损失产量增强的矮态。因此,调节植物生长和代谢的协调提高了n利用效率和产量,使得能够进行战略性育种以持续地提高全球粮食安全。
在本发明的第一方面,提供了提高植物中氮摄取和/或氮同化和/或氮利用效率的方法,所述方法包括提高生长调节因子(grf)的表达或水平或提高生长调节因子的活性。
在进一步的实施方案中,所述方法进一步包括提高植物的籽粒产量。优选地,籽粒产量的增加选自每穗或每株植物的籽粒数量的增加和/或1000籽粒重量的增加。
在另一个实施方案中,所述方法进一步包括提高c同化,如下所述。
在本发明的另一个方面,提供了产生具有提高的氮摄取和/或氮同化和/或氮利用效率的植物的方法,所述方法包括提高生长调节因子(grf)的表达或水平或提高生长调节因子的活性。优选地,所述植物还具有增加的产量和/或增加的c同化。
在一个实施方案中,所述方法进一步包括测量氮摄取和/或氮同化和/或氮利用效率和籽粒产量中至少一种的增加。
在另一个实施方案中,所述方法进一步包括使植物再生并筛选氮摄取和/或氮同化和/或氮利用效率和籽粒产量的增加。
在一个实施方案中,所述方法包括将至少一个突变引入编码grf和/或grf启动子的至少一个核酸中。优选地,突变是置换。在一个实施方案中,突变是在微小rna(mirna)结合位点中,优选在mirna396结合位点中。在备选的实施方案中,突变是在grf启动子中。
优选地,grf是grf4或其功能变体或同源物。在一个实施方案中,核酸编码grf多肽,其中grf多肽包含seqidno:3或其功能变体或同源物,或由其组成。优选地,核酸包含seqidno:1或2或其功能变体或同源物,或由其组成。在进一步的实施方案中,编码grf启动子的核酸包含seqidno:7或8或其功能变体或同源物,或由其组成。
在一个实施方案中,所述突变是使用靶向基因组修饰,优选zfn、talen或crispr/cas9引入的。
在备选的实施方案中,所述方法包括在植物中引入和表达包含grf核酸的核酸构建体。优选地,grf核酸构建体与调节序列可操作的连接。更优选地,调节序列是组成型启动子。甚至更优选地,调节序列是如seqidno:9中定义的grf启动子或其功能变体或同源物。优选地,grf核酸编码grf多肽,其中grf多肽包含seqidno:3或6或其功能变体或同源物,或由其组成。更优选地,核酸包含seqidno:1、2、4或5,或由其组成。
在一个实施方案中,所述氮摄取和/或氮同化和/或氮利用效率和/或产量和/或c代谢的增加是相对于野生型或对照植物而言的。优选地,氮摄取和/或氮同化和/或氮利用效率是在植物的枝条(shoot)和/或根中增加的。更优选地,氮摄取和/或氮同化和/或氮利用效率是在低或高氮条件下,优选在低氮条件下增加的。
在优选的实施方案中,植物高度不受影响。
在本发明的另一个方面,提供了遗传改变的植物、其部分或植物细胞,其中生长调节因子(grf)的表达或水平或grf的活性与野生型或对照植物相比增加,且其中所述植物的特征在于与野生型或对照植物相比氮摄取和/或氮同化和/或氮利用效率中至少一种的增加。优选地,所述植物还具有增加的产量和/或增加的c同化。
在一个实施方案中,植物表达包含grf核酸的核酸构建体。优选地,核酸构建体包含调节序列。更优选地,调节序列是组成型启动子。在一个实施方案中,调节序列是如seqidno:9中定义的grf启动子或其功能变体或同源物。
在备选的实施方案中,植物在至少一个编码grf多肽和/或grf启动子的核酸中包含至少一个突变。优选地,突变是置换。更优选地,所述突变是使用靶向基因组修饰,优选zfn、talen或crispr/cas9引入的。在一个实施方案中,突变是在微小rna(mirna)结合位点中,优选在mirna396结合位点中。在另一个实施方案中,突变是在grf启动子中。
在一个实施方案中,所述植物部分是籽粒或种子。
在本发明的另一个方面,提供了用于鉴定和/或选择优选与野生型或对照植物相比具有增加的氮摄取和/或氮同化和/或氮利用效率的植物的方法,该方法包括在植物或植物种质中检测至少一个grf基因或grf启动子中的至少一个多态性并选择所述植物或其后代。优选地,多态性是置换。更优选地,所述方法进一步包括将在grf基因或启动子中包含至少一个多态性的染色体区域基因渗入(introgress)至第二植物或植物种质中,以产生基因渗入的植物或植物种质。
在本发明的进一步的方面,提供了包含grf核酸和优选地调节序列的核酸构建体,其中grf核酸编码grf多肽,其中grf多肽包含seqidno:3或6或其功能变体或同源物或由其组成。优选地,调节序列是组成型启动子。在一个实施方案中,调节序列是如seqidno:9中定义的grf启动子或其功能变体或同源物。更优选地,核酸包含seqidno:1、2、4或5,或由其组成。
在本发明的另一方面,提供了包含如本文所述的核酸构建体的载体。
在本发明的进一步的方面,提供了包含本文所述的核酸构建体的宿主细胞。优选地,细胞是细菌或植物细胞。
在本发明的另一个方面,提供了表达如本文所述的核酸构建体或载体的转基因植物。
在本发明的另一个方面,提供了如本文定义的核酸构建体在增加植物中氮摄取和/或氮同化和/或氮利用效率中的用途。
在本发明的另一方面,提供了增加植物中氮摄取和/或氮同化和/或氮利用效率和/或产量和/或c同化的方法,该方法包括调节grf的表达和/或活性,其中该方法包括将至少一个突变引入grf基因,其中所述grf基因包含以下或由以下组成
a.编码如seqidno:3中所定义的多肽的核酸序列;或
b.如seqidno:1或2中所定义的核酸序列;或
c.与(a)或(b)具有至少75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或至少99%的总序列同一性的核酸序列;或
d.编码grf多肽的核酸序列,所述核酸序列能够在如本文所定义的严格条件下与(a)至(c)中任一项的核酸序列杂交。
在本发明的又进一步的方面中,提供了一种核酸构建体,其包含编码至少一个dna结合结构域的核酸序列,所述dna结合结构域能结合至少一个grf基因。
在一个实施方案中,所述核酸序列编码至少一个前间隔区元件,并且其中前间隔区元件的序列选自seqidno:50,53,58,61,66,69,74,77,82,86,91,94,99,102,107,110,115,118,123,126.131,134,139,142,147,150,155,158,163,166,171,174,179,182,187和190,或与seqidno:50,53,58,61,66,69,74,77,82,86,91,94,99,102,107,110,115,118,123,126.131,134,139,142,147,150,155,158,163,166,171,174,179,182,187和190具有至少90%同一性的序列。
在另外的实施方案中,所述构建体进一步包含编码crisprrna(crrna)序列的核酸序列,其中所述crrna序列包含所述前间隔区元件序列和额外的核苷酸。
在进一步的实施方案中,构建体进一步包含编码反式激活rna(tracrrna)的核酸序列,其中优选地,tracrrna在seqidno.46或其功能变体中定义。
在另一个实施方案中,构建体编码至少一个单向导rna(sgrna),其中所述sgrna包含tracrrna序列和crrna序列,其中sgrna包含选自seqidno51,54,59,62,67,70,75,78,83,87,92,95,100,103,108,111,116,119,124,127,132,135,140,143,148,151,156,159,164,167,172,175,180,183,188和191的序列或其变体,或由这些序列组成。
优选地,构建体可操作地连接至启动子。更优选地,启动子是组成型启动子。
在一个实施方案中,所述核酸构建体进一步包含编码crispr酶的核酸序列。优选地,所述crispr酶是cas蛋白。更优选地,所述cas蛋白是cas9或其功能变体。
在备选的实施方案中,所述核酸构建体编码tal效应子。优选地,所述核酸构建体进一步包含编码内切核酸酶或其dna切割结构域的序列。更优选地,所述内切核酸酶是fokⅰ。
在本发明的另一方面,提供了单向导(sg)rna分子,其中所述sgrna包含crrna序列和tracrrna序列,其中所述crrna序列能够结合选自seqidno:49,52,57,60,65,68,73,76,81,85,90,93,98,101,106,109,114,117,122,125,130,133,138,141,146,149,154,157,162,165,170,173,178,181,186和189或其变体的至少一个序列。
在本发明的另一方面,提供了包含dna供体核酸的核酸构建体,所述dna供体核酸选自seqidno:48,56,64,72,80,84,89,97,105,113,121,129,137,145,153,161,169,177和185或其变体,其中所述dna供体核酸与调节序列可操作地连接。优选地,构建体进一步包含至少一个sgrna,所述sgrna选自seqidno:51,54,59,62,67,70,75,78,83,87,92,95,100,103,108,111,116,119,124,127,132,135,140,143,148,151,156,159,164,167,172,175,180,183,188和191,其优选可操作地连接到调节序列。更优选地,构建体进一步包含编码crispr酶的核酸,该核酸优选地可操作地连接至调节序列。
在另一个方面,提供了用至少一个本文所述的核酸构建体或至少一个本文所述的sgrna转染的分离的植物细胞。
在本发明的进一步的方面,提供了分离的植物细胞,所述细胞用至少一种如本文所述的核酸构建体和第二核酸构建体转染,其中所述第二核酸构建体包含编码cas蛋白,优选cas9蛋白或其功能变体的核酸序列。在一个实施方案中,第二核酸构建体在本文所述的核酸构建体之前、之后或同时转染,优选仅包含sgrna核酸。
在本发明的另一个方面,提供了遗传修饰的植物,其中所述植物包含如本文所述的转染的细胞。
在本发明的进一步的方面,提供了如本文所述的遗传修饰的植物,其中编码sgrna的核酸和/或编码cas蛋白的核酸是以稳定的形式整合的。
在本发明的另一个方面,提供了增加植物中氮摄取和/或氮同化和/或氮利用效率和/或产量和/或c同化的方法,该方法包括在植物中引入和表达本文所述的核酸构建体或本文所述的sgrna,其中优选地所述增加是相对于对照或野生型植物而言的。
在另一个实施方案中,提供了如本文所定义的核酸构建体或如本文所述的sgrna在增加植物中氮摄取和/或氮同化和/或氮利用效率中的用途。优选地,核酸构建体或sgrna增加植物中grf的表达和/或活性。
在本发明的另一个方面,提供了用于获得如本文所述的遗传修饰植物的方法,所述方法包括:
a.选择植物的部分;
b.用本文所述的核酸构建体转染段落(a)所述植物的部分的至少一个细胞;
c.从所述转染的细胞再生至少一个植物;其通过选择根据段落(b)获得的一个或多个植物,其在所述植物中显示至少一种grf核酸的增加的表达。
在本发明的最后方面,提供了增加植物中碳代谢和/或细胞增殖(以及任选地,如上所述氮代谢)的方法,该方法包括增加grf4的表达或增加其水平。在一个实施方案中,碳代谢选自光合作用、碳信号传导、糖信号传导和蔗糖或韧皮部负载中的至少一种。在另一个实施方案中,细胞增殖包括细胞分裂。特别地,所述方法可以包括增加参与碳代谢和/或细胞信号传导的基因(例如细胞周期蛋白依赖性激酶)的表达。在一个实施方案中,细胞增殖的提高增加了叶和茎的宽度,但优选不增加茎的高度。
在任何上述方面的一个实施方案中,grf核酸编码grf多肽,其中grf多肽包含seqidno:3或6或其功能变体或同源物,或由其组成。优选地,核酸包含seqidno:1、2、4或5,或由其组成。在另一个实施方案中,编码grf启动子的核酸包含seqidno:7或8或其功能变体或同源物,或由其组成。
在一个实施方案中,grf是grf4或其同源物或直向同源物(orthologue)。
在任何上述方面的一个实施方案中,所述氮是硝酸盐或铵盐。
在本发明的另一个方面,提供了通过本文所述的任何方法获得的或可获得的植物。
在任何上述方面的一个实施方案中,所述植物是单子叶植物或双子叶植物。优选地,植物选自稻、玉米、小麦、大麦、高粱、马铃薯、番茄、大豆和欧洲油菜(b.napus)。更优选地,植物是稻。甚至更优选地,稻是籼稻或粳稻品种。
附图说明
在以下非限制性附图中进一步描述本发明:
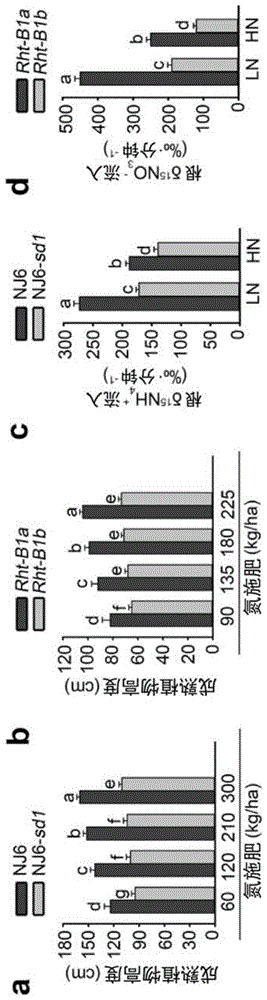
图1显示della积累抑制稻和小麦grv的生长n-应答和n-摄取。a,在不同n-供应方案中生长的稻植物的高度。b,在不同n-供应方案中生长的小麦植物的高度。数据(a,b)显示为平均值±s.e.m.(n=30)。c,在低(0.375mmnh4no3;ln)和高(1.25mmnh4no3;hn)n供应中,稻根15nh4+摄取率。d,在低(0.375mmca(no3)2;ln)和高(1.25mmca(no3)2;hn)n供应中,小麦根15no3-摄取率。数据(c,d)显示为平均值±s.e.m.(n=9)。统计学分析是使用duncan多范围检验进行的,相同的小写字母表示平均值之间的非显著性差异(p>0.05)。
图2显示osgrf4调节稻nh4+摄取和响应于n可用性的生长。a,品种的15nh4+摄取率(由低到高,从左到右排列),在高n供应(1.25mmnh4no3)中生长的植物。b,来自分布在12个稻染色体上的nj6xnh73bc1f2群体中15nh4+摄取率的qtl分析的lod得分。显示了主要峰(qngr1和qngr2)。c,15nh4+摄取率。数据显示为平均值±s.e.m.(n=9)。d,用抗osgrf4抗体显示osgrf4积累。hsp90作为负载对照。e,osgrf4mrna的相对丰度,在增加的n供应水平(0.15n,0.1875mmnh4no3;0.3n,0.375mmnh4no3;0.6n,0.75mmnh4no3;1n,1.25mmnh4no3)下生长的nj6根。丰度相对于1n(=1)表示。f,在不同水平的n供应下(如i中),抗osgrf4抗体显示的osgrf4蛋白在nj6中的积累。hsp90作为负载对照。g,功能缺失osgrf4突变体植物的可见表型。标尺,15cm。h,在增加的n-供应(如i中)中的15nh4+摄取率。数据显示为平均值±s.e.m.(n=9)。i,在增加的n供应中生长的植物的生物质(干重)。数据显示为平均值±s.e.m.(n=3)。统计学分析使用duncan多范围检验,相同的小写字母表示平均值之间的非显著性差异(p>0.05;图c,h和i)。
图3显示osgrf4是n代谢基因表达的主要协调者。a,成熟植物表型。标尺,15cm。b,15nh4+和15no3-摄取率。数据显示为平均值±s.e.m.(n=9)。统计学分析使用duncan多范围检验,相同的小写字母表示平均值之间的非显著性差异(p>0.05)。c,在具有增加的n供应的稻田条件下生长的稻植物的枝条中的谷氨酰胺合酶(gs)和硝酸还原酶(nr)活性。数据显示为平均值±s.e.m.(n=3)。d,rna-seq分析。nj6-sd1中有4883个基因的转录物丰度下调(相对于nj6),nj6-osgrf4ngr2中有5395个基因的转录丰度上调(相对于nj6),对于二者有642个共同的基因。e,相对于nj6的根mrna丰度(=1)。数据显示为平均值±s.e.m.(n=3)。f,相对于nj6的枝条mrna丰度。数据显示为平均值±s.e.m.(n=3)。g,具有flag标签的osgrf4的chip-seq富集的序列基序。h,emsa显示osgrf4-his与wt的结合,而不与核心gcgg基序的突变体(m1-m5)形式结合。i,具有flag-osgrf4的chip-pcr富集了含gcgg的启动子片段(用*标记)。j-k,在瞬时反式激活测定中的osgrf4激活启动子::萤光素酶融合构建体。数据显示为平均值±s.e.m.(n=3;图e-f,i-k)。
图4显示了竞争性osgrf4-osgif1-osslr1相互作用协调nh4+摄取和同化。a,在用100μmga(ga3)和/或2μm多效唑(pac)处理的植物中的15nh4+摄取率。数据显示为平均值±s.e.m.(n=9)。b,用ga和/或多效唑(pac)处理的植物中的相对根mrna丰度。数据显示为平均值±s.e.m.(n=3)。c,来自osamt1.1(片段5)和osgs1.2(片段2)的含gcgg启动子片段的chip-pcrosgrf4介导的富集程度(图2i所示)应答ga和/或pac处理而变化。数据显示为平均值±s.e.m.(n=3)。d,bifc测定显示slr1、osgrf4和osgif1在烟草叶表皮细胞核中的相互作用。比例尺,60μm。e,用flag标记的slr1和ha标记的osgrf4或ha标记的osgif1进行的co-ip实验。f,fret图像。供体组:仅osgif1-cfp;受体组:仅osgrf4-yfp;fret组:带有和不带有slr1的osgif1-cfp和osgrf4-yfp。标尺,200μm。g,osgif1-cfp和osgrf4-yfp通道的平均n-fret数据。数据显示为平均值±s.e.m.(n=6)。斯氏t检验产生p值。h,emsa测定显示osgrf4-his融合蛋白与来自osamt1.1启动子的含gcgg基序的dna片段的结合由osgif1促进,但被slr1抑制。i,反式激活测定。posamt1.1和posgs1.2启动子::萤光素酶融合构建体的osgrf4激活的促进是通过osgif1增强的,并通过slr1抑制。数据显示为平均值±s.e.m.(n=6)。统计学分析使用duncan多范围检验,相同的小写字母表示平均值之间的非显著性差异(p>0.05;图a-c和i)。
图5显示osgrf4-slr1拮抗作用调节碳同化和植物生长。a,从调节c固定的基因转录的mrna的相对丰度。数据显示为平均值±s.e.m.(n=3)。相对于nj6-sd1中的水平表达的丰度。b,chip-pcr测定。图中描述了用于chip-pcr的ospsbs1、ostps1和ossweet11启动子和区域。c,反式激活测定。数据显示为平均值±s.e.m.(n=9)。d,由细胞周期调节基因转录的mrna的相对丰度。数据显示为平均值±s.e.m.(n=3)。相对于nj6-sd1中的水平表达的丰度。e,chip-pcr测定。图中描述了用于chip-pcr的oscyca1.1和oscdc2os-3启动子和区域。f,反式激活测定。数据显示为平均值±s.e.m.(n=12)。统计学分析使用duncan多范围检验,相同的小写字母表示平均值之间的非显著性差异(p>0.05)。
图6显示升高的osgrf4丰度增加了稻和小麦grv的籽粒产量和n利用效率,而不增加成熟植物的高度。a,在低n供应(ln:60千克/公顷)和高n供应(hn:210千克/公顷)的稻田条件下生长的9311和9311-osgrf4ngr2稻的成熟植物表型。比例尺,15cm。b,增加n供应在稻田条件下生长的nil植物的高度。数据显示为平均值±s.e.m.(n=30)。c,响应于增加的n供应,田间生长植物的籽粒产量。数据显示为每n水平每品系六个地块(每个地块包含220株植物)的平均值±s.e.m.。统计学分析使用duncan多范围检验,相同的小写字母表示平均值之间的非显著性差异(p>0.05;图b,c)。d,b所示植物地上部分不同器官中氮的分配比例。数据显示为平均值±s.e.m.(n=30)。e,b中所示植物的c:n比。f,成熟的kn199和kn199p35s::osgrf4ngr2-gfp小麦植物。比例尺,15cm。g,(左)kn199和(右)kn199p35s::osgrf4ngr2-gfp小麦植物的最上节间的横截面。比例尺,2mm。h,kn199和kn199p35s::osgrf4ngr2-gfp小麦植物的穗长度的比较。比例尺,5cm。i,生物量积累。数据显示为平均值±s.e.m.(n=12)。j,根部15no3-摄取率,基因型如所示。数据显示为平均值±s.e.m.(n=9)。k,地上植物部分不同器官中氮的分布比较。数据显示为平均值±s.e.m.(n=9)。l,n浓度。数据显示为平均值±s.e.m.(n=20)。m,kn199和kn199p35s::osgrf4ngr2-gfp小麦植物的籽粒产量。数据显示为平均值±s.e.m.(n=30)。n,每穗的籽粒数目。数据显示为平均值±s.e.m.(n=30)。o,kn199和kn199p35s::osgrf4ngr2-gfp小麦植物的收获指数。数据显示为平均值±s.e.m.(n=6)。斯氏t检验用于生成p值(图e,i,j和l-o)。
图7显示osgrf4基因座处的等位基因变异影响osgrf4mrna的丰度和根部15nh4+的摄取。a,位置克隆表明osgrf4与qngr2的等效性(n介导的生长响应2)。连续图谱显示qngr2(灰色点,使用重组断裂点和连接的dna标记)的焦点逐渐变窄为2号染色体上的约2.7-kbp区域,其侧翼是分子标记l17和l18以及重叠的候选基因loc_os02g47280(也称为osgrf4)。显示了osgrf4的起始atg(核苷酸1)和闭合tga(核苷酸3385),以及编码蛋白质的dna序列(cds,粗黑色条)。osmir396的靶位点用*表示。b,在高氮浓度条件下(1.25mmnh4no3)生长的osgrf4ngr2或osgrf4ngr2纯合或杂合的bc2f2后代(源自nj6xnm73杂交)的根部15nh4+摄取率。数据显示为平均值±s.e.m.(n=9)。相同的小写字母表示平均值之间无显著差异(p>0.05)。c,相对于nj6(=1)中的丰度,植物中osgrf4mrna的丰度(如所示的基因型)。数据显示为平均值±s.e.m.(n=3)。d,osgrf4基因座处的天然等位基因变异。显示了相对于osgrf4起始atg的核苷酸位置。突出显示了nm73和rd23品种之间共享的snp。显示了代表osgrf4启动子单倍型a,b和c的序列(参见正文)。e,各种稻品种中osgrf4mrna的丰度。数据显示为平均值±s.e.m.(n=3)。丰度数据都是相对于稻actin2mrna的丰度。f,在高(1n)或低(0.3n)n条件下生长的所选稻品种中的osgrf4mrna。数据显示为平均值±s.e.m.(n=3)。丰度数据都是相对于1n(=1)中的数据。
图8显示,osmir396丰度没有通过增加n供应而可检测地增加。在生长于不同n供应水平(0.15n,0.1875mmnh4no3;0.3n,0.375mmnh4no3;0.6n,0.75mmnh4no3;1n,1.25mmnh4no3)的nj6植物中,稻osmir396家族成员的相对丰度,显示为相对于生长于1n条件(=1)的植物中的丰度。数据显示为平均值±s.e.m.(n=3)。
图9显示稻osgrf4功能缺失突变体(osgrf4)的crispr/cas9生成。a,osgrf4外显子-内含子结构,其显示在osgrf4突变等位基因的外显子1和内含子1中crispr/cas9生成的91-bp缺失的位置。b,osgrf4(wt)和osgrf4突变等位基因编码的蛋白质的序列。osgrf4中的缺失导致其正确编码osgrf4的前11个氨基酸,但此后编码异常序列的蛋白质。c,相对于wt中(=1)的丰度,wt(osgrf4)中与osgrf4突变体中的osgrf4mrna丰度。数据显示为平均值±s.e.m.(n=3)。d,抗osgrf4抗体揭示的osgrf4蛋白在osgrf4突变体中积累。hsp90作为负载对照。
图10显示nj6、nj6-sd1和nj6-sd1-osgrf4ngr2等基因系的表型和产量性能性状。a,成熟植物的高度。b,每株植物的分蘖数。c,每穗的籽粒数目。d,旗叶宽度。e,秆(茎)宽度,表示为最上面节间的直径。数据显示为平均值±s.e.m.(n=16,图a-e)。f,每株植物的籽粒产量。数据显示为由每品系六个地块(每个地块包含220株植物)估计的平均值±s.e.m.。相同的小写字母表示平均值之间无显著差异(p>0.05)。
图11显示osgrf4调节多个nh4+代谢基因的表达。a,nils中osamt1.2mrna的相对根丰度,如所示基因型。数据显示为平均值±s.e.m.(n=3)。b,根谷氨酰胺合酶(gs)活性。数据显示为平均值±s.e.m.(n=3)。c,osfd-gogatmrna的相对枝条丰度。数据显示为平均值±s.e.m.(n=3)。显示的丰度相对于nj6植物中的丰度(=1;图a,c)。d,枝条谷氨酰胺合酶(gs)活性。数据显示为平均值±s.e.m.(n=3)。e-h,具有flag-osgrf4的chip-pcr从osamt1.2,osgs2,osnadh-gogat2和osfd-gogat启动子中富集含gcgg的启动子片段(用*标记)。图中显示了推定的osamt1.2、osgs2,osnadh-gogat2和osfd-gogat启动子和片段(1-6)。i-l,在瞬时反式激活测定中,osgrf4激活(i)posamt1.2,(j)posgs2(k)posnadh-gogat2和(l)posfd-gogat启动子::萤光素酶融合构建体。数据显示为平均值±s.e.m.(n=3)。
图12显示osgrf4调节多个no3-代谢基因的表达。a,编码no3-摄取转运蛋白的osnrt1.1b和osnrt2.3amrna的相对根丰度。数据显示为平均值±s.e.m.(n=3)。b,编码no3-转运蛋白和同化酶的osnpf2.4,osnia1、osnia3和osprs1mrna的相对枝条丰度。数据显示为平均值±s.e.m.(n=3)。显示的丰度相对于nj6-sd1(=1;图a-b)中的丰度。c-e,具有flag-osgrf4的chip-pcr从以下富集含gcgg的片段(用*标记):(c)编码根no3-摄取转运蛋白的osnrt1.1b和osnrt2.3a基因启动子、(d)编码枝条no3-转运蛋白的osnpf2.4基因启动子和(e)编码枝条no3-同化酶的osnia1,osnia3和osprs1基因启动子。数据显示为平均值±s.e.m.(n=3)。f-g,在瞬时反式激活测定中,osgrf4激活(f)posnrt1.1b和posnrt2.3(g)posnpf2.4,posnia1,posnia3和posprs1启动子::萤光素酶融合构建体。数据显示为平均值±s.e.m.(n=3)。
图13显示ga促进gs活性。a,用100μmga(ga3)和/或2μm多效唑(pac)处理的2周龄稻植物的根中的gs活性,基因型如所示。数据显示为平均值±s.e.m.(n=3)。b,用100μmga(ga3)和/或2μm多效唑(pac)处理的植物枝条中的gs活性,基因型如所示。数据显示为平均值±s.e.m.(n=3)。
图14显示slr1-osgif1-osgrf4相互作用的bifc可视化。a,表达osgrf4和特定结构域缺失的变体的构建体的细节。osgrf4含有qlq(gln,leu,gln)和wrc(trp,arg,cys)结构域,位置如所示。b,bifc测定。将表达用yfp的n末端标记的osgrf4或缺失变体(如a中)的构建体分别与表达用yfp的c末端标记的osgif1或slr1的构建体一起共转化到烟草叶表皮细胞中。比例尺,60μm。c,bifc测定。将表达用yfp标签n末端标记的osgrf1或相关的osgrf家族蛋白的构建体与表达用yfp的c末端标记的slr1的构建体一起共转化到烟草叶表皮细胞中。比例尺,60μm。
图15显示slr1抑制osgrf4mrna的osgrf4自促进和osgrf4蛋白丰度。a,chip-pcrosgrf4介导的含gcgg的osgrf4启动子片段的富集。b,posgrf4promoter::荧光素酶融合构建体转录的osgrf4激活促进被osgrf1增强,并被slr1抑制。c,osgfr4mrna丰度,植物基因型如所示。d,osgrf4丰度(如通过抗osgrf4抗体所检测的),植物基因型如所示。e,ga和pac对osgrf4mrna丰度的影响。f,ga如何促进osgrf4丰度的图示。在没有ga的情况下,slr1抑制osgrf4-osgif1对osgrf4转录的促进。在ga的存在下,slr1通过蛋白酶体介导的降解而被破坏,这促进osgrf4-osgif1激活的osgrf4转录。
图16显示osgrf4调节多个c代谢和细胞周期调节基因的表达。a,相对于nj6-sd1植物中的丰度(=1),nj6-sd1-osgrf4ngr2植物中调节光合作用、碳信号传导和蔗糖转运/韧皮部负载的所选基因的转录物的相对枝条丰度。数据显示为平均值±s.e.m.(n=3)。b,相对于nj6-sd1植物中的丰度(=1),在nj6-sd1-osgfr4ngr2植物中调节细胞周期进程的所选基因的转录物的相对枝条丰度。数据显示为平均值±s.e.m.(n=3)。
图17显示osgrf4的天然等位基因变异与植物和籽粒形态和籽粒产量性能的变异相关。a,osgrf4的启动子区的dna多态性。绿色阴影区域表示与nm73和rd23中表型变异相关的三个独特snp变异。b,携带不同osgrf4启动子单倍型(hap.;a,b或c)的稻品种的植物高度、籽粒长度,籽粒宽度,每穗的籽粒数目和籽粒产量性能的箱形图。来自在正常稻田施肥条件21中生长的植物的所有数据。数据显示为平均值±s.e.m.(a,n=74;b,n=28;c,n=123)。相同的小写字母表示平均值之间无显著差异(p>0.05)。
图18显示了在不同n施肥水平下生长的9311和9311-osgrf4ngr2植物显示的农学性状。a,旗叶宽度。b,最上面节间的秆宽度。c,每穗的籽粒数目。d,1000籽粒重量。e,收获指数。f,每株植物的干生物量。数据显示为平均值±s.e.m.(n=30)。使用斯氏t检验来产生p值。
图19显示了在不同n施肥水平下生长的9311和9311-osgrf4ngr2植物的根部n摄取率。a,15nh4+摄取。b,15no3-摄取。数据显示为平均值±s.e.m.(n=30)。使用斯氏t检验来产生p值。
图20显示了在不同n施肥水平下生长的9311和9311-osgrf4ngr2植物地上部分不同器官中的矿质营养物分布。a,n分布。b,p(磷)分布。c,k(钾)分布。d,ca(钙)分布。e,s(硫)分布。f,mg(镁)分布。数据显示为平均值±s.e.m.(n=9)。
图21显示了在不同水平的氮施肥水平下,表达p35::osgrf4ngr2-gfp构建体的wyj7-dep1和转基因wyj7-dep1植物的生长、n摄取和籽粒产量性能。a,成熟植物的高度。比例尺,15cm。b-d,(b)15nh4+(c)15no3-以及(d)15nh4+和15no3-组合的根部摄取率。分别在低氮(0.3n,0.375mmnh4no3)和高氮(1n,1.25mmnh4no3)条件下生长的稻植物。数据显示为平均值±s.e.m.(n=9)。相同的小写字母表示平均值之间无显著差异(p>0.05)。e,成熟植物高度。f,抽穗日期。g,每株植物的分蘖数。h,每穗的籽粒数。i,每株植物的籽粒产量。数据显示为平均值±s.e.m.(n=30)。使用斯氏t检验来产生p值(图e-i)。
具体实施方式
现在将进一步描述本发明。在以下段落中,更详细地限定本发明的不同方面。除非明确地相反指示,否则如此限定的每个方面可与任何其它方面组合。特别地,任何被指示为优选或有利的特征可以与任何其它被指示为优选或有利的特征组合。
除非另有说明,本发明的实践将使用本领域技术范围内的植物学、微生物学、组织培养、分子生物学、化学、生物化学和重组dna技术、生物信息学的常规技术。这些技术在文献中有充分的说明。
如本文所用,词语“核酸”、“核酸序列”、“核苷酸”、“核酸分子”或“多核苷酸”旨在包括dna分子(例如cdna或基因组dna),rna分子(例如mrna),天然存在的、突变的、合成的dna或rna分子,以及使用核苷酸类似物产生的dna或rna的类似物。它可以是单链或双链的。这样的核酸或多核苷酸包括但不限于结构基因的编码序列、反义序列和不编码mrna或蛋白质产物的非编码调节序列。这些术语也涵盖基因。术语“基因”或“基因序列”广泛用于指与生物功能相关的dna核酸。因此,基因可以包括如基因组序列中的内含子和外显子,或者可以仅包括如cdna中的编码序列,和/或可以包括与调节序列组合的cdna。
术语“多肽”和“蛋白质”在本文中可互换使用,是指通过肽键连接在一起的任何长度的聚合形式的氨基酸,。
术语“启动子”通常指位于基因转录起点上游的核酸控制序列,其参与rna聚合酶和其它蛋白质的结合,从而指导可操作连接的核酸的转录。上述术语涵盖源自经典真核生物基因组基因的转录调节序列(包括tata盒,其是精确转录起始所需的,具有或不具有ccaat盒序列)和额外的调节元件(即上游激活序列、增强子和沉默子),所述额外的调节元件响应于发育性和/或外部刺激或以组织特异性方式改变基因表达。该术语还包括经典原核生物基因的转录调节序列,在这种情况下,它可以包括-35盒序列和/或-10盒转录调节序列。
本发明的方面涉及重组dna技术,并且排除了仅基于通过传统育种方法产生植物的实施方案。
术语“grf”指生长调节因子,一种植物特异性转录因子。优选grf是grf4。在一个实例中,grf是稻grf4(也称为osgrf4)或其直向同源物。
本文所用的术语“氮”可包括硝酸盐(no3-)和/或铵盐(nh4+)。
对于本发明的目的,“遗传改变的植物”或“突变植物”是与天然存在的野生型(wt)植物相比已经遗传改变了的植物。在一个实施方案中,突变植物是与天然存在的野生型(wt)植物相比已经使用诱变方法(诸如如本文所描述的任何诱变方法)改变了的植物。在一个实施方案中,诱变方法是靶向基因组修饰或基因组编辑。在一个实施方案中,植物基因组与野生型序列相比已经使用诱变方法改变。这种植物具有如本文所述的改变的表型,例如增加的氮代谢。因此,在这个实例中,增加的氮代谢是由改变的植物基因组的存在(例如,突变的内源grf基因或启动子)赋予的。在一个实施方案中,使用靶向基因组修饰特异性靶向内源启动子或基因序列,并且突变基因或启动子序列的存在不是由植物中表达的转基因的存在所赋予的。换言之,遗传改变的植物可描述为无转基因的。
然而,在备选的实施方案中,遗传改变的植物是转基因植物。对于本发明的目的,“转基因的”、“转基因”或“重组的”是指,关于例如核酸序列、表达盒、基因构建体或包含核酸序列的载体或用根据本发明的核酸序列、表达盒或载体转化的生物体,通过重组方法产生的所有那些构建体,其中
(a)编码在本发明方法中有用的蛋白质的核酸序列,或
(b)与根据本发明的核酸序列可操作连接的遗传控制序列,例如启动子,或
(c)a)和b)不位于其天然遗传环境中,或已经通过重组方法修饰,所述修饰可以采取例如一个或多个核苷酸残基的置换、添加、缺失、倒位或插入的形式。
天然遗传环境应理解为是指原始植物中的天然基因组或染色体基因座或基因组文库中的存在。在基因组文库的情况下,优选至少部分地保留核酸序列的天然遗传环境。该环境至少在一侧侧接核酸序列,并且具有至少50bp,优选至少500bp,特别优选至少1000bp,最优选至少5000bp的序列长度。天然存在的表达盒,例如核酸序列的天然启动子与编码用于本发明方法的多肽的相应核酸序列的天然存在的组合,如上文所定义,当该表达盒通过非天然的合成(“人工”)方法(例如诱变处理)修饰时,则成为转基因表达盒。合适的方法描述于例如us5,565,350或wo00/15815中,二者均通过引用并入本文。
根据本文所述的本发明所有方面的植物可以是单子叶植物或双子叶植物。优选地,植物是农作物植物。农作物植物是指以商业规模生长用于人或动物消耗或使用的任何植物。在优选的实施方案中,植物是谷物。在另一个实施方案中,植物是拟南芥。
在最优选的实施方案中,植物选自稻、玉米、小麦、大麦、高粱、芸苔属(brassica)、大豆、马铃薯和番茄。在一个实施方案中,植物是grv(半矮化绿色革命品种)。在最优选的实施方案中,植物是稻,优选来自于粳稻或籼稻品种。在这个实例中,籼稻品种优选携带导致生长阻抑性della蛋白(della)稳定化的突变sd1等位基因,更优选地,籼稻品种选自tq,nj11,zf802,mh63,cy1,hhz,gc2,hjx74,zs97b,mh86,gla4,wxq,gf3,skz,sh527,xaz9,faz,tzzl1,78130,93-11,sh881,ltz,ltp,qxjz,hy33,8b,ejq,qgh,xaz4,h410,ejl1,yfz,ejf和sg1。在另一个实例中,粳稻品种携带变体(dep1)gγ亚基,例如wjy7-dep1。在另一个实施方案中,植物是小麦,优选地,植物携带导致della稳定的突变rht等位基因,例如中国小麦grvkn199。
本文所用的术语“植物”涵盖全植物、植物的祖先和后代以及植物部分,包括种子、果实、枝条、茎、叶、根(包括块茎)、花、组织和器官,其中上述每一种均包含本文所述的核酸构建体或携带本文所述的突变。术语“植物”还涵盖植物细胞、悬浮培养物、愈伤组织、胚、分生组织区、配子体、孢子体、花粉和小孢子,再次,其中上述每一种均包含本文所述的核酸构建体或突变。
本发明还延伸至本文所述的本发明植物的可收获部分,但不限于种子、叶、果实、花、茎、根、根茎、块茎和鳞茎。本发明的方面还延伸至来源于(优选直接来源于)这种植物的可收获部分的产品,例如干燥的颗粒或粉末、油、脂肪和脂肪酸、淀粉或蛋白质。可来源于本发明植物的可收获部分的另一种产品是生物柴油。本发明还涉及包含本发明植物或其部分的食品和食品增补剂。在一个实施方案中,食品可以是动物饲料。在本发明的另一个方面,提供了来源于本文所述植物或其部分的产品。
在最优选的实施方案中,植物部分或可收获产品是种子或籽粒。因此,在本发明的进一步的方面,提供了由如本文所述的遗传改变的植物产生的种子。
在备选的实施方案中,植物部分是本文所述的遗传改变的植物的花粉、繁殖体或后代。因此,在本发明的进一步的方面中,提供了由本文所述的遗传改变的植物产生的花粉、繁殖体或后代。
根据本发明的所有方面,本文所用的对照植物是未根据本发明的方法进行修饰的植物。因此,在一个实施方案中,对照植物不具有如上所述的grf核酸的表达增加和/或grf多肽的活性改变。在备选的实施方案中,如上所述,植物未经过遗传修饰。在一个实施方案中,对照植物是野生型植物。对照植物通常是相同的植物物种,优选与修饰植物具有相同的遗传背景。
增加氮摄取的方法
在本发明的第一方面,提供了增加植物中氮摄取和/或氮同化和/或氮利用效率的方法,所述方法包括增加生长调节因子(grf)的表达或水平或增加生长调节因子的活性。
如本文所用,“增加”可指与对照植物相比增加至少1%、2%、3%、4%、5%、6%、7%、8%、9%、10%、20%、30%、40%、50%、60%、70%、80%、90或95%或更多。所述增加可以在植物的根和/或枝条中。
术语“氮利用效率”或nue可定义为作物的产量(例如籽粒的产量)。备选地,nue可定义为农业nue,即籽粒产量/n。植物的总n利用效率包括摄取和利用效率两者,并且可以计算为upe。在一个实施方案中,nue与对照植物相比增加5%-50%或更多。
术语“氮同化”可定义为由无机氮形成有机氮化合物。
氮摄取、氮同化和氮利用效率中至少一种的增加在本文中可以称为氮代谢的增加。
在进一步的实施方案中,所述方法进一步包括增加植物中的产量,优选籽粒产量。即,该方法包括增加植物中的氮摄取、氮同化和nue中的至少一种,和增加产量。
术语”产量”通常指可测量的经济价值的产生,通常与特定的农作物、区域和时间段有关。单个植物部分基于其数量、大小和/或重量直接贡献于产量。实际产量是农作物每年每平方米的产量,其通过将每年总产量(包括收获的和评估的产量)除以种植的平方米来确定。
本文定义的术语“增加的产量”可以认为包括以下任何一种或至少一种,并且可以通过评估以下一种或多种来测量:(a)植物的一个或多个部分、地上(可收获部分)的增加的生物量(重量),或增加的根生物量、增加的根体积、增加的根长度、增加的根直径或增加的根长度,或任何其它可收获部分的增加的生物量。增加的生物量可以表示为g/植物或kg/公顷,(b)每株植物增加的种子产量,其可以包括每株植物或在个体基础上增加种子生物量(重量)的一种或多种,(c)增加的种子饱满率,(d)增加的饱满种子数目,(e)增加的收获指数,其可以表示为可收获部分如种子的产量与总生物量的比率,(f)增加的生存力/萌发效率,(g)增加的种子或豆荚或豆或籽粒的数目或大小或重量,(h)增加的种子体积(其可以是组分(即脂质(本文中也称为油))、蛋白质和碳水化合物总含量和组分的变化,(i)增加的(个体或平均)种子面积,(j)增加的(个体或平均)种子长度,(k)增加的(个体或平均)种子周长,(l)增加的生长或增加的分枝,例如在更多分枝上的花序,(m)增加的鲜重或籽粒饱满(n)增加的穗重量(o)增加的千粒重(tkw),其可取自计算的饱满种子数和它们的总重量,并且可以是种子大小和/或种子重量增加的结果,(p)每个植物减少的无分蘖的数目和(q)更结实或更强壮的秆或茎。所有参数都是相对于野生型或对照植物。
在优选的实施方案中,所述增加的产量包括每穗或每个植物的籽粒数目中的至少一个的增加和/或1000籽粒重量的增加。相对于对照或野生型植物,产量是增加的。例如,与对照或野生型植物相比,产量增加2%、3%、4%、5%、6%、7%、8%、9%、10%、11%、12%、13%、14%、15%、16%、17%、18%、19%或20%、25%、30%、35%、40%、45%或50%。因此,籽粒产量的增加可以通过评估每穗或每个植物的籽粒数目和千粒重中的一个或多个来测量。本领域技术人员能够使用本领域已知的技术测量任何上述产量参数。
本文所用的术语“种子”和“籽粒″可以互换使用。如本文所用的术语“增加”、“改善”或“增强”也是可互换的。
在进一步的实施方案中,所述方法进一步包括增加植物中的c同化。增加是如上文所定义的。
在优选的实施方案中,grf是grf4(生长调节因子4)或其同源物或直向同源物。在一个实施方案中,grf4是稻grf4或osgrf4。
如本文所用,术语“增加表达”指核苷酸水平的增加,并且如本文所用的“增加水平”指grf的蛋白质水平的增加。
如本文还使用的,grf的“增加活性”指增加grf的生物活性,例如,增加grf的转录活性(即grf结合其靶基因并增加其靶基因转录的能力)。在一个实施方案中,grf是grf4,并且grf4在与gif1的转录复合物中起作用,它结合并促进靶基因(如参与n,c代谢和细胞增殖的基因)的转录。该复合物被slr1抑制。slr1还通过抑制grf4转录而降低grf4的积累。因此,在一个实施方案中,增加grf特别是grf4的活性可包括增加gif1或其直向同源物的表达或活性,和/或减少或消除slr1或其直向同源物的表达或活性。因此,在备选的方面,该方法可包括将至少一个突变引入slr1和/或gif1基因或其同源物中,或引入gif基因或其同源物的另一拷贝,和/或分别降低或增加slr1和gif1的活性。备选地,该方法可包括在包含编码gif1的核酸序列的核酸中引入和表达。我们还在此显示ga(赤霉酸)促进蛋白酶体介导的slr1破坏。因此,在一个实施方案中,slr1的活性可使用ga介导。
在一个实施方案中,grf的表达或水平或活性与野生型或对照植物中的水平相比增加多至或大于5%、10%、20%、30%、40%、50%、60%、70%、80%或90%。
用于确定grf表达和/或活性的水平的方法是本领域技术人员所熟知的。特别地,增加可以通过本领域技术人员已知的任何标准技术来测量。例如,grf表达和/或含量水平的增加可能损害蛋白质和/或核酸水平的量度,并且可以通过本领域技术人员已知的任何技术来测量,例如但不限于任何形式的凝胶电泳或色谱(例如hplc)。由于grf编码转录因子,在一个实施方案中,该方法可以包括使用本领域标准技术,例如但不限于rna-seq和chip-seq,来测量grf的转录谱(相对于野生型或对照)。
在优选的实施方案中,所述方法不影响植物高度。即,所述方法增加氮摄取、氮同化和nue以及任选的产量和/或c同化中的至少一种,但对植物高度没有影响。结果,所述方法不影响作为grv特征的半矮态的益处。
在一个实施方案中,所述方法包括将至少一个突变引入编码grf和/或grf的启动子的至少一个核酸中。在一个实施方案中,所述方法包括将至少一个突变引入至少一个编码grf,优选grf4或grf4启动子的内源基因。备选地,所述方法可包括插入至少一个或多个额外拷贝的编码grf多肽或其同源物或变体的核酸,使得所述序列与调节序列可操作地连接。
在一个实施方案中,核酸编码如seqidno:3中所定义的grf4或其功能变体或同源物。在进一步的实施方案中,核酸包含如seqidno:1或2中所定义的核酸序列或其功能变体或同源物,或由其组成。
“grf启动子”或“grf4启动子”是指在grf,优选grf4orf(可读框)的atg密码子上游延伸至少5kbp,优选至少2.5kbp,更优选至少1kbp的区域。在一个实施方案中,grf4启动子的序列包含或组成为如seqidno:7(单倍型a)或8(单倍型c)所定义的核酸序列、其功能变体或同源物。
在上述实施方案中,“内源”核酸可以指植物基因组中的自然或天然序列。在一个实施方案中,gse4基因的内源序列包含seqidno:1或2或由其组成,并编码如seqidno:3中所定义的氨基酸序列或其同源物。本发明范围还包括上述经鉴定的序列的功能变体(如本文所定义)和同源物。grf4同源物的实例示于seqidno:4至39和192至201中。因此,在一个实施方案中,同源物编码选自seqidno:12,15,18,21,24,27,30,33,36和39的多肽,或同源物包含选自seqidno:11,14,17,20,23,26,29,32,35和38的核苷酸序列或由其组成。在另一个实施方案中,grf启动子同源物包含选自seqidno:192至201的核酸序列或由其组成。
参考seqidno:1至201中任一项,本文所使用的术语“变体”或“功能变体”是指保留完整非变体序列的生物学功能的变体基因序列或部分该基因序列。功能变体还包含目的基因的变体,其具有不影响功能例如在非保守残基中的序列改变。还涵盖与本文所示的野生型序列相比基本同一的变体,即仅具有一些(例如在非保守残基中的)序列变异,并且具有生物学活性。导致在给定位点产生不影响所编码多肽的功能特性的不同氨基酸的核酸序列中的改变是本领域熟知的。例如,氨基酸丙氨酸(疏水氨基酸)的密码子可被编码另一较低疏水性残基(如甘氨酸)或较高疏水性残基(如缬氨酸、亮氨酸或异亮氨酸)的密码子置换。类似地,导致用一个带负电荷的残基置换另一个的变化,例如用天冬氨酸置换谷氨酸,或用一个带正电荷的残基置换另一个的变化,例如用赖氨酸置换精氨酸,也可以预期产生功能等同的产物。导致多肽分子的n-末端和c-末端部分改变的核苷酸变化预期也不会改变该多肽的活性。所提出的每一种修饰都在本领域的常规技术范围内,如编码产物的生物活性的保留所确定的。
如本文所述的本发明的任何方面中所使用的,“变体”或“功能变体”与非变体核酸或氨基酸序列具有至少25%、26%、27%、28%、29%、30%、31%、32%、33%、34%、35%、36%、37%、38%、39%、40%、41%、42%、43%、44%、45%、46%、47%、48%、49%、50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或至少99%的总序列同一性。
本文所用的术语同源物也指来自其它植物物种的grf启动子或grf基因直向同源物。同源物与由seqidno:3表示的氨基酸或与由seqidno:1或2显示的核酸序列可具有(按优选顺序递增)至少25%、26%、27%、28%、29%、30%、31%、32%、33%、34%、35%、36%、37%、38%、39%、40%、41%、42%、43%、44%、45%、46%、47%、48%、49%、50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或至少99%的总序列同一性。在一个实施方案中,总序列同一性为至少37%。在一个实施方案中,总序列同一性为至少70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、或99%,最优选为90%、91%、92%、93%、94%、95%、96%、97%、98%或至少99%。
如上所定义的grf同源物的功能变体也在本发明的范围内。
grf多肽编码特征为至少一个保守qlq结构域(谷氨酰胺、亮氨酸、谷氨酰胺)和wrc结构域(色氨酸、精氨酸和半胱氨酸)的转录因子。在一个实施方案中,同源物或变体也可具有wrc和qlq结构域中的至少一个。因此,在一个实施方案中,同源物或变体编码与seqidno:3所示的氨基酸具有至少25%,26%,27%,28%,29%,30%,31%,32%,33%,34%,35%,36%,37%,38%,39%,40%,41%,42%,43%,44%,45%,46%,47%,48%,49%,50%,51%,52%,53%,54%,55%,56%,57%,58%,59%,60%,61%,62%,63%,64%,65%,66%,67%,68%,69%,70%,71%,72%,73%,74%,75%,76%,77%,78%,79%,80%,81%,82%,83%,84%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%总序列同一性的grf4多肽,并具有wrc和qlq结构域中的至少一个。
当如下所述针对最大对应性进行比对时,如果两个序列中的核苷酸或氨基酸残基的序列分别相同,则称两个核酸序列或多肽是“同一的”。在两个或多个核酸或多肽序列的上下文中,术语“同一的”或“同一性”百分比是指当在比较窗上针对最大对应性进行比较和比对时,相同的或具有指定百分比的相同氨基酸残基或核苷酸的两个或多个序列或子序列,如使用以下序列比较算法之一或通过人工比对和目测所测量的。当序列同一性的百分比用于提及蛋白质或肽时,认识到非同一的残基位置通常因保守氨基酸置换而不同,其中氨基酸残基被具有相似化学性质(例如电荷或疏水性)的其它氨基酸残基置换,并因此不改变分子的功能性质。当序列的不同之处在于保守置换时,可以向上调整序列同一性百分比以校正置换的保守性。用于进行这种调整的手段是本领域技术人员公知的。为了进行序列比较,通常将一个序列作为参考序列,将测试序列与之进行比较。当使用序列比较算法时,将测试和参考序列输入计算机,如果需要,指定子序列坐标,并指定序列算法程序参数。可使用默认程序参数,或者可指定备选参数。然后,基于程序参数,序列比较算法计算测试序列相对于参考序列的序列同一性百分比。适于确定序列同一性百分比和序列相似性百分比的算法的非限制性实例是blast和blast2.0算法。
可通过序列比较和保守结构域的鉴定来鉴定合适的同源物。本领域存在可用于鉴定这种序列的预测因子。可以如本文所述鉴定同源物的功能,因此技术人员能够证实该功能,例如当在植物中过表达时。
因此,本发明的和本文所述的核苷酸序列也可用于从其它生物体,特别是其它植物例如农作物植物中分离相应的序列。以这种方式,可以使用诸如pcr、杂交等方法,基于这些序列与本文所述序列的序列同源性来鉴定这些序列。当鉴定和分离同源物时,也可以考虑序列的拓扑学和特征结构域结构。序列可以基于它们与整个序列或其片段的序列同一性来分离。在杂交技术中,将已知核苷酸序列的全部或部分用作探针,所述探针与来自所选植物的克隆基因组dna片段或cdna片段(即基因组或cdna文库)群体中存在的其它相应核苷酸序列选择性地杂交。杂交探针可以是基因组dna片段、cdna片段、rna片段或其它寡核苷酸,并且可以用可检测基团或任何其它可检测标记物来标记。用于制备杂交探针和构建cdna和基因组文库的方法是本领域公知的,并公开于sambrook等,(1989)molecularcloning:alibrarymanual(第2版,coldspringharborlaboratorypress,plainview,纽约)中。
这些序列的杂交可在严格条件下进行。“严格条件”或“严格杂交条件”是指这样的条件:在这个条件下,相比于与其他序列杂交,探针以可检测地更高程度(例如,至少比背景高2倍)与其靶序列杂交的条件。严格条件是序列依赖性的,并且在不同环境中将是不同的。通过控制杂交和/或洗涤条件的严格性,可以鉴定与探针100%互补的靶序列(同源探测)。备选地,可调整严格条件以允许序列中的一些错配,从而检测到较低程度的相似性(异源探测)。通常,探针长度小于约1000个核苷酸,优选长度小于500个核苷酸。
通常,严格条件是如下那些条件:其中盐浓度低于约1.5mna离子,典型地约0.01-1.0mna离子浓度(或其它盐),在ph7.0-8.3,并且温度对于短探针(例如10-50个核苷酸)至少为约30℃,对于长探针(例如大于50个核苷酸)至少为约60℃。杂交的持续时间通常小于约24小时,通常为约4至12小时。严格条件也可用去稳定剂如甲酰胺的加入来实现。
在进一步的实施方案中,如本文所用的变体可包含编码如本文所限定的grf多肽的核酸序列,其能够在如本文所限定的严格条件下与如seqidno:1或2所定义的核酸序列杂交。
在一个实施方案中,提供了如本文所述的增加植物中氮摄取和/或氮同化和/或氮利用效率的方法,该方法包括增加如本文所述的grf的表达和/或活性,其中该方法包括引入至少一个突变至grf基因和/或启动子中,其中该grf基因包含以下或由以下组成
a.编码如seqidno:3,12,15,18,21,24,27,30,33,36和39之一所定义的多肽的核酸序列;或
b.如seqidno:1,2,11,14,17,20,23,26,29,32,35,38和192至201之一所定义的核酸序列;或
c.与(a)或(b)具有至少75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或至少99%的总序列同一性的核酸序列;或
d.编码如本文所限定的grf多肽的核酸序列,其能够在如本文所限定的严格条件下与(a)至(c)中任一项的核酸序列杂交。
并且其中grf启动子包含以下或由以下组成
e.如seqidno:7,8,9和192至201之一所定义的核酸序列;
f.与(e)具有至少75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或至少99%的总序列同一性的核酸序列;或
g.能够在本文定义的严格条件下与(e)至(f)中任一项的核酸序列杂交的核酸序列。
在一个实施方案中,被引入内源grf基因或其启动子以提高grf基因或蛋白质的生物学活性和/或表达水平的突变可选自下列突变类型:
1.“错义突变”,其是核酸序列中导致一个氨基酸置换另一个氨基酸的变化;
2.“无义突变”或“stop密码子突变”,其是核酸序列中的变化,该变化导致过早stop密码子的引入,并因此终止翻译(导致截短的蛋白质);植物基因含有翻译终止密码子“tga”(rna中uga)、“taa”(rna中uaa)和“tag”(rna中uag);因此,导致在这些将要在成熟的mrna中(阅读框中)被翻译的密码子之一中的任何核苷酸置换、插入、缺失将终止翻译。
3.一个或更多个氨基酸的“插入突变”,其是由于在核酸的编码序列中已经添加了一个或更多个密码子;
4.一个或更多个氨基酸的“缺失突变”,其是由于在核酸的编码序列中已经缺失了一个或更多个密码子;
5.“移码突变”,其导致核酸序列在突变下游的不同读码框中翻译。移码突变可具有多种原因,诸如一个或更多个核苷酸的插入、缺失或重复。
6.“剪接位点”突变,其是导致在剪接位点处核苷酸插入、缺失或置换的突变。
7.“倒位”突变,其是核酸序列的一百八十度旋转。
然而,最优选地,突变是置换。即,用一个碱基置换另一个不同的碱基。
这种改变的grf多肽可导致如本文定义的功能等位基因的显性或半显性获得。
内源基因中的突变可包括在以下任一位点中的至少一个突变:grf基因的编码区,优选外显子3;微小rna(mirna)结合位点,优选在mir396结合位点;内含子序列,优选内含子2和/或内含子3;和/或在5’utr、3’utr、终止信号、剪接受体位点或核糖体结合位点中的剪接位点。
在一个实例中,mir396结合或识别位点包含如本文所定义的以下序列或其变体,或由如本文所定义的以下序列或其变体组成:
ccgttcaagaaagcctgtggaa:seqidno:45
优选地,突变是防止序列由微小rna切割并因此防止其随后降解的任何突变。这导致grfmrna和蛋白质水平的增加。在一个实施方案中,突变是置换。
在具体的实施方案中,突变是以下之一或两者:
-在seqidno:45的位置4或其同源位置处的t至a;
-在seqidno:45的位置5或其同源位置处的c至a。
在额外的或备选的实施方案中,突变是内含子2和/或内含子3中的至少以下之一:
-在seqidno:1的位置724或725或其同源位置处的a至g;
-在seqidno:1的位置1672或其同源位置处的t至c。
作为在内源基因中至少一个上述突变的备选或补充,该突变在grf启动子中。优选地,所述突变是增加grf表达的任何突变。在一个实例中,突变是以下突变中的至少一个或其任意组合。前面的位置是在单倍型a启动子(例如,包含seqidno:7或其变体或由其组成的启动子)中的位置。后面的位置是在单倍型c启动子(例如,包含seqidno:8或其变体或由其组成的启动子)中的位置。
-在grf起始密码子位置-941或-935处或在seqidno:7的位置60或seqidno:8的位置66处或其同源位置处的c至t置换;
-在grf起始密码子位置-884或位置-878处或在seqidno:7的位置118或seqidno:8的位置124处或其同源位置处的t至a置换;
-在grf起始密码子位置-855或-849处或在seqidno:7的位置148或seqidno:8的位置154处或其同源位置处的c至t置换;
-在grf起始密码子位置-847或-841处或在seqidno:7的位置157或seqidno:8的位置163处或其同源位置处的c至t置换;
-在grf起始密码子位置-801或-795处或在seqidno:7的位置204或seqidno:8的位置210处或其同源位置处的c至t置换;
-在grf起始密码子位置-522或-516处或在seqidno:7的位置484或seqidno:8的位置489处或其同源位置处的c至t置换;
-在grf起始密码子位置-157处或在seqidno:7的位置850或seqidno:8的位置516处或其同源位置处的g至c置换;
在一个实施方案中,所述突变是:
-在grf起始密码子位置-884或位置-878处或在seqidno:7的位置118或seqidno:8的位置124处或其同源位置处的t至a置换;和
-在grf起始密码子位置-847或-841处或在seqidno:7的位置157或seqidno:8的位置163处或其同源位置处的c至t置换;
-在grf起始密码子位置-801或-795处或在seqidno:7的位置204或seqidno:8的位置210处或其同源位置处的c至t置换。
包含所有三种上述多态性的grf启动子可以称为单倍型b。
在一个实施方案中,grf启动子包含至少一个以下序列,并且该方法包括将至少一个突变(优选至少一个置换)引入这些序列的至少一个中:
·caaact
·ttctaa
·ctaatt
·atacaa
·ttacag
·acatac
·acttac
·taattt
在一个实例中,grf启动子包含seqidno:192或由其组成,并且所述突变是存在于seqidno:192中的至少一个以下序列中的至少一个突变,优选至少一个置换:
·caaact
·ttctaa
·ctaatt
在另一个实例中,grf启动子包含seqidno:193或由其组成,并且所述突变是存在于seqidno:193中的至少一个以下序列中的至少一个突变,优选至少一个置换:
·atacaa
·ttctaa
在另一个实例中,grf启动子包含seqidno:194或由其组成,并且所述突变是存在于seqidno:194中的至少一个以下序列中的至少一个突变,优选至少一个置换:
·ctaatt
·atacaa
·ttctaa
在另一个实例中,grf启动子包含seqidno:195或由其组成,并且所述突变是存在于seqidno:195中的以下序列中的至少一个突变,优选至少一个置换:
·ttctaa
在另一个实例中,grf启动子包含seqidno:196或由其组成,并且所述突变是存在于seqidno:196中的以下序列中的至少一个突变,优选至少一个置换:
·atacaa
在另一个实例中,grf启动子包含seqidno:197或由其组成,并且所述突变是存在于seqidno:197中的以下序列中的至少一个突变,优选至少一个置换:
·ttcataa
在另一个实例中,grf启动子包含seqidno:198或由其组成,并且所述突变是存在于seqidno:198中的至少一个以下序列中的至少一个突变,优选至少一个置换:
·ctaatt
·atacaa
·ttacag
·ttctaa
·caaact
·acatac
在另一个实例中,grf启动子包含seqidno:199或由其组成,并且所述突变是存在于seqidno:199中的至少一个以下序列中的至少一个突变,优选至少一个置换:
·acatac
·ttctaa
·acttac
·atacaa
·caaact
在另一个实例中,grf启动子包含seqidno:200或由其组成,并且所述突变是存在于seqidno:200中的至少一个以下序列中的至少一个突变,优选至少一个置换:
·ttctaa
·ctaatt
·acttac
·ttacag
·taattt
在另一个实例中,grf启动子包含seqidno:201或由其组成,并且所述突变是存在于seqidno:201中的至少一个以下序列中的至少一个突变,优选至少一个置换:
·acatac
·caaact
·atacaa
·ttctaa
·ctaatt
“至少一个突变”是指当grf基因作为多于一个拷贝或同源体(具有相同或略微不同的序列)存在时,在至少一个基因中存在至少一个突变。优选地,所有基因都是突变的。
在一个实施方案中,突变是使用靶向基因组编辑引入的。即,在一个实施方案中,本发明涉及方法以及已经通过上述基因工程方法生成的植物,并且不涵盖天然存在的品种或通过传统育种方法生成植物。
靶向基因组修饰或靶向基因组编辑是一种基因组工程技术,其使用靶向dna双链断裂(dsbs)通过同源重组(hr)介导的重组事件来刺激基因组编辑。为了通过引入位点特异性dnadsbs实现有效的基因组编辑,可使用四种主要类别的可定制的dna结合蛋白:来源于微生物移动遗传元件的大范围核酸酶、基于真核转录因子的zf核酸酶、来自黄单胞菌属细菌的转录激活因子样效应子(tales),和来自ii型细菌适应性免疫系统crispr(成簇的规则间隔的短回文重复序列)的rna-引导的dna内切核酸酶cas9。大范围核酸酶、zf和tale蛋白都通过蛋白质-dna相互作用识别特异性dna序列。尽管大范围核酸酶整合了核酸酶和dna结合结构域,zf和tale蛋白分别由靶向dna的3个或1个核苷酸(nt)的单个模块组成。zf和tale可以以所需的组合装配,并附着于foki的核酸酶结构域,以引导针对特定的基因组基因座的溶核活性。
在通过细菌iii型分泌系统递送到宿主细胞中后,tal效应子进入细胞核,与宿主基因启动子中的效应子特异性序列结合并激活转录。它们的靶向特异性由串联的33-35个氨基酸重复序列的中心结构域决定。然后是20个氨基酸的单个截短重复序列。所检查的大多数天然存在的tal效应子具有12至27个完整重复序列。
这些重复序列仅通过两个相邻的氨基酸—它们的重复可变双残基(rvd)——而彼此不同。确定tal效应子将识别哪个单核苷酸的rvd:一个rvd对应于一个核苷酸,四个最常见的rvd各自优先与四个碱基之一相关联。天然存在的识别位点之前一致地有tal效应子活性所需的t。tal效应子可融合至fokⅰ核酸酶的催化结构域,以产生tal效应子核酸酶(talen),该核酸酶使得体内靶向dna双链断裂(dsbs)以进行基因组编辑。在本领域中充分描述了此技术在基因组编辑中的应用,例如在us8,440,431、us8,440,432和us8,450,471中。cermakt等描述了一组定制的质粒,其可以与goldengate克隆方法一起使用,以装配多个dna片段。如其中所描述的,goldengate方法使用iis型限制性内切核酸酶,该限制性内切核酸酶在它们的识别位点外切割以产生独特的4bp突出端。通过在相同的反应混合物中消化和连接使克隆加速,原因在于正确的装配消除了酶识别位点。定制的talen或tal效应子构建体的装配包括两个步骤:(i)将重复模块组装成1-10个重复序列的中间阵列,和(ii)将中间阵列连接到骨架中以制备最终构建体。因此,使用本领域已知的技术,设计靶向如本文所描述的grf基因或启动子序列的tal效应子是可能的。
根据本发明的各个方面可使用的另一种基因组编辑方法是crispr。在本领域中(例如在us8,697,359和本文引用的参考文献中)充分描述了该技术在基因组编辑中的用途。简而言之,crispr是涉及针对入侵的噬菌体和质粒的防御的微生物核酸酶系统。微生物宿主中的crispr基因座含有crispr相关(cas)基因以及能够编程crispr介导的核酸切割特异性的非编码rna元件(sgrna)的组合。已经在广泛的细菌宿主中鉴定了三种类型(i-iii)的crispr系统。每个crispr基因座的一个关键特征是存在由非重复序列的短伸展(间隔区)进行间隔的重复序列(正向重复序列)阵列。非编码crispr阵列在正向重复中被转录和切割成含有单个间隔区序列的短crrna,所述短crrna将cas核酸酶引导到靶位点(前间隔区)。ii型crispr是最良好表征的系统之一,并且在四个顺序的步骤中实施靶向dna双链断裂。首先,从crispr基因座转录两个非编码rna,前-crrna阵列和tracrrna。第二,tracrrna与前-crrna的重复区域杂交,并介导将前-crrna加工成含有单独间隔区序列的成熟crrna。第三,成熟crrna:tracrrna复合物通过crrna上的间隔区和与前间隔区邻近基序(pam)相邻的靶dna上的前间隔区之间的watson-crick碱基配对指导cas9至靶dna,这是靶向识别的额外要求。最后,cas9介导靶dna的切割,以在前间隔区内产生双链断裂。
与常规基因靶向和其它可编程的内切核酸酶相比,crispr-cas9系统的一个主要优点是易于多路化,其中多个基因能够简单地通过使用各自靶向不同基因的多个sgrna来同时突变。另外,当使用两个sgrna位于基因组区侧翼时,可以使间插区段缺失或倒位(wiles等,2015)。
cas9因此是ii型crispr-cas系统的标志蛋白,并且是一种大的单体dna核酸酶,其通过两个非编码rna(crisprrna(crrna)和反式激活crrna(tracrrna))的复合物被引导至与pam(前间隔区邻近基序)序列基序相邻的dna靶序列。cas9蛋白含有两个与ruvc和hnh核酸酶同源的核酸酶结构域。hnh核酸酶结构域切割互补dna链,而ruvc样结构域切割非互补链,并且作为结果,在靶dna中引入钝切割。cas9与sgrna的异源表达能够将位点特异性双链断裂(dsbs)引入来自各种生物体的活细胞的基因组dna中。对于在真核生物中的应用,已经使用了密码子优化形式的cas9,其最初来自于细菌酿脓链球菌(streptococcuspyogenes)。
单向导rna(sgrna)是crispr/cas系统中的第二组分,所述sgrna与cas9核酸酶形成复合物。sgrna是通过将crrna与tracrrna融合而产生的合成rna嵌合体。位于其5’末端的sgrna向导序列赋予dna靶标特异性。因此,通过修饰向导序列,可能产生具有不同靶标特异性的sgrna。向导序列的典型长度为20bp。在植物中,已经使用植物rna聚合酶iii启动子诸如u6和u3使sgrna表达。因此,使用本领域已知的技术,设计靶向如本文所描述的grf基因或启动子序列的sgrna分子是可能的。
在一个实施方案中,该方法使用下面详细定义的sgrna(和模板或供体dna)构建体将靶向snp或突变(特别是本文所述的置换之一)引入grf基因和/或启动子中。如下所述,在双链dna中由sgrna介导的剪切之后,模板dna链的引入可用于使用同源性定向修复在基因中产生特定的靶向突变(即snp)。在备选的实施方案中,可以使用技术人员已知的任何crispr技术将至少一个突变导入grf基因和/或启动子中,特别是在上述位置处。在另一个实例中,sgrna(例如,如本文所述)可以与融合至“碱基编辑器”的修饰的cas9蛋白(例如切口酶cas9或ncas9或“死亡的”cas9(dcas9))一起使用,所述“碱基编辑器”例如酶,例如脱氨酶(例如胞苷脱氨酶)或tada(trna腺苷脱氨酶)或adar或apobec。这些酶能够用一个碱基置换另一个碱基。结果,没有缺失dna,但进行了单一置换(kim等,2017;gaudelli等,2017)。
在一个实例中,如本文所述,使用以下sgrna序列和供体dna核酸序列将突变引入mirna396结合位点中:
表1:crispr构建体,其用于在mirna396识别位点中引入tc至aa突变。
在另一个实例中,如本文所述,使用以下sgrna序列和供体dna核酸序列将至少一个突变引入grf启动子中的至少一个位置:
表2:将启动子突变引入osgrf4的crispr构建体
用于本发明方法中的cas9表达质粒可以如本领域所述进行构建。
一旦进行了靶向的基因组编辑,就可以使用快速的高通量筛选程序来分析扩增产物中grf基因和/或启动子中突变的存在,特别是在上述位置处。一旦在目的基因中鉴定出突变,携带该突变的m2植物的种子就生长为成熟m3植物,并筛选与靶基因grf4相关的表型特征。因此可以鉴定出与对照相比具有增加的grf表达或水平(其结果是氮代谢增加)的突变体。
通过这种方法获得或可获得的、在内源grf基因或启动子基因座中携带功能突变的植物也在本发明的范围内。
在本发明的备选方面中,所述方法包括在植物中引入和表达包含grf核酸的核酸构建体。优选地,grf核酸与调节序列可操作的连接。
根据本发明的所有方面,包括上述方法和包括如下所述的植物、方法和用途,术语“调节序列”在本文中与“启动子”可互换使用,并且所有术语在广义上下文中被认为是指能够实现与它们连接的序列的表达的调节核酸序列。术语“调节序列”还涵盖赋予、激活或增强核酸分子在细胞,组织或器官中表达的合成融合分子或衍生物。
在一个实施方案中,启动子可以是组成型或强启动子。
“组成型启动子”指在大多数的但不必是所有的生长和发育阶段期间以及在大多数环境条件下,在至少一个细胞、组织或器官中具有转录活性的启动子。组成型启动子的实例包括花椰菜花叶病毒启动子(camv35s或19s)、稻肌动蛋白启动子、玉米泛素启动子、rubisco小亚基、玉米或苜蓿h3组蛋白、ocs、sad1或2、gos2或任何增强表达的启动子。
“强启动子”是指导致基因增加或过表达的启动子。强启动子的实例包括但不限于camv-35s、camv-35somega、拟南芥泛素ubq1、稻泛素、肌动蛋白或玉米醇脱氢酶1启动子(adh-1)。
备选地,启动子可以是grf4启动子,优选单倍型b启动子。在一个实施方案中,单倍型b启动子包含如在seqidno:9中所定义的序列或其功能变体,或由其组成。
如本文所用的术语“可操作地连接”是指启动子序列和目的基因之间的功能性连接,使得启动子序列能够启动目的基因的转录。
在一个实施方案中,grf核酸编码grf多肽,其中grf多肽包含seqidno:3或6或如上定义的其功能变体或同源物,或由其组成。更优选地,核酸包含seqidno:1,2,4或5或如上定义的其功能变体或同源物,或由其组成。
在一个实施方案中,后代植物用本文所述的核酸构建体稳定转化,并且包含可遗传地维持在植物细胞中的外源多核苷酸。该方法可以包括验证构建体被稳定整合的步骤。该方法还可以包括从所选择的后代植物收集种子的额外步骤。
在进一步的实施方案中,所述方法可以进一步包括以下至少一个或多个步骤:评估转基因或遗传改变的植物的表型,特别是测量或评估氮摄取和/或氮同化和/或氮利用效率和/或产量的增加,其中优选地所述增加是相对于对照或野生型植物。
在一个实施方案中,核酸和调节序列来自相同的植物科。在另一个实施方案中,核酸和调节序列来自不同的植物科、属或种。
在上述方法的进一步实施方案中,所述方法在低n条件下(例如180kgn/ha或更低,优选180-120kgn/ha,甚至更优选120kgn/ha或更低)增加氮摄取和/或氮同化和/或氮利用效率和/或产量。因此,在一个实施方案中,该方法增加在氮胁迫条件下的氮摄取和/或氮同化和/或氮利用效率和/或产量。在另一个实施方案中,所述方法增加在正常(例如210kg/nha)或高n(高于300kg/nha)条件下的氮摄取和/或氮同化和/或氮利用效率和/或产量。
遗传改变或修饰的植物和产生这种植物的方法。
在本发明的另一个方面,提供了遗传改变的植物、其部分或植物细胞,其特征在于与野生型或对照植物相比,所述植物具有grf核酸或多肽的增加的表达或活性。更优选地,所述植物的特征还在于植物中氮摄取、氮同化和nue中至少一种的增加。甚至更优选地,所述植物的特征还在于产量的增加。所述植物可额外或备选地以c同化的增加为特征。
在一个实施方案中,植物在grf基因和/或其启动子中包含至少一个突变。优选地,该突变是置换,甚至更优选地,该突变是上述突变之一。在进一步的实施方案中,再次如上所述,突变是使用靶向基因组编辑引入的。
在另一个实施方案中,植物表达对个体植物“外源”的多核苷酸,所述多核苷酸是通过除有性杂交以外的任何方式引入植物中的多核苷酸。下文描述了可以实现此目的的手段的实例。在该方法的一个实施方案中,在植物中表达外源核酸,所述外源核酸是这样的核酸构建体:其包含编码如seqidno:3中所定义的多肽序列的核酸或其同源物或功能变体,并且其不是所述植物内源的,而是来自另一植物物种。例如,osgrf4构建体可在不是稻的另一植物中表达。备选地,内源核酸构建体是在转基因植物中表达的。例如,osgrf4构建体可在稻中表达。因此,在一个实施方案中,植物表达这样的核酸:其包含编码如seqidno:3中定义的多肽序列的核酸或其同源物或功能变体。在这些实施方案的任一个中,植物是转基因植物。
在本发明的另一个方面,提供了制备转基因植物的方法,其特征在于所述植物表现出如本文所述的氮摄取和/或氮同化和/或氮利用效率的增加,以及任选地额外的产量和/或c同化的增加。优选地,所述方法包括在植物或植物细胞中引入和表达核酸构建体,所述核酸构建体包含编码如seqidno:3中所定义的多肽的核酸或其同源物或功能变体。在一个实施方案中,核酸构建体包含如seqidno:1或2中定义的核酸序列或其同源物或功能变体,或由其组成。
用于产生本发明的转基因植物的转化方法是本领域已知的。因此,根据本发明的各个方面,将本文定义的核酸构建体引入植物中并作为转基因表达。通过称为转化的方法将核酸构建体引入所述植物中。本文提及的术语“引入”或“转化”涵盖将外源多核苷酸转移到宿主细胞中,而与用于转移的方法无关。能够随后克隆繁殖的植物组织,无论是通过器官发生还是胚发生,都可用本发明的遗传构建体进行转化和从其再生完整植物。所选的特定组织将根据可用于并且最适合于正被转化的特定物种的克隆繁殖系统而变化。示例性的组织靶包括叶盘、花粉、胚、子叶、下胚轴、大配子体、愈伤组织、现有的分生组织(例如,顶端分生组织、腋芽和根分生组织)和诱导的分生组织(例如,子叶分生组织和下胚轴分生组织)。多核苷酸可以被瞬时或稳定地引入宿主细胞中,并且可以保持非整合,例如作为质粒。备选地,它可以整合到宿主基因组中。然后,可以用本领域技术人员已知的方法将所得的转化植物细胞用于再生转化植物。
植物的转化现在在许多物种中是常规技术。有利地,可以使用几种转化方法中的任何一种将目的基因引入合适的祖细胞中。所描述的用于从植物组织或植物细胞转化和再生植物的方法可用于瞬时或稳定转化。转化方法包括使用脂质体、电穿孔、增加游离dna摄取的化学物质、将dna直接注射到植物中、粒子枪轰击、使用病毒或花粉的转化和显微注射。方法可选自用于原生质体的钙/聚乙二醇方法、原生质体的电穿孔、显微注射到植物材料中、dna或rna包被的粒子轰击、用病毒感染(非整合)等。转基因植物,包括转基因作物植物,优选通过根癌农杆菌(agrobacteriumtumefaciens)介导的转化产生。
为了选择转化的植物,使在转化中获得的植物材料经受选择性条件,以便转化的植物可以与未转化的植物区分开。例如,可以种植以上述方式获得的种子,并且在初始生长期之后,通过喷雾进行适当的选择。另外的可能性是,如果合适,在灭菌后,在使用合适的选择剂的琼脂平板上使种子生长,以便只有转化的种子才能够生长为植物。备选地,筛选转化的植物中是否存在选择标记。在dna转移和再生之后,也可以例如使用southern印迹分析评价推定转化的植物中目的基因的存在、拷贝数和/或基因组组织。备选地或另外地,可使用northern和/或western印迹分析来监测新引入的dna的表达水平,这两种技术都是本领域普通技术人员所熟知的。
生成的转化植物可以通过多种手段繁殖,例如通过克隆繁殖或传统育种技术。例如,可以将第一代(或t1)转化的植物自交,并选择纯合的第二代(或t2)转化体,然后t2植物可通过传统育种技术进一步繁殖。产生的转化生物可采取多种形式。例如,它们可以是转化细胞和非转化细胞的嵌合体;克隆转化体(例如,所有细胞转化为含有表达盒);转化的和未转化的组织的嫁接(例如,在植物中,将转化的砧木嫁接到未转化的接穗上)。
所述方法可以进一步包括从所述植物或植物细胞再生转基因植物,其中所述转基因植物在其基因组中包含选自seqidno:1或2的核酸序列或编码如seqidno:3中定义的grf蛋白的核酸,并获得源自所述转基因植物的后代植物,其中所述后代表现出氮摄取和/或氮同化和/或氮利用效率中至少一种的增加,以及任选地额外地产量和/或c同化的增加。
在本发明的另一方面,提供了用于产生如本文所述的遗传改变的植物的方法。在一个实施方案中,所述方法包括使用本文所述的任何诱变技术,将至少一个突变引入优选至少一个植物细胞的grf基因和/或grf启动子中。优选地,所述方法进一步包括从突变的植物细胞再生植物。
所述方法可进一步包括选择一种或更多种突变植物,优选用于进一步繁殖。优选地,所述选择的植物在grf基因和/或启动子序列中包含至少一个突变。在一个实施方案中,所述植物的特征在于grf表达水平增加和/或grf多肽活性水平增加。grf的表达和/或活性水平可通过本领域技术人员已知的任何标准技术来测量。增加是如本文所描述的。
所选植物可通过多种手段繁殖,诸如通过克隆繁殖或传统育种技术。例如,可以将第一代(或t1)转化的植物自交,并选择纯合的第二代(或t2)转化体,然后t2植物可通过传统育种技术进一步繁殖。产生的转化生物可采取多种形式。例如,它们可以是转化细胞和非转化细胞的嵌合体;克隆转化体(例如,所有细胞转化为含有表达盒);转化的和未转化的组织的嫁接(例如,在植物中,将转化的砧木嫁接到未转化的接穗上)。
在本文所述的任何方法的进一步的实施方案中,所述方法可进一步包括以下步骤中的至少一个或多个:评估转基因或遗传改变的植物的表型,测量氮摄取和/或氮同化和/或氮利用效率和任选地额外地产量和/或c同化中的至少一种的增加,并比较所述表型以确定野生型或对照植物中氮摄取和/或氮同化和/或氮利用效率和任选地额外地产量和/或c同化中的至少一种的增加。换句话说,该方法可以包括筛选植物以获得所需表型的步骤。
在本发明的另外方面,提供了通过上述方法获得的或可获得的植物。
与靶向基因组修饰的方法一起使用的基因组编辑构建体
“crrna”或crisprrna是指含有前间隔区元件和与tracrrna互补的附加核苷酸的rna序列。
“tracrrna”(反式激活rna)是指这样的rna序列:其与crrna杂交并与crispr酶如cas9结合,从而激活核酸酶复合物,以便在至少一个grf1核酸或启动子序列的基因组序列内的特定位点处引入双链断裂。
“前间隔区元件”是指与基因组dna靶序列互补的crrna(或sgrna)的部分,通常长度为约20个核苷酸。这也可以称为间隔区或靶向序列。
“供体序列”是含有将特定置换引入靶序列的所有必需元件的核酸序列,优选使用同源定向修复或hdr。在一个实施方案中,供体序列包含用于引入至少一个snp的修复模板序列。优选地,修复模板序列侧翼为与靶序列相同的至少一个,优选左臂和右臂,更优选各自约100bp。更优选地,一个或多个臂进一步侧翼为两个包含pam基序的grna靶序列,使得供体序列可被cas9/grna释放。
“sgrna”(单向导rna)是指单个rna分子中的tracrrna和crrna的组合,优选还包括连接环(其将tracrrna和crrna连接成单个分子)。“sgrna”也可称为“grna”,并且在本文中,这些术语是可互换的。sgrna或grna提供了对于cas核酸酶的靶向特异性和支架/结合能力。grna可以指包含crrna分子和tracrrna分子的双rna分子。
“tal效应子”(转录激活因子样(tal)效应子)或tale是指这样的蛋白质序列:其能够结合基因组dna靶序列(grf1基因或启动子序列内的序列),并能够与内切核酸酶如fokⅰ的切割结构域融合以产生tal效应子核酸酶或talens或大范围核酸酶以产生大范围tal。tale蛋白由负责dna结合的中心结构域、核定位信号和激活靶基因转录的结构域组成。dna结合结构域由单体组成,并且每个单体能够与靶核苷酸序列中的一个核苷酸结合。单体是33-35个氨基酸的串联重复,其中位于位置12和13的两个氨基酸是高度可变的(重复可变双残基,rvd)。rvd负责识别单个特异性核苷酸。hd靶向胞嘧啶;ni靶向腺嘌呤,ng靶向胸腺嘧啶,nn靶向鸟嘌呤(尽管nn也能够以较低的特异性结合腺嘌呤)。
在本发明的另一方面,提供了核酸构建体,其中所述核酸构建体编码至少一个dna结合结构域,其中所述dna结合结构域能够结合grf基因中的序列,其中所述序列选自seqidno:49,52,57,60,65,68,73,76,81,85,90,93,98,101,106,109,114,117,122,125,130,133,138,141,146,149,154,157,162,165,170,173,178,181,186和189。在一个实施方案中,所述构建体进一步包含编码(ssn)序列特异性核酸酶(如foki或cas蛋白)的核酸。
在一个实施方案中,核酸构建体编码至少一种前间隔区元件,其中所述前间隔区元件的序列选自seqidno:50,53,58,61,66,69,74,77,82,86,91,94,99,102,107,110,115,118,123,126.131,134,139,142,147,150,155,158,163,166,171,174,179,182,187和190或其变体。
在进一步的实施方案中,核酸构建体包含crrna编码序列。如上所定义,crrna序列可包含如上所定义的前间隔区元件,优选地包含与tracrrna互补的附加核苷酸。附加核苷酸的适当序列将是本领域技术人员已知的,因为这些序列是由cas蛋白的选择来定义的。
在另一个实施方案中,核酸构建体进一步包含tracrrna序列。再次,合适的tracrrna序列是本领域技术人员已知的,因为该序列是由选择的cas蛋白来定义的。尽管如此,在一个实施方案中,所述序列包含如seqidno:46所定义的序列或其变体,或由其组成。
在进一步的实施方案中,核酸构建体包含至少一个编码sgrna(或grna)的核酸序列。再次,如已经讨论的,sgrna通常包含crrna序列、tracrrna序列,优选地针对接头环的序列。在优选的实施方案中,核酸构建体包含至少一个编码如seqidno:51,54,59,62,67,70,75,78,83,87,92,95,100,103,108,111,116,119,124,127,132,135,140,143,148,151,156,159,164,167,172,175,180,183,188和191中任一项所定义的sgrna序列或其变体的核酸序列。
在进一步的实施方案中,核酸构建体可进一步包含至少一个编码内切核糖核酸酶切割位点的核酸序列。优选内切核糖核酸酶是csy4(也称为cas6f)。当核酸构建体包含多个sgrna核酸序列时,构建体可包含相同数量的内切核糖核酸酶切割位点。在另一个实施方案中,切割位点是sgrna核酸序列的5’。因此,每个sgrna核酸序列的侧翼是内切核糖核酸酶切割位点。
术语“变体”指核苷酸序列,其中核苷酸与上述序列之一是基本上同一的。变体可通过修饰例如一个或更多个核苷酸的插入、置换或缺失来实现。在优选的实施方案中,变体与上述序列中的任何一个具有至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%的同一性。在一个实施方案中,序列同一性为至少90%。在另一个实施方案中,序列同一性为100%。序列同一性可通过本领域任何一种已知的序列比对程序来确定。
本发明还涉及包含与合适的植物启动子可操作地连接的核酸序列的核酸构建体。合适的植物启动子可以是组成型启动子或强启动子,或者可以是组织特异性启动子。在一个实施方案中,合适的植物启动子选自但不限于夜香树黄叶卷曲病毒(cmylcv)启动子或柳枝稷泛素1启动子(pvubi1)、小麦u6rna聚合酶iii(tau6)、camv35s、小麦u6或玉米泛素(例如ubi1)启动子。在一个实施方案中,启动子是p35s(seqidno:40)或pubi(seqidno:41)
本发明的核酸构建体还可进一步包含编码crispr酶的核酸序列。“crispr酶”是指可以与crispr系统相关联的rna引导的dna内切核酸酶。具体地,这种酶与tracrrna序列结合。在一个实施方案中,cripsr酶是cas蛋白(“crispr相关蛋白”),优选cas9或cpf1,更优选cas9。在具体的实施方案中,cas9是密码子优化的cas9,并且更优选地,具有seqidno:42中所述的序列或其功能变体或同源物。在另一个实施方案中,crispr酶是来自2类候选x蛋白家族的蛋白质,例如c2c1、c2c2和/或c2c3。在一个实施方案中,cas蛋白来自酿脓链球菌(streptococcuspyogenes)。在备选的实施方案中,cas蛋白可来自于金黄色葡萄球菌(staphylococcusaureus)、脑膜炎奈瑟球菌(neisseriameningitides)、嗜热链球菌(streptococcusthermophiles)或齿垢密螺旋体(treponemadenticola)中的任何一种。
本文所使用的涉及cas9的术语“功能变体”,指变体cas9基因序列或所述基因序列的一部分,它保留完整非变体序列的生物学功能,例如,作为dna内切核酸酶,或识别或/和结合dna。功能变体还包含目的基因的变体,其具有不影响功能例如非保守残基的序列改变。还涵盖与本文所示的野生型序列相比基本同一的变体,即仅具有一些(例如在非保守残基中的)序列变异,并且具有生物学活性。在一个实施方案中,seqidno:42的功能变体与seqidno:42所示的氨基酸具有至少90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的总序列同一性。在进一步的实施方案中,已修饰cas9蛋白以改善活性。
可通过序列比较和保守结构域的鉴定来鉴定合适的同源物或直向同源物。可以如本文所描述的鉴定同源物或直向同源物的功能,并且因此当在植物中表达时技术人员能够证实该功能。
在进一步的实施方案中,已修饰cas9蛋白以改善活性。例如,在一个实施方案中,cas9蛋白可包含d10a氨基酸置换,该切口酶仅切割与grna互补并被grna识别的dna链。在备选的实施方案中,cas9蛋白可以备选地或额外地包含h840a氨基酸置换,该切口酶仅切割不与srna相互作用的dna链。在该实施方案中,cas9可与一对(即两个)sgrna分子(或表达该对的构建体)一起使用,结果是能够切割相对dna链上的靶区域,具有将特异性提高100-1500倍的可能性。在进一步的实施方案中,cas9蛋白可以包含d1135e置换。cas9蛋白还可以是vqr变体。备选地,cas蛋白可以在两个核酸酶结构域hnh和ruvc样中包含突变,因此是催化失活的。不是切割靶链,而是这种催化失活的cas蛋白可用于防止转录延伸过程,导致不完全翻译的蛋白在与sgrna分子共表达时丧失功能。催化失活蛋白的实例是由ruvc和/或hnh核酸酶结构域中的点突变引起的死亡cas9(dcas9)(komor等,2016和nishida等,2016)。
在进一步的实施方案中,cas蛋白诸如cas9可进一步与阻抑效应子诸如组蛋白修饰/dna甲基化酶或碱基编辑物诸如胞苷脱氨酶(komor等,2016)融合,以实现如上所述的定点诱变。在后者中,胞苷脱氨酶不诱导dsdna断裂,但介导胞苷向尿苷的转化,从而实现c至t(或g至a)的置换。该方法对于在上述位置-855、-847、-801和-522处产生多态性可能特别有价值。
在进一步的实施方案中,核酸构建体包含内切核糖核酸酶。优选地,内切核糖核酸酶是csy4(也称为cas6f),并且更优选地是密码子优化的csy4,例如如seqidno:43中所定义的。在一个实施方案中,当核酸构建体包含cas蛋白时,核酸构建体可以包含用于表达内切核糖核酸酶的序列,例如表达为与cas蛋白(例如cas9)的5'末端p2a融合体(用作自切割肽)的csy4。
在一个实施方案中,cas蛋白、内切核糖核酸酶和/或内切核糖核酸酶-cas融合序列可以与合适的植物启动子可操作地连接。合适的植物启动子已经在上文描述,但在一个实施方案中,可以是玉米泛素1启动子。
用于产生crispr核酸和载体系统的合适方法是已知的,并且例如公开于molecularplant(ma等人,2015,molecularplant,doi:10.1016/j.molp.2015.04.007),其通过引用并入本文。
在本发明的备选方面,所述核酸构建体包含至少一种编码tal效应子的核酸序列,其中所述效应子靶向选自seqidno:40,49,52,57,60,65,68,73,76,81,85,90,93,98,101,106,109,114,117,122,125,130,133,138,141,146,149,154,157,162,165,170,173,178,181,186和189的grf序列。考虑到靶序列,用于设计tal效应子的方法是本领域技术人员熟知的。合适方法的实例在sanjana等人和cermakt等人中给出,两者均通过引用并入本文中。优选地,所述核酸构建体包含编码tal效应子的两个核酸序列,以产生talen对。在进一步的实施方案中,核酸构建体进一步包含序列特异性核酸酶(ssn)。优选地,这种ssn是内切核酸酶,例如foki。在进一步的实施方案中,talen通过goldengate克隆方法装配在单个质粒或核酸构建体中。
在本发明的另一方面,提供了sgrna分子,其中所述sgrna分子包含crrna序列和tracrrna序列,并且其中所述crrna序列能够结合选自seqidno:49,52,57,60,65,68,73,76,81,85,90,93,98,101,106,109,114,117,122,125,130,133,138,141,146,149,154,157,162,165,170,173,178,181,186和189或其变体的至少一个序列。“变体”是如本文所定义的。在一个实施方案中,sgrna分子可包含至少一个化学修饰,例如增强其对靶序列的或crrna序列对tracrrna序列的稳定性和/或结合亲和力。这种修饰将是本领域技术人员所熟知的,并且包括例如但不限于rahdar等人,2015中所述的修饰,其通过引用并入本文。在这个实例中,crrna可以包含硫代磷酸酯骨架修饰,例如2’-氟(2’-f)、2’-o-甲基(2’-o-me)和s-受限的乙基(cet)取代。
在本发明的另一个方面,提供了分离的核酸序列,其编码前间隔区元件(如在seqidno:41,50,53,58,61,66,69,74,77,82,86,91,94,99,102,107,110,115,118,123,126.131,134,139,142,147,150,155,158,163,166,171,174,179,182,187和190的任一个中所定义)或其变体,或sgrna(如在seqidno:51,54,59,62,67,70,75,78,83,87,92,95,100,103,108,111,116,119,124,127,132,135,140,143,148,151,156,159,164,167,172,175,180,183,188和191中的任一个所述或其变体)。
在本发明的另一个方面,提供了包含与如本文定义的调控序列可操作地连接的修复模板序列的核酸构建体。在一个实施方案中,修复模板序列包含选自seqidno:47,53,63,71,79,88,96,104,112,120,128,136,144,152,160,168,176和184的核酸序列或其变体。调节序列是如本文所定义的。
在本发明的另一方面,提供了另一种核酸构建体,其中所述核酸构建体包含供体dna序列。在一个实施方案中,供体dna序列包含选自seqidno:48,56,64,72,80,84,89,97,105,113,121,129,137,145,153,161,169,177和185的核酸序列或其变体。在进一步优选的实施方案中,核酸序列包含至少一个,优选两个,如本文所定义的sgrna核酸序列,更优选如本文所定义的cas核酸序列。在一个实施方案中,至少一个sgrna核酸、cas核酸和供体dna序列与相同的调节序列可操作地连接。在备选的实施方案中,至少一个sgrna核酸、cas核酸和供体dna序列与不同的调节序列可操作地连接。例如,至少一个sgrna核酸可与u3启动子可操作地连接,且cas与ubi启动子可操作地连接。在这个实施方案中,从构建体表达的sgrna核酸用于在靶序列中产生双链断裂,其然后使用hdr和供体dna序列修复,如sun等(2016)所述。该方法可用于将至少一个,但优选至少两个置换插入到靶序列中。
在本发明的另一方面,提供了用至少一个本文所述的核酸构建体转染的植物或其部分或至少一个分离的植物细胞。cas9和sgrna可以组合或在单独的表达载体(或核酸构建体,这些术语可互换使用)中。类似地,cas9、sgrna和供体dna序列可以组合或在分开的表达载体中。换句话说,在一个实施方案中,用包含sgrna和cas9二者或sgrna、cas9和供体dna序列的单个核酸构建体转染分离的植物细胞,如上文详细描述的。在备选的实施方案中,分离的植物细胞是用两种或三种核酸构建体转染的,第一核酸构建体包含至少一个如上所定义的sgrna,第二核酸构建体包含cas9或其功能变体或同源物,并且任选地第三核酸构建体包含如上所定义的供体dna序列。第二和/或第三核酸构建体可以在第一和/或第二核酸构建体之前、之后或同时转染。分开的包含cas蛋白的第二构建体的优点在于,编码至少一个sgrna的核酸构建体可与任何类型的cas蛋白配对,如本文所描述的,并且因此不限于单一cas功能(当cas和sgrna都在相同核酸构建体上编码时将是这种情况)。
在一个实施方案中,在用包含至少一个sgrna核酸的核酸构建体的第二次转染之前,首先转染包含cas蛋白的核酸构建体并将其稳定掺入基因组中。在备选实施方案中,植物或其部分或至少一个分离的植物细胞用编码cas蛋白的mrna转染,并用至少一个如本文所定义的核酸构建体共转染。
用于本发明中的cas9表达载体可以如在本领域中所描述的进行构建。在一个实例中,表达载体包含如seqidno:42所定义的核酸序列或其功能变体或同源物,其中所述核酸序列与合适的启动子可操作地连接。合适的启动子的实例包括肌动蛋白、camv35s、小麦u6或玉米泛素(例如ubi1)启动子。
本发明的范围还包括上述核酸构建体(crispr构建体)或sgrna分子在任何上述方法中的用途。例如,提供了上述crispr构建体或sgrna分子在增加如本文所述的grf表达或活性中的用途。
因此,在本发明的进一步的方面,提供了增加grf表达和/或活性的方法,该方法包括引入并表达上述任一构建体,或将也如上所述的sgrna分子引入植物中。换言之,还提供了增加grf表达和/或活性的方法,如本文所述,其中所述方法包括使用crispr/cas9,并且具体地,本文所述的crispr(核酸)构建体,将至少一个突变引入内源grf基因和/或启动子中。
因此,在本发明的进一步的方面,提供了产生具有grfngr2等位基因的植物的方法,.该方法包括在植物中引入和表达如上定义的任何核酸构建体,或引入也如上定义的sgrna分子。
在本发明的备选方面,提供了用至少一个如本文所述的sgrna分子转染的分离的植物细胞。
在本发明的另外方面,提供了包含如本文所述的转染细胞的遗传修饰或编辑的植物。在一个实施方案中,所述核酸构建体或所述多个核酸构建体可以是以稳定的形式整合的。在备选的实施方案中,所述核酸构建体或所述多个核酸构建体是未整合的(即瞬时表达)。因此,在优选的实施方案中,遗传修饰的植物不含任何sgrna和/或cas蛋白核酸。换句话说,植物是无转基因的。
本文提及的术语“引入”、“转染”或“转化”涵盖将外源多核苷酸转移到宿主细胞中,而与用于转移的方法无关。能够随后克隆繁殖的植物组织,无论是通过器官发生还是胚发生,都可用本发明的遗传构建体进行转化和从其再生完整植物。所选的特定组织将根据可用于并且最适合于正被转化的特定物种的克隆繁殖系统而变化。示例性的组织靶包括叶盘、花粉、胚、子叶、下胚轴、大配子体、愈伤组织、现有的分生组织(例如,顶端分生组织、腋芽和根分生组织)和诱导的分生组织(例如,子叶分生组织和下胚轴分生组织)。然后,可以用本领域技术人员已知的方式将所得的转化植物细胞用于再生转化植物。
外源基因向植物基因组中的转移被称为转化。植物的转化现在在许多物种中是常规技术。技术人员已知的几种转化方法中的任何一种都可用于将目的核酸构建体或sgrna分子引入合适的祖先细胞中。所描述的用于从植物组织或植物细胞转化和再生植物的方法可用于瞬时或稳定转化。
转化方法包括使用脂质体、电穿孔、增加游离dna摄取的化学物质、将dna直接注射到植物中(显微注射)、如实施例中所述的基因枪(或生物射弹颗粒递送系统(biologistics))、脂转染、使用病毒或花粉和微突出物的转化。方法可选自用于原生质体的钙/聚乙二醇方法、超声介导的基因转染、光学或激光转染、使用碳化硅纤维的转染、原生质体的电穿孔、显微注射到植物材料中、dna或rna包被的粒子轰击、感染(非整合)病毒等。转基因植物也可以通过根癌农杆菌介导的转化来产生,包括但不限于使用如clough&bent(1998)中所述的浸花法/农杆菌真空渗透方法,该方法通过引用并入本文。
因此,在一个实施方案中,使用上述方法中的任何一种,可将本文所述的至少一个核酸构建体或sgrna分子引入至少一个植物细胞中。在备选的实施方案中,本文所述的任何核酸构建体可首先转录以形成预装配的cas9-sgrna核糖核蛋白,然后用任何上述方法(如脂转染、电穿孔或显微注射)将其递送到至少一个植物细胞。
任选地,为了选择转化的植物,通常使在转化中获得的植物材料经受选择性条件,以便转化的植物可以与未转化的植物区分开。例如,可以种植以上述方式获得的种子,并且在初始生长期之后,通过喷雾进行适当的选择。另外的可能性是,如果合适,在灭菌后,在使用合适的选择剂的琼脂平板上使种子生长,以便只有转化的种子才能够生长为植物。如实例中所述,合适的标记物可以是bar-膦丝菌素或ppt。或者,筛选转化植物中是否存在选择标记,例如但不限于gfp、gus(β-葡糖醛酸糖苷酶)。其它实例对于本领域技术人员来说是容易知道的。备选地,不进行选择,种植以上述方式获得的种子并使之生长,并在合适的时间使用本领域的标准技术测量gse5表达或蛋白质水平。这种避免引入转基因的备选方法优选用于产生无转基因植物。
dna转移和再生之后,也可评价推定转化的植物,例如使用pcr检测目的基因的存在、拷贝数和/或基因组组织。备选地或另外地,可使用southern、northern和/或western分析来监测新引入的dna的整合和表达水平,这两种技术都是本领域普通技术人员所熟知的。
生成的转化植物可以通过多种手段繁殖,例如通过克隆繁殖或传统育种技术。例如,可以将第一代(或t1)转化的植物自交,并选择纯合的第二代(或t2)转化体,然后t2植物可通过传统育种技术进一步繁殖。
使用上述crispr构建体的具体方案是技术人员公知的。作为一个实例,合适的方案描述于ma&liu(“crispr/cas-basedmultiplexgenomeeditinginmonocotanddicotplants”)中,其通过引用并入本文。
在本发明的另外的相关方面,还提供了获得本文所述的遗传修饰植物的方法,所述方法包括:
a.选择植物的部分;
b.使用上述转染或转化技术,用至少一个本文所述的核酸构建体或至少一个本文所述的sgrna分子转染段落(a)的植物的部分的至少一个细胞;
c.使来源于所述转染的细胞的至少一个植物再生;
d.选择根据段落(c)获得的一个或多个植物,其显示grf,优选grf4的增加的表达或功能。
在进一步的实施方案中,所述方法还包括筛选遗传修饰植物,以获得grf基因或启动子序列中ssn(优选crispr)诱导的突变的步骤。在一个实施方案中,所述方法包括从转化的植物中获得dna样品,并进行dna扩增以检测在至少一个grf基因或启动子序列中的突变。
在进一步的实施方案中,所述方法包括生成稳定的t2植物,优选地对于突变(即在至少一个grf基因或启动子序列中的突变)是纯合的。
在至少一个grf基因和/或启动子序列中具有突变的植物也可以与在至少一个grf基因和/或启动子序列中也含有至少一个突变的另一植物杂交,以获得在grf1基因或启动子序列中具有额外突变的植物。这些组合对本领域技术人员来说是显而易见的。因此,当与如上所述转化的单个t1植物中的同源物(homoeolog)突变的数目相比时,该方法可用于产生在全部或更多数量的同源物上具有突变的t2植物。
通过上述方法获得的或可获得的植物也在本发明的范围内。
也可通过杂交(例如使用本文所述的遗传改变的植物的花粉对野生型或对照植物授粉,或用在grf基因或启动子序列的至少一个中不含突变的其它花粉对本文所述的植物的雌蕊授粉)通过转移本发明的任何序列来获得本发明的遗传改变的植物。用于获得本发明植物的方法不仅限于本段中所述的那些;例如,可以如提到的那样进行来自麦穗的胚芽细胞的遗传转化,但之后不必使再生植物。
筛选植物的天然存在的增加的氮摄取和谷物产量表型的方法
在本发明的进一步的方面,提供了一种方法,该方法用于筛选植物种群,并鉴定和/或选择携带或表达grf的ngr等位基因的植物,如本文所述。备选地,提供了用于筛选植物群体和鉴定和/或选择具有增加的氮摄取和/或氮同化和/或氮利用效率和任选地额外地增加的产量和/或c同化的植物的方法。在任一方面,该方法包括在植物或植物种质中检测grf基因和/或启动子中的至少一个多态性。优选地,所述筛选包括确定至少一个多态性的存在,其中所述多态性是至少一个插入和/或至少一个缺失和/或置换,更优选置换。
在一个具体的实施方案中,所述多态性可以包含至少一个如下置换:
-在grf起始密码子位置-941或-935处或在seqidno:7的位置60或seqidno:8的位置66处或其同源位置处的c至t置换。
-在grf起始密码子位置-884或-878处或在seqidno:7的位置118或seqidno:8的位置124处或其同源位置处的t至a置换;
-在grf起始密码子位置-855或-849处或在seqidno:7的位置148或seqidno:8的位置154处或其同源位置处的c至t置换;
-在grf起始密码子位置-847或-841处或在seqidno:7的位置157或seqidno:8的位置163处或其同源位置处的c至t置换;
-在grf起始密码子位置-801或-795处或在seqidno:7的位置204或seqidno:8的位置210处或其同源位置处的c至t置换;
-在grf起始密码子位置-522或-516处或在seqidno:7的位置484或seqidno:8的位置489处或其同源位置处的c至t置换;
-在grf起始密码子位置-157处或在seqidno:7的位置850或seqidno:8的位置516处或其同源位置处的g至c置换;
在优选的实施方案中,突变是
-在grf起始密码子位置-884或-878处或在seqidno:7的位置118或seqidno:8的位置124处或其同源位置处的t至a置换;和
-在grf起始密码子位置-847或-841处或在seqidno:7的位置157或seqidno:8的位置163处或其同源位置处的c至t置换;
-在grf起始密码子位置-801或-795处或在seqidno:7的位置204或seqidno:8的位置210处或其同源位置处的c至t置换。
如上所述,包含所有三种上述多态性的grf启动子可以称为单倍型b。
用于评定多态性存在的合适的测试是本领域技术人员熟知的,包括但不限于同工酶电泳、限制性片段长度多态性(rflp)、随机扩增多态性dna(rapd)、随机引发聚合酶链反应(ap-pcr)、dna扩增指纹(daf)、序列特定扩增区域(scar)、扩增片段长度多态性(aflp)、简单序列重复(ssr-也称为微卫星)和单核苷酸多态性(snp)。在一个实施方案中,使用kompetitive等位基因特异性pcr(kasp)基因分型。
在一个实施方案中,该方法包括:
a)从植物中获得核酸样品和
b)使用一个或多个引物对进行一个或多个grf或grf启动子等位基因的核酸扩增。
在进一步的实施方案中,所述方法可以进一步包括将包含grf多态性的染色体区域基因渐渗到第二植物或植物种质中,以产生基因渐渗的植物或植物种质。优选地,所述第二植物将显示氮摄取和/或氮同化和/或氮利用效率和任选地额外地产量和/或c同化的增加。
因此,在本发明的进一步方面,提供了用于增加植物中氮摄取和/或氮同化和/或氮利用效率以及任选地额外地产量和/或c同化的方法,该方法包括
a.筛选植物群体中的至少一种具有本文所述grf多态性的植物;
b.通过将至少一个突变引入至编码grf的核酸序列中和/或将至少一个突变引入至本文所述的grf启动子中,进一步调节本文所述的grf多肽在所述植物中的表达或活性。
grfngr构建体
如通篇所讨论的,本发明人惊奇地鉴定到grf(特别是grf4)的过表达增加植物中的氮代谢和产量。
因此,在本发明的另一方面,提供了核酸构建体,其包含编码如seqidno:3中所定义的多肽或其功能变体或同源物的核酸序列,其中所述序列与调节序列可操作地连接。优选地,所述调节序列是组织特异性启动子或组成型启动子。功能变体或同源物是如上文所定义的。合适的启动子也如上所述。然而,在一个实施方案中,启动子可以是如本文所述的单倍型b启动子。优选地,该启动子包含seqidno:9或其变体,或由其组成。
在本发明的另一方面,提供了包含上述核酸序列的载体。
在本发明的进一步的方面,提供了包含核酸构建体的宿主细胞。宿主细胞可以是细菌细胞,例如根癌农杆菌,或分离的植物细胞。本发明还涉及包含培养基和如下所述的分离的宿主细胞的培养基或试剂盒。
在另一个实施方案中,提供了表达上述核酸构建体的转基因植物。在一个实施方案中,所述核酸构建体稳定地掺入植物基因组中。
通过上述称为转化的方法将核酸序列引入所述植物中。
在另一个方面,本发明涉及如本文所述的核酸构建体在增加氮摄取和/或氮同化和/或氮利用效率的至少一种,和任选地额外地产量和/或c同化中的用途。
在本发明的进一步的方面,提供了增加氮摄取和/或氮同化和/或氮利用效率中至少一种以及任选地额外地产量和/或c同化的方法,该方法包括在所述植物中引入和表达本文所述的核酸构建体。
在本发明的另一个方面,提供了产生具有增加的氮摄取和/或氮同化和/或氮利用效率中至少一种,和任选地额外地产量和/或c同化的植物的方法,该方法包括在所述植物中引入和表达本文所述的核酸构建体,所述增加是相对于对照或野生型植物而言的。
虽然前述公开提供了对本发明范围内所包含的主题的一般描述,包括制备和使用本发明的方法及其最佳模式,但提供了以下实施例以进一步使本领域技术人员能够实施本发明并提供其完整的书面描述。然而,本领域技术人员将理解,这些实施例的细节不应被理解为对本发明的限制,本发明的范围应由本公开所附权利要求及其等同方案来理解。鉴于本公开,本发明的各种另外的方面和实施方案对于本领域技术人员将是显而易见的。
本文中“和/或”用来作为具有或不具有另一个的两个指定特征或组分中的每一个的具体公开。例如,“a和/或b”应被认为是(i)a、(ii)b和(iii)a和b中的每一个的具体公开,就像每个在本文中单独地列出一样。
除非上下文另外指出,以上陈述的特征的描述和定义不限于本发明的任何特定方面或实施方案,并且等同地应用于所描述的所有方面和实施方案。
前述申请、其中引用或在其审查期间引用的所有文件和序列登录号(“申请引用的文献”)、和在申请的引用文献中引用或参考的所有文献、以及本文引用或参考的所有文献(“本文引用的文献”)、本文引用的文献中引用或参考的所有文献、以及本文或通过引用并入本文的任何文献中提及的任何产品的任何制造商的说明书、描述、产品说明书和产品页,均通过引用并入本文,并且可以用于本发明的实施。更具体地说,所有参考文献均通过引用并入本文,其程度如同每个单独的文件被具体和单独地指出通过引用并入。
现在在以下非限制性实施例中进一步描述本发明。
实施例1
植物将代谢同化的调节与生长的调节整合。然而,对这种协调整合的分子机制了解甚少。为了增进理解,我们系统地分析了携带1960年代“绿色革命”特征的突变等位基因的谷类品种的生长和同化特性。绿色革命提高了产量,养活了不断增长的世界人口,并且部分地是由半矮化绿色革命品种(grv)的采用所推动1-3。grv半矮态是由突变性rht(小麦)4,5和sd1(籼稻)6,7等位基因赋予的生长阻抑性della蛋白(della)的积累引起的。在正常植物中,植物激素赤霉素(ga)刺激della破坏,因此促进生长8,9.然而,在grv中,della破坏被抑制。突变小麦grvdella4抵抗ga刺激的破坏,而sd1导致ga丰度降低,随后导致slr1della积累10。在两种情况下,积累的della都抑制生长,导致半矮态,并由此导致对产量减少的“倒伏”(风和雨使植物倒伏)的抗性3。
grv倒伏抗性增强,因为突变的rht和sd1等位基因赋予对增加氮(n)供应的部分生长不敏感性。例如,南京6籼稻(nj6)的高度响应于n的增加而增加,但是在nj6-sd1等基因植物中该响应减少(图1a)。rht-b1b半矮化突变体小麦具有相似的特性(与等基因rht-b1a(wt)小麦相比;图1b)。虽然grvdella积累抑制营养生长n-响应,但是n向籽粒的分配继续,使得能够提高可收获产量和减少由于增加n-供应而产生的倒伏风险。这些产量增强特性在过去的50年期间推动了grv培养的快速传播2,并确保了突变体sd1和rht等位基因在目前优良品种中的保留4,5,11。
然而,grv的部分n不敏感性与降低的n使用效率相关联12。此外,sd1和rht突变等位基因抑制n摄取。例如,nh4+是被厌氧生长的稻田稻根部同化的主要n形式13,15nh4+摄取率本身是n调节的,被高n供应降低(hn;图1c)。我们发现nj6-sd1在低n和高n条件下都表现出降低的15nh4+摄取率(图1c)。硝酸盐(no3-)是在相对有氧的土壤条件中主要摄取的n形式14,rhtb1b小麦15no3-摄取率类似地降低(图1d)。因此,除了半矮态和减少的n生长促进外,grvdella积累还赋予减少的n摄取。因此,获得高grv产量需要过量使用n肥料输入,这会不可持续地破坏环境(例如,通过农业径流15)。因此,开发尽管n供应减少但仍高产的新grv是迫切的全球可持续农业目标1,16。我们认为,grv中n代谢的系统分析可能能够发现生长和同化是如何协调的,并且这一发现可能反过来能够开发具有增加的n利用效率的新grv。
osgrf4抵消了slr1介导的对稻铵(nh4+)摄取的抑制
我们首先探讨了36个含sd1的籼稻品种中的15nh4+摄取率,发现了~3倍的差异(图2a)。有趣的是,一些产量较高的grv(例如9311)不显示最高的15nh4+摄取率,尽管多年来在中国籼稻面积上占主导地位。我们选择nm73(显示最高的15nh4+摄取率;图2a)用于数量性状基因座(qtl)分析,发现两个lod-得分峰(qngr1和qngr2,图2b)。nm73qngr1等位基因与相对低的15nh4+摄取率相关,并且在图谱位置上与sd16,7相符(参见图1c)。然而,与相对高的15nh4+摄取率相关的nm73qngr2等位基因的分子身份是未知的。
位置映射位于qngr2至osgrf417-19(图7a),暗示了nh4+摄取调节中先前未知的功能。此外,nm73(osgrf4ngr2)等位基因的杂合性赋予比nj6等位基因(osgrf4ngr2;图7b)的纯合性更高的15nh4+摄取率,表明osgrf4ngr2半显性地增加nh4+摄取。nj6-osgrf4ngr2等基因系表现出预期的更高nh4+摄取率(相对于nj6;图2c),并增加了osgrf4mrna和osgrf4蛋白的丰度(图2d;图7c),与osgrf4ngr2的半显性一致。此外,rnai降低了nj6-osgrf4ngr2的相对高的15nh4+摄取率(图2e;图7c),因此证实了qngr2和osgrf4的等效性。最后,来自天然osgrf4gngr2的osgrf4编码mrna或组成型稻肌动蛋白1启动子的转基因表达赋予nj6增加的15nh4+摄取率(图2c;图7c)。因此,osgrf4ngr2赋予nm73增加的15nh4+摄取,并抵消sd1的阻抑作用(这是由于稻della蛋白slr1的积累)。
osgrf4ngr2(nj6)和osgrf4ngr2(nm73)等位基因比较揭示了多种snp(单核苷酸多态性;图7a,d)。osgrf4ngr2snps中的两个(图7d;外显子3中位置1187t>a和1188c>a)阻止osmir396介导的osgrf4ngr2mrna的切割17-19,增加了osgrf4mrna和osgrf4丰度(图2d;图7c),并促进了nh4+摄取。然而,也显示相对高的15nh4+摄取率(图2a)的品种rd23携带缺少1187a和1188a的osgrf4等位基因。然而,rd23和nm73的确共享三个osgrf4启动子snp(-884t>a、-847c>t和-801c>t;图7d)。总之,我们检测了三种osgrf4启动子单倍型(a,如在9311中;b,具有-884a、-847t和-801t,如在nm73和rd23中;和c,常见于粳稻种质中,例如品种nipponbare;图7d)。有趣的是,osgrf4mrna丰度在品种tzzl1和rd23(均携带单倍型b)中比在携带单倍型a或c的优良品种中高(图7e)。这表明rd23nh4+摄取率相对高(图1a),原因在于启动子单倍型b赋予相对高的osgrf4mrna水平,而nm73具有更高的nh4+摄取率,原因在于它将启动子单倍型b的作用与1187a和1188a赋予的osmir396抗性结合17-19。
重要的是,我们发现尽管osgrf4调节nh4+摄取,但它本身又受n供应的调节。nj6osgrf4mrna丰度随着n的增加而减少(图2e),这可能是由于osgrf4转录的减少(osmir396丰度不随n的增加而可检测地增加;图8),从而降低了osgrf4丰度(图2f)。由于增加的osgrf4丰度增加了nh4+摄取(图2c,d),因此低n促进osgrf4丰度能够实现n稳态的反馈调节。特别地,在携带启动子单倍型b的品种(例如tzzl1和rd23)中,响应于低n供应的增加的osgrf4mrna丰度被显著扩增(图7f)。最后,crispr/cas920生成的osgrf4突变体缺乏osgrf4(图9),并表现出半矮态(图2g)、减少的15nh4+流入(图2h)、减少的15nh4+摄取率的n介导的反馈调节(图2h)和减少的n依赖性生物质积累(图2i)。因此,osgrf4是n调节的转录调节子,其促进nh4+摄取率和对n供应的生长应答,并抵消sd1(slr1)对这些过程的抑制作用。
竞争性osgrf4、slr1和osgif1相互作用调节nh4+同化
接下来,我们确定osgrf4和slr1活性如何相互抵消以调节nh4+同化作用,首先发现nj6-sd1-osgrf4ngr2等基因系保留了sd1赋予的矮态、每株植物的分蘖数和每穗的籽粒数(slr1;图3a;图10a-c),同时叶和秆的宽度增加(图10d,e)。然而,nj6-sd1-osgrf4ngr2的籽粒产量增加(图10f)。此外,多种sd1阻抑的n摄取和同化性质被osgrf4ngr2脱阻抑。首先,nj6-sd1-osgrf4ngr215nh4+摄取率大于nj6-sd1的15nh4+摄取率(与nj6的15nh4+摄取率相似),15no3-的摄取受类似的影响(图3b)。其次,在不同的n供应水平下,关键的n同化酶诸如枝条谷氨酰胺合酶(gs;nh4+同化)21和硝酸还原酶(nr;no3-同化)22的活性在nj6-sd1-osgrf4ngr2中始终高于在nj6-sd1中,并且与nj6的活性相似(图3c)。因此,osgrf4促进n摄取和同化,而slr1抑制n摄取和同化。
全转录组rna测序分析随后鉴定了642个基因,其转录物丰度在nj6-osgrf4ngr2中上调(通过osgrf4),在nj6-sd1中下调(通过slr1)(相对于nj6)(图3d),包括多个n摄取和同化基因。例如,qrt-pcr证实了在nj6-sd1-osgrf4ngr22中编码nh4+摄取转运蛋白(例如osamt1.1和osamt1.213)的mrna的根丰度升高,而在nj6-sd1中的丰度降低(图3e;图11a)。类似地,在nj6-sd1-osgrf4ngr2中,编码根和枝条nh4+同化酶(例如osgs1.223、osgs2和osnadh-gogat2)的mrna的丰度和相应的酶活性增强(图3e,f;图11b-d)。我们接着显示osgrf4通过转录激活增强n代谢。chip-seq富集了几个基序(图3g),其中多个n代谢基因启动子共有的ggcggcggcggc基序是最丰富的。emsa证明了osgrf4-his与完整但非突变的osamt1.1启动子片段的结合(图3h),并且chip-pcr证实了osgrf4与来自多个nh4+摄取和同化基因(包括osamt1.1和osgs1.2)的含gcgg的启动子片段的特异性体内缔合(图3i;图11e-h)。最后,在反式激活测定中,osgrf4激活从osamt1.1和osgs1.2启动子的转录(图3j,k;图11i-l)。接着关注no3-相关基因,qrt-pcr、chip、反式激活测定和酶活性测定证实了no3-摄取和同化是通过osgrf4介导的转录激活来促进的(图3b,c;图12)。因此,osgrf4是n代谢的总转录激活因子,并且抵消slr1的抑制作用。
因为ga促进蛋白酶体介导的slr1的破坏8,9,我们接下来研究了ga、slr1和osgrf4如何调节n代谢。ga促进nj6和nj6-sd1的15nh4+摄取率至相似的高水平(图4a)。此外,尽管ga生物合成抑制剂多效唑(pacolubutrazol)(pac)24减少nj6和nj6-sd1的15nh4+摄取,但ga消除了这种作用(图4a)。因此,slr1积累(由于sd1或pac)减少nh4+摄取,而减少的slr1积累(由于ga)增加nh4+摄取。此外,差异性slr1积累差异性调节编码nh4+摄取和同化功能的mrna的丰度:通过ga增加osamt1.1和osgs1.2mrna的丰度,通过pac降低osamt1.1和osgs1.2mrna的丰度,ga和pac的组合使所述mrna的丰度恢复到较高水平(图4b)。接下来,我们发现pac降低,而ga则促进来自osamt1.1和osgs1.2启动子的含有gcgg基序的片段的chip-pcr富集(图4c)。因此,slr1积累抑制,而减少的slr1丰度促进osgrf4结合至osamt1.1和osgs1.2启动子(图4c),从而影响osamt1.1和osgs1.2mrna水平、nh4+摄取和nh4+同化(图4a,b;图13)。
osgrf4与osgif(grf-相互作用因子)共激活因子相互作用17。bifc(图4d)和co-ip(图4e)显示osgrf4、osgif1和slr1之间的体内相互作用。osgrf4相互作用涉及保守的qlq17结构域(图14a,b),并且slr1与所有稻osgrf和osgif相互作用(图14c)。体内fret揭示了这些相互作用是竞争性的,slr1抑制osgrf4-osgif1相互作用(图4f,g)。进一步的emsa显示osgrf4-osgif1相互作用促进osgrf4与osamt1.1含有gcgg基序的启动子片段结合,并且slr1抑制这种促进(图4h)。因此,slr1抑制osgrf4-osgif1介导的从osamt1.1和osgs1.2启动子的转录激活(图4i)。此外,slr1抑制osgrf4-osgif1从osgrf4启动子的自激活转录(图15a,b),导致在nj6-sd1中osgrf4mrna和osgrf4丰度降低(相对于nj6;图15c,d),和osgrf4mrna丰度被ga调节(图15e,f)。因此,slr1以两种方式抵消osgrf4对n代谢的促进作用。首先,slr1积累减少osgrf4积累(通过抑制osgrf4转录)。其次,slr1抑制osgrf4-osgif1复合物的形成,从而减少osgrf4-激活的n代谢基因的转录。
osgrf4-slr1相互作用整合同化代谢和生长
虽然长期以来已知n摄取率与光合作用碳(c)固定的速率相偶联25,但是连接c和n代谢的平衡分子机制仍然未知。因为osgrf4-slr1相互作用调节n同化,我们确定了它是否也调节c同化。首先,nj6-sd1-osgrf4ngr2和nj6-sd1的rna-seq数据(图3d)和定量rt-pcr比较表明了osgrf4上调,而slr1下调编码光合作用的多个基因(例如oscab1、ospsbs1和其它基因;图5a;图16a)、糖信号传导(例如ostps1;图5a)和蔗糖转运/韧皮部负载(例如ossweet11和其它基因;图5a;图16a)机制组分。此外,osgrf4在体内与来自ospsbs1、ostps1和ossweet11的含gcgg的启动子片段结合(图5b),而slr1抑制从pospsbs1、postps1和possweet11启动子的转录的osgrf4-osgif1复合物激活(图5c)。因此,我们得出结论,osgrf4和slr1之间的平衡的拮抗关系调节n和c同化,并提供了它们之间的调节性协调联系。
因为slr1抑制生长,我们还测定osgrf4-slr1相互作用是否调节细胞增殖,表明osgrf4上调而slr1下调了促进细胞分裂的多个基因,包括编码细胞周期蛋白依赖性cdc2蛋白激酶的那些基因(例如oscyca1;1和oscdc20s-326,27)和其它的基因(图5d;图16b)。此外,osgrf4在体内与来自oscyca1;1和oscdc20s-3的含gcgg的启动子片段结合(图5e),以及ga促进而slr1抑制从poscyca1;1和poscdc20s-3启动子的转录的osgrf4-osgif1复合物激活(图5f)。我们得出结论,osgrf4-slr1拮抗平衡调节ga介导的细胞增殖调节,并提供整合生长、n和c代谢的协调控制连接。
osgrf4丰度的增加提高了稻和小麦grv的可持续产量
osgrf4启动子单倍型b(图7d)存在于选择的籼稻栽培品种中,但不存在于现代优良籼稻或粳稻品种中。然而,在225个成员(accession)中28,携带单倍型b的品种显示相对高的产量潜力(图17)。我们通过构建9311-osgrf4ngr2等基因系(在高产的含sd1的籼稻9311背景中;图6a),进一步评价了osgrf4等位基因变异对籼稻n利用效率和籽粒产量的可能影响。如先前在nj6-sd1(图3a)中发现的,osgrf4ngr2在低、中或高n-输入水平下不影响sd1赋予的半矮化9311表型(图6a,b),但增加了叶和秆的宽度(图18a,b)。此外,osgrf4ngr2赋予的增加的n摄取和同化(图19),在不影响植株高度(图6a、b)的同时,增加9311籽粒产量和n利用效率。在9311-osgrf4ngr2中,在n输入范围内,籽粒产量增加(相对于9311),甚至在相对低的n供应水平也观察到显著的产量提高(图6c)。这些产量增加是由于每株植物的籽粒数目增加和1,000籽粒重量的增加17-19(图18d),其中所述籽粒数目的增加程度随n输入的减少而增加(图18c)。此外,收获指数几乎没有变化(图18e),大概是因为生物量增加(图18f)抵消了籽粒产量的增加。9311-osgrf4ngr2植物地上部分的总n值实质上高于9311(图6d),而在9311-osgrf4ngr2中分配到籽粒(相对于营养器官)的n的分配比没有显著增加(图20a)。c/n平衡比同样不受影响(如预期的,因为osgrf4协调促进c和n代谢;图6e)。最后,osgrf4ngr2对其它矿质营养物在植物中的分布影响很小(图20b-f)。这些结果表明,由osgrf4ngr2赋予的osgrf4丰度的增加部分地将茎伸长(植物高度)的ga调节与n代谢调节脱离关系。因此,籼稻grv的营养同化和籽粒产量增加,特别是在低水平的n施肥下。
我们接着确定增加的osgrf4丰度是否类似地提高了粳稻和小麦grv中的籽粒产量和n利用效率。(gα、gβ和gγ亚基的)异三聚体g蛋白复合物在多种生物体中介导对多种外部刺激的应答。中国粳稻grv半矮态是由降低营养生长n响应和增加n利用效率21的变体(dep1-1)gγ亚基29赋予的。至于籼稻,我们发现增加的osgrf4丰度(表达p35s::osgrf4ngr2-gfp的转基因粳稻wjy7-dep1-1等基因植物29中的osgrf4-gfp)不抑制dep1-1赋予的半矮态(图21a),但增加了15nh4+和15no3-摄取率(图21b-d)。此外,虽然每株植物响应于不同n输入水平的植物高度、抽穗日期和分蘖数未受影响(图21e-g),p35s::osgrf4ngr2-gfp的表达增加了wjy7-dep1-1的每穗的籽粒数目(低n;图21h)和籽粒产量(图21i)。因此,提高的osgrf4丰度增加了优良籼稻和粳稻品种的n利用效率和产量。
最后,高产中国小麦grvkn199的半矮态是由突变的rht-b1b等位基因赋予的4,5。如在稻中一样,p35s::osgrf4ngr2-gfp的转基因表达不增加kn199植物高度(图6f),但增加秆直径和壁厚度(图6g)、穗长度(图6h)和生物量积累(图6i)。此外,p35s::osgrf4ngr2-gfp增加了kn19915no3-的摄取率(图6j),地上植物部分的总n(图6k)和未去壳(de-husked)籽粒中的n浓度(图6l)。p35s::osgrf4ngr2-gfp还通过增加每穗的籽粒数目(图6n)提高kn199产量(图6m),而不影响收获指数(图6o)。增加的osgrf4丰度因此提高了小麦grv的籽粒产量和n利用效率,而不影响突变的rht等位基因赋予的有益的半矮态。实际上,由p35s::osgrf4ngr2-gfp赋予的增加的秆宽度和壁厚度(图6g)可能增强突变的rht等位基因赋予的茎稳健性,因此进一步减少倒伏产量损失。总之,增加的osgrf4丰度提高了在中等n施肥水平下生长的稻和小麦grv的籽粒产量。
讨论
我们在此报道了基础植物科学和植物战略育种中的联合进展。首先,我们显示了osgrf4-della相互作用整合了植物生长和代谢的调节。osgrf4通过多个n摄取和同化基因的转录调节来调节n稳态,并且是植物n代谢的n调节协调物。重要的是,osgrf4也协调c代谢和生长。因为osgrf4丰度本身是n调节的,osgrf4将n代谢的稳态控制与生长和c代谢的控制整合。尽管长期以来认为存在,但这些整合物的身份是先前未知的。最后,osgrf4和della生长阻抑物之间的拮抗平衡调节性相互作用是osgrf4协调植物生长和代谢的机制的关键方面。基本上,物理的della-osgrf4-osgif1相互作用使得della能够抑制靶基因启动子的osgrf4-osgif1激活,因此平衡的osgrf4-della拮抗相互作用整合了植物生长和代谢的协调调节。
其次,我们显示增加grv中osgrf4的丰度改变了osgrf4-della平衡,因此部分断开della对grv生长和代谢的作用(也参见参考文献30)。特别地,增加的osgrf4丰度增加了grvn同化和细胞增殖。增加的细胞增殖增加了叶和茎的宽度,但对茎的高度几乎没有影响。这种方法的实际植物育种结果是,它能够增强grv营养同化,而不损失della积累所赋予的有益矮态。因此可以实现增强的grvn利用效率,而没有增加倒伏的产量损失代价。我们得出的结论是,osgrf4(和其它谷类直向同源物)的遗传变异现在应该成为育种者提高农作物产量和营养利用效率的主要靶标。这样的增强将使未来的绿色革命成为可能,可持续地增加产量,但减少环境恶化的农业n的使用。
方法
植物材料和田间生长条件。用于位置克隆和单倍型分析的稻种质的细节在其他地方已有描述28,21,31。使用源自nm73和籼稻品种nj6(轮回亲本)杂交产生的群体,进行qtl分析和基于图谱的克隆。通过将nm73×nj6和nm73×9311f1分别与作为轮回亲本的nj6、nj6-sd1和9311杂交六次,培育携带qngr2和sd1等位基因的不同组合的近等基因系(nil)植物。在位于陵水(海南省)、合肥(安徽省)和北京的三个遗传与发育生物学研究所实验站地点处,在标准稻田条件下以20cm的株距种植田间生长的nil和转基因稻植物。在冬季种植季节,在河北农林科学研究所(石家庄,河北省)的实验站种植田间生长的小麦植物(中国小麦grvkn199和转基因衍生物)。
水培培养条件。从liu(2004)32修改了水培培养条件。将种子在20%次氯酸钠溶液中消毒30分钟,用去离子水彻底洗涤,然后在潮湿的珍珠岩中发芽。然后选择7天龄的幼苗并移植到含有40l+n营养液(1.25mmnh4no3,0.5mmnah2po4·2h2o,0.75mmk2so4,1mmcacl2,1.667mmmgso4·7h2o,40μmfe-edta(na),19μmh3bo3,9.1μmmnso4·h2o,0.15μmznso4·7h2o,0.16μmcuso4,和0.52μm(nh4)3mo7o24·4h2o,ph5.5)的pvc盆中。含有不同水平的供应n的营养液组分如下:1n,1.25mmnh4no3;0.6n,0.75mmnh4no3;0.3n,0.375mmnh4no3;0.15n,0.1875mmnh4no3。所有营养液每周更换两次,每天将ph调节至5.5。温度保持在白天30℃和夜晚22℃,相对湿度为70%。
qngr2的位置克隆。qngr2的基于图谱的克隆是基于1,849bc2f2和3,124bc3f2群体的,所述群体源自nm73和籼稻品种nj6(以nj6作为轮回亲本)之间的回交。
转基因构建体。从nj6扩增osgrf4ngr2蛋白编码序列(连同内含子序列)。从nm73扩增osgrf4ngr2编码序列(连同位于转录起始位点上游~3-kbp的内含子和启动子区)。然后将这些扩增片段插入到pactin::nos33和pcambia1300(cambia,www.cambia.org)载体中,以便分别生成pactin::osgrf4ngr2和posgrf4ngr2::osgrf4ngr2构建体。将全长osgrf4ngr2cdna引入p35s::gfp-nos和p35s::flag-nos载体31中,以分别生成p35s::osgrf4ngr2-gfp和p35s::flag-osgrf4ngr2构建体。扩增300-bposgrf4ngr2cdna片段,并用于构建pactin::rnai-osgrf4转基因,如其他地方所述述29。如其他地方所述20,31,生成grna构建体,该构建体是在wyj7遗传背景下构建crispr/cas9使能的osgrf4功能丧失等位基因(osgrf4)所需的。如别处所述29,转基因稻和小麦植物是通过农杆菌介导的转化生成的。
定量实时pcr(qrt-pcr)分析。使用trizol试剂(invitrogen)从不同的稻植物器官提取总rna,然后根据制造商的方案用无rnase的dnasei(invitrogen)处理。然后使用cdna合成试剂盒(transgen,ae311)反转录全长cdna。随后的qrt-pcr根据制造商的说明(transgen,aq101)进行,使用三种独立的rna制备物作为生物学重复。稻actin2基因转录物用作参照。
双分子荧光互补(bifc)分析。从nj6中扩增出与slr1,osgif1,osgif2,osgif3,osgrf1,osgrf2,osgrf3,osgrf4,osgrf5,osgrf6,osgfr7,osgrf8,osgrf9,osgrf10,osgrf11和osgrf12基因对应的全长cdna,以及osgrf4cdna的缺失和非缺失形式。将得到的扩增子插入至psy-735-35s-cyfp-ha或psy-736-35s-nyfp-ee载体中34以产生融合构建体。通过农杆菌介导的渗透将构建体(例如,编码nyfp-osgrf4和cyfp-slr1的那些)共转染到烟草叶表皮细胞中,使得能够测试蛋白质-蛋白质相互作用。在黑暗中培养48小时后,使用共焦显微镜(zeisslsm710)检查yfp信号并拍照。每个bifc测定重复至少三次。
免疫共沉淀(co-ip)测定。扩增全长osgrf4,osgif1和slr1cdna,然后如前所述插入到puc-35s-ha-rbs或puc-35s-flag-rbs载体中35。用100μg质粒转染拟南芥原生质体,然后在低光强度条件下温育过夜。然后通过用50mmhepes(ph7.5)、150mmkcl、1mmedta(ph8)、0.3%trition-x100、1mmdtt以及加入的蛋白酶抑制剂混合物(rochelifescience)处理,从收获的原生质体中提取总蛋白质。将裂解物与缀合抗ddddk-标记抗体(mbl,m185-11)的磁珠在4℃温育至少4小时。然后用提取缓冲液漂洗磁珠6次,并用3×flag肽(sigma-aldrich,f4709)洗脱。免疫沉淀物通过sds-page电泳分离并转移到硝酸纤维素膜(gehealthcare)上。使用抗体抗-flag(sigma,f1804)和抗-ha(santacruzbiotechnology,sc-7392),通过免疫印迹检测蛋白质。
emsa测定。扩增全长osgrf4,osgif1和slr1cdna,并克隆到pcold-tf载体(takara)中。按照生产商的说明,用ni-nta琼脂糖(qiagen,30210)纯化osgrf4-his和slr1-his融合蛋白。人工扩增47bpdna探针,并使用生物素标记试剂盒(biosune)进行标记。使用lightshift化学发光emsa试剂盒(thermofisherscientific,20148)进行dna凝胶移位测定。
chip-qpcr测定。将约2g的转基因p35s::flag-osgrf4ngr2稻植物的两周龄幼苗用1%甲醛在20-25℃真空下固定15分钟,然后在液氮中匀浆。分离和裂解细胞核后,分离染色质并超声破碎至平均大小约500bp的片段。用抗-flag抗体(sigma,f1804)在4℃过夜进行免疫沉淀,然后将反向交联并沉淀的dna用作定量rt-pcr的模板。
fret(
体外瞬时反式激活测定。从nj6扩增各自来自osamt1.1,osamt1.2,osgs1.2,osgs2,osnadh-gogat2,osfd-gogat,oscab1,ostps1,ossweet11,oscyca1;1或oscdc2os-3的约3kb的dna启动子片段,然后亚克隆到含有由35s最小tata盒和5×gal4结合元件驱动的萤火虫luc报告基因的puc19载体中,由此产生含有与luc融合的特异性启动子的报告质粒。扩增全长osgrf4cdna,并融合至编码gal4bd的序列,从而产生效应质粒prtbd-osgrf4。如在别处所述36,瞬时反式激活测定是使用稻原生质体进行的。双荧光素酶报告基因测定系统(promega,e1960)用于进行荧光素酶活性测定,以renillaluc基因作为内部对照。
测定植物内矿质营养物的浓度。将来自各种植物器官的样品在烘箱中在80℃干燥72小时。组织匀浆后,使用元素分析仪(isoprime100;elementar)测定c和n的浓度,使用icp-oes(optima5300dv;perkinelmer)测定p和s的浓度,并使用原子吸收分光光度计(aa-6800gf;shimadzu)测定k、ca和mg的浓度。所有实验均进行至少三个重复。
15n摄取分析。在水培培养中生长4周后,稻根的15no3-和15nh4+流入测量如别处所述37,38。分离根和枝条,并在冷冻干燥前储存在-70℃。根和枝条在80℃干燥过夜,用isopreme100(elementar,德国)测定15n含量。
谷氨酰胺合酶和硝酸还原酶活性的测定。按照制造商的说明书,分别用谷氨酰胺合成酶试剂盒(solarbiolifesciences,bc0910)和硝酸还原酶试剂盒(solarbiolifesciences,bc0080)测定谷氨酰胺合酶和硝酸还原酶活性。
设计crispr的供体dna序列的方法
1.在模板序列的两端选择两个靶序列。
2.在两个靶序列的两端选择两个约100bp的序列,以产生左臂和右臂。
3.将突变基因座引入模板序列中,得到用于引入正确snp的修复模板序列。
4.将靶序列的ngg替换为nxx(确保氨基酸序列不变),得到突变的靶1和突变的靶2。
5.在臂的两端添加带有ngg的靶序列。
6.使用kpni将供体片段连接到构建体pcxun-cas9-u3-gdna。
7.使用aari将靶序列1(无ngg)连接到u3启动子和gdna之间的空间。
8.扩增u3、靶序列2(无ngg)和gdna,然后使用aari将它们连接至构建体pcxun-cas9-u3-gdna。
参考文献
1,pingali,p.l.greenrevolution:impacts,limits,andthepathahead.proc.natlacad.sci.usa109,12302-12308(2012).
2,evenson,r.e.andgollin,d.assessingtheimpactofthegreenrevolution,1960to2000.science300,758-762(2003).
3,hedden,p.thegenesofthegreenrevolution.trendsgenet19,5-9(2003).
4,peng,j.etal.greenrevolutiongenesencodemutantgibberellinresponsemodulators.nature400,256-261(1999).
5,zhang,c.,gao,l.,sun,j.,jia,jandren,z.haplotypevariationofgreenrevolutiongenerht-d1duringwheatdomesticationandimprovement.jintegrplantbiol.56,774-780(2014).
6,sasaki,a.etal.greenrevolution:amutantgibberellin-synthesisgeneinrice–newinsightintothericevariantthathelpedavertfamineoverthirtyyearsago.nature416,701-702(2002).
7,speilmeyer,w.etal.semidwarf(sd-1),greenrevolutionrice,containsadefectivegibberellin20-oxidasegene.proc.natlacad.sci.usa99,9043-9048(2002).
8,harberd,n.p.,belfield,e.andyasumura,y.theangiospermgibberellin-gid1-dellagrowthregulatorymechanism:howan“inhibitorofaninhibitor”enablesflexibleresponsetofluctuatingenvironments.plantcell21,1328-1339(2009).
9,xu,h.,liu,q.,yao,t.,fu,x.sheddinglightonintegrativegasignaling.curropinplantbiol.21,89-95(2014).
10,itoh,h.,ueguchi-tanaka,m.,sato,y.,ashikari,m.,matsuoka,m.thegibberellinsignalingpathwayisregulatedbytheappearanceanddisappearanceofslenderrice1innuclei.plantcell14,57-70(2002).
11,asano,k.etal.artificialselectionforagreenrevolutiongeneduringjaponicaricedomestication.procnatlacadsciusa.108,11034-11039(2011).
12,gooding,m.j.,addisu,m.,uppal,r.k.,snape,j.w.andjones,h.e.effectofwheatdwarfinggenesonnitrogen-useefficiency.jagricsci150,3-22(2012).
13,li,b.-z.etal.molecularbasisandregulationofammoniumtransporterinrice.ricescience16,314-322(2009).
14,hawkesford,m.j.reducingtherelianceonnitrogenfertilizerforwheatproduction.jcerealsci59,276-283(2014).
15,zhao,x.etal.nitrogenrunoffdominateswaternitrogenpollutionfromrice-wheatrotationinthetaihulakeregionofchina.agricecosystenviron156,1-11(2012).
16,conway,g.onebillionhungry.canwefeedtheworld?cornelluniv.press.usa(2012).
17,che,r.etal.controlofgrainsizeandriceyieldbygl2-mediatedbrassinosteroidresponses.natureplants2,15195(2015).
18,duan,p.etal.regulationofosgrf4byosmir396controlsgrainsizeandyieldinrice.natureplants2,15203(2015).
19,hu,j.etal.ararealleleofgs2enhancesgrainsizeandgrainyieldinrice.molplant8,1455-1465(2015).
20,ma,x.etal.arobustcrispr/cas9systemforconvenient,high-efficiencymultiplexgenomeeditinginmonocotanddicotplants.molplant8,1274-1284(2015).
21,sun,h.etal.heterotrimericgproteinsregulatenitrogen-useefficiencyinrice.natgenet.46,652-6562014).
22,somers,d.a.,kuo,t.,kleinhofsa.,warnerr.l.,oaksa.synthesisanddegradationofbarleynitratereductase.plantphysiol.72,949-952(1983).
23,tabuchi,m.,abiko,t.,yamaya,t.assimilationofammoniumionsandreutilizationofnitrogeninrice(oryzasatival.).jexpbot.58,2319-2327(2007).
24,peng,j.etal.thearabidopsisgaigenedefinesasignalingpathwaythatnegativelyregulatesgibberellinresponses.genesdev.11,3194-3205(1997).
25,nunes-nesi,a.,fernie,a.r.,stitt,m.metabolicsignallingaspectsunderpinningplantcarbonnitrogeninteractions.mol.plant3,973-996(2010).
26,fabian,t.,lorbiecke,r.,umeda,m.,sauter,m.thecellcyclegenescyca1;1andcdc2os-3arecoordinatelyregulatedbygibberellininplanta.planta211,376-383(2000).
27,sauter,m.differentialexpressionofacak(cdc2-activatingkinase)-likeproteinkinase,cyclinsandcdc2genesfromriceduringthecellcycleandinresponsetogibberellin.plantj.11,181-190(1997).
28,yu,j.etal.oslg3contributingtoricegrainlengthandyieldwasminedbyho-lamap.bmcbiol.15,28(2017).
29,huang,x.etal.naturalvariationatthedep1locusenhancesgrainyieldinrice.natgenet.41,494-4972009).
30,serrano-mislata,a.,bencivenga,s.,bush,m.,schiessl,k.,boden,s.,sablowski,r.dellagenesrestrictinflorescencemeristemfunctionindependentlyofplantheight.nat.plants3,749-754(2017).
31,wang,s.etal.non-canonicalregulationofspltranscriptionfactorsbyahumanotub1-likedeubiquitinasedefinesanewplanttypericeassociatedwithhighergrainyield.cellres.27,1142-1156(2017).
32,liu,w.j.,zhu,y.g.,smith,f.a.,smith,s.e.dophosphorusnutritionandironplaquealterarsenate(as)uptakebyriceseedlingsinhydroponicculture?newphytol.162,481-488(2004).
33,wang,s.etal.controlofgrainsize,shapeandqualitybyosspl16inrice.natgenet44,950-954(2012).
34,bracha-drori,k.etal.detectionofprotein-proteininteractionsinplantsusingbimolecularfluorescencecomplementation.plantj40,419-427(2004).
35,chen,h.etal.fireflyluciferasecomplementationimagingassayforprotein-proteininteractionsinplants.plantphysiol.146,368-376(2008).
36,wang,s.etal.theosspl16-gw7regulatorymoduledeterminesgrainshapeandsimultaneouslyimprovesriceyieldandgrainquality.nat.genet.47,949-954(2015).
37.ho,c.h.,lin,s.h.,hu,h.c.,tsay,y.f.chl1functionsasanitratesensorinplants.cell138,1184-1194(2009).
38,loqué,d.etal.additivecontributionofamt1;1andamt1;3tohigh-affinityammoniumuptakeacrosstheplasmamembraneofnitrogen-deficientarabidopsisroots.plantj48,522-534(2006).
39,sun.yetal.engineeringherbicide-resistantriceplantsthroughcrispr/cas9-mediatedhomologousrecombinationofacetolactatesynthase.mol.plant.628-630(2016).
40,nicolem.gaudellietal.programmablebaseeditingofa·ttog·cingenomicdnawithoutdnacleavage;naturevolume551,pages464–471(23november2017).
41,kimetal.highlyefficientrna-guidedbaseeditinginmouseembryos;natbiotechnol.2017may;35(5):435-437.
42,tomascermak,erinl.doyle,michellechristian,liwang,yongzhang,clariceschmidt,joshuaa.baller,nikunjv.somia,adamj.bogdanove&danielf.voytas.efficientdesignandassemblyofcustomtalenandothertal-effector-basedconstructsfordnatargeting,nucleicacidsresearch2011,39(12).
43,nevilleesanjana,,lecong,yangzhou,margaretmcunniff,guopingfeng&fengzhangatranscriptionactivator-likeeffectortoolboxforgenomeengineering,natureprotocols7,171–192(2012
44,meghdadrahdar,moiraa.mcmahon,thazhap.prakash,erice.swayze,c.frankbennettanddonw.cleveland,syntheticcrisprrna-cas9–guidedgenomeeditinginhumancellspnas2015112(51)e7110-e7117;publishedaheadofprintnovember16,2015,doi:10.1073/pnas.1520883112
序列列表
稻
seqidno:1osgrfngr2(野生型基因组序列)
seqidno:2osgrfngr2(野生型cds序列)
seqidno:3osgrfngr2(氨基酸序列)
seqidno:4osgrfngr2(基因组序列)
seqidno:5osgrfngr2(cds序列)
seqidno:6osgrfngr2(氨基酸序列)
seqidno:7:osgrf单倍型a启动子
(-941,-884,-855,-847,-801,-522和-157snp以粗体突出显示)
seqidno:8:osgrf单倍型c启动子
(-935,-878,-849,-841,-795,-516和-157snp以粗体突出显示)
seqidno:9:osgrf单倍型b启动子
(-941,-884,-855,-847,-801,-522和-157snps以粗体突出显示)
直向同源物序列
玉米
seqidno:10:rmzm2g034876(grf-转录因子6);2kb启动子
seqidno:11:cds
(mir396识别位点以粗体突出显示)
seqidno:12:氨基酸序列
seqidno:13grmzm2g041223(grf-转录因子8);2kb启动子
seqidno:14:cds
(mir396识别位点以粗体突出显示)
seqidno:15:氨基酸序列
小麦
seqidno:16:traes_6al_06a78c5202kb启动子
seqidno:17:cds
(mir396识别位点以粗体突出显示)
seqidno:18:氨基酸序列
seqidno:19:triae_cs42_6bl_tgacv1_500422_aa1604330:2kb启动子
seqidno:20:cds
(mir396识别位点以粗体突出显示)
seqidno:21:氨基酸序列
seqidno:22:triae_cs42_6dl_tgacv1_527461_aa17043702kb启动子
seqidno:23:cds
(mir396识别位点以粗体突出显示)
seqidno:24:氨基酸序列
大麦
seqidno:25:horvu2hr1g1017702kb启动子
seqidno:26:cds
(mir396识别位点以粗体突出显示)
seqidno:27:氨基酸序列
高粱
seqidno:28:sorbi_004g2699002kb启动子
seqidno:29:cds
(mir396识别位点以粗体突出显示)
seqidno:30:氨基酸序列
大豆
seqidno:31:glyma11g11826;2kb启动子
seqidno:32:cds
(mir396识别位点以粗体突出显示)
seqidno:33:氨基酸序列
油菜(brassicanapus)
seqidno:34:bnaa03g16700d:2kb启动子
seqidno:35:cds
(mir396识别位点以粗体突出显示)
seqidno:36:氨基酸序列
番茄
seqidno:37:solyc08g075950;2kb启动子
seqidno:38:cds
(mir396识别位点以粗体突出显示)
seqidno:39:氨基酸序列
seqidno:40:p35s启动子
seqidno:41pubi启动子
seqidno:42:cas9核酸
seqidno:43;cys4内切核糖核酸酶核酸序列
crispr构建体
seqidno:46;tracrrna核酸序列
稻
功能突变体g.1187-1188tc>aa(在mirna396结合位点处突变)的籽粒的信息
seqidno:47:用于引入正确snp的修复模板序列
seqidno:48:供体dna序列,其用于引入正确snp
seqidno:49:靶标1靶序列:
gaaatggcggtgctcgaaggagg
seqidno:50:靶标1前间隔区序列:
gaaatggcggtgctcgaagg
seqidno:51:靶标1完整的sgrna核酸序列:
seqidno:52:靶标2靶序列:
atctgttgtcggttctgcggcgg
seqidno:53:靶标2前间隔区序列:
atctgttgtcggttctgcgg
seqidno:54:靶标2完整的sgrna核酸序列:
用于修复hap.a/c-884t>a中启动子的信息
seqidno:55:修复模板序列,其用于引入正确snp
seqidno:56:供体dna序列,其用于引入正确snp
seqidno:57:靶标1靶序列:
atctctatggagtagtaccgagg
seqidno:58:靶标1前间隔区序列:
atctctatggagtagtaccg
seqidno:59:靶标1完整的sgrna核酸序列:
seqidno:60:靶标2靶序列:
atgtagacgcaagaagatgttgg
seqidno:61:靶标2前间隔区序列:
atgtagacgcaagaagatgt
seqidno:62:靶标2完整的sgrna核酸序列:
用于修复hap.a/c-847c>t中启动子的信息
seqidno:63:修复模板序列,其用于引入正确snp
seqidno:64:供体dna序列,其用于引入正确snp
seqidno:65:靶标1靶序列:
atctctatggagtagtaccgagg
seqidno:66:靶标1前间隔区序列:
atctctatggagtagtaccg
seqidno:67:靶标1完整的sgrna核酸序列:
seqidno:68:靶标2靶序列:
atgtagacgcaagaagatgttgg
seqidno:69:靶标2前间隔区序列:
atgtagacgcaagaagatgt
seqidno:70:靶标2完整的sgrna核酸序列:
用于修复hap.a/c-801c>t中启动子的信息
seqidno:71:修复模板序列,其用于引入正确snp
seqidno:72:供体dna序列,其用于引入正确snp
seqidno:73:靶标1靶序列:
atctctatggagtagtaccgagg
seqidno:74:靶标1前间隔区序列:
atctctatggagtagtaccg
seqidno:75:靶标1完整的sgrna核酸序列:
seqidno:76:靶标2靶序列:
atgtagacgcaagaagatgttgg
seqidno:77:靶标2前间隔区序列:
atgtagacgcaagaagatgt
seqidno:78:靶标2完整的sgrna核酸序列:
用于修复hap.a/c-884t>a,-847c>t中启动子的信息
seqidno:79:修复模板序列,其用于引入正确snp
seqidno:80:供体dna序列,其用于引入正确snp
seqidno:81:靶标1靶序列:
atctctatggagtagtaccgagg
seqidno:82:靶标1前间隔区序列:
atctctatggagtagtaccg
seqidno:83:靶标1完整的sgrna核酸序列:
seqidno:85:靶标2靶序列:
atgtagacgcaagaagatgttgg
seqidno:86:靶标2前间隔区序列:
atgtagacgcaagaagatgt
seqidno:87:靶标2完整的sgrna核酸序列:
用于修复hap.a/c-884t>a,-801c>t中启动子的信息
seqidno:88:修复模板序列,其用于引入正确snp
seqidno:89:供体dna序列,其用于引入正确snp
seqidno:90:靶标1靶序列:
atctctatggagtagtaccgagg
seqidno:91:靶标1前间隔区序列:
atctctatggagtagtaccg
seqidno:92:靶标1完整的sgrna核酸序列:
seqidno:93:靶标2靶序列:
atgtagacgcaagaagatgttgg
seqidno:94:靶标2前间隔区序列:
atgtagacgcaagaagatgt
seqidno:95:靶标2完整的sgrna核酸序列:
用于修复hap.a/c-884t>a,-847c>t,-801c>t中启动子的信息
seqidno:96:修复模板序列,其用于引入正确snp
seqidno:97:供体dna序列,其用于引入正确snp
seqidno:98:靶标1靶序列:
atctctatggagtagtaccgagg
seqidno:99:靶标1前间隔区序列:
atctctatggagtagtaccg
seqidno:100:靶标1完整的sgrna核酸序列:
seqidno:101:靶标2靶序列:
atgtagacgcaagaagatgttgg
seqidno:102:靶标2前间隔区序列:
atgtagacgcaagaagatgt
seqidno:103:靶标2完整的sgrna核酸序列:
用于修复hap.a/c-847c>t,-801c>t中的启动子的信息
seqidno:104:修复模板序列,其用于引入正确snp
seqidno:105:供体dna序列,其用于引入正确snp
seqidno:106:靶标1靶序列:
atctctatggagtagtaccgagg
seqidno:107:靶标1前间隔区序列:
atctctatggagtagtaccg
seqidno:108:靶标1完整的sgrna核酸序列:
seqidno:109:靶标2靶序列:
atgtagacgcaagaagatgttgg
seqidno:110:靶标2前间隔区序列:
atgtagacgcaagaagatgt
seqidno:111:靶标2完整的sgrna核酸序列:
用于在mirna396识别位点引入snp的crispr构建体
(i.e.ccgttcaagaaagcctgtggaa>ccgtaaaagaaagcctgtggaa)
玉米
grmzm2g034876(grf-转录因子6)
seqidno:112:修复模板序列,其用于引入正确snp
seqidno:113:供体dna序列,其用于引入正确snp
seqidno:114:靶标1靶序列:
ggcgaacggacggcaagaagtgg
seqidno:115:靶标1前间隔区序列:
ggcgaacggacggcaagaag
seqidno:116:靶标1完整的sgrna核酸序列:
seqidno:117:靶标2靶序列:
cgtctccgccgctccgcccctgg
seqidno:118:靶标2前间隔区序列:
cgtctccgccgctccgcccc
seqidno:119:靶标2完整的sgrna核酸序列:
grmzm2g041223
seqidno:120:修复模板序列,其用于引入正确snp
seqidno:121:供体dna序列,其用于引入正确snp
seqidno:122:靶标1靶序列:
aagtggcggtgctccaagggagg
seqidno:123:靶标1前间隔区序列:
aagtggcggtgctccaaggg
seqidno:124:靶标1完整的sgrna核酸序列:
seqidno:125:靶标2靶序列:
caggcccccgcgcccaccgctgg
seqidno:126:靶标2前间隔区序列:
caggcccccgcgcccaccgc
seqidno:127:靶标2完整的sgrna核酸序列:
小麦
traes_6al_06a78c520
seqidno:128:修复模板序列,其用于引入正确snp
seqidno:129:供体dna序列,其用于引入正确snp
seqidno:130:靶标1靶序列:
gaagtggcggtgcgccaaggagg
seqidno:131:靶标1前间隔区序列:
gaagtggcggtgcgccaagg
seqidno:132:靶标1完整的sgrna核酸序列:
seqidno:133:靶标2靶序列:
gcaccaacggtggtggaggcggg
seqidno:134:靶标2前间隔区序列:
gcaccaacggtggtggaggc
seqidno:135:靶标2完整的sgrna核酸序列:
triae_cs42_6bl_tgacv1_500422_aa1604330
seqidno:136:修复模板序列,其用于引入正确snp
seqidno:137:供体dna序列,其用于引入正确snp
seqidno:138:靶标1靶序列:
caagaagtggcggtgcgccaagg
seqidno:139:靶标1前间隔区序列:
caagaagtggcggtgcgcca
seqidno:140:靶标1完整的sgrna核酸序列:
seqidno:141:靶标2靶序列:
gcaccaacggtggtggaggcggg
seqidno:142:靶标2前间隔区序列:
gcaccaacggtggtggaggc
seqidno:143:靶标2完整的sgrna核酸序列:
triae_cs42_6dl_tgacv1_527461_aa1704370
seqidno:144:修复模板序列,其用于引入正确snp
seqidno:145:供体dna序列,其用于引入正确snp
seqidno:146:靶标1靶序列:
caagaagtggcggtgcgccaagg
seqidno:147:靶标1前间隔区序列:
caagaagtggcggtgcgcca
seqidno:148:靶标1完整的sgrna核酸序列:
seqidno:149:靶标2靶序列:
gcaccaacggtggtggaggcggg
seqidno:150:靶标2前间隔区序列:
gcaccaacggtggtggaggc
seqidno:151:靶标2完整的sgrna核酸序列:
大麦
horvu2hr1g101770
seqidno:152:修复模板序列,其用于引入正确snp
seqidno:153:供体dna序列,其用于引入正确snp
seqidno:154:靶标1靶序列:
gggcggtgccggcggacggacgg
seqidno:155:靶标1前间隔区序列:
gggcggtgccggcggacgga
seqidno:156:靶标1完整的sgrna核酸序列:
seqidno:157:靶标2靶序列:
tatccggcgatcgccactggcgg
seqidno:158:靶标2前间隔区序列:
tatccggcgatcgccactgg
seqidno:159:靶标2完整的sgrna核酸序列:
高粱
sorbi_004g269900
seqidno:160:修复模板序列,其用于引入正确snp
seqidno:161:供体dna序列,其用于引入正确snp
seqidno:162:靶标1靶序列:
aactggatccggagccggggcgg
seqidno:163:靶标1前间隔区序列:
aactggatccggagccgggg
seqidno:164:靶标1完整的sgrna核酸序列:
seqidno:165:靶标2靶序列:
cgtctccgccgctccgcccttgg
seqidno:166:靶标2前间隔区序列:
cgtctccgccgctccgccct
seqidno:167:靶标2完整的sgrna核酸序列:
大豆
glyma11g11826
seqidno:168:修复模板序列,其用于引入正确snp
seqidno:169:供体dna序列,其用于引入正确snp
seqidno:170:靶标1靶序列:
aaaaaagtggaggtgctccaagg
seqidno:171:靶标1前间隔区序列:
aaaaaagtggaggtgctcca
seqidno:172:靶标1完整的sgrna核酸序列:
seqidno:173:靶标2靶序列:
gacatcactcactgtcactgggg
seqidno:174:靶标2前间隔区序列:
gacatcactcactgtcactg
seqidno:175:靶标2完整的sgrna核酸序列:
欧洲油菜
bnaa03g16700d
seqidno:176:修复模板序列,其用于引入正确snp
seqidno:177:供体dna序列,其用于引入正确snp
seqidno:178:靶标1靶序列:
gtaagaagtaaagatgttcaagg
seqidno:179:靶标1前间隔区序列:
gtaagaagtaaagatgttca
seqidno:180:靶标1完整的sgrna核酸序列:
seqidno:181:靶标2靶序列:
catggcttccccagccacagcgg
seqidno:182:靶标2前间隔区序列:
catggcttccccagccacag
seqidno:183:靶标2完整的sgrna核酸序列:
番茄
solyc08g075950
seqidno:184:修复模板序列,其用于引入正确snp
seqidno:185:供体dna序列,其用于引入正确snp
seqidno:186:靶标1靶序列:
ctgagccaggaaggtgtagaagg
seqidno:187:靶标1前间隔区序列:
ctgagccaggaaggtgtaga
seqidno:188:靶标1完整的sgrna核酸序列:
seqidno:189:靶标2靶序列:
gtatgtcacaaattacagctggg
seqidno:190:靶标2前间隔区序列:
gtatgtcacaaattacagct
seqidno:191:靶标2完整的sgrna核酸序列:
直向同源物启动子序列
seqidno:192:玉米;grmzm2g034876(grf-转录因子6);2kb启动子
seqidno:193:玉米;grmzm2g041223(grf-转录因子8);2kb启动子
seqidno:194:小麦;traes_6al_06a78c520;2kb启动子
seqidno:195:小麦;triae_cs42_6bl_tgacv1_500422_aa1604330;2kb启动子
seqidno:44氨基酸序列
seqidno:196:小麦triae_cs42_6dl_tgacv1_527461_aa1704370
2kb启动子
seqidno:197:大麦;horvu2hr1g101770;2kb启动子
seqidno:198;高粱;sorbi_004g269900;2kb启动子
seqidno:199:大豆;glyma11g11826;2kb启动子
seqidno:200:欧洲油菜;bnaa03g16700d;2kb启动子
seqidno:201;番茄;solyc08g075950;2kb启动子
- 还没有人留言评论。精彩留言会获得点赞!